JEKMAL MALAU
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2015
KARAKTERISASI FRAGMEN DNA DARI BAKTERI
ANTAGONIS YANG TERLIBAT DALAM AKTIVITAS
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Karakterisasi Fragmen DNA dari Bakteri Antagonis yang Terlibat Dalam Aktivitas Antifungi Ganoderma boninense adalah benar karya saya dengan arahan dari komisi pembimbing Prof Dr Ir Antonius Suwanto, MSc serta Dr Maria Sugiharti dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir ditesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
RINGKASAN
JEKMAL MALAU. Karakterisasi Fragmen DNA dari Bakteri Antagonis yang Terlibat Dalam Aktivitas Antifungi Ganoderma boninense. Dibimbing oleh ANTONIUS SUWANTO dan MARIA SUGIHARTI.
Kelapa sawit (Elaeis guineensis jacq) merupakan salah satu tanaman komoditi yang telah digunakan secara luas dalam industri makanan, farmasi dan kosmetik. Indonesia merupakan negara yang telah dianugrahi dengan iklim, kondisi lingkungan yang cocok untuk pertumbuhan kelapa sawit dan luas areal yang menyebabkan Indonesia menjadi negara dengan perkebunan kelapa sawit terbesar di dunia. Namun demikian, budidaya kelapa sawit menghadapi beberapa ancaman yang dapat mengurangi tingkat produksi. Busuk pangkal batang (BPB) oleh Ganoderma boninense merupakan ancaman serius pada perkebunan kelapa sawit. Pengendalian penyakit tersebut hingga saat ini telah banyak dilakukan, namun belum menunjukkan hasil yang memuaskan.
Pengembangan ketersediaan agen pengendali hayati merupakan solusi yang sangat baik dalam menekan infeksi BPB. Lebih lanjut lagi bahwa pendekatan berbasis bioteknologi molekuler atau penemuan baru senyawa antifungi diperlukan untuk strategi pengendalian yang lebih baik.
Pada penelitian ini, 198 isolat bakteri berhasil diisolasi dari tiga jenis sumber isolasi yang berbeda. Empat puluh satu isolat menunjukkan efek penghambatan yang berbeda-beda terhadap miselium G. boninense. Enam belas isolat dipilih untuk diuji lebih lanjut berdasarkan kemampuan percentage inhibiton of radial growth (PIRG) lebih dari 50%. Perbedaan yang signifikan ditunjukkan pada uji kultur ganda (Dual culture assay) pada p>0.05, dimana isolat G.1.22 menunjukkan aktivitas penghambatan tertinggi yaitu 87.86%. Hasil uji filtrat menunjukkan bahwa isolat G.1.22 tahan terhadap pemanasan. Sebaliknya isolat K.3.2 sensitif terhadap pemanasan dan penambahan proteinase K sehingga diasumsikan memiliki senyawa antifungi berupa protein. Hasil identifikasi gen penyandi 16S rRNA menunjukan bahwa isolat G.1.22 dan K.3.2 memiliki tingkat kemiripan 100% dengan
Pseudomonas aeruginosa strain B13 (accession number KM588370) dan Bacillus amyloliquefaciens strain SCDB1439 (accession number KM922581).
Metode shotgun cloning digunakan untuk kloning sejumlah fargmen DNA kromosom Bacillus amyloliquefaciens JR02. Berdasarkan uji antagonis keseluruhan E. coli T0P 10 rekombinan dengan suspensi G. boninense belum menunjukkan adanya ekspresi gen antifungi. Disamping itu, karakterisasi DNA pengapit mutan transposon mini-Tn5KmR (BglK226) dengan teknik genome walking berhasil dilakukan. Berdasarkan hasil analisis pengurutan DNA dan blasting diperoleh bahwa DNA pengapit memiliki tingkat kemiripan 99% dengan sebagian gen beta-ketoacyl shyntase asal Bukholderia gladioli BSR3.
SUMMARY
JEKMAL MALAU. Characterization of DNA Fragment of Antagonistic Bacteria Contribute for Anti-fungal Activity Against Ganoderma boninense. Supervised by ANTONIUS SUWANTO and MARIA SUGIHARTI.
Elaeis guineensis Jacq. is one of the most important commodities plants that have been used widely in the food, pharmaceutical and cosmetics industry. Our country was blessed with a great climate and other suitable condition for palm oil growth as well as extensive land allows giant plantation. Indonesia has became the largest producer that supplies almost 47% palm oil in the world. Nevertheless, palm oil cultivation faces several threats that reduce production levels. Basal Stem Rot (BSR) disease caused by Ganoderma boninense is a serious threat in oil palm plantations. Many control effort was developed to solve this problem but no one was successful.
The available agent of biological control is an excellent solution to reduce the disease due to its ability to exert an effective anti-fungal compound. Furthermore, other approaches by molecular biotechnology or discovery novel anti-fungal are required for better control strategies.
In this study, 198 bacteria were isolated from bag-log medium (culture material of edible fungi), soil and oil palm empty fruit bunches. Forty-one isolates showed inhibitory effect by retarding the growth of the fungal mycelia. Sixteen isolates wereselected for further study based on their percentage inhibition ofradial growth (PIRG) ofmore than 50%. Significant difference was shown in inhibitory activity (p>0.05) for dual culture test. The highest of PIRG values showed by isolate G.1.22, with an average of 87.86 %. Culture filtrate test showed different result, i.e. G.1.22 has stable activities after boiling. Whereas with K.3.2 was sensitive to proteinase K and heating, suggesting that these isolate might have protein-based anti fungal. Full 16S rRNA gene sequences showed that G.1.22 and K.3.2 were closely related to Pseudomonas aeruginosa strain B13 (accession number KM588370) and Bacillus amyloliquefaciens strain SCDB1439 (accession number KM922581), respectively.
Shotgun cloning method was used to clone Bacillus amyloliquefaciens JR02 chromosomal DNA. Based on antagonistic assay, we found no transformant showed antifungal activity. On the other hand, genome walking method was successful to characterize flanking DNA mutant BglK226. Sequence analysis of the flanking DNA showed high similarity to beta-ketoacyl synthase gene of Burkholderia gladioli BSR3 (accession number CP0025991).
© Hak Cipta Milik IPB, Tahun 2015
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
Sebagai salah satu syarat untuk memperoleh gelar Magister Sains
Pada
Program Studi Bioteknologi
KARAKTERISASI FRAGMEN DNA DARI BAKTERI
ANTAGONIS YANG TERLIBAT DALAM AKTIVITAS
ANTIFUNGI Ganoderma boninense
JEKMAL MALAU
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PRAKATA
Puji dan syukur penulis panjatkan kepada Tuhan Yang Maha Esa atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Agustus 2014 hingga bulan Juli 2015 ini ialah Karakterisasi Fragmen DNA dari Bakteri Antagonis yang Terlibat dalam Aktivitas Antifungi Ganoderma boninense.
Terima kasih penulis ucapkan kepada Bapak Prof Dr Ir Antonius Suwanto, MSc dan Ibu Dr Maria Sugiharti selaku pembimbing atas segala arahan, saran, dan solusi selama pelaksanaan penelitian dan penyusunan karya ilmiah ini. Terima kasih kepada Ibu Dr Utut Widyastuti, MSi selaku penguji luar komisi atas saran yang diberikan pada saat ujian tesis. Di samping itu, penulis mengucapkan terima kasih kepada DIKTI melalui program beasiswa unggulan tahun 2013/2014, yang telah memberikan dana perkuliahan selama studi penulis di IPB.
Terima kasih kepada seluruh pegawai serta rekan-rekan di laboratorium R&D PT Wilmar Benih Indonesia Cikarang atas bantuan selama pelaksanaan penelitian. Terima kasih kepada teman-teman seperjuangan Natalia, Nabilah, Albert, Arif dan Tira, atas segala motivasi dan keceriaan selama masa studi. Terima kasih yang spesial buat Retno Tri Astuti telah menjadi partner terbaik dalam penelitian ini. Ungkapan terima kasih tak terhingga juga disampaikan kepada Ayah, Ibu, serta seluruh keluarga, atas segala doa dan kasih sayangnya.
Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 3
TINJAUAN PUSTAKA 4
METODE 9
Diagram Alur Penelitian 9
Waktu dan Tempat Penelitian 10
Kultur Cendawan, Bakteri, Plasmid dan Media 10
Prosedur Penelitian 10
HASIL 16
Isolasi, Penapisan dan Identifikasi Bakteri Potensial 16 Shotgun Cloning Bacillus amyloliquefaciens JR02 19 Identifikasi Fragmen DNA Pengapit Mutan BglK226 20
KESIMPULAN DAN SARAN 26
Kesimpulan 26
Saran 26
DAFTAR PUSTAKA 27
LAMPIRAN 31
DAFTAR TABEL
1 Primer yang digunakan untuk amplifikasi DNA pengapit transposon
14
2 Aktivitas penghambatan isolat potensial 16
3 Aktivitas penghambatan ekstrak kasar lima bakteri terpilih 18 4 Aktivitas penghambatan isolat B. gladioli JR01 tipe liar dan
mutan
21
DAFTAR GAMBAR
1 Penampakan serangan infeksi G. boninense 5
2 Penampakan tubuh buah G. boninense 5
3 Peta fisik plasmid pUTmini-Tn5KmR 7
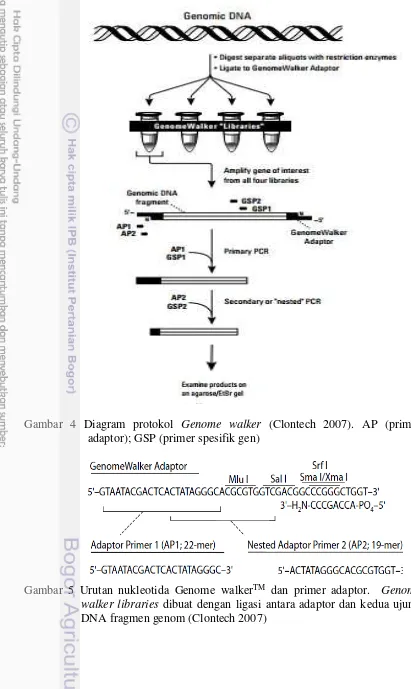
4 Diagram protokol genom walker 8
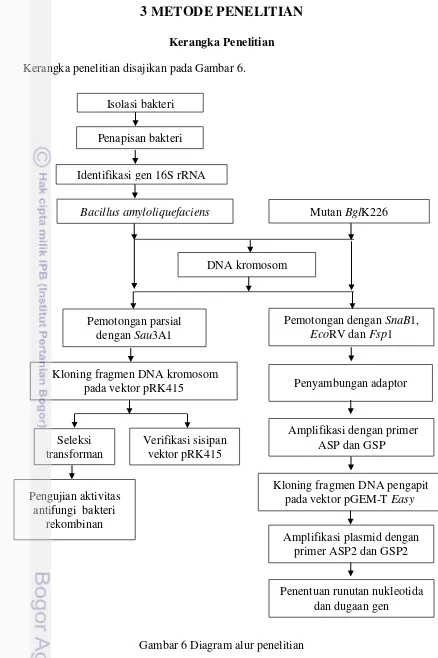
5 Urutan nukleotida Genome walkerTM dan primer adaptor 8
6 Diagram alur penelitian 9
7 Peta fisik vektor pRK415 12
8 Peta fisik vektor pGEM-T Easy 14
9 Aktivitas penghambatan bakteri antagonis pada hari ke 10 17
10 Hasil amplifikasi 16S Rdna 18
11 Pemotongan DNA kromosom JR02 secara parsial 19 12 Verifikasi sisipan plasmid pRK415 rekombinan 20 13 Penghambatan pertumbuhan miselium G. boninense oleh mutan 21 14 Strategi amplifikasi fragmen DNA pengapit transposon
mini-Tn5Kmr
21 15 Hasil amplifikasi genome walking menggunakan primer ASP2
dan GSP2
22
16 Peta plasmid rekombinan pJR226-01 22
17 Peta verifikasi fragmen DNA pengapit hasil amplifikasi genome walking
23 18 Posisi mini-Tn5KmR pada gen KAS dari Burkholderia gladioli
BSR3
23 19 Hasil analisis parsial urutan DNA mutan BglK226 pengapit 24 20 Pensejajaran sekuen asam amino KAS prokariot dan eukariot 25
DAFTAR LAMPIRAN
1 PENDAHULUAN
Latar Belakang
Kelapa sawit (Elaeis guineensis) merupakan tanaman monokotil yang memiliki nilai ekonomi penting. Tanaman ini termasuk kedalam famili Arecaceae, subfamili cocosoideae (Corley & Tinker 2003). Pada awalnya tanaman kelapa sawit berasal dari Afrika barat dan tengah, tetapi kemudian mengalami penyebaran hingga ke daerah tropis termasuk Indonesia (Ariffin et al. 2000; Darmono 2000). Sejak dua dekade terakhir terjadi perkembangan luas areal perkebunan kelapa sawit yang sangat pesat. Perkembangan tersebut tidak hanya di Sumatra, tetapi meluas hingga di Kalimantan, Sulawesi, Irian jaya (Susanto et al. 2005).
Perkembangan dan perluasan wilayah perkebunan kelapa sawit tidak selalu dibarengi dengan peningkatan produksi hasil yang signifikan. Hal ini disebabkan karena adanya serangan patogen pada tanaman. Penyakit busuk pangkal batang (BPB) merupakan penyebab utama penurunan produksi yang mengakibatkan kerugian besar pada budidaya kelapa sawit terutama di Indonesia dan Malaysia (Miller et al. 1999; Darmono 2000; Wong et al. 2012). Penyakit tersebut disebabkan oleh infeksi cendawan Ganoderma boninense (Sariah et al. 1994; Pilotti 2005).
Status infeksi penyakit busuk pangkal batang (BPB) tidak hanya menyerang tanaman tua, namun kemudian diketahui juga dapat menyerang tanaman yang masih muda. Penampakan gejala penyakit muncul lebih awal pada tanaman kelapa sawit yang lebih banyak mengalami penanaman ulang (replanting). Semakin sering suatu areal perkebunan mengalami peremajaan maka semakin tinggi persentase kejadian penyakit BPB (Soepena et al. 2000). Hal ini terjadi karena setelah cendawan menginfeksi tanaman, areal perkebunan akan terus terpapar dan inokulum patogen akan menjadi tuan rumah sejalan dengan semakin seringnya dilakukan penanaman kelapa sawit (Susanto et al. 2005).
G. boninense merupakan cendawan yang memiliki sifat patogen tular tanah (Turner 1981; Wong et al. 2012). Memiliki struktur khusus yang berdampak pada kemampuan bertahan dan menginfeksi tanaman target seperti spora istirahat berupa klamidospora dan struktur pseudosklerotia (Darmono 2000; Susanto et al. 2005; Rees et al. 2009).
2
Penggunaan agen hayati sebagai biokontrol G. boninense diharapkan menjadi langkah pengendalian yang lebih efektif dengan mengoptimalkan pengendalian alami yang ada di alam menggunakan organisme antagonis seperti bakteri yang memiliki aktivitas antifungi (Sarim 2013). Beberapa bakteri yang
telah dilaporkan memiliki kemampuan sebagai agen pengendali hayati terhadap G. boninense antara lain adalah Pseudomonas (Bivi et al. 2010; Sapak et al. 2008),
Bacillus sp. (Susanto et al. 2005; Suryanto et al. 2012) dan Burkholderia (Buana et al. 2014). Pencarian bakteri antagonis G. boninense hingga saat ini masih perlu dilakukan sebagai langkah eksplorasi mikroba potensial dari alam untuk kemudian digunakan sebagai agen pengendali hayati terhadap cendawan G. boninense dalam rangka menata kembali keseimbangan ekosistem mikroba di lapangan (Sarim 2013).
Bakteri antagonis umumnya menyandikan senyawa antifungi yang dipengaruhi oleh gen-gen tertentu. Pemahaman tentang pengaruh gen yang berkontribusi terhadap aktivitas antifungi penting dipelajari sebagai dasar pengembangan strategi pengendalian yang lebih baik, yaitu melalui pendekatan berbasis bioteknologi molekuler.
Pada penelitian ini, dilakukan pencarian kandidat bakteri antagonis G. boninense yang menghasilkan senyawa antifungi yang diduga berupa protein serta identifikasi gen yang berperan dalam aktivitas antifungi menggunakan teknik shotgun cloning. Pada waktu yang bersamaan, dilakukan karakterisasi fragmen DNA dari mutan transposon BglK226 (Koleksi lab. PT. WBE). Mutan ini merupakan hasil mutagenesis transposon dari Burkholderia gladioli JR01 yang menunjukkan peningkatan aktivitas penghambatan terhadap hifa G. boninense.
Perumusan Masalah
Indonesia merupakan negara produsen minyak kelapa sawit terbesar di dunia, namun penyakit BPB oleh Infeksi G. boninense telah menyebabkan kerugian besar terhadap produktivitas tanaman kelapa sawit. Bakteri dengan kemampuan antagonistik terhadap G. boninense memiliki potensi sebagai agen pengendali hayati terhadap penyakit BPB kelapa sawit, akan tetapi aplikasi bakteri secara langsung dilapangan relatif belum menunjukkan hasil yang menjanjikan. Telaah secara bioteknologi molekular terhadap pengembangan strategi pengendalian hayati melalui karakterisasi dan identifikasi gen yang berkontribusi dalam aktivitas antifungi G. boninense perlu dilakukan.
Tujuan Penelitian
Penelitian ini bertujuan untuk isolasi, identifikasi bakteri antagonistik G. boninense dan kloning sejumlah fragmen DNA bakteri potensial dengan teknik shotgun cloning serta karakterisasi fragmen DNA mutan BglK226 menggunakan teknik genome walking.
Manfaat Penelitian
3 pengapit mutan BglK226 yang merupakan informasi awal dalam pengembangan pengendalian hayati berbasis molekuler untuk kemudian mampu diaplikasilkan dalam mengatasi permasalahan penyakit BPB pada areal perkebunan tanaman kelapa sawit.
Ruang Lingkup Penelitian
4
2 TINJAUAN PUSTAKA
Kelapa sawit (Elaeis guineensis) merupakan tanaman monokotil, termasuk kedalam famili Arecaceae (klasifikasi sebelumnya Palmae) (Corley & Tinker 2003). Tanaman tersebut berasal dari Afrika barat dan tengah, tetapi kemudian mengalami penyebaran hingga keseluruh daerah tropis (Ariffin et al. 2000) termasuk Indonesia (Darmono 2000). Luas areal perkebunan kelapa sawit di Indonesia terus mengalami peningkatan. Pada tahun 1997 berkisar 2.463.823 ha, meningkat menjadi lebih dari 9 juta ha pada tahun 2013 (Darmono 2000; Ditjenbun 2014). Waktu yang dibutuhkan untuk setiap generasi tanaman produktif antara 25 dan 30 tahun (Darmono 2000).
Kelapa sawit telah menjadi tanaman komoditi penghasil minyak nabati terbesar di dunia. Indonesia merupakan negara terbesar penyumbang permintaan minyak sawit dunia yaitu sebesar 47%. Namun serangan berbagai penyakit pada perkebunan kelapa sawit menjadi penyebab utama penurunan produksi. Salah satu penyakit yang menyebabkan kerugian besar adalah infeksi busuk pangkal batang (BPB) oleh cendawan Ganoderma. Pada beberapa perkebunan di Indonesia, serangan BPB dapat mencapai 50% pada tanaman yang masih produktif dan status penyakit BPB tidak hanya menyerang tanaman tua, diketahui juga dapat menyerang tanaman yang masih muda. Penampakan gejala penyakit muncul lebih awal pada perkebunan generasi yang lebih banyak mengalami penanaman ulang (replanting). Semakin sering suatu areal perkebunan mengalami peremajaan maka semakin tinggi persentase kejadian penyakit BPB (Soepena et al. 2000; Susanto et al. 2005).
Busuk pangkal batang pertama kali dilaporkan pada tahun 1931 di Malaysia dan diidentifikasi disebabkan oleh Ganoderma lucidum (Thompson 1931), lebih lanjut diketahui bahwa beberapa spesies Ganoderma dapat berasosiasi dengan spesies lain pada tanaman yang terserang penyakit BPB yaitu Ganoderma boninense, G. miniatocinctum, G. zonatum, G. chalceum, G. xylonoides. Namun demikian, BPB pada areal perkebunan di Indonesia disebabkan oleh infeksi cendawan G. boninense (Utomo et al. 2005; Cooper et al. 2011).
Ganoderma boninense merupakan cendawan yang termasuk dalam kelas basidiomycetes, saprofitik dan memiliki tubuh buah, memiliki kisaran inang yang luas dan juga struktur khusus yang berdampak pada kemampuan bertahan dan menginfeksi tanaman target seperti spora istirahat berupa klamidospora dan struktur pseudosklerotia (Darmono 2000; Susanto et al. 2005; Rees et al. 2009).
Gejala dini penyakit BPB sukar dideteksi. Adanya tubuh buah yang terbentuk, daun pupus yang tidak membuka, nekrotis pada daun tua dimulai dari bagian bawah dan jatuh menggantung merupakan gejala lanjut yang dapat menyebabkan kematian pada tanaman dewasa (Gambar 1) (Cooper et al. 2011).
5 cendawan busuk putih, inokulum yang berada di tanah akan menginfeksi bagian akar dan mendegradasi komponen lignin pada akar (Paterson 2007).
Gambar 1 Penampakan serangan infeksi G. boninense pada tanaman kelapa sawit (http://ganodermadisease.blogspot.co.id/)
Penyebaran serangan pada bagian korteks, endoderm, perisikel dan jaringan pengangkut akan menyebabkan terganggunya transfortasi air dan nutrisi hingga menyebabkan munculnya gejala penyakit (Sariah et al.1994; Pilotti 2005; Rees et al. 2009; Cooper et al. 2011). Basidiospora yang menyebar melalui angin dan air juga diketahui menjadi salah satu faktor penentu yang berkontribusi terhadap penyebaran inokulum (Rees et al. 2011).
Gambar 2 Penampakan tubuh buah G. boninense pada tanaman kelapa sawit (http://pupuksawit.nasaorganicsolution.com/ancaman-penyakit-busuk-pangkal-batang-pada-tanaman-kelapa-sawit. html)
6
boninense antara lain adalah Pseudomonas (Bivi et al. 2010), Bacillus sp. (Suryanto et al. 2012) dan Burkholderia (Buana et al. 2014).
Perkembangan penemuan teknik dibidang biologi molekular menyebabkan telaah dan pemahaman tentang gen-gen yang berkontribusi terhadap aktivitas antifungi bakteri antagonistik G. boninense merupakan penelitian yang penting untuk dikembangkan. Hal ini dilakukan sebagai salah satu langkah strategi pengembangan pengendalian hayati untuk menghasilkan produk biologis yang lebih efektif. Identifikasi dan karakterisasi gen yang terlibat dalam aktivitas penghambatan hifa G. boninense dapat dilakukan menggunakan shotgun cloning dan mutagenesis dengan menggunakan transposon.
Shotgun cloning merupakan teknik yang telah diperkenalkan pada tahap awal munculnya teknologi DNA rekombinan dan bahkan ketika informasi secara bioinformatik suatu urutan protein target sangat tarbatas dan atau belum diketahui. Teknik tersebut memiliki tingkat resiko yang lebih sulit dibandingkan dengan teknik PCR, tetapi mengarah ke hasil yang mengejutkan dengan kemungkinan menemukan gen baru (Whitehurst & Oort 2010). Prinsip dasar dari teknik tersebut adalah kloning DNA kromosom, ekstraksi dengan metode standar dan pemotongan secara parsial menggunakan enzim restriksi. Pemotongan DNA kromosom akan menghasilkan sejumlah fragmen yang memiliki ukuran bervariasi dan saling tumpang tindih. Penyambungan antara DNA kromosom dan plasmid yang diintroduksikan kedalam inang tertentu akan menghasilkan pustaka DNA (Whitehurst & Oort 2010; Al-bayaty et al. 2012). Identifikasi fragmen yang mengandung gen target dilakukan dengan seleksi melalui analisis ekspresi dengan plating pada media selektif.
Optimasi kondisi pada shotgun cloning merupakan hal penting yang menentukan keberhasilan dalam memperoleh pustaka klon, baik pemilihan enzim restriksi yang digunakan untuk pemotongan secara parsial maupun pemilihan vektor dan inang. Sau3A1 merupakan enzim restriksi yang biasa digunakan untuk pemotongan DNA kromosom. Sistem vektor-inang yang digunakan adalah E. coli dengan vektor yang mengandung gen lac Z yang dapat mempermudah seleksi transforman menggunakan seleksi biru putih (Whitehurst & Oort 2010).
Teknik lain yang dapat dilakukan yaitu mutagenesis transposon. Transposon merupakan suatu fragmen DNA yang memiliki kemampuan untuk berpindah dari satu situs DNA kebagian DNA yang lain dalam satu sekuen maupun berbeda sekuen. Peristiwa penyisipan tersebut dikenal dengan transposisi yang dikatalisis oleh enzim transposase. Salah satu transposon yang sering digunakan adalah Tn5 yang memiliki ukuran 5700 pb, membawa tiga gen resistensi antibiotik yaitu kanamisin, bleomisin dan streptomisin (Vizvaryova & Valkova 2004). Daerah transposon Tn5 dibatasi oleh 19 pb direct repeat (CTGTCTCTTGATCAGATCT) insite end dan (ACTTGTGTATAAGAGTCAG) outsite end yang merupakan urutan untuk verifikasi keberhasilan identifikasi DNA pengapit (DeLorenzo et al. 1990). Salah satu turunan dari Tn5 adalah mini-Tn5, dimana gen yang menyandikan enzim transposase terletak diluar mini-transposon yang dikonstruksi pada vektor plasmid (Delorenzo et al. 1990; Herrero et al. 1990).
7 akan menempel pada bagian iteron oriR6K sebagai syarat utama jalannya replikasi DNA. Plasmid tersebut akan terdegradasi ketika bakteri inang tidak mampu melakukan replikasi (Ya-bin 1998; Kruger & Filutowicz 2000).
Penggunaan suicide plasmid dalam proses transposisi merupakan faktor penting. Hal ini disebabkan karena jika plasmid yang digunakan masih terdapat dalam sel resipien terdapat kemungkinan transposon akan menyisip berulang kali karena enzim transposase terus diproduksi yang akan sangat mengganggu kestabilan penyisipan transposon dalam DNA resipien (Herrero et al. 1990).
Gambar 3 Peta fisik plasmid pUTmini-Tn5KmR (Artiguenave et al. 1997) Mekanisme perpindahan plasmid pUT yang mengandung mini-Tn5 dari bakteri donor ke resipien terjadi melalui proses konjugasi. Bakteri yang digunakan sebagai donor harus memiliki faktor F (fertility factor), sehingga proses konjugasi dapat berlangsung dengan cara biparental mating. Faktor F terdiri dari gen tra dan oriT (origin of transfer). Komponen gen tra antara lain adalah Dtr (DNA transfer and replication) dan Mpf (Mating pair formation). Enzim yang termasuk kedalam kelompok Dtr adalah relaksase yang berfungsi dalam inisiasi pembentukan nick pada daerah oriT, kemudian membawa DNA melalui pili menuju sel donor, sementara itu primase berfungsi membantuk primer yang akan digunakan untuk proses replikasi plasmid di sel resipien dan donor. Komponen Mpf mengandung enzim yang berfungsi untuk membentuk pillus yang akan digunakan sebagai jembatan dalam proses konjugasi (Snyder & Champness 2007).
Bakteri transkonjugan yang telah mengalami transposisi dapat diseleksi dengan menggunakan biomarker yang dibawa oleh transposon yang biasanya berupa gen resistensi terhadap antibiotik (Vizvaryova & Valkova 2004). Keacakan penyisipan mini-Tn5 pada sejumlah bakteri transkonjugan dapat dianalisis dengan menggunakan teknik genome walking, merupakan teknik yang efisien dan lebih akurat untuk identifikasi daerah sekuen gen yang belum diketahui informasi urutannya. Teknik tersebut berbasis amplifikasi PCR tanpa harus membuat kunstruksi pustaka DNA. Lebih lanjut lagi bahwa teknik ini lebih cocok digunakan untuk mengamplifikasi fragmen DNA target dengan sedikit info sekuen gen yang dibutuhkan (Clontech 2007).
8
dengan PCR berbasis adaptor disajikan pada Gambar 4. Sekuen adaptor spesifik disajikan pada Gambar 5.
Gambar 4 Diagram protokol Genome walker (Clontech 2007). AP (primer adaptor); GSP (primer spesifik gen)
9
3 METODE PENELITIAN
Kerangka Penelitian
Kerangka penelitian disajikan pada Gambar 6.
Gambar 6 Diagram alur penelitian DNA kromosom Isolasi bakteri
Penapisan bakteri
Bacillus amyloliquefaciens Mutan BglK226
Seleksi transforman
Pemotongan dengan SnaB1, EcoRV dan Fsp1 Pemotongan parsial
dengan Sau3A1 Identifikasi gen 16S rRNA
Pengujian aktivitas antifungi bakteri
rekombinan
Kloning fragmen DNA kromosom pada vektor pRK415
Amplifikasi dengan primer ASP dan GSP
Kloning fragmen DNA pengapit pada vektor pGEM-T Easy Verifikasi sisipan
vektor pRK415
Amplifikasi plasmid dengan primer ASP2 dan GSP2
10
Waktu dan Tempat Penelitian
Penelitian ini dilaksanakan pada bulan Agustus 2014 hingga Juli 2015 di Laboratorium PT WBE Cikarang, Bekasi.
Kultur Cendawan, Bakteri, Plasmid dan Media
Cendawan G. boninense B29 merupakan isolat koleksi laboratorium WBE (Purnamasari et al. 2012). Kultur cendawan diremajakan setiap 15 hari pada media potato dextrose agar (PDA) dengan suhu inkubasi 28ºC. Mutan BglK226 (Koleksi WBE) digunakan untuk karakterisasi DNA pengapit mini-Tn5KmR, ditumbuhkan pada media LA dengan penambahan kanamisin 50µgmL-1 pada suhu 30 ºC. Plasmid pRK415 (Keen et al. 1988), pGEM-T Easy (Promega, Madison WI) dan pGEM-T Easy rekombinan diperbanyak pada inang E. coli Top10 (Invitrogen). Media king’s B, PDA, Soil Extract Agar (SEA) dan Luria bertani agar (LA) dengan atau tanpa penambahan antibiotik digunakan dalam skrining bakteri dan peremajaan kultur.
Isolasi dan Skrining Bakteri Antagonis
Isolasi bakteri dilakukan dengan teknik pengenceran bertingkat dengan tiga jenis sumber isolasi yaitu bag-log media tanam cendawan makroskopis (culture material of edible fungi) cendawan lingzhi (Ganoderma lucidum) cendawan kuping (Auricularia auriculata), tanah rumah kaca, dan janjangan kosong kelapa sawit. Isolasi dilakukan pada tiga jenis media agar yaitu media king’s B, king’s B 10% dan soil extract.
Pemilihan bakteri uji dilakukan berdasarkan kemampuan antagonisnya terhadap G. boninense secara in vitro pada media PDA. Penapisan bakteri dilakukan dalam empat tahapan yaitu uji tantang dalam delapan kuadran, uji tantang dalam empat kuadran dan uji tantang dua kultur sesuai dengan Jinantana dan Sariah (1997).
Tepi bagian yang aktif tumbuh pada biakan cendawan yang telah berumur 7 hari diambil dengan menggunakan cork borer, diinokulasikan pada media PDA tepat ditengah-tengah cawan petri. Selanjutnya suspensi bakteri dibuat dengan standar McFarland dengan konsentrasi ≈ 108 cfu mL-1 (Sapak et al. 2008; Samala et al. 2011). Suspensi diinokulasikan pada cawan petri yang telah ditumbuhkan G.boninense dengan teknik streak garis lurus yang berjarak 3cm dari inukulum cendawan. Akitivitas penghambatan ditentukan berdasarkan zona hambat yang terbentuk di sekitar koloni. Pengamatan dilakukan setelah sepuluh hari inkubasi. Persentase penghambatan pertumbuhan radial (PIRG) hifa G. boninense dengan nilai lebih dari 50% akan dipilih sebagai kandidat inukulum bakteri potensial untuk uji selanjutnya. Data penghambatan yang ditransformasikan dalam bentuk persentase menggunakan rumus Jinantana dan Sariah (1998):
���� =R −RR x 100%
Ket: R1= Pertumbuhan radial hifa G. boninense tanpa biokontrol
11 Uji aktivitas ekstrak bakteri dilakukan dengan mengacu pada Bivi et al. (2010) dengan modifikasi protokol WBE yaitu dengan menggunakan tiga perlakuan, supernatan dengan tanpa pemanasan (diinkubasi pada suhu ruang 28±2oC); supernatan dengan pemanasan pada suhu 950C selama 15 menit; supernatan dengan penambahan proteinase K. Isolat bakteri E. coli, media kosong dan aquades digunakan sebagai kontrol negatif sedangkan isolat CP01 (Koleksi WBE) dan atau fungisida Dithane (Dow Agroselences, Indonesia) digunakan sebagai kontrol positif.
Identifikasi Gen Penyandi 16S rRNA
Identifikasi spesies bakteri terpilih dilakukan melalui analisis gen penyandi 16S rRNA sesuai deskripsi Marchesi et al. (1998). Genom bakteri diisolasi dengan protokol standar Wizard Genomic DNA Purification Kit (Promega, USA), kemudian dilakukan visualisasi dengan elektroforesis pada gel agarosa 1% (w v-1). Konsentrasi dan kemurnian DNA dianalisis menggunakan NanoDrop 2000 (Thermo Scientific, Wilmington, DE, USA).
Amplifikasi gen penyandi 16S rRNA dilakukan dengan PCR menggunakan primer 63F (5’-CAGGCCTAACACATGCAAGTC-3’) dan primer 1387R (5-GGGCGGAA/TGTGTACAGGC-3’) (Marchesi et al. 1998). Komposisi reaksi PCR (total volume reaksi 50 µL) terdiri dari 1 µL DNA genom bakteri (500 ng/µL) sebagai cetakan, 25 µL 2x GoTaq Green PCR mastermix (Promega), 1 µL (10 picomol) masing-masing primer 63F dan 1387R dan 22 µL dH2O. Gradien temperatur yang digunakan adalah pradenaturasi pada suhu 95 oC selama 5 menit, siklus amplifikasi sebanyak 25 siklus yang terdiri dari suhu denaturasi 95 oC selama 30 detik; penempelan 55 oC selama 30 detik; pemanjangan 72 oC selama 60 detik dan pemanjangan akhir 72 oC selama 5 menit (Clontech 2007). Verifikasi dilakukan dengan elektroforesis gel agarosa 1% (wv-1), kemudian produk PCR dimurnikan mengikuti prosedur purifikasi ExoSAP-IT (ExoSAP-IT, USA).
Hasil purifikasi exosap diurutkan dengan PCR cycle dengan prinsip terminal chain reaction menggunakan tiga primer yaitu 63F, 926R dan 1387R. Purifikasi akhir hasil PCR cycle dilakukan menggunakan Xterminator purification kit. Pengurutan nukleotida dilakukan dengan perangkat ABI Prism 3030 Genetic Analyser (Applied Biosystems, USA). Analisis hasil pengurutan dilakukan dengan menggunakan software geneious ProTM5.3.4 untuk mendapatkan file fasta, kemudian dilakukan Basic Local Alignment Search Tool (BLAST) file fasta urutan nukleotida dari hasil pengurutan gen 16S rRNA dengan data base yang tersedia pada gene bank.
Shotgun CloningBacillusamyloliquefaciens JR02
Pemotongan DNA kromosom secara parsial
12
pada suhu 370C selama 10 menit, kemudian dielektroforesis pada gel agarosa 0,7%. Pita DNA kromosom hasil elektroforesis dipotong pada ukuran yang diinginkan (3-8kb), kemudian dimurnikan mengikuti protokol standar QIAquick® Gel Extraction Kit (Qiagen, Valencia, California).
Kloning fragmen DNA kromosom
Plasmid pRK415 yang merupakan turunan dari pRK404 (Ditta et al. 1985) dipakai sebagai vektor kloning sejumlah potongan DNA kromosom JR02. Plasmid tersebut dibuat linear dengan cara memotong pada daerah MCS situs pengenalan enzim restriksi BamH1 (New England Biolabs. USA). Fragmen DNA diligasikan pada daerah Multiple Cloning Site (MCS) plasmid pRK415 (Gambar 7). Reaksi ligasi terdiri dari 4 µL fragmen DNA (100 ng µL-1), 0.5 µL vektor pRK415 (100 ng µL-1), 2 µL bufer ligasi 10X, 1 µL T4 DNA ligase (NEB, USA), dan 12.5 µL ddH2O. Proses ligasi dilakukan dengan perlakuan inkubasi selama 16 jam pada suhu 4 oC. E. coli TOP10 kompeten diperoleh dengan perlakuan CaCl2 (Lampiran 1). Plasmid rekombinan diintroduksikan ke dalam sel kompeten dengan perlakuan renjatan panas (heat shock) pada suhu 42 oC selama 90 detik. Seleksi bakteri transforman dilakukan dengan seleksi biru putih (blue white selection) pada media LA+Tc (10 µgml-1) + X-gal (80 µgml-1). Tranforman dengan koloni warna putih dipanen dan ditumbuhkan pada media LA+Tc.
Gambar 7 Peta fisik vektor pRK415 (Keen et al. 1988)
Uji antagonis dilakukan menggunakan suspensi G. boninense sesuai dengan protokol laboratorium beneficial microbe PT WBE. Koloni yang menghasilkan zona penghambatan mengindikasikan bahwa transforman memiliki kemampuan dalam menghambat pertumbuhan hifa G. boninense dan mampu mengekspresikan gen antifungi asal isolat JR02.
13
Verifikasi sisipan DNA kromosom pada E. coli TOP 10 rekombinan
Isolasi plasmid rekombinan dari E. coli TOP 10 dilakukan dengan tujuan untuk melakukan verifikasi sisipan pada vektor rekombinan. Isolasi plasmid dilakukan pada koloni E. coli TOP10 yang berwarna putih di media Luria Bertani agar (LA) selektif. Bakteri E. coli TOP10 ditumbuhkan dalam 10 mL media LB yang mengandung 10 µg mL-1 tetrasiklin pada suhu 37 oC selama 12-16 jam. Plasmid rekombinan diisolasi dengan mengikuti protokol standar QIAprep Spin miniprep Kit (Qiagen, Valensia, California). Pemotongan DNA plasmid dilakukan dengan enzim restriksi Sma1 (NEB, USA) dan dilakukan analisis ukuran sisipan dengan visualisasi elektroporesis gel agarosa 1%.
Karakterisasi Mutan BglK226 Uji aktifitas mutan
Uji antagonis mutan BglK226 dan Burkholderia gladioli JR01 (tipe asal) terhadap G. boninense dilakukan untuk mengetahui peningkatan kemampuan aktivitas antifungi mutan BglK226 setelah disisipi oleh transposon mini-Tn5KmR. Metode yang digunakan adalah dual culture assay yang mengacu pada Bivi et al. (2010). Data penghambatan yang ditransformasikan dalam bentuk persentase menggunakan rumus Jinantana dan Sariah (1998).
Deteksi dan Amplifikasi Sekuen Pengapit Transposon mini-Tn5KmR
Sekuen DNA pengapit transposon diidentifikasi menggunakan teknik genome walking dengan penambahan adaptor spesifik sesuai protokol Clontech universal genome walker (Clontech Laboratories Inc, USA). Isolasi DNA kromosom mutan BglK226 dilakukan dengan metode presipitasi etanol menggunakan Wizard Genomic DNA Purification Kit (Promega, USA), kemudian dipotong menggunakan enzim restriksi SnaBI, EcoRV dan FspI (Fermentas, Ontario, Canada) secara terpisah. Komposisi masing-masing reaksi terdiri dari 5 µL DNA cetakan (1000 ng µL-1), 2 µL bufer 10X, 0.2 µL BSA, 1 µL enzim restriksi, dan 11.8 µL ddH2O, kemudian diinkubasi selama 16 jam pada suhu yang disesuaikan. Fragmen-fragmen DNA disambung dengan campuran adaptor (Clontech, USA) menggunakan T4 DNA ligase (NEB, USA) dan digunakan sebagai cetakan dalam amplifikasi PCR genome walking. Reaksi ligasi terdiri dari 4 µL fragmen DNA hasil pemotongan, 1.9 µL campuran adaptor, 1.6 µL bufer ligasi, 0,5 µL T4 DNA ligase (NEB, USA), dan 8 µL ddH2O. Proses ligasi dilakukan dengan perlakuan inkubasi selama 16 jam pada suhu 16 oC dan inaktivasi reaksi dilakukan pada suhu 70 oC selama 5 menit.
Kombinasi primer yang digunakan pada saat amplifikasi terdapat pada Tabel 1. Reaksi PCR dilakukan dalam dua tahapan. Reaksi pertama dilakukan dengan menggunakan primer spesifik gen 1 (GSP1) dan primer spesifik adaptor (ASP1). Reaksi PCR pertama dengan volume akhir 20 µL, terdiri dari 10 µL 2x GoTaq Green PCR mastermix (Promega), 1 µL 10pmol GSP1, 1 µL 10pmol ASP1 (Clontech, USA), 1 µL DNA cetakan (adaptor yang telah terligasi dengan DNA), 7 µL ddH2O. Kondisi PCR yang digunakan adalah predenaturasi 94 oC, 3 menit, diikuti dengan 7 siklus (94 oC, 25 detik, 72 oC, 3 menit) 32 siklus (94 oC, 25 detik, 67 oC, 3 menit) dan pemanjangan akhir 67 oC, 7 menit (Clontech 2007).
14
primer GSP2 dan ASP2 dengan komposisi reaksi terdiri dari 10 µL 2x GoTaq Green PCR mastermix (Promega), 1 µL 10pmol GSP1, 1 µL 10pmol ASP1 (Clontech, USA), 1 µL DNA cetakan (produk PCR pertama), 7 µL ddH2O. Kondisi PCR yang digunakan adalah predenaturasi 94 oC, 3 menit, diikuti dengan 5 siklus (94 oC, 25 detik, 72 oC, 3 menit) 20 siklus (94 oC, 25 detik, 67 oC, 3 menit) dan pemanjangan akhir 67 oC, 7 menit (Clontech 2007). Kontrol internal disertakan pada masing-masing amplifikasi untuk mengetahui adanya faktor lain yang mempengaruhi reaksi. Visualisasi elektroforesis dilakukan pada gel agarosa 1% dengan pewarnaan ethidium bromide (EtBr). Pemurnian gel dilakukan sesuai Qia Quick Gel Extraction Kit (Qiagen, Valencia, California).
Tabel 1 Primer yang digunakan untuk amplifikasi DNA pengapit transposon mini-Tn5KmR
Primer Sekuens (5’ 3’)
Asp1 GTAATACGACTCACTATAGGGC
Asp2 ACTATAGGGCACGCGTGGT
GSP_1F GCCCGATGCGCCAGAGTTGT
GSP_1R GGTCTGCGATTCCGACTCCGACTCGTCC
GSP_2F TATTGATGTTGGACGAGTCGGAATC
GSP_2R CGTTTCCCGTTGAATATGGCTCAT
Kloning Fragmen DNA Pengapit Transposon
Fragmen DNA hasil amplifikasi yang telah dimurnikan disambungkan dengan vektor pGEM-T Easy pada daerah Multiple Cloning Site (MCS) (Gambar 8). Reaksi ligasi terdiri dari 2 µL fragmen DNA (50 ng µL-1), 0.5 µL vektor pGEM-T Easy (50 ng µL-1), 2 µL bufer ligasi 10X, 1 µL T4 DNA ligase (NEB, USA), dan 14.5 µL ddH2O. Proses ligasi dilakukan dengan perlakuan inkubasi selama 16 jam pada suhu 16 oC. Plasmid rekombinan diintroduksikan kedalam sel E. coli TOP10 kompeten dengan metode renjatan panas (heat shock). Seleksi bakteri transforman dilakukan dengan seleksi biru putih seperti dideskripsikan oleh Sambrook et al. (1989). Sel E. coli kompeten ditumbuhkan pada media LA dengan dan tanpa penambahan antibiotik ampisilin sebagai kontrol transformasi.
15
Analisis pengurutan DNA pengapit
Pengurutan nukleotida DNA pengapit transposon dilakukan menggunakan perangkat ABI Prism™ 3030 Genetic Analyzer (Applied Biosystems, Foster City, California). Plasmid rekombinan pJR226-01 digunakan sebagai cetakan, GSP2 dan ASP2 digunakan sebagai primer untuk Cycle sequensing PCR dengan mengikuti protokol standar BigDyeR Terminator Cycle Sequensing Kit v3.1(Applied Biosystems, California, USA). Kondisi Cycle sequensing PCR adalah predenaturasi 96 oC, 5 menit, diikuti dengan 25 siklus PCR (96 oC, 30 detik, 50 oC, 30 detik, 61 oC, 30, detik) dan 72 oC, 10 menit pemanjangan akhir. Pemurnian produk Cycle sequensing PCR dilakukan mengikuti protokol standar BigDyeR X-Terminator Purification kit (Applied Biosystems, Foster City,
16
4 HASIL DAN PEMBAHASAN
Isolasi,Penapisan dan Identifikasi Bakteri Antagonis G. boninense
Keberadaan mikroba yang melimpah pada suatu ekosistem menjadi bagian yang sangat penting dalam menjaga keseimbangan suatu populasi organisme lain dalam habitat tertentu melalui suatu mekanisme kompleks. Ekplorasi mikroba dari berbagai habitat menjadi sangat penting untuk mempelajari efektivitasnya dalam menekan suatu organisme patogen pada tanaman budidaya. Hal ini merupakan langkah awal dalam usaha pengendalian G. boninense secara biologis (Susanto et al. 2005). Pada kondisi alami di alam, beberapa mikroba memiliki kemampuan dalam menekan busuk pangkal batang kelapa sawit yang disebabkan oleh G. boninense (Bivi et al. 2010).
Pada penelitian ini, 198 isolat bakteri berhasil diisolasi dari tiga jenis sampel yang digunakan. Perbedaan warna, tipe koloni dan ukuran koloni menjadi dasar perbandingan untuk analisis keberagaman bakteri dari setiap sumber isolasi yang digunakan (Bivi et al, 2010). Sebanyak 41 isolat bakteri menunjukkan aktifitas penghambatan dengan tingkat yang berbeda-beda. Enam belas isolat menunjukkan nilai Percentage inhibition of radial growh (PIRG) > 50% (Tabel 2). Bakteri yang memiliki potensi untuk digunakan sebagai agen biokontrol menunjukkan aktivitas penghambatan PIRG >40% (Sutanto et al. 2005).
Tabel 2 Aktivitas penghambatan isolat potensial terhadap miselium G. boninense
No Isolat Aktivitas penghambatan (%)*
1 Akuades -
* Zona hambat setelah inkubasi selama 10 hari
17 Dua isolat dengan kode G.1.22 dan G.1.7 memiliki nilai PIRG tertinggi setelah sepuluh hari masa inkubasi (Gambar 9). G.1.22 menunjukkan aktivitas penghambatan mencapai 87,86%, diikuti isolat G.1.7 dengan nilai penghambatan 82,92%. Kedua isolat tersebut berasal dari bag-log media cendawan G. lucidum. Keberadaan bakteri antagonis G. boninense pada media bag-log cendawan belum pernah dilaporkan sebelumnya.
Gambar 9 Aktivitas penghambatan bakteri antagonis pada hari ke 10 pada media PDA terhadap miselium G. boninense; (a) kontrol miselium cendawan G. boninense; (b) Isolat G.1.22; (c) Isolat 6.1.7
Efek penghambatan keseluruhan isolat potensial kemungkinan memiliki mekanisme yang berbeda-beda. Hal ini didukung dari penampakan abnormalitas (nekrotik) morfologi dari hifa G. boninense. Bivi et al. (2010) melaporkan bahwa berdasarkan hasil pengamatan hifa secara mikroskopis ditemukan adanya distorsi dan lisis pada ujung hifa, bengkak, percabangan abnormal G. boninense setelah kontak dengan Pseudomonas aeruginosa. Disisi lain, hasil pengamatan menunjukkan bahwa beberapa isolat bakteri tidak perlu kontak langsung dengan hifa untuk menunjukkan adanya aktivitas penghambatan, namun beberapa jenis lain justru sebaliknya, kontak langsung dengan hifa untuk menunjukkan adanya aktivitas penghambatan. Perbedaan tersebut menunjukan bahwa fenomena jenis dan waktu sekresi senyawa antifungi kemungkinan sangat bervariasi dan dipengaruhi oleh media pertumbuhan dan terlibat dalam regulasi ekspresi gen yang berbeda-beda.
Lima isolat terpilih diuji lanjut dengan uji filtrat untuk mengetahui aktivitas penghambatan pada ekstrak kasar filtrat bakteri. Hasil yang diperoleh disajikan pada Tabel 3. Ekstrak kasar G.1.22 menunjukkan aktivitas penghambatan tertinggi yang hampir setara dengan aktivitas fungisida dithane 0.1%, tahan terhadap pemanasan dan tidak sensitif terhadap penambahan proteinase K. Hal ini sesuai dengan Kadir et al. (2008) yang melaporkan ekstrak kasar senyawa antifungi Burkholderia cepacia tahan terhadap pemanasan, enzim proteolitik, dan senyawa pelarut organik (Quan et al. 2006). Senyawa antifungi dengan karakteristik tersebut tergolong kedalam senyawa antifungi non-protein (non- ribosomal peptide), salah satunya adalah pyrolnitrin yang telah dilaporkan dimiliki oleh bakteri Burkholderia dan Pseudomonas (Roitman et al. 1990; Quan et al. 2006).
Sebaliknya yang menarik adalah ekstrak kasar isolat K.3.2 justru sensitif terhadap pemanasan dan penambahan proteinase K sehingga diasumsikan memiliki senyawa antifungi berupa protein. Berdasarkan penelitian-penelitian yang telah dilakukan, senyawa antifungi yang berupa protein telah dilaporkan dihasilkan dari beberapa spesies Bacillus. Liu et al. (2007) melakukan pendekatan dengan isolasi protein, diketahui bahwa Bacillus subtilis memiliki senyawa
18
antifungi protein berupa bacisubin. Lebih lanjut Wong et al. (2008) juga melaporkan bahwa Bacillus amyloliquefaciens memiliki senyawa antifungi protein baciamin. Namun hingga sekarang, ketersedian sekuen secara bioinformatik gen lengkap dari senyawa-senyawa antifungi protein masih sangat terbatas.
Tabel 3 Aktivitas penghambatan ekstrak kasar lima bakteri terpilih terhadap G. boninense
Kode isolat Zona hambat (mm)
Tanpa perlakuan Pemanasan 950C selama 15 menit
* Zona hambat setelah inkubasi selama 3 hari
Kontrol negatif mengunakan E coli, Akuades, LB medium, dan kontrol positif menggunakan fungisida.
Berdasarkan hasil uji tantang kultur ganda dan uji filtrat, isolat G.1.22 dan K.3.2 dipilih untuk identifikasi lebih lanjut. Hasil amplifikasi PCR Isolat G.1.22 dan K.3.2 menggunakan primer 16S (63F dan 1387F) menghasilkan pita ukuran ±1300pb (Gambar 10).
Gambar 10 Hasil amplifikasi gen penyandi 16S rRNA; M (Marker 100pb); 1 (Isolat G.1.22); 2 (Isolat K.3.2)
Hasil identifikasi gen penyandi 16S rRNA dan analisis kesejajaran lokal berdasarkan urutan nukleotida gen 16S rRNA menggunakan program BLAST (Basic Local Alignment Search Tool) menunjukan bahwa isolat G.1.22 dan K.3.2 memiliki tingkat kemiripan 100% dengan Pseudomonas aeruginosa strain B13
(accession number KM588370) dan Bacillus amyloliquefaciens strain SCDB1439
(accession number KM922581). Berdasarkan uji antagonis yang dilakukan secara
in vitro, kedua isolat tersebut memiliki potensi sebagai agen biokontrol ±1300pb
19 G. boninense. P.aeruginosa menunjukkan kemampuan dalam menekan BPB pada bibit kelapa sawit, dengan cara menghasilkan senyawa antibiotik yang dapat menekan patogen tanaman (Harrison et al. 1993). P. aeruginosa dan Bacillus termasuk kelompok plant growth promoting rhizobacterium (PGPR), yang berperan secara langsung maupun tidak langsung pada tanaman. (Sapak et al. 2008; lugternberg & Kamilova 2009).
Bacillus amyloliquefaciens strain SCDB1439 hasil identifikasi isolat K.3.2 yang kemudian disebut sebagai B. amyloliquefaciens JR02 digunakan untuk pekerjaan shotgun cloning sebagai langkah awal untuk identifikasi gen yang berkontribusi terhadap aktivitas antifungi. Bakteri tersebut diasumsikan memiliki senyawa antifungi protein yang disandikan oleh satu gen melalui mekanisme sintesis secara ribosomal.
Shotgun CloningBacillus amyloliquefaciens JR02.
Kloning fragmen DNA kromosom JR02 dilakukan dengan metode shotgun cloning. Hasil pemotongan DNA kromosom secara parsial dengan menggunakan enzim restriksi Sau3A1 menghasilkan pola pita smear (Gambar 11). Hasil verifikasi sisipan dari plasmid rekombinan pRK415 menunjukkan adanya sisipan dengan ukuran yang bervariasi (Gambar 12).
Gambar 11 Pemotongan DNA kromosom JR02 secara parsial menggunakan ER Sau3A1. M (marker); 1(DNA kromosom dipotong secara parsial); 2 (kontrol negatif).
Fragmen DNA JR02 yang digunakan sebagai sisipan adalah hasil pemotongan Sau3A1 yang dimurnikan langsung dengan ekstraksi potongan gel DNA hasil elektroforesis ukuran 3-8 kb. Hal ini didasarkan bahwa target gen senyawa antifungi yang diharapkan berukuran ± 1-2 kb (Liu et al. 2007 ; Wong et al. 2008). Hasil transformasi antara campuran reaksi ligasi yang berisi fragmen sisipan dan pRK415 dengan E. coli TOP10 diseleksi menggunakan seleksi biru putih. Untuk meningkatkan efisisensi transformasi, inkubasi dilakukan pada suhu 40C selama 16-18 jam (Dagert & Ehrlich 1979). Total 991 koloni putih transforman Tcr diperoleh dan mengandung rekombinan pRK415.
Variasi ukuran sisipan transforman yang berada antara ± 2.7 – 5.5 kb menunjukkan bahwa ukuran tersebut mendekati kisaran yang diinginkan dan
8 kb 3 kb
20
bukan merupakan replika satu sama lain. Hasil uji antagonis keseluruhan E. coli T0P10 rekombinan dengan suspensi G. boninense belum menunjukkan adanya ekspresi gen antifungi E. coli T0P 10 rekombinan yang ditandai dengan tidak terbentuknya zona penghambatan terhadap pertumbuhan hifa G. boninense.
Gambar 12 Verifikasi sisipan plasmid pRK415 rekombinan. M (marker); 1-5 (koloni rekombinan); K- (kontrol negatif)
Plamid pRK415 merupakan plasmid yang memiliki promotor lac dari pRK404 (Ditta et al. 1985). Vektor pRK415 rekombinan yang mengandung sisipan fragmen gen tertentu dan berada pada orientasi yang sesuai dengan daerah promotor dan atau mengandung promotor dari gen asli yang dikenali oleh RNA polimerase inang akan terekspresi dan dapat diseleksi dengan seleksi biru putih (Keen et al. 1988). Kemungkinan keseluruhan rekombinan yang belum menunjukkan ekspresi dari fragmen sisipan dipengaruhi oleh banyak faktor. Faktor-faktor yang dapat mempengaruhi ekspresi dari suatu gen adalah kekuatan dan kesesuaian pembacaan promotor, oritentasi sisipan, urutan inisiasi translasi, pilihan kodon, stabilitas mRNA, jumlah kopi plasmid, stabilitas plasmid dan fisiologi sel inang (Primrose et al. 2001; Sorensen dan Mortensen 2005; Brown 2010). Dengan banyaknya kemungkinan faktor tersebut, keberhasilan dalam memperoleh klon yang memiliki aktivitas membutuhkan banyak transforman uji (Bernier et al. 1983; Tran et al. 1991).
Identifikasi Fragmen DNA Pengapit Transposon Mini-Tn5KmR pada Mutan BglK226
Mutan Bgl K226 merupakan hasil mutasi transposisi dari transposon mutagenesis mini-Tn5Kmr (pUTmini-Tn5Kmr). Mutan tersebut menarik karena menunjukkan fenomena yang unik setelah tersisipi oleh transfer elemen dari daerah pUTmini-Tn5Kmr, yaitu memiliki kemampuan lebih kuat daripada tipe liar (Burkholderia gladioli JR01) dalam menghambat pertumbuhan miselium G. boninense pada saat uji tantang in vitro pada media PDA.
21
DNA Pengapit DNA Pengapit Adaptor genom walker regulasi gen tertentu yang diketahui mempengaruhi efek kemampuan antagonistiknya terhadap G. boninense. Pembungkaman dari suatu gen dapat memberikan efek signifikan dalam jalur metabolisme dalam menghasilkan produk tertentu.
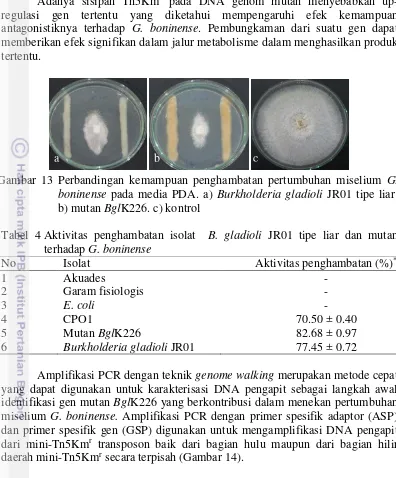
Gambar 13 Perbandingan kemampuan penghambatan pertumbuhan miselium G. boninense pada media PDA. a) Burkholderia gladioli JR01 tipe liar; b) mutan BglK226. c) kontrol
Tabel 4 Aktivitas penghambatan isolat B. gladioli JR01 tipe liar dan mutan terhadap G. boninense
No Isolat Aktivitas penghambatan (%)*
1 Akuades -
2 Garam fisiologis -
3 E. coli -
4 CPO1 70.50 ± 0.40
5 Mutan BglK226 82.68 ± 0.97
6 Burkholderia gladioli JR01 77.45 ± 0.72
Amplifikasi PCR dengan teknik genome walking merupakan metode cepat yang dapat digunakan untuk karakterisasi DNA pengapit sebagai langkah awal identifikasi gen mutan BglK226 yang berkontribusi dalam menekan pertumbuhan miselium G. boninense. Amplifikasi PCR dengan primer spesifik adaptor (ASP) dan primer spesifik gen (GSP) digunakan untuk mengamplifikasi DNA pengapit dari mini-Tn5Kmr transposon baik dari bagian hulu maupun dari bagian hilir daerah mini-Tn5Kmr secara terpisah (Gambar 14).
Gambar 14 Strategi amplifikasi fragmen DNA pengapit transposon mini-Tn5Kmr dengan genome walking PCR. PCR pertama menggunakan primer ASP1 dan GSP1 sedangkan PCR kedua mengunakan primer ASP2 dan GSP2
22
750pb
1 2 3 4 5 6
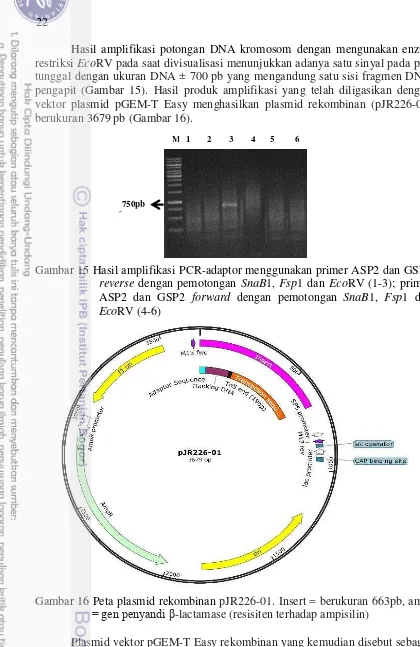
Hasil amplifikasi potongan DNA kromosom dengan mengunakan enzim restriksi EcoRV pada saat divisualisasi menunjukkan adanya satu sinyal pada pita tunggal dengan ukuran DNA ± 700 pb yang mengandung satu sisi fragmen DNA pengapit (Gambar 15). Hasil produk amplifikasi yang telah diligasikan dengan vektor plasmid pGEM-T Easy menghasilkan plasmid rekombinan (pJR226-01) berukuran 3679 pb (Gambar 16).
Gambar 15 Hasil amplifikasi PCR-adaptor menggunakan primer ASP2 dan GSP2 reverse dengan pemotongan SnaB1, Fsp1 dan EcoRV (1-3); primer ASP2 dan GSP2 forward dengan pemotongan SnaB1, Fsp1 dan EcoRV (4-6)
Gambar 16 Peta plasmid rekombinan pJR226-01. Insert = berukuran 663pb, ampr
= gen penyandi β-lactamase (resisiten terhadap ampisilin)
Plasmid vektor pGEM-T Easy rekombinan yang kemudian disebut sebagai pJR226-01 memperlihatkan bahwa terdapat sisipan DNA yang berukuran kurang lebih 663pb. Kloning sisipan tersebut dilakukan dengan tujuan untuk pemeliharaan dan perbanyakan plasmid rekombinan. Pemeliharaan dan
23 perbanyakan fragmen DNA sisipan pada suatu vektor kloning diketahui sangat membantu dalam pekerjaan telaah gen tertentu (Brown 2010).
Analisis peta rekombinan plasmid dilakukan untuk verifikasi hasil sisipan. Verifikasi daerah sisipan setelah dilakukan pengurutan menunjukkan bahwa diantara fragmen pengapit, terdapat fragmen daerah transposon mini-Tn5Kmr, 19 pb urutan CTGTCTCTTGATCAGATCT (Tn5insite) (Lorenzo et al. 1990) dan fragmen adaptor (Gambar 17). Hal ini membuktikan bahwa hasil pemotogan DNA kromosom BglK226 dengan enzim restriksi EcoRV, amplifikasi primer ASP-F/GSP-R dan kloning sisipan kedalam vektor pGEM-T Easy berhasil dilakukan dan mengandung fragmen DNA pengapit dengan ukuran 151 pb. Meskipun demikian, pemotongan DNA genom menggunakan enzim restriksi FspI dan SnaBI tidak menghasilkan fragmen amplikon. Pemotongan dengan kedua enzim ini kemungkinan menghasilkan fragmen DNA target yang berukuran sangat besar. Metode berbasis PCR dapat mengamplifikasi fragmen DNA yang terbatas, sehingga tidak optimal untuk diterapkan pada urutan DNA genom yang terlalu panjang.
Gambar 17 Peta verifikasi fragmen DNA pengapit hasil amplifikasi genome walking Mutan BglK226
Keberadaan sekuen 19 pb ini menunjukkan bahwa produk PCR genome walking diamplifikasi dengan benar menggunakan primer GSP yang didesain ke arah luar melewati 19 pb Tn5 end ke arah sekuen pengapit. Deteksi ulang yang dilakukan terhadap marker yang bisa dijadikan acuan akurasi pencirian posisi DNA pengapit menjadi landasan yang membuktikan bahwa fragmen dengan ukuran 151pb merupakan bagian potongan dari DNA kromosom BglK226 yang telah tersisipi mini-Tn5Kmr. Berdasarkan hasil analisis pengurutan DNA dan blasting diperoleh bahwa DNA pengapit memiliki tingkat kemiripan 99% dengan sebagian gen KAS penyandi enzim beta-ketoacyl synthase asal Bukholderia gladioli BSR3 (Gambar 19). Posisi mini-Tn5KmR pada gen beta-ketoacyl synthase ditunjukkan pada Gambar 18.
24
Beta-ketoacyl synthase merupakan enzim yang memainkan peranan penting sebagai bagian penting pada biosintesis asam lemak (Heath & Rock 1996; Qiu et al. 1999). Biosisntesis asam lemak terdiri dari rangkaian reaksi penting di dalam sel yang bertanggung jawab dalam pembentukan membran sel khususnya pembentukan fosfolipid. Pada membran sel bakteri dijumpai lapisan fosfolipid, baik untuk gram negatif maupun gram positif yang berfungsi membungkus sitoplasma, menjaga keluar masuknya zat dari dalam sel maupun dari luar sel. Pertumbuhan bakteri sangat tergantung pada pembentukan asam lemak. Hal menarik pada penelitian ini adalah pembungkaman gen beta-ketoacyl synthase justru tidak menunjukkan kematian pada bakteri mutan BglK226, tetapi menunjukkan perubahan fenotip (lebih berlendir, warna koloni dari putih menjadi kuning-coklat setelah inkubasi 7 hari pada media PDA serta kemampuan penghambatan pertumbuhan G. boninense yang lebih tinggi (Gambar 13).
Gambar 19 Hasil analisis parsial urutan DNA mutan BglK226 pengapit transposon (NCBI); Query = urutan sebagian DNA pengapit ; Sbjct = urutan sebagian DNA Burkholderia gladioli strain BSR3 kromosom 1 pada basis data Genebank
Diduga ada dua kemungkinan yang menyebabkan mutan BglK226 mampu bertahan hidup walaupun gen KAS telah dibungkam, yang pertama adalah kemungkinan pembungkaman gen beta-ketoacyl synthase oleh penyisipan transfer elemen dari mini-Tn5KmR bukan menyisip pada sekuen esensial yang memiliki
fungsi utama dalam aktivitas enzim. Hal ini didasarkan pada analisis posisi penyisipan mini-Tn5KmR pada gen KAS BSR3 yang bukan pada daerah situs katalitik (daerah mendekati gugus amina) melainkan berada pada daerah sekuen non-esensial (mendekati gugus karboksil) pada sekuen nukleotida ke 6085 bp dari 7653 pb total panjang gennya. Sehingga tidak menunjukkan efek yang nyata terhadap peniadaan aktivitas enzim KAS dan pada akhirnya masih tetap diproduksi dalam pembentukan posfolipid membran sel.
25 tanaman dan E. coli. sebagai bagian dari multifungsi kompleks asam lemak sintase yang setiap protein dikodekan oleh gen tertentu. Enzim KASI dan II pada E. coli merupakan produk dari gen fabB dan fabF (Knowles et al. 2000).
Gambar 20 Pensejajaran sekuen asam amino KAS beberapa organisme prokariot dan eukariot
Heath et al. (2002) melaporkan bahwa pada E. coli, diketahui KASI dan KASII merupakan enzim yang berfungsi dalam inisiasi elongasi sintesis asam lemak melalui reaksi kondensasi antara asetil-ACP dan malonil-ACP menjadi asetoacetil-ACP. Kedua enzim tersebut memiliki substrat yang sama tetapi regulasi ekspresi terjadi pada waktu dan kondisi yang berbeda. KASI (fabB) memiliki tingkat similaritas yang tinggi dengan KASII (fabF) yaitu 40% identik protein dengan triad katalitik Cys-His-His (Heath et al. 2001; Hoang et al. 2002). KASII merupakan enzim yang berperan penting dalam regulasi komposisi asam lemak pada kondisi temperatur yang fruktuatif, diekspresikan lebih tinggi pada kondisi suhu rendah untuk menjaga stabilitas fospolipid pada membran sel E. coli. Pembungkaman gen fabF pada suhu normal tidak menyebabkan kematian sel. Berbeda dengan pembungkaman fabB, menyebabkan terganggunya pertumbuhan (Garwin et al. 1980).
Pembungkaman gen beta-ketoacyl synthase menyebabkan terganggunya proses pembentukan asam lemak dari prekursor utama asetil-CoA, sehingga diduga bahwa asetil-CoA yang menumpuk lebih banyak terpakai pada jalur respirasi sel yang menyebabkan produksi ATP meningkat. Kegagalan dalam melakukan respirasi secara normal menyebabkan perubahan level ATP (Lee et al. 2012). Ketidakseimbangan level ATP seiring dengan peningkatan ketersediaan energi pada mutan BglK226 kemungkinan mengakibatkan ketidakseimbangan metabolisme yang mempengaruhi pertumbuhan maupun produksi metabolit sekunder secara dramatis, termasuk pada produksi senyawa antifungi.
26
5 SIMPULAN DAN SARAN
Simpulan
Hasil identifikasi bakteri potensial yang diperoleh adalah Pseudomonas aeruginosa dan Bacillus amyloliquefaciens JR02. Sejumlah fragmen DNA kromosom B. amyloliquefaciens JR02 berhasil di kloning pada vektor pRK415. Total 991 koloni transforman TcR yang diperoleh belum menunjukkan ekspresi gen antifungi. Disisi lain pada saat yang bersamaan, karakterisasi DNA pengapit mutan BglK226 menunjukkan tingkat kemiripan 99% dengan sebagian gen beta-ketoacyl synthase.
Saran
27
DAFTAR PUSTAKA
Al-bayaty FH. 2012. Shotgun cloning, characterization and detection of virulence gene of Staphylococcus aureus strain 125. Archives des sciences. 65 (11): 173-184.
Ariffin D, Idris AS, Singh G. 2000. Status of Ganoderma in Oil Palm. (chapter). In flood j, bridge pd, holdernes p (eds) Ganoderma Diseases of Parennial Crops. CABI Wallingfod. 49-68.
Artiguenave F, Vilagines R, Danglot C. 1997. High-efficiency transposon mutagenesis by electroporation of a Pseudomonas fluorescens strain. FEMS Microbiol. 153: 363-369.
Ashoub A, Abdalla KS. 2006. A primer based approach to genome walking. Plant Mol Biol Reporter. 24: 237-243.
Bivi MR, Farhana SN, Khairulmazmi A, Idris A. 2010. Control of Ganoderma boninense: a causal agent of basal stem rot disease in oil palm with endophyte bacteria In Vitro. Inter J Agric Biol. 12:833-839.
Bernier R, Driguez H, Desrochers M. 1983. Molecular cloning of a Bacillus subtilis xylanase in Eschericia coli. Gene. 26: 59-65.
Brown T.A. 2010. Gene Cloning and DNA Analysis: An Introduction. 6th edition. Blackwell Publishing.
Buana RFN, Wahyudi AT, Toruan-Mathius N. 2014. Control activity of potential antifungal-producing Burkholderia sp. in suppressing Ganoderma growth in oil palm. A. J Agricul Res. 8(5): 259-268.
Clontech. 2007. Genome WalkerTM Universal Kit Manual.
Cooper RM, Flood J, Rees RW. 2011. Ganoderma boninense in oil palm plantations: current thinking and epidemiology, resistence and pathology. The planter. 87(1024): 515-526.
Corley RHV, Tinker PB. 2003. The Oil Palm. Fourth Edition. Blackwell Science. Dagert M, Ehrlich SD. 1979. Prolonged incubation in calcium chloride improves
the competence of Escherichia coli cells. Gene. 6: 23-28.
Darmono TW. 2000. Ganoderma in oil palm in indonesia: current status and prospective use of antibodies for the detection of infection (chapter). In flood j, bridge pd, holdernes p (eds) Ganoderma Diseases of Parennial Crops. CABI Wallingfod. 249-266.
De Lorenzo V, Herrero M, Jacubzik U, Timmis KN. 1990. Mini-tn5 transposon derivatives for insertion mutagenesis, promoter probing, and chromosomal insertion of cloned DNA in gram-negative eubacteria. J Bacteriol. 172(11): 6568-6572.
[Ditjenbun] Direktorat Jendral Perkebunan. 2014. Pertumbuhan Areal Kelapa Sawit. [Diunduh 2015 Juli 15]. Tersedia pada:
28 synthase II of Eschericia coli. J Biol Chem. 255(8): 3263-3265.
Harrison LA, Letendre L, Kovacevich P, Pierson E, Weller DM. 1993. Purification of an antibiotic effective against Gaeumanomyces graminis var. tritici produced by a biocontrol agent, Pseudomonas aureofaciens. Soil Bio Biochem. 25: 215-221.
Heath RJ, Jackowski S, Rock CO. 2002. Fatty acid and phospholipid metabolism in prokaryotes. In vance DE, Vance JE. 2002. Biochemistry of Lipid, Lipids, Lipoproteins and Membranes. Elsevier Science BV.USA.
Heath RJ, Rock CO. 1996. Inhibition of β-ketoacyl-acyl carier protein synthase III (FaBH) by acyl-acyl carrier protein in Escherichia coli. J Biol Chem. 271(18): 10996-11000.
Heath RJ, White SW, Rock CO. 2001. Lipid biosynthesis as a target for antobacterial agents. Pro Lip Researc. 40: 467-497.
Herrero M, Lorenzo VDE, Timmis KN. 1990. Transposon vectors containing non-antibiotic resistance selection markers for cloning and stable chromosomal insertion of foreign genes in gram negative bacteria. J Bacteriol 172: 6557-6567.
Hoang TT, Sullivan, Cusick K, Schweizer HP. 2002. Beta-ketoacyl acyl carrier protein reductase (FabG) activity of the fatty acid biosynthetic pathways is a determining factor of 3-oxo-homoserine lactone acyl chain lengths. Microbiol. 148:3849-3856.
Jinantana J, Sariah M, 1997. Antagonistic effect of Malaysian isolates of Tricoderma harzianum and Gliocladium viren on Sclerotium rolfsii. pertanika. J Trop Agric Sci. 20: 35-41.
Kadir J, Rahman MA, Mahmud TMM, Rahman RA. Begum MM. 2008. Extraction of antifungal substansces from Burkholderia cepacia with antibiotic activity against Colletotrichum gloeosporioides on papaya (Carica papaya L). Int. J Agri Biol. 10: 15-20.
Keen NT, Tamaki S, Kobayashi D, Trollinger. 1988. Short communications improved broad-host-range plasmid for DNA cloning in gram-negatif bacteria. Gene. 70: 191-197.
Knowles PW, Olsen JG, Mcguire KA, Larsen S. 2000. Molecular aspects of beta-ketoacyl synthase (KAS) catalysis. Biochem S Transc. 28(6): 601-607. Kruger R, Filutowicz M. 2000. Dimer of II proten bind the A+T-rich region of the
R6K¥ origin near the leading-strand synthesis start sites: regulatory implications. J bacteriol. 182: 2461-2467.
Lee KW, Kong HG, Lee SW. 2012. Identification of a gene encoding adenylate kinase involved in antifungal activity expression of the biocontrol strain Burkholderia pyrrocinia CH-67. Plant Pathol J. 28(4): 373-380.
29 Liu Y, Chen Z, Ng TB, Zhang J, Zhou M, Song F, Lu F, Liu Y. 2007. Bacisubin an antifungal protein with ribonuclease and hemagglutinating activities from Bacillus subtilis. Peptide. 28: 553-559.
Lugternberg B, Kamilova F. 2009. Plant growth promoting rhizobacteria. Annu Rev Microbiol. 63: 541- 546.
[Major Differences] Difference between Genomic and cDNA Library. [Diunduh 2015 November 11]. Tersedia pada: http://www. Majordefferences.com/2013/11/difference-between-genomic-and cDNA. Html.
Marchesi JR, Sato T, Weightman AJ, Martin TA, Fry J, Hiom SJ, Wade WG. 1998. Design and evaluation of useful bacterium-specific PCR primers that amplify genes coding for bacterial 16s rRNA. App Environ Microbiol. 64(2): 795-799.
Miller RNG, Holderness M, Bridge PD, Chung GF, Zakaria MH. 1999. Genetic diversity of Ganoderma in oil palm plantings. Plant Pathol. 48: 595-603. Paterson RMM. 2007. Ganoderma disease of oil palm a white rot perspective
necassary for integrated control. Crop Prot. 26: 189-199.
Pilotti CA. 2005. Stem rot of oil palm caused by Ganoderma boninense: pathogen and epidemiology. Mycophatol. 159: 129-137.
Purnamasari MI, Prihatna C, Gunawan AW, Suwanto A. 2012. Isolation and molecular identification of Ganoderma spp associated with basal stem rot disease in oil palm. J Fitopatol Indon. 8(1): 9-15.
Primrose SB, Twyman RM, Old RW. 2001. Principles of Gene Manipulation. USA. Blackwell Science Ltd. Malden.
Qiu X, Janson CA, Konstantinidis AK, Nwagwu S, Silverman C, Smith WW, Khandekar S, Lonsdale J, Abdel-Meguid SS. 1999. Crystal structure of β-ketoacyl-acyl carier protein synthase III. J Biol Chem. 274(51): 36465-36471.
Quan CS, Zhen Q, Liu Y, Ohta, Fan SD. 2006. Isolation and characterization of a novel Burkholderia cepacia with strong antifungal activity against Rhizoctonia solani. Appl Microbiol Biotechnol. 72: 1276-1284.
Rees RW. Flood J, Hasan Y, Potter U, Cooper RM. 2009. Basal stem rot oil palm (Elaeis guineensis); mode of root infection and lower stem invation by Ganoderma boninense. Plant Pathol. 58: 982-989.
Rees RW. Flood J, Hasan Y, Wills MA, Cooper RM. 2011. Ganoderma boninense basidiospores in oil palm plantations: evaluation of their possible role in stem rots of Elaeis guineensis. Plant Pathol. 1-12.
Roitman J, Mahoney NE, Janisiewicz WJ. 1990. Production and composition of phenylpyrrole metabolites produced by Pseudomonas cepacia. Appl Microbiol Biotechnol. 34: 381-386.
Samala S, Sariah M, Idris AS, Radziah O. 2011. Symbiotic bacteria with arbuscular mycorrhizal fungi and its antagonistic effect on Ganoderma boninense. The J Microbiol. 49(4): 551-557.
Sambrook J, Fritsch EF, Maniatis T. 1989. Molecular Cloning. A Laboratory Manual Second Edition. Cold Spring Harbor Laboratory Press.