PENYANDI MAJOR FACILITATOR SUPERFAMILY (MFS)
DARI Melastoma affine L.
WIDYARTINI
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
SUMBER INFORMASI
Dengan ini saya menyatakan bahwa tesis Isolasi dan Pengklonan Fragmen cDNA
Gen Penyandi Major Facilitator Superfamily (MFS) dari Melastoma affine L.
adalah karya saya sendiri dan belum diajukan dalam bentuk apa pun kepada
perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari
karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah
disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir
tesis ini.
Bogor, Januari 2007
Widyartini
WIDYARTINI. Isolasi dan Pengklonan Fragmen cDNA Gen Penyandi Major Facilitator Superfamily (MFS) dari Melastoma affine L. Di bimbing oleh SUHARSONO DAN MUHAMMAD JUSUF.
Beberapa sistem transpor pada membran terbukti berperan penting bagi sifat ketahanan terhadap senyawa toksik alami dan xenobiotik pada bakteri maupun eukariot. Salah satu sistem transpor adalah Major Facilitator Superfamily (MFS) yang memiliki kisaran substrat luas yang meliputi antibiotik, obat, polisakarida, gula, asam amino, logam berat dan molekul-molekul lainnya. MFS merupakan sistem transpor sekunder aktif berupa polipeptida tunggal dan berfungsi untuk mentranspor molekul-molekul atau solut berukuran kecil dengan menggunakan gradien ion kemiosmosis.
Penelitian ini bertujuan untuk mengisolasi, mengklon dan melakukan karakterisasi gen penyandi MFS dari Melastoma affine L. yang diduga memiliki kontribusi dalam ketahanan terhadap cekaman asam dan aluminium. Penelitian dilakukan melalui beberapa tahap yaitu isolasi RNA total, sintesis cDNA total melalui transkripsi balik, isolasi fragmen cDNA Mfs dari M. affine dengan PCR,
pengklonan fragmen cDNA MfsM. Affine (MaMfs) ke dalam plasmid pGEM®–T Easy, transformasi genetik Escherichia coli DH5α dengan vektor rekombinan, analisis cDNA sisipan dan analisis urutan nukleotida MaMfs. Dalam penelitian ini kami berhasil mendapatkan fragmen cDNA Mfs M. affine berukuran 511 pb yang menyandikan 168 asam amino. Analisis situs enzim restriksi pada fragmen
MaMfs menunjukkan bahwa fragmen MaMfs mempunyai beberapa situs restriksi tetapi tidak mempunyai situs restriksi EcoR1.
Analisis kesejajaran lokal berdasarkan urutan nukleotida menggunakan program BLAST (Basic Local Alignment Search Tool) menunjukkan bahwa fragmen MaMfs mempunyai kesamaan 74% dengan bagian coding sequence
(cds) Arabidopsis thaliana (AY056374), 73% dengan klon mth2-61c11
Medicago truncantula (AC148655) pada bagian cds 29835-35720 yang menyandi Major Facilitator Superfamily (MFS_1) dan 71% dengan bagian mRNA (complete cds) yang menyandi tetracycline resistance efflux protein like protein (MFS_1) dari A. thaliana (AK230262). Analisis kesejajaran berdasarkan urutan asam amino yang dideduksi dari cDNA Mfs menunjukkan bahwa fragmen MaMFS mempunyai kesamaan 80% dengan bagian MFS_1 dari M. truncantula
(Q1S1V4), 77% dengan bagian hypotetical protein A. thaliana (O82747), 73%
dengan OsMFS_1 dari padi (Q7XTZ1), 73% dengan bagian MFS_1
WIDYARTINI. Isolation and Cloning of cDNA Fragment of Gene Encoding for Major Facilitator Superfamily (MFS) from Melastoma affine L. Under the direction of SUHARSONO and MUHAMMAD JUSUF.
Many membrane transport systems have been demonstrated to play an important role in both bacteria and eucaryotes by confering resistance to a wide range of natural toxic compounds and xenobiotic. One of them is Major Facilitator Superfamily transporters (MFS) that have a broad substrate range including unrelated chemicals such as sugars, inorganic ions, heavy metals, peptides, amino acids, oligopeptides, polysaccharides, proteins and drugs. MFS transporters are single-polypeptide secondary carriers active capable of transporting small solutes in response to chemiosmotic ion gradien.
The objective of this experiment is to isolate, clone and characterize the gene encoding MFS from Melastoma affine L. This protein is supposed to have contribution in Al and acid stress resistance in M. affine. M. affine grows well in acid soil with high level of soluble aluminum. We started our study by total RNA isolation. Total cDNA had been generated from the total RNA as template by reverse transcription. By PCR technique, the cDNA fragment of MFS from M. affine (MaMfs) had been succesfully isolated by using total RNA as template and specific primer of Mfs that designed from ZmMfs from
Zea mays and Mfs from Medicago truncantula. The MaMfs fragment was inserted into pGEM®–T Easy plasmid, then the recombinant plasmid was introduced into Escherichia coli DH5α. Sequencing of the fragment MaMfs by using ABI Prism 310 automated DNA sequencer showed that the MaMfs fragment is about 511 bp in size that encoding 168 amino acids. Enzym restriction sites analysis of the MaMfs fragment showed that the fragment MaMfs contains some restriction sites but it does not contain EcoR1 restriction site.
Nucleotide allignment analysis using BLASTn program showed that MaMfs
fragment is 74% identical to complete coding sequence (cds) Arabidopsis thaliana
(AY056374), 73% to part of MtMfs from Medicago truncantula (AC148655) and 71% to
part of tetracycline resistance efflux protein like protein (MFS_1) from
A. thaliana (AK230262). Amino acids allignment analysis using BLASTp program shows that MaMFS fragment is 80% identical to MFS_1 from M. truncantula (Q1S1V4), 77% to hypotetical protein A. thaliana (O82747), 73% to OsMFS_1 from rice (Q7XTZ1), 73% to tetracycline resistance efflux protein like protein (MFS_1) A. thaliana
PENYANDI MAJOR FACILITATOR SUPERFAMILY (MFS)
DARI Melastoma affine L.
WIDYARTINI
Tesis
sebagai salah satu syarat untuk memperoleh gelar
Magister Sains pada
Program Studi Bioteknologi
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
Major Facilitator Superfamily (MFS) dari Melastoma affine L.
Nama : Widyartini
NRP : P055030071
Disetujui
Komisi Pembimbing
Dr. Ir. Suharsono, DEA Dr. Ir. Muhammad Jusuf
Ketua Anggota
Diketahui
Ketua Program Studi Bioteknologi Dekan Sekolah Pascasarjana
Dr. Ir. Muhammad Jusuf Prof. Dr. Ir. Khairil Anwar Notodiputro, MS
Bismillaahirrahmaanirrahiim
Segala puji saya panjatkan kehadirat Allah SWT yang telah melimpahkan
rahmat dan karunia-Nya sehingga karya ilmiah yang berjudul ” Isolasi dan
Pengklonan Fragmen cDNA Gen Penyandi Major Facilitator Superfamily (MFS) dari Melastoma affine L” dapat diselesaikan. Penelitian ini dibiayai oleh Proyek Hibah Penelitian Tim Pascasarjana Angkatan III dengan judul ”Isolasi dan
karakterisasi gen-gen yang berhubungan dengan toleransi tanaman terhadap pH
rendah dan aluminium tinggi atas nama Dr. Suharsono sebagai peneliti utama.
Selama proses penulisan karya ilmiah ini penulis mendapat bantuan dan
bimbingan dari berbagai pihak baik secara langsung maupun tidak langsung.
Untuk itu penulis bermaksud menyampaikan rasa terima kasih kepada :
1. Bapak Dr. Ir. Suharsono, DEA selaku Komisi Pembimbing atas bimbingan
dan arahannya.
2. Bapak Dr. M. Jusuf selaku Komisi Pembimbing atas bimbingan dan
arahannya
3. Ibu Dr. Ir. Utut Widyastuti Suharsono atas bimbingan dan arahannya.
4. Ibu Dr. Ir. Trikoesoemaningtyas yang telah memberi masukan yang sangat
berarti dalam perbaikan tulisan ini
5. Papa, mama, adik-adik serta seluruh keluarga atas semua do’a, pengorbanan
dan kasih sayangnya.
6. Pusat Penelitian Sumberdaya Hayati dan Bioteknologi IPB yang telah
menyediakan sarana dan prasarana untuk penelitian
7. Terakhir dan tak terlupakan terima kasih kepada Eve, Firdaus, Agt, Zendi,
Pak Mul, Mbak Pepy, Huda, Bahrelvi, Didi, Pak dan Bu Elfian, Pak Muzuni,
Bu Sri Lis, Budi Hebat, Poppi, Ammay serta semua pihak yang telah banyak
membantu dan tidak dapat disebutkan satu per satu.
Semoga karya ilmiah ini dapat bermanfaat bagi kita semua.
Bogor, Januari 2007
RIWAYAT HIDUP
Penulis dilahirkan di Palembang pada tanggal 6 Juni 1979 dari ayah
I. Made Sudania, SH dan ibu Patmini A Ma Pd. Penulis merupakan putri
pertama dari empat bersaudara.
Pendidikan sarjana ditempuh di Program Studi Pemuliaan Tanaman,
Jurusan Budi Daya Pertanian, Fakultas Pertanian IPB, lulus pada tahun 2001.
Pada tahun 2003, penulis diterima di Program Studi Bioteknologi pada Program
Pascasarjana IPB.
Penulis pernah bekerja di Perkebunan Kelapa Sawit Cipta Futura pada
DAFTAR ISI
Toksisitas dan Toleransi Aluminium pada Tanaman... 5Toksisitas Al ... 5
Toleransi Al... 6
Major Facilitator Superfamily (MFS)... 9
Karakteristik Melastoma dan Toleransinya terhadap Aluminium ... 13
BAHAN DAN METODE Waktu dan Tempat Penelitian ... 15
Bahan ... 15
Metode Penelitian ... 16
Isolasi RNA total... 17
Sintesis cDNA total melalui transkripsi balik... 18
Isolasi Fragmen cDNA MaMfs melalui PCR ... 19
Pengklonan cDNA ke dalam vektor pGEM®-T Easy ... 19
Transformasi genetik bakteri E. coli DH5α dengan vektor rekombinan... 19
Analisis cDNA sisipan ... 21
Analisis urutan nukleotida MaMfs... 22
HASIL DAN PEMBAHASAN
Analisis Domain Fragmen MaMFS ... 36
Analisis Hidrofobisitas Fragmen MaMFS ... 40
SIMPULAN DAN SARAN Kesimpulan ... 42
Saran... 42
DAFTAR PUSTAKA ... 42
DAFTAR TABEL
Halaman
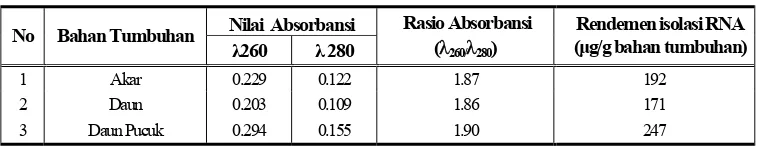
1. Rendemen Isolasi RNA Total ... 23
2. Matriks kesamaan urutan nukleotida fragmen MaMfs dan Mfs dari organisme lainnya ... 32
3. Matriks kesamaan asam amino fragmen MaMFS dan MFS dari organisme lainnya ... 34
4. Analisis domain transmembran menggunakan program
Tmpred versi 2.0 ... 37
DAFTAR GAMBAR
Halaman
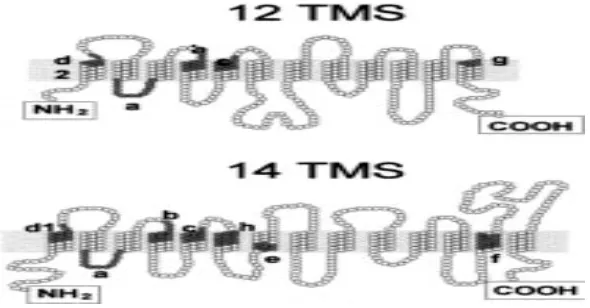
1. Topologi MFS transporter dengan 12 dan 14 domain transmembran .... 10
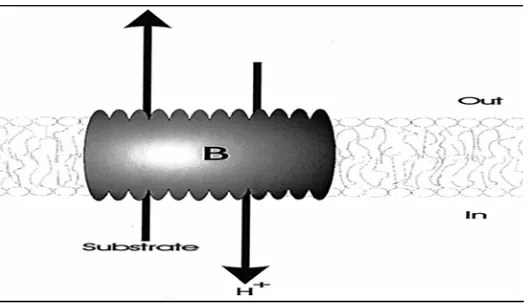
2. MFS multidrug efflux transporter menggunakan gradien elektrokimia transmembran dari proton atau ion sodium untuk mensekresi senyawa toksik dan xenobiotik dari dalam sel... 11
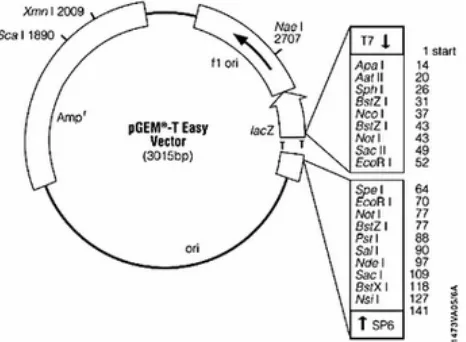
3. Melastoma affine L. ... 13 4. Vektor pengklonan pGEM® –T Easy... 15
5. Tahapan isolasi dan pengklonan fragmen cDNA Mfs dari Melastoma affine L ... 16 6. RNA total yang terdapat pada ketiga sampel yaitu akar (1), daun
tua(2) dan daun pucuk (3) ... 24
7. Pita cDNA ekson1- ekson2 dari aktin yang dihasilkan dari PCR aktin menggunakan cetakan cDNA dari sampel daun (1), daun pucuk (2) dan akar (3) ... 24
8. Pita fragmen cDNA MaMFS hasil amplifikasi dengan teknik PCR menggunakan cetakan cDNA sampel daun (1) dan sampel akar (2) ... 25
9. Seleksi biru putih koloni E. coli DH5α hasil transformasi dengan vektor pGEMT Easy pada media seleksi yang mengandung X-gal, IPTG dan antibiotik ampisilin... 26
10. Hasil PCR terhadap koloni E. coli DH5α (koloni putih) sebagai cetakan ... 27
11. Plasmid pGEMT Easy rekombinan utuh (1) dan hasil pemotongan plasmid dengan enzim restriksi EcoR1 ... 27 12. Urutan nukleotida fragmen cDNA MaMfs dan deduksi asam
aminonya ... 28
13. Situs pemotongan enzim restriksi endonuklease pada fragmen MaMfs
menggunakan program NEB Cutter... 29
14. Filogenetik berdasarkan urutan nukleotida fragmen MaMfs dengan
Mfs dari organisme lainnya (angka menunjukkan jarak) ... 33 15. Filogenetik berdasarkan asam amino fragmen MaMFS dengan MFS
dari organisme lainnya (angka menunjukkan jarak) ... 35
16. Posisi fragmen MFS Melastoma (MaMFS) pada MFS Medicago
(MtMFS) berdasarkan hasil analisis kesejajaran asam amino ... 36
17. Analisis kesejajaran transmembran TM6, TM7, TM8 dan TM9 MaMFS dengan MtMFS, AtMFS dan OsMFS ... 38
19. Profil hidrofobisitas MFS M. truncantula... 41
20. Profil hirofobisitas fragmen MaMFS dan fragmen MFS
DAFTAR LAMPIRAN
Halaman
1. Daftar asam amino dan polaritas... 49
2. Hasil sekuensing DNA sisipan (fragmen MaMfs) pada pGEM®-T Easy
menggunakan primer sp6... 50 3. Hasil analisis penyejajaran urutan nukleotida fragmen MaMfs dengan
gen pada data GenBank menggunakan program Blast... 51 4. Hasil analisis kesejajaran lokal berdasarkan urutan nukleotida antara
MaMfs dengan MtMfs... 57 5. Hasil analisis penyejajaran urutan asam amino fragmen MaMFS
dengan data GenBank menggunakan program Blast ... 58 6. Hasil analisis kesejajaran lokal berdasarkan urutan asam amino antara
Latar Belakang
Salah satu usaha untuk meningkatkan produksi pertanian secara nasional adalah melalui ekstensifikasi penanaman pada lahan marjinal yang belum dimanfaatkan secara optimal di luar pulau Jawa. Salah satu lahan marjinal adalah tanah podzolik merah kuning. Jenis tanah ini mendominasi lahan kering yang ada di Sumatera, Kalimantan dan Irian Jaya dengan penyebarannya yaitu 10.04 juta ha di Kalimantan Timur, 7.62 juta ha di Irian Jaya, 5.71 juta ha di Kalimantan Barat, 4.81 juta ha di Kalimantan Tengah dan 2.27 juta ha di Riau (Puslitbang Tanah 1999).
Permasalahan yang biasa ditemukan pada tanah podzolik merah kuning adalah tingginya tingkat keasaman, pencucian hara (leaching), defisiensi unsur hara Ca, Mg, K, P, N dan kelarutan aluminium tinggi yang dapat menyebabkan keracunan bagi tanaman serta mudah mengalami erosi. Keracunan aluminium merupakan faktor utama yang membatasi produktivitas tanaman pada tanah asam dan meliputi 40% lahan pertanaman di dunia (Kochian 1995).
Usaha untuk mengatasi permasalahan keracunan aluminium tersebut salah satunya adalah melalui perbaikan genetik tanaman. Untuk itu perlu dilakukan isolasi gen-gen yang berperan dalam toleransi terhadap aluminium terutama dari tanaman indikator tanah asam yang diharapkan akan lebih efektif dan menambah sumberdaya genetik bagi sifat ketahanan ini.
memiliki mekanisme detoksifikasi Al secara internal dan dapat menjadi sumber gen ketahanan terhadap cekaman asam dan Al tinggi.
Pada tanah dengan keasaman yang sangat tinggi, Al3+ dapat melewati membran plasma baik melalui transpor protein yang secara normal berfungsi dalam adsorpsi ion-ion mineral lainnya ataupun melalui fase-fluida dan adsorpsi endositosis. Berdasarkan beberapa penelitian sebelumnya, Al3+ menghentikan kanal ion yang terdapat pada membran plasma sel akar. Sebagai contoh adalah Al3+ yang mengikat 107 kali lebih kuat ATP daripada Mg2+. Jumlah Al3+ kurang dari nanomolar sekalipun dapat menghambat Mg2+ pada situs P (Martin 1988). Berdasarkan bukti-bukti di atas, mekanisme transpor Al3+ tidak hanya terjadi secara aktif melalui transporter primer aktif (primary active transporters) tetapi juga melalui transporter sekunder aktif (secondary active transporter). Kemungkinan bahwa tanaman akumulator Al memiliki mekanisme detoksifikasi Al secara internal.
Walaupun banyak bukti yang menunjukkan keterkaitan asam organik dalam mekanisme detoksifikasi Al secara internal dan eksternal pada tanaman, ada beberapa spesies yang menunjukkan mekanisme yang tidak berkaitan dengan asam organik. Kultivar gandum yang sangat toleran Al (Atlas) mempunyai mekanisme pelepasan fosfat sebagai mekanisme detoksifikasi Al-nya (Pellet et al. 1996). Tanaman rumput-rumputan menghasilkan asam amino non-protein yang disebut phytosiderophore yang mampu mendetoksifikasi Al secara internal dengan cara mengkelat. Phytosiderophore dapat mengkelat Fe3+, komplek divalen, kation polyvalen dan Al3+ dengan sangat efektif sehingga tidak bersifat toksik lagi bagi tanaman saat terserap ke dalam sitoplasma (Kochian 1995). Bakteri juga mempunyai siderophore yang berfungsi dalam transpor Fe3+ yaitu enterobactin dan major facilitator superfamily (MFS) merupakan transporter enterobactin pada yeast (Heymann et al. 2000)
sehingga terjadi depolarisasi di membran plasma dan secara berantai mempengaruhi aktifitas metabolisme turunannya seperti aktifitas K-channel dan Ca-transporter yang masing-masing berperan dalam proses detoksifikasi Al (Kasai et al. 1993; Kinraide et al.1994; Sasaki et al. 1995; Kasai et al. 1995; Huang et al. 1996; Larsen et al. 1998; Delhaize, 2004).
Fluconazole resistance 1 (FLR1) yang menyandi MFS transporter pada fungi Candida albicans terlibat di dalam induksi CAP1 dan YAP1 yang berperan dalam resistensi C. albicans dan S. cerevisiae terhadap senyawa toksik seperti cycloheximide (CYH), 4-nitroquinoline N-oxide (4-NQO), kadmium dan hidrogen peroksida sehingga hal ini mengindikasikan bahwa FLR1 merupakan regulator transkripsi CAP1 dan YAP1 (Alarco et al. 1997). Oksida radikal (ROS, reactive oxigen species) merupakan salah satu mekanisme yang muncul pertama kali pada tanaman untuk mengatasi cekaman oksidatif khususnya untuk reoksigenasi. Hidrogen peroksida (H2O2) dan superoksida (O2-) merupakan contoh yang dihasilkan di sejumlah reaksi selular karena adanya cekaman oksidatif. Reaktifitas Al juga dapat menyebabkan terbentuknya oksida radikal (ROS, reactive oxigen species) yang beracun bagi sel.
Major Facilitator Superfamily merupakan transporter dengan kisaran substrat yang luas sehingga memiliki spesifisitas fungsi yang luas (Van Bambeke et al. 2000). Substrat MFS meliputi antibiotik, obat anti kanker dan anti HIV, molekul amphiphilik hingga gula, asam amino, ion logam dan molekul-molekul lainnya (Pao et al. 1998). Penelitian MFS pada organisme tingkat tinggi khususnya pada tanaman masih sangat sedikit sehingga baru sebagian kecil MFS yang ditemukan. Sebagian besar MFS telah ditemukan pada mikroorganisme dan telah diketahui fungsinya. Beberapa fungsi MFS yang sudah diketahui yaitu sebagai sistem transpor gula (Henderson et al. 1990), sistem transpor senyawa toksin, obat (drug efflux) dan senyawa metabolit pada siklus Krebs (Griffith et al. 1992; Paulsen et al. 1997), sistem transpor fosfat yaitu sebagai phosphate exchanger dan oligo sakarida (H+ simport permease) (Marger et al. 1993) serta sebagai permease asam aromatik pada bakteri (Goffeau at al. 1997).
dilakukan pada MFS tanaman, maka peran MFS dalam resistensi berbagai senyawa toksik khususnya resistensi yang terkait dengan cekaman pH rendah dan aluminium pada tanaman Melastoma sangat penting untuk diteliti.
Tujuan Penelitian
Tujuan penelitian yaitu isolasi dan pengklonan fragmen cDNA gen penyandi Major Facilitator Superfamily dari Melastoma affine L. (MaMFS) melalui teknik PCR.
Hipotesis
Toksisitas dan Toleransi Aluminium pada Tanaman
Toksisitas Al. Aluminium bukan unsur esensial bagi pertumbuhan tanaman. Pada saat kelarutan aluminium meningkat seiring dengan turunnya pH hingga di bawah 5, unsur ini penting untuk diperhatikan karena menjadi toksik bagi tanaman. Bentuk-bentuk aluminium di dalam tanah dapat berupa ion trivalen yaitu Al(H2O)63+ atau disebut Al3+, bentuk hidroksida seperti Al(OH)+2, Al(OH)2+, Al(OH)3, Al(OH)4- atau berasosiasi dengan berbagai senyawa organik dan anorganik seperti PO4-3, SO4-2,F-, asam-asam organik, protein dan lipid (Delhaize dan Ryan 1995). Al3+ merupakan bentuk yang paling toksik dan mendominasi di lahan asam dengan pH di bawah 4.5 (Matsumoto 2000).
Tanah asam terjadi karena adanya pencucian kation-kation basa dari tanah yang dipicu oleh praktek-praktek pertanian dan adanya hujan asam (Kennedy 1992). Untuk melihat pengaruh fitotoksik Al sebaiknya digunakan Al dalam bentuk Al3+, pH media 4 dan kekuatan ioniknya rendah (Kinraide 1991)
Aluminium terlarut bereaksi dengan dinding dan membran sel akar serta membatasi perluasan dinding sel sehingga menghentikan pemanjangan akar. Terhentinya pemanjangan akar merupakan ciri utama dari toksisitas aluminium. Jaringan akar merupakan bagian pertama dari tanaman yang mengalami keracunan aluminium, terutama di ujung akar sehingga mengalami pemendekan dan menebal. Akar menjadi berwarna kecoklatan terutama pada akar utama serta terjadi pertumbuhan akar lateral yang gemuk dan pendek dengan percabangan yang tidak bagus (Sasaki et al. 1994; Ryan et al. 1995).
mengikat sangat kuat senyawa donor O2 seperti Pi, nukleotida RNA, DNA, protein, asam karboksilat, fosfolipid, asam poligalakturonik, heteropolisakarida, lipopolisakarida, flavanoid, antosianin dan lain-lain (Haug 1984; Martin 1986). Konsentrasi Al yang kecil saja dalam simplas berpotensi menjadi fitotoksik. Sebagai contoh yaitu Al3+ mengikat 107 kali lebih kuat ATP daripada Mg2+. Jumlah Al3+ pada tingkat nanomolar sekalipun dapat menghambat Mg2+ pada situs P (Martin 1988). Aluminium juga diduga menghambat proses pembelahan sel dan menghalangi metabolisme asam nukleat (yaitu menghalangi reproduksi bahan genetik) pada tanaman (Helyar 1998). Menurut Matsumoto (1991) Al yang berada dalam bentuk polimer (Al3+) memiliki muatan positif yang besar serta memiliki banyak situs pengikatan. Polimer ini dapat mengikat fosfat pada kedua utas DNA sehingga mengakibatkan gagalnya pemisahan DNA utas ganda saat proses replikasi.
Toleransi Al. Pada prinsipnya ada dua mekanisme toleransi tanaman terhadap cekaman Al menurut Taylor (1991), yaitu : pertama mekanisme eksternal yakni dengan mencegah Al masuk ke dalam simplas dan mencapai daerah metabolik yang peka; dan yang kedua mekanisme internal yakni dengan imobilisasi, kompartementasi atau detoksifikasi saat Al masuk ke dalam simplas. Mekanisme toleransi Al pada tanaman bervariasi baik antar maupun intra spesies. Faktor genetik berperan penting dalam menentukan toleransi tersebut. Toleransi Al pada gandum (Triticum aestivum) dikendalikan oleh sejumlah kecil gen dominan mayor dan gen-gen ini telah dimanfaatkan dalam program pemuliaan untuk membuat kultivar yang toleransi terhadap cekaman Al (Johnson et al. 1997).
Percobaan tentang mekanisme toleransi Al yang dilakukan oleh beberapa peneliti mengindikasikan beberapa hal yaitu :
(b) Peningkatan akumulasi ion nitrat yang lebih tinggi dibandingkan kation amonium dan induksi pH rizosfir lebih tinggi mendekati pH optimal untuk pertumbuhan tanaman berhubungan dengan sifat tanaman yang lebih toleran terhadap cekaman Al (Miyasaka et al. 1991)
(c) Kultivar toleran mencirikan suatu mekanisme menghilangkan aluminium dari daerah sekitar akar yang disebabkan pengeluaran senyawa-senyawa asam dikarboksilat atau pengeluaran asam organik seperti malat, oksalat, sitrat dan fulfat propanoat seperti kaffeat untuk mengkelat Al sehingga toksisitasnya menjadi rendah (Ojima et al. 1984; Ryan et al. 1995; Sopandie et al. 1996; de la Funte et al. 1997; Ma et al. 1998; Zheng 1998; Matsumoto 2000). Beberapa genotipe tanaman bersifat toleran terhadap cekaman Al karena mereka melepaskan asam organik dari ujung akar. Beberapa asam organik memiliki kemampuan untuk mengubah kompleks Al3+ menjadi bentuk yang tidak bersifat toksik bagi tanaman, seperti pengkelatan Al sehingga akar terlindung dari toksisitas Al (Hue et al. 1986)
(d) Kultivar toleran mampu meningkatkan aktifitas H+-ATPase membran plasma yang mengatur keseimbangan ion proton antara di dalam dan luar membran plasma sel sehingga terjadi depolarisasi di membran plasma dan secara berantai mempengaruhi aktifitas metabolisme turunannya seperti aktifitas K-channel dan Ca-transporter yang masing-masing berperan dalam proses detoksifikasi Al (Kasai et al.1993; Kinraide et al. 1994; Sasaki et al. 1995; Kasai et al. 1995; Huang et al. 1996; Larsen et al. 1998; Delhaize 2004)
detoksifikasi eksternal dan internal bahkan beberapa spesies tanaman menggunakan kedua bentuk mekanisme tersebut.
Beberapa tanaman dapat mengakumulasi Al pada daun dan akarnya tanpa menunjukkan gejala keracunan. Spesies tanaman toleran Al ini mempunyai mekanisme untuk mengubah Al dalam bentuk non toksik di dalam tanaman yaitu mekanisme yang membiarkan Al masuk ke dalam tanaman dan melewati membran kemudian baru mengubahnya menjadi bentuk non toksik. Teh dan hydrangea telah dikenal sebagai akumulator Al. Daun teh tua dapat mengakumulasi Al hingga 30000 mg kg-1 pada berat kering dan akumulasi Al di daun hydrangea mencapai di atas 3000 mg kg-1 (Ma et al. 1997). Beberapa tanaman seperti Melastoma malabathricum dan Vaccinium macrocarpon yang beradaptasi baik pada pH rendah mengakumulasi Al dalam level yang tinggi baik di daun maupun di akar (Osaki et al. 1997).
Pada tanah dengan keasaman yang sangat tinggi, Al3+ dapat melewati membran plasma baik melalui transpor protein yang secara normal berfungsi dalam adsorpsi ion-ion mineral lainnya ataupun melalui fase-fluida dan adsorpsi endositosis. Berdasarkan beberapa penelitian sebelumnya, Al3+menghentikan kanal ion yang terdapat pada membran plasma sel akar. Berdasarkan bukti-bukti di atas, mekanisme transpor Al3+ tidak hanya terjadi secara aktif melalui transporter aktif primer (primary active transporters) tetapi juga melalui transporter aktif sekunder (secondary active transporter) serta menunjukkan kemungkinan bahwa tanaman akumulator Al memiliki mekanisme detoksifikasi Al secara internal.
al. 1998). Kultivar gandum yang sangat toleran Al (Atlas) mempunyai mekanisme pelepasan fosfat sebagai mekanisme toleransi Al (Pellet et al. 1996). Berbeda dengan pelepasan malat, pelepasan fosfat bersifat konstitutif tanpa dipengaruhi adanya induksi Al untuk mengaktifkannya. Tanaman rumput-rumputan menghasilkan asam amino non protein yang disebut phytosiderophore yang mampu mengkelat Fe3+, komplek divalen, kation polyvalen dan Al3+ dengan sangat efektif sehingga tidak bersifat toksik lagi bagi tanaman saat terserap ke dalam sitoplasma (Kochian 1995). Pada bakteri ditemukan juga siderophore yang berfungsi dalam transpor Fe3+ yaitu enterobactin dan major facilitator superfamily (MFS) merupakan transporter enterobactin pada yeast (Heymann et al. 2000). Penemuan-penemuan ini semakin menguatkan adanya mekanisme peningkatan toleransi Al selain dari mekanisme pelepasan asam organik.
Major Facilitator Superfamily (MFS)
berulang intragenik 6 unit TM primordial sehingga dihasilkan 12 unit TM dalam bentuk polipeptida tunggal.
Gambar 1. Topologi MFS transporter dengan 12 dan 14 domain transmembran
Penelitian MFS pada organisme tingkat tinggi dan tanaman masih sedikit sehingga baru sebagian kecil MFS yang ditemukan. Sebagian besar MFS telah ditemukan pada mikroorganisme (organisme tingkat rendah) dan telah diketahui fungsinya. Beberapa fungsi MFS yang sudah diketahui adalah sebagai sistem transpor gula (Henderson et al. 1990), sistem transpor senyawa toksin, obat (drug efflux) dan senyawa metabolit pada siklus Kreb (Grifith et al. 1992; Paulsen et al. 1997), sistem transpor fosfat yaitu sebagai phosphate exchanger dan oligo sakarida (H+ simport permease) (Marger et al. 1993) serta sebagai permease asam aromatik pada bakteri (Goffeau at al. 1997).
(15) aromatic acid: H+ symporter (AAHS) family, (16) unknown major facilitator superfamily (UMF) family, (17) cyanate permease (CP) family. Masing-masing famili mengenali dan mentranspor senyawa-senyawa yang strukturnya berbeda dan menunjukkan bahwa filogenetik dari famili MFS tersebut berhubungan erat dengan fungsinya.
Major Facilitator Superfamily merupakan transporter dengan kisaran substrat yang luas sehingga memiliki spesifisitas fungsi yang luas (Van Bambeke et al. 2000). Salah satu famili MFS yaitu MFS multidrug efflux transporter diketahui berperan didalam resistensi terhadap antibiotik seperti tetrasiklin, quinolone, methicilin dan phleomycin; obat anti HIV dan anti kanker seperti triazole derivative flucanazole (FLC), methotrexate (MTX) dan benomyl; fungisida seperti oxpaconazole (Kohli et al. 2001) dan logam berat seperti kadmium (Cd2+) (Li et al. 2001).
MFS multidrug efflux transporter menggunakan gradien elektrokimia transmembran dari proton atau ion sodium untuk mensekresi senyawa toksik dan xenobiotik dari dalam sel (Gambar 2). Meskipun demikian pada tanaman diduga detoksifikasi senyawa toksik dan xenobiotik dilakukan tidak dengan mensekresinya keluar sel tetapi mengirimnya ke dalam vakuola yang bertindak sebagai kompartemen bagi senyawa -senyawa toksik dan xenobiotik tersebut.
MFS multidrug efflux transporter pada tanaman pertama kali ditemukan pada jagung (Simmons et al. 2003). Simmons et al. (2003) mengindikasikan bahwa MFS multidrug efflux transporter pada jagung berperan di dalam resistensi terhadap patogen, eksport dan re-uptake K+, integritas membran dan viabilitas sel.
Beberapa penelitian membuktikan bahwa kultivar toleran Al mampu meningkatkan aktifitas H+-ATPase membran plasma yang mengatur keseimbangan ion proton antara di dalam dan di luar membran plasma sel, sehingga terjadi depolarisasi di membran plasma dan secara berantai mempengaruhi aktifitas metabolisme turunannya seperti aktifitas K-channel dan Ca-transporter yang masing-masing berperan dalam proses detoksifikasi Al (Kasai et al. 1993; Kinraide et al. 1994; Sasaki et al. 1995; Kasai et al. 1995; Huang et al. 1996; Larsen et al. 1998; Delhaize 2004)
Fluconazole resistance 1 (FLR1) yang menyandikan MFS transporter pada fungi Candida albicans terlibat di dalam induksi CAP1 dan YAP1 yang berperan dalam resistensi C. albicans dan S. cerevisiae terhadap senyawa toksik seperti cycloheximide (CYH), 4-nitroquinoline N-oxide (4-NQO), kadmium dan hidrogen peroksida sehingga hal ini mengindikasikan bahwa FLR1 merupakan regulator transkripsi CAP1 dan YAP1 (Alarco et al. 1997). Hidrogen peroksida merupakan salah satu oksida radikal (ROS, reactive oxigen species) yang dihasilkan pada sejumlah reaksi selular dan merupakan salah satu mekanisme yang muncul pertama kali pada tanaman untuk mengatasi stress oksidatif khususnya untuk reoksigenasi. Stress oksidatif disebabkan oleh berbagai faktor lingkungan diantaranya stress UV, serangan patogen (reaksi hipersensitif), herbisida dan kekurangan oksigen (Blokhina et al. 2002). Reaktifitas Al juga dapat menyebabkan terbentuknya oksida radikal (ROS, reactive oxigen species) yang beracun bagi sel.
TM5 yang menentukan fungsi MFS sebagai antiporter. Bagian variasi diantaranya yaitu domain TM8 dan TM9 yang menentukan spesifisitas substrat (Ditty et al. 1999) dan pusat simpul yang panjang yang terletak di antara domain TM6 dan TM 7 berperan penting dalam menentukan fungsi protein (Weinglass et al. 2000).
Karakteristik Melastoma dan Toleransinya terhadap Aluminium
Melastoma affine D. Don atau disebut juga Melastoma malabathricum L. (Meyer 1999) (Gambar 3) merupakan tanaman yang termasuk dalam super divisi Spermatophyta (tanaman berbiji), divisi Magnoliophyta (tanaman berbunga), group dikotil, famili Melastomataceae dan merupakan tanaman perennial serta mempunyai kebiasaan tumbuh sebagai herba dengan tinggi mencapai 3 m dengan bunga berwarna ungu terang atau keunguan serta buah yang berisi biji-biji kecil berwarna ungu tua atau hitam yang secara alami digunakan sebagai sistem perbanyakannya (USDA 2004).
Gambar 3. Melastoma affine L.
lebih dari 7000 mg kg-1 di dalam daun-daun yang muda (Watanabe & Osaki 2001). Pada daun Melastoma, Al ditemukan berada di dalam sel epidermal atas dan juga didistribusikan di dalam sel mesofil sedangkan pada akar Al ditemukan pada semua jaringan akar terutama dalam epidermis dan endodermis. Selanjutnya Watanabe dan Osaki (2003) menemukan bahwa Al mampu menembus jaringan endodermis dan masuk ke pembuluh xilem yang kemudian ditimbun di daun Melastoma. Bukti-bukti ini menunjukkan bahwa Melastoma memiliki kemampuan menyerap Al, memobilisasi dan menimbunnya di daun tanpa menimbulkan masalah kelainan fisiologis. Bentuk Al terlarut yang ditemukan di dalam jaringan Melastoma diidentifikasikan sebagai Al monomerik, Al-oksalat, Al-(oksalat)2 dan Al-(oksalat)3 (Watanabe et al. 1998).
Selain tahan terhadap cekaman Al, pertumbuhan Melastoma juga dipacu oleh Al (Watanabe et al. 2001a). Mekanisme induksi pertumbuhan Melastoma oleh Al ini masih belum jelas. Cekaman Al pada tanaman toleran akan menginduksi sejumlah gen untuk menghindari pengaruh ion Al. Pada Melastoma, gen-gen ini diduga tidak hanya berperan dalam mendetoksifikasi Al, akan tetapi juga berperan dalam menginduksi hormon pertumbuhan. Sebagaimana telah diketahui pertumbuhan tanaman diatur oleh hormon pertumbuhan seperti auksin, sitokinin dan ABA (Wareing & Philips 1981).
Waktu dan Tempat Penelitian
Penelitian dilaksanakan mulai bulan Februari 2005 sampai Mei 2006 di
Laboraturium Biologi Molekuler dan Seluler Tanaman dan di Laboratorium
Pusat Penelitian Sumberdaya Hayati dan Bioteknologi IPB.
Bahan
Bahan yang digunakan adalah daun dan akar tumbuhan melastoma
(Melastoma affine) yang tumbuh di lahan asam Jasinga Bogor Jawa Barat.
Primer spesifik untuk gen penyandi MFS yang didesain berdasarkan cDNA
Zm-MFS dari Zea mays dan Mt-Mfs dari Medicago truncantula dengan primer
forward (MFSF) “GAAAAATGCAATTCTCTTCA” dan primer reverse
(MSFR) “GAAGCTTAGACCGATTAAC” digunakan untuk mengisolasi
fragmen MFS dari Melastoma.
Primer ActF “ATGGCAGATGCCGAGGATAT” dan ActR
“CAGTTGTGCGACCACTTGCA” digunakan untuk mengamplifikasi cDNA
ekson1 – ekson2 dari aktin Glycine max (Ac.V00450). cDNA ekson1 – ekson2
dari aktin digunakan sebagai alat untuk mengevaluasi kemurnian RNA total dan
cDNA total dari kontaminan DNA genom. Plasmid pGEM® –T Easy digunakan
sebagai vektor pengklonan (Gambar 4). E. coli galur DH5α digunakan sebagai
sel inang dari plasmid rekombinan.
Metode Penelitian
Penelitian dilakukan melalui beberapa tahap yaitu isolasi RNA total,
sintesis cDNA total melalui transkripsi balik, isolasi fragmen cDNA MaMfs
melalui PCR, pengklonan cDNA ke dalam vektor pGEM®–T Easy, transformasi
genetik E. coli DH5α dengan vektor rekombinan, analisis cDNA sisipan dan analisis urutan nukleotida MaMfs seperti ditunjukkan pada Gambar 5.
Isolasi RNA total. Isolasi RNA total dilakukan menggunakan metode CTAB (Chang et al. 1993) yang dimodifikasi. Untuk itu, 0.5 – 1 g daun segar
yang dibuang tulang daunnya atau akar digerus hingga halus di dalam mortar
yang berisi 10 ml buffer ekstraksi 2X CTAB (2% CTAB, 0.1 M Tris pH 9.5, 20
mM EDTA, 1.4 M NaCl) yang mengandung 2% PVP 25000 dan 1% β-mercapto
ethanol. Setelah itu ekstrak sel dimasukkan ke dalam tabung 20 ml dan
diinkubasi 10 menit pada suhu 65oC, kemudian didinginkan dan ditambah
dengan 1x volume Kloroform : Isoamil alkohol (24:1), divorteks dan
disentrifugasi dengan kecepatan 15000 rpm selama 10 menit pada suhu 4oC
dengan rotor SW 11, Sorvall Ultra Pro 80. Supernatan (bagian atas cairan)
dipindahkan ke tabung baru, kemudian ditambah ¼ volume LiCl 10M.
Campuran diinkubasi pada suhu -20oC selama 2.5 jam. Campuran disentrifugasi
dengan rotor SW 11, Sorvall Ultra Pro 80 pada kecepatan 15000 rpm selama 10
menit pada suhu 4oC. Fase cairan dibuang, RNA total yang mengendap
disuspensi dengan 500 μl TE 1x (10 mM Tris HCl pH 7.4 dan 1 mM EDTA) dan
dipindah ke tabung eppendorf. Selanjutnya RNA diekstraksi dengan 1x volume
fenol pH 9 dan disentrifugasi pada kecepatan 15000 rpm (Jouan BR4i) selama 10
menit pada suhu kamar (20oC). Suspensi RNA (di bagian atas) diambil dan
diekstraksi dengan 1 x volume fenol/kloroform/isoamilalkohol (25:24:1) serta
disentrifugasi pada kecepatan 15000 rpm selama 10 menit pada suhu kamar
(20oC). Cairan bagian atas diambil dan dipresipitasi dengan penambahan ¼ x V
LiCl 10M dan kemudian diinkubasi semalam pada suhu -20oC. Untuk
mengendapkan RNA total, campuran disentrifugasi pada kecepatan 15000 rpm
selama 10 menit pada suhu 4oC. Cairan dibuang dan endapan RNA total dibilas
dengan penambahan 500 μl ethanol 70% kemudian disentrifugasi pada kecepatan
15000 rpm selama 10 menit pada suhu 4oC. Endapan dikeringkan dengan
vacuum dryer selama 15 menit, kemudian diresuspensikan dengan H2O yang
telah diperlakukan dengan DEPC. RNA total diukur kuantitasnya menggunakan
spektrofotometer UV-VIS (Cecil CE 2020) pada panjang gelombang 260 nm.
Rasio absorbansi RNA pada panjang gelombang 260 dan 280 nm digunakan
untuk mengetahui kemurnian RNA total dari kontaminan protein. Keutuhan
dalam buffer MOPS 1x (4.2 g/l MOPS, 0.41 g/l Na asetat, 0.37 g/l Na2- EDTA).
Visualisasi RNA dilakukan di atas transluminator UV dan difoto menggunakan
Digidoc. Kualitas RNA murni yang baik ditunjukkan oleh rasio absorbansi
A260/280 = 1.8 sampai 2.0 dan munculnya pita ribosom 18S dan 28S pada hasil
elektroforesis (Farrel 1993).
Untuk memperkirakan konsentrasi RNA, diasumsikan bahwa satu satuan
absorban pada panjang gelombang 260 nm setara dengan 40 μg/ml RNA
(Saunders dan Parkes 1999), sehingga kuantitas (konsentrasi) RNA dapat diduga
dengan rumus berikut:
[RNA] = OD 260nm x fp x 40 µg/ml
dimana;
fp = faktor pengenceran yaitu sebesar 700x
40 = nilai satu satuan absorban pada panjang gelombang 260 nm
(40μg/ml)
Sintesis cDNA total melalui transkripsi balik. Sintesis cDNA dilakukan dengan metode transkripsi balik (reverse transcription) menggunakan
SuperScript II RT (Invitrogen). Sintesis cDNA total dilakukan dengan
mencampur 500 ng RNA total dengan 4 μl 5x First-Strand Buffer, 1 μl primer
oligo (dT) (50 pmol/ μl), 8 μl 2.5 mM dNTP mix, 0.2 μl enzim RT (10 U/μl),
2 μl DTT (0.1 M) dan air yang diperlakukan dengan DEPC sehingga mencapai
volume akhir 20 μl. RT dilakukan dengan alat PCR (MJ Research TM 100)
dengan menginkubasikan campuran pada suhu 30°C, 10 menit; 42°C, 50 menit; 95°C, 5 menit; dan 15°C, 10 menit .
Keberhasilan sintesis cDNA total diuji dengan keberhasilan amplifikasi
cDNA ekson1 – ekson2 dari aktin melalui PCR dengan menggunakan cDNA
total sebagai DNA cetakan. Untuk itu cDNA total hasil RT sebanyak 2 μl
digunakan sebagai cetakan untuk reaksi PCR aktin yang dicampurkan dengan 2 μl 10x Taq buffer, 1.6 μl 2.5 mM dNTP mix, 2 μl untuk setiap pasang forward
primer dan reverse primer spesifik aktin (10 pmol/ μl), 0.2 μl Taq polimerase (5
U/ μl) dan ditambahkan dH2O steril sampai volume akhir 20 μl. PCR dilakukan
penempelan primer pada 55°C, 30 detik; pemanjangan pada 72°C 1.5 menit dan pemanjangan akhir pada 72°C, 7 menit. PCR dilakukan sebanyak 30 siklus.
Isolasi Fragmen cDNA MaMfs melalui PCR. PCR gen Mfs dilakukan dengan membuat campuran reaksi seperti pada amplifikasi cDNA aktin dengan
mengganti primer aktin dengan primer spesifik MFSF dan MFSR. Kondisi PCR
yang digunakan adalah: pra-PCR 94°C, 5 menit; denaturasi pada 94°C, 30 detik; penempelan primer pada 35°C, 30 detik; pemanjangan pada 72°C 1.5 menit dan pemanjangan akhir pada 72°C, 7 menit. PCR dilakukan sebanyak 35 siklus.
Pengklonan cDNA ke dalam vektor pGEM® –T Easy. cDNA MaMfs
hasil amplifikasi dengan PCR diklon ke dalam vektor plasmid pGEM® –T Easy
(Promega) yang berukuran 3015 bp mengikuti prosedur Promega (2003).
Pengklonan cDNA ke dalam pGEM® –T Easy dilakukan dengan cara
mencampurkan 1 μl (50 ng/ μl) pGEM® –T Easy dengan 3 μl cDNA MaMfs, 5 μl
buffer ligasi T4 DNA ligase 2X, 1 μl T4 DNA ligase (3 Weiss Unit/ μl), dan
ddH2O sampai volume akhir 10 μl. Campuran diinkubasi pada suhu 4oC selama
semalam.
Transformasi genetik bakteri E. coli DH5α dengan vektor rekombinan.
Hasil ligasi potongan cDNA MaMfs dengan pGEM® –T Easy untuk membentuk
plasmid rekombinan kemudian dimasukkan ke dalam sel bakteri E. coli DH5α yang kompeten. Transformasi E. coli dilakukan dengan menggunakan metode
yang dipublikasikan Suharsono (2002). Untuk itu satu koloni bakteri E. coli
DH5α dikulturkan dalam 2 ml media LB cair (1% bakto-tryptona, 0.5% bakto ekstrak khamir, 1% NaCl (w/v) dan dH2O sebagai pelarut) dan diinkubasi pada
suhu 37oC selama semalam pada inkubator goyang (250 rpm). E. coli DH5α hasil kultur semalam kemudian disubkultur sebanyak 200 μl di dalam 20 ml LB
cair dengan kondisi yang sama seperti saat kultur, hingga mencapai densitas
bakteri 4-7 x 107 sel/ml (OD600 = 0.4 - 0.5). Sel bakteri diendapkan dengan
disentrifugasi pada kecepatan 5000 rpm pada suhu 4oC selama 10 menit.
Endapan bakteri disuspensikan dalam 0.33 vol buffer transformasi TB (10 mM
PIPES, 15 mM CaCl2.2H2O, 250 mM KCl, 55 mM MnCl2.4H2O, pH 6.7), dan
pada suhu 4oC selama 10 menit. Endapan bakteri (pellet) disuspensikan dalam
8ml TB, ditambah 0.6 ml DMSO dan diinkubasikan di dalam es selama 10 menit
sehingga didapatkan sel bakteri kompeten. Sel bakteri kompeten sebanyak 50 μl
dicampur dengan 10 μl (50-100 μg) DNA plasmid dan diinkubasikan di dalam es
selama 25 menit. Campuran tersebut selanjutnya diberi kejutan panas dengan
diinkubasikan pada suhu 42oC selama 45 detik dan dimasukkkan kembali ke
dalam es selama 5 menit. Campuran tersebut kemudian ditambah dengan 100 μl
media 2xYT (16 g/l bacto-tryptone, 10 g/l bacto yeast extract, 5 g/l NaCl, pH
7.0) dan diinkubasi pada suhu 37oC selama 20 menit. Bakteri disebar secara
merata pada media LB yang mengandung 100mg/l ampisilin dan 10 μl 100 mM
IPTG serta 50 μl 2% (b/v) x-gal, kemudian diinkubasikan pada suhu 37oC
selama semalam.
Hanya bakteri yang mengandung plasmid pGEM®–T Easy yang dapat
hidup pada media yang mengandung antibiotik ampisilin. Selanjutnya adanya
X-gal dan IPTG dalam media LB padat akan menghasilkan koloni bakteri yang
berwarna biru dan putih. Hanya koloni bakteri yang berwarna putih yang
kemudian diisolasi pada media LB yang mengandung ampisilin karena di
dalamnya mengandung pGEM®–T Easy rekombinan. Bakteri yang mengandung
pGEM®–T Easy rekombinan tidak mampu menghasilkan β-galaktosidase
(insertional inactivation) sehingga x-gal tidak dapat diuraikan dan koloni tetap
berwarna putih, sedangkan koloni yang mengandung pGEM® –T Easy non
rekombinan akan berwarna biru karena mampu mengurai x-gal.
Untuk verifikasi bahwa koloni putih membawa plasmid rekombinan yang
berisi cDNA sisipan (MaMfs), maka PCR dilakukan dengan menggunakan
koloni putih sebagai bahan cetakan. Untuk itu, koloni ditusuk dengan tusuk gigi
kemudian disuspensikan ke dalam ddH2O dan dipanaskan 95 °C selama 10 menit
dan didinginkan 15 °C selama 5 menit. Suspensi ini dicampur dengan 1.5 μl
buffer taq, 30 mM MgCl2, 1.2 μl 2.5 mM dNTP mix, 1.5 μl (10 pmol/ μl) primer
MFSF, 1.5 μl (10 pmol/ μl) primer MFSR, 4% DMSO, 0.15 μl (5 U/ μl) enzim
taq DNA polymerase (Fermentas Inc.) di dalam volume 15 μl. PCR dilakukan
Analisis cDNA sisipan. Isolasi DNA plasmid pGEM®–T Easy
rekombinan yang terdapat di dalam E. coli DH5α dilakukan dengan menggunakan prosedur Suharsono (2002). Untuk itu satu koloni E. coli DH5α rekombinan ditumbuhkan di dalam 2 ml media LB yang mengandung 100 mg/l
ampisilin dan diinkubasi didalam inkubator bergoyang (250 rpm) pada suhu
37oC selama semalam. Kultur bakteri disentrifugasi dengan kecepatan
13000 rpm pada suhu 4oC selama 10 menit. Endapan bakteri kemudian
disuspensikan dalam 300 μl buffer suspensi sel (50 mM tris-HCl, pH 7.5 dan 10
mM EDTA), dan tambahkan 300 μl buffer lisis (0.2 M NaOH, dan 1% SDS),
homogenkan dengan dibolak-balik perlahan beberapa kali. Setelah lisis,
tambahkan 300 μl buffer netralisasi (5 M Na-Asetat, asam asetat glasial dan H2O
dengan perbandingan 60:11.5:28.5, pH 4.8) dan campuran ini disentrifugasi pada
kecepatan 13000 rpm pada suhu 4oC selama 20 menit. Supernatan yang
mengandung DNA plasmid diambil dan kemudian diekstraksi dengan 1 x
volume fenol-kloroform-isoamil alkohol (25:24:1) divorteks dan disentrifugasi
dengan kecepatan 13000 rpm pada suhu 4oC selama 2 menit. Supernatan
diambil dan diperlakukan dengan RNase pada suhu 37oC selama semalam untuk
menghilangkan RNA. Larutan kemudian diekstraksi kembali dengan PCI dan
disentrifugasi seperti ekstraksi sebelumnya. Supernatan kemudian diambil
dengan hati-hati dan dipresipitasi dengan menambahkan 0.1 x volume 3M
sodium asetat, pH 5.2 dan 2 x volume etanol absolut. Selanjutnya larutan
diinkubasi pada suhu -35oC selama 2 jam dan disentrifugasi dengan kecepatan
13000 rpm pada suhu 4oC selama 20 menit. Endapan (DNA plasmid) dibilas
dengan 1 x volume etanol 70% dan dikeringkan dengan vakum. DNA plasmid
yang sudah kering disuspensikan di dalam 5-10 μl ddH2O.
Plasmid yang dihasilkan dari isolasi plasmid kemudian dipotong dengan
enzim restriksi EcoR1 (Promega Inc.) untuk mengeluarkan cDNA sisipan dengan
cara mencampur 100 ng DNA plasmid, 10 U enzim restriksi EcoR1, buffer 1 x
dan ddH2O sampai volumenya 20 µl. Inkubasikan campuran reaksi pada suhu
Analisis urutan nukleotida MaMfs dan protein MaMFS. Pengurutan DNA dilakukan dengan menggunakan mesin pengurut DNA otomatis
(Automated DNA Sequencer ABI Prism 310, Perkin-Elmer). Identifikasi urutan
nukleotida hasil pengurutan DNA dilakukan dengan beberapa analisis. Analisis
kesejajaran lokal (local alignment) MaMfs berdasarkan urutan nukleotida dan
asam amino dengan data yang ada di GenBank dilakukan dengan program
BLAST (Basic Local Alignment Search Tools) yang disediakan NCBI (National
Center for Biotechnology Information) melalui http://www.ncbi.nlm.nih.gov/
BLAST/ (Rashidi & Buehler 2000). Daftar sandi asam amino disajikan pada
Lampiran 1.
Analisis situs restriksi dilakukan dengan menggunakan program
NEBCutter (http://www.firstmarket.com/cutter/cut2. htm). Analisis kemiripan
atau similaritas dan filogenetik Mfs dan MFS dari berbagai spesies dilakukan
dengan menggunakan program MAFFT versi 5.8 (Katoh et al. 2005). Domain
fragmen MaMfs ditentukan dengan program motif scan
(http://www.expasy.ch/prosite/). Analisis hidrofobisitas fragmen MaMFS
menggunakan fragmen MtMFS sebagai pembanding dan dilakukan dengan
HASIL DAN PEMBAHASAN
Isolasi RNA Total
RNA total dari M. affine telah berhasil diisolasi dari bahan tumbuhan berupa daun yang telah membuka sempurna yang kemudian disebut dengan daun
tua, daun pucuk, dan akar. Kuantifikasi RNA total dengan spektrofotometer
pada panjang gelombang 260 nm menunjukkan bahwa rendemen isolasi RNA
berkisar antara 171 μg dan 247 μg tiap g bahan tumbuhan (Tabel 1). Rendemen
tertinggi diperoleh dari bahan tumbuhan berupa daun pucuk. Daun pucuk
mempunyai sel yang sangat aktif melakukan metabolisme sehingga proses
transkripsi yang menghasilkan RNA juga sangat intensif.
Tabel 1. Rendemen Isolasi RNA Total
Nilai Absorbansi
Pengujian kemurnian RNA total yang dilakukan dengan membandingkan
nilai OD260 dan OD280 menunjukkan bahwa RNA total yang diisolasi mempunyai
kemurnian yang tinggi karena mempunyai rasio berkisar antara 1.86 dan 1.9.
Menurut Farrel (1993) RNA yang mempunyai rasio absorbansi pada 260 nm dan
280 nm antara 1.8 dan 2.0 adalah murni yang bebas dari kontaminan protein.
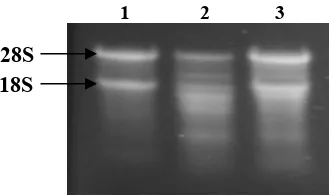
Keutuhan RNA total dianalisis dengan melakukan elektroforesis di gel
agarose yang terdenaturasi yang mengandung formaldehide (Gambar 6). Hasil
elektroforesis RNA total menunjukkan adanya 2 pita RNA yang dominan pada
RNA total yang diisolasi dari akar, daun tua dan daun pucuk yang kemungkinan
adalah RNA ribosomal (rRNA) 28S dan 18S. Selain kedua pita dominan
tersebut, RNA total dari daun tua dan daun pucuk, juga mengandung beberapa
pita RNA yang kemungkinan adalah rRNA yang terdapat pada mitokondria dan
1 2 3
28S 18S
Gambar 6. RNA total yang terdapat pada ketiga sampel yaitu akar (1), daun tua (2) dan daun pucuk (3).
Sintesis cDNA Total
cDNA total telah disintesis melalui proses transkripsi balik dengan RNA
total sebagai cetakan (template). Dengan primer oligo-dT, hanya mRNA yang dapat disintesis menjadi cDNA melalui transkripsi balik. PCR dengan primer
spesifik untuk cDNA ekson1-ekson2 dari aktin digunakan untuk mengetahui
keberhasilan sintesis cDNA total, dan kemurnian cDNA total dan RNA total dari
kontaminan DNA.
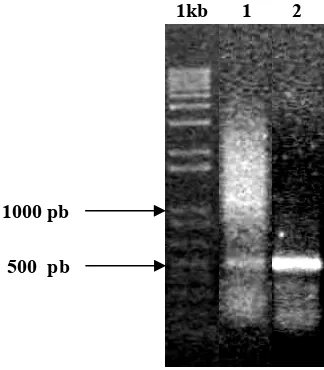
PCR dengan menggunakan primer untuk ekson1 - ekson2 dari gen aktin
(ActF dan ActR) dan dengan cetakan cDNA total, menghasilkan pita DNA yang
berukuran sekitar 450 pb (gambar 7). Hasil ini menunjukkan bahwa daerah yang
diamplifikasi adalah cDNA ekson1-ekson2 bukan DNA ekson1 - ekson2 dari gen
aktin. DNA ekson1 - ekson 2 akan berukuran lebih besar daripada 450 pb karena
diantara kedua ekson tersebut terdapat intron yang dibuang pada saat
pembentukan mRNA. DNA antara ekson1 dan ekson2 berukuran sekitar 600 pb.
M 1 2 3
1000 pb
500 pb
Teramplifikasinya cDNA dengan primer ActF dan ActR dengan ukuran
450 pb menunjukkan bahwa cDNA total telah berhasil disintesis melalui proses
transkripsi balik dan murni dari kontaminan DNA. Selain itu, hal ini juga
membuktikan bahwa RNA total yang telah diisolasi mempunyai kualitas yang
sangat bagus, karena selain dapat digunakan untuk mensintesis cDNA juga
terbebas dari DNA.
Isolasi Fragmen cDNA MaMfs Melalui PCR
PCR dengan cDNA total sebagai cetakan dan primer spesifik fragmen Mfs, menghasilkan fragmen cDNA berukuran sekitar 500 pb, yang kemudian disebut
dengan fragmen MaMfs yang merupakan fragmen cDNA dari kandidat gen penyandi MFS dari M. affine. Adanya DNA yang berukuran sama yaitu sekitar 500 pb yang dihasilkan dari PCR dengan cDNA total sebagai cetakan baik dari
akar maupun daun pucuk (gambar 8) menunjukkan bahwa Mfs diekspresikan baik pada akar maupun pada daun. Walaupun masih memerlukan konfirmasi
dengan menggunakan kontrol internal untuk ekspresi, berdasarkan intensitas pita
DNA hasil PCR menunjukkan bahwa tingkat ekspresi Mfs di akar lebih tinggi daripada di daun.
1kb 1 2
500 pb 1000 pb
Pengklonan Fragmen MaMfs ke dalam Plamid pGEM®–T Easy
Fragmen MaMfs telah diligasikan dengan plasmid pGEM-T Easy di tengah gen lacZ dan hasil ligasi kemudian diintroduksikan ke dalam E. coli galur DH5α. Seleksi terhadap E. coli yang mengandung plasmid rekombinan pembawa sisipan fragmen cDNA MaMfs dilakukan di media seleksi yang mengandung antibiotik ampisilin, X-gal dan IPTG. E. coli yang dapat bertahan hidup di media seleksi mengandung plasmid, dan koloni E. coli yang berwarna putih di media seleksi adalah E. coli yang mengandung plasmid rekombinan, dan yang berwarna biru adalah E. coli yang mengandung plasmid non-rekombinan (gambar 9).
Koloni putih
Koloni biru
Gambar 9. Seleksi bitu putih koloni E. coli DH5α yang ditransformasi dengan hasil ligasi antara pGEM®–T Easy dan MaMfs pada media seleksi yang mengandung X-gal, IPTG dan antibiotik ampisilin
Gen lacZ menyandi β-galactosidase (β-gal) yang mengubah substrat X-gal yang tidak berwarna menjadi berwarna biru bila ada IPTG yang berfungsi
menginduksi promoter lacZ. Bila fragmen MaMfs menyisip pada bagian tengah dari gen lacZ, yang berarti menghasilkan plasmid rekombinan, maka gen lacZ
tidak dapat diekspresikan sehingga koloni E. coli berwarna putih. Bila tidak ada penyisipan pada lacZ, yang berarti menghasilkan plasmid non-rekombinan, koloni yang terbentuk berwarna biru.
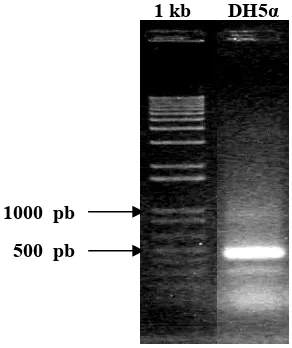
Adanya sisipan fragmen cDNA MaMfs di dalam koloni E. coli putih dikonfirmasi dengan PCR terhadap koloni yang kemudian disebut dengan
sekitar 500 pb yang menunjukkan bahwa koloni putih mengandung fragmen
MaMfs (gambar 10).
1 kb DH5α
1000 pb
500 pb
Gambar 10. Hasil PCR terhadap koloni E. coli DH5α (koloni putih) sebagai cetakan
Untuk memastikan bahwa fragmen cDNA MaMfs telah tersisip di dalam pGEM-T Easy, maka DNA plasmid rekombinan telah diisolasi dari koloni putih.
Pemotongan terhadap DNA plasmid rekombinan dengan EcoR1 yang mengapit daerah penyisipan menghasilkan dua fragmen yaitu fragmen sekitar 3000 pb
yang merupakan vektor pGEM-T Easy dan fragmen yang berukuran sama
dengan fragmen cDNA MaMfs yaitu sekitar 500 pb (gambar 11). Hal ini menunjukkan bahwa fragmen MaMfs telah tersisip di dalam pGEM-T Easy.
Vektor
Sisipan
1000 pb
500 pb 3000 pb
1 kb 1 2
Analisis Fragmen MaMfs
Pengurutan DNA terhadap fragmen MaMfs menghasilkan 511 pb yang menyandi 168 asam amino (Gambar 12). Keseluruhan hasil pengurutan DNA
termasuk posisi primer dan situs pengklonan pada pGEM®-T Easy disajikan
pada Lampiran 2. Analisis kesejajaran lokal (local alignment) berdasarkan urutan nukleotida dengan data di genBank menggunakan program BLAST
(Basic Local Alignment Search Tool) menunjukkan bahwa fragmen cDNA
MaMfs mempunyai kesamaan 74% dengan bagian coding sequence (cds) A. thaliana (AY056374), 73% dengan klon mth2-61c11 Medicago truncantula
(AC148655) pada bagian cds 29835-35720 yang menyandi Major Facilitator Superfamily (MFS_1) dan 71% dengan bagian mRNA (complete cds) yang menyandi tetracycline resistance efflux protein like protein (MFS_1) dari
Arabidopsis thaliana (AK230262). Hasil selengkapnya disajikan pada Lampiran 3. Salah satu contoh analisis kesejajaran lokal antara MaMfs dengan MtMfs
disajikan pada Lampiran 4.
Analisis kesejajaran berdasarkan urutan asam amino yang dideduksi dari
cDNA Mfs menunjukkan bahwa fragmen MaMFS mempunyai kesamaan 80% dengan bagian MFS_1 dari M. truncantula (Q1S1V4), 77% dengan bagian
hypotetical protein A. thaliana (O82747), 73% dengan OsMFS_1 dari padi (Q7XTZ1), 73% dengan bagian MFS_1 M. truncantula (Q2HV19), 73% dengan bagian tetracycline resistance efflux protein like protein (MFS_1) A. thaliana
(Q9LQ03), 30% dengan bagian putative MFS multidrug efflux pump (MFS_1) dari bakteri Yersinia pseudotuberculosis (Q66DU6) dan 30% dengan bagian
multidrug efflux transporter (MFS_1) dari bakteri Wollinela succinogenes
(Q1Q9B7). Hasil selengkapnya disajikan pada Lampiran 5. Contoh analisis
kesejajaran lokal berdasarkan urutan asam amino disajikan pada Lampiran 6.
Karena banyak kesamaan dengan MFS dari spesies lain, maka fragmen cDNA
yang diperoleh pada penelitian ini adalah termasuk fragmen cDNA penyandi
Major Facilitator Superfamily (MFS) dari Melastoma affine.
Analisis situs pemotongan untuk enzim restriksi pada suatu fragmen DNA
sangat penting untuk rekayasa genetika. Fragmen MaMfs tidak mengandung situs EcoR1 (gambar 13). Situs EcoR1 terdapat pada vektor pengklonan yang digunakan yaitu pGEM-T Easy yang mengapit daerah sisipan sehingga EcoR1 dapat digunakan untuk mengeluarkan sisipan MaMfs dari plasmid rekombinan secara utuh. Situs Pst1 terdapat di dalam MaMfs sehingga tidak dapat digunakan untuk mengeluarkan sisipan MaMfs dari pGEM®-T Easy.
Gambar 13. Situs pemotongan enzim restriksi endonuklease pada fragmen MaMfs
Analisis kesamaan berdasarkan urutan nukleotida Mfs dari berbagai spesies disajikan dalam matriks kesamaan pada Tabel 2. Fragmen MaMfs
memiliki kesamaan tertinggi dengan MtMfs yang berasal dari tanaman
M.truncantula yaitu sebesar 53.5%. Kesamaannya dengan Mfs tanaman lainnya yaitu sebesar 52.2% dengan A. thaliana Mfs dan 49.5% dengan O. sativa Mfs. Kesamaannya dengan Mfs selain tanaman yaitu Mfs yang berasal dari bakteri, yeast dan manusia berkisar dari 20.9% hingga 28.3%. Berdasarkan nilai
kesamaan nukleotida Mfs dari berbagai spesies maka pohon filogenetik disusun (Gambar 14). Pada pohon filogenetik yang berdasarkan urutan nukleotida,
MaMfs terdapat dalam satu kelompok dengan MtMfs.
Analisis kesamaan berdasarkan deduksi asam amino fragmen MaMFS
dengan asam amino fragmen MFS yang terdapat pada data Gen Bank disajikan pada Tabel 3. Fragmen MaMFS memiliki kesamaan tertinggi dengan MtMFS
yaitu sebesar 66.6%. Kesamaannya dengan MFStanaman lainnya yaitu sebesar
61.2% dengan A. thaliana MFS dan 60.7% dengan O. sativa MFS. Kesamaanya dengan MFS yang bukan berasal dari tanaman (bakteri, yeast dan manusia)
berkisar dari 3.5% hingga 8.3%. Berdasarkan kesamaan urutan asam amino,
MaMFS dari M. affine terdapat dalam satu kelompok dengan MtMFS dari
M. truncantula pada pohon filogenetik (Gambar 15). Kedekatan MaMFS dengan MtMFS karena primer yang digunakan adalah bagian terkonservasi hasil
penyejajaran antara urutan nukleotida MtMFS dan ZmMFS sehingga ada
kemungkinan teramplifikasi bagian yang sama antara Melastoma dan Medicago, namun pada dendogram terlihat ZmMFS mempunyai jarak yang jauh dengan
MaMFS dengan kesamaan yang kecil (24.5%) (Tabel 3). Ini mungkin saja
terjadi karena bagian yang kesamaannya tinggi antara Melastoma, Medicago dan
Zea mays adalah bagian terkonservasi sehingga ada kemungkinan teramplifikasi bagian yang tidak sama.
Nilai kesamaan yang ditunjukkan pada tabel matriks kesamaan dan
digambarkan dalam pohon filogenetik berbeda dengan nilai kesejajaran lokal
MaMFS dengan seluruh Mfs dan MFS secara utuh. Oleh sebab itu nilai homologi hasil BLAST lebih tinggi dari pada nilai kesamaan berdasarkan
MAFFT.
Susunan nukleotida dan asam amino MaMFS memiliki kesamaan yang
cukup tinggi dengan MFS dari spesies lain sehingga MaMFS diduga mempunyai
kesamaan peran sebagai transporter senyawa toksik dan transporter berbagai obat
(MFS multidrug efflux transporter). MFS multidrug efflux transporter berperan di dalam resistensi suatu organisme terhadap: (1) antibiotik seperti tetrasiklin,
quinolone, methicilin dan phleomycin, (2) obat anti HIV, dan (3) anti kanker
seperti triazole derivative flucanazole (FLC), methotrexate (MTX) dan benomyl,
(4) fungisida seperti oxpaconazole (Kohli et al. 2001) dan logam berat seperti cadmium (Li et al. 2001). Karena Al3+ memiliki kemiripan dengan Cd2+, kemungkinan MFS mempunyai peranan yang sama dalam detoksifikasi terhadap
Tabel 2. Matriks kesamaan urutan nukleotida fragmen MaMfs dan Mfs dari organisme lain
Gen MaMfs MtMfs AtMfs OsMfs PseMfs SerrMfs ArcMfs OceMfs SalMfs YerMfs ExiMfs BacilMfs AzoMfs ZmMfs SsacMfs SacMfs CalMfs CmalMfs HmMfs EcoMfs VibMfs CochMfs TricMfs ShewMfs MariMfs Whtpht1 Atpht15 Solpht1
MaMfs ID 0.535 0.522 0.495 0.274 0.254 0.249 0.274 0.250 0.231 0.229 0.276 0.243 0.245 0.252 0.245 0.283 0.258 0.266 0.209 0.239 0.261 0.255 0.229 0.247 0.276 0.294 0.266
MtMfs 0.535 ID 0.750 0.715 0.243 0.241 0.271 0.251 0.263 0.253 0.257 0.275 0.225 0.253 0.269 0.270 0.266 0.266 0.202 0.240 0.253 0.228 0.259 0.239 0.277 0.251 0.267 0.273
AtMfs 0.522 0.750 ID 0.719 0.259 0.235 0.278 0.214 0.257 0.280 0.253 0.275 0.245 0.239 0.278 0.249 0.256 0.272 0.217 0.248 0.267 0.219 0.250 0.253 0.298 0.247 0.255 0.249
OsMfs 0.495 0.715 0.719 ID 0.257 0.249 0.271 0.233 0.280 0.257 0.259 0.271 0.216 0.247 0.269 0.254 0.248 0.241 0.233 0.244 0.253 0.238 0.236 0.241 0.288 0.229 0.263 0.249
PseMfs 0.274 0.243 0.259 0.257 ID 0.682 0.345 0.616 0.610 0.636 0.273 0.288 0.316 0.231 0.252 0.231 0.262 0.219 0.262 0.228 0.250 0.234 0.261 0.276 0.264 0.250 0.282 0.257
SerrMfs 0.254 0.241 0.235 0.249 0.682 ID 0.369 0.600 0.730 0.766 0.293 0.286 0.298 0.224 0.272 0.222 0.264 0.229 0.270 0.201 0.260 0.213 0.250 0.280 0.249 0.232 0.282 0.255
ArcMfs 0.249 0.271 0.278 0.271 0.345 0.369 ID 0.337 0.353 0.330 0.274 0.290 0.272 0.226 0.241 0.243 0.246 0.215 0.250 0.250 0.258 0.242 0.276 0.298 0.284 0.246 0.278 0.275
OceMfs 0.274 0.251 0.214 0.233 0.616 0.600 0.337 ID 0.552 0.578 0.251 0.294 0.274 0.230 0.254 0.256 0.279 0.264 0.266 0.205 0.280 0.198 0.244 0.262 0.260 0.255 0.280 0.297
SalMfs 0.250 0.263 0.257 0.280 0.610 0.730 0.353 0.552 ID 0.698 0.303 0.276 0.285 0.226 0.284 0.212 0.248 0.249 0.289 0.228 0.287 0.225 0.253 0.305 0.245 0.222 0.273 0.240
YerMfs 0.231 0.253 0.280 0.257 0.636 0.766 0.330 0.578 0.698 ID 0.305 0.282 0.254 0.243 0.270 0.225 0.275 0.229 0.264 0.215 0.270 0.213 0.229 0.266 0.232 0.246 0.280 0.271
ExiMfs 0.229 0.257 0.253 0.259 0.273 0.293 0.274 0.251 0.303 0.305 ID 0.276 0.263 0.251 0.247 0.272 0.234 0.270 0.258 0.275 0.265 0.244 0.238 0.331 0.216 0.226 0.251 0.273
BacilMfs 0.276 0.275 0.275 0.271 0.288 0.286 0.290 0.294 0.276 0.282 0.276 ID 0.250 0.257 0.264 0.283 0.248 0.231 0.239 0.240 0.284 0.219 0.278 0.278 0.284 0.259 0.265 0.273
AzoMfs 0.243 0.225 0.245 0.216 0.316 0.298 0.272 0.274 0.285 0.254 0.263 0.250 ID 0.249 0.215 0.206 0.203 0.207 0.245 0.209 0.274 0.236 0.263 0.281 0.250 0.220 0.253 0.244
ZmMfs 0.245 0.253 0.239 0.247 0.231 0.224 0.226 0.230 0.226 0.243 0.251 0.257 0.249 ID 0.259 0.254 0.247 0.249 0.254 0.281 0.247 0.269 0.255 0.253 0.226 0.233 0.222 0.210
SsacMfs 0.252 0.269 0.278 0.269 0.252 0.272 0.241 0.254 0.284 0.270 0.247 0.264 0.215 0.259 ID 0.247 0.283 0.276 0.221 0.251 0.256 0.257 0.255 0.284 0.249 0.265 0.273 0.332
SacMfs 0.245 0.270 0.249 0.254 0.231 0.222 0.243 0.256 0.212 0.225 0.272 0.283 0.206 0.254 0.247 ID 0.285 0.277 0.254 0.276 0.229 0.265 0.257 0.218 0.216 0.243 0.266 0.274
CalMfs 0.283 0.266 0.256 0.248 0.262 0.264 0.246 0.279 0.248 0.275 0.234 0.248 0.203 0.247 0.283 0.285 ID 0.293 0.266 0.246 0.254 0.217 0.280 0.246 0.235 0.244 0.283 0.291
CmalMfs 0.258 0.266 0.272 0.241 0.219 0.229 0.215 0.264 0.249 0.229 0.270 0.231 0.207 0.249 0.276 0.277 0.293 ID 0.256 0.271 0.203 0.269 0.238 0.207 0.192 0.256 0.254 0.301
HmMfs 0.266 0.202 0.217 0.233 0.262 0.270 0.250 0.266 0.289 0.264 0.258 0.239 0.245 0.254 0.221 0.254 0.266 0.256 ID 0.230 0.249 0.253 0.240 0.250 0.230 0.315 0.266 0.274
EcoMfs 0.209 0.240 0.248 0.244 0.228 0.201 0.250 0.205 0.228 0.215 0.275 0.240 0.209 0.281 0.251 0.276 0.246 0.271 0.230 ID 0.230 0.271 0.248 0.189 0.247 0.226 0.222 0.217
VibMfs 0.239 0.253 0.267 0.253 0.250 0.260 0.258 0.280 0.287 0.270 0.265 0.284 0.274 0.247 0.256 0.229 0.254 0.203 0.249 0.230 ID 0.207 0.217 0.376 0.256 0.251 0.235 0.281
CochMfs 0.261 0.228 0.219 0.238 0.234 0.213 0.242 0.198 0.225 0.213 0.244 0.219 0.236 0.269 0.257 0.265 0.217 0.269 0.253 0.271 0.207 ID 0.238 0.211 0.266 0.246 0.261 0.238
TricMfs 0.255 0.259 0.250 0.236 0.261 0.250 0.276 0.244 0.253 0.229 0.238 0.278 0.263 0.255 0.255 0.257 0.280 0.238 0.240 0.248 0.217 0.238 ID 0.251 0.290 0.221 0.198 0.259
ShewMfs 0.229 0.239 0.253 0.241 0.276 0.280 0.298 0.262 0.305 0.266 0.331 0.278 0.281 0.253 0.284 0.218 0.246 0.207 0.250 0.189 0.376 0.211 0.251 ID 0.271 0.224 0.227 0.238
MariMfs 0.247 0.277 0.298 0.288 0.264 0.249 0.284 0.260 0.245 0.232 0.216 0.284 0.250 0.226 0.249 0.216 0.235 0.192 0.230 0.247 0.256 0.266 0.290 0.271 ID 0.267 0.256 0.281
Whtpht1 0.276 0.251 0.247 0.229 0.250 0.232 0.246 0.255 0.222 0.246 0.226 0.259 0.220 0.233 0.265 0.243 0.244 0.256 0.315 0.226 0.251 0.246 0.221 0.224 0.267 ID 0.567 0.614
Atpht15 0.294 0.267 0.255 0.263 0.282 0.282 0.278 0.280 0.273 0.280 0.251 0.265 0.253 0.222 0.273 0.266 0.283 0.254 0.266 0.222 0.235 0.261 0.198 0.227 0.256 0.567 ID 0.614
Gambar 14. Filogenetik berdasarkan urutan nukleotida fragmen MaMfs dengan Mfs
Tabel 3. Matriks kesamaan asam amino fragmen MaMFS dan MFSdari organisme lain
Gen MaMfs MtMfs AtMfs OsMfs PseMfs SerrMfs ArcMfs OceMfs SalMfs YerMfs ExiMfs BacilMfs AzoMfs ZmMfs SsacMfs SacMfs CalMfs CmalMfs HmMfs EcoMfs VibMfs CochMfs TricMfs ShewMfs MariMfs Whtpht1 Atpht15 Solpht1
Analisis Domain Fragmen MaMFS
MaMFS memiliki kesamaan tertinggi dengan MtMFS. MtMFS
memiliki 12 domain putatif transmembran. Protein MFS tersusun dari 12-14
domain putatif transmembran (TMS) yang diduga terjadi karena duplikasi
berulang intragenik dari 6 unit TMS sebelumnya (Rubin et al. 1990; Pao et al. 1998; Van Bambeke etal. 2000). Hasil analisis kesejajaran asam amino fragmen MaMFS yang terdiri dari 168 asam amino dengan MtMFS menunjukkan bahwa
fragmen MaMFS sejajar dengan MtMFS pada asam amino ke-209 sampai
dengan asam amino ke-375 yang mengandung 4 domain putatif transmembran
yaitu TM6, TM7, TM8 dan TM9 (Gambar 16) sebagaimana hasil prediksi
transmembran fragmen MaMFS dengan program TMpred 2.0 (Tabel 4). Bagian
terkonservasi dari MFS yaitu pada domain TM 2 dan TM 3 yang menentukan
fungsi transpor MFS secara umum (Yamaguchi et al. 1992) dan domain TM 5 yang menentukan fungsi MFS sebagai antiporter. Bagian variasi diantaranya
yaitu domain TM8 dan TM9 yang menentukan spesifisitas substrat (Ditty et al. 1999)
MFSMelastoma
Tabel 4. Analisis domain transmembran menggunakan program TMpred versi 2.0
Segmen
Domain TM Bagian pada Asam amino
Pusat TM
(Asam amino ke-) Nilai TMpred
TM6 (o-i) 46 - 62 54 1078
Domain TM6 dan TM 7 merupakan domain transmembran yang diketahui
menentukan berfungsinya protein (Weinglass etal. 2000) dan domain TM 8 dan TM 9 merupakan domain transmembran yang menentukan spesifisitas substrat
MFS. Berdasarkan analisis kesejajaran antara fragmen MFS pada TM6 dan TM7
(asam amino ke-46 sampai dengan asam amino ke-93 pada MaMFS) serta TM8
dan TM9 (asam amino ke-113 sampai dengan asam amino ke-152 pada MaMFS)
kemungkinan MaMFS mempunyai peran yang sama dengan MFS lainnya yaitu
dalam transpor senyawa toksik dan multidrug transporter.
Hasil analisis kemiripan terhadap TM6 – TM9 dari MaMFS dan TM MFS
dari beberapa spesies lainnya secara umum menunjukkan bahwa TM6-TM9
MaMFS mempunyai kesamaan dengan TM6-TM9 MFS dari kelompok tanaman
dan berbeda dengan TM MFS yang berasal dari kelompok bakteri, Archaea dan manusia.
Hasil analisis kemiripan TM6-TM9 MaMFS dengan MtMFS, AtMFS dan
OsMFS disajikan pada Tabel 5. TM6-TM9 dari MaMFS mempunyai kesamaan
dengan TM6-TM9 MFS dari M. truncantula (MtMFS), A. thaliana (AtMFS) dan
O. sativa (OsMFS) yang ditunjukkan dengan persentase kemiripan berkisar dari 75.00% sampai dengan 100.00%.
Hasil analisis kesejajaran transmembran dan pusat transmembran
TM6-TM9 MaMFS dengan MtMFS, AtMFS dan OsMFS disajikan pada Gambar
17. MaMFS mempunyai pusat TM6, TM7 dan TM9 yang sama dengan MtMFS,
AtMFS dan OsMFS yaitu berturut-turut asam amino tirosin (Y) pada asam
amino ke-54, asam amino isoleusin (I) pada asam amino ke-83 dan asam amino
OsMFS adalah asam amino yang berbeda. TM8 MaMFS mempunyai pusat
asam amino isoleusin (I), pusat TM8 MtMFS adalah alanin (A) dan pusat TM8
AtMFS sama dengan OsMFS yaitu asam amino leusin (L).
Tabel 5. Kemiripan TM6, TM7, TM8 dan TM9 dari MaMFS dengan MtMFS, AtMFS dan OsMFS
Kemiripan/identities dengan MaMFS (%)
Nama MFS
TM6 TM7 TM8 TM9
MtMFS 94.12 90.48 90.00 81.48
AtMFS 100.00 85.71 80.00 77.78
OsMFS 94.12 80.95 75.00 85.18
Keterangan:
Biru : Asam amino nonpolar tidak bermuatan Merah : Asam amino polar tidak bermuatan Huruf dalam lingkaran merah adalah pusat transmembran