STUDI MOLECULAR DOCKING EKSTRAK KURKUMINOID
ASAL WONOGIRI SEBAGAI INHIBITOR ENZIM
DNA TOPOISOMERASE II
TIRTA SETIAWAN
DEPARTEMEN BIOKIMIA SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa tesis berjudul Studi Molecular
Docking Ekstrak Kurkuminoid asal Wonogiri sebagai Inhibitor Enzim DNA
Topoisomerase II adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka dibagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Oktober 2015
iv
RINGKASAN
TIRTA SETIAWAN. Studi Molecular Docking Ekstrak Kurkuminoid asal Wonogiri sebagai Inhibitor Enzim DNA Topoisomerase II. Dibimbing oleh LAKSMI AMBARSARI dan TONY IBNU SUMARYADA.
Kurkuminoid adalah senyawa fitokimia yang terkandung dalam ekstrak kasar fraksi etanol dari temulawak. Penelitian ini menggunakan temulawak asal Wonogiri yang memiliki kadar kurkuminoid tertinggi dari empat sampel temulawak yang diambil dari daerah Balitro, Ciemas dan Sukabumi. Ekstrak kasar kurkuminoid asal Wonogiri diketahui memiliki efek inflamasi dan anti-kanker. Inflamasi yang bersifat kronis berkaitan erat dengan pembentukan sel kanker.
Senyawa kurkumin dilaporkan memiliki aktivitas anti-kanker. Modifikasi struktur kurkumin bertujuan untuk menghasilkan senyawa analog kurkumin yang memiliki aktifitas yang lebih spesifik terhadap target penyebab kanker. Penelitian ini bertujuan untuk mempelajari potensi ekstrak kurkuminoid asal wonogiri sebagai kandidat agen inhibitor enzim DNA topoisomerase II melalui metode penambatan molekular dengan mengamati nilai energi bebas Gibbs (∆G) dan
interaksi kimia yang terjadi seperti ikatan Pi, ikatan hidrogen, interaksi hidrofobik, interaksi elektrostatik dan gugus samping obat yang berinteraksi.
Simulasi penambatan molekular dilakukan dengan aplikasi AutoDockVina. Ligan yang digunakan adalah kurkumin, bisdemetoksikurkumin, demetoksikurkumin dan ligan pembanding etoposida sedangkan reseptor yang digunakan adalah enzim DNA topoisomerase II berkode 3qx3. Aplikasi LigPlot 4.5.3, ADT 1.5.1 (Autodock Tools) dan VMD 1.9.2 (Virtual Molecular Dinamic) digunakan untuk persiapan ligan dan reseptor.
Struktur 3D reseptor hasil kristalisasi cukup stabil, 98% (1327/1365) data asam amino yang berada pada daerah paling disukai (favorable), daerah yang diizinkan (allowed) sebesar 99.9% (1363/1365) dan daerah terlarang (disallowed region) kurang dari 1%. Berdasarkan hasil penambatan molekular dapat disimpulkan bahwa residu Asp479, Arg503, Gly478
, Gln778, DA12 (Adenin), DG13 (Guanin), DT9 (Timin), DC8 (Sitosin) dan Met782 berperan penting dalam pembentukan ikatan Pi (π), ikatan hidrogen dan interaksi elektrostatik terhadap enzim DNA topoisomerase II. Ikatan Pi dibentuk oleh cincin benzena A dari senyawa kurkuminoid terhadap basa DNA (DA12, DG13, DT9, DC8) dengan mode interkalasi, ikatan hidrogen (gugus OH2 dan OH3 dari senyawa kurkuminoid), interaksi hidrofobik (Met782) dan interaksi elektrostatik (Asp479, Arg503, Gln778, Glu477).Ekstrak kurkuminoid yang terdiri dari kurkumin, demetoksi kurkumin dan
bisdemetoksi kurkumin memiliki nilai energi bebas Gibbs (ΔGbinding) sebesar -10.1 kkal/mol, -9 kkal/mol, -8.9 sehingga berpotensi sebagai agen inhibitor enzim DNA topoisomerase II.
SUMMARY
TIRTA SETIAWAN. Molecular Docking Study of Wonogiri’s Curcuminoid
Turmeric for Inhibitor in DNA Topoisomerase II Enzyme. Supervised by
LAKSMI AMBARSARI and TONY IBNU SUMARYADA.
Curcuminoids are phytochemical compounds that contained in etanol part of Temulawak. Temulawak from Wonogiri region has highest of curcuminoids level from Balitro, Ciemas and Sukabumi region. Wonogiri’s crude turmeric curcuminoids have been known to have anti-inflamatory effect. The cronic infalamatory is related to cancer cell development.
Curcumins compound has been reported to have anti-cancer activity. Modification curcumins structure is aim to produce curcumin analogs that have more specific target. However, synthetic analog has been reported to have significan metabolism problem. Curcuminoids also reported to have similarly activity with curcumin. Curcuminoids are natural derivative compound of curcumin. Natural derivative compound more save than synthetic derivative. The
aim of this research is to study Wonogiri’s curcuminoid as inhibitor in Topoisomerase II enzyme by its Pi binding, hydrogen bond, binding energy interaction, drug function and binding mode which are formed in complex.
In this research, the docking simulations were performed using AutoDockVina program. Ligands used are curcumin, demetoxycurcumin, bisdemetoxycurcumin and etoposide, DNA topoisomerase II enzyme was used as protein target. PyMOL, LigPlot 4.5.3, ADT 1.5.1 (Autodock Tools) and VMD 1.9.2 (Virtual Molecular Dinamic) were used for ligand and receptor preparations. Three dimension structure of 3QX3 has been a stable conformation, its result considering of amount of 98% of amino acids in favorable area (1327/1365), 99.9% in allowed region (1363/1365) and 1% in disallowed region. The potential curcuminoids, the anti-cancer agent by preventing DNA topoisomerase II activity, can be predicted by molecular docking method. The molecular docking results that Asp479, Arg503, Gly478
, Gln778, DA12, DG13, DT9, DC8 and Met782 have essential residues to for Pi bond (π), hydrogen bond, and another electrostatic interaction to protein reseptor. Pi bonds (π) have been formed By intercalation mode through C-siclic in A benzena ring from curcuminoids compound to base of DNA (DA12, DG13, DT9, DC8), hydrogen bonds have been formed by Asp479 (OH2 and OH3 groups from curcuminoids), hidrofobic interations have been formed by Met782 residue and another electrostatic interaction has been formed by Asp479, Arg 503, Gln778 and Glu477. Curcuminoids compound that consist of curcumin, demetoxycurcumin and bisdemetoxycurcumin have stable free Gibbs energy, -10.1, -9, -8.9 kcal/mol, respectively so its potential as inhibitor DNA topoisomerase II enzyme.
vi
© Hak Cipta Milik IPB, Tahun 2015
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada
Program Studi Biokimia
STUDI MOLECULAR DOCKING EKSTRAK KURKUMINOID
ASAL WONOGIRI SEBAGAI INHIBITOR ENZIM
DNA TOPOISOMERASE II
TIRTA SETIAWAN
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
viii
Judul Tesis : Studi Molecular Docking Ekstrak Kurkuminoid asal Wonogiri sebagai Inhibitor Enzim DNA Topoisomerase II
Nama : Tirta Setiawan NIM : G851130101
Disetujui oleh Komisi Pembimbing
Dr Laksmi Ambarsari MS
Ketua Dr Tony Ibnu Sumaryada MSi Anggota
Diketahui oleh
Ketua Program Studi Biokimia
Prof Dr Drh Maria Bintang MS
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah MScAgr
PRAKATA
Puji dan syukur penulis panjatkan ke hadirat Allah SWT atas segala rahmat, berkah, dan karunia-Nya sehingga penulis dapat menyelesaikan tesis ini. Penelitian ini berjudul: Studi Molecular Docking Ekstrak Kurkuminoid asal Wonogiri sebagai Inhibitor Enzim DNA Topoisomerase II. Penelitian ini merupakan salah satu persyaratan dalam menyelesaikan studi di Program Pascasarjana Institut Pertanian Bogor.
Pada kesempatan ini penulis mengucapkan terima kasih yang sebesar-besarnya kepada Dr. Laksmi Ambarsari, MS dan Dr. Tony Ibnu Sumaryada, M.Si sebagai komisi pembimbing yang banyak memberi bimbingan dan arahan kepada penulis dalam menyelesaikan penelitian dan karya penelitian ini. Dr. Suryani sebagai penguji luar komisi atas saran dan masukan bagi penulisan tesis ini.
Akhirnya, semoga karya ilmiah ini dapat memberikan manfaat bagi penulis, civitas akademika, peneliti, pemerintah dan semua pihak yang terkait, sehingga mampu memperkaya khasanah keilmuan di masa mendatang.
Bogor, Oktober 2015
xii
DAFTAR TABEL
1. Filter Lipinski 7
2. Asam Amino yang Berinteraksi 12 3. Analisis Ikatan Hidrogen 20
DAFTAR GAMBAR
1. Visualisasi Struktur 3D Reseptor 6
2. Plot Ramachandran Reseptor 7
3. Validasi Metode 8
4. Energi Bebas Gibbs 9
5. Ikatan Pi (π) 9
6. Mode Ikatan 10
7. Interaksi Etoposida Jarak 5 Å 10
8. Interaksi Kurkumin Jarak 5 Å 11
9. Interaksi Demetoksikurkumin dan Bisdemetoksikurkumin Jarak 5 Å 12
10.Gugus Fungsi 22
DAFTAR LAMPIRAN
1. Diagram Alir Penelitian 31
2. Struktur Lengkap Enzim DNA Topoisomerase II 32
3. Posisi Sekuen Central Terminal Topoisomerase II dan Sekuen Domain
Toprim serta 5y-cap 33
4. Plot Ramachandran 34
5. Struktur Ligan 35
6. Parameter Penambatan 35
1 PENDAHULUAN
Latar Belakang
Temulawak (Curcuma Xanthorriza Roxb) adalah tanaman yang telah turun-temurun dimanfaatkan oleh masyarakat Indonesia sebagai minuman herbal atau jamu. Komponen yang terkandung dalam temulawak adalah kurkuminoid yaitu kurkumin, desmetoksikurkumin dan bisdesmetoksikurkumin, dengan kadar tertingginya adalah kurkumin diikuti desmetoksikurkumin dan bisdemetoksikurkumin (Maulina D & Pemasku G 2014, Anad et al. 2008, Kumar & Bora 2011). Kurkuminoid diketahui memiliki aktifitas farmakologi yang luas seperti antikanker, antiinflamasi, antioksidan, antimikroba, immunomodulator dan antidiabetes (Lin et al. 2007).
Kadar senyawa kurkuminoid dari satu daerah dengan daerah lain berbeda. Hal ini karena perbedaan faktor hara dan iklim pada daerah tersebut. Temulawak yang ditanam di Wonogiri memiliki kadar kurkuminoid tertinggi dibandingkan dengan temulawak yang ditanam di daerah Ciemas, Sukabumi dan Balitro (Pemasku G 2014). Data pada penelitian terbaru menunjukkan bahwa sediaan ekstrak kurkuminoid asal Wonogiri memiliki aktifitas antiinflamasi dan meningkat aktivitasnya ketika digunakan dalam bentuk sediaan nanopartikel (Maulina D 2014). Kasus Inflamasi kronis berkaitan dengan fase awal yang menyebabkan pertumbuhan sel kanker, adanya aktifitas antiinflamasi memberikan arti bahwa senyawa kurkuminoid asal Wonogiri juga dapat menekan laju pertumbuhan sel kanker.
Kanker merupakan penyakit yang terjadi karena pembelahan sel tidak terkontrol dan mampu menyerang sel-sel pada jaringan atau organ biologis lainnya (Lavery et al. 2010; Zhuang et al. 2008). Penelitian terbaru melaporkan bahwa hampir semua jenis kanker yang ada mengalami mutasi pada gen-gen penyandi protein supressor atau protein onkogen yang berfungsi sebagai pengatur jalannya pembelahan sel (Sakoparnig et al. 2015).
Enzim DNA topoisomerase II merupakan salah satu enzim pada proses pembelahan sel eukariot yang berfungsi mengkatalisis reaksi perubahan topologi pada DNA, termasuk relaksasi dari superkoil, katenasi atau dekatenasi dan knotting/unknotting (Bates AD et al. 1997). Kemampuannya dalam memodulasi topologi DNA membuat enzim DNA topoisomerase II memegang peran penting dalam proses replikasi, transkripsi dan penguraian/pembentukan koromosom (Wang et al. 2002; Nitis et al. 2009).
Enzim DNA topoisomerase II diketahui memiliki tingkat ekspresi yang tinggi pada semua sel-sel kanker dan paling tinggi ekspresinya pada kanker hati (Hep27) dan kanker payudara (Mcf-7) (Abu SA & Zihlif 2012). Meningkatnya level ekspresi dari enzim DNA topoisomerase II dijadikan observasi awal untuk penyakit kanker (Nitis JL et al. 1998; Fortune JM et al. 2000).
2
Osheroff et al.1989; Beck et al.1993). Akibatnya, DNA akan terpotong secara permanen yang kemudian akan mengaktifkan sinyal kematian sel terkontrol (apoptosis) melalui jalur kerusakan DNA (DNA damage). (Pommier Y et al. 1991).
Kemampuan senyawa kurkumin sebagai agen potensial antikanker telah diketahui dengan baik. Dalam upaya pengembangan kurkumin sebagai obat antikanker, telah banyak dilakukan modifikasi melalui sintesis organik dari senyawa kurkumin agar didapatkan senyawa turunan kurkumin baru yang lebih stabil dan aktifitas yang lebih efektif terhadap protein target (Fitriasari et al. 2008). Namun senyawa turunan kurkumin yang diperoleh dengan sistesis organik sering memberikan permasalahan pada sistem metabolisme dari pada senyawa yang disintesis secara alamiah. Desmetoksikurkumin dan bisdemetoksikurkumin adalah senyawa turunan kurkumin alami yang diprediksikan memiliki aktivitas yang sama dengan kurkumin sehingga perlu dilakukan penelitian mengenai potensinya sebagai anti-kanker.
Penelitian ini dirancang untuk mempelajari potensi ekstrak kurkuminoid asal wonogiri sebagai kandidat inhibitor enzim DNA topoisomerase II berdasarkan afinitas dan interaksi ligan yang terjadi melalui teknik penambatan molekular.
Metode penambatan molekular (molecular docking) adalah metode komputasi (in silico) yang menggunakan kombinasi bahasa pemrograman komputer untuk mensimulasi interaksi antara molekul kecil yang fleksibel (ligand) dan suatu molekul besar yang tetap (receptor) (Morris et al. 1998). Metode penambatan molekular menghasilkan prediksi mengenai detail koordinat dan konformasi molekul kecil yang kemudian dapat digunakan sebagai titik awal studi secara teoritis bagi molekul kecil tersebut untuk dilakukan uji pada tahap selanjutnya seperti in vitro dan in vivo (Yeturu & Nagasuma 2008).
Studi senyawa kurkumin menggunakan metode penambatan molekular telah dilakukan diantaranya studi pembuktikan potensi kurkumin sebagai kandidat obat pada pengobatan kanker payudara dengan menghambat aktifitas hormon progesteron (Pebriana et al. 2012), potensi kurkumin sebagai kandidat SARMS kanker prostat (Arwansyah 2014). Perangkat lunak yang digunakan untuk proses simulasi pada penelitian ini adalah Autodock Vina yang memiliki efektifitas dan akurasi yang tinggi serta memiliki kemampuan untuk mencari konformasi energi terendah dalam keadaan interaksi optimum pada protein-ligan dengan struktur yang hampir sama dengan keadaan struktur pada penelitian in vivo (Hetenyi et al. 2002).
Rumusan Masalah
Tujuan
Penelitian ini bertujuan untuk mempelajari potensi ekstrak kurkuminoid asal wonogiri sebagai kandidat agen inhibitor enzim DNA topoisomerase II melalui metode penambatan molekular in silico. Parameter yang diamati meliputi nilai energi ikatan (∆G), ikatan Pi (π), ikatan hidrogen, mode ikatan, interaksi hidrofobik, interaksi elektrostatik dan gugus samping kurkuminoid yang berinteraksi.
Manfaat
4
2 METODE
Alat dan Bahan
Bahan yang digunakan adalah struktur dua dimensi dari ligan uji kurkuminoid yaitu kurkumin ((1E,6E) - 1, 7 - Bis (4 - hydroxy - 3 - methoxyphenyl) - 1, 6 - heptadiene - 3, 5 - dione), demetoksikurkumin ((1E, 6E) - 1 - (4 - Hydroxy - 3 - methoxyphenyl) - 7 - (4 - hydroxyphenyl) hepta - 1, 6 - diene - 3, 5 - dione), bisdemetoksikurkumin ((1E,6E)- 1, 7 - Bis (4 - hydroxyphenyl) hepta - 1, 6 - diene - 3, 5 - dione) dan ligan pembanding senyawa etoposida (4'-Demethyl-epipodophyllotoxin 9 - [4, 6 - O - (R) - ethylidene - beta - D - glucopyranoside], 4' - (dihydrogen phosphate)) yang di dapat secara online pada database http://www.drugbank.ca. Sedangkan file makromolekul yang dipilih sebagai reseptor adalah enzim DNA topoisomerase II dengan kode 3QX3 yang dapat diunduh pada database Protein Data Bank (PDB) di situs http://www.rscb.org. Alat yang digunakan terdiri dari perangkat keras dan perangkat lunak. Perangkat keras terdiri atas komputer dengan spesifikasi laptop Hp Pavillion dv2 (Amd Athlon neo dual core, 2GB RAM ddr2, 250 seagate HDD, dan VGA ati radeon x1200). Perangkat lunak yang digunakan terdiri atas Autodock Vina, ADT 1.5.6 (The Scripps Research Institute, Amerika), VMD 1.9.2, Discovery SV 3.5, LigPlot+ 4.5.3 (Rowan Lawkowski), OS. Ubuntu 14.04 lts.
Prosedur Penelitian
Identifikasi Reseptor
Protein yang digunakan sebagai reseptor adalah enzim DNA topoisomerase II (kode PDB: 3qx3) yang diperoleh secara online melalui situs http://www.rscb.org/pdb (Gambar 1). Proses identifikasi dilakukan dengan mengunakan perangkat lunak VMD 1.9.2 dan Discovery Studio Visualizer 4.0 berdasarkan infromasi yang didapat dari data kristalisasi enzim DNA topoisomerase II berkode 3qx3 yang dilakukan oleh Wu CC et al. (2011).
Optimasi Geometri Struktur 3D Reseptor
Reseptor berkode 3qx3 tersebut memuat koordinat semua atom residu, faktor struktur kristalografi dan data eksperimen NMR dari enzim DNA topoisomerase II sehingga dapat menentukan kualitas struktur tiga dimensi protein. Kestabilan struktrur dapat dilihat dengan VMD 1.9.2 melalui plot Ramachandran.
Optimasi Geometri Struktur 3D Ligan
Ligan yang akan digunakan yaitu kurkumin, demetoksikurkumin dan bisdemetoksikurkumin dirancang menggunakan perangkat lunak Marvin sketch dengan format penyimpanan PDB (Lampiran 5). Proses optimasi dilakukan menggunakan ADT 1.5.6. Tahap awal adalah dilakukan merged nonpolar hidrogen memberikan muatan Gasteiger dan semua file disimpan dalam format PDBQT. Seluruh senyawa ligan ini dilakukan filter menurut aturan Lipinski menggunakan online access http://www.scfbio-iitd.res.in/ software/ utility/ lipinskifilters.jsp (Lipinski 2009).
Proses Penambatan Molekular
Proses Grid dan validasi parameter penambatan dilakukan dengan ADT 1.5.6, penambatan molekular dilakukan dengan AutoDock Vina (Scripps Research Institute, USA) dan diasumsikan semua rotatable bond (ikatan siklik) dari ligan dapat berotasi (fleksibel) dan reseptor adalah tetap (rigid) (Morris et al.1998). Parameter berikut adalah parameter yang telah divalidasi. Ukuran grid box yang dipilih adalah 80 x 80 x 80 Å dengan spacing centered 0.375 Å pada reseptor, center_x = 32.884, center_y = 95.413, center_z = 50.785 dan luas box melingkupi keseluruhan struktur reseptor. Exaustiveness di atur pada angka 100. Folder vina diletakkan pada drive C:\Vina kemudian di isikan file CONF.TXT dengan parameter diatas sesuai dengan angka center dan size nya.
Eksekusi perintah penambatan menggunakan perintah melalui jendela CMD, panggil Vina.exe lalu ketik perintah “C:\vina --config conf.txt --log log.txt”, lalu tekan enter dan tunggu proses sampai selesai. Kurkuminoid dalam yang memiliki energi bebas Gibbs (∆G) terkecil dari daftar disimpan dalam
format PDBQT (ADT 1.5.6) dan dikonversi ke format PDB dengan DSV 4.0. Prosedur lengkap proses penambatan dapat dilihat pada Lampiran 7.
Analisis
6
3 HASIL
Identifikasi Reseptor
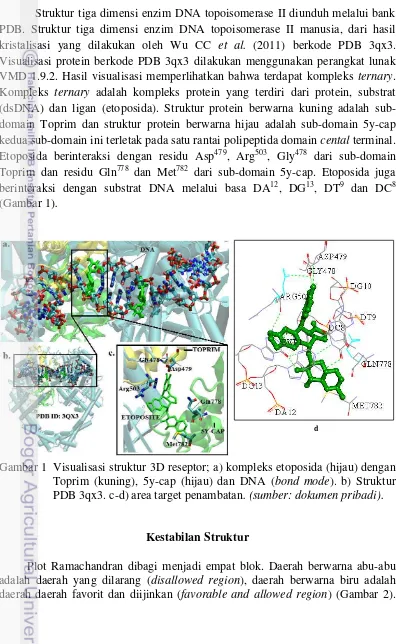
Struktur tiga dimensi enzim DNA topoisomerase II diunduh melalui bank PDB. Struktur tiga dimensi enzim DNA topoisomerase II manusia, dari hasil kristalisasi yang dilakukan oleh Wu CC et al. (2011) berkode PDB 3qx3. Visualisasi protein berkode PDB 3qx3 dilakukan menggunakan perangkat lunak VMD 1.9.2. Hasil visualisasi memperlihatkan bahwa terdapat kompleks ternary. Kompleks ternary adalah kompleks protein yang terdiri dari protein, substrat (dsDNA) dan ligan (etoposida). Struktur protein berwarna kuning adalah sub-domain Toprim dan struktur protein berwarna hijau adalah sub-sub-domain 5y-cap kedua sub-domain ini terletak pada satu rantai polipeptida domain cental terminal. Etoposida berinteraksi dengan residu Asp479, Arg503, Gly478 dari sub-domain Toprim dan residu Gln778 dan Met782 dari sub-domain 5y-cap. Etoposida juga berinteraksi dengan substrat DNA melalui basa DA12, DG13, DT9 dan DC8 (Gambar 1).
d
Gambar 1 Visualisasi struktur 3D reseptor; a) kompleks etoposida (hijau) dengan Toprim (kuning), 5y-cap (hijau) dan DNA (bond mode). b) Struktur PDB 3qx3. c-d) area target penambatan. (sumber: dokumen pribadi).
Kestabilan Struktur
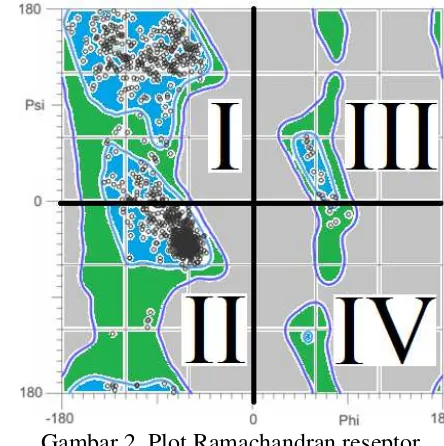
Dari data asam amino yang berada pada daerah paling disukai (favorable) sebesar 98% (1327/1365), daerah yang izinkan (allowed) sebesar 99.9% (1363/1365) dan disallowed region kecil dari 1%. (Lovell et al. 2003).
Gambar 2 Plot Ramachandran reseptor.
Aturan Lipinski
Semua ligan uji (kurkuminoid) memenuhi aturan Lipinski (the rule of five), namun etoposida tidak memenuhi aturan Lipinski (Tabel 1). Berat molekulnya besar dari 500, jumlah gugus penerima hidrogen lebih dari 10 dan molar refraktifiti diatas 130.
Tabel 1 Filter Lipinski
Lipinski’s rule Etoposida* Kurkumin Demetoksi kurkumin Bisdemetoksi kurkumin
A 588 368 338 308
B 1.1059 2.823 2.811 2.799
C 3 3 3 3
D 13 3 2 1
E 137.41 102.965 96.413 89.861
Keterangan:
* : Ligan standar (obat komersial) A : Massa atom relatif< 500
B : Log P < 5
8
Validasi Metode Penambatan
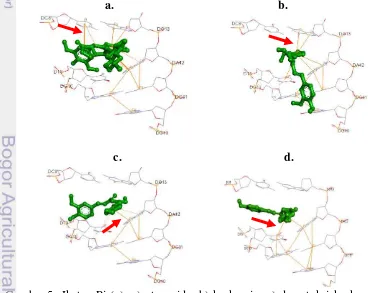
Hasil validasi metode penambatan menunjukkan nilai RMSD yang sangat kecil yaitu 0.06Å. Selain itu etoposida berinteraksi dengan asam amino yang sama dengan sebelum ligan tersebut ditambatkan ulang (redocking) yaitu Asp479, Arg503, Gly478
, Gln778, DA12, DG13, DT9, DC8 dan Met782 (Gambar 3a-3b). hasil penambatan memperlihatkan posisi ligan yang saling tumpang tindih (Gambar 3c) dan memiliki ikatan Pi dan ikatan hidrogen dengan residu Asp479 (Gambar 3a-b, 3d).
a. b.
c. d.
RMSD = 0.06
Gambar 3 Validasi metode; a) interaksi etoposida dengan asam amino pada daerah 5 Å. b) hasil validasi (self docking) dan interaksi etoposida dengan asam amino pada daerah 5 Å, c) mode ikatan etoposida terhadap DNA dan d) ikatan Pi pada etoposida. (sumber: dokumen pribadi)
Energi Ikatan (Energi bebas Gibbs)
Gambar 4 Energi bebas Gibbs.
Ikatan Pi (π)
10
Mode Ikatan
Ligan divisualisasikan dengan warna kuning, DNA di visualisasikan dengan lekukan berwarna putih sebagai backbone, basa nukleotida divisualisasikan dengan bentuk batang yang memiliki 4 warna, merah (adenin), biru (timin), ungu (sitosin) dan hijau (guanin) (Gambar 6). Pada salah satu rantai terdapat celah (gap) antara sitosin (DC8) dan timin (DT9). Etoposida memiliki nilai RMSD 0.06, kurkumin 2.491, demetoksi kurkumin 1.402, bisdemetoksi kurkumin 1.184.
a. b. c. d.
0.06 Å 2.491 Å 1.402 Å 1.184 Å
Gambar 6 Mode ikatan (Binding mode); a) etoposida. b) kurkumin. c) demetoksi kurkumin. d) bisdemetoksi kurkumin.
Ikatan Hidrogen
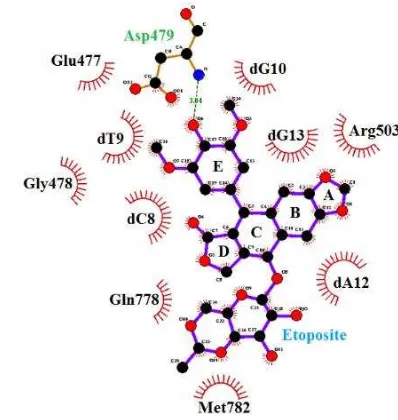
Dari hasil penambatan molekuler, etoposida berinteraksi dengan reseptor melalui residu Asp479, tampak terdapat ikatan hidrogen berjumlah satu dengan jarak donor-akseptor 3.04 Ǻ antara gugus (NH2) dari residu Asp479 terhadap gugus (OH4) dari etoposida (Gambar 7). Jarak ikatan ini termasuk dalam kategori yang memiliki energi ikatan medium.
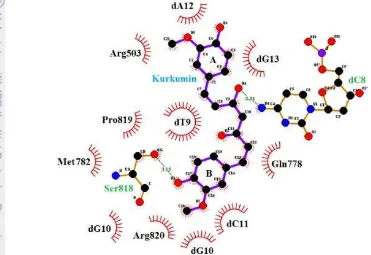
Senyawa kurkumin berinteraksi dengan reseptor melalui dua ikatan hidrogen. Atom N (biru) yang terikat pada atom C ke kempat residu DC8 berinteraksi dengan gugus OH4 pada senyawa kurkuminoid dengan jarak ikatan
3.34 Ǻ, jarak ikatan ini tergolong dalam ikatan hidrogen dengan kekuatan ikatan lemah (weak). Selanjutnya satu atom O (merah) pada residu Ser818 berinteraksi dengan satu tempat atom OH3 (merah) pada senyawa kurkumin membentuk ikatan
hidrogen dengan jarak ikatan 3.15 Ǻ (Gambar 8), jarak ikatan ini tergolong dalam ikatan hidrogen dengan kekuatan medium.
Gambar 8 Interaksi kurkumin jarak 5 Ǻ
Senyawa demetoksikurkumin dan bisdemetoksikurkumin sama-sama memiliki jumlah ikatan hidrogen dan interaksi yang sama yaitu tiga ikatan hidrogen antara atom O (merah), N (biru) pada residu Asp279 dan satu ikatan hidrogen antara atom O (merah) gugus gula pentosa pada residu DT9, ketiganya berinteraksi dengan satu atom O (merah) yang sama pada demetoksi dan bisdemetoksi kurkumin. Pada senyawa demetoksi kurkumin, jarak ikatan antara atom O dan N pada residu Asp479 dengan atom OH3 pada senyawa demetoksi kurkumin berturut-turut adalah 3.26 Ǻ dan 2.80 Ǻ dengan kekuatan ikatan lemah (weak) dan medium, sedangkan jarak ikatan antara atom O pada residu DT9 dengan atom OH3 pada senyawa demetoksi kurkumin adalah 3.13 Ǻ, termasuk kedalam kategori yang memiliki kekuatan ikatan hidrogen medium (Gambar 9). Pada senyawa bisdemetoksi kurkumin, jarak ikatan antara atom O dan N pada residu Asp479 dengan atom OH2 pada senyawa bisdemetoksi kurkumin
berturut-turut adalah 3.17 Ǻ dan 2.82 Ǻ dengan kekuatan ikatan medium, sedangkan jarak ikatan antara atom O pada residu DT9 dengan atom OH2 pada senyawa
bisdemetoksi kurkumin adalah 3.12 Ǻ, termasuk kedalam kategori yang memiliki
12
Gambar 9 Interaksi demetoksikurkumin dan bisdemetoksikurkumin jarak 5 Ǻ.
Asam Amino yang Berinteraksi
Ligan pembanding (etoposida) dan ligan uji (kurkuminoid) berinteraksi dengan berbagai jenis residu yang berada pada reseptor (Tabel 2). Semua ligan membentuk interaksi dengan residu hidrofobik Met782, residu polar bermuatan Arg503, residu polar tak bermuatan Gln778, basa DT9, DA12, DG13 dan DG10.
Tabel 2 Asam amino yang berinteraksi.
4 PEMBAHASAN
Identifikasi Reseptor
Enzim DNA topoisomerase II berkode PDB 3qx3 merupakan reseptor yang digunakan pada penelitin ini dan enzim ini berasal dari manusia. Secara umum struktur DNA topoisomerase II manusia terbagi menjadi tiga domain besar (Lampiran 2) yaitu N-terminal, Central terminal/DNA-gate dan C-terminal (Berger JM et al. 2004). DNA topoisomerase II berkode 3qx3 hanya terdiri dari domain Central terminal/DNA-gate dan C-terminal (Wu CC et al. 2011). Terdapat juga senyawa etoposida pada kompleks yang diketahui merupakan inhibitor komersial untuk kemoterapi antikanker (Gambar 1). Area yang akan dijadikan target penambatan berlokasi diantara sub-domain Toprim dan 5y-cap yang berada di dalam domain center terminal/DNA-gate (Gambar 1).
Sub-domain Toprim dan 5y-cap merupakan komponen penyusun spesifik enzim DNA topoisomerase II yang berfungsi mengkatalisis reaksi pemutusan rantai fosfat pada dsDNA dan bertindak sebagai pembuka gerbang untuk dsDNA agar dapat melewatinya sehingga terjadi perubahan topologi DNA. Sub-domain Toprim (Lampiran 3) berada pada posisi residu 455-572 (Beger JM et al. 1996; Aravind L et al. 1998) dan domain 5Y-CAP berada pada posisi residu 711-841 (Champoux JJ et al. 2001; Fass D et al. 1999).
Domain Toprim dan 5y-cap diketahui dimiliki oleh tiga jenis enzim DNA topoisomerase yaitu IA, IIA dan IIB (Aravind L et al. & Berger et al. 1998) dan dua domain tersebut berada pada satu rantai polipeptida pada bagian Central terminal/DNA-gate enzim DNA topoisomerase IA, IIA dan IIIA (Wendorf TJ et al. 2012). Domain Toprim (Lampiran 2) disebut juga topoisomerase-primase hal ini karena struktur pada domain ini mirip dengan struktur primase bakteri dan beberapa enzim nuclease (Aravind L et al. 1998). Ciri khas dari domain ini adalah
terdapat empat sampai lima struktur β-sheet dengan α -heliks terselip di antara dua
β-sheet yang tersusun secara sejajar, antara β-sheet dengan α-heliks dihubungkan dengan Rosmann fold. Selain itu domain ini juga memiliki satu struktur loop yang tersusun dari tiga sampai lima conserved residu asam (polar) asam aspartat (D) dan asam glutamat (E), ketiga residu asam ini diketahui berikatan dengan ion Mg2+ yang berada ada di rantai DNA sehingga fungsi dari residu asam tersebut adalah memegang segmen-G DNA (Aravind L et al. 1998; Bahng S et al. 2000; Lima CD et al. 1994; Osheroff N et al. 1987).
Domain 5y-cap (Lampiran 2) disebut dengan banyak istilah, yaitu CAP-like atau catabolite activator protein karena struktur nya yang mirip dengan struktur CAP pada E.coli (Brenan RG et al. 1989; Clark KL et al. 1993; Harrison SC et al. 1990; Schultz SC et al. 1991), ada juga yang menamainya dengan WHD (winged helix domain) hal ini karena residu aktif tirosin (Y) berada pada daerah tersebut (Brenan RG et al. 1989), namun secara umum domain ini disebut dengan 5y-cap (Catabolite Activator Protein in Tirosin bind and cut 5’). Ciri khusus dari domain ini adalah terdapatnya residu tirosin (Y) yang menyerang ikatan fosfodiester pada
14
langsung melakukan aktifitas pemotongan seperti tirosin, namun residu Arginin
berperan membentuk ikatan hidrogen dengan 5’ DNA sehingga menggurangi
kegagalan pemutusan yang berakibat pada mutasi DNA (Schmidt B et al. 2010). Tiga residu asam dan satu ion Mg2+ dari domain Toprim kemudian residu aktif tirosin (Y) dan arginine (R) dari domain 5y-cap akan terlokalisasi pada satu titik sesaat sebelum proses katalisis berlangsung (Changela A et al. 2001).
Enzim DNA topoisomerase II memperbaiki DNA yang mengalami superkoil melalui lima langkah yang di kenal dengan istilah memotong dsDNA. 1) protein menangkap G-segmen dsDNA sebagai substratnya dan membentuk kompleks non-kovalen DNA-Enzim yang distabilkan dengan ikatan hidrogen dan interaksi elektrostatik; 2) residu katalitik tirosin (Y) dibantu residu arginin (R) membawa nukleofilik yang menyerang satu strand dari dsDNA membentuk
kompleks kovalen protein DNA pada 5’, ion Mg2+ dibantu dengan tiga residu asam membentuk kompleks kovalen dengan satu strand dari dsDNA lainnya pada
3’; 3) DNA yang terpotong berotasi membentuk celah untuk melewatkan T-segmen dsDNA dibantu dengan sub-domain linker untuk merelaksasi superkoil; 4) dsDNA yang tepotong disambung kembali (religasi); 5) enzim melepas substrat DNA (Stewart I et al. 1998).
Kestabilan Konformasi Struktur 3D Reseptor
Penentuan struktur 3D pada reseptor berkode 3qx3 telah dilakukan dengan metode kristalografi sinar-X (X-ray cristallography) dan juga metode NMR (Nuclear Magnetic Resonance) (Wu CC et al. 2011). Kombinasi dari kedua metode tersebut dapat mempresentasikan aktivitas, stabilitas, fungsi dan mampu memberika informasi struktural tingkat atom dari protein pada keadaan unfolding yang penting dalam karakterisasi proses pelipatan protein.
Konformasi residu pada protein dapat ditentukan dengan melihat diagram pada setiap sudut dihedral dari masing-masing residu, diagram ini disebut dengan plot Ramachandran. Plot Ramachandaran digunakan untuk mengetahui kualitas struktur tiga dimensi protein hasil dari kristalisasi karena plot Ramachandran digunakan dalam visualisasi koordinat tiga dimensi protein yang telah ditentukan melalui eksperimen kedalam koordinat internal. Kluster yang terbentuk oleh residu dapat menunjukkan bentuk struktur sekunder protein tersebut. Setiap asam amino yang menyusun protein akan memiliki satu sudut phi (ɸ) dan psi (ψ),
sehingga setiap residu asam amino dapat digambarkan sebagai satu koordinat (plot). Plot-plot yang menggambarkan residu asam amino pada struktur protein inilah yang disebut Ramachandran Plot (Baxevanis & Ouellette 2001).
Selain plot Ramachandran, kualitas struktur protein dapat ditentukan dengan resolusi X-ray protein. Resolusi protein mengindikasikan terhadap detail tiap-tiap atom pada tiap residu pada saat dikristalisasi, resolusi dilambangkan dengan Angstrong (Å), semakin kecil angka resolusi maka akan semakin bagus dan besar resolusi yang dihasilkan hal semacam ini disebut dengan high resolution. Resolusi protein dikatakan bagus jika memiliki angka kecil dari 2.5 Å (Lu HM et al. 2009). Berdasarkan data kristalisasi protein, protein reseptor berkode 3qx3 memiliki resolusi kecil dari 2.5 Å yaitu 2.16 Å.
Ligan yang Memenuhi Aturan Lipinski
Obat yang diberikan pada pasien akan melintasi tubuh berdasarkan berbagai faktor yang dapat menghilangkan, merusak atau mencegahnya mencapai sisi target yang diinginkan. Empat faktor tersebut yaitu absorbsi, distribusi, metabolisme dan ekskresi, dalam farmakologi disebut farmakokinetika atau dipandang sebagai apa yang dilakukan tubuh terhadap obat (Overgarrd 2006). Sangat penting mempertimbangkan aspek farmakokinetika selama merancang suatu obat karena tidak mungkin obat bisa berinteraksi dengan target bila ia tidak bisa mencapai targetnya sehingga ditemukanlah aturan, dimana obat yang mampu mencapai target bila diberikan oral harus memenuhi syarat berikut: berat molekul kurang dari 500, jumlah gugus donor ikatan hidrogen tidak lebih dari 5, jumlah gugus penerima ikatan hidrogen tidak lebih dari 10, nilai log P kurang dari +5 dan refraktifiti molar 40-130. Kelima aturan tersebut dikenal dengan nama the rule of five atau Lipinski rule. Jika obat tidak memenuhi salah satu dari kelima aturan diatas, maka obat tersebut tidak efektif digunakan secara oral dan disarankan melalui ijeksi (Lipinski CA 2001). Filter Lipinski dilakukan dengan menggunakan tools online pada situs http://www.scfbio-iitd.res.in/ software/utility /lipinski filters.jsp.
16
Nilai Mr (berat molekul), H-donor dan H-acceptor merupakan besaran yang sangat menentukan permeabilitas suatu obat untuk dapat melewati lapisan bilayer dari sel. Nilai log P berhubungan dengan hidrofobisitas molekul obat, semakin besar nilai log P maka akan semakin hidrofobik molekul tersebut. Didalam tubuh senyawa ligan sebagai molekul obat tidak boleh terlalu hidrofobik karena dapat tertahan di lapisan lipid bilayer, oleh karena itu obat akan tertahan terlalu lama dan terdistrbusi secara luas didalam tubuh sehingga menyebabkan selektifitas ikatan terhadap protein target menjadi berkurang. Nilai log P yang terlalu negatif (kecil) juga tidak disarankan karena jika obat terlalu hidrofilik maka obat tidak mampu melewati lapisan lipid bilayer (Lipinski CA 2001; Etkins et al. 2005).
Validasi Metode Penambatan
Metode penambatan molekular adalah salah satu teknik in silico untuk mencari posisi penambatan dari struktur target (reseptor) dengan menggunakan bahasa pemprograman dari piranti lunak. Dalam molecular modelling, proses penambatan molekular adalah metode untuk memprediksikan konfigurasi ikatan yang lebih disukai antara ligan yang berukuran kecil-sedang (flexible) dengan target makromolekulnya yang rigid, yang biasa berupa protein. Penambatan molekuler dapat memprediksikan pengikatan kompleks yang terbentuk antara reseptor dengan ligan menggunakan skor seperti ΔG dan interaksi yang terjadi antara ligan dan protein (Yeturu dan Nagasuma 2008).
Validasi metode penambatan bertujuan untuk mengkalibrasi dan memvalidasi parameter penambatan molekular (Baxevanis AD & Oullette BF 2001) yang akan digunakan oleh software Autodock Vina. Parameter dikatakan valid jika parameter penambatan yang kita desain dapat menambatkan ligan alami atau kompleks ligan bawaan ke posisi semula.
Validasi metode penambatan dilakukan dengan cara menambatkan ulang etoposida terhadap enzim DNA topoisomerase II (reseptor) dengan parameter yaitu center_x = 32.884, center_y = 95.413, center_z = 50.785 namun ukuran grid box mencakup keseluruhan molekul (blind) yaitu size_x = 80, size_y = 80, size_z = 80 dan exhaustiveness = 100. Hasil validasi metode penambatan dapat dilihat pada Gambar 3.
Kualitas posisi ligan hasil penambatan sangat mempengaruhi jauh atau dekat perubahan atau konformasi suatu molekul. Perubahan posisi ini idealnya berjarak kurang dari 2.5 Å. Nilai RMSD kurang dari 2.5 Å memberikan indikasi bahwa suatu senyawa bersifat kompetitif inhibitor (Baxevanis & Oulette 2001). Apabila sebuah molekul memiliki nilai RMSD besar dari 2.5 Ǻ, menandakan
bahwa terjadi pergeseran molekul yang jauh. Pergesaran molekul ini dapat disebabkan oleh beberapa hal, yang pertama kegagalan dalam scoring yaitu energi tidak sanggup untuk berikatan dengan senyawa yang memiliki nilai RMSD rendah selain itu juga dapat disebabkan karena program yang tidak sanggup melakukan proses pada beberapa posisi ligan (Baber et al. 2009).
2009). Selain itu etoposida berinteraksi dengan asam amino yang sama pada reseptor sebelum ligan tersebut di tambatkan ulang (redocking) yaitu Asp479, Arg503, Gly478
, Gln778, DA12, DG13, DT9, DC8 dan Met782.
Dari hasil penambatan ulang (self docking) etoposida dengan reseptor berkode pdb 3qx3 terlihat bahwa parameter yang digunakan telah valid sehingga parameter ini dapat digunakan untuk penambatan molekuler ligan pembanding dan ligan uji.
Energi Bebas Gibbs (ΔGbinding)
Energi bebas Gibbs (ΔGbinding) merupakan parameter kestabilan konformasi antara ligan dengan reseptor. Secara termodinamika reaksi-reaksi metabolisme dalam tubuh berlangsung secara eksergonik dan endergonik. Reaksi eksergonik adalah reaksi yang menghasilkan energi bebas Gibbs, yaitu energi yang digunakan untuk melakukan kerja pada temperatur dan tekanan yang tetap. Reaksi eksergonik menyebabkan energi bebas molekul pereaksi menjadi turun, karena energi bebasnya dibebaskan pada saat reaksi (Nelson et al. 2008). Oleh karena itu, energi bebas produk menjadi lebih rendah dibanding energi bebas pereaksi. Semakin rendah energi bebas suatu molekul, maka molekul tersebut semakin stabil dan reaksi berjalan secara spontan. Inilah yang disebut kesetimbangan termodinamika, semakin negatif energi bebas maka reaksi akan semakin spontan atau akan cepat membentuk konformasi yang stabil (Nelson et al. 2008).
Terdapat hubungan antara nilai energi bebas ikatan (∆Gbinding) dengan konstanta inhibitor (Ki) yang nilainya mengikuti persamaan termodinamika berikut (Kitchen et al. 2004):
ikatan kompleks enzim-ligan akan semakin kuat karena kestabilan dan kekuatan interaksi nonkovalen pada kompleks enzim-ligan dapat dilihat dari besarnya energi bebas yang dilepaskan saat interaksi pada kompleks enzim-ligan terbentuk.
Analisis energi bebas ikatan (∆G) hasil penambatan molekular untuk senyawa kurkumin, demetoksi kurkumin dan bisdemetoksi kurkumin (Gambar 4) menghasilkan nilai -10.1, -9 dan -8.9 kkal/mol sedangkan etoposida memiliki nilai
18
ligan pembanding memiliki kestabilan yang lebih baik dari ligan uji. Namun nilai energi ikatan yang terjadi pada ligan uji bernilai negatif, artinya senyawa juga berhasil membentuk ikatan dengan DNA topoisomerase II namun tidak se-stabil etoposida. Data ini menunjukkan bahwa ligan uji kurkuminoid berpotensi sebagai inhibitor enzim DNA topoisomerase II.
Perbedaan nilai energi bebas Gibbs yang dihasilkan baik pada ligan uji kurkuminoid dan ligan etoposida dikarenakan oleh perbedaan karakteristik interaksi yang terjadi antara tiap-tiap ligan dengan reseptor. Perangkat lunak Autodock Vina melakukan perhitungan energi bebas Gibbs berdasarkan rumus berikut:
ΔG = ΔGvdw + ΔGhbond + ΔGelec + ΔGconform + ΔGtor + ΔGsol
Ket:
ΔGvdw = energi dari interaksi Van der waals
ΔGhbond = energi dari ikatan hidrogen
ΔGelec = energi dari interaksi elektrostatik
ΔGconfor = energi konformasi
ΔGtor = energi torsi
ΔGsol = energi solvasi (Morris et al.1996, 1998 & Wang et al. 2003) Setelah ligan diposisikan pada area grid box, perhitungan energi ikatan yang paling pertama dihitung sesaat setelah simulasi adalah ΔGvdw, selanjutnya ΔGBond sampai ΔGsol dan terakhir adalah kalkulasi total keluruhan energi bebas. Namun selama proses penambatan, tidak ada menampilkan nilai energi dari masing interaksi, yang ditampilkan pada hasil akhir adalah energi ikatan total. Oleh karena itu perbedaan nilai energi ikatan antara senyawa satu dengan senyawa yang lain dapat terjadi diakibatkan oleh perbedaan kemampuan dan kekuatan tiap senyawa tersebut melakukan tiap-tiap jenis interaksi.
Berdasarkan visualisasi interaksi yang terjadi terhadap ligan hasil dari penambatan, diketahui terdapat jenis jenis interaksi kimia yaitu ikatan Pi (π), ikatan hidrogen, interaksi hidrofobik dan interaksi elektrostatik. Berdasarkan kekuatan energi ikatannya ikatan Pi (π) merupakan jenis ikatan kovalen yang memiliki energi ikatan tertinggi dibandingkan ikatan hidrogen dan interaksi elektrostatik. Perbedaan jumlah ikatan yang terjadi inilah yang menyebabkan nilai energi bebas ikatan setiap ligan yang berbeda juga berbeda.
Ikatan Pi (π)
Ikatan Pi (π) adalah suatu ikatan (kimia) kovalen yang terbentuk antara
sehingga keberadaan ikatan Pi akan meningkatkan stabilitas kompleks dan energi ikatan total senyawa (Hof F & Pinter T 2012).
Dari hasil visualisasi pada Gambar 5, terlihat garis berwarna kuning. Garis tersebut adalah garis yang menunjukkan adanya ikatan Pi dan arah garis menunjukkan dengan struktur yang mana ikatan Pi terbentuk. Banyaknya jumlah interaksi dan residu yang terlibat akan meningkatkan stabilitas kompleks (Jones S et al. 2001 & Xiong Y et al. 2001). Ligan pembanding etoposida memiliki jumlah ikatan terbanyak yaitu tujuh ikatan Pi yang melibatkan tiga basa dari reseptor yaitu DC8, DG13 dan DA12. Sedangkan pada ligan uji, kurkumin memiliki ikatan paling banyak yaitu lima ikatan Pi yang melibatkan tiga basa yaitu DT9, DG13 dan DA12. Ligan uji demetoksi kurkumin memiliki tiga ikatan Pi dengan melibatkan dua basa yaitu DT9 dan DA12. Ligan uji bisdemetoksi kurkumin memiliki dua interaksi ikatan Pi yang melibatkan dua basa yaitu DT9 dan DA12. Oleh karena itu diantara ligan uji, kurkuminlah yang memiliki kestabilan paling tinggi.
Secara umum Ikatan Pi yang terjadi pada ligan uji lebih sedikit dibandingkan dengan ikatan Pi pada ligan pembanding, hal ini disebabkan etoposida memiliki tujuh cincin siklik yang lebih banyak dibandingkan dengan cincin siklik pada kurkuminoid yang berjumlah dua, sehingga kemungkinan terbentuknya ikatan Pi akan menjadi lebih besar. Oleh karena itu tingkat kestabilan ligan pembanding lebih tinggi dari ligan uji. Dari data tersebut, urutan kekuatan kestabilan dari tingkat kestabilan tertinggi sampai terendah yaitu etoposida, kurkumin, demetoksi kurkumin dan bisdemetoksi kurkumin.
Ikatan Pi sangat penting pada desain obat untuk menjelaskan kestabilan ikatan dan mode ikatan interkalasi yang melibatkan interaksi terhadap DNA (Synder RD et al. 2004, Jones S et al. 2001 & Xiong Y et al. 2001). DNA memiliki struktur siklik dan saling tumpang tindih, sehingga memiliki potensi untuk mendisain obat yang membentuk ikatan Pi, seperti mendisain obat yang mampu melakukan interkalasi pada pasangan basa (Miotessier et al. 2008, Grootenhuis et al. 1990 & Hannon 2007).
Ikatan Hidrogen
Ikatan hidrogen adalah gaya tarik antar molekul atau antar dipol-dipol yang terbentuk antara dua muatan parsial dengan polaritas yang berlawanan. Ikatan hidrogen terjadi ketika sebuah atom memberikan ikatan kovalen hidrogennya (donor) kepada atom yang elektronegatif (acceptor), seperti oksigen pada –OH (Ser, Thr, Tyr, karbohidrat), H2O dan Nitrogen pada –NH3+ (Lys, Arg) atau –NH- (banyak ditemukan pada ikatan peptida, Trp, His, Arg, basa pada nukleotida) kesemuanya adalah jenis donor (Jefrey & Mart 1997).
20
residu hasil validasi namun dari hasil visualisasi menunjukkan posisinya berada di sekitar area target penambatan dari reseptor, selain itu nilai RMSD hasil penambatan memperlihatkan kurkumin masih diprediksikan berinteraksi dekat dari area penambatan reseptor. Gugus hidroksil dari cincin benzena B (OH2, OH3) ligan kurkuminoid menunjukkan peran yang sangat penting pada motif pembentukan ikatan hidrogen ini, karena terlihat semua ikatan hidrogen pada ketiga ligan uji berinteraksi pada gugus hidroksil yang sama.
Banyaknya jumlah ikatan hidrogen dan jarak interaksi menentukan kekuatan interaksi, hal ini terlihat dari sifat fisik dan kimia antara H2O dan HF yang jauh berbeda walaupun sama-sama memiliki ikatan hidrogen. H2O memiliki titik didih yang lebih tinggi dari HF hal ini menunjukkan bahwa butuh energi yang lebih tinggi untuk memutuskan ikatan molekul H2O, penjelasannya karena molekul H2O memiliki jumlah ikatan hidrogen dua sedangkan HF satu selain itu Jarak antara atom donor dan akseptor menentukan kekuatan interaksi (stabilitas). (Jefrey & Mart 1997).
Interaksi Hidrofobik dan Elektrostatik
Interaksi hidrofobik juga berperan penting terhadap kestabilan ligan terhadap reseptor. Interaksi hidrofobik merupakan interaksi yang bersifat menghindari lingkungan cair dan cenderung berkelompok di bagian dalam dari struktur globular protein untuk meminimalkan interaksi dengan air yang dapat merusak struktur protein dan menyebabkan enzim kehilangan aktivitasnya (Lin & Brasseur 1995). Dari hasil validasi, Reseptor enzim DNA topoisomerase II memiliki satu residu hidrofobik yaitu Met782 dan semua ligan uji kurkuminoid membentuk interaksi hidrofobik dengan residu tersebut.
Visualisasi interaksi elektrostatik belum mampu dilakukan oleh perangkat lunak yang digunakan pada penelitian. Namun interaksi ini dapat dianalisis melalui residu-residu yang cenderung membentuk interaksi elektrostatik. Interaksi elektrostatik merupakan interaksi antara atom yang disebabkan oleh kepolarannya. Interaksi ini merupakan interaksi yang lemah dan non-kovalen sehingga mudah lepas, namun jumlahnya yang banyak, interaksi ini dapat memberikan pengaruh yang besar terhadap kestabilan (Sharp & Honig 1990). Residu polar bermuatan pada reseptor yaitu Asp479, Arg 503, Gln778 dan Glu477 (Tabel 2). Semua ligan uji kurkuminoid menunjukkan interaksi pada residu tersebut. Adanya kesamaan residu yang terlibat antara etoposida dan kurkuminoid pada area ikatan (binding site) dari reseptor menyebabkan kurkuminoid dapat menghambat aktivitas enzim DNA topoisomerase II dengan berkompetisi secara kompetitif (competitif inhibitor) dengan etoposida. Sesuai dengan data penelitian in vitro yang dilaporkan oleh Saleh E et al. (2012) bahwa kurkumin memiliki aktivitas antagonis terhadap etoposida.
Residu Asp479, Arg503, Gly478
, Gln778, DA12, DG13, DT9, DG10 dan Met782, selalu muncul pada interaksi ligan dengan reseptor sehingga residu-residu tersebut diprediksikan berperan penting pada area ikatan (binding site) pada enzim DNA topoisomerase II (reseptor).
Hubungan Aktifitas dan Gugus Fungsi
Kurkuminoid merupakan senyawa aktif bahan alam memiliki struktur dasar dari kurkumin yang terdiri dari sepasang gugus fenolik yang di hubungkan oleh tujuh rantai karbon alifatik dengan dua gugus keto-enol tautomer (bis-α,β-diketon) pada pusatnya atau dikenal dengan nama pentadienon dan sepasang gugus metoksi yang masing-masing menempel pada gugus fenolik posisi orto (Gambar 10b) (Jovanovic SV et al. 1999). Sedangkan senyawa pembanding etoposida memiliki struktur dasar yang dapat dibagi menjadi tiga posisi yaitu bawah, tengah dan atas, bagian bawah terdiri dari satu gugus fenolik, bagian tengah terdiri dari satu cincin furan dan dua cincin benzen, bagian atas terdapat dua gugus gula pentosa (Gambar 10a) (Pitts SL et al. 2011).
22
Senyawa fenolik terdiri dari sebuah cincin benzena dan satu grup hidroksil. Struktur kimia dari senyawa fenolik memberinya kemampuan untuk menangani masalah radikal bebas. Struktur fenolik dengan struktur alkohol memberikan penemuan bermakna bahwa pada alkohol, gugus samping hidroksil berikatan pada atom karbon jenuh sedangkan pada fenol, gugus hidroksil berikatan pada atom karbon tak jenuh dan ini mengindikasikan bahwa pelapasan ion hidrogen lebih mudah pada bentuk fenol dari pada bentuk alkohol, oleh karena itu membuat sifat dari fenolik lebih asam dari pada alkohol (Clark J 2007).
a. b.
Gambar 10 Gugus fungsi; a) etoposite. b) turunan alami kurkuminoid Perbedaan struktur yang terlihat antara kurkumin dan turunan alaminya dalam kurkuminoid adalah keberadaan gugus metoksi pada cincin fenolnya. Demetoksikurkumin memiliki satu gugus metoksi yang hilang pada salah satu cincin fenolnya dan bisdemetoksikurkumin kehilangan semua gugus metoksi pada kedua cincin fenolnya. Terdapat perbedaan aktifitas yang akan ditimbulkan antara fenol tanpa gugus metoksi dan fenol dengan gugus metoksi. Keberadaan gugus metoksi ini melemahkan ikatan hidrogen pada gugus hidroksil sehingga bertanggung jawab terhadap lepasnya ion H+ dimana ion ini merupakan nukleofil yang dapat menstabilkan senyawa radikal bebas (Sompram et al. 2007). Berarti, hilangnya gugus metoksi akan menyebabkan ion H+ sukar terlepas sehingga mengurangi aktifitasnya. Sehingga senyawa turunan alami yang kehilangan gugus metoksi menyebabkan melemahnya kemampuan antioksidannya. Dibandingkan dengan senyawa pembanding etoposida, gugus fenol juga tampak hadir namun memiliki perbedaan mencolok yaitu gugus fenolnya memiliki dua gugus metoksi pada posisi orto dan para sehingga menyebabkan fenol gugus fenol pada etoposida lebih aktif dari pada fenol pada senyawa kurkumin dan turunannya.
tertentu dapat diatasi oleh sistem reduktase tubuh namun pada kasus kanker, aktifitas enzim DNA topoisomerase II sangat berlebihan menghasilkan ROS yang juga berlebihan dan terlokalisasi pada daerah spesifik yaitu area DNA yang terpotong. Oleh karena itu obat yang didesain untuk menghambat enzim ini, contohnya etoposida, dibuat memiliki aktifitas antioksidan yang tinggi yang bertujuan untuk pengenalan daerah yang akan dihambat yaitu daerah yang kaya akan ROS. Karena etoposida memiliki empat struktur cincin yang kaya atom karbon pada bagian tengah tertarik kuat masuk kedalam celah DNA akibat orbital P dari karbon yang berkemampuan membentuk ikatan kovalen Pi dengan struktur basa yang juga kaya akan atom Pi, sehingga affinitas lebih tertarik untuk membentuk ikatan dengan basa. Pose yang lebih baik ditunjukkan oleh etoposida diakibatkan oleh adanya gugus pentosa yang mampu berinteraksi menyesuaikan dengan gugus gula DNA sehingga memperbaiki pose interkalasinya. Aktifitas antioksidan juga diperlihatkan oleh kurkuminoid karena memiliki gugus fenolik sehingga membuatnya mampu mengenali area target penghambatan. Keberadaan cincin benzen fenil pada kurkuminoid yang kaya akan atom karbon juga mampu membentuk ikatan Pi dengan basa.
Mode Ikatan
Mode ikatan interkalasi adalah mode ikatan yang paling sering menyebabkan kerusakan pada DNA dan biasanya berikatan secara kovalen. Ketika terdapat suatu senyawa kecil berinterkasi kuat dengan DNA secara interkalasi, jalannya replikasi dan transkripsi DNA akan terganggu bahkan dapat menyebabkan kegagalan proses tersebut. Kejadian ini akan memicu aktifnya perbaikan DNA, jika perbaikan gagal maka sel akan di arahkan pada kematian sel terprogram. Inilah tujuan dari pengembangan obat dengan kemampuan interkalasi untuk digunakan sebagai antiproliferasi (antikanker) (Grootenhuis et al. 1990 & Hannon 2007).
Visualisasi mode ikatan (binding mode) ligan hasil penambatan dengan DNA yang terdapat dalam kompleks reseptor berkode 3qx3 dapat dilihat pada Gambar 6. DNA di visualisasikan dengan lekukan berwarna putih sebagai backbone, basa nukleotida divisualisasikan dengan bentuk batang yang memiliki 4 warna, merah (adenin), biru (timin), ungu (sitosin) dan hijau (guanin). Pada salah satu rantai terdapat celah (gap) antara sitosin (DC8) dan timin (DT9), celah ini terbentuk karena aktifitas enzim DNA topoisomerase II. Celah inilah yang merupakan target dari beberapa jenis obat antikanker yang tergolong berinteraksi dalam mode interkalasi seperti etoposida. Terlihat semua ligan uji memiliki mode ikatan interkalasi yang sama dengan ligan pembanding hal ini dapat dilihat berdasarkan nilai RMSD yang kecil dari 2.5 Å.
24
cincin fonolik A kurkumin melengkung ke bawah, sedangkan dua senyawa turunannya tidak. Hal ini disebabkan oleh gugus samping metoksi dari kurkumin. Pada cincin fenolik A, senyawa demetoksikurkumin dan bisdemetoksikurkumin tidak lagi memiliki gugus samping metoksi sehingga cincin fenolik A tersebut mengalami kenaikan sifat kepolarannya dibandingkan kurkumin yang masih memiliki gugus metoksi pada cincin fenolik A. Pada visualisasi ikatan hidrogen untuk kurkumin (Gambar 8), pose kurkumin melengkung kebawah karena gugus samping metoksi tertarik oleh residu Pro819 melalui interaksi hidrofobik, ditunjukkan oleh arah garis merah mengarah ke arah prolin.
5 SIMPULAN DAN SARAN
Simpulan
Berdasarkan hasil penambatan molekular dapat disimpulkan bahwa residu Asp479, Arg503, Gly478
, Gln778, DA12, DG13, DT9, DG10 dan Met782 berperan penting dalam pembentukan ikatan Pi, ikatan hidrogen dan interaksi elektrostatik terhadap enzim DNA topoisomerase II (reseptor). Ikatan Pi terbentuk oleh struktur siklik A dari senyawa kurkuminoid terhadap DNA (DA12, DG13, DT9, DG10) dengan mode interkalasi, ikatan hidrogen (gugus OH2 dan OH3 dari senyawa kurkuminoid), interaksi hidrofobik (Met782) dan interaksi elektrostatik (Asp479, Arg 503, Gln778, Glu477). Ekstrak kurkuminoid yang terdiri kurkumin, demetoksi kurkumin dan bisdemetoksi kurkumin memiliki nilai ΔGbinding sebesar -10.1 kkal/mol, -9 kkal/mol, -8.9 kkal/mol, sehingga berpotensi sebagai agen inhibitor enzim DNA topoisomerase II.
Saran
26
DAFTAR PUSTAKA
Abu HS, Zihlif M. 2012. Gene expression alteration in doxorubicin resistan MCF-7 breast cancer All Ine. Genomic. 101: 213-220.
Anand P, Thomas SG, Kunnumakkara AB, Sundaram C, Harikumar KB. 2008. Biological activities of curcumin and its analogues (congeners) made by man and mother nature. Biochem Pharm. 76: 1590-1611.
Aggarwal BB, Bhatt ID, Ichikawa H, Ahn KS, Sethi G, Sandur SK, Natarajan C, Seeram N, Shishodia S. 2006. Turmeric: the genus gurcuma. Taylor and Francis. New York. 298–303.
Aravind L, Leipe DD, Koonin EV. 1998. Toprim: a conserved catalytic domain in type IA and II topoisomerases, dna g-type primases, OLD family nucleases and RecR proteins. Nucleic Acids Res. 26:4205–13.
Arwansah. 2014. Simulasi molecular docking senyawa kurkumin dan analognya sebagai selective androgen reseptor modulator (SARMs) pada kanker prostat. Bogor (ID): IPB Pr.
Baber JC, Thompson DC, Cross JB, Humblet C. 2009. GARD : A generally
applicable replacement for RMSD. J Chem Inf Model. 49: 1889–1900.
Bahng S, Mossessova E, Nurse P, Marians KJ. 2000. Mutational analysis of Escherichia colitopoisomerase IV. III. Identification of a region of par E involved in covalent catalysis. J Biol Chem. 275:4112–17.
Bates AD, Maxwell A. 1997. DNA topology: topoisomerases keep it simple. Curr Biol. 7: 778-81.
Baxevanis AD, Ouellette BF. 2001. Bioinformatics a practical guide to the analysis of genes and proteins second edition. 1-457.
Beck LS, Deguzman L, Lee WP, Xu Y, Siegel MW, Amento EP. 1993. One systemic administration of transforming growth factor-beta 1 reverses age or glucocorticoid impaired wound healing. J Clin Invest. 92: 2841-2849.
Bender RP, Jablonksy MJ, Shadid M, Romaine I, Dunlap N, Anklin C, Graves DE, Osheroff N. 2008. Substituents on etoposide that interact with human topoisomerase IIα in the binary enzyme-drug complex: contributions to etoposide binding and activity. Biochemistry. 47: 4501-4509.
Berger JM, Fass D, Wang JC, Harrison SC. 1998. Structural similarities between topoisomerases that cleave one or both DNA strands. Proc Natl Acad Sci. 95:7876–81.
Berger JM, Gamblin SJ, Harrison SC, Wang JC. 1996. Structure and mechanism of DNA topoisomerase II. Nature. 379:225–32.
Bosco KH, Brasseur R. 2005. The Ramachandran plots of glycine and pre-proline. BMC Struc Bio. 5:1-14.
Brennan RG, Matthews BW. 1989. The helix-turn-helix DNA binding motif. J Biol Chem. 264:1903–6.
Clark J. 2007. The Acidity of Phenol. ChemGuide.
Champoux JJ. 2001. DNA topoisomerases: structure, function, and mechanism. Annu Rev Biochem. 70: 369-413.
Chow KC, Macdonald TL, Ross WE. 1998. DNA binding by epipodophyllotoxins and N-acyl antracyclines: Implications for mechanism of topoisomerase inhbition. Mol Pharmacol. 34: 467-473.
Clark KL, Halay ED, Lai E, Burley SK. 1993. Co-crystal structure of the HNF-3/fork head DNA-recognition motif resembles histone H5. Nature. 364: 412–
20.
Corbett KD, Berger JM. 2004. Structure, molecular mechanisms, and evolutionary relationships in DNA topoisomerases. Annu Rev Bioph Biom. 33: 95-118. Ekins S, Bruno BS, Peter W, Hupcey, Maggie AZ. 2000. Towards a new age of
virtual ADME/TOX and multidimensional drug discovery. Molecular Diversity. 5(4): 255-275.
Ekins S, Nikolsky Y, Nikolskaya T. 2005. Techniques: aplications of systems biology to absorption, distribution, metabolism, excretion and toxicity. Trensin Pharm Sci. 26 (4): 2002-2009.
Fass D, Bogden CE, Berger JM. 1999. Quaternary changes in topoisomerase II may direct orthogonal movement of two DNA strands. Nat Struct Biol. 6: 322–26.
Fitriasari A, Wijayanti NK, Ismiyati N, Dewi D, Kundarto W, Sudarmanto BA,Meiyanto E. 2008. Studi potensi kurkumin dan analognya sebagai selective estrogen receptor modulators (SERMs): docking pada reseptor estrogen β. Pharmacon. 9(1): 27-32.
Fortune JM, Osheroff N. 2000. Topoisomerase II as a target for anticancer drugs: when enzymes stop being nice. Prog Nucl Acid Res. 64: 221-253.
Grotenhuis PDJ, Kollman DC, Sieble GL, Desjarlais RL, Kuntz ID. 1990. Computerized selection of potential DNA binding compounds. Anti-cancer Drug Des. 5: 237-242.
Grynkiewicz, Ślifirski. 2012. Curcumin and curcuminoids in quest for medisinal status. Acta Bio Pol. 59(2): 201–212.
Hannon MJ. 2007. Supramolecul DNA recognition by intercalator. Chem Soc Rev. 36: 280-295.
Harrold DW. 1998. Important of functional group chemistry in the drug selection process: A case study. Pharm Edu. 62: 213-218.
Hetenyi C, Spoel DV. 2002. Efficient docking of peptida to protein without prior knowledge of binding Sie. Pot Sci. 11(7): 1729-1737.
Hof T, Pinter T. 2012. Learning from protein and drug: reseptor thar mimi biomedically important binding motif. Chemical Sensors and Biosensors 12: 33–52.
Jefrey GA. 1997. An introduction to hydrogen bonding. New York: Oxford University Press.
Jovanovic SV, Steenken S, Boone CW, Simic MG. 1999. H-atom transfer is a preferred antioxidant mechanism of curcumin. J of the American Chemical Society. 121 (41): 9677–9681.
Jones S, Daley DTA, Luscombe NM, Berman HM, Thornton JM. 2001. Protein-RNA interaction: a struktur analysis. Nuc Acd Res. 29(4): 943-954.
28
Kumar A, Bora U. 2011. In silico inhibition studies of NF-kB p50 sub unit by curcumin and its natural derivative. Med Chem Res. DOI: 10.1007/s004-011-9873-0.
Lavery DN, Bevan CL. 2010. Androgen receptor signalling in prostate cancer: the Lima CD, Wang JC, Mondragon A. 1994. Three-dimensional structure of the 67K
N-terminal fragment of E. coli DNA topoisomerase I. Nature. 367: 138–46. Lins L, Brasseur R. 1995. The hydrofobic effect in protein folding. Faseb J. 9:
535-540.
Lipinski CA, Lombardo F, Beryl WD, Paul JF. 2001. Experimental and computational approaches to estimate solubility and permeability in drug discovery and development settings. Advanced Drug Delivery. 46: 3-26. Lu HM, Yin DC, Ye YJ, Luo HM, Geng LQ, Li HS, Guo WH, Shang P. 2009.
Correlation between protein sequence similarity and x-ray diffraction quality in the protein data bank. Prot Pept Lett. 16 (1): 50-5.
Moitessier N, Englebienne P, Lee D, Lawandi J, Corbeil CR. 2008. Towards the development of universal, fast and highly accurate docking/skoring method: aalong way to go. Br J Pharmaol. 153: 7-26.
Morris GM, Godsell DS, Halliday RS, Guey R, Hart WE, Belew RK, Olson AJ. 1998. Automated docking pusing lamarckian genetis algoritma and na empirical inding free energy function. J Comp Chem. 19(14): 1639-1662. Nelson D, Cox M. 2008. Principle of Biochemistry (5th Edn.). W.H. freeman and
company (USA). ISBN-13: 978-0-7176-7108-1.
Nitiss JL. 1998. Investigating the biological functions of DNA topoisomerases in eukaryotic cells. BBA-Gene Struct Expr. 1400: 63-81.
Nitiss JL. 2009 DNA topoisomerase II and its growing repertoire of biological functions. Nat Rev Cancer. 9: 327-337.
Osheroff N. 1987. Role of the divalent cation in topoisomerase II mediated reactions. Biochemistry. 26: 6402–6.
Overgaard RV. 2006. Pharmacokinetic/pharmacodynamic modelling with a stochastic perspective; Insulin secretion and interleukin-21 development as case studies. [Tesis]. Tecnical University of Denmark.
Pebriana RB, Romadhan AF, Yunianto A, Rokhman MR. 2008. Dockingkurkumin dan senyawa analognya pada reseptor progesteron: studi interaksinya sebagai selective progesterone receptor modulators (SPRMs) Pharmacon. 9(1): 14-20.
Permasku Gia. 2014. Aktivitas eskstrak kurkuminoid rimpang temulawak (Curcuma Xanthorrhiza roxb)dari berbagai aksesi terhadap inhibisi enzim alfa glukosidase secara in vitro. [skripsi]. Bogor (ID): Institute Pertanian Bogor.
Pitts SL, Jablonsky MJ, Duca M, Dauzonne D, Monneret C, Arimondo PB, Anklin C, Graves DE, Osheroff N. 2011. Contribution of the D-Ring to the activity of etoposide against human topoisomerase IIA: potential interactions with DNA in the ternary enzyme-drug-DNA complex. Biochemistry. 50(22): 5058-5066.
Ramachandran GN, Ramakrishnan C, Sasisekharan V. 1963. Conformation of polipeptides and protein. J mol Biol. 7: 95-99.
Sakoparnig T, Fried P, Beerenwinkel N. 2015. Identification of constrained cancer River genes on mutatio timing. PLoS Comput Biol. 11(1): e1004027. doi:10.1371/journal.pcbi.1004027.
Saleh EM, El-awady RA, Eissa NA, Abdel Rahman WM. 2012. Antagonism between curcumin and the topoisimerase II inhibitor etoposida: a study of DNA damage, cell cycle regulation and death pathways. Can Bio & Thr. 13 (11): 1058-1071.
Saputra G. 2014. Simulasi docking senyawa kurkumin dan analognya sebagai inhibitor enzim 12-lipoksigenase. Bogor (ID): IPB Pr.
Schmidt B, Burgin J, Dewesese J, Osheroff N, Berger J. 2010. A novel of unified two metal mechnanism for DNA cleavage by tipe II dan IA topoisomerase. Nature. 465: 461.
Schultz SC, Shields GC, Steitz TA. 1991. Crystal structure of a CAP-DNA complex: the DNA is bent by 90 degrees. Science. 253: 1001–7.
Sharp KA, Honig B. 1990. Electrostatic interactions in macromolecules: theoryand applications. Annu Rev Biophys Biophys Chem. 19: 301-322. Singh R, Chaturvedi N, Singh VK. 2012. In silico study of herbal compounds as
novel MAO inhibitors for parkinsons disease treatment. Life Scie Phar Res. 2: 81-98.
Somparn P, Phisalaphong C, Nakornchai S, Unchern S, Morales NP. 2007. Comparative antioxidant activities of curcumin and its demethoxy and hydrogenated derivatives. Biol and Pharm Bulletin. 30(1): 74–78.
Stewart I, Redinbo MR, Qiu X, Hol WG, Champoux JJ. 1998. A model for the mechanisme of human topoisomerase. Science (New York, NY) 279: 1534-1541.
Struck S, Schmidt U, Gruening B, Jaeger IS, Hossbach J, Preissner R. 2002. Toxicity vs potency: elucidation of toxicity properties discriminating between toxin, drug and natural compound. J Farm. 231-242.
Synder RD, Ewing DE, Hendry LB. 2004. Evaluation of DNA intercalation potential of pharmaceuticals and other chemical by cell-based and three-dimantional computation approach. Env and Mol Mutagenesis. 44 (2): 163 – 173.
Wang JC. 2002. Cellular roles of DNA topoisomerases: A molecular perspective. Nat Rev Mol Cell Bio. 3: 430-440.
Wendorff TJ, Schmidt BH, Heslop P, Austin CA, Berger JM. 2012. The structure of DNA-bound human topoisomerase II alpha: conformational mechanisms for coordinating inter-subunit interactions with DNA cleavage. J Mol Biol. 424: 109-24.
Wu CC, Li TK, Farh L, Lin LY, Lin TS, Yu YJ, Yen TJ, Chiang CW, Chan NL. 2011. Structural basis of type II topoisomerase inhibition by the anticancer drug etoposide. Science. 333: 459-462
Xiong Y, Sundaralingam. 2001. Protein-nucleic acid interaction: major grove recognition determinats. Ensikloedia of life Sci. 1-8
Yeturu K, Nagasuma C. 2008. Pocket match: a new algoritm to compare binding sites in protein structures. BMC Bioinformatic. 9: 543.
30
Analisis :
1. ∆G
2. RMSD
3. Ikatan Pi
4. Interaksi Hidrogen
5. Mode pengikatan
6. Gugus fungsi
Lampiran 1 Diagram Alir Penelitian
Kompeks enzim DNA Topoisomerase II-Ligan
Perancangan Ligan
Struktur 3D Central-Terminal
Kualitas struktur
Optimasi reseptor Filter Lipinski
Pencarian enzim DNA Topoisomerase II
Optimasi Struktur Reseptor
DOCKING GRID BOX
32
Lampiran 2 Struktur Lengkap Enzim DNA Topoisomera se II
Keterangan: A) Struktur enzim dan domain pada DNA topoisomerase IIα. B) Sisi
aktif pemotongan rantai fosfat pada DNA.
34
Lampiran 5 Struktur Ligan
Lampiran 6 Parameter Penambatan receptor = 3qx3eto2.pdbqt ligand = cur.pdbqt
out = out.pdbqt center_x = 32.894 center_y = 95.413 center_z = 50.785 size_x = 80 size_y = 80
36
Lampiran 7 Simulasi Penambatan Molekuler (Molecular Docking) 1. Buka file ligan (ex. Kurkumin.pdb)
Buka software ADT > klik ligan > pilih file kurkumin.pdbqt
2. Atur dan seleksi ikatan yang dapat berotasi Klik ligan > Torsion tree > Choose Torsion
3. Simpan ligan ke dalam format kurkumin.pdbqt
4. Open file protein (file 3QX3 .pdb) Open > pilih molekul 3QX3.pdb