KAJIAN KERAGAMAN GENETIK SAPI BALI DI PUSAT
PEMBIBITAN SAPI BALI MENGGUNAKAN
PENCIRI LOKUS MIKROSATELIT
WIKE ANDRE SEPTIAN
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul dalam Kajian Keragaman Genetik Sapi Bali di Pusat Pembibitan Sapi Bali Menggunakan Penciri Lokus Mikrosatelit adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Desember 2014
RINGKASAN
WIKE ANDRE SEPTIAN. Kajian Keragaman Genetik Sapi Bali Di Pusat Pembibitan Sapi Bali Menggunakan Penciri Lokus Mikrosatelit. Dibimbing oleh JAKARIA dan CECE SUMANTRI.
Sapi Bali merupakan salah satu plasma nutfah nasional yang telah banyak diketahui memiliki keunggulan sebagai potong dan tersebar hampir diseluruh wilayah Indonesia. Pemanfaatan dan perbaikan mutu genetik sapi Bali tidak bisa mengenyampingkan upaya pelestarian sapi Bali. Saat ini program persilangan dan distribusi bibit sapi Bali yang tidak terarah akan menjadi ancaman terhadap timbulnya erosi genetik pada sapi Bali. Upaya dalam mencegah terjadinya erosi genetik pada sapi Bali khususnya di pusat pembibitan sapi Bali perlu dilakukan karateristik genetik. Salah satu pendekatan untuk melakukan karakteristik genetik memalui pendekatan penciri mikrosatelit.
Penelitian ini bertujuan untuk mengetahui keragaman mikrosatelit di tiga pusat peternakan BPTU Bali, BPT-HMT Serading Sumbawa di Nusa Tenggara Barat, dan Village Breeding Center Kabupaten Barru di Sulawesi Selatan. Jumlah hewan yang digunakan dalam penelitian ini adalah 96 ekor terdiri dari 32 ekor sapi dari BPTU Provinsi Bali, 32 ekor sapi bali dari BPT-HMT Serading Sumbawa, dan 31 ekor sapi Bali dari Village Breeding Center Kabupaten Barru. Lokus mikrosatelit digunakan untuk menentukan keragaman adalah SPS115 lokus, INRA037, MM12, dan ETH185 berdasarkan metode fragmen floureschenly berlabel. Analisis data mikrosatelit pada sapi Bali di tiga lokasi yang berbeda dilakukan dengan menggunakan Program POPGEN1.2, Cervus, dan POPTREE2.
SUMMARY
WIKE ANDRE SEPTIAN. Genetic Diversity of Bali Cattle Based On Microsatellite Marker In Breeding Centre Indonesia. Supervised by JAKARIA and CECE SUMANTRI.
Bali cattle is one of Indonesian germ plasm with specific purpose for beef cattle because of its superiority and spread out in many different region of Indonesia. The utility and genetic improvement of Bali cattle must be sinergized with the conservation programme. Recently, uncontrolled of crossbreeding and breed distribution of Bali cattle has become one threat on genetic variation. In order to prevent genetic erotion of Bali cattle in breeding centre, genetic characterization is needed.
This study aims to determine diversity of microsatellites in three breeding centers BPTU Bali cattle in Bali, BPT-HMT Serading Sumbawa in West Nusa Tenggara, and Village Breeding Center in South Sulawesi. The number of animals used in this study was 96 head consists of 32 head of cattle from BPTU Bali Province, 32 head of bali cattle from BPT-HMT Serading Sumbawa, and 31 head of bali cattle from Village Barru Breeding Center Barru distric. Microsatellite locus used to determine the diversity is the locus SPS115, INRA037, MM12, and ETH185 based method fragments floureschenly labeled. Data analysis microsatellite in Bali cattle at three different locations performed using POPGEN1.2, Cervus, and POPTREE2 Program.
© Hak Cipta Milik IPB, Tahun 2014
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Ilmu Produksi dan Teknologi Peternakan
KAJIAN KERAGAMAN GENETIK SAPI BALI DI PUSAT
PEMBIBITAN SAPI BALI MENGGUNAKAN
PENCIRI MIKROSATELIT
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2014
Judul Tesis : Kajian Keragaman Genetik Sapi Bali di Pusat Pembibitan Sapi Bali Menggunakan Penciri Mikrosatelit
Nama : Wike Andre Seprtian NIM : D151114041
Disetujui oleh Komisi Pembimbing
Dr Jakaria, SPt, MSi Ketua
Prof Dr Ir Cece Sumantri, MAgrSc Anggota
Diketahui oleh
Ketua Program Studi
Ilmu Produksi dan Teknologi Peternakan
Dr Ir Salundik, MSi
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
PRAKATA
Puji syukur kehadirat Allah subhanahu wata’ala atas segala rahmat, taufiq, hidayah, serta inayah-Nya sehingga karya ilmiah ini berhasil diselesaikan. Judul penelitian yang di pilih dan dilaksanakan sejak oktober 2013 adalah “Karakteristik Keragaman Genetik Sapi Bali di Pusat Pembibitan Sapi Bali Menggunakan Pendekatan Penciri Mikrosatelit” merupakan syarat penyelesaian pendidikan program magister pada program studi Ilmu Produksi dan Teknologi Peternakan. Shalawat serta salam semoga senang tiasa tercurahkan kepada Nabi Muhammad Salllallahu alaihi wassalam keluarga, para sahabat, dan para pengikut yang diridhoi-Nya.
Penulis menyadari bahwa dalam penyelesaian tesis ini adalah hasil sumbangsih materi maupun pemikiran dari beberapa pihak. Oleh karena itu, dalam kesempatan ini penulis ingin menghaturkan terima kasih sebesar-besarnya dan penghargaan yang setinggi-tingginya kepada semua pihak yang telah membantu dalam penyelesaian karya ilmiah ini, terutama kepada Yang terhormat Bapak Dr Jakaria S.Pt, M.Si dan Prof Dr Ir Cece Sumantri MAgrSc selaku ketua dan Anggota Komisi Pembimbing yang telah memberikan bimbingan, arahan, motivasi dan saran sejak berupa sebuah ide hingga penyelesaian tesis. Bapak Dr Ir Chalid Talib MS selaku penguji luar komisi dan Dr Ir Niken Ulupi MS selaku Sekretaris Program studi ITP serta Dr Ir Salundik M.Si Ketua Program studi Pascasarjana ITP pada ujian tesis penulis atas saran dan masukan memperkaya muatan pada hasil karya penulis. Bapak Prof Dr Muladno MSA Sebagai Ketua Departemen Ilmu Produksi Teknologi Peternakan dan Prof Dr Ir Luki Abdullah MAgrSc selaku Dekan Fakultas Peternakan atas beberapa diskusi yang merupakan motivasi, arahan, wawasan baru, dan bimbingan selama masa studi. Rektor dan Dekan Sekolah Pascasarjana Institut Pertanian Bogor yang telah memberikan kesempatan kepada penulis untuk melanjutkan pendidikan S2 dengan baik.
Kolega khusus yang membantu penulis dan menjadi sahabat selama kegiatan penelitian bang Eryk Andreas SPt MSi, Ferdy Saputra SPt MSi, Irenz SPt MSi, Selvi SSi, dan Hikmawati SPt MSi serta teman-teman ITP 2011. Rekan The Helix 2012 Sri Wahyuni SPt MSi, Marlinda SPt MSi, Tya SPt MSi, Raden Jatu SPt MSi, Fuad Hasan SPt MSi, Annisa Oktavia Rini SPt MSi, Pandu SPt MSi dan tim ABGSci secara keseluruhan yang telah memberikan kesan mendalam dalam aktivitas selama studi di mayor genetika. Rekan ITP 2012 yang telah menjadi sahabat dalam menguatkan penulis untuk menyelesaikan tulisan ini. Terimakasih juga kepada abang saya Ayub Rizal SPt MSi dan Ismail SPt MSi serta teteh Irma SPt MSi dan drh Arfiani Idris MSi yang senang tiasa bersedia menjadi sahabat diskusi hangat beragam topik. Terimakasih pula penulis ucapkan kepada sahabat di Laboratorium Pemuliaan dan Genetika Molekuler LIPI Paska Partogi Agung SPt MSi, Ari Sulandari SPt MSc, Saiful Anwar SPt MSi dan Lusiana SPt yang rela tiap hari menjadi sahabat kerja, bersedia bertukar cerita dan ilmu.
Kepada semua pihak yang tidak dapat disebutkan satu persatu, yang turut membantu penulis demi suksesnya penyelesaian studi Master di Institut Pertanian Bogor ini.Semoga karya ilmiah ini bermanfaat. Salllallahu alaihi wassalam
Bogor, Desember 2014
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
1 PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 2
2 TINJAUAN PUSTAKA 3
Sumberdaya Genetik Ternak 3
Program Pemuliaan dan Pelestarian Sapi Bali 4
Sapi Bali 5
Mikrosatelit 5
3 METODE 6
Waktu dan Tempat 6
Bahan 6
Alat 6
Prosedur Penelitian 7
Analisis Data 9
4 HASIL DAN PEMBAHASAN 9
Amplifikasi dan GenotypingEmpat Lokus Mikrosatelit 9
Frekuensi Alel Lokus Mikrosatelit 13
Nilai Heterozigositas dan Keseimbangan Hardy-Weinberg 16 Laju Inbreeding dan Keragaman Genetik Sapi Bali 17
Jarak Genetik Sapi Bali 19
Strategi Pengembangan dan Pelestarian Sumberdaya Genetik Sapi Bali 20
SIMPULAN DAN SARAN 21
Simpulan 21
Saran 22
DAFTAR PUSTAKA 22
DAFTAR TABEL
1. Lokus mikrosatelit, posisi di kromosom, sekuen primer, dan panjang
alel 8
2. Frekuensi alel empat lokus mikrosatelit di tiga populasi 15 3. Keragaman genetik sapi Bali di tiga pusat pembibitan sapi Bali 16 4. Nilai Fis, Fit, dan Fst, dari empat lokus mikrosatelit 18 5. Nilai Ho, He, danPIC dari empat lokus mikrosatelit 18 6. Nilai PIC dari empat lokus mikrosatelitbeberapa bangsa sapi 18 6. Jarak genetik antar pusat pembibitan sapi Bali 19
DAFTAR GAMBAR
1. Hasil amplifikasi primer lokus mikrosatelit SPS115 pada sapi Bali menggunakan gel agaros 1,5% dan pewarnaan ethidium bromide. M= marker DNA 100 bp. Sampel 1-16 = produk amplifikasi antara
234-310 bp 9
2. Hasil amplifikasi primer lokus mikrosatelit MM12 pada sapi Bali menggunakan gel agaros 1,5% dan pewarnaan ethidium bromide. M= marker DNA 100 bp. Sampel 1-16 = produk amplifikasi antara
101-145bp 9
3. Hasil amplifikasi primer lokus mikrosatelit INRA037 pada sapi Bali menggunakan gel agaros 1,5% dan pewarnaan ethidium bromide. M= marker DNA 100 bp. Sampel 1-16 = produk amplifikasi antara
112-180 bp 9
4. Hasil amplifikasi primer lokus mikrosatelit ETH185 pada sapi Bali menggunakan gel agaros 1,5% dan pewarnaan ethidium bromide. M= marker DNA 100 bp. Sampel 1-16 = produk amplifikasi antara
214-260 bp 10
5. Hasil fragment analysis lokus mikrosatelit SPS115 dengan M13 FAM menggunakan software GeneMapper 4.0 kondisi homozigot 11 6. Hasil fragment analysis lokus mikrosatelit SPS115 dengan M13 FAM
menggunakan software GeneMapper 4.0 kondisi heterozigot 11 7. Hasil fragment analysis lokus mikrosatelit MM12 dengan M13 TET
menggunakan software GeneMapper 4.0 kondisi homozigot 11 8. Hasil fragment analysis lokus mikrosatelit MM12 dengan M13 TET
menggunakan software GeneMapper 4.0 kondisi heterozigot 12 9. Hasil fragment analysis lokus mikrosatelit INRA037 dengan M13
FAM menggunakan software GeneMapper 4.0 kondisi homozigot 12 10. Hasil fragment analysis lokus mikrosatelit INRA037 dengan M13
FAM menggunakan software GeneMapper 4.0 kondisi heterozigot 12 11. Hasil fragment analysis lokus mikrosatelit ETH185 dengan M13
FAM menggunakan software GeneMapper 4.0 kondisi homozigot 13 12. Hasil fragment analysis lokus mikrosatelit ETH185 dengan M13
1
1
PENDAHULUAN
Latar Belakang
Indonesia merupakan salah satu negara yang memiliki kekayaan sumberdaya genetik ternak. Sapi Bali merupakan salah satu sumberdaya genetik ternak yang memiliki potensi besar dalam penyediaan kebutuhan daging sapi Nasional. Populasi sapi Bali saat ini berdasarkan hasil pendataan sapi potong, sapi perah dan kerbau adalah 4 658 781 atau sekitar 31% dari jumlah total sapi potong di Indonesia. Sapi Bali tersebar di semua wilayah Indonesia dengan populasi terbanyak terdapat pada Sulawesi Selatan 954 901 ekor, Bali 637 463 ekor, Nusa Tenggara Barat 672 472 ekor, dan Nusa Tenggara Timur 683 928 ekor. Indonesia sebagai pusat sapi Bali dunia memiliki beberapa pusat pembibitan sapi Bali. Pusat pembibitan sapi Bali di Indonesia dibagi menjadi dua balai pembibitan yaitu Balai Pembibitan Ternak Unggul Pulukan Bali di Provinsi Bali dan Balai Pembibitan Ternak dan Hijauan Makan Ternak Serading di Pulau Sumbawa, Nusa Tenggara Barat, dan satu pusat pembibitan berbasis peternakan rakyat yaitu Village Breeding Center di Kabupaten Barru, Sulawesi Selatan. Minimnya informasi genetik pada sapi Bali di pusat pembibitan menjadi hal penting untuk arah kebijakan program pembibitan sapi Bali secara nasional.
Saat ini program persilangan dan distribusi bibit sapi Bali yang tidak terarah akan menjadi ancaman terhadap timbulnya erosi genetik pada sapi Bali. Persilangan dan distribusi yang tidak berdasarkan data hasil pencatatan dapat mengakibatkan laju inbreeding pada populasi sapi Bali. Hal ini merupakan salah satu bentuk ancaman kepunahan yang disebabkan oleh genetic stochasticity, yaitu perubahan genetik secara acak (genetic drift) dan meningkatnya inbreeding. Shaffer (1981) menyatakan bahwa genetic stochasticity adalah hilangnya variasi genetik terhadap alel-alel yang bersifat ekonomis atau meningkatkan alel-alel merugikan. Upaya dalam mencegah terjadinya erosi genetik dan fenomena genetic stochasticity pada sapi Bali khususnya di pusat pembibitan sapi Bali perlu dilakukan karakterisasi genetik. Salah satu pendekatan untuk melakukan karakterisasi genetik pada ternak sapi melalui pendekatan penciri mikrosatelit.
2
Penggunaan penciri mikrosatelit secara luas telah dilakukan dalam kajian keragaman genetik pada bangsa sapi di Eropa (Canon et al. 2001; Beja et al. 2003), sapi Japannese Black (Kim et al. 2002), sapi lokal China (Zhang et al. 2007), dan sapi lokal Vietnam (Lan et al. 2013). Penggunaan penciri mikrosatelit pada sapi Bali telah dilaporkan oleh Nijman et al. (2003), Maskur et al. (2007), Handiwirawan et al. (2003), Kusdiantoro et al. (2009), dan Winaya et al. (2010). Beberapa hasil penelitian tersebut masih sangat terbatas dalam analisis sapi Bali antar populasi dan metode. Penelitian terdahulu untuk mengetahui hubungan genetik antar bangsa sapi lokal Indonesia dan sebagian besar dilakukan dengan metode pewarnaan perak (PAGE). Belum ada penelitian yang berbasis demografi dari populasi – populasi sapi Bali di Indonesia. Pada saat ini penggunaan penciri mikrosatelit yang sangat populer adalah teknik fragment fluorescently berlabel. Selain itu fragment fluorescently juga dikenal dengan teknik penambahan M13 tailed forwads atau disebut juga teknik an economi method (Schuelke 2000). Metode ini dinilai lebih efektif dalam menginterprestasikan hasil analisis pada lokus mikrosatelit. Berdasarkan hal tersebut diatas maka penelitian menggunakan penciri mikrosatelit dengan fragment fluorescently berlabel perlu dilakukan pada sapi Bali di ketiga pusat pembibitan sapi Bali di Indonesia.
Perumusan Masalah
Sapi Bali yang tersebar luas di wilayah-wilayah Indonesia merupakan potensi sumberdaya genetik ternak untuk keberlangsungan ketersediaan daging di Indonesia. Akan tetapi tersebarnya sapi Bali bisa menjadi ancaman erosi genetik jika persilangan dan distribusi bibit sapi Bali tidak berlandaskan pemetaan genetik di setiap wilayah sapi Bali khususnya di pusat pembibitan sapi Bali di Indonesia. Penggunaan penciri genetik seperti mikrosatelit merupakan salah satu cara memetakan potensi genetik berdasarkan demografi. Berasarkan hal tersebut, maka penggunaan penciri mikrosatelit pada sapi Bali berdasarkan demografi menjadi sangat berperan penting dalam mengetahui karakteristik genetik sapi Bali ditiga pusat pembibitan sapi Bali serta sebagai landasan dalam penentuan arah kebijakan program pemuliaan sapi Bali.
Tujuan Penelitian
Penelitian ini bertujuan mengkaji karakteristik genetik sapi Bali di ketiga lokasi pusat pembibitan sapi Bali di BPTU Bali, BPT-HMT Serading Sumbawa, Nusa Tenggara Barat, dan Village Breeding Center Kabupaten Barru Sulawesi Selatan menggunakan pendekatan penciri mikrosatelit.
Manfaat Penelitian
3
Ruang Lingkup Penelitian
Ruang lingkup penelitian identifikasi karakteristik genetik sampel sapi Bali dari tiga pusat pembibitan sapi Bali, BPTU Bali, BPT-HMT Serading Sumbawa, Nusa Tenggara Barat, dan Village Breeding Center Kabupaten Barru, Sulawesi Selatan dengan menggunakan empat lokus penciri mikrosatelit yaitu INRA037, SPS115, MM12, dan ETH185.
2
TINJAUAN PUSTAKA
Sumberdaya Genetik Ternak
Indonesia merupakan negara di Asia Tenggara yang kaya akan sumberdaya genetik ternak. Sumberdaya genetik merupakan substansi pada individu di dalam populasi yang membentuk karakteristik genetik unik. Keunikan karakteristik genetik terbentuk melalui proses domestikasi dan pemanfaatan ternak dengan cara perkawinan yang terprogram untuk membentuk rumpun atau galur baru (Departemen Pertanian RI 2006). Salah satu sumberdaya genetik ternak lokal Indonesia adalah sapi Bali. Sapi Bali merupakan sapi lokal hasil domestikasi dari Banteng liar (bibos banteng) yang merupakan hewan kelas bovidae primitif yang terjadi sekitar 3500 SM di Indonesia (Namikawa et al. 1980; Rollinson 1984). Penyebaran sapi Bali di Indonesia dimulai pada tahun 1890 dengan dilaksanakan pengiriman ke Sulawesi, pengiriman dilakukan pada tahun 1920 dan 1927. Pada tahun 1927 sapi Bali dimasukkan ke Sulawesi Selatan (Rampi) sebanyak 5 ekor dan pada tahun 1940 jumlahnya telah mencapai 80 ekor.
Pada tahun 1947 sapi Bali disebarkan ke provinsi ini secara besar besaran. Sapi-sapi ini yang kemudian menjadi cikal bakal sapi Bali di Sulawesi Selatan. Pada tahun 1964 di Bali terjadi musibah penyakit jembrana secara besar-besaran yang menyebabkan sapi Bali tidak boleh dikeluarkan lagi dari pulau Bali sebagai ternak bibit. Mulai periode inilah sumber sapi Bali bagi daerah lain di Indonesia digantikan oleh NTT, Sulawesi dan NTB (Talib, 2002).
Pelestarian sumberdaya genetik sangat penting dilakukan karena merupakan bagian dari keragaman hayati dalam memenuhi kebutuhan pangan saat ini maupun masa yang akan datang. Perlestarian sumberdaya genetik merupakan salah satu sudut pandang akan potensi jaminan peningkatan kehidupan sosial ekonomi pada saat ini atau pada masa yang akan datang (Maskur 2012). Pada saat ini nilai ekonomis dari sumberdaya genetik ternak untuk ketahanan pangan lebih menonjol dibandingkan dalam konteks pelestarian, sehingga dalam proses peningkatan produktivitas sering kali kurang tepat dan mengancam eksistensi dari ternak lokal yang memiliki nilai ekonomis/produktivitas lebih rendah. Praktek breeding dan persilangan dilakukan hampir dibeberapa negara berkembang seperti Indonesia. Persilangan dilakukan dengan mengimpor ternak yang memiliki produktivitas lebih tinggi untuk meningkatkan produktivitas ternak lokal. Konsekuensi nyata dalam praktek persilangan adalah menurunnya keragaman hayati dan mengancam eksistensi sumberdaya genetik ternak lokal (Maskur 2012).
4
merupakan habitat asli ternak lokal seperti pulau Sumbawa dan Nusa Penida Bali sebagai lokasi konservasi genetik. Proses pemurnian yang dilakukan harus berpijak pada informasi genetik yang terdapat pada populasi tersebut, sehingga sebuah kebijakan pemurniaan tepat berfungsi sebagai tindakan pelestarian sumberdaya genetik.
Program Pemuliaan dan Pelestarian Sapi Bali
Upaya dalam melestarikan sapi Bali sebenarnya sudah tercatat dalam sejarah Indonesia bahkan sebelum Indonesia resmi menjadi negara. Hal ini menunjukkan bahwa kesadaran akan pentingnya sumberdaya genetik ternak khususnya sapi Bali sudah ada sejak dulu. National Research Council (1983) mencatat pada tahun 1913 pemerintah sudah melahirkan produk hukum yang melarang adanya persilangan pada sapi Bali untuk mempertahankan kemurnian bangsa sapi Bali. Pada saat itu konsentrasi pelestarian di arahkan ke dua tempat yaitu Pulau Bali dan Pulau Sumbawa. Program pemuliaan pada sapi Bali dengan memulai program seleksi pada sapi Bali di awali tahun 1942. Program seleksi ini terus di kembangkan dengan melibatkan peternak sapi Bali. Tahun 1949 pemerintah memberikan insentif bagi peternak yang memiliki ternak jantan baik untuk dipertahankan (Payne & Rollinson 1973).
Keseriusan pemerintah dalam upaya pengembangan dan pelestarian sapi Bali ditunjukkan dengan Undang Undang No. 6 tahun 1967. Undang Undang tersebut merupakan landasan dalam pengembangan dan pelestarian ternak lokal di Indonesia termasuk sapi Bali. Dalam hal ini semua program pengembangan harus sejalan dengan mempertahakan serta meningkatkan mutu genetik ternak di Indonesia melalui upaya pemurnian atau melalui persilangan antar bangsa (Djarsanto 1997). Soehadji (1990) menyatakan bahwa praktek program pemuliaan pada sapi Bali pada tahun 1986 melalui Proyek Pengembangan dan Pembibitan Sapi Bali (P3Bali) yang mencangkup Pulau Bali, NTB, NTT, dan Sulawesi Selatan sebagai pusat pembibitan.
Pada tahun 2013 upaya dalam pelestraian sapi Bali melalui keputusan Direktur Jendral Peternakan Nomor:18020/KPTS/PD.420/F2.3/02/201. Pemerintah mengeluarkan pedoman pelaksanaan pembibitan sapi potong asli Indonesia di Pulo Raya untuk sapi Aceh, pulau Sapudi untuk sapi Madura, dan Pulau Nusa Penida untuk sapi Bali. Selain itu melalui surat keputusan menteri pertanian indonesia Nomor:4437/Kpts/SR.120/7/2013 menetapkan Kabupaten Barru sebagai wilayah sumber bibit sapi Bali berbasis peternakan rakyat.
5
Sapi Bali
Sapi Bali yang berasal dari domestikasi banteng sangat dapat beradaptasi dengan lingkungan aslinya. Sapi Bali (Bos-bibos Banteng) merupakan domestikasi banteng liar termasuk famili bovidae, Genus bos dan Sub-Genus bibos (Williamson & Payne 1978). Menurut Hardjosubroto (1994) bahwa Sapi Bali termasuk dalam familia bovidae, Genus bos dan Sub-Genus tersebut adalah Bibos gaurus, Bibos frontalis dan Bibos sondaicus. Sapi Bali memepunyai ciri-ciri khusus antara lain: warna bulu merah bata, tetapi jantan dewasa berubah menjadi hitam, warna putih pada bagian belakang paha, pinggiran bibir atas, dan pada paha kaki bawah mulai tarsus dan carpus sampai batas pinggir atas kuku, bulu pada ujung ekor hitam, bulu pada bagian telinga putih, terdapat garis hitam pada atas punggung (Chamdi 2004).
Sapi Bali termasuk dalam kategori sapi kecil yang memiliki rata-rata tinggi badan 112.55-115.74 cm (Supriyanto et al. 2008), 109.88 cm (Tonbesi et al. 2009), 102.40-108.80 (Soares & Dryden 2011), 106.97 cm (Zurahma & The (2011). Menurut Field (2007) bahwa sapi masuk dalam kategori kecil jika tinggi badan kurang dari 117.22 cm. Ukuran tubuh pada sapi Bali yang kecil kemungkinan besar merupakan upaya adaptasi dalam mempertahankan hidup pada kondisi lingkungan yang merjinal (Talib 2002). Pertambahan bobot badan harian (PBBH) pada sapi Bali berkisar antara 0.05-0.1 kg/ekor/hari pada musim kemarau, sedangkan pada musim penghujan antara 0.2-0.4 kg/ekor/hari (Bahar & Rakhmat 2003). Rata-rata bobot lahir sapi Bali di beberapa daerah seperti Bali adalah 16-18 kg/ekor, Sulawesi Selatan 12-13 kg/ekor, dan Nusa Tenggara Timur 10.5-15 kg/ekor. Salah satu kelebihan sapi Bali adalah performans reproduksi, dimana kemampuan adaptasi reproduksi yang tinggi dibawah lingkungan marjinal membuat sapi Bali banyak diminati. Sapi Bali memiliki rata-rata panjang siklus esterus 21 hari dengan lama bunting berkisar antara 280-290 hari (Toelihere 2002; Purwantara et al. 2012).
Mikrosatelit
6
Mikrosatelit merupakan salah satu penciri genetiik yang ideal untuk analisis genom karena penanda ini bersifat polimorfik tinggi, menyebar secara acak, dan melimpah (Tautz 1993). Dalam kajian genetika populasi mikrosatelit digunakan untuk mendeteksi keragaman genetik, penetapan asal-usul keturunan, penggalian sumber-sumber genetik dan menjadi penanda molekuler penting dalam analisis genetik (Ciampolini et al. 1995). Mikrosatelit juga sering digunakan untuk mempelajari pautan (linkage), pemetaan, analisis populasi, sistem perkawinan dan struktur populasi (Silva et al. 1999). Sejumlah mikrosatelit telah diaplikasikan untuk mendeteksi sejumlah alternatif alel pada lokus genetik spesifik. Alel-alel individu mencerminkan frekuensi yang berbeda antar populasi yang berbeda. Perbedaan yang besar pada alel ini memungkinkan ketersediaan data dasar untuk pendugaan jarak genetik (Bradley et al. 1998). Perbedaan alel yang dihasilkan disebabkan oleh perbedaan jumlah pengulangan basa (Bennet 2000).
3
METODE
Waktu dan Tempat
Peneltian ini dilaksanakan selama empat bulan, yaitu mulai Oktober 2013 sampai Januari 2014. Penelitian ini dilaksanakan di Balai Pembibitan Ternak Unggul Sapi Bali (BPTU Bali) di Bali, Balai Pembibitan Ternak dan Hijauan Makan Ternak (BPT-HMT Serading) di Sumbawa, Nusa Tenggara Timur dan Village Breeding Center Kabupaten Barru Sulawesi Selatan. Laboratorium Genetika Molekuler Ternak, Bagian Pemulian dan Genetika Ternak, Departemen Ilmu Produksi dan Teknologi Peternakan, Institut Pertanian Bogor.
Bahan
Penelitian ini menggunakan sampel darah sapi Bali dari Balai Pembibitan Ternak Unggul Sapi Bali (BPTU Bali) di Bali, Balai Pembibitan Ternak dan Hijauan Pakan Ternak (BPT-HMT Serading) di Sumbawa, Nusa Tenggara Timur dan Village Breeding Center Kabupaten Barru Sulawesi Selatan. Penentuan sampel sapi di ambil 32 ekor di BPTU Bali, 32 ekor di BPT-HMT Serading, dan 31 ekor dari Village Breeding Center Kabupaten Barru sehingga diperoleh keseluruhan 95 ekor.
Alat
7
ProsedurPenelitian
Koleksi Darah dan Ekstraksi DNA
DNA genom sapi Bali diisolasi dari sel darah utuh (whole blood). Darah sapi Bali ditampung menggunakan tabung vaccutainer dari vena jugularis (+ 10 ml) dengan larutan EDTA 10%. Prosedur isolasi DNA berdasarkan pada metode standar phenol-chloroform Sambrook et al. (2001) yang dimodifikasi oleh Andreas et al. (2010). Darah yang telah diawetkan dicuci menggunakan Tris EDTA dengan konsentrasi rendah (low TE). Pada setiap tahap pencucian disentriugasi dengan kecepatan 3000 rpm selama dua menit kemudian diulang sebanyak lima kali dengan tujuan untuk menghilangkan kandungan etanol absolute yang terdapat pada sampel darah yang telah diawetkan dengan menggunakan buffer lisis sel, yaitu 40 µ l SDS 10%, 10 µl Proteinase-K 5 mg/ml, dan 1 x STE sampai 400 µ l. Larutan di goyang pelan-pelan dalam inkubator pada suhu 55 oC selama 2 jam. DNA dimurnikan dengan menambahkan 200 µ l larutan phenol 400 µl CIAA Cloroform Isoamil Aiconaol : 24:1 dan 40 µl SM NaCl diperoleh dievaluasi melalui elektroforesis 1,5% gel agarose selama kurang lebih 45 menit dengan voltase 100 V. Selanjutnya divisualisasikan dengan Ultraviolet (UV) Transiluminator. Konsentrasi DNA dihitung menggunakan Nanodrop Spectrophotometer.
Amplifikasi lokus mikrosatelit
8
Tabel 1 Lokus mikrosatelit, posisi di kromosom, sekuens primer, dan panjang alel Lokus Posisi di
Data penelitian ini dianalisis menggunakan program POPGEN1.2 (Yeh et al.1999) dan Cervus (Kalinowski et al. 2007). Data karakteristik genetik yang diamati yaitu frekuensi genotipe, jumlah alel (na), alel efektif (ne), frekuensi alel, keseimbangan Hardy-Weinberg (HW), Heteozigositas pengamatan (Ho), Heterozigositas harapan (He), laju inbreeding dalam populasi (Fis), nilai keragaman (Fst), dan pholymorpism informative content (PIC). Jarak genetik di dapat dari frekuensi alel dengan menggunakan software POPTREE2 (Takezaki et al. 2010). Dendogram menggunakan metoda UPGMA (Unweighted Pair Group Method with Arithmetic Mean) dengan bootsrap 1000 kali.
4
HASIL DAN PEMBAHASAN
Amplifikasi dan Genotyping Empat Lokus Mikrosatelit
9
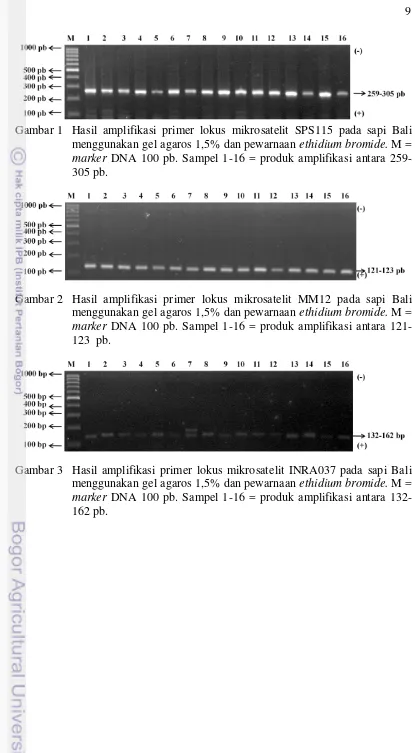
Gambar 1 Hasil amplifikasi primer lokus mikrosatelit SPS115 pada sapi Bali menggunakan gel agaros 1,5% dan pewarnaan ethidium bromide. M = marker DNA 100 pb. Sampel 1-16 = produk amplifikasi antara 259- 305 pb.
Gambar 2 Hasil amplifikasi primer lokus mikrosatelit MM12 pada sapi Bali menggunakan gel agaros 1,5% dan pewarnaan ethidium bromide. M = marker DNA 100 pb. Sampel 1-16 = produk amplifikasi antara 121-123 pb.
10
Gambar 4 Hasil amplifikasi primer lokus mikrosatelit ETH185 pada sapi Bali menggunakan gel agaros 1,5% dan pewarnaan ethidium bromide. M = marker DNA 100 pb. Sampel 1-16 = produk amplifikasi antara 230- 256 pb.
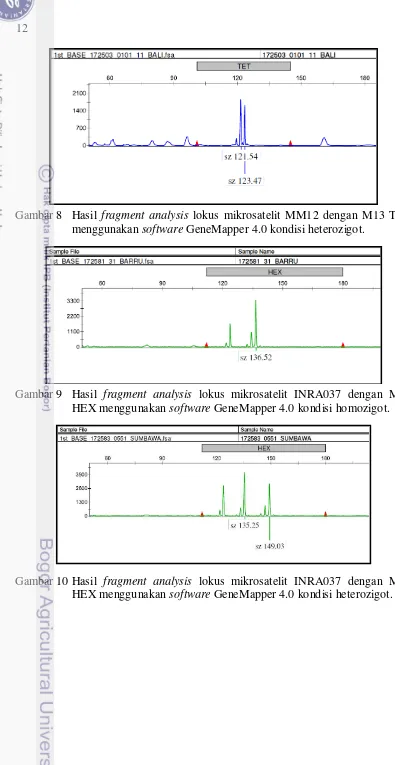
Hasil fragment analysis menampilkanke empat lokus mikrosatelit semuanya polimorfik di pusat pembibitan, yang berarti semua lokus mikrosatelit memiliki alel lebih dari satu. Nei dan Kumar (2000) menyatakan bahwa suatu alel pada populasi dikatakan polimorfik jika memiliki lebih dari satu alel dengan frekuensi lebih dari 1%. Hasil fragment analysis menunjukkan kondisi homozigot pada SPS115 (Gambar 5), MM12 (Gambar 7), INRA037 (Gambar 9), dan ETH185 (Gambar 11). Hasil fragment analysis yang menunjukkan kondisi heterozigot pada ke empat lokus mikrosatelit disajikan SPS115 (Gambar 6), MM12 (Gambar 8), INRA037 (Gambar 10), dan ETH185 (Gambar 12).
11
Gambar 5 Hasil fragment analysislokus mikrosatelit SPS115 dengan M13 FAM menggunakan software GeneMapper 4.0 kondisi homozigot.
Gambar 6 Hasil fragment analysis lokus mikrosatelit SPS115 dengan M13 FAM menggunakan software GeneMapper 4.0 kondisi heterozigot.
12
Gambar 8 Hasil fragment analysis lokus mikrosatelit MM12 dengan M13 TET menggunakan software GeneMapper 4.0 kondisi heterozigot.
Gambar 9 Hasil fragment analysis lokus mikrosatelit INRA037 dengan M13 HEX menggunakan software GeneMapper 4.0 kondisi homozigot.
13
Gambar 11 Hasil fragment analysis lokus mikrosatelit ETH185 dengan M13 NED/TAMRA menggunakan software GeneMapper 4.0 kondisi homozigot.
Gambar 12 Hasil fragment analysis lokus mikrosatelit ETH185 dengan M13 NED/TAMRA menggunakan software GeneMapper 4.0 kondisi heterozigot.
Frekuensi Alel Lokus Mikrosatelit
14
Hasil analisis menunjukkan pada lokus SPS115 ukuran alel berkisar antara 259 sampai 305 pb, sedangkan menurut FAO (2011) berkisar 234-258 pb, dengan demikian semua alel melebihi kisaran FAO. Ukuran alel lokus MM12 yang terdeteksi pada penelitian ini berkisar antara 121-123 pb, sedangkan menurut FAO (2011) berkisar 101-145 pb, dengan demikian semua alel yang ditemukan ukurannya sesuai dengan FAO (2011). Pada lokus INRA037 ukuran alelnya berkisar 132-162 pb, sedangkan menurut FAO (2011) berkisar 112-148 pb, sehingga terdapat lima alel yang berbeda yaitu alel I (150 pb), K (154 pb), L (156 pb), dan M (162 pb). Ukuran alel lokus ETH185 menunjukkan kisaran 230-256 pb, sedangkan pada rekomendasi FAO (2011) berkisar 214-246 pb, sehingga hanya terdapat satu alel yang melebihi yaitu alel F (256 pb). Total terdapat 16 alel pada sapi Bali di tiga pusat pembibitan sapi Bali yang berbeda dengan rekomendasi FAO (2011). Hal ini kemungkinkan besar merupakan alel baru yang belum pernah terdeteksi dari sapi di populasi sebelumnya. Sumantri et al. (2006) menyatakan bahwa alel berbeda yang ditemukan dipopulasi yang berbeda merupakan alel baru yang belum pernah terdeteksi pada sapi di populasi yang lain.
15 Tabel 2 Jumlah frekuensi alel empat lokus di tiga pusat pembibitan sapi Bali
Lokus Ukuran bp
Keterangan: *Alel Spesifik; n: Jumlah sampel yang teramplifikasi
16
Nilai Heterozigositas dan Keseimbangan Hardy-Weinbreg
Nilai heterozigositas merupakan cara paling akurat dalam mengukur variasi genetik, sedangkan keragaman genetik dalam populasi diukur dengan rata-rata heterozigositas jika lokus yang diamati lebih dari satu lokus (Nei dan Kumar 2000). Hasil analisis (Tabel 3) nilai heterozigositas rata-rata pada masing-masing pusat pembibitan sapi Bali termasuk rendah berkisar 0.34 sampai 0.46, sesuai dengan pendapat Javanmard et al. (2005) bahwa nilai heterozigositas menunjukkan keragaman rendah jika kurang dari 0.50.
Tabel 3 menunjukkan bahwa populasi BPTU sapi Bali merupakan populasi yang memiliki nilai heterozigositas rata-rata tertinggi di antara populasi yang lain. Nilai heterozigositas rata-rata di BPTU sapi Bali 0.46 dengan nilai heterozigositas rata-rata harapan 0.34. Hasil analisis menunjukkan BPTU sapi Bali memiliki keragaman genetik yang tinggi pada lokus SPS115 dan INRA037. Lokus INRA037 memiliki nilai heterozigositas paling tinggi (0.70) di BPTU sapi Bali sedangkan lokus mikrosatelit yang memiliki nilai heterozigositas terendah lokus MM12 (0.09). Tabel 3 di BPT-HMT Serading dan Village Breeding Center Kabupaten Barru tampak bahwa nilai heterozigositas rendahdi empat lokus mikrosatelit karena di bawah nilai heterozigositas harapan. Adapun nilai heterozigositas paling tinggi ditemukan pada lokus SPS115 dan nilai heterozigositas terendah pada lokus MM12. Rendahnya nilai heterozigositas pengamatan dibandingkan nilai heterozigositas harapan merupakan indikasi adanya derajat endogami (perkawinan dalam kelompok) dampak dari proses seleksi yang sangat intensif (Machado et al. 2003).
Tabel 3 Keragaman genetik sapi Bali di tiga pusat pembibitan sapi Bali
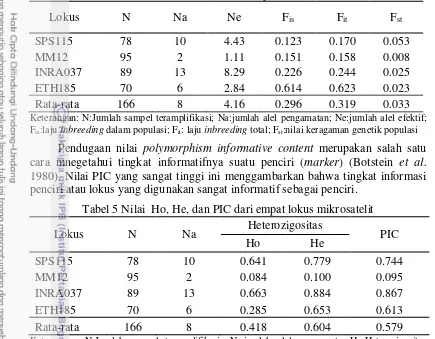
Populasi Lokus N Na Ne Ho He AS HW
Keterangan: N:Jumlah sampel yang teramplifikasi; Na:jumlah alel pengamatan; Ne:alel yang efektif; Ho:Heterozigositas pengamatan; He:Heterozigositas harapan; AS:Alel Spesifik; HW:keseimbangan Hardy-Weinberg; ns: not significant; *P<0.05
17 populasi BPT-HMT Serading dan Village Breeding Center Kabupaten Barru menunjukkan keadaan ketidakseimbangan frekuensi genotipe pada lokus ETH185. Tambasco et al. (2003) menyatakan dalam keadaan ketidakseimbangan frekuensi genotipe jika terjadi perbedaan yang besar antara nilai heterozigositas pengamatan (Ho) dan nilai heterozigositas harapan (He).
Nilai heterozigositas yang ditemukan pada sapi Bali pada penelitian ini termasuk kategori rendah dibandingkan sapi di negara berkembang lainnya, seperti (He=0.73) pada sapi lokal Cina (Zhangetal et al. 2007) dan sapi lokal Ethiopia (Dadi et al. 2008). Sapi Eropa berkisar 0.61-0.71 (Canon et al. 2001) dan menyerupai pada (He=0.4) sapi Japannese Black (Kim et al. 2002). Berdasarkan hasil tersebut (Tabel 3) bahwa populasi sapi Bali di tiga pusat pembibitan sapi Bali memiliki nilai heterozigositas yang rendah di empat lokus mikrosatelit. Hal ini dikarenakan keterbatasan jumlah sampel sapi Bali yang sedikit.
Hasil analisis keragaman genetik pada sapi Bali pada tabel 3, semua populasi pada lokus SPS115 dan MM12 berada dalam kesimbangan Hardy-Weinberg. Pada lokus INRA037 dan ETH185 hanya pada populasi BPTU sapi Bali Provinsi Bali yang berada pada keseimbangan Hardy-Weinberg. Sedangkan pada populasi BPT-HMT Serading, NTB dan VBC Kabupaten Barru menyimpang dari keseimbangan keseimbangan Hardy-Weinberg. Pusat pembibitan sapi Bali yang berada dalam keseimbangan Hardy-Weinberg berarti pada populasi tersebut tidak terjadi seleksi, migrasi, inbreeding, dan genetic drift. Menurut Lan (2013) faktor yang mempengaruhi keseimbangan Hardy-Weinberg adalah inbreeding, genetic drift, dan seleksi. Suatu populasi dinyatakan dalam keadaan keseimbangan Hardy-Weinberg secara konstan dari generasi ke generasi jika frekuensi genotipe (p2, 2pq dan q2) dan frekuensi alel (p dan q), karena akibat penggabungan gamet yang terjadi secara acak dalam populasi yang besar. Sebaliknya jika terjadi akumulasi gonotipe, populasi yang terbagi, mutasi, seleksi, migrasi dan perkawinan dalam kelompok/populasi yang sama (endogami) maka dapat menimbulkan terjadinya ketidakseimbangan frekuensi genotipe atau alel dalam populasi tersebut (Falconer & Mackay 1996).
Laju inbreeding dan Keragaman Genetik Sapi Bali
18
Keterangan: N:Jumlah sampel teramplifikasi; Na:jumlah alel pengamatan; Ne:jumlah alel efektif; Fis:laju inbreeding dalam populasi; Fit: laju inbreeding total; Fst:nilai keragaman genetik populasi
Pendugaan nilai polymorphism informative content merupakan salah satu cara mnegetahui tingkat informatifnya suatu penciri (marker) (Botstein et al. 1980). Nilai PIC yang sangat tinggi ini menggambarkan bahwa tingkat informasi penciri atau lokus yang digunakan sangat informatif sebagai penciri.
Tabel 5 Nilai Ho, He, dan PIC dari empat lokus mikrosatelit
Lokus N Na Heterozigositas PIC
Keterangan: N:Jumlah sampel teramplifikasi; Na:jumlah alel pengamatan;Ho:Heterozigositas pengamatan; He:Heterozygositas harapan; PIC: polymorphisme informative content
Seperti terlihat pada Tabel 4 nilai polymorphism informative content (PIC) pada penelitian ini berkisar antara 0.095-0.867 dengan rataan 0.579. Lokus mikrosatelit INRA037 merupakan lokus mikrosatelit dengan nilai PIC 0.867 tertinggi dengan jumlah alel teramplifikasi tertinggi 13 alel. Pada tabel 5 lokus SPS115 memiliki nilai PIC 0.744 lebih baik dari pada lokus ETH185 (0.613) dan lokus MM12 (0.095). Hal ini berarti bahwa penciri yang paling informatif digunakan pada sapi Bali adalah lokus INRA037 dan lokus MM12 memiliki tingkat informasi genetik yang rendah pada sapi Bali.
Tabel 6 Nilai PIC dari empat lokus mikrosatelit beberapa bangsa sapi
19 Lokus mikrosatelit yang digunakan pada penelitian ini memiliki tingkat polimorfisme dan PIC tinggi pada sapi lokal dinegara lain, seperti Vietnam, pada lokus INRA037 (NA=10, PIC=0.72), SPS115 (NA=14, PIC=0.80), ETH185 (NA=17, PIC=0.86), MM12 (NA=15, PIC=0.76) (Lan et al. 2013). Polimorfisme juga dilaporkan pada lokus SPS115 (NA=6, PIC=0.60) pada sapi Yugoslav Pied oleh Stavanovic et al. (2010) di Serbia. Berdasarkan nilai PIC yang diperoleh, lokus SPS115, INRA037, dan ETH185 merupakan penciri lokus mikrosatelit yang informatif untuk digunakan pada sapi Bali, sedangkan lokus MM12 kurang informatif digunakan pada sapi Bali.
Jarak Genetik Sapi Bali
Hasil analisis jarak genetik dan pohon genetik yang didasarkan atas frekuensi alel pada masing-masing populasi memperlihatkan jarak genetik terdekat antara populasi BPTU sapi Bali dan Village Breeding Center Kabupaten Barru (0.0556), sedangkan jarak genetik antara BPTU sapi Bali dan BPT-HMT Serading merupakan yang terjauh (0.0898). Nilai jarak genetik pada tiga populasi menunjukkan pula kedekatan karakteristik genetik populasi sapi Bali antar pusat pembibitan sapi Bali. Berdasarkan hal tersebut dapat dijelaskan bahwa kedekatan antara sapi Bali di BPTU sapi Bali dan Village Breeding Center Kabupaten Barru dikarenakan penggunaan semen dari penjantan BBIB Singosari yang mana merupakan pejantan terbaik dari BPTU sapi Bali. Analisis jarak genetik dilakukan untuk mengetahui secara jelas perbedaan perbedaan karakteristik antar pusat pembibitan sapi Bali di Indonesia. Berdasarkan data jarak genetik, dibuat rekonstruksi pohon genetik (fiogenetik) pada setiap pusat pembibitan sapi Bali sebagaimana disajikan pada Gambar 13.
Tabel 7 Jarak genetik antar pusat pembibitan sapi Bali
Populasi BPTU
VBC Kabupaten Barru 0.0556 0.0732
Gambar 13 Diagram filogenetik tiga populasi pusat pembibitan sapi Bali Berdasarkan data jarak genetik, dibuat rekonstruksi pohon genetik (filogenetik) pada setiap populasi pusat pembibitan sapi Bali. Hasil analisis menggunakan empat lokus mikrosatelit mampu memisahkan antar populasi sapi Bali di BPTU Bali, VBC Kabupaten Barru Sulawesi Selatan, dan BPT-HMT Serading Sumbawa, Nusa Teggara Barat. Hal ini di gambarkan juga pada pohon genetik (filogenetik) (Gambar 13), perbedaan karakteristik genetik diantara tiga
BPTU Bali
VBC Barru
BPT-HMT Serading
20
populasi pusat pembibitan sapi Bali. Pada Gambar 13 populasi sapi Bali di pusat pembibitan BPTU sapi Bali dan VBC Kabupaten Barru membentuk cluster tersendiri, yang berarti secara karakteristik VBC Barru memiliki kemiripan dengan BPTU sapi Bali. Diagram pohon genetik (filogenetik) menunjukkan populasi sapi Bali di BPTU Bali dan VBC Kabupaten Barru, Sulawesi Selatan memiliki kedekatan secara genetik dibandingkan populasi BPT-HMT Serading Sumbawa, Nusa Tenggara Barat. Hal ini mengidentifikasikan kemungkinan adanya jalur migrasi sapi Bali dari BPTU sapi Bali ke Kabupaten Barru melalui jual beli atau pengiriman pejantan.
Strategi Pengembangan dan Pelestarian Sumberdaya Genetik Sapi Bali
Sumberdaya genetik ternak merupakan salah satu aset penting sekaligus strategis yang dimiliki suatu negara. Pengelolaan sumberdaya genetik ternak secara berkelanjutan akan berdampak kepada ketersediaan pangan pada saat ini dan masa akan datang. Indonesia merupakan salah satu negara yang kaya akan sumberdaya genetik ternak. Sapi Bali pada penelitian ini merupakan sumberdaya genetik ternak yang sangat penting dan strategis menyangkut kebutuhan akan daging sapi di Indonesia. Sapi Bali sebagai sumberdaya genetik ternak juga sangat berkontribusi besar pada survivabilty dan kesejahteraan petani. Pemanfaatan dan upaya pelestarian pada sapi Bali merupakan bagian yang tidak dapat dipisahkan. Penyebaran sapi Bali di beberapa daerah menjadikan perbaikan mutu genetik harus berlandaskan keunikan karakteristik genetik pada setiap populasi sapi Bali. Diperlukan langkah-langkah yang tepat dan strategis dalam perbaikan mutu genetik sapi Bali, sehingga menghasilkan sapi Bali dengan keunggulan berbeda di setiap populasi. Indentifikasi keragaman genetik di populasi sapi Bali penghasil bibit dengan menggunakan penciri genetik mikrosatelit merupakan satu langkah evaluasi dan dokumentasi untuk strategi pengembangan sumberdaya genetik ternak.
Langkah pertama adalah strategi konservasi di populasi-populasi sapi Bali khususnya daerah panghasil bibit sapi Bali. Konservasi sapi Bali merupakan upaya dalam menjaga keragaman genetik di setiap populasi sapi Bali. Keragaman genetik di dalam populasi merupakan kunci spesies dalam mempertahankan hidup.Menjaga keragaman genetik sapi Bali dalam jangka waktu panjang akan bermanfaat pada keberadaan sapi Bali itu sendiri. Menurut Noor (2012) salah satu cara melakukan konservasi sumberdaya genetik ternak lokal adalah dengan menjaga level gen yang memiliki potensi dalam adaptasi serta produktivitas dan juga kombinasi gennya untuk tujuan pemanfaatan heterosis guna mengatasi fenomena melandainya kemajuan seleksi (selection gain plateau) termasuk di dalamnya pertimbangan kultural, penelitian, serta keamanan pangan. Terdapat langkah-langkah dalam menjaga keragaman genetik sapi Bali yaitu, melakukan inventarisasi untuk melakukan penelitian lebih lanjut dan monitoring terhadap status sapi Bali khususnya di pusat pembibitan sapi Bali. Upaya karakterisasi genetik sumberdaya genetik ternak melalui empat langkah dokumentasi, evaluasi, pengembangan program pemuliaan, dan konservasi (Turner 1981).
21 beberapa alel spesifik di masing-masing populasi pusat pembibitan. Perbedaan karakteristik genetik tergambar pada dendogram pohon filogenetik. Pusat pembibitan sapi Bali merupakan lokasi yang sangat penting dalam upaya pengembangan sekaligus pelestarian sapi Bali.
Berdasarkan hasil penelitian di atas, diperlukan program konservasi secara in situ yaitu konservasi dilakukan di lingkungan ternak tersebut. Besarnya biaya merupakan kendala selama ini dalam melakukan konservasi in situ di peternakan rakyat. Kendala biaya dalam konservasi in situ dapat diatasi dengan menggunakan pusat-pusat pembibitan di bawah kontrol pemerintah menjadi lokasi konservasi. Proses konservasi secara in situ pada Village Breeding Center Kabupaten Barru Sulawesi Selatan harus memiliki kelembagaan yang kuat sehingga semua regulasi terkontrol ditataran kelompok ternak. Strategi pengembangan melalui sistem tertutup dapat diaplikasikan untuk meningkatkan mutu genetik dengan tetap menjaga kemurnian sapi Bali. Metode Close nucleus scheme/close breeding program sangat tepat dilakukan pada pusat pembibitan sapi Bali di Indonesia. Skema close breeding program mengharuskan pusat pembibitan akan memberlakukan aturan tidak memasukkan sapi dari luar populasi. Close breeding program merupakan salah satu model yang dapat diterapkan dalam menjaga keunikan dan kemurnian sapi Bali di masing-masing pusat pembibitan. Inventasirasi meliputi penentuan jumlah pejantan dan betina dalam perkawinan, silsilah, data-data fenotipik serta produksi dan reproduksi penting dilakukan secara berkala di pusat-pusat pembibitan sapi Bali, sehingga dapat menunjang asosiasi penciri genetik dan identifikasi keunggulan sapi Bali di masing-masing populasi.
Strategi pengembangan sapi Bali kedepan diharapkan, setiap pusat pembibitan sapi Bali memiliki keunggulan masing-masing. Inventarisir data fenotipe, produksi, dan reproduksi yang akurat akan mendukung kajian genetik serta program pemuliaan yang terarah dalam meningkatkan mutu genetik sapi Bali tanpa menghilangkan keunikan sapi Bali pada setiap pusat pembibitan sapi Bali. Keunggulan dan karakteristik yang berbeda di setiap pusat pembibitan dapat menjadi rujukan distribusi sapi Bali ke beberapa daerah di Indonesia. Pejantan terbaik dari pusat pembibitan dapat dimanfaatkana untuk produksi semen dan digunakan untuk perkawinan dengan ternak sapi Bali di daerah-daerah pengembangan sapi Bali di Indonesia.
SIMPULAN DAN SARAN
Simpulan
22
Saran
Informasi hasil penelitian keragaman mikrosatelit pada sapi Bali dapat digunakan untuk mendeteksi keragaman sapi Bali berdasarkan demografi. Pada penelitian menunjukkan keragaman genetik sapi Bali di pusat pembibitan sapi Bali di Indonesia dengan menggunakan empat primer lokus mikrosatelit. Penelitian lanjut pada populasi sapi Bali yang tersebar di seluruh Indonesia perlu dilakukan menggunakan pendekatan mikrosatelit dengan jumlah populasi dan primer lokus mikrosatelit yang lebih beragam.
DAFTAR PUSTAKA
[FAO] Food and Agriculture Organization of the United Nations. 2011. Molecular Genetic Characterization Of Animal Genetic Resources. ISAG-FAO recommended microsatellite markers Hal. 68-69
Almaida SEM, Machado MSN, Stegleder CS, Gama CL, Huzt MH, Henkes MH, Moraes JCF, and Weimer TDA. 2000. Genetic diversity in a Brazilian bovine herd based on four microsatellite loci. Genet and Mol Biol. 23(2): 347-350
Barker JSF. 1994. A global protocol for determining genetic distances among domestic livestock breeds. In: Procceding of the 5th World congres on Genetic Applied to Livestock Produktion, Guelph and Ontario, Canada 21: 501-508
Beja-Pereira A, Alexandrino P, Bessa I, Carretero Y, Dunner S, Ferrand N, Jordana J, Laloe D, Moazami-Goudarzi K, Sanchez A, Canon J. 2003. Genetic characterization of southwestern Europe bovine breeds: a historical and biogeographical reassessment with a set of 16 microsatellites, J. Hered. 94: 234-250
Botstein D, White RL, Skolnick M, Davis RW. 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. Am. J. Hum. Genet. 32: 314–331
Bradley DG, Loftus RT, Cunningham P, MacHugh DE. 1998. Genetics and
Canon J, Alexandrino, P, BessaI, Carleos C, Carretero Y, Dunner S, Ferran N, Garcia D, Jordana J, Laloe D, Pereira A, Sanchez A, Moazami-Goudarzi, K. 2001. Genetic diversity measures of local European beef cattle breeds for conservation purposes. Genet.Sel. Evol. 33: 311–332
Chamdi A N. 2005. Karateristik sumberdaya genetik ternak sapi Bali (Bos-bibos banteng) dan Alternatif pola konservasinya. Biodiversitas. Vol 6. 1: 70-75. Ciampolini RK, Moazami-Goudarzi D. Vaiman C, Dillman E, Mazzanti JL, Foulley H, Levezeil, dan D Cianci. 1995. Individual multilocus genotypes using microsatellite polymorphisms to permit the analysis of the genetic variability within and between Italian beef cattle breeds. J Anim. Sci. 73: 3259-3268.
23 Departemen Pertanian. 2006. Peraturan Menteri Pertanian nomor 35/Permentan/OT.140/8/2006, Pedoman Pelestarian dan Pemanfaata Sumberdaya Genetik Ternak. Deptan, Jakarta.
Direktorat Jendral Peternakan dan Kesehatan Hewan. 2011. Pendataan Sapi Potong, Sapi Perah, dan Kerbau 2011(PSPK 2011). Kementrian Pertanian Republik Indonesia.
Djadrsanto. 1997. Kebijakan pelestarian ternak asli Indonesia dalam rangka mendukung pengembangan pembibitan ternak nasional. Prosiding seminar nasional dan Veteriner.Pusat Penelitian dan Pengembangan Peternakan. 182-185
Domestic Cattle Origin. Evolutionary Anthropology. Willey-Liss, Inc. hlm. Ellegren H. 2004. Microsatellite: Simple sequences with complex evolution:
Nature Reviews. Genetics 5: 435-445
Falconer DS, Mackay TFC. 1996. Introduction to Quantitative Genetics> Fourth Ed. Longman Inc. New York
Field TG. 2007. Beef Production and Management Decisions. Edisi ke-5. New Jersey (US): Pearson Prentice Hall.
Guichoux E. Lagache, L. Wagner S. Chaumeil P. Leger P. Lepais O. Lepoittevin, C. Malausa T. Revardel, E. Salin, F. Petit, J.R. 2011. Current trends in microsatellite genotyping. Molecular ecology resources 11: 591-611 Handiwirawan, E. Noor, R.R, Muladno, Schuler, L. 2003. The use of HEL9 and
INRA035 microsatellite as specific markers for bali cattle. Arch Tierz., Dummerstorf 46, vol 6: 503-512.
Hardjosubroto W. 1994. Aplikasi pemuliaan ternak di lapangan. Jakarta. PT Gramedia Widiasarana Indonesia.
Hart DL, Clark AG. 1997. Principle of Population Genetic. Sundarland (GB): Sinauer.
Hilmia N. 2013. Karakterisasi fenotipe dan potensi genetik serta hubunganya dengan produktivitas dan kualitas daging sapi lokal Ciamis Jawa Barat. Javanmard A, Asadazadeh N, Banabazi MH, Tavakolian J. 2005. The allele and
genotype frequencies of bovine pituitary spesific transcription factor and leptin genes in Iranian cattle and buffalo population using PCR-RFLP. Iranian J Biotechnol. 3: 104-108
Kalinowski ST, Taper ML, Marshall TC. 2007. Revising how the computer program CERVUS accommodates genotyping error increases succes in paternity assignment. Mol. Eco. 16: 1099-1006.
Kelkar YD, Tyekucheva S, Chiaromonte F, Makova KD. 2008. The genome-wide determinants of human and chimpanzee microsatellite evolution. Genome Research, 18: 30–38.
Kim KS, Yeo JS, Choi CB. 2002. Genetic diversity of north-east Asian cattle based on microsatellite data. Anim. Genet. 33: 201-204.
Kimberly AS, and Robert JT. 2006. Microsatellite for ecologist: a practical guide to using and evaluating microsatellite markers. Ecology letters 9: 615-629 Kusdiantoro M, Mia O. Helena TAVT, Sofia M, Bart HV, Goran A, Heriberto
24
Lan DP, Duy ND, Nguyen TB, Le QN, Nguyen VB, Tran TT, Tran XH, Vu CC, Haja N, Kadarmideen. 2013. Assessment of genetic dviversity and structure of Vientnam indigineus cattle population by microsatellite. Livestcok Sicenc 155: 17-22.
Lehman T, Wiliam AH, Collins FH. 1996. An evolutionry constrains on microsatellite loci using null allels. J. Genet. 144: 1155-1167.
Levinson G, and Gutman A. 1987. Slipped-strand mispairing: a major mechanism for DNA sequence evolution. Mol Biol Evol. 4: 203-221.
Machado MA, Schuster I, Martinez ML, Campos AL. 2003. Genetic diversity of four breed using microsatellite marker. Rev Bras. De Zool. 32:93-98 Maskur, Arman C, Sumantri, Gurnadi E, and Muladno. 2012. A novel singel
nucleotide polymorphism in exon 4 of insulin-like growth factor-1 associated with production traits in bali cattle. Media peternakan Agustus 2012: 96-101.
Maskur, Muladno, Tappa B. 2007. Identifikasi menggunakan marker mikrosatelit dan hubungan dengan sifat kuantitatif pada sapi. Media Peternakan Desember 2007: 147-155.
Muladno. 2006. Aplikasi teknologi molekuler dalam upaya peningkatan produktivitas hewan. pelatihan teknik diagnostik molekuler untuk peningkatan produksi peternakan dan perikanan di kawasan timur Indonesia. Kerjasama Pusat Studi Ilmu Hayati, Lembaga Penelitian dan Pemberdayaan Masyarakat Institut Pertanian Bogor dan Direktorat Jenderal Pendidikan Tinggi Depdiknas, Bogor.
Namikawa, T., Matsuda Y, Kondo K, Pangestu B, Martojo H. 1980. Blood Groups and Blood Protein Polymorphisms of Different Types of the Cattle in Indonesia. The Origin of Phylogeny of Indonesian Native Livestock (Report by Grant-in-Aid for Overseas Scientific Survey, No. 404315). Bogor :. The Research Group of Overseas Scientific Survey. 35-45.
Naoya I, Akiko T, Kazunori M, Haruka T, Mayumi S, Yasushi M, Takashi H, Tomohito I, Toshio W, Kent MR, Warren MS, Steven MK, Craig WB, Gary LB, Yoshikazu S. 2004. A Comprehensive genetic map of the cattle genom based on 3802 microsatellites. Genome Res. 14: 1987-1998.
National Research Council. 1983. Little known asian animals with a promising economic future. Wasington D.C National Academic Press.
Nei M, Kumar S. 2000> Molecular Evolution and Phylogenetics. New York (US): Oxford Univ Press.
Nei M, Tajima F, Tateno Y. 1983. Accuracy of estimated phylogenetic trees from molecular data. J. Mol. Evol. 19: 153-170.
Nijman IJ, Otsen M, Verkaar ELC, de Ruijter C, Hanekamp E, Ochieng JW, Shamshad S, Rege JEO, Hanotte O, Barwegen MW, Sulawati T, Lenstra JA. 2003. Hybridation of banteng (Bos javanicus) and zebu (Bos indicus) revealed by mitochondrial DNA, satellite DNA, AFLP and microsatellites. Heredity 90: 10-18.
Noor RR. 2012. Konservasi sumberdaya genetik ternak lokal.pemikiran guru besar IPB. Merevolusi Revolusi Hijau. IPB Press Bogor.
25 Payne WJA & Rollinson DHL. 1976. Madura cattle. Z. Tier.Züchts. 93: 89-100. Pelt-Verkul van E, Belkum van A, Hays JP. 2008. Primsiples and Technical
Aspects of PCR Amplification. Netherlands (NL): Springer.
Purwantara B, Noor RR, Anderson G, Rodriguez-Martines H. 2012. Banteng and bali cattle in Indonesia: status and forecasts. Reprod Dom Anim. 47(1):2-6, doi:10.1111/j.1439-0531.2011.01956.
Řehout V, Hradecka E, Citek J. 2006. Evaluation of parentage testing in the Czech population of Holstein cattle. Czech J. Anm. Sci. 51: 503-509.
Rollinson DHL. 1984. Bali Cattle In: Evolution of Domestication Animal Mason, I.L. editor. New York: Longman.
Sambrook J, Fritsch F, Miniatis T. 1989. Molecular Cloning Laboratory Manual. 3rd Edition. New York (US): Cold Spring Harbor Laboratory Press.
Schuelke M. 2000. An economic method for the fluorescent labeling of PCR fragments. Nature America Inc. http://biotech.nature.com.
Shaffer ML, 1981. Minimum population sizes for spesies conservation. BioScience. 31: 131-134.
Soares, FS, Dryden GMcL. 2011.A body condition scoring system for Bali cattle. Asian-Aust. J Anim. Sci. 24(11): 1587- 1594
Soehadji W. 1990. Kebijakan pemuliaan ternak (breeding policy) khususnya sapi Bali, dalam pembangunan peternakan. Proceeding Seminar Nasional Sapi Bali. Fakultas Peternakan Universitas Udayana. Hlm A-1 A-9
Stevanovic J, Stanimirovic Z, Dimitrijevic V, Maletic M. 2010. Evaluation of 11 microsatellite loci for their use in paternity testing in Yugoslav Pied cattle (YU Simmental cattle). J Anim. Sci.Vol 6: 221-226.
Sumantri C, Perwitasari D, Farajalah A, dan Anggraeni A. 2006. Keragaman DNA mikrosatelii sapi Friesian Holstein (FH) di Balai Pembibitan Ternak Unggul (BPTU) sapi perah Baturaden. Seminar Nasional Teknologi Peternakan dan Veteriner 2006.
Supriyantono A, Hakim L, Suyadi, Ismudiono. 2008. Performansi sapi bali pada tiga daerah di Provinsi Bali. Berk Penel. Hayati 13: 147-152.
Takezaki N, Nei M, Tamura K. 2010. POPTREE2: software for constructing population trees from allele frequency data and computing other population statistics with windows interface. Mol Biol. Evol. 27: 747-752. Talib C, Entwistle K, Siregar A, Budiarti-Turner S, Lindsay D. 2002. Survey of
Population and Production Dynamics ofBali Cattle and Existing Breeding Programs in Indonesia. Di dalam: Entwistle K, Lindsay DR, editor.
Proceedings of a Workshop “Strategies to Improve Bali Cattle in Eastern
Indonesia”. 3-9
Talib C. 2002. Sapi Bali di daerah sumber bibit dan peluang pengembangannya. Wartazoa 12(3): 100-107.
Tambasco DD. 2003. Candidate genes for growth traits in beef cattle crosses Bos Taurus x Bos indicus. J. An. Breed. Genet. 120:51.
26
Tonbesi TT, Ngadiyono N, Sumadi. 2009. Estimasi potensi dan kinerja sapi bali di Kabupaten Timor Tengah Utara, Provinsi Nusa Tenggara Timur. Buletin Peternakan Vol. 33(1): 30-39.
Turner HN. 1981. Animal genetic resources. Int Goat and Sheep Res. 1(4):243 Viljoen GJ, Nel LH, Crowther JR. 2005. Molecular Diagnosis PCR Handbook.
Netherlands (NL): Springer.
Winaya A, Muladno, Gurnadi RE, Saefuddin A. 2011. Y chromosome microsatellites variation in bali cattle (Bos sondaicus) population. Animal Production 11 (3): 149-154.
Xiaojuan Z, Zhaohui Z, Zhaofeng H, Pingping T, Runlin Z. 2007. Microsatellite genotpying for four expected inbred mouse strain from km mice. Science Direct 34(3): 214-222.
Yeh FC. 1999. POPGENE version 1.32, the user-friedly shaware for population genetic analysis. Molecular Biology and Biotechnology Center. University of Alberta, Canada.
Zhang GX, Wang ZG, Chen WS, Wu CX, Han X, Zan LS, Li RL, Wang JH, Song WT, Xu GF, Yang HJ, Luo YF. 2007. Genetic diversity and population structure of indigenous yellow cattle breeds of China using 30 microsatellite marker. Anim Genet. 38, 550-559.
27
RIWAYAT HIDUP
28
Lampiran 1 Genotipe yang muncul dari empat lokus mikrosatelit di tiga pusat pembibitan