KARAKTERISASI
GENOM MlTOKONDRlA
LABI-LABI,
Dogania subplana
(TRIONYCHIDAE, TESTUDINES, REPTILIA)
ACHMAD FARAJALLAH
PROGRAM PASCASARJANA
INSTITUT PERTANIAN BOGOR
ABSTRAK
ACHMAD FARAJALLAH (985098). Karakterisasi genorn rnitokondria labi-labi, Dogania subplana, (Trionychidae, Testudines, Reptilia). Komisi pernbirnbing: Prof.Dr. Nawangsari Sugiri, Prof. Osarnu Takenaka, D.Sc., Barnbang Suryobroto, D.Sc., dan Dr. M.F. Rahardjo.
Runutan nukleotida lengkap genorn rnitokondria (rntDNA) labi-labi, Dogania subplana, telah dilakukan. Ukuran rntDNA labi-labi adalah 17289 bp. Organisasi, orientasi dan ukuran setiap gen dari 13 gen penyandi protein rntDNA, 22 gen penyandi tRNA dan dua gen penyandi rRNA serta daerah kontrol adalah serupa dengan yang telah diternukan pada vertebrata lainnya. Panjang daerah kontrol adalah 1820 bp, yang di dalarnnya bisa diternukan tiga motif runutan DNA berulang. Motif pertarna dan kedua berturut-turut adalah 15 bp dan 37 bp, yang keduanya berulang secara tandem sarnpai 728 bp. Motif ketiga adalah ruas TA berulang, yaitu (TA)n dan (ATAlT)n. Motif ketiga ini disebut rnikrosatelit
rnitokondria. Analisis terhadap daerah kontrol rnenernukan adanya domain tengah yang stabil untuk sernua anggota Testudines, dan adanya tiga macarn Conserve Sequence Blocks yang hornolog dengan daerah kontrol rntDNA vertebrata.
Berdasarkan domain tengah daerah kontrol, keragarnan nukleotida D. subplana di lndonesia cukup tinggi (0.21%). Selain itu, penyebaran labi-labi di lndonesia rnengikuti penyebaran vikarian. Populasi
0.
subplana Surnatera Bagian Selatan mernpunyai afinitas yang tinggi ke populasi Jawa dibanding ke populasi Bengkulu. Trionychidae merupakan kelornpok yang rnonofiletik berdasarkan analisisparsirnoni dan neighbourjoining rnenggunakan gabungan data runutan nukleotida
Cyt b, tRNA Phe, tRNA Pro dan domain tengah daerah kontrol rntDNA.
ABSTRACT
ACHMAD FARAJALLAH (985098). Characterization of mitochondrial genome of the soft-shelled turtle, Dogania subplana, (Trionychidae; Testudines; Reptilia). Advisory committe: Prof.Dr. Nawangsari Sugiri, Prof. Osamu Takenaka, D.Sc., Bambang Suryobroto, D.Sc., and Dr. M.F. Rahardjo.
Complete nucleotide sequence of Malayan soft-shelled turtle, Dogania subplana,
mitochondrial genome (mtDNA) was determined. The total length of the sequence was 17289 bp. The organization, orientation and individual gene size of 13
protein-coding genes, 22 tRNA genes, two rRNA genes (1 2 s and 16s rRNA), and the control region were identical to the typical vertebrate arrangement. The control region is 1820 bp long that contains three repeat sequences. The first and the second repeated motifs were 15 bp and 37 bp long, repectively, and spanned 728 bp. The third repeat sequence consisted of two motifs which are (TA)n and
(ATATT)n; they are named as mitochondrial microsatellites. Analysis of this control region permitted the identification of the middle conserved domain of all
Testudines, and three types of Conserve Sequence Blocks, which have high homology to vertebrate mtDNA. Nucleotide diversity of the middle conserved domain of D. subplana in Indonesia is relatively high (0.21%). Based on the nucleotide sequences, D. subplana is shown to be vicariantly distributed in Indonesia. D. subplana population of South Sumatra has high affinity to Java compared to Bengkulu. Trionychidae as a monophyletic group was supported by parsimony and neighbor joining analyses using combined sequence data of Cyt
b,
tRNA Phe, tRNA Pro, and the middle conserved domain of mtDNA.SURAT PERNYATAAN
Dengan ini saya menyatakan bahwa disertasi yang berjudul:
"Karakterisasi genom mitokondria iabi-labi, Dogania subplana (Trionychidae,
Testudines, Reptilia)" adalah benar karya saya sendiri dan belum pernah dipublikasikan. Semua sumber data dan informasi yang digunakan telah
dinyatakan secara jelas dan dapat diperiksa kebenarannya.
Bo or, Septe ber 02
KARAKTERISASI GENOM MlTOKONDRlA
LABI-LAB1
,
Dogania
subplana
(TRIONYCHIDAE, TESTUDINES, REPTILIA)
oleh
ACHMAD FARAJALLAH
Disertasi
sebagai salah satu syarat untuk memperoleh gelar
Doktor pada
Program Studi Biologi
PROGRAM PASCASARJANA
INSTITUT PERTANIAN BOGOR
-
s . d
- ;,.*- 'em - -
Kamkterisasi Genorn M t o k o n d r i a b b b i 4 a b i ; - ~ ~ ~ i a aUbp/ana , (Trionychibae; Testudines; Reptilia)
Nama~ ' : Achmad Farajallah
..
,NRP- -:985098
P : ,
m Studi: Bidogi
n -
&Ay?-
*;i
- -4-, .;.'
Menyetujui, 1. Komisi Pembimbing
,i
-
,
Pmf.Dr. NdG;anasan
' - .
' Suairi
--; ' L .
, - * -
Prof. Osamu Takenak.
D.Sc,
AnggotaNama : Achmad Farajallah
Tempat, Tanggal Lahir : Sampang
-
Madura, 27 April 1965Pekerjaan : Dosen di Jurusan Biologi, Fakultas Matematika dan IPA, lnstitut Pertanian Bogor
Alamat Kantor : Laboratorium Zoologi Jurusan Biologi FMlPA IPB
Gedung Zoologi dan Biokimia Lantai II Kampus Gunung Gede
Jalan Raya Pajajaran Bogor 16143 : Kawin
Status Perkawinan Riwayat Peke jaan
1989
1990
-
sekarangRiwayat Pendidikan 1972
-
1984 1984-
1 985 1985-
1 989 1991-
1995: Calon Pegawai Negeri di Jurusan Biologi, FMlPA IPB
: Dosen di Jurusan Biologi, Fakultas Matematika dan IPA, IPB
: SD, SMP dan SMA di Sampang Madura
: Tingkat Persiapan Bersama IPB
: S1, Program Studi Biologi, FMlPA IPB : S2, Program Studi Biologi
Program Pascasa rjana IPB : S3, Program Studi Biologi
KATA PENGANTAR
Tulisan ini berisi hasil penelitian tentang keragaman genetik labi-labi, Dogania subplana, di Indonesia berdasarkan runutan nukleotida genom
mitokondria, dan filogeni anggota ordo Testudines. Penelitian dilaksanakan untuk menyusun disertasi sebagai syarat memperoleh gelar Doktor dalam bidang studi Biologi di Program Pascasarjana lnstitut Pertanian Bogor. Untuk itu, penulis
sampaikan terima kasih yang mendalam kepada komisi pembimbing, yaitu Prof.Dr. Nawangsari Sugiri, Prof. Osamu Takenaka D.Sc., Bambang Suryobroto, D.Sc. dan Dr. M.F. Hahardjo atas segala kesabaran, ketelitian dan dukungannya selama penulis menempuh program doktor. Terima kasih khusus kepada Prof. Osamu Takenaka, D.Sc. yang telah memperkenalkan penulis ke pendekatan molekular terbaru untuk menganalisis biodiversitas hewan. Dalam kesempatan ini pula,
penulis sampaikan terima kasih kepada Dr. Muladno dan Dr. Jatna Supriatna atas komentar dan koreksinya.
Selain itu, penulis sampaikan terima kasih yang mendalam kepada Bapak
.
Go Bang, A Cun, Benny Prakasa Putra, Kasmirudin M.Si., Nasarudin M.Si, QodriMadang M.Si., Dra. Titisari Puntorini dan Haerul Azhar atas kerjasama yang saling menguntungkan dalam menangkap dan memperoleh sampel labi-labi dan
kura-kura. Penulis menikmati interaksi yang hangat dan saling mendukung dari rekan sejawat, staf teknisi, staf administrasi dan para mahasiswa di Department of Molecular and Cellular Biology, Primate Research Institute U niversitas Kyoto dan d i
Laboratorium Zoologi FMlPA IPB, yang kalau disebutkan satu persatu terlalu banyak, terima kasih.
Untuk isteri, anak-anak dan ibu, terima kasih atas kesabaran dan dukungannya.
DAFTAR IS1
RIWAYAT HlDUP KATA PENGANTAR DAFTAR IS1
DAFTAR TABEL DAFTAR GAMBAR DAFTAR LAMPIRAN DAFTAR SINGKATAN
BAB 1. PENDAHULUAN UMUM Klasifikasi Kura-kura Labi-labi
Posisi Filogeni Kura-kura dalam Amniota Distribusi, Filogeni dan Sistematika Kura-kura Karakteristik Genom DNA Mitokondria Vertebrata Tujuan Penelitian
BAB 2. BAHAN DAN METODE
Penentuan Runutan Lengkap Nukleotida mtDNA D. subplana
Keragaman Daerah Kontrol mtDNA Labi-labi, D. subplana, di Indonesia
Filogeni Kura-kura di lndonesia Berdasarkan Runutan Cyt b, tRNA Pro, tRNA Thr dan Domain Tengah dari Daerah Kontrol
BAB 3. RUNUTAN NUKLEOTIDA LENGKAP GENOM MlTOKONDRlA LABI-LABI, DOGANIA SUBPLANA
Pendahuluan Hasil
Pembahasan Kesimpulan
BAB 4. KERAGAMAN DNA DAERAH KONTROL LABI-LABI, DOGANIA SUBPLANA, Dl INDONESIA
Pembahasan Kesimpulan
BAB 5. FlLOGENl MOLEKULAR KURA-KURA, LABI-LAB1 DAN PENYU BERDASARKAN RUNUTAN DNA CYT B, tRNA THR, tRNA PRO DAN DOMAIN TENHA DAERAH KONTROL GENOM
MlTOKONDRlA Pendahuluan Hasil
Pembahasan Kesimpulan
DAFTAR PUSTAKA
DAFTAR
TABEL
Nomor Te ks Halaman
1.1 Tabel 1.1. Klasifikasi kura-kura (Gaffney 1975; Hirayama 1984;
Meylan 1987 12
3.1 Karakteristik mtDNA labi-labi, D. subplana 36
3.2 Komposisi nukleotida mtDNA D. subplana (%) 37
3.3 Jumlah penggunaan kodon (huruf cetak tegak) dan nilai indeks
penggunaan kodon (huruf cetak miring) dalam mtDNA D. subplana 38 4.1 Sampel Labi-labi Dogania subplana dan haplotipe mtDNA 77 4.2 Jarak genetik antar haplotipe D. subplana berdasarkan domain
tengah dari daerah kontrol mtDNA (di bawah diagonal) dan standart error (di atas diagonal) yang diduga menggunakan metode
bootstrap dengan 1000 ulangan 74
4.3 Divergensi nukleotida dalam populasi (di diagonal), divergensi nukleotida antar populasi (di bawah diagonal) dan total divergensi
nuleotida antar populasi (di atas diagonal) dari D. subplana 76 5.1 Sampel spesies kura-kura dan ukuran produk PCR (bp)
menggunakan primer 512-576 (Cyt b
-
TAS), primer 577-578 (TAS-
CSBIII) dan primer 512
-
578 935.2 Jarak genetik (di bawah diagonal) menggunakan metode 2 parameter Kimura dan rasio transisi terhadap transversi (di atas diagonal) berdasarkan kombinasi data dari nukleotida pertama dan kedua gen Cyt b, gen tRNA dan domain tergah daerah kontrol
mtDNA 94
5.3 Jarak genetik (di bawah diagonal) menggunakan metode 2 parameter Kimura dan rasio transisi terhadap transversi (di atas
diagonal) berdasarkan ketiga nukleotida setiap kodcn gen Cyt b ' 95
DAFTAR GAMBAR
Nomor Te ks Halaman
1 .I Beberapa hipotesis hubungan filogeni testudines dan anggota
amniota 11
3.1 Peta pemotongan Mbol dan posisi penempelan primer (angka cetak
tebal) dalam mtDNA D. subplana 39
3.2 Peta genetik mtDNA D. subplana 40
3.3 Runutan lengkap nukleotida dan karakteristik mtDNA D. subplana 4 1 3.4 Runutan nukleotida dan struktur sekunder gen-gen tRNA mtDNA D.
subplana 56
3.5 Struktur batang dan gelung dari OnL (titik asal replikasi utas L)
mtDNA D. subplana 59
3.6 Struktur batang dan gelung dari dua motif runutan DNA berulang di
bagian ujung 5' daerah kontrol mtDNA D. subplana 60 3.7 Hubungan filogeni antar anggota amniota berdasarkan kombinasi
runutan nukleotida dari gen-gen penyandi protein (kecuali gen-gen ND3, ND4L dan ND6), penyandi tRNA (kecuali gelung DHU dan
V c )
6 13.8 Hubungan filogeni antar anggota amniota berdasarkan runutan asam amino yang ditranslasikan dari gen-gen penyandi protein
(kecuali gen-gen ND3, ND4L dan ND6) 62
4.1 Peta pengambilan sampel labi-labi D. subplana 77
4.2 Posisi runutan nukleotida yang beragam antar haplotipe mtDNA D.
subplana 78
4.3 Pohon filogeni Neighborjoining antar haplotipe D. subplana 79 4.4 Pohon filogeni yang dihasilkan metode Minimum Evolution (A) dan
UPGMA (B) antar haplotipe D. subplana 80
5.1 Organisasi gen antara Cyt b dan daerah kontrol dalam mtDNA
Testudines 97
5.2 Perbandingan runutan nukleotida gen Cyt b beberapa spesies kura-kura menggunakan ClustalW yang dibantu dengan hasil
translasi ke asam aminonya 98
5.3 Perbandingan runutan nukleotida gen-gen tRNA Thr dan tRNA Pro
5.4 Perbandingan runutan nukleotida domain tengah daerah kontrol dari
mtDNA beberapa spesies kura-kura menggunakan ClustalW 101
5.5 Hubungan filogeni antar spesies kura-kura berdasarkan kombinasi
data dari gen-gen Cyt b, tRNA Thr, tRNA Pro, dan domain tengah
DAFTAR LAMPIRAN
Halaman
Lampiran 1 Daflar kura-kura yang ada di Indonesia 111
Lampiran 2 Peta penyebaran labi-labi, Dogania subplana, di 113 lndonesia
Lampiran 3 Runutan dan perbandingan nukleotida domain tengah dari 114 daerah kontrol pada masing-masing haplotipe mtDNA D.
subplana yang diapit oleh primer 577 dan 578
Lampiran 4 Runutan nukleotida Cyt b, tRNA Thr, tRNA Pro, dan 121 domain tengah dari daerah kontrol genom mitokondria
beberapa spesies kura-kura
DAFTAR SINGKATAN
12Sr RNA
16Sr RNA
ATPase 6
ATPase 8
COI COll COlll Cyt b NDI ND2 ND3 ND4 ND4L ND5 ND6 dloop
tRNA
-
(JA
G
C T
12s ribosomal RNA 16s ribosomal RNA
Adenosine triphosphatase sub unit 6
Adenosine triphosphatase sub unit 8 Cytochrome c oxidase sub unit 1 Cytochrome c oxidase sub unit I1 Cytochrome c oxidase sub unit 111 Cytochrome b apoenzyme
Nicotinamide adenine dinucleotide dehydrogenase sub unit 1 Nicotinamide adenine dinucleotide dehydrogenase sub unit 2 Nicotinamide adenine dinucleotide dehydrogenase sub unit 3
Nicotinamide adenine dinucleotide dehydrogenase sub unit 4
Nicotinamide adenine dinucleotide dehydrogenase sub unit 4L
Nicotinamide adenine dinucleotide dehydrogenase sub unit 5 Nicotinamide adenine dinucleotide dehydrogenase sub unit 6
Displacement loop
Transfer RNA three letter amino acid abbreviation (anticodon) Adenine
BAB I.
PENDAHULUAN UMUM
Kura-kura rnemiliki tengkorak yang masih primitif, kraniurn utuh tanpa lubang
temporal dan tubuhnya berada dalam cangkang. Kura-kura diklasifikasikan seba-
gai ordo Testudines, subklas Anapsida dan Klas Reptilia (Emst & Barbour 1989).
Cangkang kura-kura terdiri atas dua bagian yang saling berhubungan, yaitu kara-
pas di sisi atas dan plastron di sisi bawah. Pada urnurnnya, cangkang kura-kura
terbuat dari bahan tanduk yang keras. Walaupun begitu, cangkang pada bebera-
pa taksa bersifat lunak, rnisalnya semua anggota farnili Trionychidae.
Dalarn tulisan ini, narna kura-kura secara urnurn rnerujuk pada semua ang-
gota ordo Testudines. Narna penyu rnerujuk pada kura-kura yang hidup di laut,
yaitu farnili Cheloniidae dan Derrnochelyidae. Sedangkan narna labi-labi rnerujuk
pada semua anggota farnili Trionychidae, yaitu kura-kura yang hidup di air tawar
dan bercangkang lunak.
Klasifi kasi Kura-kura
Klasifikasi ordo Testudines tingkat farnili ke atas rnengikuti Gaffney (1 975)
dan untuk farnili labi-labi rnengikuti Meylan (1987) (Tabel 1 .A). Ordo Testudines
dibagi rnenjadi dua subordo, yaitu Cryptodira dan Pleurodira. Subordo Cryptodira
rnemiliki leher yang bisa ditarik rnasuk ke dalarn cangkang di antara gelang bahu,
sedangkan subordo Pleurodira merniliki leher yang tidak bisa ditarik ke dalam
cangkang. Subordo Cryptodira dibagi rnenjadi tiga superfarnili, yaitu Testudinoi-
Trionychidae. Lebih lanjut Meylan (1 987) membagi Famili Trionychidae menjadi
dua subfamili, yaitu Cyclanorbinae dan Trionychinae.
Famili Emydidae dibagi menjadi dua subfamili oleh Hirayama (1 984), yaitu
Emydinae dan Batagurinae. Walaupun begitu, Hirayama (1 984) menyimpulkan
bahwa pengelompokan Emydinae dan Batagurinae dalam Emydidae dalam
pandangan kladistik bukan sebagai kelompok alami, yang keduanya mempunyai
daerah penyebaran yang sangat berbeda. Emydinae, baik fosil maupun yang
hidup sekarang, mempunyai daerah penyebaran di Eropa dan Amerika, sedany-
kan semua anggota Batagurinae mempunyai daerah penyebaran di Asia kecuali
genus neotropika, yaitu Rhinoclemmys (Ernst & Barbour 1989).
Pada saat ini ada 22 spesies labi-labi di dunia. Penyebarannya meliputi
daerah beriklim tropis sampai ke subtropis (Meylan 1987; Ernst & Barbour 1989;
lverson 1992). Nama lokal untuk setiap spesies labi-labi beragam antar daerah
(lampiran 1).
Cangkang labi-labi berbentuk pipih dorsoventral. Karapas dan plastron
labi-labi berupa kulit yang lunak. Sambungan keduanya juga lunak, yang ditunjang
oleh struktur pertulangan yang mirip dengan tulang perifer pada anggota famili
yang lain. Selain itu, labi-labi mempunyai bentuk tubuh yang memungkinkannya
untuk menggali dan membenamkan diri di dasar perairan tawar. Sebagian besar
anggota labi-labi jarang keluar dari air. Di dalam air, mereka sangat gesit dan
tersebut memungkinkan mereka bertindak sebagai karnivora (Ernst & Barbour 1989; Pritchard 1993; Lim & Das 1999).
Labi-labi melayu, Dogania subplana, merupakan spesies dengan ukuran yang paling kecil di antara semua labi-labi yag ada di Asia (Pritchard 1993). Pe-
nyebaran D. subplana meliputi seluruh wilayah Asia Tenggara, mulai dari bagian barat Thailand, Malaysia, Singapura sampai ke Sumatera, Kalimantan dan Jawa
(Ernst & Barbour 1989; lverson 1992; Lim & Das 1999). Peta penyebaran D. subplana di Indonesia yang dibuat oleh lverson (1992) disajikan dalam Lampiran 2.
Dogania subplana memiliki kepala yang besar dengan rahang yang kuat; tulang maksila bersinggungan dengan tulang frontal pada bagian sisi anterior orbit;
panjang tulang postorbital kurang dari seperlima diameter orbit; bagian posterior
foramen jugularis memanjang membentuk lengkung opisthotic yang berada di luar lubang postotic; tulang pterigoid bersinggungan dengan foramen newus trigemini
di antara tulang apipterigoid dan tulang parietal; epiplastron memanjang ke arah
anterior melebihi lebar hyohypoplastron; ada empat kalus pada plastron; tulang neural membagi sama panjang semua tulang pleural di posisi tengah; dua tulang
neural terbalik ditemukan pada posisi paling posterior dari sembilan tulang neural;
Posisi Filogeni Kura-kura dalam Amniota
Pengelompokan amniota berdasarkan kondisi lubang pada tulang temporal
diajukan oleh Laurin & Reisz (1995) sebagai berikut:
1 Anapsida: tulang temporal tidak ada lubang, yaitu pada kura-kura.
2. Diapsida: tulang temporal mempunyai dua lubang di sisi atas dan bawah, yaitu
pada arkosauria (krokodilia dan burung) dan lepidosauria (kadal, ular dan
.
Sphenodon).3. Sinapsida: tulang temporal mempunyai satu lubang di sisi bawah yang dibatasi paling tidak oleh jugal, postorbital dan skuamosal, yaitu pada mamalia.
4. Euryapsida: tulang temporal mempunyai satu lubang di sisi atas yang dibatasi
oleh parietal, postfrontal, postorbital dan skuamosal. Kelompok ini hanya
ditemukan pada reptilia jaman Mesozoikum dan Permian Bawah.
Sampai saat ini, hubungan-hubungan filogeni dari anggota amniota masih
belum disepakati, begitu juga dengan posisi kura-kura dalam amniota. Secara
luas, kura-kura dianggap berada pada posisi pangkal dari semua anggota amniota
karena kondisi anapsida dari tulang temporalnya dianggap sebagai kondisi yang
lebih primitif dibanding kondisi diapsida (Benton 1990). Gambar 1 . I memperli-
hatkan beberapa hipotesis filogeni amniota (disarikan dari Zardoya & Meyer 1998 dan Cao et a/. 2000). Menghadapi ketidakpastian filogeni amniota di atas, Wilkin-
son et a/. (1997) menyarankan pendekatan molekular. Walaupun begitu, hubung-
an filogeni akan muncul beragam tergantung pada kumpulan taksa yang dianalisis.
Hal itu berkaitan dengan ketersediaan data molekular yang sampai saat ini paling
banyak berasal dari mamalia, sedangkan yang berasal dari burung dan reptilia
data yang lebih ekstensif akan menghasilkan hubungan filogeni yang lebih bisa
dipercaya. Dengan begitu, runutan lengkap nukleotida dari genom mitokondria
(mtDNA) labi-labi melayu,
0.
subplana,
yang disajikan dalam Bab 3 diharapkan bisa menambah data yang cukup untuk memperjelas hubungan-hubungan filogeniamniota dan menempatkan posisi kura-kura pada posisi filogeni yang lebih tepat.
Distribusi, Filogeni dan Sistematika Kura-kura
Dari 260 spesies kura-kura yang sudah dikenal di dunia, 85 spesies di
antaranya bisa ditemukan di Asia (Ernst & Barbour 1989; lverson 1992) dan 39
spesies di antaranya bisa ditemukan di Indonesia (Lampiranl). Hampir semua
spesies yang ada di Asia Tenggara miskin informasi berkenaan dengan status
populasi, pola penyebaran, perikehidupan dan hubungan filogeninya (Jenkins
1995; Barzyk 1999). Barzyk (1 999) melaporkan bahwa beberapa spesies baru
berhasil dideskripsikan bukan dari spesimen yang diperoleh dari habitat aslinya,
melainkan dari pasar hewan hias. Bersamaan dengan kurangnya informasi
tersebut, saat ini terjadi penurunan populasi kura-kura yang drastis akibat perburu-
an liar yang dipicu oleh harga pasar yang tinggi. Beberapa penyebab penurunan
populasi lainnya adalah polusi perairan, fragmentasi habitat, kerusakan habitat dan
perubahan lingkur~gan akibat introduksi spesies tanaman dan hewan baru. Di sisi
yang lain, berbagai informasi biologi diperlukan sebagai pijakan dalam manajemen
konsewasi.
Sampai saat ini, penggunaan data molekular untuk mempelajari sistematika
dan filogeni kura-kura jauh tertinggal dibanding mamalia dan burung. Kalaupun
.
kan pada perspekstif yang sangat terbatas, misalnya pada tingkat proteinlisozim(Sites et a/. 1984), karyotipe (Bickham & Carr 1983), serologi (Frair 1985) dan DNA mitokondria (Lamb & Lydeard 1994; Walker et a/. 1995; Walker et a/. 1997;
Seddon et a/. 1997; Lenk et a/. 1999; Weisrock & Janzen 2000).
Dari catatan penemuan fosil, diperkirakan bahwa kura-kura pertama kali
muncul sekitar 200 juta tahun yang lalu, sedangkan famili Trionychidae muncul
pertama kali sekitar jaman Kreta Awal (sekitar 146 juta tahun yang lalu) (Meylan
1987). Meylan (1987) menunjukkan bahwa Trionychidae merupakan salah satu
famili dari 11 famili kura-kura lainnya yang fosilnya ditemukan secara kontinyu
sejak jaman Senozoikum di daerah Barat Daya Amerika. Walaupun begitu, posisi
filogeni famili Trionychidae terhadap anggota famili Testudines yang lain masih
tidak jelas berkenaan dengan daerah penyebarannya yang sangat luas, kelim-
pahannya yang tinggi dan umurnya yang relatif tua.
Berdasarkan hasil analisis morfologi oleh Gaffney (1 975), Trionychidae
merupakan famili yang unik diantara kriptodira. Disebutkan pula bahwa Trionychi-
dae justru berkerabat dekat dengan Carettochelyidae, Dermatemydidae, dan
Kinostemidae, yang kemudian dikelompokkannya menjadi satu superfamili baru,
yaitu Trionychoidea. Berbeda halnya dengan Bickham & Carr (1983) dan Frair (1985) yang mengajukan bahwa Trionychidae mempunyai hubungan yang erat
dengan Carettochelyidae dibanding dengan famili yang lain. Meylan (1 987)
berpendapat bahwa konsep Trionychoidea oleh Gaffney adalah lebih untuk
menjernihkan kontroversi yang menyebutkan bahwa Kinosternidae dan Chelydri-
dae lebih dekat ke Emydidae dan Testudinidae. Konsep Trionychoidea juga
a/. 1997). Walaupun begitu, data runutan nukleotida mtDNA kurang bisa menjelas-
kan hubungan antar anggota kriiptodira secara keseluruhan (Shaffer et al. 1997).
Kurangnya resolusi data morfologi secara parsial untuk menjelaskan hubung-
an filogeni anggota Trionychidae ditunjukkan dengan adanya kecenderungan yang
kuat memasukkan semua labi-labi ke dalam satu genus Trionyx, kecuali Chitra and
Pelochelys. Smith & Smith (1 980 dalam Meylan 1987) menghitung sebanyak 47 nama genus yang bisa dianggap sebagai sinonim. Berdasarkan data morfologi
yang ekstensif dalam rangka revisi besar-besaran terhadap semua anggota
Trionychidae, Meylan (1 987) mengusulkan pengelompokan anggota Trionychidae
menjadi empat klad, yang kemudian memunculkan klasifikasi Trionychidae yang
baru (Tabel 1.1). Lebih lanjut Meylan (1987) memberi alasan bahwa klasifikasi
baru itu bukan karena adanya perbedaan karakter morfologi, melainkan karena
perbedaan interpretasi sistematik dari sudut pandang yang menyeluruh.
Di Indonesia ada empat spesies labi-labi yang menyebar secara alami, yaitu D. subplana, Amyda cartilaginea, Pelochelys canton' dan Chitra indica (Tabel 1.1,
Lampiran I ) , dan satu spesies labi-labi introduksi, yaitu Pelodiscus sinensis (Lim &
Das 1999).
Hubungan filogeni anggota Batagurinae juga masih belum terpecahkan.
Berdasarkan kesamaan morfologi tengkorak, Batagurinae diklasifikasikan menjadi
empat kompleks spesies, yaitu kompleks spesies Hardella (Hardella, Morenia dan
Geoclemys), kompleks spesies Batagur (Batagur, Kachuga, Callagur, Ocadia,
Hieremys, Chinemys dan Malayemys), kompleks spesies Orlifia (Orlitia dan
Siebenrockiella) dan kompleks spesies Geoemyda (Geoemyda, Cyclemys,
Sacalia) (McDowell 1964). Hubungan Batagurinae seperti di atas tidak disepakati oleh Gaffney (1 975) dan Hirayama (1 984) berdasarkan data morfologi yang meli-
batkan fosil dan jumlah karakter yang lebih banyak, dan juga oleh Bickham & Carr
(1983) berdasarkan karyotipe. Hirayama (1 984) mengusulkan adanya dua jalur
evolusi di antara Batagurinae. Jalur evolusi pertama adalah kura-kura yang memi-
liki kehidupan lebih ke daratan yang diwakili oleh Geoemyda dan Testudinidae. Jalur kedua adalah kura-kura yang memiliki kehidupan lebih ke air yang dicirikan
dengan adanya langit-langit mulut sekunder yang ekstensif seperti yang ditemukan
Batagur dan Hardella.
Karakteristik Genom DNA Mitokondria Vertebrata
Sampai saat ini, runutan lengkap nukleotida genom mitokondria (mtDNA)
veAebrata telah dilaporkan untuk lebih dari seratus spesies, yang pada umumnya
mamalia (database di GenBank, EMBL
-
European Molecular Biology Laboratories, DDBJ-
DNA Database Bank of Japan ). Sedangkan untuk reptilia, runutan leng- kap nukleotida mtDNA telah dilaporkan hanya pada delapan spesies, yaitu Alligator missisippiensis (Janke & Arnason 1997), Pelomedusa subrufa (Zardoya & Meyer1998), Dinodon semicarinatus (Kumazawa et a/. 1998), Chrysemys picta (Mindell et a/. 1999), Chelonia mydas, Eumeces egregius (Kumazawa & Nishida 1999),
Iguana iguana dan Caiman crocodylus (Janke et a/. 2001).
Molekul mtDNA memiliki banyak kelebihan sebagai penanda molekular untuk
mempelajari hubungan evolusi hewan pada berbagai tingkatan. Hal ini disebabkan
oleh ukuran mtDNA vertebrata relatif kecil (sekitar 16000 bp) yang mengandung 13
menyandikan rRNA (ribosomal RNA) dan satu ruas DNA berukuran besar yang tidak menyandikan. Selain itu, mtDNA vertebrata dikemas secara ekonomis
yang ditunjukkan dengan tidak adanya intron, beberapa gennya saling tum-
pang tindih, kodon stop beberapa gennya tidak sempurna dan ujung 3'-CCA
dari gen-gen tRNA-nya tidak ada. Pola pewarisan melalui garis ibu yang
menyebabkan tidak ada rekombinasi dan laju mutasinya yang tinggi merupa-
kan keunggulan tersendiri bagi mtDNA sebagai penanda molekular tingkat
intraspesies pada sebagian besar vertebrata (Avise 1994). Penggunaan mtDNA
untuk mempelajari kekerabatan intraspesies maupun antarspesies pada beberapa
spesies kura-kura telah dilakukan Avise et al. (1992), Lamb & Lydeard (1994), Walker et a/. (1995), Walker et al. (1 997), Seddon et al. (1 997), Lenk et a/. (1 999)
dan Weisrock & Janzen (2000).
Dalam Bab 4 dibahas sejarah populasi dan kekerabatan genetik D. subplana di Indonesia berdasarkan keragaman nukleotida domain tengah dari daerah kontrol
mtDNA. Primer yang diperlukan untuk memperbanyak ruas DNA secara in vitro
didesain berdasarkan runutan nukleotida yang diperoleh dalam Bab 3. Selain itu,
beberapa primer yang lain juga didesain untuk memperbanyak ruas mtDNA labi-
labi dan kura-kura yang ada di Indonesia. Ruas mtDNA itu digunakan untuk
Tujuan Penelitian
1. Menentukan runutan lengkap nukleotida dan mengkarakterisasi mtDNA
labi-labi (D. subplana).
2. Menerangkan posisi kura-kura dalam filogeni amniota.
3.
Mempelajari keragaman intraspesies labi-labi, B. subplana menggunakanpenanda molekular mtDNA.
4. Mempelajari keragaman antarspesies labi-labi dan hubungan filogeninya
Lepidosauria
A
Archosauria
Lepidosauria
B
Testudines
u
Testudines
Lepidosauria
Archosauria
Lepidosauria
Testudines
[image:26.572.66.495.69.722.2]Mammal ia
Archosauria
Gambar 1 .I. Beberapa hipotesis hubungan filogeni testudines dan anggota amniota (disarikan dari Zardoya & Meyer 1998 dan Cao et a/. 2000).
A. Testudines adalah kelompok terdekat dari diapsida.
B. Testudines berada di dalam diapsida. Dalam ha1 ini testudines membentuk satu klad dengan lepidosauria.
C. Testudines berada di dalam diapsida. Dalam ha1 ini testudines membentuk satu klad dengan arkosauria.
D. Hipotesis bahwa testudines sebagai parareptilia, yaitu kondisi tulang temporal testudines sebagai karakter yang paling primitif.
Tabel I .I. Klasifikasi kura-kura (Gaffney 1975; Hirayama 1984; Meylan 1987)
p k l a s Anapsida
I
Ordo Testudines1
Subordo Cryptodira
semua kura-kura yang lehernya bisa ditarik ke dalam cangkang diantara kedua gelang bahu
Superfamili Testudinoidea Famili Chelydridae Famili Emydidae Subfamili Emydinae Subfamili Batagurinae Famili Testudinidae Superfamili Chelonioidea Famili Cheloniidae Famili Dermochelyidae Superfamili Tri~nychoidea Famili Dermatemydidae Famili Kinosternidae Famili Carettochelyidae Famili Trionychidae Subfamili Cyclanorbinae Tribe Cyclanorbini Tribe Lissemydini Subfamili Trionychinae Tribe Chitrini Tribe Aspideretini Tribe Trionychini Tribe Pelodiscini Subordo Pleurodira
semua kura-kura yang lehernya bisa ditekuk ke samping dan tidak bisa dimasukkan ke dalam cangkang. Tulang pelvis kura-kura ini
menyambung ke cangkang dan mereka mempunyai otot penarik rahang yang berpangkal pada penjuluran tulang pterigoid
BAB 2.
BAHAN DAN METODE
Penentuan Runutan Lengkap Nukleotida mtDNA D. subplana
A. lsolasi Molekul mtDNA Murni
Satu spesimen labi-labi, Dogania subplana, yang digunakan dalam penen-
tuan runutan lengkap mtDNA berasal dari Sungai Ciliwung Bogor, Jawa Barat.
ldentifikasi spesies mengikuti Ernst & Barbour (1989).
Molekul mtDNA murni diekstraksi dari hati, ginjal dan otot jantung. Organ-
organ itu diambil dari labi-labi yang sudah dibius (deep anaesthezised) mengguna-
kan kloroform. Hati, ginjal dan otot jantung dicuci dalam larutan NaCl0.76% dingin
untuk membersihkan sisa darah. Setiap organ (50
-
100 mg) secara terpisah dige-rus dalam larutan penggerus (Tris-HCI 30 mM, EDTA 2 mM, sukrosa 0.25 M, pH
8.0). Inti dan sisa sel dalam gerusan dipisahkan dengan sentrifugasi kecepatan
rendah (1000 g, 1 menit). Organel mitokondria dalam supernatan diendapkan de-
ngan sentrifugasi kecepatan tinggi (12000 g, 10 menit) dalam suhu dingin. Kemu-
dian organel mitokondria yang diperoleh dari ketiga organ dicampur menjadi satu.
Ekstraksi molekul mtDNA mengikuti metode lisis alkali (Tamura & Aotsuka 1988). Endapan mitokondria disuspensikan dalam 1 x STE (NaCI 1 M, Tris-HCI
10-I M, EDTA 10-2M, pH 8.0). Ke dalam suspensi ditambahkan NaOH 0.18 N dan
SDS 1 % (sodium dodesil sulfat) sebanyak 2 x volume. Setelah diinkubasi selama 5 menit dalam penangas es, larutan alkali dinetralkan dengan menambahkan
supernatan yang mengandung molekul mtDNA ditangkap dengan perlakuan fenol:
kloroform:isoamil-alkohol (25:24:1) (Sambrook et a/. 1989). Fase DNA yang me-
ngandung mtDNA dipisahkan dari fase fenol dengan sentrifugasi 1000 g selama 5
I
menit, kemudian mtDNA diendapkan dengan menambahkan NaCl5 M sebanyak
1/10 x volume dan etanol absolut sebanyak 2 x volume. Endapan mtDNA dicuci
dengan etanol 70% dingin. Molekul mtDNA murni kemudian disuspensikan dalam
larutan TE encer (Tris-HCI ~ x I O - ~ M, EDTA 2x1 0-4 MI pH 8.0).
B.
Penyiapan Fragmen Mbol Molekul mtDNA: Kloning plasmid pUC118Molekul mtDNA murni dipotong dengan enzim restriksi Mbol. Semua frag-
men kemudian disisipkan ke dalam plasmid pUCl18 pada situs BamHl (TaKaRa).
Plasmid yang sudah disisipi fragmen mtDNA ditransformasikan ke E. coli JM109
(TaKaRa) menggunakan metode kejut panas (Sambrook et al. 1989). Transfor-
man E. coli ditumbuhkan dalam medium padat Luria-Bertani (LB) dalam cawan
petri yang mengandung ampisilin 0.5 mglml, isopropiltiogalaktosidase 2.4% dan
PGalactosidase 2 %, kemudian diinkubasi pada suhu 37 OC selama 14 jam. Beberapa koloni E.coli berwarna putih diinokulasikan secara terpisah ke medium
cair 2YT dalam tabung transformasi (IWAKI), kemudian diinkubasikan pada suhu
37 OCselama 14 jam sambil dikocok secara vertikal pada kecepatan 6000 rpm.
lsolasi DNA plasmid dilakukan menggunakan pengisolasi DNA plasmid
otomatis (KURABO P100) berdasarkan prosedur lisis alkali (Sambrook et a/. 1989).
Keberadaan sisipan fragmen mtDNA dalam DNA plasmid diuji dengan mengapli-
kasikan enzim restriksi EcoRl dan Pstl. Situs pemotongan kedua enzim tersebut
Visualisasi hasil pemotongan dilakukan dengan elektroforesis gel poliakrilamida
5% dalam bufer IxTBE (Tris 17.8 mM, Borat 17.8 mM, EDTA 0.4 mM) yang dilan-
jutkan dengan pewarnaan perak (Tegelstrom 1986).
C. Penentuan Runutan Nukleotida
DNA plasmid pUCl18 yang mengandung sisipan fragmen mtDNA dijadikan
cetakan dalam siklus reaksi penentuan runutan nukleotida menggunakan primer
universal M I 3 yang diberi label floresen FITC. Reaksi itu menggunakan ABI- sequencing kit dengan TaqGold sebagai enzim polimerase yang dijalankan dalam mesin thermal cycle Perkin-Elmer tipe 9600. Runutan nukleotida kemudian dibaca menggunakan automated DNA sequencers Shimadzu DSQ-1 atau Shimadzu DSQ-2000L. Kegiatan penentuan runutan nukleotida ini dilakukan di Department of Molecular and Cellular Biology, Primate Research Institute, Kyoto University.
D.
Penyusunan Runutan Nukleotida Seluruh Genom mtDNARunutan nukleotida dari setiap DNA sisipan dalam plasmid diedit dan disu-
sun membentuk molekul mtDNA utuh. Pengeditan dilakukan dengan mengga-
bungkan hasil perunutan dari beberapa plasmid, baik yang menggunakan primer
M I 3 fonvard maupun reverse. Penyusunan itu dimulai dengan cara mencari kesa- maan antara setiap runutan nukleotida yang diperoleh dengan runutan mtDNA
pembanding, yaitu manusia (Anderson et a/. 1981), katak (Roe et a/. 1985) dan aiigator (Janke & Arnason 1997).
berdekatan. DNA hasil perbanyakan PCR-nya disisipkan ke plasmid pGEM-T easy
Vector (Promega Corp.), ditransformasikan ke E.coli , kemudian dilakukan perunut-
an nukleotida untuk dikaji ulang. Fragmen mtDNA hasil pemotongan Mbol dan
posisi setiap primer yang didesain disajikan dalam Gambar 3.1.
Dalam ha1 DNA sisipan terlalu besar dan melebihi kapasitas pembacaan
Shimadzu DSQ-1 maupun Shimadzu DSQ-2000L, maka dilakukan subkloning.
DNA sisipan diambil dari plasmid, dipotong dengan Alul, Haelll atau Rsal, kemu-
dian disisipkan kembali ke plasmid pUC118 pada situs pemotongan Smal. Tahap-
an selanjutnya sama dengan metode sebelumnya, yaitu perunutan nukleotida.
E. Analisis
DataAnalisis homologi runutan nukleotida genom mtDNA dan karakterisasi
daerah kontrol dan gen-gen penyandi RNA dan protein dilakukan dengan bantuan
Genetyx-Win versi 3.2.0 (Software). Selain berdasarkan kesamaan runutan nuk-
leotida yang tinggi dengan mtDNA pembanding, karakterisasi lebih lanjut untuk
gen-gen penyandi protein dilakukan dengan mencari adanya kodon inisiasi dan
kodon stop, kemudian menterjemahkannya ke dalam asam amino mengikuti
vertebrate mitochondria1 translation code yang ada dalam Genetyx-Win. Sedang-
kan untuk gen-gen penyandi tRNA, karakterisasi lebih lanjut dilakukan dengan
membandingkan struktur sekundernya terhadap struktur sekunder yang dilaporkan
oleh Kumazawa & Nishida (1 993).
Analisis filogeni amniota menggunakan metode maksimum parsimoni (MP)
dengan model min-mini heuristic dan metode neighborjoining (NJ) yang ada dalam
analisis filogeni dilakukan dengan ClustalW (Thompson et a/. 1994) yang kemudi-
an dikoreksi secara manual. Koreksi terhadap gen-gen penyandi protein dilakukan
dengan bantuan hasil translasi ke bentuk asam aminonya, sedangkan terhadap
gen-gen penyandi tRNA dilakukan dengan bantuan struktur sekundernya. Hasil
Keragaman Daerah Kontrol mtDNA Labi-labi,
D.
subplana, di Indonesia
A.
Sampel
D. subplanaSebanyak 52 sampel D. subplana digunakan dalam penelitian ini (Tabel 3.1). Sampel diperoleh dari Jawa (Bogor 23 ekor, Bandung 1 ekor dan Panganda-
ran 1 ekor), Sumatera Bagian Selatan (Lampung 1 ekor dan Palembang 2 ekor),
Bengkulu (Bengkulu Selatan 9 ekor dan Bengkulu Utara 10 ekor) dan Kalimantan
Selatan (Banjarbaru 5 ekor) (Gambar 4.1). ldentifikasi D. subplana mengikuti Ernst
& Barbour (1989) dan Lim & Das (1999). Spesimen sisa disimpan di Jurusan Biologi, Fakultas Matematika dan IPA, lnstitut Pertanian Bogor.
6.
Ekstraksi DNA total
DNA total diekstraksi dari darah atau otot, baik yang masih segar maupun
yang sudah disimpan dalam alkohol70%, mengikuti metode Kan
ef
a/. (1977). Setetes darah disuspensikan ke dalam 1 x STE, kemudian ditambahkan SDS 1 %.Sekitar 50 mg otot direndam dalam larutan 1 x SE (sarkosil 1 % dan EDTA 0.05 M) kemudian dipotong-potong sampai halus. Kedua sumber DNA itu kemudian
dicerna dengan proteinase-K (0.05
-
0.1 mglml) dan diinkubasi pada suhu55OC
selama 2 jam. Bahan organik yang masih tercampur dengan DNA ditangkap
dengan perlakuan fenol:kloroform:isoamil-alkohol (25:24:1). Setelah sentrifugcsi
1000 g selama 5 menit, fase DNA dipisahkan dari fase fenol kemudian dimasukkan
ke dalam kantung dialisis dan dilanjutkan dengan dialisis dalam larutan TE (Tris
I 0-2 M, EDTA M, pH 8.0) selama minimal 5 jam. Kandungan RNA kemudian
M, EDTA
l o 3
M, pH 8.0) selama minimal 5 jam. Kandungan RNA kemudiandibuang dengan menambahkan RNase A (0.05-0.1 mglml).
Domain tengah dari daerah kontrol mtDNA diperbanyak secara in vitro
menggunakan mesin PCR dengan primer 577 (5'- l T C ACG AGA GAT TAA GCA
ACC
-
3') dan primer 578 (5'- CTC GGC TTT TGG GGT l T G AC-
3'). Reaksi PCR dilakukan dalam volume 25 pI dengan komposisi sebagai berikut: sampel cetakanDNA 10
-
100 ng, dNTP 400 pM dNTP, masing-masing primer 25 pmol, MgC12 200pM, dan enzim polimerase TaqGold 0.83 unit (Perkin Elmer) beserta bufernya.
Reaksi PCR dijalankan menggunakan mesin Perkin Elmer 9600 pada kondisi
denaturasi awal 95°C 5 min yang diikuti dengan 94 "C 50 detik, 55 "C 60 detik dan
72 "C 120 detik sebanyak 30 siklus.
Produk PCR dideteksi dengan elektroforesis agarosa 1 % dalam bufer TAE pH 7.3 yang dilanjutkan dengan pewarnaan etidium bromida. Pita tunggal dari
produk PCR dipotong dari gel, kemudian dielusikan dari gel menggunakan Ultra-
Free DA (Microcon-Amicon Corp). Produk PCR yang sudah murni disisipkan ke
pGEM T-easy Vector (Promega).
Perunutan DNA dari setiap kion dilakukan dengan cara yang sama dengan
metode terdahulu.
Perbandingan runutan DNA dilakukan secara manual karena runutan DNA
yang diperoleh mempunyai kesamaan yang sangat tinggi, yang kemudian dicek
lagi dengan perbandingan yang diperoleh menggunakan Genetyx-Win versi 3.0
Filogeni Kura-kura di Indonesia Berdasarkan
Runutan DNA Cyt b, tRNA Thr, tRNA Pro dan Domain Tengah dari Daerah Kontrol
A.
Sampel Kura-kuraSebanyak 10 spesies kura-kura digunakan dalam penelitian ini, yaitu
famili Trionychidae (D. subplana
-
Bogor, A. cartilaginea-
Bengkulu, P. cantori-
Palembang dan C. indica-
Kebun Binatang Ragunan) dan subfamili Batagu-rinae (Cuora amboinensis
-
Banten, Cyclemys dentata-
Banten, Siebenrocki-ella crassicollis
-
Banten, Heosemys grandis-
Palembang, Batagur baska-
Kebun Binatang Ragunan dan Chinemys reevisii
-
Nagoya). ldentifikasispesies mengikuti Ernst & Barbour (1989) dan Lim & Das (1999). Selain itu, runutan DNA homolog juga diambil dari database GenBank, yaitu Chelonia
mydas (Cheloniidae, AB012104), Chrysemys picta (Emydinae, AF069423) dan
Pelomedusa subrufa (Pleurodira, AF039066) (Tabel 5.1).
B. Analisis Runutan DNA
Ruas antara Cyt b dan domain tengah dari daerah kontrol mtDNA
diperbanyak menggunakan primer 51 2 (5'- AGA ATG ATA TTT CCT ATT CGC
CT-3') dan 576 (5'-CTA ACA AGG GTT GCT TAC CTCS'), 577 dan 578.
Pasangan primer 512 dan 489 memperbanyak ruas Cyt b, tRNA Thr, tRNA Pro dan
motif TAS (termination associated sequence), sedangkan primer 577 dan 578
memperbanyak Domain Tengah dari Daerah Kontrol, mulai dari motif TAS sampai
motif CSB
Ill
(consenled segunce block 111). Reaksi dan kondisi PCR sama denganDeteksi produk PCR, elusi pita tunggal produk PCR dari gel dan perunutan
nukleotida dilakukan dengan cara yang sama dengan metode terdahulu.
1
Perbandingan runutan nukleotida untuk analisis filogeni dilakukan
menggunakan Clustal W (Thompson et a/. 1994) yang kemudian dikoreksi secara manual. Analisis filogeni menggunakan metode MP dan NJ yang terdapat dalam
BAB
3.
RUNUTAN NUKLEOTIDA LENGKAP GENOM MlTOKONDRlA
LABI-LABI,
DOGANlA SUBPLANA
Pendahuluan
I
Runutan nukleotida lengkap genom mitokondria (mtDNA) dari ratusan
spesies anggota vertebrata telah dilaporkan. Hampir semua mtDNA vertebrata
mempunyai kandungan gen yang sama, yaitu dua gen penyandi RNA ribosom
(rRNA), 22 gen penyandi RNA transfer (tRNA), 13 gen penyandi protein dan satu
ruas DNA yang tidak menyandikan. Biasanya, ruas DNA yang tidak menyandikan
disebut daerah kontrol atau dloop karena di dalamnya terdapat motif DNA yang
mengatur replikasi dan transkripsi (Anderson et al. 1981 ; Avise 1994). Walaupun
begitu, dilaporkan adanya perpindahan posisi beberapa gen dalam mtDNA, seperti
yang ditemukan pada ayam (Desjardins & Morais 1990), hewan berkantung
(Paabo et a/. 1991) dan beberapa spesies reptilia dan amfibia (Macey et al. 1997;
Yoneyama 1987; Kumazawa & Nishida 1995; Kumazawa et al. 1998). Avise (1994) menyebutkan bahwa mtDNA terbukti bisa digunakan sebagai penanda
genetik yang efektif untuk mempelajari berbagai fenomena populasi sampai ke
hubungan filogeni dan sistematika. Hal itu disebabkan oleh ukurannya yang relatif
kecil dengan laju mutasi tinggi, kandungan gennya relatif stabil dan diwariskan
melalui garis ibu.
Sejak ditemukannya PCR (polymerase chain reaction) dan teknik-teknik
penentuan runutan nukleotida, penggunaan mtDNA sebagai penanda dalam
filogeni itu sendiri. Di lain pihak, pada saat ini banyak dilaporkan adanya perma-
salahan yang muncul yang diakibatkan oleh perpindahan beberapa gen pada
beberapa kelompok taksa (Macey et a/. 1997) dan adanya transposisi mtDNA ke dalam DNA inti (Lopez et a/. 1996; Zhang & Hewitt 1996).
Sampai saat ini, runutan nukleotida lengkap mtDNA dari delapan spesies
Reptilia telah dilaporkan yang terdiri atas tiga spesies Testudines (Chelonia mydas,
chrysemys picta dan Pelomedusa subrufa), tiga spesies skuamata (Dinodon semicarinatus, Eumeces egregius dan Iguana iguana) dan dua spesies krokodilia
(Alligator missisippiensis dan Caiman crocodylus). Runutan nu kleotida leng kap mtDNA itu dipakai untuk mempelajari pentarikhan divergensi molekular antara
krokodilia dan burung (Janke & Arnason 1997), menempatkan kura-kura di antara spesies-spesies arkosauria dan lepidosauria (Zardoya & Meyer 1998; Kumazawa &
Nishida 1999) dan mencari hubungan filogeni amniota yang terpercaya (Janke et a/. 2001).
Bab ini mendeskripsikan runutan nukleotida lengkap genom mtDNA labi-labi
D. subplana. Penentuan runutan nukleotida lengkap mtDNA ini merupakan lang- kah awal untuk mempelajari berbagai fenomena populasi, filogeni dan sistematika
Hasil
Penentuan Runutan Nukleotida Berulang di Daerah Kontrol mtDNA
Runutan nukleotida lengkap mtDNA D. subplana disusun dari runutan
nukleotida beberapa klon yang berasal dari molekul mtDNA yang dibandingkan
dengan mtDNA manusia, Xenopus laevis dan Alligator missisippiensis. Hasil
perbandingan menunjukkan bahwa empat klon yang berasal dari pemotongan
molekul mtDNA dengan Mbol hilang selama proses penyisipan ke plasmid, yaitu
200 bp (posisi 3865
-
4065), 1 1 13 bp (posisi 4065-
51 78), 1070 bp (posisi 6032-
7102) dan 234 bp (posisi 14799
-
15033) (Gambar 3.1). Runutan nukleotida dariklon-klon yang hilang itu kemudian diperoleh dari molekul mtDNA yang
diperbanyak secara in vitro menggunakan PCR.
Klon yang menempati posisi 151 65
-
16363 (Gambar 3.1) mengandungmotif runutan berulang dengan jumlah unit ulangan yang tidak berhasil dirunlrt
semuanya. Setelah perbanyakan PCR menggunakan primer 512 dan 489
dicobakan untuk menetapkan jumlah ulangan, ditemukan bahwa perbanyakan itu
selalu menghasilkan DNA produk PCR dengan panjang yang beragam (multiple
products). Hasil perunutan nukleotida terhadap produk PCR menunjukkan bahwa
setiap DNA produk PCR memiliki perbedaan panjang kelipatan 52 bp. Ukuran 52
bp itu merupakan kornbinasi dari dua motif nukleotida berulang yang ada di daerah
kontrol (Gambar 3.3 dan 3.7). Selain itu, ukuran produk PCR terbesar yang berha-
~ i l menyisip ke dalam plasmid memiliki panjang yang berbeda dengan ukuran klon
'kan jumlah ulangan kemudian digunakan referensi ukuran klon yang berasal dari
molekul mtDNA awal, seperti yang dilakukan oleh Asakawa et al. (1 995).
Besar dan Komposisi Nukleotida mtDNA D. subplana
Ukuran panjang mtDNA D. subplana adalah 17289 bp yang setara dengan panjang mtDNA amfibia dan reptilia lainnya, tetapi sedikit lebih panjang dibanding
dengan mtDNA ikan, burung dan mamalia. Panjang mtDNA ini mencakup tiga
motif DNA berulang di daerah kontrol. Motif DNA berulang pertama berukuran 15
nt (nukleotida), motif kedua berukuran 37 nt dan motif ketiga berupa mikrosatelit
mitokondria, yaitu TA dan ATATT (Gambar 3.3).
Karakteristik mtDNA D. subplana disajikan dalam Tabel 3.1. Posisi nukleo- tida awal dalam runutan mengacu pada posisi ujung 5' dari gen tRNA Phe seperti
yang dilakukan pada spesies kura-kura lainnya (P. subrufa, Zardoya & Meyer 1998; Chelonia mydas, Kumazawa & Nishida 1999; Chrysemys picta, Mindell et a/.
1999).
Komposisi nukleotida utas H (heavy strand) didominasi oleh nukleotida A sebesar 35.3%, kemudian berturut-turut C (26.7%), T (26.0%) dan G (12.0%)
(Tabel 3.2). Komposisi nukleotida A dalam mtDNA D. subplana sedikit lebih besar dibanding P. subrufa dan Alligator. Walaupun begitu, mtDNA reptilia, amfibia, sebagian besar mamalia dan burung mempunyai pola komposisi nukleotida yang
sama, yaitu A>C>T>G.
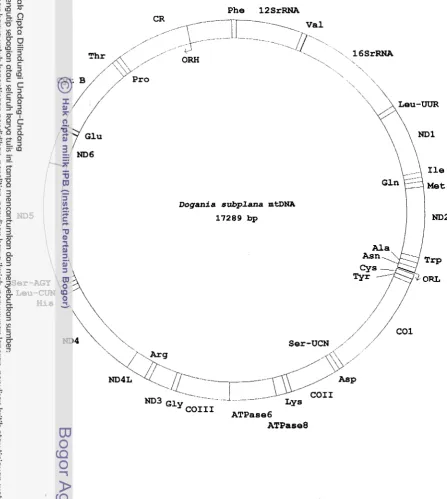
Organisasi, orientasi dan ukuran setiap gen dari mtDNA labi-labi yang
ditemukan dalam penelitian ini sama dengan yang ditemukan pada mtDNA
protein, 22 gen penyandi tRNA, dua gen penyandi rRNA (12s dan 16s rRNA) dan
satu ruas daerah kontrol.
Gen-gen Penyandi Protein Genom D. subplana
0
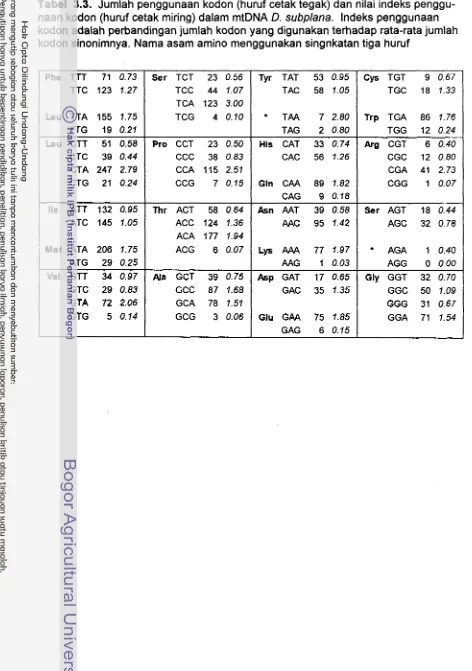
Kode genetik dan penggunaan kodon dari gen-gen penyandi protein pada
D. subplana tidak bisa dibedakan dengan yang ditemukan pada vertebrata lainnya.
Seperti halnya ciri khas gen-gen penyandi protein dalam genom mtDNA, keragam-
an terbesar ditemukan pada nukleotida ketiga dari setiap kodon. Nukleotida ketiga
A paling sering digunakan oleh setiap kodon dari gen yang disandikan utas H
(49.6%) dan nukleotida ketiga G adalah yang paling jarang digunakan (3.5%),
sedangkan nukleotida ketiga C dan T berturut-turut digunakan sebesar 30.2% dan
16.7%. Hal yang sebaliknya ditemukan untuk gen ND6 yang disandikan utas L
(light strand), yaitu kodon dengan nukleotida ketiga T paling sering digunakan
(55.4%), kemudian berturut-turut G (23.2%), A (1 5.3%) dan C (6.2%).
Semua gen penyandi protein menggunakan kodon inisiasi ATG, kecuali
gen COI menggunakan GTG seperti yang umum ditemukan pada mtDNA vertebra-
ta, terutama mamalia. Sedangkan kodon stop yang sering digunakan adalah TAA,
TAG atau TNN, kgcuali AGA yang hanya digunakan oleh gen ND6. kodon stop
TNN digunakan oleh gen-gen yang tumpang tindih dengan gen yang bersebelah-
an. Dalam mtDNA D. subplana tidak ditemukan adanya insersi satu nukleotida yang mengubah aturan kodon triplet (translational frameshifiing), seperti yang
ditemukan dalam gen ND3 dan ND4L pada P. subrufa, Chrysemys picta dan
Gengen Penyandi tRNA mtDNA D. subplana
Gen-gen penyandi tRNA mtDNA D. subplana ditemukan sebanyak 22 jenis Pada umumnya, posisi gen tRNA menyebar di antara berbagai gen penyandi pro-
tein maupun penyandi rRNA, kecuali di antara kelompok gen-gen ATPase 8
-
ATPase 6
-
COlll, ND4L-
ND4, dan ND5-
ND6. Posisi gen tRNA yang menyebaritu bisa dianggap sebagai sinyal pengenalan dalam memproses RNA mitokondria
(Ojala et a/. 1981). Selain itu, beberapa gen penyandi tRNA ditemukan berkelom- pok, yaitu Ile
-
Gin-
Met, Trp-
Ala-
Asn-
Cys-
Tyr dan His-
Ser(AGY)-
Leu(CUN),seperti yang ditemukan pada genom mtDNA yang khas vertebrata.
Ukuran gen-gen penyandi tRNA berkisar antara 62 dan 76 bp. Semua gen
penyandi tRNA dapat dilipat membentuk struktur sekunder berbentuk daun
semanggi (Gambar 3.4). Struktur sekunder yang agak menyimpang ditemukan
pada tRNA Ser(AGY). Gen ini memiliki struktur tanpa lengan DHU (dihidrouridina)
(Gambar 3.4). Runutan nukleotida paling stabil ditemukan pada bagian lengan
antikodon dan batang pembawa asam amino. Di bagian lengan pembawa asam
amino sampai ke gelung DHU dari tRNA Leu(UUR) ditemukan adanya motif
ribosomal RNA transcription termination seperti pada genom mtDNA yang khas vertebrata, sedangkan dalam genom mtDNA avertebrata motif tersebut ditemukan
berada pada posisi yang sangat beragam (Valverde et a/. 1994).
Ruas DNA yang tidak menyandikan antara tRNA Asn dan tRNA Cys pada
D. subplana mungkin bisa berfungsi sebagai titik asal replikasi utas L (ORL). Motif ORL ini secara konsisten selalu ditemukan dalam genom mtDNA mamalia
(Anderson et al. 1981) dan beberapa reptilia (Macey et a/. 1997; Kumazawa et a/.
bagian 5' daerah kontrol merupakan pembatas struktur fungsional dari proses
replikasi yang menyebabkan munculnya displacement loop, TAS (termination-
associated sequence) dan runutan kaya C.
Motif ketiga merupakan mikrosatelit mitokondria yang konsisten ditemukan
pada semua genom mtDNA anggota Testudines (Caretta caretta, Laurent et al.
1995; P. subrufa, Zardoya & Meyer 1998; C. mydas, Kumazawa & Nishida 1999;
C. picta, Mindell et al. 1999). Pada lokasi yang berbeda, daerah kaya AT juga
ditemukan pada sebagian besar genom mtDNA Arthropoda (Boore 1999).
Jika ketiga runutan nukleotida berulang dibuang maka akan diperoleh
panjang daerah kontrol setara dengan yang ada pada vertebrata umumnya, yaitu
sekitar 1000 bp (Anderson et al. 1981). Motif runutan nukleotida berulang dalam
jumlah banyak tanpa jeda sepertinya tidak sejalan dengan sifat ekonomis sebagai-
mana menjadi ciri genom mtDNA (Moritz et a1.1987). Selain itu, dalam perjalanan
evolusi biasanya akan terjadi seleksi ke jumlah ulangan yang lebih sedikit (Lopez
eta/. 1996).
Selain ketiga motif DNA berulang, dalam daerah kontrol mtDNA D.
subplana ditemukan tiga motif CSB (conserved sequence block). Runutan nukleo-
tida dan lokasi ketiga CSB tersebut mempunyai kesamaan yang tinggi dengan
yang ada pada mtDNA yang khas vertebrata, yang kemudian membentuk domain
tengah yang stabil (the middle conserved domain). Domain ini diduga terlibat
dalam membentuk struktur tersier (Boore 1999) yang berperan dalam mengontrol
Analisis Filogeni
Runutan nukleotida lengkap mtDNA Dogania dan 18 spesies vertebrata
lainnya (Lampiran 5) digunakan untuk mempelajari hubungan filogeni anggota
amniota. Data runutan nukleotida dibagi menjadi tiga himpunan, yaitu pertama
gen-gen penyandi protein kecuali ND3 dan ND4L karena adanya translational
frameshifting dan ND6 karena disandikan oleh utas L, kedua semua gen penyandi
tRNA dan ketiga dua gen penyandi rRNA.
Perbandingan situs-situs nukleotida semua gen penyandi protein mengha-
silkan 10344 situs, 477 situs di antaranya dikesampingkan karena ada senjang.
Analisis filogeni dilakukan terhadap 6578 situs dari nukleotida pertama dan kedua
dari setiap kodon triplet. Dari jumlah itu, sebanyak 3612 situs dikategorikan
sebagai beragam dan sisanya (2966 situs) sebagai situs yang seragam untuk
semua spesies yang dianalisis. Dari situs yang beragam ditemukan sebanyak 2787
situs bersifat parsimoni, yaitu satu karakter yang berbeda dalam satu situs diwakili
oleh minimal dua spesies. Perbedaan runutan nukleotida antar spesies yang
dibandingkan berkisar antara 6% dan 30%, dan rata-rata rasio transisi terhadap
transversi sebesar 0.9. Translasi ke asam amino menghasilkan 3285 situs, yang
mana 2142 situs dikategorikan sebagai beragam dan 1656 situs bersifat parsimoni.
Perbedaan asam amino tersebut berkisar antara 8% dan 44%.
Semua gen tRNA digabung, tetapi bagian gelung DHU, TyC (thymine-
pseudouridine-cytosine) dan variable loops (Gambar 3.4) dibuang. Analisis
perbandingan nukleotida menghasilkan 1162 situs, yang dikategorikan sebagai
situs beragam sebanyak 864 situs dan yang dikategorikan bersifat parsimoni
sebanyak 665 situs. Perbedaan runutan nukleotida berkisar antara 7% dan 38%,
Perbandingan runutan nukleotida gen 12Sr RNA dan 16SrRNA menghasil-
kan 2887 situs yang kemudian 937 situs di antaranya dikesampingkan karena gap.
Sebanyak 1252 situs diantaranya beragam dan 1000 situs bersifat parsimoni.
Perbedaan runutan nukleotida berkisar antara 8% dan 35%, dan rata-rata rasio
transisi terhadap tranversi adalah 1 .O.
Analisis maksimum parsimoni menggunakan kombinasi ketiga himpunan
data menghasilkan pohon filogeni tunggal (Gambar 3.7 kiri) dengan indeks konsis-
tensi untuk karakter parsimoni sebesar 0.43. Topologi pohon filogeni tersebut
sama dengan yang diperoleh menggunakan metode NJ kecuali posisi ayam dan
burung falkon saling terbalik (Gambar 3.7 kanan). Pohon filogeni MP menunjukkan
bahwa testudines berada dalam satu cabang bersama-sama dengan anggota
diapsida, walaupun tidak didukung nilai bootstrap yang tinggi (40%). Pengelom-
pokan testudines dengan burung dalam satu klad ditunjukkan oleh pohon filogeni
NJ (Gambar 3.7 kanan).
Hubungan filogeni yang berbeda diperoleh jika menggunakan data asam
amino hasil translasi gen penyandi protein (Gambar 3.8). Meskipun begitu, ada
kecenderungan testudines lebih dekat ke arkosauria dibanding ke lepidosauria
I
Pembahasan
Organisasi genom, kandungan gen, komposisi nukleotida, penggunaan
kodon dan lokasi ORL menunjukkan bahwa mtDNA D. subplana dapat mewakili genom mtDNA yang khas vertebrata (Anderson et a/. 1981 ; Macey et al. 1997). Walaupun begitu, organisasi mtDNA yang sedikit berbeda dilaporkan terjadi pada
berbagai spesies vertebrata (Desjardins & Morais 1990; Paabo et a/. 1991 ; Macey et a/. 1997; Yoneyama 1987; Kumazawa & Nishida 1995; Kumazawa et al. 1998). Sedangkan pada tingkatan taksa yang jauh berbeda, misalnya Cephalochordata,
Molluscs, Brachiopoda dan Arthropoda, transposisi gen dan penggunaan kodon
ditemukan sangat berbeda (Boore 1999). Hal ini menunjukkan bahwa laju evolusi
organisasi mtDNA vertebrata jauh lebih rendah dibanding dengan laju evolusi
runutan nukleotidanya.
Motif runutan nukleotida ORL serta lokasinya ternyata tidak konsisten
ditemukan dalam mtDNA testudines, anggota reptilia lainnya dan beberapa
spesies burung. Hal yang sebaliknya adalah beberapa motif CSB secara
konsisten ditemukan dalam daerah kontrol mtDNA vertebrata. Selain motif CSB,
dalam daerah kontrol bisa ditemukan motif TAS, the middle conserved domain, dan ORH (origin replication of H strand). Dengan begitu, pendapat bahwa replikasi mtDNA vertebrata dilakukan dengan cara yang sangat stabil (Ojala et a/. 1981 ; Moritz et al. 1987; Yoneyama 1987; Boore 1999) masih bisa dipertahankan. Keberadaan motif-motif tersebut dalam daerah kontrol diperlukan untuk memben-
tuk struktur tersier yang fungsional (Boore 1999).
Daerah kontrol mtDNA vertebrata bisa dibagi menjadi tiga domain, yaitu
dua domain perifer yang sangat beragam dan domain tengah yang sangat stabil.
dengan motif DNA berulang secara tandem yang sangat besar di bagian 5' dan
mikrosatelit mitokondria TA dan ATATT di bagian 3'. Hal yang menarik adalah
domain tengah dan mikrosatelit mitokondria ditemukan sangat stabil antar spesies
testudines (pembahasan lebih lanjut dalam Bab 5). Mikrosatelit mitokondria di
bagian 3' hanya ditemukan pada anggota testudines (Zardoya & Meyer 1998; Mindell et al. 1999; Laurent et al. 1995, Kumazawa & Nishida 1999) tetapi tidak ada pada anggota reptilia lainnya. Hal ini menunjukkan bahwa evolusi daerah
kontrol mtDNA bagi testudines masih relatif baru. Zardoya & Meyer (1998) menyarankan untuk memanfaatkan karakter mikrosatelit mitokondria sebagai
penanda genetik yang handal.
Runutan lengkap nukleotida genom mtDNA mungkin bisa digunakan untuk
memperkirakan waktu pemisahan beberapa spesies testudines menjadi lebih
tepat. Berdasarkan catatan fosil, pemisahan beberapa spesies testudines terjadi
lebih dari 100 juta tahun yang lalu. Sedangkan pemisahan anapsida dan diapsida
diperkirakan terjadi lebih dari 200 juta tahun yang lalu. Laurin & Reizs (1995) memperkirakan bahwa morfologi yang ekstrim dari kura-kura dibandingkan dengan
anggota amniota lainnya paling tidak terjadi sekitar 300 juta tahun yang lalu.
Berdasarkan catatan fosil, pemisahan kelompok-kelompok utama amniota terjadi
sekitar 250 juta tahun yang lalu.
Pada tingkat runutan nukleotida, Zardoya & Meyer (1 996) memperlihatkan
bahwa divergensi mtDNA mencapai titik yang jenuh (plateu) pada taksa-taksa yang
mempunyai waktu pemisahan di atas 200 juta tahun. Sebelum umur itu, antara
pertambahan waktu dan divergensi mtDNA masih menunjukkan hubungan yang
linear. Hal ini terlihat dari hasil analisis filogeni amniota yang melibatkan hubungan
(Gambar 3.7 dan 3.8). Selain itu, penggunaan data asam amino malah cenderung memasukkan mamalia ke dalam cabang diapsida (Gambar 3.8).
Meskipun begitu, hasil penelitian ini mendukung posisi kura-kura sebagai lebih
dekat dengan arkosauria dibanding dengan lepidosauria.
Ketersediaan runutan nukleotida lengkap mtDNA dari D. subplana sangat berguna untuk mempelajari lebih lanjut berbagai fenomena populasi dan hubungan
filogeni anggota testudines, salah satunya seperti dapat dilihat dalam Bab 4 dan 5.
Hasil penelitian ini memberi peluang yang besar untuk memanfaatkan runutan
nukleotida dari berbagai bagian mtDNA sebagai penanda genetik walaupun
beberapa laporan menyebutkan adanya masalah-masalah teknis yang mungkin
Kesimpulan
Ukuran mtDNA D. subplana adalah 17289 bp. Organisasi genom, kandungan gen, kornposisi basa, penggunaan kodon, lokasi ORL dan beberapa karakter
daerah kontrol rntDNA 0. subplana dapat mewakili mtDNA yang khas vertebrata. Adanya ruas DNA berulang secara tandem tanpa jeda di bagian 5' daerah kontrol
mtDNA D. subplana adalah unik, sedangkan rnikrosatelit mtDNA di bagian 3' daerah kontrol mtDNA merupakan ciri khas anggota testudines. Gen-gen penyandi
protein, tRNA dan rRNA, dan daerah kontrol genom rntDNA D. subplana secara kesatuan rnaupun terpisah dapat dijadikan penada molekular untuk mempelajari
Tabel 3.1. Karakteristik mtDNA labi-labi, D. subplana
- -
Gen Posisi dan Ukuran Kodon** Spasi ujung 5'
dari ke* ukuran lnisiasi Stop tRNA Phe
12Sr RNA tRNA Val
1 GSrRNA tRNA Leu(UUR) NDI
tRNA Ile
.
tRNA GlntRNA Met ND2 tRNA Trp tRNA Ala tRNA Asn tRNA Cys tRNA Tyr COI tRNA Ser(UCN) tRNA Asp COll tRNA Lys ATPase8 ATPase6 COlll tRNA Gly ND3 tRNA Arg ND4L ND4 tRNA His tRNA Ser(AGY) tRNA Leu (CUN) ND5 ND6 tRNA Glu tRNA Thr tRNA Pro ATG ATG GTG ATG ATG ATG ATG ATG ATG ATG ATG ATG ATG Tag TAA TAA TAA Taa Tac Tgg Tgg Tgt TAA AGA TAA CAACCAAAC A *** A TAACA C A T CCA TC CAA
Dloop 15470 17289 1820
* L
=
disandikan utas L [image:51.574.51.511.92.672.2]Tabel 3.2. Komposisi nukleotida mtDNA
0.
subplana (Oh)GenIKarakter
Seluruh mtDNA
12s rRNA
16s rRNA
tRNA (utas H)
tRNA (utas L)
Daerah kontrol
-
dloopG 11.95 16.37 16.01 15.70 13.80 10.57 AT 61.30 59.79 61.45 61.94 51.62 63.88 A 35.33 37.82 39.85 35.66 27.78 30.70
Penyandi protein
-
utas Hnukleotida pertama nukleotida kedua nukleotida ketiga C 26.75 23.83 22.53 22.36 34.59 25.55 T 25.97 21.97 21.60 26.28 23.84 33.18 34.79 20.39 49.56
Penyandi protein
-
utas L [image:52.576.42.511.81.760.2]Tabel 3.3. Jumlah penggunaan kodon (huruf cetak tegak) dan nilai indeks penggu- naan kodon (huruf cetak miring) dalam mtDNA
0.
subplana. lndeks penggunaan kodon adalah perbandingan jumlah kodon yang digunakan terhadap rata-rata jumlah kodon sinonimnya. Nama asam amino menggunakan singnkatan tiga hurufPhe TTT 71 0.73
TTC 123 1.27
Leu TTA 155 1.75 TTG 19 0.21
Leu CTT 51 0.58
CTC 39 0.44
CTA 247 2.79
CTG 21 0.24
lle ATT 132 0.95
ATC 145 1.05
I Met ATA 206 1.75
ATG 29 0.25
Val GTT 34 0.97
GTC 29 0.83
GTA 72 2.06
GTG 5 0.14
Ser TCT 23 0.56
TCC 44 1.07
TCA 123 3.00
TCG 4 0.10
Pro CCT 23 0.50
CCC 38 0.83
CCA 115 2.51
CCG 7 0.15
Thr ACT 58 0.64
ACC 124 1.36
ACA 177 1.94
ACG 6 0.07
Ala GCT 39 0.75
GCC 87 1.68
GCA 78 1.51
GCG 3 0.06
Tyr TAT 53 0.95
TAC 58 1.05
* TAA 7 2.80
TAG 2 0.80
His CAT 33 0.74
CAC 56 1.26
Gln CAA 89 1.82
CAG 9 0.18
Asn AAT 39 0.58
AAC 95 1.42
Lys AAA 77 1.97
AAG 1 0.03
Asp GAT 17 0.65
GAC 35 1.35
Glu GAA 75 1.85
GAG 6 0.15
Cys TGT 9 0.67
TGC 18 1.33
Trp TGA 86 1.76
TGG 12 0.24
Arg CGT 6 0.40
CGC 12 0 8 0
CGA 41 2.73
CGG I 0.07
Ser AGT 18 0.44
AGC 32 0.78
AGA 1 0.40
AGG 0 0.00 Gly GGT 32 0.70
GGC 50 1.09
GGG 31 0.67
[image:53.576.40.512.84.755.2]Dogania subplana mtDNA
17289 bp
Gambar 3.1. Situs pemotongan Mbol (angka cetak biasa) dan posisi penempelan primer (angka cetak tebal) dalam mtDNA
0.
subplana. Primer yang menempel di utas H [image:54.568.26.508.87.643.2]Phe 12SrRNA
\
\
ND2
[image:55.568.44.492.112.612.2]Dogania subplana mtDNA 17289 bp
Gambar 3.2. Peta genetik mtDNA
0.
subplana. Gen-gen yang disandikan utasH
I--tRNA P h e - - - I--12S rRNA---
G T T A T T G T A G T T T A C A C W G C A T G G C A C T G A A A A T G C C A A G A C G T A C T C A T T A T C C C T C A C G A T T T G G T C C T T C T T T G T T A C 1 0 0
...
TTTTTACTAAACCTACACATGTAAGTATCAGCAAGCCAGTGAAATGCCCTAAAAGTCACATCAGACAAAAGGAGCTGGTATCAGGCACGCCACAGCAGCC 200
...
TTAAAACGGACCTGAGGTGCGCACACACCGCCCGTCACCCCCATCAACTTTCTGTTCACCCCACAAATCAGATGGGGTGTCGT 1 0 0 0
---I I--tRNA Val---
A A C A A G G T A A G T G T A C C G G A A G G T G C A C T T G G A C T A C C G G T T A 1100
---I I--16S rRNA---
TTTTGAGCAAAACTCTCAGCTCAACCAACCAGTATCAACCCAACAAACTAAATATTCCACACAAACTAAACTAAAACATTCTAACCATAATCCAAGTATA 1200
...
G T A C C T C T T G C A T C A T G A T T T A G C C A G A A T A A T A C C T C 1400
...
C G C T A G C T T A A A T C A G A A C A G A T A A A C T A C T G A T T A T T C C A 1900
...
C A G G A G C G T A A A C A A G A A A G A T T A A A A T C T G T A A A A G G C G T A T 2000
...
TAGGGGTGATGCCTGCCCAGTGACACTGTTCAACGGCCGCGGTATCCTAACCGTGCAAAGGTAGCGTAATCACTTGTCCTTTAAATAGGGACCAGTATGA 2100
...
AAGGCTAAACGAGGCCCTATCTGTCTCTTACAGATAATCAGTGAAATTGATCTTCTCGTGCAARAGCGAGAATATAAATATAAGACGAGAAGACCCTGTG 2200
---
I I - - ~ R N A Leu(uu~)---
I ND1C C C A A A A C A C A G G G C T G A T T G G G G T G G C A G A G C C A G G T ~ T A C A T G T T T 2800
M F
A T A C T A C T A T C T A A C C T A T T