DENGAN KUALITAS KARKAS PADA SAPI ACEH
Eka Meutia Sari
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
BOGOR
2011
Dengan ini saya menyatakan bahwa disertasi Keragaman Genetik Gen Hormon Pertumbuhan dan Hubungannya dengan Kualitas Karkas pada Sapi Aceh adalah karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apapun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir disertasi ini.
Bogor, April 2011
Eka Meutia Sari
EKA MEUTIA SARI. Genetic Polymorphism of Growth Hormone (GH) in Association with Carcass Quality of Aceh Cattle. Under the supervision of RONNY RACHMAN NOOR, CECE SUMANTRI, and ENDANG TRI MARGAWATI.
This study was conducted in order to identify polymorphism of growth hormone gene and to characterize nucleotide changes and its position in the DNA sequence of the exon five in the bovine growth hormone gene. In addition, this study was done to determine the association of GH/AluI polymorphism with carcass quality and to describe D-Loop of mtDNA and microsatellite alleles in Aceh cattle. A total of 242 DNA genome samples were extracted from four Aceh cattle population, i.e., Banda Aceh (29), Aceh Besar (30), Indrapuri (129), and Saree (54), while for sample comparison was derived from the Gen Bank. The bGH gene was sequenced to identify new single nucleotide polymorphism (SNP). The sequence data were analyzed using BLAST and MEGA 4 software, and the PCR-RFLP was used to amplify 404 bp of GH gene. D-Loop sequences of mtDNA amplification were done by using specific primer with the PCR product of 980 bp, and the dnaSP program was used to build the NJ tree and to identify the haplotype. Three markers of BM1824, SPS115, and ILSTS028 were used for microsatellite DNA genotyping. The data of genotyping was analyzed using GeneMapper versi 4.0 software and Excel program. The result showed that one new SNP was found in the exon five of bGH gene, which located at position of 2.230 bp (C/T). Genotype frequencies of SNP in position 2.230 bp of Aceh cattle were 0.36; 0.14; and 0.50; for CC, TT, and CT respectively. On the contrary, the LL genotype was the only genotype which found in other Aceh cattle population. This finding indicated that there was not evidence of polymorphism of GH/AluI in Aceh cattle, and there was not correlation of GH/AluI gene with carcass quality of Aceh cattle. It could be affected by small number of sampling size. D-Loop mtDNA analyses showed that there was 27 haplotypes, and 28 Aceh cattle integrated into group of Buthan, China, India, and Zebu cattle with maternal origin of Bos indicus and one sample was from Bos taurus. These finding could be assumed that Aceh cattle has more genetic introgression of Bos indicus compared to that Bos taurus breeds. Microsatellite analyses showed that the average number of allele per locus was 7.6. The genotype of Indrapuri population was higher than those of Banda Aceh and Saree population. Based on the microsatellite alleles analyses, the frequency of C alele locus of SPS115 and ILSTS028 was higher in Indrapuri population. Nevertheless, these locus could be uses for further study to get more information about polymorphism in Indrapuri population. However, this study suggests that GH gene could be possible used as genetic marker.
EKA MEUTIA SARI. Keragaman Genetik Gen Hormon Pertumbuhan (GH) dan Hubungannya dengan Kualitas Karkas pada Sapi Aceh. Dibimbing oleh RONNY RACHMAN NOOR, CECE SUMANTRI dan ENDANG TRI MARGAWATI.
Sapi Aceh merupakan salah satu dari tujuh bangsa sapi asli yang ada di Indonesia, selain sapi Pesisir, Bali, Madura, Grati, Sumba Ongole, dan Ongole cross. Sapi Aceh jantan dan betina bertanduk, dengan warna bulu bervariasi yaitu putih, merah bata, hitam. Sapi Aceh resisten terhadap serangan parasit dan dapat menyesuaikan diri dengan lingkungan di daerah Nanggroe Aceh Darussalam (NAD). Meskipun dengan tatalaksana pemeliharaan yang sederhana, tetapi sapi Aceh dapat berproduksi dan melahirkan anak sapi yang sehat dan induknya mampu merawat anak dengan baik. Daging sapi Aceh memiliki serat daging yang halus dan warna daging merah.
Populasi sapi Aceh semakin lama semakin menurun, karena persilangan dengan bangsa sapi unggul lainnya, memiliki turunan anak sapi yang berukuran besar dan memiliki nilai jual yang tinggi. Apabila kondisi ini dibiarkan berlangsung, maka keberadaan sapi Aceh akan terancam punah, karena hilangnya sumber daya genetik sapi Aceh. Melestarikan dan mempertahankan keberadaan spesies dan bangsa sapi yang memiliki keunikan sangat diperlukan.
Berdasarkan pemikiran tersebut maka dilakukan suatu penelitian yang bertujuan untuk identifikasi polimorfisme gen hormon pertumbuhan (GH) dan karakterisasi perubahan nukleotida dan posisinya di ekson lima pada gen GH, juga membuktikan hubungan polimorfisme gen GH/AluI dengan kualitas karkas pada sapi Aceh. Penelitian ini juga menentukan keragaman genetik D-Loop mtDNA dan DNA mikrosatelit yang berguna sebagai acuan dalam pelaksanaan program pelestarian plasma nutfah sapi Aceh untuk pengembangan dan pemanfaatannya yang berkelanjutan.
Jumlah sampel sapi Aceh yang digunakan dalam penelitian ini yaitu 242 ekor yang berasal dari Banda Aceh (29), Aceh Besar (30), Indrapuri (129) dan Saree (54), serta data sampel sapi pembanding berasal dari Gen Bank. Ekstraksi dan purifikasi DNA total dilakukan menurut metode Sambrook. Primer gen GH yang digunakan berasal dari Gen Bank (kode akses M577641.1). Sekuensing gen GH dilakukan untuk identifikasi adanya perubahan single nucleotide polymorphism (SNP). Data sekuensing dianalisis dengan BLAST dan MEGA 4 software, dan PCR-RFLP digunakan untuk amplifikasi gen GH sepanjang 404 bp. Sekuensing juga dilakukan pada D-Loop dengan produk PCR sepanjang 980 bp. Program dnaSP digunakan untuk pembentukan NJ tree serta untuk identifikasi jumlah haplotipe. Analisa DNA mikrosatelit menggunakan tiga lokus yaitu BM1824, SPS115 dan ILSTS028. Ukuran-ukuran alel mikrosatelit dianalisis dengan program GeneMapper versi 4.0 dan program Excel.
GH/AluI pada sapi Aceh, dan membuktikan juga belum ada hubungan antara gen GH/AluI dengan kualitas karkas pada sapi Aceh. Keadaan ini disebabkan karena terbatasnya jumlah sampel yang dimiliki. Namun demikian, dari hasil penelitian ini dapat disimpulkan bahwa gen GH dapat digunakan sebagai marker genetik untuk sapi Aceh. Hasil analisis D-Loop mtDNA menunjukkan bahwa terdapat 27 haplotype, dengan 28 sampel sapi Aceh menyebar luas diantara sampel sapi Buthan, China, India dan Zebu yang memiliki turunan Bos indicus dan hanya satu sampel yang memiliki turunan Bos taurus. Fenomena ini dapat diasumsikan bahwa sapi Aceh memiliki introgresi genetik dari Bos indicus dan Bos taurus.
Hasil analisis DNA mikrosatelit menunjukkan, bahwa rataan alel per lokus adalah 7.6. Sapi Aceh memiliki derajat heterozigositas yang tinggi. Populasi sapi Aceh yang berasal dari Indrapuri memiliki genotipe yang lebih banyak dari populasi yang berasal dari Banda Aceh dan Saree. Hasil analisis alel mikrosatelit, frekuensi alel C pada lokus SPS115 dan ILSTS028 adalah tinggi pada populasi sapi Aceh di Indrapuri. Berdasarkan hasil penelitian, dapat disarankan bahwa lokus-lokus mikrosatelit ini dapat digunakan sebagai genetik marker pada populasi sapi Aceh di Indrapuri.
© Hak Cipta milik IPB, tahun 2011
Hak Cipta dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencamtumkan
atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan
pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan
kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan
kepentingan yang wajar IPB
Dilarang mengumumkan dan memperbanyak sebagian atau seluruh Karya tulis
DENGAN KUALITAS KARKAS
PADA SAPI ACEH
Eka Meutia Sari
Disertasi
sebagai salah satu syarat untuk memperoleh gelar
Doktor pada
Program Studi Ilmu Produksi dan Teknologi Peternakan
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
BOGOR
Nama : Eka Meutia Sari
NRP : D161080021
Program Studi/Mayor : Ilmu Produksi dan Teknologi Peternakan
Disetujui
Komisi Pembimbing
Prof. Dr. Ir. Ronny Rachman Noor, M.Rur.Sc
Ketua
Prof. Dr.Ir.Cece Sumantri, M.Agr.Sc Dr. Ir. Endang Tri Margawati, M.Agr.Sc
Anggota Anggota
Diketahui
Koordinator Mayor Dekan Sekolah Pascasarjana IPB
Ilmu dan Teknologi Peternakan Institut Pertanian Bogor
Alhamdullilah dan segala puji bagi Allah SWT atas segala karunia-Nya sehingga penulis dapat menyelesaikan karya ilmiah yang berjudul Keragaman Genetik Gen Hormon Pertumbuhan (GH) dan Hubungannya dengan Kualitas Karkas pada Sapi Aceh. Salah satu alasan yang melatarbelakangi penelitian ini adalah pengamatan penulis terhadap keberadaan sapi Aceh yang sangat sulit ditemukan. Hal ini disebabkan karena peternak lebih menyenangi memelihara ternak yang menghasilkan anak sapi yang berukuran besar, sehingga banyak peternak mengawinkan ternaknya dengan bangsa sapi unggul, sementara sapi Aceh berdasarkan pertumbuhan ukuran tubuhnya, semakin lama semakin kecil. Apabila keadaan ini berlangsung terus menerus, kemungkinan keberadaan sapi Aceh akan semakin sulit ditemukan, sehingga diperlukan suatu pemikiran untuk dapat melestarikan keberadaan sapi Aceh.
Melakukan karakterisasi keragaman genetik pada sapi Aceh merupakan suatu cara untuk dapat melestarikan keberadaan sapi Aceh. Karakterisasi genetik sapi lokal Indonesia sebenarnya telah lama dilakukan dan beberapa hasilnya telah dipublikasikan, diantaranya berdasarkan perbedaan golongan darah dan protein darah. Hasilnya bahwa sapi Bali memiliki alel HbX pada golongan darahnya yang tidak dimiliki sapi lokal lain. Tetapi, studi karakterisasi genetik di tingkat molekuler masih sangat kurang. Adanya pertimbangan perkembangan yang cepat pada sejumlah penciri genetik molekuler yang lebih akurat dibandingkan fenotipik, maka penggunaan marka ini akan sangat membantu dalam penanganan manajemen sistem seleksi pada ternak sapi potong. Informasi tentang alel-alel spesifik dari data molekuler sapi lokal Indonesia masih sangat terbatas.
Beberapa hasil penelitian menunjukkan adanya alel spesifik untuk sapi Bali pada lokus mikrosatelit INRA 023, HEL9 dan INRA 035 dibandingkan sapi Bos taurus (Simmental, Limousin, dan Brangus). Demikian pula hasil studi berdasarkan 16 lokus penciri mikrosatelit memberikan gambaran awal tentang hubungan genetik antara sapi Aceh, Bali, Madura, dan PO.
Penggunaan gen GH sebagai marker genetik telah banyak digunakan dalam penelitian untuk melihat keterkaitan polimorfisme gen GH dengan sifat produksi, karena keragaman gen GH tersebut ditunjukkan dengan adanya polimorfisme pada situs tertentu yang kemungkinan ekspresi gen GH tersebut berhubungan dengan sifat produksi. Gen GH merupakan gen yang menyandi hormon pertumbuhan sebagai produknya dan terletak pada kromosom 19 pada ternak sapi. Namun hasil penelitian tersebut belum menunjukkan adanya hubungan antara penciri gen GH dengan sifat-sifat ekonomis sapi lokal, terutama terhadap kualitas karkas dan daging.
memberikan bantuan dana penelitian. Ucapan terimakasih juga penulis ucapkan kepada Ir. Ahmady sebagai Kepala BPTU Indrapuri, Ir Asnawi (STPP Saree), Dr. Yunus M.Agr.Sc (Dinas Peternakan Kabupaten Aceh Besar), drh. Ronny (RPH Kotamadya Banda Aceh) dan Bambang S.Pt yang telah membantu dalam pengambilan sampel darah dan daging sapi Aceh. Kepada seluruh staf di BPTU Indrapuri dan STPP Saree penulis ucapkan terima kasih atas bantuannya selama pengambilan sampel darah. Terimakasih kepada Kepala Bagian dan Staff Laboratorium Genetika Molekuler Ternak Bag. Pemuliaan dan Genetika Fapet IPB, atas segala fasilitas alat dan bahan yang dapat penulis gunakan untuk ekstrasi DNA, kepada Eryk Andreas, S.Pt, MSi, Restu Misrianti, S.Pt atas bantuannya selama melakukan ekstraksi DNA. Rasa terimakasih penulis ucapkan kepada guru yang senantiasa memberi semangat, bimbingan dan diskusi yang sangat berharga Prof. Gunawan, MS, Ir. Hasan Basri, MS, dan Prof. Dr. Ir. Muladno, MSA. Kepada Dr. Ir. Rarah R.A. Maheswari, DEA sebagai Koordinator Mayor Ilmu dan Teknologi Peternakan yang senantiasa memberikan bimbingan dan pemikiran yang sangat bermanfaat bagi penulis. Ucapan terimakasih penulis sampaikan kepada Prof. Han Jianlin selaku kepala CAAS (Chinese Academy of Agriculture Sciences) – ILRI Beijing – China, yang telah mengizinkan penulis melakukan research di Lab CAAS sebagai visiting scientist. Ucapan terimakasih juga penulis ucapkan kepada Dikti atas beasiswa yang diberikan untuk program Sandwich ke Beijing, sehingga penulis dapat menyempurnakan penelitian di laboratorium. Terimakasih juga kepada Yayasan Toyota Astra atas bantuan dana penelitian dan SEARCA atas Thesis Grant yang diberikan kepada penulis. Terima kasih kepada Dr. Ir. Chalid Talid, MS dan Dr. Ir. Dedy Duryadi, DEA sebagai penguji luar komisi dan Dr. Ir. M. Yamin, M.Agr Sc sebagai pimpinan sidang pada ujian tertutup saya. Ucapan terima kasih juga penulis sampaikan kepada seluruh teman dan kolega.
Akhirnya, ungkapan terima kasih penulis sampaikan kepada Ayahanda H. Sadi Lubis (Alm), Ibunda Hj. Saadah AK (Alm) yang selalu memberikan nasehat bahwa ilmu pengetahuan merupakan harta yang sangat berharga, maka selalu belajarlah, adinda Elin Nita Sari Lubis SH dan Zul Azhar SE, adinda Drs. Nata Kurniawan Lubis dan Cut Maysarah SE, adinda Aldini Inda Sari Lubis. S.Pt (Alm), Ayahanda Husein Maun (Alm), Ibunda Nursyiah Taher (Alm), kakanda Dra. Mutmainah. MA, kakanda Dra. Ratna Sari, kakanda Dra. Mustabsyirah, MA, kakanda Ir. Ulil Azmi, kakanda Siti Bararah SH, kakanda dr. Siti Safarah, M.Sc (alm), adinda Abdul Qudus SH, dan adinda dr.Abdul Muhaimin M.Sc. Karya tulis ini penulis persembahkan kepada suami tercinta Abdul Qawi A.Md serta ananda Nabilah Putroe Agung, ananda Dwi Putroe Naulia, dan ananda Salsabilah Putroe Wieka atas pengertian, kesabaran dan doanya.
Semoga karya ilmiah ini dapat bermanfaat bagi perkembangan dan kemajuan ilmu peternakan di Indonesia dan khususnya di Nanggroe Aceh Darussalam serta para pembaca.
Halaman
DAFTAR TABEL... ... xiv
DAFTAR GAMBAR... ... xv
DAFTAR LAMPIRAN... ... xvi
PENDAHULUAN... ... 1
Latar Belakang... 1
Tujuan Penelitian... 4
Manfaat Penelitian... 4
Hipotesis... 5
TINJAUAN PUSTAKA.. ... 7
Sumber Daya Genetik Ternak Lokal Indonesia... 7
Karakteristik Sapi Aceh... 8
Gen Hormon Pertumbuhan... 10
Penanda Molekuler... 12
Faktor yang Berpengaruh pada Kualitas Daging dan Karkas... 13
METODOLOGI PENELITIAN... ... 15
Waktu dan Tempat Penelitian………...……….... 15
Sampel Darah Sapi Aceh………....……... 15
Sampel Daging Sapi Aceh………....……... 16
Ekstraksi DNA Genom (Darah)…...………....…... 17
Ekstraksi DNA Genom (Daging).………....…... 17
Identifikasi Single Nucleotide Polymorphism (SNP)...….. 18
Identifikasi D-Loop………... 20
Identifikasi Gen GH/AluI... 23
HASIL DAN PEMBAHASAN... ... 27
Identifikasi Single Nucleotide Polymorphism (SNP) Gen GH... 27
Identifikasi D-Loop... 31
Identifikasi DNA Mikrosatelit... 41
Hubungan Kualitas Karkas Sapi Aceh dengan Gen Hormon Pertumbuhan (GH)... 48
Pembahasan Umum... 52
SIMPULAN DAN SARAN.. ... 57
DAFTAR TABEL
Halaman
1 Jumlah sampel daging yang digunakan untuk analisis gen GH/SNP.... 16
2 Jumlah sampel darah yang digunakan untuk analisis D-Loop... 16
3 Jumlah sampel darah yang digunakan untuk analisis mikrosatelit... 16
4 Jumlah sampel daging yang digunakan untuk analisis gen GH/AluI.... 17
5 Basa primer GH/AluI (Gen Bank M57764.1, Gordon et al. 1983)... 19
6 Basa primer D-Loop... 20
7 Basa primer mikrosatelit lokus BM 1824, SPS 115 dan ILSTS 028.... 21
8 Basa primer GH/AluI (Gen Bank M57764.1, Gordon et al. 1983)... 23
9 Variasi sekuen di daerah promotor dan ekson dari gen GH pada sapi.. 28
10 Frekuensi alel dan genotipe SNP gen GH sapi Aceh pada posisi basa ke 2.291 bp... 29
11 Frekuensi alel dan genotipe SNP gen GH sapi Aceh pada posisi basa ke 2.230 bp... 29
12 Maternal origin dari beberapa bangsa sapi... 32
13 Persentase komposisi nukleotida D-Loop sapi Aceh... 35
14 Persentase komposisi nukleotida D-Loop sapi Aceh, Buthan, China India dan Zebu... 36
15 Rataan jarak genetik sapi Aceh, Buthan, China, India dan Zebu berdasarkan metode 2 parameter Kimura... 37 16 Genotipe, jumlah sampel dan frekuensi lokus BM1824 sapi Aceh... 41
17 Genotipe, jumlah sampel dan frekuensi lokus SPS115 sapi Aceh... 42
18 Genotipe, jumlah sampel dan frekuensi lokus ILSTS028 sapi Aceh.... 44
19 Kisaran ukuran alel, jumlah alel dan heterozigositas pada tiga lokus mikrosatelit pada sapi Aceh
20 Nilai heterozigositas lokus BM1824, SPS115, ILSTS028 sapi Aceh... 46
21 Perbandingan jumlah alel per lokus pada berbagai penelitian... 47
DAFTAR GAMBAR
Halaman
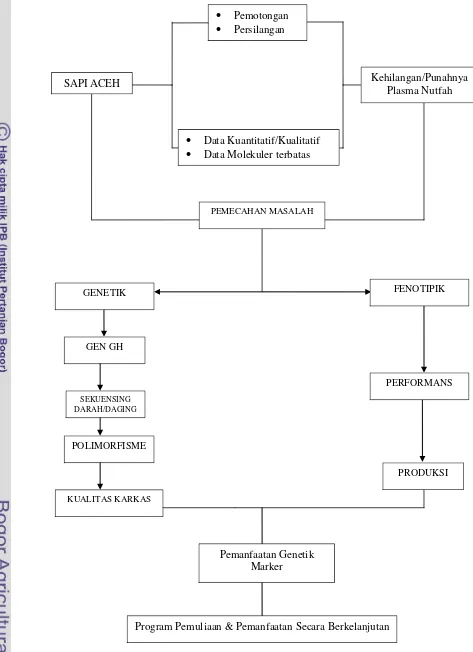
1 Kerangka pemikiran alur penelitian... 6
2 Sapi Aceh di BPTU Indrapuri NAD... 9
3 Grafik hasil elektroforesis Genetic Analyzer Applied Biosystem
3130x... 22
4 Posisi mutasi basa ke 2.230 bp pada gen GH ekson lima... 30
5 Sketsa letak penempelan primer Forward dan Reverse untuk mengamplifikasi fragmen daerah D-Loop sapi Aceh...
31
6 Sketsa daerah D-Loop hasil perunutan 230 bp sekuen sapi Aceh, Buthan, China, India dan Zebu... 33
7 Konstruksi Median Joining Network dari 230 bp D-Loop sekuen sapi Aceh (kuning), Zebu dari Indonesia (biru), Zebu dari China
(merah), India (pink) dan Buthan (pink muda)... 33
8 Dendogram Neighbour-Joining berdasarkan metode 2 parameter Kimura (230 bp) sapi Aceh, Buthan, China, India dan Zebu dengan pengolahan bootstrap 1000 ulangan... 40 9 Posisi primer forward, primer reverse dan produk PCR gen
GH... 48
10 Visualisasi hasil amplifikasi ruas gen GH pada gel agarosa 1.5%.
M: marker 100 bp, 1-11: Sampel penelitian... 48
11 Visualisai PCR-RFLP ruas gen GH|AluI pada gel agarosa 1.5%.
DAFTAR LAMPIRAN
Halaman
1 Jenis kelamin dan nomor DNA sapi Aceh untuk analisa SNP,
D-Loop dan Mikrosatelit di BPTU Indrapuri... 65
2 Jenis kelamin, lokasi dan nomor DNA sapi Aceh untuk analisa SNP, D-Loop dan Mikrosatelit dari Banda Aceh dan Saree... 67
3 Sekuen sampel dari Gen Bank yangdigunakan untuk alignments gen GH... 68
4 Sekuen dari Gen Bank yangdigunakan untuk alignments D- Loop... 69
5 Gen Bank M577641.1... 70
6 Hasil BLAST (Basic local alignment search tool) sapi Aceh... 72
7 Data Haplotype sapi Aceh (908 nt) menggunakan DnaSP Ver.4.10.3... 73
8 Data Haplotype sapi Aceh (230 nt) menggunakan DnaSP Ver.4.10.3... 74
9 Jumlah nukleotida sapi Aceh, Buthan, China, India dan Zebu setelah disejajarkan dengan nukleotida Bos Taurus (Gen Bank V00645)... 80
10 Komposisi nukleotida sapi Aceh, Buthan, China, India dan Zebu... 81
11 Jarak genetik menggunakan metode 2 parameter Kimura pada sapi Aceh, Buthan, China, India dan Zebu... 83
12 Hasil elektroforesis lokus BM1824... 86
13 Hasil elektroforesis lokus SPS115... 87
Usaha peternakan di Provinsi Nanggroe Aceh Darussalam secara umum telah dilakukan secara turun temurun meskipun dalam jumlah kecil skala rumah tangga, namun usaha tersebut telah memberikan kontribusi yang cukup besar dalam menunjang pembangunan sub sektor peternakan. Kontribusi tersebut terutama dalam memenuhi kebutuhan penyediaan bahan pangan asal ternak, baik dalam bentuk ternak hidup maupun daging, khususnya kebutuhan domestik dan daerah. Meskipun kontribusi petani peternak cukup menentukan dan memegang peranan penting dalam penyediaan kebutuhan sapi potong, namun belum sepenuhnya mampu menjawab tantangan dan peluang pasar yang cenderung meningkat yang mengakibatkan tingginya harga daging sapi di pasaran Provinsi Nanggroe Aceh Darussalam.
Pertumbuhan merupakan indikator terpenting dalam menentukan tingkat produksi daging pada sapi, sehingga memiliki nilai ekonomi penting dalam budidaya sapi pedaging. Sapi Aceh merupakan salah satu jenis sapi pedaging di Indonesia yang telah dikenal sebagai jenis sapi yang cocok untuk dikembangkan di Indonesia. Sapi Aceh ini dapat beradaptasi dengan baik pada kondisi lingkungan Indonesia, tahan terhadap iklim tropis, dapat hidup dengan kondisi pakan dan air setempat, dan tahan terhadap keberadaan bakteri dan parasit (Gunawan 1998). Meskipun sapi ini mampu beradaptasi dengan baik, namun produktivitasnya masih rendah jika dibandingkan dengan sapi impor. Melalui peningkatan produktivitas sapi lokal Indonesia, diharapkan minat para peternak untuk beternak sapi lokal akan meningkat. Dengan demikian, populasi sapi lokal akan meningkat dan dapat mengurangi ketergantungan Indonesia akan daging maupun sapi dari negara lain.
Peningkatan produktivitas sapi pedaging Indonesia akan lebih tepat bila dilakukan melalui seleksi yang tidak hanya berdasarkan pada penampakan luar (fenotip), melainkan dikombinasikan dengan seleksi langsung pada tingkat DNA yang mengkodekan fenotip yang ingin diperbaiki kualitasnya. Peta genom pada sapi (bovine genome map) yang dibuat berdasarkan marker pada DNA genom menggunakan teknik molekuler seperti RFLP, mikrosatelit, minisatelit, PCR-RFLP, dan PCR-SSCP dimungkinkan untuk mengidentifikasi lokus-lokus gen yang bertanggung jawab terhadap variasi sifat yang memiliki nilai ekonomi.
digunakan untuk membuat keputusan tentang konservasi pada ternak sapi (Sodhi et al. 2006).
Patricia et al. (2002) mengggunakan DNA mitokondria untuk membuktikan dan menganalisa variasi dalam dan antarspesies, stuktur populasi dan filogeni. Hal ini juga dibenarkan oleh Muladno (2006) bahwa DNA mitokondria terutama daerah D-loop, sangat baik digunakan untuk analisis keragaman hewan, baik intraspesies maupun antarspesies. Penelitian yang dilakukan oleh Nijman et al. (2003) dan Edwards et al. (2007), penentuan daerah D-loop mtDNA pada sapi telah dapat menunjukkan sejarah sapi, dan hibridisasi yang terjadi pada Banteng dan sapi Madura. Berdasarkan penelitian Abdulah (2008), runutan daerah D-loop DNA mitokondria sapi Aceh berada satu klaster dengan sapi Pesisir dan PO serta mempunyai jarak genetik yang lebih dekat dengan bangsa-bangsa sapi Bos indicus (zebu) dari India, sedangkan sapi Bali dan Madura membentuk klaster sendiri. Dengan menggunakan analisis DNA mikrosatelit dibuktikan bahwa sapi Aceh memiliki klaster yang sama dengan sapi PO dan Pesisir serta satu kelompok dengan sapi Madura.
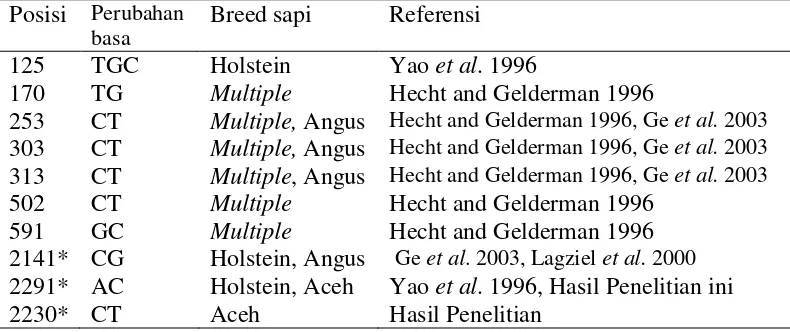
Penanda genetik molekuler yang terbaru yaitu SNP (Single Nucleotide Polymorphism), yang dapat mengidentifikasikan dengan jelas posisi perubahan hanya satu basa nukleotida pada sekuen DNA. Lucy et al. (1993) dan Zhang et al. (1992) menemukan SNP pada ekson lima (codon 127) dimana terjadi perubahan leusina ke valina (CTG – GTG) pada molekul GH. Ge et al. (2003) menggunakan metode sekuensing pada sapi Angus dan menemukan tiga SNP yang baru pada daerah promotor. Yao et al. (1996) mengidentifikasikan SNP pada ekson lima gen GH, dimana terjadi perubahan basa A menjadi basa C.
Berdasarkan uraian di atas, maka perlu dilakukan penelitian untuk mengindentifikasikan keragaman genetik gen hormon petumbuhan (GH) dan hubungannya dengan kualitas karkas sapi Aceh. Penentuan keragaman genetik ini dapat dipakai sebagai indikator untuk melakukan seleksi genetik sapi Aceh. Perbaikan kualitas karkas dan daging pada sapi Aceh melalui penggunaan marka gen hormon pertumbuhan (GH) dapat meningkatkan nilai ekonomis ternak sapi Aceh. Selanjutnya melestarikan keberadaan sapi Aceh dapat juga dilakukan dengan mengkarakterisasikan keragaman genetik dengan penanda genetik molekuler SNPs, D-Loop dan mikrosatelit, sehingga seluruh informasi yang didapat dari hasil penelitian dapat melengkapi data yang telah ada untuk dijadikan sebagai database dalam melakukan kebijakan pelestarian sapi Aceh dan penggunaannya yang berkelanjutan.
Tujuan Penelitian
a. Mengidentifikasi posisi perubahan basa nukleotida sekuen DNA gen GH pada ekson lima, dan mempelajari jumlah dan frekuensi alel SNPs pada populasi sapi Aceh.
b. Mendeterminasi polimorfisme genotipe dan alel gen GH/AluI serta pengaruh dari masing-masing genotipe terhadap kualitas karkas pada sapi Aceh
c. Mengidentifikasi keragaman pada populasi sapi Aceh dengan menggunakan marker genetik D-Loop dan Mikrosatelit
Manfaat Penelitian
a. Hasil penelitian ini dapat digunakan sebagai acuan dalam rangka perbaikan mutu genetik sapi Aceh melalui aplikasi teknik biologi molekuler
b. Dimungkinkan penciri genetik yang diperoleh dapat digunakan sebagai MAS (Marker Assisted Selection) dalam percepatan perbaikan mutu genetik sapi Aceh dan pengembangan ternak nasional pada umumnya.
Hipotesis
1. Penciri PCR-RFLP fragmen gen GH/AluI dan marker genetik D-Loop serta mikrosatelit memiliki polimorfisme yang tinggi pada sapi Aceh. 2. Terdapat korelasi yang nyata antara polimorfisme gen GH/AluI dengan
SAPI ACEH
PEMECAHAN MASALAH
Kehilangan/Punahnya Plasma Nutfah
• Data Kuantitatif/Kualitatif • Data Molekuler terbatas
GENETIK
GEN GH
FENOTIPIK
PERFORMANS
KUALITAS KARKAS
PRODUKSI
Pemanfaatan Genetik Marker
Program Pemuliaan & Pemanfaatan Secara Berkelanjutan SEKUENSING
DARAH/DAGING
POLIMORFISME
Sumber daya ternak sapi di Indonesia saat ini terdiri atas tiga kelompok, yakni (1) ternak asli, (2) ternak impor dan (3) ternak yang telah beradaptasi. Sehubungan dengan pentingnya nilai konservasi pada kelompok ternak ini, beberapa bangsa sapi menjadi target konservasi sekaligus pemanfaatannya (Utoyo 2002). Sumber daya ternak sapi tersebut diantaranya ialah sapi Aceh, Bali, Peranakan Ongole, Sumba Ongole, Madura, Jawa, Pesisir dan Grati. Keanekaragaman sapi di Indonesia menurut Utoyo (2002) terbentuk dari sumber daya genetik asli dan impor. Bos indicus Ongole dari India telah diimpor mulai pada awal abad ke-20 dan bangsa sapi ini memegang peranan penting dalam program pengembangan peternakan di Indonesia. Sapi Ongole murni pertama dibawa ke pulau Sumba kemudian disebut sebagai Sumba Ongole. Sapi ini kemudian dibawa ke tempat-tempat lain untuk disilangkan dengan sapi asli Jawa dan terbentuk Peranakan Ongole dan sapi Madura. Hasil domestikasi spesies liar Bos (bibos) banteng adalah sapi Bali (Bos sondaicus) atau Bos javanicus (Talib et al. 2002) yang sekarang telah menjadi salah satu bangsa ternak asli Indonesia (Martojo 2003).
Menurut ILRI (1995), terdapat tujuh ternak sapi asli di Indonesia, yaitu Sumba Ongole, Ongole cross, Bali, Madura, Aceh, Pesisir dan Grati. Sapi Aceh sebagai salah satu sumber daya genetik ternak lokal dan menjadi aset plasma nutfah nasional berperan sangat penting sebagai sumber daging bagi masyarakat di Nanggroe Aceh Darussalam dan sekitarnya. Selain berperan sebagai sumber daging, sapi Aceh juga sangat populer untuk kebutuhan hewan kurban dan hari besar Islam lainnya. Keunggulan sapi Aceh lainnya mempunyai daya tahan terhadap lingkungan yang buruk seperti krisis pakan, air dan pakan berserat tinggi, penyakit parasit, temperatur panas dan sistem pemeliharaan tradisional (Gunawan 1998).
Pelestarian terhadap sumber daya genetik ternak lokal sebagai bagian dari komponen keanekaragaman hayati adalah penting untuk memenuhi kebutuhan pangan di masa yang akan datang. Ada beberapa alasan untuk hal tersebut, antara lain (1) lebih dari 60 persen dari bangsa-bangsa ternak di dunia berada di negara sedang berkembang, (2) konservasi bangsa ternak lokal tidak menarik bagi petani, (3) secara umum tidak ada program monitoring yang sistematis dan tidak tersedianya informasi deskriptif dasar sumber daya genetik hewan ternak dan (4) sedikit sekali bangsa-bangsa ternak asli yang telah digunakan dan dikembangkan secara aktif (FAO 2001).
Karakteristik Sapi Aceh
Madura dan PO, namun lebih besar dari rataan bobot badan dan ukuran tubuh sapi Pesisir di Sumatera Barat.
Keragaman yang cukup tinggi pada karakter bobot badan, lebar dada, dalam dada, lebar pinggul dan lingkar dada pada semua kelompok umur, dapat digunakan sebagai suatu ukuran seleksi pada sifat-sifat tersebut. Menurut Astuti (2004), seleksi sapi lokal dengan memanfaatkan keragaman karakteristik sifat produksi dan reproduksi akan meningkatkan produktivitas, disamping itu dapat meningkatkan pelestarian terhadap sumber daya genetik ternak sapi
Gambar 2. Sapi Aceh di BPTU Indrapuri NAD
Gen Hormon Pertumbuhan
Pertumbuhan merupakan suatu proses deposisi, pemindahan substansi sel-sel, peningkatan ukuran dan jumlah pada tingkat dan titik berbeda dalam suatu waktu tertentu (Lawrence dan Fowler 2002). Pertumbuhan secara efektif dikontrol oleh hormon dan salah satu hormon yang penting dalam mengatur proses pertumbuhan adalah Growth Hormone (hormon pertumbuhan). Hormon pertumbuhan (GH) dibutuhkan untuk pertumbuhan jaringan, metabolisme lemak, metabolisme protein, pengaturan reproduksi, laktasi dan pertumbuhan tubuh normal (Akers 2006, Beauchemin et al. 2006, ThidarMyint et al. 2008)
Pertumbuhan sapi dipengaruhi oleh faktor lingkungan dan faktor genetis. Faktor lingkungan meliputi: pakan, baik hijauan maupun konsentrat, air, iklim, manajemen dan fasilitas pemeliharaan yang lain. Pengaruh pertumbuhan yang disebabkan faktor lingkungan ini tidak diturunkan kepada anaknya, sedangkan faktor genetis yang dikendalikan oleh gen akan diturunkan kepada keturunannya. Pertumbuhan dikendalikan oleh beberapa gen yang dibedakan menjadi major gene yang pengaruhnya besar/utama dan minor gene yang pengaruhnya kecil. Salah satu gen yang diduga merupakan gen utama dalam mempengaruhi pertumbuhan adalah gen pengkode hormon pertumbuhan yang berperan terhadap sekresi hormon pertumbuhan (Sutarno et al. 2005). Hormon pertumbuhan merupakan hormon anabolik yang disintesis dan disekresikan oleh sel somatotrof pada lokus anterior (Ayuk dan Sheppard 2006). Hormon pertumbuhan pada sapi memiliki ukuran sebesar 22 kilo Dalton (kDa) (Dybus 2002) yang tersusun oleh 190-191 asam amino sebagai produk dari gen hormon pertumbuhan.
meningkatkan kecepatan sintesis protein dan berdampak pada peningkatan jumlah sel sehingga mempercepat laju pertumbuhan jaringan di berbagai bagian tubuh.
Zakizadeh et al. (2006) menyatakan bahwa gen hormon pertumbuhan (GH) pada sapi Bos taurus memiliki panjang sekuens nukleotida 2856 bp dengan bagian open reading frame-nya 1800 bp. Selanjutnya dinyatakan bahwa gen GH terdiri atas lima ekson dan dipisahkan oleh empat intron dengan panjang sekuens nukleotida yang berbeda antara ekson dan intron. Gen GH merupakan gen yang menyandi hormon pertumbuhan sebagai produknya dan terletak di kromosom 19 pada ternak sapi (Tatsuda et al. 2007). Gen GH berdasarkan data PCR-RFLP telah diketahui memiliki keragaman yang tinggi (Curi et al. 2006) dan polimorfisme untuk enzim pemotong AluI di ekson lima, yaitu tranversi dari nukleotida sitosin (C) menjadi guanin (G) pada posisi asam amino 127 yang menyebabkan asam amino berubah dari leusin menjadi valin (Lucy et al. 1993).
Adanya polimorfisme pada gen GH juga berhubungan dengan sifat produksi (Pereira et al. 2005), deposisi daging (Oprzadek et al. 2003) dan bobot karkas (Beauchemin et al. 2006). Hubungan yang nyata didapatkan antara genotipe LL dan LV dari gen GH dengan rataan bobot badan hidup ternak dari bangsa sapi Alentejana, Marinhoa dan Preta asal Portugis (Reis et al. 2001) Unanian et al. (2000) melaporkan, bahwa adanya polimorfisme GH AluI dan GH MspI mungkin dapat dijadikan sebagai penciri genetik yang potensial untuk sifat pertumbuhan bobot badan pada sapi pejantan muda.
Grochowska et al. (2001) mendapatkan sapi perah bergenotipe LV memproduksi susu dengan persentase protein lebih tinggi dibandingkan genotipe LL dan VV, tetapi genotip VV mempunyai kadar lemak lebih tinggi. Di Stasio et al. (2005) melaporkan hormon pertumbuhan GH sangat berpengaruh terhadap pertumbuhan dan metabolisme melalui interaksi dengan spesifik receptor (GHR) pada permukaan sel target, sehingga GHR menjadi gen kandidat yang berpengaruh terhadap produksi daging pada sapi.
Penanda Molekuler
Aplikasi penanda molekuler dapat digunakan dan relatif lebih tepat dalam pembuatan peta genetik, selanjutnya digunakan untuk identifikasi lokasi suatu gen yang bertanggung jawab terhadap suatu sifat. Penanda molekuler ini berada pada tingkat DNA, maka penanda ini bebas dari pengaruh epistasis, lingkungan dan fenotip. Dengan demikian, penanda molekuler menyediakan informasi genetik yang definitif untuk mempelajari keragaman genetik, mendeteksi gen-gen mayor dan mempelajari sifat-sifat genetik yang komplek (Paterson et al. 1991).
Marker genetik merupakan gen atau DNA sekuen yang telah diketahui letaknya pada sebuah kromosom, yang dapat digunakan untuk identifikasi sel, individu atau spesies. Marker genetik dapat didiskripsikan sebagai sebuah variasi (yang disebabkan oleh mutasi dalam lokus genom) yang dapat diamati. Marker genetik dapat berupa sekuen DNA yang pendek, seperti SNP (single nucleotide polymorphism) dan sekuen yang panjang seperti minisatelit (Rayaa et al. 2002)
Beberapa jenis marker genetik yang biasa digunakan adalah
(RFLP) SSR
kodominan (RFLPs, mikrosatelit) hanya menganalisa satu lokus, sehingga informasi yang didapat lebih akurat, karena variasi alel pada lokus tersebut dapat dihindari. Identifikasi linkage group antara kelompok genetik yang berbeda sebagai konsekuensinya menjadi lebih mudah (Rayaa et al. 2002)
Single-nucleotide polymorphism (SNP) adalah suatu variasi yang terjadi pada
sekuen DNA ketika satu nukleotida – A, T, C, atau G- dalam genom berbeda antara satu individu dengan individu yang lain dalam suatu populasi. Sebagai contoh, ada dua sekuen fragmen DNA dari dua individu yang berbeda: AAGCCTA dan AAGCTTA, terdapat berbedaan pada satu basa nukleotidanya. Pada kasus ini terdapat dua alel yaitu alel C dan T dan biasanya SNPs hanya memiliki dua alel (Rapley dan Harbron. 2004).
kode genetik. SNP yang terbentuk dalam sekuen polipeptida yang sama disebut synonymous atau silent mutation, jika pada sekuen polipeptida yang berbeda disebut nonsynonymous. Perubahan pada nonsynonymous dapat berupa perubahan missense atau nonsense, yaitu missense mengakibatkan perubahan pada asam amino, sedangkan nonsense mengakibatkan perubahan yang tidak sempurna pada stop codon (Syvanen 2001).
DNA mikrosatelit. Penanda mikrosatelit digunakan secara luas sebagai penanda
genetik di dalam studi populasi dan verifikasi silsilah keturunan (Cervini et al. 2006). Hal ini dikarenakan mikrosatelit mengandung informasi polimorfisme yang tinggi dan tersebar luas di dalam genom eukariot. Mikrosatelit digunakan Mukesh et al. (2004) untuk mengkaji keragaman genetik dan menetapkan hubungan di antara tiga bangsa sapi zebu India (Sahiwal, Hariana dan Deoni). Penggunaan QTL dengan mikrosatelit untuk sifat pertumbuhan dan komposisi karkas pada sapi telah dilaporkan oleh sejumlah peneliti (Casas et al. 2004; Curi et al. 2006).
D-Loop DNA. DNA mitokondria telah banyak digunakan sebagai penanda
molekul untuk studi genetika populasi dan penelusuran asal-usul (Wandia 2001). DNA mitokondria hewan secara umum memiliki jumlah dan jenis gen yang sama yaitu 13 daerah yang mengkode protein dan 22 gen pengkode tRNA (Duryadi 1994). Daerah bukan pengkode hanya terdiri atas daerah kontrol yang memegang peranan penting dalam proses transkripsi dan replikasi genom mitokondria. Daerah bukan penyandi utama terletak pada wilayah displacement-loop (D-loop).
D-loop pada DNA mitokondria memiliki laju perubahan yang lebih tinggi dibandingkan dengan daerah DNA mitokondria lainnya, sehingga DNA mitokondria terutama daerah D-loop sangat baik digunakan untuk analisis keragaman hewan, baik di dalam spesies maupun antarspesies (Muladno 2006).
Faktor yang Berpengaruh pada Kualitas Daging dan Karkas
Hasil penelitian mengindentifikasikan bahwa bangsa dari suatu ternak berperan penting terhadap kualitas daging dan karkas ternak tersebut. Perbedaan kualitas daging dan karkas ditentukan oleh periode penggemukan, bobot badan, skor lemak dan kandungan lemak intramuskuler. Perbedaan ukuran pada saat mencapai dewasa merupakan komponen utama perbedaan karkas pada bangsa ternak (Sanudo et al. 2004).
Bobot potong dan umur ternak merupakan dua faktor yang berpengaruh terhadap kualitas karkas dan daging dari ternak dengan bangsa yang sama. Beberapa penelitian menunjukkan adanya pengaruh yang nyata dari umur saat pemotongan terhadap bobot potong, tebal lemak punggung (Du Plessis dan Hoffman. 2007), bobot karkas (Mojto et al. 2009), pH dan daya putus Warner Bratzler (Irurueta et al. 2008; Rao et al. 2009). Du Plesis dan Hoffman (2007) menyatakan, bahwa sapi yang lebih tua menghasilkan daging yang lebih alot. Sementara sapi yang berumur 30 bulan memiliki bobot badan dan bobot karkas yang lebih tinggi dari sapi umur 18 bulan, kandungan lemak dan persentase susut masak yang lebih tinggi serta rib eye area yang lebih besar (Mojto et al. 2009).
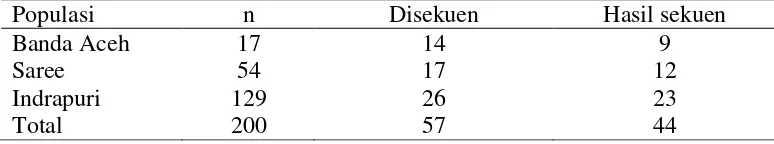
Kegiatan penelitian ini meliputi kegiatan lapang dan kegiatan laboratorium. Kegiatan lapang dilakukan melalui pengamatan dan pengambilan data di Balai Pembibitan Ternak Unggul (BPTU) Indrapuri dan Saree Kabupaten Aceh Besar, Rumah Potong Hewan (RPH) kota Banda Aceh dan RPH Kabupaten Aceh Besar. Kegiatan laboratorium ekstraksi DNA dan karakterisasi keragaman gen GH/Alu I dilakukan di Laboratorium Genetika Molekuler Ternak Bagian Pemuliaan dan Genetika Fapet IPB, sedangkan identifikasi SNP, D-Loop dan DNA Mikrosatelit dilakukan di Laboratorium Molekuler dan Genetik CAAS (Chinese Academy of Agricultural Sciences)-ILRI (International Livestock Research Institute) Beijing-China. Pengujian kualitas karkas dan daging dilakukan di Laboratorium Ruminansia Besar, Departemen Ilmu Produksi dan Teknologi Peternakan. Kegiatan penelitian ini dilaksanakan dari bulan Oktober 2009 – November 2010.
Sampel Darah Sapi Aceh
Penelitian ini menggunakan sapi Aceh yang merupakan sapi lokal yang berasal dari Propinsi Nanggroe Aceh Darussalam. Penentuan sampel penelitian di BPTU Indrapuri (Lampiran 1) dan Saree (Lampiran 2), karena kedua tempat tersebut merupakan Balai pembibitan Ternak Unggul sapi Aceh dan RPH Kotamadya Banda Aceh (Lampiran 2) mewakili populasi sapi Aceh di Kotamadya Banda Aceh. Jumlah sampel dari BPTU Indrapuri sebanyak 129 sampel, Saree Aceh Besar sebanyak 54 sampel dan RPH Kotamadya Banda Aceh sebanyak 17 sampel. Pengambilan sampel darah sapi Aceh menggunakan tabung
venojact 5 ml pada vena jugularis, kemudian disimpan pada kotak es (ice box) untuk dibawa ke Laboratorium Genetika dan Pemuliaan Ternak Fapet IPB.
Tabel 1 Jumlah sampel darah yang digunakan untuk analisis gen GH/SNP Populasi n Disekuen Hasil sekuen
Banda Aceh 17 14 9
Saree 54 17 12
Indrapuri 129 26 23
Total 200 57 44
Sampel DNA yang digunakan untuk sekuensing D-Loop (Tabel 2) berasal dari Banda Aceh (11), Saree (20) dan Indrapuri (29). Sekuen sapi pembanding yang digunakan berasal dari sekuen sapi yang berasal dari GenBank (Lampiran 4).
Tabel 2 Jumlah sampel darah yang digunakan untuk analisis D-Loop Populasi n Disekuen Hasil sekuen
Banda Aceh 17 11 7
Saree 54 20 2
Indrapuri 129 29 20
Total 200 57 29
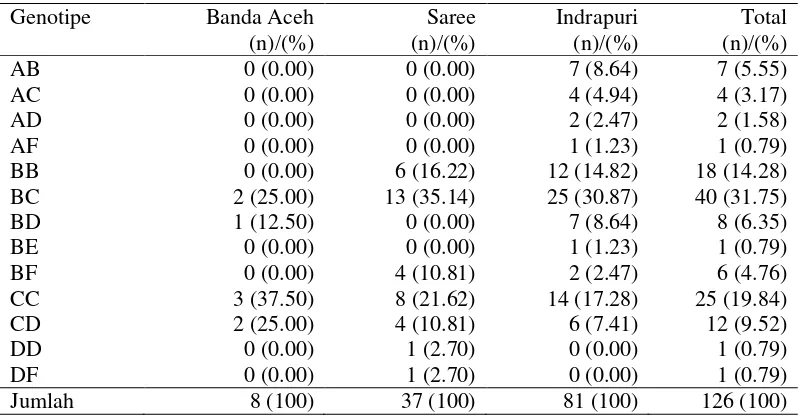
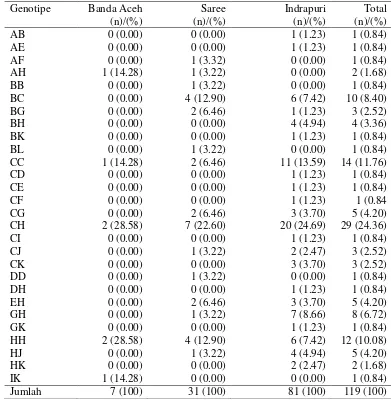
Pengambilan sampel darah sapi Aceh untuk DNA mikrosatelit (Tabel 3) berasal dari Banda Aceh (17), Saree (54) dan Indrapuri (129).
Tabel 3 Jumlah sampel darah yang digunakan untuk analisis mikrosatelit
Populasi n
Hasil sekuen
BM1824 SPS115 ILSTS028
Banda Aceh 17 8 3 7
Saree 54 37 9 31
Indrapuri 129 81 40 81
Total 200 126 52 119
Sampel Daging Sapi Aceh
digembalakan pada siang hari dan dikandangkan pada malam hari. Pemotongan dilakukan secara tradisional.
Tabel 4 Jumlah sampel daging yang digunakan untuk analisis gen GH/AluI
Populasi Jumlah sample
RPH Banda Aceh 12
RPH Aceh Besar 30
Total 42
Jumlah sapi yang dipotong sekitar 2-3 ekor/minggu. Pengambilan sampel otot sapi Aceh sebanyak 500 g. Pembekuan sampel otot dilakukan 4-5 jam setelah pemotongan, kemudian disimpan pada kotak es (ice box) dan dibawa ke Laboratorium Genetika dan Pemuliaan Ternak Fapet IPB untuk ekstrasi DNA dan Laboratorium Ruminansia Besar, Departemen Ilmu Produksi dan Teknologi Peternakan untuk pengujian kualitas karkas dan daging.
Ekstraksi DNA Genom (Darah)
Ekstraksi DNA genom yang berasal dari darah sapi Aceh dilakukan dengan metoda Sambrook et al. (1989) yang telah dimodifikasi dengan menggunakan buffer lysis sel (400 µl 1 x STE, dan 40 µl 10% SDS dan 10 µl
proteinase-K). DNA dimurnikan dengan metode fenol-kloroform, yaitu dengan menambahkan 40 µl 5 M NaCl dan 400 µl fenol dan kloroform iso amil alkohol (CIAA). DNA diendapkan dengan 40 µl 5 M NaCl dan 800 µl etanol absolut. Endapan dicuci satu kali dengan menambahkan 800 µl 70% etanol, disentrifugasi dengan kecepatan 12.000 rpm selama 5 menit, etanol (supernatan) dibuang dan pelet diuapkan. Endapan/pelet DNA dilarutkan dengan 10 µl 80% TE (Elution buffer).
Ekstraksi DNA Genom (Daging)
µl, dan dibiarkan selama 20 menit. Sampel kemudian diendapkan dengan sentrifugasi pada kecepatan 8000 rpm selama 5 menit. Sampel yang telah bersih dari alkohol ditambahkan 1x STE (sodium tris EDTA) sampai volume 340 µl, 40 µl sodium dosesil sulfat 10% dan 20 µl proteinase-K 5 mg/ml. Campuran diinkubasi pada suhu 50 ºC selama semalam sambil digoyang pelan.
Sampel yang telah diinkubasi ditambahkan 400 µl larutan fenol, 400 µl kloroform iso amil alkohol (24:1) dan 40 µl NaCl 5M. Campuran digoyang pada suhu ruang selama satu jam. Sampel selanjutnya disentrifugasi pada kecepatan 5000 rpm selama 10 menit hingga fase air terpisah dengan fase fenol. Fase air dipindahkan dalam tabung baru dengan volume terukur. Molekul DNA diendapkan dengan cara menambahkan 2x volume alkohol absolut dan 0,1x volume NaCl 5M. Campuran kemudian diinkubasi pada suhu -20 ºC selama semalam. Pengendapan DNA selanjutnya dilakukan dengan sentrifugasi pada kecepatan 12000 rpm selama 10 menit. Endapan DNA yang diperoleh dicuci dengan alkohol 70%, kemudian diendapkan lagi. Endapan DNA yang telah bersih dari alkohol dipulihkan dengan menambahkan 100 µl TE (Tris EDTA). Sampel DNA disimpan pada suhu -20 ºC dan siap untuk digunakan.
Identifikasi Single Nucleotide Polymorphism (SNP)
Amplifikasi DNA. DNA diamplifikasi dengan metode Polymerase Chain Reaction (PCR). Mesin PCR yang digunakan adalah GeneAmp PCR System 9700 Applied Biosystem. Setiap reaksi PCR dibuat dengan volume 50 µl terdiri atas 5 µl 1x buffer PCR; 4µl dNTP;1 µl Taq DNA Polymerase; 1 µl Primer Forward
Tabel 5 Basa primer GH/AluI (Gen Bank M57764.1, Gordon et al. 1983)
Lokus
Suhu
Annealing
Produk
PCR Sekuen Primer
GH 59 ºC 404 bp F:5'-TAGGGGAGGGTGGAAAATGGA-3' R:5'-GACACCTACTCAGACAATGCG-3'
Elektroforesis Produk PCR. Produk PCR dielektroforesis dengan mesin
Applied Biosystem 3130x Genetic Analyzer dan dianalisis dengan software
GeneMapper versi 4.0. Hasil analisis pada layar monitor akan menunjukkan grafik dengan puncak-puncak (peak) dalam ukuran base pairs (bp) pada masing-masing sampel. Tampilan grafik yang konsisten membentuk satu ukuran puncak yang sama menunjukkan bahwa sampel teramplifikasi dan memiliki alel yang homozigot. Sementara sampel yang memiliki dua puncakdengan ukuran berbeda menunjukkan dua alel yang heterozigot. Apabila terdapat tampilan grafik yang tidak beraturan, ini menandakan hasil yang kosong atau tidak bisa dinilai. Apabila diperoleh tanda sinyal yang lebih dari dua grafik, maka sampel tersebut tercemar dengan DNA yang lain dan tidak digunakan.
Analisis Data. Analisis data SNP dari hasil sekuen fragmen gen GH sapi Aceh di analisis kesamaan sekuensnya (homology) dengan sekuen yang terdapat di
GenBank (Lampiran 3) dengan software program Basic LocalAlignment Search Tool (BLAST) (Altchul et al. 1997) [www.ncbi.nhl.nih.gov./BLAST]. Analisis ini bertujuan untuk memastikan bahwa sekuen yang dianalisis adalah fragmen gen GH dengan melihat ukuran panjang basanya, dapat juga untuk mengetahui ada tidaknya mutasi yang terjadi pada gen GH dan sekuen basa nukleotida spesifik. Kesamaan suatu sekuen adalah tinggi apabila nilai yang didapat > 200 bits (Lampiran 6).
Xi = (2nii + ∑nij) / (2N)
Amplifikasi DNA . DNA D-Loop diamplifikasi dengan Polymerase Chain Reaction (PCR). Mesin PCR yang digunakan adalah GeneAmp PCR System 9700 Applied Biosystem. Setiap reaksi PCR memerlukan volume 50 µl dengan komposisi sebagai berikut: 5 µl 1x buffer PCR; 4µl dNTP; 1 µl Taq DNA Polymerase; 1 µl primer Forward; 1 µl primer Reverse, 4 µl DNA; dan 34 µl dH2
Primer Reff
O. Denaturasi awal pada suhu 95 ºC selama 5 menit dilakukan 1x ulangan, dilanjutkan dengan 31 x ulangan masing-masing dengan langkah denaturasi pada suhu 94 ºC selama 30 detik, 30 detik annealing pada suhu 60 ºC, elongasi pada suhu 72 ºC selama 30 detik, dilanjutkan ekstensi pada suhu 72 ºC selama 5 menit. Produk PCR disimpan pada suhu 4 ºC selama 25 menit. Susunan basa primer yang digunakan, ditampilkan pada Tabel 6 .
Tabel 6 Basa primer D-Loop Suhu
annealing
Produk
PCR Sekuen Primer Loftus et
al. 1994 60 ºC 980 bp F:5'-CTGCAGTCTCACCATCAACC-3' R:5'-GCTGGGACCAAACCTATGTC-3'
Elektroforesis Produk PCR. Produk PCR dielektroforesis dengan mesin
Applied Biosystem 3130x Genetic Analyzer dan dianalisis dengan software
GeneMapper versi 4.0. Hasil analisis pada layar monitor menunjukkan grafik dengan puncak-puncak (peak) dalam ukuran base pairs (bp) pada masing-masing sampel.
GenBank (Lampiran 4). Program dnaSP untuk mendapatkan NJ tress dan mengidentifikasikan haplotipe, kemudian digunakan software NETWORK
IdentifikasiDNA Mikrosatelit
Amplifikasi DNA Mikrosatelit . Mikrosatelit diamplifikasi melalui Polymerase Chain Reaction (PCR). Mesin PCR yang digunakan GeneAmp PCR System 9700 Applied Biosystem. Setiap reaksi PCR dibuat volume 15 µl dengan komposisi reaksi PCR mengandung 1.5 µl 1x buffer PCR; 1µl dNTP; 0.25 µl Taq DNA Polymerase; 0.25 µl primer; 1.5 µl DNA dan 10.5 µl dH2
Lokus
O. Denaturasi awal pada suhu 94 ºC selama 5 menit dilakukan 1x ulangan, dilanjutkan dengan 33 x ulangan dengan langkah denaturasi pada suhu 94 ºC selama 30 detik, 30 detik
annealing pada suhu 55.5 ºC - 62 ºC, elongasi pada suhu 72 ºC selama 30 detik, dilanjutkan ekstensi pada suhu 72 ºC selama 5 menit, dan suhu penyimpanan 4 ºC selama 25 menit. Susunan basa primer yang digunakan, ditampilkan pada Tabel 7.
Tabel 7 Basa primer mikrosatelit lokus BM1824, SPS115 dan ILSTS028
Suhu
annealing
Produk
PCR (bp) Sekuen Primer
BM1824 58 ºC 180-194 F:5'-GAGCAAGGTGTTTTTCCAATC-3' R:5'-CATTCTCCAACTGCTTCCTTG-3' SPS115 65 ºC 234-254 F:5'-AAAGTGACACAACACGTTCTCCAG-3'
R:5'-AACGAGTGTCCTAGTTTGGCTGTC-3' ILSTS028 55 ºC 131-165 F:5'-TCCAGATTTTGTACCAGACC-3'
R:5'-GTCATGTCATACCTTTGAGC-3'
Elektroforesis Produk PCR. Produk PCR dielektroforesis dengan mesin
Applied Biosystem 3130x Genetic Analyzer dan dianalisis dengan software
yang tidak beraturan, ini menandakan hasil yang kosong, bila diperoleh tanda sinyal yang lebih dari dua grafik, maka sampel tersebut tercemar dengan DNA yang lain dan tidak digunakan.
Gambar 3. Grafik hasil elektroforesis Genetic Analyzer Applied Biosystem 3130x
Analisis Data. Analisis data mikrosatelit dilakukan dengan software
GeneMapper versi 4.0 (Applied Biosystems) dan hasil yang di peroleh di masukkan ke dalam tabulasi data sheet Excel. Frekuensi masing-masing alel setiap lokus mikrosatelit dihitung berdasarkan rumus Nei dan Kumar (2000):
Xi = (2nii + ∑nij) / (2N)
Keterangan:
Xi : frekuensi alel ke-i
nii : jumlah individu untuk genotipe AiAi nij : jumlah individu untuk genotipe AiAj N : jumlah sampel
Derajat heterozigositas (ĥ) dihitung berdasarkan frekuensi alel pada tiap lokus DNA mikrosatelit dengan rumus Nei dan Kumar (2000) sebagai berikut:
ĥ= 2n (1-∑xi2)/(2n-1) Keterangan:
xi : frekuensi alel lokus ke-i n : jumlah sampel
Identifikasi Gen GH/AluI
Amplifikasi Gen GH. Amplifikasi ruas gen GH dilakukan dengan metode PCR (polymerase chain reaction). Mesin PCR yang digunakan mesin thermal cycler
(Ependorf 5332). Pereaksi yang digunakan untuk amplifikasi ruas gen target adalah 2 µl sampel DNA, masing-masing primer 25 pmol, campuran dNTP 200 µM, MgCl2
Tabel 8 Basa primer GH/AluI (Gen Bank M57764.1, Gordon et al. 1983)
1 mM, taq polymerase 0.5 unit dan bufernya dalam larutan total 25 µl. Amplifikasi in vitro dengan mesin thermal cycler dilakukan dengan kondisi denaturasi awal pada suhu 94 ºC selama 5 menit, 35 siklus yang terdiri atas denaturasi pada suhu 94 ºC selama 30 detik, penempelan primerpada suhu 63 ºC selama 45 detik dan pemanjangan DNA baru pada suhu 72 ºC selama 1 menit, dan pemanjangan akhir pada suhu 72 ºC selama 5 menit. Susunan basa primer yang digunakan dapat dilihat pada Tabel 8.
Lokus
Suhu
annealing
Produk
PCR Sekuen Primer
GH/AluI 59 ºC 404 bp F:5'-TAGGGGAGGGTGGAAAATGGA-3' R:5'-GACACCTACTCAGACAATGCG-3'
Penentuan Genotipe dengan Pendekatan PCR-RFLP
Penentuan genotipe masing-masing individu dilakukan dengan pendekatan restriction fragment length polymorphism (RFLP) yang divisualisasikan pada gel agarosa 1.5% dengan bufer 0,5x TBE (tris borat EDTA) yang difungsikan pada tegangan 100 V selama 40 menit yang diwarnai dengan etidium bromida diatas UV trans illuminator. Enzim pemotong yang digunakan untuk ruas gen target adalah AluI.
Analisa Data
Frekuensi Gen. Frekuensi genotipe merupakan rasio dari jumlah suatu genotipe terhadap jumlah populasi. Frekuensi alel merupakan rasio suatu alel terhadap keseluruhan alel pada suatu lokus dalam suatu populasi. Frekuensi masing-masing alel setiap lokus dihitung berdasarkan rumus Nei dan Kumar (2000):
Keterangan: nii : jumlah individu untuk genotipe ii Xi : frekuensi alel ke-i
nij : jumlah individu untuk genotipe ij N: jumlah sampel
Metode Penilaian Kualitas Karkas dan Daging Sapi Aceh. Penilaian kualitas karkas meliputi bobot potong dan luas urat daging mata rusuk. Prosedur penilaian sebagai berikut:
Bobot Potong: Bobot potong merupakan bobot sapi yang dihitung pada saat sapi akan dipotong. Bobot sapi diestimasi dari lingkar dada dengan menggunakan pita ukur.
Luas Urat Daging Mata Rusuk: Pengukuran luas urat daging mata rusuk dilakukan pada irisan rusuk 12 dan 13. Patron permukaan irisan luas urat daging mata rusuk dicetak pada plastik dan digambar dengan spidol. Gambar bidang permukaan penampang melintang urat daging mata rusuk ditera dengan plastik grid (satuan satu inchi2 tiap 10 titik) selanjutnya dihitung dalam satuan cm2, jumlah titik yang tercakup pada bidang penampang melintang dijadikan ukuran luas urat daging mata rusuk.
Penilaian kualitas daging meliputi nilai pH, persentase susut masak, nilai keempukan daging, daya mengikat air dan warna daging. Prosedur untuk pengukuran parameter tersebut diatas diuraikan sebagai berikut:
Nilai pH Daging: pengukuran nilai pH daging menggunakan alat pH meter. Sampel daging sebanyak 10 gram ditambahkan 100 ml aquades, kemudian dicampur dengan mixer selama 1 menit selanjutnya diukur dengan pH meter yang telah dikalibrasi pada buffer 4.0 dan 7.0 dan dicatat hasilnya.
Persentase Susut Masak (cooking loss): yaitu perbedaan antara bobot daging sebelum dan sesudah dimasak dan dinyatakan dalam persentase (%). Sampel daging sekitar 100 g dimasukkan ke dalam air rebus, namun terlebih dahulu ditancapkan termometer bimetal sampai menembus bagian dalam daging. Sampel daging harus terendam dalam air rebusan sampai suhu dalam daging menunjukkan angka 81 C lalu diangkat. Setelah itu sampel didinginkan selama 60 menit lalu ditimbang, selanjutnya setiap 30 menit ditimbang sampai sampel daging mempunyai berat konstan.
Warna daging: ditentukan standar warna daging berdasarkan skor warna yaitu dengan menggunakan Photo Graphic Colour Standard. Skor warna tersebut memiliki skala angka dari 1 – 7, semakin besar skor maka warna daging dinyatakan semakin gelap. Cara mengukur yaitu dengan menentukan kesesuaian warna daging dengan standar warna daging, namun sebelumnya sampel daging disayat terlebih dahulu.
Daya mengikat air: dilakukan dengan metode penekanan (press method) sesuai dengan petunjuk Hamm (Swatland 1984) yaitu sampel daging sebanyak 0.3 gram dibebani pada kertas saring diantara dua plat kaca dengan beban tekan sebesar 35kg selama lima menit. Daerah yang tertutup sampel daging dan telah menjadi rata dan luas daerah basah di sekitarnya ditandai dan diukur dengan planimeter. Daerah basah diperoleh dengan perhitungan selisih daerah yang tertutup daging dari daerah total yang meliputi pula area basah pada kertas saring. Jumlah air yang keluar dari daging dihitung dengan rumus:
Area basah (cm2 0.0948
)
Area basah adalah luas air yang diserap pada kertas saring akibat penjepitan selama lima menit, yaitu selisih luas lingkaran luar dan dalam pada kertas saring. Pengukuran luas lingkaran dilakukan dengan planimeter (merk Hruden). Nilai kandungan air yang diperoleh berdasarkan rumus selanjutnya dipersentasikan terhadap bobot sampel, dengan demikian besar nilai yang didapat daya mengikat air daging akan semakin rendah.
Penelitian untuk mengidentifikasi polimorfisme gen GH pada sapi Aceh menggunakan beberapa sampel DNA untuk sekuensing yang berasal dari sapi Banda Aceh (14), Saree (17) dan Indrapuri (26), tetapi sampel DNA yang berhasil disekuen berjumlah empat puluh empat sampel, masing-masing berasal dari Banda Aceh (9), Saree (12) dan Indrapuri (23) sampel. Kegagalan sekuensing terhadap sampel disebabkan amplifikasi yang tidak berhasil dan terlalu banyak duplikasi puncak pada grafik sekuen. Analisis keragaman runutan nukleotida dilakukan setelah runutan DNA sapi Aceh disejajarkan dengan sampel sekuen pembanding yang berasal dari Gen Bank (Lampiran 3).
Polimorfisme pada gen GH telah banyak diidentifikasikan pada hewan ternak, tetapi hanya beberapa yang telah tepat ditandai untuk suatu perubahan basa nukleotida dan posisinya dalam urutan DNA. Efek dari beberapa polimorfisme gen GH telah dipelajari secara luas pada ternak sapi (Switonski 2002) dan terdapat beberapa polimorfisme yang dapat ditandai dengan penggunaan enzim restriksi yang berbeda. Kehadiran situs AluI sebagai pembatas menandakan adanya asam amino leusin (L) di posisi 127 dalam rantai polipeptida GH, sedangkan tidak adanya situs ini menunjukkan adanya valin (V) pada posisi yang sama. Polimorfisme ini dapat digunakan sebagai penanda tetapi belum banyak penelitian mengenai penggunaan marker atau penanda genetik ini pada sifat produksi ternak. Oleh karena itu, gen GH adalah kandidat gen penting sebagai marker genetik untuk identifikasi pertumbuhan dan karkas pada ternak.
Posisi
Tabel 9 Variasi sekuen di daerah promotor dan ekson dari gen GH pada sapi
Perubahan basa
Breed sapi Referensi 125 TGC Holstein Yao et al. 1996
170 TG Multiple Hecht and Gelderman 1996
253 CT Multiple, Angus Hecht and Gelderman 1996, Ge et al. 2003
303 CT Multiple, Angus Hecht and Gelderman 1996, Ge et al. 2003
313 CT Multiple, Angus Hecht and Gelderman 1996, Ge et al. 2003
502 CT Multiple Hecht and Gelderman 1996
591 GC Multiple Hecht and Gelderman 1996
2141* CG Holstein, Angus Ge et al. 2003, Lagziel et al. 2000
2291* AC Holstein, Aceh Yao et al. 1996, Hasil Penelitian ini 2230* CT Aceh Hasil Penelitian
Ket: Multiple: multiple breeds ( German Friesian, German Simmental, Jersey,Galloway, Scottish Highland, Charolais dan Ceylon Dwarf Zebu).
* Ekson lima gen GH
Perunutan nukleotida telah dilakukan pada ruas gen GH pada tiga populasi sampel sapi Aceh (Banda Aceh, Indrapuri dan Saree), analisis kesamaan runutan DNA hasil sekuensing dengan runutan nukleotida gen GH yang ada di Genbank (Lampiran 3) dilakukan dengan pendekatan BLAST (basic local alignment search tool). Pada runutan nukleotida gen GH sapi Aceh tidak ditemukan adanya perubahan basa C menjadi basa G pada posisi ke 2.141 bp, tetapi ditemukan perubahan basa A menjadi basa C pada posisi ke 2.291 bp (Tabel 10). Hasil ini penelitian ini sesuai dengan Yao et al. (1996) yang telah mengidentifikasikan adanya penyisipan dari tiga pasangan basa (TGC) di daerah promotor dan terjadi berubahan basa A ke basa C pada ekson lima pada sapi Holstein.
Tabel 10 Frekuensi alel dan genotipe SNP gen GH sapi Aceh pada posisi basa ke
Pada Tabel 10 terlihat bahwa populasi sapi Aceh yang berasal dari Banda Aceh hanya memiliki genotipe CC, sementara genotipe AA, AC dan CC dimiliki oleh populasi sapi Aceh yang berasal dari Saree dan Indrapuri. Genotipe CC memiliki nilai frekuensi yang lebih tinggi dari genotipe AA dan AC pada seluruh sampel populasi sapi Aceh (Banda Aceh, Saree, Indrapuri), sehingga frekuensi alel C memiliki nilai yang lebih tinggi dari frekuensi alel A.
Populasi sampel sapi Aceh dari Banda Aceh yang mengalami mutasi pada posisi ke 2.230 bp berjumlah empat sampel, dengan dua sampel memiliki basa T dan dua sampel lainnya memiliki basa C/T (Y). Selanjutnya tujuh sampel sapi Aceh yang berasal dari Saree memiliki basa C/T (Y) dan tidak ada satu pun sapi Aceh dari Saree yang memiliki basa T. Sementara sapi Aceh dari Indrapuri, empat sampel memiliki basa T dan tiga belas sampel memiliki basa C/T (Y). Frekuensi alel dan genotipe gen GH sapi Aceh pada posisi basa 2.230 bp dapat dilihat pada Tabel 11.
Tabel 11 Frekuensi alel dan genotipe SNP gen GH sapi Aceh pada posisi basa ke 2.230 bp
dari genotipe CC dan CT. Genotipe CC memiliki nilai yang lebih tinggi dari genotipe CT pada populasi sapi Aceh di Banda Aceh, sebaliknya genotipe CT memiliki nilai yang lebih tinggi dari genotipe CC pada populasi sampel sapi Aceh yang berasal dari Indrapuri. Hasil penelitian terhadap populasi sampel sapi Aceh, mendapatkan bahwa jumlah frekuensi alel C memiliki nilai yang lebih tinggi dari frekuensi alel A (Tabel 10) dan alel T (Tabel 11). Posisi mutasi pada basa ke 2.230 bp gen GH ekson lima dapat dilihat pada Gambar 4.
Gambar 4 Posisi mutasi basa ke 2.230 bp pada gen GH ekson lima
Hasil penelitian terhadap tiga populasi sapi Aceh yang berasal dari Banda Aceh, Saree dan Indrapuri pada sekuen gen GH, terdapat satu titik mutasi baru pada ekson lima diposisi basa ke 2.230 bp yaitu perubahan basa C menjadi basa T, yang diistilahkan sebagai Silent Mutation yaitu perubahan Leusin menjadi Leusin (CTC–CTT). Fenomena ini mengidentifikasikan bahwa populasi sapi Aceh memiliki mutasi yang tinggi pada sekuen gen GH diposisi basa ke 2.230 bp. Hasil penelitian ini merupakan penemuan baru terhadap titik mutasi yang terdapat diekson lima sekuen gen GH pada sapi Aceh, setelah runutan sekuen sapi Aceh disejajarkan dengan sekuen yang berasal dari Gen Bank (Lampiran 3).
Berdasarkan hasil runutan sekuen dapat diidentifikasikan bahwa mutasi yang terjadi pada sekuen gen GH sapi Aceh diposisi basa ke 2.230 bp sama dengan titik mutasi yang ditemukan pada sapi Zebu di China (kode akses EU344985) dan sapi Sahiwal (kode akses EF451795). Sementara sekuen gen GH dari gen bank lainnya tidak memiliki mutasi diposisi basa ke 2.230 bp.
2041 ttctccaagc ctgtagggga gggtggaaaa tggagcgggc aggagggagc tgctcctgag F
2101 ggcccttcgg cctctctgtc tctccctccc ttggcaggag ctggaagatg gcaccccccg 2161 ggctgggcag atcctcaagc agacctatga caaatttgac acaaacatgc gcagtgacga 2221 cgcgctgctc aagaactacg gtctgctctc ctgcttccgg aaggacctgc ataagacgga 2281 gacgtacctg agggtcatga agtgccgccg cttcggggag gccagctgtg ccttctagtt 2341 gccagccatc tgttgtttgc ccctcccccg tgccttcctt gaccctggaa ggtgccactc 2401 ccactgtcct ttcctaataa aatgaggaaa ttgcatcgca ttgtctgagt aggtgtcatt
Identifikasi D-Loop
Sampel DNA yang digunakan untuk sekuensing berasal dari Banda Aceh (11), Saree (20) dan Indrapuri (29), tetapi sampel DNA yang berhasil disekuen berjumlah dua puluh sembilan sampel dengan rincian yang berasal dari Banda Aceh (7), Saree (2) dan Indrapuri (20) sampel. Kegagalan sekuensing terhadap sampel disebabkan jumlah DNA yang terbatas dan terlalu banyak duplikasi puncak pada grafik sekuen. Analisa keragaman runutan nukleotida dilakukan setelah runutan DNA sapi Aceh disejajarkan dengan sampel sekuen pembanding yang berasal dari Gen Bank (Lampiran 4), yang akan digunakan untuk dianalisis dengan Neighbour Joining (NJ). Berdasarkan panjang sekuen 968 bp D-Loop sapi Aceh dari 29 sampel didapat 8 haplotype (Lampiran 7). Sketsa letak penempelan priner untuk mengamplifikasi fragmen D-Loop sapi Aceh dapat dilihat pada Gambar 5.
Hasil penelitian ini menunjukkan bahwa sebagian besar sampel sapi Aceh memiliki maternal origin dari Bos indicus, hasil penelitian ini memperkuat hasil penelitian sebelumnya yang dilakukan terhadap sapi Aceh (Uggla 2008). Dua puluh delapan sampel sapi Aceh yang berasal dari Indrapuri, Banda Aceh dan
)
Gambar 5 Sketsa letak penempelan primer Forward dan Reverse untuk mengamplifikasi fragmen daerah D-Loop sapi Aceh
Setelah dilakukan perunutan sekuen sapi Aceh dengan sapi Buthan, China, India dan Zebu, dianalisa berdasarkan Neighbour Joining (NJ) tree maka seluruh sampel terbagi dalam dua kelompok yaitu Bos taurus dan Bos indicus. Pada NJ
tree, individu yang memiliki maternal origin yang sama akan membentuk klaster/kelompok bersama.
Saree memiliki maternal origin Bos indicus, sedangkan satu sampel dari Saree memiliki maternal origin Bos taurus.
Berdasarkan analisis daerah D-Loop mtDNA sapi Aceh dengan ukuran sekuen 230 bp, sapi Aceh tersebar menjadi 8 haplotypes dari 27 haplotypes yang ada (Lampiran 8), dimana penyebaran sapi Aceh pada 7 haplotypes dari 27
haplotypes memiliki persamaan dengan 8 haplotypes berdasarkan panjang sekuen 968 bp, kecuali pada haplotype 3. Penelitian yang dilakukan oleh Nijman et al.
(2003) dan Uggla (2008) menggambarkan bahwa sapi Pesisir dan sebagian besar sapi Ongole memiliki maternal origin Bos indicus, sedangkan sapi Bali yang berasal dari pulau Bali berasal dari Bos banteng (Tabel 12).
Tabel 12 Maternal origin dari beberapa bangsa sapi
Bangsa
Banteng Taurin Indicine Referensi
Aceh B.Aceh 7 7 Hasil penelitian
Aceh Saree 2 1 1 Hasil penelitian
Aceh Indrapuri 20 20 Hasil penelitian
Aceh Pidie 15 15 Abdullah (2008)
India, Zebu dan sampel referensi dari Gen Bank (Lampiran 4) untuk mengetahui hubungan antara sapi Aceh dengan sapi Buthan, China, India dan Zebu.
Program Phylip file sangat diperlukan untuk membentuk median joining network, dari NJ tree seluruh kode harus ditransformasikan. Daerah sekuen 230 bp (Gambar 6) merupakan fragmen yang sangat variabel dari mtDNA daerah
control region yang telah banyak digunakan pada penelitian lainnya (Nijman et al.
2003, Baig et al. 2005, Kim et al. 2003, Loftus et al. 1994, Uggla 2008).
D-Loop utuh 908 bp (Gen Bank)
Teramplifikasi utuh 908 bp
Gambar 6 Sketsa daerah D-Loop hasil perunutan 230 bp sekuen sapi Aceh, Buthan, China, India dan Zebu
Hasil sekuen D-Loop pada penelitian ini membentuk dua klaster yang berbentuk bintang dimana predominan haplotype berada tepat ditengah-tengah diantara dua klaster yang berbentuk bintang (Gambar 7).
Gambar 7. Konstruksi Median Joining Network dari 230 bp D-Loop sekuen sapi Aceh (kuning), Zebu dari Indonesia (biru), Zebu dari China (merah), India (pink)dan Buthan (pink muda).
Forward Reverse
230 bp 289 bp
Empat mutasi dan satu haplotype unik dari 29 sampel sapi Aceh memisahkah dua klaster yang berbentuk bintang. Sapi Zebu yang berasal dari beberapa negara terdapat pada kedua klaster yang berbentuk bintang dan tidak ada perbedaan yang nyata terhadap daerah asal sampel sapi Aceh.
Polimorfisme D-Loop sapi Aceh, Buthan, China, India dan Zebu
Hasil penelitian terhadap sapi Aceh menggunakan analisa D-Loop sekuensing menggambarkan variasi genetik dalam dan antar populasi dengan referensi populasi sampel dari Gen Bank (Lampiran 4). Pada NJ tree, korelasi genetik diantara sekuens mtDNA menggambarkan bahwa sampel yang berasal dari berbagai daerah akan membentuk klaster/kelompok yang sama, Bos taurus, Bos indicus dan Bos banteng. Hasil penelitian ini memperkuat hasil penelitian Uggla (2008) yang menggunakan referensi sekuen yang sama. Uggla (2008) menemukan 53 haplogrup dari sapi Aceh dengan hampir 53% terbagi ke dalam dua haplogrup.
Penelitian lainnya menggambarkan bahwa 240 bp dari mtDNA daerah
control regionBos indicus terbagi dalam dua klaster yang berbentuk bintang yang dipisahkan oleh empat titik mutasi (Baig et al. 2005). Pada hasil penelitian ini sekuen sapi Zebu juga terbagi dalam dua klaster dan tepat diantara dua klaster tersebut terdapat haplotypes yang dipisahkan oleh empat mutasi dan merupakan
haplotype yang khas pada sapi Aceh. Lai et al. (2006) menemukan lima titik mutasi diantara dua klaster, hal ini membuktikan bahwa jumlah titik mutasi diantara dua klaster tidak memiliki nilai yang sama.
Berdasarkan dari beberapa penelitian sebelumnya, dapat diidentifikasi bahwa satu dari beberapa mutasi yang ada pada hasil penelitian ini merupakan
Komposisi Nukleotida D-Loop
Jumlah nukleotida dari semua sampel sapi penelitian tidak memiliki frekuensi yang sama (Lampiran 9), sejalan dengan hasil penelitian Abdullah (2008). Berdasarkan ukuran runutan nukleotida sepanjang 230 bp yang dianalisis, komposisi nukleotida daerah D-Loop sapi Aceh dari tiga populasi dapat dilihat pada Tabel 13.
Tabel 13 Persentase komposisi nukleotida D-Loop sapi Aceh (228 nt) Nomor Populasi Jumlah
sampel
T C A G Total
1 Banda Aceh 7 27.0 26.0 32.4 14.6 100.0 2 Saree 2 28.0 25.3 31.0 15.7 100.0 3 Indrapuri 20 27.0 26.0 32.4 14.6 100.0
Pada Tabel 13 terlihat bahwa komposisi nukleotida A pada tiga populasi sapi Aceh memiliki persentase yang lebih tinggi dibandingkan dengan persentase nukleotida T, C dan G. Nukleotida G pada populasi Banda Aceh dan Saree memiliki persentase yang sama (14.60%).
Tabel 14 Persentase komposisi nukleotida D-Loop sapi Aceh, Buthan, China,
Frekuensi nukleotida T juga memiliki nilai tertinggi diantara sapi-sapi lokal Indonesia (Bali, Madura, Pesisir, dan PO), kemudian diikuti Buthan (26.80%), China (26.50%), serta India dan Zebu (26.30%). Nukleotida C mempunyai frekuensi terendah pada sapi Aceh (26.00%) diikuti oleh Buthan (26.30%) dan China (26.60%), frekuensi yang sama ditemukan pada sapi India dan Zebu (26.80%). Hasil penelitian ini mendukung hasil penelitian Abdulah (2008) yang menemukan frekuensi nukleotida C memiliki nilai terendah diantara sapi-sapi lokal Indonesia (Bali, Madura, Pesisir, dan PO) yang menjadi sampel pembanding.
Sapi Buthan, India dan Zebu memiliki nilai frekuensi nukleotida A yang sama (32.50%) diikuti oleh sapi China (32.40%) dan sapi Aceh (32.30%). Nukleotida G daerah D-Loop mempunyai frekuensi terendah pada sapi penelitian dibandingkan dengan komposisi nukleotida A, T dan C dengan urutan tertinggi (14.60%) ditemukan pada sapi Aceh dan China, sedangkan sapi Buthan, India dan Zebu memiliki frekuensi yang sama (14.50%). Berdasarkan Tabel 14 dapat disimpulkan bahwa komposisi nukleotida ATGC pada sapi India dan Zebu memiliki nilai frekuensi yang sama. Hasil penelitian ini membuktikan bahwa sapi India dan Zebu memiliki maternal origin yang sama. Pernyataan ini mendukung pendapat Nozawa (1979) yang menyatakan bahwa pusat gen sapi Zebu (Bos indicus) ada di India.