PERNYATAAN MENGE
ALWIYAH
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2016
IDENTIFIKASI KERAGAMAN GEN DGAT1 DAN SCD SERTA
ASOSIASINYA TERHADAP KUALITAS KARKAS
PERNYATAAN MENGENAI DISERTASI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa tesis berjudul Identifikasi Keragaman Gen DGAT1 dan SCD serta Asosiasinya terhadap Kualitas Karkas pada Sapi Bali adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Agustus 2016
Alwiyah
RINGKASAN
ALWIYAH. Identifikasi Keragaman Gen DGAT1 dan SCD serta Asosiasinya terhadap Kualitas Karkas pada Sapi Bali. Dibimbing oleh JAKARIA dan HENNY NURAINI.
Sapi Bali merupakan ternak asli Indonesia yang telah diakui oleh FAO sebagai salah satu bangsa sapi di dunia. Sapi bali memiliki potensi yang belum dimanfaatkan dengan baik khususnya ke arah kualitas karkas yang baik. Perlu dilakukan upaya perbaikan secara genetik kearah kuaitas karkas dengan menggunakan pendekatan molekuler atau Marker Assisted Selection (MAS). Gen yang dapat digunakan sebagai marker sifat kualitas karkas adalah gen Diacylglycerol Acyltransferase 1 (DGAT) dan Stearoyl-CoA desaturase (SCD). Tujuan dari penelitian ini adalah untuk mengidentifikasi keragaman gen DGAT1 dan SCD pada sapi bali di Balai Pembibitan Ternak Unggul Sapi Bali dengan menggunakan metode sekuensing, menentukan SNP pada fragmen target gen DGAT1 dan SCD dan menganalisis asosiasi antara SNP (genotipe) yang didapatkan dengan data sifat-sifat kualitas karkas.
Jumlah sampel sapi bali adalah 48 ekor terdiri atas jantan 24 ekor dan betina 24 ekor berasal dari BPTU-HMT Sapi Bali di provinsi Bali. Amplifikasi gen DGAT1 menggunakan primer forward 5’- CACCATCCTCTTCCTCAAG-3’ dan reverse 5’- GGAAGCGCTTTCGGATG-3’, SCD menggunakan primer forward 5’-ACCTGGTGTCCTGTTGTTGTGCTTC-3’ dan reverse 5’ -GATGACCCTACTCTTCTATTTATGC-3’. Keragaman gen DGAT1 dan SCD diidentifikasi dengan metode direct sequencing. Sifat kualitas daging yaitu tebal longissimus dorsi (TLD), tebal lemak punggung (TLP), tebal lemak rump (TLR), tebal rump (TR), marbling score (MS), dan persentase lemak intramuskular (PLIM) di koleksi menggunakan Veterinary Ultrasound Scanner. Frekuensi alel dan genotipe dihitung dengan GENEPOP (V3.2) untuk mengetahui apakah polmorfisme gen DGAT1 dan SCD dalam keseimbangan Hardy-Weinberg. Asosasi Single Nucleotide Polymorphism (SNP) gen SCD terhadap kualitas daging dianalisis dengan pendekatan General Linier Model (GLM).
Hasil analisis ditemukan gen DGAT1 pada SNP c.10433G>A dan c.10434C>A disapi bali bersifat monomorfik. 8 SNP yaitu 5 SNP monomorfik (c.10153A>G, c.10318C>A, c.10329C>T, g.10394G>A, g.10486A>C) dan 3 polimorfik (g.10360G>A, g.10428C>T, g.10487G>A) berada dalam keseimbangan H-W. Gen SCD Ditemukan berasosiasi nyata (P<0.05) pada SNP g.10428C>T terhadap sifat marbling score dan persentase lemak intramuskular. Berdasarkan hasil tersebut SNP g.10428C>T dapat dijadikan sebagai kandidat marker assisted selection (MAS).
SUMMARY
ALWIYAH. Identification Polymorphism of DGAT1 gene and SCD gene and Their Association with Carcass Quality in Bali Cattle. Supervised by JAKARIA and HENNY NURAINI.
Bali cattle as Indonesian native cattle has admited by FAO as a breed in the world. Potency of bali cattle didn’t utilized optimally expecially in carcass quality. Needs to be done an effort to improve genetically at carcass quality by adopting molecular or marker assisted selection ( MAS ). A gene that can used as marker of carcass quality are diacylglycerol acyltransferase 1 ( DGAT1) and stearoyl-coa desaturase (SCD) genes. The purpose of this study was to obtain the polymorphisms of the DAGT1 and SCD gene and their associations with carcass quality traits in Bali cattle.
The number of samples used were 48 heads of cattle consisted of 24 bulls and 24 cows from BPTU-HMT Bali cattle in the province of Bali. The DGAT1 gene forward primer 5’- CACCATCCTCTTCCTCAAG-3’ and reverse 5’- GGAAGCGCTTTCGGATG-3’, SCD gene has been amplified using forward primer 5’-ACC CCT TGG TGT GTG GTT GTT CTT C-3 ‘and reverses primer 5'-CCT GAC GAT ACT ATG TTT CTA CTT C-3'. The polymorphisms of the DGAT1 and SCD gene were identified by direct sequencing method. Carcass quality traits such as thick of longissimus dorsi (TLD), thick of backfat (TBF), thick of fat rump (TFR), thick of rump (TR), marbling score (MS), and the percentage of intramuscular fat (PIMF) were analyzed using the Veterinary Ultrasound Scanner. To determine Hardy-Weinberg equilibrium status, both Allele and genotype frequencies were analyzed using GENEPOP program (V3.2). Association of the DGAT1 and SCD genes SNP and carcass quality traits was analyzed by GLM.
This result showed that there were DGAT1 gene have found SNP there are c.10433G>A and c.10434C>A in Bali cattle is monomorphic, and in SCD gene 5 monomorphic SNPs (c.10153A>G, c.10318C>A, c.10329C>T, g.10394G>A, g.10486A>C) and 3 polymorphic SNPs (g.10360G>A, g.10428C>T, g. 10487G>A) were in HW equilibrium. Association anslysis showed that g.10428C>T SNP significantly affected marbling score (MS) and percentage of intramuscular fat (PIMF) (P<0.05). Based on these results, g.10428C>T SNP of the SCD gene may be used as a candidate marker to select meat quality traits in Bali cattle.
© Hak Cipta Milik IPB, Tahun 2016
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Ilmu Produksi dan Teknologi Peternakan
IDENTIFIKASI KERAGAMAN GEN DGAT1 DAN SCD SERTA
ASOSIASINYA TERHADAP KUALITAS KARKAS
PADA SAPI BALI
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
Penguji Luar Komisi pada Ujian Tesis : Dr. Ir. Chalid Talib, MS
Penguji pada Ujian Tertutup: Dr Ir Drajat Martianto, MS
Penguji pada Ujian Terbuka: Prof Dr Ir Marimin, MS
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Penelitian yang dilaksanakan sejak bulan September 2015 sampai Januari 2016 berjudul Identifikasi Keragaman Gen DGAT1dan SCD serta Asosiasinya Terhadap Sifat Kualitas Karkas pada Sapi Bali. Tesis ini disusun sebagai salah satu syarat untuk mendapatkan gelar Magister pada program studi Ilmu Produksi dan Teknologi Peternakan, Sekolah Pascasarjana, Institut Pertanian Bogor.
Penulis menyadari bahwa proses penelitian dan penyusunan karya ilmiah ini tidak dapat berjalan dengan lancar tanpa bantuan dan dukungan dari banyak pihak. Terima kasih penulis ucapkan kepada yang terhormat Dr Jakaria, SPt MSi dan Dr Ir Henny Nuraini, MSi selaku komisi pembimbing atas curahan waktu, bimbingan dan dorongan semangatnya. Terima kasih juga penulis sampaikan kepada Dr. Ir. Chalid Talib, MS selaku penguji luar komisi atas segala masukannya untuk perbaikan tesis ini.
Kepada Dr Ir Salundik, MSi selaku Ketua Program Studi ITP dan seluruh staf di sekretariat Pasca ITP, penulis menghaturkan terimakasih atas pelayanan administrasi yang diberikan selama penulis menempuh studi. Kepada teman-teman di Program Studi ITP angkatan 2014 dan ITP 2015 terima kasih atas kebersamaannya.
Penulis sampaikan terima kasih kepada kawan satu tim penelitian Himmatul Khasanah, SPt MSi dan Nurul Pratiwi SPt atas segala bimbingan dan motivasinya. Kepada Shelvi, SSi, Komang Alit P, SPt MSi, Isyana Khaerunnisa, SPt, Ahmad Furqon, SPt, Muhsinin, SPt, Rindang L Suhita, SPt MSi, Uswatun Hasanah, SPt dan teman teman ABGSci atas segala dukungan dan kebersamaannya di Laboratorium Genetika Molekuler Ternak.. Kepada yang terkhusus Aulia Rahmad Hasyim, SPt. penulis sampaikan terimakasih atas kebersamaan, canda tawa, dan motivasi yang diberikan.
Ungkapan terima kasih terdalam penulis sampaikan kepada kedua orang tua Anis Hamzah Alhiyed dan Sofiah Alatas, Kakak tercinta Fatimahtuzahro, Paman terbaik Kadzim Salim Alhiyed (Alm), serta seluruh keluarga atas doa, kasih sayang, dukungan serta motivasi yang selalu diberikan pada penulis. Semoga penulis dapat menjadi sumber kebahagiaan bagi kedua orang tua dan keluarga. Terimakasih juga kepada Yayasan Rabbithah Alawiyah atas doa dan dukungannya. Semoga hasil penelitian ini bermanfaat. Saya ucapkan terima kasih kepada semua pihak yang telah membantu, yang tidak dapat disebutkan satu persatu. Semoga karya ilmiah ini bermanfaat.
Bogor, Agustus 2016 Penyusun
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
I PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 2
2 METODE 3
Lokasi dan waktu 3
Materi 3
Prosedur 4
3 HASIL DAN PEMBAHASAN 7
Amplifikasi DNA 7
Homologi dan Deteksi Mutasi 8
Keragaman Gen 11
Heterozigositas 14
Sifat Perdagingan pada Sapi Bali 15
Asosiasi Gen SCD terhadap Sifat Perdagingan 17
4 SIMPULAN DAN SARAN 18
Simpulan 18
Saran 18
5 DAFTAR PUSTAKA 19
LAMPIRAN 23
DAFTAR TABEL
1 Primer gen DGAT1 dan SCD 3
2 Frekuensi genotipe dan alel gen DGAT1 ekson 8 11 3 Frekuensi Alel gen DGAT1 pada Berbagai Sapi di Dunia 12 4 Frekuensi Genotipe dan Frekuensi Alel gen SCD pada Sapi Bali 13 5 Frekuensi Alel gen SCD ekson 5 pada Berbagai Sapi di Dunia 14 6 Nilai Heterozigositas dan hasil uji keseimbangan Hardy-Weinberg 15 7 Karakteristik sifat perdagingan pada sapi bali 16 8 Asosiasi SNP gen SCD intron 5 terhadap sifat perdagingan sapi Bali 17
DAFTAR GAMBAR
1 Posisi penempelan primer pada Gen DGAT1 (383 bp) 3 2 Posisi penempelan primer pada Gen SCD (569 bp) 4 3 Padang Pemeliharaan sapi bali di BPTU-HMT Sapi Bali 5
4 Ilustrasi USG otot longissimus dorsi 5
5 Hasil amplifikasi gen DGAT1 dan SCD pada sapi bali 8 6 Perunutan sekuen gen DGAT1 sapi bali dengan GenBank
AJ318490
9 7 Perunutan sekuen gen SCD sapi bali dengan GenBank
AY241932
10
8 Citra USG sapi Bali 16
DAFTAR LAMPIRAN
1 Sekuen gen DGAT1 24
1
PENDAHULUAN
Latar Belakang
Kebutuhan daging sapi di Indonesia mengalami peningkatan setiap tahunnya, namun produksi daging nasional masih defisit, sehingga hal tersebut dicukupi melalui impor. Pasokan impor daging sapi di Indonesia mencapai 48 085 ton (BPS 2014). Peningkatan jumlah daging tersebut harus diimbangi dengan peningkatan kualitas daging. Salah satu hal yang digunakan untuk menentukan kualitas daging adalah marbling. Marbling merupakan komposisi lemak yang terdapat di intramuskular (Soeparno 2005).Peningkatan kualitas daging sebaiknya dilakukan pada ternak asli dan lokal Indonesia, salah satunya sapi bali.
Sapi bali (Bos javanicus) sebagai ternak asli Indonesia hasil domestikasi banteng (Bibos banteng) (Purwantara 2012) merupakan sumberdaya genetik ternak yang tidak ternilai harganya dan telah diakui oleh FAO sebagai salah satu bangsa sapi di dunia (DGLS 2003). Sapi bali memiliki beberapa keunggulan yaitu mampu beradaptasi terhadap lingkungan marjinal dan memiliki daya reproduksi yang tinggi terutama pada kondisi pakan yang buruk (Talib 2002). Di berbagai lingkungan pemeliharaan di Indonesia, sapi bali memperlihatkan kemampuannya untuk berkembang biak dengan baik yang disebabkan beberapa keunggulan yang dimiliki sapi Bali. Keunggulan sapi bali dibandingkan sapi lain yaitu memiliki persentase karkas yang tinggi (48-52%) (Ismail et al.2014), daya mengikat air 66.2% (Dewitri et al. 2015), susut masak 19-28%, keempukan 3-3.5 kg/cm2 dan memiliki komposisi kimia daging berupa kadar protein 17-21% (Eko dan Subandriyo 2004).
Salah satu upaya untuk meningkatkan mutu sumber daya genetik ternak sapi bali adalah dengan melakukan seleksi untuk memperoleh karakteristik genetik yang terdapat pada sapi bali seperti kualitas karkas yang bermutu baik. Seleksi yang biasa digunakan pada umumnya mengacu pada sifat fenotipik saja. Namun perlu dilakukan seleksi secara genetik dengan memanfaatkan Marker Assisted Selection (MAS). Pendekatan dengan MAS ini dapat dilakukan dengan menggunakan gen untuk kualitas karkas seperti gen SREBP (Barton et al. 2010), DGAT1 (Karolyid et al. 2012) dan SCD (Ohsaki et al. 2009). Penelitian sebelumnya telah dilakukan kajian mengenai gen DGAT1 menggunakan teknik Polymorphism Chain Reaction-Restriction Fragment Lenght Polymorphism (PCR-RFLP) pada sapi simmental (Karolyi et al. 2011) dan gen SCD pada sapi Jappanese Black (Ohsaki et al 2009).
2
komposis asam lemak pada daging, deposit IMF (Wu et al. 2012) dan warna daging (Reardon et al. 2010).
Informasi keragaman genetik yang berkaitan dengan gen-gen yang mengontrol kualitas karkas pada sapi bali masih belum banyak dilakukan. Oleh karena itu keragaman gen DGAT1 dan SCD pada sapi bali perlu dilakukan dengan menggunakan teknik Polymerase Chain Reaction (PCR) direct sequencing. Hasil penelitian ini diharapkan dapat menjadi informasi dasar yang dapat dimanfaatkan untuk program pemuliaan dan pelestarian sapi bali.
Perumusan Masalah
Sapi bali merupakan salah satu satu sumber daya genetik ternak asli Indonesia yang memiliki beberapa keunggulan salah satunya adalah memiliki kualitas karkas yang cukup baik. Perhatian terhadap kualitas karkas pada sapi bali masih rendah. Namun di sisi lain permintaan akan daging berkualitas meningkat setiap tahunnya. Pendekatan molekuler berupa seleksi diharapkan dapat berperan dalam memberikan informasi genetik mengenai sapi bali khususnya mengenai kualitas karkas seperti gen DGAT1 dan gen SCD masih sangat terbatas. Oleh sebab itu, untuk memperoleh informasi genetik terhadap gen-gen tersebut menjadi sangat penting untuk dilakukan pada bangsa sapi bali.
Tujuan Penelitian
Penelitian ini bertujuan untuk mengidentifikasi keragaman gen DGAT1 dan SCD pada sapi bali di Balai Pembibitan Ternak Unggul Sapi Bali dengan menggunakan metode sekuensing. Selain itu, tujuan penelitian ini adalah menentukan SNP pada fragmen target gen DGAT1 dan SCD. Dan untuk mengasosiasikan antara SNP (genotipe) yang didapatkan dengan data sifat-sifat kualitas karkas.
Manfaat Penelitian
Penelitian ini diharapkan dapat dijadikan Sebagai Informasi dasar mengenai keragaman gen DGAT1 dan SCD pada sapi Bali. Selain itu diharapkan gen yag digunakan dapat menjadi kandidat MAS untuk sifat kualitas karkas.
Ruang Lingkup Penelitian
3
2 METODE
Lokasi dan waktu
Penelitian ini akan dilakukan di Laboratorium Genetika Molekuler Ternak, Bagian Pemuliaan dan Genetika, Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor dan Balai Pembibitan Ternak Unggul (BPTU) Sapi Bali. Penelitian akan berlangsung dari bulan September 2015 hingga Januari 2016.
Materi
Ternak
Sampel darah sapi bali yang akan digunakan adalah 48 ekor yang berasal dari BPTU-HMT sapi bali di provinsi Bali dengan rataan umur 12-15 bulan dengan jumlah jantan dan betina masing-masing 24 ekor. Sedangkan untuk analisis asosiasi ternak sapi bali yang digunakan sebanyak 31 ekor, terdiri dari 18 jantan dan 13 betina.
Bahan
Bahan-bahan yang digunakan untuk ekstraksi DNA adalah sampel darah, RBC lysis buffer, Prot-K, GB buffer, Rnase A, absolute ethanol dan elution buffer. Bahan yang digunakan untuk PCR, elektroforesis adalah sampel DNA hasil ekstraksi, premix PCR master mix (PROMEGA Green Master Mix), primer forward, primer reverse, dan destilation water (DW), produk PCR, serbuk agarosa, 0.5 x Tris-Borat EDTA (TBE), Ethidium Bromide (EtBr), loading dye (0.01% Xylene Cyanol, 0.01% Bromthymol blue dan 50% gliserol), marker 100 pb.
Bahan yang digunakan untuk analisis sekuensing adalah produk PCR dan primer fragmen gen DGAT1 dan SCD pada Gambar 1 dan Gambar 2. Primer yang digunakan untuk mengamplifikasi gen-gen tersebut dapat dilihat pada Tabel 1.
Tabel 1 Primer gen DGAT1 dan SCD
Gen Kromosom Sekuen (5’-3’) Forward-Reverse Pustaka
DGAT1 14 CACCATCCTCTTCCTCAAG
GGAAGCGCTTTCGGATG
Karolyi et al. (2012)
SCD 26 ACCTGGTGTCCTGTTGTTGTGCTTC
GATGACCCTACTCTTCTATTTATGC
4
Alat
Alat-alat yang digunakan untuk ekstraksi DNA dan PCR adalah tabung eppendorf 1.5 mL, satu set mikro pipet, tip pipet, vortex, inkubator, rotary mixer, microsentrifuge, refrigerator, freezer, mesin PCR ESCO dan Eppendorf, tabung PCR 0.2 ml, dan 0.5 ml. Alat yang digunakan dalam prosedur elektroforesis adalah satu set geltray, magnetic stirrer, microwave, power supply electrophoresis 100 volt, dan UV Transiluminator. Pengukuran menggunakan alat ultrasonografi model Veterinary Ultrasound Scanner tipe WED-3000V.
Prosedur
Pemeliharaan Ternak
Sapi Bali yang digunakan untuk penelitian ini dipelihara terlebih dahulu di dalam pedok yang sama di BPTU-HMT provinsi Bali (Gambar 3). Pakan yang diberikan berupa pakan hijauan jenis Pennisetum purpureum dan Phaspalum notatum 10% dari bobot badan serta konsentrat dengan jumlah 1% dari bobot badan.
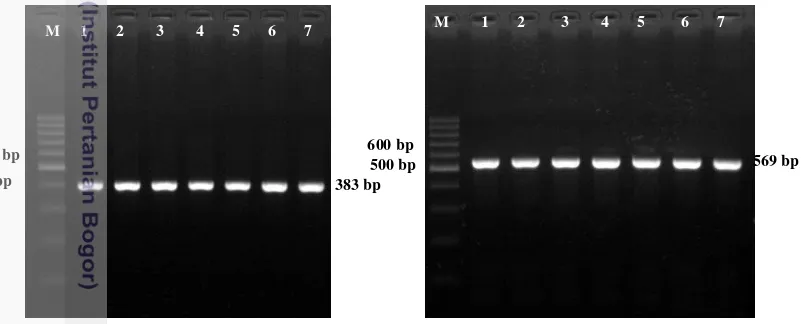
Gambar 1 Posisi penempelan primer pada Gen DGAT1 (383 pb)
5
Pengukuran Karakteristik Karkas
Tebal Longissimus dorsi dan Rump diukur menggunakan alat ultrasonografi model Veterinary Ultrasound Scanner tipe WED-3000V dengan rekuensi 6.5 Hz dan kedalaman 130 mm untuk mengetahui kualitas karkas. Pencitraan tebal lemak punggung dan tebal otot Longissimus dorsi dilakukan pada posisi tulang rusuk ke 12, dua pertiga dari medial ke sisi lateral (Gambar 4) (Ulum et al.2014) dan pengukuran tebal otot rump diantara tulang ischium dan illium (Silva et al. 2012). Data hasil pencitraan disimpan dalam format JPEG dan selanjutnya dianalisis dengan menggunakan software Image-J NIH (ImageJ®, NIH, USA).
Gambar 4 Ilustrasi USG otot longissimus dorsi pada sapi, (a) longitudinal (horizontal) (b) transversal (vertikal), c = kulit, sc = subkutan, tm = tebal otot, o = tulang
6
Pengambilan sampel
Pengambilan sampel darah dilakukan dengan jarum venoject pada bagian vena jugularis. Jarum venoject telah terhubung dengan tabung vacutainer yang berisi EDTA. Darah yang dibutuhkan sebanyak ± 5 mL dan disimpan dalam suhu ± 4 oC.
Ekstraksi DNA
Ekstraksi DNA dilakukan berdasarkan prosedur ekstraksi DNA Kit Geneaid yang dimodifikasi. Tahap pertama merupakan preparasi sampel yaitu sampel darah diambil sebanyak 300 µl dalam tabung mikrosentrifuse 1.5 mL dan ditambahkan larutan RBC lysis sebanyak 900 µl kemudian homogenkan. Setelah itu diamkan pada suhu ruang selama 10 menit kemudian sentrifuse 3 000 rpm selama 5 menit dan supernatan dibuang. Sebanyak 100 µl RBC lysis dan 200 µl GB Buffer ditambahkan kemudian dihomogenkan dengan vortex. Sampel diinkubasi pada suhu 60 °C selama 10 menit dan dibalik setiap 3 menit. Kemudian Rnase sebanyak 5 µl ditambahkan dan diinkubasi pada suhu ruang selama 5 menit. Sebanyak 200 µl ethanol asolute ditambahkan dan sampel dipindah di GD coloum kemudian sentrifuse 14 000 rpm selama 5 menit dan collection tube 2 mL dibuang. Sebanyak 400 µL larutan W1 buffer ditambahkan kedalam GD coloum yang sudah diberi collection tube baru kemudian sentrifue 14 000 rpm selama 1 menit, buang supernatan dan sentrifuse kembali GD coloum kering. Setelah itu, pindahkan tabung GD coloum ke tabung mikrosentrifuse 1.5 mL dan ditambahkan 100 µL pre-heated elution buffer dan diamkan selama 3 menit kemudian sentrifuse 14 000 rpm selama 3 menit.
Amplifikasi DNA
Fragmen gen DGAT1 dan SCD diamplifikasi dengan teknik PCR. Sampel DNA hasil ekstraksi sebanyak 1 μL dimasukkan ke dalam tabung PCR, lalu ditambahkan 49 μL larutan premix. Premix tersusun atas 0.3 μL primer, 23.4 μL DW, 25 μL Green Master Mix. Campuran ini divortex lalu di sentrifugasi menggunakan spin down, kemudian diinkubasi dalam thermocycler untuk diamplifikasi. Kondisi PCR yaitu meliputi pradenaturasi 95 °C 5 menit, dilanjutkan dengan langkah denaturasi pada 95 °C 10 detik, annealing pada suhu 50 oC selama 20 detik , elongasi pada suhu 72 oC selama 30 detik dan final
elongasi pada suhu 72 oC selama 5 menit. Proses amplifikasi DNA ini dilakukan hingga 35 siklus. Hasil ekstraksi DNA kemudian di validasi dengan elektroforesis dengan agarose 1% dan spektrofotometer.
Elektroforesis
Hasil produk PCR DGAT1 dan SCD dielektroforesis menggunakan gel agarose 1.5%. Gel agarose dibuat menggunakan 0.45 gram agarose ditambahkan 30 ml 0.5x TBE kemudian dipanaskan di dalam microwave selama 3 menit. Larutan agarose gel dihomogenisasi menggunakan magnetic stirer kemudian
ditambahkan EtBr sebanyak β.5 μl. Larutan agarose tersebut dimasukkan ke
dalam alat pencetak gel dan didiamkan hingga mengeras. Produk PCR sebanyak 5
μl dimasukkan ke dalam sumur-sumur gel dan marker DNA 100 bp dimasukkan
7
divisualisaikan menggunakan bantuan sinar UV pada mesin UV transiluminator. Hasil elektroforesis akan menunjukkan pita-pita dengan panjang fragmen DNA tersebut.
Sekuensing
Sekuensing produk PCR sampel sapi Bali akan dilakukan menggunakan jasa dari 1st Base di Selangor, Malaysia.
Analisis Data
DataFenotipik
Data kualitas perdagingan yaitu longissimus dorsi (TLD), tebal lemak punggung (TLP), tebal lemak rump (TLR), tebal rump (TR), marbling score (MS), dan persentase lemak intramuskular (PLIM) dianalisis secara deskriptif.
Analisis Sekuensing Gen DGAT1 dan SCD
Analisis dilakukan pada hasil sekuen dengan program BioEdit (Hall 1999), serta dianalisis menggunakan metode BLAST (www.ncbi.nhl.nih.gov./BLAST) untuk mengetahui kesamaan dengan gen DGAT1 dan SCD di GenBank. Keberadaan mutasi atau SNP (Single Nucleotide Polymorphism) pada sekuen fragmen gen gen DGAT1 dan SCD dianalisis menggunakan program Molecular Evolutionary Genetic Analysis 6 (MEGA6) (Tamura et al. 2011). Keragaman SNP gen SCD ditentukan berdasarkan frekuensi alel dan genotipe, keseimbangan Hardy-Weinberg dianalisis menggunakan GENEPOP (V3.2) (Raymond and Rousset 2001).
Asosiasi Gen DGAT1 dan SCD dengan Sifat Perdagingan
Hubungan antara keragaman gen DGAT1 dan SCD terhadap peubah sifat kualitas karkas (Longissimus dorsi dan tump) dianalisis dengan menggunakan metode GLM (General Liniear Model) (SAS Institute Inc.2000) pada umur sapi yang berbeda. Apabila didapatkan hasil yang berbeda, maka akan dilakukan uji lanjut LSmeans. Model matematis dirumuskan seperti berikut (Kaps and Lamberson 2004) :
Yij= µ + αi+ j + k+ Єijk
Keterangan :
Yij = nilai pengamatan
µ = nilai rata umum
αi = pengaruh genotipe ke-i j = pengaruh jenis kelamin k = pengaruh umur
Єijk = pengaruh galat
3 HASIL DAN PEMBAHASAN
Amplifikasi DNA
8
Amplifikasi terhadap dua gen yaitu DGAT1 dan SCD berhasil dilakukan pada suhu annealing 50 oC selama 20 detik. Kondisi annealing gen DGAT1 ditemukan berbeda pada penelitian Kong et al. (2007) yaitu 56 oC selama 30 detik, sedangkan menurut penelitian Wu et al. (2011) suhu annealing gen SCD adalah 65 oC selama 30 detik. Keberhasilan proses amplifikasi kedua gen tersebut sangat
bergantung pada suhu annealing. Suhu annealing adalah suhu optimum untuk proses penempelan primer sesuai dengan sekuens DNA target yang akan diperbanyak selama proses PCR. Adanya perbedaan tersebut disebabkan oleh kondisi mesin PCR dan campuran pereaksi PCR. Suhu penempelan primer (annealing) berkisar antara 36 -72 oC, namun suhu yang biasa digunakan adalah
50 - 60 oC (Muladno 2002). Menurut Pelt-Verkuil et al. (2008) waktu annealing
yang dibutuhkan supaya primer dapat berkomplemen dan menempel dengan targetnya bergantung pada kapasitas pemanasan mesin thermocycler yang digunakan, volume campuran PCR serta konsentrasi primer dan gen target. Primer yang telah menempel pada target selanjutnya mengalami pemanjangan atau ekstensi pada suhu 72 oC selama 40 detik. Kemudian dilanjutkan dengan ekstensi akhir pada suhu yang sama selama 5 menit. Tiga tahapan PCR yaitu denaturasi, annealing, dan ekstensi merupakan tahapan untuk 1 siklus termal. Pada penelitian ini dilakukan sebanyak 35 siklus.
Homologi dan Deteksi Mutasi
Gen Diacylglycerol Acyltransferase1 (DGAT1)
Sampel hasil sekuensing kemudian diverifikasi melalui perunutan dan penyetaraan hasil sekuensing Bos taurus dengan nomer akses AJ318490. Berdasarkan perbandingan tersebut didapatkan hasil bahwa sekuen gen DGAT1 sapi bali mengalami mutasi GA pada basa ke-200 (c.10433) dan CA pada basa ke-201 (c.10434). Hasil sekuensing gen DGAT1 ditampilkan pada Gambar 6. Namun dalam populasi sapi bali yang diteliti tidak ditemukan perbedaan atau SNP. Single nucleotide polymorphism (SNP) merupakan perbedaan akibat adanya subtitusi basa tunggal sehingga terjadi polimorfisme atau keragaman (Kwok dan Chen 2003). Dari gambar tersebut diketahui bahwa sapi bali memiliki kesamaan yang cukup tinggi dengan Bos taurus yang memiliki nomer akses GenBank
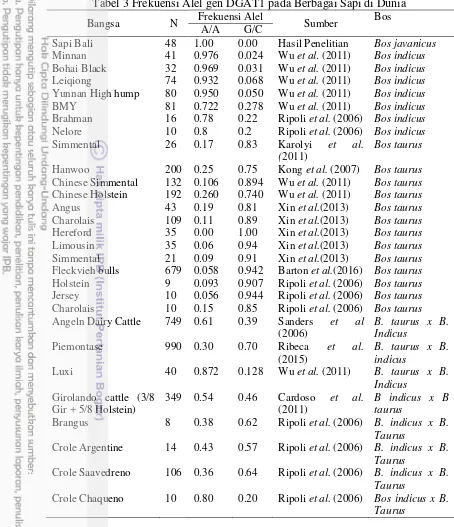
Gambar 5 Hasil amplifikasi gen DGAT1 dan SCD pada sapi bali. M = marker DNA 100 bp.
383 bp 400 bp
500 bp
M 1 2 3 4 5 6 7 9
569 bp 500 bp
600 bp
9
AJ318490. Hasil analisis MEGA.6 menyatakan bahwa SNP DGAT1 pada sapi bali yang diteliti tidak ditemukan Single nucleotide Polymorphism (SNP).
Berbeda dengan penelitian Kong et al. (2007) pada sapi Korea Ribeca et al. (2013) ditemukan dua SNP pada ekson dan titik yang sama yaitu c.10433A>G dan c.10434A>C. Penelitian Karolyi et al.(2012) menyatakan bahwa SNP pada gen DGAT1 di ekson 8 merupakan substitusi antara basa lisin (K) dengan basa alanin (A) pada posisi yang sama. Mutasi tersebut dapat dikategorikan sebagai mutasi non synonimous karena kedua kodon (GCG dan AAG) menyandikan asam amino yang berbeda yaitu alanin dan lisin. Menurut Grisart et al. (2004) bahwa adanya polimorfisme dari gen DGAT1 pada sapi Black-and-White Holstein-Friesian terjadi karena adanya mutasi pada daerah nonkonservasi K232A yang menyebabkan variasi pada deposisi marbling. Menurut Moore et al. (2003) bahwa deposisi marbling tergantung pada alel yang mengkode alanin dan lisin pada posisi basa 232 dari gen DGAT1. Keberadaan alel yang mengkode lisin terbukti berkaitan dengan meningkatnya deposisi marbling daripada keberadaan alel pengkode alanin (A) yang berhubungan dengan menurunnya deposisi marbling.
Perbedaan SNP tersebut dapat dijadikan penciri sapi pada lokasi tersebut. Efek adanya pasangan basa substitusi sederhana pada SNP dapat berhubungan juga dengan sifat atau penyakit tertentu (Schork et al. 2000). SNP yang berada di wilayah coding (ekson) berdampak langsung pada protein yang terkait SNP di intron dapat mempengaruhi splicing dan SNP di promoter dapat berpengaruh terhadap ekspresi gen (Schork et al. 2000).
Gen Stearoyl-CoA desaturase (SCD)
10
bersifat monomorfik dan lima SNP berada di intron 5 yaitu SNP g.10360G>A, g.10394G>A, g.10428C>T, g.10486A>c dan g.10487G>A (Gambar 7). Febriana et al. (2015) melaporkan bahwa mutasi yang terjadi di wilayah intron dapat memengaruhi proses splicing gen, transkripsi, dan translasi.
11
Keragaman Gen
Frekuensi Genotipe dan Frekuensi Alel Fragmen Gen DGAT1
Frekuensi genotipe gen DGAT1 berdasarkan pada sampel sapi bali yang digunakan dalam penelitian ini menghasilkan dua genotipe yang ditemukan yaitu genotipe AA (100%) pada urutan basa ke 201 dan pada urutan basa ke 202 memiliki genotipe AA (100%), sehingga frekuensi alel A pada basa ke-201 dan ke 202 adalah 1.00. Hasil penelitian ini ditunjukkan pada Tabel 2. Tabel tersebut menjelaskan bahwa tidak ada alel G pada basa ke-200 dan alel C pada urutan basa ke-201. Hal ini berarti bahwa tidak ada keragaman yang terjadi pada gen DGAT1 ekson 8 yang terdapat di sapi bali yang diteliti. Gen DGAT1 pada sapi bali yang diteliti memiliki frekuensi alel G di basa ke-200 dan C di basa ke-200 tertinggi (1.00) sehingga dapat dikatakan bahwa gen DGAT1 pada sapi bali bersifat seragam atau monomorfik.
Gen DGAT1 Frekuensi Genotipe Frekuensi alel
c.10433A>G AA (1.00) AG (0.00) GG (0.00) A (1.00) G (0.00) c.10434A>C AA (1.00) AC (0.00) CC (0.00) A (1.00) C (0.00)
Mutasi di DGAT1 telah dikonfirmasi bertanggung jawab untuk peningkatan produksi susu, lemak susu, protein susu, lemak yang terkandung dan protein yang terkandung di sapi perah (Grisart et al.2004). Menurut Thaller et al. (2003) gen diacylglycerol O-acyltransferase 1 (DGAT1) merupakan gen yang digunakan sebagai marker untuk pembentukan lemak intramuscular dan subkutan. Ribeca et al. (2014) dalam hasil penelitiannya mendapatkan hasil genotipe pada AA/AA 0.05, AA/GC 0.51 dan GC/GC 0.44, frekuensi alel minornya (AA) yaitu 0.3 dan alel GC 0.70. Karolyi et al.(2011) dalam hasil penelitiannya menyatakan bahwa sapi simmental baik bulls dan heifers memiliki frekuenssi genotipe AA yaitu 0.00. Penelitian sebelumnya menyatakan bahwa alel A/A pada umumnya dimiliki oleh bangsa Bos indicus (Winter et al. 2002) dan jika memiliki frekuensi intermediet maka itu merupakan persilangan dari Bos indicus x Bos taurus (Fortes et al. 2009). Di beberapa penelitian disebutkan frekuensi alel G/C berasosiasi dengan langsung dengan rasa yang dihasilkan (Moore et al. 2003).
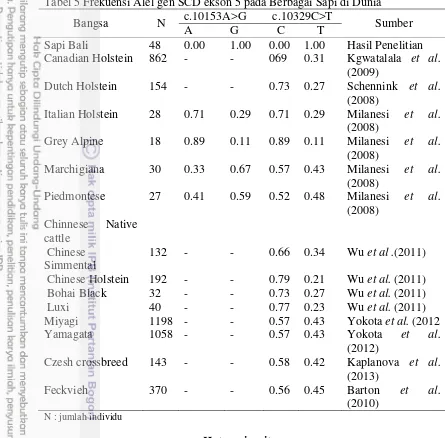
Adapun fragmen gen DGAT ekson 8 pada beberapa ternak sapi yang berbeda pada tabel 3. Dari hasil penelitian diketahui bahwa gen DGAT1 pada sapi bali bersifat seragam atau monomorfik karena hanya ditemukan satu alel saja yaitu A/A. Alel A/A dapat dijadikan sebagai alel spesifik dari sapi bali. Tabel tersebut menjelaskan bahwa Bos javanicus (sapi bali) memiliki 100% alel A/A yang mengkode asam amino lisin. Sapi yang termasuk kedalam Bos indicus memiliki frekuensi alel A/A (lisin) yang lebih tinggi jika dibandingkan dengan alel G/C (alanin). Namun hal yang berbeda terjadi pada sapi Bos taurus frekuensi alel yang tertinggi adalah G/C, sedangkan A/A sangat sedkit. Menurut Moore et al. (2003) bahwa deposisi marbling tergantung pada alel yang mengkode alanin dan lisin pada posisi basa 10433 dan 10434 dari gen DGAT1. Keberadaan alel yang mengkode Lisin (K) terbukti berkaitan dengan meningkatnya deposisi marbling daripada keberadaan alel pengkode Alanin (A) yang berhubungan dengan menurunnya deposisi marbling.
12
Frekuensi Genotipe dan Frekuensi Alel Fragmen Gen SCD
Hasil analisis keragaman berupa frekuensi genotipe dan frekuensi alel gen SCD pada sapi bali disajikan pada Tabel 4.
N : jumlah individu
13
Hasil analisis frekuensi genotipe pada SNP yang ditemukan pada Tabel 5 menjelaskan bahwa dari delapan jumlah SNP yang ditemukan lima diantaranya memiliki pola monomorfik, yaitu SNP di posisi c.10153A>G, c.10318C>A, c.10329C>T, g.10394G>A dan g.10486A>C memiliki frekuensi alel 1.00. Hal tersebut berarti SNP tersebut di gen SCD pada sapi bali yang diteliti tidak memiliki keragaman atau dapat disebut SNP tersebut bersifat monomorfik. SNP g.10360G>A genotipe GA memiliki frekuensi yang cukup tinggi, pada SNP g.10428C>T genotipe TT lebih tinggi dan pada SNP g.10487G>A frekuensi genotipe GG lebih tinggi jika dibandingkan dengan genotipe yang lain. SNP g.10360G>A memiliki frekuensi alel tertinggi pada alel A (0.52), SNP g.10428C>T alel T memiliki frekuensi tertinggi yaitu 0.80 dan SNP g.10487G>A alel G (0.73) memiliki frekuensi tertinggi. Ketiga SNP yang ditemukan (g.10360G>A, g.10428C>T dan g.10487G>A) bersifat polimorfik. Nei (2000) menyatakan bahwa suatu alel dikatakan polimorfik atauu beragam jika memiliki frekuensi alel sama dengan atau kurang dari 0.99.
Penelitian mengenai keragaman gen SCD pada berbagai sapi di dunia, umumnya hanya meneliti dua SNP yaitu 10153A>G dan 10329C>T saja. SNP 10329T>C memiliki frekuensi alel C yang tertinggi pada delapan populasi sapi dengan bangsa Bos indicus dan persilangan Bos indicus yaitu dengan kisaran 0.655-0.939, namun pada sapi Simmental China frekuensi alel C (0.663) paling rendah diantara populasi yang lain. Penelitian Kgwatalala et al. (2009) pada sapi Canadian holstein alel T memiliki frekuensi alel 0.31, Dutch holstein 0.27 (Schennink et al. 2008) dan 0.29 pada Italian Holstein (Milanesi et al.2008). Berikut ini beberpa penelitian mengenai SNP di posisi 10329C>T pada berbagai bangsa sapi (Tabel 5). Tabel 5 menjelaskan bahwa kedua SNP tersebut pada sapi lain memiliki keragaman, sedangkan pada sapi bali tidak beragam atau monomorfik.
SNP Frekensi Genotipe Frekuensi Alel c.10153A>G AA (0.00) AG (0.00) GG (1.00) A (0.00) G (1.00) c.10318C>A CC (0.00) CA (0.00) AA (1.00) C (0.00) A (1.00) c.10329C>T CC (0.00) CT (0.00) TT (1.00) C (0.00) T (1.00) g.10360G>A GG (0.19) GA (0.58) AA (0.23) G (0.48) A (0.52) g.10394G>A GG (0.00) GA (0.00) AA (1.00) G (0.00) A (1.00) g.10428C>T CC (0.04) CT (0.31) TT (0.65) C (0.20) T (0.80) g.10486A>C AA (0.00) AC (0.00) CC (1.00) A (0.00) C (1.00) g.10487G>A GG (0.52) GA (0.42) AA (0.06) G (0.73) A (0.27)
14
Tabel 5 Frekuensi Alel gen SCD ekson 5 pada Berbagai Sapi di Dunia Bangsa N c.10153A>G c.10329C>T Sumber
A G C T
Sapi Bali 48 0.00 1.00 0.00 1.00 Hasil Penelitian Canadian Holstein 862 - - 069 0.31 Kgwatalala et al.
(2009)
Dutch Holstein 154 - - 0.73 0.27 Schennink et al. (2008)
Italian Holstein 28 0.71 0.29 0.71 0.29 Milanesi et al. (2008)
Grey Alpine 18 0.89 0.11 0.89 0.11 Milanesi et al. (2008)
Marchigiana 30 0.33 0.67 0.57 0.43 Milanesi et al. (2008)
Piedmontese 27 0.41 0.59 0.52 0.48 Milanesi et al. (2008)
Chinnese Native cattle
Chinese Simmental
132 - - 0.66 0.34 Wu et al .(2011) Chinese Holstein 192 - - 0.79 0.21 Wu et al. (2011) Bohai Black 32 - - 0.73 0.27 Wu et al. (2011)
Luxi 40 - - 0.77 0.23 Wu et al. (2011)
Miyagi 1198 - - 0.57 0.43 Yokota et al. (2012
Yamagata 1058 - - 0.57 0.43 Yokota et al.
(2012)
Czesh crossbreed 143 - - 0.58 0.42 Kaplanova et al. (2013)
Feckvieh 370 - - 0.56 0.45 Barton et al.
(2010)
N : jumlah individu
Heterozigositas
Marson et al. (2005) menyatakan bahwa keragaman genetik suatu populasi dapat diukur menggunakan nilai heterozigositas yang bertujuan untuk membantu program seleksi. Gen DGAT1 memiliki keragaman yang rendah berdasarkan nilai heterozigositasnya. Hasil nilai heterozigositas gen DGAT1 dan SCD disajikan pada Tabel 5. Nilai Heterozigositas menunjukkan bahwa gen DGAT1 ekson 8 tidak beragam (0.00) pada sapi bali yang diteliti, karena gen tersebut memiliki alel monomorfik AA.
15
heterosigositas pada SNP tersebut lebih dari 0.5. SNP g.10428C>T dan g.10487G>A memiliki nilai heterosigositas yang lebih rendah dibandingkan dengan SNP sebelumnya. Nilai heterozigositas pada penelitian ini termasuk rendah, menurut Javanmard et al. (2005) apabila nilai heterozigositas rendah dibawah 0.5 maka hal tersebut mengindikasikan rendahnya suatu gen dalam sebuah populasi.
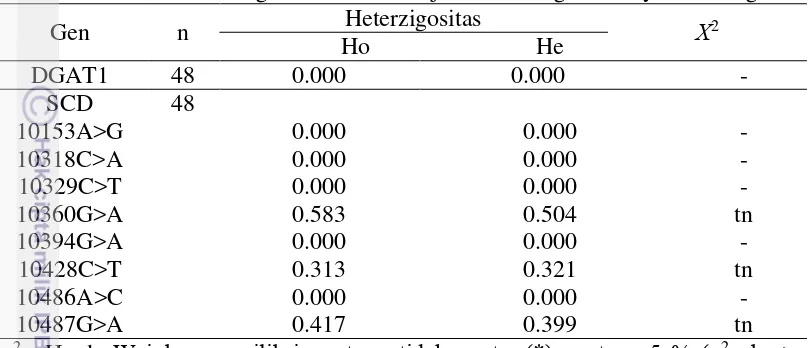
Tabel 6 Nilai Heterozigositas dan hasil uji keseimbangan Hardy-Weinberg
Gen n Heterzigositas Χ2
Ho He
DGAT1 48 0.000 0.000 -
SCD 48
10153A>G 0.000 0.000 -
10318C>A 0.000 0.000 -
10329C>T 0.000 0.000 -
10360G>A 0.583 0.504 tn
10394G>A 0.000 0.000 -
10428C>T 0.313 0.321 tn
10486A>C 0.000 0.000 -
10487G>A 0.417 0.399 tn
χ2= Hardy-Weinberg equilibrium, tn = tidak nyata, (*) nyata α 5 % (χ2 obs ≥
3.84), n = 48 ekor.
Dari tabel tersebut dapat diketahui bahwa nilai heterozigositas pengamatan (Ho) tidak terlalu berbeda jauh dengan heterozigositas harapan (He). Tombasco et al. (2003) menyatakan bahwa jika nilai Ho (heterozigositas pengamatan) lebih rendah dari He (heterozigositas harapan) maka dapat mengindikasikan adanya proses seleksi yang intensif. Menurut Tambasco et al. (2003) perbedaan antara nilai heterozigositas pengamatan (Ho) dan nilai heterozigositas harapan (He) dapat dijadikan sebagai indicator adanya ketidakseimbangan genotipe pada populasi sapi yang diamati yang diindikasikan bahwa sudah ada kegiatan seleksi yang dilakukan dan tidak adanya perkawinan acak.
Keseimbangan populasi dapat dilihat melalui keseimbangan Hardy-Weinberg yang disajikan pada tabel 5. Hasil penelitian menunjukkan bahwa gen SCD pada SNP g.10360G>A, g.10428C>T dan g.10487G>A berada dalam keadaan seimbang. Sedangkan untuk gen DGAT1 dan SCD pada SNP 10153A>G, 10318C>A, 10329C>T, 10394G>A DAN 10487G>A tidak dapat dilakukan analisis keseimbangannya karena hanya ditemukan satu alel saja dengan frekuensi alel bersifat monomorfik. Faktor-faktor yang mempengaruhi keseimbangan dalam suatu populasi adalah non-random mating, seleksi, migrasi, mutasi dan genetic drift (Noor 2010).
Sifat Perdagingan pada Sapi Bali
16
untuk memprediksi tebal lemak punggung dan lemak intramuskular (PLIM) pada berbagai ternak seperti babi (Newcom et al. 2004, Jung et al. 2015), sapi (Miar et al. 2013), kelinci (Amalianingsih et al. 2014).
Berikut ini data mengenai sifat perdagingan pada sapi bali yang diperoleh dari hasil ultrasound sebagaimana disajikan pada Tabel 7.
Berbeda dengan penelitian Rachma et al. (2010) bahwa luas longissimus dorsi pada sapi bali umur 12 bulan yaitu 16.6-18.0 cm2. Sapi bali kisaran 2.5-3.5 tahun hasil penggemukan memiliki tebal lemak punggung (TLP) sekitar 8.40 mm (Yosita 2012) lebih besar daripada hasil penelitan. Bugiwati (2005) melaporkan bahwa marbling score pada sapi bali umur 12 dan 18 bulan mendekati skor 0 berdasarkan Beef Carcass Grading Standard lebih rendah dari hasil penelitian yang didapatkan. Hasil penelitian Putri et al. (2015) menunjukkan karakteristik perdagingan tebal otot longissimus dorsi dan tebal lemak punggung sapi bali yang berumur lebih dari 3 tahun dengan genotipe GG sebesar 57.577 mm dan 2.324 mm sedangkan genotipe AG memiliki tebal otot longissimus dorsi yang lebih
Sifat Performa Kisaran Performa
Tebal longissimus dorsi (mm) 33.047±5.077 25.006-43.95 Tebal lemak punggung (TLP) (mm) 1.455±0.348 0.972-2.051 tebal otot rump (TR) (mm) 40.086±4.0895 33.781-50.496 tebal lemak rump (TLR) (mm) 1.069±0.345 0.578-2.250 persentase lemak intramuscular (PLIM)
(%)
3.509±2.114 0.617-9.00
Marbling score (MS) 2.333±1.241 0-5
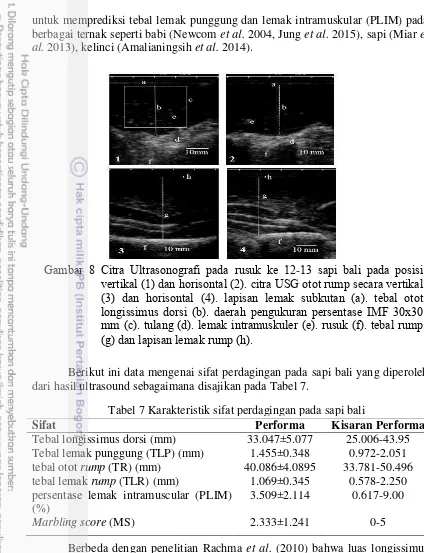
Gambar 8 Citra Ultrasonografi pada rusuk ke 1β-1γ sapi bali pada posisi
vertikal (1) dan horisontal (β). citra USG otot rump secara vertikal (γ) dan horisontal (4). lapisan lemak subkutan (a). tebal otot longissimus dorsi (b). daerah pengukuran persentase IMF γ0xγ0 mm (c). tulang (d). lemak intramuskuler (e). rusuk (f). tebal rump (g) dan lapisan lemak rump (h).
17
tinggi yaitu sebesar 63.818 mm dan tebal lemak punggung yang lebih rendah yaitu sebesar 1.935 mm. Persentase lemak intramuskular hasil USG memiliki rataan 3.509±2.114%. Pengukuran untuk sifat perdagingan juga dilakukan di bagian rump pada posisi antara antara tulang ischium dan illium (diantara penonjolan tulang hip dan hook), tebal rump dan tebal lemak rump diketahui rataannya sebesar 40.086±4.0895 dan 1.069±0.345 mm.
Asosiasi Gen SCD terhadap Sifat Perdagingan
Asosiasi antara gen SCD terhadap sifat perdagingan dilakukan pada SNP yang bersifat polimorfik, yaitu SNP g.10360C>T, SNP g.10428C>T dan SNP g.10487G>A. Tabel hasil asosiasi gen SCD terhadap perdagingan disajikan pada Tabel 5. Hasil analisis menyatakan bahwa untuk sifat perdagingan ditemukan adanya hubungan yang signifikan (P<0.05) antara SNP g.10428C>T terhadap sifat marbling score (MS) dan persentase lemak intramuskular (PLIM). Jika dibandingkan, sapi Bos taurus memiliki genotipe CC dan bersifat monomorfik, namun pada sapi bali di titik yang sama bersifat polimorfik.
Genotipe CT pada lokus g.10428C>T berpengaruh nyata terhadap sifat marbling score dan persentase lemak intramuskular pada sapi bali. Namun pada tabel tersebut genotipe CC pada lokus g.10428C>T memiliki nilai marbling dan persentase lemak intramuskular yang lebih tinggi dibandingkan genotipe CT atau TT. Sapi bali bergenotipe TT ditemukan lebih dominan dibandingkan dengan CT dan CC. Genotipe CC memiliki marbling score 4.8 yang berarti masuk kedalam kategori moderate – slightly abundant. Selain itu berdasarkan analisis persentase lemak intramuskular diketahui bahwa genotipe CC memiliki nilai persentase yang cukup tinggi jika dibandingkan dengan genotipe CT atau TT. Menurut Scott (2002) baik marbling maupun persentase lemak intramuskular berasosiasi dengan kualitas karkas. Beberapa penelitian menemukan bahwa terdapat hubungan yang sangat kuat antara marbling score dan persentase lemak intramuskular, dengan korelasi 0.7-0.9 (Scott 2011).
Tabel 8 Asosiasi SNP gen SCD intron 5 terhadap sifat kualitas karkas sapi Bali
18
Mutasi yang ditemukan dalam penelitian ini terjadi di bagian intron 5 gen SCD, intron merupakan wilayah non coding dalam DNA. Intron mungkin mengandung ikatan enhancer transcriptional yang didalamnya terdapat mutasi yang dapat mempengaruhi perubahan saat transkripsi. Intron juga mungkin mengandung urutan dari regulasi RNA yang akan berefek pada translasi dan kestabilan mRNA termasuk gen. Ketika terjadi mutasi, RNA akan mengubah produk dari gen. Mutasi di daerah ini dapat saja mempengaruhi fungsi gen tersebut. Kemudian pada saat translasi, intron terlibat dalam memproduksi protein. Sehingga terjadinya mutasi didaerah ini akan berefek pada sejumlah protein yang dihasilkan dan berpengaruh terhadap ekspresi dari gen tersebut (Perdew et al. 2006).
Penelitian gen SCD banyak dilakukan didaerah ekson, seperti penelitian Wu et al.(2011) yang menemukan SNP di titik c.10329C>T yang berasosiasi dengan persentase lemak intramuskular namun tidak berasosiasi nyata terhadap marbling score pada sapi Simmental Chinese, berpengaruh nyata terhadap aktifitas enzim SCD dan konsentrasi MUFA pada susu di sapi Italian Holstein (Conte et al. 2006) dan titik leleh dalam lemak intramuskular pada sapi Japanese Black (Taniguchi et al. 2004) serta berpengaruh nyata terhadap lemak otot dan lemak subkutan pada sapi Fleckvieh (Barton et al.2009). Selain itu, SNP c.10213T>C dilaporkan berasosiasi dengan tingginya persentase lemak intramuskular pada sapi Chinese Simmental (Wu et al.2011).Kualitas karkas dan daging dipengaruhi oleh beberapa faktor antara lain genetik, spesies, bangsa, jenis kelamin, umur, pakan termasuk bahan aditif (hormon, antibiotik atau mineral), dan stress (Soeparno 2005).
4 SIMPULAN DAN SARAN
Simpulan
Gen DGAT1 pada SNP c.10433G>A dan c.10434C>A disapi bali bersifat monomorfik. Gen SCD pada sapi bali diperoleh 8 SNP terdiri atas 5 SNP bersifat monomorfik dan 3 SNP bersifat polimorfik. SNP g.10428C>T berpengaruh nyata terhadap sifat marbling score (MS) dan persentase lemak intramuskular (PIMF), sehingga SNP tersebut dapat dijadikan sebagai salah satu kandidat MAS pada sapi bali khususnya di sifat perdagingan.
Saran
19
5 DAFTAR PUSTAKA
Amalianingsih TI, Brahmantiyo B, Jakaria. 2015. Identification of rabbit myostatin gene polymorphisms. J Med. Pet. 38(2): 77-81
Barton, L., T. Kott, D. Bures, D. Rehak, R. Zahradkova, and B. Kottova. 2010. The polymorphism of stearoyl-CoA desaturase (SCD1) and sterol regulatory element binding proyein-1 (SREBP-1) genes and their association with the fatty acid profile of muscle and subcutaneous fat in Fleckvieh bulls. Meat Sci. 85(1):15-20.
Bugiwati SRA. 2006. Seleksi pejantan unggul sapi bali melalui pendugaan sifat karkas dengan menggunakan alat bantu ultrasonografi. Laporan Hibah Bersaing XIII. Fakultas Peternakan, Universitas Hasanuddin. Makassar. Cardoso SR, Queuroz LB, Goulart VA, Mourao GB, Benedetti E, Goulart LR
2011. Productive performance of the dairy cattle Girolando breed mediated by the fat-related genes DGAT1 and LEP and their polymorphisms. J Vet. Sci 91:107-113.
Coleman R and Bell RM. 1976. Triacylglycerol synthesis in isolated fat cells. Studies on the microsomal diacylglycerol acyltransferase. J Biol Chem 251:4537-4543.
Conte G, Mele M, Castiglioni B, Serra A, Viva M, Chessa S,Pagnacco G, Secchiari P. 2006. Relationship between bovine SCD polymorphism locus and mammary gland desaturation activity. In: Proceedings of the 8th world congress on genetics applied to livestock production, Belo Horizonte, Minas Gerais, Brazil, 13–18 Aug, pp. 22–37.
[DGLS] Directorate Generale of Livestock Services. 2003. National Report on Animal Genetic Resources in Indonesia. Directorate Generale of Livestock Services, Directorate of Livestock Breeding (ID).
Dewitri J, Merthayasa, I Ketut S, Kadek KA. 2015. Daya ikat air, pH, warna, bau dan tekstur daging sapi bali dan daging wagyu. Jurnal Indonesia Medicus Veterinus 4(1) : 16 – 24.
Eko H dan Subandriyo. 2004 Potensi dan keragaman sumberdaya genetik sapi bali Proc Wartazoa vol 14 No 13. Pusat Penelitian dan Pengembangan Peternakan.
Febriana A, Farajallah A, Dyah P. 2015. Kejadian indel simultan pada intron 7 gen Branched-Chain α-Ketoacid Dehydrogenase E1a (BCKDHA) pada Sapi Madura. J Ilmu Pertanian Indonesia. Vol. 20 (2): 97-102.
Fortes MRS, Curi RA, Chardulo LAL, Silveira AC, Assumpção MEOD, Visintin JA, deOliveira H N. 2009. Bovine gene polymorphisms related to fat deposition and meat tenderness. Genetics and Molecular Biology, 32(1), 75–82.
Grisart B, Farnir F, Karim L, Cambisano N, Kim J-J, Kvasz A, Mni M, Simon P, Frère J-M, Coppieters W and Georges M .2004. Genetic and functional confirmation of the causality of the DGAT1 K232A quantitative trait nucleotide in affecting milk yield and composition. Proc Natl Acad Sci USA 101:2398-2403.
20
Javanmard A, Asadazadeh N, Banabazi MH, Tavakolian J. 2005. The allele and genotype frequencies of bovine pituitary specific transcription factor and leptin genes in Iranian cattle and buffalo populations using PCR-RFLP. J Iranian Biotechnol. 3: 104-108.
Jung JH, Shim KS, Na CS, Choe HS. 2015. Studies on Intramuscular Fat Percentage in Live Swine Using Real-time Ultrasound to Determine Pork Quality. Asian Aust J. Ani. Sci. 28 (3): 318-322.
Kaps M, Lamberson WR. 2004. Biostatistic for Animal Science. London (GB): CABI Publising.
Kgwatalala, P. M., E. M. Ibeagha-Awemu, J. F. Hayes, and X. Zhao. 2007. Single nucleotide polymorphism in the open reading frame of the stearoyl-CoA desaturase gene and resulting genetic variants in Canadian Holstein and Jersey cows. Mitochondrial DNA. 18:357-362.
Kgwatalala, P.M., Ibeagha-Awemu, E.M., Mustafa, A.F., Zhao, X., 2009. Stearoyl CoA desaturase 1 genotype and stage of lactation influences milk fatty acid composition of Canadian Holstein cows. J. Anim. Genet. 40, 609–615.
Kong JHS, Oh D, Lee JH, Yoon DH, Choi YH, Cho BW, Lee HK, Jeon GJ. 2007. Association of Sequence Variations in DGAT 1 Gene with Economic Traits in Hanwoo (Korea Cattle). Asian-Aust. J Anim.Sci 20 : 817-820. Marson EP, Ferraz JBS, Meirelles FV, Balieiro JCC, Eler JP, Figuerido LGG,
Mourao GB. 2005. Genetic characterization of European-Zebu composite bovine using RFLP markers. J Genet Mol Res. 4: 496-505.
Miar GY, Plastow H, Bruce L, Moore SS, Duruma ON, Nikrumah JD, Wang Z. 2013.Estimation of genetic and phenotypic parameters for ultrasound and carcass merit traits in crossbred beef cattle. Canad. J. Anim. Sci. 94: 273-280.
Milanesi E, Nicoloso L, Crepaldi P. 2008. Stearoyl-CoA desaturase (SCD) gene polymorphism in Italian cattle breeds. J. Anim. Breed. Genet. 125:63-67. Mizoshita K, Watanabe T, Hayashi H, Kubota C, Yamakuchi H, Todoroki J,
Sugimoto Y. 2004. Quantitative trait loci analysis for growth and carcass traits in half-sib family purebred Japanese Black (Wagyu) cattle. J. Anim. Sci. 82: 3415–3420.
Moore SS, C Li, J Basarab, WM Snelling, J Kneeland, B Murdoch, C Hansen, B Benkel. 2003. Fine mapping of quantitative trait loci and assessment of positional candidate genes for backfat on bovine chromosome 14 in a commercial line of Bos taurus. J Anim. Sci. 81:1919–1925.
Muladno. 2002. Teknik Rekayasa Genetika. Bogor (ID) : Pustaka Wirausaha Muda
Nei M, Kumar S. 2000. Molecular Evolution and Phylogenetics. New York (US): Oxford Univ Pr.
Noor R R 2010. Genetika Ternak. Jakarta (ID): Penebar Swadaya.
Oh DY, Lee YS, Yeo JS. 2011. Identification of the SNP (single nucleotide polymorphism) of the stearoyl-CoA desaturase (SCD) associated with unsaturated fatty acid in Hanwoo. Asian-Aust. J Anim Sci. 24:757-765. Ohsaki, H., A. Thnaka, S. Hoashi, S. Sasazaki, K. Oyama, M. Taniguchi, F.
21
acid composition in adipose tissue of Japanese Black cattle herds. Anim. Sci. J. 80:225-232.
Pelt-Verkuil EV, Belkum AV, Hays JP. 2007. Principles and Technical Aspects of PCR Amplification.Netherlands (GB): Springer.
Perdew GH, John PVH, Jeffrey MP. 2006. Regulation of Gene Expression: Moleculer Mechanisms. New Jersey (UK). Humana Press.
Putri R, Priyanto R, Gunawan A, Jakaria. 2015. Association of calpastatin (CAST) gene with growth traits and carcass characteristics in bali cattle. Media Petern.38(3):145-149.
Raymond, M. and F. Rousset. 2001. Genepop (3.3). Population Genetics Software
for Exact Tests and Ecumenicism (EB/OL)
(http:www:wbiomed.curtin.edu.au/ genepop).
Reardon, W., Mullen, A. M., Sweeney, T., & Hamill, R. M. 2010. Association of polymorphisms in candidate genes with colour, water-holding capacity, and composition traits in bovine M. longissimus and M. semimembranosus. J Meat Sci. 86, 270–275.
Ribeca C, Bonfatti V, Cecchinato A, Albera A, Gallo L, Camier P. 2014. Effect of polymorphisms in candidate genes on carcass and meat quality traits in double muscled Piemontese cattle. J Meat Sci. 96 : 1376-1383.
Ripoli MV, Corva P, Giovambatista G. 2006. Analysis of a polymorphism in the DGAT1 gene in 14 cattle breeds through PCR-SSCP methods. J Vet. Sci 80 : 287-290.
Schennink A, Heck JM, Bovenhuis H, Visker MH, Van Valenberg HJ, Van Arendonk JA. 2008. Milk fatty acid unsaturation: genetic parameters and effects of stearoyl-CoA desaturase (SCD1) and acyl CoA:diacylglycerol acyltransferase 1 (DGAT1). J Dairy Sci. 91(5):2135–2143. doi:10.3168/jds.2007-0825
Scott P. Greiner. 2002. The Relationship Between Marbling and Intramuscular Fat. Virginia Cooperative Extension.Virginia State University.
Silva, S.L., J.U. Tarouco, J.B.S. Ferraz R., da C. Gomes, P.R. Leme, E.A. Navajas. 2012. Prediction of retail beef yield. trim fat and proportion of high-valued cuts in Nellore cattle using ultrasound live measurements. R. Bras. Zootec.41 (9):2025-2031.
Soeparno. 2005. Ilmu dan Teknologi Daging Cetakan Keempat.Yogyakarta (ID): Gadjah Mada. University Press
Talib C. 2002. Sapi Bali di daerah sumber bibit dan peluang pengembangannya. J Wartazoa 12(3):100-107.
Tambasco DD, Paz CCP, Tambasco-Studart M, Pereira AP, Alencar MM, Freitas AR, Coutinho LL, Packer IU, Regitano CA. 2003. Candidate genes for growth traits in beef cattle crosses Bos taurus x Bos indicus. J Anim Breed Genet. 120: 51-56.
Tamura, K.,D. Peterson, N. Peterson, G. Stecher, M. Nei and S. Kumar. 2011. MEGA5: molecular evolutionary genetics analysis using maximum likelihood. evolutionary distance. and maximum parsimony methods. J Mol. Biol. Evol. 28(10):2731–2739.
22
associated with fatty acid composition in Japanese Black cattle. Mann. Genome 14, 142–148
Thaller, G., Kramer, W., Winter, A., Kaupe, B., Erhardt, G., Fries, R., 2003. Effects of DGAT1 variants on milk production traits in German cattle breeds. J. Anim. Sci. 81 (8), 1911–1918.
Tsuji S. 2008. Kobe beef as international brand. Symposium of Gyeungbuk Hanwoo Cluster.
Ulum MF, Suprapto E, Jakaria. 2014. Citra ultrasonografi otot punggung (longisimus dorsi) pada sapi bali. Proceeding. Konverensi Ilmiah Veteriner Nasional ke-13 Perhimpunan Dokter Hewan Indonesia (PDHI), 2014 Nov 23-26, Padang (ID) : KIVNAS PDHI XIII. Hlm 368-369.
Winter A, Kra¨mer W, Werner FAO, Kollers S, Kata S, Durstewitz G, Buitkamp J, Womack JE, Thaller G, Fries R. 2002. Association of a lysine-232/alanine polymorphism in a bovine gene encoding acyl CoA: diacylglycerol acyltransferase DGAT1 with variation at a quantitative trait locus for milk fat content. Proc. Natl. Acad. Sci. USA 99:9300–9305. Wu, X. X., Yang, Z. P., Shi, X. K., Li, J. Y., Ji, D. J., Mao, Y. J., Chang, L. L., & Gao, H. J. (2012). Association of SCD1 and DGAT1 SNPs with the intramuscular fat traits in Chinese Simmental cattle and their distribution in eight Chinese cattle breeds. J Molecular Biology Reports, 39, 1065– 1071.
Xin Li, Marie Ekerljung, Kerstin Lundström, Anne Lundén. 2013. Association of polymorphisms at DGAT1, leptin, SCD1, CAPN1 and CAST genes with color, marbling and water holding capacity in meat from beef cattle populations in Sweden. Sweden (SD). Meat Science 94 153–158.
Yang A, Larsen TW, Powell VH, Tume RK (1999) A comparison of fat composition of Japanese and longterm grain-fed Australian steers. J Meat Sci 51, 1–9.
Yokota S, Sugita H, Ardiyanti A, Shoji N, Nakajima H, Hosono M, Suzuki K. 2012. Contributions of FASN and SCD gene polymorphisms on fatty acid composition in muscle from Japanese Black cattle. J Anim Gen 43(6): 790–792.
23
24
Lampiran 1
Sekuen gen DGAT1 berdasarkan genbank kode akses AJ318490
Bos taurus dgat gene for Acyl-CoA:1,2 diacylglycerol O-transferase and partial hsf1 gene for Heat Shock Factor Protein 1
GenBank: AJ318490.1
FASTAGraphics
LOCUS AJ318490 14117 bp DNA linear MAM 21-NOV-2006
DEFINITION Bos taurus dgat gene for Acyl-CoA:1,2-diacylglycerol O-transferase
and partial hsf1 gene for Heat Shock Factor Protein 1. ACCESSION AJ318490
VERSION AJ318490.1 GI:21425304
KEYWORDS Acyl-CoA:1,2-diacylglycerol O-transferase; Dgat gene; Heat Shock
AUTHORS Winter,A., Kramer,W., Werner,F.A., Kollers,S., Kata,S.,
Durstewitz,G., Buitkamp,J., Womack,J.E., Thaller,G. and Fries,R.
TITLE Association of a lysine-232/alanine polymorphism in a bovine gene
29
1 ctgccccgac aggcctgaca accaacaaca agccttcctc aatgccacta gagaaatggg
30
31
32
12421 gctaaggtgt ggaagagggt tgagaatcag gctgacttga acggcagcaa agactccaag 12481 gcaaggctgc agaggtctca gaggctatgc gcacagtccc ctgctggggt gctcacctgg 12541 gctgggctct gggctgcttg gacaaagcag gtggcctggc tcagccctca ccgagggcct 12601 cccttggggg cagaggttgg cctgatgcca ggggctcccc gtttttccag gccctcagca 12661 ggtagttggg tgtggccctc aggatacctt ggtcccagag cttgccactc aaaaagcttg 12721 gcagtgaggc aagggcaacc ccgggctgtt cccccctcta ctggctctgc cgcctgggtt 12781 ggaaaccctg aggctgtgcc aggcaggtgt accctgacag ccagccatgg cccagtaaga 12841 tgggtgcccg aggtggtacc tgggcagcgg acccagctgt gctgcccccg ccccaaccag 12901 aagccgctct agcccatggt ggtcgtctgg gcgagacagg ctggttggct aggcactgtt 12961 tggtctacag caggtgtagg cagcgtctcc ctgacccctg cctcctagga agccaccacc 13021 ctgggcccta ctcatcagca aggacagcga gcagggctga gctgggggtg cgtgggctgc 13081 tacggcccgc cacctccatc acatgcacct ctgcaccccc tgctgcctga ctcaggagtg 13141 gggggggggg tcctgtgctt ccttcactcc agacccacgg tgctgaccca gtgcacccac 13201 ctggtcctct agtgcggacc tggccacagg gctcctgtgg gcccacgctg atcccgccct 13261 ggtcccttca taaagaactc ttgagcacat gcagcccagg ggagccagga ggctccagtg 13321 tgctgtgtcc atctgcctcc ctccagcccc ttccgagaca ctgcgcatca tgcccccctc 13381 cacccccacc cacactggca ggaggaacag acagggagac cacacacaga gctcgttgtt 13441 tataaatctc tgcctggctc atcggtctgt ttgtccatgt atatatctgt atatctctat 13501 ggaaggggaa agggggactc gtgtaaaaat ccaaaataca attctatgaa cacctgcatc 13561 ctggtcagtc tgagtgtggc cgtgaagccc aggtgagctg tggctcacag ggctaggccc 13621 tcggtgctgg ccgggggcca cgccccaccc cctctccccc cctccgccag ccaggggacc 13681 aggctcctgg acaccaggcc tgcccaaggc ctgctctcct cctggggctt ctacgagaca 13741 gtggggtcct tggctttggg gggttctgag cccgtcagca gggagatggt ggggtcatcc 13801 gagtagtcgt ctccctcgga gaagtaggag ccctccccca gctcgaagag caccggcagg 13861 tcgctgctcc ccacgtccac ggagcccggg tccaggagca gcaggggctg ggcggtgtag 13921 tgcaccaact gcttccctag gggtgcgact gggtcaaggt gccggtgggg ccggggggcg 13981 gggtgggggt ggggggctca gctcacctga gtctgggctg cttttctctg cctccagagg 14041 tctggggggc tcctggggag agaggagctc ctggatctgc tggggcagca ggagggagca 14101 cagtgagggc tcccgcg
//
Lampiran 2 Sekuen gen SCD
Sekuen gen SCD berdasarkan genbank kode akses AY241932
LOCUS AY241932 17088 bp DNA linear MAM 02-APR-2003
DEFINITION Bos taurus stearoyl-CoA desaturase variant A (SCD) gene, complete
AUTHORS Medrano,J.F., Islas-Trejo,A.D. and Johnson,A.M.
TITLE Genomic sequence of the bovine stearoyl-CoA desaturase JOURNAL Unpublished
REFERENCE 2 (bases 1 to 17088)
AUTHORS Medrano,J.F., Islas-Trejo,A.D. and Johnson,A.M. TITLE Direct Submission
34
35
36
37
38
14521 atcacattgc tgggggattc agaaggttac tgagtaagtt gttgggtgtc ctgatataga 14581 agctggttat acaaacaagt tagatgttgg gttcatttca ttaattccac tttctccttg 14641 gattgagaaa gcattagaag gcttctcccc acggtgttga accctttcac tcattccttc 14701 tattaccttc tagcggaaaa tacaggactg gctgggggat ggggtaggaa tctctcaact 14761 accctatcaa ttcttggctc tgccatcttt gtccactttc tcctgctggt tttatctcct 14821 tgacgtttcc ttctttttct ggacaggcaa gcctcttctg tgtgtattca gaggcagtga 14881 tggctactgc ggtccaagtc gttccctctc ttactgacag aatggtcagg gtcactgaac 14941 cactgtttct ctttacaaag ttgagcaagc tgccactttc acttggcctc cagagtctcc 15001 atctatatcc ttgtgctcct taccacactg atgactccag acaaggctgg caaagcctgc 15061 tagaaacatc ctgggcacag gcattcgcac tcatgaggca cggccaagcc gaatgctcat 15121 gttgtgccag agccagccat ggagcaaaag aggatttgtt tttagtctcc tctgtctggg 15181 tcagaaccag agagcatgct ggatgccccc ggcttactgg ataagctgcc taccctgagt 15241 cagtgctccc agcggacagt gcgaggcttg cagaagcagg gggtgcctag ccttcactgg 15301 gaagcacaag aagcaaaggc aggttccaaa gtgcctcact cagaaggtgg ccccagcccc 15361 ctggagggag ccagggtgta ccgcaagacc ttgactgagg cttaggatgt gagatgccat 15421 gaactttgct gaacagtgtc tctgttcagc aaactaacca gcattcccca caacacagtc 15481 tagggcagac gatagtatag aggagtgttg gaagaacctt gggtcccttt gtccctgtaa 15541 cctcagttgt ctaggcagaa acctggcttt attctattta aaggttgaaa atatacaata 15601 ccaaatgctc tgccactgtt gagctccaag gatggaaagg aggagaacat ttcttcctgt 15661 attaattgga tagatggagg ctacagagct taggctaaac taaaggcatc cttgtctttt 15721 gagttgttcc tctcagtagg aaaaaaaaaa aatctaatgg aagatcactg tagattagat 15781 cctctgacca agcacctacc gcttggaaat gcctgtgggg tagttttaat tccacaggtc 15841 atcagatgca tgctttacaa ctgatgatca aaaccaactt atctttctat tctaattgtg 15901 ttccgtggat ctgatctata ccatgaccct acacaaggct ggatggtgtc cttgggccca 15961 gggtacttgt acttgtgtag gtgggggttg tctactgagt aaggaatact gtttttaagg 16021 ttctaaagct aaattcaaat gatgcattaa tgacccaaaa actcagatct gatggtgtct 16081 gaatttctaa cagtccttgc tttgtgggta tgctgacaac ttatctggat gccttacatc 16141 ttttctaaac agtgttgcct ctgaacgtgc tctgctccct ccctgctccc tctttggagc 16201 ccctttgcac cccagagcct gcagaagtgg ctggtataaa agggggcctg gctagagaat 16261 gatcagtgta gctgtttgca ggattccttt ctgggcttca ttttggaaac tttgcttagg 16321 gctatttttc ttaattgccc acatttgatg gagggtagaa ggaattttga atgtatttga 16381 tttattatta tttttttttt tttagattaa aggatggttg tagcatttaa aatggaaatt 16441 tttcctcctg gttagctagt atcctgagtg tattctctgt aagtgtagct caaatgggtc 16501 atcatgaaaa gttcaagaaa gctcgatgtc aaagttatat gggtggttaa ggccagggcc 16561 tgtcctacca ctgtgccact gacttgctat gtgaccctgg gcaagtcatt taactataat 16621 gtgcctcagt tttccttctg ttaaaatggg ataataatac tgacctacct caaagggcag 16681 ttttgaggca tgactaatgc tttttataaa gcatcttgga attctcttaa gttctgagta 16741 tttttatagt agcagtatcc accatgaagt gtgtccacca cgagccacgt gtcctggatg 16801 ccgtcaggaa tctatatggt tctctctgag agatggaata aatgcatcag ataaagggtg 16861 gataactagc cggacaaaat ctgagaatgc ataaactcat tgccatggaa acatacacag 16921 gatacctttt ccttaattgg gtgggatttt tcccttttta tgtgggatag tagttatttg 16981 tgacctaaga ataattttgg aataatttct attaatatca actccaaagc tagttgtact 17041 gatctgagat tgtgtttgtt cataataaaa gtgaatctga ttgccctg
39
RIWAYAT HIDUP
Penulis lahir di Cirebon pada tanggal 20 April 1993. Penulis adalah anak dari pasangan Anis Hamzah Alhiyed dan Sofiah Alatas. Penulis merupakan anak kedua dari 2 bersaudara yaitu Fatimmah Tuzahro. Penulis mengawali pendidikan sekolah dasar pada tahun 1999 di SD Negeri Lawu Asih dan menyelesaikan sekolahnya pada tahun 2005. Selanjutnya penulis melanjutkan pendidkan ke tingkat pertama pada tahun 2005 di SMPN 2 Cirebon dan selesai pada tahun 2008. Penulis melanjutkan pendidikan di SMA Muhammadiyah Cirebon dari tahun 2008 hingga 2011. Penulis diterima di Institut Pertanian Bogor pada tahun 2011 melalui jalur SNMPTN Tulis dan diterima di Departemen Ilmu Produksi dan Teknologi Peternakan. Waktu menempuh pendidikan S1, penulis memperoleh kesempatan untuk mengikuti program percepatan pendidikan S1-S2 yaitu program sinergis sehingga penulis berkesempatan menyelesaikan studi S1-S2 dalam 5 tahun.