KARAKTERISASI MOLEKULER BOVINE HERPESVIRUS -1
GLIKOPROTEIN C (gC) ISOLAT INDONESIA
(Molecular Characterization of Bovine Herpesvirus-1
Glycoprotein C (gC) Indonesian Isolates)
MUHARAM SAEPULLOH danR.M.A.ADJID Balai Besar Penelitian Veteriner, PO Box 151, Bogor 16114
ABSTRACT
Subtypes of BHV-1.1 have been associated with respiratory desease, whereas BHV-1.2 with genital disorder. Different subtypes of Bovine Herpesvirus 1 (BHV-1) have been associated with different clinical conditions of cattle. For that reason, subtypes differentiation has become an essential tool for understanding the pathogenesis and epidemiology of BHV infections. In search for a genomic region that would allow a clear distinction between BHV-1.1 and BHV-1.2 of glycoprotein C (gC), genes of 21 Indonesian isolates were amplified and sequenced. The amino acid sequence alignments revealed that the levels of genomic gC BHV-1 similarity ranging from 75,3 to 100% among BHV-1 Indonesian isolates and the results were also similar between gC BHV-1 Indonesian isolates and gC BHV-1.1 reference, and 7.5 to 15.1% between BHV-1 Indonesian isolates and 1.2 reference. The isolates could be clearly separated into 1.1 and BHV-1.2 after phylogenetic analysis. Results showed that the Indonesian isolates were characterized as BHV-1.1 as agent caused respiratory tract infections in cattle or infectious bovine rhinotracheitis (IBR) disease. The results suggest that the phylogenetic analysis performed here can be used as a potential molecular epidemiological tool for herpesviruses.
Key Words: BHV-1.1 gC, BHV-1.2 gC, IBR, Phylogenetic
ABSTRAK
Subtipe BHV-1.1 berkaitan dengan penyakit gangguan pernapasan, sedangkan BHV-1.2 selalu dihubungkan dengan penyakit gangguan genital. Perbedaan subtipe Bovine Herpesvirus 1 (BHV-1) seringkali dihubungkan dengan adanya gejala klinis yang ditimbulkan oleh penyakit tersebut pada ternak sapi. Dalam hal ini perbedaan subtipe akan menjadi penting manakala diperlukan dalam kajian patogenesis dan epidemiologis infeksi BHV-1. Telah berhasil dibedakan antara gen glikoprotein C (gC) BHV-1.1 dan BHV-1.2 setelah melalui proses amplifikasi dan sekuensing terhadap 21 isolat BHV-1 asal Indonesia. Hasil pensejajaran terhadap urutan asam amino menunjukkan bahwa kesamaan genom antar BHV-1 gC isolat Indonesia mencapai 75,3% hingga 100%, dan ini sama dengan persentase kesamaan asam amino antara BHV-1 gC isolat Indonesia dengan BHV-BHV-1.BHV-1 gC rujukan. Sedangkan untuk BHV-BHV-1 gC isolat Indonesia dengan BHV-1.2 gC rujukan memiliki persamaan asam amino antara 7,5% hingga 15,1%. Selanjutnya, isolat-isolat tersebut dapat dikelompokkan ke dalam BHV-1.1 dan BHV-1.2 setelah dilakukan analisis filogenetik. Hasil analisis filogenetik menunjukkan bahwa semua 1 isolat Indonesia termasuk ke dalam kelompok BHV-1.1 yang merupakan agen penyebab infeksi saluran pernapasan pada ternak sapi atau biasa dikenal sebagai penyakit infectious bovine rhinotracheitis (IBR). Hasil tersebut diharapkan dapat dijadikan dalam kajian epidemiologi molekuler untuk kelompok virus herpes.
Kata Kunci: BHV-1.1 gC, BHV-1.2 gC, IBR, Filogenetik
PENDAHULUAN
Bovine Herpesvirus type 1 (BHV-1)
merupakan penyakit virus yang patogen baik bagi ruminansia domestik maupun liar yang telah tersebar ke suluruh dunia dan secara
ekonomi sangat signifikan merugikan bagi industri peternakan di berbagai negara. Virus BHV-1 juga merupakan penyebab penyakit gangguan pernapasan (Infectious bovine
rhinotracheitis, IBR), konjungtivitis, vulvovaginitis, balanopostitis, shipping fever
(demam pengapalan), dan infeksi sistemik lainnya (GIBB dan RWEYEMAMU 1977; WYLER et al., 1989; TIKOO et al., 1995).
Berdasarkan sifat antigenik dan genomik, BHV-1 dibedakan menjadi subtipe 1 (BHV-1.1) dan 2 (BHV-1.2) (MILLER et al., 1991;
ROIZMAN et al., 1992). Kedua subtipe tersebut
dapat menimbulkan penyakit dengan gejala klinis yang berbeda pada ternak sapi (ENGELS et al., 1992). Bovine herpesvirus type 1.1
(BHV-1.1) dapat menyebabkan infeksi saluran pernapasan yang lebih dikenal sebagai
Infectious bovine rhinotracheitis (IBR).
Sedangkan subtipe BHV-1.2 seringkali berhubungan dengan penyakit penyebab gangguan genital yang biasa dikenal sebagai
Infectious pustular vulvovaginitis (IPV) pada
sapi betina atau Infectious pustular
balanopostitis (IPB) pada sapi jantan.
Kemudian, varian BHV-1.2 lebih lebih lanjut dapat dibedakan sebagai 1.2a dan BHV-1.2b dimana BHV-1.2a seringkali berhubungan dengan penyakit penyebab keguguran pada sapi (MILLER et al.,1991). Secara epidemiologi
penyakit, keduanya (BHV-1.2a dan BHV-1.2b) merupakan galur yang berbeda dalam manifestasi gejala klinis, akan tetapi hingga saat ini perbedaan genetis kedua galur tersebut masih belum jelas (SPILKI et al., 2004). Kedua
subtipe (BHV-1.1 dan BHV-1.2) memiliki kemampuan dalam menimbulkan gangguan pernapasan maupun gangguan genital pada sapi, sehingga diduga bahwa masing-masing genotipe dapat beradaptasi, baik pada saluran pernapasan (BHV-1.1) maupun saluran genital (BHV-1.2) (RIJSEWIJK et al., 1999; SPILKI et al., 2004).
Berdasarkan sekuens nukleotidanya, BHV-1 paling sedikit memiliki 10 gen yang berpotensi menyandi glikoprotein yaitu gB, gC, gD, gE, gG, gH, gI, gK, gL, dan gM, yang sangat berperanan penting dalam patogenisitas, virulensi, dan replikasi virus dalam sel hospes (inang). Infeksi virus diperantarai oleh interaksi spesifik antara pelekatan virus protein (VP) dan reseptornya pada permukaan sel. Pada kelompok alphaherpesvirus, interaksi antara virus protein dan reseptor seluler diikuti dengan penetrasi virus melalui fusi sel membran. Kedua tahapan tersebut
menyebabkan proses masuknya virus pada sel inang (LI et al., 1995). Pelekatan alphaherpesvirus pada sel melalui interaksi
antara molekul gC dengan glikosaminoglikan, heparan sulfat (HS) (SPEAR et al., 1992). Untuk BHV-1, hanya virus protein pada gC yang memiliki kemampuan untuk mengikat komponen yang menyerupai haparin
(heparin-like) (OKAZAKI et al., 1991). Berdasarakan
penelitian yang menggunakan antibodi (DUBUISSON et al., 1992), fraksinasi membran
sel (KUHN et al., 1990), dan virosome
(JOHNSONet al., 1984) bahwa molekul gB dan
gD diduga kuat berperan dalam pelekatan virus. Berdasarkan laporan LIANG et al. (1991)
bahwa BHV-1 gB, gC, dan gD dapat menghambat pelekatan virus pada suhu 4°C, selain itu gC tidak saja dapat menghambat BHV-1 tipe liar (wild-type) juga dapat menghambat mutan negatif gC hingga ke tingkat yang terendah, sementara itu gB dan gD dapat menghambat BHV-1 tipe liar dan virus negatif gC saja.
Berdasarkan pemeriksaan sampel usap mukosa nasal, vagina, semen dan pheripheral
blood leucocytes (PBLs) asal sapi bibit telah
berhasil dideteksi penyebab penyakit IBR (BHV-1) dari beberapa daerah di Indonesia (SAEPULLOH et al., 2008). Akan tetapi, untuk
mengetahui bahwa isolat-isolat lapang tersebut termasuk ke dalam BH-1.1 atau BHV-1.2 diperlukan kajian lebih lanjut, yaitu dengan melakukan karakterisasi molekuler. Dengan diketahuinya karakteristik isolat-isolat BHV-1 diharapkan akan mempermudah penanggulangan dan penanganan terhadap penyakit IBR di Indonesia. Selanjutnya, isolat virus yang telah terkarakterisasi dapat dijadikan sebagai sumber kekayaan plasma nutfah genetik (genetic resources) yang pada gilirannya dapat dijadikan sebagai bahan biologik, baik untuk pengembangan vaksin IBR maupun untuk bahan diagnostik lainnya.
Tujuan penelitian ini adalah untuk karakterisasi BHV-1 glikoprotein C (gC) isolat Indonesia yang dibandingkan dengan BHV-1.1 gC rujukan serta BHV-1 gC yang telah terdeposit di GenBank melalui amplifikasi DNA, sekuensing dan analisis hubungan kekerabatan terhadap gen gC BHV-1.
MATERI DAN METODE
Virus dan isolat
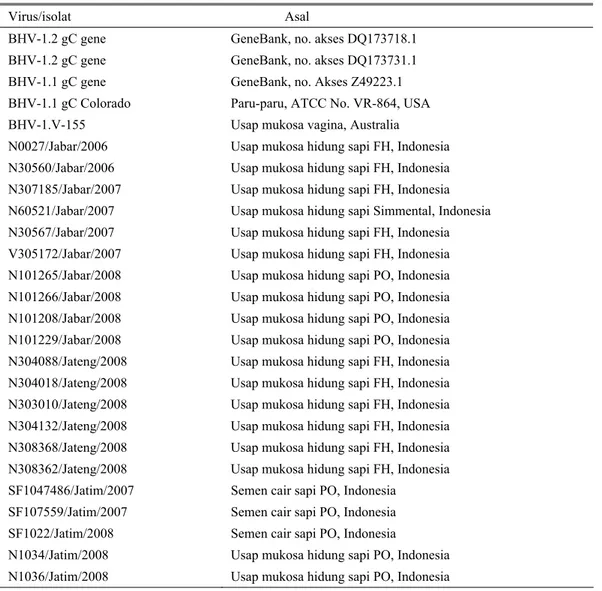
Virus rujukan dan isolat yang digunakan untuk karakterisasi molekuler dalam penelitian ini tercantum dalam Tabel 1. Semua virus telah ditumbuhkan dan beradaptasi dengan baik pada sel lestari Madin Darby Bovine Kidney (MDBK).
Ekstraksi DNA
Ekstraksi DNA virus yang dipropagasi pada sel lestari MDBK dikerjakan berdasarkan prosedur FRANCO et al. (2002) sebagai berikut:
setelah virus/isolat IBR diinfeksikan pada sel MDBK selama 36 jam atau setelah timbul CPE (Cytophatic Effect) 90 – 100%, maka supernatan disentrifugasi 5.000 x g selama 20 menit untuk menghilangkan sel lestari MDBK. Kemudian sentrifugasi dilanjutkan pada 100.000 x g selama 2 jam pada suhu 4°C. Pellet virus kemudian diresuspensikan dengan penambahan TE (Tris 10mM, EDTA 1 mM, pH 7,4) dan diberi perlakuan dengan penambahan sodium dodecyl sulphate dan
proteinase-K (konsentrasi akhir
masing-masing 1% dan 100 μg/ml) selama 1 jam pada suhu 37°C. Selanjutnya virus DNA di ekstraksi dengan menggunakan
Tabel 1. Virus rujukan dan isolat yang digunakan untuk karakterisasi molekuler BHV-1
Virus/isolat Asal BHV-1.2 gC gene GeneBank, no. akses DQ173718.1
BHV-1.2 gC gene GeneBank, no. akses DQ173731.1 BHV-1.1 gC gene GeneBank, no. Akses Z49223.1 BHV-1.1 gC Colorado Paru-paru, ATCC No. VR-864, USA BHV-1.V-155 Usap mukosa vagina, Australia N0027/Jabar/2006 Usap mukosa hidung sapi FH, Indonesia N30560/Jabar/2006 Usap mukosa hidung sapi FH, Indonesia N307185/Jabar/2007 Usap mukosa hidung sapi FH, Indonesia N60521/Jabar/2007 Usap mukosa hidung sapi Simmental, Indonesia N30567/Jabar/2007 Usap mukosa hidung sapi FH, Indonesia V305172/Jabar/2007 Usap mukosa hidung sapi FH, Indonesia N101265/Jabar/2008 Usap mukosa hidung sapi PO, Indonesia N101266/Jabar/2008 Usap mukosa hidung sapi PO, Indonesia N101208/Jabar/2008 Usap mukosa hidung sapi PO, Indonesia N101229/Jabar/2008 Usap mukosa hidung sapi PO, Indonesia N304088/Jateng/2008 Usap mukosa hidung sapi FH, Indonesia N304018/Jateng/2008 Usap mukosa hidung sapi FH, Indonesia N303010/Jateng/2008 Usap mukosa hidung sapi FH, Indonesia N304132/Jateng/2008 Usap mukosa hidung sapi FH, Indonesia N308368/Jateng/2008 Usap mukosa hidung sapi FH, Indonesia N308362/Jateng/2008 Usap mukosa hidung sapi FH, Indonesia SF1047486/Jatim/2007 Semen cair sapi PO, Indonesia
SF107559/Jatim/2007 Semen cair sapi PO, Indonesia SF1022/Jatim/2008 Semen cair sapi PO, Indonesia
N1034/Jatim/2008 Usap mukosa hidung sapi PO, Indonesia N1036/Jatim/2008 Usap mukosa hidung sapi PO, Indonesia
fenol:kloroform:isoamil alkohol, diendapkan dengan ethanol, dan akhirnya diresuspensikan dengan menggunakan TE pH 7,4 dan disimpan pada suhu 4°C atau -20°C sampai sampel siap di PCR.
Primer eksternal dan internal gC BHV-1
Nested PCR menggunakan primer gen gC ini berdasarkan prosedur ROS et al. (1999) yaitu primer gC BHV-1 eksternal: gC1 (5’GCC GAG AGM CCG CCR GMC CGA GA -3’), dan CR1 (5’-GGC CAC GCC GCA GTG CAC GTA CAG -3’), primer gC BHV-1 internal: gC1 (5’-GCC GAG AGM CCG CCR GMC CGA GA -3’) dan CR26 (5’-GCC TCG CGC TCG CAG AGC ATC CAC-3’). Nested PCR yang menggunakan primer eksternal dan internal menghasilkan fragmen 478bp.
Campuran reaksi PCR (PCR Mix) terdiri dari 5 μl 10x DNA polymerase buffer, 2 μl 10 mM campuran dNTP, 1 μl 5 mM masing-masing primer, dan 2,5 μl thermo-stable RED Taq™ DNA Polymerase, dan 5 μL DNA (10 pg/μl). Campuran reaksi tersebut kemudian ditambah dengan air deionisasi (dH2O) sehingga menjadi total volume 50μl. Proses amplifikasi DNA untuk primer eksternal yaitu : masing-masing siklus terdiri dari denaturasi 95°C selama 1 menit, pelekatan (annealing) primer pada suhu 65°C selama 1 menit dan elongasi pada suhu 72°C selama 1 menit. Total siklus 35 putaran, dan diakhiri dengan elongasi terakhir pada suhu 72°C selama 10 menit. Pada saat proses amplifikasi dengan primer internal, maka 1 μl produk PCR pertama diambil dan diamplifikasi dengan primer internal dengan proses amplifikasi DNA sama dengan yang digunakan untuk primer eksternal kecuali suhu pelekatan dinaikkan menjadi 67ºCdengan total siklus 35 putaran. Proses amplifikasi menggunakan GeneAmp®PCR Sistem 9700 (Applied Biosystem, ABI).
Analisis produk PCR
Produk PCR dianalisis dengan 2% agarose gel yang mengandung Ethidium Bromida (EtBr). Elektroporesis dilakukan pada voltage konstan yaitu pada 100 volt dalam TBE (Tris-Borate-EDTA) buffer selama 1 jam. Hasil PCR
dinyatakan positif apabila terlihat adanya produk yang spesifik dari primer yang digunakan.
Pemurnian DNA dan sekuensing
Sebanyak 50 μl masing-masing produk PCR yang diamplifikasi dari gen gC (478bp) dimurnikan dengan menggunakan QIAquick PCR Purification kit sesuai dengan prosedur dari pembuat kit.
Sekuensing (cycle sequencing method) mengikuti prosedur yang terdapat pada kit (BigDye Terminator v3.1 Cycle Sequencing Kit) yaitu: total reaksi 20 µl terdiri dari 1 µl
ready reaction premix, 3,5 µl BigDye sequencing buffer (5x), 2 µl primer internal gC
(forward dan reverse) masing-masing pada tabung terpisah (1,6 pmol/µl), 2 µl produk PCR, 0,5 µl Hot Start Taq Polymerase (5U/µl) dan 9 µl air deionisasi steril. Proses amplifikasi dilakukan sebagai berikut: Denaturasi awal suhu 95°C selama 5 menit, denaturasi suhu 95°C selama 30 detik, pelekatan suhu 50°C selama 10 menit dan ekstensi suhu 60°C selama 4 menit, dengan total siklus 25 putaran. Produk PCR dimurnikan sebagai berikut: 20 µl produk PCR dicampur dengan 2 µl EDTA 125 mM, 2 µl natrium asetat 3M dan 50 µl ethanol 100%, kemudian diinkubasikan pada suhu ruangan selama 15 menit dan disentrifugasi pada 3.000 x g selama 30 menit pada suhu 4°C. Supernatan dibuang, endapan (pelet) dicuci dengan penambahan 70 µl ethanol 70% kemudian disentrifugasi pada 1.650 x g selama 15 menit pada suhu 4°C. Akhirnya endapan dikering-udarakan dan dilarutkan denga 10 µl formamide dan dipanaskan pada suhu 95°C selama 2 menit, didinginkan secepatnya, divorteks, dan di sentrifugasi selama 2 detik. Sampel disimpan pada Es hingga siap digunakan. Sebanyak 0,5 µl sampel dipindahkan ke dalam tabung dan dimasukkan ke alat sekuenser (ABI PRISM 3130 Genetic
Analyser (Applied Biosystem, USA) melalui
kapiler elektroforesis yang mengandung polimer POP-6TM (Applied Biosystem, USA).
Analisis sekuens DNA dan filogenetik
Analisis sekuens DNA dan translasi asam amino dilakukan dengan menggunakan
software BioEdit Version 7.0.5 (HALL,1999).
Pensejajaran (alignment) sekuens menggunakan software the ClustalW version 1.83 (THOMPSON et al., 1994). Kesamaan (homologi) sekuens
antara isolat BHV-1 dianalisis dengan menggunakan The Basic Alignment Search
Tools (BLAST) dari The National Center for Biotechnolgy Information (NCBI). Sedangkan
analisis hubungan kekerabatan antar sekuens dilakukan dengan program Molecular
Evolutionary Genetic Analysis (MEGA)
version 3.1. (KUMAR et al., 2004) dimana
design konstruksi pohon kekerabatan menggunakan neighbor-joining (NJ) tree berdasarkan parameter Kimura-2 dengan 2000 replikasi.
HASIL DAN PEMBAHASAN
Hasil pemurnian produk PCR
Pemurnian terhadap produk PCR Gambar 1 dari ke-21 sampel yang terdiri dari 10 sampel isolat BHV-1 berasal dari Jawa Barat:
N0027/Jabar/06; N30560/Jabar/06;
N307185/Jabar/07; N50621T/Jabar/07, N30567/Jabar/06; V305172/Jabar/07; N101265/Jabar/08; N101266/Jabar/08; N101208/Jabar/08; N101229/Jabar/08; 6 sampel isolat BHV-1 berasal dari Jawa Tengah: (N304088/ Jateng/08; N304018/
Jateng/08; N303010/Jateng/08; N304132/Jateng/08; N308368/Jateng/08;
N308363/Jateng/08); dan 5 isolat BHV-1 berasal dari Jawa Timur: (SF1027485/Jatim/07; SF1029972/Jatim/07;
SF1022/Jatim/08; N1034/Jatim/08; N1036/Jatim/08); serta 2 sampel kontrol positif
IBR: BH1.1 galur Colorado dan BH1 V-155. Hasil pemurnian produk PCR menunjukkan bahwa pemurnian terhadap 21 isolat dan 2 kontrol positif diperoleh target yang spesifik pada 478bp tanpa adanya hibridisasi-silang (cross-hybridization) dengan target lain.
Gambar 1. Hasil pemurnian produk nPCR untuk bahan sekuensing
Lajur : 1) Kontrol positif BHV-1 galur Colorado; 2) Kontrol positif BHV-1 V-155; 3) sampai 10) isolat BHV-1 Indonesia; M) standar molekul 100bp
Sekuensing dan analisis sekuens
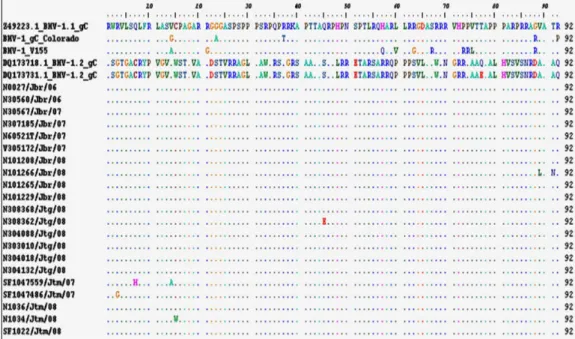
Berdasarkan hasil sekuensing yang dilanjutkan dengan analisis sekuens asam amino, Gambar 2 menunjukkan bahwa 16 isolat Indonesia (N0027; N30560; N307185; N60521T; N30567; N101208; N101229; N304088; N304018; N303010; N304132; N308368; SF1022; dan N1036 memiliki persamaan hingga 100% dengan BHV-1.1 gC (no. akses Z49223.1), sedangkan 6 isolat isolat Indonesia lainnya: V305172; N101266; N308362; SF1047486; SF107559; dan N1034 terdapat 1 sampai 2 asam amino yang berbeda dengan BHV-1.1. gC asal GeneBank (berbeda hanya 1,08 – 2,17%). Sehingga dengan demikian berdasarkan analisis sekuens asam amino ke-6 isolat Indonesia tersebut termasuk ke dalam kelompok BHV-1.1 gC. Hasil analisis sekuens antara isolat Indonesia dengan kedua BHV-1.2 gC asal GeneBank, memiliki perbedaan yang sangat mencolok hingga mencapai 84,9%
sampai 92,5% (persamaan 7,5 – 15,1%). Perbedaan asam amino antara
isolat Indonesia dengan BHV-1.2 gC asal
GeneBank sangat bervariasi. Perbedaan asam
amino yang sering terjadi (hingga 4 kali) adalah Arginina (R) Æ Serina (S) pada posisi asam amino ke-55; 63; 83; dan 85; sedangkan yang lainnya hanya 1 sampai 2 kali. Berdasarkan analisis sekuens nukleotida maupun asam amino, ke-21 isolat Indonesia termasuk ke dalam kelompok BHV-1.1 gC dan tidak termasuk ke dalam BHV-1.2 gC.
Kesamaan asam amino
Hasil kesamaan (homologi) sekuens asam amino antar isolat Indonesia yaitu mencapai 75,3 – 100%, lebih tinggi bila dibandingkan dengan kesamaan isolat Indonesia dengan BHV-1.1 gC asal GeneBank yaitu berkisar antara 63,4 – 100%. Sedangkan antara isolat Indonesia/BHV-1.1 gC dengan kedua BHV-1.2
gC memiliki kesamaan asam amino berkisar antara 7,5 – 15,1%. Hasil tersebut menunjukkan bahwa isolat Indonesia lebih dekat hubungannya dengan BHV-1.1 gC dibandingkan dengan BHV-1.2 gC.
Analisis filogenetik
Hasil analisis filogentik terhadap asam amino (Gambar 3) menunjukkan bahwa semua isolat Indonesia sangat berhubungan dekat dengan 1.1 rujukan (1.1 gC, BHV-1.1 Colorado dan BHV-1 V-155 Australia), sedangkan isolat Indonesia dengan BHV-1.2 rujukan (nomor akses DQ173718.1 dan DQ173731.1) sangat jauh hubungannya yang dibuktikan dengan tidak termasuk ke dalam kelompok yang sama (diluar kelompok BHV-1.1). Dengan demikian isolat BHV-1 Indonesia termasuk ke dalam subtipe BHV-1.1 (penyebab gangguan pernapasan/IBR), bukan subtipe BHV-1.2 (penyebab gangguan genital, IPB/IPV) dimana BHV-1.2 berada pada cluster tersendiri. Berdasarkan hasil analisis filogenetik terhadap asam amino menunjukkan bahwa semua isolat BHV-1 Indonesia tidak termasuk ke dalam subtipe BHV-1.2.
Virus herpes merupakan penyebab utama gangguan pernapasan, genital baik pada sapi jantan dan betina, aborsi, serta kematian. Akhir-akhir ini telah banyak dipelajari tentang kajian virologi molekuler pada kelompok ruminansia herpesvirus. Hal tersebut menjadikan disiplin ilmu virologi molekuler semakin maju dan terasa manfaatnya terutama dalam analisis genetik sekuens dan menjadikan dasar untuk mempelajari hubungan subtipe BHV-1.
Penelitian ini mengungkapkan karakteristik isolat BHV-1 Indonesia berdasarkan analisis sekuens nukleotida, deduksi sekuens asam amino dan analisis filogenetik terhadap BHV-1.1 gC dan BHV-1.2 gC. Dengan demikian Tabel 2. Kesamaan sekuen asam amino diantara BHV-1 gC yang dibandingkan
Sekuens asam amino yang dibandingkan Persentase Kesamaan
Antar isolat Indonesia 75,3 – 100
Isolat Indonesia vs BHV-1.1 Rujukan dan GeneBank 63,4 – 100 Isolat Indonesia vs BHV-1.2 GeneBank 7,5 – 15,1
Gambar 2. Hasil perbandingan sekuens asam amino antara isolat BHV-1 Indonesia dengan subtipe
BHV-1.1 dan BHV-1.2 gC rujukan.
Gambar 3. Analisis filogenetik terhadap asam amino pada fragmen gen gC BHV-1.
BHV-1 rujukan dari GeneBank BHV-1.1 gC (nomor akses Z49223.1), BHV-1.2. gC (nomor akses DQ173718.1 dan DQ173731.1). BHV-1 rujukan lain:BHV-1.1 gC Colorado (USA) dan BHV-1 V-155 (Australia).
akan diperoleh suatu perbandingan tipe genetik dari isolat Indonesia dengan virus BHV-1.1. dan BHV-1.2 rujukan yang terdapat di pangkalan data gen (GeneBank) yang hasilnya sangat bermanfaat untuk pengembangan diagnosis BHV-1 di Indonesia, pengembangan vaksin BHV-1, kajian epidemiologi, serta untuk meningkatkan pengendalian penyakit IBR di Indonesia.
Hasil kesamaan (homologi) sekuens asam amino antara isolat Indonesia/BHV-1.1 gC dengan kedua BHV-1.2 gC berkisar antara 7,5 – 15,1%. Dengan diperolehnya tingkat kesamaan asam amino antara isolat Indonesia dengan BHV-1.1 gC (63,4 – 100%) lebih tinggi dibandingkan dengan tingkat kesamaan asam amino antara isolat Indonesia dengan BHV-1.2 gC (7,5 – 15,1%) menunjukkan bahwa isolat Indonesia lebih cenderung masuk ke dalam subtipe BHV-1.1 gC daripada masuk dalam subtipe BHV-1.2 gC. Kesamaan asam amino antara isolat Indonesia dengan BHV-1.2 gC (7,5 – 15,1%) sangat bervariasi dan ini sangat berbeda dengan kesamaan asam amino antara isolat Indonesia dengan BHV-1.2 gD (95,1 – 96,3%) (SAEPULLOH et al., 2009). Hal tersebut menunjukkan bahwa untuk membedakan tingkat subtipe BHV-1, lebih cenderung menggunakan gen gC dibandingkan dengan menggunakan gen gD. Sebagaimana
dilaporkan oleh EL-KHOLY dan
ABDELRAHMAN (2006) bahwa gen gD adalah
conserved (memiliki posisi-posisi yang dipertahankan selama evolusi dalam sekuens protein) terhadap semua kelompok virus herpes, sehingga pemakaian gen gD sangat baik untuk digunakan dalam pembuatan vaksin sub unit dan sebagai bahan biologik untuk perangkat diagnostik. Glikoprotein D yang conserved terhadap kelompok herpesvirus menunjukkan bahwa posisi-posisi sekuens pada gD sangat penting bagi struktur atau fungsi protein tersebut. Lain halnya dengan gen gC yang memiliki variabilitas tinggi terhadap kelompok herpesvirus. Sebagaimana dilaporkan SPILKI et al. (2005) dan RIJSEWIJK et al. (1999) bahwa salah satu bagian gen gC
yang memiliki tingkat variabilitas tinggi disepanjang bagian ujung-N (N-terminal region) sehingga seringkali gen gC dijadikan sebagai target untuk membedakan subtipe virus herpes. Terjadinya perbedaan antara BHV-1.1 gC dengan BHV-1.2 gC menunjukkan adaptasi
glikoprotein terhadap perbedaan konformasi sel reseptor (ESTEVES et al., 2008).
Perbedaan sekuens asam amino antara isolat Indonesia dan BHV-1.2 gC rujukan menunjukkan adanya perbedaan struktur dan fungsi glikoprotein C itu sendiri yang pada gilirannya dapat berpengaruh terhadap patogenisitas virus tersebut. Perbedaan protein yang terkandung dalam gC akan mempengaruhi ikatan (binding) gC BHV-1 terhadap reseptor seluler spesifik dalam hal ini adalah heparan sulfate sugar moietis yang terdapat pada permukaan sel inang (LIANG et al., 1993; OKAZAKI et al., 1994; LI et al., 1995). Reseptor tersebut sebagai target potensial untuk pelekatan virus pada sel inang (SPEAR et al., 1992) dan memiliki kontribusi
yang sangat besar dalam perubahan organ/sel tropisme yang menjadi sasaran infeksi virus, sehingga dapat menyebabkan terjadinya perbedaan subtipe Bovine herpesvirus (CAMPADELLI-FUME et al., 2000; MUYLKENS et al., 2007). Hal inilah yang mungkin
menyebabkan BHV-1.1 dan BHV-1.2 dapat beradaptasi baik pada saluran pernapasan maupun genital. Akan tetapi BHV-1.1 diduga cenderung lebih mudah beradaptasi pada saluran pernapasan dibandingkan BHV-1.2.
Hasil analisis filogenetik terhadap asam amino (Gambar 3) menunjukkan bahwa semua isolat Indonesia sangat berhubungan dekat dengan 1.1 rujukan (1.1 gC, BHV-1.1 Colorado, dan BHV-1 V-155 Australia). Sedangkan isolat BHV-1 Indonesia dengan BHV-1.2 rujukan (BHV-1.2 gC) sangat jauh hubungannya yang dibuktikan dengan tidak termasuk ke dalam kelompok yang sama (di luar kelompok BHV-1.1). Analisis filogenetik tersebut mendukung hasil analisis kesamaan asam amino di atas. Dengan demikian isolat BHV-1 Indonesia termasuk ke dalam subtipe BHV-1.1 (penyebab gangguan pernapasan/ IBR), bukan subtipe BHV-1.2 (penyebab gangguan genital, IPB/IPV) dimana BHV-1.2 berada pada cluster tersendiri.
Bovine herpesvirus subtype 1.1 merupakan virus yang bersifat neurotropis yang dapat menetap pada trigeminal ganglion (CHOWDHURY et al, 2000). Sementara itu,
BHV-1.2 telah terbukti dapat menimbulkan gangguan pada saluran genital (VOGEL et al.,
2004). Akhir-akhir ini kedua subtipe virus tersebut (BHV-1.1 dan BHV-1.2) seringkali
berhubungan dengan penyakit penyebab gangguan saluran pernapasan (D’ACRE et al., 2002) dan berdasarkan hasil infeksi buatan pada hewan coba menunjukkan bahwa BHV-1.2 dapat menimbulkan penyakit gangguan pernapasan yang parah (SPILKI et al., 2004).
Oleh karena itu, untuk mengetahui kedua subtipe BHV tersebut tidak cukup hanya dengan melihat gejala klinisnya saja, akan tetapi perlu dilakukan karakterisasi molekuler untuk menentukan karakteristik kedua subtipe virus tersebut. Berdasarkan observasi di lapang, semua isolat virus yang dikarakterisasi, berasal dari ternak sapi yang menunjukkan gejala klinis gangguan pernapasan. Dengan demikian, terdapat kesesuaian antara gejala klinis pada ternak sapi dengan hasil karakterisasi molekuler, yaitu semua isolat virus yang diperoleh termasuk ke dalam subtipe BHV-1.1 sebagai agen penyebab penyakit gangguan saluran pernapasan/IBR.
Tidak termasuknya isolat asal sampel semen cair (Jawa Timur) dan usap mukosa vagina (Jawa Barat) ke dalam subtipe BHV-1.2 menunjukkan bahwa kemungkinan hewan tersebut memang terinfeksi oleh virus BHV-1.1 yang kemudian disekresikan dan bereplikasi pada mukosa genital. Menurut EDWARS et al.
(1991) bahwa pada hewan coba yang diinfeksi BHV-1.2 akan memperlihatkan klinis gangguan pernapasan dan akan mampu menyebarkan virus ke ternak sehat. Demikian pula sebaliknya bahwa BHV-1.1 seringkali berhasil diisolasi dari kasus gangguan pernapasan, aborsi dan lesi pada genital (ENGELS et al., 1992). Lebih lanjut, RIJSEWIJK et al. (1999) menyatakan bahwa sangat sulit
membedakan secara tepat sifat biologi dari kedua genotipe 1 (1.1 dan BHV-1.2), karena keduanya dapat menginfeksi baik pada saluran pernapasan maupun genital dan BHV-1.1 diduga lebih mudah beradaptasi pada saluran pernapasan dibandingkan BHV-1.2 (EDWARDS et al., 1991).
KESIMPULAN
Berdasarkan hasil karakterisasi molekuler melalui BHV-1 gC maka isolat yang berasal dari Jawa Barat, Jawa Tengah dan Jawa Timur termasuk ke dalam kelompok subtipe BHV-1.1 yaitu sebagai agen penyebab penyakit IBR dan
bukan termasuk ke dalam subtipe BHV-1.2 (penyebab gangguan genital). Oleh karena itu, kemungkinan besar penyebaran penyakit IBR yang ada di Jawa Barat, Jawa Tengah dan Jawa Timur disebabkan oleh BHV-1.1. Untuk mengetahui kajian epidemiologi molekuler virus lebih lanjut, diperlukan karakterisasi molekuler terhadap beberapa isolat virus yang berasal dari daerah lain di Indonesia
DAFTAR PUSTAKA
CAMPADELL-FUME,G.,F.COCCHI,L.MENOTTI and M. LOPEZ, 2000. The novel receptors that mediate the entry of herpes simplex viruses and animal alphaherpesviruse into cells. Rev. Med Virol 10: 305 – 319.
CHOWDHURY, S.I., B.J.LEE, A. OZKUL, and M.L. WEISS. 2000. Bovine Herpesvirus 5
glycoprotein E is important for neuroinvasiveness and neurovirulence in the olfactory pathway of the rabbit. J. Virol 74(5): 2094 – 2106.
D’ACRE,R.C. et al. 2002. Restriction endonuclease and monoclonal antibody analysis of brazillian isolates of Bovine Herpesvirus type 1 and 5. Vet Microbiol 88: 315 – 324.
DUBUISSON, J., B.A. ISRAEL and G. LETCHWORT. 1992. Mechanisms of Bovine Herpesvirus type 1 neutralization by monoclonal antinodies to glycoprotein gI, gIII, dan gIV. J. Gen Virol 73: 2031 – 2039.
EDWARDS, S.,R.H. NEWMAN and H. WHITE.1991. The virulence of british of bovid herpesvirus 1 in relationship to viral genotype. Br. Vet. J. 147: 216 – 231.
EL-KHOLY, A.A. and K.A. ABDELRAHMAN. 2006. Genetic characterization of the egyptian vaccinal strain Abu-Hammad of Bovine
Herpesvirus-1. Rev. Sci. Off. Int. Epiz. 25(3):
1081 – 1095.
ENGELS, M., M. PALATINI, A.E. METZLER, U. PROBST, U.KIHM andM. ACKERMAN. 1992. Interactions of bovine and caprine herpesviruses with the natural and foreign hosts. Vet. Microbiol 33: 69 – 78.
ESTEVES,P.A. et al., 2008. Phylogenetic comparison of the carboxy-terminal region of glycoprotein C (gC) of Bovine Herpesviruses (BoHV) 1.1, 1.2 and 5 from South America (SA). Virus Res. 131: 16 – 22.
FRANCO,A.C. et al., 2002. a Brazilian glikoprotein E-negative Bovine Herpesvirus type 1.2a (BHV-1.2a) Mutant is attenuated for cattle and induces protection against wild-type virus challenge. Pesq Vet Bras 22(4): 135 – 140. GIBBS,E.P.J. and M.M. RWEYEMAMU.1977. Bovine
Herpesviruses. part I. Bovine Herpesvirus-1.
Vet Bull 47(5): 317 – 343.
HALL,T.A. 1999. Bioedit: a User-friendly biological sequence alignment editor and analysis program for windows 95/98/NT. Nucleic acids symp. Ser. 41: 95 – 98.
JOHNSON,D.C.,M. WITTELS and P.G. SPEAR.1984. Binding to cells of virosomes containing herpes simplex virus tipe 1 glycoproteins and evidence for fusions. J. Virol 52: 238 – 247. KUMAR,S.,K.TAMURA and M. NEI,2004. MEGA3:
Integrated software for molecular evolutionary genetics analysis and sequence alignment. Brief Bioinform 5: 150 – 163.
LI, Y., VAN DRUNEN LITTLE- S.VAN DEN HURK, L.A. BABIUK and X. LIANG. 1995. Characterization of cell-binding properties of
Bovine Herpesvirus 1 glycoprotein B, C, and
D: Identification of a dual cell-binding function of gB. J. Virol 69: 4758 – 4768. LIANG,X.,M.TANG,B. MANNS,L.A. BABIUK and
T.J. ZAMB. 1993. Identification and deletion mutagenesis of the Bovine Herpesvirus-1 dUTPase gene and a gene homologous to herpes simplex virus U.L. 49.5. Virology 195: 42 – 50.
LIANG,X.P.,L.A.BABIUK,VAN DRUNKEN LITTEL-S. VAN DEN HURK , D.R.FITZPATRICK andT.J. ZAMB. 1991. Bovine Herpesvirus 1
Attachment to permissive cells is mediated by its major glycoprotein gI, gIII, and gIV. J Virol 65: 1124 – 1132.
MILLER, J.M. 1991. The effect of IBR virus infections on reproductive function of cattle. Vet. Med. 86: 95 – 98.
MILLER,J.M.,C.A. WHETSTONE,and M.J. VAN DER MAATEN. 1991. Abortifacient property of
Bovine Herpesvirus type 1 Isolates that
represent three subtypes determined by restriction endonuclease analysis of viral DNA. Am. J. Vet. Res. 52 (3): 458 – 461. MUYLKENS,B.,J.THIRY,P.KIRTEN,S.SCHYNTS,and
E. THIRY. 2007. Bovine Herpesvirus 1 Infection and infectious bovine Rhinotracheitis. Vet. Res. 38: 181 – 209.
OKAZAKI, K. et al., 1994. BHV-1 Adsorption is mediated by the interaction of glycoprotein
gIII with heparin-like moeties on the cells surface. Virology 181: 666 – 670.
OKAZAKI, K., WATANABE, M., HONDA, E. and KUMAGAI, T. 1991. Immune recognition pattern of bovid herpesvirus-1 infected rabbits against major glycoproteins. J. Vet. Med. Sci. 53:.137 – 139.
RIJSEWIJK,F.A. et al. 1999. Epitope on glycoprotein C of Bovine Herpesvirus-1 (BHV-1) that allow differentiation between BHV-1.1 and BHV-1.2 starins. J. Gen.Virol. 80(6): 1477 – 1483.
ROIZMAN,B. et al. 1992. The family herpesviridae: an up date. Arch. Virol 123: 425 – 448. ROS, C. and BELAK, S. 1999. Studies of genetic
relationships between bovine, caprine, cervine, and rangiferine alphaherpesviruses and improved molecular method for virus detection and infection. J. Clin. Microbiol: 1247 – 1253.
SAEPULLOH,M.,ADJID, R.M.A., WIBAWAN,I.W.T. danDARMINTO. 2008. Pengembangan nested PCR untuk deteksi Bovine Herpesvirus-1 (BHV-1) pada sediaan usap mukosa hidung dan semen asal sapi. JITV 13(2): 155 – 164. SAEPULLOH,M.,WIBAWAN,I.W.T.,SAJUTHI,D. dan
SETIYANINGSIH, S. 2009. Karakterisasi molekuler Bovine Herpesvirus type 1 isolat Indonesia. JITV 14(1): 66 – 74.
SPEAR,P.G.,SHIEH,M.T.,HEROLD,B.C.,WUDUNN, D., and KOSHY, T.I. 1992. Heparan sulfate glycosaminoglycan ss primary cell surface receptors for herpes simplex virus. Adv. Exp. Med. Biol. 313: 341 – 353.
SPILKI,E.R. et al., 2004. Comparative pathogenicity of Bovine Herpesvirus 1 (BHV-1) subtypes 1 (BHV-1.1) and 2a (BHV-1.2a). Pesq. Vet. Bras. 24(1): 43 – 49.
SPILKI, E.R. et al., 2005. A monoclonal antibody-based ELISA allows discrimination between responses induced by Bovine Herpesvirus subtypes 1 (BoHV-1.1) and 2 (BHV-1.2). J. Virol Meth. 129:.191 – 193.
THOMPSON, J.D., D.G. HIGGINS, and T.J. GIBSON. 1994. CLUSTAL W: Improving the sensitivity of progressive multiple squence alignment through squence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Res 22: 4673 – 4680.
TIKOO, S.K., CAMPOS and L.A. BABIUK, 1995.
Bovine Herpesvirus 1 (BHV-1): Biology,
pathogenesis and control. Adv. Virus Res. 45: 191 – 223.
VOGEL, F.S.F., E.F. FLORES, R. WEIBLEN, E.R. WINKELMANN,M.P. MORAES,and BRAGANÇA, J.F.M. 2004. Intrapreputial infection of young bulls with Bovine Herpesvirus type 1.2 (BHV-1.2): Acute balanopostitis, latent infection and detection of viral DNA in regional neural and non-neural tissues 50 days after experimental reactivation. Vet. Microbiol. 98: 185 – 196.
WYLER, R., M. ENGELS, and SCHWYZER. 1989.
Infectious bovine rhinotracheitis/vulvovaginitis (BHV-1). In:
WITTMANN G(ed.). Herpesvirus Disease of cattle, horses and pigs. Kluwer Academic publishers, Boston, Mass: 1 – 72.