B
IOLOGI
M
OLEKULER
iologi molekuler merupakan istilah umum yang mencakup rekayasa DNA dan RNA di laboratorium. Selain menyediakan perangkat penelitian canggih, teknologi molekuler telah diaplikasikan untuk memproduksi vaksin, diagnosis veteriner, membuat protein untuk pengobatan, mendeteksi pembawa gen yang berbahaya, dan untuk memperbaiki gen (terapi gen). Di masa depan, akan menjadi sangat sulit untuk menemukan bidang produksi dan kesehatan hewan yang secara substansial tidak dipengaruhi oleh teknologi ini. Dalam pembicaraan selanjutnya, kita akan menelaah keistimewaan yang paling penting tentang bidang yang sedang berkembang sangat cepat ini.
B
Enzim Restriksi
Pada tahun 1970, telah ditemukan bahwa bakteri menghasilkan enzim yang dapat mendegradasi DNA asing yang memasuki sel bakteri tersebut. Karena enzim ini memainkan peranan kunci dalam suatu fenomena yang diketahui sebagai restriksi induk semang (host restriction) (dimana bakteri melindungi dirinya sendiri dari virus dengan memotong DNA virus tersebut menjadi potongan kecil-kecil), mereka dinamakan enzim restriksi. Ratusan enzim restriksi sudah diketahui, dan metode standar untuk menamainya telah dibuat: huruf pertama dari nama genus, diikuti dengan dua huruf pertama dari nama spesies, diikuti dengan huruf yang menunjukkan galur/strain, urutan penemuan, dan seterusnya. Sebagai contoh, enzim BamHI diperoleh dari Bacillus amyloliquefaciens, dengan H menunjukkan galur tertentu, dan I menunjukkan bahwa itu merupakan enzim pertama yang diperoleh dari galur ini. Enzim restriksi mengikat sekuen basa spesifik yang disebut sekuen rekognisi, dan setiap enzim memotong DNA pada
situs pemotongan spesifik yang biasanya terletak dalam sekuen rekognisi tersebut. Dalam banyak kasus, sekuen rekognisi bersifat palindrom, yang, seperti kita lihat pada Bab 1, mengandung sekuen yang persis sama bila dibaca dari kedua arah. Daftar enzim restriksi yang umum digunakan, beserta sekuen rekognisi dan situs pemotongannya, disajikan pada Tabel 2.1. Perhatikan tabel untuk palindrom: sekuen pada rangkaian atas jika dibaca dari 5´ ke 3´ sama persis dengan sekuen pada rangkaian bawah jika dibaca dari 5´ke 3´.
Tabel 2.1. Beberapa enzim restriksi.
Sumber Enzim dan tempat pemotongan (Sekuen rekognisi, ↑↓)
Arthrobacter luteus AluI 5'...-A-G-C-T-...3' ↓ 3'...-T-C-G-A-...5' ↑
Bacillus amyloliquefaciens H BamHI
↓
5'...-G-G-A-T-C-C-...3' 3'...-C-C-T-A-G-G-...5' ↑
Escherichia coli R EcoRI
↓
5'...-G-A-A-T-T-C-...3' 3'...-C-T-T-A-A-G-...5' ↑
Haemophilus influenzae Rd HindIII
↓
5'...-A-A-G-C-T-T-...3' 3'...-T-T-C-G-A-A-...5' ↑
Haemophilus aegyptius HaeIII
↓ 5'...-G-G-C-C-...3' 3'...-C-C-G-G-...5' ↑ Providencia stuartii PstI ↓ 5'...-C-T-G-C-A-G-...3' 3'...-G-A-C-G-T-C-...5' ↑
Serratia marcescens SmaI
↓
5'...-C-C-C-G-G-G-...3' 3'...-G-G-G-C-C-C-...5' ↑
Beberapa enzim restriksi, seperti SmaI, memotong kedua rangkaian dari heliks ganda tersebut pada tempat yang sama, sehingga menghasilkan
ujung tumpul (blunt end). Beberapa lainnya, seperti EcoRI, memotong tiap rangkaian pada posisi berbeda, sehingga beberapa nukleotida tak berpasangan. Karena nukleotida komplemennya akan dengan mudah mengikat nukleotida yang tidak berpasangan tersebut, enzim seperti EcoRI dikatakan menghasilkan ujung perekat (sticky end).
Apabila enzim restriksi ditambahkan ke DNA, DNA tersebut dipotong pada banyak posisi sebanyak adanya situs pemotongan untuk enzim itu; DNA tersebut dipotong menjadi fragmen/potongan DNA. Karena situs pemotongan tidak terletak pada jarak yang teratur sepanjang molekul DNA, fragmen tersebut berbeda panjangnya dan oleh karena itu berbeda beratnya. Jika campuran fragmen tersebut ditempatkan pada salah satu bagian ujung gel dan dilakukan elektroforesis, fragmen tersebut bermigrasi sepanjang gel tersebut pada tingkat yang proporsional terbalik dengan logaritma beratnya. Dalam jangka waktu yang diberikan setelah aliran listrik pertama kali dinyalakan, terdapat banyak pita fragmen DNA yang berbeda di dalam gel sebanyak terdapatnya fragmen berbeda ukuran di dalam campuran asli tersebut di atas. Dan posisi dari setiap pita tersebut menunjukkan ukuran setiap fragmen.
Sebagai contoh, anggaplah molekul DNA yang relatif kecil seperti molekul DNA virus bakteri yang disebut bakteriofag λ (sering disingkat menjadi fag λ), yang DNA totalnya terdiri dari molekul tunggal sepanjang 49 kilobase (kb) (Gambar 2.1a). Jika DNA total dari virus tersebut didigesti dengan enzim EcoRI, dan jika fragmen yang dihasilkannya dilakukan elektroforesis dan diwarnai dengan ethidium bromide (yang mengikat DNA dan menimbulkan fluoresen di bawah sinar ultraviolet), foto seperti pada Gambar 2.1b terlihat. Setiap pita mewakili sebuah fragmen yang berbeda, yang ukurannya dalam kb disajikan di samping gel tersebut. Keberadaan enam fragmen menunjukkan bahwa terdapat lima situs pemotongan EcoRI pada DNA fag λ. Untuk menentukan urutan fragmen EcoRI tersebut, DNA tersebut harus didigesti secara terpisah dengan enzim itu dan dengan satu enzim lainnya, dan dengan campuran dari kedua enzim tersebut. Dengan membandingkan fragmen hasil dari setiap ketiga digesti tersebut, urutan fragmen untuk setiap enzim dapat ditentukan. Ini memungkinkan peta restriksi dapat digambar (Gambar 2.1c).
DNA Rekombinan, dan Kloning DNA
Satu dari proses-proses utama dalam biologi molekuler adalah memproduksi kopi segmen DNA tertentu dalam jumlah tak terbatas. Produksi masal dari suatu segmen DNA ini merupakan prasyarat yang diperlukan untuk penentuan sekuen basa segmen tersebut (yang memberikan petunjuk terhadap fungsinya) dan banyak teknik molekuler
lainnya. Itu dapat dicapai melalui dua cara. Pertama disebut kloning gen atau kloning DNA, dan ke dua dinamakan polymerase chain reaction (PCR). Kita akan mulai dengan kloning DNA; PCR akan dibahas pada bagian berikutnya.
Gambar 2.1. (a) Dua fag λ (atas) dan molekul DNA rangkaian ganda dari fag phage λ ke tiga. Dua ujung molekul DNA tersebut secara jelas dapat dilihat. (c) Peta restriksi EcoRI DNA dari fag λ. Situs-situs pemotongan ditunjukkan dengan tanda panah. Angka di bawah tiap-tiap tanda panah menunjukkan lokasi tiap situs dalam jumlah kb dari ujung kiri dari meolekul tersebut. Angka di atas garis menunjukkan ukuran tiap fragmen dalam kb, dan terkait dengan angka-angka tersebut dalam (b).
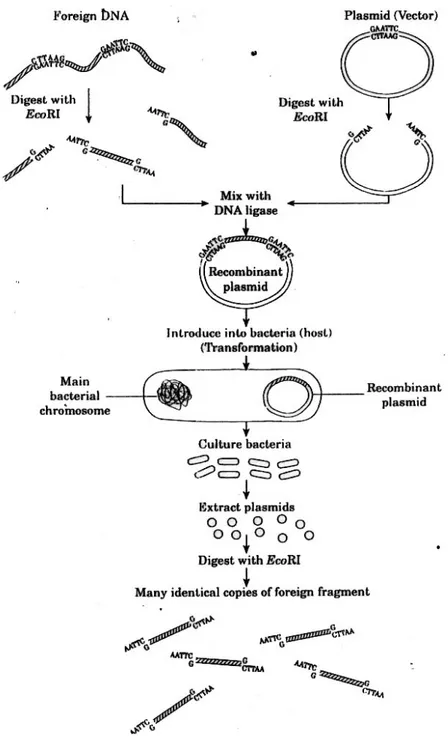
Kloning DNA dibuat dengan menggabungkan DNA yang akan diklon (disebut DNA sisipan atau DNA asing) ke vektor yang dapat mereplikasi di dalam inang. Kebutuhan awal untuk proses ini adalah bahwa DNA sisipan dan vektor masing-masing harus mempunyai ujung perekat yang sama (dihasilkan oleh enzim restriksi yang sesuai). Jika DNA sisipan dan vektor dicampur bersama dengan enzim DNA ligase (diterangkan pada Bab 1), ujung perekat dari DNA sisipan berikatan dengan ujung perekat dari DNA vektor, dalam suatu proses yang disebut penggabungan (splicing) atau ligasi (ligation). Molekul DNA yang dihasilkan merupakan satu contoh dari DNA recombinan. (Istilah ini berlaku untuk semua hasil dari penggabungan dua segmen DNA dari sumber yang berbeda.)
Satu tipe vektor yang umum digunakan adalah plasmid, yang merupakan DNA rangkaian ganda berbentuk bulatan, yang secara khas mempunyai panjang 1--3 kb, yang terdapat dalam sel bakteri tertentu (inang) dan terpisah dari kromosom utama bakteri. Plasmid sangat berguna untuk mengklon fragmen DNA yang relatif kecil--sampai sekitar 10 kb. Prosedur umum tentang kloning DNA dengan menggunakan plasmid disajikan pada Gambar 2.2.
Vektor lain yang populer adalah fag λ, yang telah kita singgung. Sampai sekitar 20 kb dari daerah bagian tengah molekul DNA yang panjangnya 49 kb dapat dihilangkan tanpa mempengaruhi kemampuan fag tersebut untuk menginfeksi sel bakteri. Jika bagian tengah ini diganti dengan sisipan yang kira-kira berukuran sama, molekul DNA rekombinan yang dihasilkan mempunyai ukuran total hampir sama dengan ukuran normal molekul DNA fag λ sehingga DNA rekombinan dapat dibungkus secara in vitro ke dalam partikel fafg yang kemudian langsung melakukan kebiasaan untuk menempel ke dinding sel bakteri dan mengeluarkan DNA-nya (dalam hal ini, DNA rekombinan) ke dalam sel bakteri (inang) tersebut. Pada saat berada di dalam inang, kedua ujung dari DNA fag bergabung bersama membentuk bulatan, yang mereplikasi di dalam inang. Biasanya, vektor fag dapat membawa sisipan yang berkuran sekitar 10--20 kb.
Fragmen yang lebih besar dapat diklon jika hanya kedua ujung DNA fag λ tersebut (dinamakan ujung cos karena mereka bersifat kohesif, yaitu mereka bergabung bersama di dalam inang) ditambahkan ke plasmid, yang membentuk vektor cosmid. Karena fag tersebut secara normal membungkus molekul DNA berukuran 49 kb dengan ujung-ujung cos-nya, dan karena panjang total ujung cos-nya plus plasmid tersebut hanya berukuran relatif kecil (sekitar 2 kb), cosmid dapat digunakan untuk mengklon fragmen berukuran antara 40--50 kb.
Fragmen DNA asing yang bahkan lebih besar lagi (dari beberapa ratus kb sampai 1 Mb) dapat diklon dalam fungi, dalam yeast artificial
chromosome(YAC), yang terdiri dari sisipan yang diapit oleh bagian-bagian penting dari kromosom fungi, yaitu kedua ujungnya (disebut telomer), sentromer, dan situs di, mana replikasi dimulai (dinamakan situs replikasi).
Dalam banyak kasus, tujuan kloning bisa saja untuk menghasilkan kopi segmen DNA tertentu dalam jumlah tak terbatas, untuk persiapan pelaksanaan sekuensing atau untuk beberapa prosedur molekul lainnya.
Akan tetapi ada penggunaan lain untuk kloning DNA. Sebagai contoh, jika semua DNA dari beberapa sel darah putih seekor sapi didigesti dengan satu enzim restriksi atau lebih, semua fragmen yang dihasilkan dapat diklon, yang menghasilkan pustaka genom (Gambar 2.3). Pada prinsipnya, pustaka semacam itu mengandung semua DNA untuk spesies tertentu itu. (Biasanya, beberapa segmen yang kecil bisa hilang.)
DNA genom (3 x 109 bp)
Partial digestion with restriction enzyme
Banyak fragmen
Fragmen diklon dalam plasmid (spt Gb. 2.2)
Pustaka Genom
Extrak mRNA dari jaringan tertentu
Tambah poly-T primer, reverse transcriptase, dan nucleotida, unt
membuat cDNA dari mRNA
Buang mRNA dg alkaline digestion
Tambah DNA polymerase untuk membuat benang baru, menggnk ujung
3’ benang pertama sbg primer
Buang loop dngn nuclease
Klon dlm plasmid (spt Gb. 2.2.) Pustaka cDNA 5’ AAAA 3’ 5’ AAAA 3’ 3’ TTTT 5’ TTTT 5’ 3’
Gambar 2.3. Konstruksi pustaka plasmid cDNA dan genom.
Satu kegunaan penting dari pustaka genom adalah untuk membuat satu rangkaian berurutan dari klon yang tumpang tindih (dinamakan contig, dari kata contiguous/berdampingan) yang secara bersama mencakup satu bagian tertentu dari satu kromosom atau bahkan seluruh kromosom atau seluruh genom. Seperti diterangkan pada Bab 11 dan 14, satu rangkaian contig seperti itu sangat bermanfaat untuk menelusuri gen tertentu, pada saat lokasi globalnya telah ditentukan melalui keterpautan dengan satu penciri DNA atau lebih.
DNA Komplementer
Ada sekelompok virus yang genomnya hanya terdiri dari RNA. Karena RNA tidak dapat mereplikasi, satu-satunya cara bagi virus ini untuk memperbanyak diri adalah mentranskripsi ‘balik’ RNA-nya menjadi DNA. Karena hal ini, mereka dinamakan retroviruses. Kemudian DNA tersebut mereplikasi dan bertindak sebagai tatakan/template untuk sintesis RNA lagi. Enzim yang melakukan transkripsi balik RNA menjadi DNA disebut reverse transcriptase. Enzim ini sangat penting dalam teknologi molekuler karena, di antara yang lain, seringkali jauh lebih mudah mengisolasi RNA daripada DNA untuk mendapatkan gen.
Suatu jaringan yang sedang menghasilkan polipeptida mengandung konsentrasi yang cukup tinggi mRNA matang bagi polipeptida itu. Jika mRNA diekstrak dari jaringan itu, dan jika nukleotida dan enzim reverse transcriptase tersebut ditambahkan ke mRNA, seuntai benang DNA komplementer (disebut DNA komplementer atau kopy DNA atau cDNA) dapat disintesis. Selanjutnya, dengan penambahan lebih banyak nukleotida dan enzim DNA polimerase, cDNA rangkaian tunggal dapat direplikasi menjadi cDNA rangkaian ganda.
cDNA ini dapat disamakan dengan gen yang aktif di dalam jaringan ketika mRNA diekstrak. Mengingat bahwa mRNA matang hanya terdiri dari exon, jelaslah bahwa cDNA ekuivalen dengan gen yang intron-nya telah dihilangkan.
Fragmen cDNA dapat diklon, yang menghasilkan pustaka cDNA (Gambar 2.3). Pustaka semacam itu mengandung kopi cDNA dari semua gen yang masih berfungsi di dalam jaringan ketika mRNA diekstrak. Akan tetapi, perlu dicatat bahwa pustaka genom mengandung semua DNA dari suatu spesies (termasuk exon dan intron dari semua gen berstruktur, plus semua DNA bukan-penyandi lainnya), sedangkan pustaka cDNA hanya
mengandung exon dari gen berstruktur yang masih sedang berfungsi (yaitu memproduksi protein) pada saat ketika jaringan diambil.
Sekuensing DNA
Satu dari terobosan utama dalam genetika molekuler adalah perkembangan metode mensekuen fragmen DNA secara cepat. Pada kenyataannya, sekuensing DNA telah menjadi kegiatan sehari-hari sehingga sekuensing seringkali merupakan satu dari langkah awal yang dikerjakan untuk mempelajari segmen DNA. Sekuensing juga digunakan untuk tujuan diagnosa.
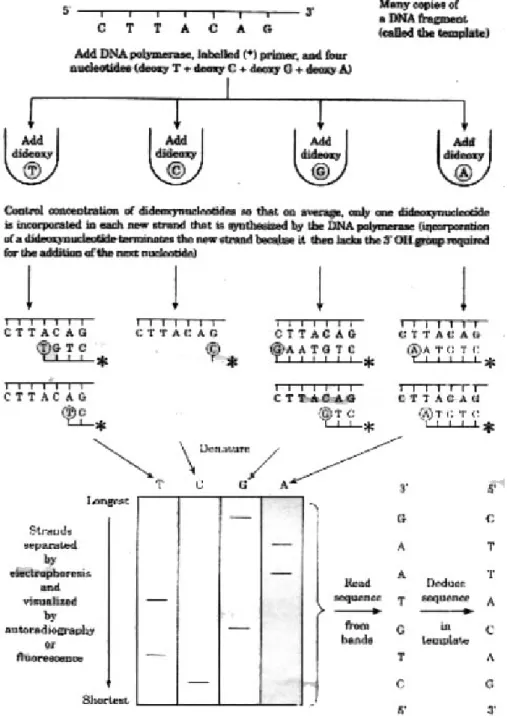
Ada dua metode: metode Sanger atau dideoxy atau chain-terminating, dan metode Maxam-Gilbert atau chemical, dengan metode yang pertama lebih umum digunakan. Tiap metode meliputi pembuatan serangkaian rangkaian tunggal berlabel dengan panjang bervariasi, yang dimulai dari salah satu ujung fragmen yang sedang disekuen. Elektroforesis dari rangkaian-rangkaian tersebut dalam gel poliakrilamid memisahkan rangkaian-rangkaian itu berdasarkan ukuran, yang menghasilkan ‘tangga pita' (ladder) berlabel, dengan tiap pita mewakili tersekuennya satu basa. Jika pelabelan bersifat radioaktif, gel tersebut kemudian dikeringkan dan dilekatkan pada film X-ray, yang mencatat keberadaan tiap pita pada autoradiograf yang dihasilkan. Semakin berkembang lagi, pelabelan fluoresen (yang diaktifkan dengan sinar laser) menggantikan radioaktif. Pada saat pita-pita tersebut telah divisualisasikan melalui kedua sistem pelabelan tersebut, sekuen basa pada fragmen dapat dibaca secara langsung dari tangga pita. Kedua metode itu diilustrasikan pada Gambar 2.4. Ukuran fragmen yang dapat disekuen berkisar 250 basa dengan metode Maxam-Gilbert dan antara 250 dan 350 dengan metode Sanger. Sekuen yang lebih panjang dihasilkan dengan menggabungkan sekuen yang diperoleh dari fragmen yang overlap.
Proses sekuensing secara keseluruhan telah berkembang menjadi otomatis, dan data sekuen terus dikumpulkan dengan kecepatan yang semakin meningkat. Database seperti Genbank (USA) dan EMBL Data Library (Eropa) telah didirikan yang secara spesifik mencatat data tersebut, dan akses on-line ke database itu tersedia di seluruh dunia. Sekarang banyak jurnal penelitian meminta agar informasi sekuen diserahkan ke salah satu dari database itu, sebagai prasyarat untuk publikasi. Dengan tiap genom mamalia yang mengandung sekitar 3 X 109 pasang basa (yaitu 3 juta kb atau 3.000 Mb), perkembangan yang cepat dari sekuensing menantang pakar-pakar informasi. Akan tetapi, banyak kemajuan telah dibuat, khususnya dalam hal pengembangan perangkat lunak yang canggih untuk mencari dan
menganalisis sekuen. Seluruh sekuen telah diketahui untuk beberapa mikroorganisme, dan untuk satu kromosom dari jamur (Saccharomyces cervisiae). Penelitian juga sedang berlangsung pada proyek genom manusia (human genome project), suatu proyek kerjasama besar dengan tujuan, di antaranya, mensekuen seluruh genom manusia.
Penting untuk dicatat bahwa tidak ada rencana untuk mensekuen seluruh genom hewan domestik, terutama karena telah disepakati bahwa dana terbatas yang tersedia untuk penelitian molekuler pada spesies ini lebih baik dihabiskan untuk mengkonsentrasikan pada gen yang menguntungkan atau yang nampak penting. Demikian juga, mengingat tingkat kesamaan yang tinggi antara genom mamalia (dibahas kemudian pada bab ini), kenyataan bahwa satu spesies mamalia (manusia) sedang disekuen, mengurangi makna memperoleh seluruh sekuen dari mamalia lainnya. Bila telah selesai, banyak penemuan yang muncul dari proyek genom manusia akan dapat diaplikasikan secara langsung ke hewan domestik.
Ada banyak aktivitas yang memerlukan informasi sekuen. Sebagai contoh, adalah mungkin untuk menelusuri data sekuen, untuk mencari open reading frame (diterangkan pada Bab 1), yang menunjukkan gen berstruktur. Oleh karenanya ini merupakan satu cara menemukan gen yang belum diketahui. Dalam situasi seperti itu, selanjutnya peneliti mempunyai sekuen gen tanpa mengetahui apapun tentang polypeptidanya. Dengan membandingkan sekuen dari gen yang baru ditemukan dengan sekuen dari gen yang fungsinya telah diketahui, seringkali mungkin untuk menentukan tipe dan fungsi dari polipeptida, jauh sebelum gen diisolasi. Misalnya, jika suatu gen yang baru ditemukan mengandung sekuen yang menyandi pembentukan zinc finger (diterangkan pada Bab 1), gen itu cenderung menyandi protein regulator.
Penggunaan penting lainnya dari data sekuen adalah dalam membandingkan sekuen dari gen yang sama pada spesies yang berbeda, yang memungkinkan pohon evolusionari dapat dibuat (lihat Bab 5).
Kepentingan yang sama adalah sekuensing genom parasit dan patogen. Pengetahuan yang didapatkan dari pekerjaan ini memberikan wawasan baru yang berharga bagi cara-cara yang mungkin dalam mengontrol organisme ini.
Polymerase chain reaction
(PCR)
Satu dari teknik yang paling ampuh dalam biologi molekuler adalah polymerase chain reaction (PCR). Dikembangkan pada tahun 1985 oleh Kary Mullis dan kawan-kawan, sekarang PCR digunakan secara ekstensif dalam penelitian. Lebih meningkat lagi, PCR juga diaplikasikan dalam diagnosis dan dalam deteksi gen tertentu (baik yang menguntungkan maupun yang membahayakan) pada ternak domestik. Di masa mendatang, PCR akan digunakan secara luas dalam banyak bidang praktek kodekteran hewan dan dalam program perbaikan ternak.
DNA tempat PCR dilakukan (DNA tatakan) dapat berupa DNA genom (diekstrak dari sel darah putih atau sampel limpa atau jaringan lain)
atau fragmen DNA dari berbagai sumber. PCR mencetak sekitar satu juta kopi segmen kecil (sampai 2 kb) dari DNA tatakan; cukup memungkinkan segmen itu diklon atau disekuen atau dianalisis dalam cara-cara lain yang dijelaskan di bawah ini. Yang diinginkan adalah untuk mengetahui sekuen dari segmen tersebut, atau, paling sedikit, sekuen pada setiap ujung dari segmen tersebut, yang ingin kita perbanyak. Dengan memanfaatkan informasi sekuen ini, dua rangkaian nukleotida (disebut oligonukleotida, atau disingkat oligo), masing-masing berukuran panjang sekitar 20 basa, disintesis: satu rangkaian berkomplementer dengan ujung 3’ dari satu rangkaian segmen yang akan diperbanyak; dan satunya lagi berkomplementer dengan ujung 3’ dari rangakain lainnya segmen tersebut. Karena peranan nukleotida ini adalah untuk memulai pembuatan benang baru DNA, mereka dinamakan primer.
Original, double-stranded DNA (Template) One million copies of fragment
Stop after 30-35 cycles
EXTENSION (700C) (New strands form) ANNEALING (50-600C)
(Primers bind) DENATURATION (700C)
(Strands separate)
Add nucleotides primers, and Taq
polymerase 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 5’ – 3’ 3’ – 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 3’ 5’ 3’
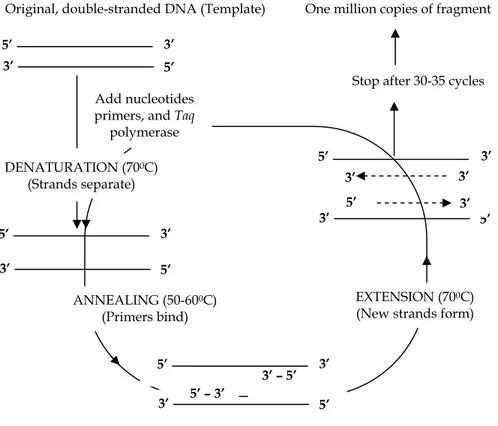
Proses penting dari PCR diilustrasikan pada Gambar 2.5. DNA tatakan ditempatkan dalam sebuah tabung, bersama dengan primer, sejumlah deoksinukleotida, dan DNA polimerase secukupnya. Campuran tersebut dipanaskan sampai sekitar 95°C untuk dapat memisahkan (denature) dua rangkaian DNA tatakan tersebut. (DNA polimerase harus stabil terhadap panas sehingga DNA polimerase tetap tahan terhadap temperatur tinggi pada fase denaturasi. Bentuk yang biasa digunakan adalah Taq polymerase, dinamakan demikian karena ini berasal dari bakteri termofilik Thermus aquaticus.) Temperatur tersebut kemudian diturunkan sampai sekitar 50--60°C, untuk memungkinkan primer mengikat (anneal) sekuen komplemennya pada DNA tatakan (yaitu pada kedua ujung dari segmen yang ingin kita gandakan). Pada saat primer telah berikatan, temperatur tersebut dinaikkan sampai sekitar 70°C, ini merupakan suhu optimum untuk DNA polimerase, yang menambahkan nukleotida yang sesuai pada ujung 3’ dari tiap primer, yang oleh karenanya memperpanjang (extending) rangkaian DNA baru. Setelah cukup waktu bagi benang baru untuk disintesis, temperatur tersebut dinaikkan lagi ke 95°C, yang kemudian memulai siklus baru yang terdiri dari denaturasi, pengikatan, dan perpanjangan. Lama setiap siklus (disebut siklus penggandaan/ amplification cycle) biasanya sekitar 5 menit, yang terdiri dari 15 detik untuk denaturasi, 30 detik untuk pengikatan, 90 detik untuk perpanjangan, plus waktu minimum yang dibutuhkan untuk merubah temperatur antar fase (30--60 detik untuk tiap perubahan).
Siklus penggandaan biasanya dilakukan antara 30 dan 35 kali di dalam mesin PCR (thermal cycler) yang dibuat sesuai pesanan, yang memungkinkan kontrol dapat diprogram secara tepat terhadap lama dan temperatur tiap tahap/fase dalam setiap siklus. Dalam teori, hasilnya antara 230 dan 235 kali jumlah kopi asli dari segmen-- setara dengan 1 miliar sampai 35 miliar segmen yang identik (dinamakan produk PCR). Dalam prakteknya, penggandaan secara eksponensial tidak sempurna. Akan tetapi, masih sangat mungkin memperoleh sedikitnya penggandaan 1 juta kali, misalnya mendapatkan 1 μg produk PCR.
Kisaran dan kekuatan PCR sungguh luar biasa. Sebagai contoh, produk PCR dapat diperoleh dari DNA dalam organisme yang masih hidup, dalam produk hewan seperti daging dan telur, atau dalam museum atau spesimen arkeologi; dan sampelnya bisa sekecil sperma tunggal atau folikel rambut tunggal. Produk PCR bahkan dapat diperoleh dari air susu, karena air susu mengandung sel somatik.
Sekarang banyak variasi mengenai pokok PCR telah digunakan. Misalnya, jika bagian tertentu terlalu besar untuk digandakan oleh hanya sepasang primer, beberapa pasang dapat dimasukkan dalam satu reaksi PCR
multipleks. Kebutuhan utamanya adalah bahwa primer dirancang sedemikian sehingga fragmen yang digandakan cukup berbeda ukurannya untuk dapat dibedakan dalam satu lajur dari gel. Dalam variasi yang lain, primer dengan sekuen acak dapat dibuat, yang menghasilkan random amplified polymorphic DNA atau RAPD. (Istilah ‘polimorfik’ menunjuk pada adanya lebih dari satu tipe atau bentuk. Dalam beberapa spesies, RAPD merupakan sumber penciri DNA acak yang bermanfaat untuk peta genetika atau (dalam hal parasit) untuk identifikasi spesies. Variasi lainnya mencakup transkripsi terbalik dari sampel RNA untuk menghasilkan cDNA, yang kemudian digandakan oleh PCR. Dikenal sebagai reverse transcription PCR (RT-PCR), ini merupakan metode canggih untuk mengidentifikasi dan mempelajari gen tanpa harus melalui proses kloning.
Aplikasi PCR yang penting adalah mengkombinasikannya dengan sekuensing DNA dideoksi, yang menghasilkan prosedur yang dinamakan cycle sequencing. Hanya satu primer digunakan, sehingga hanya satu rangkaian yang digandakan. Selain deoksinukleotida, campuran PCR juga mengandung sejumlah dideoksinukleotida, yang, seperti telah diterangkan pada Gambar 2.4, menghentikan perpanjangan dari rangkaian baru yang digabungi oleh dideoksinukleotida. Hasilnya adalah serangkaian produk PCR yang panjang masing-masing produk berbeda hanya satu basa (persis sama dengan produk yang dihasilkan pada sekuensing dideoksi), yang dapat dielektroforesis dan divisualisasikan sebagai pita-pita yang sekuennya dapat dibaca dari pita-pita tersebut, dengan cara seperti biasanya. Keuntungan cycle sequencing (dan kombinasi lain dari PCR dan sekuensing) adalah bahwa prosedur itu dapat dilakukan pada DNA dengan jumlah yang sangat kecil, dan tidak perlu mengklon DNA sebelum DNA tersebut disekuen.
Analisis
Southern
dan Teknologi Terkait
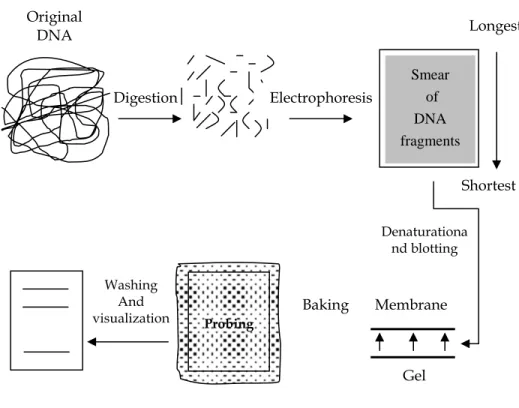
Teknik molekuler penting lainnya adalah analisis Southern, yang dinamakan demikian setelah Ed Southern mengembangkan komponen utamanya pada tahun 1975. Seperti pada PCR, kita mulai dengan DNA genom atau DNA dari beberapa sumber lain. Tahap pertama mencakup digesti DNA ini dengan enzim restriksi, yang menghasilkan ribuan fragmen dengan berbagai ukuran. Fragmen tersebut kemudian dipisahkan menurut ukurannya, dengan cara elektroforesis gel (seperti diterangkan pada Bab ini), didenaturasi menjadi rangkaian tunggal, dan ditransfer (di-blot) ke membran nitroselulose atau nilon, dengan cara kapilari (penyerapan) atau di bawah kondisi vakum. (Tahap akhir ini adalah Southern blotting.) Membran tersebut selanjutnya dipanggang di dalam oven pada suhu 80°C (agar
fragmen DNA benar-benar menempel), dan direndam dalam larutan yang mengandung DNA rangkaian tunggal berlabel. Perendaman semacam ini disebut penandaan (probing), dan DNA yang berlabel disebut probe, yang secara khas berasal dari fragmen DNA terklon, baik DNA genom atau cDNA. Dalam bentuk rangkaian tunggal, probe tersebut mengikat secara kuat ke fragmen DNA yang berkomplemen dengannya pada membran tersebut. Probe yang tidak menempel kemudian dibuang, dan fragmen yang diikat oleh probe tadi divisualisasikan dengan autoradiografi atau fluoresen (lihat Gambar 2.6).
Original DNA
Gambar 2.6. Analisis Southern.
Sejak itu tahap-tahap mendasar yang sama telah disadur ke berbagai kondisi: elektroforesis RNA yang diikuti probing dengan DNA disebut northern blotting; elektroforesis protein yang diikuti probing dengan antibodi
Denaturationa nd blotting Digestion Electrophoresis Smear of DNA fragments Longest Shortest Washing And
visualization Probing Baking Membrane
berlabel dinamakan western blotting; dan prosedur yang lebih rumit dimana protein di-probe dengan DNA dinamakan south-western blotting.
Deteksi Keragaman dalam Sekuen Basa
Teknologi molekuler dapat membedakan antara alel yang berbeda hanya sekecil substitusi basa tunggal. Untuk mengilustrasikan bagaimana deteksi ini dilakukan, kita akan menampilkan contoh praktis.
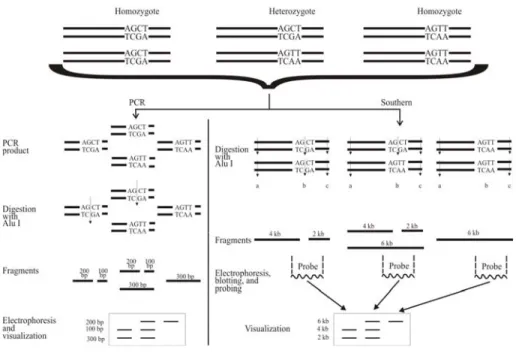
Dalam sekelompok sapi, seekor ternak bersifat homozigot untuk sekuen AGCT pada situs tertentu di dalam genom, ternak yang lain bersifat homozigot untuk sekuen AGTT pada situs yang sama, dan ternak ke tiga bersifat heterozigot pada situs ini (Gambar 2.7.). Dengan merujuk ke Tabel 2.1, anda akan melihat bahwa AGCT merupakan sekuen rekognisi untuk enzim restriksi AluI. Dalam kelompok sapi ini, semua ternak mempunyai sekuen rekognisi AluI lainnya yang posisinya 4 kb di satu sisi dari situs tadi, dan 2 kb di sisi lainnya.
Baik PCR maupun analisis Southern keduanya merupakan alat yang sangat ampuh untuk membedakan antara ketiga ternak tersebut.
Gambar 2.7. Deteksi restriction fragment length polymorphism (RFLP) dengan analisis PCR (kiri) dan (b) analisis Southern (kanan). Diagram tersebut tidak digambar berdasar skala.
Situs pemotongan untuk enzim AluI ditunjukkan dengan anak panah. Karena situs
a dan c sama pada semua ternak, sekuen nukleotidanya (AGCT) tidak ditampilkan. Untuk analisis Southern, pembaca dipersilakan untuk mencek bahwa jika probe tersebut hanya terkait dengan daerah antara a dan b, atau dengan daerah antara b dan c, di dalam kedua kasus tersebut tidak termasuk b itu sendiri, tiga ternak tersebut masih akan dapat dibedakan.
PCR
Sebagaimana disebutkan lebih awal, untuk menggunakan PCR perlu diketahui sekuen basa di sekitar empat basa tersebut. Dengan menggunakan informasi sekuen ini, sepasang primer disintesis sedemikian sehingga dapat menggandakan fragmen yang mencakup situs yang mengandung empat basa tersebut yaitu tempat perbedaan ternak tadi. Pada contoh ini, primer tersebut dipilih sehingga panjang fragmennya 300 pb dan situs tersebut terletak 100 basa dari salah satu ujung dari fragmen tersebut.
Setelah penggandaan, produk PCR dari tiap ternak didigesti dengan AluI. Semua produk PCR dari ternak pertama mengandung sekuen AGCT, dan oleh karenanya terpotong oleh enzim tersebut pada situs ini, yang menghasilkan fragmen berukuran 100 pb dan 200 pb. Ternak ke dua menghasilkan dua tipe produk PCR: tipe pertama digandakan dari kromosom yang mempunyai sekuen AGCT, tipe lainnya digandakan dari kromosom mempunyai sekuen AGTT. Produk tipe pertama terpotong oleh AluI, tetapi produk tipe ke dua tidak, karena AGTT bukan merupakan sekuen rekognisi untuk enzim itu. Digesti produk PCR dari ternak ke dua tersebut oleh karenanya menghasilkan fragmen berukuran 100 pb, 200 pb, dan 300 pb. Dengan alasan yang sama, tidak ada produk PCR dari ternak ke tiga terpotong, yang berarti bahwa ternak ke tiga hanya mempunyai fragmen berukuran 300 pb.
Setelah digesti, fragmen-fragmen tersebut dielektroforesis dan kemudian diwarnai di dalam gel yang mengandung ethidium bromide atau silver nitrat. Seperti ditunjukkan pada Gambar 2.7a, tiga ternak tersebut dapat dibedakan secara mudah.
Analisis Southern
Untuk analisis Southern, situasi yang paling sederhana adalah mempunyai probe yang mengandung situs dari empat nukleotida tersebut. (Tetapi analisis juga akan dapat dilakukan, jika probe tadi tidak menumpangi situs tersebut, dengan catatan satu ujung dari probe tersebut sangat dekat dengan situs tadi. Lihat tulisan di bawah Gambar 2.7.). Probe dalam contoh ini mempunyai panjang 3 kb, dengan situs terletak tepat di bagian tengah, yaitu 1,5 kb dari tiap ujungnya.
DNA genom diekstrak dari setiap ternak, didigesti dengan AluI, dipindahkan ke membran, dihibridisasi dengan probe, dan dilakukan
autoradiografi. Pada ternak pertama, dari semua fragmen berukuran beda yang dihasilkan akibat digesti dengan AluI, hanya fragmen yang dekat pada kedua sisi dari situs pemotongan mengandung bagian yang berkomplemen dengan probe tersebut. Karena dua situs AluI terdekat adalah 4 kb pada satu sisi dan 2 kb pada sisi lainnya, dua fragmen yang mengandung sekuen yang berkomplemen dengan probe tersebut berukuran 4 kb dan 2 kb. Semua fragmen lainnya tidak mempunyai sekuen yang sama dengan probe tersebut. Jadi, hanya fragmen berukuran 4 kb dan 2 kb yang dapat terdeteksi (Gambar 2.7b). Pada ternak ke dua, kromosom yang mengandung sekuen AGCT menghasilkan fragmen yang juga terdeteksi seperti yang diperoleh dari ternak pertama. Kromosom lainnya (yang mengandung sekuen AGTT) hanya menghasilkan satu fragmen berukuran 6 kb yang juga berhibridisasi dengan probe, karena AGTT tidak didigesti oleh AluI. Oleh karena itu ternak ini menunjukkan tiga pita. Dengan alasan yang sama, ternak ke tiga hanya menunjukkan satu pita berukuran 6 kb. Sekali lagi, ketiga ternak tersebut dapat dibedakan secara mudah.
Restriction fragment length polymorpism (RFLP)
Melalui analisis Southern dan PCR, perbedaan yang dapat dilihat pada pola pemitaan antar ternak muncul sebagai akibat dari adanya perbedaan terkecil di antara semua perbedaan genetika yang mungkin: dua alel hanya berbeda karena adanya satu substitusi basa tunggal. Ini juga mengilustrasikan kehebatan kedua teknik tersebut.
Perbedaan pola pemitaan antara ketiga ternak tersebut dinamakan restriction fragment length polymorphism atau RFLP. Mengingat bahwa kata ‘polimorfisme‘ berarti adanya lebih dari satu bentuk atau tipe, makna istilah tersebut sangat jelas: gel atau autoradiograf menunjukkan polimorfisme pada panjang fragmen DNA akibat dari digesti oleh enzim restriksi.
Walaupun contoh di atas mengenai substitusi basa tunggal, tipe lain perbedaan genetika dapat dideteksi seperti RFLP. Pada prinsipnya, semua mutasi yang menghilangkan atau menciptakan sekuen rekognisi bagi enzim restriksi dapat dideteksi sebagai RFLP. Misalnya, penyisipan (insertion), penghilangan (deletion) atau substitusi basa (base substitution) dalam sekuen rekognisi mengubah sekuen itu sehingga sekuen itu tidak dapat lagi dikenali oleh enzim restriksi. Sekuen rekognisi baru dapat diciptakan oleh substitusi basa, oleh penyisipan, atau (pada kasus langka) oleh penghilangan. Juga, penghilangan atau penyisipan antar sekuen rekognisi mengubah panjang fragmen antara dua sekuen rekognisi tersebut, dan oleh karena itu menimbulkan RFLP. Pada prakteknya, ukuran fragmen yang dapat dideteksi oleh analisis Southern biasanya berkisar antara 300 pb sampai 15
kb, dengan perbedaan ukuran sekecil 50 pb dapat terdeteksi, yang tergantung pada tipe gel dan kondisi elektroforesis yang digunakan. Kisaran untuk produk PCR biasanya 60 pb--2 kb, dengan perbedaan beberapa basa dapat terdeteksi, yang lagi-lagi tergantung pada kondisi yang digunakan.
Bagaimana dengan substitusi basa yang tidak menghilangkan atau tidak juga menciptakan sekuen rekognisi? Untungnya, ada metode untuk mendeteksi perbedaannya juga. Pada kenyataanya, ada metode yang, pada prinsipnya, dapat mendeteksi semua perbedaan pada sekuen DNA. Ini semua melibatkan PCR sebagai langkah awal dalam mengamplifikasi fragmen DNA yang diinginkan.
Metode-metode lainnya dan cycle sequencing
Dalam satu metode, produk PCR didenaturasi menjadi rangkaian tunggal, yang kemudian dielektroforesis dalam gel. Rangkaian yang berbeda hanya sekecil substitusi basa tunggal mengakibatkan bentuk yang berbeda (dan oleh karena itu migrasi pada kecepatan berbeda) ketika bergerak melalui gel tersebut. Ketika elektroforesis dihentikan, setiap rangkaian yang berbeda telah bergerak menempuh jarak yang berbeda, dan dapat divisualisasikan sebagai pita yang berbeda setelah penambahan pewarna DNA ke gel tersebut. Polimorfisme yang ditunjukkan oleh teknik ini disebut single-stranded conformational polymorphismatau SSCP.
Dalam metode lain, produk PCR rangakaian ganda dielektroforesis dalam gel, melalui gradien dari beberapa faktor yang dapat mendenaturasi DNA rangkaian ganda. (Oleh karena itu, metode ini dinamakan denaturation gradient gel electrophoresis atau DGGE.) Satu di antara bentuk yang paling praktis dari gradien pendenaturasi adalah temperatur, yang melahirkan istilah temperature gradient gel electrophoresis atau TGGE. Segera setelah rangkaian ganda tersebut mulai memisah, mereka tertahan di dalam gel. Dua fragmen DNA rangkaian ganda yang berbeda hanya sekecil substitusi basa tunggal mulai memisah pada temperatur yang berbeda, dan oleh karenanya menimbulkan pita berbeda yang dapat mudah dilihat setelah pewarnaan.
Variasi pada Southern blotting adalah dot blot. Ada berbagai tipe dot blot, tetapi satu di antara bentuk yang paling hebat adalah ketika PCR digunakan untuk mengamplifikasi segmen DNA setiap ternak dari suatu kelompok ternak atau patogen. Segmen tersebut dipilih sedemikian sehingga memasukkan satu atau lebih situs yang keragaman alelnya diketahui akan ada. Segmen yang teramplifikasi dari setiap ternak ditempatkan sebagai titik (dot) kecil di atas kertas membran, yang selanjutnya ditandai dengan oligonukleotida sintetis berlabel yang sekuennya sesuai dengan alel tertentu pada lokus tersebut. Suatu hal yang mungkin untuk mengatur kondisi sehingga probe berlabel tersebut akan menempel (hibridisasi) hanya jika
sekuennya persis sama dengan segmen yang teramplifikasi tersebut. Dengan membuat beberapa membran duplikat, masing-masing dengan titik (dot) yang sama (dari sekelompok ternak atau patogen yang sama), dan menandai tiap membran dengan oligonukleotida berlabel yang berbeda yang sesuai dengan alel berlainan, suatu hal yang mungkin untuk menentukan alel mana yang terdapat pada lokus itu pada tiap ternak atau patogen. Oligonukleotida yang digunakan dalam tipe analisis semacam ini disebut allele-specific oligo atau ASO. Jika ada banyak ASO, seringkali prosedurnya dibalik: semua ASO dititikkan (di-dot) ke sebuah membran, yang kemudian diskrining dengan DNA teramplifikasi berlabel dari seekor ternak atau patogen. Ini dinamakan reverse dot blot. Ada variasi lain yaitu merancang primer yang khas untuk setiap alel yang diketahui pada suatu lokus, dan kemudian melakukan PCR di bawah kondisi tertentu sehingga amplifikasi dari alel tertentu dapat terjadi hanya jika ada primer yang benar-benar sesuai. Ini disebut sebagai allele-specific amplification(ASA).
Dalam perkembangannya, banyak variasi tersebut diganti oleh cycle sequencing otomatis, yang dapat mendeteksi semua keragaman basa pada suatu fragmen teramplifikasi, yaitu alel diidentifikasi menurut sekuen basanya. Jika label fluoresen yang berlainan digunakan untuk setiap dari empat reaksi sekuensing (A, C, G, T), produk dari keempat reaksi tersebut dapat ditempatkan dalam satu lajur tunggal.
Diagnosis Veteriner
Teknik-teknik yang dijelaskan pada bagian sebelumnya menciptakan revolusi dalam diagnosis veteriner (kehewanan): uji diagnosis yang kurang tepat dan berlarut-larut diganti oleh uji DNA yang jauh lebih cepat dan lebih sensitif. Kebanyakan uji ini mengandalkan PCR. Sampel DNA dapat berasal dari ternak hidup (jaringan padat, darah, swab, sperma, feses), dari produk ternak (telur, susu, daging), dari jaringan post-mortem (baik produk segar maupun produk lama), atau dari air yang dicurigai terkontaminasi. Dengan primer dan/atau probe yang sesuai, uji tersebut dapat mengidentifikasi adanya spesies/galur/grup serum dari virus, retroverus, bakteri, jamur, protozoa, roundworm, atau tapeworm. Mereka juga dapat digunakan untuk membedakan antara daging dari spesies ternak yang berbeda. Uji yang paling sederhana melibatkan amplifikasi segmen DNA tertentu, diikuti dengan elektroforesis dan pewarnaan gel, yang memunculkan suatu pita diagnostik. Untuk virus RNA, langkah awal dalam transkripsi terbalik (reverse transcription) dilakukan terlebih dahulu sebelum PCR.
Kespesifikan uji-uji tersebut tergantung pada daerah yang diamplifikasi (yaitu tergantung pada pilihan primer): semakin kurang terjaga
daerah tersebut, semakin spesifik ujinya. Semua kemungkinan telah dilakukan, dari primer yang mengamplifikasi fragmen yang ditemukan hanya dalam satu galur atau grup-serum (serogroup), sampai ke primer yang mengamplifikasi fragmen yang ada hampir pada beberapa genera. Pada kasus yang terakhir, digesti produk PCR dengan enzim restriksi sering menghasilkan pola pemitaan yang bersifat diagnostik terhadap satu spesies atau galur atau grup-serum. Alternatifnya, produk PCR yang tidak didigesti dapat dititikkan (di-dot) dengan oligonukleotida yang bersifat diagnostik terhadap spesies, galur atau grup-serum. Kemungkinan tersebut tidak terbatas, dan kekuatan dari seluruh pendekatan tersebut meyakinkan bahwa PCR akan menjadi alat diagnostik yang sangat penting selama beberapa tahun ke depan.
Variable number of tandem repeat (
VNTR), sidik jari DNA,
dan mikrosatelit
Walaupun substitusi, penghilangan, dan penyisipan merupakan sumber perbedaan genetika yang sangat penting antar alel maupun antar ternak, mereka bukan satu-satunya tipe perbedaan genetika. Kategori penting lainnya melibatkan keragaman jumlah ulangan tandem dalam DNA-berulang (repetitive DNA). Sebelum mendiskusikan bagaimana DNA-berulang dapat dideteksi dengan teknik molekuler, terlebih dahulu kita harus menjabarkan informasi yang diberikan pada Bab 1.
DNA-berulang
Ada dua tipe DNA-berulang: berulang-menyebar (dispersed) dan berulang-berurutan (tandemly repeated).
DNA berulang-menyebar diklasifikasikan menurut ukuran unit ulangan: unit ulangan yang lebih panjang disebut long interspersed element (LINE), yang biasanya berukuran panjang lebih dari 1.000 basa. Ukuran yang lebih pendek disebut short interspersed element (SINE), yang biasanya berukuran lebih pendek dari 500 basa. Biasanya, LINE ditemukan sekitar 10.000 kali di dalam genom, sedangkan SINE ditemukan sekitar 100.000 kali. Secara keseluruhan, LINE dan SINE menempati sekitar 20% dari total DNA mamalia. Pada prinsipnya, LINE merupakan kopi cDNA dari gen fungsional yang terdapat di dalam genom yang sama. Dikenal sebagai processed pseudogene, segmen DNA non-fungsional ini kemungkinan besar muncul dari aksi yang tak terencana dari enzim reverse transcriptase selama infeksi retrovirus. Menariknya, beberapa LINE tampaknya juga mempunyai turunan open reading frame untuk enzim reverse transcriptase tersebut, yang
mengindikasikan bahwa pada suatu saat di masa lampau, mereka mempunyai kapasitas untuk melakukan transposisi sendiri, yaitu mereka merupakan elemen genetika yang dapat dipindahkan tempatnya (transposisi) atau transposable genetic element (TGE--diterangkan pada Bab 1). Ini dapat membantu menerangkan kejadian adanya banyak kopi tersebut. Kebanyakan SINE juga tampak berasal dari aksi tak terencana dari enzim reverse transcriptase, tetapi dalam kasus ini, DNA tersebut merupakan kopi dari molekul tRNA tertentu. Sebagai contoh, sebuah SINE yang khas adalah sekuen sepanjang 73 pb, yang terdapat pada sapi dan kambing, dan yang merupakan kopi DNA dari tRNA untuk glisin.
Pada DNA berulang-berurutan, unit ulangan terjadi secara bersambungan. Jika ada banyak kopi (biasanya ribuan) dari unit ulangan tersebut secara berurutan pada satu situs, DNA tersebut dinamakan DNA satelit. Ukuran dari unit ulangan dalam DNA satelit biasanya berkisar dari 5 sampai 500 pb, dengan jumlah total unit ulangan pada setiap situs berkisar antara 1.000 dan 50.000. Dalam beberapa kasus, unit ulangan yang lebih panjang mencakup banyak ulangan tak sempurna dari unit ulangan yang lebih pendek. Satu contoh dari DNA satelit adalah unit ulangan sepanjang 23 pb yang terdapat pada beberapa lokasi di dalam genom sapi, dengan jumlah total kira-kira 7.000.000 kopi. Contoh lainnya adalah unit ulangan sepanjang 483 pb pada kucing, yang mencakup sekitar 25 kopi tidak sempurna (yaitu sedikit beragam) dari unit ulangan sepanjang 6 pb (TAACCC), dan yang membentuk 1—2% dari genom kucing. Unit ulangan yang sedikit lebih panjang dan mirip seperti pada kucing ditemukan pada anjing.
Karena ukuran dan jumlah ulangan berurutan pada suatu situs mengecil, istilah minisatelit (minisatellite) dan mikrosatelit (microsatellite) digunakan untuk menerangkannya.
Unit ulangan minisatelit biasanya berukuran antara 10 sampai 100 basa. Mereka lebih menyebar di dalam genom daripada DNA satelit, tetapi penyebarannya cenderung masih terkonsentrasi pada daerah tertentu seperti telomer dan situs-situs yang mengalami frekuensi rekombinasi sangat tinggi (recombination hotspot). Bahkan, dipikirkan bahwa DNA minisatelit mungkin sebenarnya terlibar dalam proses dimulainya rekombinasi.
Istilah mikrosatelit mengikuti unit ulangan terkecil (sekitar lima basa atau kurang, misalnya ACCGG, ATTT, GGC, AC, T, dan lain-lain), dengan jumlah unit ulangan pada suatu situs biasanya bervariasi dari beberapa sampai 30 kali. Mikrosatelit terdapat menyebar di dalam genom. Bahkan, ada kecenderungan bahwa hampir setiap gen sedikitnya mempunyai satu mikrosatelit, yang terletak di dalam intron atau di dalam daerah 5' atau 3' yang mengapit sekuen pengkode. Sebuah contoh khas mikrosatelit adalah unit ulangan AC, yang terdapat pada sekitar 100.000 situs yang berbeda di dalam genom mamalia.
VNTR, dan sidik jari DNA
Karakteristik utama DNA-berulang adalah bahwa ada keragaman tinggi dalam jumlah unit ulangan pada suatu situs: satu situs pada satu kromosom mungkin mempunyai, katakan, 24 kopi ulangan tertentu, sedangkan pada situs yang sama pada kromosom homolognya di dalam ternak yang sama, mungkin ada 21 kopi. Di dalam ternak lain, mungkin ada 23 kopi pada situs yang sama pada satu kromosom, dan 15 kopi pada kromosom homolognya. Dengan beberapa pengecualian penting dan dapat dicatat, seperti didiskusikan pada Bab 3, keragaman dalam jumlah unit ulangan ini tampaknya tidak mempunyai efek yang membahayakan.
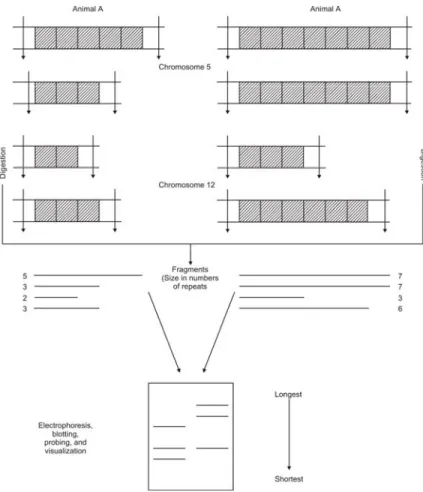
Variable number of tandemrepeat (disingkat menjadi to VNTR) dapat dideteksi melalui analisis Southern, dengan memanfaatkan enzim restriksi yang tidak memotong unit ulangan tersebut, dan di-probe dengan satu set tandem unit ulangan (Gambar 2.8). Ukuran unit ulangan yang paling cocok untuk tipe analisis seperti itu adalah sekitar 10--20 basa seperti dalam minisatelit.
Gambar 2.8. Deteksi variable number of tandem repeats (VNTR) pada dua ternak, menggunakan analisis Southern. Tanda panah menunjukkan situs-situs pemotongan untuk enzim restriksi yang tidak memotong sekuen ulangan tersebut. Pada contoh ini, sekuen ulangan tersebut terdapat pada empat situs berbeda (loci) di dalam genom. Dengan pengecualian pada lokus di bagian atas pada ternak B, tiap ternak bersifat heterosigot untuk jumlah unit ulangan (ditampilkan di sepanjang kromosom) pada tiap lokus. Pola pita tersebut dinamakan sidik jari DNA. Dalam praktek, biasanya selalu ada lebih dari empat lokus, dan oleh karenanya lebih banyak pita ditunjukkan dalam contoh ini.
Microsatellite
Seperti kita lihat di atas, mikrosatelit mencakup unit ulangan tandem dengan kisaran ukuran terkecil, biasanya dari satu sampai lima basa. Mereka dideteksi dengan menggunakan PCR, dengan primer yang sesuai dengan DNA sekuen khas yang mengapit ulangan tandem tersebut (Gambar 2.9). Juga, elektroforesis harus dilakukan dalam kondisi yang memungkinkan pita dapat dibedakan yang antara pita satu dan lainnya hanya berbeda satu
basa saja. Karena kondisi elektroforesis yang digunakan untuk merunut DNA memenuhi persyaratan ini, produk PCR mikrosatelit dielektroforesis di dalam gel perunutan. Kenyataannya memang prosedur secara keseluruhan sama dengan prosedur untuk merunut DNA: primer berlabel digunakan, dan pita-pitanya divisualisasi baik dengan auto-radiografi atau fluoresen. Seperti dengan perunutan, deteksi mikrosatelit menjadi otomatis.
Gambar 2.9. Deteksi mikrosatelit pada dua ternak, menggunakan sepasang primer, yaitu pada situs yang ditandai dengan sekuen (sequence-tagged site). Unit ulangan tersebut adalah AC. Produk PCR diidentifikasi menurut ukuran fragmen.
Sebagaimana namanya, DNA sekuen khas yang sesuai dengan primer hanya terdapat di satu tempat saja dalam genom total. Ini berarti bahwa walaupun unit ulangan mikrosatelit tertentu terdapat pada banyak situs yang berbeda di dalam genom, PCR hanya akan mengamplifikasi satu tempat saja, yaitu tempat yang sekuen pengapitnya sesuai dengan sekuen primer yang digunakan. Situs semacam itu dinamakan sequence-tagged site atau STS. Panjang produk PCR bervariasi menurut jumlah unit ulangan pada situs tersebut.
Sama halnya dengan produk PCR yang diamplifikasi dari primer bersekuen unik (khas), hanya satu atau dua pita dihasilkan dari seekor ternak; satu pita jika ternak tersebut homozigot, dan dua pita jika itu heterozigot. Ini berarti bahwa produk PCR mikrosatelit tidak menghasilkan pita dalam jumlah banyak per ternak yang merupakan karateristik sidik jari DNA. Seperti kita lihat di bawah, produk PCR mikrosatelit sangat bermanfaat dalam pemetaan gen, karena tiap pasang primer hanya sesuai dengan satu situs tertentu pada kromosom, dan karena ada banyak alel berbeda pada tiap situs, dengan kata lain mikrosatelit bersifat sangat polimorfik.
Pemetaan Gen
Barangkali terdapat antara 50.000 dan 100.000 gen dalam setiap spesies mamalia dan burung. Oleh karena itu, kita ingin dapat mengidentifikasi setiap dari gen-gen tersebut, menentukan fungsinya, dan mendokumentasi keragaman (variasi) alelnya. Tahap pertama meniti perjalanan penemuan ini adalah membuat peta gen untuk setiap spesies.
Ada dua macam peta genetika. Pertama adalah peta keterpautan (linkage map), yang terdiri atas daftar gen terpaut (linkage group) yang disusun secara linier menurut fraksi rekombinasi di antara gen-gen tersebut. Ke dua adalah peta fisik (physical map), yang menunjukkan lokasi tiap gen pada kromosomnya.
Kedua macam peta genetika telah lama ada dalam bentuk yang sangat tidak lengkap untuk beberapa spesies domestik, dan dalam bentuk yang sangat nyata untuk ayam. Hingga belum lama ini, pengembangan peta itu terhambat oleh rendahnya derajat polimorfisme yang dapat dideteksi pada kebanyakan lokus melalui cara-cara konvensional seperti penentuan sifat berdasarkan penglihatan mata atau berdasarkan uji biokimia. Semua ini telah berubah dengan perkembangan dan penggunaan secara luas teknologi molekuler, dan khususnya, dengan mikrosatelit, yang, di antara lainnya, memungkinkan pendeteksian polimorfisme DNA pada ribuan lokasi lokus di seluruh genom.
Pembuatan peta keterpautan (linkage map) melibatkan pendugaan fraksi rekombinasi antara semua kombinasi pasangan yang mungkin dari sebanyak lokus yang dapat diidentifikasi. Satu cara umum melakukan ini adalah menyilangkan dua populasi yang sedapat mungkin berbeda secara genetis (untuk membuat generasi F1 atau persilangan pertama). Generasi F1 ini kemudian dikawinkan dengan F1 lainnya, untuk menghasilkan generasi F2, atau dengan anggota dari satu di antara dua populasi tetuanya, yang menghasilkan generasi silang-balik (backcross). Alasan memilih populasi
tetua yang sedapat mungkin berbeda adalah untuk memaksimalkan peluang generasi tetua mempunyai alel yang berbeda pada tiap lokus. Dengan demikian ini akan memaksimalkan peluang generasi F1 bersifat heterozigot pada tiap lokus, yang akibatnya memaksimalkan peluang untuk dapat menduga fraksi rekombinasi untuk tiap pasang lokus. Sebagai indikasi mengenai bagaimana pendugaan semacam itu dapat diperoleh, perkawinan antara seekor ternak F1 dan seekor ternak dari salah satu populasi tetua sama dengan perkawinan yang ditunjukkan pada Gambar 1.12.
Jika fraksi rekombinasi diduga dari frekuensi genotipe pada keturunan dari satu perkawinan yang diketahui, kita benar-benar dapat menentukan frekuensi gamet yang dihasilkan oleh satu atau kedua tetua tersebut. Pada dasarnya, kita mencoba menduga proporsi gamet hasil dari rekombinasi, tetapi kita harus melakukannya secara tidak langsung, dengan melihat genotip keturunan. Belum lama ini, satu cara pemetaan keterpautan telah dikembangkan, yang kita dapat melihat gamet secara langsung, dan oleh karenanya memperoleh pendugaan langsung frekuensi rekombinasi. Diketahui sebagai single-sperm typing, metode tersebut melibatkan penyortiran sel sperma dengan cara penyortiran sel yang diaktifkan fluorescence activated cell sorting (FACS), yang menyebabkan satu sel sperma dapat ditempatkan dalam tiap sumur dari microtitre plate. Tiap sel sperma kemudian dilisis untuk mengekspos DNA-nya, dan dilakukan PCR untuk tiap lokus yang diinginkan. Identitas alel pada tiap lokus dalam tiap sperma kemudian ditentukan melalui analisa produk PCR secara tepat. Tidak seperti analisis keterpautan tradisional yang memerlukan penentuan genotipe tetua dan keturunan dari hasil perkawinan tertentu, penentuan genotipe melalui sperma tunggal tidak memerlukan perkawinan; semua yang diperlukan adalah sampel sel sperma dari seekor ternak jantan yang bersifat heterozigot pada lokus yang diinginkan.
Ketika semua metode pendeteksian polimorfisme DNA telah dicoba dalam pemetaan gen, deteksi mikrosatelit melalui PCR membuktikan yang paling efektif dan oleh karenanya metode paling populer, baik dalam gen struktural maupun pada sembarang situs di dalam genom. Ratusan penciri mikrosatelit telah dipetakan dalam setiap dari kebanyakan spesies domestik. Dalam banyak hal, sekuen basa primer untuk tiap penciri mikrosatelit tersedia untuk publik, yang berarti bahwa setiap orang dapat menghasilkan satu set penciri yang dipetakan sendiri, yang secara sederhana melalui persiapan primer PCR yang sesuai.
Jenis peta lainnya--peta fisika--dihasilkan melalui beberapa cara. Pertama melibatkan penggunaan panel persilangan sel somatik (somatic-cell hybrid), yang tiap persilangan merupakan satu galur sel yang berisi campuran kromosom yang berbeda dari dua spesies, yaitu hamster dan sapi, yang berasal dari penggabungan sel kedua spesies tersebut. Adanya lokus tertentu di dalam galur sel tertentu dapat dideteksi dengan beberapa cara,
termasuk analisis Southern atau PCR. Penciri DNA yang selalu concordant (semua ada atau semua tidak ada) dalam setiap galur sel dipastikan terletak pada kromosom yang sama; ada atau tidak adanya kelompok penciri concordant terkait dengan ada tidaknya kromosomnya. Bahkan penempatan yang lebih rinci dari penciri adalah mungkin di dalam galur sel yang hanya mengandung porsi kromosom tertentu. Lokus yang ditunjukkan dengan cara demikian dipastikan terletak pada kromosom yang sama, yang dikatakan sebagai syntenic, dan kelompok yang memilikinya disebut syntenic group. Akhirnya, tiap kelompok syntenic secara meyakinkan juga adalah kelompok keterpautan (linkage group).
Metode lain pemetaan fisika melibatkan hibridisasi in situ DNA yang sudah diklon ke situsnya sendiri pada kromosom. Awalnya, ini melibatkan pelabelan DNA klon dengan radioaktif, tetapi sekarang metode tanpa radioaktif yang dikenal sebagai fluorescence in situ hybridization (FISH) terbukti jauh lebih efektif.
Perkembangan yang menjajikan adalah persiapan pustaka DNA spesifik kromosom, yaitu pustaka yang mengandung DNA dari hanya satu kromosom. Ini dapat diperoleh dari kromosom yang dipisahkan dengan FACS atau dengan microdissection, yang melibatkan penggunaan micromanipulator atau laser untuk mengambil satu kromosom dari sekelompok kromosom seperti ditunjukkan pada Gambar 1.1. Pada semua kasus, semua DNA dari kromosom itu dapat diamplifikasi dengan PCR dan kemudian diklon dengan cara seperti biasanya. Ini dinamakan microcloning. Dengan microdissection, bahkan mungkin mengambil hanya sebagian kecil dari satu kromosom, dan oleh karenanya menghasilkan pustaka mikroklon yang hanya terkait dengan bagian kromosom itu saja. Untuk mencek bahwa tiap pustaka membawa DNA dari satu kromosom tertentu saja, atau bagian tertentu dari kromosom, sampel DNA total dari pustaka itu dapat dihibridisasikan ke sekelompok kromosom lengkap dengan menggunakan FISH. Jika pustaka tersebut mengandung apa yang seharusnya dikandung, maka kromosom yang relevan, atau bagian yang relevan dari kromosoma akan berfluoresce. Prosedur ini disebut chromosome painting.
Hasil teknologi ini adalah satu set pustaka DNA yang mencakup tiap region dari tiap kromosom. Setiap dari pustaka-pustaka itu dapat dibagi menjadi serangkaian klon YAC yang overlap, dengan hasil akhirnya berupa satu set klon YAC yang saling bersambungan (contigguous) yang menutupi genom keseluruhan. Segera setelah gen diketahui posisinya di dalam klon YAC tertentu, lokasi fisiknya secara otomatis dapat diketahui dalam daerah beberapa kilobasa (ukuran dari klon YAC tersebut). Akhirnya, sangat dimungkinkan mengisolasi gen tersebut.
Pada banyak spesies domestik, sekarang ada upaya besar, seringkali kerjasama global, untuk menciptakan peta genetika yang menggabungkan
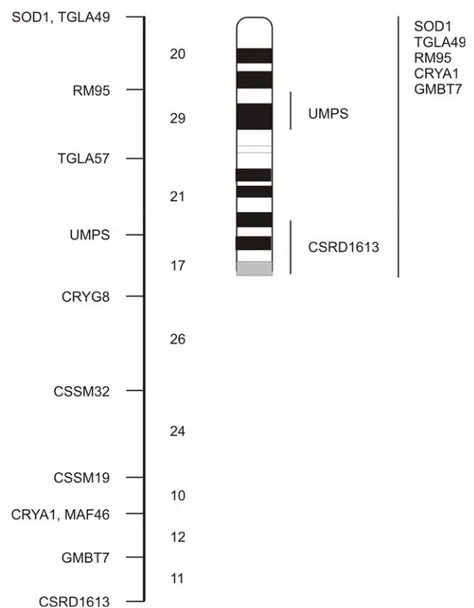
informasi dari peta fisik dan peta pautan (linkage). Satu contoh hasil kolaborasi atau kerjasama itu ditunjukkan pada Gambar 2.10, yang menyajikan peta genetik untuk kromosom 1 pada ternak sapi.
Gambar 2.10. Peta genetika kromosom 1 pada sapi. Sebelah kiri adalah peta keterpautan (linkage map), yang menunjukkan jarak peta (dalam cM) antar lokus. Sebelah kanan adalah informasi peta secara fisika: hanya dua gen telah dipetakan pada daerah spesifik.
Satu karakteristik yang paling menarik dari pembuatan peta genetika adalah kelestarian grup lokus antar spesies yang sangat berbeda. Misalnya,
banyak gen yang terletak pada kromosom 12 manusia, terletak pada kromosom 12 sapi dan pada kromosom 5 kucing. Dan kromosom X bahkan menunjukkan kelestarian yang lebih tinggi. Pada kenyataannya, jika suatu gen terpaut kromosom X dalam satu spesies mamalia, hampir dapat dipastikan gen tersebut terpaut-X dalam setiap spesies mamalia lainnya. Derajat kelestarian yang tinggi ini merangsang dibuatnya comparative gene mapping--perbandingan peta genetika antar spesies. Selain menarik dari sudut pandang evolusi, comparative gene mapping juga sangat penting artinya dari segi praktis, karena derajat kelestarian yang tinggi dari segmen kromosom antar spesies berarti bahwa daerah yang telah dipetakan dengan baik pada satu spesies dapat memberikan informasi sangat penting mengenai lokasi gen yang sama pada spesies lain, jauh sebelum mereka dipetakan pada spesies lain tersebut. Comparative mapping sangat berpotensi sebagai alat atau cara menelusuri lokus penyakit pada ternak domestik, berdasarkan informasi daru tikus dan manusia.
Mempertahankan perkembangan peta genetika yang cepat, dan juga informasi pemetaaan perbandingan, terbukti merupakan suatu tantangan. Sampai sekarang, referensi paling bermanfaat untuk informasi ini adalah publikasi Genetic Maps, yang disunting oleh Steve O'Brien, yang mengandung uraian lengkap peta keterpautan dan peta fisika semua spesies, dari virus sampai manusia. Lebih meningkat lagi, Genetic Maps juga disajikan dalam bentuk database yang menyediakan informasi terkini tentang peta genetika dari semua spesies domestik utama, yang dapat diakses secara bebas melalui internet.
Produksi Polipeptida dari Klon DNA
Apabila DNA asing diklon dalam bakteri atau mikroorganisme lain, tujuan pokoknya adalah untuk menghasilkan kopi DNA asing itu dalam jumlah tak terbatas. Jika DNA asing tersebut adalah gen, ada kemungkinan untuk melakukan satu tahap lebih jauh daripada itu; adalah mungkin merancang DNA asing itu untuk menghasilkan polipeptida yang sesuai di dalam inang tersebut. Untuk mencapainya, perlu menggabungkan promoter yang tepat, dan mungkin perlu juga beberapa daerah kontrol lainnya.
Ada satu perbedaan penting antara kontrol eukariot dan prokariot yang muncul dari tidak adanya split gene pada prokariot. Karena tidak mempunyai split gene, prokariot tidak memerlukan mekanisme penggabungan (splicing) yang dibutuhkan oleh eukariot untuk mengubah transkrip mRNA menjadi mRNA dewasa/matang. Oleh sebab itu, gen eukariot yang dapat ditranskrip dan ditranslasi di prokariot hanyalah gen yang mengandung exon saja.
Juga, karena ada perbedaan substansial dalam mekanisme kontrol antar spesies, region kontrol yang digunakan tersebut seyogyanya region kontrol dari sel inangnya.
Pada prakteknya, hal ini biasanya dicapai dengan meyakinkan bahwa DNA asing tersebut adalah hanya sekuen penyandi, dan dengan menyisipkannya dengan segera dari sekuen pemulai (leader sequence) dan promoter di dalam DNA vektor. Ini mengakali inang agar berpikir bahwa DNA asing tersebut, kenyataannya, adalah bagian dari DNA-nya sendiri. Dalam beberapa kasus, lebih mudah menyisipkan DNA asing ke tengah-tengah gen vektor, yang menghasilkan fusi polipeptida yang dari sini polipeptida asing tersebut dapat dilepas melalui perlakuan kimiawi yang sesuai. Vektor yang memungkinkan transkripsi dan translasi gen asing dinamakan expression vector, dan pustaka yang membawa vektor semacam itu disebut expression library.
Berbagai macam polipeptida dari prokariot dan eukariot yang sedang di produksi secara masal melalui cara yang diterangkan di atas, menggunakan bermacam-macam mikroorganisme (baik prokariot maupun eukariot) dan galur sel mamalia sebagai inangnya. Misalnya vaksin rekombinan (polipeptida protektif dari patogen atau parasit); protein terapi seperti insulin, faktor penggumpal darah (blood clotting), interferon, imunoglobulin, hormon pertumbuhan, dan berbagai macam enzim yang digunakan dalam teknologi molekuler. Selain itu, teknologi tersebut tidak terbatas pada gen yang ada saja: suatu hal yang mungkin, misalnya, merancang dan memproduksi polipeptida fiksi (novel) untuk tujuan-tujuan tertentu, baik sebagai varian dari polipeptida yang sudah ada, atau sebagai molekul asli secara lengkap.
Dari pemanfaatan mikroorganisme untuk memproduksi polipeptida asing di laboratorium, diperlukan satu tahap ekstra (sedikitnya secara prinsip) untuk merancang mikroorganisme tersebut menghasilkan polipeptida asingnya dimana polipeptida itu sebenarnya diperlukan, seperti di dalam tubuh ternak. Ini melahirkan konsep live recombinant vaccine, yang secara khas terdiri atas bakteri atau virus yang tidak berbahaya, beberapa gennya telah diganti dengan gen fungsional untuk polipeptida protektif dari patogen atau parasit. Daripada harus menyediakan alat untuk memindahkan polipeptida protektif melalui injeksi atau proses lain yang menyerap banyak tenaga, vektor vaksin secara alami menginfeksi dan secara otomatis menghasilkan polipetida tersebut dimana diperlukan--di dalam ternak. Pendekatan yang sama dapat digunakan sebagai alat untuk membasmi gangguan insekta. Misalnya, jika gen insekta penyandi enzim esterase hormon disisipkan ke dalam virus yang merupakan patogen alami insekta, dan jika virus rekombinan itu menginfeksi insekta tersebut, esterase menonaktifkan hormon insekta itu, yang menyebabkan penurunan tajam
dalam larval feeding, yang merupakan aspek perusak utama dari banyak gangguan insekta.
Transgenesis
Kita telah melihat bahwa sangtlah mungkin menyisipkan gen dari satu spesies, misalnya babi, ke DNA mikroorganisme atau galur sel mamalia. Kemudian, karena universitalitas kode genetika, polipeptida yang sama akan dihasilkan oleh sembarang inang. Jika ini dapat dicapai dengan mikroorganisme dan galur sel mamalia, pada prinsipnya mungkin juga memperoleh hasil yang sama melalui penyisipan gen asing ke dalam DNA ternak hidup. Proses semacam itu disebut transgenesis.
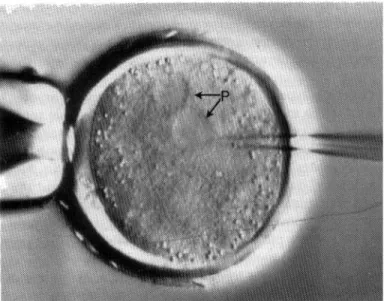
Tahap pertama dalam transgenesis adalah menggabungkan gen terklon dengan promoter yang cocok, yang kemudian menghasilkan gene construct. Tahap ke dua adalah memasukkan gen konstruk tersebut ke dalam satu atau lebih kromosom ternak. Sekarang, ini biasanya dicapai dengan mikroinjeksi dari banyak kopi gen konstruk ke dalam pronukleus betina atau jantan dari sel telur yang baru saja dibuahi (pronukleus jantan adalah nukleus haploid dari sel sperma, dan pronukleus betina adalah nukleus haploid dari sel telur), seperti ditunjukkan pada Gambar 2.11. Setelah mikroinjeksi, sel yang telah dibuahi tersebut dipindahkan ke dalam betina resipien yang telah disiapkan, yang membawa embrio yang dihasilkan sampai kelahiran dengan cara biasa. Dalam proporsi yang sangat kecil (kurang dari 1% pada ternak domestik), genom dari ternak yang dihasilkan mengandung satu atau lebih kopi gen konstruk tersebut. Ternak seperti itu disebut transgenik, dan gen konstruk yang dimasukkan dinamakan transgen. Sekitar setengah dari sejumlah ternak transgenik sebenarnya mengekspresikan (yakni menghasilkan polipeptida dari) transgen tersebut, tetapi hampir semua ternak itu menurunkan transgen tersebut ke turunannya dengan cara normal.
Ternak transgenik pertama adalah mencit (mice) yang dihasilkan tahun 1980. Aplikasi teknologi ini ke ternak domestik dilaporkan pada tahun 1985, ketika babi dan domba dibuat transgenik bagi gen hormon pertumbuhan manusia. Sejak itu, bermacam,-macam transgen telah digunakan, dari spesies bakteri (yang banyak ragamnya) dan manusia; dan spesies domestik yang dibuat transgenik telah melebar hingga termasuk ikan, ayam, kambing dan sapi. Teknik mikroinjeksi mempunyai beberapa kelemahan, misalnya tidak ada kontrol mengenai posisi transgen disisipkan, atau tidak ada kontrol juga tentang bagaimana banyak kopi disisipkan. Di masa mendatang, kita akan melihat metode yang lebih terkontrol digunakan untuk menciptakan ternak transgenik. Pada tikus, misalnya, embryonic stem cell (undifferentiated dan totipotent embryonic cell) dapat dibuat transgenik, dan kemudian diuji di laboratorium untuk ekspresi transgen. Dengan cara ini,
hanya sel yang benar-benar mengekspresikan transgen melalui cara yang diharapkan dapat dipindahkan ke tikus resipien. Ini mengatasi problem dan biaya yang berkaitan dengan tingkat keberhasilan yang sangat rendah dari mikroinjeksi ke sel telur yang dibuahi. Selanjutnya, melalui proses yang disebut targeted gene replacement, adalah mungkin untuk mengganti gen normal di dalam embryonic stem cells dengan gen sejenis yang telah dibuat mutan melalui cara spesifik. Dengan perkawinan yang sesuai antara tikus transgenik yang dihasilkan dan turunannya, adalah mungkin membentuk galur tikus transgenik yang bersifat homosigot untuk mutasi spesifik tersebut.
Gambar 2.11. Menghasilkan domba transgenik melalui mikroinjeksi trasgen yang diklon ke dalam satu di antara dua calon inti (pronuklei) dari sel telur terbuahi.
Apakah tujuan membuat ternak transgenik? Sampai sejauh ini, penggunaan paling ekstensif dari transgenesis adalah dalam penelitian dasar yang menggunakan hewan laboratorium, khususnya tikus. Teknologi tersebut membuktikan sangat bermanfaat dalam penemuan regulasi ekspresi gen dan dalam pengujian terapi gen. Jika digunakan sebagai alat untuk mengganti gen funsional dengan gen non-fungsional (yang menghasilkan mencit knockout), itu juga merupakan cara yang sangat baik untuk menemukan fungsi gen dan penyakit turunan. Demikian juga, tikus
transgenik yang gen antibodinya sendiri telah dinonaktifkan melalui penggantian gen yang ditargetkan, tetapi merupakan transgenik bagi gen antibodi dari spesies lain, dapat digunakan untuk produksi antibodi monoklonal spesisifk spesies. Berkaitan dengan ternak domestik, penggunaan transgenesis dapat dipertimbangkan dari dua sudut pandang, berasosiasi dengan dua hal utama dari transgen, yaitu promoter dan sekuen sandi.
Pemilihan yang tepat dari promoter mengakibatkan ekspresi transgen di bawah kendali manusia. Misalnya, satu dari beberapa promoter yang biasanya digunakan dalam transgenesis awal adalah dari gen metallothionein. Metallothionein adalah protein yang secara alami terdapat dalam banyak spesies; peranannya adalah untuk mengikat logam berat. Pada ternak (non-transgenik) normal, jika konsentrasi logam berat menjadi terlalu tinggi, gen metallothionein mulai menghasilkan metallothionein yang mengikat logam berat yang berlebihan, yang oleh karenanya menghilangkan ancaman keracunan. Jika promoter dari gen metallothionein ditempelkan ke sekuen sandi dari, katakan, gen hormon pertumbuhan, transgen yang dihasilkan tersebut dapat diaktifkan dengan cara menambahkan logam berat ke air minum ternak transgenik itu. Ini berarti bahwa produksi hormon pertumbuhan dari transgen dapat dihidupkan dan dimatikan melalui manipulasi sederhana terhadap air minumnya. Alternatifnya, jika tujuannya adalah mempunyai transgen yang diekspresikan hanya dalam satu jaringan saja, promoter dipilih dari gen yang secara alami diekspresikan hanya dalam jaringan itu. Misalnya, transgen yang mengandung promoter dari gen protein susu akan diekspresikan hanya di dalam kelenjar mammary ternak transgenik.
Berkaitan dengan sekuen sandi, transgenesis memberikan beberapa kemungkinan. Pertama, transgenesis dapat digunakan untuk memasukkan alel tiruan dari suatu gen yang telah ada di dalam spesies. Sebagai contoh, transgen bagi enzim yang dilibatkan dalam laktogenesis dapat dirancang dan dibuat secara spesifik untuk menghasilkan susu yang berkadar laktose rendah, yang oleh karenanya menimbulkan bentuk yang lebih baik dari produk ternak. Ke dua, transgenesis memberikan kemungkinan pemasukan gen yang benar-benar tiruan ke spesies. Misalnya, ada enzim tanaman chitinase yang mendegradasi chitin (komponen utama cuticle dan saluran pencernaan insekta). Jika gen chitinase dapat diekspresikan di dalam kulit domba atau sapi, itu dapat memberikan bentuk non-toksik dari daya tahan alami terhadap insekta. Satu contoh lainnya terjadi pada ayam, yang daya tahan alaminya terhadap penyakit virus telah dihasilkan oleh transgen yang mengekspresikan polipeptida dari virus yang menyebabkan penyakit tersebut. Ke tiga, transgenesis dapat digunakan untuk memproduksi protein manusia yang dibutuhkan untuk tujuan terapi, seperti alpha-1-antitrypsin
atau blood clotting factor. Misalnya, domba telah dibuat transgenik untuk sekuen sandi dari gen manusia (misalnya blood clotting factor IX) yang ditempelkan ke promoter dari gen penyandi suatu protein yang terdapat secara alami di dalam susu domba, misalnya β-laktoglobulin. Domba ini menghasilkan polipeptida manusia di dalam air susunya, yang dapat diekstrak dari situ secara mudah. (Satu dari keuntungan utama transgenesis sebagai alat penghasil protein terapi adalah bahwa inang eukariot cenderung dapat memproses polipeptida tersebut menjadi struktur tersier (tertiary) yang lebih tepat daripada inang alternatif lainnya, yaitu prokariot seperti bakteria.) Ke emapt, transgenesis dapat digunakan untuk menciptakan ternak yang organnya atau jaringannya cocok ditranplantasikan ke manusia. Misalnya, babi telah dibuat transgenik untuk gen yang mengekspresikan polipeptida manusia pada permukaan sel dari organ atau jaringan yang akan ditransplantasikan, dengan tujuan menghindari problem penolakan. Ini dilihat sebagai alat penting mengatasi keterbatasan organ manusia untuk transplantasi.
Penggunaan di masa mendatang dari ternak transgenik, dan kontroversi etika yang mengelilinginya, didiskusikan pada Bab 20.
Teknologi Antisens
Pada Bab 1 kita melihat bagaimana gen diatur oleh protein. Pada prokariot tertentu (dan mungkin pada eukariot), beberapa gen diatur oleh RNA: RNA dari suatu gen pengatur bersifat komplemen terhadap, dan oleh karena itu berikatan dengan, RNA dari gen ke dua, yang oleh karenanya menghambat translasi dari gen ke dua tersebut.
Ini memunculkan beberapa kemungkinan yang sangat menarik untuk kontrol buatan dari ekspresi gen. Jika promoter dari satu gen ditempatkan pada ujung lainnya dari gen itu, rangkaian non-template akan ditranskrip, yang menghasilkan mRNA yang sekuennya sama seperti sekuen rangkaian template atau rangkaian antisens, dan yang oleh karena itu berkomplemen terhadap sekuen mRNA sens atau mRNA normal untuk gen itu. RNA antisens ini akan mengikat RNA sens, mencegah translasi.
Ada banyak cara memanfaatkan RNA antisens. Misalnya, bakteri yang bertanggung jawab dalam reaksi fermentasi yang menyebabkan produk susu sapi seperti keju dan yoghurt, dapat dibuat resisten terhadap virus pengganggu yang merupakan penyebab utama kegagalan fermentasi. Ini dapat dicapai dengan memasukkan sekuen sandi virus esensial ke dalam plasmid bakteri, meyakinkan bahwa sekuen sandi tersebut ada dalam orientasi terbalik, dekat dengan promoter bakteri. Jika plasmid rekombinan dimasukkan ke dalam bakteri fermentatif tersebut, promoter akan menghasilkan RNA antisens yang dapat menonaktifkan RNA virus tersebut,