VARIASI DNA KLOROPLAS
Shorea leprosula
Miq.
DI INDONESIA MENGGUNAKAN
PENANDA PCR-RFLP
RURI SITI RESMISARI
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
PERNYATAAN
Dengan ini saya menyatakan bahwa tesis berjudul “Variasi DNA Kloroplas
Shorea leprosula Miq. di Indonesia Menggunakan Penanda PCR-RFLP ” adalah karya saya sendiri dan belum diajukan dalam bentuk apapun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam daftar pustaka dibagian akhir tesis ini.
Bogor, Agustus 2006
ABSTRAK
RURI SITI RESMISARI. Variasi DNA Kloroplas Shorea leprosula, Miq di
Indonesia Menggunakan Penanda PCR-RFLP . Dibimbing oleh ISKANDAR
ZULKARNAEN SIREGAR and ULFAH JUNIARTI SIREGAR
Shorea leprosula, salah satu jenis dari famili Dipterocarpaceae yang mendominasi hutan hujan tropis di Asia Tenggara. Dalam tesis ini Dipterocarpaceae terdiri dari banyak jenis yang mempunyai nilai ekonomis dan ekologis yang penting. Penanda
PCR-RFLP digunakan untuk mencari variasi cpDNA Shorea leprosula di Indonesia.
Sampel daun dikumpulkan dari 13 populasi yang berasal dari pulau Jawa, Sumatera dan Kalimantan. Penelitian dimulai dengan melakukan uji polimorfisme DNA untuk mencari variasi cpDNA dengan menggunakan kombinasi 4 primer dan 10 enzim
restriksi. Hasil pengujian menujukkan bahwa hanya kombinasi rbcL – Alu I yang
ABSTRACT
RURI SITI RESMISARI. Chloroplast DNA Variation of Shorea leprosula, Miq in
Indonesia assesed by PCR-RFLP Marker. Supervised by ISKANDAR
ZULKARNAEN SIREGAR and ULFAH JUNIARTI SIREGAR
Shorea leprosula is a member of Dipterocarpaceae that occurs predominately in tropical rain forests of Southeast Asia. Dipterocarps consists of many economical and ecologically important species. In this paper PCR-RFLP markers were used to
observe cpDNA variation of Shorea leprosula in Indonesia. Leaf samples from
individual plants were collected from 13 populations in Java, Sumatra and Kalimantan islands. cpDNA polymorphism was assessed using combination of 4
primers and 10 enzyme restrictions. The result showed that only rbcL-Alu I
© Hak cipta milik Ruri Siti Resmisari, Iskandar Zulkarnaen Siregar,
Ulfah Juniarti Siregar, tahun 2006 Hak cipta dilindungi
VARIASI DNA KLOROPLAS
Shorea leprosula
Miq.
DI INDONESIA MENGGUNAKAN
PENANDA PCR-RFLP
RURI SITI RESMISARI
Tesis
Sebagai salah satu syarat untuk memperoleh gelar Magister Sains
Pada Program Studi Ilmu Pengetahuan Kehutanan
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
Judul Tesis : Variasi DNA Kloroplas Shorea leprosula Miq. di Indonesia Menggunakan Penanda PCR-RFLP
Nama : Ruri Siti REsmisari
NIM : E051020291
Disetujui
Komisi Pembimbing
Dr. Ir. Iskandar Z. Siregar, M.For.Sc Ketua
Dr. Ir. Ulfah Juniarti Siregar, M.Agr Anggota
Diketahui
Ketua Program Studi Ilmu Pengetahuan Kehutanan
Dr. Ir. Dede Hermawan, M.Sc
Dekan
Sekolah Pascasarjana
Dr. Ir. Khairil Anwar Notodiputro, MS
PRAKATA
Puji dan syukur penulis panjatkan kep ada Allah SWT atas segala rahmat
dan hidayah-Nya sehingga penulis dapat menyelesaikan karya ilmiah yang berjudul
Variasi DNA Kloroplas Shorea leprosula Miq. di Indonesia Menggunakan Penanda
PCR-RFLP. Penelitian ini dilakukan mulai Maret 2005 hingga September 2005. Tesis ini di bagi dalam 5 bab, pada bab I d iuraikan latar belakang, tujuan,
serta ruang lingkup penelitian. Pada bab II disajikan tinjauan pustaka dengan tujuan
untuk memberikan gambaran singkat tentang S. leprosula, keragaman genetik , DNA
kloroplas dan penanda genetik. Bab III memberikan informasi mengenai metodologi
yang digunakan dalam penelitian termasuk waktu, tempat, bahan dan alat yang digunakan serta metode analisis data yang digunakan. Hasil dan pembahasan
disajikan pada Bab IV, pada bab ini dijelaskan tentang variasi cpDNA S. leprosula di
Indonesia, kesimpulan dan saran disajikan pada Bab V.
Penulis menyampaikan banyak terima kasih kepada Dr. Ir. Iskandar Z.
Siregar, M.For.Sc dan Dr. Ir. Ulfah Juniarti Siregar, M.Agr sebagai komisi
pembimbing yang telah banyak membantu dan mengarahkan penulis dalam penulisan
tesis ini. Ucapan terima kasih juga disampaikan kepada Departemen Silvikultur
Institut Pertanian Bogor, Inst. of Forest Genetics and Forest Tree Breeding Faculty of Forest Sciences and Forest Ecology University of Goettingen Germany dan AUNP
(Asean-Eu University Network Programe) yang telah memfasilitasi penulis dalam penelitian ini. Ungkapan terimakasih juga kami sampaikan kepada bapak, ibu, serta
suami tercinta serta seluruh keluarga atas segala doa dan kasih sayangnya, serta
semua pihak yang telah ikut membantu penulis mulai dari persiapan hingga
selesainya tesis ini.
Semoga tesis ini bermanfaat.
Bogor, Agustus 2006
DAFTAR ISI
PCR (Polymerase Chain Reaction)...10
PCR-RFLP ...14
(Polymerase Chain Reaction - Restriction Fragment Length Polymorphisms).14 METODE PENELITIAN ...16
Tempat dan waktu penelitian ...16
Bahan dan Alat Penelitian...16
Konservasi Genetik Shorea leprosula...39
SIMPULAN DAN SARAN ...41
Simpulan...41
Saran...41
DAFTAR GAMBAR
Gambar 1. Rantai DNA ...9
Gambar 2. Letak cpDNA pada sel ...10
Gambar 3. Prinsip reaksi RCR ...12
Gambar 4. Proses amplifikasi PCR...13
Gambar 5. Bagan alur penelitian di laboratorium ...17
Gambar 6. Peta pengambilan contoh S. leprosula...18
Gambar 7. Letak petB, psaA, trnLF dan rbcL pada peta plasmid cpDNA Nicotiana tabacum...21
Gambar 8. Hasil Ekstraksi DNA S. leprosula...25
Gambar 9. Elektrophoresisis cpDNA dengan berbagai pengenceran DNA dari S. leprosula...26
Gambar 10. Elektroforegram hasil PCR cpDNA dengan berbagai primer : (a) petB, (b) psaA, (c) trnLF, (d) rbcL, M = marker bond...28
Gambar 11. Elektroforegram hasil uji polimorfisme : (a) psaA-pst I (monomorfik), (b) petB-Hinf I (monomorfik), (d) rbcL-Alu I (polimorfik), M= marker bond...30
Gambar 12. Elektroforegram hasil pemotongan cpDNA dengan menggunakan kombinasi perlakuan rbcL-Alu 1 ...32
Gambar 13. Penyebaran cpDNA S. leprosula di Indonesia berdasarkan penanda PCR-RFLP dengan menggunakan rbcL-Alu1 ...35
DAFTAR TABEL
Tabel 1. Lokasi pengambilam contoh S. leprosula...18
Tabel 2. Komposisi bahan-bahan yang digunakan untuk PCR...22
Tabel 3. Pengkondisian suhu dan waktu pada mesin PCR untuk primer petB, psaA, trnLFdan rbcL...22
Tabel 4. Jenis enzim restrksi yang digunakan utuk memotong DNA...23
Tabel 5. Hasil uji polimorphisme ...31
Tabel 6. Haplotipe yang teridentifikasi pada 65 individu S. leprosula
berdasarkan PCR-RFLP terdapat dua haplotipe...33
Tabel 7. Frekuensi haplotipe dari 13 populasi ...34
Tabel 8. Hasil perhitungan AMOVA berdasarkan 65 individu yang berasal dari 13 populasi...35
Tabel 9. Matrik signifikasi nilai (P value) pada level 0.05 ...36
DAFTAR LAMPIRAN
Lampiran 1. Tahapan ekstraksi DNA...45
Lampiran 2. Elektroforegram S. leprosula di 13 populasi dengan rbcL-Alu I...46
PENDAHULUAN
Latar Belakang
Laju kerusakan hutan di Indonesia, berdasarkan data WALHI (Wahana
Lingkungan Hidup Indonesia) tahun 2004, berada dalam situasi krisis dan kondisi
yang sangat mengkhawatirkan. Pembalakan hutan, baik yang legal maupun ilegal,
telah menyebabkan kerusakan hutan yang sudah tidak terkendali di hampir
seluruh kawasan hutan Indonesia. Tingkat deforestasi saat ini telah mencapai 3,8
juta hektar per tahun (tahun 2004). Hal ini menunjukan bahwa Indonesia telah
kehilangan hutannya seluas 7,2 hektar setiap menitnya. Berdasarkan data WWF
(World Wildlife Fund) tahun 2002, pemerintah Indonesia mengakui bahwa
kerusakan hutan Indonesia selama 50 tahun terakhir sekitar 40% dari tutupan
hutannya (Harsono, 2004).
Tuntutan kebutuhan bahan baku kayu cenderung terus meningkat terutama
jenis kayu keras seperti Jati (Tectona grandis) dan Meranti (Shorea spp.)
(Handadhari, 2002). Laju pembalakan yang dilakukan sekarang kurang diikuti
oleh rehabilitasi lahan yang seimbang. Selain itu, kegiatan pembalakan hutan juga
tidak hanya menyebabkan hilangnya keanekaragaman hayati, hancurnya
habitat-habitat satwa endemik, juga menyebabkan semakin merosotnya kualitas sumber
daya alam Indonesia. Pengembangan skema pengalihan lapangan kerja
penebangan hutan sebaiknya dialihkan ke dalam program rehabilitasi hutan
dengan menggunakan dana rehabilitasi hutan. Hasil penelitian South-Central
Kalimatan Production Forest Project (SCKFP) kerjasama Dephut-Uni Eropa di
Kalimantan Selatan merekomendasikan rehabilitasi hutan yang dilakukan
diantaranya dengan menanam Meranti (Shorea spp.).
Shorea leprosula merupakan salah satu jenis Meranti Merah yang tumbuh
di hutan hujan dataran rendah. Kayunya mudah dikerjakan dan tidak mudah
mengkerut. Banyak digunakan sebagai bahan baku meubel, kayu lapis dan vinir.
Termasuk ke dalam kelas awet dan kuat III – IV. Meranti jenis ini merupakan
kayu unggulan yang memiliki nilai ekonomis tinggi, tetapi dengan eksploitasi
meranti ini akan semakin langka pada masa mendatang. Budidaya meranti dalam
skala besar (pola HTI) mempunyai kendala dalam pengadaan bibitnya (Murjahid,
2003). Ditambahkan oleh Sudarmonowati et al. (2004), bahwa kurang majunya
pembangunan sektor kehutanan di Indonesia, karena kurang ketersediaan bibit
yang bermutu. Dua diantara faktor penting yang berpengaruh pada penyediaan
bibit bermutu adalah sumber bibit yang unggul dan teknik propagasi yang mapan.
Untuk mewujudkan faktor penting ini, diperlukan satu penelitian untuk
mengetahui potensi genetik yang ada, mengingat aspek genetik meranti masih
sedikit keterangannya. Penelusuran variasi genetik penting dilakukan, sehingga
sebelum dilakukan suatu program konservasi dan perbaikan genetik dalam upaya
penyediaan bibit bermutu, informasi yang dibutuhkan sudah tersedia.
Berdasarkan pada fenomena tersebut, mutlak tersedianya kondisi genetik yang
memadai sehingga tercipta suatu sistem konservasi genetik yang mapan.
Analisis genetik merupakan cara yang dapat digunakan untuk menduga
karakteristik genetik mengkonfirmasi sifat unggul yang telah diamati berdasarkan
pengamatan morfologi di lapangan. Manfaat dari analisis genetik ini, antara lain
selain dapat mendeteksi sifat unggul pada saat kecambah atau bahkan fase embrio
untuk program pemuliaan bibit, juga menunjang program konservasi karena dapat
mendeteksi tingkat kepunahan jenis di suatu lokasi jauh hari sebelum penurunan
populasi tersebut jelas terlihat. Aplikasi nyata lainnya dari analisis genetik adalah
hubungan anak, tetua, dan kerabatnya yang ditanam di lain tempat dapat
diketahui, sehingga gambaran asal individu tersebut dapat diketahui. Teknik
analisis genetik ini, juga lebih menghemat tenaga dan biaya karena dapat
mencegah penanaman bibit yang tidak unggul. Manfaat lain dari analisis genetik
ini adalah dapat mengetahui evolusi dari suatu jenis tanaman, mendeteksi
keragaman atau keseragaman genetik suatu populasi, mendeteksi variasi
somaklonal (terutama pada tananaman hasil kultur jaringan), sertifikasi tanaman
tetua, identitas benih murni, dapat melakukan studi tentang genetik yang tahan
hama dan atau penyakit, pemetaan genetik yang memberi informasi letak gen
pada kromosom-kromosom yang mengatur sifat-sifat tertentu, sehingga dapat di
Teknik analisis yang banyak dikembangkan sekarang adala h berdasarkan
markapenanda isozim dan markapenanda DNA. Pendugaan variasi genetik
dengan teknik isozim merupakan teknik yang paling awal dikembangkan dan di
aplikasikan pada tanaman. Masing- masingBerbagai teknik yang dikembangkann,
masing- masing mempunyai kelemahan dan kelebihannya, hal ini disesuaikanjika
dikaikan dengan tujuan dan biaya yang tersedia. MarkaPenanda DNA merupakan
dasar untuk melihat adanya suatu perubahan sifat dengan mendeteksi perubahan
urutan basa dan basa nukleotida DNA khas untuk setiap jenis protein atau enzim.
MarkaPenanda DNA yang di maksud dapat berupaadalah markapenanda dominan
dan ko-dominan. MarkaPenanda dominan adalah penanda berdasarkan ada atau
tidaknya pita DNA yang muncul setelah elektroforesis, yang termasuk ke dalam
markapenanda ini adalah RAPD (random amplified polymorphic DNAdna) dan
RFLP (restriction fragment length polymorphism). Sedangkan markapenanda
kodominan adalah penanda yang menghasilkan pita heterozigot dan homozigot.
MarkaPenanda yang termasuk co-dominan adalah Mikrosatelit dan IsozimAFLP
(amplified fragment length polymorphism). .
Menurut Finkeldey (2003), sumber DNA yang diteliti dengan
markapenanda genetik ini sebagian besar terdapat dalamnukleus (99,9%). Sisanya
yang 0,1% terdapat dalam organel tertentu. Organel yang mengandung DNA ialah
terdapat pada plastida yang terdiri dari mitokondria dan kloroplas. Material
genetik yang di analisis dari plastida biasanya hanya berasal dari sifat satu
tetuanya, kalau tidak dari tetua jantannya saja atau hanya dari betinanya,
sedangkan dengan material genetik yang diambil dari inti, analisis genetiknya bisa
menunjukan dua tetuanya. Pada DNA kloroplas material genetik diturunkan dari
tetua betina, tetapi bisa mendeteksi tetua genetik jantannya.
Melihat kondisi di atas dalam usaha melengkapi data mengenai keragaman
genetik Meranti maka perlu dilakukan suatu penelitian yang dapat memberikan
infomasi mengenai hal tersebut. Penulis memilih DNA kloroplas (cpDNA)
sebagai bahan untuk analisis keragaman genetik Meranti dan teknik PCR–RFLP
sebagai markapenanda genetik. Teknik ini sangat sederhana, cepat dan ekonomis,
memiliki kisaran 50-3000 bp yang dapat dibedakan dan lebih sesuai bagi individu
Permasalahan
Kerusakan hutan dipterokarpa yang diakibatkan oleh deforestrasi seperti
pembalakan hutan secara liar, kebakaran dan lainnya dapat berdampak pada
penurunan populasi S. leprosula. Penurunan populasi ini menyebabkan terjadi
penurunan sumberdaya genetik dari S. leprosula, oleh karena itu perlu segera
dilakukan program konservasi genetik jenis ini. Penelitian tentang keragaman
genetik S. leprosula sangat diperlukan untuk memberikan landasan ilmiah dalam
penggunaan stategi konservasi genetik.
Tujuan
Tujuan dari penelitian ini adalah untuk :
1. Mengetahui variasi keragaman cpDNA S. leprosula, yaitu jumlah
haplotiype berdasarkan PCR-RLFP, yaitu titik potong (restriction site)
pada DNA fragmen hasil PCR dengan menggunakan enzim restriksi
2. Mengetahui variasi cpDNA di dalam dan antar populasi S. leprosula
Hipotesis
Hipotesis yang diuji adalah bahwa cpDNA S. leprosula di Indonesia
memiliki variasi yang rendah, dimana pola yang dijumpai dapat digunakan untuk
melacak atau membedakan populasi antar pulau.karena diwariskan secara
maternal.
Manfaat
1. Hasil dari penelitian ini diharapkan dapat memberikan informasi tetang variasi
genetik S. leprosula yang ada di Indonesia untuk kepentingan suatu program
konservasi genetik yang berkesinambungan, dan pada akhirnya dapat
digunakan untuk mendukungsuatu program pemuliaan dari jenis ini
2. Variasi cpDNA S. leprosula dapat digunakan untuk menentukan asal usul,
TINJAUAN PUSTAKA
Shorea leprosula Miq. Aspek Botanis
Shorea leprosula termasuk ke dalam famili Dipterocarpaceae, Kelas
Dicotyledone, dan sub-divisi angiospermae. Pohon jenis ini mempunyai tinggi
total mencapai 60 m, dan tinggi bebas cabang 45 m, diameter batang umumnya
mencapai 2 m dengan banir mencapai 5 m (Heyne, 1987). Samingan (1973)
menambahkan, pohon jenis ini memiliki batang yang lurus, besar, bersih dan
berbanir. Kulitnya mempunyai garis-garis halus dan lurus. Sering terlihat ada
damar yang keluar dari kulitnya, warnanya coklat sampai kuning. Bunganya
berwarna merah, kuning atau agak putih. Buah keras dengan sayap berjumlah
lima yang terdiri dari 3 sayap panjang dan 2 sayap pendek, serta bentuk buahnya
berbentuk bulat. Pada umumnya meranti menduduki strata tajuk lapisan paling
atas (strata A) atau lapisan ke-2 (strata B). Pada umumnya meranti termasuk jenis
semitoleran.
Berdasarkan keadaan dan sifat kayunya S. leprosula termasuk ke dalam
kelompok meranti merah. Jenis meranti merah terdiri dari pohon besar dan
berbanir besar, batang merah atau bersisik, pada umumnya berdamar, kulit luar
dan kulit dalam tebal, berurat- urat, warnanya merah atau kemerah- merahan,
gubalnya kuning pucat, serta isi kayu berwarna merah (Al Rasyid, et al., 1991).
Penyebaran dan Tempat Tumbuh
Shorea leprosula secara alami menyebar mulai dari Semenanjung
Thailand dan Malaysia, Sumatera dan Kalimantan Utara, biasanya ditemukan di
hutan dipterokarpa di bawah 700 m menempati ruang terbuka di hutan yang
mengalami gangguan. Tumbuh pada berbagai jenis tanah tetapi tidak toleran
terhadap genangan. Curah hujan 1500-3500 mm pertahun, dan musim kemarau
pendek perlu untuk pertumbuhan dan regenerasi. Jarang ditemukan di punggung
bukit. S. leprosula merupakan meranti merah yang pertumbuhannya paling cepat
jika dibandingkan dengan meranti jenis yang lain, namun kondisi ini hanya
sampai umur 20 tahun, selanjutnya akan terkejar oleh meranti lain. Jenis ini
mengalami penurunan populasi yang disebabkan oleh adanya penebangan liar,
Pembungaan
Pembungaan S. leprosula terjadi setiap 3 hingga 5 tahun sekali. Pada saat
mengalami pembungaan, hampir semua pohon berbunga lebat dan serempak,
bunga tersebut akan merekah pada malam hari. Jika terjadi kekeringan selama
periode ini, gugur buah tertunda dan buah tidak berkembang sempurna. Pada
sebaran alami, pengumpulan benih dilakukan pada bula Maret-Juli, terutama pada
bulan setelah musim kemarau.
Pemanenan Buah
Untuk mengurangi kerusakan oleh serangga, sebaiknya buah dipetik di
atas pohon. Pengumpulan hendaknya dilakukan ketika periode utama gugur buah,
sebab sebelum ini biasanya belum masak dan terserang serangga.
Kegunaan
Kayunya ringan, merupakan kayu berharga dan sangat baik untuk
(joinery), (meubel), panel, lantai, langit- langit dan juga untuk kayu lapis.
Mengha silkan resin yang dikenal dengan damar daging, yang dapat digunakan
obat. Kulitnya digunakan untuk produksi tannin.
Keragaman Genetik
Keragaman genetik suatu spesies adalah hasil dari perkembangbiakan
secara seksual. Pada proses perkembangbiakan seksual, terjadi peristiwa meiosis
yang mereduksi jumlah kromosom diploid (2n) dalam sel tetua menjadi haploid
(n) dalam gamet, mengikuti hukum segregasi bebas seperti diungkapkan oleh
Mendel (Hukum Mendel 1). Selanjutnya diperjelas lagi pada Hukum Mendel 2
meiosis kromosom homolog juga akan mengalami pindah silang dan
kadang-kadang terjadi perubahan susunan genetik karena mutasi yang akan menambah
keturunan (Crowder, 1986). Selain perkawinan dan mutasi, ditambahkan oleh
Finkeldey (2003) bahwa migrasi, aliran ge netik, penyimpangan genetik, dan
fenotipe yang disebabkan oleh faktor- faktor genetik. Fenotipe salah satu tanaman
akan berbeda dengan tanaman yang lainnya dalam satu atau beberapa hal.
Keragaman genetik merupakan landasan bagi pemulia untuk memulai
suatu kegiatan perbaikan tanaman. Besarnya keragaman genetik dapat menjadi
dasar untuk menduga keberhasilan perbaikan genetik di dalam program pemuliaan
(Comstock dan Moll, 1963 dalam Rachmadi 1999). Allard (1961)
mengungkapkan bahwa, keragaman genetik yang luas merupakan syarat
berlangsungnya proses seleksi yang efektif karena memberikan keleluasaan dalam
proses pemilihan suatu genotipe. Selain itu populasi dengan keragaman genetik
yang lebih luas akan memberikan peluang yang lebih besar diperolehnya
karakter-karakter yang diinginkan (Simonds, 1979).
Soerjanegara dan Djamhuri (1979) mempertegas bahwa dalam satu jenis
pohon dapat dijumpai keragaman geografis (antar provenan), keragaman lokal
(antar tempat tumbuh), dan keragaman dalam pohon serta keragaman antar pohon.
Ada dua sebab yang menimbulkan keragaman, yaitu perbedaan lingkungan dan
perbedaaan susunan genetik. Keragaman lingkungan biasanya disebabkan oleh
keadaan perbedaan tempat tumbuh, sifat tanah, atau jarak tanam. Namun adapula
keragaman yang tidak dapat diterangkan dengan perbedaan tempat tumbuh,
misalnya perbedaan bentuk batang, tebang batang, tebal cabang, dan berat jenis
kayu dari pohon-pohon dalam suatu tegakan. Dalam hal ini keragaman
dipengaruhi oleh perbedaaan genetik yang diturunkan tetua kepada keturunannya
(keragaman genetik). Adanya keragaman dalam suatu jenis perlu diketahui lebih
dahulu sebelum memulai dengan pemuliaan pohon, karenakeragaman genetik
merupakan syarat mutlak dalam pemuliaan, yaitu untuk memungkinkan seleksi
dan untuk mencegah dihasilkannya tanman yang tidak bermutu.
DNA Kloroplas
Yatim (2003) mengungkapkan bahwa unit fungsional materi genetik ialah
gen, berasal dari kata genos, artinya asal- usul. Sedangkan unit struktural atau unit
kimiawi gen ialah DNA (deoxyribo-nucleic acid, asam deoksiribo- nukleat). Gen
atau DNA itu berderet secara linier pada kromatin atau kromosom. Satu benang
kromatin dibina atas nukleoprotein, yaitu gabungan asam nukleat (DNA) dan
protein bertindak sebagai tempat melilit, protein yang jadi tempat melilit DNA
disebut histon. Protein lain dalam kromatin ada yang bertindak sebagai penyekat,
penyalut, unsur regulator, atau sebagai enzim bagi aktivitas DNA, mereka disebut
protein nonhiston. Gen menumbuhkan dan memelihara aktivitas seharian berbagai
karakter dalam tubuh. Jumlah karakter dalam satu individu ada ribuan macam.
Contoh karakter: Batang (tebal, gepeng/pipih), daun (bulat, lonjong, jarum), buah
(bulat/kriput).
Di antara gen yang banyak itu, ada karakter yang pengatur utamanya satu
gen, disebut karakter monogenik. Ada pula karakter yang diatur oleh banyak gen,
disebut karakter poligenik. Satu gen dibina atas satu molekul DNA. Antara gen
bersebelahan dalam satu kromosom ada urutan DNA seling (intervening
sequences), tidak berperan dalam menumbuhkan suatu karakter.
DNA semacam bahan organik yang memiliki BM (berat molekul) yang
terbesar dalam sel, yaitu dalam ukuran juta. Monomer DNA ialah nukleotida.
Satu gen dibina atas satu molekul DNA, dan satu molekul DNA dibina atas ribuan
sampai puluhan ribu nukleotida. Satu nukleotida terdiri dari tiga gugus senyawa:
1) gula deoksiribosa; 2) fosfat; 3) basa-N. Gula yang membina DNA tergolong
gula pentosa, yaitu gula yang atom karbonnya lima. Glukosa yang membina
sebagian besar gula dalam tubuh kita dan yang menjadi sumber utama energi,
tergolong gula heksosa, artinya gula yang atom karbonnya enam, gugus fosfat
ialah -PO4-3. Basa-N terdiri dari dua kelompok dan tiap kelompok dibina atas dua
macam basa: 1) purin; adenin (A) dan guanim (G); 2) pirimidin: timin (T) dan
citosin (C). Satu molekul DNA terdiri untaian linear nukleotida, sehingga disebut
juga satu utas. Agar sifat kimianya stabil maka DNA itu bersusun berpasangan,
disebut utas double. Kedua utas DNA yang berpasangan (double) itu berpilin
sejajar (helix) sesama, tapi arahnya berlawanan (anti-paralel). Maksudnya bagian
kepala satu utas berpasangan dengan bagian ekor utas pasangan.
Tegasnya DNA dalam inti sel disebut dalam susunan double helix
anti-paralel. Kedua utas diikat oleh ikatan hidrogen antar basa masing- masing.
Perikatan antara basa itu tertentu dan tetap, yaitu antara A dari satu utas berikatan
sepasang utas DNA yang double helix membentuk semacam tangga spiral. Induk
tangganya yang sejajar tapi berpilin ialah untaian G-P. Jadi B dari satu utas
berikatan dengan B dari utas pasangan B-B. Urutan nukleotida yang membina
satu molekul DNA membentuk semacam tangga spiral. Induk tangganya ialah
ikatan S-P dari kedua utas, sedangkan anak tangganya ialah ikatan B-B, tangga itu
berbentuk spiral, maka pegangannya kiri-kanan ialah untaian S-P, sedangkan anak
tangga yang diinjak ialah pasangan B-B.
Suatu gen diberi simbol dalam buku atau majalah menurut urutan
pasangan basa nukleotiodanya: A-T, G-C. Alasannya ia lah: 1) P (fosfat) semua
nukleotida tetap; 2) S (sugar, gula) semua nukleotida tetap, yaitu deoksiribosa; 3)
variasi antara nukleotida hanya pada basa yang empat macam; 4) mutasi yang
terjadi pada suatu gen sehingga menyebabkan kelainan atau penyakit, sela lu
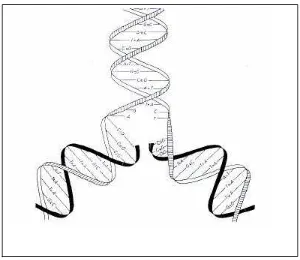
terjadi pada basa nukleotida saja. Susunan DNA disajikan seperti pada Gambar 1.
Gambar 1. Rantai DNA (Hattemer et al., 1993 dalam Finkeldey, 2003)
Finkeldey (2003) menambahkan, sampai saat ini masih diyakini bahwa
genetik merupakan hal yang mampu mengenali dan memberikan informasi suatu
dengan material yang la in yaitu DNA (deoxyribonucleic acid). Materi genetik gen
ialah DNA-nya. Asam ini disebut juga asam nukleat, berasal dari kata asam yang
terdapat dalam nukleus, karena sebagian besar (99,9 persen) asam ini terdapat
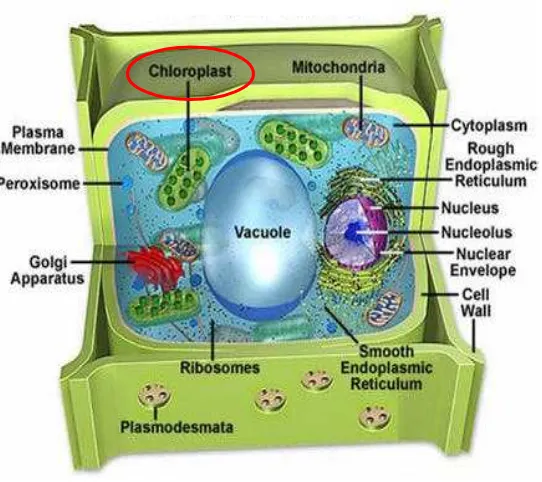
dalam inti, sisanya yang 0,1 persen terdapat dalam organel tertentu. Organel yang
mengandung DNA ialah plastida yang terdiri dari mitokondria dan kloroplas.
Material genetik yang di analisis dari plastida biasanya hanya berasal dari sifat
satu tetuanya, kalau tidak dari tetua jantannya saja atau hanya dari betinanya saja,
lain halnya dengan material genetik yang diambil dari inti analisis genetiknya,
bias menunjukan dua tetuanya. Pada DNA kloroplas material genetik diturunkan
dari induk betina nya saja.
Gambar 2. Letak cpDNA pada sel
PCR (Polymerase Chain Reaction)
Polimerase chain reaction (PCR) merupakan teknik yang mulai
berkembang pesat sekitar tahun 1987. Pada dasarnya PCR mampu mengenali
dan memperbanyak (amplifikasi) segmen DNA sasaran walupun dalam
oligonukleotida. Reaksi amplifikasi sangat tergantung dari keberadaan enzim
polimerase sebagai katalisator, terutama yang tahan pana s. Enzim yang paling
terkenal dan banyak digunakan adalah polimerase DNA Taq yang diisolasi dari
bakteri yang tahan panas Thermus aquaticus. Bahan utama lain yang diperlukan
adalah deoxynukleotide triphospates (dNTPa).
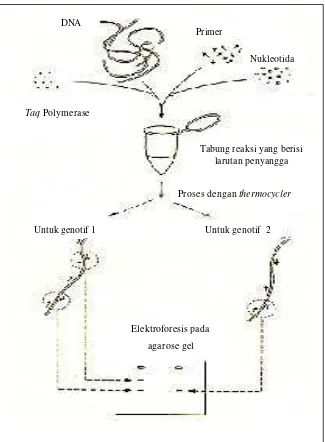
PCR adalah suatu metode untuk menggandakan atau mengamplifikasi
DNA yang diisolasi pada sebuah tabung reaksi kecil dengan melalui replikasi
berulang (Gambar 3). Titik awal dari reaksi (primers) adalah oligonukleotida,
yakni potongan kecil DNA yang dihasilkan secara buatan (biasanya terdiri
antara 10-25 nukleotida). Sekuensi basa dari primer dapat dipilih secara bebas.
DNA teramplifikasi dalam reaksi campuran mengunakan enzim thermostabil
(enzim tahan panas) yakni DNA polimerase dari titik awal seperti ditunjukkan
oleh sekuensi pada primer. Reaksi dikendalikan oleh perubahan suhu pada
thermocycler. PCR memungkinkan penggandaan potongan pendek. DNA dari
semua organisme (lebih dari 2000 hingga 3000bps). Dari satu tabung reaksi
tunggal dapat dihasilkan jutaan tiruan potongan DNA identik seperti pada
Gambar 3 (Newbury dan Fordllyoyd, 1993 dalam Finkeldey, 2003).
PCR merupakan salah satu tahapan proses dalam penentuan
keanekaragaman genetik, PCR ini berfungsi untuk mendapatkan
sekuensi-sekuensi DNA dari genom DNA. Akan tetapi menurut Gupta e t al. (2002)
penanda genetik ini tidak hanya PCR, penandaan lain yang biasa digunakan
adalah; (i) hibridisasi berdasarkan penanda, (ii) penanda molekuler berdasarkan
PCR yang dilanjutkan dengan hibridisasi, (iii) sekuensing dan chip DNA
berdasarkan penanda
Seiring dengan kemajuan dalam teknologi DNA, analisis PCR-RFLP dapat
dilakukan tanpa menggunakan pelacak DNA dan proses hibridisasi DNA.
Penemuan program PCR (polymerase chain reaction) dapat membantu
mendapatkan sekuensi-sekuensi DNA tertentu dari genom DNA, kemudian
dilakukan pemotongan dengan enzim restriksi, atau dengan kata lain analisis
PCR-RFLP dapat dilakukan terhadap sekuensi-sekuensi DNA spesifik yang telah
Gambar 3. Prinsip reaksi RCR (Rabouam et al, 1999 dalam finkeldey, 2005)
.
Dalam analisis PCR, digunakan primer spesifik yang mampu mengklon
sekuensi DNA tertentu yang dapat digunakan sebagai pengganti pelacak DNA
dalam analisis PCR-RFLP. Sekuensi DNA yang sudah diisolasi dipotongdengan
berbagai enzim restriksi, guna melihat keragaman melalui keberadaan
recognition site yang memberikan ukuran potongan DNA yang berbeda-beda.
Taq Polymerase DNA
Primer
Nukleotida
Untuk genotif 1 Untuk genotif 2 Tabung reaksi yang berisi
larutan penyangga
Elektroforesis pada
agarose gel
keragaman genetik berdasarkan sekuen DNA yang diisolasi pada berbagai jenis
tanaman yang dianalisis.
Pada saat media contoh dipanaskan hingga suhu 94o C, atau pH media
dibuat alkalis, maka DNA tersebut menjadi asam, sehingga pHnya dibawah 7, jika
ditambahkan basa (NaoH) maka media itu menjadi alkalis dan DNA mengalami
denaturasi. Pada saat suhu diturunkan hingga ke 50 oC atau pH diturunkan
menjadi asam, maka kedua utas DNA kembali berpasangan. Peristiwa ini disebut
renaturasi (re = kembali). Jika dalam media terdapat DNA lain atau RNA, dan
urutan basa mereka komplemen, maka akan terjadi perpasangan atau hibrid.
Adapun proses amplifikasi PCR adalah seperti pada Gambar 4.
Gambar 4. Proses amplifikasi PCR
(www.users.ugent.be/~avierst/principle/seq.htm)
(Gailing et al., 2003 )
Step 3 : extension
PCR:
polymerase chain reaction
Step 1 : denaturation
PCR-RFLP
(Polymerase Chain Reaction - Restriction Fragment Length Polymorphisms)
PCR-RFLP adalah penanda dominan yang merestriksi DNA secara
spesifik pada lokasi tertentu yang dikenalnya dengan enzim restriksi
endonuklease (Park dan Moran, 1995). Enzim Restriksi ini akan mengenali
sekuen tertentu dan memutus DNA jika bertemu dengan situs yang dikenalnya
dan menghasilkan sejumlah fragmen DNA.
Polimorfisme PCR-RFLP muncul karena adanya basa yang mengalami
substitusi, penambahan, pengurangan dan perpindahan (translokasi) pada genom
DNA. Perubahan tersebut menyebabkan perbedaan ukuran dari fragmen
restriksi yang dicerna oleh enzim restriksi tertentu. Fragmen yang dihasilkan
oleh enzim restriksi dapat dipilah-pilah dengan elektroforesis.
DNA genom yang dipotong oleh enzim restriksi, akan menghasilkan
beratus-ratus atau beribu-ribu potongan DNA. Untuk mempelajari pola
pemotongan DNA yang berasal dari lokus tertentu dalam kromosom, maka
ratusan atau ribuan potongan tersebut harus :
(a) Dipisahkan berdasarkan ukuran dengan elektroforesis gel agarose
(b) Potongan DNA tertentu yang diinginkan harus dapat dibedakan dari
populasi potongan DNA dengan ukuran yang sama dengan melakukan
hibridisasi menggunakan pelacak DNA (DNA probe).
Pelaksanaan analisis PCR-RFLP memerlukan penggunaan bahan
radioaktif atau bahan non-radioaktif tertentu untuk memberi label pada pelacak
DNA. Bahan ini harganya masih relatif mahal. Selain itu, bahan radioaktif juga
merupakan polutan yang berbahaya bagi lingkungan. Sebaliknya, bahan
non-radioaktif walaupun tidak berbahaya, tetapi kemampua n dan sensitifitasnya
untuk mendeteksi sekuensi DNA kopi tunggal masih belum efisien. Pemberian
label pada pelacak DNA dilakukan denganteknik nick translation atau dengan
teknik random priming DNA labelling. Dengan teknik nick translation, salah
satu untaian dari DNA yang akan dilabel diputus diberbagai titik dengan
proses sintesis DNA yang baru terjadi, nukleotida yang telah diberi label ikut
bergabung dalam untaian DNA baru yang disintesis.
Pelacak DNA yang dipakai dalam penelitian PCR-RFLP dapat berupa
DNA yang berasal dari sekerabat dengan spesies tanaman yang diteliti (pelacak
homolog), atau berasal dari tanaman yang tidak sekerabat (pelacak heterolog).
pelacak heterolog biasanya lebih sulit dalam pemakaiannya karena biasanya ada
homologi sekuensi pelacak DNA dengan DNA tanaman relatif kecil. Akibatnya,
penggunaan pelacak DNA heterolog akan memberikan komplementasi yang
kurang stabil antara DNA tanaman dan pelacak DNA. Pelacak heterologous dari
DNA yang bersifat sangat konservatif (selalu sama untuk berbagai spesies
tanaman) misalnya bagian gen small sub unit dari RUBISCO (Ribulosa Bifosfat
METODE PENELITIAN
Tempat dan waktu penelitian
Pengambilan contoh daun dilakukan pada populasi S. leprosula di 13
lokasi di Indonesia (Tabel 1). Penelitian elektroforesis dan analisis DNA
dilakukan di Laboratorium Silvikultur Fakultas Kehutanan Institut Pertanian
Bogor. Penelitian dilaksanakan pada bulan Maret-Juni 2005 untuk pengambilan
data primer selanjutnya bulan Juli-September 2005 untuk studi pustaka dan
analisis data.
Bahan dan Alat Penelitian
Bahan yang digunakan untuk penelitian ini berupa daun S. leprosula yang
telah kering. Selain itu juga digunakan silika gel, nitrogen cair, bahan-bahan kimia
untuk membuat buffer untuk proses ekstraksi DNA, PCR, dan pemotongan DNA,
agaros, serta ethidium bromida (EtBr).
Alat-alat yang digunakan untuk pengambilan contoh di lapangan meliputi:
tally sheet, label, alat tulis, plastik klip, gunting. Alat-alat yang digunakan untuk
elektroforesis dan analisis DNA di laboratorium meliputi: mortar dan pestel,
sarung tangan, pipet, pipet mikro, sentrifugasi, vortex, spatula, gelas piala, gelas
ukur, koleksi tabung, cetakan gel, bak elektroforesis, tray, microwave, power
supply, pH meter, gelas piala, gelas ukur, timbangan analitik, pengaduk magnet,
lemari pendingin, water bath, mesin PCR, ultraviolet transiluminator, kamera
digital.
Prosedur Penelitian Optimasi metode
Tahapan optimasi metode dilakukan untuk menjamin munculnya pita
dengan resolusi yang tinggi. Tahapan optimasi metode ini meliputi 3 proses
utama, yaitu ekstraksi DNA, amplifikasi PCR dan pemotongan cpDNA. Optimasi
ekstraksi DNA dari daun dimulai dari penentuan ukuran daun yang diekstrak,
komposisi bahan ekstraksi, suhu dan kepekatan gel agaros pada proses
cpDNA serta primer yang akan digunakan, pengaturan suhu pada thermocycler
sesuai dengan primer yang digunakan, suhu dan kepekatan gel agaros pada proses
elektroforesis. Optimasi pada proses pemotongan DNA yang dilakukan adalah
mencari komposisi bahan yang digunakan untuk pemotongan DNA serta mencari
jenis enzim restriksi yang akan digunakan, kondisi arus, tegangan dan persentase
agaros pada elektroforesis. Skema prosedur penelitian menggunakann penanda
PCR-RFLP dapat dilihat pada Gambar 5.
Gambar 5. Bagan alur penelitian di laboratorium
Pengambilan Contoh
Pengambilan contoh dilakukan di 13 populasi S. leprosula (Tabel 1),
dimana pada masing- masing populasi diambil 5 contoh (Gambar 6). Pengambilan
contoh daun dilakukan dengan mengambil daun ke 2 atau ke 3 dari pucuk dengan
jumlah sekitar 5 daun yang berada di lokasi dan dimasukkan ke dalam plastik klip,
kemudian dalam plastik tersebut dimasukkan silika geldengan perbandingan berat
daun contoh dan silika gelsebesar 1 : 5. Silika gel yang sudah berubah warna
diganti dengan silika gel yang baru sampai contoh daun menjadi kering. Pada
plastik klip diberi label yang meliputi: nomor pohon diameter, tinggi pohon dan Ekstraksi DNA
Ya Tidak
PCR
Ya Tidak
lokasi. Selain itu juga dilakukan pengambilan daun yang digunakan untuk
herbarium untuk keperluan identifikasi jenis.
Tabel 1. Lokasi pengambilam contoh S. leprosula
Perkiraan Lokasi No
Batch Lokasi Provinsi Garis Bujur Garis Lintang
1 Haurbentes, Bogor Jawa Barat 106041’ - 107042’ BT 6054’-7054’ LU 9 Kebun Percobaan Darmaga,
Ekstraksi DNA
Metode yang digunakan untuk ekstraksi DNA mengikuti prosedur yang
dikeluarkan oleh QIAGEN dengan menggunakan DNeasy Plant Mini Kit untuk
isolasi jaringan tanaman. Ekstraksi DNA ini meliputi tiga tahapan, yaitu tahapan
prespitasi, pencucian dan elusi (Lampiran 1).
Tahap pertama yaitu tahapan prespitasi dimulai dengan menggerus contoh
daun meranti berukuran 2 x 1 cm yang ditambahkan nitrogen cair dengan
menggunakan mortar dan pestel untuk mendapatkan serbuk. Serbuk kemudian
dipindahkan ke dalam tabung yang berkuran 2 ml. Tambahkan 400 µl buffer AP1
dan 4 µl RNAse A (100 mg/ml) yang berfungsi untuk menghilangkan RNA dalam
larutan. Larutan tersebut kemudian di kocok dengan menggunakan vortex.
Larutan diinkubasi di dalam water bath selama 10 menit dengan suhu 65oC,
kemudian dikocok 2 - 3 kali selama inkubasi dengan membalikkan tabung.
Ditambahkan 130 µl buffer AP2, vortex, kemudian diinkubasikan ke dalam es
selama 5 menit, kemudian di sentrifugasi selama 5 menit dengan kecepatan penuh.
dimasukkan cairan ke dalam QIAShedder spin column yang terdapat pada koleksi
tabung 2 ml dan dilakukan sentrifugasi selama 2 menit denga n kecepatan
maksimum. Larutan yang mengalir lewat fraksi dari dipindahkan ke tabung yang
baru (tanpa penambahan). 1,5 volume buffer AP3/E (buffer sebelumnya
ditambahkan dengan etanol) pada larutan bersih ditambahkan dan dicampur
dengan pemipetan. dimasukkan 650 µl larutan ke DNeasy mini spin column yang
di letakkan di koleksi tabung 2 ml, dilakukan sentrifugasi dengan kecepatan 8000
rpm kemudian cairan yang melewati fraksi dibuang.
Tahapan kedua yaitu tahapan pencucian (washing) dimulai dengan
menempatkan DNeasy column pada tabung koleksi 2 ml yang baru , kemudian
ditambahkan 500 µl buffer AW pada DNeasy column dan sentrifugasi selama 1
menit pada kecepatan 8000 rpm. Cairan yang melewati fraksi dibuang, kegiatan
ini dilakukan dua kali, kemudian disentrifugasi selama 2 menit dengan kecepatan
Tahapan ketiga yaitu tahapan elution dilakukan dengan menyimpan
DNeasy column pada 1,5 atau 2 ml tabung sentrifugasi dan pipet 100 µl buffer AE
hangat (65oC) ke dalam membran Dneasy dan dilakukan inkubasikan selama 5
menit pada suhu ruangan, kemudian disentrifugasi selama 1 menit dengan
kecepatan 8000 rpm, tahapan ini dilakukan dua kali, sehingga akan dihasilkan
DNA hasil ekstraksi sebanyak 200 µl.
DNA hasil ekstraksi kemudian dilakukan uji kualitas dengan
menggunakan teknik elektroforesis agaros 1%. Gel ini dibuat dengan melarutkan
agaros sebanyak 2,5 µl ke dalam 250 µl larutan TAE (tris acetate with EDTA).
Kemudian larutan dipanaskan di dalam microwave sampai mendidih. Larutan gel
dibiarkan sampai hangat (± 50oC) kemudian dituangkan ke dalam cetakan gel
dengan ketebalan 5 mm. Cetakan gel tersebut telah dipasang sisir/comb yang
berfungsi untuk membuat cetakan sumur gel/well elektroforesis. Gel didinginkan
sampai membeku. Kemudian sisir/comb dicabut dan gel beserta cetakannya
dimasukkan ke dalam bak elektroforesis yang berisi buffer elektroforesis yaitu
buffer TAE sebanyak 2300 ml. DNA hasil ekstraksi sebanyak 15 µl ditambah
3,75 µl bahan pewarna (blue juice) dimasukkan ke dalam sumur-sumur
elektroforesis. Setelah itu bak elektroforesis ditutup dan dialiri listrik dengan
tegangan 25 volt selama 3 jam.
Gel yang sudah dielektroforesis dilakukan pewarnaan dengan
merendamkan gel di dalam larutan ethidium bromida (EtBr) 25 µl dan aquadest
500 ml selama 1 jam. Kemudian dideteksi dengan mengunakan ultraviolet
transiluminator.
Amplifikasi PCR
DNA hasil ekstraksi diamplifikasi dengan menggunakan mesin PCR MJ
Reseach PTC-100 Peltier Thermal cycler. Primer yang digunakan adalah primer
universal (hampir terdapat di semua tumbuhan) yaitu petB, psaA, psbA, rbcL dan
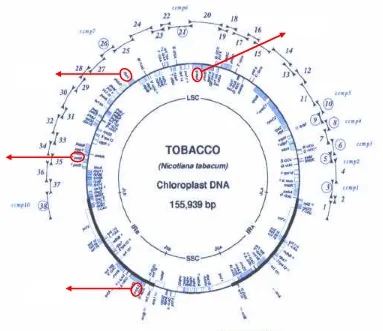
Gambar 7. Letak petB, psaA, trnLF dan rbcL pada peta plasmid cpDNA Nicotiana tabacum
Urutan nukleotida dari masing- masing primer adalah sebagai berikut :
petB : 5’-TGGGGAACTACTCCTTTGAT-3’
5’-CCCGAAATACCTTGCTTACG-3’
psaA : 5’-AAGAATGCCCATGTTGTGGC-3’
5’-TTCGTTCGCCGGAACCAGAA-3’
rbcL : 5’-TGTCACCAAAAACAGAGACT-3’
5’-TTCCATACTTCACAAGCAGC-3’
trnLF : 5’-CGAAATCGGTAGACGCTACG-3’
Primer yang akan digunakan sebelumnya dilakukan pengenceran terlebih
dahulu. Pengenceran dilakukan dengan cara mengambil primer pekat dengan
konsentrasi 50 µM sebanyak 20 µl kemudian ditambahkan H2O sebanyak 180 µl.
Konsentrasi akhir primer akan menjadi 5 µM. Komposisi bahan-bahan yang
digunakan dalam proses amplifikasi ini tersaji pada tabel 2.
Tabel 2. Komposisi bahan-bahan yang digunakan untuk PCR
Komponen Volume
Lautan PCR untuk setiap contoh merupakan campuran dari dari berbagai
komponen seperti pada Tabel 2. kemudian ditambahkan 1,9 µl destilled water
hingga volume larutan mencapai 15 µl. Larutan kemudian diaduk dengan
menggunakan vortex kemudian disentrifugasi. Larutan tersebut kemudian di
masukan kedalam mesin PCR yang sudah di program dengan pengkondisian suhu
dan waktu. Seperti pada Tabel 3.
Tabel 3. Pengkondisian suhu dan waktu pada mesin PCR untuk primer petB, psaA, trnLFdan rbcL
Hasil PCR kemudian di uji dengan menggunakan gel agaros dengan
kepekatan 2%, yang dibuat dengan melarutkan 0,4 mg agaros dalam 40 ml TAE.
Larutan hasil PCR sebanyak 3 µl dicampurkan dengan larutan pewarna (brom
sedikit atau sama dengan hasil ekstraksi maka harus dilakukan PCR ulang dengan
komposisi DNA yang lebih encer. Pengenceran ini dilakukan karena sifat DNA
dari Meranti memiliki kadar fenol yang tinggi. Kadar fenol yang tinggi ini dapat
menghambat daya kerja primer.
Analisis PCR - RFLP
Analisis polimorfik dilakukan dengan teknik PCR-RFLP untuk melihat
jumlah basa antar fragmen. DNA kloroplas hasil PCR dipotong dengan
menggunakan enzim restriksi untuk analisis keragaman genetik. Dibawah ini
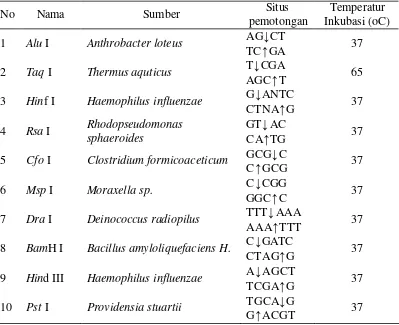
adalah jenis-jenis enzim restriksi yang digunakan dalam penelitian ini.
Tabel 4. Jenis enzim restrksi yang digunakan utuk memotong DNA
No Nama Sumber Situs
Larutan yang digunakan untuk pemotongan ini adalah 0,5 µl enzim
restriksi, 1µl bufer, 5µl bidest steril, 5 µl DNA hasil PCR. Larutan ini kemudian
diinkubasi di dalam waterbath pada suhu 370C selama 1 jam. Hasil pemotongan
dengan enzim restriksi kemudian diuji dengan elektroforesis agaros dengan
tegangan 100 volt selama 45 menit untuk memastikan fragmen DNA terpisah
dengan sempurna. Hasil pemotongan ini kemudian dideteksi dengan
menggunakan ultraviolet transiluminator dan difoto dengan menggunakan
kamera digital untuk dinalisis dikomputer.
Uji polimorfisme
Pengujian polimorfisme dilakukan dengan menggunakan teknik
PCR-RFLP. Pengujian ini dilakukan untuk mencari variasi DNA kloroplas pada S.
leprosula yang didasarkan pada pola pemotongan pita yang dihasilkan dari
penggunaan beberapa kombinasi perlakuan. Komb inasi perlakuan yang diuji
dalam penelitian ini menggunakan 4 macam primer dan 10 macam enzim
restriksi.
Hasil pengujian pada cpDNA dikatakan polimorfik apabila mempunyai
pola pita yang dihasilkan mempunyai sekurang-kurangnya lebih dari satu variasi,
Sedangkan cpDNA dikatakan monomorfik jika tidak memperlihatkan adanya
variasi pada pemotongan pola pita. Selanjutnya, untuk kombinasi perlakuan yang
memberikan hasil polimorfik dilakukan analisis lebih lanjut untuk melihat variasi
genetik pada level inter dan intra populasi, sekaligus untuk melihat
kekerabatannya.
Analisis Data
Data analisis hasil elektroforesis yaitu situs restriksi yang berupa pola pita
DNA yang digunakan untuk menghitung haplotipe. Pola pita yang muncul
(positif) diberi nilai 1 (satu) dan pola pita yang tidak muncul (negatif) diberi nilai
0 (nol). Hasil perhitungan kemudian dianalisis untuk mengetahui frekuensi dan
keragaman haplotype dalam jenis dan antar populasi S. leprosula dengan
menggunakan sofware ARLEQUIN Ver 2.000 (Schneider et al 2000), untuk
mengetahui jarak genetik antar populasi sofware GSED ver 1.1 (Gillet, 2002).
Situs restriksi digunakan juga untuk melihat kemiripan (similiarity) antar
haplotipe serta untuk melihat dendogram dengan menggunakan metoda UPGMA
(Unweighted Pair-Group Method with Averaging) dengan sofware NTSYS Ver
HASIL DAN PEMBAHASAN
Ekstraksi DNA
Analisis DNA dimulai dengan melakukan ekstraksi DNA yang berasal dari
jaringan daun. Alasan pemilihan jaringan daun, karena jaringan ini lebih mudah
diekstrak dibandingkan dengan jaringan yang lain, seperti akar, kayu dan jaringan
lainnya. Ekstrasi DNA dilakukan berdasarkan prosedur yang dikeluarkan oleh
Qiagen dengan menggunakan metode Dneasy Plant Mini Kit. Ekstraksi DNA ini
meliputi 3 tahapan penting, yaitu presipitasi (presipitation), pencucian (washing)
dan ellution. Contoh hasil ekstraksi DNA ditunjukkan pada agaros gel yang dapat
divisualisasi di bawah sinar ultraviolet (Gambar 8).
Gambar 8. Hasil Ekstraksi DNA S. leprosula
DNA hasil ekstraksi merupakan DNA total, yang terdiri dari DNA inti,
DNA mitokondria dan DNA kloroplas. Untuk menganalisis cpDNA maka
dilakukan proses amplifikasi PCR dengan menggunakan primer universal
kloroplas yang hanya dapat mengamplifikasi secara spesifik fragmen- fragmen di
cpDNA saja. CpDNA dipelajari karena lebih kecil, lebih mudah diisolasi dan
strukturnya lebih sederhana dibandingkan dengan DNA mitokondria (mtDNA)
(Banks dan Birky,1985).
DNA ekstraksi digunakan sebagai cetakan template dalam reaksi PCR.
terlebih dahulu. Hal ini dilakukan karena DNA S. leprosula mempunyai
kandungan fenol yang tinggi. Kandungan fenol yang tinggi ini dapat
menyebabkan terhambatnya kerja primer, sehingga hasil amplifikasi kurang
maksimal bahkan tidak berhasil (tidak ada pita). Pengenceran dilakukan
berdasarkan pengamatan visual dari terang tidaknya pita DNA hasil ekstraksi.
Berdasarkan hasil optimasi, pita yang terang dilakukan pengenceran sebanyak 40
kali, artinya 1 µl DNA ekstraksi diencerkan dengan menambahan H2O steril
sebanyak 39 µl. Sedangkan untuk DNA yang kurang terang dilakukan
pengenceran sebanyak 20 kali (1 µl DNA ekstrasi ditambah 19 µl H2O steril) dan
10 kali (1 µl DNA ekstraksi ditambah 9 µl H2O steril) (Gambar 7). DNA hasil
pengenceran ini yang akan dijadikan sebagai template dalam proses amplifikasi
PCR.
Gambar 9. Elektrophoresisis cpDNA dengan berbagai pengenceran DNA dari
S. leprosula
Amplifikasi dengan PCR
Keberhasilan amplifikasi dengan PCR lebih didasarkan pada kesesuaian
primer yang digunakan, efisiensi dan optimasi pelaksanaan PCR. Primer yang
tidak spesifik dapat menyebabkan teramplifikasinya daerah lain dalam genom
yang tidak dijadikan sasaran, atau sebaliknya tidak ada daerah genom yang
teramplifikasi. Optimasi PCR juga diperlukan untuk menghasilkan karakter yang
diinginkan. Optimasi ini menyangkut suhu denaturasi dan annealing DNA dalam
mesin PCR. Suhu denaturasi yang rendah, dapat menyebabkan belum terbukanya
DNA utas ganda sehingga tidak dimungkinkan terjadinya polimerisasi DNA baru.
Proses penempelan primer pada utas DNA yang sudah terbuka memerlukan suhu
optimum, sebab suhu yang terlalu tinggi dapat menyebabkan amplifikasi tidak
terjadi, atau sebaliknya suhu yang terlalu rendah menyebabkan primer menempel
pada sisi lain genom yang bukan sisi homolognya. Akibatnya dapat teramplifikasi
banyak daerah tidak spesifik dalam genom tersebut. Suhu penempelan ini
ditentukan berdasarkan primer yang digunakan dan dipengaruhi oleh panjang dan
komposisi primer. Faktor- faktor yang mempengaruhi jalannya amplifikasi PCR
diantaranya adalah karakter individual contoh DNA, komposisi premiks PCR,
kotoran, suhu dan waktu masing- masing fase, pemilihan primer spesifik dan
jumlah siklus amplifikasi.
Penggunaan primer universal petB, psaA, trnLF dan rbcL dapat
mengamplifikasi cpDNA S. leprosula dengan baik (Indrioko, 2005). Panjang
fragmen hasil amplifikasi PCR berkisar 1700 bp dengan menggunakan primer
petB, 2100 bp dengan primer psaA, 1100 bp dengan primer trnLF dan 1300
dengan menggunakan primer rbcL yang ditunjukkan melalui elektroforesis gel
Gambar 10. Elektroforegram hasil PCR cpDNA dengan berbagai primer : (a) petB, (b) psaA, (c) trnLF, (d) rbcL, M = marker bond
Uji Polimorfisme
Uji polimorfisme bertujuan untuk mencari kombinasi perlakuan dari
primer dan enzim restriksi yang menghasilkan pola pemotongan yang beragam
(variasi). Dalam uji ini cpDNA hasil amplifikasi PCR dipotong dengan
menggunakan enzim restriksi yang dikenal sebagai teknik PCR-RFLP. Teknik ini
sangat sederhana, cepat dan ekonomis, memiliki kisaran 50-3000 bp yang dapat
Pada prinsip penggunaan primernya, PCR-RFLP mirip dengan RAPD
(random amplied polymorphic DNA). Namun perbedaannya untuk melihat
polimorfisme dalam genom organisme, pada PCR-RLFP digunakan juga enzim
pemotong tertentu. Enzim ini bersifat spesifik, sehingga enzim ini akan
memotong situs tertentu yang dikenali sesuai dengan urutan basanya. Situs enzim
pemotong dari genom suatu kelompok organisme yang kemudian berubah karena
mutasi titik atau berpindah karena penyusunan kembali gentiknya (genetic
rearrangement) dapat menyebabkan situs tersebut tidak dikenali lagi oleh enzim,
atau enzim restriksi akan memotong daerah lain yang berbeda. Proses ini
menyebabkan terbentuknya fragmen- fragmen DNA yang berbeda ukurannya dari
satu organisme ke organisme lainnya. Polimorfisme ini selanjutnya digunakan
untuk membuat dendrogram untuk melihat kekerabatan kelompok.
RFLP dapat diamati bila sebagian besar dari DNA dipotong (dicerna)
menjadi fragmen- fragmen atau potongan-potongan kecil. Potongan-potongan
tersebut dapat dipisahkan secara elektroforesis. Variasi cpDNA ini ditunjukkan
oleh ada tidaknya situs pengenal (recognition sites) untuk enzim spesifik
(Finkeldey, 2005).
cpDNA dihasilkan dari amplifikasi PCR dengan menggunakan 4 macam
primer. Hasil amplifikasi tersebut kemudian dipotong dengan menggunakan 10
macam enzim restriksi. Enzim restriksi yang digunakan dalam penelitian ini
adalah Alu I, HinF I, Rsa I, Taq I, Cfo I, Msp I, Dra I, BamH I, Hind III dan Pst I.
Hasil pemotongan kemudian diinterpretasikan pada gel agaros melalui teknik
Gambar 11. Elektroforegram hasil uji polimorfisme : (a) psaA-pst I (monomorfik), (b) petB-Hinf I (monomorfik), (d) rbcL-Alu I (polimorfik), M= marker bond
Gambar 11a menujukkan bahwa dari hasil analisis dengan menggunakan
primer psaA dan dipotong dengan enzim restriksi Pst I tidak terjadi pemotongan.
Hal ini disebabkan tidak ada urutan basa TGCA↓G yang dikenali oleh enzym
Pst I sehingga tidak terjadi pemotongan. Keadaan dimana tidak adanya
pemotongan dan tidak ada variasi dinamakan monomorfik. Pada Gambar 11b
yang menggunakan primer petB dan dipotong oleh Hinf I, telah terjadi
pemotongan, karena terdapat urutan basa G↓ANTC, namun dari hasil pemotongan
dinamakan monomorfik. Gambar 11b yang menggunakan primer rbcL dan enzim
restriksi Alu I menghasilkan pola pita yang polimorfik, selain ada pemotongan
juga ada variasi dari pola potongan tersebut. Hasil uji polimorfisme secara
keseluruhan dari kombinasi perlakuan yang diuji disajikan pada Tabel 5
Tabel 5. Hasil uji polimorphisme
Primer
Hasil uji polimorfik melalui pemotongan dengan menggunakan sepuluh
enzim restriksi menunjukkan bahwa hampir di semua perlakuan mempunyai pola
pita yang sama (monomorfik). Pola pita yang sama tersebut menunjukkan tidak
terdapatnya variasi DNA dengan menggunakan perlakuan tersebut. Tidak
terdapatnya pemotongan, berarti tidak ditemukannya urutan basa yang dapat
dikenali oleh enzim restriksi tertentu. Hal ini menujukkan tidak adanya variasi
cpDNA. Pola pita yang bervariasi (polimorfik) ditemukan dengan mengunakan
kombinasi perlakuan primer rbcL yang dipotong dengan menggunakan enzim
restriksi Alu1 yang mengenali situs AG↓CT dan menghasilkan dua tipe
pemotongan (polimorf), yaitu menjadi 3 potongan dan 4 potongan (Gambar 12).
2.642 bp
M Haur Bentes Tering Asialog Pasir Manyang Bukit Tigapuluh Nanjak Makmur Sari Bumi Kusuma ITCI Darmaga Bukit Bangkirai Sumalindo Carita 1 Carita 2
M Haur Bentes Tering Asialog Pasir Manyang Bukit Tigapuluh Nanjak Makmur Sari Bumi Kusuma ITCI Darmaga Bukit Bangkirai Sumalindo Carita 1 Carita 2
Gambar 12. Elektroforegram hasil pemotongan cpDNA dengan menggunakan kombinasi perlakuan rbcL-Alu 1
Haplotipe yang ditemukan pada kombinasi rbcL- Alu 1 dalam suatu
populasi maupun antar populasi dapat disebabkan akibat terjadinya pergantian,
penambahan atau hilangnya basa tertentu pada urutan basa cpDNA S. leprosula
perbedaan urutan pasangan basa pada ind ividu yang mempunyai tipe pemotongan
basa yang berbeda. Fenomena ini mengindikasikan terdapatnya keragaman
genetik di dalam populasi dan antar populasi.
Setelah dilakukan uji polimorfik, maka dilakukan uji lanjutan pada
perlakuan yang mempunyai pola pita yang bervariasi, yaitu pada kombinasi
perlakuan rbcL dan Alu I, karena kombinasi perlakuan ini mampu membedakan
individu S. leprosula. Uji lanjutan berupa pengecekan ulang untuk memastikan
bahwa contoh yang diteliti adalah S. leprosula. Pengecekan ini menggunakan
herbarium dan dilakukan di Herbarium Bogoriense. Apabila hasil pengecekan
menyatakan bahwa contoh tersebut adalah S. leprosula, kemudian dilakukan
analisis. Analisis dimulai dengan memberikan nilai pada situs pemotongan yang
berupa pita-pita DNA berdasarkan ada tidaknya situs pemotongan. Hasil analisis
kemudian ditransformasikan dengan menggunakan angka biner, hasil transformasi
tertera pada Lampiran 3. Nilai tersebut kemudian diolah dengan bantuan program
komputer untuk mencari variasi cpDNA S. leprosula di Indonesia.
Keragaman intra populasi
Tabel 6. menunjukkan hasil dari pemotongan cpDNA dengan
menggunakan rbcL-Alu I yang teridentifikasi berdasarkan PCR-RFLP pada 65
contoh di 13 populasi yang menghasilkan 2 haplotipe Tabel 6. Keseluruhan
elektroforegram hasil analisis PCR dengan menggunakan primer rbcL dan
pemotongan dengan Alu I tertera pada Lampiran 2.
Tabel 6. Haplotipe yang teridentifikasi pada 65 individu S. leprosula berdasarkan PCR-RFLP terdapat dua haplotipe.
rbcL-Alu 1
Perbedaan situs restriksi dapat diasumsikan sebagai kejadian mutasi titik
(point mutation) yang menyebabkan berubahnya urutan DNA sehingga tidak
dikenali oleh enzim. Dari Gambar 12 dapat diketahui bahwa haplotipe 2 berasal
Dimana fragmen dengan panjang 350 bp pada haplotipe 1, terpotong menjadi 2
fragmen pada haplotipe 2, yaitu menjadi 220 bp dan 130 bp. Adapun komposisi
penyebaran haplotipe (relatipe frequencies) tersaji pada Tabel 7
Tabel 7. Frekuensi haplotipe dari 13 populasi
Haplotipe Hb Tr As Pm Bt Nm Sb It Dr Bb Sm C1 C2
1 1.0 1.0 0.4 1.0 0.8 1.0 1.0 0.4 1.0 1.0 1.0 1.0 1.0 2 0.0 0.0 0.6 0.0 0.2 0.0 0.0 0.6 0.0 0.0 0.0 0.0 0.0 Ket : Hb (Haur Bentes), Tr (Tering), As (asialog), Pm (Pasir Mayang), Bt (Bukit Tigapuluh), Nm
(Nanjak Makmur), Sb (Sari Bumi Kusuma), It (PT. ITCI), Dr (Darmaga), Bb (Bukit bangkirai), Sm (Sumalindo), C1 (Carita 1) dan C2 (Carita 2)
Ditinjau dari kondisi penyebaran haplotipe S. leprosula dari tiap populasi
(Tabel 7 ) diketahui bahwa Haplotipe 1 merupakan haplotipe yang dominan dan
tersebar di seluruh populasi, sedangkan Haplotipe 2 hanya ditemukan di tiga
populasi, yaitu di PT. Asialog dan TN. Bukit Tigapuluh di pulau Sumatera, serta PT. ITCI Karya Utama di pulau Kalimatan, sedangkan di pulau Jawa tidak
ditemukan Haplotipe 2. Hal ini menujukkan adanya keragaman dalam populasi
(intra populasi) di ketiga populasi tersebut yang ditunjukkan oleh adanya variasi
situs pemotongan pada deretan nukleotida yang teramplifikasi.
Terdapatnya Haplotipe 2 di Kalimantan dan Sumatera diduga terjadi
karena pada jaman sebelum glasial pulau Kalimantan dan Sumatera merupakan
satu daratan. Setelah terjadinya glasial (pencairan es) daratan ini terpisah menjadi
pulau Kalimantan dan Sumatra, kejadian ini menurut Polunin (1990) terjadi pada
jaman Pleistosen sekitar 1 juta tahun yang lalu.
Hasil pene litian menunjukkan tidak ditemukannya haplotipe spesifik yang
dapat dijadikan sebagai penanda (fingerprint) yang dapat diasosiasikan sebagai
asal-usul geografis tertentu di Indonesia. Haplotipe 2 tidak dapat dijadikan
sebagai penanda karena terdapat di 3 populasi di pulau Sumatera dan Kalimantan. Khusus untuk pulau Kalimatan, rbcL-Alu I dapat dijadikan sebagai penanda,
karena hanya ditemukan di satu populasi, yaitu di PT. ITCIKU.
Keragaman haplotipe pada intra spesies yang terdeteksi cukup rendah. Hal
ini menunjukkan bahwa cpDNA sangat konservatif. Sifat konservatif cpDNA ini
terjadi karena pada angiospermae hanya diturunkan oleh induk betina saja,
sehingga menyebabkan aliran gen berupa migrasi benih yang terjadi sangat rendah
dan mutasi yang terjadi juga sangat rendah. Sesuai dengan penelitian yang
dilakukan Indrioko (2005) tidak menemukan perbedaan variasi cpDNA pada S.
Gambar 13. Penyebaran cpDNA S. leprosula di Indonesia berdasarkan penanda PCR-RFLP dengan menggunakan rbcL-Alu1
Keragaman inter populasi
Berdasarkan data keragaman situs restriksi kemudian dilakukan analisis
variasi molekuler (Analysis of Molekular Variance, AMOVA) untuk melihat
keragaman akibat sumber-sumber keragaman dalam populasi dengan bantuan
program komputer Arlequin dari Schneider et al. (2000)
Tabel 8. Hasil perhitungan AMOVA berdasarkan 65 individu yang berasal dari 13 populasi
Keterangan: Derajat bebas (db), korelasi random pairs allel didapat dari nilai relatif grup dengan seluruh populasi (FCT), korelasi random pairs allel didapat dari nilai relatif populasi
dengan seluruh grup (FSC), korelasi random pairs allel didapat dari nilai relatif
populasi dengan seluruh populasi (FST), tidak berbeda nyata (ns) dan berbeda nyata
(*)
Keragaman antar pulau memberikan hasil yang tidak berbeda nyata,
sedangkan keragaman antar populasi dalam pulau dan keragaman dalam pulau
memberikan hasil yang berbeda nyata.
Hasil analisis ragam molekuler (Tabel 8) menunjukkan bahwa ragam
dalam populasi menyumbangkan persentase ragam tertinggi (62.03%) terhadap
total ragam dan berbeda nyata, kemudian diikuti dengan persentase ragam pada
level antar populasi dala m pulau (40.82%) dan antar pulau (-2.85%). Nilai negatif
yang teramati pada level antar pulau mengindikasikan contoh populasi pada
tiap-tiap pulau tidak berbeda.
Tabel 9. Matrik signifikasi nilai (P value) pada level 0.05
Hb As Tr Pm Bt Nm Sb It Dr Bb Sm C1 C2 Ket : Hb (Haur Bentes), Tr (Tering), As (asialog), Pm (Pasir Mayang), Bt (Bukit Tigapuluh), Nm
(Nanjak Makmur), Sb (Sari Bumi Kusuma), It (PT. ITCI), Dr (Darmaga), Bb (Bukit bangkirai), Sm (Sumalindo), C1 (Carita 1) dan C2 (Carita 2), berbeda nyata antar populasi (+) dan tidak berbeda nyata antar populasi (-)
Pengelompokan genetik didasarkan pada jarak genetik. Jarak genetik
digunakan untuk mengukur perbedaan struktur genetik antar dua populasi.
Semakin kecil jarak genetik, semakin dekat kekerabatan genetiknya. Sebaliknya,
semakin besar jarak genetik, maka semakin jauh kekerabatan genetiknya. Nilai
jarak genetik berkisar antara 0 sampai 1. Nilai 0 dihasilkan dari perbandingan dua
populasi yang mempunyai variasi genetik yang sama (identik). Nilai maksimum 1
dicapai jika dua populasi tersebut tidak membagi apa pun tipe genetik.
Pengukuran jarak genetik pada penelitian ini dilakukan dengan bantuan
sofware GSED ver 1.1i (Gillet, 2002). Jarak genetik S. leprosula berdasarkan
Tabel 10. Jarak genetik S. leprosula berdasarkan analisis GSED
Hb Tr As Pm Bt Nm Sb It Dr Bb Sm C1 C2 Hb 0.00
Tr 0.00 0.00
As 0.60 0.60 0.00
Pm 0.00 0.00 0.60 0.00
Bt 0.20 0.20 0.40 0.20 0.00
Nm 0.00 0.00 0.60 0.00 0.20 0.00
Sb 0.00 0.00 0.60 0.00 0.20 0.00 0.00
It 0.60 0.60 0.00 0.60 0.40 0.60 0.60 0.00
Dr 0.00 0.00 0.60 0.00 0.20 0.00 0.00 0.60 0.00
Bb 0.00 0.00 0.60 0.00 0.20 0.00 0.00 0.60 0.00 0.00
Sm 0.00 0.00 0.60 0.00 0.20 0.00 0.00 0.60 0.00 0.00 0.00
C1 0.00 0.00 0.60 0.00 0.20 0.00 0.00 0.60 0.00 0.00 0.00 0.00
C2 0.00 0.00 0.60 0.00 0.20 0.00 0.00 0.60 0.00 0.00 0.00 0.00 0.00
Ket : Hb (Haur Bentes), Tr (Tering), As (asialog), Pm (Pasir Mayang), Bt (Bukit Tigapuluh), Nm (Nanjak Makmur), Sb (Sari Bumi Kusuma), It (PT. ITCI), Dr (Darmaga), Bb (Bukit bangkirai), Sm (Sumalindo), C1 (Carita 1) dan C2 (Carita 2)
Jarak genetik adalah untuk mengetahui perbedaaan genetik antara dua
populasi. Nilai jarak genetik mempunyai interval dari 0 sampai 1. Nilai 0 (nol)
mengindikasikan bahwa kedua populasi tersebut mempunyai genetik yang identik.
Sebaliknya, nilai 1 (satu) mengindikasikan bahwa kedua populasi tersebut
mempunyai genetik yang berbeda satu sam lainnya.
Berdasarkan jarak genetik dapat diketahui bahwa terdapat 10 populasi yang
identik, yaitu Haur Bentes, Tering, Pasir Mayang, Nanjak Makmur, Sari Bumi
Gambar 14. Dendogram populasi S. leprosula di Indonesia dengan penanda RFLP
Analisis klaster UPGMA (Unweighted Pair-Group Method with
Averaging) menunjukkan bahwa populasi S. leprosula di Indonesia terbagi
menjadi dua kelompok genetik. Kelompok satu terdiri dari dua sub kelompok,
sub kelompok pertama terdiri dari 10 populasi Haur bentes, Tering, Pasir Mayang,
Nanjak Makmur, Sari Bumi kusuma, Darmaga, bukit Bangkirai, Sumalindo,
Carita 1 dan Carita 2 yang mempunyai kesamaan genetik sesuai dengan analisis
PCR-RFLP dengan menggunakan rbcL-Alu 1. Sub kelompok dua dari kelompok
satu hanya terdapat satu populasi, yaitu TN. Bukit Tigapuluh. Sedangkan
kelompok dua diduduki oleh dua populasi, yaitu PT. Asiolog dan PT. ITCI Karya
Utama. TN. Bukit Tigapuluh mempunyai kekerabatan genetik yang lebih dekat
dengan kelompok yang tidak mempunyai Haplotipe 2, fenomena ini terjadi karena
sebaran haplotipe 1 di Bukit Tigapuluh yang lebih besar dibandingkan dengan
Haplotipe 2- nya, yaitu sebesar 80%. Sehingga populasi TN. Bukit Tigapuluh
masuk ke dalam kelompok 1. Individu- individu yang mengelompok dalam satu
Kelompok I
Kelompok II
Sub kelompok 1
kelompok mempunyai kerabat yang dekat satu sama lain dibandingkan dengan
individu- individu yang berada pada kelompok yang berbeda.
Konservasi Genetik Shorea leprosula
Pentingnya penelitian populasi genetik adalah untuk mengukur variasi
genetik pada level dalam spesies dan populasi. Data tersebut sangat penting, tidak
hanya untuk pengetahuan mekanisme evolusi tapi juga untuk pengembangan
program breeding tanaman dan binatang dalam rangka konservasi plasma nuftah
spesies langka. (Banks & Birky, 1985)
Mengapa konservasi genetik? Karena munurut Hadie (2001) :
1. Gen merupakan teori fundamental dari seleksi alam
2. Kesepakatan para pakar bahwa heterozigositas atau variasi genetik
berhubungan langsung dengan fitness
3. Gene pool merupakan informasi menyeluruh dari proses biologi
4. Variasi genetik diukur pada individu dan diasumsikan pada populasi
Suatu kenyataan bahwa pembalakan hutan yang semakin tinggi
menyebabkan meningkatnya kerusakan hutan, ditambah dengan bencana alam
seperti kebakaran hutan, merupakan penyebab utama rusaknya populasi S.
leprosula. Hal ini merupakan penyebab utama menurunnya keragaman genetik S.
lerposula. Keragaman genetik diasumsikan sebagai fitness, makin tinggi
keragamannya, makin tinggi peluang untuk survive.
Kecilnya populasi tersebut akan mengarah pada terjadinya founder effect,
dimana terjadinya silang dalam yang berakibat pada rusaknya keragaman genetik.
Oleh karena itu, konservasi genetik baik secara ex situ maupun in situ perlu
dilakukan untuk mempertahankan keragaman genetik dan sejarah evolusinya.
Undang-Undang Nomor 41 Tahun 1999 mengatakan bahwa pengelolaan
hutan dilakukan dengan mengoptimalkan aneka fungsi hutan yang me liputi fungsi
konservasi, fungsi lindung, dan fungsi produksi untuk mencapai manfaat
lingkungan, sosial, budaya, dan ekonomi, yang seimbang dan lestari. Implikasi
hasil penelitian variasi genetik S. leprosula terhadap program konservasi genetik
adalah melakukan konservasi terhadap genetik yang bervariasi rendah terlebih
dahulu, yaitu pada jenis yang mempunyai Haplotipe 2. Jadi konservasi genetik
dititikberatkan di PT. Asialog, TN. Bukit Tigapuluh dan PT. ITCI Karya Utama