INTEGRASI BASIS DATA DAN
PIPELINE SINGLE
NUCLEOTIDE POLYMORPHISM
UNTUK
PEMULIAAN TANAMAN KEDELAI
MIFTAKHUL HUDA
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Integrasi Basis Data dan Pipeline Single Nucleotide Polymorphism untuk Pemuliaan Tanaman Kedelai adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
ABSTRAK
MIFTAKHUL HUDA. Integrasi Basis Data dan Pipeline Single Nucleotide Polymorphism untuk Pemuliaan Tanaman Kedelai. Dibimbing oleh WISNU ANANTA KUSUMA dan HABIB RIJZAANI.
Upaya penemuan varietas unggul dengan cara pemuliaan tanaman secara konvensional membutuhkan waktu yang lama dan biaya yang besar. Kendala tersebut dapat diatasi dengan memanfaatkan informasi yang diperoleh dari analisis genom, yaitu berupa markah single nucleotide polymorphism (SNP) beserta asosiasi dengan fenotipenya. Selama ini identifikasi SNP dapat dilakukan menggunakan beberapa program komputer. Informasi SNP disimpan dalam fail berformat variant call format (VCF). Fail dengan format tersebut sulit dimengerti dan diolah oleh peneliti pemuliaan. Penelitian ini bertujuan mengintegrasikan perangkat lunak pipeline yang dihasilkan oleh penelitian sebelumnya dan basis data yang menyimpan informasi berupa SNP dan informasi pendukung lainnya yang terkait dengan identifikasi SNP, antara lain flanking area dan ID kandidat SNP yang terintegrasi dengan data referensi SNP kedelai dari NCBI. Selain itu, sistem ini juga dilengkapi fitur pencarian yang berbasis web untuk memudahkan peneliti pemuliaan menemukembalikan informasi kandidat SNP yang relevan.
Kata kunci: basis data, kedelai, pemuliaan tanaman, pipeline, SNP
ABSTRACT
MIFTAKHUL HUDA. Integration of Database and Single Nucleotide Polymorphism Pipeline for Soybean Breeding. Supervised by WISNU ANANTA KUSUMA and HABIB RIJZAANI.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Komputer
pada
Departemen Ilmu Komputer
INTEGRASI BASIS DATA DAN
PIPELINE
SINGLE
NUCLEOTIDE POLYMORPHISM
UNTUK
PEMULIAAN TANAMAN KEDELAI
MIFTAKHUL HUDA
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
Judul Skripsi : Integrasi Basis Data dan Pipeline Single Nucleotide Polymorphism untuk Pemuliaan Tanaman Kedelai
Nama : Miftakhul Huda NIM : G64100071
Disetujui oleh
Dr Wisnu Ananta Kusuma, ST MT Pembimbing I
Habib Rijzaani, MSi Pembimbing II
Diketahui oleh
Dr Ir Agus Buono, MSi MKom Ketua Departemen
PRAKATA
Puji syukur penulis panjatkan kepada Allah atas segala rahmat dan karunia-Nya sehingga penulis mampu menyelesaikan karya ilmiah ini. Penyusunan karya ilmiah ini juga tidak lepas dari bantuan berbagai pihak. Untuk itu penulis mengucapkan terima kasih kepada:
1 keluarga tercinta: Ibunda Alfiyah dan Ayahanda Chamzawi, serta ketiga kakak saya Zuliya, Umi, dan Fadil yang selalu memberikan doa, semangat, motivasi dan kasih sayang yang tiada henti,
2 Bapak Dr Wisnu Ananta Kusuma ST MT, dan Bapak Habib Rijzaani MSi selaku dosen pembimbing, terima kasih atas segala kesabaran, ilmu, saran dan motivasinya selama membimbing penulis,
3 Bapak M Abrar Istiadi, SKomp dan semua dosen Departemen Ilmu Komputer, terima kasih atas semua ilmu yang telah diberikan,
4 Resti, Risa, Ribut, Wahyu, dan Yogi, terima kasih atas semangat, motivasi, dan doanya, serta Mira Aisyah Romliyah, terima kasih atas kasih sayang, doa, semangat, dan kebersamaannya selama ini,
5 teman-teman satu bimbingan: Alfat, Delly, Gerry, Yuda, Bang Dan yang selalu saling mengingatkan dan memberi motivasi dalam penyusunan skripsi ini, 6 keluarga Himpunan Keluarga Rembang di Bogor dan teman-teman Pondok AA
yang selalu memberikan rasa kekeluargaan dan rasa kebersamaan,
7 teman-teman mahasiswa Ilmu Komputer angkatan 47, terima kasih atas doa, semangat, serta kebersamaannya selama ini,
8 semua pihak yang telah membantu dalam penyusunan karya ilmiah ini.
Semoga karya ilmiah ini dapat bermanfaat bagi dunia ilmu pengetahuan khususnya bidang ilmu komputer dan bioinformatika, serta menjadi inspirasi bagi penelitian selanjutnya
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 2
METODE 3
Pengumpulan Data 3
Analisis 4
Perancangan 4
Implementasi 4
Pengujian 4
HASIL DAN PEMBAHASAN 5
Analisis 5
Perancangan 8
Implementasi 10
Pengujian 13
SIMPULAN DAN SARAN 16
Simpulan 16
Saran 16
DAFTAR PUSTAKA 17
DAFTAR TABEL
1 Data informasi SNP varietas kedelai berformat VCF 3
2 Kebutuhan fungsional 7
3 Rincian tabel SNP 9
4 Pengujian data varietas kedelai pada proses integrasi 14
DAFTAR GAMBAR
1 Tahapan penelitian 3
2 Format data SNP fail berformat VCF 5
3 Format data fail berformat FASTA 6
4 Ilustrasi definisi flanking area 6
5 Format data SNP kedelai dari NCBI 7
6 Use case Diagram 8
7 Rancangan antarmuka pencarian berdasarkan kromosom dan posisi SNP 9
8 Arsitektur sistem proses integrasi 10
9 Implementasi antarmuka proses integrasi basis data dan pipeline 11 10 Implementasi antarmuka fasilitas pencarian berdasarkan kromosom dan
posisi SNP 12
11 Pseudocode proses integrasi basis data dan pipeline 13
12 Contoh hasil pemrosesan data 14
13 Hasil pencarian lanjutan informasi SNP kedelai 15
DAFTAR LAMPIRAN
1 Data fail VCF hasil proses identifikasi SNP pada sistem ISNIP 18
2 Data fail VCF dari NCBI 19
3 Desain basis data 20
4 Rancangan antarmuka pencarian lanjutan 20
5 Rancangan antarmuka hasil pencarian 21
6 Rancangan detail data SNP 21
7 Implementasi antarmuka fasilitas pencarian lanjutan 22
8 Implementasi antarmuka hasil pencarian 22
9 Implementasi antarmuka detail data SNP 23
PENDAHULUAN
Latar Belakang
Menurut Tiwari et al. (2004), kedelai merupakan salah satu sumber protein nabati yang secara langsung dapat digunakan untuk pemenuhan kebutuhan gizi manusia maupun sebagai bahan penghasil produk berkualitas tinggi. Menurut Atman (2009), di Indonesia kebutuhan tanaman kedelai mencapai 2.35 juta ton per tahun dan cenderung mengalami peningkatan setiap tahunnya. Peningkatan jumlah tersebut harus diikuti dengan penemuan varietas unggul agar kualitas kedelai bisa meningkat. Usaha pemuliaan tanaman kedelai merupakan salah satu cara untuk membantu menghasilkan varietas baru kedelai yang unggul.
Upaya penemuan varietas unggul dengan cara pemuliaan tanaman secara konvensional membutuhkan waktu yang lama dan biaya yang besar. Kendala tersebut dapat diatasi dengan memanfaatkan informasi yang diperoleh dari analisis genom pada tanaman kedelai tersebut, yaitu berupa markah single nucleotide polymorphism beserta asosiasi dengan fenotipenya. Single nucleotide polymorphism atau SNP (diucapkan “snips”) didefinisikan sebagai variasi dalam urutan DNA yang terjadi ketika sebuah nukleotida tunggal A, T, C, G dalam genom berbeda antar anggota spesies (antar kromosom berpasangan dalam individu). Ketika SNP terjadi di dalam gen, tercipta varian yang berbeda (alel) pada gen tersebut. Menurut Azrai (2005), SNP adalah salah satu penanda molekuler yang dapat digunakan untuk mempercepat usaha pemuliaan tanaman. Identifikasi SNP dapat menggunakan beberapa program komputer untuk menghasilkan informasi SNP. Li et al. (2009) menggunakan SAMtools untuk membantu identifikasi SNP. Identifikasi SNP sangat penting untuk membantu proses penyilangan menjadi lebih efektif dan efisien.
Kusuma et al. (2013) telah melakukan penelitian yang menghasilkan sebuah sistem yang diberi nama ISNIP (Integrated SNP Identification Pipeline). Sistem ini mampu mengintegrasikan beberapa program dalam satu kesatuan pipeline untuk identifikasi SNP. Pipeline tersebut dilengkapi dengan antarmuka berbasis web untuk memudahkan pengguna dalam melakukan proses identifikasi SNP. Sistem ini memiliki beberapa tahapan dalam melakukan identifikasi SNP yang mengacu pada Altmann et al. (2012). Tahapan-tahapannya, antara lain sequence alignment, alignment post-process, variant calling, filtering SNP candidates, dan making sense of SNP data. Namun, kendala terjadi karena keluaran dari sistem tersebut adalah informasi SNP yang disimpan dalam fail berformat variant call format (VCF). Fail dengan format tersebut sulit dimengerti dan diolah oleh peneliti pemuliaan. Oleh karena itu, diperlukan sebuah sistem yang mampu melakukan proses penyimpanan informasi SNP kedelai dari fail VCF tersebut, termasuk informasi pendukung lainnya yaitu flanking area dan reference SNP. Flanking area adalah informasi DNA yang berada di sekitar SNP yang dapat dimanfaatkan untuk memudahkan peneliti pemuliaan menganalisa SNP lebih lanjut. Reference SNP dapat didefinisikan sebagai nomor identifikasi referensi dari basis data SNP NCBI (National Center for Biotechnology Information).
2
sistem ISNIP yang akan dirancang menjadi suatu proses tambahan dari sistem tersebut. Selain itu, sistem ini juga dilengkapi fasilitas pencarian untuk memudahkan peneliti pemuliaan menemukembalikan informasi SNP yang sudah melalui tahapan proses identifikasi SNP dan tersimpan ke basis data pada sistem tersebut.
Perumusan Masalah
ISNIP merupakan sistem yang mampu melakukan identifikasi SNP. Untuk melakukan identifikasi SNP harus melewati beberapa proses hingga akhirnya menghasilkan informasi SNP. Informasi SNP dalam sistem itu dihasilkan dalam bentuk fail berformat VCF. Namun, informasi SNP yang disimpan dalam fail berformat VCF sulit untuk dimengerti dan diolah oleh peneliti pemuliaan. Oleh karena itu, dibutuhkan suatu sistem yang mampu mengintegrasikan proses dalam sistem ISNIP. Sistem tersebut nantinya mampu mengolah dan menyimpan data informasi SNP dan informasi pendukung lainnya ke dalam basis data. Hal tersebut merupakan salah satu cara untuk membantu peneliti pemuliaan mengolah data informasi SNP. Selain itu juga diberikan fasilitas pencarian untuk menemukembalikan informasi kandidat SNP yang revelan dengan pencarian yang diinginkan peneliti pemuliaan.
Tujuan Penelitian
Tujuan dari penelitian ini adalah membangun sistem yang mampu memilah informasi yang penting dari fail VCF, menyimpannya ke dalam basis data dan mengintegrasikannya dengan pipeline sistem ISNIP serta informasi pendukung lainnya terkait identifikasi SNP.
Manfaat Penelitian
Manfaat dari penelitian ini adalah memudahkan pengolahan dan pencarian informasi pada basis data single nucleotide polymorphism tanaman kedelai serta membantu mempercepat perakitan varietas kedelai yang unggul.
Ruang Lingkup Penelitian
Lingkup dari penelitian ini, antara lain:
1 Data yang digunakan adalah data hasil dari proses identifikasi SNP pada sistem ISNIP, serta data SNP kedelai dari NCBI.
3
METODE
Dalam penelitian ini, integrasi basis data dan pipeline SNP menggunakan pendekatan metode waterfall dengan tahapan yang mengacu pada Pressman (2010) yang disesuaikan dengan penelitian ini. Tahapan penelitian ini dapat dilihat pada Gambar 1.
Gambar 1 Tahapan penelitian
Pengumpulan Data
Data yang digunakan pada penelitian ini terdiri atas tiga jenis. Data yang pertama adalah data dengan fail berformat VCF yang didapatkan dengan cara menjalankan semua proses sistem ISNIP. Untuk percobaan, pada penelitian ini digunakan empat varietas tanaman kedelai. Data informasi SNP varietas kedelai berformat VCF ini dapat dilihat pada Tabel 1.
Tabel 1 Data informasi SNP varietas kedelai berformat VCF Nama varietas kedelai Banyak informasi
kandidat SNP tanaman kedelai dengan fail berformat FASTA yang diunduh dari situs NCBI. Data
Pengumpulan Data
Analisis
Perancangan
Implementasi
4
ini untuk mendapatkan informasi pendukung, yaitu berupa flanking area untuk setiap SNP kedelai yang disimpan ke dalam basis data. Data yang ketiga adalah data SNP kedelai yang diambil dari basis data situs resmi NCBI, yang terdiri atas 20 kromosom dengan kurang lebih 12 000 000 kemunculan SNP kedelai. Data ini digunakan untuk memperbarui id data informasi SNP yang masih kosong dan diisi oleh id reference SNP. Tujuannya untuk membandingkan data informasi SNP hasil dari pipeline ISNIP dengan data informasi SNP kedelai dari NCBI.
Analisis
Tahapan awal dalam metode waterfall (Pressman 2010) pada penelitian ini adalah analisis. Pada tahapan ini akan dilakukan analisis untuk menyelesaikan permasalahan pada penelitian ini terkait proses integrasi. Di dalam tahapan analisis mencakup pendefinisian permasalahan dan pendefinisian kebutuhan fungsional. Pada penelitian ini juga terkait dengan pemrosesan data. Untuk itu, pada tahapan ini juga dilakukan analisis data fail berformat VCF, fail berformat FASTA, dan data SNP kedelai dari NCBI, karena data tersebut memiliki format atau model yang berbeda-beda. Analisis data dilakukan untuk membantu mendapatkan informasi atribut-atribut apa saja yang dibutuhkan untuk perancangan basis data.
Perancangan
Pada tahapan ini dilakukan perancangan basis data SNP, perancangan antarmuka, baik untuk fasilitas pencarian maupun proses integrasi. Tahapan ini juga mencakup perancangan arsitektur sistem integrasi basis data SNP dan sistem ISNIP. Arsitektur tersebut memperlihatkan komponen-komponen dari sistem yang dibangun beserta dengan fungsi-fungsi, antara lain: pemilahan informasi SNP dari fail VCF dan informasi pendukung SNP dari fail FASTA, hingga data tersimpan ke dalam basis data. Tahapan ini dilakukan untuk mempermudah tahap implementasi sistem.
Implementasi
Sistem yang sudah dirancang pada tahapan perancangan akan direalisasikan dalam tahapan implementasi. Pada tahapan ini mencakup penentuan perangkat lunak dan perangkat keras untuk pengembangan sistem, antarmuka sistem proses integrasi maupun fasilitas pencarian, kode program, serta implementasi proses integrasi basis data SNP dan pipeline ISNIP yang telah dirancang pada tahap perancangan.
Pengujian
5 tersebut juga mencakup pengujian data yang diintegrasikan dengan sistem ISNIP dan pengujian keyword pada fasilitas pencarian. Sebelum sistem yang dibangun digunakan oleh pengguna secara keseluruhan, hasil pengujian dapat memperlihatkan keberhasilan kinerja sistem tersebut.
HASIL DAN PEMBAHASAN
Analisis
Permasalahan pada penelitian ini adalah pengguna sulit untuk memahami dan mengolah fail berformat VCF yang diperoleh dari proses identifikasi SNP pada sistem ISNIP. Pengguna dalam sistem ini adalah peneliti pemuliaan. Permasalahan lain adalah kurangnya informasi pendukung untuk membantu identifikasi SNP tanaman kedelai. Untuk itu, perlu adanya solusi untuk mengatasi masalah tersebut yaitu dengan mengintegrasikan pipeline dan basis data yang menyimpan informasi berupa SNP kedelai dan informasi pendukung lainnya. Proses tersebut diimplementasikan dalam sistem ISNIP untuk menjadi proses tambahan dari proses-proses yang sebelumnya sudah ada dalam sistem tersebut. Kebutuhan lain pengguna adalah fasilitas untuk membantu menemukembalikan informasi SNP yang telah diproses pada sistem ISNIP. Untuk itu, penelitian ini juga dilakukan pembangunan fasilitas pencarian dalam sistem ISNIP tersebut.
Analisis Data
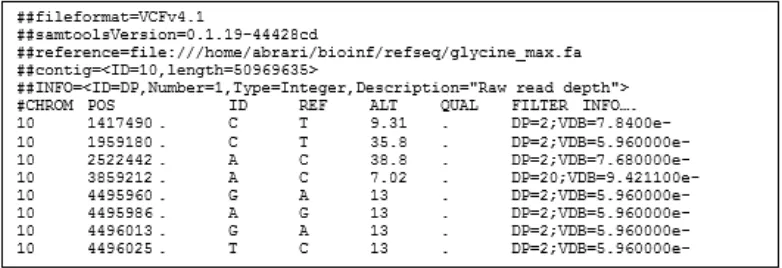
Data pertama yang dianalisis berformat VCF diperoleh dari pipeline ISNIP. Fail tersebut merupakan informasi utama mengenai SNP. Format data dengan atribut tertentu bisa dilihat pada Gambar 2.
Gambar 2 Format data SNP fail berformat VCF
Untuk data pertama dengan atribut secara lengkapnya dapat dilihat pada Lampiran 1. Dari Gambar 2 dapat dilihat berdasarkan huruf awal untuk setiap barisnya bahwa fail tersebut bisa dikelompokkan menjadi dua bagian, yaitu: diawali simbol ‘#’ dan tidak diawali simbol ‘#’.
atribut-6
atribut basis data nantinya, antara lain: chrom (kromosom), pos (posisi), id, ref (referensi), alt (alternatif), qual (kualitas), filter, info, dan format.
Bagian yang tidak diawali dengan ‘#’ merupakan informasi SNP yang dihasilkan dari proses pipeline. Bagian ini untuk setiap barisnya memiliki pemisah yang dipisahkan oleh ‘tab’, artinya untuk setiap bagian yang dipisahkan oleh ‘tab’ mengandung informasi di setiap atributnya. Sebagai contoh:
#CHROM POS ID REF ALT QUAL FILTER
10 1417490 . C T 9.31 .
- 10 : mengandung informasi atribut chrom. - 1417490 : mengandung informasi atribut pos. - . : mengandung informasi atribut id. - C : mengandung informasi atribut ref. - T : mengadung informasi atribut alt. - 9.31 : mengandung informasi atribut qual. - . : mengandung informasi aribut filter.
Pada contoh data tersebut, tanda “.” memiliki arti bahwa informasi masih kosong. Pada penelitian ini, bagian ini nantinya akan diseleksi untuk disimpan ke dalam basis data dengan memperhatikan nilai minimal kualitas SNP dan nukleotida tunggal.
Data kedua yang digunakan adalah fail berformat FASTA (.fa). Format data bisa dilihat pada Gambar 3.
Gambar 3 Format data fail berformat FASTA
Data tersebut pada intinya mengandung informasi DNA tanaman kedelai. Data ini digunakan untuk mendapatkan flanking area dengan memanfaatkan posisi SNP yang diperoleh pada informasi SNP berformat VCF. Panjang flanking area idealnya untuk memudahkan peneliti menganalisa adalah 200-300 pasang basa. Untuk memudahkan memahami definisi flanking area akan diberikan sebuah ilustrasi contoh yang dapat dilihat pada Gambar 4.
Gambar 4 Ilustrasi definisi flanking area
7 Data ketiga yang digunakan merupakan data SNP kedelai dengan fail yang diperoleh dari basis data NCBI. Fail ini berformat VCF. Format data bisa dilihat pada Gambar 5.
Gambar 5 Format data SNP kedelai dari NCBI
Data ini bentuk maupun formatnya memiliki kesamaan dengan data pertama informasi SNP yang diperoleh pada penelitian sebelumnya. Data secara keseluruhan dapat dilihat pada Lampiran 2. Pada data ini hanya atribut chrom, pos dan id yang digunakan untuk proses integrasi. Apabila data hasil percobaan proses pipeline sistem ISNIP memiliki kesamaan dalam chrom dan pos dengan salah satu data NCBI ini, maka ID pada data hasil percobaan akan diperbarui dengan diisi oleh id reference SNP dari basis data SNP NCBI. Tujuannya untuk membandingkan antara data SNP dari percobaan pipeline ISNIP dan data pada basis data SNP kedelai dari NCBI.
Analisis Kebutuhan Fungsional
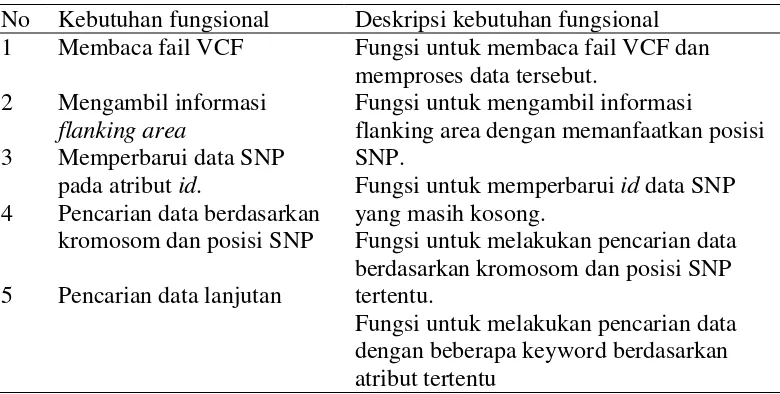
Analisis kebutuhan fungsional dilakukan untuk mengetahui fungsi-fungsi yang dibutuhkan sistem. Fungsi yang dibutuhkan dapat dilihat pada Tabel 2.
Tabel 2 Kebutuhan fungsional
No Kebutuhan fungsional Deskripsi kebutuhan fungsional 1
Fungsi untuk membaca fail VCF dan memproses data tersebut.
Fungsi untuk mengambil informasi
flanking area dengan memanfaatkan posisi SNP.
Fungsi untuk memperbarui id data SNP yang masih kosong.
Fungsi untuk melakukan pencarian data berdasarkan kromosom dan posisi SNP tertentu.
8
Analisis kebutuhan fungsional juga digambarkan dalam bentuk use case diagram. Use case diagram dapat dilihat pada Gambar 6.
Gambar 6 Use case diagram
Perancangan
Perancangan Basis Data
9 Tabel 3 Rincian tabel SNP
Atribut Deskripsi
PL List of Phred-scaled genotype likelihoods
GQ Kualitas genotipe
flank_left Flanking area sebelum posisi SNP flank_right Flanking area setelah posisi SNP Perancangan Antarmuka
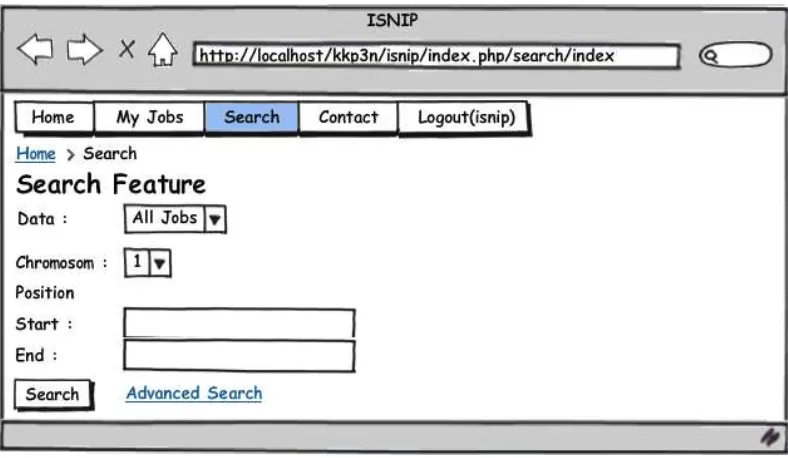
Rancangan antarmuka untuk proses integrasi menggunakan antarmuka yang sudah tersedia pada sistem ISNIP karena proses integrasi ini akan dirancang menjadi bagian proses tambahan pada sistem ISNIP. Untuk fasilitas pencarian, setelah melihat bentuk data-data yang digunakan, maka fasilitas pencarian data dibagi menjadi dua, yaitu pencarian berdasarkan kromosom dan posisi SNP serta pencarian lanjutan (advanced search). Selain itu, pengguna juga diberi kemudahan untuk menyimpan hasil pencarian ke dalam fail excel melalui fitur export to excel. Hal tersebut dibuat karena data-data yang akan dicari memiliki atribut yang banyak dan data yang dicari juga banyak. Rancangan antarmuka fasilitas pencarian berdasarkan kromosom dan posisi SNP dapat dilihat pada Gambar 7.
10
Rancangan antarmuka pencarian lanjutan dapat dilihat pada Lampiran 4, rancangan antarmuka hasil pencarian dapat dilihat pada Lampiran 5, dan rancangan detail data SNP dapat dilihat pada Lampiran 6.
Perancangan Arsitektur Sistem
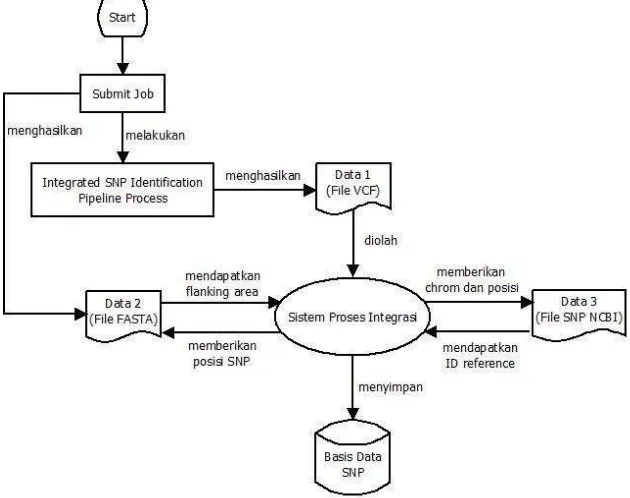
Untuk mengintegrasikan pipeline dan basis data, maka diperlukan proses integrasi tersebut. Alur proses integrasi antar data dirancang dalam bentuk arsitektur sistem. Arsitektur sistem proses integrasi dapat dilihat pada Gambar 8.
Gambar 8 Arsitektur sistem proses integrasi
Dari arsitektur sistem di atas dapat dijelaskan bahwa tahapan proses identifikasi SNP pada sistem ISNIP menghasilkan fail berformat VCF, kemudian diolah untuk setiap barisnya dengan mengabaikan baris yang diawali simbol ‘#’. Dalam pengolahan data tersebut, yang diperhatikan untuk seleksi masuk ke dalam basis data adalah nilai minimal kualitas dan nukleotida tunggal atau tidak. Kemudian sistem menggunakan pos (posisi) untuk setiap atributnya dan digunakan untuk mendapatkan flanking area yang diperoleh dari data pada fail FASTA. Di sisi lain juga menggunakan chrom dan pos untuk setiap atributnya dan digunakan untuk membandingkan dengan data dari NCBI untuk mendapatkan id references SNP. Proses ini berlangsung hingga akhirnya data saling terintegrasi dan disimpan ke dalam basis data.
Implementasi
Spesifikasi perangkat keras dan perangkat lunak yang digunakan untuk implementasi dalam penelitian ini adalah sebagai berikut:
1 Perangkat Lunak
11
MySQL sebagai perangkat lunak sistem manajemen basis data SQL (DBMS).
XAMPP untuk konfigurasi Apache, MySQL, dan bahasa pemrograman PHP.
Text Editor sebagai aplikasi untuk mengedit kode program implementasi penelitian.
2 Perangkat Keras
Processor Intel Core i5-3230M @ 2.60 GHz
Memory 4 GB RAM
VGA ATI Radeon 2GB
Hardisk: 512GB.
Implementasi Antarmuka Sistem
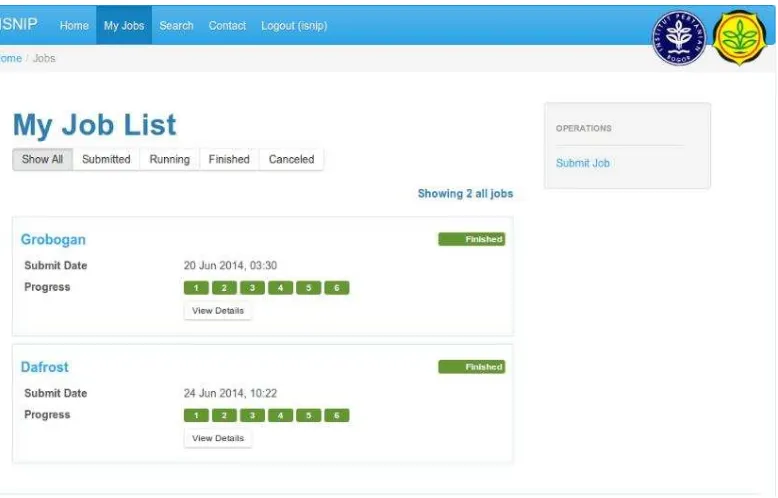
Antarmuka yang telah dirancang kemudian diimplementasikan dalam sistem ISNIP. Sistem ISNIP dibangun menggunakan Yii Framework, maka proses juga diimplementasikan menggunakan Yii Framework dengan bahasa pemrograman PHP. Implementasi antarmuka proses integrasi yang menjadi proses tambahan dalam sistem ISNIP merupakan implementasi antarmuka awal pada sistem ISNIP tersebut. Proses tersebut diimplementasikan menjadi proses ke-6 dalam proses identifikasi SNP sistem ISNIP. Implementasinya dapat dilihat pada Gambar 9 dan fasilitas pencarian berdasarkan kromosom dan posisi SNP dapat dilihat pada Gambar 10.
12
Gambar 10 Implementasi antarmuka fasilitas pencarian berdasarkan kromosom dan posisi SNP
Untuk implementasi antarmuka pencarian lanjutan, hasil pencarian dan detail data SNP tertentu dapat dilihat pada Lampiran 7, 8, dan 9.
Hasil pencarian yang diperoleh adalah data SNP yang revelan dengan keyword yang dimasukkan. Data yang ditampilkan dalam hasil pencarian berupa data singkat SNP yang bisa dilihat secara keseluruhan pada bagian detail data, serta bisa dibandingkan dengan reference SNP yang merujuk ke data SNP di situs resmi NCBI.
Kode Program
13
Gambar 11 Pseudocode proses integrasi basis data dan pipeline
Pengujian
14
Gambar 12 Contoh hasil pemrosesan data
Pada Gambar 12 terlihat data dengan id dan flanking area yang sebelumnya masih kosong. Setelah dilakukan proses integrasi, data tersebut mengalami perubahan. Ada yang id-nya tetap kosong maupun yang sudah diperbarui.
Pengujian proses integrasi ini juga dilakukan dengan beberapa data varietas kedelai yang berbeda, antara lain : B3293 5M, Grobogan, Dafrost, dan Malabar. Pada pengujian ini menggunakan nilai minimal kualitas SNP sebesar 30. Hasil pengujian dapat dilihat pada Tabel 4.
Tabel 4 Pengujian data varietas kedelai pada proses integrasi Nama varietas
Pada Tabel 4 terlihat waktu proses yang berbeda dan lama. Hal tersebut terjadi karena data yang diolah berbeda dan sangat banyak termasuk proses dimana data melakukan pencarian kromosom dan posisi pada data SNP dari NCBI yang digunakan untuk mendapatkan id reference SNP. Selain itu juga karena keterbatasan perangkat keras yang digunakan untuk menjalankan sistem ini.
15
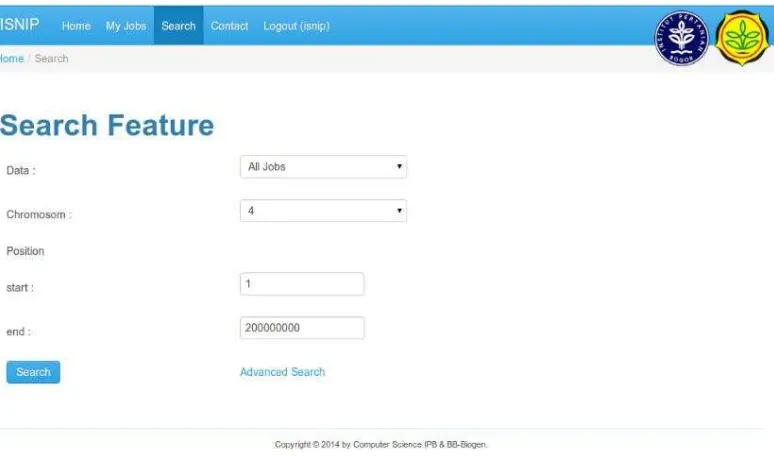
Gambar 13 Hasil pencarian lanjutan informasi SNP kedelai
Gambar 13 menunjukkan hasil pencarian lanjutan dengan beberapa keyword, antara lain:
1 Data yang dicari adalah “All Jobs”, artinya menggunakan semua data pengguna tersebut untuk dicari.
2 Posisi SNP mulai dari 1 hingga 200 000 000.
3 Keyword kromosom 10, references G, dan alternative T.
16
sebesar 0. Informasi SNP juga memberikan nilai kualitas genotipe pada atribut GQ. Salah satu contoh hasil pencarian data SNP memberikan nilai 51.
Kontribusi Penelitian
Dari semua tahapan penelitian ini, maka diperoleh beberapa keuntungan dengan adanya proses tambahan maupun fasilitas pencarian, antara lain :
1 Peneliti pemuliaan atau pengguna dapat mengolah data SNP kedelai untuk pemuliaan kedelai.
2 Dengan adanya fasilitas pencarian, maka peneliti pemuliaan akan lebih mudah untuk menemukembalikan informasi SNP kedelai yang relevan, termasuk menyimpan hasil pencariannya dalam bentuk fail excel.
3 Dengan adanya informasi pendukung, maka informasi peneliti pemuliaan tidak terbatas hanya mengidentifikasi SNP dari percobaan yang dilakukan, melainkan mampu membandingkan dengan data SNP kedelai dari NCBI yang terbukti baik kualitas SNP-nya dan mampu melihat nukleotida di sekitar SNP (flanking area).
SIMPULAN DAN SARAN
Simpulan
Penelitian ini berhasil menghasilkan suatu sistem untuk melakukan proses integrasi basis data dan pipeline SNP pada sistem ISNIP. Sistem yang dihasilkan mampu mengekstrak informasi SNP kedelai dari fail VCF dan mengintegrasikan dengan informasi data pendukung lainnya baik yang berasal dari fail FASTA maupun fail VCF yang berasal dari NCBI. Sistem ini juga mampu membantu peneliti pemuliaan untuk mengolah dan menemukembalikan data informasi SNP yang relevan. Kinerja sistem mampu memilah informasi SNP dari fail VCF, membandingkannya dengan NCBI dan menyimpannya ke dalam basis data dengan kecepatan rata-rata 0.964 SNP per detik.
Saran
17
DAFTAR PUSTAKA
Altmann A, Weber P, Bader D, Preu M, Binder EB, Myhsok BM. 2012. A beginners guide to SNP calling from high-throughput DNA-sequencing data. Human Genetics. 131(10):1541–54.
Atman. 2009. Strategi peningkatan produksi kedelai di Indonesia. Jurnal Ilmiah Tambua. 8(01):39-45.
Azrai M. 2005. Pemanfaatan markah molekuler dalam proses seleksi pemuliaan tanaman. Jurnal AgroBiogen. 1(1):26-37.
Li H, Handsaker B, Wysoker A, Fenell T, Ruan J, Homer N, Marth G, Abecasis G, Durbin R. 2009. The sequence alignment/map format and SAMtools. Bioinformatic. 25(16):2078-2079.
Pressman RS. 2010. Software Engineering A Partitioner’s Approach. Ed ke-7. New York (US): McGraw-Hill.
Tiwari S, Shanker P, Tripathi M. 2004. Effects of genotype and culture medium on in vitro androgenesis in soybean. Indian Journal of Biotechnology. 3: 441-444. Kusuma WA, Tasma IM, Buono A, Hidayat M, Rijzaani H, Haryanto T, Istiadi MA.
18
20
Lampiran 3 Desain basis data
21 Lampiran 5 Rancangan antarmuka hasil pencarian
22
Lampiran 7 Implementasi antarmuka fasilitas pencarian lanjutan
24
Lampiran 10 Pengujian black box No Kebutuhan
fungsional
Keadaan awal
25
RIWAYAT HIDUP
Penulis dilahirkan di Rembang pada tanggal 25 Oktober 1991 dari pasangan Bapak Chamzawi dan Ibu Alfiyah. Penulis adalah putra keempat dari empat bersaudara. Tahun 2010 penulis lulus dari SMA Negeri 2 Rembang dan pada tahun yang sama penulis lulus seleksi masuk Institut Pertanian Bogor (IPB) melalui Undangan Seleksi Masuk IPB dan diterima di Departemen Ilmu Komputer, Fakultas Matematika dan Ilmu Pengetahuan Alam.