PENGGUNAAN DNA
BARCODE
SEBAGAI ALTERNATIF
IDENTIFIKASI SPESIES UDANG MANTIS
RAISA AULIANE SYAFRINA
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
ABSTRAK
RAISA AULIANE SYAFRINA. Penggunaan DNA Barcode sebagai Alternatif Identifikasi Spesies Udang Mantis. Dibimbing oleh ACHMAD FARAJALLAH dan YUSLI WARDIATNO.
Udang mantis atau yang dikenal dengan udang ronggeng merupakan salah satu anggota Subfilum Crustacea Ordo Stomatopoda, Beberapa spesies udang mantis dikenal sebagai komoditi ekspor dan makanan eksotis. Beberapa udang mantis yang bernilai ekonomi tinggi adalah anggota famili Harpiosquillidae dan Squillidae. Udang-udang mantis tersebut biasa ditangkap dari Laut Jawa dan Laut Cina Selatan. Penelitian ini bertujuan untuk mengaplikasikan teknik DNA
barcoding sebagai laternatif mengidentifikasi udang mantis sampai ke tingkat spesies. Sebanyak 34 sampel udang mantis berasal dari Jambi, Cirebon, dan Aceh yang digunakan adalah spesimen koleksi yang diawetkan dalam alkohol. Identifikasi spesies dilakukan berdasarkan morfologi. Ekstraksi DNA dilakukan dari otot tungkai dan ruas gen CO1 diamplifikasi sebagai ruas DNA yang dijadikan barcode. Amplifikasi gen CO1 berhasil dilakukan untuk semua sampel yang kesemuanya menghasilkan amplikon yang multiband. Perunutan nukelotida hanya berhasil dilakukan untuk tujuh sampel, yaitu Harpiosquilla harpax (Sampel No. 34 dan No. 37 asal Jambi),
Harpiosquilla stephensoni (sampel No. 1 dan No. 2 asal Cirebon), dan Carinosquilla multicarinata
(sampel No. 9 dan No. 11 asal Cirebon, dan No. 22 asal Aceh). Berdasarkan ruas gen CO1, C. multicarinata asal Aceh memiliki kekerabatan yang berbeda dengan C. multicarinata asal Cirebon.
ABSTRACT
RAISA AULIANE SYAFRINA. DNA Barcode as an Alternative of Mantis Shrimp Species Identification. Supervised by ACHMAD FARAJALLAH and YUSLI WARDIATNO.
PENGGUNAAN DNA
BARCODE
SEBAGAI ALTERNATIF
IDENTIFIKASI SPESIES UDANG MANTIS
RAISA AULIANE SYAFRINA
Skripsi
sebagai salah satu syarat untuk memperoleh gelar
Sarjana Sains pada
Departemen Biologi
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
Judul Skripsi : Penggunaan DNA
Barcode
Sebagai Alternatif Identifikasi
Spesies Udang Mantis
Nama
: Raisa Auliane Syafrina
NIM
: G34070108
Disetujui
Dr. Ir. Achmad Farajallah, M.Si.
Dr. Ir. Yusli Wardiatno, M. Sc
Pembimbing I
Pembimbing II
Diketahui
Dr. Ir. Ence Darmo Jaya Supena, M. Si
Ketua Departemen Biologi
PRAKATA
Puji dan syukur penulis panjatkan kehadirat Allah SWT atas limpahan rahmat dan hidayah-Nya sehingga dapat menyelesaikan karya ilmiah ini. Shalawat serta salam penulis panjatkan kepada Nabi Muhammad SAW. Penelitian ini dilaksanakan mulai bulan Februari hingga Agustus 2011 sebagai salah satu syarat untuk memperoleh gelar sarjana sains di Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor yang berjudul Penggunaan DNA Barcode sebagai Alternatif Identifikasi Spesies Udang Mantis.
Terima kasih penulis ucapkan kepada Bapak Dr. Ir. Achmad Farajallah, M.Si danBapak Dr. Ir. Yusli Wardiatno, M.Sc selaku pembimbing, yang telah memberikan ilmu, pengarahan dan bimbingannya kepada penulis, serta kepada Ibu Dr. Anja Meryandini, M. Si selaku dosen penguji yang turut memberikan kritik dan saran. Ungkapan terima kasih juga disampaikan kepada keluarga tercinta, terutama almarhum papa (Drs. Syafrizal Abbas), mama (Irina Selviati), kakak (Sarah Auliani Syafrina), dan adik (Abraham Lintau) atas segala doa yang tiada henti, kasih sayang, dan dukungannya. Penulis mengucapkan terima kasih kepada semua keluarga besar zoologi (Ibu Taruni, Ibu Rika, Pak Tri Atmo, Mba Tini, Mba Ani, Pak Adi, Mba Kanthi, Mba Tetri, Kak Sarah Nila, Kak Jazi, Kak Uche, Kak Iqbal, Mas Wildan, Mba Dea, Pak Rizal, I Made K, Iqbal, Rindi, Bisri, Cahyo, Chyntia, Nishe, Renny, dan Noe) yang telah berbagi ilmu serta segala dukungannya, kepada sahabat seperjuangan (Yakub Hidayatullah, Eva Brialin Agenginardi, Soraya Puspa Jelita, Aminah, Rina Nurlia, Agessty Ika, Karina Swedianti, Nurul Ichsan, dan Gilar Cahya) atas segala bantuan, nasehat, dan semangat yang selalu diberikan selama penelitian, dan kepada teman Griya (Fatmi Harun dan Singgih Widosari). Selain itu terima kasih kepada teman-teman Biologi angkatan 44 yang telah memberikan motivasi kepada penulis.
Semoga karya ilmiah ini bermanfaat dan menambah khasanah ilmu pengetahuan kita semua. Saran dan kritik yang membangun sangat penulis harapkan agar tulisan ini menjadi lebih baik.
Bogor, September 2011
RIWAYAT HIDUP
Penulis dilahirkan di Medan pada tanggal 2 Juli 1989 dari Bapak Ir. H. Syafrizal Abbas (Alm) dan Ibu Hj. Irina Selviati. Penulis merupakan anak kedua dari tiga bersaudara.
Tahun 2007 penulis lulus dari SMA Negeri 48 Jakarta dan melanjutkan pendidikan di Institut Pertanian Bogor, Fakultas Matematika dan Ilmu Pengetahuan Alam, Departemen Biologi melalui jalur penerimaan Seleksi Penerimaan Mahasiswa Baru (SPMB).
DAFTAR ISI
DAFTAR TABEL ... viii
DAFTAR GAMBAR ... viii
DAFTAR LAMPIRAN ... viii
PENDAHULUAN ... 1
Latar Belakang ... 1
Tujuan ... 1
Waktu dan Tempat ... 1
BAHAN DAN METODE ... 1
Bahan ... 1
Identifikasi Sampel ... 2
Ekstraksi dan Isolasi DNA... 2
Amplifikasi dan Visualisasi Fragmen DNA ... 2
Perunutan Amplikon dan Analisis DNA ... 2
HASIL ... 2
Identifikasi Morfologi ... 2
Amplifikasi dan Visualisasi DNA ... 3
Analisis DNA dan Filogeni ... 3
PEMBAHASAN ... 5
SIMPULAN ... 6
SARAN ... 6
DAFTAR PUSTAKA ... 6
DAFTAR TABEL
Tabel 1 Data Hasil identifikasi spesimen udang mantis ... 3
Tabel 2 Data spesies hasil identifikasi dan barcode ... 4
Tabel 3 Jumlah perbedaan nukleotida gen CO1 antar spesies udang mantis ... 4
Tabel 4 Jarak genetik gen CO1 antar spesies udang mantis berdasarkan model subtitusi K2P ... 4
DAFTAR GAMBAR
Gambar 1 Produk PCR berupa pita tunggal yang diuji dengan menggunakan polyacrilamide gel electrophoresis (PAGE) 6% ... 3Gambar 2 Hasil rekonstruksi pohon filogeografi pengelompokan sampel berdasarkan ruas CO1 mtDNA menggunakan metode NJ dengan bootstrap 1000x ... 5
DAFTAR LAMPIRAN
Lampiran 1 Ekstraksi dan isolasi DNA dengan metode GeneAid ... 9Lampiran 2 Visualisasi fragmen DNA (penyajian produk PCR) ... 10
PENDAHULUAN
Latar Belakang
Barcode DNA adalah urutan pendek DNA yang digunakan untuk mengidentifikasi spesies secara cepat dan akurat (Meier et al.
2006). Ruas DNA yang bisa digunakan untuk sistem tersebut harus terstandardisasi.
Barcode DNA juga menjadi salah satu alternatif pelengkap atau komplemen yang dapat memperkuat identifikasi morfologi secara cepat dan akurat (Lahaye et al. 2008).
Barcode DNA menjanjikan beberapa manfaat, antara lain mengenali spesies, memastikan keamanan pangan, mengidentifikasi fase larva yang berbeda dengan fase dewasa, mengontrol hama pertanian, dan melacak asal usul vektor penyakit dan serangan hama pada suatu area.
Pada hewan, penggunaan genom mitokondria (mtDNA) dalam analisis biogeografi dan sistematik sering tidak sejalan dengan morfologi. Salah satu penyebabnya adalah karakter morfologi yang seringkali memperlihatkan fenomena species cryptic
(hampir mirip). Genom mitokondria hewan merupakan genom sitoplasmik yang diwariskan secara uniparental dan tidak mengalami rekombinasi sehingga species sibling bisa dipastikan mempunyai mtDNA dengan nilai kesamaan yang tinggi. Salah satu ruas mtDNA yang banyak digunakan sebagai
barcode adalah cytochrome oxidase 1 (CO1) genom mitokondria yang dipopulerkan oleh Hebert et al. (2003). Gen CO1 pada Crustacea berukuran sekitar 1500 pb. Dibandingkan dengan ruas-ruas gen yang lain dalam mtDNA, gen CO1 memberikan hasil yang lebih efektif dan mudah untuk diakses. Teknologi barcoding menggunakan penanda gen CO1 dari mtDNA dapat digunakan untuk mengidentifikasi hampir semua spesies hewan (Ward et al. 2005), baik interspesifik maupun intraspesifik (Hebert et al. 2003).
Udang mantis atau yang dikenal dengan udang ronggeng merupakan anggota subfilum Crustacea, Ordo Stomatopoda, yang terdiri atas empat famili, yaitu Odontodactylidae, Lysiosquillidae, Harpio-squillidae dan Squilidae. Beberapa spesies udang mantis, terutama yang bisa mencapai ukuran >30 cm, biasa dijadikan sebagai komoditi ekspor dan makanan eksotis dengan harga yang relatif mahal (Ahyong et al. 2008). Kelompok udang ini dicirikan dengan tubuh yang bersegmen, di belakang kepala terdapat karapas pendek, kaki beruas-ruas, ukuran tubuh yang besar dan mata seringkali berbentuk T (Carpenter & Niem 1998).
Beberapa udang mantis yang bernilai ekonomi tinggi adalah dari famili Harpiosquillidae dan Squilidae. Kedua famili tersebut biasa ditangkap dari Laut Jawa dan Laut Cina Selatan. Persebaran udang mantis di kedua wilayah tersebut dipengaruhi migrasi larva udang mantis mengikuti pergerakan arus air laut yang sejajar garis pantai (Barber et al.
2002). Selain itu, dari 450 spesies yang telah dideskripsikan, 118 (26%) diantaranya bisa ditemukan di perairan Indonesia (Ahyong et al. 2008, Moosa 2000). Jumlah spesies udang mantis yang sangat tinggi di Indonesia (jika dibandingkan dengan laut Brazilia dengan 35 spesies dan laut Mediterania 10 spesies) tidak sejalan dengan keterkenalannya sebagai bagian dari kekayaan biodiversitas Indonesia.
Oleh karena itu, dalam penelitian ini digunakan teknik DNA barcode untuk mengidentifikasi keanekaragaman spesies udang mantis yang terdapat di perairan Indonesia. Menurut Konvensi Keaneka-ragaman Hayati, barcode menjadi salah satu teknik yang berkontribusi cukup signifikan terhadap pelaksanaan konservasi keragaman spesies. Konvensi Keanekaragaman Hayati (Convention on Biological Diversity atau CBD) adalah perjanjian internasional yang bertujuan mengembangkan strategi nasional untuk konservasi dan pemanfaatan berkelanjutan keanekaragaman hayati. Dengan begitu, aplikasi DNA barcoding dapat menjawab klaim kepemilikan dan asal lokalitas komoditas perdagangan suatu wilayah (CBOL 2008).
Tujuan
Penelitian ini bertujuan untuk menggunakan teknik DNA barcoding sebagai alternatif identifikasi keanekaragaman spesies udang mantis yang terdapat di perairan Indonesia.
Waktu dan Tempat
BAHAN DAN METODE
Bahan
Beberapa spesimen udang mantis yang digunakan merupakan koleksi Dr. Yusli Wardiatno (Departemen Manajemen Sumber-daya Perairan IPB) yang diawetkan dalam etanol, yang berjumlah 30 ekor.
Identifikasi Sampel
Identifikasi spesies udang mantis dilakukan menggunakan buku kunci identifikasi Manning (1980).
Ekstraksi dan Isolasi DNA
Ekstraksi DNA dilakukan dari otot tungkai menggunakan DNA Extraction Kit for Fresh Blood (Geneaid) yang dimodifikasi untuk sampel yang diawetkan dalam etanol. Sekitar 50 mg otot tungkai dicuci dari etanol dengan cara merendamnya dalam air destilata selama 20 menit yang dilakukan dua kali Perendaman dalam air ini akan mengeluarkan etanol dari dalam jaringan dan sekaligus mengencerkannya sehingga etanol tidak mengganggu proses-proses berikutnya. Kemudian jaringan otot dihomogenasi dalam bufer STE (NaCl 1M, Tris-HCL 10mM, EDTA 0.1mM, pH 8) dan dilisis menggunakan proteinase K 0,125 mg/ml dan sodium dodesil sulfat 1%. Ekstraksi DNA selanjutnya dilakukan mengikuti petunjuk produsen dari kit ekstraksi DNA (Lampiran 1).
Amplifikasi dan Visualisasi Fragmen DNA
Amplifikasi ruas gen CO1 mtDNA dilakukan dengan teknik polymerase chain reaction (PCR) menggunakan forward primer
AF286 (5’-TCTACAAAYCATAAAGAYATYGG) dan/atau AF215 (5’-TTCAACAAATCATAAA GATATTGG) dan reverse primerAF287 (5’-G TGGCRGANGTRAARTARGCTCG). Pro-duk PCR atau amplikon dari kedua pasangan primer tersebut berukuran sekitar 800 pb. Reaksi PCR dilakukan dalam volume 50 μL menggunakan mesin thermocycler ESCO
MX-BLC-7. Reaksi PCR dilakukan dengan kondisi predenaturasi pada suhu 94oC selama 5 menit, kemudian dilanjutkan 30 siklus yang terdiri atas denaturasi suhu 94oC selama 1 menit, penempelan primer suhu 55oC selama 1,5 menit, pemanjangan 72oC selama 2 menit, dan diakhiri pemanjangan akhir suhu 72oC selama 5 menit.
Pengujian amplikon dilakukan menggunakan metode polyacrilamide gel electrophoresis (PAGE) 6% yang dijalankan
pada tegangan 200 V selama 50 menit atau sampai pelacak warna bromtimol blue mencapai batas bawah gel. Setelah pemisahan elektroforesis, amplikon divisualisasi dengan pewarnaan sensitif perak (Lampiran 2) (Byun
et al. 2009).
Perunutan Amplikon dan Analisis DNA
Amplikon berupa pita tunggal yang berukuran sesuai desain primer atau ada pita-pita tambahan yang tidak dominan kemudian dimurnikan untuk dijadikan cetakan dalam
PCR for sequencing. Proses PCR untuk sequencing menggunakan primer yang sama seperti amplifikasi sebelumnya dengan metode big dye terminator cycle sequencing.Runutan nukleotida yang diperoleh kemudian diedit secara manual berdasarkan kromatogram. Jika kromatogram menunjukkan runutan nukleotida yang meragukan maka dilakukan pembandingan dengan kromatogram yang diperoleh dari PCR for sequencing berikutnya. Kepastian suatu runutan nukleotida yang dipilih adalah yang memberikan puncak kurva yang lebih tinggi dan berjarak normal dari puncak-puncak kurva di sebelahnya. Selain itu, pengdeitan juga dilakukan atas bantuan translated protein sequence. Analisis homologi dari runutan nukleotida yang telah diedit terhadap runutan nukleotide sejenis yang tersimpan dalam database GenBank dilakukan dengan program BLAST (Basic Local Alignment Search Tool) (http://www.ncbi. nlm.nih.gov/ ).
Runutan nukleotida dan ruas DNA homolognya kemudian saling disejajarkan ulang (multiple alignment) menggunakan program Clustal W 1.8 yang tertanam dalam program MEGA versi 4.00 (Tamura et al. 2007).
Pairwise distance calculation dilakukan untuk menentukan jumlah perbedaan runutan nukleotida antar sampel dengan opsi model
number of differences dalam program MEGA versi 4.00 (Tamura et al. 2007). Selain itu, analisis keragaman nukleotida dan filogenetik dilakukan menggunakan model subtitusi
HASIL
Identifikasi Morfologi
Hasil identifikasi berdasarkan buku identifikasi Manning (1980) menunjukkan bahwa spesimen udang mantis koleksi terdiri atas 2 famili dan 6 spesies (Tabel 1).
Tabel 1 Hasil identifikasi spesimen udang mantis
Spesies No. Sampel
Famili Hapriosquillidae
Harpiosquilla stephensoni 1,2,3,4,5,6, 8, 10, 21
Harpiosquilla harpax 31, 34, 35, 37
Harpiosquilla raphidae 30
Harpiosquilla melanoura 23 Famili Squillidae
Carinosquilla multicarinata 9, 11, 12, 15, 16, 17, 18, 19, 20, 22, 26,
Keijia lirata 7, 13, 14, 25,
Harpiosquilla harpax memiliki ciri karapas dilengkapi dengan median carina dan distal segmen pada uropod berwarna hitam dengan garis tengah putih, carina intermediate pada thoraks tidak terlalu tajam, rostral dilengkapi dengan projeksi anterior, dan marginal carina dua kali lebih panjang dari carina gigi lateral. Harpiosquilla stephensoni
memiliki ciri karapas dilengkapi dengan median carina dan distal segmen pada uropod berwarna hitam dengan garis tengah putih, carina intermediate pada thoraks tidak terlalu tajam, rostral tidak dilengkapi dengan projeksi anterior, dan tajam pada thoraks kelima.
Carinosquilla multicarinata memiliki ciri terdapat duri ganda pada ujung thoraks kelima, thoraks dan abdomen ditutupi dengan carina longitudinal, carina pada thoraks dan abdomen terdapat duri.
Amplifikasi dan Visualisasi DNA
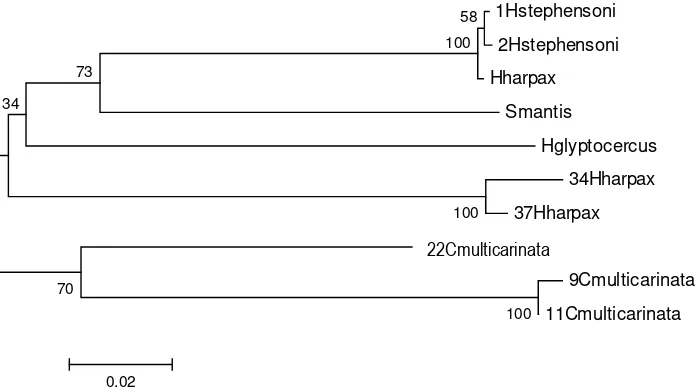
Gen CO1 target diamplifikasi menggunakan pasangan primer AF286-AF287 dan AF215-AF287 berukuran sekitar 700-800 bp (Gambar 1), dengan jumlah sampel sebanyak 34 ekor. Pada beberapa sampel suhu optimum penempelan primer pada saat amplifikasi yaitu 55°C.
M 1 2 3 4 5 6 7 8 9 10 11
Gambar 1 Amplikon gen CO1 di atas PAGE 6%. Keterangan: M. Marker; 1-11. Nomor sampel.
Hasil amplifikasi menunjukkan fragmen DNA multiband. Pita DNA target berhasil ditemukan dari pasangan primer 286-AF287 sebanyak 12 sampel, sedangkan dari pasangan primer AF215-AF287 sebanyak 14 sampel. Dari total sampel yang berhasil diamplifiaksi menampakkan pita DNA target, hanya 7 sampel dari pasangan primer AF286-AF287 dan 13 yang dijadikan cetakan dalam PCR for sequencing karena beberapa pita DNA muncul tipis pada gel poliakrilamid saat proses visualisasi. Setelah DNA target dimurnikan dan dijadikan cetakan dalam PCR for sequencing, maka diperoleh 7 sampel yang terbaca dengan jelas. Ketujuh sampel tersebut adalah 1H. stephensoni; 2H. stephensoni; 9C. multicarinata; dan 11C. multicarinata
(Cirebon), 22C. multicarinata (Aceh), dan 34H. harpax; 37 H. harpax (Jambi).
Analisis DNA dan Filogeni
Tujuh runutan DNA mantis yang diperoleh kemudian saling disejajarkan dengan ruas mtDNA yang homolog dari Haptosquilla glyptocercus (AF205239), Harpiosquilla harpax (FJ229770), dan Squilla mantis
(GQ328967). Data runutan nukleotida yang saling homolog antar sampel dengan data referensi yang bisa dilakukan analisis lebih lanjut adalah sepanjang 430 nt (Lampiran 3).
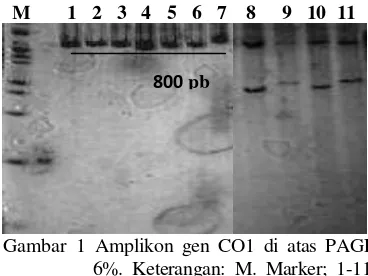
Sampel dengan jumlah perbedaan nukleotida paling kecil terjadi antara sampel Nomor 2 yang diduga sebagai Harpiosquilla stephensoni dengan Harpiosquilla harpax dari
genebank, yaitu hanya berbeda satu nukleotida. Sampel dengan jumlah perbedaan nukleotida paling tinggi terdapat antara sampel Nomor 9 yang diduga sebagai
Carinosquilla multicarinata dengan sampel Nomor 1 (Harpiosquilla stephensoni), yaitu berbeda 84 nukleotida (Tabel 3).
Jarak genetik antar sampel dibandingkan dengan sampel spesies yang ada di GeneBank
4
model substitusi K2P (Tabel 4) menghasilkan jarak genetik tertinggi sebesar 0.234 (23.4%) ditemukan antara sampel Nomor 9 (C.
multicarinata) dengan sampel Nomor 2 H. stephensoni. Jarak genetik terendah sebesar 0.002 (0.2%) antara sampel Nomor H. harpax
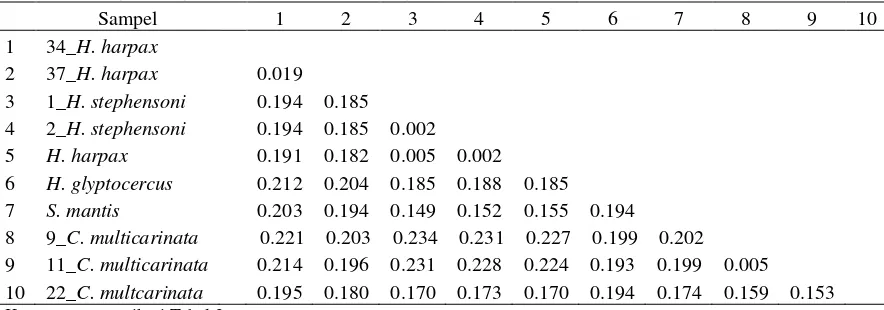
dengan sampel Nomor 1 (H. stephensoni). Topologi pohon filogeni menggunakan metode NJ dengan bootstrap1000x
mengelompokkan sampel Nomor 9, 11, dan 22 (Carinosquilla multicarinata) berada di luar percabangan. Struktur populasi yang digambarkan oleh mtDNA ini menunjukkan bahwa kekerabatan sampel Nomor 1, 2, 34, dan 37 adalah berasal dari satu nenek moyang/indukan, sedangkan populasi sampel
C. multicarinata berbeda nenek moyang/indukan (Gambar 2).
Table 2 Data spesies hasil identifikasi dan barcode
No. sampel Identifikasi Asal lokasi
Identifikasi awal Barcode
1 Harpiosquilla stephensoni Harpiosquilla harpax Cirebon 2 Harpiosquilla stephensoni Harpiosquilla harpax Cirebon 9 Carinosquilla multicarinata Carinosquilla multicarinata Cirebon 11 Carinosquilla multicarinata Carinosquilla multicarinata Cirebon 22 Carinosquilla multicarinata Carinosquilla multicarinata Aceh 34 Harpiosquilla harpax Harpiosquilla harpax Jambi 37 Harpiosquilla harpax Harpiosquilla harpax Jambi
Tabel 3 Jumlah perbedaan nukleotida gen CO1 antar spesies udang mantis
Sampel 1 2 3 4 5 6 7 8 9 10
1 34_H. harpax
2 37_H. harpax 8 3 1_H. stephensoni 72 69 4 2_H. stephensoni 72 69 1 5 H. harpax 1) 71 68 2 1 6 H. glyptocercus 2) 78 75 69 70 69 7 S. mantis 3) 75 72 57 58 59 72 8 9_C. multicarinata 80 74 84 83 82 73 74 9 11_C. multicarinata 78 72 83 82 81 71 73 2
10 22_C. multcarinata 72 67 64 65 64 72 65 60 58
1)
GenBank Accession Number FJ229770
2) GenBank Accession Number AF205239 3)
GenBank Accession Number GQ328967
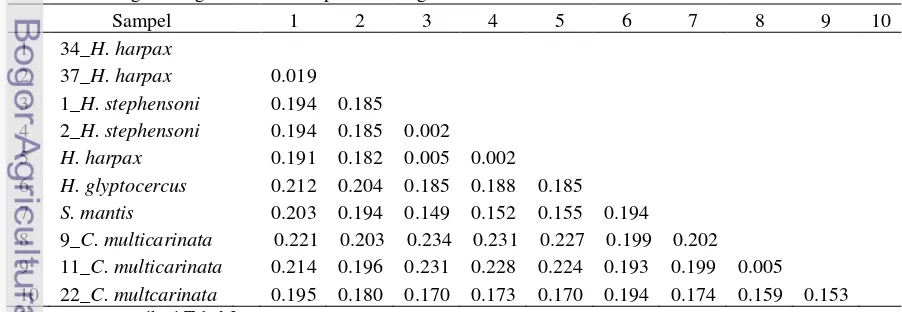
Tabel 4 Jarak genetik gen CO1 antar spesies udang mantis berdasarkan model subtitusi K2P
Sampel 1 2 3 4 5 6 7 8 9 10
1 34_H. harpax
2 37_H. harpax 0.019 3 1_H. stephensoni 0.194 0.185
4 2_H. stephensoni 0.194 0.185 0.002 5 H. harpax 0.191 0.182 0.005 0.002 6 H. glyptocercus 0.212 0.204 0.185 0.188 0.185 7 S. mantis 0.203 0.194 0.149 0.152 0.155 0.194
8 9_C. multicarinata 0.221 0.203 0.234 0.231 0.227 0.199 0.202 9 11_C. multicarinata 0.214 0.196 0.231 0.228 0.224 0.193 0.199 0.005
5
1Hstephensoni 2Hstephensoni Hharpax Smantis Hglyptocercus 34Hharpax 37Hharpax 22Cmultcarinatai 9Cmulticarinata 11Cmulticarinata 100 100 58 100 70 73 34 0.02PEMBAHASAN
Hasil amplifikasi gen CO1 menggunakan pasangan primer AF215/AF286 dan AF287 selalu menghasilkan amplikon yang multiband. Salah satu penyebab utama adalah penggunaan degenerate primer. Dalam hal ini, ada C dan T pada basa ketiga dari ujung 3’ untuk forward primer, dan ada A dan G pada basa keenam dari ujung 3’ untuk reverse primer. Menurut Innis (1990), spesifisitas tiga basa di ujung 3’ sangat menentukan hasil amplifikasi. Degenerate primer biasa digunakan untuk menjamin keberhasilan amplifikasi pada taksa yang luas (Hebert et al.
2003). Alternatif lain untuk mengakomodasi taksa yang luas adalah penggunaan beberapa primer yang disambungkan dengan runutan M13 universal. Primer M13 lebih efektif untuk barcode pada taksonomi sampel yang lebih beragam (Ivanova et al. 2007). PCR for sequencing kemudian menggunakan primer M13 universal ini. Selain masalah degenerate primer, gen CO1 dilaporkan juga ada di dalam genom inti (Perna & Kocher 1996) dan dalam kromosom beberapa spesies bakteri dengan tingkat kesamaan >70% (data analisis homologi tidak ditampilkan).
Analisis jumlah perbedaan runutan nukleotida antar sampel menggunakan opsi model No. of differences, sedangkan jarak genetik dilakukan menggunakan opsi model subtitusi Kimura-2-parameter (K2P). Model K2P lebih efektif untuk barcoding, karena opsi tersebut mempertimbangkan tingkat
substitusi transisi dan transversi. Runutan nukleotida yang digunakan dalam barcode
bisa digunakan untuk mempelajari keragaman genetik, struktur populasi, genetika populasi, filogentik, dan taksonomi.
Jumlah perbedaan nukleotida terkecil terdapat pada Harpiosquilla stephensoni
(sampel Nomor 2) dengan Harpiosquilla harpax (referensi dari genebank), yaitu berbeda satu nukleotida. Jumlah perbedaan nukleotida terbesar terdapat antara sampel Nomor 9 (Carinosquilla multicarinata) dengan sampel Nomor 1 (Harpiosquilla stephensoni), yaitu berbeda 84 nukleotida
Untuk barcoding, standardisasi data udang mantis ini mempercepat pembentukan dan konstruksi pustaka sekuens DNA yang komprehensif dan konsisten sehingga dapat menjadi teknologi yang ekonomis untuk identifikasi spesies. Harapannya adalah setiap orang kapanpun dan di manapun dapat mengidentifikasi spesies dari spesimen secara cepat dan akurat bagaimanapun kondisi spesimen tersebut.
Taksonomi atau identifikasi merupakan salah satu dasar penting bagi segala aktifitas konservasi. Pelaksanaan konservasi bertujuan agar biodiversitas tidak mengalami kerusakan yang mengakibatkan rusaknya suatu ekosistem ataupun punahnya suatu spesies. Kemampuan teknik DNA barcode untuk mengidentifikasi spesies secara cepat dan akurat perlu diterapkan sebagai upaya memantau asal usul suatu komoditas laut. Gambar 2 Hasil rekonstruksi pohon filogeografi pengelompokan sampel berdasarkan ruas CO1
mtDNA menggunakan metode NJ dengan bootstrap 1000x.
6
Menurut Choi dan Hong (2001), udang mantis betina mampu menelurkan 50.000 hingga 1 juta telur. Namun, telur yang dapat perkembang hingga tambah juvenil sangat sedikit jumlahnya. Telur yang berasal dari induk yang sama tidak memiliki jarak genetik, sehingga barcoding juga dapat digunakan untuk memastikan kandungan material genetik.
Keragaman genetik menghasilkan jarak terkecil antara H. stephensoni (sampel Nomor 2) dengan H. harpax (referensi dari genebank) sebesar 0.002 (0.2%). Jarak genetik terbesar ditemukan pada sampel Nomor 9 (C.
multicarinata) dengan sampel Nomor 2 H. stephensoni, yaitu sebesar 0.234 (23.4%)
Perbedaan nukleotida dan jarak genetik antar spesies membuktikan bahwa spesies yang semula teridentifikasi berdasarkan morfologi saja masih mungkin terdapat kesalahan. Sampel H. stephensoni Nomor sampel 1 dan 2 lebih dekat dengan H. harpax
setelah dilakukan barcode (Tabel 2). Hal tersebut sesuai dengan yang dikemukakan oleh Barber dan Boyce (2006) bahwa pada ordo Stomatopoda terdapat fenomena spesies
cryptic (hampir mirip). Pengembangan pustaka barcode dapat dijadikan suatu cara identifikasi sampai tingkat spesies dengan tingkat kebenaran yang tinggi. Ruas basa dari gen CO1 bermutasi cukup cepat sehingga dapat membedakan spesies yang hampir mirip (Hebert et al. 2004).
Kebutuhan untuk melakukan standarisasi identifikasi spesies udang mantis sangat tinggi dengan munculnya berbagai masalah dalam metode identifikasi dan determinasi spesies yang ada saat ini. Permasalahan tersebut dapat berakibat pada kesamaan nama pada dua spesies yang berbeda, yang dapat dimungkinkan karena kesamaan morfologi. Selain itu dapat juga berakibat pada perbedaan nama pada satu spesies yang memiliki tingkat kehidupan yang sulit untuk diidentifikasi secara kasat mata. Pustaka barcode udang mantis memberikan keuntungan dari standardisasi metode dan bank identifikasi spesies melalui urutan sekuens DNA yang dimilikinya. Standardisasi ini tidak membutuhkan biaya yang sangat besar dan dapat memiliki tingkat kepercayaan yang cukup tinggi.
Keragaman genetik juga memperlihatkan jarak genetik yang berbeda antar spesies C. multicarinata asal Aceh dengan asal Cirebon sebesar 0.153 (15.3%). Sampel C. multicarinata asal Cirebon memiliki perbedaan genetik yang lebih tinggi dengan C.
multicarinata asal Aceh, dibandingkan dengan
C. multicarinata sesama asal Cirebon. Perbedaan tempat asal lokalitas memperlihatkan perbedaan pada jarak genetik. Topologi pohon filogeni menggunakan metode NJ dengan bootstrap1000x mengelompokkan sampel 9, 11, dan 22 (C. multicarinata) berada di luar percabangan, serta memisahkan C. multicarinata asal Aceh dengan C. multicarinata asal Cirebon. Hal tersebut menunjukkan bahwa spesies C. multicarinata asal Aceh memiliki tingkat kekerabatan yang berbeda dengan C. multicarinata asal Cirebon, sehingga pustaka
barcode dapat digunakan untuk studi kekerabatan suatu spesies. Sebaran geografi udang mantis melalui tingkat larva yang terbawa arus dan bergerak mengikuti garis pantai mungkin saja menyebabkan terjadi mutasi sebagai proses adaptasi.
SIMPULAN
Ruas gen CO1 sebagai barcode
menunjukkan bahwa sampel H. stephensoni
Nomor 1 dan 2 lebih dekat dengan H. harpax. Perbedaan nukleotida dan jarak genetik memisahkan spesies C. multicarinata asal Aceh dengan C. multicarinata asal Cirebon. Teknik barcode dapat digunakan untuk mengidentifikasi spesimen sebagai dasar dari konservasi keragaman spesies mantis.
SARAN
Saran bagi penelitian ini adalah diperlukan penelitian terhadap spesies udang mantis yang lain dengan daerah asal yang lebih beragam dan mengunakan ruas mitokondria yang berbeda atau selain CO1.
DAFTAR PUSTAKA
Ahyong S, Chan T, dan Liao Y. 2008. A Catalog of The Mantis Shrimps (Stomatopoda) of Taiwan. Taiwan: National Taiwan Ocean University. Barber P, Moosa MK, dan Palumbi SR. 2002.
7
Barber P dan Boyce SL. 2006. Estimating diversity of Indo-Pacific coral reef stomatopods through DNA barcoding of stomatopod larvae. Proc R Soc 273: 2053-2061.
Byun SO, Fang Q, Zhou H, dan Hickford JGH. 2009. An effective method for silver-staining DNA in large numbers of polyacrylamide gels. Anal Biochem 385: 174-175.
Carpenter KE dan Niem VH. 1998. The Living Marine Resources of The Western Central Pacific Volume 2: Cephalopods, Crustacean, Holothurians and Shark.
Rome: Food and Agriculture Organization of The United Nation.
[CBOL] The Consortium for the Barcode of Life. 2008. DNA Barcoding: A New Tool for Identifying Biological Specimens and Managing Spesies Diversity. Washington: CBOL.
Choi HJ dan Hong SY. 2001. Larval development of the kishi velvet shrimp, metapenaeopsis dalei (rathbun) (decapoda: penaeidae), reared in the laboratory. Fish Bull 99: 275-291.
Hebert PDN, Cywinska A, Ball SL, dan deWaard JR. 2003. Biological identification through DNA barcodes.
Proc R Soc 270: 313-321.
Hebert PDN, Ratnasingham S, dan deWaard JR. 2003. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species.
Proc R Soc 270: 96–99.
Hebert PDN, Pento EH, Burns JM, Janzen DH, Hallwachs W. 2004. Ten species in one: DNA barcoding reveals cryptic species in the neotropical skipper butterfly
Astraptes fulgerator. PNAS 101(41): 14812-14817.
Innis MA dan Gelfand DH. 1990. PCR Protocols: A Guide to Methods and Applications. San Diego: Academic Press. Ivanova NV, Zemlak TS, Hanner RH, dan
Hebert PDN. 2007. Barcoding: universal primer cocktails for fish DNA barcoding.
Mol Eco 10:1111-1115.
Lahaye et al. 2008. DNA barcoding the floras of biodiversity hotspots. PNAS 105(8): 2923-2928.
Manning RB. 1980. The superfamilies, families and genera of recent stomatopod Crustacea with diagnoses of six new families. Proc Biol Soc Wash 93: 362-372.
Meier R, Shiyang K, Vaidya G, dan Peter. 2006. DNA barcoding and taxonomy in
diptera: a tale of high intraspecific variability and low identification success.
Syst Biol 55(5): 715-728.
Moosa M. 2000. Marine biodiversity of the South China Sea: a checklist of stomatopod Crustacea. The Raffles Billetin of Zoology 8: 405-457.
Perna NT dan Kocher TD. 1996. Mitochondrial DNA: molecular fossils in the nucleus. Current Biology 6(2): 128-129.
Tamura K, Dudley J, Nei M dan Kumar S. 2007. MEGA4: Molecular evolutionary genetics analysis (MEGA) software version 4.0. Molecular Biology and Evolution 24: 1596-1599.
9
Satu ruas tungkai udang mantis dalam etanol dipotong dan dimasukkan ke dalam tube 1,5 ml. Ruas tungkai yang telah dipotong (otot tungkai) kemudian dicuci dengan air destilata sebanyak
2 kali ulangan menggunakan mesin sentrifuse.
Sampel yang telah dicuci kemudian dihomogenasi menggunkana grinder dan ditambahkan 200 µl buffer STE (NaCl 1M, Tris-HCL 10mM, EDTA 0.1mM, pH 8) ke dalam tube.
Sebanyak 50 mg/ml proteinase K 20 µl ditambahkan ke dalam tabung dan vortex selama beberapa detik.
Inkubasi pada suhu 600C selama 30 menit. Setiap 5 menit sekali tabung dibolak-balik secara perlahan.
Sebanyak 200 µl buffer GB ditambahkan ke dalam tabung dan vortex selama 5 detik.
Inkubasi pada suhu 700C selama 20 menit. Setiap 5 menit sekali tabung dibolak-balik secara perlahan. Pada saat yang bersamaan, inkubasi buffer elusi pada suhu yang sama untuk step elusi DNA.
Sebanyak 200 µl etanol ditambahkan ke dalam tabung dan vortex selama 10 detik. Sampel dipindahkan ke dalam kolom GD pada tube 2 ml.
Sentrifugasi 13.000 rpm selama 2 menit.
Kolom GD dipindahkan ke dalam tabung koleksi yang baru dan supernatan dibuang. Sebanyak 400 µl buffer W1 ditambahkan ke dalam tabung.
Sentrifugasi 13.000 rpm selama 30 detik.
Supernatan dibuang dan kolom GD diletakkan kembali ke dalam tabung.
Sebanyak 600 µl wash buffer (mengandung etanol) ditambahkan ke dalam kolom GD. Sentrifugasi 13.000 rpm selama 30 detik.
Supernatan dibuang dan kolom GD diletakkan kembali ke dalam tabung. Sentrifugasi 13.000 rpm selama 3 menit.
Kolom GD dipindahkan ke dalam tube 1,5 ml yang baru.
Sebanyak 100 µl buffer elusi yang telah diinkubasi ditambahkan ke dalam kolom GD (tepat bagian tengah matriks kolom GD).
Diamkan selama 5 menit.
Sentrifugasi 13.000 rpm selama 30 detik.
10
Gel yang telah dielektroforesis kemudian dikeluarkan dari kaca dan dibilas dengan DW (air destilata) sebanyak 200 ml.
Air destilata dibuang*, gel direndam dalam larutan A.
Gel direndam dalam larutan A selama 8 menit. Pada saat yang bersamaan, larutan B dipanaskan pada suhu 550C.
Kemudian air larutan A dibuang ke dalam botol khusus Ag. Gel dibilas dengan DW sebanyak 200 ml, kemudian DW dibuang. Gel direndam dalam larutan B yang telah ditambahkan formaldehid. Gel direndam hingga muncul pita.
Air larutan B dibuang
Gel direndam dalam larutan C selama 2 menit.
Catatan:
Larutan A: DW 200 ml
AgNO3 0,2 gram
NaOH 10N 80 µl (sebaiknya dibuat fresh)
amonia 0,8 ml Larutan B: DW 200 ml
NaOH 6 gram
Formaldehid 100 µl ( ditambahkan sesaat sebelum dituang) Larutan C: DW 100 ml
Asetat 100 µl
11
Lampiran 3 Hasil pensejajaran tujuh runutan DNA mantis dengan referensi genbank sepanjang 430 nt
1 100
1 1_H. stephensoni TTGATTATTCGTGCAGAATTAGGACAGCCTGGTAGGCTAATTGGAGATGATCAAATTTACAACGTTATTGTTACAGCACACGCTTTCGTTATAATTTTTT
2 2_H. stephensoni TTGATTATTCGTGCAGAATTAGGACAGCCTGGTAGGCTAATTGGAGATGATCAAATTTACAACGTTATTGTTACAGCACACGCTTTCGTTATAATTTTTT
3 9_C. multicarinata TTTATCATTCGAGCAGAACTAGGACAACCAGGTAGTTTAATTGGAGACGACCAAATTTATAATGTTATCGTTACAGCCCATGCTTTCATTATGATTTTTT
4 11_C. multicarinata TTAATCATTCGAGCAGAACTAGGACAACCAGGTAGTTTAATTGGAGACGACCAAATTTATAATGTTATCGTTACAGCCCATGCTTTCATTATGATTTTTT 5 22_C. multicarinata TTAATTATTCGAGCAGAATTAGGACAACCAGGTAGATTAATTGGAGATGATCAAATCTACAACGTTATTGTTACAGCACATGCTTTTATTATAATTTTTT
6 34_H. harpax TTAATCATTCGAGCCGAATTAGGGCAACCCGGTAGGTTAATTGGAGATGATCAAATTTATAATGTTATTGTCACAGCCCACGCCTTTATTATAATTTTTT
7 37_H. harpax TTAATCATTCGAGCCGAATTAGGGCAACCCGGTAGGTTAATTGGAGATGATCAAATTTATAATGTTATTGTCACAGCCCACGCCTTTATTATAATTTTTT
8 H. glyptocercus TTAATTATTCGAGCAGAATTAGGACAACCCGGTAGATTAATTGGAGACGATCAAATCTACAACGTTGTAGTCACAGCCCATGCCTTCATTATAATTTTTT
9 H. harpax TTGATTATTCGTGCAGAATTAGGACAGCCTGGTAGGCTAATTGGAGATGATCAAATTTACAACGTTATTGTTACAGCACACGCTTTCGTTATAATTTTTT
10 S. mantis TTGATTATTCGAGCTGAGCTAGGTCAACCAGGTAGGTTAATTGGAGATGACCAAATCTACAATGTTATCGTTACAGCACACGCTTTTGTTATAATTTTTT
101 200
201 300
1 TTTTTGACTACTACCGCCTGCACTTACTTTACTTTTATGTAGTGGATTAGTAGAAAGAGGGGTAGGAACAGGATGAACAGTTTATCCTCCTTTATCAGCG
2 TTTTTGACTACTACCGCCTGCACTTACTTTACTTTTATGTAGTGGATTAGTAGAAAGAGGGGTAGGAACAGGATGAACAGTTTATCCTCCTTTATCAGCG
3 ATTCTGATTATTACCACCTGCTCTCACGCTTTTACTCTCAAGTGGCTTAGTAGAAAGAGGAGTAGGAACAGGATGAACGGTTTACCCTCCTTTATCTGCA
1 TCATGGTTATACCAATTATAATTGGAGGTTTTGGAAACTGATTAGTTCCTTTAATGTTAGGGGCCCCAGATATAGCCTTCCCCCGTATAAACAACATAAG
2 TCATGGTTATACCAATTATAATTGGAGGTTTTGGAAACTGATTAGTTCCTTTAATGTTAGGGGCCCCAGATATAGCCTTCCCTCGTATAAACAACATAAG
3 TTATGGTAATGCCAATTATAATTGGAGGGTTTGGGAATTGACTAGTCCCTCTTATACTAGGAGCTCCTGATATAGCTTTCCCTCGAATAAACAATATGAG
4 TTATGGTAATGCCAATTATAATTGGAGGGTTTGGGAATTGACTAGTCCCTCTTATACTAGGAGCTCCTGATATAGCTTTCCCTCGAATAAACAATATGAG
5 TTATGGTTATACCAATCATAATTGGAGGTTTCGGGAATTGATTAGTACCACTTATATTAGGAGCCCCTGATATGGCATTTCCCCGTATAAACAACATAAG
6 TTATGGTAATACCAATTATAATTGGAGGTTTCGGAAACTGATTAGTTCNCTTGATATTGGGGGCCCCAGATATAGCCTTCCCACGAA-AAATAATATAAG
7 TTATGGTAATACCAATTATAATTGGAGGTTTCGGAAACTGATTAGTTCCCTTGATATTGGGAGCCCCAGATATAGCCTTCCCACGAATAAATAATATAAG
8 TTATAGTTATACCAATTATAATTGGAGGATTTGGAAACTGGCTAGTGCCTTTAATGCTAGGGGCACCTGATATGGCTTTCCCCCGAATAAATAACATAAG
9 TCATGGTTATACCAATTATAATTGGAGGTTTTGGAAACTGATTAGTTCCTTTAATGTTAGGGGCCCCAGATATAGCCTTCCCTCGTATAAACAACATAAG
12
4 ATTCTGATTATTACCACCTGCTCTCACGCTTTTACTCTCAAGTGGCTTAGTAGAAAGAGGAGTAGGAACAGGATGAACGGTTTACCCTCCTTTATCTGCA
5 ATTTTGATTATTACCACCAGCTCTTACTCTCCTTTTATCAAGAGGCCTAGTAGAAAGAGGAGTTGGAACAGGATGAACTGTTTACCCTCCTTTGTCTGCA
6 ATTTTGGTTACTACCCCCAGCTCTCACACTTCTTTTATCAAGAGGTCTAGTAGAAA-AGGAGTTGGGACCGGATGAACCGTTTATCCCCCACTATCTGCT
7 ATTTTGGTTACTACCCCCAGCTCTCACACTTCTTTTATCAAGAGGTCTAGTAGAAAGAGGAGTTGGGACCGGATGAACCGTTTATCCCCCACTATCTGCT
8 ATTTTGATTACTTCCCCCCGCACTTACTTTATTACTTTCAAGAGGTATAGTAGAAAGAGGAGTAGGAACAGGATGAACAGTTTATCCTCCTTTAGCCGCC
9 TTTTTGACTACTACCGCCTGCACTTACTTTACTTTTATGTAGTGGATTAGTAGAAAGAGGAGTAGGAACAGGATGAACAGTTTATCCTCCTTTATCAGCG
10 ATTTTGATTACTACCTCCCGCACTCACCTTATTACTATCTAGGGGCTTAGTTGAAAGAGGGGTTGGTACTGGATGAACAGTTTATCCCCCTTTATCAGCA
301 400
1 GGAATTGCTCATGCTGGGGCTTCAGTAGACATGGGTATTTTTTCTTTACACTTAGCCGGAGCTTCATCAATTTTAGGAGCTGTTAACTTCATTACAACAG
2 GGAATTGCTCATGCTGGGGCTTCAGTAGACATGGGTATTTTTTCTTTACACTTAGCCGGAGCTTCATCAATTTTAGGAGCTGTTAACTTCATTACAACAG
3 GGAATTGCACACGCAGGAGCGTCTGTGGATATGGGTATTTTTTCTTTACATCTAGCAGGGGCCTCTTCGATTTTAGGGGCAGTAAACTTTATTACTACCG
4 GGAATTGCACACGCAGGAGCGTCTGTGGATATGGGTATTTTTTCTTTACATCTAGCAGGGGCTTCTTCGATTTTAGGGGCAGTAAACTTTATTACTACCG
5 GGAATTGCTCATGCAGGAGCTTCTGTAGACATGGGTATTTTTTCGTTACATTTAGCAGGAGCTTCTTCTATTTTAGGTGCCGTAAACTTCATTACTACTG
6 GGAATCGCACACGCAGGGGCTTCAGTAGATATGGGTATTTTTTCTCTACACATAGACGGAGGT-CATCTAATGTAGGAGCTGTAA-TTTTATTACAACCG
7 GGAATCGCACACGCAGGGGCTTCAGTAGATATGGGTATTTTTTCTCTACACCTAGCCGGAGCTTCATCTATCTTAGGAGCTGTAAATTTTATTACAACCG
8 GGAATTGCCCACGCAGGAGCGTCTGTAGATTTAGGAATTTTTTCATTGCACATAGCAGGAGCTTCATCAATCCTAGGAGCAGTAAACTTTATTACAACAG
9 GGAATTGCTCATGCTGGGGCTTCAGTAGACATGGGTATTTTTTCTTTACACTTAGCCGGAGCTTCATCAATTTTAGGAGCTGTTAACTTCATTACAACAG
10 GGAATTGCGCATGCCGGGGCTTCTGTAGATATGGGTATTTTCTCTTTACATTTAGCAGGAGCTTCTTCAATTTTAGGAGCTGTAAATTTCATTACTACGG
401 430
1 TTATTAATATACGATCAAATGGGATAACTA
2 TTATTAATATACGATCAAATGGGATAACTA
3 TAATCAATATACGATCTAATGGAATAACTA
4 TAATCAATATACGATCTAATGGAATAACTA
5 TAATCAACATACGATCTAACGGAATAACTA
6 TAATTAATATCCG-TCCAACGGAATAACTA
7 TAATTAATATACGATCCAACGGAATAACTA
8 TTATTAATATACGATCTAACGGAATAACAA
9 TTATTAATATACGATCAAATGGGATAACTA
ABSTRAK
RAISA AULIANE SYAFRINA. Penggunaan DNA Barcode sebagai Alternatif Identifikasi Spesies Udang Mantis. Dibimbing oleh ACHMAD FARAJALLAH dan YUSLI WARDIATNO.
Udang mantis atau yang dikenal dengan udang ronggeng merupakan salah satu anggota Subfilum Crustacea Ordo Stomatopoda, Beberapa spesies udang mantis dikenal sebagai komoditi ekspor dan makanan eksotis. Beberapa udang mantis yang bernilai ekonomi tinggi adalah anggota famili Harpiosquillidae dan Squillidae. Udang-udang mantis tersebut biasa ditangkap dari Laut Jawa dan Laut Cina Selatan. Penelitian ini bertujuan untuk mengaplikasikan teknik DNA
barcoding sebagai laternatif mengidentifikasi udang mantis sampai ke tingkat spesies. Sebanyak 34 sampel udang mantis berasal dari Jambi, Cirebon, dan Aceh yang digunakan adalah spesimen koleksi yang diawetkan dalam alkohol. Identifikasi spesies dilakukan berdasarkan morfologi. Ekstraksi DNA dilakukan dari otot tungkai dan ruas gen CO1 diamplifikasi sebagai ruas DNA yang dijadikan barcode. Amplifikasi gen CO1 berhasil dilakukan untuk semua sampel yang kesemuanya menghasilkan amplikon yang multiband. Perunutan nukelotida hanya berhasil dilakukan untuk tujuh sampel, yaitu Harpiosquilla harpax (Sampel No. 34 dan No. 37 asal Jambi),
Harpiosquilla stephensoni (sampel No. 1 dan No. 2 asal Cirebon), dan Carinosquilla multicarinata
(sampel No. 9 dan No. 11 asal Cirebon, dan No. 22 asal Aceh). Berdasarkan ruas gen CO1, C. multicarinata asal Aceh memiliki kekerabatan yang berbeda dengan C. multicarinata asal Cirebon.
ABSTRACT
RAISA AULIANE SYAFRINA. DNA Barcode as an Alternative of Mantis Shrimp Species Identification. Supervised by ACHMAD FARAJALLAH and YUSLI WARDIATNO.
PENDAHULUAN
Latar Belakang
Barcode DNA adalah urutan pendek DNA yang digunakan untuk mengidentifikasi spesies secara cepat dan akurat (Meier et al.
2006). Ruas DNA yang bisa digunakan untuk sistem tersebut harus terstandardisasi.
Barcode DNA juga menjadi salah satu alternatif pelengkap atau komplemen yang dapat memperkuat identifikasi morfologi secara cepat dan akurat (Lahaye et al. 2008).
Barcode DNA menjanjikan beberapa manfaat, antara lain mengenali spesies, memastikan keamanan pangan, mengidentifikasi fase larva yang berbeda dengan fase dewasa, mengontrol hama pertanian, dan melacak asal usul vektor penyakit dan serangan hama pada suatu area.
Pada hewan, penggunaan genom mitokondria (mtDNA) dalam analisis biogeografi dan sistematik sering tidak sejalan dengan morfologi. Salah satu penyebabnya adalah karakter morfologi yang seringkali memperlihatkan fenomena species cryptic
(hampir mirip). Genom mitokondria hewan merupakan genom sitoplasmik yang diwariskan secara uniparental dan tidak mengalami rekombinasi sehingga species sibling bisa dipastikan mempunyai mtDNA dengan nilai kesamaan yang tinggi. Salah satu ruas mtDNA yang banyak digunakan sebagai
barcode adalah cytochrome oxidase 1 (CO1) genom mitokondria yang dipopulerkan oleh Hebert et al. (2003). Gen CO1 pada Crustacea berukuran sekitar 1500 pb. Dibandingkan dengan ruas-ruas gen yang lain dalam mtDNA, gen CO1 memberikan hasil yang lebih efektif dan mudah untuk diakses. Teknologi barcoding menggunakan penanda gen CO1 dari mtDNA dapat digunakan untuk mengidentifikasi hampir semua spesies hewan (Ward et al. 2005), baik interspesifik maupun intraspesifik (Hebert et al. 2003).
Udang mantis atau yang dikenal dengan udang ronggeng merupakan anggota subfilum Crustacea, Ordo Stomatopoda, yang terdiri atas empat famili, yaitu Odontodactylidae, Lysiosquillidae, Harpio-squillidae dan Squilidae. Beberapa spesies udang mantis, terutama yang bisa mencapai ukuran >30 cm, biasa dijadikan sebagai komoditi ekspor dan makanan eksotis dengan harga yang relatif mahal (Ahyong et al. 2008). Kelompok udang ini dicirikan dengan tubuh yang bersegmen, di belakang kepala terdapat karapas pendek, kaki beruas-ruas, ukuran tubuh yang besar dan mata seringkali berbentuk T (Carpenter & Niem 1998).
Beberapa udang mantis yang bernilai ekonomi tinggi adalah dari famili Harpiosquillidae dan Squilidae. Kedua famili tersebut biasa ditangkap dari Laut Jawa dan Laut Cina Selatan. Persebaran udang mantis di kedua wilayah tersebut dipengaruhi migrasi larva udang mantis mengikuti pergerakan arus air laut yang sejajar garis pantai (Barber et al.
2002). Selain itu, dari 450 spesies yang telah dideskripsikan, 118 (26%) diantaranya bisa ditemukan di perairan Indonesia (Ahyong et al. 2008, Moosa 2000). Jumlah spesies udang mantis yang sangat tinggi di Indonesia (jika dibandingkan dengan laut Brazilia dengan 35 spesies dan laut Mediterania 10 spesies) tidak sejalan dengan keterkenalannya sebagai bagian dari kekayaan biodiversitas Indonesia.
Oleh karena itu, dalam penelitian ini digunakan teknik DNA barcode untuk mengidentifikasi keanekaragaman spesies udang mantis yang terdapat di perairan Indonesia. Menurut Konvensi Keaneka-ragaman Hayati, barcode menjadi salah satu teknik yang berkontribusi cukup signifikan terhadap pelaksanaan konservasi keragaman spesies. Konvensi Keanekaragaman Hayati (Convention on Biological Diversity atau CBD) adalah perjanjian internasional yang bertujuan mengembangkan strategi nasional untuk konservasi dan pemanfaatan berkelanjutan keanekaragaman hayati. Dengan begitu, aplikasi DNA barcoding dapat menjawab klaim kepemilikan dan asal lokalitas komoditas perdagangan suatu wilayah (CBOL 2008).
Tujuan
Penelitian ini bertujuan untuk menggunakan teknik DNA barcoding sebagai alternatif identifikasi keanekaragaman spesies udang mantis yang terdapat di perairan Indonesia.
Waktu dan Tempat
HASIL
Identifikasi Morfologi
Hasil identifikasi berdasarkan buku identifikasi Manning (1980) menunjukkan bahwa spesimen udang mantis koleksi terdiri atas 2 famili dan 6 spesies (Tabel 1).
Tabel 1 Hasil identifikasi spesimen udang mantis
Spesies No. Sampel
Famili Hapriosquillidae
Harpiosquilla stephensoni 1,2,3,4,5,6, 8, 10, 21
Harpiosquilla harpax 31, 34, 35, 37
Harpiosquilla raphidae 30
Harpiosquilla melanoura 23 Famili Squillidae
Carinosquilla multicarinata 9, 11, 12, 15, 16, 17, 18, 19, 20, 22, 26,
Keijia lirata 7, 13, 14, 25,
Harpiosquilla harpax memiliki ciri karapas dilengkapi dengan median carina dan distal segmen pada uropod berwarna hitam dengan garis tengah putih, carina intermediate pada thoraks tidak terlalu tajam, rostral dilengkapi dengan projeksi anterior, dan marginal carina dua kali lebih panjang dari carina gigi lateral. Harpiosquilla stephensoni
memiliki ciri karapas dilengkapi dengan median carina dan distal segmen pada uropod berwarna hitam dengan garis tengah putih, carina intermediate pada thoraks tidak terlalu tajam, rostral tidak dilengkapi dengan projeksi anterior, dan tajam pada thoraks kelima.
Carinosquilla multicarinata memiliki ciri terdapat duri ganda pada ujung thoraks kelima, thoraks dan abdomen ditutupi dengan carina longitudinal, carina pada thoraks dan abdomen terdapat duri.
Amplifikasi dan Visualisasi DNA
Gen CO1 target diamplifikasi menggunakan pasangan primer AF286-AF287 dan AF215-AF287 berukuran sekitar 700-800 bp (Gambar 1), dengan jumlah sampel sebanyak 34 ekor. Pada beberapa sampel suhu optimum penempelan primer pada saat amplifikasi yaitu 55°C.
M 1 2 3 4 5 6 7 8 9 10 11
Gambar 1 Amplikon gen CO1 di atas PAGE 6%. Keterangan: M. Marker; 1-11. Nomor sampel.
Hasil amplifikasi menunjukkan fragmen DNA multiband. Pita DNA target berhasil ditemukan dari pasangan primer 286-AF287 sebanyak 12 sampel, sedangkan dari pasangan primer AF215-AF287 sebanyak 14 sampel. Dari total sampel yang berhasil diamplifiaksi menampakkan pita DNA target, hanya 7 sampel dari pasangan primer AF286-AF287 dan 13 yang dijadikan cetakan dalam PCR for sequencing karena beberapa pita DNA muncul tipis pada gel poliakrilamid saat proses visualisasi. Setelah DNA target dimurnikan dan dijadikan cetakan dalam PCR for sequencing, maka diperoleh 7 sampel yang terbaca dengan jelas. Ketujuh sampel tersebut adalah 1H. stephensoni; 2H. stephensoni; 9C. multicarinata; dan 11C. multicarinata
(Cirebon), 22C. multicarinata (Aceh), dan 34H. harpax; 37 H. harpax (Jambi).
Analisis DNA dan Filogeni
Tujuh runutan DNA mantis yang diperoleh kemudian saling disejajarkan dengan ruas mtDNA yang homolog dari Haptosquilla glyptocercus (AF205239), Harpiosquilla harpax (FJ229770), dan Squilla mantis
(GQ328967). Data runutan nukleotida yang saling homolog antar sampel dengan data referensi yang bisa dilakukan analisis lebih lanjut adalah sepanjang 430 nt (Lampiran 3).
Sampel dengan jumlah perbedaan nukleotida paling kecil terjadi antara sampel Nomor 2 yang diduga sebagai Harpiosquilla stephensoni dengan Harpiosquilla harpax dari
genebank, yaitu hanya berbeda satu nukleotida. Sampel dengan jumlah perbedaan nukleotida paling tinggi terdapat antara sampel Nomor 9 yang diduga sebagai
Carinosquilla multicarinata dengan sampel Nomor 1 (Harpiosquilla stephensoni), yaitu berbeda 84 nukleotida (Tabel 3).
Jarak genetik antar sampel dibandingkan dengan sampel spesies yang ada di GeneBank
4
model substitusi K2P (Tabel 4) menghasilkan jarak genetik tertinggi sebesar 0.234 (23.4%) ditemukan antara sampel Nomor 9 (C.
multicarinata) dengan sampel Nomor 2 H. stephensoni. Jarak genetik terendah sebesar 0.002 (0.2%) antara sampel Nomor H. harpax
dengan sampel Nomor 1 (H. stephensoni). Topologi pohon filogeni menggunakan
metode NJ dengan bootstrap1000x
mengelompokkan sampel Nomor 9, 11, dan 22 (Carinosquilla multicarinata) berada di luar percabangan. Struktur populasi yang digambarkan oleh mtDNA ini menunjukkan bahwa kekerabatan sampel Nomor 1, 2, 34, dan 37 adalah berasal dari satu nenek moyang/indukan, sedangkan populasi sampel
C. multicarinata berbeda nenek moyang/indukan (Gambar 2).
Table 2 Data spesies hasil identifikasi dan barcode
No. sampel Identifikasi Asal lokasi
Identifikasi awal Barcode
1 Harpiosquilla stephensoni Harpiosquilla harpax Cirebon 2 Harpiosquilla stephensoni Harpiosquilla harpax Cirebon 9 Carinosquilla multicarinata Carinosquilla multicarinata Cirebon 11 Carinosquilla multicarinata Carinosquilla multicarinata Cirebon 22 Carinosquilla multicarinata Carinosquilla multicarinata Aceh 34 Harpiosquilla harpax Harpiosquilla harpax Jambi 37 Harpiosquilla harpax Harpiosquilla harpax Jambi
Tabel 3 Jumlah perbedaan nukleotida gen CO1 antar spesies udang mantis
Sampel 1 2 3 4 5 6 7 8 9 10
1 34_H. harpax
2 37_H. harpax 8 3 1_H. stephensoni 72 69 4 2_H. stephensoni 72 69 1 5 H. harpax 1) 71 68 2 1 6 H. glyptocercus 2) 78 75 69 70 69 7 S. mantis 3) 75 72 57 58 59 72 8 9_C. multicarinata 80 74 84 83 82 73 74 9 11_C. multicarinata 78 72 83 82 81 71 73 2
10 22_C. multcarinata 72 67 64 65 64 72 65 60 58
1)
GenBank Accession Number FJ229770
2) GenBank Accession Number AF205239 3)
GenBank Accession Number GQ328967
Tabel 4 Jarak genetik gen CO1 antar spesies udang mantis berdasarkan model subtitusi K2P
Sampel 1 2 3 4 5 6 7 8 9 10
1 34_H. harpax
2 37_H. harpax 0.019 3 1_H. stephensoni 0.194 0.185
4 2_H. stephensoni 0.194 0.185 0.002 5 H. harpax 0.191 0.182 0.005 0.002 6 H. glyptocercus 0.212 0.204 0.185 0.188 0.185 7 S. mantis 0.203 0.194 0.149 0.152 0.155 0.194
8 9_C. multicarinata 0.221 0.203 0.234 0.231 0.227 0.199 0.202 9 11_C. multicarinata 0.214 0.196 0.231 0.228 0.224 0.193 0.199 0.005
5
1Hstephensoni 2Hstephensoni Hharpax Smantis Hglyptocercus 34Hharpax 37Hharpax 22Cmultcarinatai 9Cmulticarinata 11Cmulticarinata 100 100 58 100 70 73 34 0.02PEMBAHASAN
Hasil amplifikasi gen CO1 menggunakan pasangan primer AF215/AF286 dan AF287 selalu menghasilkan amplikon yang multiband. Salah satu penyebab utama adalah penggunaan degenerate primer. Dalam hal ini, ada C dan T pada basa ketiga dari ujung 3’ untuk forward primer, dan ada A dan G pada basa keenam dari ujung 3’ untuk reverse primer. Menurut Innis (1990), spesifisitas tiga basa di ujung 3’ sangat menentukan hasil amplifikasi. Degenerate primer biasa digunakan untuk menjamin keberhasilan amplifikasi pada taksa yang luas (Hebert et al.
2003). Alternatif lain untuk mengakomodasi taksa yang luas adalah penggunaan beberapa primer yang disambungkan dengan runutan M13 universal. Primer M13 lebih efektif untuk barcode pada taksonomi sampel yang lebih beragam (Ivanova et al. 2007). PCR for sequencing kemudian menggunakan primer M13 universal ini. Selain masalah degenerate primer, gen CO1 dilaporkan juga ada di dalam genom inti (Perna & Kocher 1996) dan dalam kromosom beberapa spesies bakteri dengan tingkat kesamaan >70% (data analisis homologi tidak ditampilkan).
Analisis jumlah perbedaan runutan nukleotida antar sampel menggunakan opsi model No. of differences, sedangkan jarak genetik dilakukan menggunakan opsi model
subtitusi Kimura-2-parameter (K2P). Model
K2P lebih efektif untuk barcoding, karena opsi tersebut mempertimbangkan tingkat
substitusi transisi dan transversi. Runutan nukleotida yang digunakan dalam barcode
bisa digunakan untuk mempelajari keragaman genetik, struktur populasi, genetika populasi, filogentik, dan taksonomi.
Jumlah perbedaan nukleotida terkecil terdapat pada Harpiosquilla stephensoni
(sampel Nomor 2) dengan Harpiosquilla
harpax (referensi dari genebank), yaitu berbeda satu nukleotida. Jumlah perbedaan nukleotida terbesar terdapat antara sampel Nomor 9 (Carinosquilla multicarinata) dengan sampel Nomor 1 (Harpiosquilla stephensoni), yaitu berbeda 84 nukleotida
Untuk barcoding, standardisasi data udang mantis ini mempercepat pembentukan dan konstruksi pustaka sekuens DNA yang komprehensif dan konsisten sehingga dapat menjadi teknologi yang ekonomis untuk identifikasi spesies. Harapannya adalah setiap orang kapanpun dan di manapun dapat mengidentifikasi spesies dari spesimen secara cepat dan akurat bagaimanapun kondisi spesimen tersebut.
Taksonomi atau identifikasi merupakan salah satu dasar penting bagi segala aktifitas konservasi. Pelaksanaan konservasi bertujuan agar biodiversitas tidak mengalami kerusakan yang mengakibatkan rusaknya suatu ekosistem ataupun punahnya suatu spesies. Kemampuan teknik DNA barcode untuk mengidentifikasi spesies secara cepat dan akurat perlu diterapkan sebagai upaya memantau asal usul suatu komoditas laut. Gambar 2 Hasil rekonstruksi pohon filogeografi pengelompokan sampel berdasarkan ruas CO1
mtDNA menggunakan metode NJ dengan bootstrap 1000x.
6
Menurut Choi dan Hong (2001), udang mantis betina mampu menelurkan 50.000 hingga 1 juta telur. Namun, telur yang dapat perkembang hingga tambah juvenil sangat sedikit jumlahnya. Telur yang berasal dari induk yang sama tidak memiliki jarak genetik, sehingga barcoding juga dapat digunakan untuk memastikan kandungan material genetik.
Keragaman genetik menghasilkan jarak terkecil antara H. stephensoni (sampel Nomor 2) dengan H. harpax (referensi dari genebank) sebesar 0.002 (0.2%). Jarak genetik terbesar ditemukan pada sampel Nomor 9 (C.
multicarinata) dengan sampel Nomor 2 H. stephensoni, yaitu sebesar 0.234 (23.4%)
Perbedaan nukleotida dan jarak genetik antar spesies membuktikan bahwa spesies yang semula teridentifikasi berdasarkan morfologi saja masih mungkin terdapat kesalahan. Sampel H. stephensoni Nomor sampel 1 dan 2 lebih dekat dengan H. harpax
setelah dilakukan barcode (Tabel 2). Hal tersebut sesuai dengan yang dikemukakan oleh Barber dan Boyce (2006) bahwa pada ordo Stomatopoda terdapat fenomena spesies
cryptic (hampir mirip). Pengembangan pustaka barcode dapat dijadikan suatu cara identifikasi sampai tingkat spesies dengan tingkat kebenaran yang tinggi. Ruas basa dari gen CO1 bermutasi cukup cepat sehingga dapat membedakan spesies yang hampir mirip (Hebert et al. 2004).
Kebutuhan untuk melakukan standarisasi identifikasi spesies udang mantis sangat tinggi dengan munculnya berbagai masalah dalam metode identifikasi dan determinasi spesies yang ada saat ini. Permasalahan tersebut dapat berakibat pada kesamaan nama pada dua spesies yang berbeda, yang dapat dimungkinkan karena kesamaan morfologi. Selain itu dapat juga berakibat pada perbedaan nama pada satu spesies yang memiliki tingkat kehidupan yang sulit untuk diidentifikasi secara kasat mata. Pustaka barcode udang mantis memberikan keuntungan dari standardisasi metode dan bank identifikasi spesies melalui urutan sekuens DNA yang dimilikinya. Standardisasi ini tidak membutuhkan biaya yang sangat besar dan dapat memiliki tingkat kepercayaan yang cukup tinggi.
Keragaman genetik juga memperlihatkan jarak genetik yang berbeda antar spesies C. multicarinata asal Aceh dengan asal Cirebon sebesar 0.153 (15.3%). Sampel C. multicarinata asal Cirebon memiliki perbedaan genetik yang lebih tinggi dengan C.
multicarinata asal Aceh, dibandingkan dengan
C. multicarinata sesama asal Cirebon. Perbedaan tempat asal lokalitas memperlihatkan perbedaan pada jarak genetik. Topologi pohon filogeni menggunakan
metode NJ dengan bootstrap1000x
mengelompokkan sampel 9, 11, dan 22 (C. multicarinata) berada di luar percabangan, serta memisahkan C. multicarinata asal Aceh dengan C. multicarinata asal Cirebon. Hal tersebut menunjukkan bahwa spesies C. multicarinata asal Aceh memiliki tingkat kekerabatan yang berbeda dengan C. multicarinata asal Cirebon, sehingga pustaka
barcode dapat digunakan untuk studi kekerabatan suatu spesies. Sebaran geografi udang mantis melalui tingkat larva yang terbawa arus dan bergerak mengikuti garis pantai mungkin saja menyebabkan terjadi mutasi sebagai proses adaptasi.
SIMPULAN
Ruas gen CO1 sebagai barcode
menunjukkan bahwa sampel H. stephensoni
Nomor 1 dan 2 lebih dekat dengan H. harpax. Perbedaan nukleotida dan jarak genetik memisahkan spesies C. multicarinata asal Aceh dengan C. multicarinata asal Cirebon. Teknik barcode dapat digunakan untuk mengidentifikasi spesimen sebagai dasar dari konservasi keragaman spesies mantis.
SARAN
Saran bagi penelitian ini adalah diperlukan penelitian terhadap spesies udang mantis yang lain dengan daerah asal yang lebih beragam dan mengunakan ruas mitokondria yang berbeda atau selain CO1.
DAFTAR PUSTAKA
Ahyong S, Chan T, dan Liao Y. 2008. A Catalog of The Mantis Shrimps (Stomatopoda) of Taiwan. Taiwan: National Taiwan Ocean University. Barber P, Moosa MK, dan Palumbi SR. 2002.
6
Menurut Choi dan Hong (2001), udang mantis betina mampu menelurkan 50.000 hingga 1 juta telur. Namun, telur yang dapat perkembang hingga tambah juvenil sangat sedikit jumlahnya. Telur yang berasal dari induk yang sama tidak memiliki jarak genetik, sehingga barcoding juga dapat digunakan untuk memastikan kandungan material genetik.
Keragaman genetik menghasilkan jarak terkecil antara H. stephensoni (sampel Nomor 2) dengan H. harpax (referensi dari genebank) sebesar 0.002 (0.2%). Jarak genetik terbesar ditemukan pada sampel Nomor 9 (C.
multicarinata) dengan sampel Nomor 2 H. stephensoni, yaitu sebesar 0.234 (23.4%)
Perbedaan nukleotida dan jarak genetik antar spesies membuktikan bahwa spesies yang semula teridentifikasi berdasarkan morfologi saja masih mungkin terdapat kesalahan. Sampel H. stephensoni Nomor sampel 1 dan 2 lebih dekat dengan H. harpax
setelah dilakukan barcode (Tabel 2). Hal tersebut sesuai dengan yang dikemukakan oleh Barber dan Boyce (2006) bahwa pada ordo Stomatopoda terdapat fenomena spesies
cryptic (hampir mirip). Pengembangan pustaka barcode dapat dijadikan suatu cara identifikasi sampai tingkat spesies dengan tingkat kebenaran yang tinggi. Ruas basa dari gen CO1 bermutasi cukup cepat sehingga dapat membedakan spesies yang hampir mirip (Hebert et al. 2004).
Kebutuhan untuk melakukan standarisasi identifikasi spesies udang mantis sangat tinggi dengan munculnya berbagai masalah dalam metode identifikasi dan determinasi spesies yang ada saat ini. Permasalahan tersebut dapat berakibat pada kesamaan nama pada dua spesies yang berbeda, yang dapat dimungkinkan karena kesamaan morfologi. Selain itu dapat juga berakibat pada perbedaan nama pada satu spesies yang memiliki tingkat kehidupan yang sulit untuk diidentifikasi secara kasat mata. Pustaka barcode udang mantis memberikan keuntungan dari standardisasi metode dan bank identifikasi spesies melalui urutan sekuens DNA yang dimilikinya. Standardisasi ini tidak membutuhkan biaya yang sangat besar dan dapat memiliki tingkat kepercayaan yang cukup tinggi.
Keragaman genetik juga memperlihatkan jarak genetik yang berbeda antar spesies C. multicarinata asal Aceh dengan asal Cirebon sebesar 0.153 (15.3%). Sampel C. multicarinata asal Cirebon memiliki perbedaan genetik yang lebih tinggi dengan C.
multicarinata asal Aceh, dibandingkan dengan
C. multicarinata sesama asal Cirebon. Perbedaan tempat asal lokalitas memperlihatkan perbedaan pada jarak genetik. Topologi pohon filogeni menggunakan
metode NJ dengan bootstrap1000x
mengelompokkan sampel 9, 11, dan 22 (C. multicarinata) berada di luar percabangan, serta memisahkan C. multicarinata asal Aceh dengan C. multicarinata asal Cirebon. Hal tersebut menunjukkan bahwa spesies C. multicarinata asal Aceh memiliki tingkat kekerabatan yang berbeda dengan C. multicarinata asal Cirebon, sehingga pustaka
barcode dapat digunakan untuk studi kekerabatan suatu spesies. Sebaran geografi udang mantis melalui tingkat larva yang terbawa arus dan bergerak mengikuti garis pantai mungkin saja menyebabkan terjadi mutasi sebagai proses adaptasi.
SIMPULAN
Ruas gen CO1 sebagai barcode
menunjukkan bahwa sampel H. stephensoni
Nomor 1 dan 2 lebih dekat dengan H. harpax. Perbedaan nukleotida dan jarak genetik memisahkan spesies C. multicarinata asal Aceh dengan C. multicarinata asal Cirebon. Teknik barcode dapat digunakan untuk mengidentifikasi spesimen sebagai dasar dari konservasi keragaman spesies mantis.
SARAN
Saran bagi penelitian ini adalah diperlukan penelitian terhadap spesies udang mantis yang lain dengan daerah asal yang lebih beragam dan mengunakan ruas mitokondria yang berbeda atau selain CO1.
DAFTAR PUSTAKA
Ahyong S, Chan T, dan Liao Y. 2008. A Catalog of The Mantis Shrimps (Stomatopoda) of Taiwan. Taiwan: National Taiwan Ocean University. Barber P, Moosa MK, dan Palumbi SR. 2002.
PENGGUNAAN DNA
BARCODE
SEBAGAI ALTERNATIF
IDENTIFIKASI SPESIES UDANG MANTIS
RAISA AULIANE SYAFRINA
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
7
Barber P dan Boyce SL. 2006. Estimating diversity of Indo-Pacific coral reef stomatopods through DNA barcoding of stomatopod larvae. Proc R Soc 273: 2053-2061.
Byun SO, Fang Q, Zhou H, dan Hickford JGH. 2009. An effective method for silver-staining DNA in large numbers of polyacrylamide gels. Anal Biochem 385: 174-175.
Carpenter KE dan Niem VH. 1998. The Living Marine Resources of The Western Central Pacific Volume 2: Cephalopods, Crustacean, Holothurians and Shark.
Rome: Food and Agriculture Organization of The United Nation.
[CBOL] The Consortium for the Barcode of Life. 2008. DNA Barcoding: A New Tool for Identifying Biological Specimens and Managing Spesies Diversity. Washington: CBOL.
Choi HJ dan Hong SY. 2001. Larval development of the kishi velvet shrimp, metapenaeopsis dalei (rathbun) (decapoda: penaeidae), reared in the laboratory. Fish Bull 99: 275-291.
Hebert PDN, Cywinska A, Ball SL, dan deWaard JR. 2003. Biological identification through DNA barcodes.
Proc R Soc 270: 313-321.
Hebert PDN, Ratnasingham S, dan deWaard JR. 2003. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species.
Proc R Soc 270: 96–99.
Hebert PDN, Pento EH, Burns JM, Janzen DH, Hallwachs W. 2004. Ten species in one: DNA barcoding reveals cryptic species in the neotropical skipper butterfly
Astraptes fulgerator. PNAS 101(41): 14812-14817.
Innis MA dan Gelfand DH. 1990. PCR Protocols: A Guide to Methods and Applications. San Diego: Academic Press. Ivanova NV, Zemlak TS, Hanner RH, dan
Hebert PDN. 2007. Barcoding: universal primer cocktails for fish DNA barcoding.
Mol Eco 10:1111-1115.
Lahaye et al. 2008. DNA barcoding the floras of biodiversity hotspots. PNAS 105(8): 2923-2928.
Manning RB. 1980. The superfamilies, families and genera of recent stomatopod Crustacea with diagnoses of six new families. Proc Biol Soc Wash 93: 362-372.
Meier R, Shiyang K, Vaidya G, dan Peter.
2006. DNA barcoding and taxonomy in
diptera: a tale of high intraspecific variability and low identification success.
Syst Biol 55(5): 715-728.
Moosa M. 2000. Marine biodiversity of the South China Sea: a checklist of stomatopod Crustacea. The Raffles Billetin of Zoology 8: 405-457.
Perna NT dan Kocher TD. 1996.
Mitochondrial DNA: molecular fossils in the nucleus. Current Biology 6(2): 128-129.
Tamura K, Dudley J, Nei M dan Kumar S. 2007. MEGA4: Molecular evolutionary genetics analysis (MEGA) software version 4.0. Molecular Biology and Evolution 24: 1596-1599.
9
Satu ruas tungkai udang mantis dalam etanol dipotong dan dimasukkan ke dalam tube 1,5 ml. Ruas tungkai yang telah dipotong (otot tungkai) kemudian dicuci dengan air destilata sebanyak
2 kali ulangan menggunakan mesin sentrifuse.
Sampel yang telah dicuci kemudian dihomogenasi menggunkana grinder dan ditambahkan 200 µl buffer STE (NaCl 1M, Tris-HCL 10mM, EDTA 0.1mM, pH 8) ke dalam tube.
Sebanyak 50 mg/ml proteinase K 20 µl ditambahkan ke dalam tabung dan vortex selama beberapa detik.
Inkubasi pada suhu 600C selama 30 menit. Setiap 5 menit sekali tabung dibolak-balik secara perlahan.
Sebanyak 200 µl buffer GB ditambahkan ke dalam tabung dan vortex selama 5 detik.
Inkubasi pada suhu 700C selama 20 menit. Setiap 5 menit sekali tabung dibolak-balik secara perlahan. Pada saat yang bersamaan, inkubasi buffer elusi pada suhu yang sama untuk step elusi DNA.
Sebanyak 200 µl etanol ditambahkan ke dalam tabung dan vortex selama 10 detik. Sampel dipindahkan ke dalam kolom GD pada tube 2 ml.
Sentrifugasi 13.000 rpm selama 2 menit.
Kolom GD dipindahkan ke dalam tabung koleksi yang baru dan supernatan dibuang. Sebanyak 400 µl buffer W1 ditambahkan ke dalam tabung.
Sentrifugasi 13.000 rpm selama 30 detik.
Supernatan dibuang dan kolom GD diletakkan kembali ke dalam tabung.
Sebanyak 600 µl wash buffer (mengandung etanol) ditambahkan ke dalam kolom GD. Sentrifugasi 13.000 rpm selama 30 detik.
Supernatan dibuang dan kolom GD diletakkan kembali ke dalam tabung. Sentrifugasi 13.000 rpm selama 3 menit.
Kolom GD dipindahkan ke dalam tube 1,5 ml yang baru.
Sebanyak 100 µl buffer elusi yang telah diinkubasi ditambahkan ke dalam kolom GD (tepat bagian tengah matriks kolom GD).
Diamkan selama 5 menit.
Sentrifugasi 13.000 rpm selama 30 detik.
10
Gel yang telah dielektroforesis kemudian dikeluarkan dari kaca