i
ISOLASI DAN KLONING GEN
SITOKININ OKSIDASE
(
OsCKX
)
PENGENDALI SIFAT PRODUKTIVITAS DARI PADI
VARIETAS CIHERANG
SISCA RESHA SAPUTRI
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
iii
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa skripsi berjudul Isolasi dan Kloning Gen Sitokinin Oksidase (OsCKX) Pengendali Sifat Produktivitas dari Padi Varietas Ciherang adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Juli 2013
ABSTRAK
SISCA RESHA SAPUTRI. Isolasi dan Kloning Gen Sitokinin Oksidase (OsCKX) Pengendali Sifat Produktivitas dari Padi Varietas Ciherang. Dibimbing oleh SURYANI dan TRI JOKO SANTOSO.
Gen Gn1a/OsCKX (Grain nomor 1a/Sitokinin oksidase) dari tanaman padi (Oryza sativa L.) yang mengkode sitokinin oksidase, telah diidentifikasi sebagai lokus sifat kuantitatif yang berkontribusi terhadap peningkatan jumlah biji padi. Studi ini bertujuan mengisolasi gen OsCKX dari padi varietas Ciherang. Amplifikasi gen OsCKX dilakukan dengan teknik PCR (Polymerase Chain Reaction) menggunakan pasangan primer spesifik dan cetakan cDNA padi cv. Ciherang. Fragmen gen OsCKX produk PCR selanjutnya diklon pada vektor kloning pGEM-T Easy dengan bantuan enzim T4-ligase dan ditransformasikan ke sel kompeten bakteri Escherichia coli Dh5α melalui metode kejut panas, serta ditumbuhkan pada media LB agar yang mengandung antibiotik ampisilin. Hasil studi menunjukkan bahwa amplifikasi gen OsCKX dengan primer spesifik menghasikan amplikon berukuran ± 2000 pb. Fragmen gen tersebut berhasil diklon ke vektor pGEM-T dan ditransformasikan ke sel E. coli Dh5α. Verifikasi klon rekombinan dengan pemotongan menggunakan dua enzim restriksi SmaI dan KpnI diperoleh 2 pita yang berukuran 3015 pb (vektor pGEM-T Easy) dan 2000 pb (fragmen gen OsCKX).
Kata kunci : Enzim restriksi, gen OsCKX, isolasi gen, kloning, padi (Oryza sativa L.), vektorpGEM-T Easy
ABSTRACT
SISCA RESHA SAPUTRI. Isolation and Cloned Cytokinin Oxidase gene (OsCKX) as a Gene Controlling Productivity Character from Rice cv. Ciherang. Supervised by SURYANI and TRI JOKO SANTOSO.
Gn1a/OsCKX gene (grain number 1a/Cytokinin Oxidase) of rice (Oryza sativa L.) that encodes cytokinin oxidase has been identified as a quantitative trait loci that contribute to an increase in the number of grains of rice. This study aimed to isolate an OsCKX gene from rice cv. Ciherang. Amplification of OsCKX gene was done by PCR (Polymerase Chain Reaction) technique using specific primer pairs and cDNA template of rice cv. Ciherang. Subsequently, OsCKX gene fragment was cloned into a cloning vector pGEM-T Easy using T4-ligase enzyme and transformed into competent cell Escherichia coli Dh5α through heat shock method, and grown on LB agar medium containing ampicillin. Result showed that amplification with specific primers of OsCKX gene generated an amplicon fragment with size of ± 2000 bp. The gene fragment was succesfully cloned into pGEM-T vector and transformed into cells of E. coli Dh5α. Verification of recombinant clone by cutting using two restriction enzymes SmaI and KpnI regenerated two bands of 3015 bp (pGEM-T Easy vector) and 2000 bp (OsCKX gene fragment).
v
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Sains
pada
Departemen Biokimia
ISOLASI DAN KLONING GEN
SITOKININ OKSIDASE
(
OsCKX
)
PENGENDALI SIFAT PRODUKTIVITAS DARI PADI
VARIETAS CIHERANG
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
vii
Judul Skripsi : Isolasi dan Kloning Gen Sitokinin Oksidase (OsCKX) Pengendali Sifat Produktivitas dari Padi Varietas Ciherang
Nama : Sisca Resha Saputri
NIM : G84090041
Disetujui oleh
Dr. Suryani, S.P, M.Sc Pembimbing I
Dr. Tri Joko Santoso, S.P, M.Si Pembimbing II
Diketahui oleh
Dr. Ir. I Made Artika, M.App.Sc Ketua Departemen
PRAKATA
Puji syukur penulis panjatkan kehadirat Allah SWT atas segala limpahan rahmat, taufik, serta hidayah-Nya sehingga penulis dapat menyelesaikan dan penyusunan laporan penelitian dengan baik dan lancar. Shalawat dan salam semoga selalu tercurahkan kepada Nabi Muhammad SAW dan keluarga dan sahabatnya.
Penulis mengucapkan terima kasih kepada semua pihak yang telah memberikan bantuan dalam pembuatan laporan ini. Ucapan terima kasih terutama ditujukan kepada ibu Suryani selaku dosen pembimbing utama dan bapak Tri Joko Santoso selaku pembimbing lapangan yang telah membimbing serta memberi saran dan kritik yang membangun. Ucapan terima kasih penulis juga sampaikan kepada para peneliti, teknisi, staf, Dewi Praptiwi, Falin, Mira Sitepu, Dendi, Siska, Hilda, Clara, Vita, Sari, Kiki, Tuhfah, serta sesama mahasiswa yang juga melakukan penelitian di Laboratorium Biologi Molekuler, Balai Besar Penelitian dan Pengembangan Bioteknologi dan Sumber Daya Genetik Pertanian, Jl. Tentara Pelajar No.3A, Cimanggu, Bogor. Penulis juga mengucapkan terima kasih kepada kedua orang tua atas dukungan yang selalu diberikan.
Penulis menyadari bahwa tulisan ini masih jauh dari kesempurnaan, maka saran dan kritik yang konstruktif dari semua pihak sangat diharapkan demi pelaksanaan penelitian. Akhir kata penulis berharap semoga tulisan ini dapat bermanfaat bagi semua pihak, khususnya bagi penulis dan pembaca pada umumnya.
ix
DAFTAR ISI
DAFTAR TABEL x
DAFTAR GAMBAR x
DAFTAR LAMPIRAN x
PENDAHULUAN 1 METODE 2 Bahan 2 Alat 2
Prosedur Analisis Data 2
HASIL DAN PEMBAHASAN 5
Hasil 5 Pembahasan 8
SIMPULAN DAN SARAN 13
Simpulan 13 Saran 13
DAFTAR PUSTAKA 13
DAFTAR TABEL
1 Komposisi larutan ligasi vektor pGEM-T Easy 4
2 Komposisi larutan digest pGEM-T OsCKX dengan enzim restriksi
SmaI dan KpnI 5
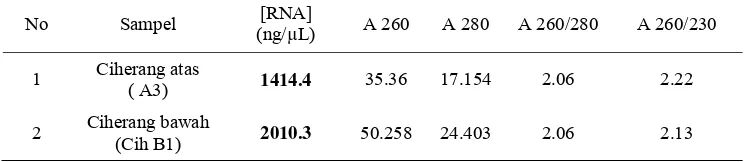
3 Kuantitas dan kemurnian RNA padi 5
DAFTAR GAMBAR
1 Elektroforegram hasil amplifikasi gen OsCKX menggunakan
primer spesifik dengan cetakan cDNA 6
2 Hasil transformasi gen OsCKX ke plasmid pGEM-T Easy melalui
bakteri Escherichia coli 6
3 Elektroforegram hasil amplifikasi pGEM-T 9 CKX dan pGEM-T 10 CKX 7
4 Elektroforegram hasil digest dengan enzim restriksi SmaI dan KpnI 7
5 Tanaman padi varietas Ciherang 8
DAFTAR LAMPIRAN
1 Diagram alir penelitian 15
2 Peta plasmid vektor pGEM-T Easy 15
3 Koloni sel transforman Escherichia coli yang ditumbuhkan di media
LB cair 16
4 Isolasi plasmid metode lisis alkali 16
5 Komposisi larutan sediaan 17
1
PENDAHULUAN
Padi memegang peranan paling penting dalam penyediaan pangan yang mendukung ke arah ketahanan pangan nasional dan pemberdayaan ekonomi petani. Bukan hanya dari segi kuantitas, tetapi kualitas padi yang menyangkut selera pasar, rasa, aroma, dan kandungan nutrisi menjadi hal penting yang perlu diperhatikan dalam pengembangan padi ke depan (Krishnan & Puepke 1983). Hal tersebut dipengaruhi tingkat kebutuhan padi masyarakat yang semakin tinggi seiring bertambahnya penduduk sehingga produktivitas serta kualitas perlu ditingkatkan (Kibria et al. 2008).
Upaya yang dapat dilakukan untuk mengatasi permasalahan produktivitas adalah dengan mengintensifkan teknik peningkatan produktivitas padi melalui teknologi DNA rekombinan dengan cara mengisolasi gen pengendali karakter jumlah biji per malai (gen OsCKX) pada tanaman padi. Berdasarkan penelitian yang dilakukan Li Shuyu et al. (2012) menunjukkan bahwa protein sitokinin oksidase positif mengatur kegiatan dan fungsi meristem apikal tunas (SAM) yang merupakan parameter utama penentu produksi benih. Peningkatan aktivitas meristem menyebabkan peningkatan produksi malai bercabang sebagai akibat peningkatan jumlah gabah. Pertama kali sitokinin ditemukan sebagai hormon tanaman yang mengatur pembelahan sel yang mempengaruhi berbagai aspek pertumbuhan dan perkembangan tanaman, termasuk perkecambahan biji, dominasi apikal, perluasan daun, dan perkembangan alat reproduksi. Gen ini diidentifikasi dapat mengkode protein sitokinin oksidase yang berpengaruh pada pembelahan sel sehingga pemanfaatan gen OsCKX tersebut berguna untuk memperbaiki sifat tanaman padi agar produktivitas semakin meningkat (Ashikari et al. 2005).
Perbaikan sifat tanaman padi seperti jumlah dan kualitas tidak kalah penting dari usaha perbaikan karakter lain, karena pengaruhnya terhadap hasil panen cukup signifikan. Pemanfaatan gen OsCKX ini selain untuk perbaikan sifat, diharapkan pula dapat membantu usaha penyediaan gen-gen penting pada tanaman padi. Molekul DNA yang mengandung gen OsCKX dapat diperbanyak melalui amplifikasi dan ditransformasikan ke dalam sel inang Escherichia coli (Tsen et al. 2002). Penyisipan molekul DNA rekombinan ke dalam sel inang ini menggunakan vektor berupa plasmid seperti pGEM-T Easy. Peristiwa ini akan menghasilkan sel transforman yang mengandung gen OsCKX untuk diaplikasikan pada tanaman padi guna mengetahui tingkat produktivitas padi Ciherang sebagai hasil rekayasa teknologi DNA rekombinan melalui tahap kloning gen.
2
dapat menambah koleksi penyediaan gen-gen penting pada padi. Hasil isolasi gen OsCKX tersebut dapat diaplikasikan pada tanaman padi varietas lain untuk meningkatkan produktivitas.
METODE
Bahan
Bahan yang digunakan dalam penelitian ini adalah tanaman padi varietas Ciherang, akuades, nitrogen cair, mini kit RNeasy Qiagen (buffer RLT[Qiagen], buffer RPE[Qiagen], buffer RW1[Qiagen]), kit Transcriptor First Strand Synthesis [Roche] (dNTP 0.2 mM, oligo dT, ddH2O DEPC (dietil pirokarbonat),
transcriptor RT reaction buffer 5x, MgCl2 1.5 mM, protector RNase inhibitor (40
U/ l), dan transcriptor reverse transcriptase 0.02 U/ L),buffer PCR, primer gen OsCKX (forward dan reverse), enzim Taq DNA Polimerase, agarosa, buffer TAE 1x (asam borat 0.83 M, Tris HCl 1 M pH 8.3, etilendiamina tetra-asetat 10 mM), akuades, marker DNA invitrogen 1 kb, loading dye, gel red (BIOTIUM), DNA plasmid pGEM-T Easy, enzim restriksi (SmaI dan KpnI), buffer restriksi, kultur bakteri Escherichia coli, media LB (Luria bertani), media agar, dan antibiotik ampisilin.
Alat
Alat yang digunakan dalam penelitian ini adalah cawan Petri, autoklaf, mortar, tabung mikrosentrifugasi 1.5 mL dan 2 mL, vortex, Microwave [Sanyo], neraca analitik, labu Erlenmeyer, pipet volumetrik, QIAshredder spin column, tabung RNeasy mini column, sentrifus, Nanodrop 2000/200c Spectrophotometer V1.0 User Manual with package Laptop [Thermo Scientific], mesin PCR DNA Engine Tetrad 2 Peltier Thermal Cycler [Bio-Rad], stopwatch, perangkat alat elektroforesis, komputer, UV Illuminator ChemiDocTM Gel System EQ [Bio-Rad] (Sistem XRS, Image Lab™ Software, dan kamera CCD), laminar, dan alat-alat gelas.
Prosedur Penanaman Padi
3
dalam bak penanaman, padi dipindahkan ke dalam ember dan disimpan di rumah kaca. RNA padi diisolasi pada umur sekitar 2 bulan setelah malai padi keluar.
Isolasi RNA (Qiagen 2010)
Jaringan yang digunakan untuk isolasi RNA padi adalah jaringan malai. Isolasi RNA dilakukan berdasarkan metode Qiagen 2010. Semua peralatan yang akan digunakan terlebih dahulu disterilisasi dengan autoklaf. Isolasi RNA diawali dengan pemanenan sampel malai padi varietas Ciherang. Sebanyak 0.3 gram malai dimasukkan ke dalam mortar, dan ditambahkan nitrogen cair secukupnya. Malai digerus hingga halus dan ditambahkan dengan 450 µL buffer RLT yang mengandung merkaptoetanol 5 µL. Hasil gerusan dimasukan ke dalam tabung mikro 2 mL dan divorteks selama 1 menit hingga homogen lalu diinkubasi pada suhu 56 ºC selama 3 menit. Kemudian suspensi dipindahkan ke dalam tabung QIAshredder spin column. Sampel disentrifus selama 2 menit dengan kecepatan 12000 rpm. Supernatan yang dihasilkan dipindahkan ke dalam tabung mikro 1.5 mL yang baru dan ditambahkan etanol absolut (96%) sebanyak setengah dari volume total. Selanjutnya sebanyak 650 µL supernatan dipindahkan ke dalam tabung RNeasy mini column disentrifus kembali selama 15 detik pada kecepatan 1200 g.
Supernatan yang dihasilkan pada tahap sentrifus sebelumnya dibuang dan ditambahkan dengan larutan RW1 sebanyak 700 µL ke dalam kolom RNeasy. Suspensi disentrifus kembali dengan kecepatan 1200 g selama 15 detik. Supernatan yang dihasilkan dibuang, ditambahkan buffer RPE sebanyak 500 µL dan disentrifus selama 2 menit dengan kecepatan 1200 g. Kolom RNeasy dipindahkan ke dalam tabung mikro 1.5 mL kemudian ditambahkan 50 µL air bebas RNase, didiamkan selama 15 menit, kemudian disentrifus pada kecepatan 1200 g selama 1 menit sehingga diperoleh RNA total tanaman padi.
Uji Kuantitas dan Kemurnian RNA Padi dengan Spektrofotometer (Thermo Fisher Scientific 2009)
Sebanyak 2 µL sampel RNA hasil isolasi diukur konsentrasinya dengan menggunakan spektrofotometer nanodrop.Blanko yang digunakan adalah larutan akuades. Larutan akuades dimasukkan ke dalam lubang optik sebanyak 2 µL dan bersihkan dengan tisu. Selanjutnya sampel RNA sebanyak 2 µL dimasukkan ke dalam lubang optik. Hasil pengukuran berupa konsentrasi ng/µL dan kemurnian RNA dapat dilihat langsung berdasarkan nilai rasio absorbansi pada panjang gelombang 230, 260, dan 280 nm.
Sintesis cDNA dengan Kit Transcriptor first strand synthesis (RT-PCR)
Sintesis cDNA dilakukan berdasarkan protokol kit Roche untuk mendapatkan cDNA dari RNA total hasil isolasi yang akan digunakan sebagai cetakan. Sampel RNA yang digunakan, yaitu RNA yang berasal dari malai padi Ciherang bagian atas (Cih A3) dan bagian bawah (Cih B1). Campuran reaksi untuk masing-masing sampel terdiri atas 1 L dNTP mix 0.2 mM, 2 L RNA total (2 g/mL), 1 L oligo dT, dan ddH2O DEPC sehingga total larutan menjadi 13 L.
4
diinkubasi dalam es selama 10 menit. Selanjutnya ditambahkan 4 L transcriptor RT reaction buffer 5X, 4 L MgCl2, 2 L dNTP, dan 0.5 L protector RNAse
inhibitor (40 U/ L), kemudian campuran diinkubasi kembali pada suhu 42°C selama 2 menit. Setelah itu ditambahkan 1 L transcriptor reverse transcriptase (20 U/ L), diinkubasi kembali pada suhu 55°C selama 30 menit. Inaktivasi transcriptor reverse transcriptase pada suhu 85°C selama 5 menit dan disimpan pada suhu -20°C.
DNA komplementer yang diperoleh digunakan sebagai cetakan untuk reaksi PCR. Tabung mikro diisi dengan campuran reaksi PCR (total volum reaksi 20 µL ) yang terdiri atas 2 µL buffer PCR, 0.6 µL MgCl2 50 mM, 0.4 µL dNTP
mix 10 Mm, 2 µL campuran primer gen OsCKX (Forward 5’- GCC CCG GGA CAC ACA CAC TGA CAC ACA -3’, sedangkan Reverse 5’- GCG GTA CCT TCA TGC GAG TGG TGA CGT G -3’), 0.16 U/µL Taq polymerase, 1 µL DNA 50 ng/µL, dan 13.24 µL ddH2O. Reaksi amplifikasi dilakukan dengan tahap
denaturasi pada suhu 94ºC selama 30 detik , penempelan primer pada suhu 55ºC selama 1 menit, dan pemanjangan pada suhu 72ºC selama 2 menit sebanyak 35 siklus dan diikuti proses pemanjangan akhir pada suhu 72ºC selama 7 menit. Selanjutnya produk PCR dipisahkan dengan elektroforesis gel agarosa 1% yang telah ditambahkan gel red dan divisualisasi menggunakan UV Illuminator ChemiDoc EQ (Bio-rad). Fragmen gen OsCKX berukuran 2000 pb yang muncul pada gel agarosa selanjutnya dielusi dan diekstrak untuk memisahkan DNA fragmen dari matriks gel dengan metode ekstraksi gel menggunakan kit komersial Gene Clean.
Ligasi Gen OsCKX pada Vektor pGEM-T Easy (Promega 2010)
Gen OsCKX (2000 pb) yang didapatkan dari pemurnian hasil PCR selanjutnya diligasikan pada vektor pGEM-T Easy dan diinkubasi pada suhu 4ºC selama semalam. Larutan mix dalam ligasi memiliki komposisi seperti pada Tabel 1.
Tabel 1 Komposisi larutan mix ligasi vektor pGEM-T Easy
Larutan Volume
Transformasi Plasmid Rekombinan OsCKX Padi ke dalam Sel Escherichia coli (Sambrook & Rusell 2001)
5
yang tumbuh kemudian diperbanyak dalam 5 mL LB cair yang mengandung 5 µL ampisilin (100 mg/mL) dan diinkubasi pada shaker incubator 37ºC selama 16 jam. Transforman yang tumbuh diisolasi DNA plasmid dengan metode lisis alkali (Lampiran 4).
Restriksi Plasmid Rekombinan OsCKX E. coli (Takara 2012)
Plasmid hasil isolasi plasmid kemudian dipotong dengan enzim restriksi SmaI dan KpnI. Campuran reaksi enzimatis disajikan pada Tabel 2. Campuran tersebut diinkubasi pada suhu 37ºC semalam. Selanjutnya hasil restriksi diverifikasi dengan elektroforesis pada gel agarosa 1%.
Tabel 2 Komposisi larutan digest pGEM-T OsCKX dengan SmaI dan KpnI Kuantitas dan Kemurnian RNA Padi
RNA padi Ciherang dari jaringan malai yang telah diisolasi diuji secara kuantitatif dan kualitatif melalui pengukuran absorban pada panjang gelombang 230, 260 dan 280 nm. Sampel RNA yang diukur merupakan hasil isolasi RNA malai padi bagian atas dan bagian bawah seperti yang ditunjukkan pada Tabel 3. Konsentrasi RNA dihitung dengan perbandingan bahwa nilai 1 pada serapan 260 nm merupakan serapan RNA dengan konsentrasi 40 ng/µL. Hasil konsentrasi yang diperoleh pada masing-masing sampel cukup tinggi, yaitu sebesar 1414.4 ng/µL untuk sampel Ciherang atas (Cih A) dan 2010.3 ng/µL untuk sampel Ciherang bawah (Cih B1). Adapun nilai kemurnian RNA hasil isolasi diukur pada rasio 260/280 dan 260/230.
Tabel 3 Kuantitas dan kemurnian RNA padi
6
Amplikon Gen OsCKX Hasil Sintesis cDNA
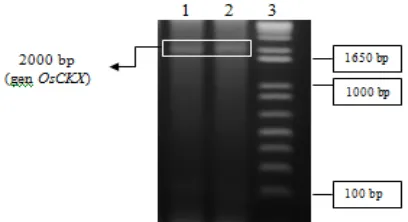
Sintesis cDNA dilakukan untuk mendapatkan cDNA dari RNA total hasil isolasi yang akan digunakan sebagai cetakan untuk isolasi gen OsCKX menggunakan PCR. Sampel yang digunakan untuk sintesis cDNA adalah RNA dari sampel Cih A3 dan Cih B1. Produk sintesis berupa cDNA tersebut selanjutnya diamplifikasi dengan menggunakan pasangan primer spesifik untuk gen OsCKX. Pasangan primer spesifik akan menghasilkan amplikon DNA yang berukuran sekitar 2000 pb. Elektroforegram hasil amplifikasi cDNA dapat dilihat pada Gambar 1. Gen OsCKX berhasil diamplifikasi dari cDNA sampel Ciherang atas (Cih A3) yang diindikasikan dengan dihasilkannya fragmen berukuran sekitar 2000 pb yang merupakan ukuran dari gen tersebut setelah dibandingkan dengan marker 1 kb plus (kisaran 100 pb hingga 12000 pb).
Gambar 1 Elektroforegram amplikon gen OsCKX menggunakan primer spesifik dengan cetakan cDNA. (1) Marker 1 kb plus ladder, (2) Sampel Cih B1, dan (3) Sampel Cih A3
Transforman Plasmid Rekombinan OsCKX
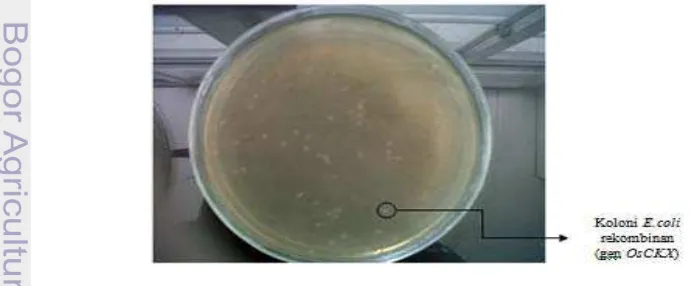
Fragmen gen OsCKX hasil amplifikasi dan elusi selanjutnya diligasikan dengan vektor kloning pGEM-T Easy dan selanjutnya ditransformasikan ke sel bakteri E. coli Dh5α. Hasil menunjukkan bahwa transformasi ke E. coli telah menghasilkan koloni-koloni tunggal bakteri. Koloni hasil transformasi gen OsCKX pada sel E.coli yang tumbuh pada media LB agar yang mengandung ampisilin dapat dilihat pada Gambar 2.
7
Karakterisasi Plasmid Rekombinan OsCKX
Verifikasi gen OsCKX pada plasmid rekombinan dapat dianalisis melalui ampifikasi PCR. Sebanyak 10 koloni tunggal bakteri diisolasi DNA plasmidnya dan diberi kode pGEM-T CKX 1 sampai pGEM-T CKX 10. Hasil isolasi menunjukkan bahwa DNA plasmid hanya diperoleh dari koloni 9 (pGEM-T CKX 9) dan koloni 10 (pGEM-T CKX 10). Untuk menentukan apakah kedua DNA plasmid tersebut merupakan plasmid rekombinan yang membawa gen OsCKX maka dilakukan verifikasi dengan amplifikasi PCR. Hasil verifikasi menunjukkan bahwa kedua plasmid tersebut membawa gen OsCKX dan menghasilkan pita DNA berukuran 2000 pb setelah amplifikasi. Elektroforegram hasil verifikasi DNA plasmid rekombinan dengan PCR dapat dilihat pada Gambar 3. Pita tersebut menunjukan fragmen gen OsCKX telah berhasil disisipkan ke dalam plasmid pGEM-T Easy.
Gambar 3 Elektroforegram amplikon gen OsCKX dari plasmid pGEM-T 9 CKX dan pGEM-T 10 CKX. (1) pGEM-T 9 CKX, (2) pGEM-T 10CKX, dan (3) Marker 1 kb
Selain itu, verifikasi gen OsCKX pada plasmid rekombinan dapat dilakukan dengan pemotongan DNA plasmid rekombinan pGEM-T CKX 9 dan pGEMT-T CKX 10 menggunakan enzim restriksi SmaI dan KpnI. Hasil restriksi dari plasmid pGEM-T CKX 9 dan pGEMT-T CKX menghasilkan 2 fragmen, yaitu fragmen pertama merupakan plasmid pGEM-T Easy sebagai vektor (3015 pb) dan fragmen kedua merupakan fragmen gen OsCKX (2000 pb) (Gambar 4).
8
Pembahasan
Padi Ciherang yang digunakan sebagai bahan penelitian merupakan padi unggul yang telah dikembangkan oleh Balai Besar Tanaman Padi, berasal dari persilangan IR 18349-53-1-3-1-3/IR19661-131-3-1//IR19661-131-3-1-///IR64//// IR64 (Hermanto 2006). Tanaman padi varietas Ciherang memiliki karakteristik umur tanam yang cukup singkat, yaitu 116 hingga 125 hari, menghasilkan anakan produktif 14 hingga 17 batang, warna gabah kuning bersih, tekstur nasi pulen, potensi hasil 8.5 ton/ha dilihat dari jumlah malai padi tersebut seperti pada Gambar 5. Padi Ciherang termasuk dalam sub-spesies Sinica yang tahan terhadap penyakit bakteri hawar daun strain III dan IV (Balitbang-Pertanian 2000). Peningkatan produktivitas padi dapat dilakukan salah satunya diawali dengan pencarian gen yang berperan terhadap tingginya jumlah malai padi. Jumlah malai per tanaman merupakan komponen hasil yang secara langsung mempengaruhi hasil padi. Identifikasi gen yang mengendalikan jumlah malai akan berperan penting dalam produktivitas tanaman padi. Salah satu gen target untuk tujuan tersebut adalah gen OsCKX. Gen ini berperan dalam meningkatkan jumlah bulir pada padi. Gen OsCKX ini diidentifikasi dapat mengkode protein sitokinin oksidase/dehidrogenase yang berpengaruh pada pembelahan sel (Ashikari et al. 2005).
Padi (Oryza sativa L.) memiliki gen Gn1a/OsCKX (Grain nomor 1a/Sitokinin oksidase) yang mengkode oksidase sitokinin. Gen tersebut telah diidentifikasi sebagai lokus sifat kuantitatif utama yang berkontribusi terhadap peningkatan jumlah biji pada tiap pemuliaan padi yang dilakukan (Li Shuyu et al. 2012). Sitokinin fitohormon (CK) positif mengatur aktivitas dan fungsi dari meristem apikal tunas (SAM), yang merupakan parameter utama yang menentukan produksi benih. Ekspresi gen OsCKX mengatur akumulasi CK di SAM sehingga dapat mengontrol jumlah organ reproduksi. Peningkatan aktivitas gen OsCKX ini menyebabkan aktivitas meristem meningkat yang ditandai dengan peningkatan malai bercabang sebagai akibat peningkatan jumlah gabah. Tahap pertama dari isolasi gen OsCKX tersebut adalah melakukan isolasi RNA dari malai padi Ciherang .
(a) (b)
9
RNA diperlukan untuk digunakan sebagai materi dalam mengisolasi gen spesifik pada jaringan tertentu. Pada penelitian ini dilakukan isolasi RNA total dari jaringan malai padi dengan harapan akan didapatkan gen OsCKX penyandi sitokinin oksidase yang berperan dalam meningkatkan jumlah malai padi. Gen OsCKX banyak diekspresikan pada jaringan malai padi. Isolasi RNA dilakukan pada bagian malai tanaman padi yang berumur 2 bulan. Secara umum isolasi RNA lebih sulit dibandingkan dengan DNA karena RNA sangat mudah terdegradasi oleh RNase (ribonuklease) sehingga membutuhkan penanganan khusus dalam mengisolasinya. Metode yang digunakan dalam isolasi RNA pada penelitian ini mengacu pada protokol kit Qiagen.
Prinsip dasar metode isolasi RNA tersebut adalah pemecahan sel (lisis sel) dibantu dengan mekanik melalui penggerusan, dilanjutkan dengan penjerapan menggunakan kolom yang menyebabkan asam nukleat berupa DNA dan RNA akan terjerap dalam kolom, dan adanya pencucian kolom yang mengakibatkan hanya RNA saja yang kita peroleh. Penghancuran dinding sel bertujuan untuk membebaskan sitoplasma dan DNA dalam sel (Brown 1991). Penggunaan nitrogen cair merupakan cara efektif untuk membekukan jaringan sehingga jaringan mudah untuk dihancurkan dan kondisi suhu yang dingin dapat menjaga RNase berada dalam keadaan tidak aktif . Penggunaan merkaptoetanol dan senyawa pengekstrak seperti fenol : klorofom: isoamilalkohol dapat menyebabkan denaturasi senyawa nukleoprotein, dan selanjutnya membuang kontaminan DNA serta protein (Promega 2009).
Kuantitas dan Kemurnian RNA Padi
Adanya ikatan rangkap terkonjugasi pada basa heterosiklik purin dan pirimidin menyebabkan nukleosida, nukleotida, dan polinukleotida dapat menyerap sinar ultra violet (UV) (Murray et al. 2003). Konsentrasi dan kemurnian RNA ditentukan melalui pengukuran absorbansi sampel RNA pada panjang gelombang 260 nm, 280 nm, dan 230 nm. Konsentrasi suatu RNA ditentukan dengan nilai absorban yang diperoleh pada panjang gelombang ( ) 260 nm. Nilai tersebut kemudian dimasukan ke dalam rumus perhitungan yaitu, A260 × faktor konversi × faktor pengenceran. Faktor konversi yang digunakan merupakan nilai 1 unit absorbansi pada 260 nm yang sebanding dengan 40 g/ml RNA (Wilson & Walker 2000). Konsentrasi RNA padi yang diperoleh dari hasil isolasi cukup tinggi, yaitu sebesar 1414.4 ng/µL untuk sampel Ciherang atas (Cih A) dan 2010.3 ng/µL untuk sampel Ciherang bawah (Cih B1). Perbedaan nilai konsentrasi tersebut dapat dipengaruhi oleh beberapa faktor yaitu, perbedaan jenis sampel, hasil gerusan sampel yang kurang halus, dan pengocokan yang kurang kuat saat melakukan ekstraksi sehingga seluruh isi sel tidak dapat terlisis dengan sempurna (Couch & Fritz 1990).
10
sampel Cih A3 dan Cih B1 pada A260/230, yaitu 2.22 dan 2.13. Rasio A260/230 digunakan sebagai indikator tingkat kontaminasi oleh polisakarida dan polifenol (Loulakakis et al. 1996 dan Schultz et al. 1994).
Amplikon Gen OsCKX Padi Varietas Ciherang
Gen OsCKX diisolasi menggunakan teknik RT-PCR melalui sintesis cDNA dari RNA total yang digunakan sebagai cetakan (Watson et al 1987). Hal tersebut dilakukan karena untuk mengisolasi gen-gen yang diekspresikan secara spesifik pada jaringan tertentu dari kromosom eukariot diperlukan cDNA yang merupakan DNA yang komplemen dengan mRNA (Dale 1994), sedangkan mRNA adalah transkrip dari gen-gen yang terekspresi yang mebawa pesan berupa informasi genetik dari DNA ke dalam bentuk protein (Darnell et al 1990). cDNA digunakan sebagai cetakan (template) untuk mengampifikasi gen OsCKX dengan teknik RT-PCR yang banyak digunakan untuk menganalisis ekspresi gen tunggal (Campbell et al. 2008). Untuk mendapatkan gen tersebut digunakan primer spesifik gen OsCKX yang dapat menempel dan mengamplifikasi gen tersebut secara spesifik sehingga cDNA dapat diketahui mengandung gen OsCKX. Keuntungan penggunaan cDNA untuk isolasi gen adalah bahwa cDNA untai ganda merupakan manifestasi dari gen yang diharapkan, tetapi gen tersebut tidak mengandung intron.
Amplifikasi gen OsCKX menggunakan sepasang primer OsCKX forward dan OsCKX reverse yang dirancang spesifik mengamplifikasi gen CKX berukuran 2000 pb. Primer yang dirancang harus dapat menghasilkan produk yang diinginkan dengan konsentrasi tinggi, dan mengurangi amplifikasi sekuens yang tidak diinginkan (Sambrook & Russell 2001). Hasil amplifikasi gen OsCKX dengan optimasi suhu annealing pada mesin PCR, dianalisis dengan elektroforesis gel agarosa 1%. Berdasarkan hasil verifikasi pada elektroforesis gel agarosa terlihat adanya gen OsCKX yang diperoleh dari ukuran fragmen yang terbentuk 2000 pb (Gambar 1). Namun, terdapat fragmen lain yang terbentuk dari pita yang dihasilkan pada gel agarosa. Hal tersebut dapat disebabkan karena penggunaan suhu annealing yang belum optimal dan primer yang digunakan kurang spesifik. Suhu annealing adalah suhu dimana primer akan menempel pada template DNA. Pencarian kondisi optimal dari suhu annealing sangat penting, karena berkaitan dengan spesifitas dan sensitifitas produk PCR. Apabila primer yang digunakan kurang spesifik dan nilai suhu annealing yang digunakan terlalu rendah maka primer tidak hanya menempel pada gen target tetapi juga menempel pada gen lain sehingga akan terbentuk dua pita atau lebih. Sedangkan jika nilai suhu annealing yang digunakan saat PCR lebih besar maka akan dihasilkan produk yang sedikit atau pita yang muncul akan tipis karena kemampuan primer untuk menempel pada target menjadi lebih rendah (Sasmito Bayu 2010).
11
diamplifikasi, sementara sekuens fragmen yang tidak memiliki homologi dengan sekuens primer tidak akan teramplifikasi. Hal tersebut dapat memaksimalkan keberhasilan amplifikasi untuk memperoleh sekuens target sehingga kemungkinan terjadinya kesalahan penempelan primer (mispriming) tidak dapat terjadi, yaitu penempelan primer di luar sekuens target. Urutan basa dari primer yang komplementer setidaknya memiliki homologi cukup tinggi dengan urutan basa kedua daerah ujung fragmen yang akan diamplifikasi untuk mempermudah penempelan pada gen target. Primer yang dirancang tidak boleh mengamplifikasi gen atau daerah lain yang homolog dengan gen target yang dapat menyebabkan produk PCR tidak spesifik dan menghasilkan lebih dari satu produk (Retnoningrum DS 1997).
Selain itu, struktur sekunder pada primer dapat menyebabkan masalah selama proses PCR, sehingga perlu dihindari sekuens yang saling berkomplemen di dalam primer (hairpin) atau sekuen yang saling berkomplemen antara primer forward dan reverse (primer-dimer) itu sendiri (Newton & Graham 1997; McDowell 1999; Birt 2000; Rapley 2000). Penentuan urutan basa pasangan primer perlu dianalisis menggunakan program komputer untuk mengetahui kemungkinan terjadinya primer-dimer akibat homologi sendiri (self-homology) atau homologi silang (cross-homology) sehingga tidak terbentuk produk-produk non spesifik yang muncul pada saat visualisasi produk PCR dengan elektroforesis gel agarosa. Oleh karena itu, perancangan primer yang spesifik sangat dibutuhkan untuk memperoleh gen target yang diinginkan. Fragmen yang terbentuk pada gel agarosa dengan ukuran 2000 pb yang menunjukkan gen OsCKX selanjutnya dipotong dan dimurnikan.ang spesifik.
Pemurnian dilakukan dengan pemotongan fragmen pita 2000 pb pada gel agarosa dengan menggunakan kit Gene Clean. Hal tersebut bertujuan memurnikan fragmen DNA yang diinginkan (gen OsCKX) dan menghilangkan pengotor-pengotor lain berupa nukleotida, sisa-sisa garam, dan enzim yang dapat mengganggu proses selanjutnya (Dobhal et al. 2010). Fragmen DNA yang sudah murni diligasikan dengan plasmid pGEM-T Easy. Plasmid pGEM-T Easy digunakan sebagai vektor kloning yang merupakan agen pembawa fragmen DNA masuk ke dalam sel makhluk hidup yang berfungsi untuk memperbanyak fragmen DNA (Primrose dan Twiman 2006). Gen OsCKX yang telah dimurnikan tersebut kemudian diligasikan ke vektor pGEM-T Easy agar dapat ditransformasikan ke dalam sel inang.
Transforman yang Membawa Gen OsCKX
12
Fragmen yang sudah diligasikan kemudian ditransformasikan ke dalam sel bakteri Escherichia coli menggunakan metode heatshock, yaitu dengan pemberian kejut panas pada suhu 42°C selama 90 detik. Prinsip dari proses tersebut adalah terjadinya lonjakan suhu dari 0°C ke 42°C terhadap sel kompeten yang telah diberi perlakuan CaCl2. Garam CaCl2 akan mempengaruhi porositas membran sel
sehingga pada saat terjadi lonjakan suhu membran menjadi tidak selektif terhadap molekul asing dan produk ligasi dapat masuk ke dalam sel. Sel bakteri yang digunakan dalam proses transformasi umumnya adalah sel E.coli ini merupakan sel inang yang paling umum digunakan dalam teknologi DNA rekombinan. Setiap sel memiliki efisiensi yang berbeda dalam menyerap molekul DNA. Kemampuan sel dalam menyerap DNA dapat ditingkatkan dengan perlakuan khusus, sehingga sel tersebut menjadi kompeten.
Sel inang yang telah mengandung DNA rekombinan dibiakkan dalam medium padat, sehingga membentuk koloni seperti pada Gambar 2. Koloni yang terbentuk merupakan sekumpulan sel yang identik karena hasil pembiakan sebuah sel. Media seleksi yang digunakan pada penelitian ini adalah media LB agar yang mengandung antibiotik ampisilin. Hasil yang diperoleh dari pembiakan sel E.coli rekombinan, yaitu terbentuknya koloni-koloni berwarna putih pada media agar. Terbentuknya koloni tersebut disebabkan adanya marka seleksi pada vektor pGEM-T Easy dan koloni tunggal yang terbentuk akan dianalisis selanjutnya untuk menentukan keberadaan gen OsCKX yang diinginkan. Marka seleksi biasanya berupa gen yang membawa sifat resisten antibiotik tertentu dan vektor pGEM-T Easy memiliki marka seleksi berupa gen resisten ampisilin dapat dilihat pada peta plasmid pGEM-T Easy pada Lampiran 2. Setelah diperoleh transforman yang membawa gen OsCKX pada koloni yang tumbuh dalam media agar, dilakukan konfirmasi pada plasmid pembawa gen tersebut melalui amplifikasi PCR dan pemotongan enzim restriksi.
Plasmid rekombinan pembawa gen OsCKX berhasil dikonstruksi dengan menyisipkan gen tersebut ke dalam plasmid pGEM-T Easy. Teknik ini dilakukan dengan mengisolasi plasmid pGEM-T yang membawa gen OsCKX dari kultur bakteri Escherichia coli yang ditumbuhkan pada media LB cair sebelumnya. Isolasi DNA plasmid dilakukan dengan menggunakan metode lisis alkali. Adapun prinsip dari metode ini adalah merusak dinding sel dan membran sel dari E.coli (Sambrook dan Russel 2001). Penggunaan etilen diamin tetra asetat (EDTA) bertujuan untuk merusak dinding sel dengan menghilangkan ion magnesium pada selubung sel sehingga dinding sel bakteri pecah (Zhiwen et al. 2006). Sodium Deodosil Sulfat (SDS) berfungsi untuk menghilangkan molekul lipid, sehingga akan menyebabkan kerusakan pada membran sel (Davis et al. 2004).
13
bertujuan untuk memastikan arah orientasi gen OsCKX ketika berligasi dengan vektor pGEM-T Easy. Hasil elektroforegram (Gambar 3) menunjukan bahwa plasmid pGEM-T Easy yang membawa gen OsCKX berhasil diisolasi dari kultur E.coli, dibuktikan dengan adanya 2 pita DNA yang dihasilkan dari pemotongan dengan SmaI dan KpnI dengan ukuran pita 1 vektor pGEM-T Easy (3015 pb) dan pita 2 fragmen gen OsCKX (2000 pb) .Verifikasi jumlah fragmen yang diperoleh dari hasil pemotongan dengan kedua enzim restriksi tersebut dapat dilihat pada peta restriksi pGEM-T Easy Lampiran 2.
SIMPULAN DAN SARAN
Simpulan
Gen OsCKX dari jaringan malai padi telah berhasil diisolasi. Amplifikasi gen OsCKX hasil isolasi dan verifikasi gen pada plasmid rekombinan berukuran 2000 pb dan pemotongan plasmid rekombinan dengan enzim restriksi SmaI dan KpnI menghasilkan 2 fragmen DNA yang berukuran 3015 pb (vektor pGEM-T Easy) dan 2000 pb (gen OsCKX).
Saran
Perlu dilakukan penentuan spesifisitas primer dan penentuan tingkat homologi dari urutan basa primer yang digunakan menggunakan program komputer agar dapat diperoleh sekuen yang spesifik dari gen target OsCKX yang diinginkan.
DAFTAR PUSTAKA
Ashikari M, Sakakibara H, Lin S, Yamamoto T, Takashi T, Nishimura A, Angeles ER, Qian Q, Kitano H, dan Matsuoka M. 2005. Cytokinin Oxidase Regulates Rice Grain Production. J Science AAAS 309: 741.
Balitbang-Pertanian. 2000. Varietas ciherang. [terubung berkala]. http://www.litbang.deptan.go.id/ varietas/one/130. (Diakses pada 10 Februari 2013)
Birt TP, Baker AJ, Baker AJ. 2000. Molecular Methods in Ecology. London: Blackwell Science Ltd.
Brown TA. 1991. Pengantar Kloning Gen. Yogyakarta: Esentica Medica.Faatih M. 2009. Isolasi dan Digesti DNA Kromosom. Jurnal Penelitian Sains & Teknologi 10: 61 – 67.
Campbell NA, Reece JB, Urry LA, Chain ML, Wasserman SA, Minorsky PV, dan Jackson RB. 2008. Biology, Eight Edition. Pearson Benjamin Cummings. San Francisco.
14
Couch JA, Fritz PJ. 1990. Isolation of DNA from plants high in polyphenolics. Plant Mol Biol Rep 8: 8-12 Dale JW. 1994. Molecular Genetic of Bacteria England: John Wiley and Sons.
Darnell et al. 1990. Molecular CellBiology. New York:Scientific American Books. Davis L, Kuehl M, dan Battey J. 1994. Basic methods: Molecular biology 2nd ed.
Appleton dan Lange, Norwola.
Dobhal S, P Dinesh, K Anil, A Sanjeev. 2010. Studies on plant regeneration and transformation efficiency of Agrobacterium mediated transformationusing neomycin phospotransferase II (nptII) dan glucuronidase (GUS) as a reporter gene. Afr J Biotech 9(41):6853-6859.
Hermanto. 2006. Padi Ciherang makin populer. Warta Penelitian dan Pengembangan Pertanian 28:14-15. [terhubung berkala]. http://crifc@indo.net.id/journal/warta [25 Juni 2013].
Kibria et al. 2008. Screening of aromatic rice lines by phenotypic and molecular markers. J Bot. 37:141-147.
Krishnan HB, Puepke SG. 1983. Nucleotide sequence of an abundant rice seed globulin. J Biophys. 193:460-466.
Lee MD, Fairchild A. 2006. Sample Preparation for PCR. In: Maurer, J (ed.). Food Microbiology and Food Safety: PCR Methods in Foods. USA: Springer.
Li Shuyu et al. 2012. Rice zinc finger protein DST enhances grain production through controlling Gn1a/OsCKX2 expression. Journal of lant Biology, PNAS Early Edition 5:1-6.
[LITBANG] Balai Penelitian Bahan Pangan. 2006. Mengenal padi VUTB Fatmawati. http://jakarta.litbang.deptan.go.id/klinikagribisnis. J Litbang 12:1-6.
Loulakakis KA. 1996. Isolation of functional RNA from grapevine tissues poor in nucleic acid content. J EnolVitic 47:181-185.
McDowell D, Saunders, Parkes. 1999. Analytical Molecular Biology Quality and Validation. Cambridge: Royal Society of Chemistry.
Murray RK, Graner DK, Mayes PA, Rodwell VW. 2003. Biokimia Harper. Andry H, penerjemah; Anna PB, Tiara MN, editor. Jakarta: EGC. Terjemahan dari: Harper’s Biochemistry.
Nanodrop. 2000. NanoDrop 2000 Packaged with Laptop Computer [Themo Scientific]. US.
Newton CM, Graham A. 1997. PCR Ed ke-2. New York: Springer-verlag.
Pestana EA, Belak S, Diallo A, Crowther JR, Viljoen GJ. 2010. Early, Rapid, and ensitive Veterinary Molecular Diagnostics Real-Time PCR Application. Dordrecht: Springer.
Primrose SB, Twyman RM. 2006. Induksi kalus dan optimasi regenerasi empat varietas padi melalui kultur In Vitro. J Agro Biogen 2 (2): 74-80.
Promega. 2009. Technical Manual. Woods Hollow Road: Promega.
Promega. 2010. pGEM-T and pGEM-T Easy Vector Systems. Promega Corporation, Madison.
Rapley R, Walker JM & Rapley R. 2002. Molecular Biology and Biotechnology. Cambridge: Royal Society of Chemistry.
15
Retnoningrum DS. 1997. Penerapan Polymerase Chain Reaction (PCR) untuk diagnosis penyakit infeksi. JurusanFarmasi FMIPA. Bandung: ITB. Sambrook J, Russel DW. 2001. Molecular Cloning: A Laboratory manual. 3rd
Edition. New York: Cold Spring Harbor Laboratory Pr, Cold Spring Harbor.
Sasmito Bayu. 2010. Ekspresi Gen Aminocyclopropane Carboxylic Synthase pada Klon Tanaman Karet Akibat Pemberian Etefon [skripsi]. Bogor: Institut Pertanian Bogor.
Suharsono dan Widyastuti, Utut. 2006. Pelatihan Singkat Teknik Dasar Pengklonan Gen. Pusat Penelitian Sumberdaya Hayati dan Bioteknologi Lembaga Penelitian dan Pemberdayaan Masyarakat IPB dengan DIKTI DIKNAS, Bogor.
Takara. 2012. Buffer Activity Chart: relative restriction enzyme activity in universal and basal buffers. [terhubung berkala] http://www.clontech.com/takara/US/Support/Applications/Restriction_En zymes/ Bufer_Activity_Chart [03 Juli 2013]
Thermo Fisher Scientific. 2009. Nanodrop 2000/200c Spectrophotometer V1.0 User Manual. Wilmington: Thermo Fisher Scientific.
Tsen et al. 2002. Natural plasmid transformation in Escherichia coli. Journal of Biomedical Science 9:246-252.
Watson et al. 1987. Molecular Biology of the Gene. USA: The Benjamin Publishing Company.
Wilson K, Walker J. 2000. Principles and Techniques of Practical Biochemistry. Ed ke-4. United Kingdom: Cambridge University.
16
Isolasi plasmid rekombinan dengan metode lisis alkali Ligasi ke pGEM-T Easy dan transformasi ke sel E.coli
Sintesis cDNA Isolasi RNA malai padi
Amplifikasi plasmid rekombinan
Pemotongan dengan enzim restriksi SmaI dan KpnI
LAMPIRAN
Lampiran 1 Diagram alir penelitian
Lampiran 2 Peta plasmid pGEM-T Easy
16
15 Lampiran 3 Koloni sel transforman Escherichia coli yang ditumbuhkan di
media LB cair
Hasil perbanyakan bakteri transforman E.coli
Lampiran 4 Isolasi plasmid dengan metode lisis alkali (Sambrook & Rusell 2001)
17 16
Lampiran 5 Komposisi larutan sediaan Komposisi larutan isolasi plasmid
Stok Larutan 1 Pembuatan 100 mL Konsentrasi akhir
Glukosa 0.9 gram 50 mM
1 M Tris HCl pH 8.0 2.5 mL 25 mM
0.5 M EDTA pH 8.0 2 mL 10 mM
Larutan diautoklaf selama 15 menit dan disimpan pada suhu 4 °C
Stok Larutan 2 Pembuatan 1 mL Konsentrasi akhir
10 N NaOH 20 µl 0.2 N
10% SDS 100 µl 1%
NF water 880 µl
Stok Larutan 3 Pembuatan 100 mL Konsentrasi akhir
5 M Potasium asetat 60 mL 50 mM
Asan asetat glacial 11.5 mL
NF water 28.5 mL
Komposisi media Luria Bertani (LB)
LB cair Pembuatan 100 mL
Trypton 1.0 gram
NaCl 1.0 gram
Ekstrak yeast 0.5 gram
Akuades ditera hingga 100 mL (pH 7.0)
LB padat
Akuades ditera hingga 100 mL (pH 7.0)
Komposisi bufer TAE 50x
Bahan Pembuatan 1000 mL
Tris242 gram
ditera dengan akuades hingga 1000 mL Asam asetat glacial 57.1 mL
0.5 EDTA (pH 8.0) 100 mL
Pembuatan bufer TAE 1x (dari stok bufer TAE 50x) :
V1 x M1 = V2 x M2
V1 x 50 = 1 x 2000 mL
V1 = 40 mL (stok bufer TAE 50x ditera dengan akuades hingga 2000 mL)
16
15 Lampiran 6 Ekstaksi DNA dengan menggunakan kit Gene Clean
Gel agarosa yang mengandung DNA ditimbang, dan NF water sebanyak 20 µL dipanaskan pada
water bath padasuhu 56-60°C
100 mg gel agarosa ditambahkan 300 µL buffer agarose solubiluation
Gel diresuspensi dengan ditambahkan 20 µL glass milk hingga homogen,
Campuran diinkubasi pada water bath suhu 56-60°C selama 10 menit dan kocok setiap 3 menit
Campuran disentrifugasi 1800 g selam 30 detik
Supernatan dibuang dan pelet ditambahkan larutan nucleic acid binding
buffer sebanyak 500 µL, disentrifus 1800 g selama 30 detik
Supernatan dibuang dan pelet dicuci dengan larutan washing
buffer sebanyak 500 µL, disentrifus 1800 g selama 30 detik
Supernatan dibuang dan pelet dicuci kembali dengan larutan washing
buffer sebanyak 500 µL, disentrifus 1800 g selama 30 detik
Pelet dikeringkan pada suhu ruang sekitar 3 jam
Setelah kering tambahkan NF water yang sudah dipanaskan sebelumya, divortex dan diinkubasi pada suhu 56-60°C selama 10 menit dan kocok setiap 3 menit
Larutan disentrifus 1800 g selama 30 detik, dan supernatant diambil dan disimpan pada suhu -20°C
16
15
RIWAYAT HIDUP
Penulis dilahirkan di Jakarta pada tanggal 25 September 1991 dari ayah Hasan M dan ibu Ida Kurniali. Penulis adalah putri pertama dari empat bersaudara. Tahun 2009 penulis lulus dari SMA Negeri 1 Cianjur dan pada tahun yang sama penulis lulus seleksi masuk Institut Pertanian Bogor (IPB) melalui jalur PMDK IPB dan diterima di Departemen Biokimia, Fakultas Matematika dan Ilmu Pengetahuan Alam.