ISOLASI DAN KLONING GEN SELULASE
DARI BAKTERI RUMEN SAPI
Rahadian Pratama
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN SUMBER INFORMASI
Dengan ini saya menyatakan bahwa tesis yang berjudul “Isolasi dan Kloning Gen Selulase dari Bakteri Rumen Sapi” merupakan karya saya dengan arahan dari komisi pembimbing dan belum pernah dipublikasikan dalam bentuk apapun kepada perguruan tinggi mana pun. Tesis ini merupakan bagian dari proyek penelitian Balai Penelitian Bioteknologi Perkebunan Indonesia (BPBPI). Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Bogor, September 2013
RINGKASAN
RAHADIAN PRATAMA. Isolasi dan Kloning Gen Selulase dari Bakteri Rumen Sapi. Dibimbing oleh I MADE ARTIKA dan TETTY CHAIDAMSARI.
Selulase adalah enzim yang dapat menghidrolisis biomassa selulosa dan dihasilkan oleh mikroorganisme yang tumbuh dengan menggunakan selulosa sebagai sumber karbonnya. Tujuan dari penelitian ini adalah mengisolasi dan melakukan kloning gen selulase dari bakteri penghasil selulosa berasal dari rumen sapi. Bakteri pemecah selulosa diisolasi dari cairan rumen menggunakan media selektif. RNA total diisolasi dari koloni yang memiliki aktivitas enzim selulase dan digunakan sebagai cetakan untuk konstruksi cDNA menggunakan teknik
Reverse Transcriptase–Polymerase Chain Reaction (RT-PCR). Teknik tersebut
menghasilkan cDNA sebagai produk dan digunakan sebagai cetakan untuk amplifikasi gen selulase menggunakan teknik PCR dengan primer spesifik. Kandidat gen selulosa yang diperoleh dikloning ke dalam vektor pGEM-T Easy, dilanjutkan dengan penentuan urutan nukleotida nya. Urutan sekuen nukleotida yang dihasilkan lalu dibandingkan dengan urutan gen selulase yang ada pada database GenBank.
SUMMARY
RAHADIAN PRATAMA. Isolation and Cloning of Cellulase Gene from Bovine Rumen Bacteria. Under the direction of I MADE ARTIKA and TETTY CHAIDAMSARI.
Cellulases are the enzymes that hydrolize cellulosic biomass and are produced by the microorganisms that grow over cellulosic matters. The objective of this research was to isolate and clone cellulase gene from cellulose-degrading bacteria of bovine rumen. Cellulose-degrading bacteria were isolated from rumen fluid using a selective medium. Total RNA was isolated from selected colony having cellulose degrading activity and it was used as a template for cDNA construction using reverse transcriptase polymerase chain reaction (RT-PCR) technique. The resulted cDNA was used as a template for PCR amplification of cellulose gene using specific primers. The cellulose gene candidate obtained was cloned into the pGEM-T-Easy vector followed by determination of its nucleotide sequence. The nucleotide sequence generated was aligned with sequences of cellulase genes from GenBank.
A number of isolates of rumen bacteria showed cellulase activity and the isolate CR-8 was selected for further analysis. Total RNA was successfully isolated from isolate CR-8 indicated by the presence of two intense bands of ribosomal RNA (23S and 16S). The reverse transcription process was successful and amplification of cellulase gene using the specific primer F1 and R1 resulted in a DNA fragment of 1900 bp as a candidate of cellulase gene. The fragment was successfuly cloned into the pGEM-T-Easy vector, and the resulted recombinant plasmid was successfully introduced into the E. coli cells. Nucleotide sequence analysis suggested that the isolated cellulase gene shares 99% homology with the endo-1,6-beta-glucanase of T. harzianum.
© Hak Cipta Milik IPB, Tahun 2013
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
ISOLASI DAN KLONING GEN SELULASE
DARI BAKTERI RUMEN SAPI
Rahadian Pratama
Tesis
sebagai salah sat u syarat unt uk memperoleh gelar M agist er Sains pada
Program St udi Biokimia
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
Judul Tesis : Isolasi dan Kloning Gen Selulase dari Bakteri Rumen Sapi Nama : Rahadian Pratama
NRP : G851110031
Disetujui oleh Komisi Pembimbing
Dr I Made Artika, MAppSc Dr Tetty Chaidamsari, MSi
Ketua Anggota
Diketahui oleh
Ketua Program Studi Dekan Sekolah Pascasarjana Biokimia
Prof Dr Drh Maria Bintang, MS Dr Dahrul Syah, MScAgr
PRAKATA
Puji dan syukur penulis panjatkan khadirat Allah SWT atas segala rahmat dan karunia-Nya sehingga penulisan tesis yang berjudul “Isolasi dan Kloning Gen Selulase dari Bakteri Rumen Sapi” dapat diselesaikan. Tesis ini merupakan salah satu syarat untuk memperoleh gelar Magister Sains pada Program Studi Biokimia, Sekolah Pascasarjana, Institut Pertanian Bogor.
Terima kasih penulis ucapkan kepada Bapak Dr I Made Artika, MAppSc dan Ibu Dr Tetty Chaidamsari, Msi selaku pembimbing atas bimbingan dan arahannya yang diberikan selama ini. Disamping itu, penulis sampaikan terima kasih kepada Balai Penelitian Bioteknologi Perkebunan Indonesia yang mendanai proyek penelitian ini.
Ucapan terima kasih juga disampaikan kepada Ibu dan Bapak tercinta, serta seluruh keluarga atas doa dan kasih sayangnya. Semoga Allah senantiasa membalas kebaikan semuanya dengan pahala yang berlipat ganda, amin.
Semoga hasil penelitian ini bermanfaat.
Bogor, September 2013
DAFTAR GAMBAR
Halaman
1. Bagan alir kegiatan penelitian 4
2. Pertumbuhan isolat CR-8 dalam media CCRA 9
3. Mekanisme hidrolisis selulosa 10
4. Hasil isolasi RNA dan amplifikasi gen selulase 11 5. Hasil PCR koloni menunjukkan amplifikasi gen sisipan 13
6. Kromatogram sekuen gen selulase 14
7. Perakitan DNA contig 15
8. Conserved domain selulase dari DNA contig 16
DAFTAR LAMPIRAN
Halaman
1. Inkubasi kultur bakteri pada inkubator anaerob 22
2. Seleksi bakteri pada media CCRA 23
3. Tabel nilai absorbansi larutan standar glukosa 24
4. Grafik linear kurva standar 25
5. Uji aktivitas selulase CR-8 (kualitatif) 26
6. DNA komplemen (cDNA) CR-8 (volume cDNA 2 µL) 27 7. Seleksi hasil transformasi (White/Blue Screen) 28 8. Hasil BLASTx gen selulase dari bakteri rumen sapi 29 9. Pensejajaran sekuen gen selulase asal bakteri rumen sapi dengan
1
PENDAHULUAN
Latar Belakang
Kehidupan manusia di bumi ini tidak terlepas dari peran tumbuhan sebagai penghasil oksigen bagi manusia untuk bertahan hidup. Tumbuhan hijau maupun non-hijau menempati hampir setengah dari daratan dengan beragam jenis. Sebagian besar tumbuhan dapat dimanfaatkan oleh manusia sebagai persediaan kayu untuk bahan bangunan, bahan bakar, pangan, obat, dan untuk keperluan lainnya. Namun ada juga sampah tumbuhan (biomass) yang belum dapat dimanfaatkan sepenuhnya oleh manusia.
Penyusun utama tumbuhan ialah selulosa dan hemi-selulosa. Selulosa merupakan senyawa organik polisakarida yang terdiri atas rantai linier dari beberapa ratus hingga lebih dari sepuluh ribu ikatan unit D-glukosa. Selulosa menjadi komponen utama penyusun dinding sel tumbuhan, bersama dengan senyawa lain seperti lignin yang menopang kekuatan dinding sel tumbuhan sehingga menjadi rigid dan kokoh. Tumbuhan menjadi sumber selulosa yang paling berlimpah di bumi namun pemanfaatannya belum maksimal sehingga masih banyak biomassa tumbuhan yang berakhir sebagai sampah. Pada zaman modern ini manusia dihadapkan pada krisis energi akibat kurangnya cadangan minyak bumi, sedangkan permintaan semakin meningkat. Berbagai sumber energi alternatif terus dicari dan banyak inovasi bermunculan seperti bioetanol, biosolar, dan sebagainya. Bahan-bahan tersebut umumnya berasal dari tumbuhan seperti jarak, dan sebagainya. Selulosa sebagai salah satu sumber karbohidrat yang paling berpotensi untuk dijadikan sumber energi alternatif belum banyak dikembangkan. Mengingat kelimpahan selulosa yang sangat tinggi di alam, selulosa cocok untuk dijadikan sebagai sumber energi alternatif, tetapi masih harus dikembangkan metode pemanfaatannya.
Salah satu kendala yang ditemui ialah cara memecah selulosa menjadi unit penyusunnya, D-glukosa, sehingga dapat dimanfaatkan untuk berbagai macam keperluan manusia. Metode degradasi selulosa yang paling aman, cepat, dan tidak memerlukan energi yang besar ialah dengan menggunakan bio-katalis atau enzim, yaitu enzim selulase.
Selulase ialah suatu grup enzim yang berkerja secara bersama-sama untuk menghidrolisis selulosa. Selulase terdiri atas eksoglukanase, endoglukanase dan β -glukosidase (kompleks selulase). Lebih detilnya, selulase dibagi menjadi β -1,4-endoglukanase (EG I, II, III dan V), β-1,4-selobiohidrolase (CBH I dan II), xilanase (XYN I dan II), β-glukosidase, α-L-arabinofuranosidase, asetil xilan esterase, β-mannase dan α-glukuronidase (Lenting and Warmoeskerken 2001). Molekul selulosa umumnya memiliki struktur yang mirip, seperti domain katalitik, domain pengikatan selulosa dan jembatan penghubungnya (linker). Dampaknya, selulosa dapat didegradasi menjadi glukosa secara sinergis dengan kompleks enzim ini. Sejumlah besar bakteri, fungi dan actinomycetes diketahui memiliki kemampuan untuk mendegradasi selulosa (Nagaraju et al. 2009).
jeans), yang dapat memberikan warna khas pada bahan jeans. Penggunaan selulase menghilangkan polusi lingkungan yang terjadi akibat penggunaan asam pada teknik pencucian jeans secara tradisional (Bai et al. 2012).
Para peneliti akhirnya mencoba untuk memproduksi enzim selulase dalam jumlah besar agar dapat digunakan untuk memecah selulosa dalam skala industri. Salah satu caranya ialah dengan kloning dan ekspresi gen penyandi selulase. Sumber selulase bisa berasal dari lambung sapi atau hewan pemakan rumput lainnya, bakteri dan fungi. Walaupun enzim selulase dari berbagai bakteri dan fungi telah diteliti, namun kemampuan selulase dari berbagai sumber isolasi dapat berbeda-beda. Oleh karena itu, diperlukan penelitian terhadap enzim selulase yang diisolasi dari isolat lokal Indonesia. Pada penelitian ini sumber enzim selulase diperoleh dari isolasi bakteri selulolitik yang terdapat dalam caian rumen sapi. Beberapa penelitian terdahulu sudah meneliti enzim selulase yang dihasilkan dari hewan ruminansia lainnya seperti pada kerbau yang sudah diakukan oleh
Sapi memiliki kemampuan untuk mencerna dan bertahan hidup dengan mengkonsumsi diet tinggi serat tetapi rendah protein. Kemampuan sapi tersebut dimungkinkan berkat adanya bakteri selulolitik, fibrolitik fungi dan protozoa (Chen et al. 2008: Lee et al. 2004). Gen penyandi enzim selulase yang diisolasi dapat diklon dan diekspresikan pada sel mikroba seperti Eschericia coli untuk produksi enzim selulase dalam skala besar.
Tujuan Penelitian
Penelitian ini bertujuan:
1. Mengisolasi gen penyandi selulase dari bakteri selulolitik yang hidup di dalam rumen sapi.
2. Mengklon gen penyandi selulase yang telah diisolasi ke dalam sel E. coli. 3. Menentukan gen penyandi selulase yang telah diisolasi apakah berupa gen
utuh atau fragmen.
Hipotesis
Hipotesis pada penelitian ini yaitu:
1. Gen penyandi selulase dari bakteri selulolitik dapat diisolasi dan diklon ke bakteri E. coli.
2. Gen yang didapat berupa gen utuh sehingga dapat diaplikasikan langsung.
2
BAHAN DAN METODE
Tempat dan Waktu
Bahan
Sumber bakteri selulolitik berasal dari rumen sapi segar yang diperoleh di rumah potong hewan. Bakteri inang E. coli XL1-Blue merupakan milik BPBPI. Kit yang digunakan pada penelitian berupa kit komersil yang diperoleh dari masing-masing vendor (Invitrogen, Promega).
Metode
Penelitian terdiri atas beberapa tahapan seperti pada Gmbar 1. Tahap pertama ialah skrining bakteri penghasil enzim selulase dari cairan rumen sapi (media selektif CCRA), tahap kedua ialah pengujian aktivitas enzim selulase (metode DNS dan substrat CMC), tahap ketiga ialah isolasi RNA total bakteri, tahap keempat ialah studi bioinformatika dan desain primer spesifik selulase, tahap kelima ialah amplifikasi dan kloning gen selulase, tahap keenam ialah sekuensing kandidat gen selulase, tahap terakhir ialah analisis bioinformatika kandidat gen selulase (DNASTAR, Bioedit, BLAST)
Gambar 1 Bagan alir kegiatan penelitian
Isolasi Bakteri Rumen Sapi
Rumen sapi diperoleh dari rumah potong hewan (RPH). Rumen sapi yang masih segar diambil cairannya menggunakan syringe yang sudah disiapkan sebelumnya dan cairan segera dimasukkan ke dalam tabung koleksi anaerob (berisi resazurin agar). Tabung koleksi anaerob kemudian dimasukkan ke dalam anaerobic gloves box (85% N2, 5% CO2, 10% H2, dan kurang dari 10 ppm O2) (Dowell 1972). Cairan rumen sapi tersebut kemudian ditumbuhkan dalam media agar selektif (Cellulose Congo-Red Agar, CCRA) sehingga yang tumbuh ialah bakteri yang memiliki kemampuan mendegradasi selulosa. Komposisi CCRA yaitu: KH2PO4 0.5 g, MgSO4 0.25 g, Congo-Red 0.2 g, agar 7.5 g, gelatin 2 g,
carboxymethyl cellulose 1.88 g; akuades1 L dan pH 6.8 – 7.2 (Gupta et al. 2012).
Bakteri yang ditumbuhkan dalam cawan petri media CCRA diberi kode CR. Koloni bakteri yang menghasilkan zona bening atau pertanda memiliki aktivitas selulase, diisolasi dan ditumbuhkan pada media Luria Broth (LB) dan disimpan untuk uji selanjutnya.
Uji Aktivitas Enzim Selulase
Kultur bakteri dipipet 50 µl ke dalam tabung reaksi, kemudian ditambahkan bufer sitrat 950 µl, dan substrat CMC 1%. CMC merupakan substrat yang baik untuk menginduksi produksi enzim selulase pada bakteri (Ahmad et al. 2003: Dashtban et al. 2011). Campuran kemudian dikocok lalu diinkubasi 50oC selama 10 menit, kemudian dididihkan selama 15 menit. Ke dalam tabung reaksi ditambahkan larutan DNS sebanyak 3 ml. Campuran kemudian di-vortex dan dididihkan selama 10 menit lalu diukur absorban pada λ540 nm. Sebagai standar, digunakan glukosa dengan komposisi glukosa-akuades, CMC dan DNS dengan perbandingan 1:1:1. Konsentrasi standar yang digunakan ialah 100, 200, 400, 800 dan 1600 ppm.
Isolasi RNA
Isolasi RNA dilakukan menggunakan peqGOLD Bacterial RNA Kit dari Invitrogen. Setelah bakteri hasil isolasi ditumbuhkan pada media LB, RNA diisolasi dan dilanjutkan dengan elektroforesis gel terhadap hasil isolasi RNA dengan konsentrasi agarosa 1%.
Sintesis cDNA
Sintesis cDNA (transkripsi balik) dilakukan menggunakan kit Superscripttm First-Strand dari Invitrogen dan sistem PCR. Setelah cDNA di sintesis, kemudian dilakukan elektroforesis terhadap cDNA dengan kepadatan gel 0.8%. Sebagai marker digunakan 1Kb DNA Ladder dari Invitrogen.
Desain Primer Selulase
Primer selulase didesain menggunakan cetakan dari sekuen selulase yang telah ada di website National Center for Biotechnology Information (www.ncbi.nlm.nih.gov). Beberapa sekuen selulase dipilih (exoglucanase, endoglucanase, cellobiase) sebagai referensi. Sekuen selulase bersumber dari
Trichoderma reesei. Desain primer menggunakan bantuan software Lasergene
DNASTAR versi 7.
Amplifikasi Gen Selulase
Amplifikasi gen penyandi selulase dilakukan dengan metode Polymerase
berdasarkan cetakan gen selulase. Enzim yang digunakan dalam PCR ialah Taq DNA polymerase dari Invitrogen beserta kit PCR seperti bufer dan dNTP. Untuk Mix PCR 1X berisi bufer Taq Polimerase 2,5 µl, dNTP 1 µl, Primer forward 1 µ l, primer reverse 1 µ l, Taq polimerase 1 µ l dan molecular water (akuades steril, MW) 13,5 µl. Untuk amplifikasi gen selulase, komposisi pereaksi adalah cDNA target 1 µl, MW 4 µ l dan Mix 20 µ l. PCR dilakukan sebanyak 35 siklus dengan setiap siklus diatur suhu denaturasi 94 oC, suhu annealing dibuat bervariasi untuk mencari suhu annealing yang tepat, dan suhu elongasi 72 oC. setelah PCR, hasilnya diuji dengan elektroforesis gel 0,8% dan marker 1 Kb DNA Ladder.
Recovery DNA
DNA yang telah di elektroforesis dan menghasilkan pita yang cukup tebal dipulihkan kembali (recovery). Pemulihan DNA menggunakan kit Quick Gel
Extraction dari Invitrogen. DNA yang telah dipulihkan kemudian dielektroforesis
kembali sebanyak 1 µl untuk memastikan keberadaan DNA tersebut.
Pembuatan Sel Kompete n
Sel kompeten yang digunakan ialah bakteri E.coli strain XL1-blue. Bakteri dikulturkan pada media SOB + tetrasiklin pada suhu 37oC hingga mencapai OD600=0.6. Lalu didinginkan dalam es selama 10 menit, disentrifugasi 4000 rpm 2oC 10 menit, diresuspensi dalam bufer TB, lalu didiamkan dalam es selama 10 menit. Selanjutnya kultur disentrifugasi kembali 3000 rpm 2oC 10 menit, resuspensi kembali dalam TB buffer, ditambahkan DMSO dalam es selama 10 menit lalu dibekukan dengan nitrogen cair dan disimpan dalam suhu -70oC.
Ligasi
Kandidat gen selulase kemudian diligasikan dengan vektor pGEM-T-Easy dari Invitrogen. cDNA dicampurkan dengan komponen ligasi (buffer, vektor, enzim ligasi, kontrol DNA) dan dibiarkan selama satu jam pada suhu ruang (25 oC). Hasil ligasi lalu digunakan untuk mentranformasi sel bakteri kompeten.
cawan petri lain. Petri diinkubasi semalam dan dicek koloni yang tumbuh keesokan paginya.
Seleksi Transforman
Seleksi transforman dilakukan menggunakan metode PCR Koloni. Koloni berwarna putih yang tumbuh setelah transformasi kemudian diuji dengan PCR koloni menggunakan primer khusus (M13). Produk PCR lalu dielektroforesis. Teknik PCR koloni digunakan untuk mendeteksi koloni yang membawa DNA insert dengan cara mengamplifikasi DNA insert tersebut. Koloni yang membawa DNA insert akan menunjukkan pita yang jelas pada gel elektroforesis dengan ukuran yang telah diketahui.
Analisis Sekuen Kandidat Gen Selulase
Kandidat gen selulase yang telah disekuen kemudian di analisis dengan program Bioedit (www.mbio.ncsu.edu/bioedit/bioedit.html) dan DNASTAR untuk menentukan kualitas sekuen DNA dengan memeriksa grafik kromatogram. Selanjutnya sekuen DNA dibersihkan dari sisa primer dan vektor lalu diperiksa secara keseluruhan untuk melihat kesatuan DNA nya (DNA contig). Setelah dikonfirmasi apakah sekuen DNA contig yang didapat berupa fragmen DNA atau DNA utuh, dilakukan pencarian sekuen terhadap database DNA dengan program BLASTx (blast.ncbi.nlm.nih.gov/Blast.cgi).
3
HASIL DAN PEMBAHASAN
Isolat Bakteri Rumen Penghasil Selulase
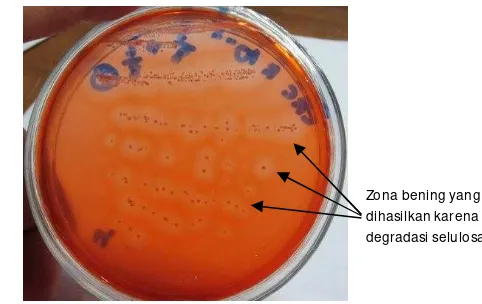
Gambar 2 Pertumbuhan isolat CR-8 menghasilkan zona bening dalam media CCRA.
Beberapa isolat lain menunjukkan zona bening namun tidak sejelas zona bening dari isolat CR-8. Beberapa bakteri selulolitik yang berhasil diidentifikasi dari rumen sapi ialah Fibrobacter Succinogenes, Ruminococcus albus dan Ruminococcus flavefaciens (Koike and Kobayashi 2001: Deng et al. 2007). Bakteri Fibrobacter sp yang telah diisolasi mengindikasikan terdapat dua spesies yang berbeda, F. succinogenes S85 dan F. intestinalis NR9 dengan tingkat homologi yang rendah diantara spesies tersebut (Qi et al. 2004: Bera-Maillet et al. 2004). Selain bakteri selulolitik, beberapa bakteri yang ditemukan dalam jumlah besar pada cairan rumen ialah Prevotella sp dan Eubacterium sp, diikuti dengan
Ruminococcus sp, Clostridium sp, Roseburia sp dan beberapa bakteri lain hingga
mencapai total 25 bakteri yang diketahui (Broadway et al. 2012). Hasil penelitian tersebut sedikit berbeda dengan yang dilaporkan oleh Ozutsumi et al. (2005) yang menyebutkan bahwa bakteri terbanyak pada cairan rumen sapi ialah Bacteroides sp dan Prevotella sp. Wang et al. (2011) mengisolasi dan mengkarakterisasi enzim glikosil hidrolase dari Neocallimastix patriciarum, semacam fungi yang hidup di rumen sapi. Penelitian tersebut menunjukkan bahwa selain bakteri, fungi juga membentuk simbiosis mutualisme dengan sapi.
Enzim selulase terdiri dari beberapa jenis yang ditandai dengan di bagian mana enzim tersebut memotong ikatan beta 1-4 pada selulosa. endoselulase (EC 3.2.1.4), memotong ikatan beta secara acak pada bagian dalam rantai polimer selulosa, menghasilkan ujung rantai yang baru. Eksoselulase (EC 3.2.1.91), memotong dua sampai empat unit ikatan beta dari ujung rantai polimer yang dihasilkan oleh endoselulase, menghasilkan tetrasakarida, disakarida maupun selobiosa. Selobiase (EC 3.2.1.21) atau beta-glukosidase menghidrolisis produk eksoselulase menjadi unit-unit monosakarida (Zverlov et al. 2005). Mekanisme hidrolisis selulosa menjadi monomer monosakaridanya diilustrasikan seperti pada gambar 3 di bawah.
Gambar 3 Mekanisme hidrolisis selulosa
(Sumber: http://en.wikipedia.org/wiki/File:Types_of_Cellulase2.png)
Beberapa bakteri rumen yang dapat mendegradasi selulola meliputi Fibrobacter sp, Ruminococcus sp, Clostridium sp, dan bakteri lainnya. Selain memiliki fungsi selulolitik, bakteri rumen juga memiliki beberapa fungsi hidrolisis lainnya seperti xilanolitik, amilolitik, lipolitik, degradasi oksalat, dan beberapa fungsi lainnya (Chiba 2009). Isolat CR-8 selanjutnya ditumbuhkan dalam media LB untuk keperluan analisis selanjutnya.
Isolat CR-8 yang diduga menghasilkan enzim selulase, dalam uji aktivitas enzim juga memperlihatkan aktivitas selulase dengan memecah CMC menjadi glukosa-glukosa bebas yang ditandai dengan perubahan warna larutan DNS. Berdasarkan kurva standar glukosa yang dibuat dan pengukuran nilai absorbansi sampel CR-8, diketahui konsentrasi glukosa yang dihasilkan dari pemecahan CMC 1% ialah 2432 ppm. Nilai tersebut cukup tinggi mengingat batas atas konsentrasi standar glukosa ialah 1600 ppm.
Kandidat Gen Selulase
A B
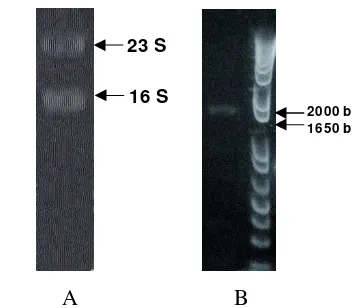
Gambar 4 Hasil isolasi RNA dan amplifikasi gen selulase, (A) dua pita RNA, 23S dan 16S, (B) amplifikasi menggunakan cetakan cDNA dan pasangan primer F1-R1.
Pada Gambar 4A hasil elektroforesis, terlihat dua pita, pita tersebut menunjukkan subunit 23S dan 16 dari 70S RNA ribosom bakteri. RNA ribosom merupakan jenis RNA yang jumlahnya paling besar, sehingga bila kedua pita tersebut terdeteksi menunjukkan kualitas isolasi yang cukup baik. RNA selanjutnya diubah menjadi cDNA melalui proses transkripsi balik menggunakan kit Superscripttm First-Strand dari Invitrogen. Hasil yang didapat ialah cDNA dengan volume total 20 µL.
Untuk mengamplifikasi sekuen cDNA penyandi selulase, primer khusus didesain berdasarkan cetakan gen enzim selulase dari fungi Trichoderma reesei. T. reesei dipilih karena enzim selulase fungi tersebut sudah umum diteliti sehingga diharapkan primer yang didesain khusus tersebut dapat mengamplifikasi gen penyandi selulase pada bakteri dengan baik. T. reesei banyak dimanfaatkan dalam industri karena kemampuannya untuk mensekresikan selulase dan hemi-selulase dalam jumlah besar (Oinonen 2004). Pembuatan primer mengacu pada kriteria yang ditetapkan oleh Rozen & Skaletsky (1999). Cetakan gen selulase T.
reesei didapatkan dari bank gen digital di situs NCBI. Primer didesain
menggunakan bantuan program primerselect (Lasergene DNASTAR v7). Karena muncul berbagai open reading frame (ORF) dari cetakan DNA, maka disiapkan dua set primer, F1-R1 dan F2-R2. Untuk meminimalisir keambiguan karena beberapa reading frame, dilakukan perbandingan beberapa sekuen penyandi selulase dari spesies kerabat T. reesei seperti T. virens, T. harzianum, dan beberapa bakteri pendegradasi selulosa lainnya. ORF yang paling sering muncul dijadikan dasar dalam mendesain primer. Persentase GC dan melting point (Tm) diatur agar dapat seefisien mungkin menempel pada cetakan (Dieffenbach et al. 1993). Primer yang digunakan untuk amplifikasi gen selulase tercantum dalam Tabel 1.
Tabel 1 Primer yang didesain dari cetakan sekuen DNA penyandi selulase T.reesei.
Amplifikasi cDNA dilakukan menggunakan pasangan primer F1 dan R1, F2 dan R2. Data menunjukkan bahwa pasangan primer F1-R1 berhasil mengamplifikasi gen penyandi selulase, sedangkan kombinasi primer lainnya gagal mengamplifikasi. Kegagalan kombinasi primer lainnya dalam proses amplifikasi diduga karena proses annealing primer tidak cukup baik, sehingga proses amplifikasi tidak berjalan.
Hasil amplifikasi gen selulase menggunakan pasangan primer F1-R1 dikonfirmasi menggunakan gel elektroforesis. Hasil yang didapat ialah pita DNA dengan ukuran sekitar 1800 – 1900 pasang basa (bp, Gambar 4B). Sebagai referensi, gen cellobiohydrolase I (eksoglukanase) T. reesei memiliki ukuran 2220 bp (NCBI 2005). Selulase pada sel eukariot (dalam hal ini fungi T.reesei) umumnya memiliki sistem yang lebih kompleks dibanding selulase pada bakteri, sehingga perbedaan ukuran sekuen DNA penyandi umum terjadi.
Kandidat gen selulase yang telah dipurifikasi dari gel elektroforesis dan diklon ke dalam vektor pGEM-T-Easy. Plasmid rekombinan yang dihasilkan digunakan untuk mentransformasi sel kompeten E. coli strain XL1-Blue. Penggunaan E. coli strain XL1-Blue sebagai sel kompeten dikarenakan strain tersebut yang paling optimal dalam transformasi plasmid ke dalam sel (Zhiming et al. 2005). Strain XL1-Blue memiliki efisiensi transformasi dan colony forming unit (cfu) paling tinggi dibanding strain DH5α dan TG1. Hasil PCR koloni mempertegas keberhasilan transformasi sel E. coli. Pita yang cukup intens pada hasil elektroforesis PCR koloni (Gambar 5) menandakan koloni E. coli membawa gen target. Sekuensing gen kandidat selulase yang berhasil di klon dilakukan oleh perusahaan 1st Base di Singapura.
Gambar 5 Hasil PCR koloni menunjukkan amplifikasi gen sisipan (selulase).
Data Bioinformatika Kandidat Gen Selulase
Hasil sekuensing kandidat gen selulase dari 1st Base berupa file sekuen (*.seq) dan file kromatogram (*.ab1). Hasil sekuensing yang baik dapat dilihat dari grafik kromatogram yang bagus, yaitu puncak setiap basa terlihat jelas dan berjarak sama dari satu basa ke basa lainnya.
Kualitas profil kromatogram sekuen DNA hasil 1st Base secara
keseluruhan terlihat baik dengan noise yang rendah dan puncak yang jelas. Namun pada beberapa sekuen, kekuatan sinyal kromatogram terlihat rendah. Hal tersebut dapat terjadi salah satunya karena jumlah DNA sampel yang kurang, adanya kontaminan, atau proses annealing primer yang kurang (Kingdon et al. 2010).
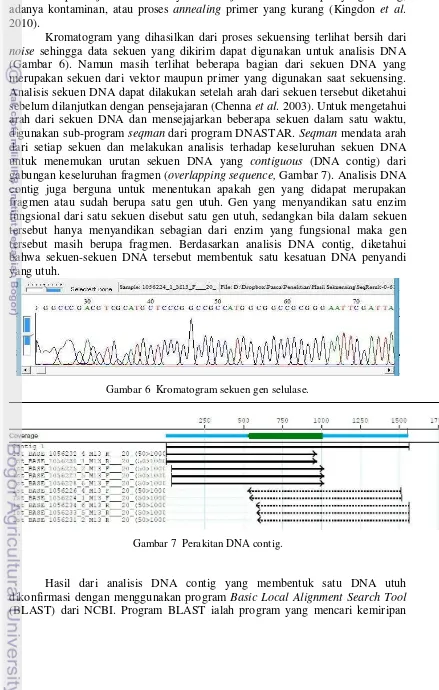
Kromatogram yang dihasilkan dari proses sekuensing terlihat bersih dari noise sehingga data sekuen yang dikirim dapat digunakan untuk analisis DNA (Gambar 6). Namun masih terlihat beberapa bagian dari sekuen DNA yang merupakan sekuen dari vektor maupun primer yang digunakan saat sekuensing. Analisis sekuen DNA dapat dilakukan setelah arah dari sekuen tersebut diketahui sebelum dilanjutkan dengan pensejajaran (Chenna et al. 2003). Untuk mengetahui arah dari sekuen DNA dan mensejajarkan beberapa sekuen dalam satu waktu, digunakan sub-program seqman dari program DNASTAR. Seqman mendata arah dari setiap sekuen dan melakukan analisis terhadap keseluruhan sekuen DNA untuk menemukan urutan sekuen DNA yang contiguous (DNA contig) dari gabungan keseluruhan fragmen (overlapping sequence, Gambar 7). Analisis DNA contig juga berguna untuk menentukan apakah gen yang didapat merupakan fragmen atau sudah berupa satu gen utuh. Gen yang menyandikan satu enzim fungsional dari satu sekuen disebut satu gen utuh, sedangkan bila dalam sekuen tersebut hanya menyandikan sebagian dari enzim yang fungsional maka gen tersebut masih berupa fragmen. Berdasarkan analisis DNA contig, diketahui bahwa sekuen-sekuen DNA tersebut membentuk satu kesatuan DNA penyandi yang utuh.
Gambar 6 Kromatogram sekuen gen selulase.
Gambar 7 Perakitan DNA contig.
suatu region dalam beberapa sekuen. BLAST membandingkan sekuen nukelotida atau protein target dengan database yang dimiliki NCBI dan menghitung persamaan yang dimiliki secara statistik. Jenis BLAST yang digunakan ialah BLASTx, yaitu program BLAST yang akan menerjemahkan input sekuen DNA target ke dalam sekuen protein lalu membandingkannya dengan database protein yang dimilikinya. Berdasarkan hasil pencarian BLASTx, sekuen DNA contig memiliki kemiripan dengan sekuen glucan endo-1,6-beta-glucanase
[Trichoderma harzianum] (Cruz et al. 1995) dengan tingkat kemiripan mencapai
99%. Sekuen glucan endo-1,6-beta-glucanase tersebut terdiri atas 430 asam amino, bits score 722 dan E-value 0 yang menunjukkan sekuen DNA contig memiliki kemiripan yang tinggi dengan sekuen pada database GenBank (Claverie & Notredame 2007: Davidson and Blaxter 2005).
Sebagai perbandingan, enzim selulase yang dihidrolisis dari rumen kerbau (famili glikosil hidrolase, GHF), memiliki ukuran sekitar 1200 bp dan masih berupa fragmen gen (Rungrattanakasin et al. 2011). Enzim serupa lainnya yang berhasil diisolasi dari rumen ialah GHF 45 dari Fibrobacter succinogenes, sayangnya gen yang didapat belum utuh karena terdapat beberapa domain yang absen (Park JS et al. 2007).
Pada sekuen DNA contig juga ditemukan conserved domain yaitu domain selulase, sehingga menegaskan bahwa sekuen DNA contig menyandi enzim selulase (Gambar 8).
Gambar 8 Conserved domain selulase dari sekuen DNA contig.
4
SIMPULAN DAN SARAN
Simpulan
Gen penyandi selulase dari rumen sapi berhasil diisolasi dan telah diklon ke dalam vektor pGEM-T-easy dan dimasukkan ke dalam sel E. coli strain XL1-Blue. Sekuen-sekuen DNA yang didapat merupakan satu kesatuan DNA utuh yang menyandikan enzim endoglukanase yang berdasarkan hasil BLASTx, memiliki kemiripan mencapai 99% dengan endoglukanase T. harzianum, dan terbukti mengandung conserved domain enzim selulase pada sekuennya.
Saran
DAFTAR PUSTAKA
Ahmad S, Qurrat-ul-Ain N, Aslam S, Naeem S, Rahman, Jamil A. 2003. Induction of Xylanase and Cellulase genes from Trichoderma harzianum with different carbon sources. Pak. Jour. Biol. Sci. 6(22): 1912-1916. Bai S, Kumar MR, Kumar DJM, Balashanmugam P, Balakumaran MD,
Kalaichelvan PT. 2012. Cellulase production by Bacillus subtilis isolated from cow dung. Archives of Applied Science Research 4(1): 269-279. Bera-Maillet C, Ribot Y, Forano E. 2004. Fiber-degrading systems of different
strains of the genus Fibrobacter. Appl. Environ. Microb. 70: 2172–2179. Broadway PR, Callaway TR, Carroll JA, Donaldson JR, Rathmann RJ, Johnson
BJ, Cribbs JT, Durso LM, Nisbet DJ, Schmidt TB. 2012. Evaluation of the ruminal bacterial diversity of cattle fed diets containing citrus pulp pellets. Agric. Food Anal. Bacteriol. 2(4): 297-308.
Chenna R, Sugawara H, Koike T, Lopez R, Gibson TJ, Higgins DG, Thompson JD. 2003. Multiple sequence alignment with the Clustal series of programs. Nucleic Acid Research. 131(13):3497-3500. DOI:10.1093/nar/gkg500.
Chen XL, Wang JK, Wu YM, Liu JX. 2008. Effects of chemical treatments of rice straw on rumen fermentation characteristics, fibrolytic enzyme activities and populations of liquid-and solid associated ruminal microbes in vitro. Animal Feed Sci. Technol. 141: 1-14.
Claverie JM, Notredame C. 2007. Bioinformatics for Dummies, 2nd Edition. USA: Wiley Publishing, Inc.
Cruz J, Toro JAP, Benitez T, Llobell A. 1995. Purification and characterization of an endo-beta-1,6-glucanase from Trichoderma harzianum that is related to its mycoparasitism. J Bacteriol. 177(7):1864-1871.
Dashtban M, Buchkowski R, Qin W. 2011. Effect of different carbon sources on cellulase production by Hypocrea jecorina (Trichoderma reesei) strains. Int. J. Biochem Mol. Biol. 2(3): 274-286.
Davidson A, Blaxter M. 2005. Ancient origin of glycosyl hydrolase family 9 cellulase gene. Molecular Biology and Evolution 22(5): 1273-1284. Deng W, Wang L, Ma S, Jin B, Bao He T, Yang Z, Mao H, Wanapat M. 2007.
Comparison of Gayal (Bos frontalis) and Yunnan Yellow cattle (Bos taurus): rumen function, digestibilities and nitrogen balance during feeding of pelleted lucerne (Medicago sativum). Asian-Aust. J. Anim. Sci. 20: 900-907.
Dowel VR. 1972. Comparison of techniques for isolation and identification of anaerobic bacteria. The American Journal of Clinical Nutrition 25: 1335-1343.
Gupta P, Samant K, Sahu A. 2011. Isolation of cellulose-degrading bacteria and determination of their cellulolytic potential. International Journal of
Microbiology. 2012:1-5. doi:10.1155/2012/578925.
Hendricks CW, Doyle JD, Hugley B. 1995. A new solid medium for enumerating cellulose-utilizing bacteria in soil. Applied and Environmental
Kingdon PG, Gedge F, Donahue WW, Schrijver I, Weck KE, Kant JA, Oglesbee D, Toydemir PB, Lyon E. 2012. Design and analytical validation of clinical DNA sequencing assays. Arch Pathol Lab Med. 136: 41-46. doi:10.5858/arpa.2010-0623-OA.
Koike, S. and Y. Kobayashi. 2001. Development and use of competitive PCR assays for the rumen cellulolytic bacteria: Fibrobacter succinogenes,
Ruminococcus albus and Ruminococcus flavefaciens. FEMS Microbiol.
Lett. 204: 361-366.
Lee SS, Choi CK, Ahn BH, Moon YH, Kim CH, Ha JK. 2004. In vitro stimulation of rumen microbial fermentation by a rumen anaerobic fungal culture. Animal Feed Sci. Technol. 115: 215-226.
Lenting HBM, Warmoeskerken MMCG. 2001. Mechanism of interaction between cellulase action and applied shear force, an hypothesis. J Biotechnol. 89: 217-226.
Nagaraju M, Narasimha G, Rangaswamy V. 2009. Impact of sugar industry effluents on soil cellulase activity. Int Biodeter Biodegr. 63:1088-1092. Oinonen AM. 2004. Trichoderma reesei Strains for Production of Cellulases for
the Textile Industry. Helsinki: VTT Publications 550.
Ozutsumi Y, Tajima K, Takenaka A, Itabashi H. 2005. The effect of protozoa on the composition of rumen bacteria in cattle using 16S rRNA gene clone libraries. Biosci. Biotechnol. Biochem. 69(3): 499-506.
Park JS, Russel JB, Wilson DB. 2007. Characterization of a family 45 glycosyl hydrolase from Fibrobacter succinogenes S85. Anaerobe 13: 83-88.
Qi M, Nelson KE, Daugherty SC, Nelson WC, Hance IR, Morrison M, Forsberg CW. 2004. Novel molecular features of the fibrolytic intestinal bacterium Fibrobacter intestinalis not shared with Fibrobacter succinogenes as determined by suppressive subtractive hybridization. J. Bacteriol 187: 3739–3751.
Rozen S, Skaletsky H. 1999. Primer3 on the www for general users and for biologist programmers. Method In Mol Biol. 132:365-385.
Rungrattanakasin B, Jirajaroenrat K, Maneewan K, Chaowarat M. 2011. Molecular cloning of glycoside hydrolase family 9 cellulase gene from buffalo rumen. 2011 International Conference on Bioscience,
Biochemistry and Bioinformatics Vol.5. Singapore: IACSIT Press.
Wang TY, et al. 2011. Functional characterization of cellulases identified from the cow rumen fungus Neocallimastix patriciarum W5 by transcriptomic and secretomic analyses. Biotechnology for Biofuels 4:24. doi:10.1186/1754-6834-4-24.
Lampiran 2 Seleksi bakteri pada media CCRA.
CR-1 CR-2
CR-3 CR-4
CR-5 CR-6
Lampiran 3 Tabel nilai absorbansi larutan standar glukosa.
Konsentrasi
(ppm) Absorbansi
100 0.053
200 0.254
400 0.798
800 1.586
Lampiran 4 Grafik linear kurva standar.
Y = 0,1412 + 1,2x10-3X
Lampiran 5 Uji aktivitas selulase CR-8 (kualitatif).
Ulangan ke- A
1 2.432
2 2.432
3 1.931
4 1.920
Lampiran 6 DNA komplemen (cDNA) CR-8 (volume cDNA 2 µL).
1000 bp
400 bp
cDNA CR-8
RIWAYAT HIDUP
Penulis dilahirkan di Bogor 5 Februari 1989 dari pasangan Prasodjo Waluyo dan Iis Arifiantini. Penulis merupakan putra ke 1 dari 4 bersaudara.
Tahun 2006 penulis lulus dari SMA Negeri 7 Bogor dan pada tahun yang sama melanjutkan ke Institut Pertanian Bogor, penulis memilih Mayor Biokimia dan lulus tahun 2010. Pada tahun 2011 penulis diterima di Program Studi Biokimia Program Pascasarjana IPB melalui sponsor Beasiswa Unggulan DIKTI.
Pada tahun 2010 penulis bekerja sebagai tenaga honorer di kantor Program Internasional (International Collaboration Office), IPB hingga saat ini.