IDENTIFIKASI MOLEKULER, KERAGAMAN GENETIK
DAN KARAKTERISTIK HABITAT SIPUT LAUT
(NUDIBRANCHIA) DARI BEBERAPA POPULASI
DI INDONESIA
INDRI VERAWATI
DEPARTEMEN ILMU DAN TEKNOLOGI KELAUTAN FAKULTAS PERIKANAN DAN ILMU KELAUTAN
INSTITUT PERTANIAN BOGOR BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa skripsi berjudul Identifikasi Molekuler, Keragaman Genetik dan Karakteristik Habitat Siput Laut (Nudibranchia) dari Beberapa Pupulasi di Indonesia adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
ABSTRAK
INDRI VERAWATI. Identifikasi Molekuler, Keragaman Genetik dan Karakteristik Habitat Siput Laut (Nudibranchia) dari Beberapa Populasi di Indonesia. Dibimbing oleh HAWIS MADDUPPA dan BEGINER SUBHAN.
Nudibranchia berasal dari “Nudus” berarti telanjang dan “Branchia” yang
berarti insang. Spesies Nudibranchia yang telah teridentifikasi di kawasan perairan Indonesia sebanyak 59 spesies dan terdiri dari 15 famili. Namun, eksplorasi di studi tentang jenis-jenis nudibranch di Indonesia masih jarang dilakukan. Tujuan dari penelitian ini adalah untuk mengidentifikasi serta menganalisis keragaman genetik nudibranch di Indonesia dengan menggunakan metode molekuler, menganalisis karakteristik habitat dan hubungannya dengan spesies nudibranch di Indonesia. Metode yang digunakan adalah DNA Barcoding dengan marka mitokondria lokus Cytochrom oxidase 1 (CO1). Hasil BLAST yang diinterpretasikan dalam pohon filogenetik menghasilkan 14 spesies nudibranch dari 30 sampel dengan tujuh lokasi di Indonesia. Keragaman haplotipe untuk setiap spesies nudibranch berbeda-beda dengan kategori rendah hingga sedang serta memiliki nilai keragaman genetik 0 - 0.5. Nilai keragaman genetik rendah terdapat pada spesies Phyllidiella pustulosa di Gosong pramuka, Phyllidiella pustulosa di Papua, Phyllidia ocellata di Papua, Phyllidiella pustulosa, Phyllidia ocellata, Chromodoris annae and Elysia cf marginata di Papua, Phyllidia elegans dan Phyllidia coelestis di Luwuk, Phyllidia varicosa di Nusa Penida, Taringa caudata di Pulau Dapur, Doriprismatica atromarginata, Mexichromis multituberculata, Risbecia tyroni, Chromodoris striatella di Teluk Jakarta. Nilai keragaman genetik sedang terdapat pada Phyllidiella pustulosa di Luwuk, Nusa Penida dan Biak, Chromodoris magnifica di Nusa Penida, serta Plakobranchus sp. di Papua. Hubungan karakteristik habitat dengan spesies nudibranch didapatkan melalui Correspondence Analysis (CA) yang menjelaskan spesies nudibranch yang termasuk kedalam famili Phyllidiidae habitatnya adalah terumbu karang dan spesies dalam famili Chromodorididae banyak mendiami rubbel sebagai habitatnya.
Kata kunci: Correspondence Analysis (CA), DNA Barcoding, hubungan karakteristik habitat dengan spesies, keragaman genetik, Nudibranch
ABSTRACT
INDRI VERAWATI. Molecular Identification, Genetic Diversity and Habitat Characteristics of Sea Slug (nudibranch) from Many Populations in Indonesia. Supervised by HAWIS MADDUPPA and BEGINER SUBHAN.
relationship between the habitat characteristic of nudibranch and species in study site. DNA Barcoding with markers mitochondrial Cytochrom oxidase 1 (CO1) locus was used. A total of 14 species was succesfully identified from 30 tissue samples. The range of maximum identification in BLAST was ranged from 87% - 99%. The haplotype diversity was categorized low and medium (0 - 0.5). The low genetic diversity was observed in Phyllidiella pustulosa at Gosong pramuka, Phyllidiella pustulosa, Phyllidia ocellata, Chromodoris annae and Elysia cf marginata at Papua, Phyllidia elegans and Phyllidia coelestis at Luwuk, Phyllidia varicosa at Nusa Penida, Taringa caudata at Dapur Island, Doriprismatica atromarginata, Mexichromis multituberculata, Risbecia tyroni and Chromodoris striatella at Jakarta Bay. The medium genetic diversity value was founded in Phyllidiella pustulosa at Luwuk, Nusa Penida and Biak, Chromodoris magnifica at Nusa Penida, Plakobranchus sp. at Papua. The relationship between habitat characteristic and species of nudibranch is founded by Correspondence Analysis (CA), which explains the species for family Phyllidiidae was mainly observed in corals and species for family Chromodorididae was mainly observed oftenly in rubble as its habitat.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Ilmu Kelautan
pada
Departemen Ilmu dan Teknologi Kelautan
IDENTIFIKASI MOLEKULER, KERAGAMAN GENETIK
DAN KARAKTERISTIK HABITAT SIPUT LAUT
(NUDIBRANCHIA) DARI BEBERAPA POPULASI
DI INDONESIA
INDRI VERAWATI
DEPARTEMEN ILMU DAN TEKNOLOGI KELAUTAN FAKULTAS PERIKANAN DAN ILMU KELAUTAN
INSTITUT PERTANIAN BOGOR BOGOR
PRAKATA
Puji dan syukur penulis ucapkan kepada Allah subhanahu wata’ala atas
segala rahmat dan karunia-Nya sehingga penulis dapat menyelesaikan skripsi yang berjudul “Identifikasi Molekuler, Keragaman Genetik dan Karakteristik Habitat Siput Laut (Nudibranchia) dari Beberapa Populasi di Indonesia”. Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Ilmu Kelautan pada Departemen Ilmu dan Teknologi Kelautan.
Pada kesempatan ini penulis mengucapkan terima kasih kepada semua pihak yang telah membantu dalam penulisan dan penyusunan skripsi ini terutama kepada:
1. Tuhan Yang Maha Esa atas segala rahmat yang diberikan kepada penulis hingga dapat menyelesaikan penulisan skripsi ini.
2. Bapak Dr. Hawis Madduppa, SPi, MSi dan Bapak Beginer Subhan, SPi MSi selaku pembimbing, yang telah banyak memberi segala saran, bimbingan, dan nasihat selama penelitian berlangsung hingga karya ilmiah ini selesai.
3. Laboratorium Biosistematik dan Biodiversitas Kelautan ITK IPB atas penyediaan fasilitas dalam proses pengolahaan data dalam dan penulisan skripsi ini.
4. Ayah Iwan Kuswandi, Ibu Fenti Hamidah dan seluruh keluarga besar atas kasih sayang yang diberikan
5. Mbak Lita, Hari Putra Setiawan dan “Genetic Team” atas ketersediaannya menemani dan membantu dalam proses pengerjaan.
6. Keluarga besar Ilmu dan Teknologi Kelautan, ITK 48, atas dukungan dan semangat yang diberikan selama penyelesaian skripsi ini.
Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 2
METODE 2
Waktu dan Tempat 2
Bahan 4 Alat 4
Pengumpulan Sampel 4
Ekstraksi 5
Amplifikasi DNA(PCR) 5
Elektroforesis 6
Sekuensing DNA 6
Prosedur Analisis Data 6
Rekonstruksi Filogenetik dan Analisis Keragaman Genetik 6 Hubungan Karakteristik Habitat dengan Spesies Nudibranch 7
HASIL DAN PEMBAHASAN 8
Rekonstruksi Filogenetik 8
Struktur genetik 12
Keragaman Genetik 14
Karakteristik Habitat 16
SIMPULAN DAN SARAN 19
Simpulan 19 Saran 19
DAFTAR PUSTAKA 20
LAMPIRAN 22
DAFTAR TABEL
1 Pengumpulan sampel penelitian 3
2 Matriks komposisi nukleotida per spesies 12 3 Matriks komposisi nukleotida per individu 12
4 Matriks probabilitas nukleotida 13
5 Deskripsi statistik keragaman genetik nudibranch 14
6 Deskripsi habitat famili nudibranch 16
7 Deskripsi habitat spesies nudibranch 17
DAFTAR GAMBAR
1 Peta lokasi pengumpulan sampel 4
2 Pohon filogenetik 8
3 Peta persebaran spesies nudibranch di Nusa Penida (Bali) 9 4 Peta persebaran spesies nudibranch di Teluk Jakarta 10 5 Peta persebaran spesies nudibranch di Luwuk (Sulawesi Timur) 10 6 Peta persebaran spesies nudibranch di Papua 11
7 Haplotype network spesies nudibranch 14
8 Plot simetrik hubungan antara habitat dengan spesies dalam famili
nudibranch 17 9 Plot simetrik hubungan antara habitat dengan spesies nudibranch 18
DAFTAR LAMPIRAN
1 Komposisi Master Mix (MM) 23
2 Gambar spesies nudibranch dan tipe substrat 24
PENDAHULUAN
Latar Belakang
Nudibranchia merupakan ordo terbesar dari Opisthobranch dan terdiri dari 3000 spesies yang telah teridentifikasi. Nudibranchia berasal dari “Nudus” berarti telanjang dan “Branchia” yang berarti insang. Istilah jika kedua kata ini
digabungkan berarti insang telanjang. Istilah ini mengarah pada organ respirasi eksternal yang terdapat di nudibranchia (Behrens 2005). Bentuk yang beragam, warna, pola tubuh dan sisi ekologi yang terdapat di Nudibranchia menarik untuk dipelajari. Sebagian besar nudibranchia memiliki warna cukup menarik yang digunakan dalam sistem pertahanan diri. Spesies Nudibranchia yang telah teridentifikasi di kawasan perairan Indonesia sebanyak 59 spesies dan terdiri dari 15 famili (Dayrat 2006). Ordo Nudibranchia terdiri dari empat subordo, 66 famili, dan 116 genus. Keempat subordo tersebut adalah Dendronotacea (10 famili, 14 genus), Doridacea (26 famili, 56 genus), Aeolidacea (21 famili, 35 genus), dan Arminacea (9 famili, 11 genus) (Brunckhorst 1993).
Nudibranch hanya terdiri dari kulit, otot dan organ tubuh karena telah menanggalkan cangkang pada jutaan tahun yang lalu (Debelius 2004). Nudibranch hidup pada perairan dangkal, terumbu karang, hingga dasar laut yang gelap dengan kedalaman lebih dari satu kilometer dan dapat dijumpai di berbagai tipe habitat mulai dari substrat bersedimen lumpur lunak sampai substrat keras berbatu (Aiken 2003).
Menurut Lubis (2006), Kepulauan Indonesia merupakan gabungan dari 5 pulau utama dan sekitar 30 kelompok kepulauan, lokasi strategis dari kepulauan yang sangat luas ini yaitu diantara lautan pasifik di timur, lautan Hindia di barat, daratan Asia di Utara dan daratan Australia di selatan, mempengaruhi sirkulasi global baik atmosfir maupun laut. Indonesia bagian barat merupakan bagian dari Benua Asia, Indonesia bagian timur merupakan bagian dari Benua Australia, sedangkan Indonesia bagian tengah merupakan peralihan yang disebut daerah Wallace. Batas kondisi geografis Indonesia yang sangat luas dan kompleks tersebut masih perlu eksplorasi di DNA barcoding, untuk mengidentifikasi spesies sehingga dapat melihat keragaman genetik.
Penentuan spesies siput laut (Nudibranch) masih banyak dilakukan dengan pengamatan morfologi, padahal dalam satu spesies Nudibranch yang sama bisa saja memiliki beberapa keanekaragaman genetik yang berbeda. Teknik yang digunakan dalam penelitian ini, yaitu analisis sekuensing DNA dengan menggunakan marka mitokondria. Penggunaan DNA sebagai ciri suatu spesies yang memiliki beberapa kelebihan, yaitu lebih termostabil dari pada protein, lebih sensitif, tidak dipengaruhi oleh lingkungan dan faktor pertumbuhan serta hampir semua jaringan dapat digunakan sebagai sumber material genetik (Teletchea et al. 2005). Selain pengukuran dengan genetika memiliki kelebihan yang akurat, penggunaan genetika molekular untuk menentukan suatu spesies mulai berkembang dan masih jarang dilakukan.
2
CO1. Marka CO1 memiliki tingkat keragaman yang tinggi sehingga mampu mengidentifikasi spesies dengan tingkat taksonomi yang luas. Komponen-komponen dalam DNA barcoding yaitu spesimen, laboratorium, basis data, dan analisis data. Kegiatan laboratorium dan analisis data terdiri dari ekstraksi DNA, amplifikasi (PCR), elektroforesis, sekuensing, koreksi dan pengolahan data (Madduppa 2014).
Keanekaragaman Nudibranchia dapat diketahui dengan melihat faktor-faktor yang mempengaruhi keberadaannya di lautan antara lain perbedaan habitat, seperti tutupan karang, ketersediaan dan jenis makanan (Jensen 2005). Ketiga hal ini berkaitan karena diketahui bahwa banyak Nudibranchia makan dan hidup dalam asosiasi yang dekat dengan spesies karang (Godfrey 2001). Nudibranchia pada umumnya memakan algae, sponge, karang keras dan lunak, bryozoans dan hydroids (Allen dan Steene 1999).
Bell dan Galzin (1984) menyebutkan dalam penelitiannya bahwa, terdapat hubungan langsung antara tutupan karang hidup dan keanekaragaman spesies organisme bentik. Diduga bahwa ditempat dimana tutupan karang baik, maka makin banyak jumlah nudibranchia dan makin baik keanekaragaman spesiesnya. Terumbu karang di Indonesia yang mencapai 51 persen dari luas 99.513 km2 terumbu karang di Asia Tenggara, saat ini hampir 85% terancam rusak, sedangkan 50% nya mendapat ancaman kerusakan yang tinggi (Indraswati 2006). Oleh karena itu spesies nudibranch terancam mengalami penurunan jumlah dan terjadi penurunan keanekaragaman spesiesnya.
Tujuan Penelitian
Penelitian ini bertujuan untuk mengidentifikasi serta menganalisis keragaman genetik nudibranch di Indonesia dengan menggunakan metode molekuler, menganalisis karakteristik habitat dan hubungannya dengan spesies nudibranch di Indonesia.
METODE
Waktu dan Tempat
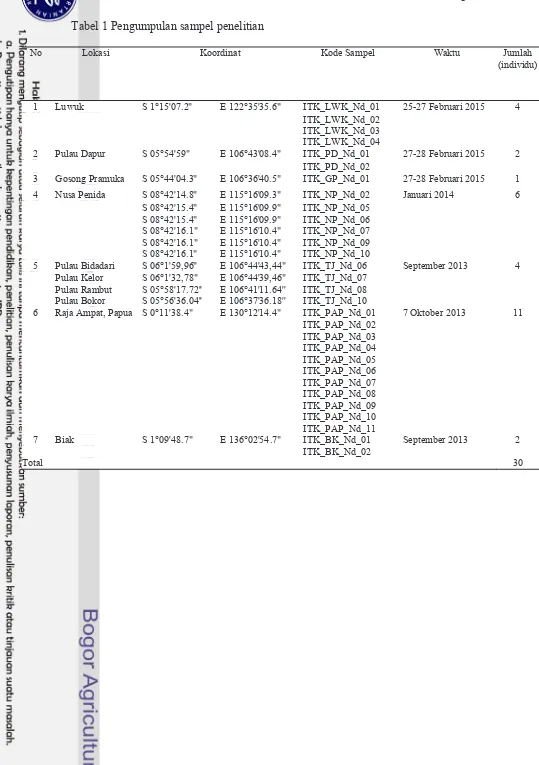
3 Tabel 1 Pengumpulan sampel penelitian
No Lokasi Koordinat Kode Sampel Waktu Jumlah
(individu)
1 Luwuk S 1°15'07.2" E 122°35'35.6" ITK_LWK_Nd_01 25-27 Februari 2015 4
ITK_LWK_Nd_02 ITK_LWK_Nd_03 ITK_LWK_Nd_04
2 Pulau Dapur S 05°54'59" E 106°43'08.4" ITK_PD_Nd_01 27-28 Februari 2015 2
ITK_PD_Nd_02
3 Gosong Pramuka S 05°44'04.3" E 106°36'40.5" ITK_GP_Nd_01 27-28 Februari 2015 1
4 Nusa Penida S 08°42'14.8" E 115°16'09.3" ITK_NP_Nd_02 Januari 2014 6
S 08°42'15.4" E 115°16'09.9" ITK_NP_Nd_05 S 08°42'15.4" E 115°16'09.9" ITK_NP_Nd_06 S 08°42'16.1" E 115°16'10.4" ITK_NP_Nd_07 S 08°42'16.1" E 115°16'10.4" ITK_NP_Nd_09 S 08°42'16.1" E 115°16'10.4" ITK_NP_Nd_10
5 Pulau Bidadari S 06°1'59,96" E 106°44'43,44" ITK_TJ_Nd_06 September 2013 4
Pulau Kelor S 06°1'32,78" E 106°44'39,46" ITK_TJ_Nd_07
Pulau Rambut S 05°58'17.72'' E 106°41'11.64'' ITK_TJ_Nd_08
Pulau Bokor S 05°56'36.04'' E 106°37'36.18'' ITK_TJ_Nd_10
6 Raja Ampat, Papua S 0°11'38.4" E 130°12'14.4" ITK_PAP_Nd_01 7 Oktober 2013 11
ITK_PAP_Nd_02
7 Biak S 1°09'48.7" E 136°02'54.7" ITK_BK_Nd_01 September 2013 2
ITK_BK_Nd_02
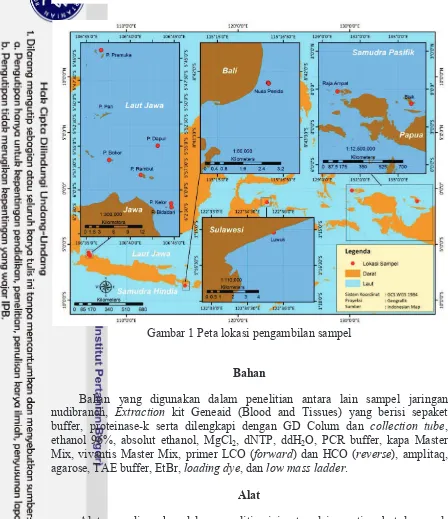
4
Gambar 1 Peta lokasi pengambilan sampel
Bahan
Bahan yang digunakan dalam penelitian antara lain sampel jaringan nudibranch, Extraction kit Geneaid (Blood and Tissues) yang berisi sepaket buffer, proteinase-k serta dilengkapi dengan GD Colum dan collection tube, ethanol 96%, absolut ethanol, MgCl2, dNTP, ddH2O, PCR buffer, kapa Master
Mix, vivantis Master Mix, primer LCO (forward) dan HCO (reverse), amplitaq, agarose, TAE buffer, EtBr, loading dye, dan low mass ladder.
Alat
Alat yang digunakan dalam penelitian ini antara lain gunting, botol sampel, timbangan digital, alat tulis, bunsen, cawan petri, pinset, tray, sarung tagan karet, vortex, heating block, alat sentrifuge, microtube, micropippet, pippet tips, thermo cycler, kalkulator, gelas ukur, labu erlenmeyer, microwave, dan UV transluminator.
Pengumpulan Sampel
5 merasakan tekstur setelah itu pada kertas sabak dan kemudian difoto. Analisis substrat berikutnya melalui foto yang telah diambil dan disesuaikan dengan catatan tipe substrat. Seluruh sampel merupakan koleksi Laboratorium Biosistematika dan Biodivesitas Kelautan, Ilmu dan Teknologi Kelautan, Fakultas Perikanan dan Ilmu Kelautan, IPB.
Ekstraksi
Tahap ekstraksi dilakukan dengan menggunakan dua metode yaitu, chelex 10% dan extraction kit Geneaid (Blood and Tissues). Metode ekstraksi menggunakan chelex 10% yaitu metode yang digunakan untuk menyimpan DNA kedalam raissin, sedangkan metode extraction kit Geneaid (Blood and Tissues) yaitu metode yang digunakan untuk menyaring DNA dengan menggunakan beberapa jenis buffer. Pengekstraksian metode chelex 10% dilakukan dengan cara pemanasan 105 °C selama 60 menit. Tahapan ini harus dalam keadaan steril untuk mencegah kontaminasi pada sampel.
Pada tahapan ekstraksi menggunakan kit, hal yang pertama dilakukan adalah mengambil sedikit bagian perut nudibranch dengan menggunakan pinset, kemudian sampel dikoyak agar sel mudah untuk lisis. Setelah dikoyak, sampel dimasukkan ke dalam tube berukuran 1.5 mL kemudian ditambahkan 200 μL GST Buffer dan 20 μL Proteinase-K. Campuran tersebut kemudian diinkubasi dengan menggunakan heatingblock pada suhu 60 oC selama 1 jam atau hingga sampel lisis. Setelah sampel lisis, sentrifuse selama 2 menit pada 14.000 rpm, pindahkan supernatan pada tube 1.5 mL baru. Tambahkan 200 μL GSB Buffer lalu vortex selama 10 detik. Tambahkan 200 μL absolut ethanol, segera vortex selama 10 detik. Tempatkan GD Column dalam 2 ml Collection Tube, dan pindahkan semua campuran ke dalam GD Column. Sentrifuse selama 1 menit pada 14,000 rpm. Buang 2 mLCollection Tube beserta cairan di dalamnya. Tempatkan GD Column dalam 2 mL Collection Tube baru. Tambahkan 400 μLW1 Buffer ke dalam GD Column kemudian sentrifus selama 30 detik pada 14,000 rpm. Buang cairan dalam Collection Tube dan tempatkan kembali pada GD Column. Tambah 600 μL Wash Buffer ke GD Column. Sentrifus selama 30 detik pada 14,000 rpm.Buang cairan pada Collection Tube dan tempatkan kembali. Sentrifus selama 3 menit pada 14.000 rpm untuk mengeringkan GD Collumn. Pindahkan GD Colum kering ke tube berukuran 1.5 mL. Tambah 100 μL Elution Buffer dan 100 μL ddH2O,
diamkan selama 3 menit kemudian sentrifus selama 30 detik pada 14.000 rpm untuk elusi DNA yang sudah dimurnikan.
Amplifikasi DNA(PCR)
6
dilakukan dengan menggunakan alat thermocycler yang diprogram untuk melakukan 35 siklus. Setiap siklusnya terdiri dari proses penempelan utas ganda (pre denaturation) pada suhu 95 °C selama 3 menit, denaturation pada suhu 94 °C selama 45 detik, annealling pada suhu 45 °C selama 45 detik dan extention pada suhu 72 °C selama 2 menit. Kemudian dilanjutkan dengan final elongated pada suhu 72 °C selama 10 menit.
Elektroforesis
Elektroforesis adalah metode untuk memisahkan senyawa kimia berdasarkan laju pergerakan molekul dalam aliran listrik (Madduppa 2014). Metode ini biasanya digunakan untuk tujuan analitik, tetapi dapat digunakan sebagai teknik preparasi untuk memurnikan molekul sebelum menggunakan metode lain untuk karakterisasi lanjut seperti spektrometri massa, PCR, kloning, pengurutan DNA. Elektroforesis ini bertujuan untuk mengetahui ada tidaknya DNA dalam produk PCR kita. Sebelum melakukan elektroforesis, terlebih dahulu dibuat media elektroforesis yaitu agarosa 1% (agarose 0.5 g dan 50 mL TAE Buffer) dengan 4 μL Etidium Bromida (EtBr) sebagai pewarna. Langkah selanjutnya adalah mencampurkan 3 μLsampel hasil PCR dengan 1 μL loading dye, campuran tersebut kemudian dimasukkan ke dalam sumur agarosa. Elektroforesis menggunakan mesin elektroforesis tegangan 220 V dan arus 400 mA dengan waktu 25 menit. Panjang untaian basa DNA dapat diukur dengan menggunakan 4μL Lowmass ladder yang dimasukkan pada sumur pertama pada agarosa. Hasil elektroforesis berupa pita-pita yang dapat dilihat dengan menggunakan alat UV transluminator.
Sekuensing DNA
Siklus pengurutan Nukleotida (Sequencing DNA) adalah metode untuk menentukan urutan nukleotida yang terdapat dalam DNA. Urutan DNA berhubungan dengan informasi genetik turunan dalam nukleus (inti), plasmid, mitokondria, dan kloroplas yang membentuk dasar pengembangan semua makhluk hidup. Prinsip sequencing menggunakan metode PCR sebagai pijakkannya. Hasil berupa fragmen DNA dengan panjang yang bervariasi. Produk PCR dikirimkan ke sequencing facilty UC Berkeley, Dept. of Moleculler and Cell Biology Sequencing Facility, USA dengan menggunakan mesin sequencer ABI 1377.
Prosedur Analisis Data
Rekonstruksi Filogenetik dan Analisis Keragaman Genetik
Rekonstruksi filogenetik dan analisis keragaman genetik dilakukan dengan cara mengedit data hasil sekuensing dengan menggunakan perangkat lunak MEGA 6.0 (Molecular Evolutionary Genetic Analysis) untuk pembacaan urutan nukleotida dan penjajaran (aligment) menggunakan ClustalW pada program tersebut untuk melihat adanya keragaman nukleotida (Tamura et al. 2007).
7 (http://blast.ncbi.nlm.nih.-gov). Analisis DNA barcoding dilakukan dengan membuat pohon filogeni berdasarkan hasil penjajaran nukleotida yang telah dicocokkan dengan data pada GeneBank. Pohon filogeni dibuat dengan menjajarkan terlebih dahulu urutan nukleotida menggunakan piranti lunak Clustal W2, lalu dihitung jarak genetik dari setiap sampel dan pembuatan pohon filogeni dengan metode pengkelasan Neigbour-joining (NJ).
Rekonstruksi filogenetik bertujuan untuk mengetahui kekerabatan melalui komposisi nukleotida yang digambarkan dalam pohon filogenetik.Pembuatan Pohon filogenetik menggunakan metode Neighbor Joining Tree dengan jumlah Bootstrap sebanyak 1000. Nilai Bootstrap menggambarkan ulangan yang dilakukan oleh perangkat lunak MEGA 6.0 dalam merekonstruksi pohon filogenetik tersebut. Metode Neighbor Joining Tree dapat menganalisa jarak genetik antar spesies maupun intra spesies. Dalam pembuatan pohon filogenetik perlu dimasukkan outgroup sebagai perbandingan. Dalam penelitian digunakan Stichopus ocellatus sebagai outgroup. Analisis Keragaman genetik menggunakan software dnasp dan arlequin yang menghasilkan nilai keragaman genetik. Nei (1987) nilai keragaman genetik berkisar dari 0.1 – 0.4 masuk dalam kategori rendah, sementara nilai 0.5 – 0.7 tergolong dalam kategori sedang, dan 0.8 – 1.0 merupakan kategori tinggi.
Hubungan Karakteristik Habitat dengan Sepesies Nudibranch
8
Phyllidia ocellata gi614469624 phyllidia ocellata
62-LWKND01 IPB LCO
Phyllidia elegans gi4741707 Phyllidia elegans
58-NPND06 IPB LCO
Phyllidia varicosa gi614469622 Phyllidia varicosa
64-LWKND03 IPB LCO
Phyllidia coelestis gi614469620 Phyllidia coelestis
66-PDND02 IPB LCO
Taringa caudata gi595650141 Taringa caudata
69-TJND06 IPB LCO
Doriprismatica atromarginata gi389941677 Doriprismatica atromarginata
70-TJND07 IPB LCO
Mexichromis multituberculata gi389941763 Mexichromis multituberculata
72-TJND10 IPB LCO
Risbecia tryoni gi156047644 Risbecia tryoni
71-TJND08 IPB LCO
Chromodoris striatella gi614469614 Chromodoris striatella
75-PAPND01 IPB LCO
Chromodoris annae gi389941607 Chromodoris annae
77-PAPND03 IPB LCO
Elysia cf. marginata 3 JV-2013 gi469735901 Elysia cf. marginata
Plakobranchus sp. 1 JV-2013 gi469735971
78-PAPND04 IPB LCO
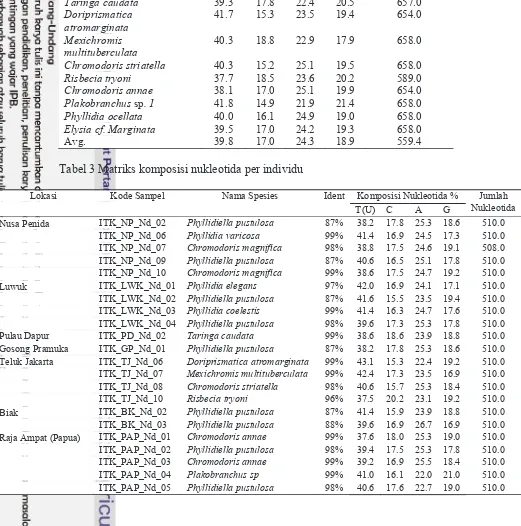
Hasil BLAST yang diinterpretasikan ke dalam pohon filogenetik seperti dibawah ini.
Gambar 2 Rekonstruksi pohon filogeni DNA nudibranch
9
Data urutan basa yang sudah di BLAST didapatkan 14 spesies nudibranch dari 30 sampel dengan tujuh lokasi di Indonesia. Gambar spesies nudibranch serta habitat nudibranch yang ditemukan dapat dilihat pada Lampiran 2. Gambar 2 merupakan hasil pohon filogenetik nudibranch yang terdiri dari 15 clade besar, 14 clade merupakan spesies nudibranch dan 1 clade Stichopus ocellata yang merupakan outgroup. Setiap clade yang terbentuk menandakan perbedaan spesies. Spesies yang didapatkan dari hasil BLAST adalah Phyllidiella pustulosa, Phyllidia ocellata, Phyllidia elegans, Phyllidia varicosa, Phyllidia coelestis, Taringa caudata, Doriprismatica atromarginata, Mexichromis multituberculata, Risbecia tryoni, Chromodoris striatella, Chromodoris annae, Chromodoris magnifica, Elysia cf. Marginata, dan Plakobranchus sp. Keragaman genetik dapat terjadi tidak hanya pada intraspesies namun juga dapat terjadi pada interspesies. Pada setiap clade besar masih terdapat clade kecil yang menandakan adanya perbedaan komposisi basa nukleotida antar individu. Hal ini dapat terjadi karena tiap-tiap individu berasal dari populasi yang berbeda.
10
Gambar 4 Peta persebaran spesies nudibranch di Teluk Jakarta
11
Gambar 6 Peta persebaran spesies nudibranch di Papua
12
Struktur Genetik
Urutan rantai basa nukleotida menggunkan locus CO1 menghasilkan 645-689 bp, Tabel 2 dan Tabel 3 menunjukan Hasil analisis komposisi basa nukleotida dari individu nudibranch.
Tabel 2 Matriks komposisi nukleotida per spesies
Nama Spesies Komposisi Nukleotida % Jumlah
Nukleotida
Tabel 3 Matriks komposisi nukleotida per individu
Lokasi Kode Sampel Nama Spesies Ident Komposisi Nukleotida % Jumlah
Nukleotida
T(U) C A G
Nusa Penida ITK_NP_Nd_02 Phyllidiella pustulosa 87% 38.2 17.8 25.3 18.6 510.0
ITK_NP_Nd_06 Phyllidia varicosa 99% 41.4 16.9 24.5 17.3 510.0
ITK_NP_Nd_07 Chromodoris magnifica 98% 38.8 17.5 24.6 19.1 508.0
ITK_NP_Nd_09 Phyllidiella pustulosa 87% 40.6 16.5 25.1 17.8 510.0
ITK_NP_Nd_10 Chromodoris magnifica 99% 38.6 17.5 24.7 19.2 510.0
Luwuk ITK_LWK_Nd_01 Phyllidia elegans 97% 42.0 16.9 24.1 17.1 510.0
ITK_LWK_Nd_02 Phyllidiella pustulosa 87% 41.6 15.5 23.5 19.4 510.0
ITK_LWK_Nd_03 Phyllidia coelestis 99% 41.4 16.3 24.7 17.6 510.0
ITK_LWK_Nd_04 Phyllidiella pustulosa 98% 39.6 17.3 25.3 17.8 510.0
Pulau Dapur ITK_PD_Nd_02 Taringa caudata 99% 38.6 18.6 23.9 18.8 510.0
Gosong Pramuka ITK_GP_Nd_01 Phyllidiella pustulosa 87% 38.2 17.8 25.3 18.6 510.0
Teluk Jakarta ITK_TJ_Nd_06 Doriprismatica atromarginata 99% 43.1 15.3 22.4 19.2 510.0
ITK_TJ_Nd_07 Mexichromis multituberculata 99% 42.4 17.3 23.5 16.9 510.0
ITK_TJ_Nd_08 Chromodoris striatella 98% 40.6 15.7 25.3 18.4 510.0
ITK_TJ_Nd_10 Risbecia tryoni 96% 37.5 20.2 23.1 19.2 510.0
Biak ITK_BK_Nd_02 Phyllidiella pustulosa 87% 41.4 15.9 23.9 18.8 510.0
ITK_BK_Nd_03 Phyllidiella pustulosa 88% 39.6 16.9 26.7 16.9 510.0
Raja Ampat (Papua) ITK_PAP_Nd_01 Chromodoris annae 99% 37.6 18.0 25.3 19.0 510.0
ITK_PAP_Nd_02 Phyllidiella pustulosa 98% 39.4 17.5 25.3 17.8 510.0
ITK_PAP_Nd_03 Chromodoris annae 99% 39.2 16.9 25.5 18.4 510.0
ITK_PAP_Nd_04 Plakobranchus sp 99% 41.0 16.1 22.0 21.0 510.0
13
ITK_PAP_Nd_06 Plakobranchus sp. 99% 39.2 17.6 25.7 17.5 510.0
ITK_PAP_Nd_07 Elysia cf marginata 93% 39.6 17.3 25.5 17.6 510.0
ITK_PAP_Nd_08 Chromodoris annae 99% 41.0 16.1 22.0 21.0 510.0
ITK_PAP_Nd_10 Phyllidia ocellata 88% 39.4 17.1 23.1 20.4 510.0
ITK_PAP_Nd_11 Phyllidiella pustulosa 98% 39.6 16.5 25.7 18.2 510.0
Avg. 40.0 17.0 24.4 18.6 509.9
DNA makhluk hidup terdapat kuantitas yang sama antara basa Adenin (A) dengan Timin (T), dan Guanin (G) dengan Sitosin (C). Komposisi basa inilah yang membedakan antar spesies organisme (Jusuf 2001). Tabel 2 menunjukkan bahwa komposisi A dan T pada tiap spesies nudibranch lebih banyak dibandingkan C dan G dengan rata-rata pasang basa 559.4. Tabel 3 menunjukkan hal yang sama bahwa komposisi A dan T pada setiap individu nudibranch lebih banyak dibandingkan C dan G dengan rata-rata pasang basa 509.9. Jumlah A dan T yang lebih banyak dibandingkan C dan G berkaitan dengan ikatan hidrogennya. Ikatan hidrogen merupakan ikatan lemah yang mudah lepas dan mudah berikatan kembali. Sifat ini yang memudahkan DNA untuk melakukan replikasi. Jarak genetik dapat terlihat dengan jelas dalam tabel jarak genetik antar spesies (Lampiran 3).
Pada tabel jarak genetik, jarak genetik terkecil ditunjukkan antara Chromodoris striatella dengan Chromodoris magnifica sebesar 0.098 yang artinya dalam 100 urutan basa terdapat 9.8 basa yang berbeda, sedangkan untuk jarak genetik dengan nilai terbesar ditunjukkan antara outgroup dengan Mexichromis multituberculata sebesar 0.455 yang artinya dalam 100 urutan basa terdapat 45.5 basa yang berbeda. Semakin kecil nilai matriksnya, menunjukkan bahwa kekerabatan setiap spesies semakin dekat dan dibuktikan oleh bentuk pohon filogenik Gambar 2 dan Haplotype network Gambar 7.
Tabel 4 Matriks probabilitas substitusi nukleotida
From\To Nukleotida
Keterangan : Transisi = cetak tebal, Transversi = cetak normal
14
Burleugh, 2001). Salah satu jenis mutasi adalah mutasi substitusi yang terdiri dari transisi dan transversi. Transisi merupakan perubahan antara A dengan T (purin) atau C dengan G (pirimidin). Transversi perupakan perubahan antara purin dengan pirimidin (Karmana 2009). Kemungkinan mutasi substitusi jenis transisi lebih banyak terjadi dibandingkan dengan transversi, seperti terlihat pada Tabel 4 (Nei and Kumar 2000). Hal ini terjadi karena molekul yang mempunyai kemiripan struktur lebih mudah tersubstitusi seperti pada transisi yang terjadi antara purin dengan purin dan pirimidin dengan pirimidin (Wijana dan Mahardika 2010).
Keragaman Genetik
Nilai keragaman genetik memberikan informasi untuk beradaptasi terhadap perubahan lingkungan dan iklim serta penyakit (Lande 1988). Berikut adalah nilai keragaman genetik nudibranch per lokasi penelitian.
Tabel 5 Keragaman genetik spesies nudibranch
Keterangan: NP (Nusa Penida), LWK (Luwuk), PD (Pulau Dapur), GP (Gosong Pramuka), TJ (Teluk Jakarta), BK (Biak), PAP (Papua), n (Jumlah sampel), h (Jumlah haplotype), Hd (Haplotype diversity)
Spesies NP LWK PD GP TJ BK PAP
15
Spesies Phyllidiella pustulosa ditemukan pada lima lokasi penelitian dengan nilai keragaman haplotipe yang berbeda-beda. Phyllidiella pustulosa di Nusa Penida memiliki nilai keragaman haplotipe (Hd) 0.5, Luwuk memiliki nilai keragaman haplotipe 0.5, Gosong Pramuka nilai keragaman haplotipe 0, Biak nilai keragaman haplotipe 0.5, dan Phyllidiella pustulosa Papua memiliki nilai keragaman haplotipe 0.27. Menurut Nei (1987) nilai keragaman haplotipe Phyllidiella pustulosa di Nusa Penida, Luwuk, dan Biak termasuk kedalam kategori sedang, sedangkan Phyllidiella pustulosa Papua termasuk kedalam kategori rendah, nilai keragaman haplotipe pada lokasi Gosong pramuka 0 karena individu yang didapatkan hanya satu dan tidak mewakili populasi. Phyllidia ocellata hanya ditemukan pada lokasi Papua dengan jumlah sampel sebanyak satu sampel dengan nilai keragaman haplotipe 0. Phyllidia elegans memiliki nilai keragaman haplotipe 0 yang hanya ditemukan pada satu lokasi saja yaitu, Luwuk. Phyllidia varicosa hanya ditemukan sebanyak satu individu pada lokasi Nusa Penida dengan nilai keragaman haplotipe 0. Taringa caudata memiliki nilai keragaman haplotipe 0 pada lokasi Pulau Dapur. Teluk Jakarta ditemukan sebanyak empat spesies nudibranch yaitu, Doriprismatica atromarginata, Mexichromis multituberculata, Risbecia tyroni, dan Chromodoris striatella dengan nilai keragaman haplotipe untuk masing-masing spesies adalah 0. Chromodoris annae hanya ditemukan pada lokasi penelitian Papua dengan nilai haplotipe 0.3 dan termasuk kedalam kategori rendah. Chromodoris magnifica memiliki nilai keragaman haplotipe 0.5 pada lokasi Nusa Penida dan termasuk kedalam kategori sedang. Papua juga ditemukan Elysia cf. marginata dan Plakobranchus sp. dengan nilai keragaman haplotipe 0 dan 0.5 serta termasuk kedalam kategori rendah dan sedang. Nuryanto (2009) menyebutkan bahwa keragaman genetik yang tinggi mencerminkan besarnya ukuran populasi, sedangkan penurunan ukuran populasi akan mengurangi keragaman genetik. Nilai keragaman genetik yang rendah menandakan kecilnya ukuran populasi dan akan mengurangi keragaman genetik.
Keragaman genetik dalam populasi yang tinggi didasarkan pada asumsi bahwa tingkat evolusi dalam lingkungan yang berubah dibatasi oleh variasi genetik (Soule and Simberloff 1986). Keragaman genetik penting bagi kelangsungan hidup spesies karena spesie-spesies yang memiliki keragaman genetik kecil mungkin lebih rentan terhadap penyakit atau efek dari perubahan lingkungan. Peningkatan keragaman genetik menghasilkan keturunan dengan berbagai karakteristik yang dapat menjamin untuk menahan perubahan lingkungan dan mengurangi kemungkinan kerusakan gen (seperti penyakit) muncul dalam populasi. Pada penelitian ini tidak ada yang masuk kedalam kategori tinggi dikarenakan jumlah sampel yang ditemukan belum terlalu banyak sehingga belum mewakili populasi.
16
dengan spesies nudibranch yang lainnya. Chromodoris annae dan Chromodoris magnifica merupakan satu keturunan dengan jumlah haplotipe pada spesies Chromodoris annae lebih banyak dibandingkan dengan Chromodoris magnifica. Doriprismatica atromarginata dan Mexichromis multituberculata merupakan satu keturunan dengan genus yang sama dengan spesies yang berbeda. Sedangakan Plakobranchus sp dan Taringa caudata memiliki jaringan haplotipe yang sangat jauh, hal ini dikarenakan keduanya tidak termasuk kedalam famili, genus, dan spesies yang sama.
Konektivitas genetik yang terjadi akan mempengaruhi tinggi rendahnya keragaman genetik pada suatu populasi. Keragaman genetik suatu populasi akan meningkat jika terdapat suatu masukan genetik dari populasi lain atau biasa disebut dengan migrasi genetik. Migrasi yang besar akan menyebabkan terjadinya perkawinan silang dan percampuran gen antar populasi yang berbeda, sehingga akan diperoleh variasi gen yang berbeda-beda (Kusuma 2014).
Karakteristik Habitat
Setiap spesies nudibranch memiliki hubungan dengan habitatnya sebagai tempat tinggal, mencari makan, dan kawin. Pada penelitian ini dilakukan Correspondence Analysis (CA) antara karakteristik habitat dengan spesies nudibranch untuk melihat hubungan antar keduanya. Dibawah ini merupakan tabel deskripsi habitat famili nudibranch:
Tabel 6 Deskripsi habitat famili nudibranch
Famili
Habitat
Terumbu Karang Rubbel Substrat Berpasir Pasir Berbatu
Phyllidiidae 7 2 2 2
Chromodorididae 2 4 0 3
Discodorididae 1 0 0 0
17
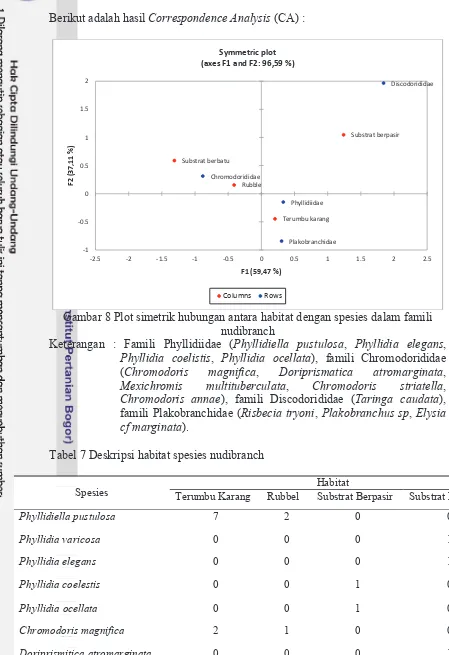
Berikut adalah hasil Correspondence Analysis (CA) :
Gambar 8 Plot simetrik hubungan antara habitat dengan spesies dalam famili nudibranch
Keterangan : Famili Phyllidiidae (Phyllidiella pustulosa, Phyllidia elegans, Phyllidia coelistis, Phyllidia ocellata), famili Chromodorididae (Chromodoris magnifica, Doriprismatica atromarginata, Mexichromis multituberculata, Chromodoris striatella, Chromodoris annae), famili Discodorididae (Taringa caudata), famili Plakobranchidae (Risbecia tryoni, Plakobranchus sp, Elysia cf marginata).
Tabel 7 Deskripsi habitat spesies nudibranch
Spesies Terumbu Karang Rubbel Habitat Substrat Berpasir Substrat Berbatu
18
Berikut adalah hasil Correspondence Analysis (CA) :
Gambar 9 Plot asimetrik hubungan antara habitat dengan spesies nudibranch Nudibranch dapat hidup di perairan dangkal, terumbu karang, hingga dasar laut yang gelap dengan kedalaman lebih dari satu kilometer dan dapatdijumpai di berbagai tipe habitat mulai dari substrat bersedimen lumpur lunak sampai substrat keras berbatu (Aiken 2003). Habitat nudibranch erat kaitannya dengan makanan yang dibutuhkannya. Berbagai jenis nudibranch pada umumnya memilih jenis makanan yang spesifik (Todd 1981). Gambar 8 dan 9 merupakan plot hubungan dua kategori peubah hasil yang dapat dilihat pada dari analisis korespondensi. Berdasarkan gambar 8 dan 9 dengan total informasi 96.59% menunjukkan bahwa habitat berhubungan erat dengan spesies. Tabel 6 memperlihatkan spesies nudibranch yang termasuk kedalam famili Phyllidiidae habitatnya adalah terumbu karang dan spesies dalam famili Chromodorididae banyak mendiami rubbel sebagai habitatnya. Bell dan Galzin (1984) menyebutkan dalam penelitiannya bahwa, terdapat hubungan langsung antara tutupan karang hidup dan keanekaragaman spesies organisme bentik. Diduga bahwa ditempat dimana tutupan karang baik, maka makin banyak jumlah nudibranchia dan makin baik keanekaragaman spesiesnya. Hal ini berbanding lurus terhadap spesies dalam
Terumbu karang
Chromodoris magnifica Doriprismatica atromarginata Plakobranchus sp Elysia cf marginata
19 famili Phyllidiidae lebih banyak ditemukan pada karang hidup dibandingakan dengan spesies nudibranch dalam famili Chromodorididae.
Jumlah individu nudibranch dari famili Phyllidiidae ditemukan lebih banyak dibandingakan dengan famili nudibranch lainnya (Tabel 7). Hal ini berbanding lurus dengan penelitian sebelumnya Yasman (2002) memaparkan mengenai distribusi vertikal siput anggota ordo Nudibranchia pada ekosistem terumbu karang di Kepulauan Seribu. Hasil observasinya menunjukkan bahwa spesies nudibranch dari famili Phyllidiidae yang palin sering ditemui dan paling banyak jumlah individunya, serta tersebar di berbagai kedalaman dari 6 meter hingga 24 meter.
SIMPULAN DAN SARAN
Simpulan
Identifikasi spesies nudibranch dengan metode molekuler menghasilkan pohon filogenetik yang menerangkan hubungan kekerabatan antar spesies. Analisis keragaman genetik menggunakan marka mitokondria menghasilkan empat belas spesies nudibranch dari tiga puluh sampel yang teridentifikasi dengan kesamaan (homologi) sebesar 87% - 99%. Panjang basa rata-rata per spesies yang didapatkan adalah 559.4 dan panjang basa rata-rata per individu adalah 509.9. Keragaman genetik untuk setiap spesies nudibranch berbeda-beda dengan kategori rendah hingga sedang, serta memiliki nilai keragaman genetik 0 hingga 0.5. Nilai keragaman genetik rendah terdapat pada spesies Phyllidiella pustulosa di Gosong pramuka, Phyllidiella pustulosa di Papua, Phyllidia ocellata di Papua, Phyllidia elegans di Luwuk, Phyllidia varicosa di Nusa Penida, Phyllidia coelestis di Luwuk, Taringa caudata di Pulau Dapur, Doriprismatica atromarginata, Mexichromis multituberculata, Risbecia tyroni, Chromodoris striatella di Teluk Jakarta, Chromodoris annae di Papua dan Elysia cf marginata di Papua. Nilai keragaman genetik sedang terdapat pada Phyllidiella pustulosa di Luwuk, Nusa Penida dan Biak, Chromodoris magnifica di Nusa Penida, serta Plakobranchus sp di Papua. Hubungan karakteristik habitat dengan spesies nudibranch didapatkan melalui Correspondence Analysis (CA) yang menjelaskan spesies nudibranch yang termasuk kedalam famili Phyllidiidae habitatnya adalah terumbu karang dan spesies dalam famili Chromodorididae banyak mendiami rubbel sebagai habitatnya.
Saran
20
DAFTAR PUSTAKA
Aiken RB. 2003. Some aspects of the life history of an intertidal population of the Nudibranch Dendronotus frondosus (Ascanius, 1774) (Opisthobranchia : Dendronotoidea) in the bayof fundy. Veliger 46 (2): 169-175.
Allen GR, Steene R. 1999. Indo-Pacific Coral Reef Guide. Singapore : Tropical Reef Research.
Behrens DW. 2005. “Nudibranch Behavior”. New World Publications, Inc. Jacksonville, FL. 176 pp.
Bell JD, Galzin R. 1984. “Influence of Coral Cover on Coral-Reef Fish Communities”. Marine Ecology Progress Series15: 265-274.
Bengen DG. 2000. Teknik Pengambilan Contoh dan Analisis Data Biofisik Sumberdaya Pesisir. Pusat Kajian Sumberdaya Pesisir dan Lautan Institut Pertanian Bogor. Bogor. 86 hal.
Brunckhorst DJ. 1993. The systematic and phylogeny of phyllidiid nudibranch (Doridoidea). Records of the Australian Museum Supplement 16:107 hlm. Dayrat B. 2006. A Taxonomic Revision Of Paradoris Sea Slugs (Mollusca:
Gastropoda: Nudibranchia: Doridina). Zoological journal of the linnaean society. 147:125-238.
Debelius H. 2004. Nudibranch and Sea Snails Indo-Pacific Field Guide. IKAN-Unterwasserarchiv, Frankfurt: 320 pp.
English S, Wilkinson C and Baker V. 1997. Survey Manual for Tropical Marine Resources. 2nd Edition. Australian Institute of Marine Science. Townsville. Australia.
Folmer O, Black M, Hoeh W, Lutz R and Vrijenhoek R. 1994. DNA Primers for Amplification of Mitochondrial Cytochrome C Oxidase Subunit I from Diverse Metazoan Invertebrates. Mol Mar Bio Biotech. 3: 294–299.
Freeland R. 2005. Molecular Ecology. England(GB): John Wiley&Sons. Ltd. Godfrey S. 2001. Factors Affecting Nudibranch Diversity in The Wakatobi
Marine National Park, URL : http//www.opwall.com/.../Invertebrates/Godfrey,%20S%20Factors%affect
ing%20nudibranch%20distribution.pdf.
Graur D, Hsiung Li. 2000. Fundamentals of Molecular Evolution. Sunder-land, MA, USA : Sinauer Associates.
Indraswati E. 2006. INCL Indonesia: Indonesian Nature Conservation news Letter 9-44b.
Jensen KR. 2005. “ Distribution and Zoogeographic affinities of The Nudibranch Fauna (Mollusca, Ophistobranchia, Nudibranchia) of The Faroe Islands”. BIOFAR Project, DK-210.
Jusuf M. 2001. Genetika 1 : Struktur dan Ekspresi Gen. Jakarta (ID) : CV. Sagung Seto.
Karmana IW. 2009. Kajian evolusi berbasis urutan nukleotida. Gene C Swara. 3:75-81.
Kusuma AB. 2014. Konektivitas dan keragaman genetik pada karang lunak Sarcophyton trocheliophorum serta implikasi terhadap kawasan konservasi laut [Tesis]. Bogor (ID) Institut Pertanian Bogor.
Lande R. 1988. Genetic and Demography in Biological Conservation. Science. 241: 1455-1460
21 Lubis SM. 2006. Oseanografi Indonesia. Program Studi Oseanografi. ITB :
Bandung
Madduppa H. 2014. Bioekologi dan Biosistematika Ikan Terumbu. Bogor (ID) : IPB Press.
Mattjik A, Sumertajaya. 2011. Sidik Peubah Ganda. Bogor (ID): IPB Press
Nei M. 1987. Moleculer evolutionary genetics. Columbia University. Press. New York. 512 hal.
Nei M, Kumar S. 2000. Molecular Evolution and Phylogenetics. New York (GB) : Oxford University Press.
Nuryanto A & M. Kochzius. 2009. Highly restricted gene flow and deep evolutionary linieages in the giant clam Tridacna maxima. Coral Reefs, 28: 607-619.
Puterbaugh MN, Burleugh JG. 2001. Investigating Evolutionary Question Using Online Molecular Databases. American Biology Teacher. 63(6):422-431. Soule ME, Simberloff D. 1986. Conservation Biology on Evolutionary
Ecologycal Perspective. Biol Conserv. 35: 19.
Tamura K, Dudley J, Nei M, and Kumar S. 2007. MEGA: Molecular Evolutionary Genetic Analysis (MEGA) software version 4.0. Advance Access published May 7. Oxford University Press. Mol Bio 10. 1093/molbev/msm092.
Teletchea F, Celia M, and Catherine H. 2005. Food and forensic molecular identification : update and challenges. Trends in Biotecnology. 23(7):359-366.
Todd C. 1981. The ecology of Nudibranch Molluscs. Ocean Mar Biol. Ann. Rev. 19: 141-234.
Wijana IMS, Mahardika IGN. 2010. Struktur genetik dan filogeni Yellowfin Tuna (Thunnus albacares) berdasarkan sekuen DNA mitokondria Control Region sitokrom oksidase I pada diversitas zone biogeografi. Jurnal Bumi Lestari. 10(2): 270-274.
22
23 Lampiran 1 Master Mix (MM)
Komposisi Master Mix (MM) pada PCR Master mix ... tabung
STANDAR PROTOCOL ( 1μL DNA template)
MM 1 MM 2
ddH2O 5,5 9
10x Buffer PCR (PE-II) 1,5 1
dNTPs (8 mM) 2,5 ....
MgCl2 (25 mM) 2 ....
Primer 1 (10 mM) 1,25 ....
Primer 2 (10 mM) 1,25 ....
Amplitaq polymerase ( 5 unit/ μL) ... 0,125
Total 14 10,125
Komposisi Kapa Master Mix Master mix ….. tabung
STANDAR PROTOCOL ( 1μL DNA template)
MM
Kapa MM 12,5 μL
ddH2O 12,9 μL
HCO 1,25 μL
LCO 1,25 μL
Komposisi 2x Taq Master Mix (Vivantis) Master mix ….. tabung
STANDAR PROTOCOL ( 1μL DNA template)
MM
2x Taq Master Mix 25 μL
ddH2O 5,5 μL
HCO 1,25 μL
24
Lampiran 2 Gambar spesies nudibranch dan tipe substrat Phyllidia coelestis; substrat berpasir
Choromodoris annae; Rubbel
25 Phyllidia varicosa; Substrat berbatu
Phyllidia elegans; Substrat berbatu
Phyllidia ocellata; Substrat berpasir
26
Plakobranchus sp; Terumbu karang
Chromodoris magnifica; Terumbu karang
27 Doriprismatica atromarginata; Substrat berbatu
Mexichromis multituberculata; Substrat berbatu
28
Risbecia tyroni; Substrat berbatu
Elysia cf marginata
30
RIWAYAT HIDUP
Penulis dilahirkan di Bogor pada tanggal 5 Juli 1993 sebagai anak pertama dari dua bersaudara dari orang tua bernama Iwan Kuswandi dan Fenti Hamidah. Penulis lulus dari Sekolah Menengah Pertama Negeri 6 Bogor pada tahun 2008, dan Madrasah Aliyah Negeri 1 Bogor tahun 2011. Pada tahun 2011, penulis diterima sebagai mahasiswa Institut Pertanian Bogor, Fakultas Perikanan dan Ilmu Kelautan, Departemen Ilmu dan Teknologi Kelautan melalui jalur SNMPTN Undangan dan memperoleh beasiswa BIDIKMISI tahun 2011-2015.
Selama kuliah di Institut Pertanian Bogor penulis pernah menjadi asisten pelatihan Molecular Genetic Technique tahun 2015 di Laboratorium Biosistematik dan Biodiversitas Kelautan, Departemen Ilmu dan Teknologi Kelautan, Fakultas Perikanan dan Ilmu Kelautan, Institut Pertanian Bogor.