Enterobacter sakazakii ISOLAT ASAL SUSU FORMULA DAN
MAKANAN BAYI: KARAKTERISASI GEN 16S rRNA DAN
PERILAKU BAKTERI PASCA REKONSTITUSI
YULIASRI RAMADHANI MEUTIA
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN SUMBER
INFORMASI
Dengan ini saya menyatakan bahwa tesis Enterobacter sakazakii Isolat Asal Susu Formula dan Makanan Bayi: Karakterisasi Gen 16S rRNA dan
Perilaku Bakteri Pasca Rekonstitusi adalah karya saya sendiri dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Bogor, Agustus 2008
Yuliasri Ramadhani Meutia
RINGKASAN
YULIASRI RAMADHANI MEUTIA. Enterobacter sakazakii Isolat Asal Susu Formula dan Makanan Bayi: Karakterisasi Gen 16S rRNA dan Perilaku Bakteri Pasca Rekonstitusi. Dibimbing oleh RATIH DEWANTI-HARIYADI dan SRI ESTUNINGSIH
Enterobacter sakazakii merupakan bakteri patogen yang dalam 20 tahun
terakhir ini dilaporkan menyebabkan beberapa kasus kematian serta penyakit pada bayi-bayi yang lahir prematur. Data penelitian di Indonesia menyebutkan bahwa dari 74 kemasan makanan bayi, 10 di antaranya (13.5%) ditemukan mengandung
E.sakazakii.Karena informasi mengenai E.sakazakii di Indonesia sangat terbatas,
tidak diketahui resiko bakteri ini terhadap bayi di Indonesia, sementara angka kematian bayi yang tinggi di Indonesia tidak selalu diketahui penyebabnya. Oleh karena itu diperlukan lebih banyak informasi mengenai keberadaan dan karakteristik E.sakazakii dalam susu formula dan makanan bayi, agar dapat dilakukan upaya-upaya dalam meminimalkan resiko bakteri ini.
Penelitian ini bertujuan untuk (1) mengisolasi E.sakazakii dari beberapa produk susu formula dan makanan bayi, (2) mempelajari karakteristik fenotip dan genotip isolat E.sakazakii, (3) mempelajari perilaku isolat setelah direkonstitusi dengan air berbagai suhu.
Tahap-tahap yang dilakukan pada penelitian ini meliputi isolasi
E.sakazakii dari susu formula dan makanan bayi dengan menggunakan media
selektif E.sakazakii yang bersifat kromogenik (DFI), dilanjutkan dengan identifikasi secara biokimiawi dengan menggunakan perangkat API 20E (Biomerieux) dan karakterisasi gen 16S rRNA dari isolat E.sakazakii yang diperoleh melalui metode PCR dengan menggunakan 2 pasang primer yakni 16SUNI-L/Saka-2b untuk mengamplifikasi segmen pertama dengan amplikon berukuran 977 bp dan ESA1/16SUNI-R untuk mengamplifikasi segmen kedua dengan amplikon berukuran 408 bp, setelah itu dilakukan perunutan asam nukleat (sekuensing) pada produk PCR yang diperoleh. Selain itu dipelajari juga perilaku isolat setelah direkonstitusi dengan air berbagai suhu, serta selama hang time
setelahrekonstitusi dengan air bersuhu 70oC.
Sebanyak 8 isolat E.sakazakii yang memiliki koloni tipikal pada DFI berhasil diisolasi dari 4 sampel uji (n=25). Identifikasi dengan API 20E menunjukkan bahwa 4 dari 8 isolat memiliki kemiripan secara biokimia sebesar 98.4% dengan E.sakazakii, sementara 2 isolat hanya memiliki sedikit kemiripan, dan 2 isolat lainnya tidak memiliki kemiripan dengan E.sakazakii secara biokimia. Setelah dikonfirmasi secara genotipik dengan menggunakan PCR dan sekuensing disimpulkan bahwa semua isolat yang diperoleh merupakan E.sakazakii.
Analisis hasil sekuensing menunjukkan bahwa isolat-isolat yang diperoleh memiliki kemiripan yang bervariasi antara 92 – 97% dengan genom lengkap E.
sakazakii ATCC BAA-894 (nomor akses CP000783). Seluruh isolat
Rekonstitusi susu dengan air bersuhu 4oC dan 40oC tidak banyak mengurangi jumlah E.sakazakii, sementara air bersuhu 100oC menginaktifkan
E.sakazakii hingga jumlah yang tak terdeteksi lagi. Suhu rekonstitusi 70oC dapat
mengurangi 2.74 hingga 6.72 log CFU/ml sehingga efektif jika digunakan sebagai suhu untuk merekonstitusi susu formula dan makanan bayi jika jumlah awal bakteri ini maksimal 5 log CFU/ml. Isolat YR t2a yang merupakan outgroup dari isolat E.sakazakii lainnya ditemukan memiliki kecenderungan lebih sensitif terhadap suhu air rekonstitusi dibandingkan isolat-isolat lainnya.
ABSTRACT
YULIASRI RAMADHANI MEUTIA. Enterobacter sakazakii Isolated From Powdered Infant Formula and Weaning Food: Characterization of the Gene Encoding the 16S rRNA and Its Behaviour Upon Reconstitution. Under the direction of RATIH DEWANTI-HARIYADI and SRI ESTUNINGSIH
Recently there has been increasing concern related to the presence of
Enterobacter sakazakii in powdered infant formula which was linked to severe
systemic infection in neonates. The occurrence of E.sakazakii in Indonesia was previously described but more information about E.sakazakii is required to evaluate the risk of this bacterium for infants in Indonesia.
Eight isolates of E.sakazakii were isolated from 4 out of 25 packages of powdered infant formula (PIF) and weaning food. Those isolates were identified by API 20E kit based on their biochemical reactions and followed by PCR test for confirmation. Further characterization was done by amplification of their 16S rRNA gene using 2 primer pairs, 16 SUNI-L/Saka 2b to amplify the first segment with amplicon size of 977 bp and ESA1/16 SUNI-R to amplify the second segment with amplicon size of 408 bp. Then The PCR products were sequenced to show their genetic variability. Isolates were also tested for their ability to survive during reconstitution with water having various temperatures, and their fate during hang time.
The results showed that the isolates had 92 to 97% similarity with
Enterobacter sakazakii ATCC BAA-894 based on their complete genome
(accesion number CP000783). YR t2a isolate was found to have more similarity with Citrobacter koseri (95%) and phylogenic tree analysis showed that YR t2a was located on outgoup of E.sakazakii.
Reconstitution with 100oC water decreased the number of bacteria of most isolates to undetectable level, while those with 40oC and 4oC water did not reduce the bacterial number significantly. Using water of 70oC, reconstitution reduced the number of bacteria in the range of 2.74 to 6.72 log CFU/ml. Therefore, temperature 70oC can be used for reconstitution of PIF and weaning food to reduce the risk of E.sakazakii. YR t2a isolate, which has lower genetic similarity with E.sakazakii than other isolates obtained in this study, tends to be more sensitive to the reconstitution water temperature than other E.sakazakii isolates.
© Hak cipta milik Yuliasri Ramadhani Meutia, tahun 2008
Hak cipta dilindungi
Enterobacter sakazakii ISOLAT ASAL SUSU FORMULA DAN
MAKANAN BAYI: KARAKTERISASI GEN 16S rRNA DAN
PERILAKU BAKTERI PASCA REKONSTITUSI
YULIASRI RAMADHANI MEUTIA
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada
Departemen Ilmu dan Teknologi Pangan
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
BOGOR
Judul Tesis : Enterobacter sakazakii Isolat Asal Susu Formula dan Makanan Bayi: Karakterisasi Gen 16S rRNA dan Perilaku Bakteri Pasca Rekonstitusi
Nama : Yuliasri Ramadhani Meutia NIM : F251040171
Disetujui Komisi Pembimbing
Dr.Ir. Ratih Dewanti-Hariyadi, MSc Dr.drh. Sri Estuningsih, MSi Ketua Anggota
Diketahui .
Ketua Program Studi Ilmu Pangan Dekan Sekolah Pascasarjana IPB
Dr.Ir. Ratih Dewanti-Hariyadi, MSc Prof. Dr. Ir. Khairil Anwar Notodiputro, MS
PRAKATA
Puji dan syukur tak terhingga penulis panjatkan kepada Allah SWT atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak Agustus 2006 ini ialah keamanan pangan, dengan judul Enterobacter sakazakii Isolat Asal Susu Formula dan Makanan Bayi: Karakterisasi Gen 16S rRNA dan Perilaku Bakteri Pasca Rekonstitusi.
Terimakasih penulis ucapkan kepada Ibu Dr. Ir. Ratih Dewanti-Hariyadi, MSc dan Ibu Dr. drh. Sri Estuningsih MSi selaku pembimbing, serta Ibu Dr. Ir. Sri Hendrastuti Hidayat, MSc yang telah banyak memberi saran khususnya dalam bidang analisis genetik.
Terima kasih penulis ucapkan kepada mbak Ari, Pak Taufik, Pak Karna, serta para teknisi baik di laboratorium Departemen ITP maupun di laboratorium SEAFAST Center atas bantuan dan kerjasamanya selama penulis melaksanakan penelitian. Terima kasih kepada Ibu Siti Nurjanah, STP, MSi., Yassier Anwar, Mbak Pepy, Suhanda Jaya serta rekan-rekan di laboratorium BIORIN IPB, serta Mbak Tuty, Pak Rai, Mbak Sat dari Laboratorium Virologi Tumbuhan Departemen Proteksi Tanaman IPB yang telah banyak memberikan masukan dalam hal analisis genetika. Tak lupa penulis ucapkan terima kasih kepada rekan-rekan seperjuangan di laboratorium SEAFAST IPB, Reno, Agnani, Pak Soenar, Anggi, Fitri, Desty, dan rekan-rekan lainnya yang tak dapat penulis ungkapkan satu persatu.
Ungkapan terima kasih juga disampaikan kepada mama, ayah, abang, dan adik-adikku, atas segala dukungan, doa, dan kasih sayangnya. Juga kepada Ibu mertua atas dukungan dan doanya Terutama sekali kepada sumber inspirasi penulis Muhammad Syafiq Abdurrahman yang telah menceriakan hari-hari penulis, serta Rusdiansah Usni yang tidak pernah lelah mendengarkan keluh-kesah penulis. Serta semua pihak yang tidak dapat disebutkan satu persatu.
Semoga karya ilmiah ini bermanfaat.
Bogor, Agustus 2008
RIWAYAT HIDUP
Penulis dilahirkan di Lhokseumawe pada tanggal 16 Juli 1980 dari ayah Ir. Aknasio Sabri dan ibu Hana Rohana. Penulis merupakan putri kedua dari empat bersaudara.
Tahun 1998 penulis lulus dari SMU Bina Insani Bogor dan pada tahun yang sama lulus seleksi masuk IPB melalui jalur Undangan Seleksi Masuk IPB (USMI). Penulis memilih Program Studi Teknologi Pangan (yang kini bernama Ilmu dan Teknologi Pangan), Fakultas Teknologi Pertanian. Selama mengikuti program S1, penulis pernah menjadi asisten mata kuliah Kimia Dasar 2 dan Mikrobiologi Pangan 2 pada tahun ajaran 2001/2002.
DAFTAR ISI
Karakteristik Pertumbuhan dan Kematian E.sakazakii ... 5
Sumber E.sakazakii... 6
Penyakit karena E.sakazakii... 8
Dosis Infeksi E.sakazakii... 8
Faktor Virulensi E.sakazakii ...9
Sifat Resistensi E.sakazakii Terhadap Antibiotik...10
Media Isolasi E.sakazakii...11
Gen...14
Konstruksi Pustaka Gen 16S rRNA...15
Polymerase Chain Reaction (PCR)...15
Perunutan Basa Nukleotida (Sekuensing)...18
Analisis Keragaman Genetik...19
Analisis Neighbor-Joining...20
Keragaman Genetik E.sakazakii...22
METODOLOGI
Amplifikasi Gen 16S rRNA dan Analisis Sekuensing...44
Pengaruh Suhu Rekonstitusi...51
Persiapan Kultur...51
Simulasi Rekonstitusi dalam Penyiapan Susu Formula...53
DAFTAR TABEL
Halaman 1. Waktu yang dibutuhkan pada dosis infeksi (1000 sel) untuk dapat diraih
pada susu formula yang direkonstitusi...9
2. Pasangan primer oligonukleotida yang digunakan...25
3. Sifat biokimia dari spesies Enterobacter...29
4. Hasil isolasi E.sakazakii pada beberapa media...35
5. Hasil analisis uji biokimia E.sakazakii dengan menggunakan program apiweb™...41
6. Perbandingan tingkat homologi isolat E.sakazakii hasil isolasi dengan menggunakan program BLAST...47
DAFTAR GAMBAR
Halaman
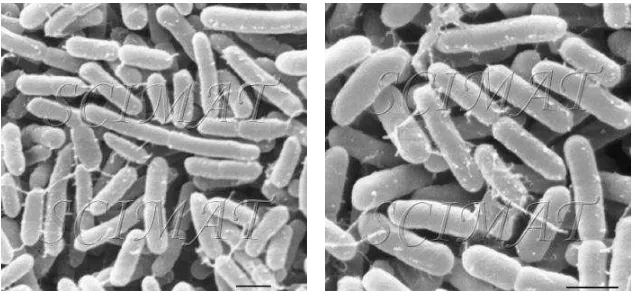
1. E.sakazakii , bar= 1μm...3
2. E.sakazakii yang positif pada media VRBG...27
3. Koloni yang tumbuh pada media DFI...27
4. Koloni positif E.sakazakii pada media TSA...28
5. Penampakan E.sakazakii secara marfologi di bawah mikroskop dengan pembesaran 1000x...40
6. Elektroforesis DNA genom E.sakazakii dengan buffer 1xTAE...43
7. Hasil PCR segmen 1 dengan ukuran amplikon 977 bp...45
8. Hasil PCR segmen 2 dengan ukuran amplikon 407 bp...45
9. Dendogram neighbour joiningE.sakazakii...50
10. Pengaruh rekonstitusi dengan air bersuhu 40oC terhadap E.sakazakii...53
11. Perubahan jumlah E.sakazakii setelah rekonstitusi dengan air bersuhu 40oC...54
12. Pengaruh rekonstitusi dengan air bersuhu 4oC terhadap E.sakazakii...56
13. Perubahan jumlah E.sakazakii setelah direkonstitusi dengan air bersuhu 4oC...56
14. Pengaruh rekonstitusi dengan air bersuhu 70oC terhadap E.sakazakii...57
15. Penurunan jumlah E.sakazakii setelah direkonstitusi dengan air bersuhu 70oC...58
16. Pertumbuhan E.sakazakii selama hang time pasca rekonstitusi dengan air bersuhu 70oC...61
17. Pertumbuhan E.sakazakii selama hang time pasca rekonstitusi dengan air bersuhu 70oC...62
18. Pertumbuhan E.sakazakii selama hang time yang tidak terdeteksi pasca rekonstitusi dengan air bersuhu 70o C...63
DAFTAR LAMPIRAN
Halaman
1. Bagan alir teknik isolasi E.sakazakii dari susu formula dan makanan bayi....74
2. Cara pembuatan larutan untuk isolasi DNA dan buffer...75
3. Hasil pengujian API 20E yang dianalisis dengan program apiweb™...76
4. Absorbansi sampel DNA pada λ 260 / 280 nm...80
5. Urut-urutan basa nukleotida hasil sekuensing...81
6. Pengamatan plating susu bubuk terinokulasi...85
7. Pengamatan Plating Rekonstitusi dengan air bersuhu 100oC...86
8. Pengamatan Plating Rekonsitusi dengan air bersuhu 40oC...87
9. Pengamatan Plating Rekonstitusi dengan air bersuhu 4oC...88
10. Pengamatan Plating Rekonstitusi dengan air bersuhu 70oC ...89
11. Pengamatan PlatingHang Time Setelah direkonstitusi dengan air bersuhu 70oC...90
PENDAHULUAN
Latar Belakang
Enterobacter sakazakii (E.sakazakii) merupakan bakteri patogen yang dilaporkan menyebabkan beberapa kasus kematian serta penyakit pada bayi-bayi yang lahir prematur dalam kurun waktu 20 tahun terakhir. Beberapa laporan mengidentifikasikan bahwa susu formula dan makanan bayi merupakan sumber infeksi dan sarana penularan E.sakazakii. E.sakazakii bersifat berbahaya pada bayi yang baru lahir (neonatal) dengan status kesehatan yang rendah, termasuk di dalamnya bayi baru lahir berusia 28 hari, bayi prematur, bayi yang berbobot lahir rendah, bayi yang secara spesifik mengalami immuno-compromised, serta bayi dari ibu yang terinfeksi HIV. Review kasus antara tahun 1961 hingga 2003 menemukan bahwa 25 kasus (52 persen) terjadi pada bayi yang berbobot rendah yang mengkonsumsi susu formula (Anonim, 2004).
pertumbuhan E.sakazakii ini dibiarkan selama penanganan serta penyimpanan yang tidak layak dapat menjadikannya tumbuh semakin banyak dan membahayakan. (Anonim, 2004). Angka mortalitas dari kejadian infeksi oleh
E.sakazakii ini tinggi yaitu bervariasi antara 10 hingga 80% (van Acker et al.
2001, Lai et al. 2001). Pasien biasanya meninggal atau jika sembuh akan mengalami sequele pada otak yang menyebabkan hydrocephalus, quadriplegia,
dan keterbelakangan dalam perkembangan mental.
Identifikasi Masalah
Keberadaan E.sakazakii dalam produk pangan di Indonesia pertama kali dilaporkan oleh Estuningsih et al. (2006) yang melakukan penelitian terhadap 74 kemasan makanan bayi. E.sakazakii berhasil diisolasi dari 10 kemasan makanan bayi yang berasal dari 2 manufaktur. Data mengenai patogenisitas isolat tersebut serta karakteristiknya dalam bahan pangan khususnya susu formula dan makanan bayi juga belum tersedia. Oleh karena itu perlu diketahui keberadaan E.sakazakii
dalam susu formula dan makanan bayi, serta pengaruh suhu rekonstitusi pada isolat lokal E.sakazakii yang berguna dalam manajemen resiko bakteri ini.
Tujuan Penelitian
Penelitian ini bertujuan untuk:
1. Mendapatkan isolat E.sakazakii dari produk susu formula dan makanan bayi yang beredar di pasaran, terutama yang diperuntukkan untuk bayi berusia 6 bulan ke bawah (untuk susu formula) atau tahap 1 (untuk makanan bayi).
2. Melakukan karakterisasi sifat genotip E.sakazakii yang diperoleh dengan menggunakan isolat yang diperoleh sebelumnya oleh Estuningsih (2006) dan isolat ATCC E.sakazakii sebagai pembanding.
Manfaat Penelitian
Manfaat penelitian ini adalah menambah basis data mengenai E.sakazakii , khususnya untuk kondisi Indonesia yang berguna dalam meningkatkan jaminan keamanan produk makanan bayi.
Hipotesis
Hipotesis dalam penelitian ini adalah, E. sakazakii dapat ditemukan di dalam susu formula dan makanan bayi dan kemungkinan memiliki karakteristik genotip berdasarkan gen 16S rRNA yang homolog dengan isolat lokal E.sakazakii
atau isolat E.sakazakii yang terdapat pada GenBank, serta memilki ketahanan yang bervariasi pada kondisi suhu rekonstitusi tertentu.
TINJAUAN PUSTAKA
Enterobacter sakazakii
Secara biologis E. sakazakii merupakan bakteri yang bersifat motil, tidak membentuk spora, Gram negatif fakultatif anaerob. E.sakazakii yang diamati dengan mikroskop elektron dapat dilihat pada Gambar 1.
Pertama kali dikenal sebagai ‘Enterobacter cloacae berpigmen kuning’ hingga tahun 1980 dimana telah diidentifikasi sebagai spesies baru dan diberi nama Enterobacter sakazakii oleh Farmer, et al. (1980) sebagai penghargaan terhadap bakteriologis dari Jepang, Riichi Sakazaki.
Akhir-akhir ini E. sakazakii ditemukan sebagai kontaminan yang terkadang terdapat pada susu bubuk formula untuk bayi yang menyebabkan
neonatal meningitis, bacterimia, necrotizing enterocolitis (NEC), dan
meningoencephalitis yang mengancam hidup bayi (Muytjens & Kollee, 1990). Meskipun baru-baru ini saja mendapat perhatian dari industri dan masyarakat dari jumlah kejadian dan produk yang di-recall, namun kasus yang menyebabkan kematian pada bayi ini telah diutarakan sejak tahun 1958 oleh Urmenyi & Franklin (1961). ICMSF (International Commision for Microbiological Specification for Foods (2002) juga menggolongkan organisme ini sebagai mikroba yang sangat berbahaya untuk populasi yang terbatas, mengancam kehidupan atau substansi kronis lanjutan atau berdurasi lama. Organisme ini dinyatakan mempunyai peringkat yang sama sebagaimana patogen pada makanan dan air yang sudah cukup dikenal seperti Listeria monocytegenes, Clostridium botulinum tipe A dan tipe B, serta Cryptosporodium parvum.
Secara fenotipik bakteri ini mempunyai kemiripan dengan E.cloacae,
sehingga Farmer et al. 1980 menempatkan bakteri ini ke dalam genus
Enterobacter. Secara umum organisme Enterobacter bertanggung jawab pada sekitar 50% infeksi nosocomial yang hampir semuanya terjadi pada pasien yang mengalami immunocompromised (Leclerc et al. 2001).
Farmer et al. (1980) melaporkan studi hibridisasi DNA-DNA dan menyimpulkan bahwa perbandingan antara E.sakazakii dengan spesies
Enterobacer dan Citrobacter memiliki homologi sebesar 41% dengan C.freundii
Karakteristik Pertumbuhan dan Kematian E.sakazakii
E.sakazakii tumbuh pada media yang digunakan untuk mengisolasi mikroba enterik seperti MacConkey, Eosin Methylene Blue, dan Deoxycholate Agar. Pada agar cawan bakteri ini dapat membentuk dua tipe koloni yaitu glossy
(mengkilat) dan matt (tidak mengkilat), tergantung pada media yang digunakan dan galurnya. Pertumbuhan pada Tryptone Soya Agar pada 25oC selama 48 jam dapat memproduksi pigmen kuning yang tidak dapat berdifusi. Bakteri ini memproduksi reaksi ekstraseluler DNase yang tertunda pada Toluidine Blue Agar
(36oC, 7 hari). E.sakazakii bersifat α-glukosidase positif yang dapat terdeteksi dengan menggunakan 4-nitrophenyl-α-D-glucopyranoside setelah 4 jam pada 36oC. Mikroba ini memproduksi D-lactic acid dan bersifat mucate negatif. Hampir seluruh isolat tidak memfermentasi sorbitol, dan dapat tumbuh pada kisaran suhu yang lebar (6 – 47oC). Pada suhu ruang atau 21oC bakteri ini mempunyai waktu penggandaan (doubling time) sekitar 75 menit pada susu formula bayi yang direkonstitusi (Iversen et al. 2003). Skladal et al. (1993) menemukan bahwa susu UHT dalam karton yang diinokulasi dengan 10-15 sel
E.sakazakii / 500 ml dan diinkubasi pada 30oC sangat mendukung pertumbuhan bakteri ini, dan susu tersebut menjadi asam dikarenakan produksi D-lactate.
Waktu penggandaan pada suhu rendah kira-kira 10 jam pada susu formula dan kemudian dapat tumbuh secara lambat di bawah kondisi refrigerasi. Batasan dalam pertumbuhannya yang dikarenakan aktivitas air (aw) dan pH belum diketahui (Iversen & Forsythe, 2003).
Nazarowec-White (1997) dan Farber (1999) menerangkan bahwa mikroba pada susu formula memiliki waktu reduksi desimal dan nilai z tertentu yaitu nilai D52 adalah 54,8 menit dan D60 nya adalah 2,5 menit. Data kemudian
E.sakazakii memproduksi kapsul heteropolisakarida yang mengandung 29-32% glucuronic acid, 23-30% glukosa, 19-24% galaktosa, 13-22% D-fukosa, dan 0-8% mannosa (Harris & Oriel, 1989). Produksi optimalnya adalah di bawah kondisi pertumbuhan yang terbatas nitrogennya (rasio C/N 20:1) (Scheepe-Leberkϋhne & Wagner, 1986).
Kapsul terlibat dalam kemampuan bakteri ini untuk bertahan pada masa simpan susu formula yang panjang (24 bulan). Kapsul juga menjadikan bakteri ini dapat menempel pada permukaan dan membentuk biofilm yang bersifat sangat resisten terhadap bahan pembersih dan desinfektan (Harris & Oriel, 1989).
Iradiasi dengan sinar gamma dengan dosis di atas 1 kGy untuk susu formula yang telah direkonstitusi dan dosis di atas 9kGy untuk susu formula bubuk dapat membunuh E.sakazakii dengan nilai D10 berkisar antara 0.21 hingga
0.29 kGy, 0.24 hingga 0.37 kGy, dan 1.06 hingga 1.71 kGy berturut-turut pada
Brain Hearth Infusion Broth, RIMF, dan DIMF (Osaili et al. 2007). Penelitian yang serupa dilakukan oleh Lee et al. (2007) yang menyatakan bahwa nilai D10
dari E.sakazakii yang diberi iradiasi sinar gamma adalah 0.22 – 0.27 kGy pada
broth dan 0.76 kGy pada susu bubuk formula. Lee et al. (2007) juga menyatakan bahwa iradiasi pada 5.0 kGy dapat mengeliminasi E.sakazakii yang diinokulasi sebesar 8.0 hingga 9.0 log CFU/g pada susu bubuk formula.
Metode pulsed electric field (PEF) dengan intensitas sebesar 40 kV/cm pada paparan selama 360 µs dapat mereduksi E.sakazakii pada BPW maksimum sebesar 2.7 siklus log CFU/ml, dan paparan yang sama pada susu formula bubuk dapat mereduksi E.sakazakii sebesar 1.2 log CFU/ml (Perez et al. 2007).
Sumber E.sakazakii
organisme ini terdapat secara luas namun Muytjens & Kollee (1990) tidak berhasil mengisolasi organisme ini dari susu sapi mentah, ternak, tikus, padi-padian, kotoran burung, hewan peliharaan, permukaan air, tanah, lumpur, atau akar kayu (Iversen & Forsythe, 2003).
Organisme ini dapat diisolasi dari berbagai makanan termasuk keju, roti, tahu, teh asam, daging yang digaramkan (curing), minced beef, dan sosis.
E.sakazakii juga ditemukan pada khamir roti dikarenakan organisme ini merupakan bagian dari flora permukaan biji sorgum (Gassem 1999). Organisme ini juga ditemukan pada biji padi (Cottyn et al. 2001).
Sebagian besar wabah E.sakazakii yang terjadi dilaporkan berasal dari susu formula yang terkontaminasi, karena susu formula tidak dirancang sebagai produk dengan hasil akhir steril. Muytjens et al. (1988) menemukan bahwa 52.2% dari 141 sampel susu formula dari 35 negara telah terkontaminasi dengan
Enterobacteriaceae, dimana 25% mengandung E.agglomerans, 21% mengandung
E.cloacae, dan 14% mengandung E.sakazakii. E.sakazakii juga berhasil diisolasi dari produk susu formula bayi yang tidak terpakai dari 13 negara, dengan level kontaminasi 0.36 hingga 66.0 CFU/ 100 g. Nilai tersebut serupa dengan nilai 8 sel /100g yang dilaporkan oleh Simmons et al. (1989) untuk susu bubuk formula yang telah terbuka kalengnya yang digunakan selama waktu terjadinya wabah pada ruang intensive care unit (ICU) untuk bayi yang baru lahir. Nazarowec-White & Farber (1997) melakukan pengujian terhadap 120 kaleng susu formula dari lima perusahaan yang berbeda di Kanada dan menemukan bahwa 6.7% mengandung E.sakazakii. Jumlah E.sakazakii pada sampel yang positif umumnya adalah 0.36 CFU /100 g. Heuvelink et al. (2001) menggunakan uji
present/absence dalam 25 g susu bubuk, mendeteksi E.sakazakii pada 1 dari 40 susu bubuk formula untuk bayi dan 7 dari 170 susu bubuk. Estuningsih et al.
Penyakit karena E.sakazakii
Jenis-jenis penyakit yang disebabkan oleh E.sakazakii umumnya adalah meningitis, sepsis, seizure, bacterimia, brain cyst (Lai et al. 2001). Meningitis adalah suatu infeksi dan inflamasi pada meninges (lapisan penutup otak), sepsis adalah beredarnya bakteri pembentuk nanah atau toksinnya mengikuti sirkulasi darah yang dapat berada dalam darah atau jaringan, seizure adalah kejang, sedangkan brain cyst adalah munculnya kista pada otak. Van Acker et al.(2001) melaporkan bahwa E.sakazakii dapat menyebabkan penyakit necrotizing enterocolitis (NEC).
Dosis Infeksi E.sakazakii
Meskipun tidak ada bukti secara epidemiologis tentang dosis infeksinya, Iversen & Forsythe (2003) memperkirakan 1000 sel sebagai konsentrasi awal
E.sakazakii yang dapat menyebabkan infeksi. Hal ini cukup beralasan karena sama dengan dosis infeksi pada Neiserria meningitidis, E.coli O157, dan
L.monocytogenes 4b. Dosis infeksi E.sakazakii dapat bervariasi bergantung pada respon bakteri ini terhadap stres, kondisi kesehatan inang (sehat atau bersifat
immuno-compromised), serta bergantung pada komponen-komponen dalam makanan (Iversen & Forsythe, 2003). Pada kasus bayi yang baru lahir yang diberikan susu formula, bila dilihat dari sisi mikrobanya, E.sakazakii telah mengalami kondisi stres selama pengeringan semprot (spray drying) dan penyimpanan. Bila dilihat dari sisi inangnya, dalam hal ini bayi yang baru lahir, bayi merupakan golongan individu yang memiliki daya tahan tubuh yang masih lemah karena bayi yang baru lahir belum mampu membentuk antibodi dalam dirinya hingga berusia 2 bulan, sehingga bayi yang baru lahir dapat dikatakan bersifat immuno-compromised. Nazarowec-White & Farber (1997) melaporkan bahwa E.sakazakii dapat menimbulkan infeksi pada mencit bila diinokulasikan dengan bakteri ini secara oral sebesar 105 CFU dan secara intraperitonial sebanyak 103 CFU.
yang diperlukan oleh organisme ini untuk menggandakan diri hingga 14 generasi pada dosis infeksi (1000 sel) pada kondisi inkubasi yang berbeda-beda yang mana dapat dilihat pada Tabel 1 berikut.
Tabel 1. Waktu yang dibutuhkan pada dosis infeksi (1000 sel) untuk dapat diraih pada susu formula yang direkonstitusi
Suhu (oC) Waktu penggandaan
(jam)
Waktu yang dibutuhkan untuk meraih dosis infeksi (14 generasi)*
10
* Penghitungan mengasumsikan rata-rata E.sakazakii pada susu bubuk formula untuk bayi adalah 0.36 sel /100 g dan untuk sekali makan adalah 18 g (direkonstitusi menjadi 115 ml) dengan tidak ada yang mati selama persiapan dan tidak ada yang bertambah selama dalam lambung. Waktu lag pada 10oC adalah 2 jam, untuk semua suhu lainnya waktu lag tidak signifikan. Dosis infeksi diasumsikan berasal dari 1000 sel E.sakazakii yang ditelan sebagai dosis tunggal dan tidak pada paparan yang terakumulasi. Namun perlu dipertimbangkan bahwa bayi makan selama 4 hingga 6 kali pada periode 24 jam.
Berdasarkan Tabel 1 di atas, dapat dilihat bahwa peluang tercapainya dosis infeksi E.sakazakii rendah, karena diperlukan waku yang relatif lama, kematian
E.sakazakii selama persiapan akibat penambahan air panas, dan tidak terjadi penggandaan bakteri dalam lambung. Sebagai tambahan, penghitungan ini mengasumsikan bahwa meskipun bayi makan sebanyak 4 hingga 6 kali selama periode 24 jam, dosis infeksi diperoleh pada sekali makan, tidak dalam bentuk kumulatif (Forsythe, 2002). Meskipun keterbatasan pada penghitungan ini merupakan bukti bahwa susu formula pada level normal yang rendah (≤ 0.36 sel
E.sakazakii /100 g) kemungkinan tidak menyebabkan infeksi jika tidak terdapat penyimpangan suhu atau kontaminasi melalui preparasi yang bersanitasi buruk seperti blender yang terkontaminasi atau sendok pencampur. Tingkat sanitasi yang buruk merupakan salah satu sumber yang memungkinkan dari terjadinya wabah (Block et al. 1988, Clark et al. 1990).
Faktor Virulensi E.sakazaki
Laporan mengenai virulensi E.sakazakii meyebutkan bahwa seluruh galur
kemampuan penempelan Esakazakii lebih besar daripada E.coli galur K1 (NMEC) yang bisa menyebabkan meningitis pada bayi yang baru lahir. Selain itu, berbeda dengan E.cloacae dan NMEC, E.sakazakii mampu memperahankan diri dari serangan makrofage (Ownsend et al. 2008).
Manget et al.(2006) menyebutkan bahwa tahap-tahap patogenisitas dari
E.sakazakii ini meliputi kesuksesan bakteri ini dalam kolonisasi, keberhasilan dalam menimbulkan penyakit melalui penempelan pada permukaan sel inang seperti membran mukosa, lambung, epitelial usus, dan jaringan endotelial pada otak. Penelitian ini juga menyimpulkan bahwa penempelan E.sakazakii pada sel epitelial dan endotelial bukan berdasarkan fimbrae bakteri ini.
Sifat Resistensi E.sakazakii Terhadap Antibiotik
Farmer et al. (1980) menemukan bahwa seluruh galur E.sakazakii rentan terhadap gentamycin, kanamycin, chloramphenicol, dan amphicilin; lebih dari 87% E.sakazakii bersifat rentan terhadap nalidixic acid, streptomycin, tetracycline, dan carbenicilin; 71 dan 67% bersifat rentan terhadap sulfadiazine,
dan colistin; hanya 13% yang bersifat rentan terhadap cephalothin. Seluruh galur bersifat resisten terhadap penicillin; hanya 1 dari lebih dari 100 galur yang diuji menunjukkan resistensi terhadap antibiotik yang berganda. Muytjens & van der Ros-van der Repe (1986) menemukan bahwa MIC untuk 90% dari 195 galur
E.sakazakii yang diuji terhadap 25 antibiotik sedikitnya setengah dari E.cloacae. Namun terjadi juga resistensi terhadap cephalothine dan sulfamethoxazole. Nazarowec-White & Farber (1999) menemukan bahwa tipe galur (ATCC-29544), 5 dari 8 sampel makanan dan 8 dari 9 galur dari sampel klinis hanya resisten terhadap sulphisoxazole dan cephalothin. Galur klinis lainnya ditunjukkan bersifat rentan terhadap seluruh antibiotik sedangkan tiga dari isolat makanan juga bersifat resistan terhadap chloramphenicol. Dua dari sampel makanan yang resisten terhadap chloramphenicol tersebut juga mempunyai sifat resisten terhadap
tetracyclin, serta satu dari sampel makanan tersebut juga resisten terhadap
seluruh isolat E.sakazakii bersifat resisten terhadap ampicillin,cefazoline, dan spektrum yang luas dari penicillin, namun bersifat rentan terhadap
aminoglycoside dan trimetophrim-sulfamethoxazole, dimana sensitivitas terhadap generasi ketiga dari cephalosporine dan quinolone bervariasi. Dennison & Morris (2002) melaporkan bahwa infeksi E.sakazakii resisten terhadap berbagai antibiotik, termasuk ampicillin, gentamycin, dan cefotazamine. Pada studi resistensi bakteri ini terhadap antibiotik yang dilakukan oleh Burgos & Varela (2002), ditemukan bahwa DNA genomik dari E.sakazakii mengandung operon yang resisten terhadap berbagai antibiotik (multiple antibiotic resistance=mar).
Berdasarkan beberapa keterangan di atas dapat dikatakan bahwa
E.sakazakii mempunyai sifat resistensi yang berbeda-beda terhadap beberapa jenis antibiotik.
Media Isolasi E.sakazakii
FDA (2002) telah mengembangkan media serta reagen-reagen yang digunakan dalam isolasi E.sakazakii. Pada metode ini dibutuhkan beberapa tahapan dalam pengisolasian E.sakazakii antara lain susu formula bubuk perlu direkonstitusi terlebih dahulu dengan air destilata steril selama 24 jam pada 6oC, diikuti dengan tahap pengkayaan pada media Enterobacteriaceae enrichment (EE)
broth selama 24 jam pada 36oC, dilanjutkan dengan plating permukaan dan penggoresan pada Violet Red Bile Glucose (VRBG) Agar dan diinkubasi selama 24 jam pada 36oC, koloni positif yang tumbuh diseleksi kembali pada media
Tryptic Soy Agar (TSA) dan diinkubasi selama 48 hingga 72 jam pada 25oC. Koloni positif E.sakazakii pada TSA yang berpigmen kuning kemudian dikonfirmasi dengan menggunakan sistem identifikasi biokimia, API 20E yang memerlukan tambahan waktu selama 18 hingga 24 jam. EE broth dan VRBG Agar mengandung bahan-bahan yang menjadikannya sebagai media selektif yaitu oxgall dan brilliant green pada EE broth, dan bile salt no.3 dan kristal violet pada VRBG) yang mampu mencegah resusitasi dari sel E.sakazakii yang mengalami luka.
mengemukakan beberapa kelemahan metode FDA yaitu pada VRBG dan TSA. Media VRBG dinyatakan kurang selektif karena mikroba lain dapat juga tumbuh dan menghasilkan koloni yang berwarna ungu juga yang dikelilingi dengan halo
berwarna ungu yang diakibatkan oleh presipitasi garam empedu, sehingga agak sulit untuk membedakan E.sakazakii dari bakteri lainnya. Media TSA juga memiliki beberapa kekurangan yaitu waktu inkubasi yang diperlukan terlalu lama yaitu hingga 72 jam, selain itu terdapat spesies Enterobacteriaceae yang berpigmen kuning lainnya selain E.sakazakii yaitu E. hermanii dan E. vulneris
yang dapat menimbulkan kerancuan dalam pendeteksiannya. Sehingga Oh & Kang (2004) mengembangkan media isolasi E.sakazakii yang selektif dan berdasarkan sifat fluorogenik yaitu Oh & Kang (OK) Agar. Media ini dibuat berdasarkan sifat fluorogenik dari senyawa fluorogen yang juga merupakan substrat dari enzim α-glukosidase yang diproduksi secara spesifik oleh
E.sakazakii yaitu 4-methyl-umbelliferyl α-D-glucoside. Bile salt no.3 yang terdapat dalam media ini digunakan untuk mengisolasi bakteri enterik, ferric citrate dan sodium tiosulfat digunakan untuk mengisolasi Enterobacteriaceae
yang mampu memproduksi H2S. Senyawa fluorogen tersebut juga terdapat pada
media yang dikembangkan oleh Leuschner et al. (2003) yang mengembangkan media non selektif, Leuschner-Baird-Donald-Cox (LBDC) Agar untuk deteksi awal E.sakazakii pada susu formula.
Iversen & Forsythe (2004) mengembangkan media chromogenic selektif untuk mendeteksi E.sakazakii pada susu formula yaitu Druggan-Forsythe-Iversen (DFI) agar. Bahan selektif yang terdapat pada media ini adalah suatu senyawa
chromogen yaitu 4-chloro-indolyl-α-D-glucopyranoside. Senyawa ini akan berikatan dengan enzim α-glukosidase pada E.sakazakii yang akan membentuk koloni berwarna hijau-biru. Selain itu pada media ini terdapat sodium desoxycholate yang bersama-sama dengan sodium thiosulphate dan ferric ammonium citrate yang bertindak sebagai senyawa selektif.
awal dibandingkan dengan metode konvensional. Di samping itu, sebanyak 67 sampel positif E.sakazakii dapat terisolasi dengan menggunakan DFI, sedangkan dengan menggunakan metode konvensional, dari sampel-sampel yang sama hanya 19 sampel yang terdeteksi positif E.sakazakii. Isolasi bakteri ini dengan menggunakan metode konvensional mengalami kesalahan positif hingga 72.9%, dibandingkan dengan apabila diisolasi dengan DFI, yaitu hanya sekitar 38.5%. Tidak ada sampel positif pada metode konvensional yang terdeteksi negatif pada media DFI.
Restaino (2004) juga telah mengembangkan media chromogenic untuk isolasi E.sakazakii. Medium ini mengandung suatu senyawa chromogen yang menyebabkan koloni positif E.sakazakii berwarna biru-hitam hingga biru-abu-abu, sementara bakteri enterik lainnya menunjukkan warna hijau, kuning, atau koloni yang bening. Media ini juga mengandung pewarna tertentu dan garam bile
sebagai senyawa penghambat dan selektif.
Lehner et al. (2006) membandingkan dua media chromogenic yaitu
Enterobacter sakazakii Isolation Agar (ESIA) dengan DFI, dimana dapat disimpulkan bahwa DFI lebih baik dari pada ESIA karena terdapat salah satu galur yang pada ESIA tidak menunjukkan koloni tipikal, sedangkan pada DFI terlihat koloni tipikal berwarna biru-hijau.
Gen
Gen merupakan bagian dari DNA yang membawa informasi genetik atau menentukan sifat suatu organisme. Gen adalah fragmen DNA atau kromosom yang menyandi satu rantai polipeptida fungsional atau molekul RNA. Enzim RNA polimerase akan membaca basa-basa yang terdapat pada ruas DNA dan untuk setiap basa akan dicari padanan nukleotidanya yang kemudian akan dirangkaikan menjadi RNA. Pembacaan oleh RNA polimerase dimulai dari tanda awal promotor sampai tanda akhir terminator. Gen mempunyai peran dalam proses kehidupan melalui pengendalian pembentukan enzim dan protein. Enzim memegang peranan penting dalam kehidupan organisme yaitu sebagai katalisator dalam menjalankan reaksi kimia dalam proses metabolisme seluler. Ekspresi gen adalah proses penterjemahan informasi genetik yang dibawa pada DNA atau gen menjadi sifat biologis yang terekspresi. Ekspresi gen merupakan proses sintesis polipeptida berdasarkan sekuen nukleotida pada gen tersebut menjadi protein yang berfungsi (Yusuf, 2001).
Ekspresi gen terbagi menjadi dua tahapan yaitu transfer informasi genetik dari DNA ke dalam RNA (transkripsi) berdasarkan sekuen DNA dan penterjemahan informasi genetik yang terdapat pada RNA ke dalam polipeptida (translasi). Proses transfer atau penterjemahan informasi dilakukan dengan menggunakan molekul sumber informasi sebagai model cetakan dalam sintesis molekul penerima informasi. Di dalam transkripsi DNA akan dijadikan model cetakan untuk sintesis RNA, dan dalam translasi RNA akan menjadi model dalam sintesis protein. Di dalam transkripsi gen dibutuhkan faktor-faktor seperti promotor, RNA polimerase, ribonukleotida, signal terminasi, faktor transkripsi dan aktivitas protein. Dalam proses translasi, sintesis polipeptida terjadi berdasarkan kodon pada mRNA. Kodon merupakan tiga ribonukleotida pada mRNA yang menyandi asam amino. Selama proses translasi dibutuhkan mRNA, tRNA, ribosom, asam amino, dan beberapa faktor protein (Yusuf, 2001).
poliadenilasi yaitu proses penambahan ekor poli-A pada ujung 3’ utas mRNA,
capping atau penambahan 7-metil guanosin di ujung 5’ utas mRNA (Yusuf, 2001).
Konstruksi Pustaka Gen 16S-rRNA
Kunci untuk mengerti keragaman mikroba adalah sistem klasifikasi yang dapat diandalkan. Metode molekuler terutama klasifikasi dan identifikasi berbasis filogenetik, menggunakan parameter yang tidak bergantung pada kondisi pertumbuhan dan media yang digunakan. Pendekatan yang banyak dipakai saat ini adalah analisis sekuen gen 16S rRNA atau 23S rRNA. 16S rRNA merupakan gen yang bersifat spesies spesifik teradap prokariotik (Amann et al. 1994).
RNA bekerja membawa informasi genetik dari DNA kepada proses biosintesis protein di dalam ribosoma, dan RNA ribosom (rRNA) merupakan komponen utama ribosom yang menyusun sampai 65 persen berat ribosom. 16S dan 23S rRNA merupakan bagian dari subunit 30S dan 50S pada ribosom. Bila 16S rRNA diisolasi dalam bentuk murni kemudian dicampur dalam urutan spesifik yang benar pada suhu yang sesuai, maka molekul ini secara spontan menyusun diri kembali membentuk subunit 30S yang identik dalam struktur dan aktivitasnya dengan subunit 30S yang asli (Lehninger, 1982).
Polymerase Chain Reaction (PCR)
PCR atau reaksi polimerisasi berantai adalah teknik amplifikasi DNA yang spesifik dengan melakukan proses pemanjangan nukleotida dari primer yang merupakan pasangan komplementer dari utas DNA secara simultan. Proses pemanjangan nukleotida merupakan proses polimerisasi yang dilakukan oleh DNA polimerase berdasarkan atas sampel DNA (DNA template). Proses pemanjangan terjadi karena adanya primer. PCR dapat mengamplifikasi sampai sejuta kali sehingga dapat menghasilkan DNA dalam jumlah besar dan DNA
denaturasi, tahap penempelan primer pada DNA template(annealing) serta tahap pemanjangan primer melalui reaksi polimerisasi nukleotida (extention).
1. Denaturasi
Tahap denaturasi merupakan tahap dimana DNA utas ganda dipisahkan menjadi utas tunggal. Dalam keadaan ini masing-masing untai dapat mencetak pasangannya. Tahap ini berlangsung pada suhu 90-95oC.
2. Penempelan primer
Tahap kedua adalah menurunkan suhu reaksi agar primer dapat menempel pada utas DNA cetakan yang telah terdenaturasi menjadi utas tunggal. Penempelan terjadi karena adanya kecocokan pasangan basa. Umumnya penempelan terjadi pada suhu 55-57oC untuk primer 20 mer dan 34-40oC untuk primer 10 mer (Uphoff & Wreeke, 1992). Sambrook et al. (1989) menyatakan bahwa suhu penempelan primer yang ideal umumnya adalah 5oC di bawah suhu leleh (Tm) dari tiap primer.
3. Pemanjangan DNA
Setelah primer menempel pada utas tunggal DNA template, maka DNA polimerase akan mensintesis DNA yang baru berdasarkan utas DNA cetakan. Sintesis DNA ini dilakukan pada suhu cukup tinggi yaitu sekitar 72oC pada saat enzim Taq DNA polimerase bekerja optimal.
Reaksi PCR merupakan reaksi amplifikasi DNA dimana teknik pelaksanaannya harus teliti dan cermat. Kegagalan reaksi PCR dapat disebabkan karena tidak sempurnanya denaturasi atau suhu annealing yang terlalu tinggi. Selain itu reaksi PCR juga dipengaruhi oleh beberapa faktor diantaranya:
1. Konsentrasi DNA template
2. Pemicu reaksi (primer)
Primer adalah rantai utas tunggal DNA yang pendek dan terdiri dari beberapa nukleotida primer yang umumnya dipakai, terdiri atas 10-25 nukleotida (oligonukleotida), sedangkan primer yang biasa digunakan dalam percobaan adalah primer acak dan primer spesifik. Primer acak adalah primer yang susunan basa nukleotida seimbang sehingga primer ini biasanya digunakan untuk analisis DNA dengan sampel yang belum diketahui susunan nukleotidanya, sedangkan primer spesifik adalah primer yang susunan basanya telah diketahui merupakan komponen dari utas DNA yang akan dianalisis.
3. Enzim Taq DNA Polimerase
Dalam proses replikasi DNA diperlukan adanya enzim untuk polimerisasi jalinan DNA. Enzim yang mampu mengkatalis replikasi DNA disebut DNA polimerase. DNA polimerase yang biasa digunakan adalah Taq. DNA polimerase bersifat termostabil yang didapatkan dari bakteri termofilik
Thermus aquaticus yang dapat bertahan pada suhu 94oC. Enzim ini bekerja secara optimum pada suhu 75-80oC (Sambrook et al. 1989). Taq DNA polimerase ini digunakan untuk membantu amplifikasi potongan primer dan proses pemanjangan DNA dan karena Taq DNA polimerase tetap stabil pada suhu 94oC, maka ketika berlangsung proses denaturasi, enzim ini tidak menjadi rusak tetapi dalam keadaan tidak aktif. Aktivitas enzim ini akan terhambat oleh bufer fosfat tetapi akan aktif bila ditambahkan 10 mM tris dalam bufer pada temperatur ruang dengan pH 8,3 (Sambrook et al. 1989). Taq DNA polimerase mulai aktif pada pH 8,2 – 9 dan suhu 65 – 72oC.
4. dNTP
5. Mg2+
Mg2+ mempengaruhi aktivitas enzim Taq DNA polimerase karena ion Mg2+ berfungsi sebagai kofaktor yang dapat membentuk kelat dengan larutan EDTA. Mg2+ berperan dalam kestabilan primer pada tahap penempelan primer.
6. Bufer PCR
Bufer PCR terdiri atas larutan Tris-HCl dengan konsentrasi 10-50 mM dan pH 8.3 – 8.8 dan berperan dalam keberhasilan proses amplifikasi (Innis dan Gelfand, 1990). Untuk membantu proses penempelan primer pada bufer PCR dapat ditambahkan KCl dengan konsentrasi sampai 50 mM.
Perunutan Basa Nukleotida (Sekuensing)
Sekuensing DNA adalah proses penentuan urutan basa suatu DNA. Pada proses ini digunakan prinsip reaksi polimerisasi DNA secara enzimatis. Reaksi yang dilakukan secara in vitro ini dikembangkan oleh Sanger dengan memasukkan satu nukleotida ddNTP (dideoksi nukleosidatrifosfat) yang berbeda ke dalam masing-masing 4 reaksi untuk menghentikan reaksi polimerisasi. Teknik dideoksi sekuensing hanya mampu membaca dengan teliti urutan basa sepanjang 400 sampai dengan 500 bp (Brown 1992).
Sekuen DNA adalah informasi penting untuk mengetahui identitas, fungsi, dan modifikasi suatu fragmen DNA atau gen dalam rekayasa genetik atau bioteknologi secara umum. Sebagai contoh, dengan mengetahui sekuen suatu DNA target kita dapat menyisipkan atau menghilangkan daerah restriksi tertentu sehingga potong-sambung DNA dapat dilakukan sesuai dengan keinginan.
Analisis Keragaman Genetik
Informasi genetik dari sel prokariot dan eukariot saat ini telah banyak diketahui. Suatu organisme telah dapat diuji berdasarkan organisasi genomnya, ekspresi global gen, dan struktur serta fungsi dari seluruh proteinnya. Bioinformatika dapat didefinisikan sebagai cabang komputasi dari biologi molekuler, yang mencakup teknologi pengumpulan, penyimpanan, analisis, interpretasi, penyebaran, dan aplikasi dari informasi biologi. Internet dan server world wide web (www) merupakan dua hal yang sangat diperlukan dalam aplikasi bioinformatika. Bioinformatika menggunakan program komputer untuk analisis data biologi dan penyimpanan sejumlah data biologi yang dihasilkan oleh proyek genom. Bioinformatika banyak berhubungan dengan sekuen, struktur, fungsi, dan perbandingan seluruh genom dan struktur 3 dimensi protein, serta manajemen data (Claverie & Notredame,2007).
Beberapa program komputer dan database untuk bioinformatika yang dapat digunakan dari internet antara lain : GeneMArk, NCBI (National Center for Biotechnology Information), Expasy, dan lain sebagainya. Pada NCBI dapat diakses program PubMed, Entrez, BLAST, Blankit, OMIM, Taxonomy, dan penelusuran struktur. Salah satu program yang umum digunakan adalah BLAST (Basic Local Alignment Search Tool) yang merupakan program untuk pencarian kesamaan yang didesain dalam mengeksplorasi semua database sekuen yang diminta, baik berupa DNA ataupun protein. Program BLAST juga dapat digunakan untuk mendeteksi hubungan antar sekuen yang hanya berbagi daerah tertentu yang memiliki kesamaan (Claverie & Notredame,2007).
Ada beberapa variasi BLAST yang masing-masing dibedakan dari tipe sekuen (DNA atau protein) yang dicari dengan sekuen pada database. Berikut ini beberapa jenis program BLAST (Claverie & Notredame, 2007):
BLASTP : membandingkan sekuen asam amino dengan sekuen protein dalam database
BLASTN : membandingkan sekuen nukleotida dengan sekuen nuklotida dalam database
Secara garis besar, orang menggunakan bioinformatika melalui beberapa tahapan, diantaranya menggunakan PubMed untuk mencari pengetahuan tentang subjek biologi secara cepat, mendapatkan sekuen protein maupun DNA yang relevan, membandingkan sekuen protein maupun DNA yang tersedia di database
menggunakan BLAST, dan melakukan analisis multiple alignment sekuen protein maupun DNA dengan ClustaIW serta membangun pohon filogenetik (Claverie & Notredame, 2007).
BLAST merupakan alat pembanding suatu sekuen yang dicari dengan sekuen yang telah diketahui dengan cepat yang dapat menjelaskan apakah sekuen tersebut memiliki similaritas cukup signifikan. Informasi ini dapat digunakan untuk bermacam-macam tujuan yaitu meliputi perkiraan fungsi protein, struktur tiga dimensi, dan organisasi domain atau identifikasi homologi dengan organisme lain. Sekuen yang serupa sering diperoleh dari sekuen keturunan nenek moyang yang sama. Bila ditemukan sekuen serupa mungkin memiliki nenek moyang yang sama, bagian struktur yang sama, dan memiliki fungsi biologi serupa. Prinsip ini bahkan bekerja ketika sekuen berasal dari organisme yang sangat berbeda (Claverie & Notredame, 2007).
Hasil BLAST meliputi tiga bagian yang berbeda yaitu grafik yang menunjukkan bagaimana porsi similaritas sekuen yang dibandingkan, daftar hits yang berisi nama sekuen yang serupa dengan yang dicari urut berdasarkan similaritas dan penjajaran (alignment) antara sekuen yang dicari dengan sekuen yang ada pada database (Claverie & Notredame, 2007).
keragaman makhluk hidup. Prinsip filogeni adalah mencoba mengelompokkan makhluk hidup menurut tingkat similaritas (Claverie & Notredame, 2007).
Analisis Neighbor-Joining
Neighbor-Joining merupakan salah satu media pengelompokan dalam bioinformatika yang dapat membentuk suatu pohon filogeni atau dendogram.
Neighbor-joining biasanya digunakan pada pohon filogeni yang berdasarkan pada data sekuen DNA atau protein. Metode ini menggunakan algoritma yang membutuhkan pengetahuan mengenai jarak antara tiap pasang taxa (spesies atau sekuen) pada pohon filogeni.
Neighbor-joining merupakan algoritma yang berulang-ulang. Tiap tahap pengulangan terdiri atas hal-hal berikut ini:
1. Berdasarkan jarak langsung matriks dengan menghitung matriks Q. Matriks Q merupakan suatu matriks yang dibuat berdasarkan pada matriks jarak yang relatif terhadap r taxa.
2. Menemukan pasangan taxa pada matriks Q dengan nilai yang paling rendah. Menciptakan simpul pada pohon filogeni yang terdiri atas dua taxa (mengikutkan dua kekerabatan terdekat)
3. Menghitung jarak antar taxa pada pasangan taxa di simpul yang baru 4. Menghitung jarak seluruh taxa diluar pasangan taxa di simpul yang baru
5. Mengulang kembali algoritma, dengan mempertimbangkan pasangan yang berkerabat dekat (joined neighbors) sebagai taxon tunggal dan menggunakan penghitungan jarak seperti yang dilakukan sebelumnya.
Keragaman Genetik E.sakazakii
Berdasarkan perbandingan sekuen gen 16S rRNA, Iversen et al. (2004) menyatakan bahwa tipe galur E.sakazakii lebih dekat sebagai Citrobacter koseri
(similaritas sebesar 97.8%) dibandingkan dengan Enterobacter lainnya, meskipun E.sakazakii memiliki similaritas yang cukup besar juga dengan
E.cloacae dan C.freundii yakni berturut-turut sebesar 97.0% dan 96.0%. Namun Iversen et al. (2004) menyatakan bahwa diperlukan studi lebih lanjut untuk memperjelas hubungan kekerabatan bakteri ini.
Berdasarkan analisis 16S rRNA-nya, galur E.sakazakii dapat dibagi menjadi 4 kelompok (cluster). Cluster pertama yang merupakan mayoritas dari galur-galur E.sakazakii (110 galur) dimana memiliki perbedaan antar galur sebesar 0.1 hingga 1.2%. Kelompok 1 ini meliputi 17 galur klinis dan 3 galur yang tidak berpigmen kuning. Sebanyak 9 galur (termasuk 1 galur klinis) memiliki keragaman sekuen sebesar 1.6 hingga 1.9% dan membentuk suatu
cluster kedua yang dekat dengan E.sakazakii. Cluster ketiga yang terdiri dari 5 galur (termasuk 1 galur klinis), dimana pada pengujian biokimia dengan API20 E dan ID 32E lebih memiliki kedekatan (97.5% hingga 97.8%) sebagai
Enterobacter pyrinus, Enterobacter hormaechei, dan C.koseri. Cluster ketiga ini memiliki keragaman sekuen sebesar 3.0% dan isolat E.sakazakii ATCC 51329 termasuk ke dalam cluster ini. Cluster yang keempat terdiri dari dua galur, dimana satu diantaranya adalah isolat klinis. Galur ini teridentifikasi sebagai
E.sakazakii dengan menggunakan perangklat biokimia API 20E dan ID 32E, namun similaritas 16S rRNA nya hanya sebesar 96.5% sebagai E.sakazakii. Analisis sekuen 16S rRNA tersebut mengkonfirmasi bahwa beberapa galur yang teridentifikasi sebagai E.sakazakii, bila dianalisis dengan perangkat biokimia komersial dapat dibedakan sebagai spesies yang berbeda, yaitu sebagai
E.amnigenus atau E.cloacae, dimana hasil ini berpotensi untuk membagi
E.sakazakii ke dalam taxa yang berbeda (Iversen et al. (2004).
Berdasarkan fenomena tersebut, Iversen et al. (2008a) memperkenalkan
sp.nov., C.dublinensis sp. nov., dan C.genomospecies I. Pembedaan spesies
E.sakazakii sebagai genus Cronobacter ini berdasarkan pada perbedaan reaksi-reaksi biokimia pada perangkat API 20E dan ID 32E, serta berdasarkan reaksi-reaksi pada methyl-α-D-glucopyranoside.
METODOLOGI
Tempat dan Waktu Penelitian
Penelitian ini dilakukan di Laboratorium SEAFAST (South East Asia for Food and Agricultural Science and Technology) Center, Kampus IPB Darmaga, Bogor dan Laboratorium Biokimia Pangan dan Mikrobiologi Pangan Departemen ITP, Fakultas Teknologi Pertanian, Institut Pertanian Bogor. Penelitian ini dilaksanakan mulai Agustus 2006 sampai Februari 2008.
Alat dan Bahan
Bahan yang digunakan adalah 25 kemasan susu formula dan makanan bayi berbagai merk yang dibeli dari supermarket atau toko di wilayah Bogor. Media untuk isolasi E.sakazakii yang digunakan antara lain; Bufered- Peptone Water (BPW) (CM 509 Oxoid Ltd., Basingstoke, UK); Enterobacter enrichment
(EE) broth (CM 317 Oxoid Ltd.,UK) ; Violet Red Bile Glucose (VRBG) Agar (CM 485 Oxoid Ltd.,UK); Druggan-Forsythe-Iversen (DFI) Agar (Oxoid,UK);
Trypticase (Tryptic) Soy Agar (TSA) (CM 131 Oxoid Ltd.,UK); serta API 20E
Biochemical Strips (bioMĕrieux, Perancis).
Beberapa bahan yang digunakan untuk isolasi DNA antara lain media pertumbuhan bakteri yaitu Nutrient Broth (NB), bahan-bahan untuk ekstraksi DNA antara lain Sodium dodecyl sulphate (SDS) (Merck, Darmstadt, Germany), proteinase K (AppliChem, Darmstadt, Germany), Cethyiltrimethyl ammonium
bromide (CTAB) (Merck, Darmstadt, Germany), natrium chloride (NaCl) (Merck, Darmstadt, Germany); Larutan TE yang terdiri dari Tris (hydroxymethyl )-amino methan (Tris) (Amersham Bioscience, Sweden) dan Di natrium ethylene tetra acetat (Na2-EDTA) (Amersham Bioscience, Sweden); phenol, chloroform,
dan bufer berdasarkan metode Sambrook et al. (1989) dapat dilihat pada Lampiran 2.
Bahan-bahan untuk amplifikasi gen 16S-rRNA antara lain PCR Master Mix (Fermentas) yang terdiri dari 0.05 U/µl Taq DNA polimerase, PCR bufer, MgCl2, dan dNTP (masing-masing 0.4 mM dATP, dCTP, dGTP, dan dTTP);
akuabides steril; DNA cetakan, primer, dan akuabides steril. Primer yang digunakan merupakan primer untuk analisis gen 16S rRNA E.sakazakii (Hassan
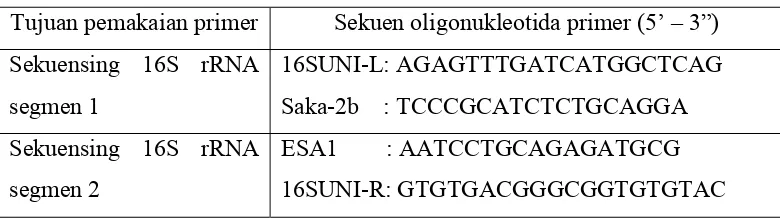
et al. 2007)antara lain; 16 SUNI-L (AGAGTTTGATCATGGCTCAG) dan Saka-2b (TCCCGCATCTCTGCAGGA) untuk megamplifikasi segmen pertama yang berukuran 977 bp. Untuk amplifikasi segmen berikutnya (408 bp), digunakan primer ESA-1 (AATCCTGCAGAGATGCG) dan 16SUNI-R (GTGTGACGGGCGGTGTGTAC). Primer dipesan dari Alpha DNA (Notre-Dame St.W., Montreal, Quebec). Urut-urutan basa primer dapat dilihat pada Tabel 2.
Tabel 2. Pasangan primer oligonukleotida yang digunakan
Tujuan pemakaian primer Sekuen oligonukleotida primer (5’ – 3”) Sekuensing 16S rRNA
segmen 1
16SUNI-L: AGAGTTTGATCATGGCTCAG Saka-2b : TCCCGCATCTCTGCAGGA Sekuensing 16S rRNA
segmen 2
ESA1 : AATCCTGCAGAGATGCG 16SUNI-R: GTGTGACGGGCGGTGTGTAC
Alat-alat utama yang digunakan pada penelitian ini adalah penangas air yang bertutup, termometer, inkubator 37oC, pipet volumetrik 1, 5, dan 10 ml; pipet mikro berikut tip 1 ml, 0.1 ml, dan 2 – 20 µl; 0.2 -2 µl; batang gelas pengaduk; jarum ose; labu takar 50 ml, 500 ml, dan 1000 ml; erlenmeyer berukuran 250 ml, 125 ml, dan 500 ml; tabung reaksi bertutup, gelas ukur, cawan petri, plastik steril, pH meter, penangas air, sentrifusi (berkekuatan sampai 18.000 rpm), pengaduk magnet, wadah gelas tertutup, oven, vorteks, freeze dryer, perangkat elektroforesis (Bio-Rad), perangkat PCR Applied Biosystem 2720
Beberapa software juga digunakan pada penelitian ini antara lain apiweb™ (Biomeireux) untuk menganalisis hasil uji biokimia dengan API 20E, Program BLAST (Basic Local Alignment Search Tool) dari situs NCBI (www.ncbi.nih.nlm.gov) untuk menganalisis hasil sekuensing, serta program MEGA4 (www.megasoftware.net) untuk membentuk pohon filogeni.
Pelaksanaan Penelitian
Tahapan penelitian yang akan dilakukan meliputi (1) isolasi E.sakazakii
dari susu formula dan makanan bayi, (2) karakterisasi sifat fenotipik isolat berdasarkan identifikasi isolat secara biokimiawi, (3) karakterisasi sifat genotipik isolat dengan membandingkan hasil amplifikasi gen 16S rRNA isolat serta sekuen parsialnya dengan sekuen-sekuen parsial E.sakazakii yang ada di GenBank (situs NCBI), termasuk sekuen parsial 16S rRNA isolat Estuningsih et al. (2006), yang sekaligus merupakan konfirmasi terhadap hasil analisis fenotipik isolat, (4) karakterisasi survival isolat setelah direkonstitusi dengan air pada berbagai suhu.
Isolasi Enterobacter sakazakiidari Susu Formula dan Makanan Bayi
Metode isolasi E.sakazakii yang digunakan merupakan metode FDA (2002) yang telah dimodifikasi oleh Iversen dan Forsythe (2004). Pengujian awal yang dilakukan adalah tahap enrichment agar jumlah yang sangat kecil dari bakteri ini dapat dideteksi. Wadah dan sendok yang digunakan untuk mengambil sampel harus dalam kondisi steril. Triplikat sampel masing-masing sebanyak 25 g susu formula ditimbang secara aseptis dan dimasukkan ke dalam gelas Erlenmeyer berukuran 250 ml. Ke dalam Erlenmeyer ditambahkan sembilan bagian (larutan 1:10) BPW steril, dipanaskan hingga 45oC, kemudian diaduk perlahan hingga bubuk tersuspensi secara seragam. Suspensi tersebut kemudian diinkubasi pada 37oC selama 24 jam.
kuadran goresan untuk mendapatkan koloni terpisah. Penggoresan untuk masing-masing isolat dilakukan sebanyak duplo. Kemudian masing-masing-masing-masing cawan diinkubasikan pada 37oC selama 24 jam. Cawan yang positif mengandung
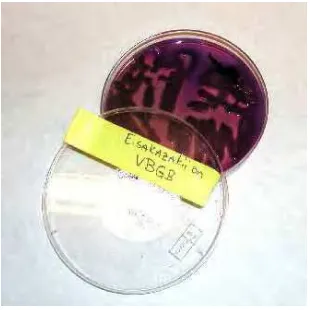
E.sakazakii pada VRBG memperlihatkan koloni berbentuk gumpalan garam bile
seperti dapat dilihat pada Gambar 2 berikut.
Gambar 2. E.sakazakii yang positif pada VRBG (Sharon Edelson Mammel)
Dari total 5 koloni presumtif E.sakazakii pada VRBG masing-masing diisolasi kembali dengan menggoreskan pada cawan yang berisi DFI Agar yang kemudian diinkubasi pada 37oC selama 24 jam. Koloni positif pada DFI berwarna hijau-biru seperti dapat dilihat pada Gambar 3. Koloni positif pada DFI kemudian digoreskan kembali pada media TSA dan diinkubasi pada 37oC selama 48 – 72 jam. Koloni positif pada TSA merupakan koloni yang berpigmen kuning seperti yang dapat dilihat pada Gambar 4.
Gambar 4. Koloni positif E.sakazakii pada TSA
Pada masing-masing isolat E.sakazakii yang diperoleh, sebagian diuji lebih lanjut dan sebagian lainnya disimpan dalam bentuk kultur stok dengan menambahkan gliserol dan disimpan di suhu freezer (0 hingga -20 oC). Diagram alir isolasi E.sakazakii dapat dilihat pada Lampiran 1.
Isolat E.sakazakii yang diperoleh juga diamati secara morfologi di bawah mikroskop dengan proses pewarnaan Gram untuk memastikan bahwa isolat yang diperoleh sudah homogen. Pengamatan mikroskopik dilakukan dengan pembesaran 1000x.
Karakterisasi Sifat Fenotipik Isolat
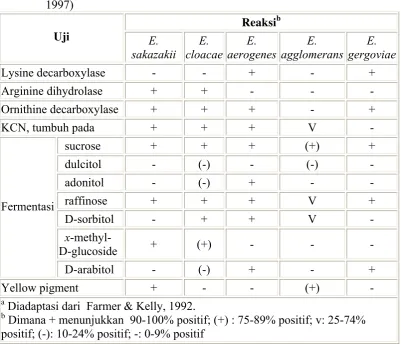
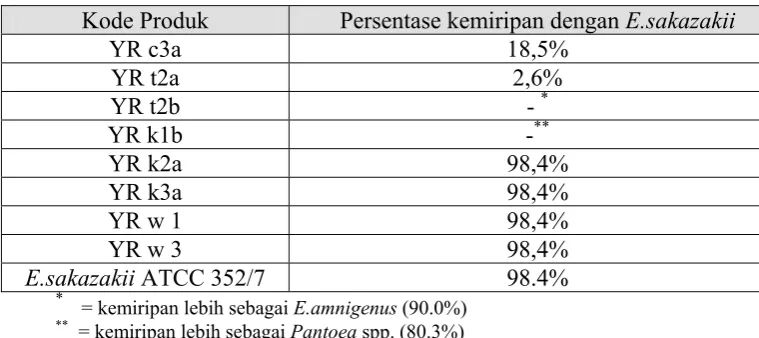
Setelah diinkubasi pada wadah yang telah diberi suasana lembab selama 18 – 24 jam pada 37oC, reaksi-reaksi warna pada masing-masing sumur dibaca. Beberapa sumur dibaca setelah ditambahkan reagen terlebih dahulu. Hasil pembacaan positif atau negatif kemudian diinterpretasikan dengan menggunakan perangkat lunak (software) apiweb™ sehingga dapat dihitung persentase kemiripan isolat yang memiliki koloni tipikal pada DFI dengan E.sakazakii secara biokimiawi. Sifat biokimia dari spesies Enterobacter dapat dilihat pada Tabel 3.
Tabel 3. Sifat biokimia dari spesies Enterobactera (Nazarowec-White dan Farber; 1997)
Diadaptasi dari Farmer & Kelly, 1992.
b
Dimana + menunjukkan 90-100% positif; (+) : 75-89% positif; v: 25-74% positif; (-): 10-24% positif; -: 0-9% positif
dan arabinosa. Fermentasi ditunjukkan dengan reaksi asam (warna kuning pada indikator). Produksi Hidrogen disulfida (H2S) dan hidrolisis gelatin (GEL) terlihat dari terbentuknya warna hitam di seluruh sumur. Reaksi positif untuk triptopan deaminase (TDA) ditunjukkan dengan warna coklat kemerahan bila ditambahkan perak klorida; dimana hasil yang positif berkorelasi dengan reaksi lisin dan fenilalanin deaminase yang merupakan karakteristik dari Proteus, Morganella, dan Providencia (Lindquist, 2001). Untuk dapat memunculkan angka persentase kemiripan, analisis tambahan seperti uji oksidase (OX), reaksi terhadap NO2, N2, motilitas (MOB), kemampuan tumbuh pada Mc Conkey agar
(McC), oksidasi glukosa (OF-O), dan fermentasi glukosa (OF-F) ditentukan berdarkan sifat E.sakazakii pada literatur (Nazarowec-White & Farber 1997; FDA, 2002).
Analisis Keragaman Genetik E.sakazakii
Isolasi dan Pemurnian DNA Genom. Untuk analisis keragaman genetik ini sebagai pembanding digunakan isolat E.sakazakii ATCC 352/7, dan 7 isolat hasil isolasi dari beberapa susu formula yang dilakukan oleh Estuningsih et al. (2006). Masing-masing isolat ditumbuhkan pada media NB selama 48 jam. Sel bakteri dipanen dengan cara melakukan sentrifugasi kultur biakan bakteri (2 ml) pada 18000 rpm selama 3 menit. Pelet yang diperoleh diresuspensi dalam 200 µl bufer TE dengan menggunakan vorteks, kemudian ditambahkan 50 µl SDS 10% dan dicampur dengan hati-hati dengan cara membalikkan tabung beberapa kali sampai suspensi terlihat jernih. Sejumlah 10 µl proteinase-K (10 mg/ml) ditambahkan kemudian diinkubasi pada 37oC selama 1 jam. Setelah 1 jam, ditambahkan 80 µl CTAB/NaCl (10% CTAB dalam 0.7 M NaCl) kemudian inkubasi pada 65oC selama 20 menit.
(24:1) dengan volume yang sama. Pencampuran dilakukan dengan membolak-balik tabung beberapa kali, kemudian disentrifugasi kembali pada 13500 rpm selama 10 menit hingga diperoleh kembali fase terpisah. Fase cairan (top layer) dipindahkan kembali ke tabung eppendorf 1.5 ml yang baru. Kemudian ditambahkan 0.1 volume sodium asetat (3M) dan isopropanol dengan volume yang sama dan dilakukan pencampuran dengan membolak-balikkan tabung beberapa kali. Tabung diinkubasi pada –20oC selama 1 jam atau pada -80o C selama 15 menit, kemudian presipitasi DNA dilakukan dengan sentrifugasi pada 13500 rpm selama 10 menit.
Supernatan hasil sentrifugasi dibuang, kemudian ditambahkan 500 µl etanol (70%) dan tabung dibolak-balikkan beberapa kali. Setelah itu dilakukan sentrifugasi kembali pada 13500 rpm selama 10 menit. Pelet DNA dikeringkan dengan meletakkannya dengan kondisi tabung terbuka pada laminar air flow
selama 15 menit kemudian diresuspensi dalam100 µl TE atau akuades steril. Untuk penyimpanan jangka panjang, larutan DNA disimpan pada suhu -20oC. Verifikasi DNA dilakukan dengan elektroforesis dengan gel agarosa 1.5% pada 120 V dengan menggunakan 1x bufer TAE. Gel kemudian diwarnai dengan Ethidium Bromida (EtBr) dan divisualisasikan pada 302 nm (Geldoc XR, Bio-rad).
Visualisasi DNA dilakukan dengan mewarnai DNA yang ada pada gel dengan EtBr (0.5 µg/ml). Gel hasil elektroforesis direndam dalam larutan EtBr selama 30 menit, kemudian gel dibilas dengan akuabides maksimal selama tak lebih dari 5 menit. Setelah itu gel diamati di bawah sinar ultra violet dengan menggunakan GelDoc-XR (Bio-rad). EtBr dapat berinteraksi diantara pasangan basa pada DNA dan menangkap sinar ultra violet sehingga pendaran dari ultra violet dapat terlihat. Pita DNA dapat dilihat dengan menyinari gel dengan sinar UV pada UV transiluminator (Brown 1992).
Amplifikasi dan Sekuensing Gen 16S rRNA. Gen 16S rRNA
E.sakazakii diamplifikasi dengan menggunakan dua tahapan PCR (Polymerase Chain Reaction). Untuk mengamplifikasi segmen pertama yang berukuran 977 pasang basa (bp) digunakan primer 16 SUNI-L dan Saka-2b. Untuk amplifikasi segmen berikutnya (408 bp), digunakan primer ESA-1 dan 16SUNI-R. Primer 16SUNI-L dan 16SUNI-R (Kuhner et al., 1996) digunakan untuk basa 8-28 dan basa 1392-1411 dimana merupakan sekuen gen 16S rRNA dari Escherichia coli
(akses NCBI no. J01859).
Proses amplifikasi dengan PCR dilakukan menggunakan 50 µl campuran reaktan yang masing-masing mengandung masing-masing 1 µl primer forward
dan reverse (20 pmol/µl), 25 µl PCR Master Mix (Fermentas) yang terdiri dari 0.05 U/µl Taq DNA polimerase, PCR bufer, MgCl2, dan dNTP (masing-masing
0.4 mM dATP, dCTP, dGTP, dan dTTP); akuabides steril; DNA template yang merupakan hasil isolasi DNA genom, dan akuabides steril. Volume DNA cetakan bervariasi tergantung pada konsentrasi DNA yang diperoleh.
Protokol PCR yang digunakan adalah pre-PCR (94oC, 4 menit), denaturasi (94oC, 50 detik), penempelanprimer (57oC, 1 menit), elongasi atau pemanjangan primer (72oC, 50 detik) dan post-PCR (72o C, 4 menit) dengan siklus sebanyak 30 kali. Sebanyak 9 μl hasil PCR dielektroforesis pada gel agarosa 1.5% (w/v), dengan menggunakan bufer 1x TAE pada voltase 110.
dapat mengganggu proses perunutan basa nukleotida sehingga dapat menimbulkan kesalahan dalam pembacaan hasil sekuensing (Applied Biosystem, 2002). Gen penyandi 16S rRNA yang telah diisolasi dan dimurnikan selanjutnya di-sekuensing dengan menggunakan ABI PRISM™ 3100-Avant Genetic Analyzer dengan 4-Capillary System (Applied Biosystem), dimana tahapan ini dilakukan di PT Charoen Pokphand Indonesia. Hasil sekuensing diolah dengan program BioEdit kemudian dibandingkan dengan data sekuen yang ada di Bank Gen. Analisis sekuen dilakukan dengan menggunakan program BLAST dari NCBI (the National Center of Biotechnology Information) pada situs
http://www.ncbi.nih.gov dan program MEGA (Molecular Evolutionary Genetics Analysis)) 4 untuk membentuk pohon filogeni.
Pengaruh Suhu Air Rekonstitusi terhadap E.sakazakii
Rekonstitusi adalah proses persiapan susu formula atau makanan bayi yang berbentuk bubuk dengan cara mencampurkannya dengan air sehingga susu bubuk atau makanan bayi tersebut siap dikonsumsi. Uji pengaruh rekonstitusi terhadap keberadaan E.sakazakii dilakukan pada berbagai suhu air yakni 4oC, 40oC, 70oC, dan 100oC. Setelah rekonstitusi juga dilakukan pengujian terhadap pertumbuhan E.sakazakii selama hang time atau jeda antara rekonstitusi hingga susu dikonsumsi pada susu yang direkonstitusi dengan air bersuhu 70oC. Tahapan pada penelitian ini terdiri dari persiapan kulur kerja, simulasi rekonstitusi, dan
hang time.
Persiapan kultur kerja. Masing-masing isolat E.sakazakii yang diperoleh ditumbuhkan pada 10 ml BHI dan diinkubasi pada 37oC selama 24 jam. Isolat sel
yang berisi susu bubuk dan kultur kering beku tersebut ditutup dengan rapat. Selanjutnya dilakukan pengocokan (shaking) selama 2 menit. Sebanyak masing-masing 10 g susu yang diinokulasi dicampur dengan 90 ml media BPW. Jumlah
E.sakazakii pada susu terinokulasi ini dihitung dengan plating pada DFI sebagai jumlah susu bubuk sebelum direkonstitusi. Masing-masing cawan diinkubasi pada 37oC selama 24 jam.
Pengaruh Suhu Air Rekonstitusi dalam Penyiapan Susu Formula.
Susu skim yang telah dicampur dengan isolat E.sakazakii pada tahap persiapan direkonstitusi dengan menggunakan akuades steril dengan suhu yang berbeda-beda. Suhu rekonstitusi yang digunakan adalah 100oC, 70oC, 40oC, dan 4oC. Sebanyak 10 g susu terinokulasi direkonstitusi dengan akuades steril sehingga volume larutan adalah 100 ml. Masing-masing tahapan rekonstitusi dienumerasi dengan menggunakan media DFI.
HASIL DAN PEMBAHASAN
Isolasi Enterobacter sakazakiidari Susu Formula dan Makanan Bayi
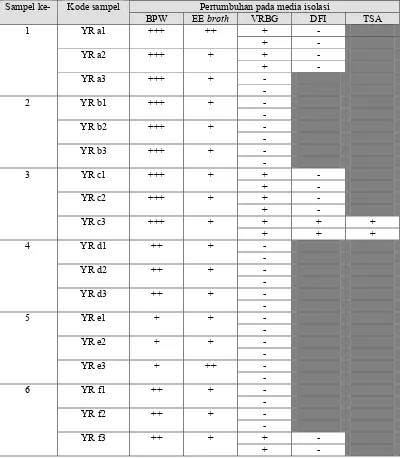
Isolasi E.sakazakii dilakukan dari beberapa sampel susu formula dan makanan bayi yang ditujukan untuk bayi yang berusia di bawah 6 bulan. Penggunaan sampel yang ditujukan untuk bayi di bawah 6 bulan dikarenakan bayi pada usia ini merupakan kelompok beresiko tinggi terhadap E.sakazakii. Hasil isolasi bakteri ini dapat dilihat pada Tabel 4.
Tabel 4. Hasil isolasi E.sakazakii pada beberapa media Pertumbuhan pada media isolasi Sampel ke- Kode sampel
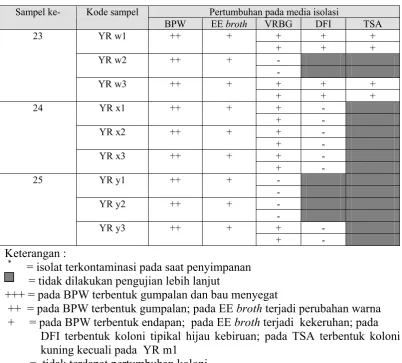
Tabel 4. Hasil isolasi E.sakazakii pada beberapa media (lanjutan)
Pertumbuhan pada media isolasi Sampel ke- Kode sampel
Tabel 4. Hasil isolasi E.sakazakii pada beberapa media (lanjutan)
Pertumbuhan pada media isolasi Sampel ke- Kode sampel