KERAGAMAN GENETIK TARUM (
Indigofera tinctoria
L.)
DI PULAU JAWA DAN MADURA SEBAGAI PEWARNA
ALAMI BATIK BERDASARKAN MARKA
INTER-SIMPLE SEQUENCE REPEATS
MUHAMMAD RIFQI HARIRI
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa tesis berjudul Keragaman Genetik Tarum (Indigofera tinctoria L.) di pulau Jawa dan Madura sebagai Pewarna Alami Batik berdasarkan Marka Inter-Simple Sequence Repeats adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Februari 2016
Muhammad Rifqi Hariri
RINGKASAN
MUHAMMAD RIFQI HARIRI. Keragaman genetik tarum (Indigofera tinctoria
L.) di pulau Jawa dan Madura sebagai pewarna alami batik berdasarkan marka
inter-simpe sequence repeats. Dibimbing oleh TATIK CHIKMAWATI dan ALEX HARTANA.
Tarum (Indigofera tinctoria L.) merupakan anggota dari suku Fabaceae, yang dimanfaatkan sebagai pewarna indigo alami. Saat ini, tarum telah banyak dimanfaatkan kembali sebagai bahan pewarna alami, baik untuk tekstil maupun kosmetik. Penelitian mengenai marga Indigofera di Indonesia masih terbatas pada eksplorasi jenis dan pemanfaatannya untuk peningkatan produksi pakan ternak maupun kualitas warna pada tekstil. Akan tetapi, penelitian mengenai kajian keragaman genetik marga Indigofera, khususnya jenis I. tinctoria L. belum pernah dilakukan. Penelitian ini dilakukan untuk mempelajari keragaman genetik I. tinctoria L. di pulau Jawa dan Madura sebagai pewarna alami batik berdasarkan marka Inter-Simple Sequence Repeats.
Penelitian ini dilaksanakan pada bulan April 2014-April 2015. Pengambilan sampel dilakukan di 6 lokasi yang tersebar di pulau Jawa (Cirebon, Serang, Bantul, Gunung Kidul, Kulonprogo, dan Tuban) dan 4 lokasi di pulau Madura (Bangkalan, Sampang, Pamekasan, dan Sumenep). Bahan tumbuhan yang digunakan adalah daun segar dan sehat yang dimasukkan ke dalam plastik berisi silika gel. DNA diisolasi berdasarkan metode CTAB, kemudian diamplifikasi menggunakan GoTaq® Green PCR Mix dengan 15 primer ISSR. Produk hasil PCR dielektroforesis dan difoto menggunakan GelDoc. Pita polimorfik diskor sebagai data biner. Data disusun dalam matriks dan digunakan untuk menghitung koefisien kemiripan simple matching, mengontruksi PCA, dan dendrogram dengan metode UPGMA menggunakan NTSys-PC versi 2.1.1a. Analisis struktur populasi diakukan menggunakan GenAlex versi 6.501.
Kelima belas primer yang digunakan menghasilkan 123 pita dan 84 pita diantaranya bersifat polimorfik. Panjang pita yang dihasilkan berkisar 250-2500 pasang basa dan persentase polimorfisme pita berkisar 50-90% dengan rata-rata 68.3%. Pola pita yang dihasilkan bersifat unik sehingga dapat digunakan untuk pembeda antar aksesi.
Analisis gugus mengelompokkan 50 aksesi tarumke dalam empat kelompok berdasarkan 84 pita polimorfik yakni Kelompok I (Bangkalan, Sampang, Pamekasan, dan Sumenep), II (Tuban), III (Bantul, Gunung Kidul, dan Kulonprogo), dan IV (Cirebon dan Serang). Keragaman genetik tarum di pulau Jawa dan Madura tergolong dalam kategori yang tinggi (68.3%), tetapi variasi molekuler di dalam populasi (37%) lebih kecil dari antar populasi (63%). Nilai keragaman (I) paling tinggi dimiliki oleh populasi yang berasal dari Bantul dan Tuban (I=0.35) sedangkan paling rendah dimiliki oleh populasi yang berasal dari Serang (I=0.06). Berdasarkan hasil yang diperoleh, primer ISSR tidak hanya dapat digunakan untuk mempelajari keragaman genetik, tetapi juga dapat digunakan untuk membedakan setiap aksesi tarum (I. tinctoria L.) di pulau Jawa dan Madura. Kata kunci: Indigofera tinctoria, Jawa dan Madura, keragaman genetik, marka
SUMMARY
MUHAMMAD RIFQI HARIRI. Genetic diversity of I. tinctoria L. in Java and Madura islands as natural batik dye based on Inter-simple sequence repeats markers. Supervised by TATIK CHIKMAWATI and ALEX HARTANA.
Indigo (Indigofera tinctoria L.) is a member of Fabaceae family that can be utilized as natural indigo dye. Indigo has been frequently used for natural dye, both for textile or cosmetics. Previous research on Indigofera in Indonesia was conducted on species exploration and utilization as animal feed or improvement on dye quality. However, genetic diversity assessment on
Indigofera, especially I. tinctoria L. has not been conducted yet. The aim of this research was to determine the genetic diversity and population structure of I. tinctoria L. in Java and Madura islands as natural batik dye based on inter-simple sequence repeats (ISSR) markers.
This research was conducted from April 2014 to April 2015. The samples were collected from Java and Madura island, from 6 locations representing Java island (Cirebon, Serang, Bantul, Gunung Kidul, Kulonprogo, and Tuban) and 4 locations representing Madura island (Bangkalan, Sampang, Pamekasan, and Sumenep). Fresh and healthy leaves were collected in plastic bag containing silica gel. The total DNA was extracted from leaves based on CTAB method. PCR amplification used GoTaq® Green PCR Mix and 15 ISSR primers. The PCR product was observed using electrophoresis and photographed using GelDoc. The polymorphic bands were scored into binary data and compiled in a data matrix to be used to calculate simple matching similarity coefficient, construct dendrogram and principle component analysis using UPGMA method. The population structure was analyzed using GenAlex 6.501.
All ISSR primers produced a total of 123 bands and 84 of them were polymorphic. The bands length varied from 250 to 2500 bp and the percentage of polymorphic bands was 50-90% with average about 68.3% for every primer. Band patterns generated from PCR were unique and could be used for identifying each accession.
The clustering analysis di vi ded 50 indigo accessions into 4 groups based 84 polymorphic bands. Group I grouped all accessions from Madura (Bangkalan, Sampang, Pamekasan, and Sumenep), group II only consisted of accessions from East Java (Tuban), group III consisted of accessions from Central Java (Bantul, Gunung Kidul, and Kulonprogo), and group IV grouped all accessions from West Java (Cirebon and Serang). The indigo genetic diversity in Java and Madura islands was relatively high (%P=68.3), but the molecular variation within population (37%) was lower than among population (63%). The highest diversity (I) value was from Bantul and tuban population (I=0.35), while the lowest was from Serang (I=0.06). Based on the result, ISSR primers were not only able to assess genetic diversity but also identify each indigo accession in Java and Madura islands.
© Hak Cipta Milik IPB, Tahun 2016
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Biologi Tumbuhan
KERAGAMAN GENETIK TARUM (
Indigofera tinctoria
L.)
DI PULAU JAWA DAN MADURA SEBAGAI PEWARNA
ALAMI BATIK BERDASARKAN MARKA
INTER-SIMPLE SEQUENCE REPEATS
MUHAMMAD RIFQI HARIRI
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
11%4. .4 4 ,')4 )/!$4,1'4
#!!#
41%14 24)41-4. 42(4&' 40"$4,.,$)4,$4
!#"#! #
'4 4
4 1''4 + 4 , , 4
,4,40 $4 4
014
014 ,*'401 4 *%* 4 1'1)4
,4,4 4
)%4# )4
.',4
.01#1 4 *&4
*' . 4 ' ' )4
$01 4*%4
,*4,4,4%34 4
)*04
PRAKATA
Puji dan syukur penulis ucapkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan April 2014 ini yaitu Keragaman Genetik Tarum (Indigofera tinctoria L.) di pulau Jawa dan Madura sebagai Pewarna Alami Batik berdasarkan Marka Inter-Simple Sequence Repeats.
Penelitian ini didanai oleh Dana Hibah Penelitian PUPT nomor kontrak 339/UN27.11/PL/2015 atas nama Dra. Muzzazinah, M.Si.
Terima kasih penulis ucapkan kepada Dr. Ir. Tatik Chikmawati, M.Si dan Prof. Dr. Ir. Alex Hartana, M.Sc selaku dosen pembimbing yang telah banyak memberikan nasihat, motivasi, saran, serta bimbingan. Penulis juga mengucapkan terima kasih kepada bapak dan ibu pengajar di program studi Biologi Tumbuhan (BOT) atas semua ilmu, pengalaman, bimbingan, dan nasihat selama ini, Direktorat Jenderal Pendidikan Tinggi (DIKTI) atas Beasiswa Program Pascasarjana Dalam Negeri (BPPDN) tahun 2013, Laboratorium Taksonomi Tumbuhan, Laboratorium Terpadu, serta Laboratorium Fisiologi dan Biologi Molekuler Tumbuhan Departemen Biologi FMIPA IPB atas perizinan penelitian, serta Dra. Muzzazinah, M.Si atas penyediaan dan perizinan menggunakan bahan penelitian. Kepada Herbarium Bogoriense (BO) atas perizinannya untuk pengamatan spesimen koleksi tarum (Indigofera tinctoria L.) di Indonesia. Ungkapan terima kasih juga disampaikan kepada orang tua, seluruh keluarga dan sahabat atas segala doa, dukungan, serta kasih sayangnya.
Semoga karya ilmiah ini bermanfaat.
Bogor, Februari 2016
DAFTAR ISI
DAFTAR TABEL x
DAFTAR GAMBAR xi
1 PENDAHULUAN
Latar Belakang 1
Tujuan Penelitian 3
2 TINJAUAN PUSTAKA
Tumbuhan Tarum (Indigofera tinctoria L.) 4
Struktur Populasi 6
Marka Molekuler ISSR 8
3 METODE
Waktu dan Tempat Penelitian 11
Koleksi Sampel 11
Isolasi DNA Total 12
Amplifikasi dan Visualisasi DNA 13
Analisis Data 13
4 HASIL DAN PEMBAHASAN
Seleksi dan Profil Primer ISSR 14
Analisis Gugus Tarum (I. tinctoria L.) di pulau Jawa dan Madura 16 Keragaman Genetik Tarum (I. tinctoria L.) di pulau Jawa dan Madura 21 5 SIMPULAN DAN SARAN
Simpulan 23 Saran 23
DAFTAR PUSTAKA 24
DAFTAR TABEL
1 Identitas aksesi tarum (I. tinctoria L.), asal lokasi, dan nama populasi 12 2 Sekuen primer ISSR dan profil pita hasil amplifikasi DNA tarum (I.
tinctoria L.) di pulau Jawa dan Madura 14 3 Perbandingan aksesi dan profil pita hasil amplifikasi DNA tarum (I.
tinctoria L.) di pulau Jawa dan Madura terhadap hasil penelitian rujukan 15 4 Jarak genetik Nei 10 populasi tarum (I. tinctoria L.) di pulau Jawa dan
Madura 19 5 Pita spesifik untuk identifikasi asal populasi tarum (I. tinctoria L.) di
pulau Jawa dan Madura 20
DAFTAR GAMBAR
1 Profil tumbuhan tarum (I. tinctoria L.). a. Perawakan, b. Buah dan Biji,
c. Bunga 5
2 Reaksi pembentukan warna indigo (Glowacki et al. 2012) 6 3 Struktur Populasi dan divergensi frekuensi alel yang disebabkan oleh
terbatasnya aliran gen (Hamilton 2009) 7
4 Wilayah amplifikasi marka Inter-Simple Sequence Repeats (ISSR). Primer tunggal dengan inti (CA)n yang telah dilekatkan dengan
nukleotida tambahan pada ujung 3- (panah putih) atau ujung 5- (panah
hitam) (Zietkiewicz et al. 1994) 9
5 Lokasi pengambilan sampel tarum (I. tinctoria L.) di pulau Jawa dan Madura. A=Cirebon, B=Serang, H=Tuban, J=Kulonprogo, K=Bantul, L=Gunung Kidul, P=Sumenep, Q=Pamekasan, S=Bangkalan,
T=Sampang 11 6 Profil pita primer (AG)8T. M=DNA Ladder 1 kb, A2,3=Cirebon, B3=Serang,
H1,2=Tuban, J1,2=Kulonprogo, K1,3,8=Bantul, L1,2,3=Gunung Kidul, P2=Sumenep, Q1,3=Pamekasan, S1,6=Bangkalan, T1,3=Sampang. CB=pita
umum, SB=pita khusus 16
7 Pola pengelompokan tarum (I. tinctoria L.) berdasarkan analisis
komponen utama. □ = Bangkalan, ♯ = Bantul, ● = Cirebon, × = Gunung Kidul, + = Kulonprogo, ◊ = Pamekasan, = Sampang, ■ = Serang,
○ = Sumenep, = Tuban 17
8 Dendrogram 50 aksesi tarum (I. tinctoria L.) berdasarkan 84 pita ISSR dengan metode UPGMA. A1-6=Cirebon, B1-3=Serang, H1-3=Tuban,
J1-4=Kulonprogo, K1-9=Bantul, L1-6=Gunung Kidul, P1-4=Sumenep,
Q1-3=Pamekasan, T1-3=Sampang, S1-9=Bangkalan 18
9 Pola pita spesifik dari primer (GA)9T (di dalam kotak) untuk populasi Tuban,
Sumenep, dan Pamekasan. M=DNA Ladder, A2,3=Cirebon, B3=Serang, H1,2=Tuban, J1,2=Kulonprogo, K1,3,8=Bantul, L1,2,3=Gunung Kidul,
P2=Sumenep, Q1,3=Pamekasan, T1,3=Sampang, S1,6=Bangkalan. Pita di dalam kotak menunjukkan pita spesifik yang dapat digunakan untuk identifikasi asal populasi: Tuban (GA)9T 10 Persentase variasi molekuler di dalam dan antar populasi tarum (I.
1
PENDAHULUAN
Latar Belakang
Marga Indigofera L. merupakan kelompok tumbuhan polong dari suku
Fabaceae. Sebanyak 700 jenis Indigofera terdapat di dunia yang tersebar dan tumbuh di daerah Asia, Afrika dan Amerika. Sesuai dengan namanya, Indigofera
yang berarti penghasil indigo merupakan kelompok tumbuhan penghasil warna indigo alami meskipun tidak semua jenis di dalam marga tersebut menghasilkan pewarna (Gao & Schrire 2010). Indigofera mudah dikenali dari perawakannya yang dapat berupa pohon, perdu, atau herba dengan ciri daun tunggal atau majemuk menyirip gasal dengan anak daun tersusun berseling. Pada batang dapat ditemukan trikoma uniseluler. Daun penumpu berbentuk segitiga memanjang dengan ujung lancip atau kadang menyegitiga. Perbungaan bertipe tandan dan muncul di ketiak daun. Bunga berbentuk kupu-kupu dengan lunas yang selalu tertutup (Kort & Thijsse 1984; Gao & Schrire 2010; Muzzazinah 2012).
Indonesia sebagai salah satu negara yang terletak di kawasan tropis memiliki 17 jenis Indigofera yang tumbuh dan telah tercatat di dalam Flora of Java (Backer & Bakhuizen van den Brink 1963). Tiga jenis Indigofera telah dimanfaatkan sebagai sumber pewarna indigo alami, yakni tom atal atau tom katemas (I. arrecta Hochst. Ex A. Rich.), tom jawa atau tarum (I. tinctoria L.), dan tom cantik (I. suffruticosa Mill.) (Kasmudjo & Saktianggi 2011; Muzzazinah 2012; Herdiawan & Krisnan 2014). Jenis yang paling banyak ditanam dan dimanfaatkan sebagai pewarna alami adalah I. tinctoria L. yang berasal dari India. Tarum (I. tinctoria L.) memiliki bentuk anak daun membundar telur sungsang hingga melonjong-membundar telur sungsang dengan ujung anak daun membundar atau bergubang yang merupakan ciri pembeda utama dari jenis I. arrecta Hochst. Ex A. Rich. maupun I. suffruticosa Mill. (Gao & Schrire 2010).
Tumbuhan penghasil indigo alami ini telah ditanam dan dimanfaatkan oleh penduduk lokal di Indonesia sejak masa kerajaan Tarumanegara. Tingginya tingkat budi daya dan pemanfaatan tarum (I. tinctoria L.) menyebabkan Indonesia dikenal sebagai negara pengekspor indigotin terbesar di Asia. Indigotin dalam bentuk serbuk dan blok pasta diekspor dari Indonesia ke negara-negara industri tekstil di kawasan Eropa (FAO 1995; Arsenault et al. 2008).
Pemanfaatan tarum sebagai penghasil indigo alami mengalami penurunan seiring dengan ditemukan dan dikembangkannya indigo sintetik pada tahun 1883. Pewarnaan menggunakan indigo sintetik memerlukan biaya lebih murah dan waktu yang diperlukan lebih cepat dibandingkan dengan indigo alami. Indigo sintetik mulai diperkenalkan pada tahun 1913 dan terus meningkat. Pada tahun 2002, sebanyak 17000 ton indigo sintetik diproduksi di seluruh dunia (Glowacki
2
Pembatik di daerah Jawa Tengah dan Sumenep, sebagai salah satu pengguna warna indigo, memanfaatkan tarum (I. tinctoria L.) sebagai penghasil indigo alami untuk mewarnai batik. Pembatik di Magelang memanfaatkan tarum (I. tinctoria L.) yang dibudidayakan di kebun sedangkan pembatik di Sumenep masih mengambilnya secara langsung dari alam. Proses budi daya tarum yang dilakukan oleh pembatik di Magelang dapat melahirkan adanya kultivar yang memiliki karakteristik berbeda dibandingkan dengan tarum yang tumbuh secara liar di alam.
Untuk pengembangan tarum di masa depan, perlu dilakukan konservasi mengingat bahwa pemanfaatan tarum (I. tinctoria L.) mengalami peningkatan sedangkan kondisi habitatnya terancam oleh pertambahan penduduk dan aktivitas modernisasi. Langkah pertama yang dapat dilakukan dalam upaya memulai konservasi tarum di pulau Jawa dan Madura adalah meninjau keragaman genetik tarum sehingga dapat diketahui status keragaman genetiknya (Narain 2000). Selain itu, kajian keragaman genetik tarum (I. tinctoria L.) penting untuk dilaksanakan karena dibutuhkan metode yang tepat untuk dapat membedakan varian-varian yang terdapat pada jenis tarum (I. tinctoria L.). Hasil kajian dapat digunakan sebagai dasar pertimbangan untuk menyeleksi kultivar unggul. Ketersediaan informasi kultivar unggul mengenai keragaman jenis atau sumber plasma nutfah tumbuhan akan menjadikan program pemuliaan lebih terarah sehingga menghasilkan kultivar dengan sifat-sifat unggul yang diinginkan.
Kajian keragaman pada tingkat di bawah jenis dapat dilakukan melalui pengamatan morfologi jika seluruh organ vegetatif maupun generatif tersedia dan memungkinkan untuk diamati. Namun, ketersediaan organ lengkap tidak selalu mudah diperoleh karena perbedaan masa pertumbuhan di setiap daerah dan sedikitnya aksesi tarum (I. tinctoria L.) yang ditemukan tumbuh secara liar di alam. Pengamatan ciri morfologi juga sulit dilakukan pada beberapa tumbuhan yang berkerabat dekat sehingga sangat sedikit ciri yang bisa didapatkan. Selain itu, pengamatan ciri morfologi memerlukan waktu yang cukup lama dan tidak semua ciri dapat diamati perbedaannya secara visual. Penanda morfologi juga bersifat kompleks karena merupakan hasil interaksi antar gen dan lingkungan sehingga sulit untuk membedakan antara ciri yang diturunkan secara genetik atau hasil adaptasi terhadap lingkungan (Babić et al. 2012). Kendala yang muncul pada pengamatan morfologi dapat diatasi dengan menggunakan marka molekuler berbasis DNA (Lin et al. 2010). Keunggulan marka molekuler dalam mempelajari keragaman di bawah jenis (infraspecies) adalah variasi pada tingkat DNA dapat ditunjukkan dalam waktu yang lebih cepat, efisien, dan biaya yang rendah dibandingkan dengan pengamatan ciri morfologi (Babićet al. 2012).
3 untuk mempelajari keragaman genetik, kekerabatan filogenetik, penandaan gen, pemetaan genom, dan evolusi suatu jenis yang berkerabat sangat dekat (Pardhe & Satpute 2011; Sadeghi & Cheghamirza 2012). Marka ISSR hingga saat ini telah berhasil digunakan untuk mempelajari keragaman genetik pada beberapa marga dalam kelompok suku Fabaceae, misalnya marga Cicer (Amirmoradi et al. 2013).
Tujuan Penelitian
4
2
TINJAUAN PUSTAKA
Tumbuhan Tarum (Indigofera tinctoria L.)
Suku Fabaceae merupakan kelompok tumbuhan berbunga yang bersifat kosmopolit dengan perawakan berupa pohon, perdu, atau herba. Anggota suku
Fabaceae terdiri atas 440 marga dan 11.000 jenis. Banyak anggota dari suku ini memiliki nilai ekonomi dan kemanfaatan tinggi, baik untuk bahan makanan, industri tekstil, bahan bakar, tanaman hias, dan penutup lahan terbuka. Ciri yang dimiliki oleh suku ini adalah perbungaan tandan; bunga aktinomorf hingga zigomorf, banci; polen bertipe monad, tetrad, atau poliad; karpel beruang satu; ovarium menumpang; buah berbentuk polong; biasanya tidak memiliki endosperma; umumnya embrio berwarna hijau; stipula kadang termodifikasi menjadi duri halus hingga tebal; elemen vasal memiliki lubang perforasi sederhana; umumnya akar bernodul; mengandung ellagitannin (Balgooy 1998; Kubitzki 2007).
Salah satu marga yang bernilai ekonomi tinggi adalah Indigofera. Marga ini umum dimanfaatkan sebagai pewarna tekstil dan pakan ternak. Indigofera
merupakan tumbuhan dengan perawakan berupa pohon, perdu, atau herba dengan ciri daun tunggal atau majemuk menyirip gasal dengan anak daun tersusun berseling. Trikoma uniseluler dapat ditemukan di batang pada semua jenis
Indigofera. Daun penumpu berbentuk segitiga memanjang dengan ujung lancip atau kadang menyegitiga dengan panjang bervariasi. Perbungaan bertipe tandan dan muncul di ketiak daun. Bunga berbentuk kupu-kupu dengan lunasyang selalu tertutup. Buah berbentuk polong bulat atau memanjang dengan jumlah biji yang bervariasi (Kort & Thijsse 1984; Gao & Schrire 2010; Muzzazinah 2012).
Anggota marga Indigofera mencapai lebih dari 700 jenis yang tersebar di daerah tropis dan subtropis (Schrire et al. 2009). Tujuh belas jenis ditemukan di pulau Jawa sebagaimana tercantum di dalam Flora of Java, yakni I. arrecta
Hochst. ex A. Rich, I. colutea (Burm. f.) Merr., I. cordifolia Heyne ex Roth, I. galegoides DC, I. glandulosa J.C. Wendl, I. guatemalensis Moc. & Sessé ex Prain & Backer, I. hirsuta L., I. linnaei Ali, I. longeracemosa Boiv. ex Baill., I. nigrescens Kurz ex King & Prain, I. oblongifolia Forssk., I. trifoliata L., I. trita L., I. tinctoria L., I. spicata Forssk., I. suffruticosa Mill., dan I. zollingeriana
Miq. (Backer & Bakhuizen van den Brink 1963).
Indigofera arrecta Hochst. ex A. Rich, I. suffruticosa Mill., dan I. tinctoria L. merupakan jenis yang dapat dimanfaatkan sebagai pewarna batik (Muzzazinah 2012; Herdiawan & Krisnan 2014). Akan tetapi, jenis Indigofera
5 Sri Lanka, India, Pakistan, Cina, Taiwan, Jepang, Vietnam, dan Filipina (Lemmens & Wessel-Riemens 1992; Mattapha & Chantaranothai 2012).
Tarum (I. tinctoria L.) memiliki perawakan perdu pendek, tegak, dengan tinggi mencapai 50-150 cm (Gambar 1a). Pada bagian akar terdapat nodul. Daun bertipe majemuk menyirip gasal dengan anak daun berjumlah 9-17 helai berwarna hijau kebiruan. Anak daun tersusun berhadapan, berbentuk membundar telur sungsang hingga melonjong dengan ujung membundar atau bergubang. Perbungaan bertipe tandan yang terdapat di ketiak daun (Gambar 1c). Bentuk bunga menyerupai kupu-kupu yang terbagi menjadi bendera, sayap, dan lunas. Bagian bendera berwarna hijau pada bagian tengah sedangkan bagian tepi berwarna kemerahan. Bagian sayap berwarna merah muda menyolok. Buah bertipe polong tersusun dalam tandan dan berbentuk lurus atau semi-sirkular di bagian ujungnya tetapi tidak pernah melancor (Gambar 1b). Polong muda berwarna hijau dan menjadi coklat ketika tua (Backer & Bakhuizen van den Brink 1963; Kort & Thijsse 1984; Gao & Schrire 2010). Bentuk anak daun dan tipe ujung anak daun merupakan ciri pembeda utama I. tinctoria L. dengan I. arrecta Hochst. Ex A. Rich. dan I. suffruticosa Mill.
Gambar 1 Profil tumbuhan tarum (I. tinctoria L.). a. Perawakan, b. Buah dan Biji, c. Bunga
6
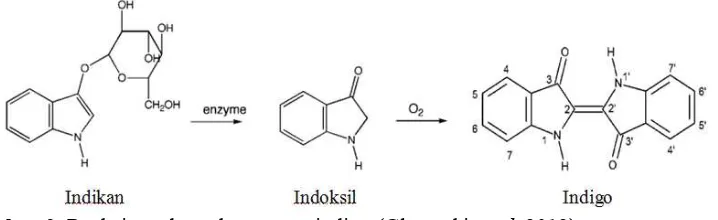
senyawa indoksil akan teroksidasi ketika terpapar udara sehingga muncul warna biru (Gambar 2) (Chanayath et al. 2002; Kasmudjo & Saktianggi 2011).
Gambar 2 Reaksi pembentukan warna indigo (Glowacki et al. 2012)
Indigo telah digunakan sebagai pewarna alami tekstil sejak sebelum pewarna sintetis ditemukan. Ekstraksi indigo dari tumbuhan tarum (I. tinctoria
L.) dimulai di India, Mesir, dan Cina kemudian disebarluaskan ke negara tropis lain termasuk Indonesia, sedangkan indigo di Thuringia (Eropa) berasal dari tumbuhan woad (Isatis tinctoria L.). Indigo yang diekstrak dari tarum memiliki tingkat kualitas warna yang lebih baik dibandingkan dengan indigo yang diekstrak dari Isatis tinctoria L., sehingga industri tekstil Eropa mulai mengimpor indigo dari India dan Indonesia (Glowacki et al. 2012). Perkembangan pewarna alami mengalami fluktuasi dengan kemasyhuran Indigofera di Indonesia yang tercatat pada tahun 1918-1925. Nilai ekspor tertinggi terjadi pada tahun 1921 mencapai 69.777 kg berat kering (Heyne 1987). Impor indigo yang berasal dari tarum menurunkan aktivitas industri pewarna alami Eropa, sehingga pada tahun 1865 Johann Friedrich Wilhelm Adolf von Baeyer, ahli kimia dari Jerman, mulai meneliti struktur kimia indigo dan berhasil membuat indigo sintesis pada tahun 1880. Pewarna indigo alami perlahan mulai digantikan oleh indigo sintetik dan pada tahun 1913 pewarna indigo yang digunakan untuk keperluan tekstil berasal dari indigo sintetik (Sequin-Frey 1981; Glowacki et al. 2012).
Struktur Populasi
Setiap organisme hidup membentuk populasi di alam. Keberadaan suatu populasi ditandai dengan adanya interaksi kerjasama atau penghambatan antar anggota sehingga laju pertumbuhan populasi tergantung pada ukuran atau kepadatan populasi disamping faktor lingkungan fisik tempat populasi itu berada. Pada organisme dengan jenis kelamin terpisah, perkawinan merupakan faktor penting untuk menjaga kelangsungn hidup populasi. Interaksi antar individu tidak terbatas hanya sesama jenis saja tetapi juga dapat terjadi antar jenis yang berlainan. Kelangsungan hidup suatu jenis umumnya tergantung pada keberadaan jenis lain, baik berfungsi sebagai makanan, perantara kawin, maupun perlindungan dari bahaya (Nei 1975).
7 genetika populasinya harus dijabarkan berdasarkan persebaran geografi dari frekuensi gen atau genotipenya. Struktur genetika suatu populasi ditentukan berdasarkan sejumlah besar lokus yang ada. Selain itu, genetika populasi juga mengamati kodrat suatu gen yang dimiliki oleh suatu jenis sehingga dapat digunakan untuk menghitung atau memperkirakan hasil akhir dari seleksi alam karena seleksi alam muncul dari bagaimana individu-individu berinteraksi dengan lingkungan yang cenderung mengalami perubahan. Oleh karena itu, genetika populasi bertujuan mempelajari struktur genetika populasi dan hukum yang mengubah struktur genetik, yakni tipe dan frekuensi gen atau genotipe yang terdapat di dalam populasi (Nei 1975; Templeton 2006).
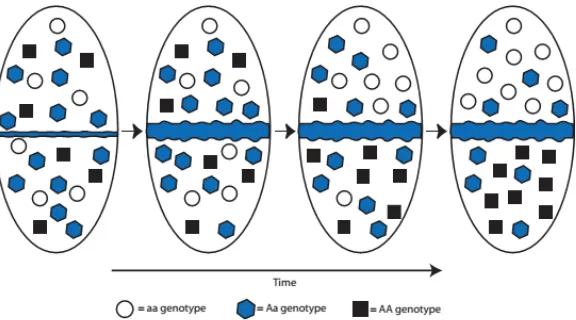
Ketidakseragaman sangat umum terjadi di dalam sebuah populasi. Pada populasi yang besar, terjadi ketidakseragaman kesempatan untuk kawin sebagaimana diasumsikan oleh Hardy-Weinberg. Ketidakseragaman kesempatan untuk kawin umumnya disebabkan oleh adanya jarak pemisah, baik ruang ataupun waktu, yang terjadi secara terus-menerus sehingga anggota dalam populasi menjadi terisolasi dan terbagi menjadi populasi yang lebih kecil (subpopulasi) (Gambar 3). Pada populasi yang terisolasi, peluang terjadinya kawin antar keluarga (inbreeding) menjadi tinggi dan aliran gennya menjadi rendah. Tanpa adanya aliran gen yang cukup untuk menjaga perkawinan acak, maka pergeseran genetik menyebabkan divergensi frekuensi alel antar subpopulasi (Hamilton 2009). Keadaan seperti ini, jika terus terjadi akan menyebabkan variasi genetik dalam populasi yang telah terpisah tersebut menjadi rendah.
Gambar 3 Stuktur populasi dan divergensi frekuensi alel yang disebabkan oleh terbatasnya aliran gen (Hamilton 2009)
8
maupun antar populasi. Seleksi dapat menurunkan atau meningkatkan keragaman di dalam maupun antar populasi (Hamilton 2009).
Perbedaan ketinggian tempat dapat mempengaruhi tingkat keragaman genetic di dalam populasi, seperti pada Pinus brutia dan Sophora moorcroftiana. Meningkatnya keragaman disebabkan oleh clinal variation terhadap frekuensi alel (Kara et al. 1997; Liu et al. 2006). Rendahnya keragaman genetik di dataran yang lebih rendah dimungkinkan karena dampak negatif aktivitas manusia dan fragmentasi habitat seperti pembukaan dan perluasan lahan untuk kebun atau sawah. Sebaliknya, di dataran yang lebih tinggi kurang dapat dijangkau oleh aktivitas manusia karena hanya dimanfaatkan sebagai tempat menggembala ternak sehingga populasi S. moorcroftiana tidak mendapat banyak gangguan seperti halnya di dataran rendah (Liu et al. 2006).
Pada tumbuhan Dillwynia tenuifolia, keragaman genetik di dalam populasi sangat tinggi dibandingkan dengan yang umumnya ditemui pada tumbuhan suku Fabaceae sedangkan keragaman genetik antar populasi sangat rendah (Hamrick & Godt 1996). Pada D. tenuifolia diperkirakan bahwa aliran gen yang tinggi terjadi melalui perpindahan polen yang efektif antar populasi dengan bantuan agen penyerbukan seperti lebah.
Keragaman genetik di dalam populasi pada kacang lima menunjukkan adanya korelasi antar jumlah populasi yang diamati. Mekanisme inbreeding pada kacang lima menghasilkan sejumlah besar alel homozigot dan penyebaran gen di dalam populasi tidak cukup kuat untuk menjaga pemaduan gamet secara acak (Bi
et al. 2003). Pada populasi dengan jumlah individu yang sedikit, hanya sedikit jumlah a lel dapat diamati sehingga tingkat heterozigositasnya lebih rendah yang disebabkan oleh fiksasi alel. Korelasi antar heterozigositas dan ukuran populasi efektif juga diharapkan untuk lokus yang termasuk dalam kategori lokus dengan tingkat heterozigositas rendah pada populasi yang kecil (Montgomery et al. 2000; Hedrick 2001).
Struktur genetika populasi memiliki peran penting dalam penyusunan strategi konservasi. Sebelum menentukan tindakan konservasi, status struktur populasi perlu diketahui terlebih dahulu. Informasi mengenai struktur genetika populasi diperlukan sebagai dasar untuk menentukan apakah status populasi yang dikaji berada dalam keadaan aman atau terancam. Penentuan status populasi sangat penting untuk diketahui agar dapat ditentukan apakah suatu populasi akan dikelola sebagai unit yang berbeda atau tidak sehingga tidak menimbulkan dampak negatif terhadap populasi yang sedang dikaji (Rao & Hodgkin 2002).
Marka Molekuler ISSR
Pengembangan teknik-teknik molekuler berbasis DNA merupakan alat untuk menganalisis genom tumbuhan. Teknik ini dapat mengurangi adanya keterbatasan sifat dari marka morfologi seperti rendahnya polimorfisme, pengaruh
9
polymorphism DNA (RAPD), dan mikrosatelit atau Simple Sequence Repeat
(SSR) (Spooner et al. 2005).
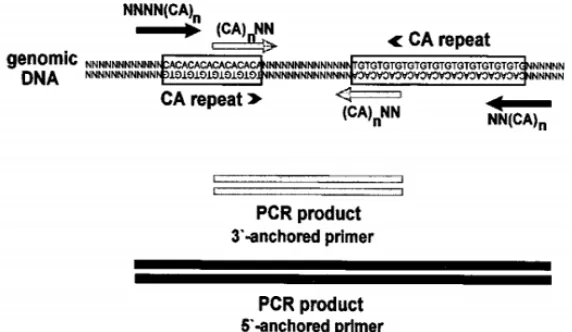
Inter-simple sequence repeat (ISSR) merupakan marka semi acak yang diamplifikasi oleh primer yang komplementer terhadap suatu target mikrosatelit. Marka ini dikembangkan dari daerah yang terletak di antara dua lokus mikrosatelit. Teknik genotyping menggunakan marka ISSR didasarkan pada variasi yang terdapat dalam sekuen yang terletak di antara duan sekuen mikrosatelit yang berdekatan (Zietkiewics et al. 1994). Awalnya Zietkiewics et al. (1994) menggunakan primer tunggal dengan ulangan (CA)n dengan
menambahkan nukleotida spesifik pada ujung pelekatan 3- atau 5- (Gambar 4). Primer tunggal yang telah didesain dapat berperan sebagai primer forward yang akan mengamplifikasi cetakan DNA ke arah downstream dan primer reverse yang akan mengamplifikasi cetakan DNA ke arah upstream. Karakteristik primer ISSR yang memiliki sekuen tambahan dengan inti mikrosatelit umumnya bersifat sama di dalam genom seluruh organisme, yakni memiliki tingkat variasi alel yang tinggi dan potensial untuk analisis yang dapat diulang sehingga menjadikan daerah ini sebagai marka molekuler yang unggul (Trojanwska & Balibok 2004).
Gambar 4 Wilayah amplifikasi marka Inter-simple sequence repeats (ISSR). Primer tunggal dengan inti (CA)n yang telah dilekatkan dengan nukleotida tambahan pada ujung 3- (panah putih) atau ujung 5- (panah hitam) (Zietkiewicz et al. 1994)
Marka ISSR merupakan daerah di dalam DNA yang panjangnya sangat bervariasi dalam suatu spesies yang sama (Salimath et al. 1995). Amplifikasi menggunakan marka ISSR tidak membutuhkan informasi sekuen genom dan pola pita multilokus yang dihasilkan sangat polimorfik. Ukuran pita DNA yang diamplifikasi oleh primer ISSR berkisar 100-3000 pasang basa (Nagaoka & Ogihara 1997). Metode ISSR sering digunakan untuk mempelajari sidik jari genetik, penandaan gen, deteksi variasi klon, identifikasi kultivar, analisis filogenetik, deteksi ketidakstabilan genom, dan pengujian hasil hibridisasi pada tumbuhan maupun hewan yang berkerabat dekat (Song 2005).
10
(AFLP), random amplified polymorphism DNA (RAPD) dan restriction fragment length polymorphism (RFLP). Amplifikasi dengan primer ISSR tetap dapat dilakukan meskipun jumlah DNA yang dimiliki sedikit dan biaya yang dibutuhkan juga lebih terjangkau karena tidak perlu dilakukan klon maupun karakterisasi sekuen terlebih dahulu (Spooner et al. 2005; LTC 2010).
Marka ISSR saat ini telah berhasil digunakan untuk mempelajari keragaman genetik baik untuk identifikasi jenis maupun infra-jenis. Pada tingkat jenis, marka ISSR dapat digunakan untuk mengidentifikasi kompleks
11
3
METODE
Waktu dan Tempat Penelitian
Penelitian ini dilaksanakan pada bulan April 2014 sampai dengan April 2015. Isolasi dan amplifikasi DNA dilaksanakan di Laboratorium Fisiologi dan Biologi Molekuler Tumbuhan serta Laboratorium Terpadu, Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Koleksi Sampel
Aksesi tarum (I. tinctoria L.) dikoleksi berdasarkan metode jelajah yaitu mengunjungi lokasi yang diketahui dan memungkinkan dijumpainya tumbuhan tersebut tumbuh (Widjaja & Poerba 2004). Penentuan lokasi pengambilan sampel didasarkan pada catatan koleksi spesimen di Herbarium Bogoriense (BO) dan informasi langsung dari pembatik di Jawa dan Madura. Sampel yang diambil berupa cabang dengan organ lengkap yang terdiri atas daun, bunga, buah dan biji yang akan diproses menjadi herbarium kering (Vogel 1987). Sebagian daun segar, bersih, dan sehat dimasukkan ke dalam plastik klip yang berisi silika gel untuk isolasi DNA.
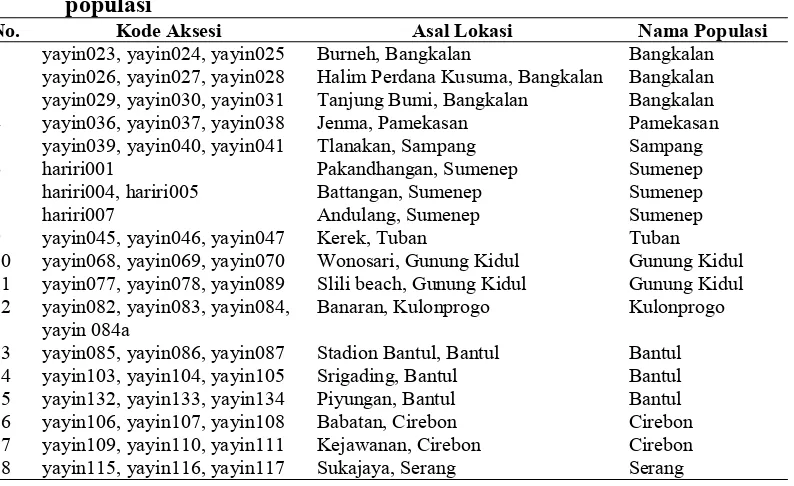
Sebanyak 50 aksesi daun tarum (I. tinctoria L.) diperoleh dari 10 lokasi yang tersebar di pulau Jawa dan Madura, yakni 6 lokasi yang mewakili pulau Jawa (Cirebon, Serang, Tuban, Kulonprogo, Bantul, dan Gunung Kidul) dan 4 lokasi yang mewakili pulau Madura (Sumenep, Pamekasan, Sampang, dan Bangkalan) (Gambar 5, Tabel 1). Setiap lokasi dianggap sebagai satu populasi sehingga terdapat 10 populasi tarum (I. tinctoria L.) yang dianalisis.
12
Tabel 1 Identitas aksesi tarum (I. tinctoria L.), asal lokasi, dan nama populasi
No. Kode Aksesi Asal Lokasi Nama Populasi
1 yayin023, yayin024, yayin025 Burneh, Bangkalan Bangkalan 2 yayin026, yayin027, yayin028 Halim Perdana Kusuma, Bangkalan Bangkalan 3 yayin029, yayin030, yayin031 Tanjung Bumi, Bangkalan Bangkalan 4 yayin036, yayin037, yayin038 Jenma, Pamekasan Pamekasan 5 yayin039, yayin040, yayin041 Tlanakan, Sampang Sampang
6 hariri001 Pakandhangan, Sumenep Sumenep
7 hariri004, hariri005 Battangan, Sumenep Sumenep
8 hariri007 Andulang, Sumenep Sumenep
9 yayin045, yayin046, yayin047 Kerek, Tuban Tuban
10 yayin068, yayin069, yayin070 Wonosari, Gunung Kidul Gunung Kidul 11 yayin077, yayin078, yayin089 Slili beach, Gunung Kidul Gunung Kidul 12 yayin082, yayin083, yayin084,
yayin 084a
Banaran, Kulonprogo Kulonprogo
13 yayin085, yayin086, yayin087 Stadion Bantul, Bantul Bantul 14 yayin103, yayin104, yayin105 Srigading, Bantul Bantul 15 yayin132, yayin133, yayin134 Piyungan, Bantul Bantul 16 yayin106, yayin107, yayin108 Babatan, Cirebon Cirebon 17 yayin109, yayin110, yayin111 Kejawanan, Cirebon Cirebon 18 yayin115, yayin116, yayin117 Sukajaya, Serang Serang
Isolasi DNA Total
13 Amplifikasi dan Visualisasi DNA
DNA tarum diamplifikasi menggunakan ESCO Swift™ Maxi model SWT-MY-BLC-7 (USA). Lima belas primer ISSR yang digunakan pada penelitian ini telah berhasil digunakan untuk menganalisis keragaman genetik tumbuhan Sesbania, V. radiata, dan V. savi (Bisoyi et al. 2010; Pardhe & Satpute 2011; Khajudparn et al. 2012) (Tabel 1). Setiap reaksi ISSR-PCR diamplifikasi dalam volume 25 μl yang terdiri dari 2.5 μl DNA, 1 μl primer ISSR, 12.5 μl 2X GoTaq® Green PCR Mix pH 8.5 (mengandung 400 μM dATP, 400 μM dGTP, 400 μM dCTP, 400 μM dTTP, 3 mM MgCl2), and 9 μl
air bebas nuklease (Promega, USA). Proses amplifikasi terdiri dari denaturasi awal pada suhu 94 ᵒC selama 3 menit, kemudian diikuti dengan 30 siklus denaturasi pada 94 ᵒC selama 1 menit, penempelan primer pada 45-51 ᵒC selama 45-60 detik, pemanjangan primer pada 72 ᵒC selama 2 menit, dan selanjutnya pemanjangan primer akhir selama 10 menit pada suhu 72 ᵒC (Bisoyi et al. 2010; Pardhe & Satpute 2011; Khajudparn et al. 2012). Produk hasil PCR dielektroforesis pada gel agarosa 1% dalam 1X buffer TBE yang ditambahkan dengan 1 μl ethidium bromida (1 μg/ml), pada tegangan 80 V selama 90 menit. Pola pita diamati di bawah cahaya UV dan difoto menggunakan uv-transilluminator (WiseDoc,
Germany).
Analisis Data
Pita polimorfik dari hasil elektroforesis dinilai nol (0) jika tidak terdapat pita dan satu (1) jika terdapat pita pada posisi yang sama untuk setiap aksesi yang dibandingkan. Data yang diperoleh disusun dalam satu matriks data dan digunakan untuk menghitung koefisien kemiripan simple matching (SM) (Kosman & Leonard 2005). Koefisien kemiripan SM digunakan untuk mengkonstruksi diagram analisis komponen utama atau principal component analysis (PCA) dan dendrogram menggunakan analisis Sequential Agglomerative Hierarchial and Nested Clustering (SAHN) dengan metode Unweighted Pair Group Method with Arithmatic Average (UPGMA) melalui program Numerical Taxonomy and Multivariate Analisys System for PC (NTSys- PC) versi 2.1.1a (Exeter Software, New York) (Rohlf 1998). Jarak genetik Nei, analisis variasi molekuler (AMOVA), dan struktur populasi yang meliputi indeks Shannon, keragaman genetik, persentase lokus polimorfik dihitung menggunakan program
14
4
HASIL DAN PEMBAHASAN
Seleksi dan Profil Primer ISSR
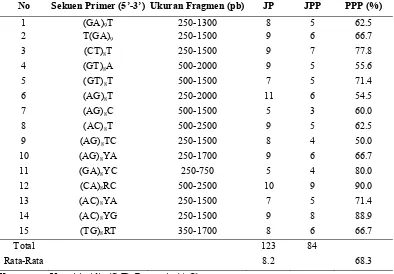
Lima belas primer ISSR yang digunakan untuk mendeterminasi keragaman 50 aksesi tarum (I. tinctoria L.) berhasil mengamplifikasi sekuen genom tarum (I. tinctoria L.) dengan pola, jumlah dan ukuran pita yang bervariasi antar aksesi dan primer. Sebanyak 123 pita dihasilkan dari 15 primer ISSR yang digunakan dan 84 pita bersifat polimorfik. Jumlah pita yang diamplifikasi oleh masing-masing primer berkisar 5-11 pita dengan panjang pita berkisar 250-2500 pasang basa (pb) dan persentase polimorfisme pita berkisar 50-90% dengan rata-rata 68.3% (Tabel 2).
Tabel 2 Sekuen primer ISSR dan profil pita hasil amplifikasi DNA tarum (I. tinctoria L.) di pulau Jawa dan Madura
No Sekuen Primer (5’-3’) Ukuran Fragmen (pb) JP JPP PPP (%)
Keterangan: Y = pirimidin (C,T); R = purin (A,G)
JP=Jumlah pita; JPP=Jumlah pita polimorfik, PPP=Persentase pita polimorfik
Primer (AG)8C dan (GA)8YC menghasilkan lebih sedikit pita (5 pita)
sedangkan primer (AG)8T menghasilkan lebih banyak pita (11 pita) dibandingkan
dengan primer lainnya. Sedikitnya jumlah pita yang diamplifikasi oleh primer (AG)8C dan (GA)8YC mungkin disebabkan oleh sedikitnya sekuen komplemen
dari pola (AG)8C dan (GA)8YC dibandingkan dengan pola (AG)8T pada genom
tarum (I. tinctoria L.).
Kelimpahan sekuen komplementer suatu primer yang diamati tidak selalu berhubungan dengan tingginya persentase polimorfisme pita. Primer (AG)8T
15 (GA)8YC yang hanya menghasilkan 5 pita tetapi persentase polimorfisme tinggi
(80%). Sekuen komplementer primer (AG)8T dimungkinkan melimpah dalam
genom tarum (I. tinctoria L.), tetapi tidak bersifat spesifik karena juga ditemukan cukup melimpah pada sekuen milik Vigna radiata hibrida,
Pandanus, dan Freycinetia (Rahayu et al. 2007; Khajudparn et al. 2012).
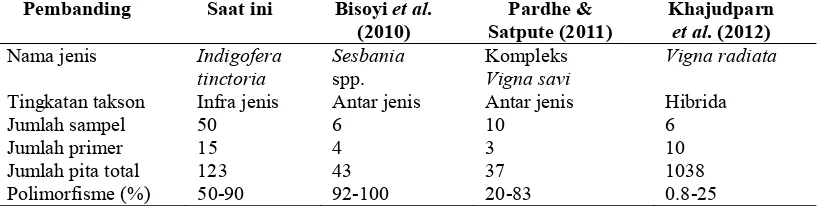
Jumlah pita yang berhasil diamplifikasi pada penelitian ini lebih sedikit dibandingkan Khajudparn et al. (2012), tetapi lebih banyak dibandingkan Bisoyi
et al. (2010) serta Pardhe dan Satpute (2011). Persentase pita polimorfik yang dihasilkan pada penelitian ini lebih besar dibandingkan penelitian Khajudparn
et al. (2012), tetapi relatif sama dengan penelitian Bisoyi et al. (2010) dan Pardhe dan Satpute (2011) (Tabel 3).
Tabel 3 Perbandingan aksesi dan profil pita hasil amplifikasi DNA tarum (I. tinctoria L.) di pulau Jawa dan Madura terhadap hasil penelitian rujukan
Tingkatan takson Infra jenis Antar jenis Antar jenis Hibrida
Jumlah sampel 50 6 10 6
Jumlah primer 15 4 3 10
Jumlah pita total 123 43 37 1038
Polimorfisme (%) 50-90 92-100 20-83 0.8-25
Perbedaan jumlah pita yang teramplifikasi dan persentase polimorfisme pita antara penelitian ini dan sebelumnya dimungkinkan oleh adanya perbedaan pada jenis tumbuhan, tingkatan takson, jumlah sampel, maupun jumlah primer yang digunakan. Bisoyi et al. (2010) menganalisis 6 jenis Sesbania menggunakan 4 primer ISSR, Pardhe dan Satpute (2011) menganalisis 10 genotipe V. savi
menggunakan 3 primer ISSR, dan Khajudparn et al. (2012) menganalisis 6 aksesi kacang hijau hibrida menggunakan 10 primer ISSR.
Pada analisis keragaman genetik antar jenis, persentase pita polimorfik yang dihasilkan umumnya lebih besar (20-83% dan 92-100%) dibandingkan dengan infra jenis (0.8-25% dan 50-90%). Perbedaan tersebut disebabkan oleh ada tidaknya sekuen komplementer untuk setiap primer ISSR yang digunakan serta ukuran fragmen yang diamplifikasi menggunakan setiap primer ISSR juga cukup bervariasi pada tumbuhan yang berbeda. Selain itu, dapat diketahui juga bahwa terdapat primer ISSR yang bersifat umum (universal) yang dapat digunakan untuk jenis berbeda, seperti primer (AG)8T yang juga berhasil digunakan dalam
menganalisis keragaman genetik kacang hijau hibrida dan pandan-pandanan (Rahayu et al. 2007; Khajudparn et al. (2012).
16
karena dimungkinkan pita monomorfik yang merupakan akumulasi hasil hibridisasi dari kedua tetuanya lebih banyak teramplifikasi.
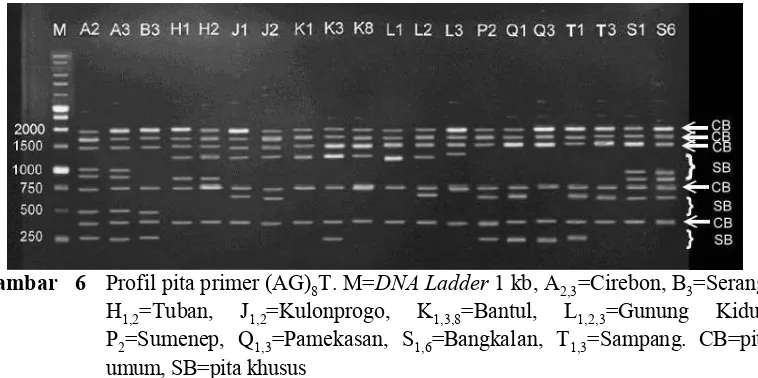
Berdasarkan visualisasi pola pita amplifikasi primer (AG)8T (Gambar 6),
dapat diketahui dua macam pita, yakni pita umum (300, 750, 1500, 1700, dan 2000 pb) dan pita khusus (250, 500, 600, 900, 1000, dan 1300 pb). Pola pita khusus yang menjadi penanda populasi dari Cirebon (A2 dan A3) adalah pita 250,
500, 900 dan 1000 pb yang tidak ditemukan pada populasi lainnya, misalnya saja B3 yang memiliki pita 250 pb dan 500 pb tetapi tidak memiliki pita 900 pb dan
1000 pb. Populasi dari Cirebon dan Serang tidak memiliki pita 1300 pb, sebagaimana dimiliki populasi dari pulau Jawa lainnya yakni Tuban, Kulonprogo, Bantul, dan Gunung Kidul.
Gambar 6 Profil pita primer (AG)8T. M=DNA Ladder 1 kb, A2,3=Cirebon, B3=Serang, H1,2=Tuban, J1,2=Kulonprogo, K1,3,8=Bantul, L1,2,3=Gunung Kidul, P2=Sumenep, Q1,3=Pamekasan, S1,6=Bangkalan, T1,3=Sampang. CB=pita umum, SB=pita khusus
Tingginya persentase polimorfisme pita yang teramplifikasi merupakan keunggulan marka ISSR. Primer ISSR menempel pada sekuen simple sequence repeats yang melimpah pada genom eukariot dengan laju evolusi yang tinggi sehingga dapat menunjukkan tingkat polimorfisme yang tinggi (Zietkiewics et al. 1994).
Analisis GugusTarum (I. tinctoria L.) di pulau Jawa dan Madura
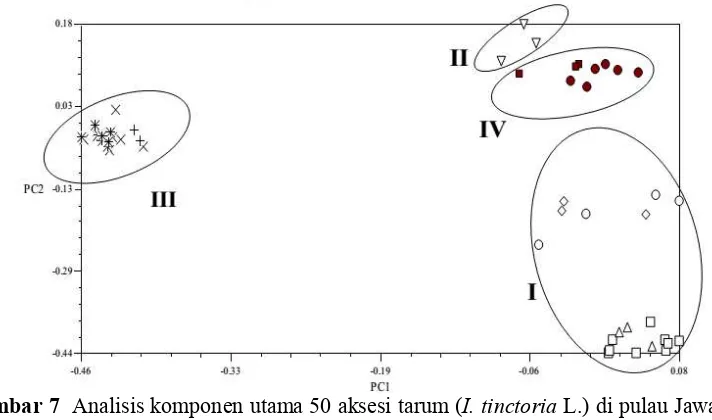
Pita polimorfik yang dihasilkan oleh 15 primer dianalisis menggunakan analisis komponen utama (PCA) untuk mengetahui pola pengelompokan berdasarkan ciri pita yang telah diperoleh sehingga memunculkan sebaran data dari masing-masing aksesi dengan lebih sederhana dalam bentuk plot (Sebola & Balkwill 2013). Lima puluh aksesi tarum terbagi menjadi empat kelompok berdasarkan PC1 dan PC2 dengan sumbangan kumulatif 51.72% (30.75% untuk
PC1 dan 20.97% untuk PC2) (Gambar 7).
17 dan Kulonprogo). Kelompok IV menyatukan semua aksesi yang berasal dari Jawa Barat (Cirebon dan Serang).
Gambar 7 Analisis komponen utama 50 aksesi tarum (I. tinctoria L.) di pulau Jawa dan Madura. □=Bangkalan, ♯=Bantul, ●=Cirebon, ×=Gunung Kidul, +=Kulonprogo, ◊=Pamekasan, △=Sampang, ■=Serang, ○=Sumenep,
▽=Tuban
Pola pengelompokan aksesi tarum menggunakan PCA (Gambar 7) belum dapat menunjukkan hubungan antar aksesi tarum (I. tinctoria L.) sehingga dilanjutkan dengan analisis gugus menggunakan metode UPGMA. Dendrogram yang dikonstruksi menunjukkan pola hubungan antar aksesi secara lebih jelas (Gambar 8).
Lima puluh aksesi tarum (I. tinctoria L.) yang berasal dari pulau Jawa dan Madura tergabung dalam satu kelompok pada koefisien kemiripan 0.51 kemudian dibagi ke dalam dua kelompok besar yakni kelompok A dan B pada koefisien 0.53. Kelompok A merupakan aksesi tarum (I. tinctoria L.) yang berasal dari pulau Madura dan kelompok B merupakan aksesi tarum (I. tinctoria L.) yang berasal dari pulau Jawa. Pada koefisien kemiripan 0 . 60, kelompok A tetap menjadi kelompok yang terpisah (Kelompok I) sedangkan kelompok B terbagi menjadi tiga kelompok yang lebih spesifik (Kelompok II, III dan IV), sebagaimana pengelompokan pada PCA. Kelompok I (aksesi dari pulau Madura) dapat dikelompokkan menjadi dua kelompok yang lebih kecil yang tersusun atas aksesi dari Pamekasan-Sumenep dan Bangkalan-Sampang; kelompok II merupakan aksesi yang berasal dari Tuban; kelompok III merupakan aksesi yang berasal dari Bantul, Gunung Kidul, dan Kulonprogo; serta Kelompok IV terdiri dari aksesi yang berasal dari Cirebon dan Serang.
18
Gambar 8 Dendrogram 50 aksesi tarum (I. tinctoria L.) berdasarkan 84 pita ISSR
dengan metode UPGMA. A1-6=Cirebon, B1-3=Serang, H1-3=Tuban,
J1-4=Kulonprogo, K1-9=Bantul, L1-6=Gunung Kidul, P1-4=Sumenep,
Q1-3=Pamekasan, S1-9=Bangkalan, T1-3=Sampang
19 memiliki sedikit perbedaan (Gambar 6), yakni pada pita dengan ukuran 900 pb dan 1000 pb untuk primer (AG)8T.
Tabel 4 Jarak genetik Nei 10 populasi tarum (I. tinctoria L.) di pulau Jawa dan Madura
Primer ISSR yang digunakan dalam penelitian ini tidak hanya dapat digunakan untuk mempelajari keragaman genetik tarum (I. tinctoria L.) di pulau Jawa dan Madura, tetapi juga mampu digunakan sebagai alat untuk mengidentifikasi asal populasi (Tabel 5). Hampir seluruh populasi dapat diidentifikasi, tetapi populasi dari Gunung Kidul belum dapat diidentifikasi oleh primer ISSR yang digunakan karena belum diperoleh pita spesifik untuk populasi tersebut. Identifikasi asal populasi dapat dilakukan dengan mengamati pita yang diamplifikasi oleh setiap primer, sebagai contoh adalah pola pita pada primer (GA)9T. Populasi aksesi tarum (I. tinctoria L.) yang berasal dari Tuban dapat
diidentifikasi oleh pita tunggal berukuran 1200 pb, aksesi tarum (I. tinctoria L.) yang berasal dari Sumenep ditunjukkan oleh pita tunggal berukuran 650 pb, dan aksesi tarum (I. tinctoria L.) yang berasal dari Pamekasan ditunjukkan oleh dua pita berukuran 650 pb dan 1200 pb (Gambar 9).
Gambar 9 Pola pita spesifik dari primer (GA)9T (di dalam kotak) untuk populasi
Tuban, Sumenep, dan Pamekasan. M=DNA Ladder 1kb, A2,3=Cirebon,
B3=Serang, H1,2=Tuban, J1,2=Kulonprogo, K1,3,8=Bantul, L1,2,3=Gunung
Kidul, P2=Sumenep, Q1,3=Pamekasan, T1,3=Sampang, S1,6=Bangkalan. Pita di dalam kotak menunjukkan pita spesifik yang dapat digunakan untuk identifikasi asal populasi: Tuban (GA)9T
20
Tabel 5 P ita spesifik untuk identifikasi asal populasi tarum (I. tinctoria L.) di pulau Jawa dan Madura
Asal Popula si Pita spesifik untuk identifika si
Sumenep (GA)9T6 5 0
Keterangan: Identifikasi asal populasi tarum (I. tinctoria L.) dapat dilakukan dengan mengamati satu atau lebih pita spesifik primer tertentu, misalnya saja populasi yang berasal dari Sumenep dapat diidentifikasi menggunakan primer (GA)9T yang dicirikan
dengan adanya pita spesifik berukuran 650 pb atau dapat pula diidentifikasi menggunakan primer (AG)8T yang dicirikan dengan adanya
pita spesifik berukuran 250 pb dan 600 pb
21 Keragaman Genetik dan Struktur PopulasiTarum (I. tinctoria L.)
di pulau Jawa dan Madura
Keragaman genetik tarum (I. tinctoria L.) di pulau Jawa dan Madura secara umum termasuk dalam kategori tinggi dengan nilai rata-rata persentase pita polimorfik yang teramati bernilai 68.3% (Tabel 2). Akan tetapi, tingkat keragaman di dalam setiap populasinya rendah karena nilai rata-rata persentase pita polimorfik yang teramati pada setiap populasi bernilai 29.40% (Tabel 6). Hasil analisis variasi molekuler (AMOVA) juga menunjukkan bahwa variasi genetik antar populasi lebih tinggi (63%) dibandingkan variasi genetik di dalam populasi (37%) (Gambar 10).
Gambar 10 Persentase variasi molekuler di dalam dan antar populasi tarum (I. tinctoria L.) di pulau Jawa dan Madura
Tabel 6 Keragaman genetik 10 populasi tarum (I. tinctoria L.) di pulau Jawa dan Madura
Populasi N Na Ne I h P(%)
Sumenep 4 0.90 (0.10) 1.21 (0.03) 0.19 (0.03) 0.13 (0.02) 33.33 Bangkalan 9 1.07 (0.09) 1.14 (0.03) 0.16 (0.02) 0.10 (0.02) 36.90 Pamekasan 3 0.65 (0.08) 1.10 (0.03) 0.08 (0.02) 0.06 (0.02) 13.10 Sampang 3 0.76 (0.09) 1.17 (0.04) 0.14 (0.03) 0.10 (0.02) 21.43 Tuban 3 1.40 (0.08) 1.44 (0.04) 0.35 (0.03) 0.24 (0.02) 54.76 Gunung Kidul 6 0.81 (0.09) 1.14 (0.03) 0.13 (0.03) 0.09 (0.02) 23.81 Kulonprogo 4 0.81 (0.09) 1.15 (0.03) 0.14 (0.03) 0.09 (0.02) 23.81 Bantul 9 1.27 (0.10) 1.42 (0.04) 0.35 (0.03) 0.24 (0.02) 58.33 Cirebon 6 0.70 (0.08) 1.11 (0.03) 0.10 (0.02) 0.07 (0.02) 19.05 Serang 3 0.52 (0.07) 1.08 (0.03) 0.06 (0.02) 0.04 (0.01) 9.52
Rata-rata 29.40
Keterangan: N=jumlah aksesi, Na=jumlah alel yang berbeda, Ne=jumlah alel efektif, I=indeks Shannon, h=keragaman, P=persentase lokus polimorfik. Angka di dalam tanda kurung menunjukkan nilai standar deviasi
22
variasi genetik di dalam populasi pada tumbuhan yang menyerbuk sendiri cenderung memiliki nilai yang lebih rendah dibandingkan dengan variasi antar populasinya (Persson & Bothmer 2000; Huang et al. 2009; Wright et al. 2013).
Struktur populasi tarum (I. tinctoria L.) di pulau Jawa dan Madura menunjukkan bahwa keragaman genetik dan indeks Shannon maksimum secara berurutan dimiliki oleh populasi yang berasal dari Tuban dan Bantul dengan nilai 0.24 dan 0.35, sedangkan nilai keragaman genetik dan indeks Shannon minimum dimiliki oleh populasi yang berasal dari Serang dengan nilai 0.04 dan 0.06.
Aksesi tarum (I. tinctoria L.) yang berasal dari Bantul memiliki nilai keragaman paling tinggi (0.24) karena dikoleksi dari kebun budi daya sehingga aksesi yang dikoleksi lebih berlimpah dibandingkan dengan lokasi lainnya. Kondisi lingkungan dan habitat tempat tumbuh di Bantul cukup bervariasi dengan ketinggian lokasi berkisar 44-129 m dpl. Perbedaan kondisi lingkungan menyebabkan tumbuhan menyesuaikan diri dengan kondisi lingkungan tempat hidupnya. Penyesuaian diri yang dilakukan dapat memicu terjadinya perubahan-perubahan biokimia maupun fisiologis pada tumbuhan tersebut sehingga mempengaruhi keragaman genetik populasi tersebut (Indriani et al. 2008).
Populasi yang berasal dari Bangkalan memiliki jumlah aksesi yang sama dengan Bantul (9 aksesi), tetapi nilai keragaman pada populasinya lebih rendah (0.10). Aksesi yang berasal dari Bangkalan dikoleksi dari beberapa lokasi yang berjauhan dengan jumlah aksesi yang ditemukan di setiap lokasi hanya sedikit.Kondisi habitat dan lingkungan dari 3 lokasi pengambilan sampel di Bangkalan menunjukkan profil yang hampir sama (data tidak ditampilkan), yakni berada di sepanjang jalan di daerah dekat pantai dengan ketinggian 5-51 m dpl. Adanya kesamaan habitat dan kondisi lingkungan tempat hidup aksesi dari Bangkalan menyebabkan rendahnya keragaman yang dimiliki karena tumbuhan tidak perlu melakukan adaptasi untuk menyesuaikan terhadap lingkungan tempatnya hidup sehingga tidak terjadi perubahan dan perbedaan signifikan meskipun aksesi yang dikaji berasal dari lokasi yang berbeda (Hartati
et al. 2007).
Hal menarik yang ditemukan adalah aksesi yang berasal dari Tuban memiliki nilai keragaman yang tinggi (h=0.24) dibandingkan dengan aksesi yang tumbuh liar dari daerah lain (h=0.06 hingga 0.13) (Tabel 6). Tingginya nilai keragaman populasi tarum di daerah Tuban dimungkinkan berasal dari populasi yang besar dengan keragaman genetik yang tinggi tetapi kemudian mengalami penurunan jumlah anggota sebagai akibat kerusakan lingkungan yang disebabkan oleh alih fungsi lahan maupun pelebaran jalan (Luan et al. 2006). Selain itu,
aksesi yang berasal dari Tuban dimungkinkan mampu melakukan reproductive
23
5
SIMPULAN DAN SARAN
Simpulan
Lima belas primer ISSR berhasil mengamplifikasi DNA dari 50 aksesi tarum (I. tinctoria L.) dan menghasilkan 123 pita dengan 84 pita polimorfik dan persentase polimorfisme 50-90%. Tarum (I. tinctoria L.) di pulau Jawa dan Madura yang berasal dari lokasi yang sama atau berdekatan cenderung untuk mengelompok menjadi satu yang mengindikasikan tingginya nilai koefisien kemiripan antar anggota di dalam kelompok tersebut. Keseluruhan aksesi tarum (I. tinctoria L.) terbagi menjadi empat kelompok pada koefisien kemiripan 0.61, yakni kelompok I berasal dari pulau Madura (Bangkalan, Sampang, Pamekasan, dan Sumenep), kelompok II berasal dari Jawa Timur (Tuban), kelompok III berasal dari Jawa Tengah (Bantul, Gunung Kidul, dan Kulonprogo), dan kelompok IV berasal dari Jawa Barat (Cirebon dan Serang).
Pita spesifik hasil amplifikasi setiap primer ISSR dapat digunakan untuk mengidentifikasi hampir keseluruhan asal populasi tarum di pulau Jawa dan Madura, kecuali populasi yang berasal dari Gunung Kidul belum diperoleh pita spesifik. Keragaman genetik tarum (I. tinctoria L.) di pulau Jawa dan Madura secara umum termasuk dalam kategori tinggi (68.3%), akan tetapi keragaman di dalam populasi rendah (37%) sedangkan antar populasi tergolong tinggi (63%). Nilai keragaman (h) paling tinggi dimiliki oleh populasi yang berasal dari Bantul dan Tuban (h=0.24) sedangkan nilai keragaman (h) paling rendah dimiliki oleh populasi yang berasal dari Serang (h=0.04).
Saran
24
DAFTAR PUSTAKA
Amirmoradi B, Talebi R, Karami E, Shirvani H. 2013. Evaluation of genetic diversity within and between Cicer species using ISSR molecular markers.
Int J Agr Crop Sci. 5(22):2683-2688.
Anand KK, Srivastava RK, Chaudhary LB, Singh AK. 2010. Delimitation of species of the Astragalus rhizanthus complex (Fabaceae) using molecular markers RAPD, ISSR, and DAMD. Taiwania. 55(3):197-207.
Angelini LG, Pistelli L, Belloni P, Bertoli A, Panconesi S. 1997. Rubia tinctorium as a sources of natural dyes: agronomic evaluation, quantitative analysis of alizarin and industrial assays. Industr Crops Prod. 6:303-31. Arsenault N, Rose C, Azulay A, Meyer R. 2008. Explorers, traders & immigrants:
tracking the cultural and social impacts of the global commodity trade. Texas (US): Hemispheres, The International Outreach Consortium.
Babić M, Babić V, Prodanović S, Filipović M, Andjelković V. 2012. Comparison of morphological and molecular genetic distances of maize inbreds.
Genetika. 44(1):119-128.
Backer CA, Bakhuizen van den Brink RC. 1963. Flora of Java (Spermatophytes Only) Vol I. Groningen-The Netherlands (NL): NVP Noordhoff.
Balgooy MMJ van. 1998. Malesian Seed Plant. Volume 2 – Portraits of Tree Families. Leiden (NL). Rijksherbarium/Hortus Botanicus.
Bi IZ, Maquet A, Baudoin JP. 2003. Population genetic structure of wild
Phaseolus lunatus (Fabaceae), with special reference to population sizes.
Am J Bot. 90(6): 897–904.
Bisoyi MK, Acharya L, Mukherjee AK, Panda CP.2010. Study of inter-specific relationship in six species of Sesbania Scop. (Leguminosae) through RAPD and ISSR markers. Int J Plant Physiol Biochem. 2(2):11-17.
Chanayath N, Lhieochaiphant S, Phutrakul S. 2002. Pigment extraction techniques from the leaves of Indigofera tinctoria Linn. and
Baphicacanthus cusia Brem. and chemical structure analysis of their major components. CMU J. 1(2):149-160.
Ellstrand NC, Elam DR. 1993. Population genetic consquences of small population size: implications for plant conservation. Ann Review Ecol Syst. 24:217–242.
[FAO] Food and Agriculture Organization of the United Nations. 1995. Natural colourants and dyestuffs. United States of America (US): FAO Corporate Document Repository.
Gao X, Schrire BD. 2010. Indigofera. FOC. 10:137-164.
Głowacki ED, Voss G, Leonat L, Irimia-Vladu M, Bauer S, Sariciftci NS. 2012. Indigo and tyrian purple – from ancient natural dyes to modern organic semiconductors. Isr J Chem. 52:1-12.
Hamilton MB. 2009. Population genetics. Hoboken (US): Wiley-Blackwell. Hamrick JL, Godt M J. 1996. Effects of life history traits on genetic diversity in
25 Hartati D, Rimbawanto A, Taryono, Sulistyaningsih E, Widyatmoko. 2007.
Pendugaan keragaman genetik di dalam dan antar provenan pulai (Alstonia scholaris (L.) R. Br.) menggunakan penanda RAPD. J Pemulia Tanaman Hut. 1(2):1-9.
Hedrick PW. 1985. Genetics of populations. Boston (US): Jones and Bartlett. Hedrick PW. 2001. Conservation genetics: where are we now? Trends Ecol
Evol. 16:629-636.
Herdiawan I, Krisnan R. 2014. Produktivitas dan pemanfaatan tanaman
Leguminosa pohon Indigofera zollingeriana pada lahan kering. J Wartazoa.
24(2):75-82.
Heyne K. 1987. Tumbuhan Berguna Indonesia III. Jakarta (ID): Yayasan Sarana Wana Jaya.
Huang Y, Zhang CQ, Li DZ. 2009, Low genetic diversity and high genetic differentiation in the critically endangered Omphalogramma souliei
(Primulaceae): implications for its conservation. J Syst Evol. 47(2):103–109. Indriani FC, Sudjindro AN, Sugiharto, Soetopo L. 2008. Keragaman genetik
plasma nutfah kenaf (Hibiscus cannabinus L.) dan beberapa spesies yang sekerabat berdasarkan analisis isozim. Agritek. 6(9):1793:1802.
Kara N, Korol L, Isik K, Schiller G. 1997. Genetic diversity in Pinus brutia
Ten.: altitudinal variation. Silvae Genetic. 46:155-161.
Kasmudjo, Saktianggi PP. 2011. Pemanfaatan daun Indigofera sebagai pewarna alami batik. Di dalam Prosiding Seminar Nasional Masyarakat Peneliti Kayu Indonesia (MAPEKI) XIV Pengolahan Hasil Hutan [Internet]. [University Club, Universitas Gadjah Mada 2 November 2011]. Yogyakarta (ID). Hlm 542-548; [diunduh 2014 Jan 3]. Tersedia pada: http://teknologihutan.fkt.ugm.ac.id/userfiles/download/pemanfaatan
_daun_indigofera_sebagai_pewarna_alami_batik.pdf
Khajudparn P, Prajonhjai T, Poolsawat O, Tantasawat PA. 2012. Application of ISSR markers for verification of F1 hybrids in mungbean (Vigna radiata).
Genet Mol Res. 11(3):3329-3338.
Kort ID de, Thijsse G. 1984. Revision of the genus Indigofera (Leguminosae- Papilionoideae) in Southeast Asia. Blumea. 30:89-151.
Kosman E, Leonard KJ. 2005. Similarity coefficients for molecular markers in studies of genetic relationships between individuals for haploid, diploid, and polyploid species. Mol Ecol. 14:415-424.
Kubitzki K. 2007. Introduction to Fabales. Di dalam: Kubitzki K, editor. The Families and Genera of Vascular Plants. Volume ke-9. New York (US): Springer.
Lemmens RHMJ, Wessel-Riemens PC. 1992. Indigofera L. in: Lemmens RHMJ, Wulijarni-Soetjipto N, editor. Plant Resources of South-East Asia. No 3. Dye and Tannin-Producing Plants. Bogor (ID): PROSEA.
Lin XC, Lou YF, Liu J, Peng JS. 2010. Crossbreeding of Phyllostachys species (Poaceae) and identification of their hybrids using ISSR markers. Genet Mol Res. 9:1398-1404.
Liu ZM, Zhao AM, Kang XY, Zhou SL, López-Pujol J. 2006. Genetic diversity, population structure, and conservation of Sophora moorcroftiana
26
[LTC] Life Technologies Corporation. 2010. ISSR genotyping of endangered plants using an optimized workflow. USA: O-090404-r202/2010 Publication 106AP31-01.
Luan S, Chiang T, Gong X. 2006. High genetic diversity vs. low genetic differentiation in Nouelia insignis (Asteraceae), a narrowly distributed and endemic species in China, revealed by ISSR fingerprinting. Ann Bot. 98:583-589.
Mattapha S, Chantaranothai P. 2012. The genus Indigofera L. (Leguminosae) in Thailand. Trop Nat Hist. 12(2): 207-244.
Mohanty S, Das AB, Ghosh N, Panda BB, Smith DW. 2010. Genetic diversity of 28 wild species of fodder legume Cassia using RAPD, ISSR, and SSR markers: a novel breeding strategy. J Biotech Res. 2: 44-55.
Montgomery M E, Woodworth LM, Nurthen RK, Gilligan DM, Briscoe DA, Frankham R. 2000. Relationships between population size and loss of genetic diversity: comparisons of experimental results with theoretical predictions. Conserv Genet. 1: 33-43.
Muzzazinah. 2012. Jejak evolusi dan spesiasi marga Indigofera. J Bioedukasi. 5(2):1-12.
Nagaoka T, Ogihara Y. 1997. Applicability of inter-simple sequence repeat polymorphism in wheat for use as DNA markers in comparison to RFLP and RAPD markers. Theor Appl Genet. 94:597-602.
Narain P. 2000. Genetic diversity – conservation and assessment. Curr Sci. 79(2):170-175.
Nei M. 1975. Molecular population genetics and evolution. Amsterdam (NL): North-Holland Publishing Company.
Niessen S. 2009. Batak Textile in Indonesia. Netherlands (NL): Kitlv Leiden. Pardhe DD, Satpute RA. 2011. A comparative analysis of genetic diversity in
genus Vigna savi genotypes using ISSR. Int J PharmTech Res. 3(1):464-470.
Peakall R, Smouse PE. 2012. GenAlEx 6.5: genetic analysis in excel. Population genetic software for teaching and research–an update. Bioinformatics. 28: 2537-2539.
Persson K, Bothmer R von. 2000. Assessing the allozyme variation in cultivars and Swedish landraces of rye (Secale cereale L.). Hereditas. 132:7-17. Rahayu SE, Hartana A, Chikmawati T. 2007. Genetic diversity of Pandanus and
Freycinetia from Java based on ISSR marker. Floribunda. 3(4):95-103. Rao VR, Hodgkin T. 2002. Genetic diversity and conservation and utilization of
plant genetic resources. J Plant Biotech. 68(1):1-19.
Rohlf FJ. 1998. NTSys-pc. Numerical taxonomy and mutivariate analysis system. Version 2.02. New York (US): Exeter Software.
Sadeghi A, Cheghamirza K. 2012. Efficiency of RAPD and ISSR marker system for studying genetic diversity in common bean (Phaseolus vulgaris L.) cultivars. Ann Biol Res. 3(7):3267-3273.
27 Schrire BD, Lavin M, Barker NP, Forest F. 2009. Phylogeny of the tribe
Indigofereae (Leguminosae-Papilioniodeae): geographically structure more in succulent-rich and temperate setting than in grass-rich environments. AmJ Bot. 96(4):816-825.
Sebola RJ, Balkwill K. 2013. Calibration, verification and stepwise analysis for numerical phenetics: Olinia (Oliniaceae) as an example. J Bot South Afr. 88:42-55.
Sequin-Frey M. 1981. The chemistry of plant and animal dyes. J Chem Ed. 58(4):301-305.
Song J. 2005. Genetic diversity and flowering in Clianthus and New Zealand
Sophora (Fabaceae) [disertasi]. New Zealand (NZ): Massey University. Spooner D, Treuren R van, Vicente MC de. 2005. Molecular markers for
genebank management. Maccarese-Rome (IT): International Plant Genetic Resources Institute.
Syahrudin K, Sobir, Khumaida N. 2012. Analisis keragaman beberapa genotipe Durian (Durio zibethinus Murr.) menggunakan penanda morfologi dan molekuler (ISSR). [tesis]. Indonesia (ID): Institut Pertanian Bogor.
Templeton AR. 2006. Population genetics and microevolutionary theory. New Jersey (US): John Wiley & Sons, Inc.
Tiwari KL, Jadhav SK, Gupta S. 2012. Modified CTAB Technique for Isolation of DNA from some Medicinal Plants. Res J Med Plant. 6(1):65-73.
Trojanowska MR, Balibok H. 2004. Characteristic and comparison of three classes of microsatellite-based markers and their application in plants.
Cell Moll Biol Lett. 9:221-238.
Vogel EA de. 1987. Guideline for the preparation of revision. Di dalam: EA de Vogel, editor. Manual Herbarium Theory and Practice edisi 9. Jakarta (ID): UNESCO.
Weising K, Nybom H, Wolff K, Kahl G. 2005. DNA fingerprinting in plants: principle, methods, and applications. New York (US): CRC press.
Widjaja EA, Poerba YS. 2004. Pengumpulan Data Lapangan Plasma Nutfah dan Genetik Tumbuhan. Dalam: Rugayah, Widjaja EA, Praptiwi, editor.
Pedoman Pengumpulan data Keanekaragaman Flora. Bogor (ID): Puslit-LIPI.
Wright SI, Kalisz S, Slotte T. 2013. Evolutionary consequences of selffertilization in plants. Proc R Soc B. 280:20130133.
Zietkiewicz E, Rafalski A, Labuda D. 1994. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification.
28
RIWAYAT HIDUP
Muhammad Rifqi Hariri merupakan anak ketiga dari tiga bersaudara, dilahirkan di Situbondo, Jawa Timur pada tanggal 21 Mei 1990 sebagai putra dari pasangan Bapak Syafi’i dan Ibu Huzaimah. Penulis lulus dari SMA Negeri 1 Asembagus pada tahun 2008. Pada tahun yang sama, penulis melanjutkan pendidikan sarjana Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam (FMIPA) di Universitas Negeri Malang (UM) melalui jalur SNMPTN. Penulis pernah menerima beasiswa Supersemar, PPA, dan Pertamina. Selama kuliah di strata S1, penulis pernah menjabat sebagai ketua LSO Biology Scientist Club