MORFOGENETIK POPULASI DUA SPESIES GASTROPODA
(Turbo sparverius DAN Turbo bruneus) DI BENTANG LAUT
KEPALA BURUNG PAPUA, INDONESIA
DANDI SALEKY
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Morfogenetik Populasi Dua Spesies Gastropoda (Turbo sparverius dan Turbo bruneus) di Bentang Laut Kepala Burung Papua, Indonesia adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Maret 2016
RINGKASAN
DANDI SALEKY. Morfogenetik Populasi Dua Spesies Gastropoda (Turbo sparverius dan Turbobruneus) di Bentang Laut Kepala Burung Papua, Indonesia. Dibimbing oleh HAWIS H. MADDUPPA dan ISDRAJAD SETYOBUDIANDI.
Turbo sparverius dan Turbo bruneus merupakan spesies gastropoda herbivora yang hidup di daerah intertidal berbatu yang berperan dalam menjaga populasi alga pada ekosistem intertidal. Gastropoda tersebut juga telah dimanfaatkan oleh masyarakat di Papua untuk dikonsumsi dan juga digunakan sebagai souvenir. Meskipun eksploitasi ini dapat mempengaruhi keragaman genetik dan populasi, studi tentang struktur populasi kedua spesies tersebut masih sangat sedikit.
Penelitian ini dilakukan pada bulan Oktober 2014 sampai Juni 2015. Tujuan penelitian ini adalah untuk menganalisis pola pertumbuhan, keragaman genetik, struktur populasi dan konektivitas genetik dari T. sparverius (tiga populasi) dan T. bruneus (dua populasi) di perairan Papua Barat, Indonesia. Analisis pola pertumbuhan T. sparverius dan T. bruneus dilakukan dengan mengukur panjang cangkang dan berat total. Sementara itu, analisis genetik dilakukan dengan beberapa tahapan yaitu ekstraksi, Polymerase Chain Reaction (PCR), elektroforesis dan sekuensing DNA.
Hasil penelitian menunjukkan bahwa pola pertumbuhan T. sparverius dan T. bruneus adalah allometrik negatif. Nilai keragaman haplotipe dari semua populasi T. sparverius dan T. bruneus secara berturut-turut adalah 0.657 - 0.705 dan 0.739 - 0.816. Selain itu. setiap populasi menunjukkan keragaman haplotipe yang tinggi. Struktur genetik ditemukan dalam semua populasi T. sparverius dan T. bruneus dengan nilai Fst masing-masing 0.037 - 0.201 dan 0.031. Struktur populasi dan pohon filogenetik menunjukkan kedekatan genetik akibat aliran gen dari T. sparverius dan T. bruneus. Nilai jarak genetik antara populasi T. sparverius dan T. bruneus sangat rendah masing-masing sebesar 0.002 dan 0.003 - 0.004. Kemiripan genetik terjadi karena kondisi dan arah aliran arus sebagai media transportasi gen antar populasi. Kemiripan genetik juga disebabkan oleh kesamaan habitat di setiap populasi. Studi ini menunjukkan bahwa pola pertumbuhan T. sparverius dan T. bruneus adalah allometrik negatif. Populasi T. sparverius dan T. bruneus di Papua Barat menunjukkan tingkat keragaman genetik yang tinggi.
SUMMARY
DANDI SALEKY. MorphogeneticPopulation of Two Gastropods Species (Turbo sparverius and T.bruneus) in the Bird Seascape Papua, Indonesia. Supervised by HAWIS H. MADDUPPA and ISDRAJAD SETYOBUDIANDI.
Turbo sparverius and T. bruneus are the species of herbivorous grazers gastropod that lived on the rocky intertidal area, which have a role on controlling algal population on the intertidal ecosystem. They have also been exploited by local peoples in Papua for food and souvenirs. Even though this exploitation might affect their genetic diversity and population, study on the population structure of these species are still lacking.
This study was conducted on Oktober 2014 to June 2015. The aims of this study were to analyze the growth pattern, genetic diversity, population structure and genetic connectivity of T. sparverius (three populations) and T.bruneus (two populations) in coastal water of West Papua,Indonesia. The growth pattern of T. sparverius and T. bruneus was inferred from the measurement of shell length and total weight. Meanwhile, genetic analysis was performed by some steps such as DNA extraction, Polymerase Chain Reaction (PCR), electrophoresis, and DNA sequencing.
The results of this study showed that the growth pattern of T. sparverius and T. bruneus was negative allometric. Haplotype diversity values of all population of T. sparverius and T. bruneus were 0.657 - 0.705 and 0.739 - 0.816, respectively. In addition, the haplotype diversity of each population showed a high level of diversity. The genetic structure was found in all population of T. sparverius and T. bruneus with Fst values -0.037 - 0.201 and 0.031, respectively.
Population structure and phylogenetic tree showed the closeness genetic relationship due to gene flow between both T. sparverius and T. bruneus. Genetic distance values between populations of T. sparverius and T. bruneus were very low ranged from 0.002 and 0.003 - 0.004, respectively. Genetic similarity occurs because of condition and direction of current, which act as the media for gene transport among population. The genetic similarity found here was also caused by the similarity of habitats in each population. This study showed that the growth pattern of T. sparverius and T. bruneus was negative allometric. The population of T. sparverius and T. bruneus in West Papua showed a high level of genetic diversity.
© Hak Cipta Milik IPB, Tahun 2016
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Ilmu Kelautan
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2016
DANDI SALEKY
MORFOGENETIK POPULASI DUA SPESIES GASTROPODA
(Turbo sparverius DAN Turbo bruneus) DI BENTANG LAUT
PRAKATA
Puji dan syukur penulis panjatkan kepada Tuhan Yang Maha Kuasa atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Oktober 2014 ini ialah Morfogenetik populasi dua spesies gastropoda (Turbo sparverius dan Turbo bruneus) di Bentang Laut Kepala Burung Papua, Indonesia
Terima kasih penulis ucapkan kepada Bapak Dr Hawis H. Madduppa, M.Si dan Bapak Dr Ir Isdrajad Setyobudiandi, M.Sc selaku pembimbing yang telah banyak memberikan masukan dalam penyusunan tesis ini. Penulis juga mengucapkan terima kasih kepada Prof Dr Ir Dedi Soedharma, DEA dan Dr Ir Agus Admadipoera, DESS yang telah bersedia menjadi dosen penguji dan moderator dalam sidang tesis yang juga memberikan banyak saran dan masukkan kepada penulis dalam perbaikan tesis ini. Ucapan terima kasih juga kepada Direktorat Jenderal Perguruan Tinggi yang telah bersedia memberikan beasiswa kepada penulis sepanjang masa studi. Penghargaan yang sama juga penulis berikan kepada seluruh dosen Ilmu dan Teknologi Kelautan atas bekal ilmu yang diberikan. Penulis juga mengucapkan banyak terima kasih kepada Dr Abdul Hamid A. Toha, M.Sc, Ir Muhammad Takdir, MP, teman-teman di Marine Biodiversity And Biosystematics Laboratory ITK IPB, Laboratorium Genetika UNIPA, Pascasarjana IKL angkatan 2013, Andika Ariwibowo, S.IK, Muhammad Dailami, S.Si.,M.Si, Matheos Rayar, S.IK, Bentara Papua (Meki, Maros, dan Yanu), Ikatan Mahasiswa Papua (IMAPA) dan sahabat terkasih (Alin, Amelius, Eca dan Ida). Penulis juga mengucapkan terima kasih kepada Balai Taman Nasional Teluk Cenderawasih (BTNTC) yang memberikan ijin dalam pengambilan sampel. Ungkapan terima kasih yang paling dalam saya sampaikan kepada keluarga tercinta. Semoga apa yang tertuang di dalam karya ilmiah ini bermanfaat bagi seluruh pembaca.
Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL xii
DAFTAR GAMBAR xii
DAFTAR LAMPIRAN xii
1 PENDAHULUAN
Latar belakang 1
Perumusan masalah 2
Hipotesis 3
Tujuan penelitian 3
Manfaat 3
2 METODE PENELITIAN
Waktu dan Lokasi Penelitian 3
Deskripsi Sampel 4
Pengambilan Sampel dan Pengukuran Panjang-Berat 5
Analisis Molekuler 5
Analisis Data 6
3 HASIL DAN PEMBAHASAN
Distribusi Frekuensi Panjang Cangkang 8
Hubungan Panjang-Berat 10
Rekonstruksi Filogenetik 11
Keragaman Genetik 13
Struktur Populasi 14
Distribusi Haplotipe 16
4 SIMPULAN DAN SARAN
Simpulan 18
Saran 19
DAFTAR PUSTAKA 19
LAMPIRAN 26
RIWAYAT HIDUP 38
DAFTAR TABEL
1 Total jumlah individu T. sparverius dan T. bruneus yang teramplifikasi
positif 7
3 Keragaman genetik dari spesies T. sparverius dan T. bruneus yang dilihat dari jumlah haplotipe (Hn), keragaman haplotipe (Hd), keragaman nukleotida ( ), dengan jumlah sampel (n) dan total
polimorfisme genetik dari setiap populasi 14
4 Analisis jarak genetik (D) dalam dan antar populasi T. sparverius dan
T. bruneus 15
5 Analisis uji jarak berpasangan (Fst) pada populasi T. sparverius dan T.
bruneus 15
DAFTAR GAMBAR
1 Lokasi pengambilan sampel T. sparverius dan T. bruneus di empat populasi di Papua Barat, Indonesia: Raja Ampat, Sorong, Manokwari,
dan Teluk Wondama 4
2 Bentuk morfologi T. sparverius (A) dan T. bruneus (B) 5 3 Grafik distribusi frekuensi panjang T. sparverius 8 4 Grafik distribusi frekuensi panjang T. bruneus 9 5 Analisis regresi linier terhadap panjang total dan berat total dari 105
individu T. sparverius (A) dan dari 74 individu T. bruneus (B) 11 6 Rekonstruksi pohon filogenetik T. sparverius dan T. bruneus
menggunakan metode Neighbour-Joining (NJ) dengan model Kimura
2-parameter, nilai bootstrap 1000x 12
7 Jaringan haplotipe dari T. sparverius (kiri) dan T. bruneus (kanan) dari lokasi yang berbeda di Papua Barat. Setiap haplotipe diwakili oleh lingkaran, sedangkan ukuran lingkaran menunjukkan frekuensi
haplotipe. 17
8 Arus yang melewati Pesisir Utara Papua yang diturunkan dari data
keluaran model INDESO (Atmadipoera Pers.Comm 2016). 18
DAFTAR LAMPIRAN
1 Lokasi pengambilan sampel T. sparverius dan T. bruneus 26 2 Hasil analisis regresi T. sparverius dan T. bruneus 27 3 Distribusi frekuensi panjang cangkang T. sparverius dan T. bruneus
yang digunakan pada analisis molekuler 29
4 Visualisasi Hasil PCR 29
1
PENDAHULUAN
Latar Belakang
Bentang Laut Kepala Burung Papua termasuk Teluk Cenderawasih dan Kepulauan Raja Ampat, merupakan bagian dari Coral Triangle yang memiliki keanekaragaman ikan karang tertinggi di dunia, juga terdapat sejumlah besar moluska (Veron et al. 2009; Allen & Erdmann 2009; Kartikasari et al. 2012). Berbagai jenis moluska hidup di daerah tersebut diantaranya Turbo sp. yang dikenal dengan nama lokal bia mata bulan. Bia mata bulan tersebut juga telah dimanfaatkan oleh masyarakat di Papua untuk dikonsumsi dan juga cangkang maupun operculum digunakan sebagai souvenir. T. sparverius dan T. bruneus merupakan jenis gastropoda herbivora pemakan alga, hidup pada intertidal berbatu, sering berada di dalam lubang dan celah-celah batu maupun pada rataan terumbu (Lee & Chao 2004; Quinones & Michel-Morvin 2006; Wernberg et al. 2008). T. sparverius dan T. bruneus dicirikan memiliki sebuah operculum tebal dengan variasi warna seperti hitam, hijau tua, putih dan kecoklatan (Dharma 2005). Operculum tersebut berbentuk lempeng yang bermanfaat dalam melindungi hewan tersebut ketika menarik diri ke dalam cangkangnya (Quinones & Michel-Morvin 2006). Beberapa jenis Turbo sp. seperti T. marmoratus, T. setosus dan T. argyrostomus menjadi target penangkapan di kawasan Pasifik Selatan (Kikutani et al. 2002). T. bruneus dimanfaatkan baik daging maupun cangkangnya di Kepulauan Soccoro, Meksiko (Yamaguchi 1993; Quinones & Michel-Morvin 2006).
Morfologi cangkang memiliki berbagai variasi bentuk yang memberikan kontribusi dalam identifikasi jenis, klasifikasi, informasi taksonomi dan untuk mengetahui kondisi lingkungan (Chiu et al. 2002; Urra et al. 2007; Moneva et al. 2012; Caill-Milly et al. 2012). Analisis karakter morfologi seperti hubungan panjang-berat suatu spesies dilakukan dalam usaha manajemen pengelolaan sumber daya baik ikan dan organisme lainnya, juga untuk mengetahui kondisi populasi (Turan 1999; Udo 2013). Perubahan morfologi cangkang gastropoda dapat dipengaruhi oleh beberapa faktor seperti perubahan komposisi substrat (Tan 2009), adaptasi terhadap paparan gelombang (Boulding et al.1999), pencemaran (Chiu et al. 2002; Urra et al. 2007), perlindungan diri dari predator dan perbedaan kedalaman (Olabarria & Thurston 2003; Marquez et al. 2011). Hubungan panjang-berat dan faktor kondisi banyak dianalisis pada gastropoda untuk mendapatkan indeks kondisi fisik populasi dan mengevaluasi kualitas habitat (Albuquerque et al. 2009). Analisis morfometrik dari karakter cangkang dapat mengalami kesulitan dalam membedakan spesies, hal ini terjadi dikarenakan perubahan faktor lingkungan yang dapat mempengaruhi pewarnaan dan pola morfologi, sehingga diperlukan pula pendekatan genetik (Mauro et al. 2003). Oleh karena itu, beberapa identifikasi gastropoda berdasarkan analisis genetik dilakukan untuk memperjelas identifikasi yang telah dilakukan sebelumnya (Marquez et al. 2011), dan juga pada organisme laut lainnya (Madduppa et al. 2014, 2016; Prehadi et al. 2015; Sembiring et al. 2015; Jefri et al. 2015).
2
alam dan juga dalam pemantauan keanekaragaman hayati dan konservasi (Frankham 1996; Schwartz et al. 2006). Keragaman genetik menentukan kapasitas populasi untuk beradaptasi dengan kondisi lingkungan, spesies dengan dengan kemampuan beradaptasi yang baik akan menghasilkan variasi morfologi dan genetik guna merespon perubahan lingkungan (Taylor & Aarssen 1988). Suatu populasi dengan keragaman genetik tinggi memiliki peluang lebih besar untuk kelangsungan hidup (Bonde et al. 2012). Pengetahuan tentang tingkat keragaman genetik dalam suatu populasi sangat berguna dalam usaha konservasi dan pemulihan suatu populasi, karena perubahan ukuran populasi dapat menyebabkan menurunnya variasi genetik yang dapat meningkatkan resiko kepunahan (Allentoft &O’Brien 2010;Jena et al. 2011).
Analisis struktur populasi dilakukan dalam pemahaman tentang dinamika yang terjadi pada populasi alami (Hoffman et al. 2009). Karakteristik sejarah hidup dan aliran gen suatu organisme sangat berpengaruh dalam membentuk struktur populasi suatu spesies (Storfer 1999; Hoffman et al. 2010), namun studi tersebut sering mengalami hambatan dalam melacak aliran dan kondisi larva organisme di laut (Weersing & Toonen 2009). Hal tersebut terjadi karena pola penyebaran setiap organisme berbeda akibat dari respon yang diberikan tiap organisme terhadap kondisi lingkungan dan faktor oseanografi (Crandall et al. 2008). Keanekaragaman habitat, geografis dan seleksi alam juga menjadi faktor penting dalam membentuk struktur dan diferensiasi populasi (Colson & Hughes 2004; Gaspari et al. 2013).
Konektivitas genetik menjadi kunci konservasi karena berperan penting dalam mempertahankan populasi dan pemulihan dari kerusakan (Neel 2008). Konektivitas juga menjadi perhatian penting dalam hampir semua rencana konservasi (Luque et al. 2012). Organisme bentik dengan siklus hidup yang kompleks, pertukaran individu yang menyebabkan konektivitas antar populasi terjadi terutama selama tahap larva pelagis (Cowen & Sponaugle 2009). Pemahaman tentang kompleksitas penyebaran larva sangat penting dalam usaha konservasi ekosistem laut karena tahapan larva pelagis dan kapasitas penyebaran larva bersama-sama membentuk struktur genetik suatu organisme (Avise 1998; Grantham et al. 2003; Madduppa et al. 2014).
Penelitian tentang morfometrik, keragaman genetik, struktur populasi dan konektivitas populasi T. sparverius dan T. bruneus di daerah Bentang Laut Kepala Burung Papua belum pernah dilakukan, yang mana penelitian tersebut dapat memberikan gambaran dalam manajemen potensi dan juga area konservasi suatu spesies (Gaspari et al. 2013; Japaud et al. 2013).
Perumusan Masalah
panjang-3
berat dan keragaman genetik Turbo sparverius dan Turbo bruneus masih sedikit dilakukan, dan hal ini dapat dilihat dari jumlah jurnal yang dipublikasi serta juga jumlah data genetik pada GeneBank di NCBI (National Center for Biotechnology Information). Sementara itu penelitian mengenai struktur populasi dan konektivitas genetik Turbo sparverius dan Turbo bruneus di Kepala Burung Papua belum pernah dilakukan sebelumnya. Data genetik menjadi sangat penting dalam sistem kehidupan di laut karena memungkinkan kesimpulan yang akan dibuat mengenai konektivitas dan aliran gen organisme laut lebih mudah didapatkan melalui pendekatan molekuler (Hellberg 2007). Berdasarkan hal tersebut maka ada beberapa permasalahan yang perlu dijawab dalam penelitian ini yang dirumuskan sebagai berikut:
1. Bagaimana hubungan panjang-berat dan genetik populasi Turbo sparverius dan Turbo bruneus di Bentang Laut Kepala Burung Papua.
2. Apakah kompleksitas oseanografi dan kondisi geografis memberikan pengaruh terhadap aliran gen dan kekerabatan Turbo sparverius dan Turbo bruneus di Bentang Laut Kepala Burung Papua.
Hipotesis
Hipotesis dalam penelitian ini adalah:
1. Kepulauan Raja Ampat dan Teluk Cenderawasih merupakan pusat dan sumber keanekaragaman genetik Turbo sparverius dan Turbo bruneus di Bentang Laut Kepala Burung Papua.
2. Kompleksitas oseanografi dan kondisi geografis memberikan pengaruh terhadap aliran gen dan kekerabatan Turbo sparverius dan Turbo bruneus di Bentang Laut Kepala Burung Papua.
Tujuan Penelitian
Penelitian ini bertujuan untuk menganalisis hubungan panjang-berat, keragaman genetik, struktur populasi, konektivitas genetik antar populasi Turbo sparverius dan Turbo bruneus di daerah Kepala Burung Papua, Indonesia.
Manfaat Penelitian
Hasil dari penelitian ini dapat dijadikan sebagai basis data dalam pengelolaan lestari sumber daya kelautan khususnya Turbo sparverius dan Turbo bruneus termasuk manajemen dan desain strategi konservasi dalam menghadapi eksploitasi yang berlebihan dan resiko kepunahan.
2
METODE
Waktu dan Lokasi Pengambilan Sampel
4
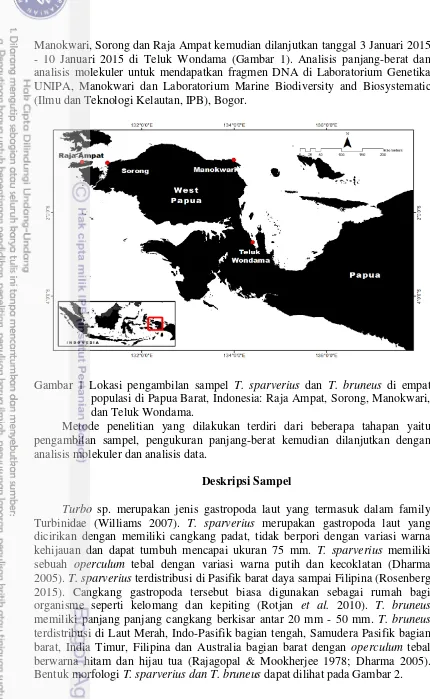
Manokwari, Sorong dan Raja Ampat kemudian dilanjutkan tanggal 3 Januari 2015 - 10 Januari 2015 di Teluk Wondama (Gambar 1). Analisis panjang-berat dan analisis molekuler untuk mendapatkan fragmen DNA di Laboratorium Genetika UNIPA, Manokwari dan Laboratorium Marine Biodiversity and Biosystematic (Ilmu dan Teknologi Kelautan, IPB), Bogor.
Gambar 1 Lokasi pengambilan sampel T. sparverius dan T. bruneus di empat populasi di Papua Barat, Indonesia: Raja Ampat, Sorong, Manokwari, dan Teluk Wondama.
Metode penelitian yang dilakukan terdiri dari beberapa tahapan yaitu pengambilan sampel, pengukuran panjang-berat kemudian dilanjutkan dengan analisis molekuler dan analisis data.
Deskripsi Sampel
5
Gambar 2 Bentuk morfologi T. sparverius (A) dan T. bruneus (B) Pengambilan Sampel dan Pengukuran Panjang-Berat
Pengambilan sampel dilakukan saat kondisi surut pada daerah intertidal berbatu (Lampiran 1). Sampel diidentifikasi secara morfologi dengan menggunakan buku identifikasi, yaitu Indonesian Shells II (Dharma 1992) dan Recent & Fossil Indonesian Shell (Dharma 2005). Sampel yang didapatkan kemudian diukur panjang cangkang dan berat total dengan menggunakan jangka sorong digital dan timbangan digital, kemudian dilanjutkan dengan mengambil sampel jaringan. Sampel jaringan T. sparverius dan T. bruneus yang didapatkan kemudian disimpan dalam tabung sampel yang berisi ethanol 96% sampai pada tahapan ekstraksi. Ukuran sampel yang digunakan dalam ekstraksi DNA memiliki ukuran yang bervariasi (Lampiran 3), sebagian besar sampel yang digunakan memiliki ukuran relatif besar karena sampel tersebut selain diekstraksi dan juga disimpan sebagai sub sampling dan dapat digunakan lagi jika diperlukan.
Analisis Molekuler
Analisis molekuler dilakukan untuk mendapatkan fragmen DNA. Analisis molekuler di laboratorium terdiri atas beberapa tahapan yaitu ekstraksi DNA, PolymeraseChainReaction (PCR), elektroforesis dan sekuensing DNA.
Ekstraksi DNA
Ekstraksi DNA menggunakan sampel jaringan kaki perut T. sparverius dan T. bruneus dengan menggunakan extraction kit (Qiagen kit, Cat No. 69504) dan larutan Chelex 10% (Walsh et al. 1991). Hasil ekstraksi selanjutnya dapat dimanfaatkan untuk proses amplifikasi.
Polymerase Chain Reaction (PCR)
Primer yang digunakan dalam penelitian ini mengacu pada penelitian Folmer et al. (1994) dengan menggunakan primer forward (LCO1490) yaitu 5’- GGTCAACAAATCATAAAGATATTGG-3’ dan primer reverse (HCO2198)
6
dalam total volume 25 µl dengan jumlah template DNA 1 - 4 µl. Dalam setiap reaksi PCR terdapat 2.5 µl 10x PCR buffer, 2 µl MgCl2 (25 mM), 2.5 µl dNTP (8 mM), 0.125 µl AmplyTaq, 1.25 µl (10 mM) masing-masing primer, 1 µl 1x BSA dan 13.5 µl ddH2O. Profil PCR meliputi denaturasi awal 94 °C selama 15 detik, denaturasi 94 °C selama 30 detik, annealing pada 50 °C selama 30 detik dan extention pada 72 °C selama 45 detik dan final extention pada 72 °C selama 10 menit, semua proses tersebut dilakukan dengan pengulangan sebanyak 40 siklus. Elektroforesis
Elektroforesis bermanfaat untuk melihat keberhasilan proses PCR. Elektroforesis dilakukan dengan menggunakan gel agarosa 1% dengan penambahan etidium bromida (EtBr). Proses elektroforesis dilakukan dengan menggunakan power supplay elektroforesis dengan tegangan 200 V, arus 400 mA selama 15 menit. Hasil elektroforesis kemudian direndam dalam larutan EtBr selama 20 menit dan dibilas dengan aquades. Selain perendaman pada larutan EtBr, dapat juga dilakukan penambahan EtBr pada gel agarosa yang sudah dipanaskan, kemudian dielektroforesis tanpa perendaman pada larutan EtBr. Selanjutnya gel divisualisasikan menggunakan lampu UV transluminator dan didokumentasikan menggunakan kamera digital.
Sekuensing DNA
Hasil PCR yang telah diamplifikasi positif kemudian akan disekuensing untuk mendapatkan urutan pasang basa sekuen. Sekuensing dilakukan dengan mengirim produk PCR ke lembaga Berkeley Sequencing Facility USA dengan menggunakan metode Sanger et al. (1977) untuk mendapatkan urutan pasang basa sekuen nukleotida.
Analisis Data
Distribusi Frekuensi Panjang
Pendugaan kelompok ukuran dilakukan dengan menganalisis frekuensi panjang. Distribusi frekuensi panjang dikelompokkan ke dalam beberapa kelompok panjang yang diasumsikan menyebar normal. Distribusi frekuensi panjang didapatkan dengan menentukkan selang kelas, nilai tengah kelas, dan frekuensi dalam setiap kelompok panjang.
Perbandingan Panjang-Berat
Hubungan panjang-berat adalah pendekatan yang paling banyak digunakan untuk memperkirakan biomassa invertebrata bentik, analisis ini dapat menduga ada tidaknya perbedaan ukuran akibat perbedaan kondisi lingkungan (Schefler 1987; Benke et al. 1999). Variabel yang diukur dalam penelitian ini adalah panjang total dan berat total, dan untuk mengetahui hubungan panjang-berat T. sparverius dan T. bruneus digunakan analisis dengan menggunakan uji regresi linear yang diproses dalam software Microsoft Excel 2010 yang mengacu rumus sebagai berikut (Efendie 1979):
W = aLb Keterangan :
7
L = Panjang tubuh (mm). a dan b = konstanta
Persamaan tersebut dapat dirubah dalam bentuk linear, yaitu sebagai berikut: Log y = a log x + log b
Nilai b = 3 menggambarkan pertumbuhan isometrik atau pertambahan panjang seimbang dengan pertambahan bobotnya. Nilai b ≠ menggambarkan pertumbuhan allometrik. Jika b kurang dari 3 menunjukkan keadaan yang kurus dimana pertambahan panjang lebih cepat dibandingkan pertambahan bobotnya. Jika b lebih dari 3 menunjukkan pertambahan bobot lebih cepat dibandingkan degan pertambahan panjangnya (Effendie 1979).
Identifikasi Spesies, Jarak Genetik dan Rekonstruksi Pohon Filogenetik Hasil sekuensing yang didapatkan kemudian akan dianalisis. Beberapa analisis yang dilakukan adalah identifikasi, keragaman genetik, struktur populasi dan konektivitas genetik antar populasi. Komposisi jumlah individu yang berhasil teramplifikasi pada tiap lokasi dapat dilihat pada Tabel 1.
Tabel 1 Total jumlah individu T. sparverius dan T. bruneus yang teramplifikasi positif
Spesies Lokasi Waktu koleksi Jumlah sampel
Turbo sparverius Manokwari 2014 20 Turbo sparverius Sorong 2014 3 Turbo sparverius Teluk wondama 2015 15 Turbo bruneus Manokwari 2014 18 Turbo bruneus Raja Ampat 2014 17
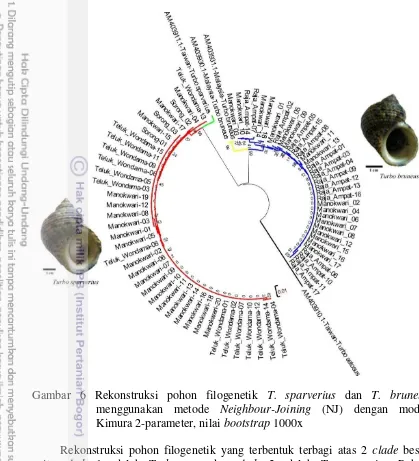
Hasil sekuensing yang didapat kemudian diedit dan diurutkan dengan menggunakan model ClustalW (1.6) (Tamura et al. 2013) pada program MEGA 6 (Moleculer Evolutionary Genetic Analysis). Data hasil sekuen nukleotida yang telah diedit kemudian dicocokkan dengan data yang tersedia pada GeneBank di NCBI (National Center for Biotechnology Information) dengan menggunakan BLAST (Basic Local Alignment Search Tool). Dilanjutkan dengan menghitung jarak genetik (D) dalam dan antar populasi dan juga merekonstruksi pohon filogenetik dengan menggunakan aplikasi Mega 6 (Tamura et al. 2013). Rekonstruksi pohon filogenetik T. sparverius dan T. bruneus menggunakan metode Neighbour-Joining (NJ) dengan model Kimura 2-parameter, nilai bootstrap 1000x.
Analisis Keragaman Genetik dan Struktur Populasi
8
lokasi. Analisis ini menggunakan aplikasi DnaSP 5.10 (Rozaz et al. 2003) sebagai dasar pengukuran tingkat keragaman genetik pada data sekuen contoh region dan analisis struktur populasi menggunakan aplikasi Arlequin 3.5 (Excoffer & Lischer 2009). Deskripsi analisis statistik adalah dengan menghitung keragaman haplotipe (Hd) dan keragaman nukleotida ) (Nei 1987). Analisis perbedaan level jarak antar populasi dilakukan dengan menggunakan Fixation Index (Fst) (Excoffier et al.1992) dengan menggunakan aplikasi Arlequin 3.5 (Excoffer & Lischer 2009). Analisis Konektivitas Antar Populasi
Konektivitas genetik yang terjadi antara masing-masing populasi dianalisis menggunakan software Network 4.6.1 (http://www.fluxusengineering.com). Analisis konektivitas genetik dilakukan untuk melihat sebaran haplotipe T. sparverius dan T. bruneus pada tiap populasi. Jumlah sampel yang digunakan dalam analisis ini adalah 38 sampel T. sparverius dari populasi Manokwari, Teluk Wondama dan Sorong. Jumlah sampel T. bruneus sebanyak 35 sampel dari populasi Manokwari dan Raja Ampat.
3
HASIL DAN PEMBAHASAN
Distribusi Frekuensi Panjang
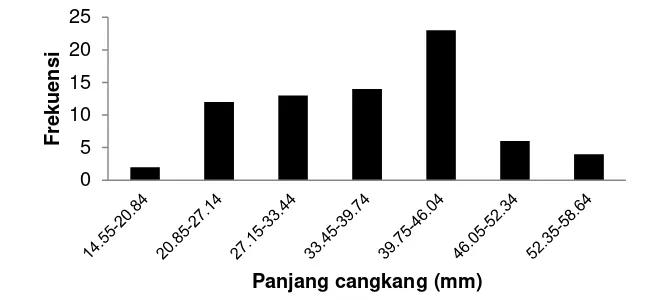
Pengukuran secara keseluruhan T. sparverius dikelompokkan kedalam 8 kelas ukuran panjang, sedangkan T. bruneus terdiri dari 7 kelas ukuran panjang. Ukuran cangkang terpanjang pada T. sparverius yaitu 66.19 dengan frekuensi tertinggi diperoleh pada kisaran ukuran 52.75 - 57.80 berasal dari populasi Manokwari sebanyak 16 individu. Ukuran cangkang terkecil T. sparverius yaitu 27.45 dengan frekuensi terendah pada kelas ukuran 27.55 - 32.50 dan 32.51 - 37.56 tersebar pada populasi Manokwari, Sorong dan Teluk Wondama. Grafik distribusi frekuensi panjang cangkang T. sparverius terlihat pada Gambar 3.
9
populasi Manokwari. Grafik ukuran frekuensi panjang T. bruneus terlihat pada Gambar 4.
Gambar 4 Grafik distribusi frekuensi panjang cangkang T. bruneus Grafik distribusi frekuensi panjang menunjukan bahwa T. sparverius memiliki jumlah kelas ukuran lebih banyak dan ukuran cangkang lebih panjang dari T. bruneus. Cangkang gastropoda T. sparverius seringkali digunakan sebagai tempat hidup bagi organisme seperti kelomang dan kepiting dengan panjang aperture dapat mencapai 6 - 7.5 cm (Rotjan et al. 2010). Ukuran cangkang T. bruneus dapat berkisar antara 31.9 – 35.1 mm (Tan 2009). Ramesh et al. (2010) mengemukakan bahwa panjang cangkang T. bruneus dapat mencapai 53 mm. Perbedaan ukuran cangkang gastropoda pada tiap lokasi dapat berbeda diakibatkan faktor habitat. Kondisi habitat yang berubah dapat mempengaruhi ukuran cangkang dan kepadatan T. bruneus (Tan 2009),
10
Hubungan Panjang-Berat
Hubungan panjang-berat merupakan faktor-faktor yang digunakan dalam penelitian biologi perikanan dalam menggambarkan perubahan ukuran individu, menunjukkan pola pertumbuhan dari suatu organisme dan untuk mendapatkan indeks kondisi fisik populasi dan mengevaluasi kualitas habitat (Gayon 2000; Albuquerque et al. 2009). Hasil analisis pola pertumbuhan T. sparverius dan T. bruneus dapat dilihat pada Tabel 2.
Tabel 2 Hasil perhitungan hubungan panjang-berat T. sparverius dan T. bruneus dengan jumlah sampel (n), Nilai Konstanta (a), Nilai Indeks Pertumbuhan (b), Nilai Korelasi (r), dan Nilai Determinasi (R2)
Jenis Lokasi n a b r R2 menunjukkan hubungan linier dengan persamaan garis Y = 2.4672x-2.6569 dengan nilai R² = 0.799. Grafik regresi linear terhadap panjang dan berat total T. sparverius dan T. bruneus disajikan pada Gambar 5.
11
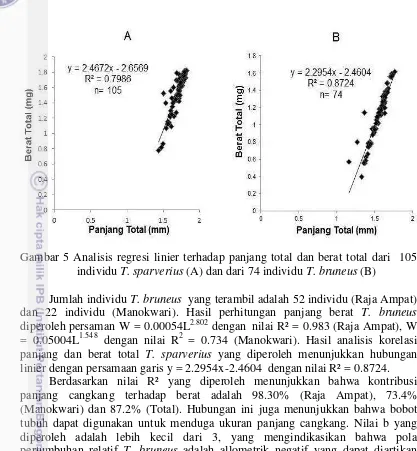
Gambar 5 Analisis regresi linier terhadap panjang total dan berat total dari 105 individu T. sparverius (A) dan dari 74 individu T. bruneus (B)
Jumlah individu T. bruneus yang terambil adalah 52 individu (Raja Ampat) dan 22 individu (Manokwari). Hasil perhitungan panjang berat T. bruneus diperoleh persaman W = 0.00054L2.802 dengan nilai R² = 0.983 (Raja Ampat), W = 0.05004L1.548 dengan nilai R2 = 0.734 (Manokwari). Hasil analisis korelasi panjang dan berat total T. sparverius yang diperoleh menunjukkan hubungan linier dengan persamaan garis y = 2.2954x-2.4604 dengan nilai R² = 0.8724.
Berdasarkan nilai R² yang diperoleh menunjukkan bahwa kontribusi panjang cangkang terhadap berat adalah 98.30% (Raja Ampat), 73.4% (Manokwari) dan 87.2% (Total). Hubungan ini juga menunjukkan bahwa bobot tubuh dapat digunakan untuk menduga ukuran panjang cangkang. Nilai b yang diperoleh adalah lebih kecil dari 3, yang mengindikasikan bahwa pola pertumbuhan relatif T. bruneus adalah allometrik negatif yang dapat diartikan bahwa penambahan berat lebih lambat dari pada panjang.
Hasil penelitian Ramesh et al. (2009) menunjukan pola pertumbuhan T. bruneus adalah allometrik negatif. Pola pertumbuhan allometri negatif juga didapat pada beberapa jenis gastropoda seperti Lambis-lambis (Jaykhumar et al. 2011), Achatina fulica (Albuquerque et al. 2009), Litorina sp. (McKinney et al. 2004), Tympanotonus fuscatus (Udo 2013). Berbagai faktor yang berpengaruh terhadap perubahan morfometrik moluska adalah variasi pasang surut, availability makanan, perubahan musim dan pematangan seksual (Ramesh et al. 2009).
Rekonstruksi Filogenetik
12
Gambar 6 Rekonstruksi pohon filogenetik T. sparverius dan T. bruneus menggunakan metode Neighbour-Joining (NJ) dengan model Kimura 2-parameter, nilai bootstrap 1000x
Rekonstruksi pohon filogenetik yang terbentuk terbagi atas 2 clade besar yaitu clade 1 adalah T. bruneus dan clade 2 adalah T. sparverius. Pohon filogenetik T. bruneus dengan nilai bootstrap 99 menunjukkan T. bruneus terbagi atas 2 clade yaitu clade 1 terdiri dari T. bruneus asal Papua Barat sedangkan clade kedua adalah T. bruneus asal Malaysia. Pohon filogenetik T. sparverius terbagi atas 2 clade yaitu clade T. sparverius asal Papua Barat dan clade T. sparverius asal Taiwan dengan nilai bootstrap 99.
Secara keseluruhan hasil analisis filogenetik memperlihatkan terjadinya percampuran antar individu dari seluruh populasi di Papua Barat baik populasi T. sparverius maupun populasi T. bruneus (Gambar 6). Hal ini juga didukung oleh nilai jarak genetik antar populasi (Tabel 4) dan nilai Fst yang rendah (Tabel 5).
13
Brown et al. (1982) mengemukakan jarak genetik antara spesies yag berbeda berkisar 9 - 19%.
Jarak antar lokasi dan faktor oseanografi diduga mempengaruhi jarak genetik antar lokasi baik Papua Barat, Malaysia dan Taiwan. Karakteristik sejarah hidup dan aliran gen suatu organisme sangat berpengaruh dalam membentuk struktur genetik populasi suatu spesies (Storfer 1999; Hoffman et al. 2010). Faktor habitat juga diduga berpengaruh terhadap perbedaan genetik antar populasi papua barat dan populasi lainnya. Perbedaan kondisi lingkungan dapat mengakibatkan terjadinya perubahan karakter morfologi, anatomi dan filogenetik dari suatu populasi (Twindiko et al. 2013).
Keragaman Genetik
Hasil analisis panjang fragmen DNA dari semua sampel T. sparverius dan T. bruneus diperoleh panjang fragmen 656 bp pada lokus Cytocrom Oksidase I (COI) dengan kemiripan sebesar 98 - 99% (Lampiran 5), ukuran panjang basa tersebut juga ditemukan pada jenis - jenis gatropoda Turbinidae (Williams 2007). Komposisi nukleotida yang banyak ditemukan pada fragmen Gen COI T. sparverius adalah adalah nukleotida T (Timin) sebesar 39.9% dan terendah nukleotida C (Cytosine) sebesar 16.1%. Nukleotida terbanyak yang ditemukan pada fragmen COI T. bruneus adalah T sebesar 40.4% dan terendah adalah C sebesar 15.6%. Kandungan G+C dari seluruh sampel T. sparverius dan T.bruneus memiliki jumlah rata-rata 36.8% dan 35.3%. Jumlah ini lebih rendah jika dibandingkan dengan kandungan A+T yang memiliki jumlah rata-rata 63.2 % dan 64.7%. Rendahnya kandungan G+C ini memudahkan kita dalam proses amplifikasi (Mamedov et al. 2008).
Terdapat beberapa perbedaan nukleotida (polimorfisme) dalam fragmen DNA T. sparverius dan T. bruenus yang diakibatkan adanya mutasi titik (Lampiran 6), disebut mutasi titik dikarenakan setiap mutasi hanya terjadi pada satu nukleotida (Xiao et al. 2007). Subtitusi transisi merupakan subtitusi antara basa A dan G (Purin) atau C dan T (Pyrimidin), sedangkan mutasi transversi terjadi ketika suatu nukleotida kelompok purin termutasi menjadi nukleotida kelompok pirimidin atau sebaliknya (Graur 2003). Jumlah mutasi pada T. sparverius sebanyak 5 titik mutasi dan jenis mutasi yang terjadi adalah transisi, sedangkan jumlah mutasi yang terjadi pada T. bruneus sebanyak 15 titik mutasi dengan komposisi 14 jenis mutasi adalah transisi dan 1 jenis mutasi tranversi. Subtitusi transisi lebih sering terjadi daripada subtitusi transversi (Santos et al. 2003) dan tingkat transisi antara Pirimidin (C dan T) yang lebih tinggi daripada antar Purin (A dan G) (Castro et al. 1998).
14
Tabel 3 Keragaman genetik dari spesies T. sparverius dan T. bruneus yang dilihat dari jumlah haplotipe (Hn), keragaman haplotipe (Hd), keragaman nukleotida ( ), dengan jumlah sampel (n) dan total polimorfisme genetik dari setiap populasi
Spesies Populasi n Keragaman Genetik
Hn Hd
Keragaman haplotipe (Hd) pada tiap populasi menunjukkan tingkat keragaman haplotipe yang relatif tinggi dengan kisaran 0.657 - 0.816. Keragaman T. sparverius terendah pada populasi Manokwari sebesar 0.657 sedangkan tertinggi berasal dari populasi Teluk Wondama sebesar 0.705. Keragaman terendah T.bruneus berasal dari populasi Manokwari sebesar 0.739 dan tertinggi adalah populasi Raja Ampat sebesar 0.816. Keragaman nukleotida ( T. sparverius berkisar antara 0.0018 - 0.0021 dengan nilai terendah pada populasi Manokwari dan tertinggi pada populasi Teluk Wondama dan Sorong. Keragaman nukleotida ( T. bruneus berkisar 0.0027 – 0.0037 dengan nilai terendah pada populasi Raja Ampat dan tertinggi berasal dari populasi Manokwari. Hasil penelitian ini menemukan bahwa keragaman nukleotida T. sparverius dan T. bruneus lebih rendah daripada spesies gastropoda laut lainnya seperti Lunnela gradulata = 0.0046). Keragaman genetik dapat ditentukan oleh 2 faktor yaitu eksploitasi yang berlebihan dan kondisi habitat (Chiu et al. 2013). T. sparverius dan T. bruneus banyak dimanfaatkan oleh masyarakat sekitar, baik dikonsumsi maupun untuk kerajinan tangan. Tingkat ekspoitasi yang tinggi tersebut dapat menurunkan tingkat keanekaragaman genetik spesies tersebut.
Struktur Populasi
15
di daerah tersebut karena penyebaran tahap planktonik dan sirkulasi arus laut sangat penting dalam proses pertukaran genetik di antara populasi (Chiu et al. 2013).
Tabel 4 Analisis jarak genetik (D) dalam dan antar populasi T. sparverius dan T. bruneus
Spesies Populasi Manokwari Teluk wondama Sorong T. sparverius
16
Aliran gen berhubungan dengan isolasi geografis yang dipengaruhi oleh jarak geografis dan kompleksitas keanekaragaman lingkungan (Arnaud et al. 1999). Kedekatan secara genetik ini juga dapat terjadi karena semua populasi memiliki jarak lokasi yang cukup dekat juga didukung dengan adanya arus New Guinea Coastal Current (NGCC) yang melewati daerah utara Papua yang dapat menjadi media tansport larva T. sparverius dan T. bruneus. Kesamaan secara genetik juga dapat diakibatkan karena kesamaan habitat pada tiap populasi, gastropoda yang berasal dari kondisi habitat yang berbeda dapat memiliki morfologi dan genetik yang berbeda pula (Urra et al. 2003).
Konektivitas Populasi
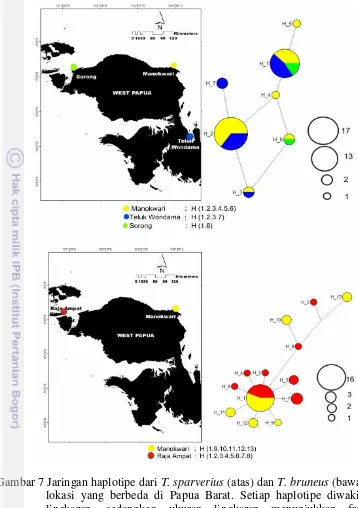
Hubungan (network) antar haplotipe T. sparverius yang didapatkan dari 3 populasi (Gambar 7) terlihat bahwa populasi Manokwari memiliki jumlah haplotipe tertinggi dari populasi Teluk Wondama dan Sorong. Terdapat haplotipe yang sama yang terdapat diantara populasi-populasi terbebut yaitu H 1, H2, H3 dan H6, sedangkan H5, H4, dan H7 merupakan haplotipe yang hanya terdapat pada Teluk Wondama maupun Manokwari. Jaringan haplotipe T. bruneus dari populasi Raja Ampat dan Manokwari (Gambar 7) terlihat bahwa populasi Raja Ampat memiliki jumlah haplotipe tertinggi dan hanya haplotipe 1 yang terdapat pada kedua populasi.
Terjadinya percampuran haplotipe dapat diakibatkan karena larva dispersal yang terbawa oleh arus laut. Arus New Guinea Coastal Curret (NGCC) (Gambar 8) mengalir sepanjang pesisir utara Papua yang merupakan arus permukaan yang disebabkan oleh pengaruh musim (Kuroda 2000). Wyrtki (1961) menyatakan bahwa pola sirkulasi di perairan utara Papua memiliki variabilitas musiman yang kuat yang mengalir terus sepanjang tahun. Keadaan tersebut sangat berpengaruh terhadap penyebaran organisme laut diwilayah pesisir utara Papua. Organisme bentik dengan siklus hidup yang kompleks, pertukaran individu yang menyebabkan konektivitas antar populasi terjadi terutama selama tahap larva pelagis (Cowen & Sponaugle 2009).
17
Gambar 7 Jaringan haplotipe dari T. sparverius (atas) dan T. bruneus (bawah) dari lokasi yang berbeda di Papua Barat. Setiap haplotipe diwakili oleh lingkaran, sedangkan ukuran lingkaran menunjukkan frekuensi haplotipe.
18
yang berhubungan satu dengan yang lain (Noreen et al. 2009). Isolasi ekologi dan struktur geografis turut mempengaruhi struktur populasi genetik (Crispo & Chapman 2008). Jarak antar populasi cenderung menyebabkan populasi yang berdekatan memiliki kemiripan daripada populasi yang jauh (Palumbi 2003).
Gambar 8 Arus yang melewati Pesisir Utara Papua yang diturunkan dari data keluaran model INDESO (Atmadipoera Pers.Comm 2016).
Variasi genetik antar populasi tersebut memiliki nilai relatif tinggi dan tidak berbeda jauh antar satu populasi dengan populasi yang lain. Isolasi geografis. sejarah populasi, dinamika oseanografi dan sifat-sifat ekologi dapat menyebabkan tingginya variasi keragaman genetik di antara populasi (Chiu et al. 2013; Silva et al. 2013). Kondisi mikrohabitat yang relatif sama yaitu intertidal berbatu turut mendukung terjadinya percampuran gen diantara populasi T. sparverius dan T. bruneus. Albaina et al. (2012) menyatakan kondisi mikrohabitat menjadi faktor yang penting dalam penyebaran diantara spesies yang berbeda, meskipun memiliki pola daur hidup dan potensi penyebaran yang sama.
4
SIMPULAN DAN SARAN
Simpulan
19
haplotipe dan nilai keragaman genetik yang tinggi. Struktur populasi dan pohon filogenetik memperlihatkan kedekatan secara genetik akibat adanya aliran gen antar populasi baik spesies T. sparverius maupun T. bruneus hal ini didukung pula dengan pola rekonstruksi pohon yang memperlihatkan keeratan individu-individu dari populasi T. sparverius dan T. bruneus.
Saran
Perlunya penelitian lebih lanjut pada jenis-jenis Turbo sp. lain seperti T. marmoratus yang dilindungi dan menurut masyarakat setempat jenis tersebut sudah sangat jarang ditemui di alam. Penulis juga menyarankan dilakukan penelitian lanjut dengan menambahkan jumlah sampel dan jumlah lokasi di Papua khususnya. Analisis morfometrik juga dapat dilakukan dengan menambahkan karakter-karakter terukur lainnya.
DAFTAR PUSTAKA
Atmadipoera AS. 2016. Rataan (2008-2015) suhu dan vektor arus pada kedalaman 5 m, yang diturunkan dari data keluaran model INDESO. Pers.Comm.
Albaina N, Olsen JL, Couceiro L, Ruiz JM, Barreiro R. 2012. Recent history of the European Nassarius nitidus (Gastropoda): phylogeographic evidence of glacial refugia and colonization pathways. Mar Biol. 159: 1871–1884. Albuquerque FS, Peso-Aguiar MC, Assuncao-Albuquerque MJT, Galvez L. 2009.
Do climate variables and human density affect Achatina fulica (Bowditch) (Gastropoda: Pulmonata) shell length, total weight and condition factor?. Braz J Biol. 69: 879-885.
Allen GR, Erdmann MV. 2009. Reef fishes of the Bird’s Head Peninsula. West Papua, Indonesia. Check List: J Spec List Dist. 5(3): 587-628.
Allentoft ME, O’Brien. 2010. Global Amphibian Declines, Loss of Genetic Diversity and Fitness: A Review. Diversity. 2: 47-71.
ArnaudJF, MadecL, Bellido A, Guiller A. 1999. Microspatial genetic structure in the land snail Helix aspersa (Gastropoda: Helicidae). Heredity. 83: 110– 119; doi:10.1046/j.1365-2540.1999.00565.x.
Avise JC. 1998. The history and purview of phylogeography: A personal reflection. Mol Ecol. 7: 371–379.
Benke AC, Huryn AD, Smock LA, Wallace JB. 1999. Length-mass relationships for freshwater macro invertebrates in North America with particular reference to the southeastern United States. J N Am Benthol Soc. 18 (3): 308-343.
Bonde RK, Mc Guire PM, Hunter ME. 2012. A review of the key genetic tools to assist imperiled species conservation: analyzing West Indian manatee populations. JMATE. 5: 1.
20
Brown WM, Prager EM, Wang A, Wilson AC. 1982. Mitochondrial DNA sequences of primates: tempo and mode of evolution. J Mol Evol. 18: 225-39.
Caill-Milly N, Bru N, Mahe K, Borie C, D’amico F. 2012. Shell Shape Analysis and Spatial Allometry Patterns of Manila Clam (Ruditapes philippinarum) in a Mesotidal Coastal Lagoon. J Mar Biol. 2012: 11.
Castro JA, Picornell A, Ramon M. 1998. Mitochondrial DNA: a tool for populational genetics studies. Int Micro 1: 327–332.
Chiu Y, Bor H, Tan M, Lin H, Jean C. 2013. Phylogeography and Genetic Differentiation among Populations of the Moon Turban Snail Lunella granulata Gmelin. 1791 (Gastropoda: Turbinidae). Int J Mol Sci. 14: 9062-9079.
Chiu Y, Chen H, Lee S, Chen CA. 2002. Morphometric Analysis of Shell and Operculum Variations in the Viviparid Snail. Cipangopaludina chinensis (Mollusca: Gastropoda). in Taiwan. Zoo Stud. 41(3): 321-331.
Colson I, Hughes RN. 2004. Rapid recovery of genetic diversity of dogwhelk (Nucella lapillus L.) populations after local extinction and recolonization contradicts predictions from life-history characteristics. Mol Ecol. 13: 2223– 2233.
Cowen RK, Sponaugle S. 2009. Larval Dispersal and Marine Population Connectivity. Ann Rev MarSci. 1: 443–466.
Crandall ED, Frey MA, Grosberg RK, Barber PH. 2008. Contrasting demographic history and phylogeographical patterns in two Indo-Pacific gastropods. Mol Ecol. 17: 611–626.
Crispo E, Chapman LJ. 2008. Population Genetic Structure Across Dissolved Oxygen Regimes In An African Cichlid Fish. Mol Ecol. 17: 2134–2148. Crowe T. 1996. Different effects of microhabitat fragmentation on patterns of
Dispersal of an intertidal gastropod in two habitats. J Exp Mar Biol Ecol. 206 : 83-107.
Dharma B. 1992. Indonesian Shells II . Sarana Graha. Jakarta.
Dharma B. 2005. Recent & Fossil Indonesian Shell. PT. Ikrar Mandiriabadi. Indonesia.
Diaz-Ferguson E, Haney R, Wares J, Silliman B. 2010. Population Genetics of a Trochid Gastropod Broadens Picture of Caribbean Sea Connectivity. PloS ONE. 5: e12675. doi:10.1371/journal.pone.0012675
Diaz-Ferguson E, Haney RA, Wares JP, Silliman BR. 2011. Genetic Structure And Connectivity Patterns Of Two Caribbean Rocky Intertidal Gastropods. J Mollusc Stud. 0: 1–7.
Dwiono SPA, Pradina, Makatipu PC. 2001. Spawning and seed production of the green snail (Turbo marmoratus L.) in Indonesia. SPC Trochus Information Bulletin. 7 January 2001.
Effendi MI. 1979. Metoda biologi Perikanan. Yayasan Agromedia. IPB. Bogor. Excoffier L, Smouse PE, Quattro JM. 1992. Analysis of molecular variance
inferred from metric distance among DNA haplotype; application to human mitochondrial DNA restricion data. Genetic.131: 479-491.
21
Folmer O, Black M, Hoeh W, Lutz R, Vrijenhoek R. 1994. DNA primers for amplification of mitochondrial cytochrome oxidase subunit I from diverse metazoan invertebrates. Mol Mar Bio Biotech. 3: 294–299.
Frankham R. 1996. Relationship of Genetic Variation to Population size in Wildlife. Con Biol. 6: 1500-1508.
GaspariS, Holcer D, Mackelworth P, Fortuna C, Frantzis A, GenovT, Vighi M, Natali C, Rako N, BanchiE, Chelazzi G, Ciofi C. 2013. Population genetic structure of common bottlenose dolphins (Tursiops truncatus) in the adriatic sea and contiguous regions: implications for international conservation. Aquatic Conserv: Mar Freshw Ecosyst. 2013.
Gayon J. 2000. History of the consept of allometry. American Zoologist. 40: 748-758.
Grande C, Templado J, Cervera JL, Zardoya R. 2004. Molecular Phylogeny of Euthyneura (Mollusca: Gastropoda). Mol Biol Evol. 21: 303–313.
Grantham BA, Eckert GL, Shanks AL. 2003. Dispersal potential of marine invertebrates in diverse habitats. Ecol App. 13(1): 108–116.
Graur D. 2003. Single-base Mutation. Nature Encyclopedia Of The Human Genome. Macmillan Publishers Ltd, Nature Publishing Group.
Hebert PDN, Ratnasingham S, deWaard JR. 2003. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species. Proceedings of the Royal Society of London Series B-Biological Sciences. 270: 313-322.
Helmuth B, Veit RR, Holberton R. 1994. Long-distance dispersal of a subantarctic brooding bivalve (Gaimardia trapesina) by kelp-rafting. Mar Biol. 120: 421-426.
Hellberg ME. 2007. Footprints on water: the genetic wake of dispersal among reefs. Coral Reefs. 26: 463–473.
Hendrich L, Pons J, Ribera I, Balke M. 2010. Mitochondrial Cox1 Sequence Data Reliably Uncover Patterns of Insect Diversity But Suffer from High Lineage- Idiosyncratic Error Rates. PLoS ONE. 5: e14448.
Hickman CS, Porter SS. 2007. Nocturnal swimming. aggregation at light traps. and mass spawning of scissurellid gastropods (Mollusca: Vetigastropoda). Inv Biol. 126 (1): 10–17.
Hoffman JI, Dasmahapatra KK, Amos W, Phillips CD, Gelatt TS, Bickham JW. 2009. Contrasting patterns of genetic diversity at three differentmgenetic markers in a marine mammal metapopulation. Mol Ecol. doi: 10.1111/j.1365-294X.2009.04246.x.
Hoffman JI, Clarke A, Linse K, Peck LS. 2010. Evects of brooding and broadcasting reproductive modes On the population genetic structure of two antarctic gastropod Molluscs. Mar biol. 158: 287–296.
Japaud A, Fauvelot C, Bouchon C. 2013. Populations Genetic Study of the Corals Acropora palmata and Acropora cervicornis of Guadeloupe (French West Indies) in View of Their Preservation. Proceedings of the 66 th Gulf and Caribbean Fisheries Institute. November 4 – 8, 2013 Corpus Christi, Texas USA.
22
Jefri E, Zamani NP, Subhan B, Madduppa HH. 2015. Molecular phylogeny inferred from mitochondrial DNA of the grouper Epinephelus spp. in Indonesia collected from local fish market. BIODIVERSITAS. 16: 254-263. Jena SN, Srivastava A, Singh UM, Roy S, Banerjee N, Rai KM, Singh SK, Kumar
V, Chaudhary LB, Roy JK, Tuli R, Sawant SV. 2011. Analysis of genetic diversity, population structure and linkage disequilibrium in elite cotton (Gossypium L.)germplasm in India. Crop & Past Sci. 62: 859–875.
Jusmaldi, Duryadi D, Affandi R, Rahardjo MF, Gustiano R. 2014. Kode batang DNA ikan hias genus Kryptopterus asal Sungai Mahakam Kalimantan Timur. J Iktiologi Ind. 14: 191-199.
Kashino J, Ueki I, Kuroda Y, Purwandani A. 2007. Ocean Variability North of New Guinea Derived from TRITON Buoy Data. J OCEAN. 63: 545-559. Kashino Y, Atmadipoera A, Kuroda Y, Lukijanto. 2013. Observed features of the
Halmahera and Mindanao Eddies. J GEOPHYS RES: OCEANS. 118: 6543– 6560.
Kartikasari SN, Marshall AJ, Beehler BM. 2012. Ekologi Papua. Yayasan Pustaka Obor Indonesia dan Conservation International. Jakarta.
Kikutani K, Ohba H, Yamakawa H. 2002. Distribution and Gut Contents of the Green Snail Turbo marmoratus in Tokunoshima Island. Ryukyus (Southern Japan). J Tokyo Uni Fish. 88: 47-52.
Kuroda Y. 2000. Variability of the currents off the northern coast of New Guinea. J Ocean. 56: 103–116.
Lee S, Chao S. 2004. Shallow-water Marine Shells from Kenting National Park. Taiwan. Coll and Res. 17: 33-57 33.
Lin T, Liu L. 2008. Low Levels of Genetic Differentiation among Populations of the Coral-Inhabiting Snail Coralliophila violacea (Gastropoda: Coralliophilidae) in Regions of the Kuroshio and South China Sea. Zoo Stud. 47(1): 17-24.
Luque S, Saura S, Fortin M. 2012. Landscape connectivity analysis for conservation: insights from combining new methods with ecological and genetic data. Landscape Ecol. 27: 153–157.
Madduppa HH, Timm J, Kochzius M. 2014. Interspecific, Spatial and Temporal Variability of Self-Recruitment in Anemone fishes. PLoS ONE. 9: e90648. Madduppa H, Ayuningtyas RU, Subhan B, Arafat D, Prehadi. 2016. Exploited but
unevaluated: DNA Barcoding reveals skates and stingrays (Chordata, Chondrichthyes) species landed in the Indonesian fish market. IJMS. 00(0): 000-000. DOI: 10.14710/ik.ijms.0.0. identifying the Mediterranean limpets Patella caerulea, Patella aspera and Patella rustica. J Exp Mar Biol Ecol. 295: 131– 143.
23
McKinney RA, Glatt SM, McWilliams SR. 2004. Allometric lenght-weight relationship for benthic prey of aquatic wildlife in coastal marine habitat. Wildlife biol. 10: 4.
Moneva CSO, Torres MAJ, Demayo CG. 2012. Relative Warp and Correlation Analysis based on Distances of the Morphological shell shape patterns among freshwater gastropods (Thiaridae: Cerithimorpha). AACL BIOFLUX 5: 3.
Neel MC. 2008. Patch connectivity and genetic diversity conservation in the federally endangered and narrowly endemic plant species Astragalusalbens (Fabaceae). Biol Cons. 141: 938-955.
Nei M. 1987. Molecular Evolutionary Genetics. Columbia Univ Press. New York. Newcomb JM, Lawrence KA, Watson WH. 2004. The Influence of Light On Locomotion In the Gastropod Melibe leonina. Mar Fresh Behav Physiol. 37 (4): 253–269.
Noreen AME, Harrison PL, Van Oppen MJH. 2009. Genetic diversity and connectivity in a brooding reef coral at the limit of its distribution. Proc R Soc B. 276: 3927–3935.
Olabarria C, Thurston MH. 2003. Patterns of morphological variation of the deep-sea Gastropod Troschelia berniciensis (King. 1846) (Buccinidae) from the Northeastern Atlantic Ocean. J Moll Stud. 70: 59–66.
Palumbi SR. 2003. Population Genetics, Demographic Connectivity, and the Design of marine reserves. Eco App. 13: 46–158.
Prehadi, Sembiring A, Kurniasih EM, Rahmad, Arafat D, Subhan B, Madduppa HH. 2015. DNA barcoding and phylogenetic reconstruction of shark species landed in Muncar fisheries landing site in comparison with Southern Java fishing port. Biodiversitas. 16 : 55-61.
Quinones OEH, Michel-Morfin JE. 2006. Population structure and accompanying biota of the snail Turbo (Callopoma) funiculosus (Gastropoda: Turbinidae). on Socorro Island. Revillagigedo Archipelago. Mexico. Int J Trop Biol. 54 (4): 1079-1084.
Rajagopal AS, Mookherjee HP. 1978. Contributions to the molluscan fauna of India. Pt. I. Marine molluscs of the Coromandel Coast, Palk Strait and Gulf of Mannar - Gastropoda: Archaeogastropoda. Rec Zoo Surv India. 12: 1-48. Ramesh R, Ravichandran S, Rameshkumar G. 2009. Analysis of age and growth
rate of Turbobruneus. World J Of Dairy & Food Sci. 4: 56-64. Coenobita clypeatus hermit crabs. Behav Ecol. doi:10.1093/beheco/arq027 Rozas JJ, Sanchez-DelBarrio C, Messequer X, Rozas R. 2003. DnaSP. DNA
polymorphism analyses by the coalescent and other methods. Bioinformatics. 19: 2496-2497.
Sanger F, Nicklen S. Coulson AR. 1977. DNA sequencing with chain-terminating inhibitors. National Academical Science, USA. 74 (12): 5463-5467.
24
of South America as revealed by mtDNA analysis. Gen & Mol Biol 26: 151-161.
Schefler WC. 1987. Statistika untuk biologi. farmasi. kedokteran. dan ilmu yang bertautan. Edisi Bahasa Indonesia. Institut Teknologi Bandung. Bandung. 279 pp.
Sembiring A, Pertiwi NPD, Mahardini A, Wulandari R,Kurniasih EM, Kuncoro AW, Cahyani NKD, Anggoro AW, Ulfa M, Madduppa H, Carpenter KE, Barber PH, Mahardika GH. 2015. DNA barcoding reveals targeted fisheries for endangered sharks in Indonesia. Fish Res. 164: 130-134.
Schwartz MK, Luikart G, Waples RS. 2006. Genetic monitoring as a promising tools for conservation and management. Tren Eco Evo. 22: 1.
Silva SE, Silva IC, Madeira C,Sallema R, Paulo OS, Paula J. 2013. Genetic and morphological variation in two littorinid gastropods: evidence for recent population expansions along the East African coast. Biol J Linn Soc.108: 494–508.
Storfer A. 1999. Gene Flow and Endangered Species Translocations: a Topic Revisited. Biol Cons. 87:173-180.
Sun Y, Li Q, Kong L, Zheng X. 2012. DNAbarcoding of Caenogastropoda along coast of China based on the COI gene. Mol Eco Res. 12: 209–218.
Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. 2013. MEGA5: Molecular Evolutionary Genetics Analysis Using Maximum Likelihood. Evolutionary Distance. and Maximum Parsimony Methods. Mol Biol Evol. 28 (10): 731-2739.
Tan CKW. 2009. Effects of trenching on shell size and density of Turbo brunneus (Gastropoda: Turbinidae) and Monodonta labio (Gastropoda: Trochidae) at Labrador Beach. Singapore. Nat Singapore. 2: 421–429.
Taylor DR, Aarssen LW. 1988. An interpretation of phenotypic plasticity in agropyron repens (Gramminae). Am J Bot. 75: 401-413.
Turan C. 1999. A Note on the examination of morphometric differentiation among fish populations: the truss system. Tr J of Zoology . 23: 259-263.
Twindiko AFS, Wijayanti DP, Ambariyanto. 2013. Studi Filogenetik Ikan Karang Genus Pseudochromis dan pictichromis di Perairan Indo-Pasifik. Bul Osea Marina. 2: 29–37.
Udo PJ. 2013. Length-Weight / Girth Relationship and Condition Factor of the Periwinkle Tympanotonus fuscatus. (Cerithidae: Gastropoda) of the Cross River. Nigeria. Int J Fish Aqua Stud. 1: 26-28.
Urra A, Oliva D, Sepulveda M. 2007. Use of a Morphometric Analysis to Differentiate Adelomelon ancilla and Odontocymbiola magellanica (Caenogastropoda: Volutidae) of Southern Chile. Zool Stud. 46(3): 253-261. Veron JEN, Devantier LM, Turak E, Green AL, Kininmonth S, Stafford-Smith M,
Peterson N. 2009. Delineating the coral triangle. Gal J Coral ReefStud. 11: 91-100.
Walsh PS, Metzger DA, Higuchi R. 1991. Chelex-100 as a medium for simple extraction of DNA for PCR based typing from forensic material. Biotechniques. 10: 506–513.
25
Wernberg T, White M, Vanderklift MA. 2008. Population structure of Turbinid gastropods on wave-exposed subtidal reefs: effects of density. body size and algae on grazing behaviour. Mar Ecol Prog Ser. 362: 169–179.
Williams ST. 2007. Origins and diversification of Indo-West Pacific marine fauna: evolutionary history and biogeography of Turban shells (Gastropoda. Turbinidae). Biol J Linn Society. 92: 573–592.
Wolf HD, Verhagen R, Backeljau T. 2000. Large scale population structure and gene flow in the planktonic developing periwinkle, Littorina striata in Macaronesia (Mollusca: Gastropoda). J Exp Mar Biol Ecol. 246: 69–83. Wyrtki K. 1961. Physical oceanography of the southeast Asian waters. Scientific
results of marine investigations of the South China Sea and Gulf of Thailand 1959–1961. NAGA Rep. 2, Scripps Inst. Oceanogr., La Jolla, CA., 195 pp. Xiao M, Gordon PM, Phong A, Ha C, Chan TF, Cai D, Selvin PR, Kwok Y. 2007.
Determination of haplotype for single DNA molecules : A Method for single molecules barcoding. Hum Mut. 0: 1-9.
26
Lampiran 1. Lokasi pengambilan sampel T. sparverius dan T. bruneus
Kampung Arevi Kabupaten Raja Ampat
Nuni Kabupaten Manokwari
Kampung Dusner, Kabupaten teluk Wondama
27
Lampiran 2. Hasil analisis regresi T. sparverius dan T. bruneus 1. Hasil analisis regresi T. sparverius
SUMMARY OUTPUT Regression Statistics Multiple R 0.89367
R Square 0.798646 Adjusted R
Square 0.796691 Standard
Error 0.100266 Observations 105
ANOVA
df SS MS F
Significance F Regression 1 4.107141 4.107141 408.5359 1.25E-37
Residual 103 1.035492 0.010053
Total 104 5.142632
Coefficients
Standard
Error t Stat P-value Lower 95%
Upper 95%
Lower 95.0%
Upper 95.0% Intercept -2.65686 0.208743 -12.7279 7.29E-23 -3.07085 -2.24287 -3.07085 -2.24287 X Variable 1 2.467161 0.122063 20.21227 1.25E-37 2.225079 2.709244 2.225079 2.709244
28
2. Hasil analisis regresi T. bruneus SUMMARY OUTPUT
Regression Statistics Multiple R 0.934023 R Square 0.872399 Adjusted R
Square 0.870627
Standard
Error 0.104035
Observations 74
ANOVA
df SS MS F
Significance F Regression 1 5.327833 5.327833 492.2592 6.48E-34 Residual 72 0.779272 0.010823
Total 73 6.107105
Coefficients
Standard
Error t Stat P-value Lower 95%
Upper 95%
Lower 95.0%
Upper 95.0% Intercept -2.46039 0.161024 -15.2797 2.71E-24 -2.78139 -2.1394 -2.78139 -2.1394 X Variable 1 2.295437 0.103459 22.18691 6.48E-34 2.089195 2.501679 2.089195 2.501679
29
Lampiran 3. Distribusi frekuensi panjang T. sparverius dan T. bruneus yang digunakan dalam analisis molekuler
1. T. sparverius
Range Frekuensi 27.45 - 32.50 2 32.51 - 37.56 1 37.57 - 42.62 3 42.63 - 47.68 4 47.69 - 52.74 8 52.75 - 57.80 13 57.81 - 62.86 7 62.87 - 67.92 0
2. T. bruneus
Range Frekuensi
14.55 - 20.84 0
20.85 - 27.14 2
27.15 - 33.44 9
33.45 - 39.74 8
39.75 - 46.04 11
46.05 - 52.34 3
52.35 - 58.64 2
Lampran 4. Visualisasi Hasil PCR
Keterangan :
No gel 1 : DNA ladder No gel 2 : Negatif No gel 3 : Tipis No gel 4 : Positif
1
4 3 2
30
Lampiran 5. Hasil identifikasi T.sparverius dan T. bruneus dengan program Basic Local Aligment Search Tools (BLAST)
1. Hasil identifikasi T. sparverius program Basic Local Aligment Search Tools (BLAST)
No ID Site BLAST
Spesies Ident (%) 1 Manokwari-01 Manokwari Turbo sparverius 99% 2 Manokwari-02 Manokwari Turbo sparverius 99% 3 Manokwari-03 Manokwari Turbo sparverius 99% 4 Manokwari-04 Manokwari Turbo sparverius 99% 5 Manokwari-05 Manokwari Turbo sparverius 99% 6 Manokwari-06 Manokwari Turbo sparverius 99% 7 Manokwari-07 Manokwari Turbo sparverius 99% 8 Manokwari-08 Manokwari Turbo sparverius 99% 9 Manokwari-09 Manokwari Turbo sparverius 99% 10 Manokwari-10 Manokwari Turbo sparverius 99% 11 Manokwari-11 Manokwari Turbo sparverius 99% 12 Manokwari-12 Manokwari Turbo sparverius 99% 13 Manokwari-13 Manokwari Turbo sparverius 99% 14 Manokwari-14 Manokwari Turbo sparverius 99% 15 Manokwari-15 Manokwari Turbo sparverius 98% 16 Manokwari-16 Manokwari Turbo sparverius 99% 17 Manokwari-17 Manokwari Turbo sparverius 99% 18 Manokwari-18 Manokwari Turbo sparverius 99% 19 Manokwari-19 Manokwari Turbo sparverius 99% 20 Manokwari-20 Manokwari Turbo sparverius 99% 21 Teluk Wondama-01 Teluk Wondama Turbo sparverius 99% 22 Teluk Wondama-02 Teluk Wondama Turbo sparverius 99% 23 Teluk Wondama-03 Teluk Wondama Turbo sparverius 99% 24 Teluk Wondama-04 Teluk Wondama Turbo sparverius 99%
31
25 Teluk Wondama-05 Teluk Wondama Turbo sparverius 99% 26 Teluk Wondama-06 Teluk Wondama Turbo sparverius 99% 27 Teluk Wondama-07 Teluk Wondama Turbo sparverius 99% 28 Teluk Wondama-08 Teluk Wondama Turbo sparverius 99% 29 Teluk Wondama-09 Teluk Wondama Turbo sparverius 99% 30 Teluk Wondama-10 Teluk Wondama Turbo sparverius 99% 31 Teluk Wondama-11 Teluk Wondama Turbo sparverius 99% 32 Teluk Wondama-12 Teluk Wondama Turbo sparverius 99% 33 Teluk Wondama-13 Teluk Wondama Turbo sparverius 99% 34 Teluk Wondama-14 Teluk Wondama Turbo sparverius 99% 35 Teluk Wondama-15 Teluk Wondama Turbo sparverius 99%
36 Sorong-01 Sorong Turbo sparverius 99%
37 Sorong-02 Sorong Turbo sparverius 99%
38 Sorong-01 Sorong Turbo sparverius 99%
2. Hasil identifikasi T. bruneus program Basic Local Aligment Search Tools (BLAST)
No ID Site BLAST
32
11 Raja Ampat-11 Raja Ampat Turbo bruneus 99% 12 Raja Ampat-12 Raja Ampat Turbo bruneus 99% 13 Raja Ampat-13 Raja Ampat Turbo bruneus 99% 14 Raja Ampat-14 Raja Ampat Turbo bruneus 99% 15 Raja Ampat-15 Raja Ampat Turbo bruneus 99% 16 Raja Ampat-16 Raja Ampat Turbo bruneus 99% 17 Raja Ampat-17 Raja Ampat Turbo bruneus 99% 18 Manokwari_01 Manokwari Turbo bruneus 99% 19 Manokwari_02 Manokwari Turbo bruneus 99% 20 Manokwari_03 Manokwari Turbo bruneus 99% 21 Manokwari_04 Manokwari Turbo bruneus 99% 22 Manokwari_05 Manokwari Turbo bruneus 99% 23 Manokwari_06 Manokwari Turbo bruneus 99% 24 Manokwari_07 Manokwari Turbo bruneus 99% 25 Manokwari_08 Manokwari Turbo bruneus 99% 26 Manokwari_09 Manokwari Turbo bruneus 99% 27 Manokwari_10 Manokwari Turbo bruneus 99% 28 Manokwari_11 Manokwari Turbo bruneus 99% 29 Manokwari_12 Manokwari Turbo bruneus 99% 30 Manokwari_13 Manokwari Turbo bruneus 99% 31 Manokwari_14 Manokwari Turbo bruneus 99% 32 Manokwari_15 Manokwari Turbo bruneus 99% 33 Manokwari_16 Manokwari Turbo bruneus 99% 34 Manokwari_17 Manokwari Turbo bruneus 99% 35 Manokwari_18 Manokwari Turbo bruneus 99%
33
Lampiran 6. Subtitusi nukleotida pada spesies T. sparverius dan T.bruneus 1. Subtitusi nukleotida pada spesies T. sparverius