Konfirmasi Pewarisan Alel pada Generasi F
1
Hasil Persilangan Calcutta-4 (Musa acuminata
ssp
.
burmannicoides) dan Musa acuminata ssp. microcarpa
Berdasarkan Marka SSR
(Confirmation of Alleles Inheritance in F
1Progenies Derived from a Cross of
Calcutta-4 [Musa acuminata ssp. burmannicoides] and Musa acuminata
ssp. microcarpa Based on SSR Markers)
Dea Rosalia1*, Puji Lestari2,3, Andy Soegianto1, Darmawan Saptadi1, Agus Sutanto4, Kristianto Nugroho2,
Rerenstradika T. Terryana2, I Made Tasma2, dan Ika Roostika2
1Jurusan Budidaya Pertanian, Fakultas Pertanian, Universitas Brawijaya, Jl. Veteran, Ketawanggede, Lowokwaru, Malang 65145 Indonesia
Telp. (0341) 551665; Faks. (0341) 560011; *Email: dearosa99@gmail.com
2Balai Besar Penelitian dan Pengembangan Bioteknologi dan Sumber Daya Genetik Pertanian, Jl. Tentara Pelajar 3A, Bogor 16111 Indonesia 3Balai Besar Penelitian Tanaman Padi, Kebun Percobaan Muara, Jl. Ciapus No. 25A, Pasir Jaya, Bogor 16119 Indonesia
4
Balai Penelitian Tanaman Buah Tropika, Jl. Raya Solok Aripan Km. 8, Solok 27365 Indonesia Diajukan: 6 Agustus 2019; Direvisi: 14 April 2020; Diterima: 25 April 2020
ABSTRACT
Banana breeding to produce improved varieties with disease resistance characters and other desired traits could sustain its yield. Alleles harbored by parents could be passed on to the offsprings through hybridization, but need to be confirmed using molecular markers. This study aimed to confirm allele inheritance in F1 progenies derived from a cross of Calcutta-4 (Musa
acuminata ssp. burmannicoides) and M. acuminata ssp. microcarpa based on SSR markers. Eleven pairs of SSR primers were used to amplify DNA of 44 progenies using the PCR technique. The results showed that six SSR markers (MaSSR 1.1, MaSSR 5.1, MaSSR 6.1, MaSSR 7.1, MaSSR 8.1, and MaSSR 11.1) were polymorphic in both parents. Four markers (MaSSR 1.1, MaSSR 5.1, MaSSR 6.1, and MaSSR 8.1) had PIC >0.7, indicating their informativeness to distinguish these progenies and other genetic studies of banana germplasms. A total of 44 F1 individuals were confirmed to harbor alleles inherited from their parents,
suggesting as true progenies from the cross of Calcutta-4 and M. acuminata ssp. microcarpa. This population demonstrated 100% success of hybridization performed. Chi-Square analysis revealed that segregation of all markers did not match to Mendelian ratio 1:2:1, except for MaSSR 1.1 (x2
= 5,62) and MaSSR 6.1 (x2
= 3,77) markers. The genetic traceability of banana F1 progenies demonstrating the usefulness and feasibility of SSR markers in this study provided information on selection of true
progenies which may be valuable for breeders to assist selection process in future banana breeding program in Indonesia. Keywords: Banana, cross, allele, F1 population.
ABSTRAK
Pemuliaan pisang bertujuan menghasilkan varietas unggul dengan karakter ketahanan terhadap penyakit dan karakter lain yang dapat mempertahankan hasil. Alel tetua dapat diturunkan melalui persilangan, namun perlu dilakukan konfirmasi dengan marka molekuler. Tujuan penelitian ini ialah mengonfirmasi pewarisan alel pada generasi F1 hasil persilangan tetua
Calcutta-4 (Musa acuminata ssp. burmannicoides) dan M. acuminata ssp. microcarpa berdasarkan marka SSR. Sebelas pasang primer SSR digunakan untuk mengamplifikasi DNA kedua tetua dan 44 progeni menggunakan teknik PCR. Hasil penelitian menunjukkan bahwa enam marka (MaSSR 1.1, MaSSR 5.1, MaSSR 6.1, MaSSR 7.1, MaSSR 8.1, dan MaSSR 11.1) menunjukkan polimorfisme pada kedua tetua. Sebanyak empat marka, yaitu MaSSR 1.1, MaSSR 5.1, MaSSR 6.1, dan MaSSR 8.1, memiliki PIC >0,7, yang mengindikasikan keinformativannya dalam membedakan progeni hasil persilangan dan dapat diaplikasikan dalam studi genetik plasma nutfah pisang. Seluruh individu FI terkonfirmasi sebagai hasil persilangan antara
Calcutta-4 dan M. acuminata ssp. microcarpa sehingga keberhasilan persilangan yang telah dilakukan mencapai 100%. Analisis Chi-Square menunjukkan bahwa segregasi hampir semua marka tidak sesuai dengan rasio Mendel 1:2:1, kecuali marka MaSSR 1.1 (x2 = 5,62) dan MaSSR 6.1 (x2 = 3,77). Keterlacakan genetik pada progeni F
1 pisang telah menunjukkan
manfaat dan kelayakan penggunaan marka SSR dalam seleksi progeni F1 murni. Informasi ini akan sangat berharga bagi
pemulia pada proses seleksi dalam program pemuliaan pisang di Indonesia. Kata kunci:Pisang, persilangan, alel, populasi F1.
Hak Cipta © 2020, BB Biogen
PENDAHULUAN
Pisang (Musa spp.) termasuk tanaman
hortikul-tura asli Indonesia yang sebaran populasinya cukup luas. Buah pisang memiliki kandungan gizi yang tinggi, kaya akan mineral, vitamin, dan kalium yang bermanfaat bagi tubuh. Pisang menjadi kontributor utama dalam produksi buah unggulan nasional dengan persentase mencapai 31%, mengungguli jeruk (16%), mangga (10%), durian (5%), dan buah-buahan lainnya (38%) (Balitbu 2009).
Komoditas pisang memiliki prospek untuk di-kembangkan dalam mendukung sektor pertanian Indonesia. Permasalahan dalam upaya peningkatan produktivitas pisang berupa serangan penyakit. Penyakit layu Fusarium yang disebabkan oleh
cenda-wan Fusarium oxysporum f. sp. cubense E.F. Smith
(Foc) dianggap sebagai penyakit paling merugikan
dan mengancam industri pisang dunia. Penyakit layu Fusarium telah menyebar ke berbagai provinsi dan menyerang banyak varietas pisang lokal serta men-jadi penyebab utama turunnya produksi pisang di
Indonesia (Molina etal. 2010; Hermanto et al. 2011).
Pengembangan varietas unggul dengan karakter ketahanan terhadap penyakit dapat diupayakan, salah satunya melalui program pemuliaan tanaman. Pengembangan varietas pisang tahan layu Fusarium memerlukan populasi hasil persilangan tetua tahan
dan rentan. Pisang Calcutta-4 (M. acuminata ssp.
burmannicoides) banyak digunakan dalam
pemulia-an karena memiliki ketahpemulia-anpemulia-an terhadap penyakit layu Fusarium (Zuo et al. 2018). Persilangan antara pisang
Calcutta-4 (betina) yang bersifat tahan dan M.
acuminata ssp. microcarpa (jantan) yang bersifat
rentan namun memiliki viabilitas dan fertilitas polen
yang tinggi diharapkan akan menghasilkan progeni F1
dengan sifat gabungan kedua tetua, yang selanjutnya dapat digunakan sebagai tetua jantan untuk per-silangan berikutnya. Terdapat berbagai faktor yang dapat memengaruhi keberhasilan persilangan
sehing-ga progeni persilansehing-gan belum tentu merupakan F1.
Karakterisasi secara morfologi kurang efektif karena dipengaruhi oleh lingkungan dan waktu yang dibutuhkan terlalu lama sehingga diperlukan pende-katan melalui marka molekuler. Marka molekuler merupakan teknik yang efektif untuk karakterisasi karena didasarkan pada fragmen pada genom yang tidak dipengaruhi oleh variasi lingkungan, presisi, dan lebih cepat menghasilkan informasi (De Langhe et al. 2005; Christelová et al. 2011). Karakterisasi molekuler tidak hanya bermanfaat untuk analisis keragaman genetik, pemetaan, ataupun studi genetik lainnya, tetapi dapat digunakan untuk mengonfirmasi progeni
hasil persilangan (Lestari et al. 2017). Progeni hasil persilangan dapat diidentifikasi berdasarkan alel yang diturunkan oleh salah satu atau kedua tetua meng-gunakan marka molekuler yang bersifat kodominan.
Simple Sequence Repeat (SSR) atau dikenal
sebagai marka mikrosatelit merupakan satu marka molekuler yang sering digunakan dalam berbagai studi genetik pada tanaman termasuk pisang. Pemilihan SSR didasarkan pada sifatnya yang kodominan, memiliki polimorfisme tinggi, dan mudah dalam aplikasinya (Changadeya et al. 2012). Marka SSR telah banyak diaplikasikan untuk mengakses keragaman genetik pada famili Musaceae (Li et al.
2010), keragaman genotipe Musa bergenom B
(Oriero et al. 2006; Wahyuningtyas et al. 2009),
analisis keragaman genetik pisang Thailand
(Rotchanapreeda et al. 2016), dan genotyping pisang
(Christelová et al. 2011). Saat ini, populasi F1 hasil
persilangan antara Calcutta-4 dan M. acuminata ssp.
microcarpa telah terbentuk dan diperlukan upaya
konfirmasi alel yang diwariskan pada progeni hasil persilangan kedua tetua tersebut. Analisis molekuler untuk menguji progeni hasil persilangan pisang belum pernah dilakukan sebelumnya di Indonesia se-hingga penelitian ini dilakukan untuk mengonfirmasi
pewarisan alel pada generasi F1 hasil persilangan
tetua Calcutta-4 dan M. acuminata ssp. microcarpa
berdasarkan marka SSR.
BAHAN DAN METODE Materi Genetik
Materi genetik yang digunakan pada penelitian ini terdiri atas 44 individu progeni hasil persilangan
antara Calcutta-4 dan M. acuminata ssp. microcarpa.
Sebanyak 19 individu merupakan hasil perbanyakan
secara in vitro di Balai Penelitian Tanaman Buah
(Balitbu), Solok, Sumatra Barat dan 25 individu
lain-nya merupakan hasil embryo rescue di Balai Besar
Penelitian dan Pengembangan Bioteknologi dan Sumber Daya Genetik Pertanian (BB Biogen), Bogor, Jawa Barat yang merupakan hasil penelitian Konsorsium Genom lingkup Badan Penelitian dan
Pengembangan Pertanian (2010–2015). Materi
genetik kedua tetua persilangan ini diperoleh dari Balitbu.
Isolasi DNA
Isolasi DNA dilakukan dengan menggunakan
metode Cetyltrimethylammonium bromide/CTAB
(Doyle dan Doyle 1990) yang dimodifikasi, yaitu
dengan penambahan Polyvinylpyrrolidone (PVP) 2%
(w/v), dan asam askorbat 40 mM pada bufer ekstraksi. Bahan-bahan pembuatan bufer ekstraksi, yaitu CTAB 2% (w/v), Tris-HCl 100 mM (pH 8,0), NaCl 1,4 M, dan EDTA 20 mM (pH 8,0). Sebanyak 0,5 gram potongan daun pisang digerus menggunakan bufer ekstraksi, lalu sampel diekstraksi dua kali mengguna-kan pelarut kloroform:isoamil alkohol (24:1). Pelet DNA yang telah dikeringanginkan kemudian dilarut-kan dengan bufer TE (Tris 10 mM [pH 8,0] dan EDTA 1 mM). Larutan DNA stok selanjutnya disimpan pada suhu -20°C hingga digunakan. Larutan DNA stok yang diperoleh diuji secara kualitatif dan kuantitatif untuk mengetahui kualitas dan konsentrasinya. Uji
kuantita-tif dilakukan dengan spektrofotometer NanoDrop™
2000 (Thermo Scientific, AS), sedangkan uji kualitatif
dilakukan dengan teknik elektroforesis pada gel agarosa 1%. Hasil elektroforesis kemudian diamati di
bawah sinar UV pada UV TransIlluminator (UVP, UK).
Amplifikasi PCR
Urutan DNA sebelas pasang primer SSR yang digunakan pada penelitian ini (Tabel 1) dapat diakses pada laman Pusat Genom Pertanian Indonesia (2015). Setiap sampel diamplifikasi dalam total reaksi
10 μl yang mengandung 10 ng DNA template
sebanyak 2 μl, 2 MyTaq™ Red Mix (Bioline, UK)
sebanyak 5 μl, primer forward dan reverse dengan
konsentrasi 10 μM masing-masing sebanyak 0,5 μl,
dan ddH2O steril sebanyak 2 μl. Reaksi PCR dilakukan
dengan mesin PCR T1 Thermocycler (Biometra,
Jerman) dengan program PCR sebagai berikut: denaturasi awal pada 95°C selama 5 menit, dilanjut-kan dengan 35 siklus denaturasi pada 94°C selama 30
detik, penempelan primer (annealing) pada 52°C
se-lama 1 menit, pemanjangan basa pada 72°C sese-lama 1
menit, dan proses akhir perpanjangan basa (final
extension) pada 60°C selama 15 menit. Hasil PCR
selanjutnya dielektroforesis pada gel poliakrilamida 6% untuk analisis lebih lanjut. Hasil elektroforesis diwarnai dengan etidium bromida dan divisualisasi
dengan ChemiDoc EQ™ System (Bio-Rad, AS).
Analisis Data
Analisis data dilakukan berdasarkan skoring pita DNA polimorfik pada tetua persilangan. Skoring
di-lakukan dengan membandingkan pola pita progeni F1
dengan kedua tetuanya. Hasil skoring disajikan dalam persentase untuk mengetahui konfirmasi hasil per-silangan. Analisis tingkat polimorfisme dilakukan
dengan perangkat lunak PowerMarker 3.25 (Liu dan
Muse 2005) untuk mengetahui nilai Polymorphic
Information Content (PIC), heterozigositas, dan
diver-sitas gen. Analisis sebaran populasi progeni F1 hasil
persilangan berdasarkan jarak genetik ditampilkan
dalam bentuk Principal Coordinate Analysis (PCoA)
menggunakan perangkat lunak XLSTAT. Analisis
Chi-Square alel tiap marka SSR dilakukan untuk
menge-tahui segregasi marka berdasarkan rasio Mendel 1:2:1.
Tabel 1. Daftar primer marka SSR pisang yang digunakan pada penelitian ini*.
Nama primer Sekuen Tm (°C)
MaSSR 1.1 F: TCATCTGTAGACAGGAGATGC 53,6 R: TCTACTGTGATTTCGATGGAT 50,9 MaSSR 2.1 F: AGACAGTCCAATTGTCACATC 52,6 R: AGGATCCATGTCTTAGATTCG 51,5 MaSSR 3.1 F: CCCACTTCAGCAATTAAGAT 50,2 R: CGGAGATCGATTAGGGTATAA 50,7 MaSSR 4.1 F: TTAAGAAGCAGGTATGCAATC 50,5 R: AAGCTTCTACCTTGTGTGCTA 53,7 MaSSR 5.1 F: AAGATGAAACCTCGAAAAGTT 50,1 R: GGTCTCTCTGTCGAACTCAAT 53,9 MaSSR 6.1 F: TCCTCCATGAAGATTTGTAGA 50,6 R: GACTTCTCTGCTGGTCTGATA 53,5 MaSSR 7.1 F: GAAACAAGCTCCTCAAAGAGT 52,8 R: ATTGCAATGTAGCACTGTAGC 53,4 MaSSR 8.1 F: CTCAAGAAGTGTGGGAGGT 54,1 R: GGTCACTATCGTGTGTGTGT 54,5 MaSSR 9.1 F: ATGATCAGCAGAAAAACAAGA 50,2 R: GACTCGGTTATTATCGAACCT 51,6 MaSSR 10.1 F: GAAGTCGAACGTGCAGTC 53,6 R: CAACCTCTGCTCTAAATGCTA 52,1 MaSSR 11.1 F: AATCGTTACTCCCACGTAAAT 51,6 R: AAGCGAGCTTCCAAGTAACTA 53,7
HASIL DAN PEMBAHASAN Analisis Polimorfisme Marka SSR
Berdasarkan tingkat polimorfisme terhadap
ke-dua tetua persilangan (Calcutta-4 dan M. acuminata
ssp. microcarpa), terseleksi sebanyak 6 marka SSR
yang polimorfik dari total 11 marka yang digunakan (Tabel 2). Enam marka tersebut (MaSSR 1.1, MaSSR 5.1, MaSSR 6.1, MaSSR 7.1, MaSSR 8.1, dan MaSSR 11.1) digunakan pada analisis lanjut dalam mengon-firmasi progeni hasil persilangan pisang. Keenam marka SSR tersebut mampu mendeteksi adanya alel
heterozigot pada tetua dan individu F1 dengan kisaran
nilai heterozigositas antara 0,59 dan 0,90 dengan rata-rata 0,79. Tingginya nilai heterozigositas pada
pene-litian ini mengindikasikan bahwa progeni F1 yang
di-peroleh memiliki tingkat keragaman genetik yang tinggi dan berpotensi membawa gen-gen unggul yang berasal dari kedua tetua (Santoso et al. 2014). Sayangnya, di sisi lain tingginya tingkat heterozigositas justru menjadi salah satu faktor yang menjadi kendala dalam kegiatan persilangan tanaman pisang selain kompleksnya sistem genetik, rendahnya fertilitas polen, adanya inkompatibilitas, dan tingkat ploidi yang berlainan (Handayani et al. 2017). Teknik
embryo rescue melalui kultur in vitro dapat menjadi
alternatif perbanyakan pisang dalam mengatasi per-masalahan tersebut seperti yang diaplikasikan pada penelitian ini.
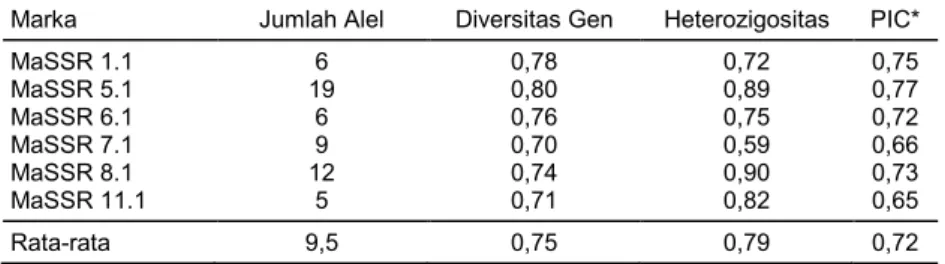
Tingkat polimorfisme yang tinggi keenam marka direpresentasikan dengan nilai PIC dan diversitas gen. Nilai PIC keenam marka berkisar antara 0,65 (MaSSR 11.1) dan 0,75 (MaSSR 1.1) dengan rata-rata 0,72 (Tabel 2). Sementara itu, pada penelitian Ravishankar et al. (2013) diperoleh nilai PIC berkisar antara 0,164 dan 0,912 dengan rata-rata 0,771, sedangkan pada penelitian Rotchanapreeda et al. (2016) berkisar antara 0,4111 dan 0,9392 dengan rata-rata 0,836. Perbedaan nilai PIC ini dipengaruhi oleh jumlah dan tipe marka molekuler, serta seberapa besar variasi materi genetik termasuk inter dan intraspesies yang diobservasi. Hildebrand et al. (1992) menyatakan bahwa marka dengan nilai PIC >0,7 merupakan marka yang informatif. Berdasarkan kriteria tersebut, sebanyak empat marka (MaSSR1.1, MaSSR5.1, MaSSR6.1, dan MaSSR8.1) merupakan marka infor-matif yang bermanfaat untuk analisis keragaman genetik dan studi genetik lainnya pada plasma nutfah pisang termasuk identifikasi progeni hasil persilangan.
Tabel 2. Ringkasan statistik polimorfisme yang diobservasi pada total progeni pisang berdasarkan multialel yang dihasilkan dari enam marka SSR.
Marka Jumlah Alel Diversitas Gen Heterozigositas PIC*
MaSSR 1.1 6 0,78 0,72 0,75 MaSSR 5.1 19 0,80 0,89 0,77 MaSSR 6.1 6 0,76 0,75 0,72 MaSSR 7.1 9 0,70 0,59 0,66 MaSSR 8.1 12 0,74 0,90 0,73 MaSSR 11.1 5 0,71 0,82 0,65 Rata-rata 9,5 0,75 0,79 0,72
*PolymorphismInformation Content.
Gambar 1. Elektroforegram kedua tetua dan total progeni F1 pada gel poliakrilamida 6% menggunakan marka MaSSR 5.1. M = marker 100 bp,
A = Musa acuminata ssp. microcarpa, B = Calcutta-4,1–44 = progeni.
200 bp 300 bp
M A B 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44
Pola pita yang dihasilkan dari setiap marka digu-nakan sebagai dasar analisis konfirmasi pewarisan alel pada progeni persilangan. Contoh pola pita poli-morfik ditunjukkan oleh primer MaSSR 5.1 (Gambar 1). Keberhasilan penggunaan marka SSR dalam
menganalisis keragaman genetik dan genotyping
pisang telah banyak dilakukan. Hal ini tidak terlepas dari sifat SSR yang polimorfik, multialelik, kodominan, reproduksibilitas tinggi, mudah untuk diinterpretasi-kan, dan diamplifikasi dengan PCR (Changadeya et al. 2012; Doloiras-Larano et al. 2018) sehingga pemilihan marka SSR pada penelitian ini sudah tepat.
Berdasarkan pola pita polimorfik yang dihasilkan pada kedua tetua terlihat bahwa enam marka SSR polimorfik mampu menganalisis keragaman genetik pisang sebagaimana hasil penelitian sebelumnya (Li et al. 2010; Changadeya et al. 2012; Rotchanapreeda et al. 2016; Nyine et al. 2017). Sementara itu, peng-gunaan marka SSR untuk seleksi populasi persilangan telah banyak diaplikasikan pada beberapa tanaman, seperti kedelai (Santana et al. 2014; Lestari et al. 2017), jagung (Elci dan Hancer 2015), singkong (Vincent et al. 2014), kapas (Dongre et al. 2011), dan
Eucalyptus (Subashini et al. 2013). Konfirmasi
pewa-risan alel pada pisang dengan menggunakan marka SSR belum pernah dilakukan sebelumnya, akan tetapi keberhasilan yang ditunjukkan marka SSR pada tanaman lain dapat digunakan sebagai acuan untuk konfirmasi progeni hasil persilangan dalam studi genetik plasma nutfah pisang.
Konfirmasi Pewarisan Alel dari Tetua
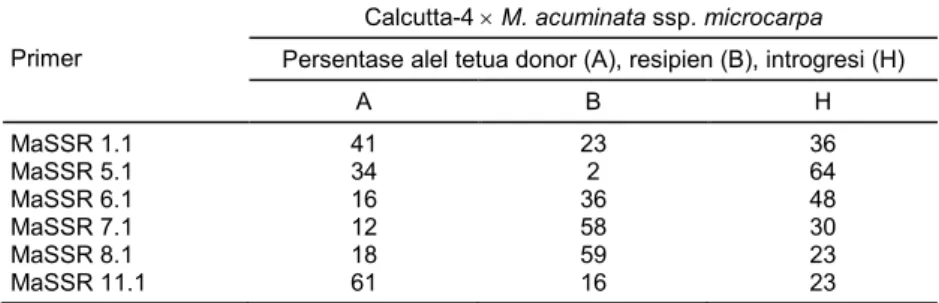
Keberhasilan persilangan diketahui dengan adanya introgresi alel yang ditunjukkan oleh pita DNA berasal dari kedua tetua sehingga terkonfirmasi sebagai hasil persilangan. Enam marka SSR tersebut berhasil mengonfirmasi keberhasilan persilangan kedua tetua dalam bentuk persentase. Persentase introgresi yang beragam dari setiap primer erat kait-annya dengan nilai heterozigositas yang dihasilkan dari skoring pita DNA target (Tabel 3) dan nilai PIC dari skoring berdasarkan multialel (Tabel 2). Marka-marka yang digunakan menunjukkan kemampuan yang berbeda dalam mengonfirmasi pewarisan alel. Persentase nilai introgresi yang dihasilkan dari ke-enam marka berkisar 23–64%. Persentase introgresi tertinggi pada marka MaSSR 5.1 yang berhasil mengonfirmasi bahwa 64% dari total progeni merupa-kan hasil persilangan, persentase alel tetua pemulih sebesar 2%, dan persentase alel tetua donor sebesar 34%. Marka MaSSR 8.1 dan MaSSR 11.1 menunjukkan nilai introgresi terendah dengan persentase 23%.
Persentase pewarisan alel dari tetua M. acuminata
ssp. microcarpa sebagai tetua donor dan Calcutta-4
sebagai tetua pemulih serta introgresi pada generasi
F1 menggunakan keenam marka disajikan pada Tabel
4. Nomor-nomor individu hasil persilangan yang ber-hasil dikonfirmasi oleh setiap marka secara detil di-sajikan pada Tabel 5.
Hasil penelusuran alel pada tiap marka
menun-jukkan bahwa semua nomor individu F1 merupakan
hasil introgresi kedua tetua persilangan. Sebanyak 44
Tabel 3. Ringkasan statistik polimorfisme yang diobservasi pada total progeni F1
berdasarkan pita DNA target yang dihasilkan dari enam marka SSR. Marka Jumlah alel Diversitas gen Heterozigositas PIC*
MaSSR 1.1 2 0,483 0,358 0,366 MaSSR 5.1 2 0,449 0,636 0,348 MaSSR 6.1 2 0,479 0,477 0,364 MaSSR 7.1 2 0,391 0,302 0,315 MaSSR 8.1 2 0,416 0,227 0,329 MaSSR 11.1 2 0,396 0,227 0,318 Rata-rata 2 0,436 0,371 0,340
*PolymorphismInformation Content.
Tabel 4. Persentase pewarisan alel pada generasi F1.
Primer
Calcutta-4 M. acuminata ssp. microcarpa
Persentase alel tetua donor (A), resipien (B), introgresi (H)
A B H MaSSR 1.1 41 23 36 MaSSR 5.1 34 2 64 MaSSR 6.1 16 36 48 MaSSR 7.1 12 58 30 MaSSR 8.1 18 59 23 MaSSR 11.1 61 16 23
nomor individu F1 terkonfirmasi membawa alel dari
kedua tetua dengan pewarisan alel sebesar 100% merupakan hasil persilangan. Individu-individu yang telah terkonfirmasi sebagai hasil persilangan ke-mudian dikelompokkan untuk melihat kedekatannya dengan tetua persilangan.
Sebaran populasi progeni F1 dan kedua tetua
disajikan pada diagram PCoA (Gambar 2). Populasi
progeni F1 cenderung menyebar pada semua
kuadran. Terdapat individu-individu yang berada pada posisi berdekatan dengan tetuanya. Individu
nomor 10 paling dekat dengan tetua M. acuminata
ssp. microcarpa, sedangkan individu nomor 29 paling
dekat dengan tetua Calcutta-4. Sebaran progeni F1
pada semua kuadran menunjukkan variasi sebagai akibat dari penggabungan alel kedua tetua secara acak yang menandakan keberhasilan persilangan seperti hasil penelitian Pancoro et al. (2016). Penggabungan alel secara acak diharapkan mem-bawa karakter unggul kedua tetua ke dalam satu individu.
Analisis Chi-Square
Analisis Chi-Square dilakukan untuk mengetahui
kemampuan marka SSR dalam mendiskriminasi
progeni populasi F1 berdasarkan kesesuaiannya
dengan rasio Mendel. Berdasarkan analisis enam marka yang polimorfik pada kedua tetua persilangan, hampir semua marka tidak mampu mengonfirmasi
bahwa progeni F1 sesuai dengan rasio Mendel 1:2:1,
kecuali MaSSR 1.1 (x2 = 5,62; P <0,10) dan MaSSR 6.1
(x2 = 3,77; P <0,5) (Tabel 6). Segregasi marka
molekuler dikatakan tidak sesuai dengan rasio
Mendel jika nilai x2 hitung lebih besar daripada nilai x2
tabel. Keempat marka yang tidak sesuai dengan rasio
segregasi Mendel memiliki nilai x2 hitung berkisar
antara 12,18 (MaSSR 5.1) dan 31,27 (MaSSR 11.1). Rasio segregasi Mendel tidak selalu terpenuhi pada suatu populasi, penyimpangan tersebut lebih dikenal
sebagai Segregation Distortion (SD). Terdapat banyak
faktor yang memengaruhi terjadinya SD, seperti genetik, fisiologis, dan lingkungan. Faktor genetik me-megang peran penting. Hal ini erat kaitannya dengan
female function (pengaruh embryo-sac), male
function (pengaruh polen), dan zygotic selection yang
dapat memengaruhi terjadinya SD pada suatu populasi (Reflinur et al. 2014). Terkait rasio Mendel ini, MaSSR 1.1 berpotensi sebagai marka seleksi progeni secara genotipe hingga generasi lanjut.
Hasil penelitian ini menunjukkan bahwa analisis SSR dapat secara efektif menyeleksi individu-individu
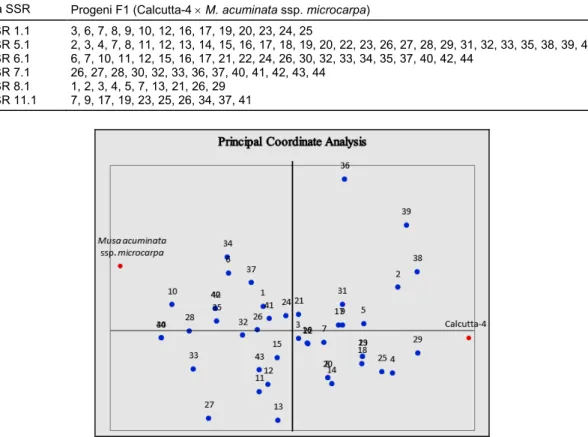
Tabel 5. Nomor-nomor individu progeni F1 hasil persilangan yang berhasil dikonfirmasi dengan 6 marka SSR.
Marka SSR Progeni F1 (Calcutta-4 M. acuminata ssp. microcarpa) MaSSR 1.1 3, 6, 7, 8, 9, 10, 12, 16, 17, 19, 20, 23, 24, 25 MaSSR 5.1 2, 3, 4, 7, 8, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 22, 23, 26, 27, 28, 29, 31, 32, 33, 35, 38, 39, 43 MaSSR 6.1 6, 7, 10, 11, 12, 15, 16, 17, 21, 22, 24, 26, 30, 32, 33, 34, 35, 37, 40, 42, 44 MaSSR 7.1 26, 27, 28, 30, 32, 33, 36, 37, 40, 41, 42, 43, 44 MaSSR 8.1 1, 2, 3, 4, 5, 7, 13, 21, 26, 29 MaSSR 11.1 7, 9, 17, 19, 23, 25, 26, 34, 37, 41
hasil persilangan. Namun demikian, karakterisasi
morfologi dan keragaan fenotipe progeni F1 yang
telah terkonfirmasi secara molekuler perlu dilakukan untuk memastikan informasi fenotipe pada generasi awal yang sangat berguna bagi kegiatan pemuliaan selanjutnya. Akuisisi polimorfisme dan marka SSR yang mampu membedakan progeni hasil persilangan berdasarkan penurunan alel dari tetua persilangan sangatlah penting dan perlu diaplikasikan untuk pembentukan populasi dalam program pemuliaan (Demore et al. 2009). Dengan demikian, penambahan jumlah marka molekuler agar tingkat diskriminatif lebih tinggi perlu dilakukan pada studi lanjutan agar proses seleksi progeni hasil persilangan dapat lebih efektif dan mempercepat proses pemuliaan pisang.
KESIMPULAN
Sebanyak 6 marka dari 11 marka SSR
menun-jukkan polimorfisme pada Calcutta-4 dan M.
acuminata ssp. microcarpa. Marka (MaSSR 1.1,
MaSSR 5.1, MaSSR 6.1, dan MaSSR 8.1) dapat
digunakan untuk mengonfirmasi F1 hasil persilangan
kedua tetua dan mampu membedakan antargenotipe
F1. Marka-marka tersebut informatif dan potensial
untuk diaplikasikan pada berbagai studi genetik serta dapat digunakan untuk seleksi berbasis marka
sete-lah divalidasi. Semua individu populasi F1 (44 nomor)
yang diuji terkonfirmasi sebagai hasil persilangan
antara tetua Calcutta-4 dan M. acuminata ssp.
microcarpa.
UCAPAN TERIMA KASIH
Penelitian ini didanai dari APBN TA 2010–2016 BB Biogen, Badan Penelitian dan Pengembangan Pertanian. Penulis menyampaikan terima kasih kepada Balitbu, Solok yang telah menyediakan materi genetik untuk kegiatan penelitian ini. Penulis juga menyampaikan terima kasih kepada Meddy Saputra yang telah membantu analisis di laboratorium.
KONTRIBUTOR PENULISAN
DR: kontributor utama, melakukan percobaan, analisis data, menulis manuskrip. PL: kontributor utama, desain dan supervisi penelitian, interpretasi data, konsep dan memfinalkan manuskrip. ANS: kontributor utama, supervisi interpretasi data dan pe-nulisan manuskrip. DS: kontributor utama, supervisi interpretasi data dan penulisan manuskrip. AGS: kontributor anggota, membuat populasi persilangan sebagai materi genetik penelitian. KN: kontributor anggota, membantu kegiatan percobaan dan revisi manuskrip. RTT: kontributor anggota, membantu analisis data. IMT: kontributor anggota, ide desain pe-nelitian dan pengembangan populasi, revisi ma-nuskrip. IR: kontributor anggota, penyediaan sebagian
materi genetik penelitian melalui embryo rescue.
DAFTAR PUSTAKA
Balai Penelitian Tanaman Buah Tropika (2009) Teknologi pengendalian penyakit layu pisang dan penerapannya di lapangan. Pertemuan Rehabilitasi Kebun Pisang dan POKJA Penanggulangan Penyakit Layu Pisang. Tanjungkarang, 28 Juli.
Changadeya, W., Kaunda, E. & Ambali, A.J.D. (2012) Molecular characterisation of Musa L. cultivars cultivated in Malawi using microsatellite markers.
African Journal of Biotechnology, 11 (18), 4140–4157. Christelová, P., Valárik, M., Hřibová, E., Van den Houwe, I.,
Channelière, S., Roux, N. & Dolezel, J. (2011) A platform for efficient genotyping in Musa using microsatellite markers. AoB Plants, 11 (1), 1–14. De Langhe, E., Pillay, M., Tenkouano, A. & Swennen, R.
(2005) Integrating morphological and molecular taxonomy in Musa: The African plantains (Musa spp. AAB group). Plant Systematics and Evolution, 255, 225–236.
Demore, P.D.S., Uneda-Trevisoli, S.H., Di Mauro, A.O., Morceli, T.G.S., Muniz, F.R.S., Costa, M.M., Sarti, D.G.P. & Mancini, M.C. (2009) Validation of microsatellite markers for assisted selection of soybean genotypes resistant to powdery mildew. Crop Breeding and Applied Biotechnology, 9, 45–51.
Doloiras-Larano, A., Garcia, R.N., Sandoval, C.M.C., Lalusin, A.G., Gueco, L.S., Huelgas, V.C. & Tecson-Mendoza, E.M. (2018) DNA fingerprinting and genetic
Tabel 6. Hasil analisis Chi-Square pada enam marka SSR yang digunakan.
Marka x2 hitung x2 tabel Nilai P
MaSSR 1.1 5,62 5,99 0,060 MaSSR 5.1 12,18 5,99 0,002 MaSSR 6.1 3,77 5,99 0,152 MaSSR 7.1 25,33 5,99 0,000 MaSSR 8.1 27,82 5,99 0,000 MaSSR 11.1 31,27 5,99 0,000
diversity analysis using of Philippine saba and other cultivars of Musa balbisiana Colla using simple sequence repeat markers. Philippine Journal of Crop Science, 43 (2), 1–11.
Dongre, A.B., Raut, M.P., Bhandarkar, M.R. &. Meshram, K.J. (2011) Identification and genetic purity testing of cotton F1 hybrid using molecular markers. Indian Journal of Biotechnology, 10, 301–306.
Doyle, J.J. & Doyle, J.L. (1990) Isolation of plant DNA from fresh tissue. Coccus, 12, 13–15.
Elci, E. & Hancer, T. (2015) Genetic analysis of maize (Zea mays L.) hybrids using microsatellite markers. Tarim Bilimeri Dergisi-Journal of Agricultural Sciences, 21, 192–198.
Handayani, T., Martanti, D., Poerba, Y.S. & Witjaksono (2017) Deteksi awal ketahanan beberapa aksesi pisang lokal dan hasil persilangan terhadap penyakit layu Fusarium (Foc VCG 01213/16 TR4). Jurnal Hortikultura Indonesia, 8 (2), 88–96.
Hermanto, C., Sutanto, A., Jumjunidang, Edison, H.S., Daniells, J.W., O'Neil, W., Sinohin, V.G., Molina, A.B. & Taylor, P. (2011) Incidence and distribution of Fusarium wilt disease of banana in Indonesia. Acta Horticulturae, 897, 313–322.
Hildebrand, C.E., Torney, D. & Wagner, R.P. (1992) Informativeness of polymorphic DNA markers. Los Alamos Science, 17 (3), 233–238.
Lestari, P., Sustiprijatno & Asadi (2017) Konfirmasi pe-nurunan alel tetua persilangan kedelai pada generasi F1 berdasarkan marka SSR. Prosiding Seminar
Nasional III Tahun 2017. Biologi, Pembelajaran, dan Lingkungan Hidup Perspektif Interdisipliner. 29 April 2017: Prodi Pendidikan Biologi-FKIP bekerja sama dengan Pusat Studi Lingkungan dan Kependudukan (PSLK), Malang.
Li, L.F., Häkkinen, M., Yuan, Y.M., Hao, G. & Ge, X.J. (2010) Molecular phylogeny and systematics of the banana family (Musaceae) inferred from multiple nuclear and chloroplast DNA fragments, with a special reference to the genus Musa. Molecular Phylogenetics and Evolution, 57 (1), 1–10.
Liu, K. & Muse, S.V. (2005) PowerMarker: An integrated analysis environment for genetic marker analysis.
Bioinformatics, 21 (9), 2128–2129.
Molina, A.B., Williams, R.C., Hermanto, C., Suwanda, Komolong, B. & Kokoa, P. (2010) Mitigating the threat of banana Fusarium wilt: Understanding of agroecological distribution of pathogenic forms and developing disease management strategies. ACIAR Publication.
Nyine, M., Uwimana, B., Swennen, R., Batte, M., Brown, A., Christelova, P., Hribova, E., Lorenzen, J. & Dolozel, J. (2017) Trait variation and genetic diversity in a banana genomic selection training population. PLoS ONE, 12 (6), 1–23.
Oriero, C. E., Odunola, O.A., Lokko, Y. & Ingelbrecht I. (2006) Analysis of B-genome derived simple sequence
repeat (SSR) markers in Musa spp. Journal of Biotechnology, 5 (2), 126–128.
Pancoro, A., Septiyani, T.A., Indriyani, N.L.P. & Santoso, P.J. (2016) Analisis progeni F1 hasil persilangan intra
dan interspesies durian (Durio sp.) menggunakan marka mikrosatelit. Jurnal Hortikultura, 26 (2), 171–180. Pusat Genom Pertanian Indonesia (2015) Pusat Genom Pertanian Indonesia (PGPI). [Online] Tersedia pada: http://genom.litbang.pertanian.go.id/hortikultura/index/ php/primer/index/4/ssr) [Diakses 27 November 2018]. Ravishankar, K.V., Raghavendra, K.P., Athani, V., Rekha,
A., Sudeepa, K., Bhavya, D., Srinivas, V. & Ananad, L. (2013) Development and characterisation of microsatellite markers for wild banana (Musa balbisiana). Journal of Horticultural Science and Biotechnology,88 (5), 605–609.
Reflinur, Kim, B., Jang, S.M., Chu, S.H., Bordiya, Y., Akter, Md.B., Lee, J., Chin, J.H. & Koh, H.J. (2014) Analysis of segregation distortion and its relationship to hybrid barriers in rice. Rice, 7 (3), 1–12.
Rotchanapreeda, T., Wongniam, S., Swangpol, S.C., Chareonsap, P.P., Sukkaewmanee, N. & Somana, J. (2016) Development of SSR markers from Musa balbisiana for genetic diversity analysis among Thai bananas. Plant Systematics and Evolution, 302 (7), 739–761.
Santana, F.A., da Silva, M.F., Guimaraes, J.K.F., Ferreira, M.F.S., Pereira, W.D., Piovesan, N.D. & de Barros, E.G. (2014) Marker-assisted selection strategies for developing resistant soybean plants to cyst nematode.
Crop Breeding and Applied Biotechnolog, 14 (3), 180– 186.
Santoso, P.J., Djamas, N., Rebin & Pancoro, A. (2014) Analisis diversitas dan paternitas progeni F1 hasil
per-silangan Arumanis 143 Mangga Merah mengguna-kan marka mikrosatelit. Jurnal Hortikultura, 24 (3), 210– 219.
Subashini, V., Shanmugapriya, A. & Yasodha, R. (2013) Hybrid purity assessment in Eucalyptus F1 hybrids
using microsatellite markers. Biotechnology, 4 (4), 367– 373.
Vincent, K., Robert, K., Morag, F., Tadeo, K., Yona, B. & Peter, V. (2014) Identification of F1 cassava (Manihot esculenta) progeny using microsatellite markers and capillary electrophoresis. American Journal of Plant Science, 5 (1), 119–125.
Wahyuningtyas, W., Retnoningsih, A. & Rahayu, E.S. (2009) Keanekaragaman genetika pisang bergenom B berdasarkan penanda mikrosatelit. Biosaintifika: Journal of Biology & Biology Education, 1 (1), 1–10. Zuo, C., Deng, G., Li, B., Huo, H., Li, C., Hu, C., Kuang, R.,
Yang, Q., Dong, T., Sheng, O. & Yi, G. (2018) Germplasm screening of Musa spp. for resistance to
Fusarium oxysporum f. sp. cubense tropical race 4 (Foc
TR4). European Journal of Plant Pathology, 151, 723– 734.