Docking Molekular Terbalik dari Senyawa Zerumbon
Reverse Molecular Docking of Zerumbone
Broto Santoso*, Dedi Hanwar, Andi Suhendi, Ika Trisharyanti Dian Kusumowati, Rosita Melannisa
Faculty of Pharmacy, Universitas Muhammadiyah Surakarta *Broto.Santoso@ums.ac.id
ABSTRACT
Background: Main compounds contained in Zingiber zerumbet are zerumbone and two
of its derivates, humulen and epoxy-humulen. Previous research for its antioxidant activity using DPPH did not showed the better potency from the activity of known compounds. However zerumbone has stated several researchers have a variety of biological activities. Reverse molecular docking can be used to determine how close the predictions are given for a variety of biological activities claimed.
Methods: The study was conducted in a way, all protein targets acquired from
www.pdb.org and can represent the chosen biological activity and target ligand (zerumbone and its derivatives gained from PubChem) were prepared using Chimera, including protein spheres calculations. Dock6 software that achieved from http://dock.compbio.ucsf.edu was used for computing the gridbox of binding site pockets of protein and binding activity of the target protein-ligand. Molecular docking was performed to the native ligand of the target protein and three compounds of zerumbone using Dock6 to 59-target protein.
Results: The results showed that RMSD value of native ligands obtained from
molecular docking have suited the requirement compared to its crystallography product. All of zerumbone have the ligand-protein binding score improved sequentially in 2QAK protein (protein of HIV-1 PR mutant is one of the successful crystallography result of HIV protease ligands with nelfinavir), 4NOS (protein of the human inducible nitric oxide synthase inhibitor with antioxidant activity that represented the mechanism of enzyme inhibition nitric oxide synthase), 3D9C (crystal structure of PTP1B complex acids with aryl Seleninic representing anti-diabetic activity through the mechanism of inhibition of tyrosine phosphorylation) and 1D6N (ternary complex structure of human
HGPRTase, PRPP, Mg2+ and the inhibitor HPP reveals the involvement of the flexible
loop in substrate binding is the enzyme hypoxanthine phosphoribosyltransferase (HGPRTase) which is responsible for rheumatic arthritis).
Conclusions: The research proves that zerumbone allegedly still has antioxidant
activity, anti-HIV, anti-diabetic and anti-arthritic with a particular mechanism in accordance with the target protein. Further research on the four modeling activities must be carried out in the laboratory to validate the reverse approach to molecular docking.
Keywords: Zerumbone, Reverse Molecular Docking, Dock6, Chimera, Ligand-Protein.
PENDAHULUAN
Zerumbon (ZER) beserta dua molekul ikutannya, yaitu hidroksi humulen (HH) dan epoksi humulen (EPH) dapat diperoleh dari ekstraksi Zingiber zerumbet. Beberapa peneliti menyatakan bahwa zerumbon memiliki aktivitas biologis yang berguna untuk pengobatan, diantaranya adalah sebagai antikanker, antioksidan, anti inflamasi, antipiretik, antibakteri, antimalaria dan antiviral (Sriphana et al., 2013; Singh et al., 2012; Sutthanont et al., 2010).
Hasil penelitian sebelumnya untuk sifat antioksidan zerumbon melalui mekanisme penangkapan radikal bebas menggunakan metode DPPH diperoleh hasil yang kurang baik, yaitu % inhibisi untuk ekstrak rimpang dan daun berturut-turut adalah 2123 dan 326,24 ppm (Hanwar et al., 2012). Hal ini dimungkinkan terjadi karena reaksi penangkapan radikal bebas DPPH oleh zerumbon tidak dapat mewakili aktivitas antioksidan zerumbon yang telah diteliti sebelumnya oleh Giang et al. (2009). Mekanisme antioksidan zerumbon yang dilaporkan tersebut adalah melalui penghambatan pembentukan NO dalam lipopolisakarida.
Aktivitas antioksidan zerumbon yang tidak terekspresikan oleh uji DPPH menjadi landasan diperlukan studi lainnya,
dalam hal ini dipilih secara in silico. Studi ini pun diharapkan dapat menjembatani pendekatan mekanisme aksi dari beberapa aktivitas biologis yang dimiliki oleh zerumbon atau didapatkan aktivitas biologis lainnya. Studi in silico dilakukan dengan metode molecular docking menggunakan perangkat lunak Dock6 (diperoleh dari http://dock.compbio.ucsf.edu) dalam sistem operasi Ubuntu (virtual).
SUBJEK DAN METODE
Perangkat keras yang digunakan dalam penelitian ini adalah seperangkat komputer dengan spesifikasi prosesor Intel QuadCore Q6600 2,4GHz dengan random access memory (RAM) 8 GB dan video graphic adapter (VGA) NVidia GT520 dengan sistem operasi Linux Ubuntu secara virtualisasi. Perangkat lunak yang digunakan adalah Dock6, Chimera, OpenBabel, LigPlot+ dan PyMOL.
Metodologi penelitian yang dilakukan secara singkat dapat diuraikan sebagai berikut: semua protein target yang diperoleh dari www.pdb.org (maksimal resolusi kristal adalah 2,5 Å) dipreparasi menggunakan perangkat lunak Chimera untuk memisahkan antara protein dan ligannya kemudian dilakukan penambahan hidrogen dan muatan menggunakan script DockPrep. Chimera pun digunakan untuk melakukan preparasi dalam proses
kalkulasi sphere dari protein untuk mendapatkan file DMS yang digunakan pada tahap molecular docking. Molekul zerumbon, hidroksi humulen dan epoksi humulen diperoleh dari database PubChem dan dilakukan preparasi menggunakan Chimera dan digabungkan menjadi satu file menggunakan bantuan OpenBabel.
Perhitungan sphere dilakukan menggunakan Chimera dan dilanjutkan dengan penentuan gridbox (area terpilih untuk melakukan docking berdasarkan posisi dari ligan asli) dari protein target dan dilakukan validasi proses molecular docking menggunakan perangkat lunak Dock6. Sistem terpilih adalah jika nilai RMSD dari konformasi 3 dimensi (3D) ligan asli dari protein hasil molecular docking yang diperoleh mendekati nilai 0 atau dengan kata lain posisi 3D ligan hasil molecular docking mendekati posisi 3D dari ligan kristalnya. Nilai RMSD diperoleh dengan melakukan alignment kedua molekul dengan PyMOL.
Zerumbon dan turunannya dilakukan molecular docking dengan sistem terpilih tersebut menggunakan Dock6.
Visualisasi 3D hasil menggunakan Chimera (Gambar 1), untuk interaksi ligan zerumbon dan turunannya terhadap protein terpilih secara 3D menggunakan PyMOL (Gambar 2, 6, 8, 10 dan 12) dan interaksi 2D ligan-protein menggunakan LigPlot+ (Gambar 5, 7, 9, 11, 13) menunjukan residu-residu protein yang terlibat.
HASIL
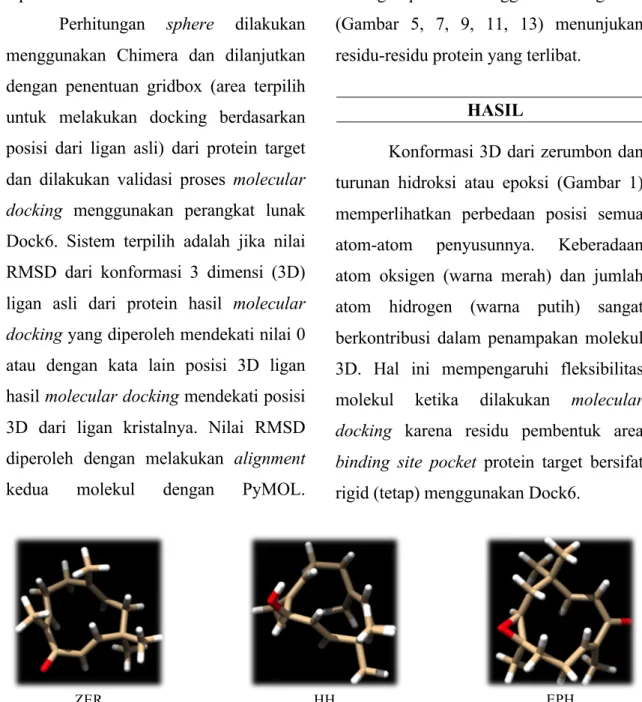
Konformasi 3D dari zerumbon dan turunan hidroksi atau epoksi (Gambar 1) memperlihatkan perbedaan posisi semua atom-atom penyusunnya. Keberadaan atom oksigen (warna merah) dan jumlah atom hidrogen (warna putih) sangat berkontribusi dalam penampakan molekul 3D. Hal ini mempengaruhi fleksibilitas molekul ketika dilakukan molecular docking karena residu pembentuk area binding site pocket protein target bersifat rigid (tetap) menggunakan Dock6.
ZER HH EPH
Gambar 1. Konformasi 3D zerumbon (ZER), hidroksi humulen (HH) dan epoksi humulen (EPH) dari
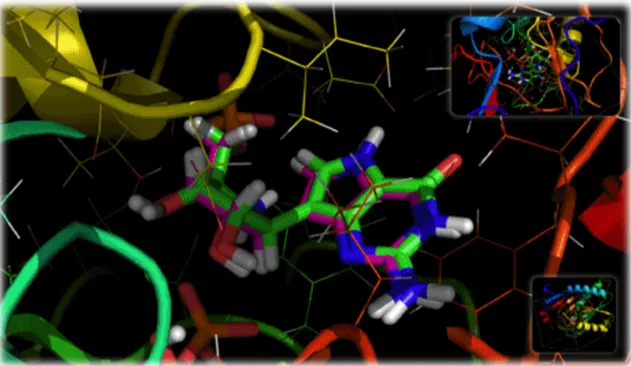
Gambar 2. Contoh hasil validasi ligan-protein: 1BZY, hijau=ligan hasil kristalografi dan merah
muda=ligan hasil molecular docking dengan Dock6 (HA_RMSD = 0.20217, RMSD = 0.067). Validasi konformasi 3D telah
dilakukan terhadap molekul ligan asli yang menyertai protein target hasil kristalografi dengan konformasi 3D hasil dari perhitungan molecular docking perangkat
lunak Dock. Salah satu contoh terdapat pada Gambar 2 yang merupakan ligan kuanosin-5-fosfat modifikasi dari protein target hipoksantin-guanin fosforibosil-transferase pada manusia.
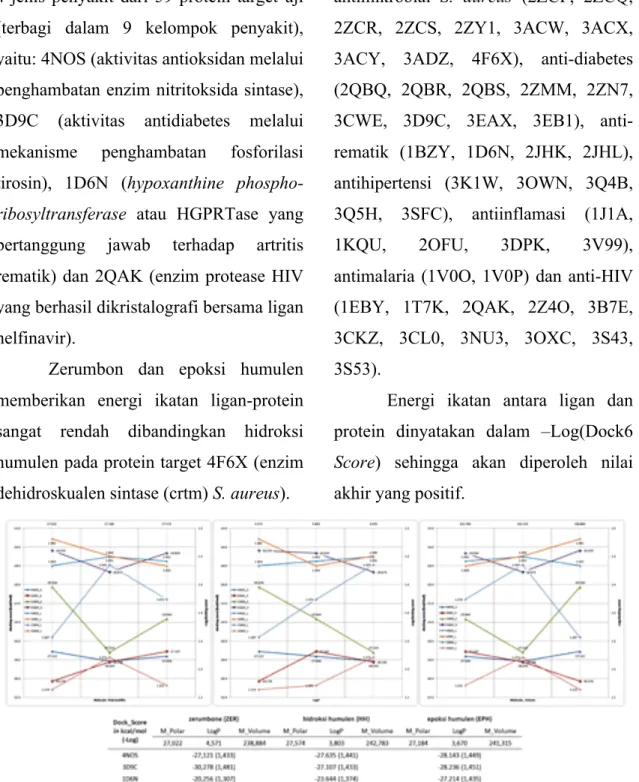
Gambar 3. Hasil molecular docking (-Log10 Dock6 Score (kkal/mol)) antara ligan (L_Protein, biru)
dalam kristal protein target (diperoleh dari www.pdb.org), zerumbone (ZER, merah), hidroksi-humulen (HH, hijau) dan epoksi-humulen (EPH, ungu) yang diperoleh dari database PubChem dengan berbagai protein yang mewakili mekanisme obat sebagai antioksidan (6 protein), analgesik (7 protein), antibakteri terhadap Staphylococcus aureus (10 protein), anti-diabetes (9 protein), anti-rematik (4 protein), antihipertensi (5 protein), anti inflamasi (5 protein), antimalaria (2 protein) dan antiviral (11 protein).
Skrining molecular docking memberikan hasil seperti pada Gambar 3. Zerumbon dan dua senyawa turunannya menunjukkan energi ikatan yang terbaik hanya pada 4 protein target yang mewakili 4 jenis penyakit dari 59 protein target uji (terbagi dalam 9 kelompok penyakit), yaitu: 4NOS (aktivitas antioksidan melalui penghambatan enzim nitritoksida sintase), 3D9C (aktivitas antidiabetes melalui mekanisme penghambatan fosforilasi tirosin), 1D6N (hypoxanthine phospho-ribosyltransferase atau HGPRTase yang bertanggung jawab terhadap artritis rematik) dan 2QAK (enzim protease HIV yang berhasil dikristalografi bersama ligan nelfinavir).
Zerumbon dan epoksi humulen memberikan energi ikatan ligan-protein sangat rendah dibandingkan hidroksi humulen pada protein target 4F6X (enzim dehidroskualen sintase (crtm) S. aureus).
Kelompok penyakit dari protein target yang digunakan adalah analgesik (1S2A, 2BXG, 2BXK, 2BXM, 2ZB8, 3ADS, 3ADX), antioksidan (1JNK, 1TDI, 1YVL, 3LJR, 4NOS, 18GS), antimikrobial S. aureus (2ZCP, 2ZCQ, 2ZCR, 2ZCS, 2ZY1, 3ACW, 3ACX, 3ACY, 3ADZ, 4F6X), anti-diabetes (2QBQ, 2QBR, 2QBS, 2ZMM, 2ZN7, 3CWE, 3D9C, 3EAX, 3EB1), anti-rematik (1BZY, 1D6N, 2JHK, 2JHL), antihipertensi (3K1W, 3OWN, 3Q4B, 3Q5H, 3SFC), antiinflamasi (1J1A, 1KQU, 2OFU, 3DPK, 3V99), antimalaria (1V0O, 1V0P) dan anti-HIV (1EBY, 1T7K, 2QAK, 2Z4O, 3B7E, 3CKZ, 3CL0, 3NU3, 3OXC, 3S43, 3S53).
Energi ikatan antara ligan dan protein dinyatakan dalam –Log(Dock6 Score) sehingga akan diperoleh nilai akhir yang positif.
Gambar 4. Keterkaitan antara deskriptor ligan (molecule polarizability, LogP dan molecule volume)
dengan hasil docking (Dock6 Score beserta nilai –Log-nya) ligan (zerumbon, hidroksi humulen dan epoksi humulen) dan protein (4NOS, 3D9C, 1D6N dan 2QAK).
Hubungan antara besaran tiga deskriptor ligan ketiga molekul uji dengan nilai perolehan energi ikatan ligan-protein (kkal/mol dan –Log-nya) pada keempat protein target terpilih dipaparkan dalam Gambar 4. Interaksi kimia antara
molekul-molekul uji dengan residu-residu (asam amino) dari protein-protein target terpilih ditunjukkan pada Gambar 5, 7, 9 dan 11. Beberapa interaksi kimia yang terjadi dapat berupa interaksi hidrofobik dan interaksi ikatan hidrogen.
Gambar 5. Interaksi ligan ZER, HH dan EPH dengan residu protein 4NOS.
Gambar 6. Konformasi 3D interaksi antara ligan ZER, HH dan EPH dengan protein 4NOS.
Gambar 8. Konformasi 3D interaksi antara ligan ZER, HH dan EPH dengan protein 3D9C.
Gambar 9. Interaksi ligan ZER, HH dan EPH dengan residu protein 1D6N.
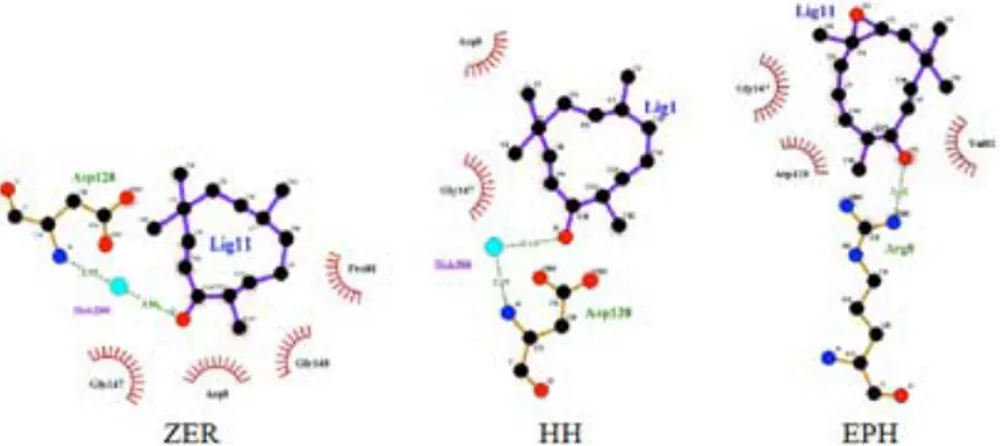
Gambar 11. Interaksi ligan ZER, HH dan EPH dengan residu protein 2QAK.
Gambar 12. Konformasi 3D interaksi antara ligan ZER, HH dan EPH dengan protein 2QAK. Tabel 1. Residu-residu protein target yang berinteraksi dengan ligan uji (ikatan hidrogen=garis bawah,
residu sama=ditebalkan, residu mirip ligan asli=latar gelap dan selainnya adalah interaksi hidrofobik).
PDB-id ZER HH EPH
4NOS Hem422, Gln181, Arg299, Asp300, Asp303, Arg306,
H2b423
Hem422, Gln181, Val270, Asp300, Arg306
Hem422, Gln181, Arg184,
Tyr291, Glu295, Arg299,
Asp300, Arg306
Phe369, Trp372, Glu377; Data PoseView*: Glu377, Trp372
3D9C Glu114, Lys115, Lys119, Gly182, Cys214, Ser215,
Arg220, Gln261, Gln265
Tyr45, Lys115, Lys119, Cys214, Ser215, Arg220,
Gln261, Thr262, Gln265
Glu114, Lys115, Lys119, Trp178, Gly182, Ser215,
Arg220, Gln261, Gln265
Asp48, Cys215, Ala217, Arg221, Gln262; Data PoseView*: Tyr45, Arg220
1D6N Asp104, Gln105, Lys162, Arg166, Asp181, Lys182
Asp104, Gln105, Lys162, Arg166, Asp181, Lys182,
Phe183
Asp104, Asp134, Lys162, Arg166, Asp181, Lys209
Ile135, Asp137, Lys165, Phe186, Val187, Asp193; Data PoseView*: Lys165, Phe186
2QAK Arg8, Pro81, Asp128, Gly147, Gly148,
Arg8, Asp128, Gly147 Arg8, Val82, Asp128,
Gly147
Asp25, Asp29, Asn30, Ile50, Pro81, Data PoseView*: Asp25, Gly27, Ala28, Asn30, Val32, Ile50, Pro81, Val82, Ile84
*diperoleh dari www.pdb.org
Penampakan warna residu yang memiliki interaksi hidrofobik dengan ligan oleh perangkat lunak LigPlot+ ditampakkan berupa kode dari residu dengan lingkaran berupa sisir berwarna merah, sedangkan interaksi ikatan hidrogen ditunjukkan dengan struktur dari residu secara utuh dengan garis interaksi ikatan berwarna hijau.
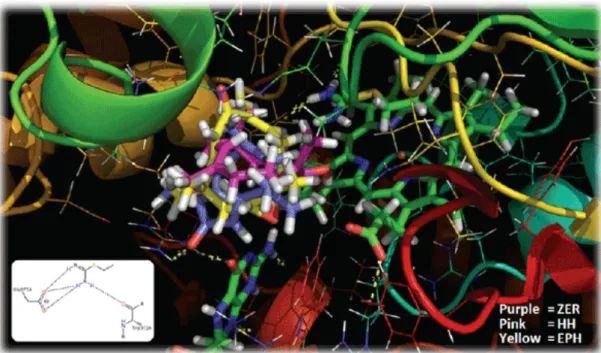
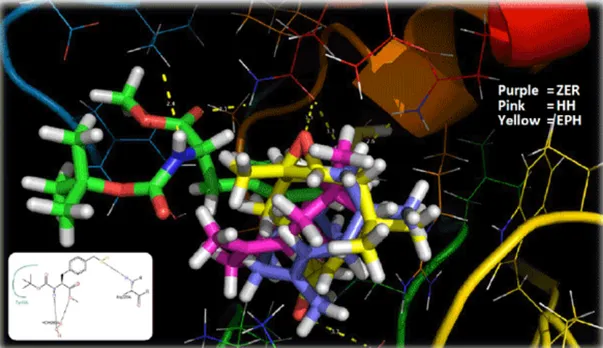
Visualisasi 3D dari interaksi yang terjadi antara ligan uji dengan protein target terpilih diperlihatkan pada Gambar 6, 8, 9 dan 11 disertai dengan visualisasi oleh perangkat lunak PoseView dari
keempat ligan asli protein target tersebut. Zerumbon ditampilkan dengan kerangka karbon berwarna ungu, sedangkan hidroksi humulen dan epoksi humulen secara berurutan berwarna merah muda dan kuning.
Residu-residu protein yang terlibat dalam interaksi kimia antara ligan dan protein disimpulkan dalam Tabel 1. Tabel ini pula menampilkan residu-residu dari protein target terpilih yang berinteraksi dengan ligan aslinya (Gambar 13). LigPlot+ yang digunakan telah dilakukan beberapa modifikasi.
PEMBAHASAN
Reverse atau inverse molecular docking (RMD) merupakan teknik rancang obat yang relatif baru. RMD dapat digunakan untuk melakukan skrining protein target yang potensial terhadap ligan. Teknik ini dapat diaplikasikan untuk mengenali aktivitas biologis yang belum diketahui atau aktivitas terapetik kedua dari suatu obat, senyawa penuntun, produk alam dan ligan-ligan lainnya (Zheng et al., 2011; Chen and Zhi, 2001).
Metode ini telah dikembangkan oleh banyak akademisi dan praktisi peneliti di kalangan industri obat baik ditujukan untuk penelitian murni atau bahkan untuk dikomersialkan. Strategi spesifik untuk reverse docking dapat menggunakan perangkat lunak yang sudah ada, seperti Dock, MDock, Vina, Gold, FlexX, Maestro secara offline atau Tarfisdock, PharmMapper, idTarget yang dapat diakses secara online (Chen and Ren, 2014). Chen and Ung (2001) berhasil menemukan strategi yang efektif dalam memprediksi potensi ketoksikan dan efek samping molekul kecil obat menggunakan metode RMD.
Zerumbon merupakan molekul kecil yang dihasilkan oleh Z. zerumbet dan dalam kuantitas yang dominan oleh
karenanya senyawa ini merupakan marker dari spesis tanaman ini. Pengembangan metode isolasi dari sumber tanaman Indonesia telah dikembangkan (Hanwar et al., 2013) untuk memperoleh konstituen zerumbon murni. Zerumbon yang diperoleh dapat digunakan untuk mengidentifikasi aktivitas biologi lainnya yang dimiliki dikarenakan zerumbon tidak menunjukkan aktivitas penangkap radikal melalui mekanisme penangkapan radikal bebas DPPH (Hanwar et al., 2012).
Zerumbon beserta dua senyawa ikutannya yaitu hidroksi humulen dan epoksi humulen merupakan senyawa utama Z. zerumbet. Ekstrak tanaman ini sendiri telah banyak diteliti dan memiliki berbagai khasiat. Hal inilah yang mendasari dalam pemilihan protein target yang digunakan dalam melakukan RMD ketiga senyawa penanda Zingiber zerumbet. Beberapa peneliti bahkan telah mengembangkan turunan senyawa dari zerumbon dan mengujicobakan beberapa aktivitas biologi yang ditemukan ketika masih berbentuk ekstrak tanaman (Kitayama et al., 2013; Santosh Kumar et al., 2013; Pitchuanchom et al., 2011).
Protein target sebanyak 59 buah dikelompokkan dalam 9 jenis macam penyakit berdasarkan kajian yang telah dilakukan terhadap ekstrak tanaman penghasil zerumbon. Hasil interaksi ikatan
kimiawi secara virtual melalui perhitungan docking menunjukkan bahwa hanya terdapat 4 protein target yang berhasil dilampaui nilai aktivitas ikatan ligan-protein dibandingkan dengan ligan aslinya. Besaran afinitas ikatan ligan uji dibandingkan dengan 3 jenis deskriptor ketiganya (Gambar 4) akan diperoleh hubungan keterkaitan, yaitu semakin kecil nilai logP yang dimiliki ligan uji akan menaikkan nilai aifinitas ikatan ligan-protein 4NOS secara perlahan dan 1D6N dengan signifikan. Kedua protein lainnya, 3D9C dan 2QAK tidak memberikan profil keterkaitan yang cukup baik. Polaritas dan volume 3D molekul akan mempengaruhi hasil ikatan ligan-protein hanya pada 3D9C walaupun nilai kedua deskriptor untuk ligan uji tidak terlalu berbeda. Besaran kedua deskriptor cenderung ditentukan oleh jumlah atom oksigen dan jenis gugus fungsional dari atom tersebut yang bisa berupa karbonil keton, epoksi atau hidroksil.
Interaksi ikatan antara residu protein dengan ligan uji menunjukkan bahwa atom oksigen berperan dalam interaksi ikatan hidrogen baik secara langsung atau melalui perantara komponen air pada protein 2QAK (Gambar 11), zerumbon dan hidroksi humulen pada 4NOS (Gambar 5). Ligan
uji akan mengalami interaksi hidrofobik pada protein 3D9C (Gambar 7), kedua humulen pada 1D6N (Gambar 9) serta epoksi humulen pada 4NOS. Interaksi yang terjadi memperlihatkan bahwa atom O pada ligan menjadi faktor penentu afinitas ikatan ligan-protein yang juga telah dibuktikan oleh Kitayama et al. (2013) dalam mengembangkan senyawa novel turunan zerumbon.
Gambar 6, 8, 10 dan 12 memperlihatkan bahwa tidak ada ligan uji yang memiliki konformasi 3D yang saling berhimpitan satu sama lainnya diantara atom-atom penyusunnya. Ini menjelaskan perbedaan awal yang terjadi pada Gambar 1 sebelum ketiganya dilakukan docking bahwa ketiga ligan memiliki konformasi 3D dasar yang berbeda. Namun ligan-ligan uji ini memiliki kemiripan residu-residu yang berinteraksi dengan ligan seperti yang terlihat pada Tabel 1 (ditandai dengan ditebalkan).
Hal menarik lainnya yang perlu dikonfirmasi melalui penelitian lanjutan adalah ada beberapa residu protein yang menentukan nilai afinitas ikatan ligan-protein sama dengan hasil pemotretan interaksi ligan-proetin yang terjadi dengan menggunakan LigPlot+ dan PoseView pada Tyr45 untuk hidroksi humulen-3D9C dan Pro81 untuk zerumbon-2QAK. Hal ini menjadi landasan kuat bahwa ada
kemiripan interaksi yang terjadi antara ligan uji dengan ligan asli hasil kristalografi dari protein target dan mendukung bahwa ligan dapat memiliki aktivitas yang sama atau bahkan lebih baik.
SIMPULAN
Hasil penelitian membuktikan bahwa zerumbon diduga masih memiliki aktivitas antioksidan, anti-HIV, antidiabetes dan anti-rematik dengan mekanisme tertentu sesuai dengan protein targetnya.
SARAN
Penelitian lanjutan terhadap keempat pemodelan aktivitas tersebut perlu dilakukan di laboratorium untuk membuktikan kebenaran pendekatan reverse molecular docking (RMD).
UCAPAN TERIMA KASIH Penulis berterima kasih kepada Lembaga Penelitian dan Pengabdian Masyarakat (LPPM) Universitas Muhammadiyah Surakarta yang telah mendanai Penelitian Unggulan Program Studi (PUPS) ini.
DAFTAR PUSTAKA
Chen SJ and Ren JL (2014). Identification
of a Potential Anticancer Target Of Danshensu by Inverse Docking. Asian Pac J Cancer Prev, 15(1): 111-6.
Chen YZ and Zhi DG (2001). Ligand-Protein Inverse Docking and Its Potential Use in The Computer Search of Protein Targets of A Small Molecule. Proteins, 43(2: 217-26.
DOCK6.5 (2011). University of California at San Francisco; San Francisco, CA.
Giang PM, Son PT, Jin HZ, Lee JH and Lee JJ (2009). Comparative Study on Inhibitory Activity of Zerumbone and Zerumbone 2,3-Epoxide on NF-κB Activation and NO Production. Sci. Pharmaceutica, 77(3): 589-95.
Hanwar D, Suhendi A, Santoso B, Kusumowati ITD. and Melannisa R (2013). Isolation and Purification of Chemical Marker from Zingiber zerumbet Rhizome from Indonesia.
International Conference on
Natural Products. Shah Alam, Malaysia, 4-6 March 2013, 11. Hanwar D, Suhendi A, Santoso B,
Kusumowati ITD. dan Melannisa R (2012). Pengembangan Lempuyang Gajah (Zingiber zerumbet) sebagai Obat Herbal untuk Antioksidan. Laporan Tahun Pertama Penelitian Unggulan Program Studi (PUPS). Fakultas Farmasi, Universitas Muhammadiyah Surakarta, Desember 2012, 38.
Kitayama T, Nakahira M, Yamasaki K, Inoue H, Imada C, Yonekura Y, Awata M, Takaya H, Kawai Y, Ohnishi K and Murakami A (2013). Novel Synthesis of Zerumbone-pendant Derivatives
and Their Biological Activity.
Tetrahedron, 69(47:
10152-10160.
O'Boyle N, Banck M, James C, Morley C, Vandermeersch T and Hutchison G (2011). Open Babel: An Open Chemical Toolbox. Journal of Cheminformatics, 3(1): 33.
Pettersen EF, Goddard TD, Huang CC, Couch GS, Greenblatt DM, Meng EC and Ferrin TE (2004). UCSF Chimera--a Visualization System for Exploratory Research and Analysis. J. Comput. Chem., 25(13): 1605-12.
Pitchuanchom S, Songsiang U, Weerapreeyakul N and Yenjai C (2011). Anticancer Activity of the Bioreductive and Non-Bioreductive Zerumbone Derivatives. Letters in Drug Design and Discovery, 8(6): 536-543.
PyMOL (2012). Molecular Graphics System, Version 1.6.0 Schrödinger, LLC.
Santosh Kumar SC, Srinivas P, Negi PS and Bettadaiah BK (2013). Antibacterial and Antimutagenic Activities of Novel Zerumbone Analogues. Food Chemistry, 141(2): 1097-1103.
Singh CB, Nongalleima K, Brojendrosingh S, Ningombam S, Lokendrajit N and Singh LW (2012). Biological and Chemical Properties of Zingiber zerumbet Smith: a Review. Phytochemistry Reviews, 11(1): 113-125.
Sriphanaa U, Pitchuanchoma S, Kongsaereeb P and Yenjai C (2013). Antimalarial Activity and Cytotoxicity of Zerumbone Derivatives. ScienceAsia, 39(1): 95-99.
Sutthanont N, Choochote W, Tuetun B, Junkum A, Jitpakdi A, Chaithong U, Riyong D and Pitasawat B (2010). Chemical Composition and Larvicidal Activity of Edible Plant-Derived Essential Oils Against the Pyrethroid-Susceptible and -Resistant Strains of Aedes aegypti (Diptera: Culicidae). J. Vector Ecology, 35(1): 106-115.
Zheng R, Chen TS and Lu T (2011). A Comparative Reverse Docking Strategy to Identify Potential Antineoplastic Targets of Tea Functional Components and Binding Mode. Int. J. Mol. Sci., 12(8): 5200-12.