IDENTIFIKASI
Melastoma
sp. DI SEKITAR KAMPUS IPB
MENGGUNAKAN DNA BARKODE GEN
maturase K
(
matK
)
RHADITYA EKA PRATAMA
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa skripsi berjudul Identifikasi Melastoma
sp. di Sekitar Kampus IPB Menggunakan DNA Barkode Gen maturase K (matK) adalah benar karya saya bersama dengan komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, November 2015
Rhaditya Eka Pratama
ABSTRAK
RHADITYA EKA PRATAMA. Identifikasi Melastoma sp. di Sekitar Kampus IPB Menggunakan DNA Barkode Gen maturase K (matK). Dibimbing oleh UTUT WIDYASTUTI dan SUHARSONO.
Melastoma sp. merupakan tumbuhan liar yang banyak tumbuh di Indonesia dan dapat tumbuh sampai ketinggian 1650 mdpl. Tujuan penelitian ini adalah identifikasi spesies Melastoma sp. di Kampus IPB menggunakan DNA barkode gen
matK. Primer yang digunakan untuk PCR dan sekuensing adalah primer matK-3F dan matK-3R menghasilkan fragmen nukleotida sekitar 750 pb. Selain identifikasi secara molekular, juga diamati morfologi daun dan bunganya. DNA genom berasal dari daun Melastoma sp. digunakan sebagai DNA cetakan dalam PCR. Fragmen gen matK hasil PCR digunakan untuk sekuensing. Hasil urutan nukleotida kemudian dianalisis menggunakan BLAST pada situs NCBI. Hasil identifikasi
Melastoma sp. menunjukkan bahwa morfologi bunga sampel asal Cikabayan, Pool Bus IPB, Rektorat, dan Jasinga Hijau adalah Melastoma malabathricum L. Hasil BLAST pada situs NCBI juga menunjukkan kemiripannya dengan Melastoma malabathricum subsp. normale KP093758.1, meskipun nilai max score sampel tersebut menunjukkan kemiripan terhadap Melastoma saigonense. Sampel asal
Fapet adalah Melastoma saigonense.
Kata kunci: DNA barkode, gen matK, sekuensing
ABSTRACT
RHADITYA EKA PRATAMA. Identification of Melastoma sp. in Around IPB Campus Using DNA Barcode of maturase K (matK) Gene. Supervised by UTUT WIDYASTUTI and SUHARSONO.
Melastoma sp. is one of the wild plant that grows in Indonesia and can grow at altitude of 1650 meters above sea level. The objective of this research is to identify species of Melastoma sp. in IPB campus using DNA barcodes of matK
gene. The primer sequence that was used were matK-3F and matK-3R than produced about 750 bp of nucleotide for sequencing. Additionally, the iden-tification of Melastoma sp. was also carried out by observing the morphology of leaves and flowers. DNA which is from Melastoma sp. leaves were used as DNA template on PCR process. Fragment of matK gene as the result of PCR was used for sequencing. The sequence then was analyzed using BLAST on NCBI site. The result showed that the flowers morphology of sample from Cikabayan, Pool Bus IPB, Rektorat, and Jasinga Hijau were Melastoma malabathricum L. The result of BLAST on NCBI site also showed their similarity with Melastoma malabathricum
subsp. normale KP093758.1 though their max score were similar to Melastoma saigonense. The sample from Fapet was Melastoma saigonense.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Sains
pada
Departemen Biologi
IDENTIFIKASI
Melastoma
sp. DI SEKITAR KAMPUS IPB
MENGGUNAKAN DNA BARKODE GEN
maturase K
(
matK
)
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
BOGOR 2015
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah SWT atas segala karunia-Nya sehingga skripsi ini berhasil diselesaikan. Tema yang dipilih dalam usulan penelitian yang telah dilaksanakan sejak bulan Februari 2015 hingga April 2015 ini adalah DNA barkode, dengan judul Identifikasi Melastoma sp. di Sekitar Kampus IPB Menggunakan DNABarkode Gen maturase K (matK).
Terima kasih penulis ucapkan kepada Dr Ir Utut Widyastuti, MSi selaku pembimbing pertama dan Prof Dr Suharsono, DEA selaku pembimbing kedua. Terima kasih penulis ucapkan kepada Dr Sri Listiyowati selaku dosen penguji yang telah memberikan saran dan masukan terhadap karya ilmiah penulis. Penelitian ini menggunakan dana penelitian mandiri atas nama Dr Ir Utut Widyastuti, MSi. Terima kasih juga penulis ucapkan kepada seluruh staf di Laboratorium
Biotechnology Research Indonesia-The Netherlands (BIORIN), Pusat Penelitian Sumber Daya Hayati dan Bioteknologi (PPSHB) IPB, khususnya untuk Bapak Abdul Mulya, Ibu Pepi Elvavina, Ibu Nia Dahniar, SP, dan Bapak Sairi atas segala bantuan. Ucapan terimakasih juga penulis sampaikan kepada rekan-rekan seperjuangan S-1 Silmi Aziza Azmi, Nurul Khafidoh, Zakiyyatul Maftuhah, Santi Murtiningsih, Zani Umami, Amanda Dwikarina, Abdullah Adzky Rabbani, Muhamad Junaidi, Vestika Iskawati WH, Gia Permasku, dan rekan-rekan Pascasarjana Kak Luthfi, Kak Ari, Kak Fatah, Kak Yusdar, Kak Fajri, Kak Holifah, Kak Nurul, Kak Win, Kak Seni, Kak Nadea, Kak Tiwi, Kak Ivan, Ibu Ifa, Ibu Idha, Pak Asri, dan Pak Ilyas dan teman-teman Biologi 48 yang ikut mendukung dalam penelitian ini. Penghargaan terbesar penulis sampaikan kepada kedua orang tua Bapak Kamijan dan Ibu Rodiyah, SPd, dan Adik M. Rayhan Kurniawan, serta
seluruh keluarga atas do’a, semangat, dan kasih sayangnya. Semoga karya ilmiah ini bermanfaat.
Bogor, November 2015
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
PENDAHULUAN 1
METODE 2
Waktu dan Tempat 2
Alat dan Bahan 2
Metode 2
HASIL DAN PEMBAHASAN 4
Morfologi Daun Melastoma sp. 4
Identifikasi Fragmen Gen matK 6
Analisis Urutan Nukleotida Fragmen Gen matK 7
SIMPULAN DAN SARAN 14
Simpulan 14
Saran 14
DAFTAR PUSTAKA 14
LAMPIRAN 17
DAFTAR TABEL
1 Morfologi daun Melastoma sp. di Kampus IPB yang telah diidentifikasi 6 2 Homologi tertinggi fragmen gen matK dari 5 sampel dengan
membandingkan data di GeneBank menggunakan BLAST (www.ncbi.
nlm.nih.gov/blastn) 8
3 Morfologi bunga Melastoma sp. di Kampus IPB yang telah
diiden-tifikasi 9
4 Variasi posisi nukleotida fragmen gen matK dari subtitusi 5 sampel dan 2 spesies pembanding dari GeneBank pada urutan basa 1 sampai
ke-31 11
5 Variasi posisi nukleotida fragmen gen matK dari subtitusi 5 sampel dan 2 spesies pembanding dari GeneBank pada urutan basa 32 sampai
ke-64 11
DAFTAR GAMBAR
1 Morfologi daun Melastoma sp. di Kampus IPB yang akan diidentifikasi; (a) Cikabayan, (b) Gymnasium, (c) GOR Lama, (d) Leuwiliang, (e)
Gunung Walat, (f) Fapet, (g) FKH, (h) Gerbang Utama IPB, (i) Astra, (j) Pool Bus IPB, (k) Green House 2, (l) Rektorat, dan (m) Jasinga
Hijau 5
2 PCR untuk fragmen gen matK; (M) marker 1 kb, (1) Cikabayan, (2)
Fapet, (3) Pool Bus IPB, (4) Rektorat, dan (5) Jasinga Hijau 6 3 Morfologi bunga Melastoma sp. di Kampus IPB yang akan diidentifikasi;
(a) Cikabayan, (b) Fapet, (c) Pool Bus IPB, (d) Rektorat, dan (e) Jasinga
Hijau 9
4 Urutan nukleotida fragmen gen matK dari sampel asal Cikabayan dan
deduksi asam amino 12
5 Urutan nukleotida fragmen gen matK dari sampel asal Fapet dan deduksi
asam amino 12
6 Urutan nukleotida fragmen gen matK dari sampel asal Pool Bus IPB dan
deduksi asam amino 13
7 Urutan nukleotida fragmen gen matK dari sampel asal Rektorat dan
deduksi asam amino 13
8 Urutan nukleotida fragmen gen matK dari sampel asal Jasinga Hijau dan
deduksi asam amino 14
DAFTAR LAMPIRAN
1 Diagram alir penelitian 18
2 Cara membuat larutan bufer TAE 50x dan TAE 1x 19
3 Komposisi larutan CTAB 2% 20
4 Hasil analisis BLAST menggunakan urutan nukleotida fragmen gen
5 Hasil analisis BLAST menggunakan urutan nukleotida fragmen gen
matK sampel asal Fapet 22
6 Hasil analisis BLAST menggunakan urutan nukleotida fragmen gen
matK sampel asal Pool Bus IPB 23
7 Hasil analisis BLAST menggunakan urutan nukleotida fragmen gen
matK sampel asal Rektorat 24
8 Hasil analisis BLAST menggunakan urutan nukleotida fragmen gen
matK sampel asal Jasinga Hijau 25
9 Penyejajaran berganda fragmen gen matK dari 5 sampel dan 2 spesies
pembanding dari GeneBank 26
10 Ciri-ciri morfologi Melastoma malabathricum (Backer dan Bakhuizen 1968; BOLDS 2014; Giesen et al. 2006; Joffry et al. 2012) 28 11 Ciri-ciri morfologi Melastoma candidum, Melastoma affine, dan
Melastoma saigonense (BOLDS 2014; Giesen et al. 2006; Gross 1993;
Motooka et al. 2003; Starr et al. 2003) 29
PENDAHULUAN
Indonesia sebagai negara megabiodiversitas memiliki berbagai macam sumber daya. Pendataan terhadap keragaman jenis tumbuhan di Indonesia harus terus dilakukan untuk memenuhi kebutuhan penelitian dan tujuan praktis di masa depan, serta untuk mengimbangi laju hilang keragaman tumbuhan. Tumbuhan di Indonesia yang memiliki banyak potensi diantaranya adalah Melastoma sp. Tumbuhan tersebut memiliki banyak manfaat, seperti antimikroba, antioksidan, antiinflamasi, antivirus, dan sitotoksisitas (Rajenderan 2010). Salah satu contoh spesies dari genus Melastoma adalah Melastoma malabathricum atau sinonimnya yaitu Melastoma candidum dan Melastoma affine (TPL 2012). Melastoma malabathricum tumbuh liar pada tempat-tempat yang mendapat cukup sinar matahari, semak belukar, atau di daerah objek wisata sebagai tanaman hias.
Melastoma malabathricum dapat tumbuh sampai ketinggian 1650 mdpl (Simanjuntak 2008). Melastoma malabathricum merupakan tumbuhan semak dengan tinggi batang 0.5-5 meter. Daun Melastoma malabathricum letaknya berhadapan berbentuk bulat telur, runcing, elips, atau elips lanset (Joffry et al. 2012). Pertulangan daun 3 dengan tulang daun yang melengkung (Backer dan Bakhuizen 1968).
Metode identifikasi spesies makhluk hidup telah berkembang dari identifikasi morfologi sampai pada identifikasi molekular berdasarkan potongan DNA pendek
yang disebut “barcode DNA” (Hebert et al. 2003). Menururt Vijayan dan Tsou (2010) Barcode DNA merupakan proses identifikasi spesies berdasarkan kera-gaman nukleotida. The Consortium for the Barcode of Life (CBOL) me-rekomendasikan penggunaan dua gen plastida yaitu ribulose-1,5-bisphosphate carboxylase/oxygenase (rbcL) dan maturase K (matK) sebagai barcode standar untuk tumbuhan (Bafeel et al. 2011). Gen rbcL merupakan bagian ribosom sub unit besar ribulose-1,5-bisphosphate carboxylase/oxygenase (Rubisco) untuk foto-sintesis (Zurawski et al. 1981). Gen rbcL banyak digunakan dalam studi filogentik (Chase et al. 2007). Menurut Hollingsworth et al. (2011) gen rbcL memiliki tingkat keberhasilan amplifikasi yang tinggi, tetapi resolusinya rendah dalam membedakan spesies yang berkerabat dekat, sedangkan gen matK sulit untuk diamplifikasi, tetapi memberikan resolusi yang tinggi dalam membedakan spesies yang berkerabat dekat. Menurut Kalangi et al. (2014) tingkat keakuratan gen matK lebih spesifik pada tingkat spesies, dibandingkan gen rbcL. Oleh karena itu, gen matK lebih banyak digunakan untuk berbagai penelitian dibandingkan gen rbcL. Gen matK merupakan gen pengkode enzim maturase bagian sub unit K yang terdapat dalam kloroplas tumbuhan. Gen matK memiliki panjang basa nukleotida sekitar 1500 pb. Gen matK
telah banyak digunakan dalam studi pengelompokkan dan cocok dalam mem-bedakan spesies antar tumbuhan (Vogel et al. 1997).
2
METODE
Waktu dan Tempat
Penelitian dilaksanakan pada bulan Februari sampai April 2015 di Laboratorium Biotechnology Research Indonesia-The Netherlands (BIORIN), Pusat Penelitian Sumberdaya Hayati dan Bioteknologi (PPSHB), Institut Pertanian Bogor.
Alat dan Bahan
Alat yang digunakan adalah mortar, penggerus, sentrifus merk BACKMAN dan Jouan BR4, timbangan merk GF-1200, tabung mikro merk Thermo Scientific, pipet mikro merk GILSON, Microwave merk the Genius 1200W, corong plastik, labu erlenmeyer, cetakan agar dengan sisirnya, bak elektroforesis, elektroforesis merk Mupid-exu, inkubator merk Multi-Blok Heater, oven merk BINDER, autoklaf,
vacuum merk Jouan RC10.09., PCR merk Applied Biosystem, Geldoc merk Labquip dan tip berbagai ukuran.
Bahan yang digunakan adalah 5 sampel daun Melastoma sp. yang berasal dari 13 plot yaitu plot 1: Cikabayan, plot 2: Gymnasium, plot 3: Gedung Olahraga Lama
(GOR Lama), plot 4: Leuwiliang, plot 5: Gunung Walat, plot 6: Fapet, plot 7: FKH, plot 8: Gerbang Utama IPB, plot 9: Asrama Putra IPB (Astra), plot 10: Pool Bus IPB, plot 11: Rumah Kaca PPSHB (Green House 2), plot 12: Rektorat, dan plot 13:
Jasinga Hijau. Bahan lain yang digunakan adalah nitrogen (N2) cair, cetyl trimethyl amonium bromide (CTAB) 2% (b/v), polivinil-pirolidon (PVP) 2% (b/v), β -merkaptoetanol 0.5% (v/v), Chloroform:Isoamylalcohol (C:I) (24:1), Phenol: Chloroform:Isoamylalcohol (P:C:I) (25:24:1), 2 M natrium asetat pH 5.2, etanol 100%, etanol 70%, ddH2O, RNAse 100 mg/ml, loading dye (blue/orange 6x), etidium bromida 1 mg/ ml, agarosa 1% (b/v), bufer Tris-Asetat EDTA (TAE) 1x, DNA cetakan 100 ng/µl, DNA ladder 10 ng/µl, PCR master mix (Fermentas), primer matK-3F, dan primer matK-3R.
Metode
Morfologi Daun Melastoma sp.
3
Isolasi DNA Melastoma sp.
DNA diekstrak dari potongan kecil lembaran daun tumbuhan Melastoma sp. menggunakan metode isolasi DNA Doyle dan Doyle (1990) dengan modifikasi PVP 2% (b/v) dan β-merkaptoetanol 0.5% (v/v). Sampel daun kering (≈ 0.15 gr) digerus dengan menggunakan nitrogen cair hingga halus. Setelah halus, sampel dimasukkan ke dalam tabung 1500 µl dan ditambahkan 600 µl CTAB 2% (b/v) dan PVP 2 % (b/v), serta ditambahkan 3 µl β-merkaptoetanol 0.5% (v/v). Sampel dihomogenkan dan diinkubasi pada suhu 60oC selama 30 menit. Setelah diinkubasi, sampel ditambahkan 600 µl C:I (24:1), kemudian sampel dihomogenkan dan disetrifugasi dengan kecepatan 10.000 rpm pada suhu 25oC selama 10 menit. Supernatan yang terbentuk dipindahkan ke dalam tabung baru dan ditambahkan P:C:I (25:24:1) sebanyak 1 kali volume sampel. Sampel dihomogenkan kembali dan disentrifugasi dengan kecepatan 10.000 rpm pada suhu 25oC selama 10 menit. Supernatan yang terbentuk dipindahkan ke dalam tabung baru dan ditambahkan 0.1 kali volume 2 M natrium asetat pH 5.2 dan ditambahkan etanol absolut (100%) sebanyak 2 kali volume sampel. Setelah itu, sampel disimpan semalam pada suhu -20oC. Sampel disentrifugasi kembali dengan kecepatan 10.000 rpm pada suhu 4oC selama 25 menit. Supernatan yang terbentuk dibuang, sedangkan peletnya ditambahkan 500 µl etanol 70%. Sampel disentrifugasi kembali dengan kecepatan 10.000 rpm pada suhu 4oC selama 10 menit. Supernatan yang terbentuk dibuang dan pelet dikeringkan menggunakan vacuum selama 15 menit, kemudin pelet ditambahkan ddH2O sebanyak ± 20 μl sampai 30 μl dan ditambahkan RNAse 100 mg/ml sebanyak 0.2 kali volume. Selanjutnya, sampel diinkubasi pada suhu 37oC selama 10 menit dan diinkubasi kembali pada suhu 70oC selama 10 menit. Setelah diinkubasi, sampel dilakukan uji kualitatif DNA menggunakan elektroforesis.
Elektroforesis DNA
DNA yang telah diisolasi kemudian dianalisis secara kualitatif menggunakan elektroforesis. Agarosa 1% (b/v) dimasukkan ke dalam labu erlenmeyer, kemudian, labu erlenmeyer ditutup dengan menggunakan plastik penutup. Setelah ditutup, labu erlenmeyer dipanaskan di dalam microwave selama 1 menit hingga agarose 1% (b/v) larut. Setelah larut, agarosa 1% (b/v) dimasukkan ke dalam cetakan dan didiamkan hingga mengeras, sehingga terbentuk gel agarosa 1% (b/v). Bak elektroforesis ditambahkan bufer TAE 1x dan gel agarosa 1% (b/v) dimasukkan ke dalam bak elektroforesis hingga terendam. Sebanyak 3 μl DNA ladder (10 ng/µl) dimasukkan ke dalam sumur pertama. Sebanyak 5 μl DNA (100 ng/μl) sampel yang telah diresuspensi dengan loading dye (blue/orange 6x) kemudian dimasukkan ke dalam sumur berikutnya. DNA dielektroforesis dalam gel agarosa 1% (b/v) pada tegangan listrik 100 volt selama 30 menit. Setelah dielektroforesis, gel agarosa 1% (b/v) diwarnai menggunakan etidium bromida 1 mg/ml selama 15 menit, kemudian gel agarosa 1% (b/v) dibilas menggunakan air dan gel agarosa 1% (b/v) dimasukkan ke dalam Geldoc untuk melihat pita DNA dengan menggunakan sinar ultraviolet (UV) pada panjang gelombang 260 nm.
Polymerase Chain Reaction (PCR)
4
Proses PCR dilakukan dengan cara: pradenaturasi DNA selama 5 menit pada suhu 94oC, kemudian dilanjutkan dengan siklus PCR sebanyak 35 kali yang terdiri atas denaturasi DNA pada suhu 94oC selama 30 detik, kemudian annealing pada suhu 50oC selama 1 menit dan proses pemanjangan pada suhu 72oC selama 2 menit. Proses PCR diakhiri dengan proses pemanjangan pada suhu 72oC selama 5 menit dan suhu 25oC selama 10 menit.
Analisis Urutan Nukleotida
Pita DNA hasil PCR divisualisasi menggunakan elektroforesis gel agarosa 1% (b/v) dan dikirim untuk sekuensing ke penyedia jasa sekuensing menggunakan primer yang sama dibaca dari dua arah (forward dan reverse). Selanjutnya, data urutan basa hasil sekuensing dianalisis menggunakan program Basic Local Alignment Search Tool (BLAST) pada situs GenBank National Center for Biotechnology Information (NCBI). Sebanyak 5 sampel dan 6 spesies pembanding berdasarkan gen matK disejajarkan menggunakan program ClustalW2 pada situs
European Molecular Biology Laboratory (EMBL) dan BioEdit.
HASIL DAN PEMBAHASAN
Morfologi Daun Melastoma sp.
Sebanyak 13 sampel daun Melastoma sp. (Gambar 1) diambil untuk di-identifikasi secara morfologi. Karakter morfologi sangat penting untuk mengenal identitas spesies (Zein dan Prawiradilaga 2013). Karakter yang digunakan untuk identifikasi adalah karakter vegetatif yaitu daun. Karakter awal yang digunakan untuk pengelompokkan sampel daun dilakukan dengan karakter jumlah tulang daun, pangkal daun, ujung daun, warna tangkai, dan daun berbulu atau tidak (Tabel 1). Sampel yang memiliki kesamaan dalam karakter dibuat ke dalam satu kelompok.
Hasil pengamatan sampel daun Melastoma sp. secara morfologi (Tabel 1) menunjukkan bahwa seluruh sampel mempunyai jumlah tulang daun 3. Kelompok 1 mempunyai ciri yaitu pangkal daun membundar dan ujung daun meruncing, sedangkan kelompok 2 mempunyai ciri yang sama dengan kelompok 1 kecuali ujung daunnya runcing. Kelompok 3 mempunyai ciri yaitu pangkal daun tumpul dan ujung daun runcing, sedangkan kelompok 4 dan kelompok 5 mempunyai ciri yang sama dengan kelompok 3 kecuali ujung daunnya meruncing. Seluruh sampel mempunyai tangkai berwarna merah dan daunnya berbulu, kecuali kelompok 5 warna tangkainya hijau dan daunnya berbulu. Secara keseluruhan, kelompok yang memiliki banyak perbedaan morfologi, yaitu kelompok 5. Masing-masing ke-lompok diambil 1 sampel untuk diidentifikasi morfologi bunga dan sampel daun diidentifikasi menggunakan DNA barkode. Sampel yang diambil dari kelompok 1 sampai kelompok 5 berturut-turut adalah sampel asal Cikabayan, Fapet, Pool Bus IPB, Rektorat, dan Jasinga Hijau.
5 fragmen DNA dalam satu untaian. Selain rekombinasi, yang menyebabkan variasi adalah mutasi. Mutasi merupakan perubahan struktur genetik (DNA maupun RNA) pada makhluk hidup yang menyebabkan terjadi perubahan fenotipe. Mutasi terjadi karena faktor luar, seperti perlakuan senyawa kimia atau perlakuan fisik, radiasi, atau terjadi secara alami, misalnya karena kesalahan dalam replikasi DNA (Yuwono 2005).
Gambar 1 Morfologi daun Melastoma sp. di Kampus IPB yang akan diiden- tifikasi; (a) Cikabayan, (b) Gymnasium, (c) GOR Lama, (d) Leuwiliang, (e) Gunung Walat, (f) Fapet, (g) FKH, (h) Gerbang Utama IPB, (i) Astra, (j) Pool Bus IPB, (k) Green House 2, (l)
6
Tabel 1 Morfologi daun Melastoma sp. di Kampus IPB yang telah diidentifikasi Kelompok Plot/
Gymnasium 3 Membundar Meruncing Merah Tidak
GOR Lama 3 Membundar Meruncing Merah Tidak
Leuwiliang 3 Membundar Meruncing Merah Tidak
Gunung Walat 3 Membundar Meruncing Merah Tidak
2
Sampel dari 5 kelompok, yaitu sampel asal Cikabayan, Fapet, Pool Bus IPB,
Rektorat, dan Jasinga Hijau diisolasi DNAnya dan di PCR. Hasil pita DNA dari PCR terlihat jelas dan terang, serta berukuran sekitar 750 pb untuk fragmen gen
matK sesuai dengan posisi primer yang digunakan (Gambar 2). Faktor-faktor yang menentukan keberhasilan PCR yaitu dNTP (deoksiribonukleotida triphospat), oligonukleotida primer, DNA cetakan, komposisi larutan bufer, jumlah siklus reaksi, enzim yang digunakan, pemisahan pekerjaan PCR dari reaksi yang lain, penyimpanan reagensia, pipet, kecermatan dalam teknik laboratorium, penggunan kontrol, dan sumber kontaminasi yang lain. Kontaminasi fragmen dalam jumlah sangat sedikit sekalipun dapat menyebabkan terjadi kesalahan yaitu dengan didapatkan produk amplifikasi yang tidak diinginkan (Yuwono 2006).
Gambar 2 PCR untuk fragmen gen matK; (M) marker 1 kb, (1) Cikabayan, (2) Fapet, (3) Pool Bus IPB, (4) Rektorat, dan (5) Jasinga Hijau
7
matK adalah primer matK-3F dan matK-3R yang berukuran 20 nukleotida. Menurut Yuwono (2006) primer yang umum digunakan untuk PCR berkisar antara 18 sampai 28 nukleotida, bergantung pada jenis primer dan komposisinya. Suhu
annealing yang digunakan dalam penelitian ini adalah 50oC. Menurut Handoyo dan Rudiretna (2001) suhu optimum annealing berkisar antara 37oC sampai 60oC.
Annealing adalah proses hibridisasi antara primer dengan urutan nukleotida (Bintang 2010). Primer akan menempel pada rantai DNA yang telah terpisah menjadi rantai tunggal selama proses annealing (Yuwono 2006).
Analisis Urutan Nukleotida Fragmen Gen matK
Gen matK merupakan gen kloroplas yang dikodekan dalam kelompok II intron maturase, sehingga gen matK berimplikasi pada pengolahan pasca-transkripsi. Gen matK yang digunakan sebagai gen penyandi protein memiliki model evolusi yang tidak biasa karena tingkat substitusi nukleotida gen matK tiga kali lebih tinggi dibandingkan gen rbcL dan tingkat substitusi asam amino enam kali lebih tinggi dibandingkan gen rbcL (Barthet dan Hilu 2007). Hasil dari pembacaan urutan nukleotida fragmen gen matK menghasilkan runutan DNA yang kemudian dianalisis menggunakan BLAST pada situs NCBI. BLAST merupakan teknik untuk menyelaraskan urutan nukleotida sampel dengan membandingkan urutan nuk-leotida yang terdapat di dalam database GeneBank. Program BLAST bertujuan mencari skor tertinggi penyejajaran urutan nukleotida yang tidak bercelah (ungapped alignment) dengan urutan nukleotida pembandingnya (Xiong 2006).
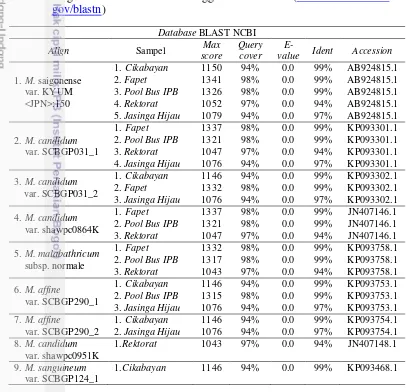
Hasil analisis urutan nukleotida masing-masing sampel, yaitu sampel asal
Cikabayan, Fapet, Pool Bus IPB, Rektorat, dan Jasinga Hijau dapat dilihat pada Tabel 2. Hal ini menunjukkan bahwa sampel asal Cikabayan, Fapet, Pool Bus IPB,
Rektorat, dan Jasinga Hijau memiliki tingkat homologi yang tinggi, identik, dan mempunyai kecocokan urutan nukleotida dengan align yang terdapat pada Tabel 2. Sampel asal Fapet dengan align M. saigonense var. KYUM<JPN>:150 memiliki nilai max score yang lebih tinggi dibandingkan dengan align M.candidum var. SCBGP031_1, M. candidum var. SCBGP031_2, M. candidum var. shawpc0864K, dan M. malabathricum subsp. normale. Hal ini menunjukkan bahwa sampel asal
Fapet lebih cenderung memiliki kesamaan urutan nukleotida dengan align M. saigonense var. KYUM<JPN>:150. Sebaliknya, sampel asal Cikabayan, Pool Bus IPB, Rektorat, dan Jasinga Hijau memiliki kesamaan urutan nukleotida berdasarkan max score dengan align M. candidum var. SCBGP031_1, M. candidum
var. SCBGP031_2, dan M. candidum var. shawpc0864K. M. candidum merupakan sinonim dari M. malabathricum (TPL 2012).
Nilai max score merupakan nilai kesamaan (identik) pasang basa antar sampel dengan spesies pembanding di GeneBank (Nugraha et al. 2014). Semakin tinggi nilai skor, maka semakin tinggi tingkat homologi kedua urutan nukleotida (Altschul
et al. 1990). Nilai query cover menunjukkan persentase sampel yang terpakai di dalam analisis BLASTn (Nugraha et al. 2014). Urutan nukleotida dikatakan identik apabila memiliki nilai minimal query cover sebesar 40% (Daniels et al. 2013). Nilai
8
0 menunjukkan bahwa kedua urutan nukleotida identik. Semakin tinggi nilai E-value maka semakin tinggi tingkat homologi antar nukleotida. Nilai E-value tinggi adalah 0, sedangkan nilai E-value rendah yaitu di bawah 0, misalnya nilai E-value
di bawah 0 adalah 1e-174. Menurut Altschul et al. (1990) nilai ident merupakan nilai tertinggi dari persentase identitas atau kecocokan antara urutan nukleotida
query dengan urutan nukleotida database yang disejajarkan. Semakin tinggi nilai
ident maka semakin cocok urutan nukleotida sampel dengan urutan nukleotida
database. Menurut Nugraha et al. (2014) accession merupakan nomor aksesi atau penomoran khusus bagi tiap record.
Tabel 2 Homologi tertinggi fragmen gen matK dari 5 sampel dengan memban-dingkan data di GeneBank menggunakan BLAST (www.ncbi.nlm.nih. gov/blastn)
9 mempunyai kesamaan morfologi dengan M.saigonense (Lampiran 11). Morfologi bunga Melastoma sp. dari ke-5 sampel yaitu sampel asal Cikabayan, Fapet, Pool Bus IPB, Rektorat, dan Jasinga Hijau menunjukkan bahwa ke-5 sampel mempunyai 5 petal yang berwarna ungu atau pink magenta, 5 sepal berwarna ungu, 1 putik berwarna ungu dengan kepala putik berwarna ungu, 10 benang sari yang terdiri atas 5 benang sari kecil berwarna kuning dan 5 benang sari besar berwarna ungu, serta terdapat hipantium (Tabel 3). Hal ini menunjukkan bahwa morfologi bunga sampel asal Cikabayan, Pool Bus IPB, Rektorat, dan Jasinga Hijau memiliki kesamaan dengan morfologi bunga M. malabathricum, sedangkan morfologi bunga sampel asal Fapet memiliki kesamaan dengan morfologi bunga M. saigonense
(Gambar 3).
Gambar 3 Morfologi bunga Melastoma sp. di Kampus IPB yang akan didentifikasi; (a) Cikabayan, (b) Fapet, (c) Pool Bus IPB, (d) Rektorat, dan (e) Jasinga Hijau
Tabel 3 Morfologi bunga Melastoma sp. di Kampus IPB yang telah diidentifikasi Kelompok Nama sampel Jumlah
petal
Jumlah sepal
Jumlah benang sari
Jumlah
putik Hipantium
1 Cikabayan 5 5 10 1 Ada
2 Fapet 5 5 10 1 Ada
3 Pool Bus IPB 5 5 10 1 Ada
4 Rektorat 5 5 10 1 Ada
5 Jasinga Hijau 5 5 10 1 Ada
10
Molekul tRNA membawa polipeptida yang dibentuk melalui proses pembentukan ikatan peptida antar asam amino (Lewin 2007). Urutan nukleotida yang berbeda akan berpengaruh terhadap asam amino yang dihasilkan, maka asam amino yang berbeda akan menghasilkan fenotipe yang berbeda pula pada suatu tumbuhan. Oleh karena itu, perwujudan fisik suatu karakter atau fenotipe pada tumbuhan di-pengaruhi oleh asam amino yang berbeda, sehingga menghasilkan morfologi yang berbeda antar tumbuhan yang satu dengan yang lainnya. Perbedaan asam amino ke-5 sampel ditunjukkan pada Gambar 4 untuk sampel asal Cikabayan, Gambar 5 untuk sampel asal Fapet, Gambar 6 untuk sampel asal Pool Bus IPB, Gambar 7 untuk sampel asal Rektorat, dan Gambar 8 untuk sampel asal Jasinga Hijau.
Keakuratan dalam DNA barkode dilakukan dengan cara penerjemahan urutan nukleotida menjadi asam amino menggunakan program ORF (Open Reading Frame) pada situs NCBI. ORF merupakan kumpulan ekson yang membentuk rangkaian nukleotida baru (Agoes dan Suryadi 2005). Urutan nukleotida sampel asal Cikabayan, Fapet, Pool Bus IPB, Rektorat, dan Jasinga Hijau dapat langsung diterjemahkan, karena fragmen gen matK terdapat di dalam kloroplas dan secara evolusi kloroplas berasal dari prokariot. Fragmen gen matK langsung ditranskrip-sikan dan diterjemahkan menjadi mRNA. Penerjemahan urutan nukleotida sampel asal Cikabayan, Fapet, dan Pool Bus IPB menjadi asam amino tidak menunjukkan keberadaan kodon stop ditengah urutan asam amino. Panjang asam amino yang dihasilkan dari ke-3 sampel yaitu sampel asal Cikabayan, Fapet, dan Pool Bus IPB
berturut-turut adalah 220 asam amino (Gambar 4), 245 asam amino (Gambar 5), dan 245 asam amino (Gambar 6). Penerjemahan urutan nukleotida sampel asal
Rektorat dan Jasinga Hijau menunjukkan terdapat kodon stop ditengah urutan asam amino. Panjang asam amino yang dihasilkan sampel asal Rektorat adalah 242 asam amino (Gambar 7). Panjang asam amino yang dihasilkan sampel asal Jasinga Hijau
adalah 233 asam amino (Gambar 8).
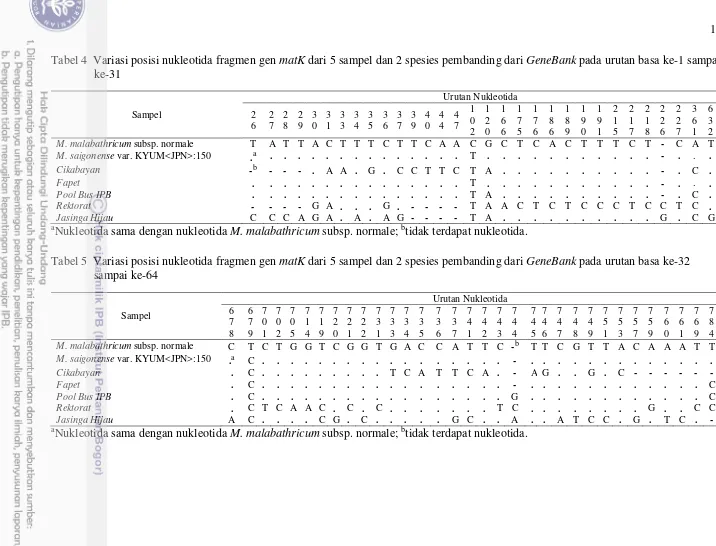
11 Tabel 4 Variasi posisi nukleotida fragmen gen matK dari 5 sampel dan 2 spesies pembanding dari GeneBank pada urutan basa ke-1 sampai
ke-31
aNukleotida sama dengan nukleotida M. malabathricum subsp. normale; btidak terdapat nukleotida.
Tabel 5 Variasi posisi nukleotida fragmen gen matK dari 5 sampel dan 2 spesies pembanding dari GeneBank pada urutan basa ke-32 sampai ke-64
12
20 ttggataggtttattcttccaaagaatggtttcttttcaaaaact L D R F I L P K N G F F S K T 65 aatccaagattcttcttattcatatataattttcatatatgtgaa
N P R F F L F I Y N F H I C E 110 tacgaatacatctttttttttctgcgtaatcaatcttttcattta
Y E Y I F F F L R N Q S F H L 155 tgttcacaattttctggattcttttttcaacgaatatattttttt
C S Q F S G F F F Q R I Y F F 200 cgaaaaatcaaaaatcttgtgaaagtgtttattcataatgaattt
R K I K N L V K V F I H N E F 245 caagacatcttggagttatgcaaagatccttttatgcattatgtt
Q D I L E L C K D P F M H Y V 290 agatatcaaggaaaattcattatttcttcaaataatacgcctatt
R Y Q G K F I I S S N N T P I 335 ctcattaataaatggaaatattattttcttcatttatggcaatat
L I N K W K Y Y F L H L W Q Y 380 cattattctatgtggtctcaacccgaaaggatcgacagaaatcaa
H Y S M W S Q P E R I D R N Q 425 ttatggaagcggtctcttaactttttgggttatcgttcaagagta
L W K R S L N F L G Y R S R V 470 ctaaccacttcttccgtggtacggagtcaaatgttagaaaattca
L T T S S V V R S Q M L E N S 515 tttctactcgataatgctatgccgaagctcgaaacacgagttaca
F L L D N A M P K L E T R V T 560 cttagtccaatgattgattcattgtctaaatcgaaattttgtaac
L S P M I D S L S K S K F C N 605 ccattaggacatcccattggtaagccggcctgggctgatttatcc
P L G H P I G K P A W A D L S 650 gattcggatattatctcattatcacagcggatc 682 D S D I I S L S Q R I
Gambar 4 Urutan nukleotida fragmen gen matK dari sampel asal Cikabayan dan deduksi asam amino
18 ttgcatttattacgtttctttctctatgagtattgtaattggaat L H L L R F F L Y E Y C N W N 63 aggtttattcttccaaagaatggtttcttttcaaaaactaatcca R F I L P K N G F F S K T N P 108 agattcttcttgttcatatataattttcatatatgtgaatacgaa R F F L F I Y N F H I C E Y E 153 tacatctttttttttctgcgtaatcaatcttttcatttatgttca Y I F F F L R N Q S F H L C S 198 caattttctggattcttttttcaacgaatatatttttttcgaaaa Q F S G F F F Q R I Y F F R K 243 atcaaaaatcttgtgaaagtgtttattcataatgaatttcaagac I K N L V K V F I H N E F Q D 288 atcttggagttatgcaaagatccttttatgcattatgttagatat I L E L C K D P F M H Y V R Y 333 caaggaaaattaattatttcttcaaataatacgcctattctcatt Q G K L I I S S N N T P I L I 378 aataaatggaaatattattttcttcatttatggcaatatcattat N K W K Y Y F L H L W Q Y H Y 423 tctatgtggtctcaacccgaaaggatcgacagaaatcaattatgg S M W S Q P E R I D R N Q L W 468 aagcggtctcttaactttttgggttatcgttcaagagtactaacc K R S L N F L G Y R S R V L T 513 acttcttccgtggtacggagtcaaatgttagaaaattcatttcta T S S V V R S Q M L E N S F L 558 ctcgataatgctatgccgaagctcgaaacacgagttacacttagt L D N A M P K L E T R V T L S 603 ccaatgattgattcattgtctaaatcgaaattttgtaacccatta P M I D S L S K S K F C N P L 648 ggacatcccattggtaagccggcctgggctgatttatccgattcg G H P I G K P A W A D L S D S 693 gatattatcgaccaatttcttcgtatatgccgaaatctttctcat D I I D Q F L R I C R N L S H 738 tatcacagcggatcaaa 754
Y H S G S
13 19 ttgcatttattacgtttctttctctatgagtattgtaattggaat
L H L L R F F L Y E Y C N W N 64 aggtttattcttccaaagaatggtttcttttcaaaaactaatcca R F I L P K N G F F S K T N P 109 agattcttcttattcatatataattttcatatatgtgaatacgaa R F F L F I Y N F H I C E Y E 154 tacatctttttttttctgcgtaatcaatcttttcatttatgttca Y I F F F L R N Q S F H L C S 199 caattttctggattcttttttcaacgaatatatttttttcgaaaa Q F S G F F F Q R I Y F F R K
244 atcaaaaatcttgtgaaagtgtttattcataatgaatttcaagac I K N L V K V F I H N E F Q D 289 atcttggagttatgcaaagatccttttatgcattatgttagatat I L E L C K D P F M H Y V R Y 334 caaggaaaattcattatttcttcaaataatacgcctattctcatt Q G K F I I S S N N T P I L I 379 aataaatggaaatattattttcttcatttatggcaatatcattat N K W K Y Y F L H L W Q Y H Y 424 tctatgtggtctcaacccgaaaggatcgacagaaatcaattatgg S M W S Q P E R I D R N Q L W 469 aagcggtctcttaactttttgggttatcgttcaagagtactaacc K R S L N F L G Y R S R V L T 514 acttcttccgtggtacggagtcaaatgttagaaaattcatttcta T S S V V R S Q M L E N S F L 559 ctcgataatgctatgccgaagctcgaaacacgagttacacttagt L D N A M P K L E T R V T L S 604 ccaatgattgattcattgtctaaatcgaaattttgtaacccatta P M I D S L S K S K F C N P L 649 ggacatcccattggtaagccggcctgggctgatttatccgattcg G H P I G K P A W A D L S D S 694 gatattatcgaccaatttcgttcgtatatgccgaaatctttctca D I I D Q F R S Y M P K S F S 739 ttatcacagcggatcaaa 756
L S Q R I K
Gambar 6 Urutan nukleotida fragmen gen matK dari sampel asal Pool Bus IPB dan deduksi asam amino
4 atgcctcttctttgcatgagtttgtattggataggtttattcttc
M P L L C M S L Y W I G L F F
49 caaagaatggtttcttttcaaaaactaatccaagattcttcttat
Q R M V S F Q K L I Q D S S Y
94 tcatacctataattttcatatatgtgaatacgaatacaatctatt
S Y L * F S Y M * I R I Q S I
139 ttttttcttgcgtaatccattccttctcaatttatgtttcacaat F F L A * S I P S Q F M F H N 184 tcttctgggattctttttttcaaccgaatatatttttttcgaaaa S S G I L F F N R I Y F F R K 229 atcaaaaatcttgtgaaagtgtttattcataatgaatttcaagac I K N L V K V F I H N E F Q D 274 atcttggagttatgcaaagatccttttatgcattatgttagatat I L E L C K D P F M H Y V R Y 319 caaggaaaattcattatttcttcaaataatacgcctattctcatt Q G K F I I S S N N T P I L I 364 aataaatggaaatattattttcttcatttatggcaatatcattat N K W K Y Y F L H L W Q Y H Y 409 tctatgtggtctcaacccgaaaggatcgacagaaatcaattatgg S M W S Q P E R I D R N Q L W 454 aagcggtctcttaactttttgggttatcgttcaagagtactaacc K R S L N F L G Y R S R V L T 499 acttcttccgtggtacggagtcaaatgttagaaaattcatttcta T S S V V R S Q M L E N S F L 544 ctcgataatgctatgccgaagctcgaaacacgagttacgacttag L D N A M P K L E T R V T T * 589 tccaatgattgattcattgtctaaatcgaaattttgtaacccaat S N D * F I V * I E I L * P N
634 taggacatcccattggataagcccggcctgggtctgaatttatcc * D I P L D K P G L G L N L S 679 aaattcccgatattaatccgaccaaattttcttcgtattatgccg K F P I L I R P N F L R I M P 724 gaatcactttctctcattatcacagcggatc 754
E S L S L I I T A D
14
1 aagatgcctcttctttgcattcccagagtatagtattggataggt K M P L L C I P R V * Y W I G 46 ttattcttccaaagaatggtttcttttcaaaactaatccaagatt L F F Q R M V S F Q N * S K I 91 cttcttattcatatataattttcaattatatgtgaatacgaatac L L I H I * F S I I C E Y E Y 136 atctttttttttctgcgtaatcaatcttttcatttatgttcacaa I F F F L R N Q S F H L C S Q 181 ttttctggattgcttttttcaacgaatatatcttttttcgaaaaa F S G L L F S T N I S F F E K 226 tcaaaaatcttgtgaaagtgtttattcataatgaatttcaagaca S K I L * K C L F I M N F K T 271 tcttggagttatgcaaagatccttttatgcattatgttagatatc S W S Y A K I L L C I M L D I 316 aaggaaaattcattatttcttcaaataatacgcctattctcatta K E N S L F L Q I I R L F S L 361 ataaatggaaatattatttttcttcatttatggcaatatcattat I N G N I I F L H L W Q Y H Y 406 tctatgtggtctcaacccgaaaggatcgacagaaatcaattatgg S M W S Q P E R I D R N Q L W 451 aagcggtctctttaactttttgggttatcgttcaagagtactaac K R S L * L F G L S F K S T N 496 cacttcttccgtggtacggagtcaaatgttagaaaattcatttct H F F R G T E S N V R K F I S 541 actcgataatgctatgccgaaagctcgaaacacgagttacactta T R * C Y A E S S K H E L H L 586 gtccaatgatggattcattgtctaaatccgaaattttgtaaccca V Q * W I H C L N P K F C N P 631 ttaggacatcacattggtaagccggcctgggctgatttatccgat L G H H I G K P A W A D L S D 676 cggcatatatcgaccgatctcattatcacagcggatc 712 R H I S T D L I I T A D
Gambar 8 Urutan nukleotida fragmen gen matK dari sampel asal Jasinga Hijau dan deduksi asam amino
SIMPULAN DAN SARAN
Simpulan
Hasil analisis menggunakan fragmen gen matK untuk Melastoma sp. menunjukkan bahwa sampel asal Cikabayan, Pool Bus IPB, Rektorat, dan Jasinga Hijau adalah Melastoma malabathricum, sedangkan sampel asal Fapet adalah
Melastomasaigonense.
Saran
Saran dari penelitian ini yaitu perlunya dilakukan penambahan jumlah sampel dan jumlah fragmen DNA barkode supaya nama spesies yang dihasilkan lebih tepat.
DAFTAR PUSTAKA
Agoes S, Suryadi. 2005. Simulasi identifikasi daerah coding pada deoxyribonucleic acid dengan menggunakan discrete fourier transform. JETri. 4(2):45-60. Lenght: 233 aa
Keterangan: *: kodon stop
15 Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. 1990. Basic local
alignment search tool. J Mol Biol. 215(3):403-410.
Backer CA, Bakhuizen VBJ. 1968. Flora of Java Volume 3. Netherlands (NL): Netherlands Organisation for the Advancement of Research.
Bafeel SO, Arif IA, Bakir MA, Khan HA, Al-Farhan AH, Al-Homaidan AA, Ahmed A, Jacob T. 2011. Comparative evaluation of PCR success with universal primers of maturase K (matK) and ribulose-1,5-bisphosphate carboxylase oxygenase large subunit (rbcL) for barcoding of some arid plants. Plant Omics. 4(4):195-198.
Barthet MM, Hilu KW. 2007. Expression of matK: functional and evolutionary implications. American Journal of Botany. 94(8):1402-1412.
Bintang M. 2010. Biokimia Teknik Penelitian. Jakarta (ID): Erlangga.
[BOLDS] Barcode of Life Data Systems. 2014. Taxonomy of Genus Melastoma
[internet]. [diunduh 2015 Ags 8]. Tersedia pada: http://www.boldsystems.org. [BOLDS] Barcode of Life Data Systems. 2014. Taxonomy of Genus Dissotis
[internet]. [diunduh 2015 Okt 10]. Tersedia pada: http://www.boldsystems.org. Chase MW, Cowan RS, Hollingsworth PM, van der Berg C, Madrinan S, Petersen G, Seberg O, Jorgsensen T, Cameron KM, Carine M et al. 2007. A proposal for a standardised protocol to barcode all land plants. Taxon. 56(2):295-299. Daniels NM, Gallant A, Peng J, Cowen LJ, Baym M, Berger B. 2013. Compressive
genomics for protein databases. Bioinformatics. 29:1283-1290.
Doyle JJ, Doyle JL. 1990. Isolation of plant DNA from fresh tissue. Focus. 12:13-15.
Handoyo D, Rudiretna A. 2001. General principles and implementation of polymerase chain reaction. Unitas. 9(1):17-29.
Hebert PDN, Cywinska A, Ball SL, deWaard JR. 2003. Biological identification through DNA barcodes. Proc. R. Soc Lond. 270: 313-321.
Hollingsworth PM, Forest LL, Spouge JL, Hajibabaei M, Ratnasingham S, van der Bank M, Chase MW, Cowan RS, Erickson DL, Fazekas AJ et al. 2009. A DNA barcode for land plants. Proc Natl Aca Sci USA. 106(31):12794-7.
Hollingsworth PM, Graham SW, Little DP. 2011. Choosing and using a plant DNA barcode. PloS One. 6(5):e9254.
Giesen W, Wulffraat S, Zieren M, Scholten L. 2006. Mangrove Guidebook for Southeast Asia. Bangkok (TH): Food and Agriculture Organization of the United Nations Regional Office for Asia and the Pacific.
Gross CL. 1993. The breeding system and pollinators of Melastoma affine
(Melastomatacae); a pioneer shrub in tropical Australia. Biotropica. 25(4):468-474.
Joffry SM, Yob NJ, Rofiee MS, Affandi MMRMM, Suhaili Z, Othman F, Akim, Desa MNM, Zakaria ZA. 2012. Melastoma malabathricum (L.) Smith ethno-medicinal uses, chemical constituents, and pharmalogical properties: a review [ulasan]. eCAM. 2012:48.
Kalangi C, Kamu VS, Kumaunang M. 2014. DNA tanaman Leilem (Clerodendrum minahassae L.) berdasarkan gen matK. MIPA UNSRAT. 3(2):108-109.
Lehninger AL. 1982. Principle of Biochemistry, Third Edition. New York (US): Worth Publisher Inc.
16
Motooka P, Castro L, Nelson D, Nagai G, Ching L. 2003. Weeds of Hawaii’s
Pastures and Natural Areas; An Identification and Management Guide. Hawaii (US): College of Tropical Agriculture and Human Resources, University of Hawaii.
Nugraha F, Roslim DI, Ardilla YP, Herman. 2014. Analysis of partial gene sequence Ferritine2 on Rice Plants (Oryza sativa L.) Indragiri Hilir, Riau.
Biosaintifika. 6(2):70-79.
Rajenderan MT. 2010. Ethno medicinal uses and antimicrobial properties of
Melastoma malabathricum. SEGi Review. 3(2):34-44.
Simanjuntak M. 2008. Ekstraksi dan fraksinasi komponen ekstrak daun tumbuhan senduduk (Melastoma malabathricum L.) serta pengujian efek sediaan krim terhadap penyembuhan luka bakar [skripsi]. Medan (ID): Universitas Sumatera Utara.
Starr F, Starr K, Loope L. 2003. Eradications of Potentially Invasive Plant Species. Hawaii (US): Maui Invasive Species Commite.
[TPL] The Plant List (2012). A Working List of All Plant Species. [internet]. [diunduh 2015 Okt 26]. Tersedia pada: http://www.theplantlist.org/tpl1.1/ record/tro-20302081.
Vijayan K, Tsou CH. 2010. DNA Barcoding in plants: taxonomy in a new perspective. Current Science. 99(11):1531-1541.
Vogel J, Hubschmann T, Borner T, Hess WR. 1997. Splicing and intron-internal RNA editing of trnK-matK transcripts in barley plastids: support for matK as an essential splice factor. J Mol Biol. 270(2):179.
Xiong J. 2006. Essential Bioinformatics. Cambridge (GB): Cambridge University Pr.
Yuwono T. 2005. Biologi Molekular. Jakarta (ID): Erlangga.
Yuwono T. 2006. Teori dan AplikasiPolymerase Chain Reaction. Yogyakarta (ID): Andi.
Zein MSA, Prawiradilaga DM. 2013. DNA Barcode Fauna Indonesia. Jakarta (ID): Kencana.
17
18
Lampiran 1 Diagram alir penelitian
Morfologi Daun Melastoma sp.
Isolasi DNA Melastoma sp.
Elektroforesis DNA
Polymerase Chain Reaction
(PCR)
19 Lampiran 2 Cara membuat larutan bufer TAE 50x dan TAE 1x
Komposisi : - Tris base 242 gr - Acetic acid glacial 57.1 ml - EDTA 0.5 M pH 8 100 ml
- Aquades 1000 ml
Cara membuat:
1. Buat larutan stok bufer TAE 50x dengan cara mencampurkan Tris base 242 gr, Acetic acid glacial 57.1 ml, dan EDTA 0.5 M pH 8 100 ml, kemudian tambahkan aquades hingga ± 700 ml.
2. Ukur pH smpai 8 (+ asam/basa). 3. Tambahkan aquades sampai 1000 ml. 4. Sterilisasi dengan autoklaf.
20
Lampiran 3 Komposisi larutan CTAB 2% Komposisi :
- 100 ml 1 M TRIS pH 7.5 - 140 ml 5 M NaCl
21 Lampiran 4 Hasil analisis BLAST menggunakan urutan nukleotida fragmen gen
22
Lampiran 5 Hasil analisis BLAST menggunakan urutan nukleotida fragmen gen
23 Lampiran 6 Hasil analisis BLAST menggunakan urutan nukleotida fragmen gen
24
Lampiran 7 Hasil analisis BLAST menggunakan urutan nukleotida fragmen gen
25 Lampiran 8 Hasil analisis BLAST menggunakan urutan nukleotida fragmen gen
26
27
M.malabathricum subsp.normale T-ATGCCGAAATC----TTTCTCATTATCACAGTGGATC--- 746 bp
M.saigonense var.KYUM<JPN>:150 T-ATGCCGAAATC----TTTCTCATTATCACAGTGGATC--- 746 bp
Cikabayan T-C--- 682 bp
Fapet T-ATGCCGAAATC----TTTCTCATTATCACAGCGGATCAAA 754 bp
Pool Bus IPB T-ATGCCGAAATC----TTTCTCATTATCACAGCGGATCAAA 756 bp
Rektorat TTATGCCGGAATCACTTTCTCTCATTATCACAGCGGATC--- 754 bp
28
Lampiran 10 Ciri-ciri morfologi Melastoma malabathricum (Backer dan Bakhuizen 1968; BOLDS 2014; Giesen et al. 2006; Joffry et al. 2012)
Melastoma malabathricum L. (Melastomataceae) merupakan salah satu dari 22 spesies yang ditemukan di kawasan Asia Tenggara. Menurut BOLDS (2014) M. malabathricum masuk ke dalam dunia Plantae, filum Magnoliophyta, kelas
Magnoliopsida, ordo Myrtales, famili Melastomataceae, dan genus Melastoma.
Melastoma malabathricum merupakan tumbuhan semak dengan tinggi batang 0.5 m sampai 1.5 m dan jarang tingginya hingga 3 m sampai 4 m (Giesen et al. 2006). Daun M. malabathricum tunggal, bertangkai, dan letaknya berhadapan. Bentuk daunnya adalah bulat telur, runcing, elips, atau elips lanset. Panjang daun 4 cm sampai 14 cm dan lebar daun 1.7 cm sampai 3.5 (sampai 6) cm. Panjang tangkai daun (petiole) 0.5 cm sampai 1.9 cm. Tekstur daun papery (kaku tipis), bagian abaksial strigose (halus) dan puberulous (ditutupi dengan rambut tegak dan halus), dan bagian adaksial strigose (halus) (Joffry et al. 2012). Pertulangan daun 3 dengan tulang daun yang melengkung (Backer dan Bakhuizen 1968). Pertulangan daun (venasi) sekunder berjumlah 2 atau 3 pada setiap sisi midvein (tulang daun utama). Pertulangan daun tersier sejajar (paralel), bentuk pangkal daun subcordate (bentuk antara jantung dan membundar), dan bentuk apeks atau ujung daun acuminate
(meruncing) (Joffry et al. 2012).
29
Lampiran 11 Ciri-ciri morfologi Melastoma candidum, Melastoma affine, dan Melastoma saigonense (BOLDS 2014; Giesen et al. 2006; Gross 1993; Motooka et al. 2003; Starr et al. 2003)
Menurut BOLDS (2014) Melastoma candidum masuk ke dalam dunia
Plantae, filum Magnoliophyta, kelas Magnoliopsida, ordo Myrtales, famili
Melastomataceae, dan genus Melastoma. Melastoma candidum merupakan tum-buhan semak dengan tinggi batang 1.5 m sampai 5 m (Starr et al. 2003). Daun M. candidum saling berhadapan (Motooka et al. 2003). Bentuk daun elips dan bulat telur. Panjang daun 4 cm sampai 11 cm dan lebar daun 1.3 cm sampai 4 cm (Starr
et al. 2003). Pertulangan daun 5 dengan tulang daun yang melengkung (Motooka
et al. 2003). Panjang tangkai daun (petiole) 5 mm sampai 12 mm. Pertulangan daun tersier sejajar (paralel), bentuk pangkal daun bulat, dan bentuk apeks atau ujung daun runcing. Bunga M. candidum mempunyai 5 mahkota berwarna ungu, 5 kelopak berwarna merah muda, 1 putik berwarna ungu, dan 10 benang sari yang terdiri atas 5 benang sari berwarna kuning dan 5 benang sari berwarna ungu. Hipantium M.candidum berwarna hijau (Starr et al. 2003).
Menurut BOLDS (2014) Melastoma affine masuk ke dalam dunia Plantae, filum Magnoliophyta, kelas Magnoliopsida, ordo Myrtales, famili Melastomata-ceae, dan genus Melastoma. Melastoma affine merupakan tumbuhan semak dengan tinggi batang 2 m sampai 7 m. Daun M. affine bulat telur dengan panjang daun 6 cm sampai 12 cm dan lebar daun 2 cm sampai 4 cm. Pertulangan daun 3 dengan tulang daun yang melengkung dan pertulangan daun (venasi) sekunder berjumlah 2 pada setiap sisi midvein (tulang daun utama). Panjang tangkai daun (petiole) 10 mm. Pertulangan daun tersier sejajar (paralel), bentuk pangkal daun subcordate, dan bentuk apeks atau ujung daun acuminate (meruncing). Bunga M.affine mempunyai 5 sampai 6 mahkota berwarna ungu, 5 kelopak berwarna ungu muda, 1 putik berwarna ungu, dan 10 benang sari yang terdiri atas 5 benang sari berwarna kuning dan 5 benang sari berwarna ungu. Hipantium M. affine berwarna hijau (Gross 1993).
Melastoma saigonense merupakan tumbuhan semak dengan tinggi batang 1 m sampai 3 m. Melastoma saigonense termasuk ke dalam famili Melastomataceae
30
RIWAYAT HIDUP
Penulis dilahirkan di Serang pada tanggal 11 Oktober 1993 dari pasangan Bapak Kamijan dan Ibu Rodiyah, S.Pd. Penulis merupakan anak pertama dari dua bersaudara. Penulis lulus dari SDN IX Kota Cilegon pada tahun 2005, kemudian lulus dari SMPN 1 Kramatwatu pada tahun 2008, dan lulus dari SMAN 1 Kramatwatu pada tahun 2011. Penulis diterima di jurusan Biologi Institut Pertanian Bogor pada tahun 2011 melalui jalur SNMPTN (Seleksi Nasional Masuk Perguruan Tinggi Negeri) Undangan.
Penulis aktif sebagai anggota Biosains Himabio (Himpunan Mahasiswa Biologi) sejak tahun 2012 sampai 2014. Penulis juga aktif dalam beberapa kepanitiaan seperti Maulid Nabi Muhammad SAW 1430 H, BIONIC (Biology On Science and Application) dan BOX (Biology On Experiment) 2013 dan 2014, LCTB (Lomba Cepat Tepat Biologi) – PSN (Pesta Sains Nasional) 2012 dan 2013, MGF (Mipa Go Field) 2013, Klinik PKM (Program Kreativitas Mahasiswa) 2013, MPF (Masa Perkenalan Fakultas) FMIPA 2013 dan 2014, MPD (Masa Perkenalan Departemen) – Morfologi 49 tahun 2013, dan Himabio Go Field 2013. Penulis menjadi asisten praktikum Biologi Dasar pada tahun ajaran 2013/2014 sampai tahun ajaran 2015/2016. Penulis menjadi asisten praktikum Genetika Molekular pada tahun ajaran 2014/2015.
Penulis pernah mengikuti PKM (Program Kreativitas Mahasiswa) dibidang
kewirausahaan tahun 2012 yang berjudul “MORENPO: Inovasi Bakpao Berbahan Dasar Kimpul (Xanthosoma sagittifolium) dengan Varian Isi Keong Tutut (Belamnya javanica Van Den Bush) sebagai Alternatif Jajanan yang Kaya Gizi, Sehat, dan Murah”. Penulis pernah mengikuti PKM (Program Kreativitas
Mahasiswa) dibidang penelitian tahun 2013 yang berjudul “Efektivits Ekstrak
Trichoderma sp. Daun Mahkota Dewa (Phaleria marcocarpa) sebagai Anti Kanker Payudara”. Penulis telah melakukan studi lapangan di Taman Wisata Alam Telaga Warna, Puncak, Bogor, Jawa Barat pada tahun 2013 mengenai “Keragaman Ordo
Lepidoptera di Kawasan Taman Wisata Alam Telaga Warna dan Sekitarnya”.