BAB VII
PENGEMBANGAN MARKA SNAP BERBASIS
RESISTANCE GENE ANALOGUE (RGA) DAN DEFENSE
GENE ANALOGUE (DGA) UNTUK KETAHANAN TERHADAP
LAYU FUSARIUM PADA TANAMAN PISANG (Musa spp.)
Abstrak
Pengembangan kultivar pisang tahan penyakit secara konvensional menghadapi kendala lamanya waktu yang diperlukan untuk seleksi dan evaluasi tanaman hasil persilangan, oleh karena itu pendekatan bioteknologi melalui pengembangan marka molekuler merupakan salah metode alternatif yang dapat digunakan. Berdasarkan substitusi basa nukleotida (SNP) yang diidentifikasi pada fragmen resistance gene analogue (RGA), β-1,3-glucanase dan chitinase asal tanaman pisang, dikembangkan marka SNAP untuk seleksi ketahanan tanaman pisang terhadap penyakit. Tahapan penelitian meliputi (1) penentuan situs SNP asal gen MNBS, β-1,3-glucanase dan chitinase untuk pembuatan primer SNAP, (2) pembuatan primer spesifik alel, (3) pengujian efektifitas primer SNAP dalam mengamplifikasi produk PCR pada DNA genom pisang, (4) pengujian dan pemilihan primer SNAP untuk marka ketahanan terhadap FOC pada 10 kultivar referensi dan beberapa kultivar/aksesi pisang lainnya. Primer SNAP dibuat berdasarkan 14 sekuen MNBS, 4 sekuen β-1,3-glucanase dan 8 sekuen chitinase. Berdasarkan fragmen gen MNBS dihasilkan sebanyak 14 pasang primer SNAP dan 10 diantaranya terbukti efektif mengamplifikasi produk PCR pada kultivar Klutuk Wulung dan Barangan, sedangkan dari fragmen gen β-1,3-glucanase diperoleh 6 pasang primer dan 22 pasang primer dihasilkan dari fragmen gen chitinase. Semua pasangan primer asal fragmen gen β-1,3-glucanase dan chitinase telah terbukti efektif mengamplifikasi produk PCR. Berdasarkan hasil pengujian menggunakan kultivar referensi terpilih 2 pasang primer berdasarkan fragmen gen MNBS, 2 pasang primer berdasarkan fragmen gen β-1,3-glucanase dan 10 pasang primer berdasarkan fragmen gen chitinase, yang digunakan sebagai marka SNAP untuk ketahanan terhadap layu FOC. Marka SNAP tersebut berhasil mengelompokkan kultivar pisang berdasarkan karakter ketahanan terhadap FOC. Hasil penelitian ini merupakan langkah awal dalam pembuatan marka SNAP berdasarkan gen yang bertanggung jawab pada ketahanan dan pertahanan terhadap penyakit tanaman pisang.
Kata kunci: Pisang, SNAP, marka molekuler, RGA, DGA, ketahanan penyakit
1
Bagian bab ini telah diterbitkan di Jurnal Hortikultura Vol. 23 No. 4 dengan judul: Pengembangan Marka SNAP Berbasis Resistance Geneanalogue (RGA) pada Tanaman Pisang (Musa spp.)
106
THE DEVELOPMENT OF SNAP MARKER FOR FUSARIUM
WILT RESISTANCE BASED ON RESISTANCE GENE
ANALOGUE (RGA) AND DEFENSE GENE ANALOGUE (DGA)
ON BANANA (Musa spp.)
Abstract
The development of resistance banana cultivars through conventional breeding methods encounters some obstacles, because breeding program require a long time for selecting and evaluating of the new hybrids. Therefore, the support of biotechnology approaches and molecular markers in banana breeding will be beneficial. SNAP markers for banana disease resistance were developed based on SNPs identified from RGAs, β-1,3-glucanase dan chitinase genes isolated from banana. The activities include (1) the identification of putative SNPs from RGAs, β-1,3-glucanase dan chitinase genes for generating of SNAP primers, (2) allel specific primer design, (3) the evaluation of effectivity of primers on PCR reactions, (4) selection and evaluation of SNAP primers for FOC resistance marker using 12 banana cultivars. SNAP primers were generated based on 14 MNBS, 4 β-1,3-glucanase dan 8 chitinase sequences. Fourteen allelic specific primers were obtained from MNBS sequences and 10 of them were able to amplified PCR products from genomic DNA of banana cv. Klutuk Wulung and Barangan. Six allelic primers from β-1,3-glucanase gene and 22 primers from chitinase gene were effectively able to amplified PCR products from genomic DNA of the same banana cultivars. Two primers pairs based on MNBS sequences, 2 primers based on β-1,3-glucanase gene sequences, and 10 primers based on chitinase gene sequences were selected for SNAP marker for FOC resistance. This study was the initial effort of SNAP marker development using RGAs and defense genes and potentially can be used for banana disease resistance markers in general.
Keywords: Banana, SNAP, molecular marker, RGA, DGA, disease resistance
1Part of this chapter has been published in the Jurnal Hortikultura Vol. 23 No. 4, entitled: Pengembangan
107
Pendahuluan
Marka molekuler seperti RAPD (Howell et al.1994), RFLP (Faure et al. 1993), SSR (Crouch et al. 1998), ISSR (Venkatachalam et al. 2008) dan AFLP (Ude et al. 2002) telah digunakan sebagai alat untuk analisis genetik tanaman pisang. Marka RAPD dan SCAR telah digunakan sebagai indikator seleksi secara tidak langsung untuk ketahanan pisang terhadap penyakit bercak daun sigatoka (Nwauzoma et al. 2011) dan layu Fusarium (Wang et al. 2012). Single nucleotide polymorphism (SNP) adalah marka molekular generasi baru yang telah dikembangkan dan digunakan pada kedokteran, ternak dan tanaman (Gupta et al. 2001).
Marka molekuler berbasis SNP telah mulai banyak digunakan untuk studi genotipe (Till et al. 2010), pembuatan peta tautan genetik beresolusi tinggi (Drenkard et al. 2000; Rabbi et al. 2012), identifikasi varietas ginseng (Sun et al. 2011), penentuan spesies dari kerabat jeruk (Jiang et al. 2010), identifikasi Canabis yang menghasilkan produk narkotika atau tidak (Rotherham & Harbison 2011), dan seleksi untuk karakter tertentu (MAS) dalam proses pemuliaan tanaman (Gupta et al. 2001).
Metode identifikasi SNP yang masih sering digunakan untuk skala kecil adalah Cleaved Amplified Polymorphic Sequence (CAPS), derived CAPS (dCAPS) (Thiel et al. 2004; Nasu et al. 2002) dan Allel Specific-PCR (AS-PCR) atau Single Nucleotide Amplified Polymorphism (SNAP) (Drenkard et al. 2000; Liu et al. 2012). Dalam aplikasinya marka CAPS dan dCAPS mempunyai keterbatasan yaitu memerlukan enzim nuclease sehingga memerlukan biaya yang masih relatif tinggi, sedangkan marka AS-PCR atau SNAP hanya memerlukan pasangan primer dan teknik PCR dan elektroforesis gel agarose standar (Drenkard et al. 2000).
Salah satu contoh pengembangan marka SNAP untuk marka yang berhubungan dengan karakter aroma dan supernodulasi pada tanaman kedelai (Kim et al. 2005; Juwattanasomran et al. 2011), sehingga proses seleksi yang berhubungan dengan kedua karakter tersebut bisa lebih cepat dan efisien.
Secara umum penelitian yang dilakukan berusaha mengembangkan marka SNAP untuk seleksi tidak langsung sifat ketahanan tanaman pisang terhadap penyakit. Secara khusus penelitian yang dilakukan bertujuan untuk: (1) menentukan situs SNP non synonymous pada RGA dan DGA asal tanaman pisang untuk pengembangan marka SNAP, (2) mendisain pasangan primer spesifik alel untuk menghasilkan marka SNAP pada situs-situs SNP yang teridentifikasi, (3) mengevaluasi efektivitas pasangan primer yang didapat untuk menghasilkan marka SNAP, dan (4) mengevaluasi marka SNAP berasis RGA dan DGA sebagai marka ketahanan terhadap FOC pada beberapa kultivar pisang.
Bahan dan Metode Waktu dan Tempat
Kegiatan penelitian dilaksanakan mulai bulan November 2012 sampai Juli 2013, di laboratorium Pemuliaan dan Biologi Molekular Tanaman, Program Studi Agronomi dan Hortikultura, Fakultas Pertanian, Institut Pertanian Bogor.
108
Bahan Penelitian dan Isolasi DNA
Pengujian efektivitas primer SNAP dilakukan menggunakan DNA genom yang diisolasi dari daun pisang cv. Klutuk Wulung (BB) dan Barangan (AAA). Penentuan primer SNAP untuk marka ketahanan terhadap penyakit layu FOC, digunakan DNA genom asal kultivar pisang yang mempunyai ketahanan terhadap FOC yang bervariasi sebagai kultivar referensi. Sebagian kultivar yang digunakan sebagai referensi adalah kultivar yang telah diuji ketahanannya terhadap penyakit layu FOC pada Percobaan 1, dan sebagian lagi berdasarkan informasi hasil penelitian sebelumnya (Molina et al. 2010). Untuk tanaman sangat rentan sampai rentan digunakan kultivar Barangan, Ambon Kuning dan Ambon Hijau. Kultivar yang mewakili rentan sampai tahan adalah Ketan, Kepok, Jawaka dan Awak, sedangkan yang mewakili kultivar tahan sampai sangat tahan adalah Calcuta, Rejang dan Klutuk Wulung.
Pengujian marka SNAP selanjutnya dilakukan pada 10 kultivar/aksesi yang belum diketahui ketahanannya, yang mewakili spesies liar M. acuminata: HlbS, Slk-29 dan yang kultivar, yaitu: MDO-01, MDO-04, MLU-07, SUM-01, SUM-04, HyKC-01 dan Muli, serta seksi Australimusa AMB-01.
Bahan tanaman yang digunakan adalah daun muda dan isolasi DNA berdasarkan metode CTAB (Doyle & Doyle 1987) yang dimodifikasi oleh Das et al. (2009). Selanjutnya stok DNA disimpan dalam lemari pendingin bersuhu -20 °C sampai siap digunakan.
Identifikasi Situs SNP yang Non-synonymous
Diantara sejumlah situs SNP yang teridentifikasi, dipilih situs SNP pada coding region yang menyebabkan terjadinya perubahan residu asam amino atau disebut synonymous. Identifikasi situs SNP yang non-synonymous berbasis RGA dilakukan pada sekuen DNA dari 14 fragmen RGA asal tanaman pisang (MNBS) yang telah diisolasi dari Percobaan 2, sedangkan SNP berbasis DGA dilakukan pada sekuen chitinase hasil dari Percobaan 3, dan sekuen β-1,3-glucanase hasil dari Percobaan 4. Identifikasi situs SNP dengan melakukan pensejajaran sekuen DNA dan prediksi asam amino dari menggunakan perangkat lunak Geneious Pro 5.6.6 versi percobaan (Biomatters, USA).
Disain Primer Spesifik Alel untuk Marka SNAP
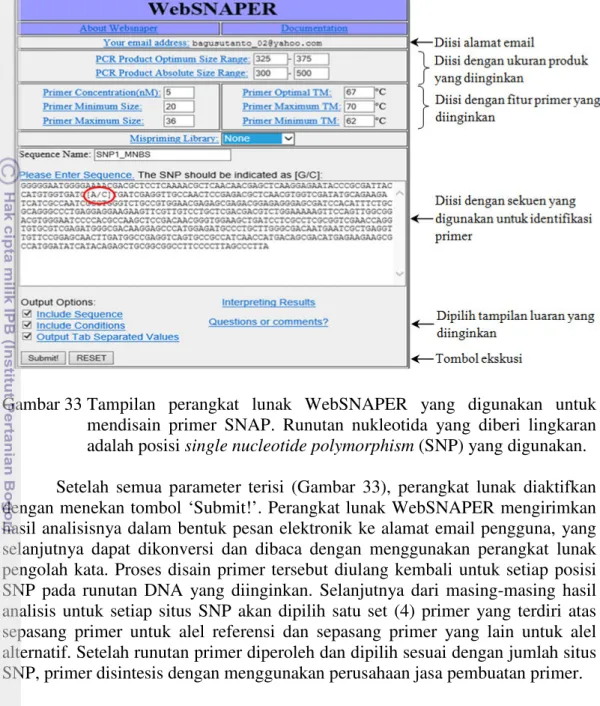
Pasangan primer SNAP didisain berdasarkan situs SNP terpilih dengan menggunakan perangkat lunak WebSNAPER yang tersedia di situs http://ausubellab.mgh.harvard.edu/. Ketika mengakses situs tersebut, pada layar WebSNAPER dipilih menu ‘SNAP Program’. Pengguna program yang baru pertama kali mengakses harus melakukan ‘Sign in’ dan memasukkan alamat email pengguna pada tempat yang tersedia. Setelah ‘Sign in’, muncul kotak dialog (Gambar 1) untuk memasukkan runutan nukleotida dengan situs SNP target yang dievaluasi. Situs SNP target ditandai sebagai [X/Y], dimana X adalah nukleotida untuk alel referensi dan Y adalah nukleotida untuk alel alternatif. Beberapa parameter yang perlu diisi/disesuaikan adalah ukuran produk amplifikasi PCR, suhu TM dan nama primer yang diinginkan. Screen shot perangkat lunak WebSNAPER dan sejumlah isian yang perlu diisi terkait dengan disain marker SNAP disajikan pada Gambar 33.
109
Gambar 33 Tampilan perangkat lunak WebSNAPER yang digunakan untuk mendisain primer SNAP. Runutan nukleotida yang diberi lingkaran adalah posisi single nucleotide polymorphism (SNP) yang digunakan. Setelah semua parameter terisi (Gambar 33), perangkat lunak diaktifkan dengan menekan tombol ‘Submit!’. Perangkat lunak WebSNAPER mengirimkan hasil analisisnya dalam bentuk pesan elektronik ke alamat email pengguna, yang selanjutnya dapat dikonversi dan dibaca dengan menggunakan perangkat lunak pengolah kata. Proses disain primer tersebut diulang kembali untuk setiap posisi SNP pada runutan DNA yang diinginkan. Selanjutnya dari masing-masing hasil analisis untuk setiap situs SNP akan dipilih satu set (4) primer yang terdiri atas sepasang primer untuk alel referensi dan sepasang primer yang lain untuk alel alternatif. Setelah runutan primer diperoleh dan dipilih sesuai dengan jumlah situs SNP, primer disintesis dengan menggunakan perusahaan jasa pembuatan primer. Evaluasi Efektivitas Primer SNAP
Untuk menentukan posisi SNP mana saja yang dapat digunakan untuk menghasilkan marka SNAP, setelah disain primer dan pemesanan primer diselesaikan, primer SNAP yang didapat diuji kemampuannya untuk menghasilkan produk amplifikasi dengan menggunakan cetakan DNA genom pisang. Dalam penelitian ini, pengujian efektivitas primer SNAP dilakukan dengan menggunakan DNA genom pisang cv. Klutuk Wulung dan Barangan.
Reaksi PCR dilaksanakan dengan volume 25 µl menggunakan KAPA2GTM
PCR Kit (Kapa Biosystems Inc., USA), yang komposisinya terdiri atas 5.0 µl 5 X buffer PCR (di dalamnya terkandung 1.5 mM Mg2+), 0.5 µl MgCl2 25 mM, 0.5 µl
dNTPs 10 mM, 1.0 µl masing-masing primer dengan konsentrasi 10 µM (primer forward dan reverse), 30 ng DNA genom, 0.1 µl Taq DNA polymerase (5 U µl-1) dan 15.4 µl ddH2O. Proses PCR menggunakan mesin GeneAmp® PCR System
110
2400 (Perkin Elmer). Denaturasi cetakan DNA pada awal reaksi pada suhu 95 °C selama 3 menit, diikuti dengan 35 kali siklus 95 °C selama 10 detik, 60 sampai 62 °C selama 10 detik dan 72 °C selama 3 detik, dan diakhiri dengan satu siklus 72 °C selama 10 menit. Produk PCR dipisahkan berdasarkan ukuran dengan menggunakan elektroforesis gel agarose 1 % pada mesin elektroforesis dengan tegangan 80 V selama 25 menit.
Analisis Data
Berdasarkan hasil elektroferogram, data keberadaan pita amplifikasi dikonversi menjadi data binary untuk membuat data genotipe. Bila dalam satu lokus hanya primer reference yang menghasilkan produk amplifikasi, disandi dengan ‘1-0-1-0’, bila hanya primer alternate yang menghasilkan produk, disandi dengan ‘0-1-0-1’, sedangkan bila kedua primer menghasilkan produk disandi dengan ‘1-0-0-1’. Selanjutnya dari data genotipe yang diperoleh, dilakukan analisis filogenetik menggunakan perangkat lunak NTSYS ver 2.02 (Exeter Software, New York, USA).
Hasil dan Pembahasan
Single nucleotide polymorphism (SNP) adalah polimorfisme yang disebabkan oleh proses substitusi satu basa pada nukleotida dalam genom tanaman (Syvänen 2001). Keberadaan SNP diketahui menyebar di seluruh bagian dari genom tanaman sehingga berpotensi untuk digunakan sebagai marka molekuler (Gupta et al. 2001).
Identifikasi Situs SNP yang Non-synonymous
SNP berbasis sekuen RGA. Fragmen MNBS yang digunakan dalam penelitian ini berukuran 524 pasang basa (pb). Seluruh runutan nukleotida MNBS tersebut merupakan bagian exon dan tidak terdapat intron di dalamnya. Fragmen MNBS menyandi 174 asam amino yang merupakan bagian dari domain motif p-loop sampai dengan GLPL. Berdasarkan hasil pensejajaran runutan DNA diantara 14 MNBS, berhasil diidentifikasi keberadaan 30 situs SNP.
Nukleotida pada urutan nomor 215 merupakan situs SNP dengan varian nukleotida [G/A]. Pada posisi tersebut, keberadaan SNP menyebabkan terjadinya substitusi residu asam amino dari arginin (R) menjadi lisin (K) (Gambar 34). Situs SNP seperti pada posisi 215 yang dipilih untuk pengembangan marka SNAP dalam tahap-tahap penelitian selanjutnya.
Dari 30 situs SNP yang berhasil diidentifikasi pada fragmen MNBS, diperoleh 20 situs SNP yang menyebabkan terjadinya substitusi asam amino. Selanjutnya, 20 situs SNP tersebut dipilih untuk mendisain pasangan primer SNAP. Meskipun tergolong menyebabkan substitusi residu asam amino, belum tentu dari setiap situs SNP yang terpilih dapat didisain pasangan primer SNAP, karena tidak semua situs bisa menghasilkan pasangan primer. Kegagalan mendisain primer dari situs yang teridentifikasi antara lain disebabkan oleh kurangnya jumlah basa di sekitar situs SNP terutama untuk situs SNP ada di ujung 5’ ataupun 3’ runutan nukleotida fragmen DNA yang digunakan atau karena komposisi nukleotida di sekitar target SNP tidak dapat memenuhi kriteria yang ditentukan di awal proses disain primer.
111
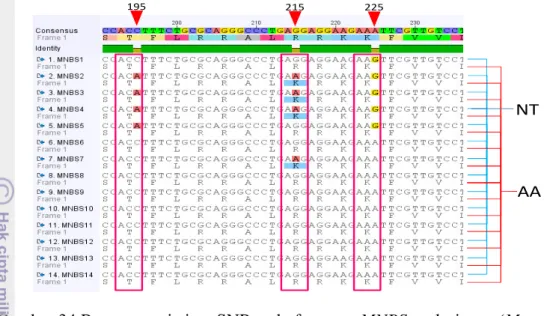
Gambar 34 Representasi situs SNP pada fragmen MNBS asal pisang (Musa spp.). Situs SNP 195 dan 225 tidak menyebabkan terjadinya substitusi asam amino sedangkan situs SNP 215 merubah residu asam amino arginin menjadi lisin. NT: runutan nukleotida DNA dan AA: runutan asam amino yang diprediksi dari runutan NT, segitiga merah: posisi SNP.
Gambar 35 Representasi situs SNP pada fragmen MNBS. Situs SNP dengan latar belakang kuning adalah situs SNP yang menyebabkan terjadinya substitusi asam amino sedangkan situs SNP di dalam kotak merah adalah situs SNP terpilih untuk pembuatan primer SNAP. Nomor di atas runutan DNA mengindikasikan posisi nukleotida dalam fragmen MNBS. EU855841 dan EU239821 adalah RGA asal Musa yang terdeposit dalam GenBank NCBI.
Untuk mendisain primer, di sekitar situs SNP disyaratkan keberadaan sebanyak minimal 25 nukleotida. Selain itu untuk mengkonfirmasi apakah situs SNP teridentifikasi benar-benar SNP atau kesalahan proses sekuensing (sequencing error), dalam pemilihan SNP perlu dilibatkan juga aksesi dari gen serupa yang diambil dari GenBank. Untuk mendisain primer spesifik alel, dari alternatif 20 situs SNP pada
195 215 225
NT
112
fragmen MNBS, hanya 8 situs SNP saja yang dipilih (Gambar 35).
SNP berbasis sekuen β-1,3-glucanase. Analisis pensejajaran 4 sekuen MaGlu yang berukuran 788 pb yang diperoleh dalam penelitian ini menemukan adanya 8 SNP, tetapi hanya 4 SNP yang menyebabkan perubahan asam amino atau non-synonymous SNP, yaitu nukleotida pada urutan nomor 91, 538, 677 dan 778. Keberadaan situs SNP ditampilkan pada Gambar 36.
SNP berbasis sekuen gen chitinase. Berdasarkan analisis pensejajaran 8 sekuen gen MaChi yang berukuran 596 pb yang diperoleh dalam penelitian ini ditemukan sebanyak 22 situs SNP pada coding region dan 6 situs SNP pada non-coding region. Namun demikian hanya 14 situs SNP yang menyebabkan substitusi 11 residu asam amino (Gambar 37), dan 11 situs di antaranya digunakan untuk mendisain primer SNAP.
Gambar 36 Keberadaan situs SNP pada fragmen MaGlu asal pisang (Musa spp.). Situs SNP 30, 480, 618 dan 753 tidak menyebabkan terjadinya substitusi residu asam amino sedangkan situs SNP 91, 538, 677 dan 778 yang dapat merubah residu asam amino (segitiga merah). NT: runutan nukleotida DNA dan AA: runutan asam amino yang diprediksi dari runutan NT. Huruf di atas segitiga merah adalah varian nukleotida pada situs non-synonymous SNP.
Gambar 37 Representasi situs SNP pada fragmen gen MaChi dari 5 kultivar pisang. MaChi-Rjg asal Rejang, MaChi_Klt#1 & #2 asal Klutuk Wulung, MaChi_Kpk#1 & #2 asal Kepok, MaChi_AH#1 & #2 asal Ambon Hijau, dan MaChi_Br asal Barangan. Tanda bintang di bawah sekuen adalah posisi SNP, dan angka di atas sekuen adalah posisi basa nukleotida dalam fragmen MaChi asal DNA genom. Basa nukleotida dalam kotak adalah SNP yang digunakan untuk mengembangkan marka SNAP.
[G/C] [A/C] [A/G] [C/A]
MaGlu_Klt : MaGlu_Rjg: MaGlu_AH :
MaGlu_Br : AA
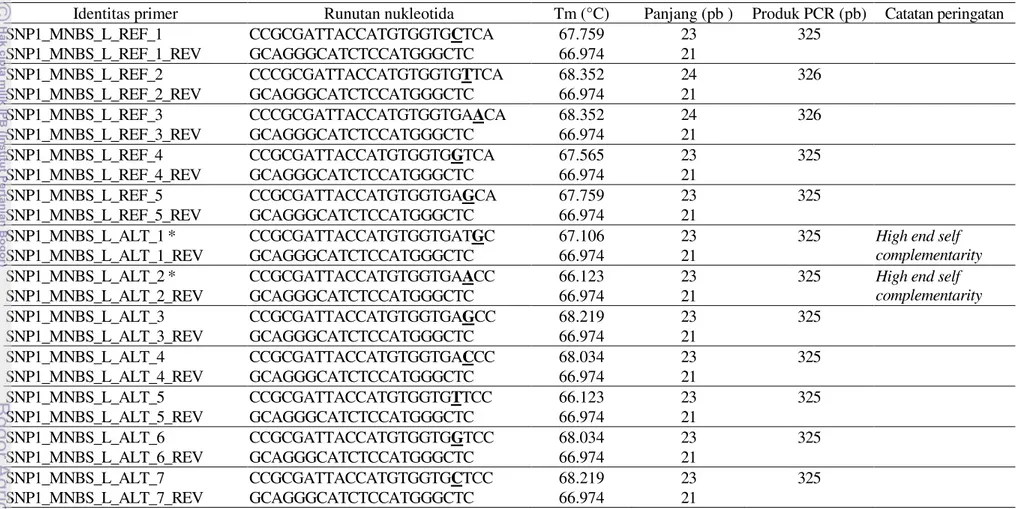
Tabel 22 Primer alternatif sebagai luaran yang didapat dari proses mendisain primer dengan menggunakan WebSNAPER untuk situs SNP1 dari fragmen gen MNBS
Identitas primer Runutan nukleotida
SNP1_MNBS_L_REF_1 CCGCGATTACCATGTGGTG SNP1_MNBS_L_REF_1_REV GCAGGGCATCTCCATGGGCTC SNP1_MNBS_L_REF_2 CCCGCGATTACCATGTGGT SNP1_MNBS_L_REF_2_REV GCAGGGCATCTCCATGGGCTC SNP1_MNBS_L_REF_3 CCCGCGATTACCATGTGGTGA SNP1_MNBS_L_REF_3_REV GCAGGGCATCTCCATGGGCTC SNP1_MNBS_L_REF_4 CCGCGATTACCATGTGGTG SNP1_MNBS_L_REF_4_REV GCAGGGCATCTCCATGGGCTC SNP1_MNBS_L_REF_5 CCGCGATTACCATGTGGTGA SNP1_MNBS_L_REF_5_REV GCAGGGCATCTCCATGGGCTC SNP1_MNBS_L_ALT_1 * CCGCGATTACCATGTGGTGAT SNP1_MNBS_L_ALT_1_REV GCAGGGCATCTCCATGGGCTC SNP1_MNBS_L_ALT_2 * CCGCGATTACCATGTGGTGA SNP1_MNBS_L_ALT_2_REV GCAGGGCATCTCCATGGGCTC SNP1_MNBS_L_ALT_3 CCGCGATTACCATGTGGTGA SNP1_MNBS_L_ALT_3_REV GCAGGGCATCTCCATGGGCTC SNP1_MNBS_L_ALT_4 CCGCGATTACCATGTGGTGA SNP1_MNBS_L_ALT_4_REV GCAGGGCATCTCCATGGGCTC SNP1_MNBS_L_ALT_5 CCGCGATTACCATGTGGTG SNP1_MNBS_L_ALT_5_REV GCAGGGCATCTCCATGGGCTC SNP1_MNBS_L_ALT_6 CCGCGATTACCATGTGGTG SNP1_MNBS_L_ALT_6_REV GCAGGGCATCTCCATGGGCTC SNP1_MNBS_L_ALT_7 CCGCGATTACCATGTGGTG SNP1_MNBS_L_ALT_7_REV GCAGGGCATCTCCATGGGCTC
Keterangan: nukleotida yang diberi garis bawah pada primer marka SNAP karena “high end self complementarity
Primer alternatif sebagai luaran yang didapat dari proses mendisain primer dengan menggunakan WebSNAPER untuk situs SNP1
Runutan nukleotida Tm (°C) Panjang (pb ) Produk PCR (p
CCGCGATTACCATGTGGTGCTCA 67.759 23 GCAGGGCATCTCCATGGGCTC 66.974 21 CCCGCGATTACCATGTGGTGTTCA 68.352 24 GCAGGGCATCTCCATGGGCTC 66.974 21 CCCGCGATTACCATGTGGTGAACA 68.352 24 GCAGGGCATCTCCATGGGCTC 66.974 21 CCGCGATTACCATGTGGTGGTCA 67.565 23 GCAGGGCATCTCCATGGGCTC 66.974 21 CCGCGATTACCATGTGGTGAGCA 67.759 23 GCAGGGCATCTCCATGGGCTC 66.974 21 CCGCGATTACCATGTGGTGATGC 67.106 23 GCAGGGCATCTCCATGGGCTC 66.974 21 CCGCGATTACCATGTGGTGAACC 66.123 23 GCAGGGCATCTCCATGGGCTC 66.974 21 CCGCGATTACCATGTGGTGAGCC 68.219 23 GCAGGGCATCTCCATGGGCTC 66.974 21 CCGCGATTACCATGTGGTGACCC 68.034 23 GCAGGGCATCTCCATGGGCTC 66.974 21 CCGCGATTACCATGTGGTGTTCC 66.123 23 GCAGGGCATCTCCATGGGCTC 66.974 21 CCGCGATTACCATGTGGTGGTCC 68.034 23 GCAGGGCATCTCCATGGGCTC 66.974 21 CCGCGATTACCATGTGGTGCTCC 68.219 23 GCAGGGCATCTCCATGGGCTC 66.974 21
nukleotida yang diberi garis bawah pada primer forward adalah mismatch. Primer yang yang diberi tanda asterik [*] tidak dipilih untuk meng
high end self complementarity”
Primer alternatif sebagai luaran yang didapat dari proses mendisain primer dengan menggunakan WebSNAPER untuk situs SNP1
Produk PCR (pb) Catatan peringatan 325
326 326 325 325
325 High end self complementarity
325 High end self complementarity 325 325 325 325 325
Disain Primer untuk Marka SNAP
Pada Tabel 22 disajikan contoh luaran dari tahapan disain primer SNAP menggunakan perangkat lunak WebSNAPER. Contoh luaran hasil analisis situs SNP posisi #1 tersebut telah dikonversi ke dalam bentuk tabel. Dalam disain primer SNAP dengan menggunakan WebSNAPER, dari satu situs SNP dapat diperoleh sejumlah alternatif pasangan primer untuk menghasilkan marka SNAP. Sebagai contoh, untuk SNP yang dievaluasi posisi #1 telah dihasilkan 12 pilihan pasangan primer SNAP (5 pasang primer reference dan 7 pasang primer alternate) seperti yang ditampilkan pada Tabel 22. Namun demikian 2 primer alternate tidak bisa dipilih karena berpotensi membentuk utas ganda antar pasangan primer (high end self complementarity), yaitu pasangan primer SNP1_MNBS_L_ALT_1 dan SNP1_MNBS_L_ALT_2. Selanjutnya dari 12 alternatif primer yang dihasilkan, hanya akan dipilih sepasang primer untuk alel referensi dan sepasang primer untuk alel alternatif.
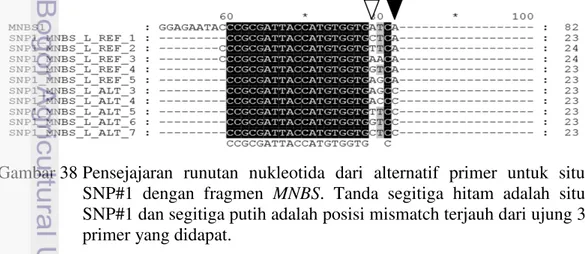
Salah satu hal penting dalam pemilihan primer SNAP adalah Tm. Pasangan primer harus mempunyai Tm yang tidak jauh berbeda. Pada Tabel 22 terlihat bahwa terdapat satu nukleotida yang berbeda selain pada situs SNP-nya (ujung 3’) dari primer forward terhadap sekuen aslinya, yang disebut ‘mismatch’. Posisi mismatch bisa berjarak satu sampai 4 nukleotida dari situs SNP. Untuk lebih jelasnya bisa dilihat pada Gambar 38 yang menunjukkan pensejajaran runutan nukleotida semua primer forward dari Tabel 22 dengan fragmen aslinya (MNBS). Menurut Bru et al. (2008), posisi mismatch dari ujung 3’ sangat berpengaruh terhadap keberhasilan mendapatkan produk PCR. Semakin dekat mismatch dengan ujung 3’ semakin besar kegagalan mendapat produk PCR. Berdasarkan hal tersebut, maka primer SNAP yang dipilih adalah yang mempunyai posisi mismatch dengan jarak paling jauh dari ujung. Gambar 38 adalah contoh pensejajaran runutan nukleotida primer forward untuk SNP1 dari Tabel 22 terhadap fragmen MNBS untuk melihat jarak nukleotida mismatch terhadap ujung 3’ (situs SNP1).
Berdasarkan hasil disain primer SNAP, dari 8 situs SNP fragmen MNBS yang dipilih, satu situs SNP (posisi #215) tidak menghasilkan primer SNAP karena primer yang didapat mempunyai peluang membentuk utas ganda dengan dirinya sendiri pada ujung-ujungnya (high end self complementarity). Hal ini dapat mengakibatkan terbentuknya hairpin atau terbentuknya primer dimer sehingga berpotensi mengganggu amplifikasi fragmen target dan produk PCR yang diinginkan tidak dapat dihasilkan (Brownie et al. 1997).
Gambar 38 Pensejajaran runutan nukleotida dari alternatif primer untuk situs SNP#1 dengan fragmen MNBS. Tanda segitiga hitam adalah situs SNP#1 dan segitiga putih adalah posisi mismatch terjauh dari ujung 3’ primer yang didapat.
Tabel 23 Primer SNAP terpilih dari 7 situs SNP
SNP Identitas primer Sekuen primer forward
1 SNP1_MNBS_Ref CCGCGATTACCATGTGGTG SNP1_MNBS_Alt CCGCGATTACCATGTGGTG 2 SNP2_MNBS_Ref GGCGAGGATCAGCTTCCACC SNP2_MNBS_Alt GGCGAGGATCAGCTTCCACC 3 SNP3_MNBS_Ref GCGAGGCGAGGATCAGA SNP3_MNBS_Alt GCGAGGCGAGGATCAGA 4 SNP4_MNBS_Ref TGGTTCGACCGCGAGGC SNP4_MNBS_Alt TGGTTCGACCGCGAGGC 5 SNP5_MNBS_Ref AACCTCAGCGATTCATTG SNP5_MNBS_Alt ACAACCTCAGCGATTCATTG 6 SNP6_MNBS_Ref CGGAACAACCTCAGCGATTC SNP6_MNBS_Alt CCGGAACAACCTCAGCGATTCA 7 SNP7_MNBS_Ref CTGTATGATATCCATGGCG SNP7_MNBS_Alt TCTGTATGATATCCATGGCG
Keterangan: Nukleotida yang diberi garis bawah adalah
Tabel 24 Primer SNAP terpilih dari 3 situs SNP asal fragmen gen
SNP Identitas primer Sekuen primer forward
1 SNP1_MGlu_Ref GGATGTCCCCCGATCCG SNP1_MGlu_Alt GGATGTCCCCCGATCCG 2 SNP2_MGlu_Ref CGATGGCGTCGAACAGGT SNP2_MGlu_Alt GACGATGGCGTCGAACAGGTT 3 SNP3_MGlu_Ref GCCTGATCAAGTTCTGGTT SNP3_MGlu_Alt CCTGATCAAGTTCTGGTTGTACG
Keterangan: Nukleotida yang diberi garis bawah adalah
situs SNP asal fragmen gen MNBS yang dapat digunakan untuk menghasilkan marka SNAP
forward (5’ - 3’) Sekuen primer reverse (5’ - 3’) Suhu TM (ºC)
GCGATTACCATGTGGTGCTCA GCAGGGCATCTCCATGGGCTC CCGCGATTACCATGTGGTGCTCC GCAGGGCATCTCCATGGGCTC GGCGAGGATCAGCTTCCACCCTTT GGTGATCATGATCGAGGTTGCCAACT GGCGAGGATCAGCTTCCACCCTTA GGTGATCATGATCGAGGTTGCCAACT GCGAGGCGAGGATCAGATTC GGAATGGGGAAAACGACGCT GCGAGGCGAGGATCAGATTA GGAATGGGGAAAACGACGCT TGGTTCGACCGCGAGGCCAG GGAATGGGGAAAACGACGCTCCT GTTCGACCGCGAGGCCAC GGAATGGGGAAAACGACGCTCCT AACCTCAGCGATTCATTGGCGC ATGCAGAAGATCATCGCCAATCGG ACAACCTCAGCGATTCATTGGCGT ATGCAGAAGATCATCGCCAATCGG CGGAACAACCTCAGCGATTCCTTG GGTGATCATGATCGAGGTTGCCAACT CCGGAACAACCTCAGCGATTCACTC GGTGATCATGATCGAGGTTGCCAACT CTGTATGATATCCATGGCGGTTC ATGCAGAAGATCATCGCCAATC TCTGTATGATATCCATGGCGGTTT ATGCAGAAGATCATCGCCAATC
rangan: Nukleotida yang diberi garis bawah adalah mismatch
situs SNP asal fragmen gen β-1,3-glucanase yang dapat digunakan untuk menghasilkan marka SNAP
forward (5’ - 3’) Sekuen primer reverse (5’ - 3’) Suhu TM (
GGATGTCCCCCGATCCGTCG TTACTCGCCAAGAACTGCACGATGG GGATGTCCCCCGATCCGTCC TTACTCGCCAAGAACTGCACGATGG CGATGGCGTCGAACAGGTGCTT TACATCCTCCCCGCCATGCG GACGATGGCGTCGAACAGGTTTTG TACATCCTCCCCGCCATGCG GCCTGATCAAGTTCTGGTTGTACGCCT GCCCAGGCGTACCTGAGCCC CCTGATCAAGTTCTGGTTGTACGGCC GCCCAGGCGTACCTGAGCCC
Keterangan: Nukleotida yang diberi garis bawah adalah mismatch
yang dapat digunakan untuk menghasilkan marka SNAP
Suhu TM (ºC) Produk (pb) 67 325 68 325 68 250 67 250 60 325 59 325 69 335 69 325 66 287 66 289 66 342 67 343 59 372 60 373
yang dapat digunakan untuk menghasilkan marka SNAP
Suhu TM (ºC) Produk (pb) 67 339 66 339 67 334 67 336 67 337 66 336
Tabel 25 Primer SNAP terpilih dari 11 situs SNP
SNP Identitas primer Sekuen primer forward
1 SNP1_Mchi_Ref GCGTGGGGTTACTGCTTC SNP1_Mchi_Alt GCGTGGGGTTACTGCTTC 2 SNP2_Mchi_Ref CGCGTGGGGTTACTGCTTCAT SNP2_Mchi_Alt CGCGTGGGGTTACTGCTTCAT 3 SNP3_Mchi_Ref GCGTGGGGTTACTGCTTCATC SNP3_Mchi_Alt CGTGGGGTTACTGCTTCATC 4 SNP4_Mchi_Ref GGGGTTACTGCTTCATCAGTG SNP4_Mchi_Alt TGGGGTTACTGCTTCATCAGTG 5 SNP5_Mchi_Ref CTTCATCAGTGAACGGAACC SNP5_Mchi_Alt GCTTCATCAGTGAACGGAACC 6 SNP6_Mchi_Ref CATCAGTGAACGGAACCCCC SNP6_Mchi_Alt TCATCAGTGAACGGAACCCCC 7 SNP7_Mchi_Ref CCCAAAGGACTACTGCGTCGC SNP7_Mchi_Alt CCCAAAGGACTACTGCGTCGC 8 SNP8_Mchi_Ref GGTGGCCACCAGGTCTGGG SNP8_Mchi_Alt GTGGCCACCAGGTCTGGG 9 SNP9_Mchi_Ref CGACGGCTTGGGCGACT SNP9_Mchi_Alt GACGGCTTGGGCGACTG 10 SNP10_Mchi_Ref CGGCGTTGGTTGGCTT SNP10_Mchi_Alt CGGCGTTGGTTGGCTTC 11 SNP11_Mchi_Ref CCGGTCGGCGTTGGTTG SNP11_Mchi_Alt CCGGTCGGCGTTGGTTGG Keterangan: Nukleotida yang diberi garis bawah adalah
situs SNP asal fragmen gen chitinase yang dapat digunakan untuk menghasilkan marka SNAP
forward (5’ - 3’) Sekuen primer reverse (5’ - 3’)
GCGTGGGGTTACTGCTTCGTCA GTGGTGACACCGTAGCCCGGAA CGTGGGGTTACTGCTTCGTCC GTGGTGACACCGTAGCCCGGAA CGCGTGGGGTTACTGCTTCATGAG GTGGTGACACCGTAGCCCGGAA CGCGTGGGGTTACTGCTTCATGAC GTGGTGACACCGTAGCCCGGAA GCGTGGGGTTACTGCTTCATCGGT GTGGTGACACCGTAGCCCGGAA CGTGGGGTTACTGCTTCATCGGG GTGGTGACACCGTAGCCCGGAA GGGGTTACTGCTTCATCAGTGCACG GTGGTGACACCGTAGCCCGGAA TGGGGTTACTGCTTCATCAGTGCACA GTGGTGACACCGTAGCCCGGAA CTTCATCAGTGAACGGAACCGCC GTGGTGACACCGTAGCCCGGAA GCTTCATCAGTGAACGGAACCGCT GTGGTGACACCGTAGCCCGGAA CATCAGTGAACGGAACCCCCAAAA GTGGTGACACCGTAGCCCGGAA ATCAGTGAACGGAACCCCCAAAC GTGGTGACACCGTAGCCCGGAA CCCAAAGGACTACTGCGTCGCAAA GTGGTGACACCGTAGCCCGGAA CCCAAAGGACTACTGCGTCGCAAG GTGGTGACACCGTAGCCCGGAA GGTGGCCACCAGGTCTGGGATC CGGCTTTCTTGGCCCAGTTATCTCA GTGGCCACCAGGTCTGGGATG CGGCTTTCTTGGCCCAGTTATCTCA CGACGGCTTGGGCGACTCAGT CGCGTGGGGTTACTGCTTCATCA GACGGCTTGGGCGACTGTGG CGCGTGGGGTTACTGCTTCATCA CGGCGTTGGTTGGCTTTCAA TGCGCTGCAGGCAAGAAATACTACG CGGCGTTGGTTGGCTTCGAC TGCGCTGCAGGCAAGAAATACTACG CCGGTCGGCGTTGGTTGTCT TGCGCTGCAGGCAAGAAATACTACG CCGGTCGGCGTTGGTTGGTG TGCGCTGCAGGCAAGAAATACTACG
Keterangan: Nukleotida yang diberi garis bawah adalah mismatch
yang dapat digunakan untuk menghasilkan marka SNAP
Suhu TM (ºC) Produk (pb) 65 344 65 344 67 345 67 345 67 344 67 343 66 340 67 341 65 330 66 331 66 327 67 328 67 309 66 309 67 263 66 262 67 262 67 261 67 234 66 232 66 237 68 237
Disain primer menggunakan 7 situs SNP menghasilkan total 64 alternatif primer SNAP. Untuk setiap situs SNP diperlukan 2 pasangan primer, yang terdiri atas sepasang primer forward dan reverse untuk alel referensi dan sepasang primer forward dan reverse untuk alel alternatifnya. Dengan demikian, dari 7 situs SNP pada fragmen gen MNBS, dihasilkan 14 pasang primer SNAP seperti yang ditampilkan pada Tabel 23.
Cara yang sama dalam mendisain primer SNAP untuk fragmen gen MNBS juga diterapkan untuk mendisain primer SNAP fragmen gen β-1,3-glucanase (MaGlu) dan chitinase (MaChi). Di dalam fragmen gen β-1,3-glucanase (MaGlu) dari 4 situs SNP yang menyebabkan substitusi residu asam amino, hanya 3 situs yang dipilih untuk mendisain primer SNAP. Satu situs SNP yang berada pada urutan nukleotida nomor 778 tidak dapat digunakan untuk mendisain primer SNAP, karena letaknya terlalu dekat dengan ujung dari fragmen MaGlu (berjarak 9 basa nukleotida), sehingga tidak memenuhi kriteria untuk perancangan primer SNAP menggunakan WebSNAPER. Untuk mendisain primer, di sekitar situs SNP disyaratkan keberadaan sebanyak minimal 25 basa nukleotida.
Berdasarkan 3 situs SNP yang digunakan untuk mendisain primer, diperoleh 22 alternatif pasangan primer SNAP. Berdasarkan beberapa kriteria dalam pemilihan primer seperti yang telah dijelaskan sebelumnya, dipilih 6 pasang primer SNAP untuk 3 situs SNP yang terdapat dalam fragmen MaGlu. Pasangan primer terpilih ditampilkan pada Tabel 24.
Di dalam fragmen gen chitinase (MaChi), dari 14 situs SNP yang teridentifikasi dipilih 11 situs SNP yang digunakan untuk merancang primer SNAP. Disain primer menggunakan 11 situs SNP menghasilkan total 124 alternatif pasangan primer SNAP. Berdasarkan kriteria tertentu dalam pemilihan primer seperti yang dijelas sebelumnya pada perancangan primer SNAP untuk RGA, diperoleh 22 pasang primer untuk 11 situs SNP yang teridentifikasi pada fragmen gen chitinase, seperti yang ditampilkan pada Tabel 25.
Evaluasi Efektivitas Primer SNAP untuk Menghasilkan Marka
Kemampuan primer SNAP yang dikembangkan untuk menghasilkan marka SNAP dengan menggunakan genom tanaman pisang perlu dievaluasi. Untuk evaluasi primer dalam penelitian, dari 14 pasangan primer SNAP yang dihasilkan berdasarkan fragmen gen MNBS, ini hanya dievaluasi 10 pasang primer SNAP. Pada Gambar 39 disajikan hasil PCR yang dilakukan dengan menggunakan 10 pasang primer SNAP dan DNA genom pisang cv. Klutuk Wulung atau Barangan. Produk amplifikasi PCR difragmentasi menggunakan teknik elektroforesis gel agarose, di-staining dengan ethidium bromide (EtBr), dan divisualisasi di bawah penyinaran UV.
Berdasarkan elektroferogram (Gambar 39), pasangan primer yang diuji mampu menghasilkan produk PCR. Semua pasangan primer yang diuji menghasilkan produk PCR pada kedua pisang Klutuk Wulung dan Barangan yang diuji. Untuk primer SNP6_MNBS_Alt, hasil PCR-nya negatif dan tidak ada produk PCR ketika diuji menggunakan genom pisang Barangan. Hal tersebut menunjukkan bahwa pada Klutuk Wulung 5 lokus SNP yang diuji mempunyai kombinasi alel heterozigot, sedangkan pada Barangan, 4 lokus SNP mempunyai kombinasi alel heterozigot dan satu lokus homozigot (lokus SNP6_MNBS).
118
Gambar 39 Produk PCR hasil amplifikasi dari genom tanaman pisang cv. Klutuk Wulung (A) dan Barangan (B) menggunakan 10 pasang primer SNAP berdasarkan situs SNP pada gen MNBS. R= primer reference, A= primer alternate
Produk PCR hasil amplifikasi menggunakan 6 pasang primer SNAP yang dihasilkan untuk fragmen gen MaGlu terhadap DNA genom kultivar Klutuk Wulung dan Barangan ditampilkan pada Gambar 40. Berdasarkan hasil PCR menggunakan DNA genom pisang kultivar Klutuk Wulung dan Barangan sebagai cetakan DNA, semua pasang primer (6 pasang) yang didisain berdasarkan 3 situs SNP dari fragmen MaGlu yang diperoleh dari penelitian ini, mampu menghasilkan produk PCR. Berdasarkan hasil elektroferogram (Gambar 40) terlihat pasangan primer SNP1_MGlu_Ref memberikan hasil negatif pada kultivar Klutuk Wulung, tetapi hasil positif pada kultivar Barangan. Hasil sebaliknya diperoleh apabila menggunakan pasangan primer SNP1_MGlu_Alt. Hal ini menunjukkan lokus SNP1_MGlu kedua kultivar tersebut mengandung alel homozigot, dan sesuai dengan situs SNP yang teridentifikasi pada masing-masing kultivar (Gambar 39). Primer SNP1_MGlu_Ref adalah primer yang mengamplifikasi fragmen MaGlu yang mengandung nukleotida ‘G’ sebagai situs SNP1 pada nukleotida nomor 91, yang terdapat pada Barangan, sedangkan primer SNP1_MGlu_Alt mengamplifikasi fragmen yang mengandung nukleotida ‘C’ (nukleotida nomor 91) pada Klutuk Wulung. Semua pasangan primer SNP2_MGlu dan SNP3_MGlu (reference dan alternate) menghasilkan produk PCR pada kedua kultivar yang diuji. Hal ini menunjukkan baik pada Klutuk Wulung maupun Barangan, kedua lokus tersebut mengandung alel heterozigot.
Gambar 40 Produk PCR hasil amplifikasi dari genom tanaman pisang cv. Klutuk Wulung (A) dan Barangan (B) menggunakan 6 pasang primer SNAP berdasarkan situs SNP pada gen MaGlu. R= primer reference, A= primer alternate R A SNP1_MGlu A. B. R A R A SNP2_MGlu SNP3_MGlu SNP1_MNBS SNP2_MNBS SNP4_MNBS SNP5_MNBS SNP6_MNBS R A R A R A R A R A
A.
B.
119
Gambar 41 Produk PCR hasil amplifikasi dari genom tanaman pisang cv. Klutuk Wulung (A) dan Barangan (B) menggunakan 22 pasang primer SNAP berdasarkan situs SNP pada gen MaChi. R= primer reference, A= primer alternate
Hasil evaluasi 22 pasang primer SNAP berdasarkan fragmen gen chitinase menggunakan DNA genom pisang Klutuk Wulung dan Barangan ditampilkan pada Gambar 41. Berdasarkan elektroferogram (Gambar 41), pasangan primer yang diuji mampu menghasilkan produk PCR dan terdapat polimorfisme antar primer maupun antar kultivar pisang. Primer SNP4_MChi, SNP5_MChi, SNP7_MChi dan SNP11_MChi mampu menghasilkan produk PCR baik primer reference maupun alternate pada genom kedua kultivar pisang. Hal ini menunjukkan keempat lokus tersebut mempunyai kombinasi alel heterozigot pada kedua kultivar pisang. Lokus SNP1_MChi, SNP8_MChi dan SNP10_MChi mempunyai kombinasi alel heterozigot pada Klutuk Wulung, dan alel homozigot pada Barangan, sebaliknya primer SNP6_MChi dan SNP9_MChi mempunyai kombinasi alel heterozigot pada Barangan dan alel homozigot pada Klutuk Wulung. Lokus SNP2_MChi dan SNP3_MChi mengandung alel homozigot pada kedua kultivar pisang. Berdasarkan hasil pengujian tersebut dapat diketahui bahwa dari 11 lokus SNP yang diuji, Klutuk Wulung mempunyai 7 lokus heterozigot dan 4 lokus homozigot, sedangkan Barangan mempunyai 6 lokus heterozigot dan 5 lokus homozigot. Berdasarkan hasil pengujian tersebut dapat disarankan bahwa primer spesifik alel yang didisain bisa digunakan sebagai marka SNAP untuk keperluan analisis lebih lanjut.
Sehubungan dengan pengembangan marka SNAP untuk marka ketahanan terhadap penyakit, marka molekuler berbasis SNP sebaiknya dikembangkan dari beberapa gen yang mempunyai peranan dalam ketahanan terhadap penyakit. Sebagai contoh implementasi marka SNAP berbasis SNP yang berasal dari gen MNBS dapat digabungkan dengan SNP yang berasal dari gen chitinase dan β-1,3-glucanase tanaman pisang.
Pemanfaatan SNP berbasis RGA dan DGA untuk Marka Ketahanan terhadap Penyakit
Adanya keragaman SNP yang terkait (linkage) dengan fenotipe tertentu telah dilaporkan pada tanaman padi, kedelai dan bawang putih (Gupta et al. 2001). Mutasi akibat substitusi satu basa bisa merubah susunan asam amino dari polipeptida yang disandi oleh gen dan dapat merubah fungsi dari polipeptidanya (Kowarsch et al. 2010). Marka molekuler berbasis SNP dengan pendekatan PCR adalah lebih mudah dan efisien dalam pelaksanaannya bila dibandingkan dengan pendekatan enzim restriksi (Liu et al 2012). Salah satu marka molekular berbasis SNP yang menggunakan pendekatan teknik PCR adalah allel-specific PCR (AS-PCR) atau dikenal juga dengan nama SNAP (Drenkard et al. 2000).
R A R A R A R A R A R A R A R A R A R A R A
SNP1_MChi SNP2_MChi SNP3_MChi SNP4_MChi SNP5_MChi SNP6_MChi SNP7_MChi SNP8_MChi SNP9_MChi SNP10_MChi SNP11_MChi
A. B.
120
Agar dapat dimanfaatkan sebagai marka ketahanan terhadap penyakit, marka SNAP berbasis RGA dan DGA perlu dievaluasi polimorfismenya dan keterkaitannya dengan sifat resistensi. Evaluasi tersebut perlu dilakukan untuk seluruh marka SNAP berbasis RGA dan DGA yang telah teridentifikasi. Apabila marka SNAP yang diuji mampu mengelompokkan kultivar atau spesies pisang yang berbeda-beda responnya terhadap penyakit, maka marka SNAP tersebut berpotensi digunakan sebagai marka untuk sifat resistensi penyakit.
Evaluasi marka SNAP berbasis RGA pada 10 kultivar referensi. Hasil amplifikasi PCR menggunakan primer SNAP berbasis RGA pada 10 kultivar pisang ditampilkan pada Gambar 42. Dengan menggunakan primer-primer SNAP tersebut terlihat adanya polimorfisme pada produk amplifikasi PCR. Primer SNP1_MNBS (reference dan alternate) menghasilkan produk amplifikasi pada 10 kultivar yang diuji, hal ini berarti bahwa lokus SNP1_MNBS yang ada pada semua kultivar tersebut mengandung alel heterozigot, sedangkan primer SNAP yang lain menghasilkan produk amplifikasi yang beragam pada kesepuluh kultivar yang diuji (Gambar 42). Berdasarkan elektroferogram hasil amplifikasi PCR menggunakan primer SNAP berbasis RGA, diperoleh data genotipe seperti ditampilkan pada Tabel 26.
Berdasarkan hasil analisis filogenetik terlihat bahwa semua primer SNAP berbasis RGA belum bisa mengelompokkan kultivar pisang berdasarkan sifat ketahanan terhadap penyakit layu FOC. Dalam dendogram tersebut, kultivar rentan berada dalam kelompok yang terpisah dan salah satu kultivar rentan Ambon Kuning menjadi satu kelompok dengan kultivar tahan FOC, Klutuk Wulung. Oleh karena itu untuk menjadikan primer SNAP sebagai marka ketahanan, tidak semua pasangan primer yang digunakan, tetapi dipilih salah satu atau beberapa pasang, serta dikombinasi dengan primer SNAP lain berbasis gen yang berperanan dalam ketahanan terhadap layu cendawan patogen.
Gambar 42 Representasi analisis 5 primer SNAP berbasis RGA pada 10 kultivar pisang. R= primer reference, A= primer alternate, 1= Rejang, 2= Calcuta-4, 3= Klutuk Wulung, 4= Kepok, 5= Jawaka, 6= Ketan, 7= Ambon Hijau, 8= Ambon Kuning, 9= Barangan, 10= Awak. R= primer reference, A= primer alternate R A R A R A R A R A R A R A R A R A R A 1 2 3 4 5 6 7 8 9 10 SNP1_MNBS SNP2_MNBS SNP4_MNBS SNP5_MNBS SNP6_MNBS
121
Tabel 26 Data genotipe hasil konversi dari elektroferogram produk PCR menggunakan primer SNAP berbasis RGA (MNBS) pada 10 kultivar pisang Kultivar SNP1_MNBS SNP2_MNBS SNP4_MNBS SNP5_MNBS SNP6_MNBS R A R A R A R A R A Rejang 1 0 0 1 1 0 1 0 1 0 1 0 1 0 0 1 1 0 1 0 Calcuta-4 1 0 0 1 0 1 0 1 1 0 1 0 1 0 0 1 1 0 1 0 Klutuk Wulung 1 0 0 1 1 0 0 1 1 0 0 1 1 0 0 1 1 0 0 1 Kepok 1 0 0 1 1 0 1 0 1 0 0 1 1 0 0 1 1 0 0 1 Jawaka 1 0 0 1 1 0 1 0 1 0 1 0 1 0 0 1 1 0 0 1 Ketan 1 0 0 1 1 0 1 0 1 0 0 1 1 0 0 1 1 0 1 0 Ambon Hijau 1 0 0 1 1 0 1 0 1 0 0 1 1 0 0 1 1 0 1 0 Barangan 1 0 0 1 1 0 0 1 1 0 0 1 1 0 0 1 1 0 1 0 Ambon Kuning 1 0 0 1 1 0 0 1 1 0 0 1 1 0 0 1 1 0 0 1 Awak 1 0 0 1 1 0 1 0 1 0 1 0 0 1 0 1 1 0 0 1 Keterangan: Jika (+)= ada produk, (-)= tidak ada produk, R(+)-A(-)= 1-0-1-0, R(-)-A(+)= 0-1-0-1,
R(+)-A(+)= 1-0-0-1
Pemilihan primer untuk marka SNAP dapat dilakukan dengan menganalisis setiap lokus. Analisis setiap lokus ini bertujuan untuk mendapatkan gambaran kemampuan setiap situs SNP dalam mengelompokan kultivar yang diuji berdasarkan sifat ketahanan terhadap penyakit layu FOC. Hasil analisis filogenetik setiap lokus untuk SNP berbasis RGA terhadap 10 kultivar pisang ditampilkan pada Lampiran 1. Berdasarkan hasil analisis terlihat bahwa primer SNP4_MNBS mampu mengelompokkan kultivar rentan (Ambon Kuning, Ambon Hijau dan Barangan) dalam satu kelompok, meskipun kultivar tahan Kepok dan Klutuk Wulung juga terikut dalam kelompok kultivar rentan. Kultivar tahan lainnya (Rejang, Calcuta-4, Jawaka dan Awak) sudah terkelompokkan menjadi satu. Dibandingkan dengan primer lainnya, primer SNP4_MNBS adalah primer yang lebih representatif, sehingga bisa digabungkan dengan primer SNAP berbasis gen chitinase dan β-1,3-glucanase sebagai marka ketahanan terhadap penyakit.
Gambar 43 Dendogram hasil analisis filogenetik pengelompokkan kultivar berdasarkan hasil amplifikasi PCR menggunakan primer SNAP berbasis RGA (MNBS). Huruf dalam kurung di belakang kultivar menunjukkan karakter ketahanan masing-masing kultivar: R= tahan, S= rentan, R-S= rentan sampai tahan.
Coefficient 0.70 0.77 0.85 0.93 1.00 Rejang Jawaka Awak Klutuk_Wlg Amb_Kng Kepok Ketan Amb_Hj Barangan Calcuta-4 (R) (R) (R) (R) (S) (R) (R-S) (S) (S) (R)
122
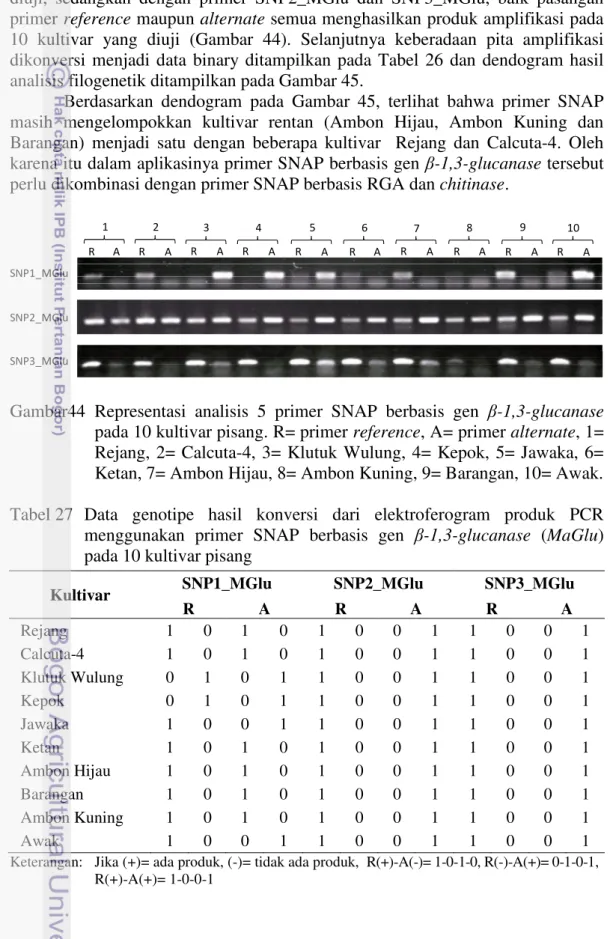
Evaluasi marka SNAP berbasis gen β-1,3-glucanase pada 10 kultivar referensi. Elektroferogram hasil amplifikasi primer SNAP berbasis gen β-1,3-glucanase ditampilkan pada Gambar 44. Berdasarkan hasil amplifikasi terlihat bahwa primer SNP1_MaGlu (reference dan alternate) menghasilkan polimorfisme pada produk amplifikasi PCR asal DNA genom 10 kultivar yang diuji, sedangkan dengan primer SNP2_MGlu dan SNP3_MGlu, baik pasangan primer reference maupun alternate semua menghasilkan produk amplifikasi pada 10 kultivar yang diuji (Gambar 44). Selanjutnya keberadaan pita amplifikasi dikonversi menjadi data binary ditampilkan pada Tabel 26 dan dendogram hasil analisis filogenetik ditampilkan pada Gambar 45.
Berdasarkan dendogram pada Gambar 45, terlihat bahwa primer SNAP masih mengelompokkan kultivar rentan (Ambon Hijau, Ambon Kuning dan Barangan) menjadi satu dengan beberapa kultivar Rejang dan Calcuta-4. Oleh karena itu dalam aplikasinya primer SNAP berbasis gen β-1,3-glucanase tersebut perlu dikombinasi dengan primer SNAP berbasis RGA dan chitinase.
Gambar44 Representasi analisis 5 primer SNAP berbasis gen β-1,3-glucanase pada 10 kultivar pisang. R= primer reference, A= primer alternate, 1= Rejang, 2= Calcuta-4, 3= Klutuk Wulung, 4= Kepok, 5= Jawaka, 6= Ketan, 7= Ambon Hijau, 8= Ambon Kuning, 9= Barangan, 10= Awak. Tabel 27 Data genotipe hasil konversi dari elektroferogram produk PCR menggunakan primer SNAP berbasis gen β-1,3-glucanase (MaGlu) pada 10 kultivar pisang
Kultivar SNP1_MGlu SNP2_MGlu SNP3_MGlu
R A R A R A Rejang 1 0 1 0 1 0 0 1 1 0 0 1 Calcuta-4 1 0 1 0 1 0 0 1 1 0 0 1 Klutuk Wulung 0 1 0 1 1 0 0 1 1 0 0 1 Kepok 0 1 0 1 1 0 0 1 1 0 0 1 Jawaka 1 0 0 1 1 0 0 1 1 0 0 1 Ketan 1 0 1 0 1 0 0 1 1 0 0 1 Ambon Hijau 1 0 1 0 1 0 0 1 1 0 0 1 Barangan 1 0 1 0 1 0 0 1 1 0 0 1 Ambon Kuning 1 0 1 0 1 0 0 1 1 0 0 1 Awak 1 0 0 1 1 0 0 1 1 0 0 1
Keterangan: Jika (+)= ada produk, (-)= tidak ada produk, R(+)-A(-)= 1-0-1-0, R(-)-A(+)= 0-1-0-1, R(+)-A(+)= 1-0-0-1 R A R A R A R A R A R A R A R A R A R A 1 2 3 4 5 6 7 8 9 10 SNP1_MGlu SNP2_MGlu SNP3_MGlu
123
Gambar 45 Dendogram hasil analisis filogenetik pengelompokkan kultivar berdasarkan hasil amplifikasi PCR menggunakan primer SNAP berbasis gen β-1,3-glucanase (MaGlu). Huruf dalam kurung di belakang kultivar menunjukkan karakter ketahanan masing-masing kultivar: R= tahan, S= rentan, R-S= rentan sampai tahan.
Dalam proses analisis, data genotipe yang dianalisis hanya berdasarkan amplifikasi dari primer SNP1_MGlu saja, karena primer SNP lainnya menghasilkan produk yang monomorfik, sehingga kalaupun digunakan data tersebut, tidak akan mempengaruhi luaran, dalam hal ini hasil pengelompokkan dalam dendogram.
Evaluasi marka SNAP berbasis gen chitinase pada 10 kultivar referensi. Amplifikasi PCR menggunakan primer SNAP berbasis gen chitinase yang diperoleh dari Percobaan 4 (Tabel 25) terhadap 10 kultivar pisang ditampilkan pada Gambar 46. Berdasarkan hasil amplifikasi PCR, terlihat adanya polimorfisme antar primer SNAP dan juga antar kultivar yang diuji. Primer SNP4_MChi, SNP5_MChi dan SNP7_MChi menghasilkan produk amplifikasi baik primer reference maupun alternate. Hal ini menunjukkan pada lokus tersebut, kesepuluh kultivar mengandung alel heterozigot, sedangkan primer SNAP yang lain menghasilkan produk amplifikasi yang beragam pada kesepuluh kultivar yang dievaluasi (Gambar 46) dan dikonversi menjadi data genotipe seperti ditampilkan dalam Tabel 28.
Seperti halnya yang terjadi pada analisis filogenetik data genotipe berdasarkan gen β-1,3-glucanase, hasil skoring data monomorfik tidak digunakan untuk analisis data karena tidak mempengaruhi luaran yang tampak dalam dendogram.
Hasil analisis filogenetik memperlihatkan semua primer SNAP berbasis gen chitinase belum bisa mengelompokkan kultivar pisang berdasarkan sifat ketahanan terhadap penyakit layu FOC. Kultivar-kultivar rentan seperti Ambon Hijau, Ambon Kuning dan Barangan berada dalam kelompok terpisah dan masing-masing bersama dengan kultivar tahan seperti Rejang dan Calcuta-4.
Coefficient 0.71 0.78 0.85 0.93 1.00 Rejang Calcuta-4 Ketan Amb_Hj Barangan Amb_Kng Jawaka Awak Klutuk_Wlg Kepok (R) (R) (R) (R) (S) (R) (R-S) (S) (S) (R)
124
Gambar 46 Representasi analisis 5 primer SNAP berbasis gen chitinase pada 10 kultivar pisang. R= primer reference, A= primer alternate, 1= Rejang, 2= Calcuta-4, 3= Klutuk Wulung, 4= Kepok, 5= Jawaka, 6= Ketan, 7= Ambon Hijau, 8= Barangan, 9= Ambon Kuning, 10= Awak.
Hal yang sama juga terjadi pada kultivar tahan FOC. Oleh karena itu untuk menjadikan primer SNAP sebagai marka ketahanan, tidak semua pasangan primer yang digunakan dan dipilih salah satu atau beberapa pasang, serta dikombinasi dengan primer SNAP lain berbasis gen yang berperanan dalam ketahanan terhadap patogen.
Pemilihan primer untuk marka SNAP dapat dilakukan dengan menganalisis setiap lokus. Analisis setiap lokus ini bertujuan untuk mendapatkan gambaran kemampuan setiap situs SNP dalam mengelompokan kultivar yang diuji berdasarkan sifat ketahanan terhadap penyakit layu FOC. Keragaan hasil analisis setiap lokus ditampilkan pada Lampiran 2.
Dari hasil analisis tiap lokus terpilih kombinasi lokus SNP2_MChi, SNP6_MChi, SNP8_MChi, SNP10_MChi dan SNP11_MChi yang digunakan sebagai marka SNAP. Hasil analisis filogenetik yang menggabungkan kelima lokus tersebut ditampilkan pada Gambar 48. Namun demikian pengelompokkan kultivar yang diperoleh masih belum sempurna mewakili pengelompokkan berdasarkan karakter ketahanan masing-masing kultivar terhadap FOC. Oleh karena itu dalam aplikasinya primer SNAP berbasis gen chitinase ini perlu dikombinasi dengan primer SNAP berbasis RGA dan β-1,3-glucanase R A R A R A R A R A R A R A R A R A R A 1 2 3 4 5 6 7 8 9 10 SNP1_MChi SNP2_MChi SNP3_MChi SNP4_MChi SNP5_MChi SNP6_MChi SNP7_MChi SNP8_MChi SNP9_MChi SNP10_MChi SNP11_MChi
125
Tabl 28 Data genotipe hasil konversi dari elektroferogram produk PCR menggunakan primer SNAP berbasis gen chitinase (MaChi) pada 10 kultivar pisang
Kultivar SNP1_MChi SNP2_MChi SNP3_MChi SNP4_MChi SNP5_MChi SNP6_MChi R A R A R A R A R A R A Rejang 1 0 0 1 1 0 0 1 1 0 1 0 1 0 0 1 1 0 0 1 1 0 0 1 Calcuta-4 1 0 0 1 1 0 0 1 1 0 1 0 1 0 0 1 1 0 0 1 1 0 0 1 Klutuk Wlg 1 0 0 1 1 0 1 0 1 0 1 0 1 0 0 1 1 0 0 1 1 0 1 0 Kepok 1 0 0 1 1 0 0 1 1 0 1 0 1 0 0 1 1 0 0 1 1 0 0 1 Jawaka 1 0 0 1 1 0 0 1 1 0 1 0 1 0 0 1 1 0 0 1 1 0 0 1 Ketan 0 1 0 1 1 0 1 0 1 0 1 0 1 0 0 1 1 0 0 1 1 0 0 1 Ambon Hj 1 0 0 1 1 0 0 1 1 0 1 0 1 0 0 1 1 0 0 1 1 0 0 1 Barangan 0 1 0 1 1 0 1 0 1 0 1 0 1 0 0 1 1 0 0 1 1 0 0 1 Ambon Kng 1 0 0 1 1 0 0 1 1 0 1 0 1 0 0 1 1 0 0 1 1 0 0 1 Awak 1 0 0 1 1 0 0 1 1 0 1 0 1 0 0 1 1 0 0 1 1 0 0 1
Kultivar SNP7_MChi SNP8_MChi SNP9_MChi SNP10_MChi SNP11_MChi R A R A R A R A R A Rejang 1 0 0 1 1 0 0 1 1 0 0 1 1 0 1 0 1 0 0 1 Calcuta-4 1 0 0 1 1 0 0 1 1 0 1 0 1 0 1 0 1 0 1 0 Klutuk Wlg 1 0 0 1 1 0 0 1 1 0 1 0 1 0 0 1 1 0 0 1 Kepok 1 0 0 1 1 0 0 1 1 0 0 1 1 0 0 1 1 0 0 1 Jawaka 1 0 0 1 1 0 0 1 1 0 1 0 1 0 0 1 1 0 0 1 Ketan 1 0 0 1 1 0 0 1 1 0 0 1 1 0 1 0 1 0 0 1 Ambon Hj 1 0 0 1 1 0 0 1 1 0 0 1 1 0 1 0 1 0 0 1 Barangan 1 0 0 1 0 1 0 1 1 0 0 1 1 0 1 0 1 0 0 1 Ambon Kng 1 0 0 1 1 0 0 1 1 0 1 0 1 0 1 0 1 0 0 1 Awak 1 0 0 1 1 0 0 1 1 0 0 1 1 0 0 1 1 0 0 1
Keterangan: Jika (+)= ada produk, (-)= tidak ada produk, R(+)-A(-)= 1-0-1-0, R(-)-A(+)= 0-1-0-1, R(+)-A(+)= 1-0-0-1
Gambar 47 Dendogram hasil analisis filogenetik pengelompokkan kultivar berdasarkan hasil amplifikasi PCR menggunakan primer SNAP untuk 10 lokus berbasis gen chitinase (MaChi). Huruf dalam kurung di belakang kultivar menunjukkan karakter ketahanan masing-masing kultivar: R= tahan, S= rentan, R-S= rentan sampai tahan.
Coefficient 0.84 0.88 0.92 0.96 1.00 Rejang Amb_Hj Kepok Awak Jawaka Calcuta-4 Amb_Kng Ketan Barangan Klutuk_Wlg (R) (R) (R) (R) (S) (R) (R-S) (S) (S) (R)
126
Gambar 48 Dendogram hasil analisis filogenetik
berdasarkan hasil amplifikasi PCR menggunakan primer SNP6_MChi, SNP8_MChi,
tahan, S= rentan, R
Gambar 49 Dendogram hasil analisis filogenetik
berdasarkan hasil amplifikasi PCR menggunakan primer
SNP4_MNBS, SNP2_MChi, SNP6_MChi, SNP8_MChi, SNP10_
SNP11_MChi yang melibatkan primer SNP1_MGlu (A) dan yang
tidak (B). R= tahan, S= rentan, R
(A)
(B)
0.80 0.85
hasil analisis filogenetik pengelompokkan
berdasarkan hasil amplifikasi PCR menggunakan primer SNP2_MChi,
SNP6_MChi, SNP8_MChi, SNP10_MChi dan SNP11_MChi.
tahan, S= rentan, R-S= rentan sampai tahan.
hasil analisis filogenetik pengelompokkan
berdasarkan hasil amplifikasi PCR menggunakan primer
_MNBS, SNP2_MChi, SNP6_MChi, SNP8_MChi, SNP10_ hi yang melibatkan primer SNP1_MGlu (A) dan yang R= tahan, S= rentan, R-S= rentan sampai tahan.
Coefficient 0.90 0.95 1.00 Rejang Ketan Amb_Hj Amb_Kng Calcuta-4 Barangan Klutuk_Wlg Kepok Jawaka Awak pengelompokkan kultivar SNP2_MChi,
SNP10_MChi dan SNP11_MChi. R=
pengelompokkan kultivar
berdasarkan hasil amplifikasi PCR menggunakan primer
_MNBS, SNP2_MChi, SNP6_MChi, SNP8_MChi, SNP10_MChi,
hi yang melibatkan primer SNP1_MGlu (A) dan yang
Rejang Ketan Amb_Hj Amb_Kng Calcuta-4 Barangan Klutuk_Wlg Kepok Jawaka Awak (R) (R) (R) (R) (S) (R) (R-S) (S) (S) (R)
127
Dengan menggabungkan primer-primer SNAP terpilih dari ketiga kelompok gen yang diperoleh dari penelitian ini, yaitu RGA (MNBS), β-1,3-glucanase dan chitinase, diperoleh pengelompokkan kultivar berdasarkan karakter ketahanan terhadap FOC yang lebih baik, seperti terlihat pada Gambar 49A dan 49B. Gambar 49A adalah melibatkan primer SNP1_MGlu sedangkan Gambar 49B tanpa melibatkan primer SNP1_MGlu. Dendogram yang dihasilkan baik tanpa atau dengan melibatkan primer SNP1_MGlu dapat mengelompokkan kultivar berdasarkan ketahanan terhadap FOC. Namun demikian dengan tanpa melibatkan primer SNP1_MGlu pengelompokkan yang diperoleh menjadi lebih baik (Gambar 49B) karena dapat memisahkan cabang kelompok kultivar rentan dengan kultivar tahan (pada koefisien kemiripan 83 %). Oleh karena itu untuk pengujian marka menggunakan kultivar/aksesi lain, tanpa melibatkan primer SNP1_MGlu.
Evaluasi marka SNAP pada 10 kultivar/aksesi yang belum diketahui karakter ketahanan terhadap FOC. Dendogram hasil evaluasi yang dilakukan menggunakan DNA genom 10 kultivar/aksesi yang belum diketahui karakter ketahanan terhadap FOC bersama dengan 10 kultivar referensi, ditampilkan pada Gambar 50. Berdasarkan koefisien kemiripan sebesar 80 %, kultivar/aksesi tersebut dibagi menjadi 3 kelompok besar, yaitu R-I (kultivar resisten I) yang terdiri atas 3 kultivar, 2 kultivar adalah Rejang dan Calcuta-4 yang tahan FOC dan satu aksesi AMB-01 (Tongka Langit dari Maluku). Kelompok kedua adalah R-II (kultivar resisten II) yang terdiri atas 10 kultivar/aksesi. Yang termasuk dalam kelompok ini adalah Klutuk Wulung, Kepok, Jawaka dan Awak yang diketahui tahan FOC, dan beberapa kultivar seperti Muli, MDO-01, MBO-04, MLU-07, SUM-04 serta satu spesies liar HlbS-01.
Kelompok ketiga adalah S (rentan) yang beranggotakan beberapa kultivar rentan seperti Ambon Hijau, Ambon Kuning, Barangan dan yang rentan sampai tahan yaitu Ketan. Kultivar Ketan sebetulnya termasuk kultivar yang toleran atau mempunyai ketahanan terhadap FOC sedang. Namun demikian hal ini juga mengkonfirmasi hasil Percobaan 1 (BAB II) bahwa meskipun Ketan dikategorikan kultivar tahan (Tabel 2), tetapi sebanyak 42.% tanaman menunjukkan gejala RDI dan LSI sedang (LSI= 2-3 dan RDI= 2-3), jadi sebetulnya Ketan adalah kultivar yang mempunyai tingkat ketahanan rentan sampai tahan terhadap FOC. Dalam kelompok ketiga ini juga terdapat satu spesies liar Slk-29 dan 2 kultivar (SUM-01 dan HyKC).
Walaupun berdasarkan hasil analisis filogenetik marka SNAP terpilih bisa mengelompokkan kultivar berdasarkan ketahanan terhadap FOC, tetapi marka-marka tersebut perlu diuji pada populasi bersegregasi. Selain itu untuk validasi marka, diperlukan pengujian lebih lanjut terhadap aksesi-aksesi lain yang juga diuji ketahanannya terhadap cendawan FOC. Namun demikian metode ini merupakan tahapan awal pada pemilihan marka SNAP berdasar situs SNP yang terdapat dalam gen yang bertanggung jawab pada ketahanan dan pertahanan tanaman terhadap patogen.
Selain itu, dari penelitian ini dapat diketahui bahwa terdapat perbedaan susunan basa nukleotida antara kultivar pisang yang rentan dan tahan terhadap penyakit layu FOC, dan perbedaan nukleotida tersebut bisa merubah residu asam amino.
128
Gambar 50 Dendogram hasil analisis filogenetik pengelompokkan kultivar referensi dan 10 kultivar/aksesi lain berdasarkan hasil amplifikasi PCR menggunakan primer SNP4_MNBS, SNP2_MChi, SNP6_MChi, SNP8_MChi, SNP10_MChi dan SNP11_MChi. R1= kultivar tahan I, R2= kultivar tahan II, S= kultivar rentan
Simpulan
Pada fragmen RGA (MNBS) asal pisang yang berukuran 524 pb, berhasil diidentifikasi adanya 30 situs SNP, tetapi hanya sebanyak 8 SNP saja yang bisa digunakan untuk mendisain primer SNAP. Selanjutnya dari 8 SNP terpilih, hanya 7 SNP saja yang dapat menghasilkan primer sebanyak 14 pasang untuk pengembangan marka SNAP. Pada fragmen gen β-1,3-glucanase yang berukuran 788 pb diidentifikasi sebanyak 8 situs SNP, tetapi hanya 3 SNP saja yang digunakan untuk pengembangan marka SNAP dan menghasilkan 6 pasang primer SNAP, sedangkan dari fragmen gen chitinase (MaChi) yang berukuran 596 pb berhasil diidentifikasi 22 situs SNP dan sebanyak 11 situs yang menghasilkan 22 pasang primer digunakan untuk pengembangan marka SNAP.
Sebanyak 14 pasang primer yang didapat dari fragmen MNBS, 10 pasang primer yang terbukti efektif untuk mengamplifikasi PCR dengan menggunakan genom pisang Klutuk Wulung dan Barangan. Begitu juga dengan 6 pasang primer SNAP yang berasal dari fragmen gen β-1,3-glucanase dan 22 pasang primer asal fragmen gen chitinase, telah terbukti efektivitasnya dalam mengamplifikasi produk PCR asal kedua kultivar pisang tersebut.
Penggunaan secara bersama-sama marka SNP4_MNBS dengan SNP2_ MChi, SNP6_ MChi, SNP8_ MChi, SNP10_ MChi dan SNP11_MChi dapat mengelompokkan kultivar pisang berdasarkan taraf ketahanannya terhadap penyakit layu FOC.
Coefficient 0.76 0.82 0.88 0.94 1.00 Rejang Calcuta-4 AMB-01 Klutuk_Wlg MDO-01 MND-04 MLU-07 SUM-04 Kepok Jawaka Awak HlbS-01 Muli Ketan Amb_Hj Amb_Kng SUM-01 Barangan Slk-29 HyKC
R1
R2
S
129
Daftar Pustaka
Brownie J, Shawcross S, Theaker J, Whitcombe D, Ferrie R, Newton C, Little S. 1997. The elimination of primer-dimer accumulation in PCR. Nuc Acids Res 25(6):3235–3241.
Bru D, Martin-Laurent F, Philippot L. 2008. Quantification of the detrimental effect of a single primer-template mismatch by Real-Time PCR using the 16s rrna gene as an example. App Env Microbiol 74(5):1660-1663.
Crouch HK, Crouch JH, Jarret RL, Cregan PB, Ortiz R. 1998. Segregation at microsatellite loci in haploid and diploid gametes of Musa. Crop Sci 38:211-217.
Das BK, Jena RC, Samal KC. 2009. Optimization of DNA isolation and PCR protocol for RAPD analysis of banana/plantain (Musa spp.). Int J Agric Sci 1(2):21-25.
Davis RW, Ausubel FM. 2000. A simple procedure for the analysis of single nucleotide polymorphisms facilitates map-based cloning in Arabidopsis. Am Soc Plant Physiol 124(4):1483-1492.
Faure S, Noyer JL, Horry JP, Bakry F, Lanaud C, de Leon Gonzales D. 1993. A molecular marker-based linkage map of diploid bananas (Musa acuminata). Theor Appl Gen. 87:517-526.
Gupta PK, Roy JK, Prasad M. 2001. Single nucleotide polymorphisms: A new paradigm for molecular marker technology and DNA polymorphism detection with emphasis on their use in plants. Current Sci 80:524–535. Howell EC, Newburry HJ, Swennen RL, Withers LA, Ford-Llyod BV. 1994. The
use of RAPD for identifying and classifying Musa germplasm. Genome 37:328-332.
Jiang D, Ye Q-L, Wang F-S, Cao L. 2010. The mining of citrus EST-SNP and its application in cultivar discrimination. Agric Sci China 9(2):179-190. Juwattanasomran R, Somta P, Kaga A, Chankaew S, Shimizu T, Sorajjapinun W,
Srinives Peerasak. 2012. Identification of a new fragrance allele in soybean and development of its functional marker. Mol Breed 29:13-21. Kim MY, Van K, Lestari P, Moon J-K, Lee S-H. 2005. SNP identification and
SNAP marker development for a GmNARK gene controlling supernodulation in soybean. Theor App Genet 110:1003-1010.
Kowarsch A, Fuchs A, Frishman D, Pagel P. 2010. Correlated Mutations: A Hallmark of Phenotypic Amino Acid Substitutions. PLoS Comput Biol 6(9): e1000923. doi:10.1371/journal.pcbi.1000923.
Liu J, Huang S, Sun M, Liu S, Liu Y, Wang W, Zhang X, Wang H, Hua W. 2012. An improved allele-specific PCR primer design method for SNP marker analysis and its application. Plant Methods 8:34.
Molina AB, Williams RC, Hermanto C, Suwanda, Komolog B, Kokoa, P. 2010. Mitigating the threat of banana Fusarium wilt: understanding the agroecological distribution of pathogenic forms and developing disease management strategies. Australia: ACIAR 76 hlm..
Nasu S, Suzuki J, Ohta R, Hasegawa K, Yui R, Kitazawa N, Monna L, Minobe Y. 2002. Search for and analysis of single nucleotide polymorphisms (SNPs) in Rice (Oryza sativa, Oryza rufipogon) and establishment of SNP markers. DNA Res 9:163-171.
130
Nwauzoma, Barth A, Uma S, Saraswathi MS, Mustaffa M. 2011. Developing markers for Sigatoka leaf spot disease(Mycosphaerella musicola Leach) resistance in banana (Musa spp.). Afr J Biotech 10(33):6213-6219.
Rabbi IY, Kulembeka HP, Masumba E, Marri PR, Ferguson M. 2012. An EST-derived SNP and SSR genetic linkage map of cassava (Manihot esculenta Crantz). Theor Appl Genet 125:329–342.
Rotherham D, Harbison SA. 2011. Differentiation of drug and non-drug Cannabis using a single nucleotide polymorphism (SNP) assay. Forensic Sci Int 207:193–197
Sun H, Wang H-T, Kwon W-S, Kim Y-J, In J-G, Yang D-C. 2011. A simple and rapid technique for the authentication of the ginseng cultivar, Yunpoong, using an SNP marker in a large sample of ginseng leaves. Gene 487:75–79. Syvänen AC. 2001. Acessing genetic variation: genotyping SNPs. Nature Rev.
Genet 2:930-942.
Thiel T, Kota R, Grosse I, Stein N, Graner A. 2004. SNP2CAPS: a SNP and INDEL analysis tool for CAPS marker development. Nuc Acids Res 32(1) e5.
Till BJ, Jankowicz-Cieslak J, Sági L, Huynh AO, Utsushi H, Swennen R, Terauchi R, Mba C. 2010. Discovery of nucleotide polymorphisms in the Musa gene pool by Ecotilling. Theor Appl Genet 121:1381–1389.
Ude G, Pillay M, Nwakanma D, Tenkouano A. 2002. Analysis of genetic diversity and sectional relationships in Musa using AFLP markers. Theor Appl Gen 104:1239-1245.
Venkatachalam L, Sreedhar RV, Bhagyalakshmi N. 2008. The use of genetic markers for detecting DNA polymorphism, genotype identification and phylogenetic relationships among banana cultivars. Mol Phylog Evol 47:974–985.
Wang W, Hu Y, Sun D, Staehekin C, Xin D, Xie J. 2012. Identification and evaluation of two diagnostic markers linked to Fusarium wilt resistance (race 4) in banana (Musa spp.). Mol Biol Rep 39(1):451-459.
BAB VIII
PEMBAHASAN UMUM
Pengembangan tanaman pisang di Indonesia masih terus berlangsung walaupun menghadapi beberapa kendala baik kendala teknis maupun non teknis. Kendala non teknis berupa makin berkurangnya lahan pertanian karena beralih fungsi menjadi areal pemukiman, pengembangan sarana umum dan lain-lain merupakan kendala yang tidak bisa dihindari. Kendala teknis yang dihadapi adalah penurunan produksi karena masalah cekaman abiotik maupun biotik. Cekaman abiotik seperti kekeringan, kemasaman tanah, kandungan Al yang tinggi merupakan cekaman abiotik yang masih bisa diatasi walaupun kadang memerlukan biaya yang sangat besar karena melibatkan tindakan modifikasi lingkungan. Cekaman biotik adalah cekaman yang ditimbulkan oleh adanya hama dan penyakit tanaman pisang.
Masalah penyakit tanaman pisang sudah merupakan masalah nasional, karena penyakit pisang seperti layu yang disebabkan oleh cendawan Fusarium oxysporum f.sp. cubense, bakteri Ralstonia solanacearum, dan banana bunchy top virus (BBTV) sudah ditemukan di hampir seluruh wilayah Indonesia dengan intensitas serangan yang beragam (Hermanto et al. 2011).
Sebagai daerah pusat keragaman pisang, Indonesia mempunyai keragaman genetik pisang yang sangat besar, baik spesies liar maupun kultivar, dan tentunya menyimpan sejumlah besar sumber gen termasuk gen ketahanan terhadap penyakit tanaman. Usaha perbaikan kultivar tanaman untuk menghasilkan kultivar pisang tahan penyakit telah dilakukan antara lain dengan induksi mutasi (Sulyanti 2008; Yanti et al. 2009; Indrayanti 2012) maupun persilangan konvensional yang dilakukan oleh Balai Penelitian Tanaman Buah Tropika yang mengadopsi teknik yang digunakan oleh FHIA (Rowe & Rosales, 1993). Namun demikian sampai saat ini mutan maupun hibrida yang komersial masih belum ditemukan di pasaran karena tanaman masih dalam tahap evaluasi dan perbaikan karakter komersial lainnya. Sementara itu kegiatan inventarisasi dan eksplorasi spesies liar dan kultivar pisang masih terus dilakukan untuk mengumpulkan sumber daya genetik yang nantinya bisa digunakan untuk materi perbaikan kultivar pisang komersial (Sutanto et al. 2004).
Walaupun saat ini telah beredar kultivar pisang hibrida hasil persilangan secara konvensional yang tahan terhadap layu FOC, bercak daun sigatoka dan nematoda, yaitu ‘Gold Finger’ yang berasal dari FHIA, Honduras, yang dilepas pada tahun 1988 (Rowe & Rosales 1993), tetapi persilangan secara konvensional menghadapi kendala berupa sterilitas bunga yang tinggi dan kompatibilitas persilangan yang sangat rendah. Untuk menghasilkan kultivar komersial seperti Gold Finger, upaya perbaikan kultivar dimulai dari pengumpulan materi plasma nutfah yang berasal dari Asia Tenggara oleh Dr. Paul Allen pada tahun 1959-1961 (Rosales et al. 1999), dan kegiatan pemuliaan pada tahun 1970-an oleh Dr. Phillip Rowe (Rowe & Rosales 1993). Selain kendala sterilitas bunga dan kompatibilitas persilangan, lamanya proses perbaikan kultivar pisang juga disebabkan proses evaluasi dan seleksi hibrida yang memerlukan waktu yang cukup lama.