DNA BARCODE KERAGAMAN GENETIK, DAN KONSERVASI FAUNA INDONESIA
Oleh
M. Syamsul Arifin Zein
Peneliti Madya Bidang Genetika Molekuler
Laboratorium Genetika Bidang Zoologi
Pusat Penelitian Biologi
LIPI
All Birds DNA barcoding Initiative (ABBI) of Inaugural
Workshop (USA, 2005)
Second International Barcode of Life
Conference (Taiwan, 2007)
Zein MSA et al. 2007. Initiative DNA Barcoding Research in Indonesia.
Second International Barcode of Life Conference, Taipei, Taiwan.
Sutrisno H. 2007. Molecular Phylogeny of Indo-Australian Glyphodes and its Allied Genera (Lepidoptera: Crambidae) Inferred from Mitochondrial COI and COII and Nuclear EF-Iα Gene Sequences. Second International Barcode of Life
Conference.Taipei, Taiwan.
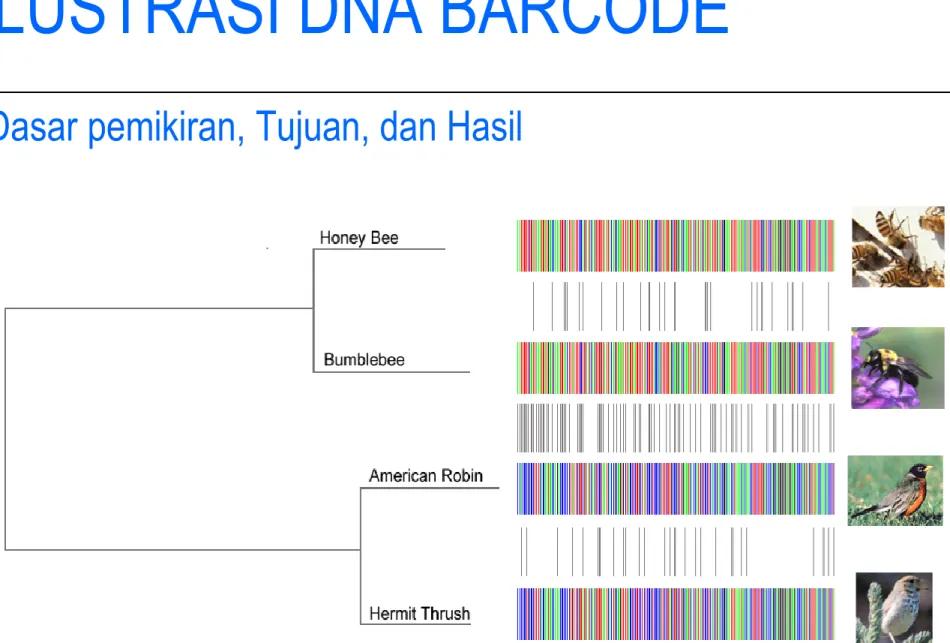
ILUSTRASI DNA BARCODE
Dasar pemikiran, Tujuan, dan Hasil
Apa yang disebut DNA Barcode ???
Barcoding: adalah pendekatan standar untuk
mengidentifikasi tumbuhan dan hewan dengan urutan minimal sekuen DNA, yang disebut barcode DNA.
(Barcoding is a standardized approach to identifying plants and animals by minimal sequences of DNA, called DNA barcodes)
Barcode DNA:
Sebuah urutan DNA pendek, dari sebuah wilayah yang seragam pada genom, yang digunakan untuk mengidentifikasi spesies.
(A short DNA sequence, from a uniform locality on the genome, used
for identifying species).
DASAR PEMIKIRAN STANDARISASI DNA BARCODE
Mempercepat pembangunan perpustakaan referensi urutan DNA yang konsisten dan komprehensif
Mempercepat pengembangan teknologi yang
ekonomis untuk identifikasi spesies
TUJUAN
AGAR SIAPAPUN, DIMANAPUN, KAPANPUN DAPAT
MENGIDENTIFIKASI DENGAN CEPAT DAN AKURAT SPESIES
DARI SPESIMEN APAPUN
KONDISINYA
DNA BARCODE FAUNA....?????
DNA GENOME INTI
DNA MITOKONDRIA
DNA INTI: Exon dan intron
Exon adalah sekuens DNA yang
ditranskripsi menjadi RNA dan ditranslasi menjadi polypeptida (coding region)
Intron adalah sekuens DNA yang tidak
diubah menjadi RNA dan protein (Non
coding region )
DNA MITOKONDRIA
Genom DNA mitokondria berbentuk sirkuler, berisi 13 gen penyandi protein, 22 gen
transfer RNA (tRNA), 2 gen ribosoma (rRNA), dan daerah kontrol (control region/D-
Loop) dengan panjang sekitar 16.775 pasang basa (D ESJARDIN dan M ORAIS , 1990).
Mengapa barcode DNA Fauna dengan DNA mitokondria ?????
Mitokondria:
Merupakan organel yang memproduksi energi dalam sel tumbuhan dan
hewan, Dua puluh tahun penelitian telah menetapkan utilitas urutan DNA
mitokondria dalam membedakan antara spesies hewan yang berhubungan
erat.
Perbedaan antara spesies lebih besar
Jumlah copy:
Ada 100-10,000 lebih banyak salinan mitokondria dibandingkan DNA inti per sel, membuat pemulihan, terutama dari sampel kecil atau
sebagian rusak, lebih mudahdan lebih murah
Relatif sedikit perbedaan dalam spesies
Dalam kebanyakan kasus, kecilnya perbedaan intraspesifik dan
besarnya perbedaan interspesifik merupakan sinyal batas genetik yang berbeda antara sebagian besar spesies, memungkinkan identifikasi yang tepat dengan barcode DNA mitokondria
Gen di DNA mitokondria
Semua merupakan daerah coding ( tidak diselingi intron dan extron
seperti pada gen inti), sehingga dapat amplifikasi langsung. Sedangkan
pada gen inti sering terganggu dengan daerah non coding (intron) yang
sering amplifikasi sulit dan tak terduga.
Results so far suggest that a mitochondrial gene will enable
identification of most animal species.
Mengapa Standarisasi DNA Barcode Fauna menggunakan COI ?
Menggunakan Gen Protein DNA Mitokondria:
Mendefinisikan wilayah standar dan membuat perbandingan antara urutan gen penyandi protein yang mudah, karena gen protein umumnya tidak memiliki insersi dan delesi seperti yang ada di dalam gen ribosom.
Mitokondria Gen penyandi protein pada DNA mitokondria umumnya mengandung
perbedaan lebih tinggi dari gen ribosom dan dengan demikian lebih mungkin untuk
membedakan antara spesies yang berhubungan erat.
KESEPAKATAN INTERNASIONAL DNA BARCODE FAUNA
Menggunakan Gen COI DNA Mitokondria:
Gen Cytochrome c oxidase subunit I (COI)
merupakan reprensentatif dari semua gen penyandi protein DNA mitokondria
Segmen dekat terminus 5’ dari COI sepanjang sekitar 650 pasang basa merupakan daerah
yang digunakan sebagai barcode DNA untuk
fauna (Herbert et al. 2003)
COI terbukti memiliki variasi intraspesifik
rendah, tetapi interspesifik divergensinya tinggi antara taksa yang berdekatan (closely allied
taxa) (Ward et al. 2005; Hajbabaei et al. 2006a).
Primer Universal Standard: (amplifikasi muda
dan hasil sekuen baik).
An Internal ID System for All Animals
Typical Animal Cell
Mitochondrion
DNA
mtDNA
D-Loop
ND5
H-strand ND4
ND4L
ND3 COIII L-strand ND6
ND2 ND1
COII Small ribosomal RNA
ATPase subunit 8 ATPase subunit 6 Cytochrome b
CO I
CO I
The Mitochondrial Genome
DNA BARCODE PADA
MAMALIA
Universal Primer Mamalia:
FORWARD:
LepF1-tl: 5”TGT AAA ACG ACG GCC AGT ATT CAA CCA ATC ATA AAG ATATTG G3”
VF1-tl: 5”TGT AAA ACG ACG GCC AGT TCT CAA CCA ACC ACA ARG AYA3”
VF1d-tl: 5”TGT AAA ACG ACG GCC AGT TCT CAA CCA ACC ACA ARG AYA TYG G3”
VFli-tl: 5”TGT AAA ACG ACG GCC AGT TCT CAA CCA ACC AAA GAA TGG3”
REVERSE:
LepR1-tl: 5”cag gaa aca gct atg cta aac ttc tgg atg tcc aaa aaa tca3”
VR1-tl: 5”cag gaa aca gct atg act aga ctt ctg ggt ggc cra ara ayc a3”
VR1d-tl: 5”cag gaa aca gct atg act aga ctt ctg ggt ggc caa aga atc a3”
VRli-tl: 5”cag gaa aca gct atg act aga ctt ctg ggt gcc aaa ac3”
(Ivanova et al. 2006). Mix 1:1:1:3
Primer untuk sekuensing: (Messing 1983) M13F: 5”tgt aaa acg acg gcc agt3”
M13R:5”cag gaa aca gct atg ac3”
Hasil amplifikasi primer universal
Capillary Sequence Read
Barcode menegaskan kesatuan dari spesies Homo sapiens
Perbandingan menunjukkan bahwa kita berbeda satu sama lain dengan hanya 1 atau 2
nukleotida dari 648, sementara kita berbeda dari simpanse
pada 60 lokasi dan gorila di 70
lokasi.
Teknik Molekuler untuk Identifikasi Spesies Ordo Cetartiodactyla Menggunakan DNA Barcode (Zein MSA&Fitriana YS,2012)
ZooIndonesia:21(02):1-8.
112 spesimen, 4 famili, 10 marga and 15 spesies
Jarak genetik:
intraspesies : 0-0,7% (0,13±0,05%) interspesies : 2-28%
intragenera : 8,8-27,4 (1,36±0,037%) intergenera : 8,8-27,4%
intrafamily : 5,8-11,9% (7,8±2,85) interfamili : 18,6-26,3%
Hasil rekonstruksi pohon filogeni Cetartiodactyla menunjukkan semua
spesies membentuk sebuah cluster kohesif yang jelas berbeda.
BARCODING DNA PADA SURVEI KOMUNITAS KELELAWAR PEMAKAN SERANGGA DI INDONESIA
(DNA Barcoding in Surveys Microbat Communities in Indonesia) (Zein and Fitriana)
Evaluasi pada 136 individu 6 Famili
45 spesies
Sekuen divergensi intraspesifik gen CO1 0.000 -
0,260.
Hipposideros papua.MG 3540 Hipposideros papua.MG 3542 Hipposideros papua.MG 3539 Hipposideros papua.MG 3538 Hipposideros papua.MG 3537 Hipposideros muscinus.MG 2967 Hipposideros muscinus.MG 2968 Hipposideros sabanus.MG 2745
Hipposideros cervinus.MG 2844 Hipposideros ater.MG 1687 Hipposideros ater.MG 1685 Hipposideros ater.MG 1686 Hipposideros diadema.MG 1237
Hipposideros diadema.MG 1556 Hipposideros diadema.MG 3545 Hipposideros diadema.MG 3650 Hipposideros larvatus.MG 2839 Hipposideros larvatus.MG 2840 Hipposideros larvatus.MG 2847 Hipposideros larvatus.MG 3299
Hipposideros
Pipistrellus Pipistrellus javanicus.MG 113
Asseliscus tricuspidatus.MG 3551 Asseliscus tricuspidatus.MG 3547 Asseliscus tricuspidatus.MG 3529 Asseliscus tricuspidatus.MG 3530
Asseliscus
Megaderma spasma.MG 2793 Megaderma spasma.MG 2794 Megaderma spasma.MG 106
Megaderma spasma.MG 2897 Megaderma spasma.MG 2902
Megaderma
Miniopterus pusillus.MG 3378 Miniopterus pusillus.MG 3379 Miniopterus schreibersi.MG 3367 Miniopterus australis.MG 1710
Miniopterus
Miniopterus australis.MG 3533 Miniopterus australis.MG 3534 Miniopterus australis.MG 3711 Miniopterus australis.MG 3532 Miniopterus australis.MG 2845
Miniopterus
Miniopterus schreibersi.MG 3536 Miniopterus schreibersi.MG 3699 Miniopterus schreibersi.MG 3700
Miniopterus
Pipistrellus Pipistrellus javanicus.MG 152
Miniopterus Miniopterus schreibersi.MG 90 Miniopterus schreibersi.MG 1018 Miniopterus schreibersi.MG 1709 Miniopterus magnater.MG 3247 Miniopterus medius.MG 2843 Miniopterus medius.MG 2841 Miniopterus medius.MG 2842
Miniopterus
Mosia Mosia nigrescens.MG 3681
Scotophilus kuhlii.MG 761 Scotophilus kuhlii.MG 763 Scotophilus kuhlii.MG 765 Scotophilus kuhlii.MG 796 Scotophilus kuhlii.MG 805
Scotophilus
Mops sarasinorum.MG 1528 Mops sarasinorum.MG 1529 Mops sarasinorum.MG 1513 Mops sarasinorum.MG 1519 Mops sarasinorum.MG 1511
Mops
Hipposideros Hipposideros diadema.MG 543 Chaerephon plicata.MG 2851
Chaerephon plicata.MG 2852 Chaerephon plicata.MG 2850 Chaerephon plicata.MG 2853
Chaerephon
Otomops Otomops formosus.MG 2539
Emballonura alecto.MG 2895 Emballonura alecto.MG 2894 Emballonura alecto.MG 2482 Emballonura alecto.MG 2498 Emballonura alecto.MG 2521 Emballonura alecto.MG 2795 Emballonura alecto.MG 2801
Emballonura
Taphozous Taphozous melanopogon.MG 2368 Phoniscus atrox.MG 3787 Phoniscus atrox.MG 3788 Phoniscus
Harpiocephalus Kerivoula hardwickei.MG 3759
Kerivoula intermedia.MG 3798 Kerivoula hardwickei.MG 4127 Kerivoula papillosa.MG 3797
Kerivoula
Pipistrellus Pipistrellus javanicus.MG 2469
Tylonycteris Tylonycteris robustula.MG 1081 Pipistrellus javanicus.MG 3295 Pipistrellus javanicus.MG 3301 Pipistrellus
Nyctophilus Nyctophilus sp.MG 2671
Tylonycteris Tylonycteris robustula.MG 1653
Harpiocephalus Harpiocephalus harpia.MG 3767 Myotis muricola.MG 3290 Myotis muricola.MG 2462 Myotis muricola.MG 1712 Myotis muricola.MG 2439 Myotis muricola.MG 472
Myotis muricola.MG 2747 Myotis muricola.MG 2748 Myotis muricola.MG 474 Myotis muricola.MG 464 Myotis muricola.MG 471
Myotis 100
69 100 100
100 75 100
99 76 92 100
100
100 100
66 100
100 66 88 100
100
100
100 100
70 48 100 48 41 81 100
62 86 100
84
79 100 100 92
100 100 100
100 97
78
99 100
100 100
93
73 78 51 100 48 100 58
100 98
96 96
98
100
99 100
100 58 50
64 59
82
70 51 57
44 70
44
51
25 47
19 35
27 18
13
26 15
2
6
0.02