PERBANDINGAN INHIBISI BRUSEIN D TERHADAP PROTEIN
NFKB PADA SEL KANKER PANKREAS SECARA FLEKSIBEL DAN
RIGID DOCKING
Harry Noviardi
1, Armi Wulanawati
2, Muhamad Sholehuddin Malik Ibrohim
21Program Studi Farmasi, Sekolah Tinggi Teknologi Industri dan Farmasi , Bogor 2Departemen Kimia, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian
Bogor
ABSTRAK
Kanker pankreas merupakan jenis tumor ganas yang sangat mematikan. Penderita kanker pankreas umumnya ditangani dengan kemoradioterapi dan obat gemsitabin, namun masih belum memberikan hasil klinis yang baik. Penelitian ini bertujuan menentukan perbandingan potensi senyawa bahan alam brusein D sebagai inhibitor protein transkripsi NFĸB sel kanker pankreas berdasarkan energi bebas Gibbs (∆G), tetapan inhibisi, interaksi hidrogen secara fleksibel dan rigid
docking. Berdasarkan hasil analisis docking, menunjukkan bahwa docking secara fleksibel
memberikan hasil yang lebih baik dibandingkan rigid. Selain itu, senyawa brusein D memberikan potensi inhibisi yang lebih baik dibandingkan standar gemsitabin.
Kata kunci: brusein D, docking, kanker pankreas, protein NFKB
ABSTRACT
Pancreatic cancer is a malignant tumor types are particularly deadly. Patients with pancreatic cancer are generally treated with chemoradiotherapy and gemcitabine, but still provide a good clinical outcome. This study aims to determine the ratio of the potential of bruceine D as inhibitors of protein NFĸB transcription of pancreatic cancer cells by the Gibbs free energy (ΔG), inhibition constants, hydrogen interactions are flexible and rigid docking. Based on the analysis docking, docking flexibly deliver better results than rigid. Additionally, bruceine D provides the potential for better inhibition than gemcitabine.
Keywords : bruceine D, docking, pancreatic cancer, NFĸB protein
PENDAHULUAN
Salah satu jenis kanker paling mematikan di dunia adalah kanker pankreas. Kasus kanker pankreas terus meningkat dalam 2 dekade terakhir dengan tingkat morbiditas dan mortalitas penderita yang sangat tinggi [1]. Penderita kanker pankreas yang dapat bertahan hidup selama 5 tahun berjumlah kurang dari 5%[2]. Hal ini terjadi karena proliferasi atau pembelahan sel yang bermutasi secara tidak terkendali sehingga sel kanker mengurangi proses terjadinya apoptosis [3] (proses bunuh diri pada sel) sangat penting karena berguna untuk menghilangkan sel-sel abnormal atau sel kanker [4]. Oleh karena itu, protein yang berperan dalam proses pembelahan dan metabolisme sel kanker (protein antiapoptosis) harus dihambat [5]. Salah satu jenis protein antiapoptosis yang akan dihambat pada penelitian ini adalah NFĸB [6].
Protein NFĸB merupakan jenis protein yang berperan sebagai faktor transkripsi pada sel kanker payudara, kolon, prostat dan kanker pankreas. Aktivasi protein NFĸB terkait dengan tahap inisiasi dan perkembangan gen sel kanker yang terlibat pada pertumbuhan sel, proliferasi, antiapoptosis, angiogenesis, dan metastasis [7]. Beberapa jenis protein NFĸB yang terdapat pada mamalia adalah NFĸB1 (p50), dan NFĸB2 (p52). Protein tersebut berbentuk homodimer maupun heterodimer [8]. Beberapa faktor yang dapat mengaktivasi dari protein NFĸB adalah ROS, TNFα, interleukin (IL)-1β, bakteri lipopolisakarida, virus DNA beruntai ganda, dan radiasi7. Protein NFĸB bersifat spesifik hanya terdapat jaringan yang terkena sel kanker dan tidak terdapat pada jaringan sel normal [9].
Gemsitabin merupakan analog dari pirimidina yang bekerja merusak susunan dari asam nukleat sehingga menghambat proses pembelahan sel. Penghambatan tersebut mendorong sel untuk mengaktifkan mekanisme apoptosis atau sel melakukan bunuh diri [10]. Namun, dalam dosis yang tinggi, obat ini dapat menyebabkan keracunan pada hati dan ginjal, anemia, dan trombositopenia [11]. Oleh karena itu, telah banyak diteliti obat kanker yang berbasis bahan alam. Obat kanker yang banyak dikembangkan saat ini berasal dari bahan alam yang digabung dengan kemoterapi dan radioterapi konvensional [6].
Brucea javanica (L.) Merr. atau biasa
dikenal dengan nama buah makasar merupakan
tumbuhan obat yang biasa digunakan sebagai antimalaria, antipiretik, dan efek homeostatis [12]. Kandungan senyawa aktif dari buah ini adalah brusein D yang telah diketahui memiliki aktifitas antiproliferasi pada sel kanker. Pada penelitian sebelumnya diketahui bahwa ekstrak etanol dari tanaman ini dapat menghambat proliferasi sel adenokarsinoma pankreas manusia dan menyebabkan apoptosis secara in vitro [13]. Brusein D menghambat protein p38 atau dikenal juga sebagai MAPK dan mengaktifkan jalur sinyal apoptosis pada sel kanker pankreas [14].
Analisis penghambatan menggunakan metode komputasi atau in silico yang merupakan metode docking atau penambatan ligan pada suatu protein. Metode ini lazim digunakan dalam tahap pencarian suatu kandidat molekul obat, karena metode ini sangat cepat, ekonomis, andal, dan ramah lingkungan [15]. Pada penelitian ini bertujuan menentukan potensi inhibisi dari Brusein D terhadap protein NFĸB berdasarkan parameter energi bebas Gibbs, tetapan inhibisi, serta interaksi antara protein dan senyawa ligan kandidat obat.
METODE PENELITIAN
Bahan: Perangkat lunak Autodock Tools 1.5.6,
Autogrid 4.2, Autodock 4.2, Pymol 1.7.
Alat: Seperangkat komputer dengan chip
processor AMD A10-6800K quadcore4.1GHz, random access memory (RAM) 8 gigabit, dan video graphics array AMD Radeon HD 8670D,
ditunjang dengan akses internet 4G LTE menggunakan modem ZTE mf825a untuk mengunduh data-data protein dan substrat.
Metode
Preparasi Fail Protein dan Ligan
Protein-protein yang berperan dalam pertumbuhan dan pembelahan sel kanker (antiapoptosis), ditelusuri pada situs Protein
Data Bank (http://www.rscb.org/pdb/) dengan
menggunakan perangkat komputer yang terhubung ke internet. Protein yang ditelusuri adalah protein transkripsi (NFĸB1, NFĸB2) [6]. Fail protein tersebut diunduh dan dikumpulkan dengan format .pdb.
Ligan yang akan digunakan untuk menginhibisi protein antiapoptosis juga ditelusuri dan dikumpulkan dalam format .pdb. Ligan-ligan tersebut brusein D dan gemsitabin.
Optimasi Grid
Protein .pdb diolah menggunakan Perangkat Lunak Autodock Tools 1.5.6 dengan melakukan read molecule. Protein disimpan dengan ekstensi .pdbqt. Kemudian ligan dibuka dengan input ligand, diatur agar sehingga memiliki torsi yang bebas dan disimpan juga dengan ekstensi .pdbqt. Ukuran grid ditentukan pada setiap protein dengan nilai sumbu x, y, dan
z yang berbeda-beda berdasarkan hasil optimasi. Jarak antar grid juga ditetapkan berbeda-beda pada setiap protein dengan satuan angstrom (Å). Grid di buat sebesar mungkin sampai tepat menutupi seluruh sisi aktif permukaan protein. Fail grid disimpan dalam bentuk grid.gpf.
Docking
Autogrid merupakan bagian dari proses docking untuk menentukan area interaksi antara
protein dan ligan. Grid yang digunakan merupakan hasil dari proses optimasi sebelumnya. Setelah ukuran grid disesuaikan dengan grid optimum, disimpan dalam bentuk grid.gpf. Setelah itu, Autogrid 4.2 diolah menggunakan terminal ubuntu pada direktori fail tersebut.
Prosedur docking secara flesibel pada umumnya sama dengan docking secara rigid. Perbedaannya hanya terdapat pada ikatan σ, pada kondisi rigid ikatan σ dibuat tidak dapat berotasi. Docking dilakukan pada terminal Ubuntu menggunakan Autodock 4.2 sehingga menghasilkan fail docking.dlg. Fail docking.dlg dianalisis menggunakan Autodock Tools 1.5.6 untuk mengetahui nilai ∆G. Fail tersebut kemudian disimpan dalam bentuk kompleks untuk divisualisasikan menggunakan Pymol 1.7.
Visualisasi Fail Docking
Fail keluaran docking dengan ekstensi .pdbqt divisualisasikan menggunakan Pymol 1.7 untuk melihat tapak pengikatan, jumlah interaksi hidrogen, dan jarak antar atom yang berikatan.
HASIL DAN PEMBAHASAN
Pertumbuhan sel kanker disebabkan protein-protein yang meregulasi kelangsungan hidup sel mengalami malafungsi sehingga sel tersebut terus tumbuh secara abnormal. Protein tersebut harus diinaktivasi dengan cara inhibisi melalui pada tapak aktifnya dengan menggunakan ligan obat. Oleh karena itu, pencarian obat baru masih terus dilakukan
untuk memberikan tingkat harapan hidup yang lebih tinggi. Kandidat obat harus mengandung ligan yang dapat menginhibisi protein antiapoptosis sehingga menghambat terjadinya proliferasi, diferensiasi, apoptosis, dan metastasis [3]. Salah satu ligan yang dapat menginhibisi protein antiapoptosis adalah senyawa ligan bahan alam yang memiliki aktivitas antioksidan yang baik dan tanpa efek samping.
Pencarian kandidat obat tersebut dapat dilakukan secara in silico dengan metode
docking. Simulasi docking ini melibatkan
beberapa protein antiapoptosis sel kanker pankreas serta ligan-ligan yang berasal dari senyawa bahan alam. Protein dan ligan tersebut diinteraksikan untuk menentukan senyawa bahan alam terbaik yang dapat menginhibisi protein antiapoptosis berdasarkan beberapa parameter seperti energi bebas Gibbs (∆G), tetapan inhibisi, dan interaksi hidrogen. Interaksi kemudian dibandingkan dengan hasil
docking antara protein antiapoptosis dan standar
gemsitabin.
Tapak Aktif Protein
Sebelum dilakukan docking, terlebih dahulu ditentukan daerah pada protein yang akan diinhibisi atau grid. Grid merupakan daerah tempat terjadinya interaksi pada proses
docking, yang berbentuk kubus atau balok
dengan sebuah titik pusat x,y,z dan dimensi x,y,
z (Gambar 1). Pada proses docking, grid harus
diarahkan pada tapak aktif protein. Biasanya tapak aktif protein berupa seperti lubang atau
cave yang akan diisi oleh ligan. Pada protein
biasanya terdapat lebih dari 1 lubang. Oleh karena itu, ketertarikan ligan terhadap lubang-lubang protein tersebut diuji secara acak menggunakan algoritma, untuk mengetahui daerah tapak aktif protein. Optimasi grid seperti ini juga biasa dikenal dengan uji cavity [16]. Untuk mendapatkan grid pada lubang yang tepat, protein diinteraksikan dengan ligan menggunakan dimensi grid yang besarnya tepat menutupi seluruh permukaan protein target. Parameter algoritma yang digunakan adalah
Lamarckian Genetic Algorithm [17]. Total konformasi terbaik yang ditampilkan oleh Autodock Tools adalah 10 interaksi ligan. Daerah dengan banyak ligan menempel diduga merupakan tapak aktif protein yang selanjutnya akan digunakan untuk docking.
Tabel 1 menunjukkan hasil dari optimasi
grid. Hasil optimasi grid tapak aktif protein
tersebut kemudian digunakan untuk docking secara fleksibel dan rigid.
Interaksi Protein-Ligan
Docking atau penambatan protein-ligan
dilakukan untuk mencari interaksi terbaik antara protein-ligan dengan parameter tertentu seperti energi bebas Gibbs (∆G), tetapan inhibisi, dan interaksi hidrogen. Docking dilakukan 2 macam perlakuan terhadap ligan,
yaitu rigid dan fleksibel. Perlakuan ini bertujuan untuk membandingkan interaksi yang lebih baik antara ligan rigid dan fleksibel.
Docking secara rigid berbasis pada teori enzim
Emil Fischer. Substrat akan menginduksi enzim tepat pada tapak aktif untuk mencapai reaksi.
Docking secara fleksibel berbasis pada teori
enzim Induced Fit, teori ini mengasumsikan bahwa substrat berperan dalam menentukan bentuk akhir dari enzim dan sebagian dari enzim tersebut bersifat fleksibel. Hal ini menjelaskan mengenai senyawa tertentu dapat
a
b
mengikat enzim, tetapi tidak bereaksi karena enzim tersebut terdistorsi. Senyawa tersebut mungkin telalu kecil untuk menginduksi enzim,
sehingga enzim hanya dapat bereaksi dengan substrat yang tepat.
Tabel 1. Grid tapak aktif protein transkripsi NFĸB.
Protein
Titik Pusat Tapak aktif (Å) Dimensi Tapak aktif (Å)
x y z x Y Z
NFĸB1 5.048 4.968 1.404 50 44 52
NFĸB2 9.334 -9.326 -11.197 46 46 46
Interaksi Protein-Ligan secara Fleksibel dan Rigid
Docking secara fleksibel memungkinkan
ligan berotasi bebas pada rantai tunggalnya sehingga ligan memiliki banyak konformasi. Ikatan σ yang berbentuk silinder simetris memungkinkan rotasi antar karbon-karbon dalam molekul rantai terbuka. Pada dasarnya prosedur docking protein-ligan secara fleksibel sama dengan saat menentukan tapak aktif protein, akan tetapi menggunakan grid yang lebih spesifik dan terarah pada tapak aktif protein.
Docking secara rigid pada umumnya sama
dengan docking secara fleksibel, perbedaannya hanya terdapat pada ikatan σ ligan. Pada
docking secara rigid, ikatan σ dibuat menjadi
kaku sehingga tidak dapat berotasi.
Fail protein diolah menggunakan Autodock Tools, kemudian protein dibersihkan dari molekul-molekul air dan dilakukan penambahan hidrogen. Penambahan atom hidrogen dilakukan karena terdapat kemungkinan hilangnya atom hidrogen pada
saat kristalisasi yang dapat memengaruhi interaksi.
Setelah grid terbentuk, dilakukan autogrid menggunakan Autogrid 4.2 dan docking menggunakan Autodock 4.2 melalui Terminal Ubuntu. Kemudian akan diperoleh nilai ∆G paling rendah atau negatif. Kriteria ∆G ˂ 0 pada suhu dan tekanan konstan, memiliki arti bahwa reaksi kimia tersebut bersifat spontan dengan penurunan energi berbas Gibbs. Jika pada hasil reaksi ∆G menurun, maka reaksi memiliki kecenderungan spontan untuk mengkonversi reaktan menjadi produk.
Indikator proses docking yang baik dapat dilihat dengan membandingkan nilai energi bebas Gibbs (ΔG), tetapan inhibisi, dan jumlah interaksi hidrogen terhadap standar inhibitor. Ikatan pembentukan kompleks yang kuat ditandai dengan nilai ΔG yang rendah, tetapan inhibisi rendah, dan banyaknya jumlah interaksi hidrogen. Berdasarkan nilai ∆G tersebut menggunakan persamaan Gibbs maka akan diperoleh nilai tetapan inhibisi (Ki) [18].
𝐾 = 1 𝐾𝑖⁄ ……… {i}
∆𝐺 = −𝑅𝑇𝐿𝑛𝐾 ………... {ii}
∆𝐺 = −𝑅𝑇𝐿𝑛 1 𝐾𝑖⁄ ………. {i} dan {ii}
∆𝐺 = 𝑅𝑇𝐿𝑛𝐾𝑖………. {i} dan {ii}
Hasil analisis energi bebas Gibbs , tetapan inhibisi rendah, dan banyaknya jumlah interaksi hidrogen dapat dilihat pada Tabel 2. Jika dibandingkan antara docking secara rigid dan fleksibel, maka docking secara fleksibel memberikan lebih banyak kemungkinan interaksi, karena pada docking secara rigid, ikatan antara karbon-karbon sp3 dibuat kaku sehingga tidak dapat berputar. Ligan-ligan yang masuk ke dalam lubang protein membutuhkan penyesuaian dengan mengubah konformasi ligan agar ligan bisa menginhibisi dengan baik. Senyawa ligan bahan alam memiliki struktur
yang cukup besar, rerata terdiri lebih dari 20 karbon sehingga cukup sulit bagi ligan tersebut untuk masuk ke dalam protein jika tidak melakukan penyesuaian. Kesulitan tersebut membuat interaksi protein-ligan menjadi kurang spontan dibandingkan dengan docking secara fleksibel, terlihat dari nilai ΔG yang menurun pada hampir seluruh interaksi protein-ligan secara rigid. Jumlah interaksi hidrogen juga mengalami penurunan akibat sulitnya ligan berinteraksi dengan protein (Gambar 2 dan Gambar 3).
Tabel 2. Hasil Docking protein-ligan secara fleksibel dan rigid.
Protein Ligan Fleksibel Rigid ΔG (kkal/mol) KInhibisi ∑Interaksi Hidrogen ΔG (kkal/mol) KInhibisi ∑Interaksi Hidrogen NFĸB1 Brusein D -5.49 94.05 5 -4.55 460.05 4 Gemsitabin -3.75 1776.50 6 -3.03 5993.05 4 NFĸB2 Brusein D -6.14 31.38 6 -5.93 44.73 4 Gemsitabin -5.33 123.23 7 -3.54 2532.73 4
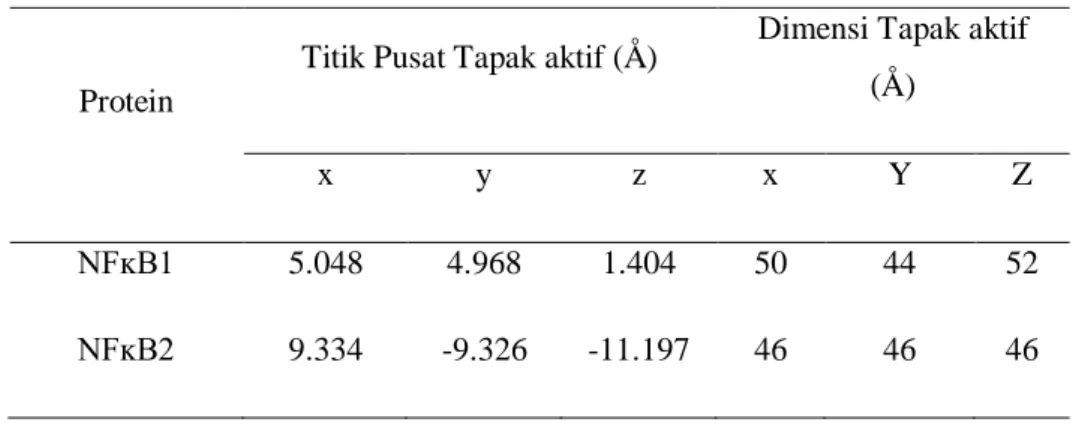
Gambar 2. Visualisasi interaksi docking protein NFĸB1dengan ligan, brusein D rigid (a), brusein D fleksibel (b), gemsitabin rigid (c), gemsitabin fleksibel (d).
a
c d
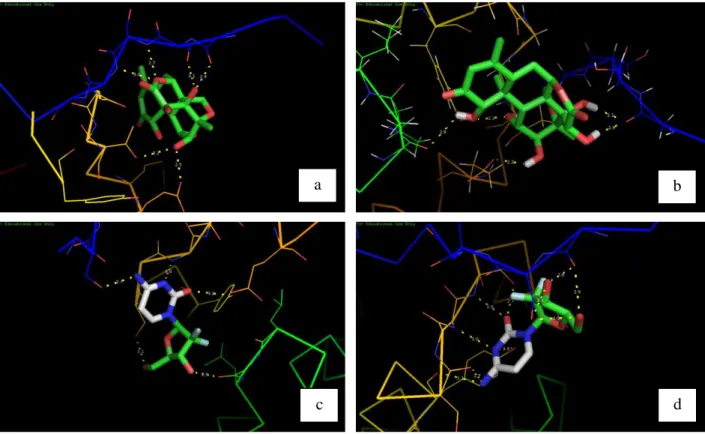
Gambar 3. Visualisasi interaksi docking protein NFĸB2 dengan ligan, brusein D rigid (a), brusein D fleksibel (b), gemsitabin rigid (c), gemsitabin fleksibel (d).
SIMPULAN
Berdasarkan pada hasil analisis
docking, docking secara fleksibel memberikan
hasil yang lebih baik dibandingkan rigid. Brusein D memberikan potensi inhibisi lebih baik dibandingkan dengan gemsitabin berdasarkan nilai energi bebas Gibbs, tetapan inibisi dan interaksi ikatan hidrogen antara protein dan ligan.
DAFTAR PUSTAKA
[1] Nakamura T, Masuda K, Harada S, Akioka K, Sako H. 2013. Pancreatic cancer: Slow progression in the early stages. Int JSurgery Case Rep.
4:693-696.
[2] Zhao G, Deng S, Zhu S, Wang B, Li X, Liu Y, et al. 2014.Chronic pancreatitis and pancreatic cancer demonstrate active epithelial-mesenchymal transition profile, regulated by miR-217-SIRT1 pathway. Cancer Lett. 355:184-191.
[3] Pratheeshkumar P, Sreekala C, Budhraja A, Ding S, Son Y, Wang X, et
al. 2012. Cancer prevention with
promising natural products: mechanisms of action and molecular targets. Anti-Cancer Agents in MedChem. 12:1-26.
[4] Kuno T, Tsukamoto T, Hara A, Tanaka T. 2012. Cancer chemoprevention trough the introduction of apoptosis by natural compound. Journal of Biophysical Chemistry. 2:156-173
[5] Yan C, Siegel D, Newsome J, Chiloux A, Moody CJ, Ross D. 2012. Antitumor indolequinones induced apoptosis in human pancreatic cancer cells via inhibition of thioredoxin reductase and activation of redox signaling.
Molecular Pharmacology.
81(3):401-410.
[6] Lin L, Leung PS. 2014.Use herbal medicines and natural product: an alternative approach to overcoming the apoptotic resistance of pancreatic cancer. Int JBiochem & Cell Bio. 53:224-236.
a
c d
[7] Acharya A, Das I, Chandhok D, Saha T. 2010. Redox regulation in cancer: a double-edged sword with therapeutic potential. Oxid Med Cell Longev. 3:23-34.
[8] Carbone C, Melisi D. 2012. NF-kappaB as a target for pancreatic cancer therapy.Expert Opin Ther Targets. 16(2): S1-10.
[9] Wang W, Abbruzzese JL, Evans DB, Larry L, Cleary KR, Chiao PJ. 1999. The nuclear factor-kappa B RelA transcription factor is constitutively activated in human pancreatic adenocarcinoma cells. Clin Cancer Res. 5:119-127.
[10] Cerqueira NMFSA, Fernandes PA, Ramos MJ. 2007. Understanding ribonucleotide reductase inactivation by gemcitabine. Chem Eur J. 13:8507-8515.
[11] Moysan E, Bastiat G, Benoit JP. 2013. Gemcitabine versus modified gemcitabine: A review of several promising chemical modifications. J
Am Chem Soc. 10:430-444.
[12] Wagih ME, Alam G, Wiryowidagdo, Attia K. 2008. Improved production of the indole alkaloid canthin-6-one from cell suspension culture of Brucea javanica(L.) Merr. Indian Journal of
Science and Technology. 1(7):1-6.
[13] Lau ST, Lin ZX, Zhao M, Leung PS. 2008. Brucea javanica fruit induces
cytotoxicity and apoptosis in pancreatic adenocarcinoma cell lines. Phytother.
Res. 22:477-486.
[14] Lau ST, Lin ZX, Liao Y, Zhao M, Cheng HK, Leung PS. 2009. Brucein D induces apoptosis in pancreatic adenocarcinoma cell line PANC-1 through the activation of p38-mitogen activated protein kinase. Cancer Letters. 281:42-52.
[15] Ördög R dan Grolmusz V. 2008.
Evaluating genetic algorithms inprotein-ligand docking. Lecture Notes in Comp Sci. 4983:402-413.
[16] Oliveira SHP, Ferraz FAN, Honorato RV, Xavier-Neto J, Sobreira TJP, Oliveira PSL. 2014. KVFinder: steered identification of protein cavities as a PyMOL plugin. BMC Bioinformatics. 15:197-204.
[17] Morris GM, Huey R, Lindstrom W, Sanner MF, Belew RK, Goodsell DS, Olson AJ. 2009. AutoDock4 and AutoDockTools4: automated docking with selective receptor flexibility. J
Comput Chem. 30(16):2785-2791.
[18] Morris GM, Goodsell DS, Halliday RS, Huey R, Hart WE, Belew RK, Olson AJ. 1998. Automated docking using a lamarckian genetic algorithm and an empirical binding free energy function. J Comput Chem. 19(14):1639-1662.