BAB III
ISOLASI DAN KARAKTERISASI RESISTANCE GENE
ANALOGUE (RGA) ASAL 3 KULTIVAR PISANG YANG
TAHAN PENYAKIT LAYU FUSARIUM
Abstrak
Isolasi dan karakterisasi resistance gene analogue dari kultivar pisang lokal sangat bermanfaat untuk mendukung pengembangan kultivar tahan penyakit layu
Fusarium oxysporum f.sp. cubense (FOC). Resistance gene analogue (RGA) diisolasi dan dikarakterisasi tiga kultivar pisang tahan FOC menggunakan
degenerate primer berdasarkan domain NBS. Dari 91 fragmen yang diproses runutan basa nukleotidanya, 17 fragmen positif mengandung sekuen NBS dan disandi dengan MNBS1-MNBS17. Analisis filogenetik prediksi asam amino
MNBS menghasilkan empat kelompok, yaitu kelompok pertama mempunyai 14 anggota (MNBS1-MNBS14) dengan sequence identity sebesar 97.4 %, sedangkan ketiga kelompok yang lain masing-masing mempunyai satu anggota, yaitu
MNBS15, MNBS16 dan MNBS17 dengan sequence identity sebesar 28.5 %. Semua MNBS termasuk dalam kategori non-TIR-NBS-LRR. Analisis filogenetik
MNBS dengan RGA dan R-gene yang terdeposit pada GenBank menunjukkan bahwa prediksi asam amino MNBS mempunyai sequence identity sebesar 91.7-98.8 % dengan Musa NBS-LRR dan 19.9-35.5 % dengan R-gene tanaman lain yang telah diketahui. Diantara 17 MNBS tersebut, MNBS17 mempunyai sequence
identity sebesar 50.5 % dengan RGC2 (ABY75802) asal Musa acuminata ssp.
malaccensis, yang berasosiasi dengan ketahanan terhadap layu FOC.
Kata kunci: ketahanan penyakit, NBS, pisang, RGA 1
Bab ini telah dikirim ke Emirates Journal of Food and Agriculture (EJFA) untuk dipublikasi dengan judul: Isolation and Characterization of Resistance Gene Analogue (RGA) from Fusarium Resistance Banana Cultivars
ISOLATION AND CHARACTERIZATION OF
RESISTANCE GENE ANALOGUE (RGA) FROM 3 FUSARIUM
WILT RESISTANT BANANA CULTIVARS
Abstract
Isolation and characterization of resistance genes from local banana cultivars should be useful to support development of future FOC resistance cultivars. Resistance gene analogues (RGAs) were isolated and characteized form 3 fusarium wilt resistant banana cultivars using degenerate primers based on NBS domains. Out of 91 fragments sequenced, 17 fragments were positively NBS-type sequences and encoded as MNBS1-MNBS17. Phylogenetic analysis of MNBS deduced amino classified into 4 groups; the first group consisted of 14 members (MNBS1-MNBS14) with 97.4 % sequence identity, the other three groups consisted of one member (MNBS15, MNBS16 and MNBS17, respectively) with 28.5 % identity. All MNBS sequences were chategorized as non-TIR-NBS-LRR. Comparison and phylogenetic analysis of MNBS with other known RGA and R genes showed that deduced amino acid MNBSs shared 91.7-98.8 % identity with
Musa NBS-LRR and 19.9-35.5 % identity with known R genes. Among them MNBS17 shared 50.5 % identity with RGC2 (ABY75802) that assosiated to FOC race 4 resistance Musa species.
Keywords: banana, disease resistance, NBS, RGA
1
This chapter has been submitted to the Emirates Journal of Food and Agriculture (EJFA), entitled: Isolation and Characterization of Resistance Gene Analogue (RGA) from Fusarium Resistance Banana Cultivars
Pendahuluan
Penyakit layu Panama yang disebabkan oleh cendawan Fusarium
oxysporum f.sp. cubense (FOC) adalah salah satu penyakit yang sangat mematikan dan sulit dikendalikan, karena selain mudah disebarkan oleh air irigasi, tanah, peralatan pertanian dan bahan tanaman, juga keberadannya di dalam tanah mampu bertahan lebih dari 20 tahun tanpa kehilangan daya virulensinya (Agrios 2005). Penyakit layu FOC mempengaruhi 80 % produksi pisang dunia, termasuk pisang Cavendish dan subgrup plantain (Ploetz 2006). Pengendalian layu FOC menggunakan kultur praktis seperti penggunaan fungisida, perlakuan tanah, rotasi tanaman, pemanfaatan bahan organik, dapat mengurangi intensitas serangan, tetapi relatif sulit untuk diterapkan pada skala komersial (Pegg et al. 1993), oleh karena itu penggunaan kultivar tahan merupakan alernatif terbaik untuk mengatasi masalah penyakit layu tersebut. Pengembangan kultivar pisang dapat dilakukan melalui program pemuliaan konvensional yang melibatkan spesies atau kultivar tahan. Namun demikian hal tersebut menghadapi kendala sterilitas bunga dan ploidi dari tanaman pisang (Roux et al. 2004). Program pemuliaan melalui teknik rekayasa genetika merupakan teknik yang menjanjikan, tetapi sampai saat ini gen ketahanan pada tanaman pisang untuk ketahan terhadap layu FOC ras 4 tropika masih belum ditemukan.
Sebagian besar gen ketahanan (R gene) yang telah teridentifikasi menyandikan protein reseptor yang mengandung domain nucleotide binding site dan leucine rich repeat (NBS-LRR) yang menghasilkan ketahanan terhadap hama dan penyakit tanaman yaitu serangga hama, cendawan, bakteri, virus dan nematoda (Dangl & Jones 2001). Protein reseptor NBS-LRR mengenali protein efektor patogen dan menghasilkan sinyal transduksi yang akan menstimulir ekspresi pertahanan terhadap patogen (Caplan et al. 2008). Komposisi gen NBS-LRR terdiri dari beberapa domain yaitu N-terminal, nucleotide binding site (NBS), dan C-terminal LRR. Khusus domain NBS mengandung motif p-loop atau kinase-1, kinase-2, kinase-3a and hydrophobic (GLPL), yang sangat terkonservasi pada berbagai ATP-/GTP-binding protein dan berbagai organisme (Traut 1994).
Berdasarkan motif yang ada pada bagian N-terminal, gen R kelas NBS-LRR terbagi dalam dua subkelas. Subkelas I, TIR-NBS-NBS-LRR, dimana pada bagian
N-terminal berhomologi dengan protein Drosophila Toll dan mammalian
Interleukin-1 receptor (TIR) (Meyers et al. 1999), hanya ditemukan pada spesies tanaman dikotil dan gymnospermeae (Tarr & Alexander 2009). Subkelas II yaitu non-TIR-NBS-LRR, mempunyai motif coiled-coil (CC) pada bagian N-terminal, terdapat pada spesies tanama dikotil dan monokotil (McHale et al. 2006).
Berdasarkan domain NBS-LRR dari gen R yang telah teridentifikasi, dapat didisain primer degenerate untuk mengisolasi resistance gene analoque (RGA) dari berbagai tanaman seperti kedelai (Kanazin et.al. 1996), slada (Shen et al. 1998), apel liar (Baek & Choi 2013), dan lentil (Yaish et al. 2004) menggunakan pendekatan teknik PCR.
Secara umum tujuan dari penelitian ini adalah untuk mengisolasi dan mengkarakterisasi resistance gene analogue (RGA) yang berasal dari tiga kultivar tahan penyakit layu FOC. Secara khusus masing-masing kegiatan bertujuan untuk: (1) mengamplifikasi fragmen RGA dari DNA genom tanaman pisang tahan layu FOC menggunakan degenerate primer, (2) mengkloning dan sekuensing
produk PCR hasil amplifikasi, dan (3) menganalisis sekuen produk PCR berupa analisis BLAST, pensejajaran dan filogenetik.
Bahan dan Metode Waktu dan Tempat
Penelitian dilaksanakan mulai bulan November 2011 sampai September 2012 di laboratorium Pemuliaan dan Biologi Molekuler Tanaman, Departemen Agronomi dan Hortikultura, Fakultas Pertanian IPB.
Bahan Penelitian dan isolasi DNA
Kultivar yang digunakan adalah dua kultivar tahan hasil kegiatan “Uji Dini Ketahanan Beberapa Kultivar Pisang Terhadap Penyakit Layu Fusarium
oxysporum f.sp. cubense VCG 01213/16 (TR4)”, yaitu Calcuta-4 (AAw) dan Klutuk Wulung (BB), dan hasil penelitian sebelumnya yang dilakukan oleh Balai Penelitian Tanaman Buah Tropika, yaitu Rejang (AA) (Molina et al. 2010). Bahan tanaman yang digunakan adalah daun muda tanaman hasil perbanyakan kultur jaringan dan isolasi DNA berdasarkan metode CTAB (Doyle & Doyle 1987) yang dimodifikasi oleh Das et al. (2009). Prosedur isolasi DNA secara detail ditampilkan pada Lampiran 2. Selanjutnya stok DNA disimpan dalam lemari pendingin bersuhu -20 °C sampai siap digunakan.
Perancangan Primer dan PCR
Empat pasang kombinasi degenerate primer dirancang berdasarkan NBS-LRR yang mengandung domain P-loop/kinase-1, kinase-2, kinase-3a dan
hydrophobic digunakan untuk mengamplifikasi fragmen NBS-LRR asal DNA genom tanaman pisang. Kombinasi pasangan primer tersebut adalah F5(F)+F6(R), F9(F)+F6(R), F5(F)+F10(R), dan F9(F)+F10(R). Informasi sekuen dari degenerate
primer tersebut ditampilkan pada Tabel 3.
Reaksi PCR dilaksanakan dengan volume 25 µl menggunakan KAPA2GTM PCR Kit (Kapa Biosystems Inc., USA), yang komposisinya terdiri atas 5.0 µl 5 X buffer PCR (di dalamnya terkandung 1.5 mM Mg2+), 0.5 µl MgCl2 25 mM, 0.5 µl
dNTPs 10 mM, 1.0 µl masing-masing primer 10 µM (primer forward dan reverse), 30 ng DNA genom, 0.1 µ l Taq DNA polymerase (5 U µl-1) dan15.4 µl ddH2O.
Tabel 3 Degenerate primer yang digunakan untuk mengamplifikasi NBS-LRR asal DNA genom tanaman pisang
Kode Sekuen Domain
terkonservasi Pustaka F5(F) GGIGGIGTIGGIAAIACIAC P-loop Peraza-Echeverria
et al. 2008 F6(R) AAGIGCTAAGIGGIAAGICC hydrophobic
domain/GLPL
Peraza-Echeverria
et al. 2008 F9(F) GGNGGNRTIGGIAARACIAC P-loop Sun et al. 2009 F10(R) GAGGGCNARNGGNAAICC hydrophobic
domain/GLPL
Sun et al. 2009 Keterangan : I= inosine; R= A/G; N=A/C/G/T
Proses PCR menggunakan mesin GeneAmp® PCR System 2400 (Perkin Elmer). Denaturasi cetakan DNA pada awal reaksi pada suhu 95 °C selama 3 menit, diikuti dengan 35 kali siklus 95 °C selama 10 detik, 55 °C selama 10 detik dan 72 °C selama 3 detik, dan diakhiri dengan satu siklus 72 °C selama 10 menit. Produk PCR dipisahkan berdasarkan ukuran dengan menggunakan elektroforesis gel agarose 1 % pada mesin elektroforesis dengan tegangan 80 V selama 25 menit.
Purifikasi Produk PCR
Sebelum dilakukan kloning, produk PCR terlebih dahulu dipurifikasi menggunakan Gel/PCR DNA Fragments Extraction Kit (Geneaid, Taiwan) dengan prosedur standar dari perusahaan tersebut. Sebanyak 100 μl produk PCR dimasukkan ke dalam tabung mikrosentrifus 1.5 ml. Kedalam tabung tersebut ditambah larutan DF buffer dengan volume lima kali volume sampel, dikocok dengan baik menggunakan vortex, kemudian dimasukkan ke dalam DF column dan disentrifus 1.1 x 104 rpm selama 30 detik. Filtrat yang terkumpul dibuang. Ke dalam bagian tengah DF column ditambah 600 μl wash buffer dan dibiarkan selama satu menit. Selanjutnya tabung disentrifus 1.1 x 104 rpm selama 30 detik dan filtrat yang terkumpul dibuang. Tabung disentrifus lagi dengan kecepatan 1.1 x 104 rpm selama tiga menit untuk mengering matrik kolom. DF column dipindah ke tabung mikrosentrifus 1.5 ml yang baru, diberi 50 μl elution buffer dan dibiarkan selama 20 sampai 30 menit. Selanjutnya tabumg disentrifus dengan kecepatan 1.1 x 104 rpm selama dua menit untuk mendapatkan produk PCR murni.
Kloning Fragmen RGA
Sebelum dilakukan proses kloning, terlebih dahulu harus disiapkan sel kompeten dari E. coli strain DH5α. Pembuatan sel kompeten mengadopsi metode modifikasi buffer CaCl2 yang dikembangkan oleh Li et al. (2010).
Produk PCR dikloning ke dalam vektor pGEM-T® EasyVector (Promega, Madison, WI, USA), dengan protokol standar sesuai dengan petunjuk perusahaan. Reaksi ligasi berisi: 5 μl buffer ligase, 1 μl pGEM-T® Easy Vector, 1 μl T4 DNA ligase, 2.5 μl produk PCR (10 ng μl-1), 0.5 μl air bebas nuclease dicampur dan disimpan pada suhu 4 °C selama satu malam.
Selanjutnya vektor ditransformasi ke sel kompeten bakteri E. coli strain DH5-α pada keesokan harinya. Tabung berisi sel kompeten yang sebelumnya disimpan dalam freezer, di-thawing dengan meletakkan pada wadah berisi es selama lebih kurang lima menit. Sebanyak 50 μl sel kompeten tersebut secara hati-hati diambil dan dituang ke dalam tabung yang berisi reaksi ligasi. Dengan menggunakan jari, tabung tersebut dijentik agar larutan tercampur dengan sempurna, dan diletakkan dalam es selama 20 menit.
Selanjutnya dilakukan heat shock dengan meletakkan tabung ke dalam air hangat dengan suhu 42 °C selama 45 sampai 50 detik, dan segera dipindah ke wadah berisi es dan dibiarkan selama dua menit. Sebanyak 900 μl media SOB dituangkan ke dalam tabung yang berisi sel yang telah ditransformasi, kemudian digoyang (shaker) pada kecepatan 150 rpm dan diinkubasi pada tempat yang bersuhu 37 °C selama 45 sampai 90 menit.
Setiap 100 μl larutan berisi sel yang telah ditransformasi, disebar secara merata ke cawan petri yang berisi media padat LB/ampicillin/IPTG/X-Gal dan diinkubasi pada suhu 37 °C selama satu malam.
Seleksi koloni dan purifikasi plasmid
Koloni bakteri transforman dipilih yang berwarna putih dan disubkultur ke tabung Falcon yang berisi media LB cair yang ditambah ampicilin (100 mg l-1) sebanyak 4 sampai 5 ml, diinkubasi pada suhu kamar dan digoyang (shaker) selama satu malam. Sebelum dikirim ke 1stBASE (Malaysia) untuk proses perunutan basa nukleotida (DNA sequencing), plasmid dipurifikasi menggunakan High-Speed Plasmid Mini Kit (Geneaid, Taiwan). Suspensi bakteri dipindah ke tabung 1.5 ml dan disentrifus 1.1 x 104 rpm selama satu menit. Supernatan dibuang dan pelet yang terbentuk ditambah lagi dengan sisa suspensi dari tabung Falcon, dan disentrifus lagi. Pelet yang terbentuk ditambah dengan 200 μl PD1
buffer dan divortex agar homogen. Selanjutnya ditambah dengan 200 μl PD2
buffer dan tabung dibalik sebanyak 10 kali agar larutan tercampur dengan sempurna dan didiamkan selama dua menit. Ke dalam tabung dimasukkan PD3
buffer sebanyak 300 μl dan segera dibalik sebanyak 10 kali. Tabung disentrifus 11000 rpm selama tiga menit, supernatan diambil secara hati-hati dan dipindahkan ke PD column yang diletakkan pada tabung 2 ml, dan disentrifus kembali dengan kecepatan 1.1 x 104 rpm selama 30 detik. Filtrat yang terkumpul pada tabung 2 ml dibuang dan PD column diletakkan kembali, ditambah 600 μl wash buffer dan disentrifus 1.1 x 104 rpm selama 30 detik. Selanjutnya dikeringkan dengan cara disentrifus lagi selama tiga menit. Kemudian PD column yang telah kering diletakkan pada tabung 2 ml yang baru. Tepat di bangian tengah PD column ditambah 50 μl
elution buffer, didiamkan selama 20 menit dan disentrifuse 1.1 x 104 rpm selama dua menit untuk mendapatkan DNA plasmid yang murni.
Konfirmasi adanya insersi
Sebelum dikirim ke 1stBASE (Malaysia) untuk proses perunutan DNA, dilakukan konfirmasi adanya insersi dengan melakukan PCR menggunakan pasangan primer F9+F6. Sebelum PCR, plasmid DNA yang sudah dipurifikasi perlu diencerkan sepuluh kali, untuk digunakan sebagai cetakan DNA. Hasil yang positif ditandai dengan terbentuknya pita tunggal dengan ukuran tertentu pada hasil elektroforesis, dikirim ke 1stBASE (Malaysia) untuk proses perunutan basa (DNA sequencing).
Analisis Sekuen DNA
Sekuen DNA dari vektor dipisahkan secara manual dari hasil proses perunutan DNA klon produk PCR. Sekuen produk PCR yang telah teridentifikasi dianalisis dengan cara membandingkan sekuen DNA dan prediksi asam amino produk PCR tersebut dengan aksesi yang terdeposit dalam pangkalan data GenBank menggunakan alogaritma BLASTn dan BLASTp (http://blast.ncbi.nlm. nih.gov/Blast.cgi). Translasi runutan DNA produk PCR menjadi asam amino menggunakan perangkat lunak Geneious Pro 5.6.6 versi percobaan (Biomatters, USA). Perangkat lunak ClustalW2
(Larkin et al. 2007) digunakan untuk analisis pensejajaran runutan prediksi asam amino
dari produk PCR dengan R-gene yang berasal dari tanaman lain, yaitu Arabidopsis thaliana: RPP1 (AAC72977) dan RPP5 (AAF08790), tomat: Prf (LEU65391) dan Mi-1,2 (AAC67238), lada: Bs2 (AF202179), kentang: Gpa2 (AF195939), gandum: Yr10 (AF149114), flax: L6 (Q40253), M (AAB47618), tembakau: N (Q40253), dan RGA asal pisang, yaitu RGC3 (ABY75803), RGA-J (ACK44408), RGC2 (ABY75802),
perangkat lunak GeneDoc (Nicholas
metode Neighbour-Joining dengan bantuan perangkat lunak
2011). Nilai bootstrap
filogram.
Amplifikasi PCR Menggunakan
Dari empat pasang
daerah conserved domain
terbaik adalah F9(F)+F6
tunggal yang cukup jelas
Calcuta-4 dan Klutuk Wulung) menghasilkan pita lebih dari satu
~1100 pb (Gambar 10
ukuran spesifik untuk
kombinasi pasangan primer F5
produk amplifikasi. Produk PCR ketiga kultivar yang dihasilkan oleh pasangan primer F9(F)+F6(R) digunakan untuk proses anal
Analisis Sekuen Fragmen Produk PCR
Dari 95 sampel produk PCR yang telah dikloning, sebanyak 91 klon mengandung insersi yang berukuran antara 68
asam amino, ditemukan sebanyak 16 sekuen dengan ukuran 523
mengandung stop codon
mengandung motif terkonservasi P
hydrophobic (GLPL). Selanjutnya 16 sekuen tersebut disandi dengan MNBS16.
Untuk mengetahui identitas 75 sekuen sisanya, dilakuka BLAST terhadap genom pisang (
diperoleh sebanyak 18 klon yang beberapa basanya berhomolog dengan basa cDNA yang ada pada pangkalan data genom pisang (data tidak dita
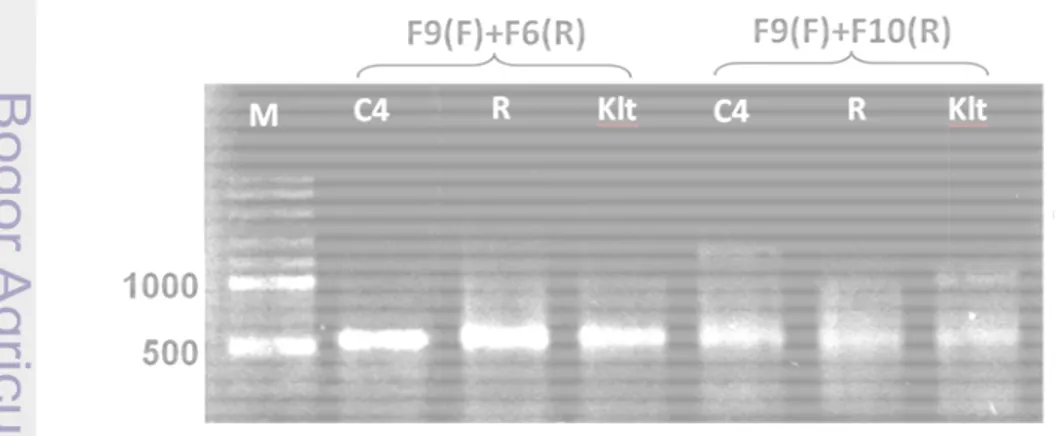
Gambar 10 Elektroferogram primer pada ladder 100 pb, C4=
GeneDoc (Nicholas et al. 1997). Analisis filogenetik menggunakan Joining dengan bantuan perangkat lunak MEGA 5 (
bootstrap 1000 kali pengulangan digunakan untuk menguji keakuratan
Hasil dan Pembahasan
Menggunakan Degenerate Primer
Dari empat pasang degenerate primer yang dicoba untuk mengamplifikasi
conserved domain p-loop sampai GLPL, diperoleh kombinasi pasangan
+F6(R). Pasangan primer tersebut menghasilkan
yang cukup jelasdengan ukuran ~550 pb untuk ketiga kultivar pisang (Rejang, 4 dan Klutuk Wulung), sedangkan pasangan primer F9
menghasilkan pita lebih dari satu, dengan produk berukuran antara ~550 pb sampai 10). Satu pita amplikon yang berukuran ~550 pb
motif P-loop sampai GLPL dari NBS. Namun demikian, dua asangan primer F5(F)+F6(R) dan F5(F)+F10(R) tidak menghasilkan Produk PCR ketiga kultivar yang dihasilkan oleh pasangan primer F9(F)+F6(R) digunakan untuk proses analisis selanjutnya.
Sekuen Fragmen Produk PCR dari DNA Genom Pisang
Dari 95 sampel produk PCR yang telah dikloning, sebanyak 91 klon mengandung insersi yang berukuran antara 68-959 pb. Berdasarkan analisis predisksi asam amino, ditemukan sebanyak 16 sekuen dengan ukuran 523-638
stop codon dan mempunyai ciri RGA kelas NBS
mengandung motif terkonservasi P-loop (GG[V/M]GKTT), kinase
-(GLPL). Selanjutnya 16 sekuen tersebut disandi dengan Untuk mengetahui identitas 75 sekuen sisanya, dilakuka BLAST terhadap genom pisang (http://banana-genome.cirad.fr/
diperoleh sebanyak 18 klon yang beberapa basanya berhomolog dengan basa cDNA yang ada pada pangkalan data genom pisang (data tidak dita
Elektroferogram hasil amplifikasi menggunakan dua pasang
primer pada pada cetakan DNA genom 3 kultivar pisang. M=DNA 100 pb, C4=Calcuta-4, R=Rejang dan Klt=Klutuk Wulung
nalisis filogenetik menggunakan MEGA 5 (Tamura et al.
an digunakan untuk menguji keakuratan
yang dicoba untuk mengamplifikasi loop sampai GLPL, diperoleh kombinasi pasangan primer
(R). Pasangan primer tersebut menghasilkan pita amplikon
dengan ukuran ~550 pb untuk ketiga kultivar pisang (Rejang, ngkan pasangan primer F9(F)+F10(R)
, dengan produk berukuran antara ~550 pb sampai
yang merupakan
dari NBS. Namun demikian, dua tidak menghasilkan Produk PCR ketiga kultivar yang dihasilkan oleh pasangan primer
dari DNA Genom Pisang
Dari 95 sampel produk PCR yang telah dikloning, sebanyak 91 klon 959 pb. Berdasarkan analisis predisksi 638 pb yang tidak
dan mempunyai ciri RGA kelas NBS-LRR, yaitu
-2, kinase-3a dan
(GLPL). Selanjutnya 16 sekuen tersebut disandi dengan MNBS1-Untuk mengetahui identitas 75 sekuen sisanya, dilakukan analisis
genome.cirad.fr/) dan
diperoleh sebanyak 18 klon yang beberapa basanya berhomolog dengan basa cDNA yang ada pada pangkalan data genom pisang (data tidak ditampilkan).
menggunakan dua pasang kombinasi
kultivar pisang. M=DNA Klutuk Wulung.
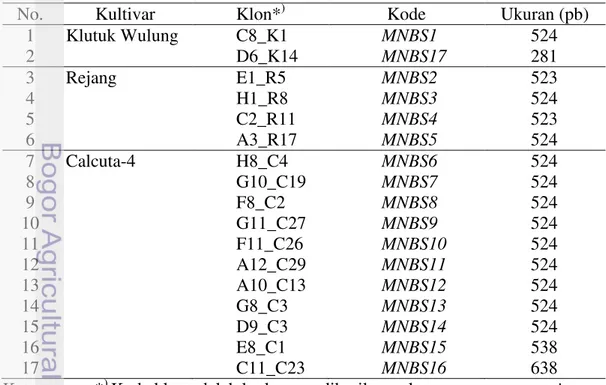
Terdapat satu klon dari 18 klon tersebut, yaitu D6_K14 berukuran 281 pb, dengan 90.78 % nukleotida penyusunnya (query coverage) mempunyai identity sebesar 98.44 % terhadap putative disease resistance protein RGA3, dan mempunyai motif terkonservasi P-loop dan kinase2a. Tidak lengkapnya struktur klon D6_K14 untuk sebuah fragmen NBS kemungkinan disebabkan ligasi fragmen DNA pada plasmid pGem-T® kurang sempurna akibat proses pengkloningan terjadi secara random. Untuk keperluan analisis selanjutnya klon D6_K14 diberi kode MNBS17. Tujuh belas fragmen RGA tersebut mempunyai identity sekuen DNA sebesar 52.2 % sampai 98.5 % dan sekuen prediksi asam amino sebesar 18.3 % sampai 100 %. Runutan basa nukleotida dan prediksi asam amino dari MNBS1, MNBS15, MNBS16 dan MNBS17 ditampilkan pada Gambar 11, 12, 13 dan 14.
Berdasarkan asal klon, dua fragmen RGA berasal dari DNA genom Klutuk Wulung, yaitu MNBS1 dan MNBS17, empat RGA berasal dari Rejang, yaitu MNBS2-MNBS5, dan 11 RGA berasal dari Calcuta-4 (Tabel 4).
Analisis Keragaman RGA yang Berasal dari DNA Genom Pisang
Berdasarkan hasil analisis filogenetik sekuen prediksi asam amino fragmen RGA yang didapatkan dari penelitian ini, menunjukan bahwa 17 RGA tersebut terbagi menjadi empat kelompok. Fragmen dengan identity >97 % disebut identik atau berada dalam satu kelompok. Kelompok pertama beranggotakan 14 RGA yaitu MNBS1–MNBS14 yang mempunyai identity 98 %, dimana satu RGA berasal dari Klutuk Wulung (MNBS1), empat RGA berasal dari Rejang (MNBS2-MNBS5), dan sembilan RGA berasal dari Calcuta-4
(MNBS6-MNBS14). Tiga fragmen sisanya yang mempunyai identity sebesar 28.5 % dibagi menjadi tiga kelompok yang masing-masing beranggotakan satu fragmen. Tabel 4 Pengelompokan fragmen RGA dari produk PCR berdasarkan asal DNA
genom diisolasi
No. Kultivar Klon*) Kode Ukuran (pb)
1 Klutuk Wulung C8_K1 MNBS1 524 2 D6_K14 MNBS17 281 3 Rejang E1_R5 MNBS2 523 4 H1_R8 MNBS3 524 5 C2_R11 MNBS4 523 6 A3_R17 MNBS5 524 7 Calcuta-4 H8_C4 MNBS6 524 8 G10_C19 MNBS7 524 9 F8_C2 MNBS8 524 10 G11_C27 MNBS9 524 11 F11_C26 MNBS10 524 12 A12_C29 MNBS11 524 13 A10_C13 MNBS12 524 14 G8_C3 MNBS13 524 15 D9_C3 MNBS14 524 16 E8_C1 MNBS15 538 17 C11_C23 MNBS16 638
Kelompok kedua dengan anggota MNBS16 dari Calcuta
disebut kelompok keempat. Hasil analisis ditampilkan pada Gambar 15.
Gambar 11 Sekuen DNA dan prediksi asam amino fragmen kelompok I)
Wulung. Huruf kapital di bawah sekuen DNA adalah sekuen prediksi asam amino. Residu asam amino di dalam kotak adalah motif terkonservasi dari
hydrophobic domain
Gambar 12 Sekuen DNA dan prediksi asam amino fragmen hasil amplifikasi PCR asal DNA genom pisang Calcuta
di bawah sekuen DNA adalah sekuen prediksi asam amino. Residu asam amino di dalam kotak adalah motif terkonservasi dari
P-loop, kinase
Kelompok kedua dengan anggota MNBS15 dan kelompok ketiga yang anggotanya Calcuta-4, sedangkan MNBS17 yang berasal dari Klutuk Wulung disebut kelompok keempat. Hasil analisis filogenetik 17 fragmen RGA tersebut ditampilkan pada Gambar 15.
Sekuen DNA dan prediksi asam amino fragmen MNBS1
kelompok I) hasil amplifikasi PCR asal DNA genom pisang Klutuk Wulung. Huruf kapital di bawah sekuen DNA adalah sekuen prediksi asam amino. Residu asam amino di dalam kotak adalah motif terkonservasi dari NBS, yaitu P-loop, kinase-2, kinase
hydrophobic domain (GLPL)
Sekuen DNA dan prediksi asam amino fragmen MNBS15 hasil amplifikasi PCR asal DNA genom pisang Calcuta
di bawah sekuen DNA adalah sekuen prediksi asam amino. Residu asam amino di dalam kotak adalah motif terkonservasi dari
loop, kinase-2, kinase-3a dan hydrophobic domain (GLPL
yang anggotanya yang berasal dari Klutuk Wulung filogenetik 17 fragmen RGA tersebut
MNBS1 (mewakili
hasil amplifikasi PCR asal DNA genom pisang Klutuk Wulung. Huruf kapital di bawah sekuen DNA adalah sekuen prediksi asam amino. Residu asam amino di dalam kotak adalah motif , kinase-3a dan
MNBS15 (kelompok II)
hasil amplifikasi PCR asal DNA genom pisang Calcuta-4. Huruf kapital
di bawah sekuen DNA adalah sekuen prediksi asam amino. Residu asam amino di dalam kotak adalah motif terkonservasi dari NBS, yaitu
Gambar 13 Sekuen DNA dan prediksi asam amino fragmen
hasil amplifikasi PCR asal DNA genom pisang
di bawah sekuen DNA adalah sekuen prediksi asam amino. Residu asam amino di dalam kotak adalah motif terkonservasi dari
kinase-2, kinase
Gambar 14 Sekuen DNA dan prediksi asam
IV) hasil amplifikasi PCR asal DNA genom pisang Klutuk Wulung. Huruf kapital di bawah sekuen DNA adalah sekuen prediksi asam amino. Residu asam amino di dalam kotak adalah
terkonservasi dari
Analisis Sekuen DNA dan Prediksi Asam Amino Fragmen RGA
Berdasarkan hasil analisis BLASTn (
bahwa sekuen DNA dari 17 fragmen RGA yang dihasilkan dari penelitian ini
mempunyai identity yang tinggi dengan sekuen DNA resistance protein yang terdapat pada pangkalan data
dengan persentase identity
MNBS14 mempunyai identity
EU855841 dan DQ119306,
EF488472, MNBS16 mempunyai
mempunyai identity 99 % dengan aksesi
Sekuen DNA dan prediksi asam amino fragmen MNBS16 (kelompok III) amplifikasi PCR asal DNA genom pisang Calcuta-4. Huruf kapital di bawah sekuen DNA adalah sekuen prediksi asam amino. Residu asam amino di dalam kotak adalah motif terkonservasi dari NBS, yaitu P
, kinase-3a dan hydrophobic domain (GLPL).
Sekuen DNA dan prediksi asam amino fragmen MNBS17 (kelompok hasil amplifikasi PCR asal DNA genom pisang Klutuk Wulung. Huruf kapital di bawah sekuen DNA adalah sekuen prediksi asam amino. Residu asam amino di dalam kotak adalah sebagian terkonservasi dari NBS, yaitu P-loop dan kinase-2.
Analisis Sekuen DNA dan Prediksi Asam Amino Fragmen RGA
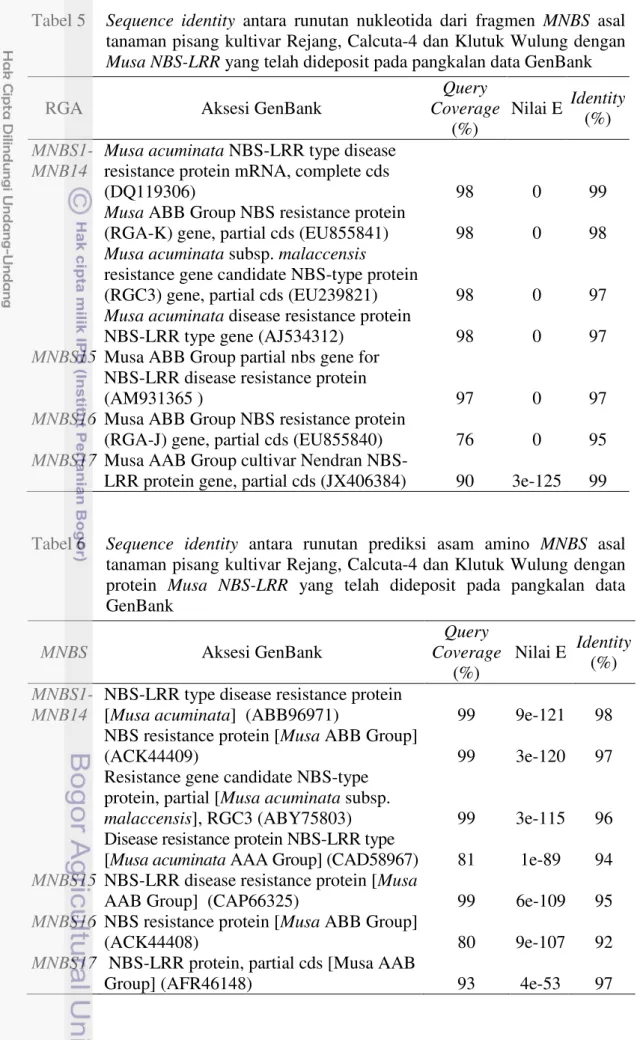
Berdasarkan hasil analisis BLASTn (Altschul et al. 1997) menunjukkan bahwa sekuen DNA dari 17 fragmen RGA yang dihasilkan dari penelitian ini yang tinggi dengan sekuen DNA Musa NBS-LRR disease
yang terdapat pada pangkalan data GenBank, ditunjukkan
identity dalam Tabel 5. Sekuen DNA fragmen
identity 97-99 % dengan aksesi AJ534312, EU239
EU855841 dan DQ119306, MNBS15 mempunyai identity 97 % dengan aksesi mempunyai identity 95 % aksesi EU855840, dan
% dengan aksesi JX406384 (Tabel 5).
(kelompok III) . Huruf kapital di bawah sekuen DNA adalah sekuen prediksi asam amino. Residu asam NBS, yaitu P-loop,
(kelompok hasil amplifikasi PCR asal DNA genom pisang Klutuk Wulung. Huruf kapital di bawah sekuen DNA adalah sekuen prediksi asam sebagian motif
) menunjukkan bahwa sekuen DNA dari 17 fragmen RGA yang dihasilkan dari penelitian ini
LRR disease
, ditunjukkan dalam Tabel 5. Sekuen DNA fragmen MNBS1
-EU239821,
% dengan aksesi dan MNBS17
Gambar 15 Dendogram hasil analisis filogenetik sekuen prediksi asam amino
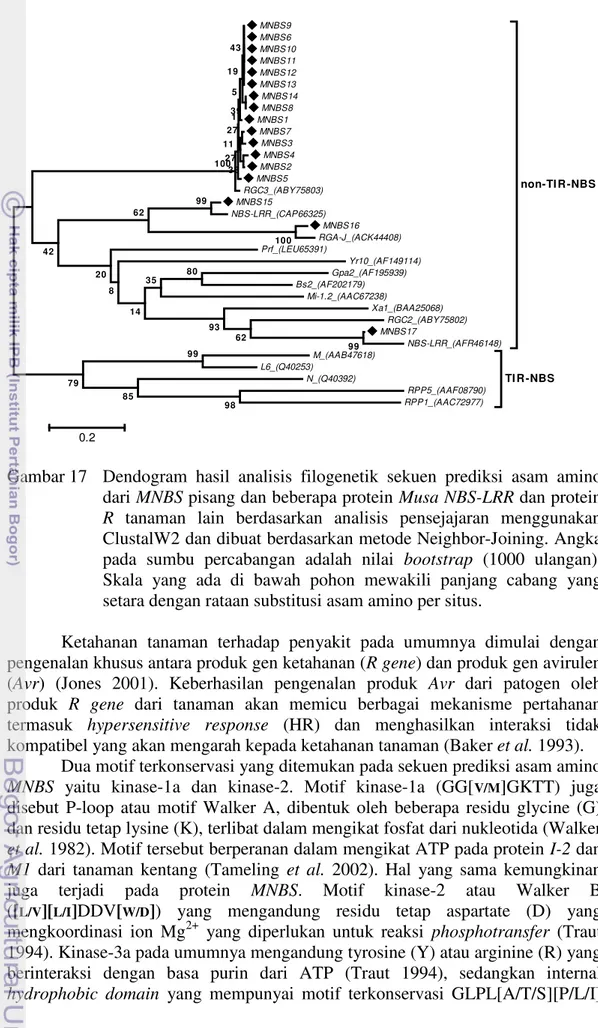
MNBS berdasarkan metode Neighbor-Joining. Angka pada sumbu percabangan adalah nilai bootstrap (1000 ulangan). Cabang yang diberi label I, II, III dan IV mewakili kelompok dari RGA. Skala yang ada di bawah pohon mewakili panjang cabang yang setara dengan rataan substitusi asam amino per situs.
Hal yang sama juga diperoleh dari hasil analisis BLASTp (Altschul et al. 1997) pada residu prediksi asam amino asal 17 fragmen RGA (MNBS1-MNBS17). Prediksi asam amino MNBS1-MNBS14 mempunyai identity sebesar 94 % sampai 98 % dengan aksesi CAD58967, ABY75803, ACK44409 dan ABB96971, sedangkan prediksi asam amino dari MNBS15 mempunyai identity 95 % dengan aksesi CAP66325, prediksi asam amino MNBS16 mempunyai identity sebesar 92 % dengan aksesi ACK44408, dan prediksi asam amino MNBS17 mempunyai
identity sebesar 97 % dengan aksesi AFR46148 (Tabel 6).
Dari 17 sekuen RGA yang dihasilkan dari penelitian ini, 16 RGA yaitu
MNBS1–MNBS16 telah dideposit di pangkalan data GenBank dengan nomor aksesi KF034945, KF034946, KF034947, KF034948, KF034949, KF034950, KF034951, KF034952, KF034953, KF034954, KF034955, KF034956, KF034957, KF034958, KF034959, KF034960.
Analisis Pensejajaran Sekuen Prediksi Asam Amino Fragmen MNBS
Untuk mengetahui konsensus asam amino MNBS, dilakukan analisis pensejajaran runutan prediksi asam amino 17 fragmen MNBS dengan protein Musa
NBS-LRR dan protein R yang telah terdeposit pada pangkalan data GenBank.
MNBS2 MNBS4 MNBS3 MNBS7 MNBS5 MNBS8 MNBS14 MNBS13 MNBS12 MNBS11 MNBS6 MNBS9 MNBS10 MNBS1 MNBS15 MNBS16 MNBS17 53 84 36 40 39 9 19 100 13 36 7 3 12 4 0.1 I II III IV
Tabel 5 Sequence identity antara runutan nukleotida dari fragmen MNBS asal tanaman pisang kultivar Rejang, Calcuta-4 dan Klutuk Wulung dengan
Musa NBS-LRR yang telah dideposit pada pangkalan data GenBank
RGA Aksesi GenBank
Query Coverage (%) Nilai E Identity (%) MNBS1-MNB14
Musa acuminata NBS-LRR type disease resistance protein mRNA, complete cds
(DQ119306) 98 0 99
Musa ABB Group NBS resistance protein
(RGA-K) gene, partial cds (EU855841) 98 0 98
Musa acuminata subsp. malaccensis
resistance gene candidate NBS-type protein
(RGC3) gene, partial cds (EU239821) 98 0 97
Musa acuminata disease resistance protein
NBS-LRR type gene (AJ534312) 98 0 97
MNBS15 Musa ABB Group partial nbs gene for NBS-LRR disease resistance protein
(AM931365 ) 97 0 97
MNBS16 Musa ABB Group NBS resistance protein
(RGA-J) gene, partial cds (EU855840) 76 0 95
MNBS17 Musa AAB Group cultivar Nendran
NBS-LRR protein gene, partial cds (JX406384) 90 3e-125 99
Tabel 6 Sequence identity antara runutan prediksi asam amino MNBS asal tanaman pisang kultivar Rejang, Calcuta-4 dan Klutuk Wulung dengan protein Musa NBS-LRR yang telah dideposit pada pangkalan data GenBank MNBS Aksesi GenBank Query Coverage (%) Nilai E Identity (%) MNBS1-MNB14
NBS-LRR type disease resistance protein
[Musa acuminata] (ABB96971) 99 9e-121 98
NBS resistance protein [Musa ABB Group]
(ACK44409) 99 3e-120 97
Resistance gene candidate NBS-type protein, partial [Musa acuminata subsp.
malaccensis], RGC3 (ABY75803) 99 3e-115 96
Disease resistance protein NBS-LRR type
[Musa acuminata AAA Group] (CAD58967) 81 1e-89 94
MNBS15 NBS-LRR disease resistance protein [Musa
AAB Group] (CAP66325) 99 6e-109 95
MNBS16 NBS resistance protein [Musa ABB Group]
(ACK44408) 80 9e-107 92
MNBS17 NBS-LRR protein, partial cds [Musa AAB
Hasil pensejajaran asam amino MNBS bersama lima RGA pisang dan sepuluh R-gene tanaman yang telah diketahui, yaitu gen N dari tembakau, Prf dan
Mi-1.2 dari tomat, Gpa2 dari kentang, Bs2 dari lada, L6 dan M dari flax, RPP1 dan RPP2 dari Arabidopsis dan Yr10 dari gandum, ditampilkan pada Gambar 16.
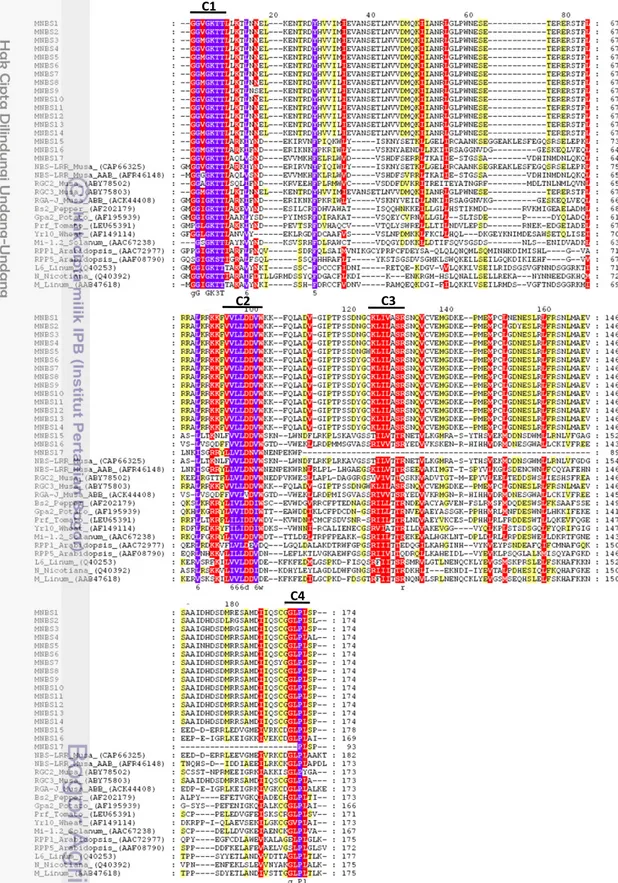
Berdasarkan hasil pensejajaran prediksi asam amino fragmen MNBS dan protein R tanaman lain terlihat bahwa motif utama NBS-LRR seperti P-loop/kinase-1a (G[V/M/I]GKTT), kinase-2 ([L/V][L/I]DDVW/D), RNBS-B/kinase-3a dan
hydrophobic domain (GLPL) sangat terkonservasi pada semua sekuen prediksi asam amino MNBS, Musa NBS-LRR dan protein R tanaman lain, seperti: protein
Prf , Mi-1,2, Bs2, Gpa2, Yr10, RPP1, RPP5, L6, M, dan N. Diantara
protein-protein tersebut RPP1, RPP5, L6, M dan N termasuk dalam kelas TIR-NBS-LRR,
sedangkan yang lainnya termasuk dalam kelas non-TIR-NBS-LRR. Perbedaan antara TIR-NBS-LRR dan non-TIR-NBS-LRR terdapat pada motif sekuen asam amino yang ada pada daerah N-terminal dan residu asam amino terakhir dari motif kinase-2. Residu terkahir dari kinase-2 pada TIR-NBS-LRR adalah tryptophan (W), sedangkan pada non-TIR-NBS-LRR adalah aspartate (D) (Tarr & Alexander 2009). Selain itu analisis pensejajaran juga menghasilkan matrik genetic identity yang ditampilkan pada Tabel 7. Matrik tersebut menunjukkan sequence identity satu aksesi terhadap aksesi lainnya.
Analisis Filogenetik Prediksi Asam Amino MNBS dengan Protein RGA dan
R-Gene Tanaman Lain
Hasil analisis filogenetik sekuen prediksi asam amino MNBS yang diperoleh dari penelitian ini, protein Musa NBS-LRR dan protein R tanaman lain yang terdeposit pada pangkalan data GenBank ditampilkan pada Gambar 17.
Hasil analisis menunjukkan bahwa protein R dan RGA (termasuk MNBS) terbagi dalam dua kelas, yaitu non-TIR-NBS-LRR dan TIR-NBS-LRR. Semua
MNBS dan tanaman monokotil lainnya (gandum dan padi) termasuk dalam kelas non-TIR-NBS-LRR. Dari hasil penelitian sebelumnya tentang NBS-LRR, pada tanaman monokotil hanya ditemukan non-TIR-NBS-LRR (Bai et al. 2002), sedangkan pada tanaman dikotil selain non-NBS-LRR juga ditemukan TIR-NBS-LRR (Pan et al. 2000b).
Selanjutnya kelas non-TIR-NBS-LRR dibagi menjadi dua kelompok. Bersama dengan RGC3 Musa (ABY78503), MNBS1-MNBS14 berada dalam kelompok I dengan identity sebesar 96 % sampai 98 %, sedangkan MNBS15,
MNBS16 dan MNBS17 berada dalam kelompok II bersama-sama dengan NBS-LRR
Musa (CAP66325) dan RGA-J Musa (ACK44408) serta BS2, GPA2, Mi-1,2, Prf dan Yr10 dengan identity sebesar 18 % sampai 95 %. Salah satu hal yang menarik adalah MNBS17 mempunyai identity sebesar 50.5 % dengan RGC2 (ABY75802), dimana RGC2 adalah RGA yang diisolasi dari tanaman pisang tahan FOC ras 4 dan terekspresi pada saat tanaman pisang terinfeksi FOC ras 4 (Peraza-Echeverria et al. 2008).
Tanaman telah berhasil mengembangkan mekanisme pengenalan dan perlindungan terhadap patogen. Interaksi antara inang dan patogen akan memicu respon lokal maupun sistemik (Meyer et al. 2003). Gen ketahanan tanaman (R gene) menyandi protein R yang merupakan pertahanan pertama dari tanaman melawan serangan dari patogen Patogen yang dikenali oleh protein R biasanya bersifat spesifik terhadap inang spesifik (Friedman & Baker 2007).
Gambar 16 Analisis pensejajaran sekuen prediksi asam amino MNBS dengan beberapa protein Musa NBS-LRR dan protein R yang terdeposit pada GenBank. Daerah domain terkonservasi ditandai dengan garis di atas sekuen (C1=kinase-1a atau P-loop, C2=kinase-2, C3=kinase-3a, dan C4=hydropobic domain atau GLPL)
C1
C2
C4
Gambar 17 Dendogram hasil analisis filogenetik sekuen prediksi asam amino dari MNBS pisang dan beberapa protein Musa NBS-LRR dan protein
R tanaman lain berdasarkan analisis pensejajaran menggunakan ClustalW2 dan dibuat berdasarkan metode Neighbor-Joining. Angka pada sumbu percabangan adalah nilai bootstrap (1000 ulangan). Skala yang ada di bawah pohon mewakili panjang cabang yang setara dengan rataan substitusi asam amino per situs.
Ketahanan tanaman terhadap penyakit pada umumnya dimulai dengan pengenalan khusus antara produk gen ketahanan (R gene) dan produk gen avirulen (Avr) (Jones 2001). Keberhasilan pengenalan produk Avr dari patogen oleh produk R gene dari tanaman akan memicu berbagai mekanisme pertahanan termasuk hypersensitive response (HR) dan menghasilkan interaksi tidak kompatibel yang akan mengarah kepada ketahanan tanaman (Baker et al. 1993).
Dua motif terkonservasi yang ditemukan pada sekuen prediksi asam amino
MNBS yaitu kinase-1a dan kinase-2. Motif kinase-1a (GG[V/M]GKTT) juga disebut P-loop atau motif Walker A, dibentuk oleh beberapa residu glycine (G) dan residu tetap lysine (K), terlibat dalam mengikat fosfat dari nukleotida (Walker
et al. 1982). Motif tersebut berperanan dalam mengikat ATP pada protein I-2 dan
M1 dari tanaman kentang (Tameling et al. 2002). Hal yang sama kemungkinan juga terjadi pada protein MNBS. Motif kinase-2 atau Walker B
([L/V][L/I]DDV[W/D]) yang mengandung residu tetap aspartate (D) yang
mengkoordinasi ion Mg2+ yang diperlukan untuk reaksi phosphotransfer (Traut 1994). Kinase-3a pada umumnya mengandung tyrosine (Y) atau arginine (R) yang berinteraksi dengan basa purin dari ATP (Traut 1994), sedangkan internal
hydrophobic domain yang mempunyai motif terkonservasi GLPL[A/T/S][P/L/I]
MNBS9 MNBS6 MNBS10 MNBS11 MNBS12 MNBS13 MNBS14 MNBS8 MNBS1 MNBS7 MNBS3 MNBS4 MNBS2 MNBS5 RGC3_(ABY75803) MNBS15 NBS-LRR_(CAP66325) MNBS16 RGA-J_(ACK44408) Prf_(LEU65391) Yr10_(AF149114) Gpa2_(AF195939) Bs2_(AF202179) Mi-1.2_(AAC67238) Xa1_(BAA25068) RGC2_(ABY75802) MNBS17 NBS-LRR_(AFR46148) non-TI R -NBS M_(AAB47618) L6_(Q40253) N_(Q40392) RPP5_(AAF08790) RPP1_(AAC72977) TI R -NBS 99 100 98 99 99 62 93 85 80 62 35 14 79 8 20 42 33 27 10027 39 11 1 5 19 43 0.2
masih belum diketahui fungsinya. Walaupun demikian, melihat keberadaannya sangat terkonservasi di dalam sekuen R-gene kelas NBS-LRR, diduga domain tersebut mempunyai fungsi penting yang berhubungan dengan aktifitas gen yang bersangkutan (Jones & Jones 1997).
Tidak adanya TIR-NBS-LRR tidak hanya terjadi pada tanaman pisang (Musa spp.), tetapi juga dilaporkan pada tanaman monokotil lainnya seperti padi (Zhou et
al. 2004), gandum (Zhang et al. 2011), jagung (Xiao et al. 2006), barley (Madsen
et al. 2003), sorghum (Cheng et al. 2010), dan tebu (Que et al. 2009). Proses eliminasi TIR-NBS-LRR dari sebagian besar tanaman monokotil kemungkinan terjadi selama masa evolusi, walaupun belum ada informasi yang dapat menggambarkan bagaimana penjelasan dari proses eliminasi tersebut. Pan et al. (2000b) mengusulkan sebuah model yang menunjukkan evolusi NBS-LRR melibatkan dua tahapan. Selama tahap pertama, genom tanaman mempunyai sedikit NBS-LRR, dan selama tahap kedua baik TIR maupun non-TIR mengalami perkembangan dan diversifikasi pada spesies tanaman dikotil. Berdasarkan analisis sekuen yang dilakukan oleh Cannon et al. (2002), gen non-TIR-NBS tampaknya lebih beragam dari pada kelas TIR-NBS, dan hal ini memunculkan spekulasi bahwa non-TIR-NBS lebih dulu keberadaannya dibanding TIR-NBS. Namun demikian, hasil penelitian terbaru tentang evolusi NBS yang dilakukan oleh Yue et al. (2012) menunjukkan bahwa NBS yang ditemukan pada tanaman darat purba (Coleochaetales dan lumut-lumutan) lebih mirip dengan tipe TIR pada level sekuen dan hal ini dapat disimpulkan bahwa NBS tipe TIR telah berkembang terlebih dahulu bila dibandingkan dengan tipe non-TIR.
Berdasarkan matrik genetic identity pada Tabel 7, semua prediksi asam amino MNBS yang dihasilkan pada penelitian ini mempunyai identity sebesar 91.7.% sampai 98.8 % dengan protein Musa NBS-LRR yang terdeposit pada pangkalan data bank gen, dan 19.9 % sampai 35 % dengan protein R tanaman seperti Bs2, Gp2, Prf, Yr10 dan Mi-1.2. Hal tersebut mengindikasikan bahwa
MNBS kemungkinan mempunyai fungsi sebagai gen ketahanan. Diantara 17
MNBS tersebut, MNBS17 mempunyai identity sebesar 50.5 % dengan RGC2 (ABY75802). Ekspresi RGC2 berhubungan dengan ketahanan layu FOC ras 4 salah satu galur M. acuminata spp. malaccensis (Peraza-Echeverria et al. 2008). Hal ini dapat diasumsikan bahwa MNBS17 kemungkinan mempunyai fungsi ketahanan terhadap layu FOC pada tanaman pisang.
Pseudogenitas MNBS dalam Kromosom Pisang
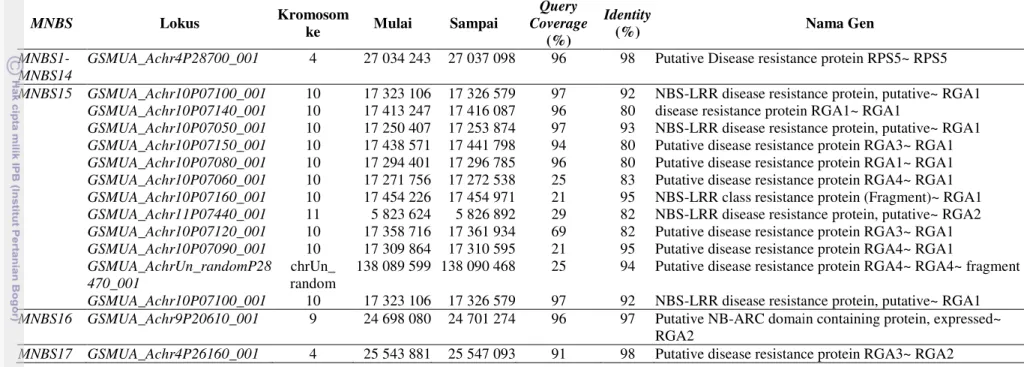
Perkembangan teknologi sekuensing telah menghasilkan informasi lengkap tentang runutan DNA genom beberapa jenis tanaman termasuk pisang (D’Hont et al. 2012). Informasi tersebut telah dapat diakses secara gratis dan dapat digunakan untuk menganalisis sekuen DNA yang berasal dari pisang, salah satunya adalah fasilitas BLAST. Hasil yang diperoleh dapat memperlihatkan letak suatu fragmen dalam kromosom pisang maupun jumlah salinan fragmen DNA yang bersangkutan. Hasil analisis BLAST fragmen MNBS ditampilkan pada Tabel 8. Berdasarkan hasil analisis BLAST terhadap DNA genom pisang yang tersedia pada situs http://banana-genome.cirad.fr/blast
terlihat bahwa MNBS1-MNBS14, MNBS16 dan MNBS17 mempunyai satu salinan, sedangkan MNBS15 mempunyai 11 salinan yang tersebar dalam dua kromosom pisang, yaitu kromosom nomor 10 dan 11, dan satu salinan secara acak.
Tabel 7 Matrik genetic identity (%) hasil dari analisis pensejajaran sekuen prediksi asam amino Aksesi 1 2 3 4 5 6 7 8 9 MNBS1 95.4 96.0 96.0 96.0 96.6 94.8 96.0 96.0 MNBS2 ... 97.1 97.7 95.4 96.0 95.4 95.4 95.4 MNBS3 ... ... 97.1 97.7 96.6 97.1 97.1 96.0 MNBS4 ... ... ... 96.6 97.1 96.6 96.6 96.6 MNBS5 ... ... ... ... 97.1 96.6 97.7 96.6 MNBS6 ... ... ... ... ... 98.3 99.4 99.4 100.0 MNBS7 ... ... ... ... ... ... 98.9 97.7 MNBS8 ... ... ... ... ... ... ... 98.9 MNBS9 ... ... ... ... ... ... ... ... MNBS10 ... ... ... ... ... ... ... ... ... MNBS11 ... ... ... ... ... ... ... ... ... ... MNBS12 ... ... ... ... ... ... ... ... ... ... MNBS13 ... ... ... ... ... ... ... ... ... ... MNBS14 ... ... ... ... ... ... ... ... ... ... MNBS15 ... ... ... ... ... ... ... ... ... ... MNBS16 ... ... ... ... ... ... ... ... ... ... MNBS17 ... ... ... ... ... ... ... ... ... ... CAP66325 ... ... ... ... ... ... ... ... ... ... AFR46148 ... ... ... ... ... ... ... ... ... ... ABY78502 ... ... ... ... ... ... ... ... ... ... ABY75803 ... ... ... ... ... ... ... ... ... ... ACK44408 ... ... ... ... ... ... ... ... ... ... AF202179 ... ... ... ... ... ... ... ... ... ... AF195939 ... ... ... ... ... ... ... ... ... ... LEU65391 ... ... ... ... ... ... ... ... ... ... AF149114 ... ... ... ... ... ... ... ... ... ... AAC67238 ... ... ... ... ... ... ... ... ... ... AAC72977 ... ... ... ... ... ... ... ... ... ... AAF08790 ... ... ... ... ... ... ... ... ... ... Q40253 ... ... ... ... ... ... ... ... ... ... Q40392 ... ... ... ... ... ... ... ... ... ... AAB47618 ... ... ... ... ... ... ... ... ... ...
Keterangan: Angka pada baris pertama adalah aksesi Musa RGA d
MNBS10, 11: MNBS11, 12: MNBS12, 13: MNBS13, 14: MNBS14
(ABY78502), 21: RGC3 Musa (ABY75803), 22: RGA-J Musa ABB (ACK44408), 23:
Mi-1.2 Tomat (AAC67238), 28: RPP1 Arabidopsis (AAC72977), 29:
(%) hasil dari analisis pensejajaran sekuen prediksi asam amino Musa RGA dan protein R mengguna
10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 96.6 96.6 96.6 96.6 96.0 24.7 19.5 20.4 25.3 20.2 23.1 96.0 19.7 22.5 19.9 25.2 96.0 96.0 96.0 96.0 95.4 24.1 18.9 19.4 24.7 19.7 20.2 96.0 19.7 23.7 20.5 26.9 96.6 96.6 96.6 96.6 97.1 23.6 18.9 19.4 23.6 20.2 20.8 97.1 18.5 23.7 20.5 26.3 97.1 97.1 97.1 97.1 96.6 23.6 18.9 18.3 25.3 20.2 20.8 98.3 19.7 23.7 21.1 25.7 97.1 97.1 97.1 97.1 97.7 23.6 18.9 19.4 23.6 20.2 21.4 98.8 18.5 23.1 19.9 26.3 100.0 100.0 100.0 100.0 99.4 24.7 18.3 20.4 24.7 20.2 21.4 97.1 18.5 23.1 19.9 26.3 98.3 98.3 98.3 98.3 98.9 23.6 18.3 19.4 23.6 19.7 20.8 96.5 17.9 23.1 19.9 25.7 99.4 99.4 99.4 99.4 100.0 24.1 18.9 19.4 24.1 20.2 21.4 97.7 18.5 23.1 19.9 26.3 99.4 99.4 99.4 99.4 98.9 24.1 17.8 19.4 24.1 19.7 20.8 96.5 17.9 22.5 20.5 25.7 100.0 100.0 100.0 99.4 24.7 18.3 20.4 24.7 20.2 21.4 97.1 18.5 23.1 19.9 26.3 ... 100.0 100.0 99.4 24.7 18.3 20.4 24.7 20.2 21.4 97.1 18.5 23.1 19.9 26.3 ... ... 100.0 99.4 24.7 18.3 20.4 24.7 20.2 21.4 97.1 18.5 23.1 19.9 26.3 ... ... ... 99.4 24.7 18.3 20.4 24.7 20.2 21.4 97.1 18.5 23.1 19.9 26.3 ... ... ... ... 24.1 18.9 19.4 24.1 20.2 21.4 97.7 18.5 23.1 19.9 26.3 ... ... ... ... ... 49.1 35.5 94.4 30.6 25.4 23.1 47.4 31.2 31.3 33.3 ... ... ... ... ... ... 35.5 51.5 32.0 32.0 19.5 91.7 30.8 33.7 31.4 ... ... ... ... ... ... ... 32.3 94.6 50.5 17.2 33.3 32.3 34.4 29.0 ... ... ... ... ... ... ... ... 30.1 26.6 24.9 51.5 31.8 32.5 32.8 ... ... ... ... ... ... ... ... ... 43.4 20.2 31.8 30.1 35.5 28.1 ... ... ... ... ... ... ... ... ... ... 21.4 31.8 25.4 27.7 27.5 ... ... ... ... ... ... ... ... ... ... ... 19.1 23.1 20.5 25.7 ... ... ... ... ... ... ... ... ... ... ... ... 30.1 33.7 31.0 ... ... ... ... ... ... ... ... ... ... ... ... ... 54.8 41.5 ... ... ... ... ... ... ... ... ... ... ... ... ... ... 47.0 ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ...
dan R gene dari GenBank. 1: MNBS1, 2: MNBS2, 3: MNBS3, 4: MNBS4, 5: MNBS5, 6: MNBS6
MNBS14, 15: MNBS15, 16: MNBS16, 17: MNBS17, 18: NBS-LRR Musa (CAP66325), 19: NBS-LRR
ABB (ACK44408), 23: Bs2 Lada (AF202179), 24: Gpa2 Kentang (AF195939), 25: Prf Tomat (LEU65391), 26: (AAC72977), 29: RPP5 Arabidopsis (AAF08790), 30: L6 Linum (Q40253), 31: N Tembakau
RGA dan protein R mengguna kan ClustalW2
25 26 27 28 29 30 31 32 25.2 22.5 22.2 17.2 19.2 23.0 19.0 25.3 26.9 22.0 23.4 17.2 18.6 23.6 19.5 25.3 26.3 21.4 22.8 16.7 18.0 23.0 18.4 24.7 25.7 22.0 22.8 17.2 18.6 23.6 20.1 25.3 26.3 22.0 22.8 17.2 18.0 23.0 19.0 24.7 26.3 22.0 22.8 17.2 18.0 23.0 19.0 24.7 25.7 20.8 22.2 16.7 18.0 23.0 17.8 24.7 26.3 22.0 22.8 17.2 18.0 23.0 18.4 24.7 25.7 22.0 22.2 16.7 18.6 22.4 19.0 24.1 26.3 22.0 22.8 17.2 18.0 23.0 19.0 24.7 26.3 22.0 22.8 17.2 18.0 23.0 19.0 24.7 26.3 22.0 22.8 17.2 18.0 23.0 19.0 24.7 26.3 22.0 22.8 17.2 18.0 23.0 19.0 24.7 26.3 22.0 22.8 17.2 18.0 23.0 18.4 24.7 33.3 28.9 29.9 21.1 18.6 22.6 26.3 23.4 31.4 28.4 31.7 18.3 20.7 24.3 24.9 21.9 29.0 25.8 35.5 17.2 16.1 26.9 21.5 29.0 32.8 29.5 29.3 21.7 19.2 23.7 28.0 24.0 28.1 28.9 34.7 20.2 18.0 23.7 22.0 24.9 27.5 23.7 32.9 18.5 17.4 20.2 21.4 22.0 25.7 22.5 22.8 17.3 18.0 23.1 19.7 24.9 31.0 28.9 29.9 20.8 19.8 25.4 24.9 23.7 41.5 33.0 42.5 20.2 18.6 25.4 24.3 25.4 47.0 31.3 42.8 19.3 19.9 23.5 24.1 25.3 31.6 49.7 19.3 18.7 21.1 22.2 21.1 ... 32.3 21.4 19.2 19.7 25.4 21.4 ... ... 22.2 22.2 22.2 26.4 22.8 ... ... ... 44.2 29.1 38.3 28.6 ... ... ... ... 30.8 40.7 30.8 ... ... ... ... ... 40.0 81.7 ... ... ... ... ... ... 37.7 ... ... ... ... ... ... ... MNBS6, 7: MNBS7, 8: MNBS8, 9: MNBS9, 10:
LRR Musa AAB (AFR46148), 20: RGC2 Musa Tomat (LEU65391), 26: Yr10 Gandum (AF149114), 27: Tembakau (Q40392), 32: M Linum (AAB47618)
Tabel 8 Hasil analisis BLASTN antara sekuen fragman MNBS Lokus Kromosom ke MNBS1-MNBS14 GSMUA_Achr4P28700_001 4 MNBS15 GSMUA_Achr10P07100_001 10 GSMUA_Achr10P07140_001 10 GSMUA_Achr10P07050_001 10 GSMUA_Achr10P07150_001 10 GSMUA_Achr10P07080_001 10 GSMUA_Achr10P07060_001 10 GSMUA_Achr10P07160_001 10 GSMUA_Achr11P07440_001 11 GSMUA_Achr10P07120_001 10 GSMUA_Achr10P07090_001 10 GSMUA_AchrUn_randomP28 470_001 chrUn_ random GSMUA_Achr10P07100_001 10 MNBS16 GSMUA_Achr9P20610_001 9 MNBS17 GSMUA_Achr4P26160_001 4
N antara sekuen fragman MNBS dengan data genom pisang global (http://banana
Kromosom Mulai Sampai Query Coverage (%) Identity (%)
27 034 243 27 037 098 96 98 Putative Disease resistance protein RPS5~ RPS5 17 323 106 17 326 579 97 92 NBS-LRR disease resistance
17 413 247 17 416 087 96 80 disease resistance protein RGA1~ RGA1
17 250 407 17 253 874 97 93 NBS-LRR disease resistance protein, putative~ RGA1 17 438 571 17 441 798 94 80 Putative disease resistance protein RGA3~ RGA1 17 294 401 17 296 785 96 80 Putative disease resistance protein RGA1~ RGA1 17 271 756 17 272 538 25 83 Putative disease resistance protein RGA4~ RGA1 17 454 226 17 454 971 21 95 NBS-LRR class resistance protein (Fragment)~ RGA1
5 823 624 5 826 892 29 82 NBS-LRR disease resistance protein, putative~ RGA2 17 358 716 17 361 934 69 82 Putative disease resistance protein RGA3~ RGA1 17 309 864 17 310 595 21 95 Putative disease resistance protein RGA4~ RGA1
138 089 599 138 090 468 25 94 Putative disease resistance protein RGA4~ RGA4~ fragment 17 323 106 17 326 579 97 92 NBS-LRR disease resistance protein, putative~ RGA1 24 698 080 24 701 274 96 97 Putative NB-ARC domain containing protein, expressed~
RGA2
25 543 881 25 547 093 91 98 Putative disease resistance protein RGA3~ RGA2
http://banana-genome.cirad.fr/blast)
Nama Gen
Putative Disease resistance protein RPS5~ RPS5 LRR disease resistance protein, putative~ RGA1 disease resistance protein RGA1~ RGA1
LRR disease resistance protein, putative~ RGA1 Putative disease resistance protein RGA3~ RGA1 Putative disease resistance protein RGA1~ RGA1 Putative disease resistance protein RGA4~ RGA1
LRR class resistance protein (Fragment)~ RGA1 LRR disease resistance protein, putative~ RGA2 Putative disease resistance protein RGA3~ RGA1 Putative disease resistance protein RGA4~ RGA1
Putative disease resistance protein RGA4~ RGA4~ fragment LRR disease resistance protein, putative~ RGA1
ARC domain containing protein, expressed~ Putative disease resistance protein RGA3~ RGA2
Kemungkinan besar MNBS1-MNBS14, MNBS16, dan MNBS17 bukan
pseudogene, karena salah satu kriteria gen yang termasuk pseudogene adalah mengalami duplikasi (Yao 2006). Fragmen MNBS15 mempunyai identity sebesar 78% dengan pseudogene NBS-LRR yang terdeposit dalam GenBank NCBI (EF488470). Ada kemungkinan MNBS15 adalah pseudogene, tetapi harus dibuktikan melalui uji ekspresi, apakah terjadi translasi protein atau tidak.
Hubungan Gen MNBS dengan RGA Pisang yang Terdeposit pada Pangkalan Data GenBank
Sampai saat ini sudah terdaftar lebih dari 180 fragmen gen RGA dari kelas NBS-LRR yang diisolasi dari tanaman pisang dalam pangkalan data NCBI. Namun demikian setelah periksa kembali sekuen nukleotida dan asam aminonya, hanya 168 sekuen yang mempunyai ciri-ciri NBS-LRR yaitu yang mengandung motif p-loop/kinase-1a, kinase-2, kinase-3 dan GLPL.
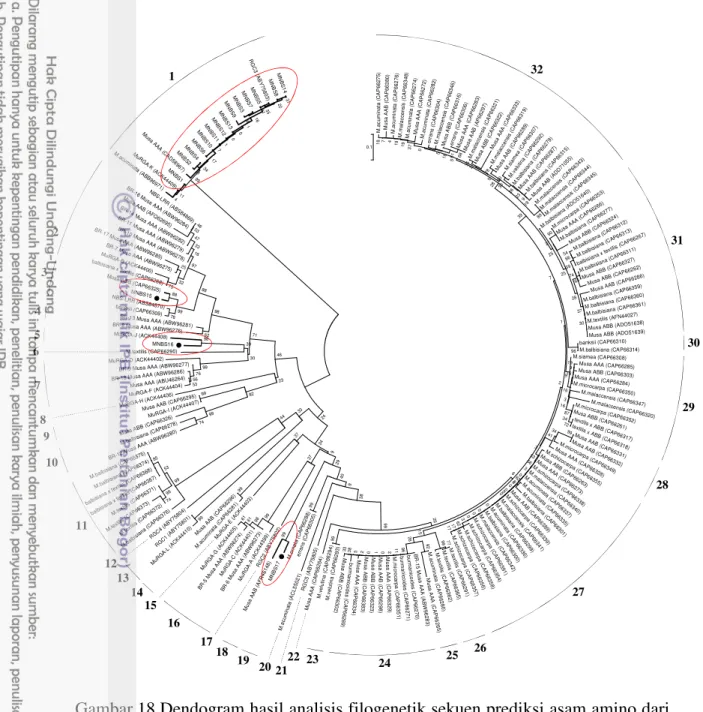
Dari hasil analisis filogenetik sekuen prediksi asam amino 17 gen MNBS yang dihasilkan dalam penelitian ini bersama dengan 168 sekuen asam amino RGA pisang, didapatkan 32 group RGA (Gambar 18). Gen MNBS1-MNBS14 berada pada satu group tersendiri bersama dengan empat RGA dengan nomor aksesi ABB96971, ACK44409, ABY75803 dan CAD58967, dengan identity sebesar 94% sampai 97 %, sedangkan
MNBS15 berada satu group dengan aksesi CAP66325, ABS84870 dan CAP66309 dengan identity sebesar 90% sampai 94 %. Gen MNBS16 berada dalam satu group dengan satu RGA ACK44408 dengan identity sebesar 92 %. Begitu juga dengan
MNBS17 yang hanya berada dalam satu group dengan satu RGA AFR46148, dengan
identity sebesar 97 %. Terdapat delapan group yang hanya beranggotakan satu RGA, dengan anggota masing-masing adalah aksesi CAP66290, ACK44406, ABY75801, ACK44403, ABY75802, ACL55021, ABY75805 dan CAP66264.
Berdasarkan hasil dari penelitian ini sebetulnya masih banyak RGA yang bisa dieksplorasi, terutama dari kultivar pisang lokal maupun spesies liar asal Indonesia. Penelitian sebelumnya juga telah berhasil mengamplifikasi sejumlah RGA dari tanaman pisang dan kerabatnya (Pei et al. 2007; Peraza-Echeverria et al. 2008; Azhar & Heslop-Harrison 2008; Sun et al. 2009). Banyaknya RGA tipe NBS-LRR LRR dari tanaman pisang yang teridentifikasi, akan dapat memberikan gambaran besarnya famili NBS dalam tanaman pisang.
Namun demikian perlu ditekankan bahwa tidak semua RGA mempunyai fungsi atau disebut pseudogene. Pan et al. (2000a) menemukan bahwa 10 % RGA yang telah teridentifikasi dari tanaman tomat adalah pseudogene. Hasil serupa juga ditemukan oleh Liu & Ekramoddoullah (2003) pada tanaman pinus, bahwa hampir setengah RGA yang teridentifikasi adalah pseudogene dan bahkan beberapa diantaranya ditranskripsi menjadi mRNA.
Oleh karena itu karakterisasi RGA perlu dilanjutkan dengan pengujian ekspresi gen terhadap kondisi cekaman patogen, sehingga pada akhirnya akan diperoleh gen ketahanan yang spesifik untuk penyakit tertentu. Selain itu gen ketahanan tersebut bisa dimanfaatkan untuk program perbaikan kultivar pisang tahan penyakit ataupun mengembangkan marka molekuler yang dapat digunakan untuk seleksi ketahanan terhadap penyakit.
Gambar 18 Dendogram hasil analisis filogenetik sekuen prediksi asam amino dari
MNBS pisang dan Musa NBS-LRR yang terdeposit pada GenBank dan dibuat berdasarkan metode Neighbour Joining. Angka pada sumbu percabangan adalah nilai bootstrap (1000 ulangan). Aksesi yang diberi tanda elip berwarna merah adalah RGA yang diperoleh dari penelitian ini.
Simpulan
Dari tiga kultivar pisang tahan layu Fusarium yaitu Rejang, Calcuta-4 dan Klutuk Wulung diperoleh 17 sekuen RGA (MNBS1-MNBS17) yang mengandung domain terkonservasi NBS-LRR dan terbagi ke dalam empat kelompok. Kelompok I beranggotakan 14 sekuen (MNBS1-MNBS14), dan ketiga kelompok lainnya masing-masing beranggotakan satu sekuen, yaitu MNBS15, MNBS16 dan MNBS17.
M .a cu m in at a (C A P 66 27 5) M us a AAB (C A P 6638 0) 18 M .acu min at a (CA P 6627 6) 7 M .m alac cens is (CA P 6634 8) 4 M .acu m inat a (C A P66 27 4) 19 Mus a A AA (C AP66 272) 19 M.a cum inat a (C AP6 6282 ) 37 er rans (CAP 6630 4) M.m alac cens is (C AP66 346) Mus a AB B (C AP66 316) erra ns (C AP66 306) Mus a AA A (C AP66 283) Mus a AA B (A P662 97) 68 61 18 5 0 M.m alac cens is (C AP66 321) Mus a AB B (C AP66 322) 35 Mus a AA A (C AP66 333) 2 M .mal acce nsis (CAP 6631 9) Mus a AA B (C AP66 289) 78 M. siam ea (C AP66 307) M.ve lutina (CAP 6629 2) 60 M.balbi siana (CAP 6627 9) Musa AAB (CAP 6628 7) M.ba lbisia na (C AP66 315) Musa AAB (ADD 7105 5) 19 9 23 26 30 0 5 M.ma lacce nsis (C AP66 343) M.ma lacce nsis (C AP66 344) M.ma lacce nsis ( CAP6 6345) 89 28 M.balbis iana ( ADO5 1640) M.micro carpa (CAP6 6353) Musa AAA ( CAP66 266) M.balb isiana (CAP66 277) 90 Musa ABB (CAP66 324) 21 M.balbi siana (C AP6631 2) M.balbis iana (CA P66313 ) 54 balbisian a x textilis (C AP66267) 66 M.balbisia na (CAP6 6311) Musa ABB (CAP663 27) 65 39 Musa ABB (CAP6626 2) 74
Musa AAB (CAP6 6286)
M.balbisiana (CAP6 6359) M.balbisiana (C
AP66360)
M.balbisiana (CAP 66361) M.textilis (AFN44027) Musa ABB (ADO51638) Musa ABB (ADO51639) 30 37 28 25 25 39 banksii (CAP66310) M.balbisiana (CAP66314) 96 7 M.siamea (CAP66308) Musa AAA (CAP66285) Musa ABB (CAP66303)
19
Musa AAA (CAP66284) 9
M.microcarpa (CAP66350)
16
M.malaccensis (CAP66347)
M.malaccensis (CAP66
320)
M.microcarpa (CAP6
6352) Musa AB B (CAP66261) textilis x ABB (C AP66317) textilis x ABB (C AP66318) 72 3487 163 16 9 1 Musa AAB (C AP66331) Musa AAB (C AP66332) 99 M.mic rocarpa (CA P66349) 34 Musa AAA (CA P66328) 41 M.sc hizocarpa (CA P66355) Musa ABB (CA P66263) Musa AA A (CAP6 6273) M.schizo carpa (C AP6633 8) M.m alaccen sis (CA P66340 ) M.acum inata (CA P66337 ) M.acum inata (C AP 66335) M.balb isia na (CA P66301 ) 62 Mu sa AAB (CAP 663 89) 2 M.s chizoca rpa (C AP663 39) 0 M.m ala cce nsis (C AP663 41) 0 M.balb isiana (C AP66 299) 0 M .acum ina ta (CA P663 36) M .ba lbisian a (C AP66 300) M .m ala cce nsis (C AP66 342) M .schiz oc arp a (CA P663 91) M .schiz ocarp a (C AP66 354) M .sc hizoc arp a (C AP 6635 8) M .sc hizoc arp a (CA P6 63 56) M .schiz ocarp a (C AP 6635 7) 17 1762 3 00 0 0 0 0 03 5653 3 2 M .te xtilis ( C AP 6629 1) M .te xtilis ( C AP 66 36 5) 77 M .te xtilis (C AP 6636 2) 99 M .a cum inata (CA P6 62 80) M us a A AA (C AP 66 26 5) 92 BR -1 5 M us a A AA (A BW 9628 3) bu rm an icoid es (CA P6 6270 ) bu rm an ico id es (CA P6 62 71 ) M .m icroc arpa ( C AP 66 35 1) M us a A AA (C A P 66 32 9) M us a A AB (C A P 66 29 8) M us a A B B ( C A P 66 32 3) M us a A BB (C AP 66 38 3) M us a A AA (C AP 66 33 4) bu rm an ico id es (C AP 66 26 9) M us a A BB (C AP 66 30 2) 33 38 5 2 0 1 0 2 11 96 26 15 6 M.v elut ina (CAP 6629 3) M.v elut ina (CAP 6629 4) 99 26 Mus a AA A (C AP66 264) 99 RGC 5 (A BY75 805) 38 M.a cum inat a (A CL5 5021 ) 9 erra ns (C AP66 305) M.o rnat a (C AP66 288) 99 40 MNB S17 Mus a AA B (A FR46 148) 99 29 RGC2 (ABY 7580 2) MuR GA-A (ACK 4439 9) BR-6 Mus a AA A (A BW96 273) MuR GA-C (ACK 4440 1) BR-5 Mus a AA A (A BW96 274) MuRG A-G (ACK 4440 5) 61 61 98 99 37 9 MuRG A-E (A CK44 403) M.ac umina ta (C AP66 281) Musa AAB (CAP 6629 6) 99 37 34 MuRG A-L ( ACK4 4410 ) RGC1 (ABY 7580 1) 99 RGC4 (ABY 75804) M.balbis iana ( CAP6 6376) M.text ilis (CA P6637 2) M.text ilis (CA P6637 3) 74 M.text ilis (CA P66371 ) 98 balbisi ana x te xtilis (C AP663 67) balbisia na x tex tilis (CA P66368 ) M.balbi siana (C AP6637 4) M.balbis iana (CA P66375) 85 53 99 44 33 24 BR-12 Mu sa AAA (ABW 96280) M.balbisia na (CAP66 278) 74 Musa ABB (CAP6632 6) 99 MuRGA-I (A CK44407) Musa AAB (CA P66295)99 82 MuRGA-H (ACK 44406) 23 MuRGA-F (ACK44404)
Musa AAA (ABU46264) 53 BR-18 Musa AAA (ABW96286) 56 BR-9 Musa AAA (ABW96277)
76 MuRGA-D (ACK44402) 99 M.textilis (CAP66290) MNBS16 MuRGA-J (ACK44408) 98 39 30
BR-8 Musa AAA (ABW96276)BR-13 Musa AAA (ABW96281) 62 banksii (CAP66309) NBS-LRR (ABS84870) 78 MNBS15 Musa ABB (CAP66325) 88 99 balbisiana x textilis (CAP66268) MuRGA-B (ACK 44400) 75 BR-7 Musa AA A (ABW 96275) BR-17 Musa A AA (AB W96285) 25 32 BR-10 Musa AAA (ABW 96278) BR-11 Musa AAA (ABW 96279) BR-14 Mu sa AA A (ABW962 82) Musa AAB (AFQ 62695) BR-16 M usa A AA (ABW 96284) NBS-LR R (ABS8 4869) 46 53 47 33 16 97 88 98 71 46 M.acum inata (AB B96971 ) MuR GA -K (A CK4440 9) 4 MN BS1 11 Musa A AA (C AD 589MN67)BS2 MNBS 4 31 MN BS6 MN BS10 M NBS1 1 M NB S12 M NB S13 M NB S9 M NB S3 M NB S7 36 M NB S5 RG C3 (A BY 7580 3) 41 M NB S8 MN BS 14 31 22 35 16 1 0 0 1 7 17 34 99 0.1 1 2 4 8 9 12 5 6 7 10 11 13 14 15 16 17 18 19 20 21 3 22 23 24 25 26 27 28 29 30 31 32
Prediksi asam amino 17 MNBS mempunyai identity sebesar 91.7 % sampai 98.8 % dengan protein Musa NBS-LRR dan 19.9 % sampai 35 % dengan protein dari gen ketahanan tanaman lain yang telah teridentifikasi. MNBS17 mempunyai
identity yang relatif tinggi (50.5 %) dengan RGC2 asal galur M. acuminata ssp.
malaccensis yang berasosiasi dengan ketahanan terhadap layu FOC ras 4.
Daftar Pustaka
Agrios GN. 2005. Plant Pathology. Ed ke-5. Burlington: Elsevier Academic Pr. hlm 125-174.
Altschul SF, Madden TL, Schaffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ. 1997. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nuc Acids Res 25:3389-3402.
Azhar M, Heslop-Harrison JS. 2008. Genomes, diversity and resistance gene analogues in Musa species. Cytogenet Genom Res 121(1):59-66.
Baek DE, Choi C. 2013. Identification of resistance gene analogs in Korean wild apple germplasm collections. Genet Mol Res 12(1):483-493.
Bai J, Pennill LA, Ning J, Lee SW, Ramalingam J, Webb CA, Zhao B, Sun Q, Nelson JC, Leach JE, Hulbert SH. 2002. Diversity in nucleotide binding site--leucine-rich repeat genes in cereals. Genome Res 12:1871-1884. Baker CJ, Mock N, Glazener J, Orlandi E. 1993. Recognitian responses in
pathogen/non-host and race/cultivar interactions involving soybean (Glycine max) and Pseudomonas syringae pathovars. Physiol Mol Plant
Pathol 43:81-94.
Cannon SB, Zhu H, Baumgarten AM, Spangler R, May G, Cook DR, Young ND. 2002. Diversity, distribution, and ancient taxonomic relationships within the TIR and non-TIR NBS-LRR resistance gene subfamilies. J Mol Evol 54:548-562.
Caplan J, Padmanabhan M, Dinesh-Kumar SP. 2008. Plant NB-LRR immune receptors: From recognition to transcriptional reprogramming. Cell Host
Microbe 3:126-135.
Cheng X, Jiang H, Zhao Y, Qian Y, Zhu S, Cheng B. 2010. A genomic analysis of disease-resistance genes encoding nucleotide binding sites in Sorghum
bicolor. Genet Mol Biol 33(2):292-297.
Dangl L, Jones J. 2001. Plant pathogens and integrated defence responses to infection. Nature 411:826-833.
Das BK, Jena RC, Samal KC. 2009. Optimization of DNA isolation and PCR protocol for RAPD analysis of banana/plantain (Musa spp.). Int J Agricul
Sci 1(2):21-25.
Doyle JJ, Doyle JL. 1987. A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochem Bull 19:11-15.
Friedman AR, Baker BJ. 2007. The evolution of resistance genes in multiprotein plant resistance systems. Curr Opin Genet Dev 17:493–499.
Jones DA, Jones JDG. 1997. The role of leucine-rich repeat proteins in plant defences. Adv Bot Res 24:89-167.
Jones JDG. 2001. Putting knowledge of plant disease resistance genes to work.
Kanazin V, Marek LF, Shoemaker RC. 1996. Resistance gene analogs are conserved and clustered in soybean. Proc Nat Acad Sci USA 931:11746-11750
Larkin MA, Blackshields G, Brown NP, Chenna R, McGettigan PA, McWilliam H, Valentin F, Wallace IM, Wilm A, Lopez R et al. 2007. Clustal W and Clustal X version 2.0. Bioinformatics 23(21):2947-2948.
Li X, Sui X, Zhang Y, Sun Y, Zhao Y, Zhai Y, Wang Q. 2010. An improved calcium chloride method preparation and transformation of competent cells. Afr J Biotechnol 9(50):8549-8554.
Liu JJ, Ekramoddoullah AKM. 2003. Isolation, genetic variation and expression of TIR-NBS-LRR resistance gene analogs from western white pine (Pinus
monticola Dougl. ex. D. Don.). Mol Genet Genom 270:432–441.
Madsen LH, Collins NC, Rakwalska M, Backes G, Sandal N, Krusell L, Jensen J, Waterman EH, Jahoor A, Ayliffe M et al. 2003. Barley disease resistance gene analogs of the NBS-LRR class: identification and mapping. Mol
Genet Genomics 269:150-161.
McHale L, Tan X, Koehl P, Michelmore RW. 2006. Plant NBS-LRR proteins: adaptable guards. Genom Biol 7:212 (doi:10.1186/gb-2006-7-4-212). Meyers BC, Dickerman AW, Michelmore RW, Sivaramakrishnan S, Sobral BW,
Young ND. 1999. Plant disease resistance genes encode members of an ancient and diverse protein family within the nucleotide-binding superfamily. Plant 20(3):317-332.
Meyers BC, Kozik A, Griego A, Kuang H, Michelmore RW. 2003. Genome-wide analysis of NBS-LRR-encoding genes in Arabidopsis. Plant Cell 15:809-834. Molina AB, Williams RC, Hermanto C, Suwanda, Komolog B, Kokoa, P. 2010.
Mitigating the threat of banana Fusarium wilt: understanding the agroecological distribution of pathogenic forms and developing disease management strategies. Australia: ACIAR 76 hlm.
Nicholas KB, Nicholas HB Jr., Deerfield DW II. 1997. GeneDoc: analysis and visualization of genetic variation. Embnet News 4:1-4.
Pan Q, Liu YS, Budai-Hadrian O, Sela M, Carmel-Goren L, amir D, Fluhr R. 2000a. Comparative genetics of nucleotide binding site-leucine rich repeat resistance gene homologues in the genomes of two dicotyledons: tomato and Arabidopsis. Genet 155:309–322.
Pan Q, Wendel J, Fluhr R. 2000b. Divergent evolution of plant NBS-LRR resistance gene homologues in dicot and cereal genomes. J Mol Evol 50:203-213.
Pegg KG, Moore NY, Sorensen S. 1993. Fusarium wilt in the Asian Pacific region. Di dalam: Valmayor RV, Hwang SC, Ploetz RC, Lee SW, Roa NV, editor. Procceeding of International Symposium Recent Developments
in Banana Cultivation Technology; Pingtung (TWN), 4-18 Des 1992. Los Baños: INIBAP-ASPNET. hlm 225-269.
Pei X, Li S, Jiang Y, Zhang Y, Wang Z, Jia S. 2007. Isolation, characterization and phylogenetic analysis of the resistance gene analogues (RGAs) in banana (Musa spp.). Plant Sci 172:1166–1174.
Peraza-Echeverria S, Dale JL, Harding RM, Smith MK, Collet C. 2008. Characterization of disease resistance gene candidates of the nucleotide binding site (NBS) type from banana and correlation of a transcriptional