PHYLOGENETIC ANALYSIS OF AQUILARIA AND GYRINOPS MEMBER BASED ON TRNL-TRNF GENE SEQUENCE OF CHLOROPLAST
Oleh:
I Gde Adi Suryawan Wangiyana
Forestry Faculty of Nusa Tenggara Barat University
Abstract: Aquilaria and Gyrinops are main agarwood producers that only could be distinguished based on their flower structure. Molecular characteristic analysis is needed to support morphological character for breeding and conservation purpose. Several molecular analyses were conducted based on similarity characteristic of Aquilaria and Gyrinops. However phylogenetic analysis of these genera was so few to support the polyphasic classification. The purpose of this study is to do phylogenetic analysis based on trnL-trnF sequence. Thirteen sequences of Aquilaria and six sequences of Gyrinops were downloaded from NCBI. ClustalX 2.1 program were used for multiple alignment of sequences. Three phylogenetic trees were reconstructed with MEGA 5.1 by different method including: Neighbor Joining, Minimum and Maximum Liklihood. Phydit program were used to construct similarity matrix between sequences. Based on phylogenetic analysis, all Aquilaria members were clustered in the same clade. All sister taxa also composed with Aquilaria member except for A.hirta. On the other hand, G.salicifora, G.Podocarpa and G.Ladermanii are the only Gyrinops members that form same clade. Other Gyrniops members were spread to different clade. It could be concluded that trnL-trnF sequence is a useful phylogenetic marker for Aquilaria although have several limited for Gyrinops. Moreover, based on phylogenetic analysis and similarity analysis it could be suggested that Aquilaria and Gyrinops sould not be separated into different genera.
Keyword: Phylogenetic Analysis, Aquilaria-Gyrinops, trnL-trnF Chloroplast
PENDAHULUAN
Gaharu adalah hasil hutan bukan kayu dengan nilai ekonomis tinggi yang dihasilkan terutama oleh dua genus famili thymelaeaceae yaitu: Aquilaria dan Gyrinops. Dengan nilai ekonomis yang tinggi menyebabkan pesatnya eksploitasi terhadap anggota kedua genus tersebut. Akibatnya kedua genus tersebut terancam kelestariannya sehingga perlu tindakan konservasi (Schmidt, 2011).
Genus Aquilaria dan Gyrinops selama ini dibedakan secara morfologis berdasarkan struktur bunga. Genus Aquilaria mempunyai 10 buah stamen sementara genus Gyrinops hanya memiliki 5 stamen (Compton and Zich, 2002). Di era taksonomi modern saat ini, membedakan dua kelompok hanya berdasarkan satu karakter tunggal kurang relevan. Terlebih lagi seringkali terjadi penyimpangan jumlah stamen pada Gyrinops seperti yang dilaporkan oleh Koherani (2013), bahwa jumlah stamen pada G. versteegii bervariasi antara 3 – 8 tetapi tidak pernah berjumlah 10.
Perkembangan biologi molekular yang pesat belakangan ini mendorong pesatnya perkembangan penerapan data molekular untuk menyokong proses identifikasi dan klasifikasi. Hal ini berlaku juga untuk genus Aquilaria dan Gyrinops. Tercatat beberapa proses identifikasi kedua genus ini dengan menggunakan data molekular diantaranya: studi keragaman genetic anggota genus Aquilaria berdasarkan analisis Isoenzim (Situmorang, 2000),
studi keragaman genetis pada anggota genus Aquilaria dan Gyrinops versteegii dengan analisis Amplified Fragment Length Polymorphism (Toruan-Mathius et al., 2009) dan studi gen δ-Guanine synthase untuk keperluan identifikasi anggota genus Aquilaria (Kumeta and Ito, 2011). Study – study tersebut menekankan pada analisis similaritas untuk melakukan klasifikasi molekular.
Gambar 1. Struktur bunga Gyrinops (kiri) dan Aquilaria (kanan) (S=stamen) (Koherani, 2013)
Untuk memperoleh klasifikasi molekular yang bersifat polyphasic dibutuhkan dukungan data pendekatan dengan analisis similaritas dan analisis phylogenetic (Lengeler et al., 1999). Pendekatan analisis similaritas pada genus Aquilaria dan Gyrinops telah banyak dilakukan sebelumnya namun tidak demikian halnya dengan analisis filogenetik. Untuk itu perlu dilakukan lebih banyak studi filogenetik terhadap kedua genus ini.
Untuk analisis filogenetik diperlukan marker molekular yang representatif. Hasil dan kualitas
dari analisis filogenetik sangat ditentukan oleh marker yang digunakan (Patwardhan et al., 2014). Gen kloroplast telah banyak digunakan sebagai marker molekular untuk analisis filogenetik tumbuhan (Small et al., 2004). Komplit genom dari kloroplast digunakan untuk analisis filogenetik berbagai macam tanaman diantaranya: sebagai marker molekular untuk studi anggota genus Oncidium (Wu et al., 2010), analisis phylogenetic dan studi genetika populasi anggota genus Cymbidium (Yang et al., 2013), marker molekular hubungan filogenetik antar anggota genus Eleusine (Agrawal et al., 2013).
Belakangan ini, Gen kloroplast sudah mulai digunakan sebagai marker molekular sekaligus DNA barcoding anggota genus Aquilaria. Sekuen Gen kloroplast yang digunakan diantaranya adalah: matK, rbcL, rpoB, rpoC1, psbA-trnH, ITS dan trnL-trnF (Lee et al., 2016). Akan tetapi , analisis filogenetik yang secara langsung membandingkan antara anggota genus Aquilaria dan Gyrinops belum pernah dilakukan sebelumnya. Diantara sekuen gen kloroplast, sekuen gen trnL-trnF mempunyai primer yang sangat conserve dengan amplifikasi yang sangat bisa diandalkan sehingga cocok digunakan sebagai marker molekular (Gambar 1). Oleh karena itulah Studi ini bertujuan untuk melakukan analisis filogenetik anggota genus Aquilaria dan Gyrinops berdasarkan sekuen trnL-trnF kloroplast
Gambar 2. Posisi sekuen Non-Coding spacer kloroplast (trnL-trnF) serta primer yang digunakan untuk amplifikasi METODE
a. Data sekuen
Sekuen gen trnL-trnF kloroplast dari anggota genus Aquilaria dan Gyrinops diunduh dari web NCBI (http://www.ncbi.nlm.nih.gov/) menggunakan mode Nucleotide Search. Sebanyak 13 sekuen anggota genus Aquilari dan 6 sekuen anggota genus Gyrinops digunakan untuk merekonstruksi pohon filogenetik. Sebagai outgroup untuk melakukan root dipilih sekuen Gonystylus bancanus karena masih merupakan
anggota Famili thymelaeaceae sebagaimana Aquilaria dan Gyrinops. Keterangan tentang sekuen yang digunakan dalam studi ini dapat dilihat pada tabel 1.
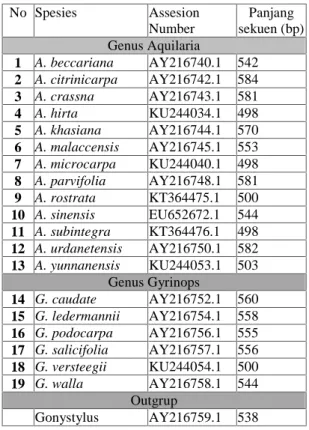
Tabel 1. Keterangan sekuen trnF-trnL anggota genus Aquilaria dan Gyrinops
No Spesies Assesion Number Panjang sekuen (bp) Genus Aquilaria 1 A. beccariana AY216740.1 542 2 A. citrinicarpa AY216742.1 584 3 A. crassna AY216743.1 581 4 A. hirta KU244034.1 498 5 A. khasiana AY216744.1 570 6 A. malaccensis AY216745.1 553 7 A. microcarpa KU244040.1 498 8 A. parvifolia AY216748.1 581 9 A. rostrata KT364475.1 500 10 A. sinensis EU652672.1 544 11 A. subintegra KT364476.1 498 12 A. urdanetensis AY216750.1 582 13 A. yunnanensis KU244053.1 503 Genus Gyrinops 14 G. caudate AY216752.1 560 15 G. ledermannii AY216754.1 558 16 G. podocarpa AY216756.1 555 17 G. salicifolia AY216757.1 556 18 G. versteegii KU244054.1 500 19 G. walla AY216758.1 544 Outgrup Gonystylus AY216759.1 538
b. Multiple Sequence Alignment
Multiple Sequence Alignment dilakukan dengan menggunakan program ClustalX 2.1. (Larkin et al., 2007). Alignment bertujuan untuk menata sequence trnL-trnF agar satu sama lain diletakkan sesuai dengan posisi homologi antar sequence sehingga dapat dibandingkan. File FASTA digunakan sebagai data input. Alignment total (do complete alignment) dilakukan agar diperoleh file yang kompatibel untuk program MEGA dan PHYDIT.
c. Rekonstruksi Phylogenetic tree dengan MEGA
Rekonstruksi pohon filogenetik pada MEGA 5.1(Tamura et al. 2011) menggunakan data sequence hasil alignment ClustalX 2.1. Karena terdapat perbedaan format file Clustal X dan Mega, file sekuen harus dikonversi terlebih dahulu agar kompatibel. Rekonstruksi Phylogenetic tree dilakukan dengan menggunakan 2 algoritme filogenetik, yaitu: Neighbor Joining dan Maximum Liklihood.
Neighbor Joining tree (Saitou and Nei, 1987) direkonstruksi dengan menggunakan Test of
Phylogeny bootstrap 1000 replikasi.Model evolusi yang digunakan adalah Tamura 3 Parameter. Laju evolusi (Rates among sites) menggunakan Gamma Distributed. Perlakuan terhadap data jika ada yang hilang (missing data treatment) menggunakan mode complete deletion.
Maximum Likelihood tree (Felsenstein,1981) direkonstruksi dengan menggunakan Test of Phylogeny bootstrap 1000 replikasi. Model evolusi yang digunakan adalah Tamura 3 Parameter. Laju evolusi (Rates among sites) menggunakan Gamma Distributed. Perlakuan terhadap data jika ada yang hilang (missing data treatment) menggunakan mode use all sites. Untuk optimasi pohon filogenetik digunakan ML Heuristic Method Nearest-Neighbor-Interchange (NNI) dengan Initial tree for ML adalah Neighbor Joining. d. Pembuatan Matriks Similaritas dengan
PHYDIT
Program Phydit menggunakan data input berupa hasil alignment dengan format “gde” sehingga harus dilakukan konversi terlebih dahulu agar data kompatibel. Matriks similaritas nukleotida dibuat dengan mode Generating Similarity Table. Output berupa Matrix similaritas selanjutnya di copy ke MS Excel untuk pengaturan tampilan yang lebih baik (Chun, 1995). Setelah data matriks similaritas diperoleh, dilakukan analisis hubungan similaritas antara anggota genus Gyrinops dengan Gyrinops, Aquilaria dengan Aquilaria serta Gyrinops dengan Aquilaria dengan nilai cut of value sebesar 99%.
HASIL
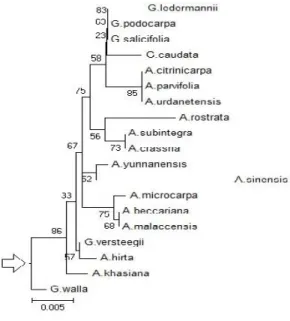
Pohon filogenetik hasil rekonstruksi dengan menggunakan program MEGA disajikan pada gambar 3 dan gambar 4
Gambar 3. Maximum Likelihood tree Tamura 3 Parameter 1000 bootstrap
Gambar 4. Neighbor Joining tree Tamura 3 Parameter 1000 bootstrap
Sementara itu, untuk matriks similaritas ditunjukkan oleh tabel 2. Matriks similaritas ditampilkan dalam bentuk jauh lebih sederhana dibandingkan dengan matriks similaritas output dari program PHYDIT. Hanya terdapat dua kelompok nilai similaritas antar spesies yang dibandingkan yaitu nilai similaritas lebih dari 99 dan nilai similaritas kurang dari 99.
Tabel 2. Matriks similaritas PHYDIT yang dimodifikasi 1 7 13 18 4 9 11 10 15 5 2 8 12 3 17 14 16 6 19 1 7 13 18 4 9 11 10 15 5 2 8 12 3 17 14 16 6 19
Keterangan:
1 – 13 : Anggota genus Aquilaria 14 – 15 : Anggota genus Gyrinops
(masing – masing spesies anggota genus Aquilaria dan Gyrinops dapat dilihat pada table 1)
: Similaritas > 99
: Similaritas <99
Untuk menentukan kecenderungan similaritas antar sesama anggota genus Gyrinops, sesama anggota genus Aquilaria dan antar genus Gyrinops dan Aquilaria, maka dibuat tabel yang mampu menerangkan hubungan tersebut seperti tertera pada tabel 3, 4 dan 5
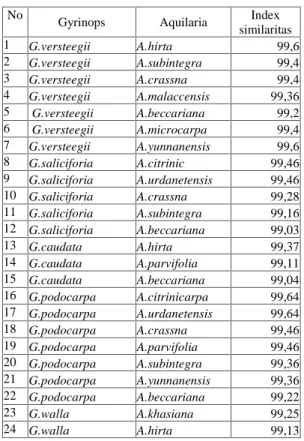
Tabel 3. Nilai similaritas antar Anggota Genus Gyrinops dan Aquilaria
No
Gyrinops Aquilaria Index similaritas 1 G.versteegii A.hirta 99,6 2 G.versteegii A.subintegra 99,4 3 G.versteegii A.crassna 99,4 4 G.versteegii A.malaccensis 99,36 5 G.versteegii A.beccariana 99,2 6 G.versteegii A.microcarpa 99,4 7 G.versteegii A.yunnanensis 99,6 8 G.saliciforia A.citrinic 99,46 9 G.saliciforia A.urdanetensis 99,46 10 G.saliciforia A.crassna 99,28 11 G.saliciforia A.subintegra 99,16 12 G.saliciforia A.beccariana 99,03 13 G.caudata A.hirta 99,37 14 G.caudata A.parvifolia 99,11 15 G.caudata A.beccariana 99,04 16 G.podocarpa A.citrinicarpa 99,64 17 G.podocarpa A.urdanetensis 99,64 18 G.podocarpa A.crassna 99,46 19 G.podocarpa A.parvifolia 99,46 20 G.podocarpa A.subintegra 99,36 21 G.podocarpa A.yunnanensis 99,36 22 G.podocarpa A.beccariana 99,22 23 G.walla A.khasiana 99,25 24 G.walla A.hirta 99,13
Tabel 4. Nilai similaritas antar anggota genus Gyrinops.
NO Gyrinops Gyrinops Index similaritas 1 G.versteegii G.podocarpa 99,57 2 G.versteegii G.walla 99,35 3 G.ledermanii G.podocarpa 99,62 4 G.ledermanii G.salicifo 99,44 5 G.salicifolia G.podocarpa 100 6 G.salicifolia G.caudata 99,46 7 G.salicifolia G.walla 99,07 8 G.caudata G.podocarpa 99,46 9 G.podocarpa G.walla 99,07
Tabel 5. Nilai similaritas antar anggota genus Aquilaria
No Aquilaria Aquilaria Index Similaritas 1 A. beccariana A.microcarpa 99,8 2 A.beccariana A,malaccensis 99,81 3 A.beccariana A,hirta 99,2 4 A,beccariana A.crassna 99,08 5 A.microcarpa A.hirta 99,4 6 A.microcarpa A.malaccensis 99,57 7 A.yunnanensis A.hirta 99,2 8 A,subintegra A.crassna 100 9 A,subintegra A.malaccensis 99,15 10 A,citrinicarpa A.urdanetensis 100 11 A,citrinicarpa A.parvifolia 99,83 12 A,parvifolia A.urdanetensis 99,83 13 A.crassna A.malaccensis 99,28 PEMBAHASAN
Selama ini satu – satunya karakter yang digunakan untuk membedakan antara genus Gyrinops dan Aquilaria adalah karakter bunga. Kedua genus tersebut terkenal sebagai penghasil komoditi gaharu yang memiliki nilai ekenomis tinggi. Berdasarkan karakter morfologi bunga, Gyrinops mempunyai jumlah stamen sama dengan jumlah mahkota yaitu sebanyak 5 stamen dan 5 mahkota. Sementara itu, Gyrinops mempunyai jumlah stamen dua kali jumlah mahkota, yaitu: 10 stamen dan 5 mahkota (Koherani, 2013).
Membedakan takson hanya berdasarkan 1 karakter merupakan hal yang kurang relevan untuk dilakukan diera taksonomi modern. Kelompok takson sebaiknya diklasifikasikan berdasarkan banyak karakter (politetik) bukan hanya dari karakter tunggal (monotetik). Karakter molekular merupakan salah satu karakter kunci yang sangat dapat diandalkan sebagai dasar klasifikasi diera taksonomi modern. Dengan memanfaatkan karakter molekar, dapat dilakukan klarifikasi apakah karakter morfologi bunga sudah cukup dijadikan sebagai acuan untuk membedakan anggota genus Aquilaria dan Gyrinops. Salah satunya adalah analisis filogenetik.
Resolusi marker yang digunakan dalam analisis filogenetik merupakan hal yang sangat esensial. Marker yang memiliki resolusi bagus akan mampu membedakan taksa berbeda baik pada level spesies maupun genus. Marker tersebut akan menempatkan taksa satu spesies atau satu genus kedalam clade yang sama dalam phylogenetic tree (Patwardhan et al., 2014)
Phylogenetic tree yang direkonstruksi menggunakan algoritme berbeda mempunyai topologi yang tidak jauh berbeda. Semua anggota genus Aquilaria tergabung dalam clade yang sama. Hal ini menunjukkan bahwa sekuen gen trnL-trnF
mempunyai resolusi cukup bagus untuk analisis filogenetik anggota genus Aquilaria.
Untuk anggota genus Gyrinops, tidak semua anggota menempati satu clade. hanya G. salicifolia, G. podocarpa dan G. ladermanii selalu tergabung dalam satu clade. Sementara itu anggota genus Gyrinops lainnya tergabung dalam clade bersama – sama dengan anggota genus Aquilaria. Hal ini menunjukkan bahwa sekuen gen trnL-trnF relatif memiliki resolusi yang rendah untuk analisis filogenetik anggota genus Gyrinops
Dalam Neighbor Joining tree dan Maximum likelihood tree, terdapat anggota genus Gyrinops yang tergabung dalam clade yang sama dengan anggota genus Aquilaria. Hal ini menunjukkan bahwa pembagian genus Aquilaria dan Gyrinops pada dasarnya tidak bersifat monofiletik karena terdapat anggota genus Aquilaria dan Gyrinops yang membagi nenek moyang bersama. Hal ini juga mengindikasikan bahwa berdasarkan hubungan kekerabatan, Aquilaria dan Gyrinops sebaiknya tidak dipisahkan menjadi genus yang berbeda.
Pasangan sister taksa antara Neighbor Joining tree dan Maximum Likelihood tree cukup konsisten meskipun terdapat beberapa perbedaan. Sister taksa merupakan kerabat terdekat terakhir yang tidak berbagi nenek moyang bersama dengan taksa lainnya dalam phylogenetic tree (Gregory, 2008). Sister taksa yang terbentuk kebanyakan adalah antara Anggota genus Aquilaria dengan anggota genus aquilaria serta anggota genus Gyrinops dengan anggota genus Gyrinops. Penyimpangan sister taksa terdapat pada pasangan sister taksa G. versteegii dan A. Hirta. Hal ini mengindikasikan bahwa keduanya taksa tersebut seharusnya secara filogenetik menempati genus yang sama.
Meskipun direkonstruksi dengan metode statistik dan algoritme berbeda, namun Neighbor Joining tree dan Maximum likelihood tree menunjukkan topology yang tidak jauh berbeda. Neighbor Joining adalah algoritme yang menggunakan metode distance similarity sehingga terdapat indeks similaritas yang disusun menggunakan model evolusi tertentu (Saitou and Nei, 1987). Sementara itu algoritme Maximum likelihood menggunakan metode character state yang berusaha mencari Phylogenetic tree optimum dari replikasi yang dihasilkan (Felsenstein,1981). Perbedaan keduanya hanya terlihat dari skala divergensi yang sedikit berbeda antar taksa yang dibandingkan. Skala divergensi taksa pada Neighbor Joining tree sedikit lebih besar dibandingkan dengan skala divergensi taksa Maximum likelihood tree.
Pasangan taksa yang menjadi sister taksa memiliki skala divergensi yang berbeda – beda.
Hal ini menunjukkan bahwa dalam sejarah evolusinya terdapat perbedaan laju evolusi antara taksa yang ada sekarang dengan nenek moyang bersama sister taksa tersebut. Baik pada Neighbor Joining tree dan Maximum likelihood tree pasangan sister taksa A. sinensis dan A. yunnanensis selalu memiliki skala divergensi yang berbeda secara signifikan. Dalam hal ini, A. yunnanensis cenderung memiliki laju evolusi yang jauh lebih tinggi dibandingkan dengan A. sinensis. Selain A. yunnanensis, A. rostrata juga memiliki skala divergensi yang lebih tinggi dibandingkan anggota genus Aquilaria lainnya. Sementara itu untuk anggota genus Gyrinops yang memiliki laju divergensi signifikan berbeda dengan anggota lainnya adalah: G. caudata dan G. ladermanii.
Berdasarkan matriks similaritas secara umum tiap anggota genus Aquilari dan Gyrinops memiliki indeks similaritas yang tinggi. Nilai similaritas yang tinggi ini mengindikasikan bahwa antara anggota genus Aquilaria dan Gyrinops memiliki similaritas sekuen trnL-trnF yang tinggi. Hal ini semakin mempertegas bahwa sebenarnya kedua genus ini secara genetis tidak perlu dipisahkan. Analisis similaritas antar taksa selanjutnya dilakukan dengan menggunakan cut of value sebesar 99. Untuk menjaring agar tidak terlalu banyak ditemukan pasangan taksa dengan indeks similaritas tinggi.
Berdasarkan hasil pembandingan nilai similaritas antar taksa, diperoleh pasangan taksa antar genus Gyrinops dan Aquilaria lebih banyak dibandingkan dengan pasangan taksa Gyrinops-Gyrinops dan Aquilaria-Aquilaria. Berdasarkan data tabel 3, 4 dan 5 Terdapat 24 pasangan sister taksa Gyrinops-Aquilaria, jauh lebih banyak dibandingkan dengan pasangan taksa Gyrinops-Gyrinops yang hanya 9 pasang, dan Aquilaria-Aquilaria yang hanya 13 pasang. Hal ini menunjukkan bahwa dari segi genetis, similaritas antara anggota Genus Gyrinops dan Aquilaria sangat tinggi sehingga seharusnya tidak dipisahkan menjadi genus berbeda. Similaritas yang tinggi antar anggota genus Aquilaria dan Gyrinops sesuai dengan penelitian Toruan-Mathius et al. (2009) bahwa berdasarkan analisis Amplified Fragment Length Polymorphysm (AFLP) antara anggota Aquilaria dan Gyrinops versteegii memiliki nilai similaritas yang tinggi.
PENUTUP
a. Simpulan
Sekuen gen trnL-trnF cocok digunakan untuk analisis filogenetik anggota genus Aquilaria meskipun memiliki keterbatasan jika digunakan untuk genus Gyrinops
b. Saran
Berdasarkan analisis filogenetik dan analisis hubungan similaritas, Aquilaria dan Gyrinops sebaiknya tidak dipisahkan menjadi genus berbeda.
DAFTAR PUSTAKA
Agrawal, R., N. Agrawal, R. Tandon and S. N. Raina. 2013. Chloroplast genes as genetic markers for inferring patterns of change, maternal ancestry and phylogenetic relationship among Elusine spescies. AoB PLANT 6: plt056; doi:10.1093/aobpla/ plt056
Chun, J. 1995. Computer-assisted classification and identification of actinomycetes. Ph. D. Thesis. University of Newcastle, United Kingdom.
Felsenstein, J. 1981. Evolutionary trees from DNA sequences: a maximum likelihood approach. J Mol Evol 17, 368–376
Gregory, R. T. 2008. Understanding Evolutionary Tree. Evo Edu Outreach (2008) 1:121–137 Koherani (2013). Studi pembungaan dan
perkembangan buah serta viabilitas poleh pohon gaharu (Gyrinops versteegii). Skripsi. Fakultas Matematika dan Ilmu Pengetahuan Alam Institut Pertanian Bogor. Bogor
Kumeta, Y. and M. Ito, 2011. Genomic organization of δ-Guanine synthase genes in Aquilaria crassna and its possible use for the identification of Aquilaria species. Journal of Natural Medicine, 65, 508-513 (2011).
Larkin M. A., G. Blackshields, N. P. Brown, R. Chenna, P. A. McGettigan, H. McWilliam, F. Valentin, I. M. Wallace, A. Wilm, R. Lopez, J. D. Thompson, T. J. Gibson, D. G. Higgins, 2007. ClustalW and ClustalX version 2. Bioinformatics23(21): 2947-2948.
Lee S. Y., W. L. Ng, M. N. Mahat, M. Nazre, R Mohammed. 2016. DNA Barcoding of the endangered Aquilaria (Thmelaeaceae) and its application in species authentication of agarwood products traded in the market. PLos ONE 11(4): e0154631. Doi:10.1371/ journal.pone.0154631.
Lengeler, J. W., G. Drews, H. G. Schlegel. 1999. Biology of Prokaryotes. Georg Thieme Verlag, Stuttgart.
Patwardhan, A., S. Ray and A. Roy., 2014. Molecular markers in phylogenetic studied-a review. J Phylogen Evolution Biol (2014), 2: 131
Saitou. N and M. Nei, 1987. The Neighbor-joining Method: A New Method for Reconstructing Phylogenetic Trees. Mol. Biol. Evol. 4(4):406-425. 1987
Schmidt, M. S., 2011. Introduction To CITES And Agarwood Overview. PC20 Inf. 7 Annex 9. Situmorang, J. 2000. Mikropropagasi Kayu Gaharu (Aquilaria spp) Asal Riau serta Identifikasi Sifat Genetiknya Berdasarkan Analisis Isoenzim. Tesis. Program Pascasarjana Institut Pertanian Bogor. Bogor.
Small R. L., R. C. Cronn and J. F. Wendel, 2004. Use of nuclear genes for phylogeny reconstruction in plants. Australian Systematic Botany 17, 145.
Tamura, K., Peterson, D., Peterson, N., Stecher, G., Nei, M., and S. Kumar.2011. MEGA5: Molecular Evolutionary GeneticsAnalysis using Maximum Likelihood, Evolutionary Distance,and Maximum Parsimony Methods. Mol Biol Evol.28, 2731-9. Toruan-Mathius, N., D. Rahmawati and Anidah,
2009. Genetic variation among Aquilaria species and Gyrinops versteegii using Amplified Fragment Length Polymorphysim Markers. BIOTROPIA. Vol. 16 No. 2, 2009: 88 – 95.
Wu, F. H., M. T. Chan, D. C. Liao, C. T. Hsu, Y.W. Lee, H. Daniell, M. R. Duvall and C. S. Lin., 2010. Complete chloroplast genome of Oncidium Gower Ramsey and evaluation of molecular markers for identification and breeding in Oncidiinae. BMC Plant Biology 2010, 10: 68
Yang, J. B., M. Tang, H. T. Li, Z. R. Zhang and D. Z. Li, 2013. Complete chloroplast genome of the genus Cymbidium: light into the species identification, phylogenetic implications and population genetic analyses. BMC Evolutionary Biology 2013, 13: 84.
Zich, F. And J. Compton. 2002. Agarwood (Gaharu) harvest and trade in Papua New Guinea: a preliminary Assessment .