BAB II
TINJAUAN PUSTAKA
2.1 Hutan Mangrove Sebagai Sumber Keanekaragaman Mikroorganisme Ekosistem mangrove adalah lingkungan yang unik, menyimpan beragam kelompok mikroorganisme yang melakukan peran penting dalam siklus nutrisi dan mengatur lingkungan kimia dari ekosistem. Berbagai kelompok bakteri seperti pemecah nitrogen, pelarut fosfat, pengurai selulosa, pengoksidasi belerang, pengoksidasi dan pereduksi besi lazim terdapat di ekosistem ini (Thatoi, et al., 2012).
Gambar 2.1 Hutan Mangrove di Taman hutan raya Ngurah Rai, Bali(Yusuf, 2010)
Keragaman mikroorganisme dan penyebarannya di hutan mangrove merupakan sumber daya yang potensial untuk dieksploitasi dalam bidang bioteknologi. Beberapa penelitian dengan hutan mangrove sebagai objeknya telah banyak dilakukan, diantaranya Verheyden, et al., (2005) meneliti tentang hutan mangrove di Belgia; Couto, et al., (2010) mengisolasi lipase baru dari sedimen mangrove Pantai Selatan Brazil; dan Zakaria, et al., (2010) mengisolasi spesies
Fusarium dari tanah mangrove Kampung Pantai Acheh-Malaysia. Selain itu, telah diuji aktivitas selulase dari Aspergillus fumigates pada tanah hutan mangrove di Kerala-India oleh Gilna dan Khaleel (2011).Mangamuri, et al., (2012) meneliti Actinomycetes langka dari ekosistem mangrove di Nizampatnam-India. Penelitian adanya aktivitas selulase pada tanah hutan Mangrove Pantai Suwung Bali telah dilakukan (Wirajana, et al., 2012; Kurniawati, et al., 2013).
2.2 DNA (Deoxyribose Nucleic Acid)
DNA (Deoxyribose Nucleic Acid) terutama ditemui dalam inti sel merupakan makromolekul polinukleotida yang tersusun atas polimer nukleotida yang berulang-ulang. Setiap nukleotida terdiri dari tiga gugus molekul, yaitu gula 5 karbon (2-deoksiribosa), basa nitrogen yang terdiri golongan purin, yaitu adenin (Adenin = A) dan guanin (guanini = G), serta golongan pirimidin, yaitu sitosin (cytosine = C) dan timin (thymine = T), gugus fosfat.
Adapun fungsi DNA adalah sebagai pembawa informasi genetik dari generasi ke generasi berikutnya, menyimpan dan mentransmisi informasi genetik, mengontrol aktivitas hidup secara langsung dan tidak langsung, mensintesis RNA dan berperan dalam proses sintesis protein (Aryanti, 2010).
Pada tahun 1953, Frances Crick dan James Watson menemukan model molekul DNA sebagai suatu struktur heliks beruntai ganda, atau yang lebih dikenal dengan heliks ganda Watson-Crick tersusun rangkap, membentuk DNA heliks ganda dan berpilin ke kanan. Susunan struktur kimia komponen penyusun DNA adalah sebagai berikut : baik purin ataupun pirimidin yang berkaitan dengan deoksiribosa membentuk suatu molekul yang dinamakan nukleotida atau
deoksiribonukleotida yang merupakan prekursor elementer untuk sintesis DNA. Prekursor merupakan suatu unsur awal pembentukan senyawa deoksiribonukleotida yang berkaitan dengan gugus fosfat. DNA tersusun dari empat jenis monomer nukleotida. Keempat basa nitrogen nukleotida tidak sama jumlahnya di dalam DNA tetapi jumlah adenin (A) selalu sama dengan jumlah timin (T) dan jumlah guanin (G) dengan sitosin (C) selalu sama. Fenomena ini dinamakan ketentuan Chargaff. Adenin (A) selalu berpasangan dengan timin (T) dan membentuk dua ikatan hidrogen (A = T), sedagkan sitosin (C) selalu berpasangan dengan guanin (G) dan membentuk 3 ikatan hirogen (C ≡ G).
Stabilitas DNA heliks ganda ditentukan oleh susunan basa dan ikatan hidrogen yang terbentuk sepanjang rantai tersebut. Perubahan jumlah hidrogen menyebabkan ikatan C ≡ G memerlukan tenaga yang lebih besar untuk memisahkannya. Antara mononukleotida satu dengan yang lainnya berhubungan secara kimia melalui ikatan fosfodiester.
2.3 Isolasi DNA Metagenomik
Handelsman, et al., (2004) mengemukakan bahwa metagenomik merupakan bidang penelitian baru yang memungkinkan studi tentang genom diperoleh dari sampel lingkungan. Metagenomik dapat didefinisikan sebagai penerapan teknik genomik modern untuk studi komunitas mikroorganisme secara langsung dari lingkungan alami.
Penelitian menunjukkan bahwa hanya 0,001-0,1% dari total mikroorganisme air laut, 0,25% di air tawar, 0,25% dalam sedimen dan hanya 0,3% dari mikroorganisme tanah bisa diolah in vitro (Amann, et al., 1995; Singh, et al., 2008). Penelitian metagenomik saat ini sebagian besar telah berkembang karena pembangunan vektor kloning gen efisien seperti kromosom bakteri buatan (BAC) atau kosmid, (Xu, 2006; Babcock, et al., 2007) yang memungkinkan kloning dan ekspresi dari segmen DNA yang lebih besar dan kompleks (Singh, et al., 2008; Ghazanfar dan Azim, 2009).
Banyak mikroorganisme yang mampu mendegradasi limbah, membuat obat baru untuk aplikasi medis, memproduksi plastik ramah lingkungan, maupun sebagai bahan baku produk pangan tertentu. Dengan mengisolasi DNA dari organisme, memberikan kesempatan untuk mengoptimalkan proses dan mengadaptasi mikroorganisme untuk digunakan oleh masyarakat. Penerapan berharga lain dari metagenomik adalah bahwa metagenomik menyediakan kemampuan untuk secara efektif mengkarakterisasi keragaman genetik yang hadir dari sampel air, tanah dan rumen terlepas dari ketersediaan teknik kultur laboratorium (Ghazanfar, et al., 2010).
Metode analisis genom membutuhkan DNA bebas dari berbagai jenis pengotor, umumnya ditemukan dalam sampel lingkungan. DNA harus mewakili keseluruhan keragaman mikroorganisme dan mengandung DNA genomik utuh (Robe, et al., 2003; Schloss dan Handelsman, 2003). Metode isolasi DNA harus benar-benar disesuaikan dengan jenis bahan biologis yang terisolasi. Selain mengisolasi DNA, ukuran DNA yang diperoleh sangat penting. Fragmen DNA sisipan yang kecil cukup untuk analisis kelompok gen tunggal atau kecil, sedangkan fragmen DNA sisipan yang besar dibutuhkan untuk analisis jalur multigenik, organisasi genom atau analisis urutan fragmen DNA panjang (Gillespie, et al., 2005; Lorenz dan Schleper, 2002).
Kesulitan utama yang terkait dengan pendekatan metagenomik biasanya berhubungan dengan kontaminasi DNA yang telah dimurnikan oleh senyawa fenolik yang termurnikan bersama DNA. Senyawa-senyawa ini sulit dihilangkan. Selain itu, polifenol juga diketahui dapat mengganggu proses modifikasi enzimatis pada DNA yang telah dimurnikan (Tsai dan Olson, 1992).
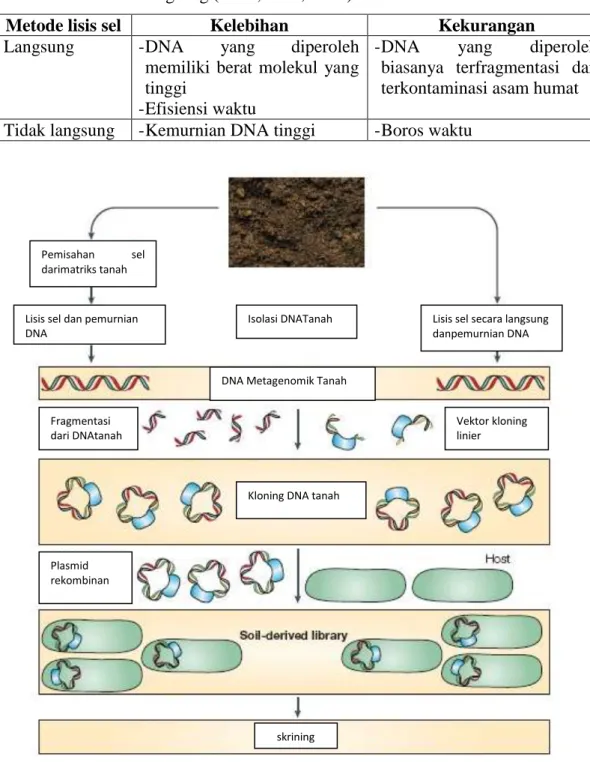
Saat ini, ada dua pendekatan untuk isolasi DNA genom. Pendekatan pertama didasarkan pada lisis sel langsung dari sampel lingkungan (lisisin situ). Pendekatan kedua didasarkan pada pemisahan sel-sel bakteri dari sampel lingkungan lalu lisis suspensi sel (lisis ex situ) (Lorenz dan Schleper, 2002).Tujuan dari pendekatan pertama adalah untuk mencapai lisis lengkap dari semua sel mikroorganisme yang ditemukan dalam sampel lingkungan, in situ. Umumnya, lisis in situ memberikan kualitas DNA yang lebih tinggi dibandingkan dengan metode tidak langsung (Robe, et al., 2003). Penerapan metode langsung meliputi dua langkah dasar, yaitu lisis sel dan ekstraksi DNA serta pemurnian.
Langkah pertama (lisis) sangat penting dalam hal efisiensi proses isolasi DNA metagenomik, dan karena berbagai mikroorganisme hadir dalam sampel, sulit untuk melakukan proses ini dengan benar (Roose-Amsaleg, et al., 2001). Faktor-faktor yang mempengaruhi efisiensi proses isolasi DNA metagenomik antara lain kerentanan dinding sel mikroba untuk lisis, dari bahan mana DNA terisolasi, dan interaksi dengan partikel lingkungan, misalnya tanah (Robe, et al., 2003). Faegri, et al., (1977) dan kemudian Torsvik dan Goksoyr (1978) telah mengusulkan metode isolasi DNA genom dari sampel lingkungan setelah pemisahan awal sel mikroorganisme (ex situ). Metode isolasi DNA metagenomik secara tidak langsung terdiri atas langkah-langkah berikut, yaitu dispersi dari sampel lingkungan, pemisahan sel, lisis sel, isolasi DNA dan pemurnian (Robe, et al., 2003).
Prosedur lisis langsung lebih tepat digunakan bila diperlukan hasil DNA dengan jumlah yang besar, mikroorganisme terbatas, dan bila keanekaragaman seluruh sampel lingkungan harus diteliti dengan bias minimal. Salah satu kelemahan metode ini adalah ekstrak DNA yang dihasilkan biasanya terfragmentasi dan terkontaminasi dengan asam humat. Selain itu, ekstrak sering mengandung DNA ekstraseluler dan/atau eukariotik dengan jumlah yang tidak diketahui. Metode ekstraksi tidak langsung, meskipun memakan waktu, lebih dipilih bila yang menjadi sasaran adalah sel-sel prokariotik, bila diperlukan kemurnian DNA yang tinggi, dan bila diperlukan DNA dengan berat molekul yang tinggi (tidak terfragmentasi) (Robe, et al., 2003).
Tabel 2.1 Kelebihan dan Kekurangan Metode Lisis Sel Secara Langsung dan Tidak Langsung (Robe, et al., 2003)
Gambar 2.3.1 Langkah-langkah penting untuk mengeksplorasi dan mengeksploitasi keragaman genom dari komunitas mikroorganisme tanah dengan metagenomik (Daniel, 2005)
Saat ini, ada tiga metode dasar lisis sel mikroorganisme yang digunakan, yaitu fisik, kimia dan enzimatik. Metode-metode ini dapat digunakan sebagai
Metode lisis sel Kelebihan Kekurangan
Langsung -DNA yang diperoleh memiliki berat molekul yang tinggi
-Efisiensi waktu
-DNA yang diperoleh biasanya terfragmentasi dan terkontaminasi asam humat Tidak langsung -Kemurnian DNA tinggi -Boros waktu
Pemisahan sel darimatriks tanah
Lisis sel dan pemurnian DNA
Lisis sel secara langsung danpemurnian DNA Isolasi DNATanah
DNA Metagenomik Tanah
Fragmentasi dari DNAtanah
Vektor kloning linier
Kloning DNA tanah
Plasmid rekombinan
prosedur independen, tetapi paling sering digunakan dalam berbagai kombinasi, sehingga meningkatkan efisiensi proses (Urban and Adamczak, 2008).
Penerapan metode fisik pada lisis sel umumnya memudahkan akses ke setiap sel tunggal yang akan menghasilkan efisiensi yang lebih tinggi dari isolasi DNA. Perlakuan fisik secara radikal akan menghasilkan DNA dengan ukuran yang lebih kecil. Metode fisik meliputi: pembekuan-pencairan dan siklus pembekuan-pendidihan, yang memanfaatkan efek pemecahan dari kristal es dan suhu pada dinding sel mikroorganisme (More, et al., 1994). Metode thermal shock adalah metode lisis yang lebih lembut daripada metode mekanis, meskipun sama-sama efisien. Jumlah siklus, waktu inkubasi dalam nitrogen cair atau es dan pemanasan pada suhu 50°C, 60°C, 100°C semua dapat bervariasi (Picard, et al., 1992). Metode lisis sel yang dimodifikasi oleh Kauffmann, et al., (2004) meliputi tiga siklus pembekuan sampel tanah dicampur dengan buffer ekstraksi dalam nitrogen cair dan pencairan di water bath pada 65°C. Metode ini relatif lembut untuk isolasi DNA sehingga tidak efektif untuk pelepasan asam nukleat dari sampel lingkungan. Metode yang lebih mudah untuk lisis sel adalah bead-beating, yang dapat digunakan untuk memperoleh sisipan ukuran yang sangat berbeda, misalnya 0,1-0,5 kb (Picard, et al., 1992), 2-5 kb (Gillespie, et al., 2005) bahkan 20 kb (Yeates, et al., 1997). Metode fisik lainnya yang digunakan dalam mengisolasi DNA genomik meliputi penggilingan pada mortar, penggilingan dalam nitrogen cair, dan penerapan ultrasound (Robe, et al., 2003). Orsini dan Romano-Spica (2001) berhasil menggunakan microwave untuk memisahkan asam nukleat dari sampel tanah. DNA yang diperoleh menunjukkan kemurnian yang lebih tinggi dengan ukuran 20-23 kb. Sebuah hasil DNA yang lebih tinggi
dibandingkan dengan sampel DNA yang diperoleh setelah lisis enzimatik sel. Keuntungan dari metode ini juga memerlukan biaya yang rendah, penyelesaian cepat, dan kemungkinan menggunakannya untuk isolasi mRNA (Urban and Adamczak, 2008).
Metode lisis sel secara kimia dapat digunakan secara terpisah atau kombinasi dengan metode fisik atau enzimatik yang sangat umum digunakan untuk isolasi DNA. Deterjen yang paling populer digunakan untuk isolasi DNA adalah satrium dodesil sulfat (SDS), yang berfungsi untuk mengeluarkan lemak yang ditemukan dalam membran sel mikroorganisme (Roose-Amsaleg, et al., 2001). Deterjen ini dapat digunakan dalam berbagai konsentrasi (0,1% - 20%), pada suhu tinggi (65°C), dan paling sering digunakan dengan senyawa pengkelat seperti EDTA (ethylenediaminetetraacetic acid) atau Chelex 100 (chelating resin dalam bentuk natrium) (Robe, et al., 2003). Peningkatan konsentrasi EDTA memiliki efek menguntungkan pada intensitas lisis namun mengurangi kemurnian DNA. Oleh karena itu, konsentrasi EDTA harus dipilih sangat hati-hati untuk mencapai kesesuaian antara jumlah dan kemurnian DNA (Krsek dan Wellington, 1999). Penerapan metode kimia dengan kombinasi getaran lembut dapat secara signifikan mengurangi kerusakan DNA, sehingga diperolah fragmen DNA hingga 80 kb, tetapi ukuran DNA yang diperoleh tergantung pada jenis mikroorganisme yang ditemukan dalam yang sampel diperiksa (Rondon, et al., 2000). Niemi, et al., (2001) menemukan bahwa metode isolasi DNA yang terdiri atas lisis sel dalam larutan SDS dengan penambahan isothiosianat guanidin, dan pembekuan-pencairan atau homogenisasi dengan manik-manik, tidak memiliki pengaruh yang signifikan terhadap kandungan DNA tetapi mengurangi ukuran DNA,
dibandingkan dengan lisis lembut hanya dengan SDS. Jumlah yang lebih besar dari DNA diperoleh dengan menggunakan lisis termal dan mekanik, tetapi kemurnian DNA rendah (Zhou, et al., 1996). Dalam rangka meningkatkan kemurnian DNA selama lisis kimia dan/atau lisis fisik, digunakan senyawa seperti, CTAB (Cetyl trimethylammonium bromide) atau PVPP (polivinilpolipirolidon), yang sebagian dapat menghilangkan senyawa humus yang ditemukan dalam sampel lingkungan (Roose-Amsaleg, et al., 2001). CTAB lebih sering digunakan untuk isolasi DNA karena PVPP menyebabkan kerugian dalam hasil DNA (Zhou, et al., 1996) namun penggunaan PVPP memungkinkan untuk hasil yang lebih baik, sementara DNA dimurnikan dalam kolom berputar (Krsek dan Wellington, 1999).
Berbagai metode isolasi DNA didasarkan pada lisis enzimatik. Enzim yang paling sering digunakan adalah lisozim yang menghidrolisis polisakarida yang ditemukan pada dinding sel mikroorganisme (Niemi, et al., 2001; Roose-Amsaleg, et al., 2001). Enzim lain yang juga sering digunakan dalam lisis sel adalah proteinase K untuk menghilangkan kontaminasi protein (Robe, et al., 2003).
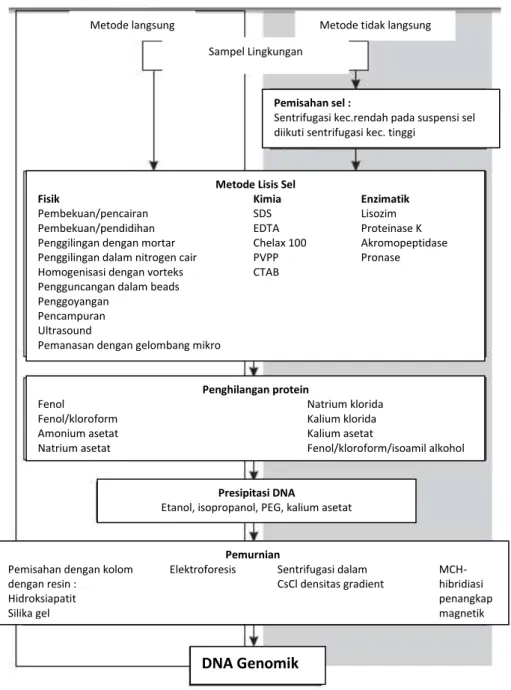
Gambar 2.3.2 Skema Isolasi DNA Metagenomik dengan Metode Langsung dan Tidak Langsung (Urban and Adamczak, 2008)
2.4 Elektroforesis DNA
Elektroforesis DNA merupakan teknik untuk memisahkan sampel DNA berdasarkan ukuran (berat molekul) sehingga molekul DNA dengan ukuran berbeda dapat dipisahkan. Gel yang biasa digunakan adalah agarosa yang merupakan suatu polisakarida yang diekstraksi dari rumput laut sehingga
Metode langsung Metode tidak langsung Sampel Lingkungan
Metode Lisis Sel
Fisik Kimia Enzimatik
Pembekuan/pencairan SDS Lisozim Pembekuan/pendidihan EDTA Proteinase K Penggilingan dengan mortar Chelax 100 Akromopeptidase Penggilingan dalam nitrogen cair PVPP Pronase Homogenisasi dengan vorteks CTAB
Pengguncangan dalam beads Penggoyangan
Pencampuran Ultrasound
Pemanasan dengan gelombang mikro
Penghilangan protein
Fenol Natrium klorida
Fenol/kloroform Kalium klorida Amonium asetat Kalium asetat
Natrium asetat Fenol/kloroform/isoamil alkohol
Presipitasi DNA
Etanol, isopropanol, PEG, kalium asetat
Pemurnian
Pemisahan dengan kolom Elektroforesis Sentrifugasi dalam MCH- dengan resin : CsCl densitas gradient hibridiasi
Hidroksiapatit penangkap
Silika gel magnetik
DNA Genomik
Pemisahan sel :
Sentrifugasi kec.rendah pada suspensi sel diikuti sentrifugasi kec. tinggi
elektroforesis ini dikenal dengan elektroforesis gel agarosa. Teknik ini sederhana, cepat, dan mampu memisahkan fragmen DNA sesuai dengan ukurannya. Elektroforesis gel agarosa dapat memisahkan sampel DNA dari ukuran beberapa ratus hingga 20 kilo pasang basa (pb). Gel agarosa digunakan sebagai media pergerakan (running) DNA.
Prinsip kerja dari elektroforesis ini adalah memisahkan molekul berdasarkan muatan listrik. Ion positif akan tertarik ke arah kutub negatif. Sebaliknya, ion negatif akan tertarik ke arah kutub positif serta akan saling tolak menolak jika muatannya sama. Molekul DNA bermuatan negatif (rangka gula fosfat) sehingga di dalam medan listrik akan bermigrasi melalui matriks gel ke arah kutub positif (anoda). Semakin besar ukuran molekulnya maka laju migrasi semakin rendah.
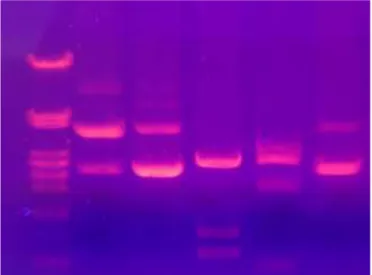
Elektroforesis gel agarosa terdiri atas beberapa komponen, antara lain : sampel DNA, gel agarosa (konsentrasi 0,5 – 2%), EtBr (agen pengkelat DNA) yang akan berpendar ketika terpapar dengan sinar UV, loading buffer, running buffer (memfasilitasi hantaran arus, biasanya TAE atau TBE), marker (penanda DNA) yang merupakan serangkaian fragmen DNA standar yang berguna untuk memperkirakan ukuran fragmen yang berbeda-beda pada DNA template yang dielektroforesis, serta lampu UV transilluminator yang membantu dalam visualisasi DNA (Fatchiyah, 2006).
Gambar 2.4.1 Rangkaian Alat Elektroforesis (Prasetya, 2011)
Gambar 2.4.2 Pita DNA hasil elektroforesis di bawah sinar UV (Anonim, 2006)
2.5 Analisis DNA Menggunakan Spektrofotometer UV-Vis
Penentuan DNA secara kuantitatif dapat dilakukan dengan menggunakan spektrofotometer UV-Vis. Besarnya absorbansi DNA pada pengukuran secara spektrofotometri akan berbanding lurus dengan konsentrasi DNA yang terkandung di dalam sampel. Untuk mengetahui jumlah DNA, maka DNA hasil
isolasi harus dianalisis dengan spektrofotometer UV-Vis dengan panjang gelombang 256 nm (260 nm).
Analisis kemurnian DNA pada panjang gelombang 230 nm dilakukan untuk mengetahui ada tidaknya kontaminasi dari asam humat. Asam humat diketahui dapat menganggu proses kloning ataupun ekspresi gen (Harry, et al., 1999). Analisis pada panjang gelombang 260 nm ditujukan untuk mengetahui tingkat kemurnian dari DNA linear untai ganda, sedangkan analisis pada panjang gelombang 280 dimaksudkan untuk mengetahui adanya kontaminasi protein. Hal ini dikarenakan asam nukleat dari DNA maupun RNA memiliki serapan yang kuat pada panjang gelombang 260 nm, namun protein memiliki serapan yang lebih lemah pada panjang gelombang tersebut. Sebaliknya, pada panjang gelombang 280 nm, protein memiliki serapan yang lebih kuat jika dibandingkan dengan asam nukleat dari DNA maupun RNA (Boyer, 1993).
Konsentrasi DNA dihitung dengan perbandingan bahwa pada panjang gelombang 260 nm, 1 unit absorban sebanding dengan 50 μg/ml DNA (Brown, 2003). Sehingga konsentrasi DNA bisa didapat melalui perkalian nilai absorban dengan faktor pengenceran dan 50 μg/ml. Kemurnian DNA ditentukan dengan indeks kemurnian. DNA dikatakan murni bila memiliki indeks kemurnian antara 1,6 - 1,8. Kemurnian tersebut dihitung berdasarkan kemurnian terhadap protein (A260/A280) dan asam humat (A260/A230).
Kadar Asam Nukleat (µg/mL) = 𝐴𝑏𝑠𝑜𝑟𝑏𝑎𝑛𝑠𝑖 𝜆260. 50 µ𝑔/𝑚𝐿