KARAKTERISASI GEN
nif
D BAKTERI METANOTROF ASAL SAWAH
RESTI RACHMAWATI
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
ABSTRAK
RESTI RACHMAWATI. Karakterisasi gen nifD Bakteri Metanotrof Asal Sawah. Dibimbing oleh IMAN RUSMANA dan ALINA AKHDIYA.
Bakteri metanotrof adalah bakteri pengoksidasi CH4. Beberapa metanotrof mampu memfiksasi nitrogen dengan menggunakan enzim nitrogenase. Enzim nitrogenase tersusun atas dua komponen protein besi. Komponen I mengandung situs aktif untuk mereduksi nitrogen, dikodekan oleh gen nifD dan nifK. Isolat metanotrof BGM 3 dan BGM 9 memiliki akumulasi amonium yang tinggi pada media Nitrat Mineral Salt (NMS) tanpa nitrogen. Fragmen gen nifD dari BGM 3 dan BGM 9 di amplifikasi dan disekuensing. Sekuen yang diperoleh dianalisis dengan menggunakan perangkat bioinformatik seperti BLAST-N, BLAST-X, Expasy Translate Tools, Conserved Domain (CDD) dan Scan PROSITE. Kontruksi filogenetik dibuat dengan menggukan program MEGA 4. Analisis sekuen menunjukkan bahwa sekuen gen nifD dari BGM 3 dan BGM 9 mirip dengan gen nifD pada Xanthobacter autotropicus PY2. Nilai kemiripan dari sekuen gen nifD tersebut cukup tinggi ( 92%-91% ). Gen nifD BGM 3 dan BGM 9 mengkodekan protein FeMo yang didalamnya terdapat klaster 4Fe-4S dan 2Fe-2S. Gen nifD dari BGM 3, BGM 9 diduga dimiliki oleh nenek moyang yang sama dengan Xanthobacter autotropicus PY2 dan Bradyhizobium japonicum USDA 110.
Kata kunci: Bakteri metanotrof, fiksasi nitrogen, nitrogenase, nifD
ABSTRACT
RESTI RACHMAWATI. Characterization nifD gene of Methanotrophic Bacteria from Ricefields. Under supervision of IMAN RUSMANA and ALINA AKHDIYA.
Methanotrophic bacteria are methane oxidizing bacteria. Some methanotrophs able of fix nitrogen using nitrogenase enzyme. Nitrogenase is composed of two multisubunit metallo protein. Component I contains the active site to nitrogen reduction, encoded by the nifD and nifK genes. Methanotrophic isolates of BGM 3 and BGM 9 had high ammonium accumulation cultured in Nitrate Mineral Salts (NMS) free nitrogen medium. nifD gene fragmens from BGM 3 and BGM 9 were amplified and sequenced. Sequences analyzed using bioinformatics tools such as Blast-N, Blast-X, Expasy Translate Tools, Conserved Domain (CDD) and Scan PROSITE. Phylogenetic construction made using MEGA 4 software. Sequence analysis showed that the nifD sequences of BGM 3 and BGM 9 most closely related to nifD fragmen of Xanthobacter autotropicus PY2. The Identity values of nifD sequences was 92% and 91% respectively. nifD genes from these isolates encode FeMo protein, which was 4Fe4S and 2Fe2S clusters. The nifD gene of BGM 3 and BGM 9 suspected to have same ancestor with the nifD gene of Xanthobacter autotropicus PY2 and Bradyhizobium japonicum USDA 110.
KARAKTERISASI GEN
nif
D BAKTERI METANOTROF ASAL SAWAH
RESTI RACHMAWATI
Skripsi
sebagai salah satu syarat untuk memperoleh gelar
Sarjana Sains pada
Departemen Biologi
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
Judul Skripsi :
Karakterisasi Gen
nif
D Bakteri Metanotrof Asal Sawah
Nama :
Resti Rachmawati
NIM :
G34062348
Menyetujui :
Pembimbing I,
Pembimbing II,
Dr. Ir. Iman Rusmana, M.Si Alina Akhdiya
NIP 196507201991031002
NIP 196812082001122001
Mengetahui:
Ketua Departemen Biologi
Fakultas Matematika dan Ilmu Pengetahuan Alam
Institut Pertanian Bogor
(Dr. Ir. Ence Darmo Jaya Supena, M.Sc)
NIP 196410021989031002
PRAKATA
Alhamdulillahirobbil’alamin, puji syukur penulis panjatkan kehadirat Allah SWT yang telah memberi rahmat dan kemudahan dalam menyelesaikan karya ilmiah ini. Tema penelitian penulis yaitu tentang kemampuan fiksasi nitrogen bakteri metanotrof, dengan judul Karakterisasi gen nifD Bakteri Metanotrof Asal Sawah. Penelitian ini dilaksanakan mulai bulan Februari sampai November 2010 di Laboratorium Mikrobiologi Departemen Biologi, Institut Pertanian Bogor dan Laboratorium Biologi Tanah Departemen Tanah, Institut Pertanian Bogor.
Penulis mengucapkan terima kasih kepada Bapak Dr. Ir. Iman Rusmana, M.Si. dan Ibu Alina Akhdiya, M.Si. selaku pembimbing atas saran dan bimbingannya dalam pelaksanaan penelitian dan penyusunan karya ilmiah ini. Di samping itu, penulis sampaikan terima kasih dan syukur kepada keluarga atas segala doa dan kasih sayangnya, kepada Yohan aripin dan teman-teman Biologi angkatan 43 khususnya yang melakukan penelitian di laboratorium Mikrobiologi. Terima kasih penulis ucapkan kepada Bu Ratna, Mba Ari, Ka Fina, Bang Jo, Dana, Tea, Vina, Rio dan Magda Serta pihak-pihak yang secara tidak langsung telah membantu dalam pengumpulan data karya ilmiah ini.
Semoga karya ilmiah ini bermanfaat.
Bogor, 23 Desember 2010
RIWAYAT HIDUP
Penulis dilahirkan di Bogor Jawa Barat pada tanggal 7 Juli 1988 dari ayahanda Ence Abdurahman dan ibunda Siti Rogayah. Penulis merupakan anak pertama dari tiga bersaudara. Tahun 2006 penulis lulus dari SMU Kornita IPB dan lolos seleksi masuk IPB melalui jalur Undangan Seleksi Masuk IPB (USMI) pada Departemen Biologi Fakultas Matematika dan Ilmu Pengetahuan Alam.
DAFTAR ISI
Halaman
DAFTAR TABEL ... vii
DAFTAR GAMBAR ... vii
DAFTAR LAMPIRAN ... vii
PENDAHULUAN ... 1
Latar Belakang ... 1
Tujuan Penelitian ... 1
Waktu dan Tempat ... 1
BAHAN DAN METODE ... 1
Bahan dan Alat ... 1
Metode Penelitian ... 1
Pemurnian dan peremajaan isolat. ... 1
Isolasi DNA, Amplifikasi Gen nifD, dan Visualisasi Amplikon ... 1
Sekuensing DNA dan Analisis Bioinformatika ... 2
HASIL DAN PEMBAHASAN ... 2
Hasil... ... 2
Isolasi DNA, Amplifikasi Gen nifD, dan Visualisasi Amplikon ... 2
Sekuensing DNA dan Analisis Bioinformatika ... 2
Pembahasan ... 4
SIMPULAN ... 5
DAFTAR PUSTAKA ... 6
DAFTAR TABEL
Halaman
1. Hasil analisis sekuen gen nifD dengan menggunakan program BLAST-N ... ...3
2. Hasil analisis sekuen gen nifD dengan menggunakan program BLAST-X ... 3
3. Analisis bioinformatik protein hasil translasi gen nifD BGM 3 dan BGM 9 dengan menggunakan program SCAN PROSITE ... 4
DAFTAR GAMBAR
Halaman 1. Amplifikasi gen nifD menghasilkan pita DNA berukuran ~1.9 kb . ... 2
2. Filogenetik gen nifD bakteri metanotrof BGM 3 dan BGM 9 yang dibandingkan dengan gen nifD dari beberapa spesies bakteri diazotrof menggunakan metode Neighbor Joining (NJ) dan boostrop 100x ... 3
3. Analisis bioinformatik protein turunan sekuen gen nifD BGM 3 dan BGM 9 dengan menggunakan program CDD (Conserved Domain) ... 4
4. Struktur kuartener nitrogenase MoFe Alpa dengan menggunakan program Cn3D 4.1 ... 5
DAFTAR LAMPIRAN
Halaman 1. Sekuensing DNA ... 9
Urutan nukleotida hasil sekuen gen nifD BGM 3 ... 9
Urutan nukleotida hasil sekuen gen nifD BGM9 ... 9
Protein hasil translasi gen nifD BGM 3 dan BGM 9 ... 9
2. Hasil BLAST-N dan BLAST-X ... 10
Hasil BLAST-N sekuen gen nifD BGM 3 ... 10
PENDAHULUAN
Latar Belakang
Bakteri metanotrof adalah bakteri gram negatif, bersifat aerob dan menggunakan metan sebagai sumber karbon penghasil energi (Hanson & Hanson 1996). Bakteri metanotrof tipe II dan tipe X telah diketahui memiliki kemampuan dalam menambat nitrogen sedangkan hanya beberapa bakteri metanotrof tipe I yang dapat memfiksasi nitrogen (Auman et. al 2001). Kemampuan tersebut terkait dengan keberadaan enzim nitrogenase (Dedysh et al. 2004).
Enzim nitrogenase merupakan kompleks enzim yang bertanggung jawab dalam proses fiksasi ntrogen. Nitrogenase terdiri atas dua komponen, yaitu komponen I (dinitrogenase atau protein Fe-Mo) dan komponen II (dinitrogenase reduktase atau protein Fe). Komponen I merupakan heterodimer dengan berat molekul 250 kDA yang dikodekan oleh gen nifD dan nifK, dan komponen II merupakan homodimer dengan berat molekul 70kDA yang dikodekan oleh gen nifH (Zehr et al. 1989).
Terdapat sekitar 20 gen nif yang mengkodekan nitrogenase yaitu nifA , nifB, nifZ, nifH, nifD, nifK, nifE, nifN, nifX, nifO, nifU, nifS, nifV, nifW, nifT, nifY, nifE, nifM, nifF, nifL (Lee et al. 2000) dan diatara 20 gen tersebut nifH dan nifD merupakan gen terpenting untuk mengenali adanya nitrogenase karena mengkodekan sub unit pembentuk nitrogenase. Gen struktural nifH, D, dan K pada dan proteobakteria terletak berdampingan dalam satu operon sehingga membentuk operon nifHDK, sedangkan diazotrof simbiotik yang tumbuh lambat mempunyai gen nifH, D, dan K yang terpisah membentuk operon nifH dan nifDK (Choo et al. 2003)
Diantara 40 isolat bakteri metanotrof yang diisolasi dari sedimen sawah di Bogor dan Sukabumi, tiga diantaranya (BGM 1, BGM 3, dan BGM 9) memiliki aktivitas oksidasi metan dan fiksasi nitrogen yang tinggi (Hapsary 2008, Sagala 2009). Isolat-isolat tersebut belum dikarakterisasi gen pengkode enzim nitrogenasenya.
Informasi tentang karakter enzim nitrogenase bakteri metanotrof asal Indonesia masih sangat sedikit, sehingga penelitian tersebut sangat diperlukan. Informasi tersebut bermanfaat untuk
mendukung pemanfaatan bakteri tersebut sebagai penambat nitrogen di lahan pertanian.
Tujuan Penelitian
Penelitian ini bertujuan mengkarakterisasi gen nifD pada isolat bakteri metanotrof BGM 3 dan BGM 9
Waktu dan Tempat
Penelitian ini dilaksanakan mulai bulan Februari 2010 sampai dengan bulan November 2010 di Laboratorium Mikrobiologi, Departemen Biologi, FMIPA, IPB.
BAHAN DAN METODE
Bahan dan AlatBahan-bahan yang digunakan, yaitu dua isolat metanotrof BGM 3 dan BGM 9, medium Nitrate Mineral Salt (NMS), Primer spesifik gen nifD dan komponen PCR kit KAPA Hifi Polimerase, 1 kb Ladder (Promega).
Peralatan yang digunakan, yaitu mesin PCR (Perkin Elmer Biosystem, USA), Laminar Air Flow, otoklaf, sentrifuse, elektroforesis mini-gel (BioRad Mini-Sub Cell GT, CA, USA), UV Transluminator (Hoefer Scientific Instruments, San Fransisco, USA), dan peralatan lainnya yang biasanya digunakan di laboratorium mikrobiologi
Metode Penelitian
Pemurnian dan peremajaan isolat. Pemurnian dan peremajaan isolat dilakukan dengan menggunakan metode cawan gores pada medium NMS, kemudian inkubasi dilakukan selama 14 hari pada suhu ruang (Hanson 1998).
Isolasi DNA, Amplifikasi gen nifD, dan Visualisasi Amplikon
sebanyak 5,0 µ L, 10mM dNTP mix (konsentrasi akhir 0,3mM) 0,75µ l, 10µ M primer (konsentrasi akhir 0,3µ M) 0,75µl, Template DNA (100ng) 0,5µ l, dan 0,5µl. KAPAHifi DNA Polymerase (1U/µl).
Sekuensing DNA dan Analisis Bioinformatika
Pengurutan DNA hasil amplifikasi dilakukan dengan memanfaatkan jasa perusahaan sekuensing 1st Base di Singapura. Sekuen yang diperoleh dijajarkan dengan data pada GenBank menggunakan program Blast-N, Blast-X dari situs NCBI (National Center for Biotechnology Information) melalui http://www.ncbi.nlm.nih.gov. untuk mengetahui tingkat kemiripan gen nifD dari isolat yang dianalisa. Penurunan sekuen DNA menjadi protein dilakukan menggunakan Expasy translate tools. Konstruksi pohon filogenetik dilakukan dengan menggunakan program MEGA 4.0 (Tamura et al. 2007), metode Neighbor Joining (NJ) dengan bootstrap 100x, sedangkan analisis protein dilakukan menggunakan Conserved Domain (CDD) dan SCAN PROSITE. Visualisasi struktur protein menggunakan program Cn3D 4.1.
HASIL DAN PEMBAHASAN
Hasil
Isolasi, Amplifikasi Gen nifD, dan Visualisasi Amplikon
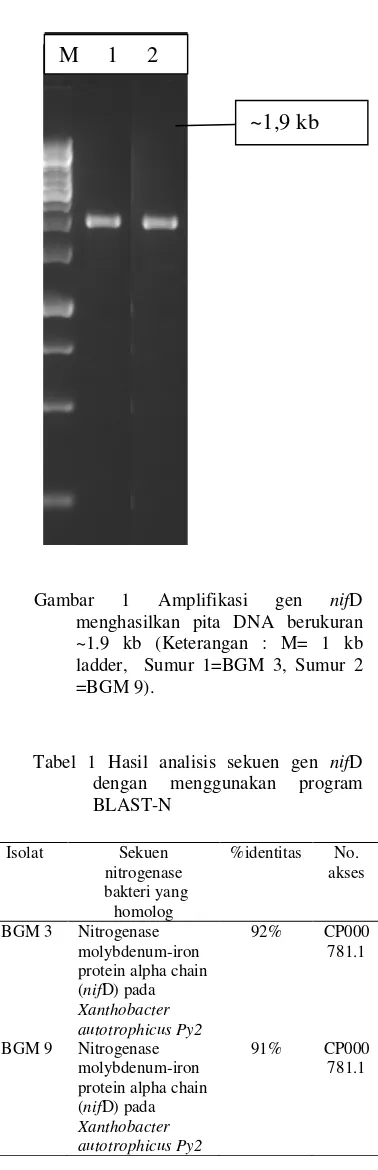
Amplifikasi gen nifD dari kedua isolat Metanotrof dengan menggunakan primer nifHD-F dan nifD-R dapat dilakukan. Hasil visualisasi amplikon pada gel elekroforesis yang diamati di atas UV transluminator memperlihatkan adanya pita DNA berukuran ~1.9 kb (Gambar 1).
Sekuensing DNA dan Analisis Bioinformatika
Produk amplifikasi gen nifD disekuen untuk mengetahui urutan nukleotidanya. Sekuen yang diperoleh (Lampiran 1) dianalisis kemiripannya dengan data di GenBank menggunakan program BLAST-N dan Blast-X (Tabel 1, Tabel 2, dan Lampiran 2). Berdasarkan analisis program tersebut diketahui homologi nitrogenase dari isolat yang diuji.
Gambar 1 Amplifikasi gen nifD menghasilkan pita DNA berukuran ~1.9 kb (Keterangan : M= 1 kb ladder, Sumur 1=BGM 3, Sumur 2 =BGM 9).
Tabel 1 Hasil analisis sekuen gen nifD dengan menggunakan program BLAST-N
Isolat Sekuen
nitrogenase bakteri yang
homolog
%identitas No.
akses
BGM 3 Nitrogenase
molybdenum-iron protein alpha chain (nifD) pada Xanthobacter autotrophicus Py2
92% CP000
781.1
BGM 9 Nitrogenase
molybdenum-iron protein alpha chain (nifD) pada Xanthobacter autotrophicus Py2
91% CP000
781.1
M 1 2
Tabel 2 Hasil analisis sekuen gen nifD dengan menggunakan program BLAST-X
Isolat Sekuen
nitrogenase bakteri yang
homolog
%identitas No.
akses
BGM 3 Nitrogenase
molybdenum-iron protein alpha chain (nifD) pada Xanthobacter autotrophicus Py2
72% YP_00
14150 05.1
BGM 9 Nitrogenase
molybdenum-iron protein alpha chain (nifD) pada Xanthobacter autotrophicus Py2
71% YP_00
14150 05.1
Analisis sekuen gen nifD menunjukkan bahwa BGM 3 dan BGM 9 memiliki gen nifD yang homolog dengan gen nifD Xanthobacter autotrophicus Py2 (Lampiran 2). Berdasarkan analisis filogenetik, gen nifD BGM 3 dan BGM 9 berkerabat dekat dengan Xanthobacter autotrophicus Py2 dan Bradyhizobium japonicum USDA 110 (Gambar 2). Gen nifD BGM 3 dan 9 termasuk superfamili protein Oxidoreductase nitrogenase dan sub famili Nitrogenase MoFe (Gambar 3).
Pada Nitrogenase MoFe alpa BGM3 dan BGM9 memiliki gugus 4Fe-4S ferredoxin-type iron-sulfur binding region (4Fe4S_FER_1) dan gugus 2 Fe-2S ferredoxin-type iron-sulfur binding region (Tabel 3).
Gambar 3 Analisis bioinformatik protein turunan sekuen gen nifD BGM 3 dan BGM 9 dengan menggunakan program CDD (Conserved Domain)
Tabel 3 Analisis bioinformatik protein hasil translasi gen nifD BGM 3 dan BGM 9 dengan menggunakan program SCAN PROSITE
Klaster pada domain
Motif sekuen Urutan sekuen No. Akses
4Fe4S_FER_1 CgGCGgCGtGCG 62-73 PS00198
2FE2S_FER_1 CTGGGCGGC, CTGATCTAC, CTGGTCGTC, CTGAACGTC, CTGGTCGGC
75-83, 159-167, 1236-1244, 1339-1347, 1792-1800
PS00197
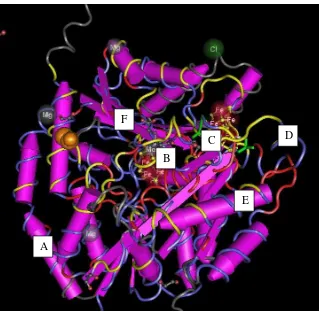
Gambar 4 Struktur kuartener nitrogenase Fe-Mo Alpa dengan menggunakan program Cn3D 4.1 (Keterangan : Warna kuning = Interaksi Fe-Mo sub unit alpa and
beta, A = dimer protein Fe-Mo, B = residu FeMoCo terikat, C = residu kluster P sub unit alpa terikat , D = Fe-Mo sub unit alpa/ kontak protein Fe, E = helix alpa, dan F = lembar beta ).5
A
B
C
F
D
Pembahasan
Amplifikasi gen nifD dengan primer nifHD-F dan nifD-R menghasilkan amplikon berukuran 1,9kb untuk BGM 3 dan BGM 9 (Gambar 1). Primer tersebut didesain dari sekuen nifH ke 436 dan sekuen nifD Bradyrhzobium japonicum USDA 110 sehingga menghasilkan amplikon rata-rata sebesar 1,9 kb. Amplikon ini didapat jika nifD, nifH, dan nifK terletak dalam satu operon (Dedysh et al. 2004). Transkripsi enzim nitrogenase pada dan proteobakteria dikodekan oleh operon nifHDK. Sedangkan pada diazotrof simbiotik yang tumbuh lambat enzim ini dikodekan oleh dua operon yang berbeda yaitu operon nifH dan nifDK (Choo et al. 2003).
Analisis Filogenetik menunjukkan bahwa BGM 3, BGM 9 dan Xanthobacter autotrophicus py2 dan Bradyrhizobium japonicum USDA 110 berkerabat dekat bila dilihat dari perkembangan gen nifD nya. (Gambar 2). BGM 3, BGM 9 dan Xanthobacter autotrophicus Py2 merupakan diazotrof yang hidup bebas sedangkan Bradyrhzobium japonicum USDA 110 merupakan diazotrof yang bersimbiosis dan memiliki operon nifH yang terpisah dari nifDK. Berdasarkan analisis gen 16S- rRNA isolat BGM 3 memiliki kemiripan terdekat dengan Methylocystisparvus strain 57 (69%), sedangkan BGM 9 memiliki kemiripan dengan Methylococcus capsulatus strain texas (83% dan 85%) (Astuti 2009). Nilai kemiripan sekuen nifD BGM 3 dan BGM 9 lebih tinggi dari analsis 16S-rRNA tersebut. Sehingga kemungkinan BGM 3 dan BGM 9 memiliki nenek moyang bakteri pemfiksasi nitrogen yang sama dengan Xanthobacter autotrophicus py2 dan Bradyrhizobium japonicum USDA 110.
Penelitian yang dilakukan Dedysh et al. (2004) juga menunjukkan bahwa nilai kemiripan (identity values) dari sekuen nifH dan nifD pada Methylocapsa acidiphila B2 dan Beijerinckia lebih tinggi (98.5 % dan 96.6 %) dibandingkan dengan hubungan kekerabatannya berdasarkan 16S rRNA sehingga kemungkinan dua bakteri tersebut berasal dari nenek moyang bakteri pemfiksasi nitrogen yang sama.
Nitrogenase oxidoreduktase merupakan sekelompok famili protein yang mengkatalisis reduksi nitrogen menjadi amonium dengan menggunakan ATP.
Selain ATP, proses tersebut membutuhkan feredoksin tereduksi sebagai donor elektron, sitokrom sebagai protein pembawa elektron, dan koenzim. Proses fiksasi satu mol nitrogen ini memerlukan energi sel sebanyak 16 ATP.
Oxidoreduktase memiliki sub unit alpa dan sub unit beta dari komponen I yang secara genetik terbagi atas nitrogenase yang tergantung pada atom molibdenum (Mo-nitrogenase), nitrogenase yang tergantung pada atom vanadium (V-nitrogenase), dan nitrogenase besi (Fe-nitrogenase). Nitrogenase alternatif vanadium dan nitrogenase yang hanya mengandung besi saja masing-masing dikodekan oleh gen vnf dan anf dan mempunyai struktur heksamer yang dikodekan oleh operon vnfDGK dan anfDGK. (Caton 2007). Selain itu terdapat beberapa sub unit Protochlorophyllide (Pchlide) reductase and chlorophyllide (chlide) reductase (Gambar 3).
SIMPULAN
Amplifikasi gen nifD BGM 3 dan BGM 9 dengan menggunakan primer nifHD-F dan nifD-R menghasilkan amplikon berukuran 1,9kb. Gen nifD, nifH dan gen nifK pada isolat bakteri metanotrof BGM 3 dan BGM 9 terletak dalam satu operon. Sekuen gen nifD BGM 3 dan BGM 9 memiliki kemiripan terdekat dengan sekuen gen nifD Xanthobacter autotrophicus Py2.
DAFTAR PUSTAKA
Alhapel A et al. 2006. Molecular and functional analysis of nicotinate catabolism in Eubacterium barkeri. Proc Natl Acad Sci 103: 12341-12346.
Astuti DD. 2009. Karakterisasi fisiologi dan identifikasi molekuler isolat-isolat bakteri metanotrof asal sawah wilayah Bogor dan Sukabumi. [Skripsi]. Bogor: Departemen Biologi, Institut Pertanian Bogor.
Auman AJ, Speake CC, Lidstrom ME. 2001. nifH sequences and nitrogen fixation in type I and type II methanotrophs. Appl Environ Microbiol 67(9): 4009-4016.
Caton IR. 2007. Abundance of nifH genes in urban, agricultural, and pristine prairie streams exposed to different levels of nitrogen loading [thesis]. Wichita State University.
Choo QC, Samian MR, Najimudin N. 2003. Phylogeny and characterization of three nifH-homologous genes from Paenibacillus azotofixans. Appl Environ Microbiol 69(6): 3658-3662.
Dedysh SN, Ricke P, Liesack W. 2004. NifH and NifD phylogenies: an evolutionary basis for understanding nitrogen fixation capabilities of methanotrophic bacteria. Microbiol 150; 1301– 1313.
Hanson RS. 1998. Ecology of methylotrophic bacteria. Di dalam:
Burlage RS, Atlas R, Stahl, Geesey G, Dayler G, editor. Techniques in microbial Ecology. Oxford: Oxford University Press. 137- 162.
Hanson R, Hanson TE. 1996. Metanotrophic Bacteria. J Microbiol Reviews 60 : 439-471
Hapsary W. 2008. Isolasi dan karakterisasi bakteri metanotrof asal sawah di Bogor dan Sukabumi [Skripsi]. Bogor: Departemen Biologi, Institut Pertanian Bogor.
Lazo GR, Roffey R, Gabriel DW. 1987. Conservation of plasmid DNA sequences and pathovar identification of strain
Xanthomonas campestris.
Pytopathology 77: 1461-1467.
Lee S, Reth A, Meletzus D, Sevilla M, Kennedy C. 2000. Characterization of major cluster of nif, fix, and associated genes in sugarcane endophyte, Acetobacter diazotrophicus. J Bacteriol. 182(24): 7088-7091.
Sagala BT. 2009. Seleksi dan uji aktivitas fiksasi nitrogen (N2) bakteri metanotrof asal sawah pada konsentrasi oksigen (O2) berbeda [skripsi]. Bogor : Departemen Biologi, Institut Pertanian Bogor.
Tamura K, Dudley J, Nei M, Kumar S. 2007. MEGA4: Molecular evolutionary genetics analysis (MEGA) software version 4.0. Mol Biol Evol 24:1596–1599.
Lampiran 1. Hasil sekuen gen nifD
A. Urutan nukleotida hasil sekuen gen nifD BGM 3
CGCGAAGAGGCATGTATGCGGCCAACAACATCTCCAAGGGCATCCTGAAGTATGCGAACTCCGGCGGCGTG CGCCTGGGCGGCCTGGTGTGCAACGAGCGCCAGACCGACAAGGAGTACGAGCTGGCGGAGTCGCTGGCGAA GAAGCTCGGCACGCAGCTGATCTACTTCGTGCCGCGCGACAACATCGTGCAGCACGCCGAACTGCGCCGCAT GACGGTGATCGAGTATGCGCCCGATTCCGCGCAGGCCCAGCACTACCGGAACCTTGCGACCAAGGTGCACGG CAACTCGGGCAACGGCATCATCCCGACCCCGATCACCATGGACGAGCTCGAAGACCTGCTCATGGAGCACGG CATCATGAAGGCCGTGGACGAGAGCATCGTCGGCAAGACCGCCGCAGAGCTGGCCGTCGGCTGAAGCCATC GCGTCGATTGACCTGGGGCCGGCCTCAATTGCGGGGCCGGCCTGCCGAACGGCAACCCAACGAGAGCGAGA TTGCGAAATGAGCTTGGCCCAACCGCAAAACGTTGCTGAAATCAAGGCGCGGAACAAGGAACTCATCGCGG AAGTCCTCAAGGTTTATCCCGAGAAGACCGCGAAGCGCCGCGCGAAGCACCTCAACGTCCATGAGTCCGGCA AGTCCGACTGCGGCGTGAAGTCGAACATCAAGTCCATCCCGGGCGTGATGACGATCCGCGGCTGCGCCTACG CCGGTTCCAAGGGCGTGGTGTGGGGTCCGATCAAGGACATGATCCACATCTCCCACGGCCCCGTCGGCTGCG GCCAGTACAGCTGGGCCGCCCGCCGCAACTACTACATGGGCAGGACCGGAATGGACACCTTGGTGAGGATG
CAGTTCACGTCCGACTTCCAGGAAAAGGACATCGTTTTCGGCGGGGACAAGAAGCTCGCCAAGATCATGGAC
GAAATCCAGGAGCTCTTCCCGCTCAACAACGGCATCACTGTCCAGTCCGAGTGTCCGATCGGCCTCATCGGC
GACGACATCGAGGCCGTGTCCAAGTCGAAGTCCAAGGAATACGACGGCAAGACCATCGTTCCGGTCCGCTGC GAGGGCTTCCGCGGCGTGTCGCAGTCCCTCGGCCACCACATCGCCAACGACGCGGTGCGGGACTGGGTGTTC GACAAGCTGGACCCGAACAAGGCTCCGCCCTTCGAGCCCTCGCCGTATGACGTCGCCATCATCGGCGACTAC AACATCGGCGGCGATGCCTGGTCGTCCCGTATCCTCCTCGAGGAGATGGGCCTGCGCGTCATCGCCCAGTGG TCGGGCGACGGCTCGCTCGCCGAGCTCGAGAACACCCCGCGGGCGAAGCTGAACGTCCTGCACTGCTACCGC TCCATGAACTACATCTCCCGCCACATGGAAGAGAAGTTCGGGATCCCGTGGTGCGAGTACAACTTCTTCGGT CCCTCCAAGATCGCGGAGTCGCTGCGCAAGATCGCCGCTTACTTCGACGACAAGATCAAGGAAGGCGCCGA GCGCGTCATCGCCAAGTACGCGCCCCTCATGGACGCGGTGATCGCCAAGTATCGTCCGCGCCTCGAGGGCAA GACGGTCATGCTGTACGTGGGCGGCCTGCGTCCCCGTCACGTGATCGGCGCGTACGAAGATCTCGGCATGGA AGTGGTGGGCACGGGTTACGAGTTCGCCCACAACGACGACTATCAGCGCACCGCCCAGCACTACGTCAAGG ATGGCACGCTCATCTATGACGACGTGACCGGCTACGAGTTCGAGAAGTTCGTCGAGAAGGTCCAGCCGGATC TGGTCGGCTCGGGCATCAAGGAAAAGTACGTCTCCAGAAGATGCGCT
B. Urutan nukleotida hasil sekuen gen nifD BGM9
CCGCGAAGAGGCATGTTGCGGCCAACAACATCTCCAAGGGCATCCTGAAGTATGCGAACTCCGGCGGCGTG CGCCTGGGCGGCCTGGTGTGCAACGAGCGCCAGACCGACAAGGAGTACGAGCTGGCGGAGTCGCTGGCGA AGAAGCTCGGCACGCAGCTGATCTACTTCGTGCCGCGCGACAACATCGTGCAGCACGCCGAACTGCGCCGC ATGACGGTGATCGAGTATGCGCCCGATTCCGCGCAGGCCCAGCACTACCGGAACCTTGCGACCAAGGTGCA CGGCAACTCGGGCAACGGCATCATCCCGACCCCGATCACCATGGACGAGCTCGAAGACCTGCTCATGGAGC AGGCATCATGAAGGCCGTGGACGAGAGCATCGTCGGCAAGACCGCCGCAGAGCTGGCCGTCGGCTGAAGC CATCGCGTCGATTGACCTGGGGCCGGCCTCAATTGCGGGGCCGGCCTGCCGAACGGCAACCCAACGAGAGC GAGATTGCGAAATGAGCTTGGCCCAACCGCAAAACGTTGCTGAAATCAAGGCGCGGAACAAGGAACTCAT CGCGGAAGTCCTCAAGGTTTATCCCGAGAAGACCGCGAAGCGCCGCGCGAAGCACCTCAACGTCCATGAGT CCGGCAAGTCCGACTGCGGCGTGAAGTCGAACATCAAGTCCATCCCGGGCGTGATGACGATCCGCGGCTGC GCCTACGCCGGTTCCAAGGGCGTGGTGTGGGGTCCAATCAAGGACATGATCCACATCTCCCACAAATTTTCC CAAGGGCCCCGTTGGGTGGGGCCAGTAAAGGTGGGCCGCCCGCCGGAAAATTATACATTGGCACGACCGG
CATCGACCCCTTCGGGACGATGCAGTTCACGTCCGATTTCCAGGAAAAGGACATCGTTTTCGGCGGCGACA
AGAAGCTCGCCAAGATCATGGACGAAATCCAGGAGCTCTTCCCGCTCAACAACGGCATCACTGTCCAGTCC
GAGTGCCCGATCGGCCTCATCGGCGACGACATCGAGGCCGTGTCCAAGTCGAAGTCCAAGGAATACGACGG
CAAGACCATCGTTCCGGTCCGCTGCGAGGGCTTCCGCGGCGTGTCGCAGTCCCTCGGCCACCACATCGCCA ACGACGCGGTGCGGGACTGGGTGTTCGACAAGCTGGACCCGAACAAGGCTCCGCCCTTCGAGCCCTCGCCG TATGACGTCGCCATCATCGGCGACTACAACATCGGCGGCGATGCCTGGTCGTCCCGTATCCTCCTCGAGGAG ATGGGCCTGCGCGTCATCGCCCAGTGGTCGGGCGACGGCTCGCTCGCCGAGCTCGAGAACACCCCGCGGGC GAAGCTGAACGTCCTGCACTGCTACCGCTCCATGAACTACATCTCCCGCCACATGGAAGAGAAGTTCGGGA TCCCGTGGTGCGAGTACAACTTCTTCGGTCCCTCCAAGATCGCGGAGTCGCTGCGCAAGATCGCCGCTTACT TCGACGACAAGATCAAGGAAGGCGCCGAGCGCGTCATCGCCAAGTACGCGCCCCTCATGGACGCGGTGATC GCCAAGTATCGTCCGCGCCTCGAGGGCAAGACGGTCATGCTGTACGTGGGCGGCCTGCGTCCCCGTCACGT GATCGGCGCGTACGAAGATCTCGGCATGGAAGTGGTGGGCACGGGTTACGAGTTCGCCCACAACGACGACT ATCAGCGCACCGCCCAGCACTACGTCAAGGATGGCACGCTCATCTATGACGACGTGACCGGCTACGAGTTC GAGAAGTTCGTCGAGAAGGTCCAGCCGGACCTGGTCGGCTCGGGCTCAAGGAAAAGTACTCTCCAGAAGAT GCGGGCC
C. Protein hasil translasi gen nifD BGM 3 dan BGM 9 >5’3’ Frame 2 (BGM3)
DKKLAKIMDEIQELFPLNNGITVQSECPIGLIGDDIEAVSKSKSKEYDGKTIVPVRCEGFRGVSQSLGHHIANDAVR DWVFDKLDPNKAPPFEPSPYDVAIIGDYNIGGDAWSSRILLEEMGLRVIAQWSGDGSLAELENTPRAKLNVLHCY RSMNYISRHMEEKFGIPWCEYNFFGPSKIAESLRKIAAYFDDKIKEGAERVIAKYAPLMDAVIAKYRPRLEGKTVM LYVGGLRPRHVIGAYEDLGMEVVGTGYEFAHNDDYQRTAQHYVKDGTLIYDDVTGYEFEKFVEKVQPDLVGSG IKEKYVSRRCA
>5’3’ Frame 3 (BGM 9)
REEACCGQQHLQGHPEVCELRRRAPGRPGVQRAPDRQGVRAGGVAGEEARHAADLLRAARQHRAARRTAPHD GDRVCARFRAGPALPEPCDQGARQLGQRHHPDPDHHGRARRPAHGAGMKAVDESIVGKTAAELAVG*SHRVD* PGAGLNCGAGLPNGNPTRARLRNELGPTAKRC*NQGAEQGTHRGSPQGLSREDREAPREAPQRP*VRQVRLRREV EHQVHPGRDDDPRLRLRRFQGRGVGSNQGHDPHLPQIFPRAPLGGASKGGPPAGKLYIGTTGIDPFGTMQFTSDFQ EKDIVFGGDKKLAKIMDEIQELFPLNNGITVQSECPIGLIGDDIEAVSKSKSKEYDGKTIVPVRCEGFRGVSQSLGH HIANDAVRDWVFDKLDPNKAPPFEPSPYDVAIIGDYNIGGDAWSSRILLEEMGLRVIAQWSGDGSLAELENTPRA KLNVLHCYRSMNYISRHMEEKFGIPWCEYNFFGPSKIAESLRKIAAYFDDKIKEGAERVIAKYAPLMDAVIAKYRP RLEGKTVMLYVGGLRPRHVIGAYEDLGMEVVGTGYEFAHNDDYQRTAQHYVKDGTLIYDDVTGYEFEKFVEKV QPDLVGSGSRKSTLQKMRA
Keterangan : A=alanin B=aspartat/asparagin C=cystine D=aspartat E=glutamat F=Fenilalanin G=glysin
H=histidin I=isoleusin M=metionin N=asparagin P=prolin Q=glutamin R=arginin S=serin T=threonine V=valin W=tryptophan Y=tyrosin Z=glutamat X=any *=kodon stop
Lampiran 2. Hasil BLAST-N
A. Hasil BLAST-N sekuen gen nifD BGM3
gb|CP000781.1| Xanthobacter autotrophicus Py2, complete genome Features in this part of subject sequence:
nitrogenase iron protein
nitrogenase molybdenum-iron protein alpha chain
Score = 2529 bits (1369), Expect = 0.0
Identities = 1688/1838 (92%), Gaps = 37/1838 (2%)
Query 11 CATGTATGCGGCCAACAACATCTCCAAGGGCATCCTGAAGTATGCGAACTCCGGCGGCGT 70 ||||||||||||||||||||||||||||||||| ||||||||||| |||||||||||||| Sbjct 96890 CATGTATGCGGCCAACAACATCTCCAAGGGCATTCTGAAGTATGCCAACTCCGGCGGCGT 96949
Query 71 GCGCCTGGGCGGCCTGGTGTGCAACGAGCGCCAGACCGACAAGGAG-TACGAGCTGGCGG 129 |||||||||||| ||||||||||||||||||||||||||||||||| | |||||||| | Sbjct 96950 GCGCCTGGGCGGGCTGGTGTGCAACGAGCGCCAGACCGACAAGGAGCT-GGAGCTGGCCG 97008
Query 130 AG-TCGCTGGCGAAGAAGCTCGGCA-CGCAGCTGATCTACTTCGTGCCGCGCGACAACAT 187 || | ||||| ||||||||||||| || ||||||||||||||||||||||||||||||| Sbjct 97009 AGAAC-CTGGCCAAGAAGCTCGGCACCG-AGCTGATCTACTTCGTGCCGCGCGACAACAT 97066
Query 188 CGTGCAGCACGCCGAACTGCGCCGCATGACGGTGATCGAGTATGCGCCCGATTCCGCGCA 247 ||||||||||||||| |||||||||||||| ||||||||||||||||||||||| | || Sbjct 97067 CGTGCAGCACGCCGAGCTGCGCCGCATGACCGTGATCGAGTATGCGCCCGATTCGGAACA 97126
Query 248 GGCCCAGCACTACCGGAACCTTGCGACCAAGGTGCACGGCAACTCGGGCAACGGCATCAT 307 |||||||||||||||||||||||| |||||||| |||| |||| ||||||||||||||| Sbjct 97127 GGCCCAGCACTACCGGAACCTTGCCACCAAGGTTCACGCCAACAAGGGCAACGGCATCAT 97186
Query 308 CCCGACCCCGATCACCATGGACGAGCTCGAAGACCTGCTCATGGAGCACGGCATCATGAA 367 ||||||||| ||||| |||||||||||||| ||| |||| |||||||||||||||||||| Sbjct 97187 CCCGACCCCCATCACGATGGACGAGCTCGAGGACATGCTGATGGAGCACGGCATCATGAA 97246
Query 368 GGCCGTGGACGAGAG-CATCGTCGGCAAGACCGCCGCAGAGCTGGCCGTCGGCTGAAGCC 426 |||||| |||||||| || ||||||||||||||||| ||||| ||| | ||| || | Sbjct 97247 GGCCGTCGACGAGAGCCA-GGTCGGCAAGACCGCCGCCGAGCTCACCG-C--CTG-AGGC 97301
Query 427 ATCG-CGTCGATTGACCTGGGGCCGGCCTCAATTGCGGGGCCGGCCTGCCGAAC-GGCAA 484 | | || | | | | | |||||||| ||||||||||| |||||| | | | ||| | Sbjct 97302 -T-GACG-C-A--G--C-GAGGCCGGCCCCAATTGCGGGGTCGGCCT-TC-ATCTGGC-A 97349
Query 485 CCC--AACGAGAGCGAGATTGCGAAATGAGCTTGGCCCAACCGCAAAACGTTGCTGAAAT 542 ||| |||||||||||||||||||||||||||||||||||||||||| |||||||||||| Sbjct 97350 CCCAAAACGAGAGCGAGATTGCGAAATGAGCTTGGCCCAACCGCAAAGCGTTGCTGAAAT 97409
Sbjct 97410 CAAGGCGCGTAACAAAGAACTCATCGCGGAAGTTCTCAAGGTTTATCCCGAGAAGACCGC 97469
Query 603 GAAGCGCCGCGCGAAGCACCTCAACGTCCATGAGTCCGGCAAGTCCGACTGCGGCGTGAA 662 |||||||||||||||||||||||||||||| ||||||||||||||||| ||||||||||| Sbjct 97470 GAAGCGCCGCGCGAAGCACCTCAACGTCCACGAGTCCGGCAAGTCCGATTGCGGCGTGAA 97529
Query 663 GTCGAACATCAAGTCCATCCCGGGCGTGATGACGATCCGCGGCTGCGCCTACGCCGGTTC 722 |||||||||||||||||||||||||||||||||||||||||||||||| || |||||||| Sbjct 97530 GTCGAACATCAAGTCCATCCCGGGCGTGATGACGATCCGCGGCTGCGCGTATGCCGGTTC 97589
Query 723 CAAGGGCGTGGTGTGGGGTCCGATCAAGGACATGATCCACATCTCCCACGGCCCCGTCGG 782 ||||||||||||||||||||| |||||||||||||||||||||||||||||||| || || Sbjct 97590 CAAGGGCGTGGTGTGGGGTCCCATCAAGGACATGATCCACATCTCCCACGGCCCGGTGGG 97649
Query 783 CTGCGGCCAGTACAGCTGGGCCGCCCGCCGCAACTACTACATGGGCAGGACCGGAATGGA 842 |||||||||||||||||||||||||||||||||||||||||| |||| ||| || || || Sbjct 97650 CTGCGGCCAGTACAGCTGGGCCGCCCGCCGCAACTACTACATCGGCACGACGGGCATCGA 97709
Query 843 CACCTTGGTGAGGATGCAGTTCACGTCCGACTTCCAGGAAAAGGACATCGTTTTCGGCGG 902 |||||| |||| ||||||||||| || ||||||||||||||||||||||| |||||||| Sbjct 97710 CACCTTCGTGACCATGCAGTTCACCTCGGACTTCCAGGAAAAGGACATCGTCTTCGGCGG 97769
Query 903 GGACAAGAAGCTCGCCAAGATCATGGACGAAATCCAGGAGCTCTTCCCGCTCAACAACGG 962 |||||||||||||||||||||||||||||||||||||| || |||||||| |||||||| Sbjct 97770 CGACAAGAAGCTCGCCAAGATCATGGACGAAATCCAGGACCTGTTCCCGCTGAACAACGG 97829
Query 963 CATCACTGTCCAGTCCGAGTGTCCGATCGGCCTCATCGGCGACGACATCGAGGCCGTGTC 1022 |||||| |||||||||||||| |||||||||||||||||||||||||||||||||||||| Sbjct 97830 CATCACCGTCCAGTCCGAGTGCCCGATCGGCCTCATCGGCGACGACATCGAGGCCGTGTC 97889
Query 1023 CAAGTCGAAGTCCAAGGAATACGACGGCAAGACCATCGTTCCGGTCCGCTGCGAGGGCTT 1082 |||| |||||||||||||||||||| |||||||||||| |||||||||||||||||||| Sbjct 97890 CAAGGCGAAGTCCAAGGAATACGACAACAAGACCATCGTGCCGGTCCGCTGCGAGGGCTT 97949
Query 1083 CCGCGGCGTGTCGCAGTCCCTCGGCCACCACATCGCCAACGACGCGGTGCGGGACTGGGT 1142 |||||||||||| ||||| |||||||| |||||||||||||| ||| | ||||||||||| Sbjct 97950 CCGCGGCGTGTCCCAGTCGCTCGGCCATCACATCGCCAACGATGCGATCCGGGACTGGGT 98009
Query 1143 GTTCGACAAGCTGGACCCGAACAAGGCTCCGCC-CTTCGAGCCCTCGCCGTATGACGTCG 1201 |||||||||| |||||||||| ||| || || ||||||||| || ||||||||||||| Sbjct 98010 GTTCGACAAGATGGACCCGAATGCGGC-CCCCCGCTTCGAGCCGTCCCCGTATGACGTCG 98068
Query 1202 CCATCATCGGCGACTACAACATCGGCGGCGATGCCTGGTCGTCCCGTATCCTCCTCGAGG 1261 ||||||||||||||||||||||||| ||||| |||||||| || || ||||| ||||||| Sbjct 98069 CCATCATCGGCGACTACAACATCGGTGGCGACGCCTGGTCTTCGCGCATCCTGCTCGAGG 98128
Query 1262 AGATGGGCCTGCGCGTCATCGCCCAGTGGTCGGGCGACGGCTCGCTCGCCGAGCTCGAGA 1321 | |||||||||||||| ||||| |||||||| |||||||||||||| |||||||||||| Sbjct 98129 AAATGGGCCTGCGCGTGATCGCACAGTGGTCCGGCGACGGCTCGCTTGCCGAGCTCGAG- 98187
Query 1322 AC-ACCCCGCGGGCGAAGCTGAACGTCCTGCACTGCTACCGCTCCATGAACTACATCTCC 1380 | || ||| |||||||||||||||||||||||||||||| |||||||||||||||||| Sbjct 98188 GCGACTCCGAAGGCGAAGCTGAACGTCCTGCACTGCTACCGGTCCATGAACTACATCTCC 98247
Query 1381 CGCCACATGGAAGAGAAGTTCGGGATCCCGTGGTGCGAGTACAACTTCTTCGGTCCC-TC 1439 ||||||||||||||||||||||| ||||||||||||||||||||||||||||| ||| || Sbjct 98248 CGCCACATGGAAGAGAAGTTCGGTATCCCGTGGTGCGAGTACAACTTCTTCGG-CCCGTC 98306
Query 1440 CAAGATCGCGGAGTCGCTGCGCAAGATCGCCGCTTACTTCGACGACAAGATCAAGGAAGG 1499 ||||||||| ||||||||||| |||||||||||||||||||||||||||||||||||||| Sbjct 98307 CAAGATCGCCGAGTCGCTGCGTAAGATCGCCGCTTACTTCGACGACAAGATCAAGGAAGG 98366
Query 1500 CGCCGAGCGCGTCATCGCCAAGTACGC-GCCCCTCATGGACGCGGTGATCGCCAAGTATC 1558 ||||||||||||||||||||||||| | |||||||||||||||||| || |||||||| | Sbjct 98367 CGCCGAGCGCGTCATCGCCAAGTAC-CAGCCCCTCATGGACGCGGTCATTGCCAAGTACC 98425
Query 1559 GTCCGCGCCTCGAGGGCAAGACGGTCATGCTGTACGTGGGCGGCCTGCGTCCCCGTCACG 1618 ||||||| |||||||||||||| ||||||||||||||||||||||||||||||||||||| Sbjct 98426 GTCCGCGGCTCGAGGGCAAGACCGTCATGCTGTACGTGGGCGGCCTGCGTCCCCGTCACG 98485
Query 1679 ACAACGACGACTATCAGCGCACCGCCCAGCACTACGTCAAGGATGGCACGCTCATCTATG 1738 |||||||||||||||||||||||||||||||||||||||||||||||||| ||||||||| Sbjct 98546 ACAACGACGACTATCAGCGCACCGCCCAGCACTACGTCAAGGATGGCACGATCATCTATG 98605
Query 1739 ACGACGTGACCGGCTACGAGTTCGAGAAGTTCGTCGAGAAGGTCCAGCCGGATCTGGTCG 1798 |||||||||||||||| |||||||||||||||||||||||||||||||| ||||| |||| Sbjct 98606 ACGACGTGACCGGCTATGAGTTCGAGAAGTTCGTCGAGAAGGTCCAGCCCGATCTCGTCG 98665
Query 1799 GCTCGGGCATCAAGGAAAAGTACGTCT-CCAGAAGATG 1835 ||||||||||||||||||||||||| | |||||||||| Sbjct 98666 GCTCGGGCATCAAGGAAAAGTACGTGTTCCAGAAGATG 98703
B. Hasil BLAST-N sekuen gen nifD BGM9
gb|CP000781.1| Xanthobacter autotrophicus Py2, complete genome Length=5308934
Features in this part of subject sequence: nitrogenase iron protein
nitrogenase molybdenum-iron protein alpha chain
Score = 2410 bits (1305), Expect = 0.0
Identities = 1681/1854 (91%), Gaps = 59/1854 (3%)
||||| ||||||||||||||||||||||||||| ||||||||||| |||||||||||||| Sbjct 96890 CATGTATGCGGCCAACAACATCTCCAAGGGCATTCTGAAGTATGCCAACTCCGGCGGCGT 96949
Query 71 GCGCCTGGGCGGCCTGGTGTGCAACGAGCGCCAGACCGACAAGGAG-TACGAGCTGGCGG 129 |||||||||||| ||||||||||||||||||||||||||||||||| | |||||||| | Sbjct 96950 GCGCCTGGGCGGGCTGGTGTGCAACGAGCGCCAGACCGACAAGGAGCT-GGAGCTGGCCG 97008
Query 130 AG-TCGCTGGCGAAGAAGCTCGGCA-CGCAGCTGATCTACTTCGTGCCGCGCGACAACAT 187 || | ||||| ||||||||||||| || ||||||||||||||||||||||||||||||| Sbjct 97009 AGAAC-CTGGCCAAGAAGCTCGGCACCG-AGCTGATCTACTTCGTGCCGCGCGACAACAT 97066
Query 188 CGTGCAGCACGCCGAACTGCGCCGCATGACGGTGATCGAGTATGCGCCCGATTCCGCGCA 247 ||||||||||||||| |||||||||||||| ||||||||||||||||||||||| | || Sbjct 97067 CGTGCAGCACGCCGAGCTGCGCCGCATGACCGTGATCGAGTATGCGCCCGATTCGGAACA 97126
Query 248 GGCCCAGCACTACCGGAACCTTGCGACCAAGGTGCACGGCAACTCGGGCAACGGCATCAT 307 |||||||||||||||||||||||| |||||||| |||| |||| ||||||||||||||| Sbjct 97127 GGCCCAGCACTACCGGAACCTTGCCACCAAGGTTCACGCCAACAAGGGCAACGGCATCAT 97186
Query 308 CCCGACCCCGATCACCATGGACGAGCTCGAAGACCTGCTCATGGAGCA-GGCATCATGAA 366 ||||||||| ||||| |||||||||||||| ||| |||| |||||||| ||||||||||| Sbjct 97187 CCCGACCCCCATCACGATGGACGAGCTCGAGGACATGCTGATGGAGCACGGCATCATGAA 97246
Query 367 GGCCGTGGACGAGAG-CATCGTCGGCAAGACCGCCGCAGAGCTGGCCGTCGGCTGAAGCC 425 |||||| |||||||| || ||||||||||||||||| ||||| ||| | ||| || | Sbjct 97247 GGCCGTCGACGAGAGCCA-GGTCGGCAAGACCGCCGCCGAGCTCACCG-C--CTG-AGGC 97301
Query 426 ATCG-CGTCGATTGACCTGGGGCCGGCCTCAATTGCGGGGCCGGCCTGCCGAAC-GGCAA 483 | | || | | | | | |||||||| ||||||||||| |||||| | | | ||| | Sbjct 97302 -T-GACG-C-A--G--C-GAGGCCGGCCCCAATTGCGGGGTCGGCCT-TC-ATCTGGC-A 97349
Query 484 CCC--AACGAGAGCGAGATTGCGAAATGAGCTTGGCCCAACCGCAAAACGTTGCTGAAAT 541 ||| |||||||||||||||||||||||||||||||||||||||||| |||||||||||| Sbjct 97350 CCCAAAACGAGAGCGAGATTGCGAAATGAGCTTGGCCCAACCGCAAAGCGTTGCTGAAAT 97409
Query 542 CAAGGCGCGGAACAAGGAACTCATCGCGGAAGTCCTCAAGGTTTATCCCGAGAAGACCGC 601 ||||||||| ||||| ||||||||||||||||| |||||||||||||||||||||||||| Sbjct 97410 CAAGGCGCGTAACAAAGAACTCATCGCGGAAGTTCTCAAGGTTTATCCCGAGAAGACCGC 97469
Query 602 GAAGCGCCGCGCGAAGCACCTCAACGTCCATGAGTCCGGCAAGTCCGACTGCGGCGTGAA 661 |||||||||||||||||||||||||||||| ||||||||||||||||| ||||||||||| Sbjct 97470 GAAGCGCCGCGCGAAGCACCTCAACGTCCACGAGTCCGGCAAGTCCGATTGCGGCGTGAA 97529
Query 662 GTCGAACATCAAGTCCATCCCGGGCGTGATGACGATCCGCGGCTGCGCCTACGCCGGTTC 721 |||||||||||||||||||||||||||||||||||||||||||||||| || |||||||| Sbjct 97530 GTCGAACATCAAGTCCATCCCGGGCGTGATGACGATCCGCGGCTGCGCGTATGCCGGTTC 97589
Sbjct 97590 CAAGGGCGTGGTGTGGGGTCCCATCAAGGACATGATCCACATCT-CC-C--A---C- 97638
Query 782 AGGGCCCCGTTGGG-TGGGGCCAGTAAAGGTGGGCCGCCCGCCGGAAAATTA-TACATTG 839 || |||| |||| || |||||||| || ||||||||||||| | || || ||||| | Sbjct 97639 --GG-CCCGGTGGGCTGCGGCCAGTACAGCTGGGCCGCCCGCC-G-CAACTACTACATCG 97693
Query 840 GCACGACCGGCATCGACCCCTTCGGGACGATGCAGTTCACGTCCGATTTCCAGGAAAAGG 899 ||||||| ||||||||| |||||| ||| ||||||||||| || || ||||||||||||| Sbjct 97694 GCACGACGGGCATCGACACCTTCGTGACCATGCAGTTCACCTCGGACTTCCAGGAAAAGG 97753
Query 900 ACATCGTTTTCGGCGGCGACAAGAAGCTCGCCAAGATCATGGACGAAATCCAGGAGCTCT 959 ||||||| ||||||||||||||||||||||||||||||||||||||||||||||| || | Sbjct 97754 ACATCGTCTTCGGCGGCGACAAGAAGCTCGCCAAGATCATGGACGAAATCCAGGACCTGT 97813
Query 960 TCCCGCTCAACAACGGCATCACTGTCCAGTCCGAGTGCCCGATCGGCCTCATCGGCGACG 1019 ||||||| |||||||||||||| ||||||||||||||||||||||||||||||||||||| Sbjct 97814 TCCCGCTGAACAACGGCATCACCGTCCAGTCCGAGTGCCCGATCGGCCTCATCGGCGACG 97873
Query 1020 ACATCGAGGCCGTGTCCAAGTCGAAGTCCAAGGAATACGACGGCAAGACCATCGTTCCGG 1079 |||||||||||||||||||| |||||||||||||||||||| |||||||||||| |||| Sbjct 97874 ACATCGAGGCCGTGTCCAAGGCGAAGTCCAAGGAATACGACAACAAGACCATCGTGCCGG 97933
Query 1080 TCCGCTGCGAGGGCTTCCGCGGCGTGTCGCAGTCCCTCGGCCACCACATCGCCAACGACG 1139 |||||||||||||||||||||||||||| ||||| |||||||| |||||||||||||| | Sbjct 97934 TCCGCTGCGAGGGCTTCCGCGGCGTGTCCCAGTCGCTCGGCCATCACATCGCCAACGATG 97993
Query 1140 CGGTGCGGGACTGGGTGTTCGACAAGCTGGACCCGAACAAGGCTCCGCC-CTTCGAGCCC 1198 || | ||||||||||||||||||||| |||||||||| ||| || || ||||||||| Sbjct 97994 CGATCCGGGACTGGGTGTTCGACAAGATGGACCCGAATGCGGC-CCCCCGCTTCGAGCCG 98052
Query 1199 TCGCCGTATGACGTCGCCATCATCGGCGACTACAACATCGGCGGCGATGCCTGGTCGTCC 1258 || |||||||||||||||||||||||||||||||||||||| ||||| |||||||| || Sbjct 98053 TCCCCGTATGACGTCGCCATCATCGGCGACTACAACATCGGTGGCGACGCCTGGTCTTCG 98112
Query 1259 CGTATCCTCCTCGAGGAGATGGGCCTGCGCGTCATCGCCCAGTGGTCGGGCGACGGCTCG 1318 || ||||| |||||||| |||||||||||||| ||||| |||||||| |||||||||||| Sbjct 98113 CGCATCCTGCTCGAGGAAATGGGCCTGCGCGTGATCGCACAGTGGTCCGGCGACGGCTCG 98172
Query 1319 CTCGCCGAGCTCGAGAAC-ACCCCGCGGGCGAAGCTGAACGTCCTGCACTGCTACCGCTC 1377 || |||||||||||| | || ||| |||||||||||||||||||||||||||||| || Sbjct 98173 CTTGCCGAGCTCGAG-GCGACTCCGAAGGCGAAGCTGAACGTCCTGCACTGCTACCGGTC 98231
Query 1378 CATGAACTACATCTCCCGCCACATGGAAGAGAAGTTCGGGATCCCGTGGTGCGAGTACAA 1437 ||||||||||||||||||||||||||||||||||||||| |||||||||||||||||||| Sbjct 98232 CATGAACTACATCTCCCGCCACATGGAAGAGAAGTTCGGTATCCCGTGGTGCGAGTACAA 98291
Query 1438 CTTCTTCGGTCCC-TCCAAGATCGCGGAGTCGCTGCGCAAGATCGCCGCTTACTTCGACG 1496 ||||||||| ||| ||||||||||| ||||||||||| |||||||||||||||||||||| Sbjct 98292 CTTCTTCGG-CCCGTCCAAGATCGCCGAGTCGCTGCGTAAGATCGCCGCTTACTTCGACG 98350
Query 1497 ACAAGATCAAGGAAGGCGCCGAGCGCGTCATCGCCAAGTACGC-GCCCCTCATGGACGCG 1555 ||||||||||||||||||||||||||||||||||||||||| | |||||||||||||||| Sbjct 98351 ACAAGATCAAGGAAGGCGCCGAGCGCGTCATCGCCAAGTAC-CAGCCCCTCATGGACGCG 98409
Query 1556 GTGATCGCCAAGTATCGTCCGCGCCTCGAGGGCAAGACGGTCATGCTGTACGTGGGCGGC 1615 || || |||||||| |||||||| |||||||||||||| ||||||||||||||||||||| Sbjct 98410 GTCATTGCCAAGTACCGTCCGCGGCTCGAGGGCAAGACCGTCATGCTGTACGTGGGCGGC 98469
Query 1616 CTGCGTCCCCGTCACGTGATCGGCGCGTACGAAGATCTCGGCATGGAAGTGGTGGGCACG 1675 |||||||||||||||||||||||||| |||||||| ||||||||||||||||| ||||| Sbjct 98470 CTGCGTCCCCGTCACGTGATCGGCGCCTACGAAGACCTCGGCATGGAAGTGGTCGGCACC 98529
Query 1676 GGTTACGAGTTCGCCCACAACGACGACTATCAGCGCACCGCCCAGCACTACGTCAAGGAT 1735 || |||||||||| |||||||||||||||||||||||||||||||||||||||||||||| Sbjct 98530 GGCTACGAGTTCGGCCACAACGACGACTATCAGCGCACCGCCCAGCACTACGTCAAGGAT 98589
Query 1736 GGCACGCTCATCTATGACGACGTGACCGGCTACGAGTTCGAGAAGTTCGTCGAGAAGGTC 1795 |||||| ||||||||||||||||||||||||| ||||||||||||||||||||||||||| Sbjct 98590 GGCACGATCATCTATGACGACGTGACCGGCTATGAGTTCGAGAAGTTCGTCGAGAAGGTC 98649
KARAKTERISASI GEN
nif
D BAKTERI METANOTROF ASAL SAWAH
RESTI RACHMAWATI
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
ABSTRAK
RESTI RACHMAWATI. Karakterisasi gen nifD Bakteri Metanotrof Asal Sawah. Dibimbing oleh IMAN RUSMANA dan ALINA AKHDIYA.
Bakteri metanotrof adalah bakteri pengoksidasi CH4. Beberapa metanotrof mampu memfiksasi nitrogen dengan menggunakan enzim nitrogenase. Enzim nitrogenase tersusun atas dua komponen protein besi. Komponen I mengandung situs aktif untuk mereduksi nitrogen, dikodekan oleh gen nifD dan nifK. Isolat metanotrof BGM 3 dan BGM 9 memiliki akumulasi amonium yang tinggi pada media Nitrat Mineral Salt (NMS) tanpa nitrogen. Fragmen gen nifD dari BGM 3 dan BGM 9 di amplifikasi dan disekuensing. Sekuen yang diperoleh dianalisis dengan menggunakan perangkat bioinformatik seperti BLAST-N, BLAST-X, Expasy Translate Tools, Conserved Domain (CDD) dan Scan PROSITE. Kontruksi filogenetik dibuat dengan menggukan program MEGA 4. Analisis sekuen menunjukkan bahwa sekuen gen nifD dari BGM 3 dan BGM 9 mirip dengan gen nifD pada Xanthobacter autotropicus PY2. Nilai kemiripan dari sekuen gen nifD tersebut cukup tinggi ( 92%-91% ). Gen nifD BGM 3 dan BGM 9 mengkodekan protein FeMo yang didalamnya terdapat klaster 4Fe-4S dan 2Fe-2S. Gen nifD dari BGM 3, BGM 9 diduga dimiliki oleh nenek moyang yang sama dengan Xanthobacter autotropicus PY2 dan Bradyhizobium japonicum USDA 110.
Kata kunci: Bakteri metanotrof, fiksasi nitrogen, nitrogenase, nifD
ABSTRACT
RESTI RACHMAWATI. Characterization nifD gene of Methanotrophic Bacteria from Ricefields. Under supervision of IMAN RUSMANA and ALINA AKHDIYA.
Methanotrophic bacteria are methane oxidizing bacteria. Some methanotrophs able of fix nitrogen using nitrogenase enzyme. Nitrogenase is composed of two multisubunit metallo protein. Component I contains the active site to nitrogen reduction, encoded by the nifD and nifK genes. Methanotrophic isolates of BGM 3 and BGM 9 had high ammonium accumulation cultured in Nitrate Mineral Salts (NMS) free nitrogen medium. nifD gene fragmens from BGM 3 and BGM 9 were amplified and sequenced. Sequences analyzed using bioinformatics tools such as Blast-N, Blast-X, Expasy Translate Tools, Conserved Domain (CDD) and Scan PROSITE. Phylogenetic construction made using MEGA 4 software. Sequence analysis showed that the nifD sequences of BGM 3 and BGM 9 most closely related to nifD fragmen of Xanthobacter autotropicus PY2. The Identity values of nifD sequences was 92% and 91% respectively. nifD genes from these isolates encode FeMo protein, which was 4Fe4S and 2Fe2S clusters. The nifD gene of BGM 3 and BGM 9 suspected to have same ancestor with the nifD gene of Xanthobacter autotropicus PY2 and Bradyhizobium japonicum USDA 110.
PENDAHULUAN
Latar Belakang
Bakteri metanotrof adalah bakteri gram negatif, bersifat aerob dan menggunakan metan sebagai sumber karbon penghasil energi (Hanson & Hanson 1996). Bakteri metanotrof tipe II dan tipe X telah diketahui memiliki kemampuan dalam menambat nitrogen sedangkan hanya beberapa bakteri metanotrof tipe I yang dapat memfiksasi nitrogen (Auman et. al 2001). Kemampuan tersebut terkait dengan keberadaan enzim nitrogenase (Dedysh et al. 2004).
Enzim nitrogenase merupakan kompleks enzim yang bertanggung jawab dalam proses fiksasi ntrogen. Nitrogenase terdiri atas dua komponen, yaitu komponen I (dinitrogenase atau protein Fe-Mo) dan komponen II (dinitrogenase reduktase atau protein Fe). Komponen I merupakan heterodimer dengan berat molekul 250 kDA yang dikodekan oleh gen nifD dan nifK, dan komponen II merupakan homodimer dengan berat molekul 70kDA yang dikodekan oleh gen nifH (Zehr et al. 1989).
Terdapat sekitar 20 gen nif yang mengkodekan nitrogenase yaitu nifA , nifB, nifZ, nifH, nifD, nifK, nifE, nifN, nifX, nifO, nifU, nifS, nifV, nifW, nifT, nifY, nifE, nifM, nifF, nifL (Lee et al. 2000) dan diatara 20 gen tersebut nifH dan nifD merupakan gen terpenting untuk mengenali adanya nitrogenase karena mengkodekan sub unit pembentuk nitrogenase. Gen struktural nifH, D, dan K pada dan proteobakteria terletak berdampingan dalam satu operon sehingga membentuk operon nifHDK, sedangkan diazotrof simbiotik yang tumbuh lambat mempunyai gen nifH, D, dan K yang terpisah membentuk operon nifH dan nifDK (Choo et al. 2003)
Diantara 40 isolat bakteri metanotrof yang diisolasi dari sedimen sawah di Bogor dan Sukabumi, tiga diantaranya (BGM 1, BGM 3, dan BGM 9) memiliki aktivitas oksidasi metan dan fiksasi nitrogen yang tinggi (Hapsary 2008, Sagala 2009). Isolat-isolat tersebut belum dikarakterisasi gen pengkode enzim nitrogenasenya.
Informasi tentang karakter enzim nitrogenase bakteri metanotrof asal Indonesia masih sangat sedikit, sehingga penelitian tersebut sangat diperlukan. Informasi tersebut bermanfaat untuk
mendukung pemanfaatan bakteri tersebut sebagai penambat nitrogen di lahan pertanian.
Tujuan Penelitian
Penelitian ini bertujuan mengkarakterisasi gen nifD pada isolat bakteri metanotrof BGM 3 dan BGM 9
Waktu dan Tempat
Penelitian ini dilaksanakan mulai bulan Februari 2010 sampai dengan bulan November 2010 di Laboratorium Mikrobiologi, Departemen Biologi, FMIPA, IPB.
BAHAN DAN METODE
Bahan dan AlatBahan-bahan yang digunakan, yaitu dua isolat metanotrof BGM 3 dan BGM 9, medium Nitrate Mineral Salt (NMS), Primer spesifik gen nifD dan komponen PCR kit KAPA Hifi Polimerase, 1 kb Ladder (Promega).
Peralatan yang digunakan, yaitu mesin PCR (Perkin Elmer Biosystem, USA), Laminar Air Flow, otoklaf, sentrifuse, elektroforesis mini-gel (BioRad Mini-Sub Cell GT, CA, USA), UV Transluminator (Hoefer Scientific Instruments, San Fransisco, USA), dan peralatan lainnya yang biasanya digunakan di laboratorium mikrobiologi
Metode Penelitian
Pemurnian dan peremajaan isolat. Pemurnian dan peremajaan isolat dilakukan dengan menggunakan metode cawan gores pada medium NMS, kemudian inkubasi dilakukan selama 14 hari pada suhu ruang (Hanson 1998).
Isolasi DNA, Amplifikasi gen nifD, dan Visualisasi Amplikon
PENDAHULUAN
Latar Belakang
Bakteri metanotrof adalah bakteri gram negatif, bersifat aerob dan menggunakan metan sebagai sumber karbon penghasil energi (Hanson & Hanson 1996). Bakteri metanotrof tipe II dan tipe X telah diketahui memiliki kemampuan dalam menambat nitrogen sedangkan hanya beberapa bakteri metanotrof tipe I yang dapat memfiksasi nitrogen (Auman et. al 2001). Kemampuan tersebut terkait dengan keberadaan enzim nitrogenase (Dedysh et al. 2004).
Enzim nitrogenase merupakan kompleks enzim yang bertanggung jawab dalam proses fiksasi ntrogen. Nitrogenase terdiri atas dua komponen, yaitu komponen I (dinitrogenase atau protein Fe-Mo) dan komponen II (dinitrogenase reduktase atau protein Fe). Komponen I merupakan heterodimer dengan berat molekul 250 kDA yang dikodekan oleh gen nifD dan nifK, dan komponen II merupakan homodimer dengan berat molekul 70kDA yang dikodekan oleh gen nifH (Zehr et al. 1989).
Terdapat sekitar 20 gen nif yang mengkodekan nitrogenase yaitu nifA , nifB, nifZ, nifH, nifD, nifK, nifE, nifN, nifX, nifO, nifU, nifS, nifV, nifW, nifT, nifY, nifE, nifM, nifF, nifL (Lee et al. 2000) dan diatara 20 gen tersebut nifH dan nifD merupakan gen terpenting untuk mengenali adanya nitrogenase karena mengkodekan sub unit pembentuk nitrogenase. Gen struktural nifH, D, dan K pada dan proteobakteria terletak berdampingan dalam satu operon sehingga membentuk operon nifHDK, sedangkan diazotrof simbiotik yang tumbuh lambat mempunyai gen nifH, D, dan K yang terpisah membentuk operon nifH dan nifDK (Choo et al. 2003)
Diantara 40 isolat bakteri metanotrof yang diisolasi dari sedimen sawah di Bogor dan Sukabumi, tiga diantaranya (BGM 1, BGM 3, dan BGM 9) memiliki aktivitas oksidasi metan dan fiksasi nitrogen yang tinggi (Hapsary 2008, Sagala 2009). Isolat-isolat tersebut belum dikarakterisasi gen pengkode enzim nitrogenasenya.
Informasi tentang karakter enzim nitrogenase bakteri metanotrof asal Indonesia masih sangat sedikit, sehingga penelitian tersebut sangat diperlukan. Informasi tersebut bermanfaat untuk
mendukung pemanfaatan bakteri tersebut sebagai penambat nitrogen di lahan pertanian.
Tujuan Penelitian
Penelitian ini bertujuan mengkarakterisasi gen nifD pada isolat bakteri metanotrof BGM 3 dan BGM 9
Waktu dan Tempat
Penelitian ini dilaksanakan mulai bulan Februari 2010 sampai dengan bulan November 2010 di Laboratorium Mikrobiologi, Departemen Biologi, FMIPA, IPB.
BAHAN DAN METODE
Bahan dan AlatBahan-bahan yang digunakan, yaitu dua isolat metanotrof BGM 3 dan BGM 9, medium Nitrate Mineral Salt (NMS), Primer spesifik gen nifD dan komponen PCR kit KAPA Hifi Polimerase, 1 kb Ladder (Promega).
Peralatan yang digunakan, yaitu mesin PCR (Perkin Elmer Biosystem, USA), Laminar Air Flow, otoklaf, sentrifuse, elektroforesis mini-gel (BioRad Mini-Sub Cell GT, CA, USA), UV Transluminator (Hoefer Scientific Instruments, San Fransisco, USA), dan peralatan lainnya yang biasanya digunakan di laboratorium mikrobiologi
Metode Penelitian
Pemurnian dan peremajaan isolat. Pemurnian dan peremajaan isolat dilakukan dengan menggunakan metode cawan gores pada medium NMS, kemudian inkubasi dilakukan selama 14 hari pada suhu ruang (Hanson 1998).
Isolasi DNA, Amplifikasi gen nifD, dan Visualisasi Amplikon
sebanyak 5,0 µ L, 10mM dNTP mix (konsentrasi akhir 0,3mM) 0,75µ l, 10µ M primer (konsentrasi akhir 0,3µ M) 0,75µl, Template DNA (100ng) 0,5µ l, dan 0,5µl. KAPAHifi DNA Polymerase (1U/µl).
Sekuensing DNA dan Analisis Bioinformatika
Pengurutan DNA hasil amplifikasi dilakukan dengan memanfaatkan jasa perusahaan sekuensing 1st Base di Singapura. Sekuen yang diperoleh dijajarkan dengan data pada GenBank menggunakan program Blast-N, Blast-X dari situs NCBI (National Center for Biotechnology Information) melalui http://www.ncbi.nlm.nih.gov. untuk mengetahui tingkat kemiripan gen nifD dari isolat yang dianalisa. Penurunan sekuen DNA menjadi protein dilakukan menggunakan Expasy translate tools. Konstruksi pohon filogenetik dilakukan dengan menggunakan program MEGA 4.0 (Tamura et al. 2007), metode Neighbor Joining (NJ) dengan bootstrap 100x, sedangkan analisis protein dilakukan menggunakan Conserved Domain (CDD) dan SCAN PROSITE. Visualisasi struktur protein menggunakan program Cn3D 4.1.
HASIL DAN PEMBAHASAN
Hasil
Isolasi, Amplifikasi Gen nifD, dan Visualisasi Amplikon
Amplifikasi gen nifD dari kedua isolat Metanotrof dengan menggunakan primer nifHD-F dan nifD-R dapat dilakukan. Hasil visualisasi amplikon pada gel elekroforesis yang diamati di atas UV transluminator memperlihatkan adanya pita DNA berukuran ~1.9 kb (Gambar 1).
Sekuensing DNA dan Analisis Bioinformatika
Produk amplifikasi gen nifD disekuen untuk mengetahui urutan nukleotidanya. Sekuen yang diperoleh (Lampiran 1) dianalisis kemiripannya dengan data di GenBank menggunakan program BLAST-N dan Blast-X (Tabel 1, Tabel 2, dan Lampiran 2). Berdasarkan analisis program tersebut diketahui homologi nitrogenase dari isolat yang diuji.
Gambar 1 Amplifikasi gen nifD menghasilkan pita DNA berukuran ~1.9 kb (Keterangan : M= 1 kb ladder, Sumur 1=BGM 3, Sumur 2 =BGM 9).
Tabel 1 Hasil analisis sekuen gen nifD dengan menggunakan program BLAST-N
Isolat Sekuen
nitrogenase bakteri yang
homolog
%identitas No.
akses
BGM 3 Nitrogenase
molybdenum-iron protein alpha chain (nifD) pada Xanthobacter autotrophicus Py2
92% CP000
781.1
BGM 9 Nitrogenase
molybdenum-iron protein alpha chain (nifD) pada Xanthobacter autotrophicus Py2
91% CP000
781.1
M 1 2
sebanyak 5,0 µ L, 10mM dNTP mix (konsentrasi akhir 0,3mM) 0,75µ l, 10µ M primer (konsentrasi akhir 0,3µ M) 0,75µl, Template DNA (100ng) 0,5µ l, dan 0,5µl. KAPAHifi DNA Polymerase (1U/µl).
Sekuensing DNA dan Analisis Bioinformatika
Pengurutan DNA hasil amplifikasi dilakukan dengan memanfaatkan jasa perusahaan sekuensing 1st Base di Singapura. Sekuen yang diperoleh dijajarkan dengan data pada GenBank menggunakan program Blast-N, Blast-X dari situs NCBI (National Center for Biotechnology Information) melalui http://www.ncbi.nlm.nih.gov. untuk mengetahui tingkat kemiripan gen nifD dari isolat yang dianalisa. Penurunan sekuen DNA menjadi protein dilakukan menggunakan Expasy translate tools. Konstruksi pohon filogenetik dilakukan dengan menggunakan program MEGA 4.0 (Tamura et al. 2007), metode Neighbor Joining (NJ) dengan bootstrap 100x, sedangkan analisis protein dilakukan menggunakan Conserved Domain (CDD) dan SCAN PROSITE. Visualisasi struktur protein menggunakan program Cn3D 4.1.
HASIL DAN PEMBAHASAN
Hasil
Isolasi, Amplifikasi Gen nifD, dan Visualisasi Amplikon
Amplifikasi gen nifD dari kedua isolat Metanotrof dengan menggunakan primer nifHD-F dan nifD-R dapat dilakukan. Hasil visualisasi amplikon pada gel elekroforesis yang diamati di atas UV transluminator memperlihatkan adanya pita DNA berukuran ~1.9 kb (Gambar 1).
Sekuensing DNA dan Analisis Bioinformatika
Produk amplifikasi gen nifD disekuen untuk mengetahui urutan nukleotidanya. Sekuen yang diperoleh (Lampiran 1) dianalisis kemiripannya dengan data di GenBank menggunakan program BLAST-N dan Blast-X (Tabel 1, Tabel 2, dan Lampiran 2). Berdasarkan analisis program tersebut diketahui homologi nitrogenase dari isolat yang diuji.
Gambar 1 Amplifikasi gen nifD menghasilkan pita DNA berukuran ~1.9 kb (Keterangan : M= 1 kb ladder, Sumur 1=BGM 3, Sumur 2 =BGM 9).
Tabel 1 Hasil analisis sekuen gen nifD dengan menggunakan program BLAST-N
Isolat Sekuen
nitrogenase bakteri yang
homolog
%identitas No.
akses
BGM 3 Nitrogenase
molybdenum-iron protein alpha chain (nifD) pada Xanthobacter autotrophicus Py2
92% CP000
781.1
BGM 9 Nitrogenase
molybdenum-iron protein alpha chain (nifD) pada Xanthobacter autotrophicus Py2
91% CP000
781.1
M 1 2
Tabel 2 Hasil analisis sekuen gen nifD dengan menggunakan program BLAST-X
Isolat Sekuen
nitrogenase bakteri yang
homolog
%identitas No.
akses
BGM 3 Nitrogenase
molybdenum-iron protein alpha chain (nifD) pada Xanthobacter autotrophicus Py2
72% YP_00
14150 05.1
BGM 9 Nitrogenase
molybdenum-iron protein alpha chain (nifD) pada Xanthobacter autotrophicus Py2
71% YP_00
14150 05.1
Analisis sekuen gen nifD menunjukkan bahwa BGM 3 dan BGM 9 memiliki gen nifD yang homolog dengan gen nifD Xanthobacter autotrophicus Py2 (Lampiran 2). Berdasarkan analisis filogenetik, gen nifD BGM 3 dan BGM 9 berkerabat dekat dengan Xanthobacter autotrophicus Py2 dan Bradyhizobium japonicum USDA 110 (Gambar 2). Gen nifD BGM 3 dan 9 termasuk superfamili protein Oxidoreductase nitrogenase dan sub famili Nitrogenase MoFe (Gambar 3).
Pada Nitrogenase MoFe alpa BGM3 dan BGM9 memiliki gugus 4Fe-4S ferredoxin-type iron-sulfur binding region (4Fe4S_FER_1) dan gugus 2 Fe-2S ferredoxin-type iron-sulfur binding region (Tabel 3).
Gambar 3 Analisis bioinformatik protein turunan sekuen gen nifD BGM 3 dan BGM 9 dengan menggunakan program CDD (Conserved Domain)
Tabel 3 Analisis bioinformatik protein hasil translasi gen nifD BGM 3 dan BGM 9 dengan menggunakan program SCAN PROSITE
Klaster pada domain
Motif sekuen Urutan sekuen No. Akses
4Fe4S_FER_1 CgGCGgCGtGCG 62-73 PS00198
2FE2S_FER_1 CTGGGCGGC, CTGATCTAC, CTGGTCGTC, CTGAACGTC, CTGGTCGGC
75-83, 159-167, 1236-1244, 1339-1347, 1792-1800
PS00197
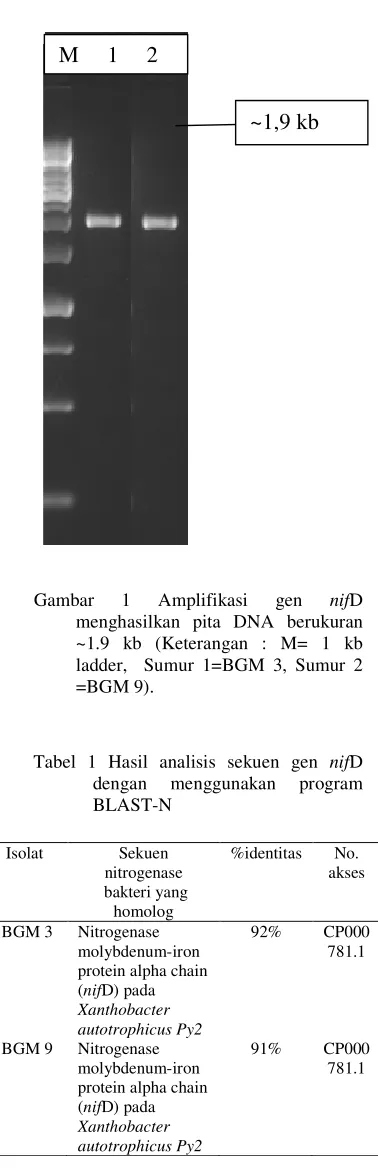
Gambar 4 Struktur kuartener nitrogenase Fe-Mo Alpa dengan menggunakan program Cn3D 4.1 (Keterangan : Warna kuning = Interaksi Fe-Mo sub unit alpa and
beta, A = dimer protein Fe-Mo, B = residu FeMoCo terikat, C = residu kluster P sub unit alpa terikat , D = Fe-Mo sub unit alpa/ kontak protein Fe, E = helix alpa, dan F = lembar beta ).5
A
B
C
F
D
[image:30.792.241.563.102.415.2]Pembahasan
Amplifikasi gen nifD dengan primer nifHD-F dan nifD-R menghasilkan amplikon berukuran 1,9kb untuk BGM 3 dan BGM 9 (Gambar 1). Primer tersebut didesain dari sekuen nifH ke 436 dan sekuen nifD Bradyrhzobium japonicum USDA 110 sehingga menghasilkan amplikon rata-rata sebesar 1,9 kb. Amplikon ini didapat jika nifD, nifH, dan nifK terletak dalam satu operon (Dedysh et al. 2004). Transkripsi enzim nitrogenase pada dan proteobakteria dikodekan oleh operon nifHDK. Sedangkan pada diazotrof simbiotik yang tumbuh lambat enzim ini dikodekan oleh dua operon yang berbeda yaitu operon nifH dan nifDK (Choo et al. 2003).
Analisis Filogenetik menunjukkan bahwa BGM 3, BGM 9 dan Xanthobacter autotrophicus py2 dan Bradyrhizobium japonicum USDA 110 berkerabat dekat bila dilihat dari perkembangan gen nifD nya. (Gambar 2). BGM 3, BGM 9 dan Xanthobacter autotrophicus Py2 merupakan diazotrof yang hidup bebas sedangkan Bradyrhzobium japonicum USDA 110 merupakan diazotrof yang bersimbiosis dan memiliki operon nifH yang terpisah dari nifDK. Berdasarkan analisis gen 16S- rRNA isolat BGM 3 memiliki kemiripan terdekat dengan Methylocystisparvus strain 57 (69%), sedangkan BGM 9 memiliki kemiripan dengan Methylococcus capsulatus strain texas (83% dan 85%) (Astuti 2009). Nilai kemiripan sekuen nifD BGM 3 dan BGM 9 lebih tinggi dari analsis 16S-rRNA tersebut. Sehingga kemungkinan BGM 3 dan BGM 9 memiliki nenek moyang bakteri pemfiksasi nitrogen yang sama dengan Xanthobacter autotrophicus py2 dan Bradyrhizobium japonicum USDA 110.
Penelitian yang dilakukan Dedysh et al. (2004) juga menunjukkan bahwa nilai kemiripan (identity values) dari sekuen nifH dan nifD pada Methylocapsa acidiphila B2 dan Beijerinckia lebih tinggi (98.5 % dan 96.6 %) dibandingkan dengan hubungan kekerabatannya berdasarkan 16S rRNA sehingga kemungkinan dua bakteri tersebut berasal dari nenek moyang bakteri pemfiksasi nitrogen yang sama.
Nitrogenase oxidoreduktase merupakan sekelompok famili protein yang mengkatalisis reduksi nitrogen menjadi amonium dengan menggunakan ATP.
Selain ATP, proses tersebut membutuhkan feredoksin tereduksi sebagai donor elektron, sitokrom sebagai protein pembawa elektron, dan koenzim. Proses fiksasi satu mol nitrogen ini memerlukan energi sel sebanyak 16 ATP.
Oxidoreduktase memiliki sub unit alpa dan sub unit beta dari komponen I yang secara genetik terbagi atas nitrogenase yang tergantung pada atom molibdenum (Mo-nitrogenase), nitrogenase yang tergantung pada atom vanadium (V-nitrogenase), dan nitrogenase besi (Fe-nitrogenase). Nitrogenase alternatif vanadium dan nitrogenase yang hanya mengandung besi saja masing-masing dikodekan oleh gen vnf dan anf dan mempunyai struktur heksamer yang dikodekan oleh operon vnfDGK dan anfDGK. (Caton 2007). Selain itu terdapat beberapa sub unit Protochlorophyllide (Pchlide) reductase and chlorophyllide (chlide) reductase (Gambar 3).
SIMPULAN
Amplifikasi gen nifD BGM 3 dan BGM 9 dengan menggunakan primer nifHD-F dan nifD-R menghasilkan amplikon berukuran 1,9kb. Gen nifD, nifH dan gen nifK pada isolat bakteri metanotrof BGM 3 dan BGM 9 terletak dalam satu operon. Sekuen gen nifD BGM 3 dan BGM 9 memiliki kemiripan terdekat dengan sekuen gen nifD Xanthobacter autotrophicus Py2.
DAFTAR PUSTAKA
Alhapel A et al. 2006. Molecular and functional analysis of nicotinate catabolism in Eubacterium barkeri. Proc Natl Acad Sci 103: 12341-12346.
Astuti DD. 2009. Karakterisasi fisiologi dan identifikasi molekuler isolat-isolat bakteri metanotrof asal sawah wilayah Bogor dan Sukabumi. [Skripsi]. Bogor: Departemen Biologi, Institut Pertanian Bogor.
Auman AJ, Speake CC, Lidstrom ME. 2001. nifH sequences and nitrogen fixation in type I and type II methanotrophs. Appl Environ Microbiol 67(9): 4009-4016.
Caton IR. 2007. Abundance of nifH genes in urban, agricultural, and pristine prairie streams exposed to different levels of nitrogen loading [thesis]. Wichita State University.
Choo QC, Samian MR, Najimudin N. 2003. Phylogeny and characterization of three nifH-homologous genes from Paenibacillus azotofixans. Appl Environ Microbiol 69(6): 3658-3662.
Dedysh SN, Ricke P, Liesack W. 2004. NifH and NifD phylogenies: an evolutionary basis for understanding nitrogen fixation capabilities of methanotrophic bacteria. Microbiol 150; 1301– 1313.
Hanson RS. 1998. Ecology of methylotrophic bacteria. Di dalam:
Burlage RS, Atlas R, Stahl, Geesey G, Dayler G, editor. Techniques in microbial Ecology. Oxford: Oxford University Press. 137- 162.
Hanson R, Hanson TE. 1996. Metanotrophic Bacteria. J Microbiol Reviews 60 : 439-471
Hapsary W. 2008. Isolasi dan karakterisasi bakteri metanotrof asal sawah di Bogor dan Sukabumi [Skripsi]. Bogor: Departemen Biologi, Institut Pertanian Bogor.
Lazo GR, Roffey R, Gabriel DW. 1987. Conservation of plasmid DNA sequences and pathovar identification of strain
Xanthomonas campestris.
Pytopathology 77: 1461-1467.
Lee S, Reth A, Meletzus D, Sevilla M, Kennedy C. 2000. Characterization of major cluster of nif, fix, and associated genes in sugarcane endophyte, Acetobacter diazotrophicus. J Bacteriol. 182(24): 7088-7091.
Sagala BT. 2009. Seleksi dan uji aktivitas fiksasi nitrogen (N2) bakteri metanotrof asal sawah pada konsentrasi oksigen (O2) berbeda [skripsi]. Bogor : Departemen Biologi, Institut Pertanian Bogor.
Tamura K, Dudley J, Nei M, Kumar S. 2007. MEGA4: Molecular evolutionary genetics analysis (MEGA) software version 4.0. Mol Biol Evol 24:1596–1599.
Lampiran 1. Hasil sekuen gen nifD
A. Urutan nukleotida hasil sekuen gen nifD BGM 3
CGCGAAGAGGCATGTATGCGGCCAACAACATCTCCAAGGGCATCCTGAAGTATGCGAACTCCGGCGGCGTG CGCCTGGGCGGCCTGGTGTGCAACGAGCGCCAGACCGACAAGGAGTACGAGCTGGCGGAGTCGCTGGCGAA GAAGCTCGGCACGCAGCTGATCTACTTCGTGCCGCGCGACAACATCGTGCAGCACGCCGAACTGCGCCGCAT GACGGTGATCGAGTATGCGCCCGATTCCGCGCAGGCCCAGCACTACCGGAACCTTGCGACCAAGGTGCACGG CAACTCGGGCAACGGCATCATCCCGACCCCGATCACCATGGACGAGCTCGAAGACCTGCTCATGGAGCACGG CATCATGAAGGCCGTGGACGAGAGCATCGTCGGCAAGACCGCCGCAGAGCTGGCCGTCGGCTGAAGCCATC GCGTCGATTGACCTGGGGCCGGCCTCAATTGCGGGGCCGGCCTGCCGAACGGCAACCCAACGAGAGCGAGA TTGCGAAATGAGCTTGGCCCAACCGCAAAACGTTGCTGAAATCAAGGCGCGGAACAAGGAACTCATCGCGG AAGTCCTCAAGGTTTATCCCGAGAAGACCGCGAAGCGCCGCGCGAAGCACCTCAACGTCCATGAGTCCGGCA AGTCCGACTGCGGCGTGAAGTCGAACATCAAGTCCATCCCGGGCGTGATGACGATCCGCGGCTGCGCCTACG CCGGTTCCAAGGGCGTGGTGTGGGGTCCGATCAAGGACATGATCCACATCTCCCACGGCCCCGTCGGCTGCG GCCAGTACAGCTGGGCCGCCCGCCGCAACTACTACATGGGCAGGACCGGAATGGACACCTTGGTGAGGATG
CAGTTCACGTCCGACTTCCAGGAAAAGGACATCGTTTTCGGCGGGGACAAGAAGCTCGCCAAGATCATGGAC
GAAATCCAGGAGCTCTTCCCGCTCAACAACGGCATCACTGTCCAGTCCGAGTGTCCGATCGGCCTCATCGGC
GACGACATCGAGGCCGTGTCCAAGTCGAAGTCCAAGGAATACGACGGCAAGACCATCGTTCCGGTCCGCTGC GAGGGCTTCCGCGGCGTGTCGCAGTCCCTCGGCCACCACATCGCCAACGACGCGGTGCGGGACTGGGTGTTC GACAAGCTGGACCCGAACAAGGCTCCGCCCTTCGAGCCCTCGCCGTATGACGTCGCCATCATCGGCGACTAC AACATCGGCGGCGATGCCTGGTCGTCCCGTATCCTCCTCGAGGAGATGGGCCTGCGCGTCATCGCCCAGTGG TCGGGCGACGGCTCGCTCGCCGAGCTCGAGAACACCCCGCGGGCGAAGCTGAACGTCCTGCACTGCTACCGC TCCATGAACTACATCTCCCGCCACATGGAAGAGAAGTTCGGGATCCCGTGGTGCGAGTACAACTTCTTCGGT CCCTCCAAGATCGCGGAGTCGCTGCGCAAGATCGCCGCTTACTTCGACGACAAGATCAAGGAAGGCGCCGA GCGCGTCATCGCCAAGTACGCGCCCCTCATGGACGCGGTGATCGCCAAGTATCGTCCGCGCCTCGAGGGCAA GACGGTCATGCTGTACGTGGGCGGCCTGCGTCCCCGTCACGTGATCGGCGCGTACGAAGATCTCGGCATGGA AGTGGTGGGCACGGGTTACGAGTTCGCCCACAACGACGACTATCAGCGCACCGCCCAGCACTACGTCAAGG ATGGCACGCTCATCTATGACGACGTGACCGGCTACGAGTTCGAGAAGTTCGTCGAGAAGGTCCAGCCGGATC TGGTCGGCTCGGGCATCAAGGAAAAGTACGTCTCCAGAAGATGCGCT
B. Urutan nukleotida hasil sekuen gen nifD BGM9
CCGCGAAGAGGCATGTTGCGGCCAACAACATCTCCAAGGGCATCCTGAAGTATGCGAACTCCGGCGGCGTG CGCCTGGGCGGCCTGGTGTGCAACGAGCGCCAGACCGACAAGGAGTACGAGCTGGCGGAGTCGCTGGCGA AGAAGCTCGGCACGCAGCTGATCTACTTCGTGCCGCGCGACAACATCGTGCAGCACGCCGAACTGCGCCGC ATGACGGTGATCGAGTATGCGCCCGATTCCGCGCAGGCCCAGCACTACCGGAACCTTGCGACCAAGGTGCA CGGCAACTCGGGCAACGGCATCATCCCGACCCCGATCACCATGGACGAGCTCGAAGACCTGCTCATGGAGC AGGCATCATGAAGGCCGTGGACGAGAGCATCGTCGGCAAGACCGCCGCAGAGCTGGCCGTCGGCTGAAGC CATCGCGTCGATTGACCTGGGGCCGGCCTCAATTGCGGGGCCGGCCTGCCGAACGGCAACCCAACGAGAGC GAGATTGCGAAATGAGCTTGGCCCAACCGCAAAACGTTGCTGAAATCAAGGCGCGGAACAAGGAACTCAT CGCGGAAGTCCTCAAGGTTTATCCCGAGAAGACCGCGAAGCGCCGCGCGAAGCACCTCAACGTCCATGAGT CCGGCAAGTCCGACTGCGGCGTGAAGTCGAACATCAAGTCCATCCCGGGCGTGATGACGATCCGCGGCTGC GCCTACGCCGGTTCCAAGGGCGTGGTGTGGGGTCCAATCAAGGACATGATCCACATCTCCCACAAATTTTCC CAAGGGCCCCGTTGGGTGGGGCCAGTAAAGGTGGGCCGCCCGCCGGAAAATTATACATTGGCACGACCGG
CATCGACCCCTTCGGGACGATGCAGTTCACGTCCGATTTCCAGGAAAAGGACATCGTTTTCGGCGGCGACA
AGAAGCTCGCCAAGATCATGGACGAAATCCAGGAGCTCTTCCCGCTCAACAACGGCATCACTGTCCAGTCC
GAGTGCCCGATCGGCCTCATCGGCGACGACATCGAGGCCGTGTCCAAGTCGAAGTCCAAGGAATACGACGG
CAAGACCATCGTTCCGGTCCGCTGCGAGGGCTTCCGCGGCGTGTCGCAGTCCCTCGGCCACCACATCGCCA ACGACGCGGTGCGGGACTGGGTGTTCGACAAGCTGGACCCGAACAAGGCTCCGCCCTTCGAGCCCTCGCCG TATGACGTCGCCATCATCGGCGACTACAACATCGGCGGCGATGCCTGGTCGTCCCGTATCCTCCTCGAGGAG ATGGGCCTGCGCGTCATCGCCCAGTGGTCGGGCGACGGCTCGCTCGCCGAGCTCGAGAACACCCCGCGGGC GAAGCTGAACGTCCTGCACTGCTACCGCTCCATGAACTACATCTCCCGCCACATGGAAGAGAAGTTCGGGA TCCCGTGGTGCGAGTACAACTTCTTCGGTCCCTCCAAGATCGCGGAGTCGCTGCGCAAGATCGCCGCTTACT TCGACGACAAGATCAAGGAAGGCGCCGAGCGCGTCATCGCCAAGTACGCGCCCCTCATGGACGCGGTGATC GCCAAGTATCGTCCGCGCCTCGAGGGCAAGACGGTCATGCTGTACGTGGGCGGCCTGCGTCCCCGTCACGT GATCGGCGCGTACGAAGATCTCGGCATGGAAGTGGTGGGCACGGGTTACGAGTTCGCCCACAACGACGACT ATCAGCGCACCGCCCAGCACTACGTCAAGGATGGCACGCTCATCTATGACGACGTGACCGGCTACGAGTTC GAGAAGTTCGTCGAGAAGGTCCAGCCGGACCTGGTCGGCTCGGGCTCAAGGAAAAGTACTCTCCAGAAGAT GCGGGCC
C. Protein hasil translasi gen nifD BGM 3 dan BGM 9 >5’3’ Frame 2 (BGM3)
DKKLAKIMDEIQELFPLNNGITVQSECPIGLIGDDIEAVSKSKSKEYDGKTIVPVRCEGFRGVSQSLGHHIANDAVR DWVFDKLDPNKAPPFEPSPYDVAIIGDYNIGGDAWSSRILLEEMGLRVIAQWSGDGSLAELENTPRAKLNVLHCY RSMNYISRHMEEKFGIPWCEYNFFGPSKIAESLRKIAAYFDDKIKEGAERVIAKYAPLMDAVIAKYRPRLEGKTVM LYVGGLRPRHVIGAYEDLGMEVVGTGYEFAHNDDYQRTAQHYVKDGTLIYDDVTGYEFEKFVEKVQPDLVGSG IKEKYVSRRCA
>5’3’ Frame 3 (BGM 9)
REEACCGQQHLQGHPEVCELRRRAPGRPGVQRAPDRQGVRAGGVAGEEARHAADLLRAARQHRAARRTAPHD GDRVCARFRAGPALPEPCDQGARQLGQRHHPDPDHHGRARRPAHGAGMKAVDESIVGKTAAELAVG*SHRVD* PGAGLNCGAGLPNGNPTRARLRNELGPTAKRC*NQGAEQGTHRGSPQGLSREDREAPREAPQRP*VRQVRLRREV EHQVHPGRDDDPRLRLRRFQGRGVGSNQGHDPHLPQIFPRAPLGGASKGGPPAGKLYIGTTGIDPFGTMQFTSDFQ EKDIVFGGDKKLAKIMDEIQELFPLNNGITVQSECPIGLIGDDIEAVSKSKSKEYDGKTIVPVRCEGFRGVSQSLGH HIANDAVRDWVFDKLDPNKAPPFEPSPYDVAIIGDYNIGGDAWSSRILLEEMGLRVIAQWSGDGSLAELENTPRA KLNVLHCYRSMNYISRHMEEKFGIPWCEYNFFGPSKIAESLRKIAAYFDDKIKEGAERVIAKYAPLMDAVIAKYRP RLEGKTVMLYVGGLRPRHVIGAYEDLGMEVVGTGYEFAHNDDYQRTAQHYVKDGTLIYDDVTGYEFEKFVEKV QPDLVGSGSRKSTLQKMRA
Keterangan : A=alanin B=aspartat/asparagin C=cystine D=aspartat E=glutamat F=Fenilalanin G=glysin
H=histidin I=isoleusin M=metionin N=asparagin P=prolin Q=glutamin R=arginin S=serin T=threonine V=valin W=tryptophan Y=tyrosin Z=glutamat X=any *=kodon stop
Lampiran 2. Hasil BLAST-N
A. Hasil BLAST-N sekuen gen nifD BGM3
gb|CP000781.1| Xanthobacter autotrophicus Py2, complete genome Features in this part of subject sequence:
nitrogenase iron protein
nitrogenase molybdenum-iron protein alpha chain
Score = 2529 bits (1369), Expect = 0.0
Identities = 1688/1838 (92%), Gaps = 37/1838 (2%)
Query 11 CATGTATGCGGCCAACAACATCTCCAAGGGCATCCTGAAGTATGCGAACTCCGGCGGCGT 70 ||||||||||||||||||||||||||||||||| ||||||||||| |||||||||||||| Sbjct 96890 CATGTATGCGGCCAACAACATCTCCAAGGGCATTCTGAAGTATGCCAACTCCGGCGGCGT 96949
Query 71 GCGCCTGGGCGGCCTGGTGTGCAACGAGCGCCAGACCGACAAGGAG-TACGAGCTGGCGG 12