PENGELOMPOKAN SEKUEN DNA MENGGUNAKAN
ALGORITME
SINGLE LINK
DAN
FEATURE VECTORS
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
BOGOR 2013
iii
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Pengelompokan Sekuen DNA Menggunakan Algoritme Single Link dan Feature Vectors adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, September 2013
iv
ABSTRAK
AL HARIS TAMSIN. Pengelompokan Sekuen DNA Menggunakan Algoritme Single Link dan Feature Vectors. Dibimbing oleh WISNU ANANTA KUSUMA.
Pengelompokan merupakan teknik pembelajaran untuk menemukan kelompoknya secara otomatis berdasarkan ciri dan karakteristik yang dimiliki. Pengelompokan dilakukan untuk memisahkan data ke dalam kelompok sehingga data yang dimiliki menjadi mudah dimengerti. Pengelompokan sekuen DNA dengan feature vectors merupakan proses penggabungan sekelompok sekuen DNA yang memiliki kemiripan jumlah nukleotida, susunan, dan penyebaran nukleotidanya, akan digabungkan ke dalam sebuah kelompok yang sama. Pengelompokan sekuen DNA terdapat empat tahap utama yaitu feature vectors, min max normalization, cosine similarity, dan pengelompokan single link. Penelitian terdiri dari 8 studi kasus dan 5 percobaan di setiap studi kasus yang mendapatkan akurasi rata-rata sebesar 86.7%, dan akurasi pengelompokan terbaik terdapat pada studi kasus 3 sekuen percobaan 4 sebesar 100%. Faktor yang paling berpengaruh terhadap hasil pengelompokan sekuen DNA adalah ukuran dan jumlah sekuen yang digunakan dalam penelitian.
Kata kunci: feature vectors, single link, min max normalization, sekuen DNA
ABSTRACT
AL HARIS TAMSIN. Clustering DNA Sequences Using Single Link Algorithms and Feature Vectors. Supervised by WISNU ANANTA KUSUMA.
Clustering is a learning technique to find the the group automatically based on the characteristics. By separating the data into groups so the data will easy to understand. Clustering DNA sequences with feature vectors is the process of combining a group of DNA sequences with the same amount of nucleotides, the composition and distribution of nucleotide, will combine into the same group. There are 4 main stages of clustering DNA sequence: feature vectors, min max normalization, cosine similarity and single link clustering. This research consist of the 8 case and 5 experiment in each case, with the result of least average 86,7% and the best cluster found 100% in case 3 experiment 4. The most affecting for the result of DNA sequence clustering is the size and volume used in the research.
v
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Ilmu Komputer
pada
Departemen Ilmu Komputer
PENGELOMPOKAN SEKUEN DNA MENGGUNAKAN
ALGORITME
SINGLE LINK
DAN
FEATURE VECTORS
AL HARIS TAMSIN
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
vi
Penguji :
vii
Judul Skripsi : Pengelompokan Sekuens DNA Menggunakan Algoritme Single Link dan Feature Vectors
Nama : Al Haris Tamsin
NIM : G64104050
Disetujui oleh
Dr Wisnu Ananta Kusuma, ST MT Pembimbing
Diketahui oleh
Dr Ir Agus Buono, MSi M Kom Ketua Departemen Ilmu Komputer
viii
PRAKATA
Puji dan syukur penulis panjatkan ke hadirat Allah subhanahu wa ta’ala atas rahmat dan karunia-Nya sehingga penulis bisa menyelesaikan karya ilmiah ini. Penelitian yang dilakukan sejak bulan September 2012 mengangkat tema pengelompokan sekuen DNA menggunakan algoritme single link dan ekstraksi ciri feature vectors.
Penulis mengucapkan terima kasih kepada dosen pembimbing Bapak Wisnu Ananta Kusuma yang telah memberikan arahan, saran, dan masukan untuk selesainya penelitian ini. Ucapan terima kasih juga penulis sampaikan kepada kedua orang tua, kakak, adik, dan keluarga atas doa, semangat, dan kasih sayangnya sehingga penulis bisa menyelesaikan penelitian ini.
Penulis menyadari bahwa pada karya ilmiah ini masih banyak kekurangan dan jauh dari kesempurnaan. Oleh karena itu, penulis mengharapkan saran dan kritik yang membangun demi penyempurnaan karya ilmiah berikutnya. Semoga karya ilmiah ini dapat bermanfaat bagi penulis khususnya dan bagi semua pihak pada umumnya.
Semoga karya ilmiah ini bermamfaat.
Bogor, September 2013
ix
DAFTAR ISI
DAFTAR TABEL ... vi
DAFTAR GAMBAR ... vi
DAFTAR LAMPIRAN vi
PENDAHULUAN ... 1
Latar Belakang ... 1
Tujuan ... 2
Ruang lingkup ... 2
METODE PENELITIAN ... 2
Tahap pengumpulan data ... 3
Tahap pengelompokan ... 3
Feature Vectors ... 3
Normalisasi Min-max ... 5
Cosine Similarity ... 5
Single link ... 5
Tahap Evaluasi ... 7
Confusion Matrix ... 7
Lingkungan implementasi ... 7
HASIL DAN PEMBAHASAN ... 8
Tahap Pengumpulan Data ... 8
Tahap Pengelompokan ... 8
Feature Vectors ... 8
Normalisasi Min-max ... 9
Cosine Similarity ... 9
Single Link ... 10
Tahap Pengujian ... 11
Analisis Hasil Percobaan ... 11
SIMPULAN DAN SARAN ... 15
Simpulan ... 15
Saran ... 15
LAMPIRAN ... 18
x
DAFTAR TABEL
1 Contoh perhitungan feature vectors ... 4
2 Hasil cosine similarity untuk studi kasus 2 sekuen percobaan 1 ... 10
3 Confusion matrix untuk studi kasus 3 sekuen percobaan 4 ... 11
4 Hasil akurasi rata-rata 8 studi kasus ... 12
5 Confusion matrix studi kasus 4 sekuen percobaan 4 ... 13
6 Hasil penjajaran sekuen Borelliaburgdorferi ... 13
7 Hasil penjajaran sekuen Borelliaduttoni ... 14
8 Hasil penjajaran sekuen Borelliagarinii ... 14
9 Hasil penjajaran sekuen Borelliaspielmanii ... 14
DAFTAR GAMBAR
1 Metode penelitian ... 22 Proses dalam tahap pengelompokan ... 3
3 Contoh pengelompokan single link ... 6
4 Contoh dendrogram ... 7
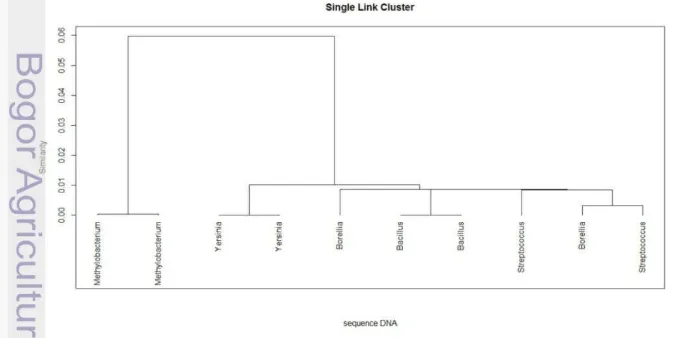
5 Dendrogram dari studi kasus 2 sekuen percobaan 1 ... 10
6 Grafik rata-rata akurasi 8 studi kasus ... 12
DAFTAR LAMPIRAN
1 Data yang digunakan dalam penelitian 17
2 Tampilan awal pengelompokan single link 19
3 Dendrogram studi kasus 3 percobaan 4 akurasi 100% 19
4 Feature vectors untuk 50 data sekuen DNA 20
5 Hasil normalisasi min-max untuk 50 data sekuen DNA 21
6 Confusion matrix studi kasus 2 sekuen 25
7 Confusionmatrix studi kasus 3 sekuen 25
8 Confusion matrix studi kasus 4 sekuen 25
9 Confusion matrix studi kasus 5 sekuen 25
10 Confusion matrix studi kasus 6 sekuen 25
11 Confusion matrix studi kasus 7 sekuen 26
12 Confusion matrix studi kasus 8 sekuen 26
1
PENDAHULUAN
Latar Belakang
Analisis data dapat menggali informasi yang lebih detail dari data yang dimiliki, sehingga bisa dimanfaatkan untuk berbagai keperluan. Hasil dari analisis data dipengaruhi oleh banyak hal antara lain objek yang dianalisis, peubah yang diamati, tingkat kesamaan atau jarak yang dipakai, skala ukuran yang dipakai, serta metode yang akan digunakan untuk analisis data. Pada bidang biologi molekuler saat ini sudah banyak ditemukan teknik-teknik analisis data yang mendukung proses dalam bidang ini. Pengelompokan sekuen Deoxyribo nucleic acid (DNA) merupakan salah satu contoh proses analisis data yang diterapkan pada bidang biologi molekuler.
DNA merupakan asam nukleat yang terdapat pada inti sel pada sel eukariotik, pada sel prokariotik DNA terdapat dalam sitoplasma. Satu asam nukleat terdiri dari satu molekul gula ribosa, satu basa nitrogen, dan fosfat. Satu asam nukleat dengan asam nukleat yang lain dirangkai dengan ikatan fosfodiester. Basa nitrogen terdiri atas dua jenis yaitu purin dan pirimidin. Basa purin terdiri atas adenin (A) dan guanin (G), sedangkan pirimidin terdiri atas sitosin (C) dan timin (T). DNA berfungsi untuk menyimpan informasi genetik pada suatu organisme (Yuwono 2008).
Sekuen DNA berbentuk sebuah urutan huruf-huruf mewakili struktur primer dari molekul DNA yang dapat digunakan sebagai parameter dalam pengelompokan DNA. Sekuen DNA dalam bentuk digital disimpan dalam sebuah file berbasis teks berformat Fasta.
Pengelompokan adalah suatu proses pembelajaran tidak terlatih (unsupervised) terhadap suatu pattern (data, feature vectors) menjadi beberapa kelompok berdasarkan kemiripannya (Jain et al 1999). Pengelompokan sekuen DNA dilakukan untuk mempartisi data berdasarkan kemiripan sekuen DNA yang dimiliki. Pengelompokan seringkali disamakan dengan klasifikasi, dalam hal ini pengelompokan memisahkan sekelompok data ke dalam beberapa kelompok menurut kemiripannya, sedangkan klasifikasi yaitu memberikan kelompok kepada sebuah data berdasarkan kemiripan dengan data pada setiap kelompok tertentu.
2
Tujuan
Tujuan dilakukan penelitian ini adalah untuk:
1 Menerapkan feature vectors dengan perhitungan tingkat kesamaan cosine similarity dalam pengelompokan sekuen DNA menggunakan metode single link.
2 Visualisasi hasil pengelompokan sekuen DNA.
3 Dapat memberikan informasi yang akurat dalam pengelompokan sekuen DNA.
Ruang lingkup
1 Data sekuen DNA yang digunakan dalam format Fasta.
2 DNA sekuen yang digunakan adalah DNA bakteri complete sequence.
METODE PENELITIAN
Penelitian ini melakukan pengelompokan otomatis terhadap sekuen DNA berformat fasta dengan menggunakan metode hierarki clustering single link. Metode penelitian ini dilakukan dalam beberapa tahap, yaitu pengumpulan data, pengelompokan, dan tahap evaluasi seperti yang ditampilkan pada gambar 1.
Penelitian terdiri dari 8 studi kasus dan masing-masing studi kasus terdiri dari 5 percobaan. Studi kasus 2 sekuen menggunakan 2 sekuen setiap genus dengan 5 percobaan menggunakan data sekuen yang berbeda di setiap percobaan. Hal yang sama juga berlaku bagi studi kasus yang lain studi kasus 3 sekuen hingga 9 sekuen setiap genus.
Mulai
Pengumpulan data
Tahap Evaluasi
Selesai Pengelompokan
3
Tahap Pengumpulan Data
Dalam penelitian ini menggunakan 50 data sekuen DNA yang didapatkan dari situs National Centre of Biotechnology Information, US National Library of Medicine, yang beralamat di http://www.ncbi.nlm.nih.gov/. Semua data tersebut disimpan dalam format standar fasta. Format fasta adalah format berbasis teks untuk mewakili urutan nukleotida. Nukleotida tersebut diwakili menggunakan huruf tunggal. Format ini juga menyimpan informasi spesies dari sekuen DNA tersebut. Format fasta awalnya merupakan hasil dari perangkat lunak fasta, tapi sekarang telah menjadi standar dalam bidang bioinformatika. Data tersebut terdiri dari lima genus nukleotida, yang akan dikelompokkan menurut kelasnya masing-masing. Data yang digunakan pada penelitian ini ditampilkan pada Lampiran 1.
Tahap Pengelompokan
Dalam tahap ini akan dilakukan perhitungan terhadap sekuen DNA yang sudah didapatkan pada tahap sebelumnya. Proses pada tahap pengelompokan dapat dilihat pada Gambar 2. Dimulai dari ekstraksi fitur dengan feature vectors dari setiap sekuen, normalisasi, perhitungan tingkat kesamaan dengan cosine similarity, dan pengelompokan sekuen DNA menggunakan single link. Hasil dari tahap ini nantinya akan digunakan pada tahap pengujian.
Gambar 2 Proses dalam tahap pengelompokan
Feature Vectors
Pada dasarnya sebuah sekuen DNA terdiri dari rangkaian huruf, sehingga akan sangat sulit untuk melakukan perbandingan dan perhitungan dari masing-masing sekuen. Oleh karena itu perlu ditentukan identitas yang dapat digunakan sebagai penciri suatu sekuen DNA, sehingga setiap sekuen DNA dapat dilakukan perbandingan melalui penciri sekuen tersebut.
4
Jumlah setiap nukleotida pada sekuen DNA (n)
Setiap sekuen DNA terdiri atas empat nukleotida yang menyusunnya, n merupakan parameter yang menunjukkan jumlah total satu nukleotida pada sebuah sekuen DNA, suatu parameter na merupakan jumlah nukleotida A pada
sekuen DNA tersebut. Hal tersebut juga berlaku pada tiga nukleotida lainnya yaitu C, G, dan T. sehingga nanti akan didapatkan empat nilai yaitu nA, nC, nG, dan nT. Jumlah total jarak antar nukleotida dengan nukleotida pertama (t)
Untuk mendapatkan nilai dari parameter t yang merupakan perhitungan total jarak antara satu nukleotida sejenis, jika sebuah nukleotida A maka jarak antar nukleotida ke n dengan nukleotida pertama akan dijumlahkan sehingga mendapatkan sebuah nilai t. Nilai parameter t dapat dihitung dengan menggunakan Persamaan 1:
∑ (1)
dengan nilai i adalah nukleotida A, T, G, C, dan j = jarak antar nukleotida. Varians nukleotida (d)
Jika dua DNA memiliki ukuran yang sama, dan memiliki total jarak sama, maka jika dilakukan perbandingan akan terjadi kesalahan. Jika dalam satu sekuen DNA terdapat nukleotida A pada posisi 4 dan 6, dan pada suatu sekuen DNA lain juga memiliki nukleotida A pada posisi 5 dan 7, maka kedua sekuen DNA tersebut memiliki jumlah A yang sama, total jarak dari nukleotida pertama sama, sehingga dibutuhkan parameter ketiga yaitu d. Parameter d merupakan parameter yang melakukan analisis distribusi dari setiap nukleotida, perhitungan parameter d untuk setiap nukleotida dilakukan untuk menggambarkan distribusi dari nukleotida dengan menggunakan Persamaan 2:
∑ ( - )
(2) dengan i= A,T,G,C dan tj = jarak nukleotida pertama dengan nukleotida ke n
Nilai didapatkan dari Persamaan 3:
(3)
Tabel 1 Contoh perhitungan feature vectors
Sekuen DNA Parameter n Parameter t Parameter d
CAATTAACCCCTT
Setelah dilakukan perhitungan tiga parameter seperti ditampilkan pada Tabel 1, akan didapatkan dua belas nilai yang akan dijadikan sebuah vektor, sehingga setiap sekuen DNA ditransformasikan menjadi sebuah vektor yang memiliki 12 nilai.
<n
5
Normalisasi Min-Max
Untuk mendapatkan hasil pengelompokan yang baik, maka harus menggunakan data yang baik, lengkap, dan terstruktur. Sebelum melakukan pengelompokan sekuen DNA perlu dilakukan normalisasi untuk memastikan data yang akan digunakan adalah data yang bagus untuk dikelompokkan karena data sangat berpengaruh terhadap hasil pengelompokan.
Normalisasi merupakan proses penskalaan nilai atribut dari data, sehingga bisa berkisar pada range nilai tertentu, karena oleh dimensi data yang terlalu jauh atau terlalu dekat, sehingga akan sulit untuk melakukan pengelompokan data.
Min-max melakukan transformasi linear pada data, menggunakan nilai minimum dan nilai maksimum. Normalisasi min-max mempertahankan hubungan antara nilai data asli (Han dan Kamber 2006). Proses normalisasi min-max dilakukan dengan mengurangkan nilai data dengan nilai minimal, kemudian dibagi dengan nilai maksimal kurang nilai minimal. Normalisasi min-max didapatkan dari persamaan (4):
Min Max(x) = - - ( - ) (4)
Cosine Similarity
Pada analisis terhadap objek, terdapat dua konsep yaitu perhitungan jarak antar objek atau dissimilarity dan perhitungan tingkat kesamaan antar objek atau similarity. Metode cosine similarity merupakan metode yang digunakan untuk menghitung similarity antara dua buah objek (Han dan Kamber 2006). Pada penelitian ini objek yang dimaksud adalah vektor sekuen DNA. Berikut adalah persamaan cosine similarity :
|| |||| ||
∑
√∑ √∑
Perhitungan tingkat kesamaan vektor dilakukan dengan membandingkan setiap vektor sekuen DNA menggunakan persamaan cosine similarity, dari perhitungan tersebut akan didapatkan nilai yang merupakan tingkat kesamaan antara setiap vektor sekuen DNA.
Single Link
6
dengan klasifikasi, dalam hal tidak ada variabel target untuk clustering. Clustering tidak mengklasifikasikan, meramalkan, atau memprediksi nilai dari sebuah variabel target. Algoritme clustering digunakan untuk menentukan segmen keseluruhan himpunan data menjadi subgrup yang relatif sama atau cluster, dengan kesamaan record dalam cluster dimaksimumkan dan kesamaan record di luar cluster diminimumkan (Larose 2005).
Gambar 3 Contoh pengelompokan single link
Secara umum metode utama clustering dapat diklasifikasikan menjadi kategori-kategori berikut (Han dan Kamber 2006):
Metode partisi. Misalkan ada sebuah basis data berisi n objek. Metode partisi membangun k partisi pada basis data tersebut, dengan tiap partisi merepresentasikan cluster dan k ≤ n. Partisi yang terbentuk harus memenuhi syarat yaitu setiap cluster harus berisi minimal satu objek dan setiap objek harus termasuk tepat satu cluster.
Metode hirarkhi, yaitu membuat sebuah dekomposisi berhirarki dari himpunan data (atau objek) menggunakan beberapa kriteria. Metode ini memiliki dua jenis pendekatan yaitu :
oAgglomerative, dimulai dengan titik-titik sebagai cluster individu. Pada setiap tahap dilakukan penggabungan setiap pasangan titik pada cluster sampai hanya satu titik (atau cluster) yang tertinggal.
oDivisive, dimulai dengan satu cluster besar yang berisi semua titik data. Pada setiap langkah, dilakukan pemecahan sebuah cluster sampai setiap cluster berisi sebuah titik (atau terdapat k cluster).
Metode berdasarkan kepekatan, merupakan pendekatan yang berdasarkan pada konektivitas dan fungsi kepadatan.
Metode berdasarkan grid, merupakan pendekatan yang berdasarkan pada struktur multiple-level granularity.
Metode berdasarkan model, yaitu: sebuah model yang dihipotesis untuk tiap cluster dan ide dasarnya adalah untuk menemukan model yang cocok untuk tiap cluster.
7
Dendrogram akan menampilkan gambaran penggabungan dan pembagian pada tingkat yang berurutan. Contoh dendrogram ditampilkan pada Gambar 3.
Gambar 4 Contoh dendrogram
Single link memberikan hasil bila kelompok-kelompok digabungkan menurut jarak antara anggota-anggota yang paling dekat. Input dari metode single link bisa berupa jarak atau tingkat kesamaan antara pasangan dari objek, kemudian dibentuk kelompok-kelompok dari entitas setiap objek dengan menggabungkan jarak paling pendek, atau tingkat kesamaan yang paling besar, diambil nilai yang terkecil terlebih dahulu, lalu dilakukan penggabungan dua vektor, dan membandingkan nilai vektor yang digabungkan dengan vektor lain, selanjutnya dilakukan pengambilan nilai yang terdekat untuk dibandingkan kembali. Cara perhitungan single link direpresentasikan pada Gambar 4.
Tahap Evaluasi
Confusion Matrix
Untuk melakukan pengujian terhadap perhitungan yang telah dilakukan dibutuhkan sebuah metode yang akan menguji ketepatan dan keakuratan perhitungan yang dilakukan sebelumnya.
Confusion matrix adalah sebuah matrik yang menyimpan nilai aktual dan nilai prediksi dari klasifikasi atau pengelompokan yang dilakukan (Kohavi dan Provost 1998). Kinerja sebuah sistem pengelompokan dievaluasi menggunakan data dalam matrik.
Perhitungan Akurasi
Kinerja dari pengelompokan single link ditentukan dengan menghitung besaran akurasi yang berhasil diperoleh, akurasi dihitung dengan persamaan berikut:
Akurasi = ∑
∑
Lingkungan Implementasi
8
Processor Intel Core i3 @2.4 GHz, RAM kapasitas 2 GB,
Harddisk kapasitas 320 GB,
Layar dengan resolusi 1366×768 piksel. Perangkat lunak berupa:
Sistem operasi Microsoft Windows 7 Ultimate, Program pengolahan data statistik R,
Microsoft Excel 2010
HASIL DAN PEMBAHASAN
Tahap Pengumpulan Data
Data yang telah dikumpulkan berupa sekuen DNA berformat standar fasta sebanyak 50 data. Data tersebut terdiri dari rangkaian huruf yang merepresentasikan nukleotida adenin (A), guanin (G), timin (T) dan sitosin (C). Urutan dalam format fasta dimulai dengan deskripsi sekuen DNA tersebut, dan diikuti oleh barisan data sekuen. Diawali oleh simbol lebih besar (>) dan deskripsi dari sekuen DNA, sisanya merupakan barisan huruf nukleotida yang panjangnya tidak melebihi delapan puluh karakter per baris dan tanpa mengandung spasi. Pada 50 data tersebut memiliki paling banyak 97 452 karakter yaitu Bacillus, sedangkan data paling sedikit adalah Streptococcus thermophilus dengan 3 090 karakter. Semua data sekuen DNA terdiri dari 5 genus yaitu Borellia (10 sekuen), Bacillus (10 sekuen), Methylobacterium (10 sekuen), Streptococcus (10 sekuen), dan Yersinia (10 sekuen).
Pada awalnya akan dilakukan pengelompokan dengan jumlah sekuen yang tidak sama setiap genus nya, tapi setelah dilakukan penelitian jumlah data sekuen setiap genus diputuskan sama yaitu 10 sekuen setiap genus, dengan 8 studi kasus pengelompokan menggunakan 2 sekuen setiap genus hingga menggunakan 9 sekuen setiap genus. Hal ini dilakukan untuk mengetahui jumlah sekuen dalam setiap genus akan mempengaruhi hasil pengelompokan.
Tahap Pengelompokan
Feature Vectors
Tahapan feature vectors merupakan proses ekstraksi ciri dari sebuah sekuen DNA, dari sebuah sekuen DNA yang terdiri dari urutan huruf ditransformasi menjadi sebuah vektor. Sehingga dari 50 sekuen DNA yang memiliki beragam ukuran diubah menjadi 50 vektor yang merepresentasikan urutan huruf dari sekuen DNA tersebut. Feature vectors dilakukan karena dari sebuah sekuen DNA tersebut harus didapatkan nilai-nilai yang bisa dijadikan sebagai identitasnya, sehingga bisa diteruskan pada tahap selanjutnya.
9
perhitungan parameter kedua yaitu parameter t, yaitu total jarak setiap nukleotida sejenis pada sekuen DNA dengan nukleotida pertama. Hasil dari perhitungan parameter t akan disimpan pada variabel tA, tC, tG, dan tT. Tahap terakhir dari feature vectors adalah parameter d, yaitu varians nukleotida. Parameter d didapatkan dari persamaan matematika yang telah ditentukan. Sehingga akan menghasilkan dA, dC, dG, dan dT.
Hasil perhitungan tiga parameter n, t, d untuk setiap nukleotida A, C, G dan T digabungkan kedalam sebuah vektor. Sehingga setiap vektor terdiri dari dua belas nilai yang masing-masing menyimpan informasi DNA tersebut.
Normalisasi Min-max
Normalisasi merupakan tahapan yang sangat penting untuk dilakukan, karena keberagaman nilai yang didapatkan pada proses feature vectors, sehingga nilai data harus diskalakan ke dalam range nilai tertentu agar tidak terdapat dimensi data yang terlalu besar ataupun terlalu kecil yang akan sangat mempengaruhi hasil pengelompokan. Data dengan kualitas yang rendah juga akan berdampak terhadap hasil pengelompokan yang rendah juga. Proses normalisasi menggunakan algoritme min-max melibatkan nilai terkecil dan nilai terbesar dari data. Setiap nilai dalam data dikurangkan dengan nilai paling kecil dan dibagi dengan nilai paling besar kurang nilai paling kecil, sehingga nilai yang didapatkan berada pada 0.0 hingga 1.0. Setiap data dilakukan perhitungan terhadap semua data vektor yang dimiliki.
Nilai dari vektor yang sebelumnya memiliki dimensi data yang terlalu jauh atau terlalu dekat, setelah dilakukan normalisasi data akan menjadi lebih terstruktur dan berkisar antar 0 sampai 1, sehingga akan berdampak kepada akurasi dan efisiensi pengelompokan.
Cosine Similarity
Perhitungan tingkat kesamaan dengan cosine similarity dilakukan antar tiap vektor, satu vektor Borellia akan dibandingkan dengan vektor tersebut dan vektor lain dari genus yang berbeda. Pada studi kasus 2 sekuen percobaan menggunakan 2 vektor setiap genus, dilakukan perhitungan tingkat kesamaan antara Borellia 1 terhadap Borellia 1, Borellia 1 terhadap Streptococcus 1, Borellia 1 terhadap Yersinia 1, Borellia 1 terhadap Methylobacterium 1, dan Borellia 1 terhadap Bacillus 1. Hal tersebut juga dilakukan pada Borellia 2 dan sekuen yang lain, sehingga akan menghasilkan matrik tingkat kesamaan seperti yang terdapat pada Tabel 2.
10
Tabel 2 Hasil cosine similarity untuk studi kasus 2 sekuen percobaan 1
Borellia 1 Borellia 2
Streptococc us 1
Streptococc
us 2 Yersinia 1 Yersinia 2
Methylobact
cus 1 0.9966989 0.9802347 Streptococ
cus 2 0.9891670 0.9747853 0.9913926
Yersinia 1 0.9706462 0.9456844 0.9763492 0.9898386
Yersinia 2 0.9684138 0.9434951 0.9738889 0.9888656 0.9998738 Methyloba
cterium 1 0.8135433 0.7577499 0.8350892 0.8729222 0.9280086 0.9307786 Methyloba
cterium 2 0.8293263 0.7751865 0.8501796 0.8863459 0.9378593 0.9403285 0.9995218
Bacillus 1 0.9912748 0.9879127 0.9845589 0.9875234 0.9785964 0.9770640 0.8384712 0.8524595
Bacillus 2 0.9908064 0.9877019 0.9838709 0.9872081 0.9783980 0.9768986 0.8384772 0.8524241 0.9999912
Single Link
Pengelompokan single link dilakukan dengan data tingkat kesamaan yang didapatkan dari cosine similarity, dimulai dari pengelompokan menggunakan 1 data tiap genus, hingga 9 data sekuen setiap genus. Studi kasus 2 sekuen percobaan 1 dengan 2 vektor setiap genus menggunakan data dari Tabel 2, langkah pertama adalah menentukan nilai terbesar selain 1, nilai terbesar merupakan vektor yang mempunyai tingkat kesamaan yang paling mirip. Vektor dengan tingkat kesamaan paling besar adalah antara Bacillus 1 dan Bacillus 2 yaitu 0.99999912, sehingga kedua vektor tersebut digabungkan.
Setelah Bacillus 1 dan Bacillus 2 digabungkan, kembali dilakukan perhitungan tingkat kesamaan antara vektor Bacillus1-Bacillus2 dengan vektor lain, contohnya antara Yersinia dengan Bacillus1-Bacillus2 didapatkan dengan membandingkan tingkat kesamaan antara Yersinia1-Bacillus1 dengan Yersinia 1-Bacillus2, yaitu 0.9770640 dengan 0.9768986, diambil nilai terbesar karena menggunakan single link, yaitu tingkat kesamaan yang paling mirip. Sehingga didapatkan tingkat kesamaan antara Bacillus1-Bacillus2 dengan Yersinia1 adalah 0.9770640. Hal yang sama juga dilakukan terhadap Methylobacterium dan Bacillus, sehingga akan didapatkan nilai tingkat kesamaan yang baru.
11
Setelah didapatkan nilai tingkat kesamaan yang baru kembali dilakukan pengelompokan single link, dilakukan pemilihan cara pemilihan yang sama, hingga menyisakan dua vektor terakhir. Dari nilai-nilai penggabungan semua vektor dibentuk ke dalam sebuah dendrogram yang akan digunakan untuk menentukan kelas dari setiap vektor.
Tahap Pengujian
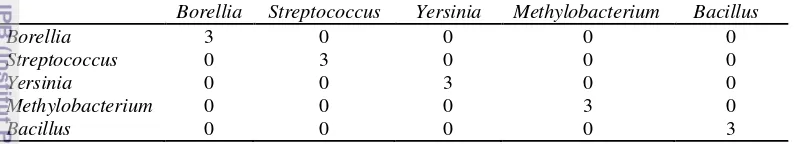
Tahap pengujian menggunakan confusion matrix, dilakukan perbandingan dengan nilai asli dan nilai setelah dilakukan pengelompokan. Pada studi kasus 3 sekuen percobaan 4 dengan menggunakan 3 vektor setiap genus, didapatkan akurasi paling tinggi dari semua percobaan sebesar 100%. Semua data dikelompokkan dengan benar sesuai kelompoknya. Hasil confusion matrix dari studi kasus 3 sekuen percobaan 4 dapat dilihat pada Tabel 3.
Tabel 3 Confusion matrix untuk studi kasus 3 sekuen percobaan 4
Borellia Streptococcus Yersinia Methylobacterium Bacillus dimana pada setiap studi kasus dilakukan 5 percobaan terhadap data yang berbeda menghasilkan akurasi rata-rata sebesar 86.7%. Hasil pengelompokan dengan akurasi studi kasus rata-rata paling tinggi didapatkan pada studi kasus 6 sekuen 89.2%. Adapun pengelompokan dengan akurasi paling rendah pada studi kasus 2 sekuen dengan 84%.
12
Tabel 4 Hasil akurasi rata-rata 8 studi kasus Studi
Dari 8 studi kasus yang dilakukan terjadi kenaikan akurasi dari studi kasus 2 sekuen dan mencapai akurasi tertinggi pada studi kasus 6 sekuen, namun pada studi kasus 7 sekuen hingga studi kasus 9 sekuen terjadi penurunan akurasi rata-rata.
Gambar 4 Grafik rata-rata akurasi 8 studi kasus
Dari Gambar 6 terlihat bahwa semakin banyak sekuen yang digunakan akurasi cenderung menurun, hal ini disebabkan karena semakin banyak jumlah data yang digunakan, menyebabkan selisih kemiripan antara vektor akan semakin kecil, sehingga mengakibatkan data yang berbeda genus dikelompokkan ke dalam kelas yang sama. Hal ini menunjukkan bahwa jumlah sekuen yang digunakan akan mempengaruhi hasil pengelompokan, semakin banyak data yang digunakan maka hasil pengelompokan akan menunjukkan grafik penurunan akurasi.
Analisis Kesalahan
13
menampilkan confusion matrix yang didapatkan pada studi kasus 4 sekuen percobaan 4.
Tabel 5 Confusion matrix studi kasus 4 sekuen percobaan 4
Borellia Streptococcus Yersinia Methylobacterium Bacillus semua vektor Yersinia dan Methylobactrium ditempatkan sesuai dengan genusnya, 4 vektor Borellia digabungkan ke dalam genus Bacillus, dan 1 vektor Bacillus dimasukkan ke dalam genus Borellia, sedangkan 3 vektor Streptococcus yang ditempatkan sesuai dengan kelompoknya, dan 1 vektor lain digabungkan ke dalam kelompok Borellia.
Untuk mengetahui penyebab terjadinya kesalahan dalam pengelompokan studi kasus di atas, dilakukan penjajaran antara sekuen yang salah dalam pengelompokannya. Penjajaran dilakukan untuk mencari kecocokan (kesamaan) antar karakter pada setiap sekuen. Penjajaran menggunakan data sekuen pada studi kasus 4 sekuen percobaan 4 karena mendapatkan akurasi terendah dalam penelitian. Data yang digunakan dalam penjajaran adalah genus Borellia karena pada percobaan tersebut semua vektor Borellia dikelompokan tidak pada kelasnya. Penjajaran dilakukan menggunakan Basic Local Alignment Search Tools (BLAST).
Tabel 6 Hasil penjajaran sekuen Borelliaburgdorferi
No Deskripsi Max
1 Borrelia bissettii DN127 plasmid lp25, complete sequence
16289 33883 87% 0.0 100%
2 Borrelia garinii Far04 plasmid Far04_lp36, complete sequence
5299 7800 28% 0.0 90%
3 Borrelia afzelii PKo plasmid lp28-3, complete sequence
5104 9367 30% 0.0 89%
4 Borrelia spielmanii A14S plasmid A14S_lp28-3, complete sequence
5068 8527 30% 0.0 88%
14
Tabel 7 Hasil penjajaran sekuen Borelliaduttoni
No Deskripsi Max
1 Borrelia crocidurae str. Achema plasmid clone 3 genomic sequence
6803 35436 73% 0.0 97%
2 Borrelia garinii PBr plasmid PBr_lp28-4, complete sequence
2168 2833 15% 0.0 82%
3 Borrelia recurrentis A1 plasmid pl33, complete sequence
2073 14715 45% 0.0 88%
4 Borrelia burgdorferi Bol26 plasmid Bol26_lp28-4, complete sequence
1496 1496 7% 0.0 78%
Penjajaran yang dilakukan terhadap Borellia duttoni mengasilkan total score tertinggi pada Borellia crocidurae sebesar 2246 dengan query cover 73% seperti yang ditampilkan pada Tabel 7. Sedangkan pada studi kasus 4 percobaan 4 Borellia duttoni dikelompokkan ke dalam genus Streptococcus.
Tabel 8 Hasil penjajaran sekuen Borelliagarinii
No Deskripsi Max
1 Borrelia burgdorferi 297 plasmid 297_lp38, complete sequence
11021 11071 29% 0.0 97%
2 Borrelia valaisiana VS116 plasmid VS116_lp28-3, complete sequence
10368 13149 38% 0.0 93%
3 Borrelia afzelii PKo plasmid lp17, complete sequence
8091 13527 47% 0.0 90%
4 Borrelia spielmanii A14S plasmid A14S_lp17, complete sequence
8074 14800 50% 0.0 89%
Tabel 8 di atas merupakan hasil penjajaran Borellia garinii menggunakan BLAST yang menghasilkan spesies dari genusnya sendiri, Borellia burgdorferi dengan total score sebesar 11 071 dengan query cover sebesar 29%. Pada studi kasus 4 percobaan 4 Borellia garinii dikelompokkan kedalam genus Bacillus. Tabel 9 merupakan hasil penjajaran Borellia spielmanii, dan total score terbesar adalah 14 909 dan query cover 56% Bacillus afzelii.
Tabel 9 Hasil penjajaran sekuen Borelliaspielmanii
No Deskripsi Max
15
pengelompokan pada studi kasus 4 sekuen percobaan 4 berbeda dengan hasil penjajaran sekuen menggunakan BLAST. Perbedaaan tersebut disebabkan karena kurang kayanya informasi yang digunakan pada saat ekstraksi fitur menggunakan feature vectors sehingga terjadi beberapa kesalahan dalam pengelompokan yang dilakukan.
SIMPULAN DAN SARAN
Simpulan
Single link merupakan algoritme yang digunakan untuk pengelompokan data, sehingga biasa menjadi sebuah informasi yang dapat digunakan untuk berbagai keperluan. Dari sekumpulan data yang tidak berhubungan digabungkan ke dalam kelompok berdasarkan tingkat kemiripan atau jarak dari data tesebut.
Penelitian ini dilakukan untuk mengelompokkan 50 data sekuen DNA berformat Fasta. Data sekuen DNA tersebut diekstraksi untuk mendapatkan ciri dari setiap data menggunakan feature vectors, dilakukan normalisasi dengan min-max dan perhitungan tingkat kemiripan menggunakan cosine similarity, selanjutnya dilakukan pengelompokan dengan algoritme single link. Dari 8 studi kasus yang masing-masing terdiri dari 5 percobaan menggunakan 50 data sekuen DNA didapatkan akurasi rata-rata 86.7%. Dengan akurasi terbesar didapatkan pada studi kasus 3 sekuen percobaan 4 mendapatkan akurasi 100%, sedangkan nilai akurasi terkecil pada studi kasus 4 sekuen percobaan 4 yang mendapatkan akurasi 70%.
Untuk analisis pengaruh jumlah sekuen terhadap hasil pengelompokan didapatkan bahwa semakin banyak jumlah sekuen yang digunakan hasil akurasi akan cenderung semakin menurun, yang disebabkan oleh semakin kecilnya selisih tingkat kesamaan antar vektor sehingga pada saat pemotongan threshold vektor yang beda genus dikelompokkan pada kelas yang sama.
Faktor yang paling berpengaruh terhadap hasil pengelompokan sekuen DNA adalah ukuran sekuen dan jumlah sekuen yang digunakan dalam penelitian. Setelah dilakukan perbandingan hasil pengelompokan dengan penjajaran sekuen menggunakan BLAST diketahui bahwa kemiripan antara vektor yang salah dalam pengelompokan sangat kecil. Perbedaan hasil antar pengelompokan dengan BLAST disebabkan karena kurang kayanya informasi yang digunakan pada saat ekstraksi fitur menggunakan feature vectors sehingga terjadi beberapa kesalahan dalam pengelompokan yang dilakukan.
Saran
Untuk pengembangan dari penelitian ini disarankan untuk melakukan hal-hal berikut:
1 Menambah data sekuen DNA yang digunakan.
2 Menggunakan algoritme pengelompokan lain seperti k-means dan complete link sehingga dapat dilakukan perbandingan.
16
DAFTAR PUSTAKA
Han H, Kamber M. 2006. Data Mining: Concepts and Techniques. San Francisco (US): Morgan Kaufmann.
Jain AK, Murty MN, Flynn PJ. 1999. Data Clustering: a Review. New York (US): ACM Computing Surveys.
Kohavi R, Provost F. 1998. Machine Learning. Boston (US): Springer Netherlands. 30:271-274.
Larose DT. 2005. Discovering Knowledge in Data: an Introduction to Data Mining. New Jersey (US): J Wiley.
Liu L, Yeo YK, Yau S. 2006. Molecular Phylogenetics and Evolution, Detroit (US): Academic Press. 41:64-69.
17
Lampiran 1 Data yang digunakan dalam penelitian
No Nama sekuen Genus
1. Borrelia afzelii PKo plasmid lp25 Borellia
2. Borrelia bissettii DN127 plasmid lp25 Borellia
3. Borrelia burgdorferi B31 plasmid lp25 Borellia
4. Borrelia burgdorferi ZS7 plasmid ZS7_lp28-1 Borellia
5. Borrelia duttonii Ly plasmid pl23b Borellia
6. Borrelia garinii Far04 plasmid Far04_lp17 Borellia
7. Borrelia sp. SV1 plasmid SV1_lp28-2 Borellia
8. Borrelia spielmanii A14S plasmid A14S_lp38 Borellia
9. Borrelia recurrentis A1 plasmid pl23 Borellia
10. Borrelia valaisiana VS116 plasmid VS116_cp32-5 Borellia 11. Streptococcus agalactiae plasmid pGB3634 Streptococcus 12. Streptococcus agalactiae plasmid pLS1 Streptococcus 13. Streptococcus dysgalactiae subsp. equisimilis plasmid
pSdyT132
Streptococcus
14. Streptococcus mutans UA140 plasmid pUA140 Streptococcus 15. Streptococcus pneumoniae D39 plasmid pDP1 Streptococcus 16. Streptococcus pneumoniae plasmid pSMB1 Streptococcus
17. Streptococcus suis plasmid pSSU1 DNA Streptococcus
18. Streptococcus pyogenes 71-724 plasmid pDN571 Streptococcus 19. Streptococcus pyogenes plasmid pDN281 Streptococcus 20. Streptococcus thermophilus LMD-9 plasmid 2 Streptococcus 21. Yersinia enterocolitica subsp. enterocolitica 8081
plasmid pYVe8081
Yersinia
22. Yersinia enterocolitica subsp. palearctica 105.5R(r) plasmid 105.5R(r)p
Yersinia
23. Yersinia pestis Angola plasmid new_pCD Yersinia
24. Yersinia pestis Antiqua plasmid pCD Yersinia
25. Yersinia pestis biovar Microtus str. 91001 plasmid pCD1
Yersinia
26. Yersinia pestis CO92 plasmid pCD1 Yersinia
27. Yersinia pestis Z176003 plasmid pCD1 Yersinia
28. Yersinia pseudotuberculosis IP 32953 plasmid pYV Yersinia 29. Yersinia pestis strain KIM5 plasmid pCD1 Yersinia 30. Yersinia pseudotuberculosis PB1/+ plasmid pYPTS01 Yersinia
31. Methylobacterium extorquens AM1 plasmid p1META1 Methylobacterium 32. Methylobacterium extorquens AM1 plasmid p2META1 Methylobacterium 33. Methylobacterium extorquens DM4 plasmid p2METDI Methylobacterium 34. Methylobacterium nodulans ORS 2060 plasmid
pMNOD03
Methylobacterium
35. Methylobacterium nodulans ORS 2060 plasmid pMNOD04
Methylobacterium
36. Methylobacterium radiotolerans JCM 2831 plasmid pMRAD02
Methylobacterium
37. Methylobacterium radiotolerans JCM 2831 plasmid pMRAD04
18
Lanjutan lampiran 1
38. Methylobacterium radiotolerans JCM 2831 plasmid pMRAD05
Methylobacterium
39. Methylobacterium radiotolerans JCM 2831 plasmid pMRAD03
Methylobacterium
40. Methylobacterium radiotolerans JCM 2831 plasmid pMRAD06
Methylobacterium
41. Bacillus anthracis str. A0248 plasmid pXO2 Bacillus 42. Bacillus anthracis str. 'Ames Ancestor' plasmid pXO2 Bacillus 43. Bacillus anthracis str. CDC 684 plasmid pX02 Bacillus
44. Bacillus cereus E33L plasmid pE33L54 Bacillus
45. Bacillus cereus Q1 plasmid pBc53 Bacillus
46. Bacillus megaterium QM B1551 plasmid pBM400 Bacillus 47. Bacillus anthracis str. H9401 plasmid BAP2 Bacillus 48. Bacillus thuringiensis str. Al Hakam plasmid pALH1 Bacillus
49. Bacillus anthracis plasmid pXO2 Bacillus
50. Bacillus weihenstephanensis KBAB4 plasmid pBWB404
19
Lampiran 2 Tampilan awal pengelompokan single link
20
Lampiran 4 Feature vectors untuk 50 data sekuen DNA
nA tA dA nC tC dC nG tG dG nT tT dT
Borellia 9052 116894478 48144439 3829 42598667 50027915 3948 49946834 46716741 7965 97918842 56423500
Borellia 8566 100141185 44452323 2744 28495274 41422686 2691 33591237 44092917 9028 102928210 44112983
Borellia 8884 108985242 48047662 2816 30664859 46210859 2828 36750924 48288288 9642 115752321 49455219
Borellia 8354 102209626 40861795 2737 32998356 45659548 4652 40450974 48162132 7679 98624375 42402181
Borellia 8287 97419542 42166069 2298 24509026 48804127 4372 56006276 37195289 8079 87382286 47167258
Borellia 8903 96621123 42463097 3211 38501270 42100836 2204 25339703 39422997 8115 91146432 41572869
Borellia 9128 102305008 41320639 3220 32165029 38933898 2685 35956259 44103542 7912 92798744 46456998
Borellia 8041 87409628 41056263 2567 24433732 37007496 2826 31371149 39863335 8274 92393269 37360429
Borellia 8320 86274582 34801643 2843 27894542 36575991 2097 21873992 37751136 7672 83020730 37710163
Borellia 9181 108364065 44643015 2915 31978201 44181546 3775 43787455 49141656 7548 90083350 45585095
Streptococcus 2244 6639429 2901026 901 2569281 2862049 1062 3086348 2706481 1635 4766503 2840612
Streptococcus 1414 3160950 1597320 672 1484976 1738264 969 2134727 1732975 1353 2932375 1499066
Streptococcus 1139 1970928 1097983 539 1007213 1104365 724 1370346 1149789 1176 2050766 953513
Streptococcus 2072 5818357 2511431 766 2218711 2852391 1078 3052201 2803021 1724 4812711 2629893
Streptococcus 1116 1799493 827778 365 567721 837588 653 1040996 794227 1027 1586170 857901
Streptococcus 1113 1794724 830722 365 569451 843048 653 1041326 795001 1031 1592040 853842
Streptococcus 954 1384230 785456 457 730081 808011 603 952351 863245 1027 1555658 676677
Streptococcus 1753 4396562 2119689 744 1863397 1929570 1090 2615816 1849436 1388 3497050 2221286
Streptococcus 1080 1790584 892712 537 894581 994214 601 964008 1008942 1133 1963752 903908
Streptococcus 1096 1882634 944624 539 891283 940580 640 1154104 891779 1086 1718459 947402
Yersinia 19032 646486399 379253428 14799 505427784 385684578 14945 518495121 380414398 18945 622623756 382150109
Yersinia 19335 668294570 407264727 15150 535079705 403584020 15671 560950215 396932191 19548 664964466 408261528
Yersinia 18583 626558942 390014962 15319 526745063 401150575 15122 528271416 380893141 19166 643328534 378285993
21
Lanjutan lampiran 4
Yersinia 19675 688852716 397373478 15631 536434730 419683066 15833 558655972 426248998 19020 677164143 401490572
Yersinia 19135 667507579 412730268 15863 559404596 429064248 15659 560652642 411066779 19648 683796543 397354611
Yersinia 19783 695845998 403023973 15709 543016633 422240368 15910 565063619 429376601 19157 685324711 408333451
Yersinia 18626 630557171 391659760 15355 528773895 403051059 15154 530227286 383171323 19207 645721959 379578906
Yersinia 18670 645606503 384775930 15384 506884687 390837056 15177 520692615 402705637 19294 674619745 387063708
Yersinia 19047 663346515 404097128 15704 550016066 389078553 15494 533869676 417800433 19567 689590509 412308479
Methylobacterium 6886 151375560 159776466 15220 339324165 164764386 14802 324688276 164358950 7287 161188914 158037005
Methylobacterium 6482 119923569 114035718 12403 240836972 119568270 12299 233629915 124157046 6674 122204697 114720920
Methylobacterium 7178 136797911 123811636 12113 233908012 124533878 12476 243827900 120736044 6812 129616508 129142872
Methylobacterium 7150 142909935 134682544 13315 269346909 137845345 12657 257317015 135673597 7341 149033094 136832683
Methylobacterium 7095 135350686 119711464 11466 211529747 115944113 11646 216523966 117577923 7335 141277712 116870331
Methylobacterium 8793 204248878 178251135 14234 336494809 185403716 15133 354265529 183684185 8843 209608287 188405150
Methylobacterium 7881 175509098 150837112 13826 286761133 157414967 13355 282272927 154372973 7923 179290462 147333771
Methylobacterium 6652 124720304 116554083 12201 230261434 116714137 11837 227360554 119641721 7053 129905861 122229678
Methylobacterium 7130 126061407 109844184 11286 208902685 110004908 11294 210303986 113211690 6700 117557767 106144293
Methylobacterium 5539 80788348 64729916 8471 113812166 64528729 8511 115370288 61862546 5315 77436728 67335779
Bacillus 29846 1458062894 692252541 17847 816464376 799479209 13488 643040426 738952368 33649 1578749339 774116806
Bacillus 29846 1450166866 690289584 17847 818048745 803126640 13488 641869322 738524478 33649 1586232102 775047011
Bacillus 29847 1442035324 688586669 17858 822596585 809060256 13501 640552073 738655057 33669 1595401393 776276833
Bacillus 18130 485505494 236239173 8901 237521735 241985218 8190 217849694 245348982 18280 490274827 236045751
Bacillus 15895 407389410 246397472 10403 287731771 209648085 8109 201281351 236523003 18359 495696463 227392660
Bacillus 17546 451220844 231725301 9718 277791751 267586589 9927 273312236 243562048 16674 448367349 232915835
Bacillus 30179 1486300802 708196565 18134 843826558 826719332 13698 662375093 761717420 34220 1637652112 799993945
Bacillus 29829 1449025030 690512713 17849 818129957 802811394 13494 642173136 738120390 33652 1586419953 774899948
22
Lanjutan lampiran 4
Bacillus 18179 477382314 226526793 8289 220143562 236983303 10397 278296030 210249049 15965 419656129 251607371
Lampiran 5 Hasil normalisasi min-max untuk 50 data sekuen DNA
Borellia 0.000005304547 0.071379103167 0.029398236889 0.000002115224 0.026011819716 0.030548344660 0.000002187889 0.030498834133 0.028526441037 0.000004640791 0.059792002286 0.034453683516
Borellia 0.000005007780 0.061149032560 0.027143718487 0.000001452690 0.017399858701 0.025293729925 0.000001420326 0.020511608809 0.026924254244 0.000005289891 0.062850874851 0.026936507155
Borellia 0.000005201961 0.066549482941 0.029339141907 0.000001496655 0.018724673336 0.028217534091 0.000001503983 0.022441009859 0.029486075467 0.000005664819 0.070681667340 0.030198639052
Borellia 0.000004878327 0.062412085590 0.024951232809 0.000001448415 0.020149577626 0.027880886815 0.000002617773 0.024700373003 0.029409040773 0.000004466151 0.060222822209 0.025891839384
Borellia 0.000004837414 0.059487114509 0.025747662210 0.000001180349 0.014965734348 0.029801062460 0.000002446796 0.034198913843 0.022712352653 0.000004710403 0.053358060503 0.028801540429
Borellia 0.000005213563 0.058999575567 0.025929036547 0.000001737854 0.023509824400 0.025707828955 0.000001122949 0.015472971006 0.024072658960 0.000004732386 0.055656562616 0.025385436236
Borellia 0.000005350955 0.062470328742 0.025231416921 0.000001743350 0.019640722796 0.023774000224 0.000001416663 0.021955763224 0.026930742193 0.000004608428 0.056665514613 0.028367834056
Borellia 0.000004687199 0.053374756361 0.025069980889 0.000001344608 0.014919757540 0.022597680531 0.000001502762 0.019155955506 0.024341542744 0.000004829476 0.056417919237 0.022813192163
Borellia 0.000004857565 0.052681662727 0.021250719552 0.000001513142 0.017033033458 0.022334190445 0.000001057612 0.013356702388 0.023051769748 0.000004461877 0.050694761662 0.023026750388
Borellia 0.000005383318 0.066170173359 0.027260160826 0.000001557108 0.019526639933 0.026978373809 0.000002082250 0.026737729850 0.030007167940 0.000004386158 0.055007412391 0.027835423547
Streptococcus 0.000001147375 0.004054014544 0.001771231891 0.000000327298 0.001568658297 0.001747431348 0.000000425609 0.001884395144 0.001652436792 0.000000775501 0.002910348924 0.001734341263
Streptococcus 0.000000640551 0.001929949396 0.000975149328 0.000000187464 0.000906548662 0.001061214024 0.000000368821 0.001303306398 0.001057984399 0.000000603303 0.001790374544 0.000915152445
Streptococcus 0.000000472628 0.001203285744 0.000670238958 0.000000106250 0.000614812033 0.000674136001 0.000000219216 0.000836552095 0.000701873278 0.000000495221 0.001252037256 0.000582021179
Streptococcus 0.000001042346 0.003552642991 0.001533333326 0.000000244863 0.001354589585 0.001741533879 0.000000435380 0.001863543947 0.001711387055 0.000000829847 0.002938564935 0.001605669829
Streptococcus 0.000000458583 0.001098602315 0.000505243561 0.000000000000 0.000346444842 0.000511233845 0.000000175862 0.000635440961 0.000484756299 0.000000404237 0.000968340798 0.000523637581
Streptococcus 0.000000456752 0.001095690218 0.000507041257 0.000000000000 0.000347501232 0.000514567888 0.000000175862 0.000635642469 0.000485228927 0.000000406680 0.000971925199 0.000521159032
Streptococcus 0.000000359661 0.000845030088 0.000479400459 0.000000056178 0.000445586799 0.000493173229 0.000000145330 0.000581311626 0.000526900790 0.000000404237 0.000949709242 0.000412976691
Streptococcus 0.000000847555 0.002684451690 0.001294123738 0.000000231429 0.001137624042 0.001178031290 0.000000442707 0.001597073984 0.001129099031 0.000000624675 0.002135182285 0.001356161958
Streptococcus 0.000000436601 0.001093162208 0.000544894238 0.000000105028 0.000546035506 0.000606874448 0.000000144109 0.000588429745 0.000615867813 0.000000468964 0.001198903860 0.000551730856
23
Lanjutan lampiran 5
Yersinia 0.000011398638 0.394764048696 0.231583463148 0.000008813840 0.308629365142 0.235510519075 0.000008902992 0.316608678829 0.232292386765 0.000011345513 0.380192792601 0.233352264729
Yersinia 0.000011583659 0.408080781658 0.248688014864 0.000009028171 0.326735730585 0.246440463144 0.000009346309 0.342533051381 0.242378653903 0.000011713724 0.406047318802 0.249296691893
Yersinia 0.000011124465 0.382595736943 0.238154783344 0.000009131368 0.321646344508 0.244954527564 0.000009011073 0.322578382106 0.232584721811 0.000011480463 0.392835760215 0.230992717892
Yersinia 0.000011817531 0.421505794663 0.250122925555 0.000009340203 0.333734537884 0.262345569983 0.000009459276 0.346803302986 0.247834711955 0.000011418179 0.406786367871 0.246674001197
Yersinia 0.000011791274 0.420634211310 0.242648117176 0.000009321884 0.327563150091 0.256271030620 0.000009445232 0.341132116778 0.260280388539 0.000011391311 0.413496812885 0.245162140080
Yersinia 0.000011461533 0.407600221001 0.252025440547 0.000009463550 0.341589249378 0.261999465873 0.000009338982 0.342351344251 0.251009663534 0.000011774787 0.417546758188 0.242636596412
Yersinia 0.000011857222 0.424904522146 0.246098481401 0.000009369513 0.331582260389 0.257832597055 0.000009492250 0.345044821059 0.262190198122 0.000011474967 0.418479904079 0.249340610266
Yersinia 0.000011150722 0.385037177260 0.239159147064 0.000009153350 0.322885211077 0.246115020937 0.000009030614 0.323772695856 0.233975849079 0.000011505499 0.394297258366 0.231782209921
Yersinia 0.000011177590 0.394226757418 0.234955670951 0.000009171059 0.309518994456 0.238656778962 0.000009044658 0.317950535548 0.245904095750 0.000011558624 0.411943126025 0.236352657828
Yersinia 0.000011407798 0.405059348677 0.246753782506 0.000009366460 0.335856327212 0.237582983508 0.000009238228 0.325996850049 0.255121437611 0.000011725326 0.421084730171 0.251767883346
Methylobacterium 0.000003981921 0.092434301296 0.097564150188 0.000009070915 0.207201439880 0.100609925951 0.000008815672 0.198264320601 0.100362354390 0.000004226784 0.098426633926 0.096501982360
Methylobacterium 0.000003735226 0.073228758324 0.069633457302 0.000007350769 0.147062162295 0.073011801941 0.000007287264 0.142661313938 0.075813848230 0.000003852467 0.074621684509 0.070051862498
Methylobacterium 0.000004160225 0.083532745134 0.075602930371 0.000007173686 0.142831128430 0.076043953318 0.000007395345 0.148888513963 0.073724880287 0.000003936734 0.079147561890 0.078858345333
Methylobacterium 0.000004143128 0.087264933013 0.082241037661 0.000007907664 0.164471197551 0.084172340214 0.000007505869 0.157125378135 0.082846204786 0.000004259758 0.091003920261 0.083553977975
Methylobacterium 0.000004109543 0.082649025501 0.073099240555 0.000006778608 0.129166278720 0.070798781372 0.000006888522 0.132215900845 0.071796435485 0.000004256094 0.086268247971 0.071364358273
Methylobacterium 0.000005146393 0.124720358510 0.108845345371 0.000008468834 0.205473748992 0.113212929025 0.000009017790 0.216325091491 0.112162931061 0.000005176925 0.127992976763 0.115045695976
Methylobacterium 0.000004589498 0.107170974123 0.092105508559 0.000008219696 0.175104852741 0.096122147024 0.000007932089 0.172364217555 0.094264612902 0.000004615145 0.109479990070 0.089966261917
Methylobacterium 0.000003839034 0.076157790708 0.071171247619 0.000007227422 0.140604416917 0.071268981463 0.000007005152 0.138833051298 0.073056653357 0.000004083896 0.079324249639 0.074636938668
Methylobacterium 0.000004130915 0.076976709017 0.067073979069 0.000006668695 0.127562114706 0.067172122035 0.000006673580 0.128417791747 0.069130280725 0.000003868344 0.071784127618 0.064814713015
Methylobacterium 0.000003159402 0.049331601269 0.039525833938 0.000004949770 0.069496949647 0.039402983032 0.000004974196 0.070448386363 0.037774930545 0.000003022621 0.047285000087 0.041117053197
Bacillus 0.000018001996 0.890337357543 0.422710247932 0.000010675041 0.498557774872 0.488186114944 0.000008013303 0.392659832701 0.451226583646 0.000020324223 0.964032174052 0.472699059747
Bacillus 0.000018001996 0.885515802524 0.421511606643 0.000010675041 0.499525238805 0.490413347326 0.000008013303 0.391944720955 0.450965300988 0.000020324223 0.968601376884 0.473267071232
Bacillus 0.000018002607 0.880550435489 0.420471754915 0.000010681758 0.502302288327 0.494036593850 0.000008021241 0.391140368624 0.451045036500 0.000020336436 0.974200425043 0.474018037975
Bacillus 0.000010847850 0.296464208516 0.144254606288 0.000005212341 0.145037777681 0.147763316250 0.000004778183 0.133025430711 0.149817332928 0.000010939444 0.299376508405 0.144136496928
24
Lanjutan lampiran 5
Bacillus 0.000010491242 0.275528957745 0.141498298661 0.000005711226 0.169627875102 0.163396292582 0.000005838848 0.166892546905 0.148726176641 0.000009958772 0.273786526849 0.142225274956
Bacillus 0.000018205336 0.907580283612 0.432446154256 0.000010850292 0.515265955992 0.504819763124 0.000008141536 0.404466168838 0.465127617270 0.000020672893 1.000000000000 0.488500428413
Bacillus 0.000017991615 0.884818562710 0.421647856002 0.000010676263 0.499574829324 0.490220848523 0.000008016967 0.392130239031 0.450718552557 0.000020326055 0.968716084422 0.473177270088
Bacillus 0.000009837867 0.283160306732 0.148222926788 0.000006773113 0.196213642851 0.172930601099 0.000005129906 0.153037804563 0.152751959907 0.000011525649 0.322954021188 0.163183490928
25
Lampiran 6 Confusion matrix studi kasus 2 sekuen
Borellia Streptococcus Yersinia Methylobacterium Bacillus
Borellia 8 2 0 0 0
Streptococcus 1 8 0 0 1
Yersinia 0 0 10 0 0
Methylobacterium 0 0 0 10 0
Bacillus 2 1 1 0 6
Lampiran 7 Confusion matrix studi kasus 3 sekuen
Borellia Streptococcus Yersinia Methylobacterium Bacillus
Borellia 10 3 0 0 2
Streptococcus 1 12 0 0 2
Yersinia 0 0 15 0 0
Methylobacterium 0 0 0 15 0
Bacillus 2 0 0 0 13
Lampiran 8 Confusion matrix studi kasus 4 sekuen
Borellia Streptococcus Yersinia Methylobacterium Bacillus
Borellia 12 5 0 0 3
Streptococcus 1 18 0 0 1
Yersinia 0 0 20 0 0
Methylobacterium 0 0 0 20 0
Bacillus 4 0 0 0 16
Lampiran 9 Confusion matrix studi kasus 5 sekuen
Borellia Streptococcus Yersinia Methylobacterium Bacillus
Borellia 20 5 0 0 0
Streptococcus 0 19 0 0 6
Yersinia 0 0 25 0 0
Methylobacterium 0 0 0 25 0
Bacillus 4 0 0 0 21
Lampiran 10 Confusion matrix studi kasus 6 sekuen
Borellia Streptococcus Yersinia Methylobacterium Bacillus
Borellia 24 6 0 0 0
Streptococcus 0 22 0 0 8
Yersinia 0 0 30 0 0
Methylobacterium 0 0 0 30 0
26
Lampiran 11 Confusion matrix studi kasus 7 sekuen
Borellia Streptococcus Yersinia Methylobacterium Bacillus
Borellia 29 6 0 0 0
Streptococcus 0 22 0 0 13
Yersinia 0 0 35 0 0
Methylobacterium 0 0 0 35 0
Bacillus 0 2 0 0 33
Lampiran 12 Confusion matrix studi kasus 8 sekuen
Borellia Streptococcus Yersinia Methylobacterium Bacillus
Borellia 34 6 0 0 0
Streptococcus 0 25 0 0 15
Yersinia 0 0 40 0 0
Methylobacterium 0 0 0 40 0
Bacillus 0 2 0 0 32
Lampiran 13 Confusion matrix studi kasus 9 sekuen
Borellia Streptococcus Yersinia Methylobacterium Bacillus
Borellia 40 5 0 0 0
Streptococcus 0 32 0 0 13
Yersinia 0 0 45 0 0
Methylobacterium 0 0 0 45 0
27