PEMODELAN
SUPPORT VECTOR MACHINE
UNTUK KLASIFIKASI
BAKTERI PATOGEN DAN NON PATOGEN BERDASARKAN
DATA SEKUENS GENOM
ESKAWATI KURNIA DWIMARDYASTUTI
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Pemodelan Support Vector Machine untuk Klasifikasi Bakteri Patogen dan Non Patogen Berdasarkan Data Sekuens Genom adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tulisan ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
ABSTRAK
ESKAWATI KURNIA DWIMARDYASTUTI. Pemodelan Support Vector Machine untuk Klasifikasi Bakteri Patogen dan Non Patogen Berdasarkan Data Sekuens Genom. Dibimbing oleh MUHAMMAD ASYHAR AGMALARO.
Bakteri merupakan mikroorganisme yang dapat dibedakan menjadi 2 domain yaitu patogenik (bakteri berbahaya) dan bakteri non patogenik (bakteri tidak berbahaya). Tujuan penelitian ini adalah membuat pemodelan klasifikasi bakteri patogen dan non patogen berdasarkan data sekuens genom dan menguji pengaruh kernel dan panjang fragmen terhadap hasil akurasi. Data sekuens genom diperoleh dari NCBI dengan panjang fragmen 100 bp, 400 bp, 800 bp, 1000 bp, dan 5000 bp yang kemudian dilakukan ekstraksi ciri menggunakan metode K-Mers dan metode Support Vector Machine (SVM) dengan 3 kernel utama yaitu Kernel Linear, Radial Basic Function (RBF), dan Polynomial sebagai metode klasifikasinya. Dari proses tersebut didapatkan panjang fragmen 5000 bp dengan kernel RBF merupakan akurasi tertinggi yaitu mencapai 96.61%.
Kata kunci : K-Mers, non patogenik, patogenik, SVM,
ABSTRACT
ESKAWATI KURNIA DWIMARDYASTUTI. Modeling Support Vector Machine for Pathogenic and Non-Pathogenic Bacteria Based on Data from the Sequence Genome. Supervised by MUHAMMAD ASYHAR AGMALARO.
Bacteria are microorganisms that can be divided into two domains, pathogenic (harmful bacteria) and non-pathogenic bacteria (bacteria are harmless). The purpose of this research is making modeling clasifications of pathogenic bacteria and non pathogenic based on the sequence genom and test the effect of kernels and fragment length of the accuration result. The genome sequence obtained from NCBI with long fragments 100 bp, 400 bp, 800 bp, 1000 bp, and then 5000 bp extraction features done using methods K-Mers and methods of Support Vector Machine (SVM) with 3 main kernel, that is Linear, Radial Basic Function (RBF) and a Polynomial as a method of classifier. From this process, 5000 bp fragment length is obtained with RBF is the highest accuracy reached 96.61%.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Komputer
pada
Departemen Ilmu Komputer
PEMODELAN
SUPPORT VECTOR MACHINE
UNTUK KLASIFIKASI
BAKTERI PATOGEN DAN NON PATOGEN BERDASARKAN
DATA SEKUENS GENOM
ESKAWATI KURNIA DWIMARDYASTUTI
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
Penguji :
Judul Skripsi : Pemodelan Support Vector Machine untuk Klasifikasi Bakteri Patogen dan Non Patogen Berdasarkan Data Sekuens Genom Nama : Eskawati Kurnia Dwimardyastuti
NIM : G64124045
Disetujui oleh
Muhammad Asyhar Agmalaro, SSi MKom Pembimbing
Diketahui oleh
Dr Ir Agus Buono, MSi MKom Ketua Departemen
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Juni 2014 ini ialah sekuens genom, dengan judul Pemodelan Support Vector Machine untuk Klasifikasi Bakteri Patogen dan Non Patogen Berdasarkan Data Sekuens Genom.
Terima kasih penulis ucapkan kepada Bapak M. Asyhar Agmalaro, SSi MKom selaku pembimbing, Ibu Wiwin Imro’atun Khoiriyah, MSi yang telah banyak memberi saran, Bapak Auzi Asfarian, SKom, MKom serta Bapak Aziz Kustiyo, SSi, MKom selaku dosen penguji. Ungkapan terima kasih juga disampai-kan kepada ayah, ibu, seluruh keluarga, teman – teman Vilbar dan Riverside, seluruh teman-teman Alih Jenis Ilmu Komputer IPB angkatan 7, serta keluarga besar SMK Pembangunan Bogor Utara atas segala doa dan kasih sayangnya.
Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL ix
DAFTAR GAMBAR ix
DAFTAR LAMPIRAN ix
PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 3
METODE 3
Studi Literatur 4
Pengumpulan Data 4
Praproses 4
Ekstraksi Ciri K-Mers 4
Normalisasi 5
Pembagian Data 5
Grid Search 6
Pelatihan SVM 6
Pengujian 7
Analisis 8
HASIL DAN PEMBAHASAN 8
Pengumpulan Data 8
Praproses 8
Ekstraksi Ciri 9
Normalisasi 9
K-Fold Cross Validation 9
Grid Search 10
Pelatihan SVM 10
Pengujian SVM 10
SIMPULAN DAN SARAN 13
Simpulan 13
Saran 14
DAFTAR PUSTAKA 14
LAMPIRAN 15
DAFTAR TABEL
1 Ilustrasi 5-fold cross validation 5
2 Pembanding parameter 8
3 Akurasi pada tiap panjang fragmen dan kernel pada 3-Mers 10
DAFTAR GAMBAR
1 Metode penelitian 3
2 Ilustrasi K-Mers 5
3 Support Vector Machine oleh Nugroho. et al (2003) 6 4 Contoh hasil ekstraksi ciri K-Mers dengan nilai K = 3 pada fragmen
100 bp 9
5 Normalisasi dengan nilai 3-Mers pada fragmen 100 bp 9
6 Perbandingan akurasi kernel pada 3-Mers 11
7 Pengaruh nilai K terhadap akurasi pada kernel RBF 12
8 Perbedaan akurasi kernel pada 4-Mers 12
9 Perbedaan akurasi kernel pada 5-Mers 13
DAFTAR LAMPIRAN
1 Nama organisme 15
2 Hasil proses grid search 16
3 Akurasi pada tiap panjang fragmen dan kernel pada 4-Mers 16 4 Akurasi pada tiap panjang fragmen dan kernel pada 5-Mers 16 5 Pengaruh nilai K terhadap akurasi pada kernelPolynomial 17 6 Pengaruh nilai K terhadap akurasi pada kernelLinear 17
7 Selisih akurasi kernel pada 3-Mers 18
8 Selisih akurasi kernel pada 4-Mers 18
PENDAHULUAN
Latar Belakang
Bakteri dapat dibedakan menjadi 2 jenis yaitu patogen dan non patogen. Bakteri patogen adalah bakteri yang merugikan bagi tubuh manusia maupun makhluk hidup lainnya, sedangkan bakteri non patogen merupakan bakteri yang tidak merugikan bagi makhluk hidup lainnya. Menurut (Harahap LH 2013) identifikasi bakteri patogen dan non patogen dapat dilakukan dengan cara konvensional. Namun, cara konvensional memerlukan waktu yang lama karena memerlukan proses isolasi, uji fisiologi dan biokimia untuk mengetahui ada atau tidak ada enzym tertentu dan reaksi hipersensitif, uji morfologi dengan menggunakan mikroskop untuk melihat struktur eksternal dari bakteri tersebut, seperti: kapsul, slime (lapisan lendir), fimbriae, dan pili. Sedangkan menurut (Louws dan Cuppels 2001) identifikasi bakteri dapat menggunakan molekuler berbasis DNA. Deoxyribo nucleic acid (DNA) adalah rantai ganda molekul sederhana (nukleotida) yang diikat bersama-sama. Nukleotida DNA ini terdiri atas adenin (A), guanin (G), sitosin (S), dan timin (T) (de Carvalho 2003). Urutan nukleotida DNA dalam tubuh suatu organisme disebut sekuen genom, secara sederhana sekuen genom dapat dianalogikan berupa susunan huruf yang memiliki makna yang penting dan spesifik tetapi tidak langsung dapat memberikan informasi genetik dalam suatu spesies.
Identifikasi berbasis DNA memiliki keuntungan karena keakuratan identifikasi tidak tergantung pada kondisi lingkungan, umur, atau sifat fisiologi dari organisme tersebut, tetapi lebih tergantung pada kualitas DNA yang diekstraksi. Oleh karena itu, alternatif pengujian dengan metode yang modern, cepat dan akurat dapat dilakukan dengan pelatihan DNA. Salah satu metode pelatihan DNA yang dapat digunakan untuk analisis deteksi bakteri patogen dengan menggunakan teknik Polymerase Chain Reaction (PCR). Teknik ini digunakan untuk menelaah profil DNA gen 16S ribosomal Ribonucleic Acid (16S-rRNA). Penggunaan 16S-rRNA sebagai parameter sistematik molekuler universal, representatif, dan praktis untuk mengkonstruksi kekerabatan filogenetik pada tingkat spesies. Salah satu faktor penting yang mempengaruhi kualitas deteksi molekuler berbasis PCR ialah pemilihan primer yang tepat. Primer PCR merupakan oligonukleotida yang berperan sebagai inisiasi amplifikasi molekul DNA dan analisis PCR dengan primer spesifik merupakan langkah terbaik untuk kepentingan deteksi bakteri patogen karena cukup sensitif dan mudah digunakan dalam kegiatan rutin. Identifikasi isolat bakteri yang memberikan tingkat patogenisitas tertinggi dilakukan berdasarkan hasil sequencing gen 16S-rRNA. Sequencing gen 16S-rRNA terdiri atas tahapan ekstraksi DNA, amplifikasi gen 16S-rRNA dengan PCR dan sequencing dengan mesin Sequencer (Aris M et al 2013). Dengan mesin tersebut para ilmuwan masih harus menerjemahkan hasil sequencing untuk memahami bagaimana genom tersebut bekerja. Oleh karena itu, penerjemah hasil sequencing ini dibutuhkan untuk membantu kerja para ilmuwan.
2
suatu data. Salah satu metode ekstraksi ciri yang dapat digunakan untuk melakukan klasifikasi sekuen DNA adalah metode K-Mers. Berdasarkan penelitian yang telah dilakukan oleh (McHardy et al. 2007), penelitian klasifikasi terhadap 340 organisme tersebut menggunakan metode ekstraksi ciri K-Mers dan metode klasifikasi Support Vector Machine (SVM). Hasil akurasi yang didapat dari penelitian tersebut untuk panjang fragmen ≥ 5 Kilobasepair (Kbp) mencapai 90% disetiap tingkat takson.
Berdasarkan latar belakang di atas dan kelebihan SVM dibanding dengan metode pelatihan lain yaitu menggunakan strategi Structural Risk Minimization (SRM) serta berbagai studi empiris menunjukkan bahwa pendekatan SRM pada SVM memberikan error generalisasi yang lebih kecil dari pada yang diperoleh dari strategi Empirical Risk Minimization (ERM) pada Neural Network (NN) maupun metode lain (Nugroho et al. 2003). Dari penelitian ini diharapkan dapat mengelompokkan bakteri menjadi 2 jenis bakteri, yaitu bakteri patogen dan non patogen berdasarkan rantai DNA secara otomatis dengan menggunakan metode klasifikasi SVM dan K-Mers sebagai ekstraksi ciri. Dalam pelatihan SVM diguna-kan 3 fungsi kernel yang berbeda untuk mengetahui kernel yang dapat menghasil-kan model terbaik untuk pengklasifikasian jenis bakteri. Adapun data bakteri yang akan digunakan pada penelitian ini menggunakan data yang dikembangkan oleh National Center for Biotechnology Information (NCBI).
Perumusan Masalah
Adapun permasalahan yang akan menjadi bahan analisis pada penelitian ini ialah :
1 Berapa akurasi yang diperoleh dari hasil klasifikasi dengan metode SVM ? 2 Bagaimana pengaruh kernel yang digunakan terhadap hasil akurasi ?
3 Bagaimana pengaruh panjang fragmen yang digunakan terhadap hasil akurasi ? Tujuan Penelitian
Tujuan penelitian ini adalah:
1 Mengklasifikasikan bakteri berdasarkan jenisnya, yaitu patogen dan non patogen dengan membuat pemodelan Support Vector Machine (SVM) dengan ekstraksi ciri menggunakan metode K-Mers.
2 Menguji pengaruh kernel yang digunakan untuk klasifikasi terhadap hasil akurasi.
3 Menguji pengaruh panjang fragmen yang digunakan terhadap hasil akurasi. Manfaat Penelitian
3 Ruang Lingkup Penelitian
Ruang lingkup penelitian ini meliputi :
1 Data sekuens DNA terdiri atas bakteri patogen dan non patogen dengan panjang fragmen 100 bp, 400 bp, 800 bp, 1 Kbp, dan 5 Kbp.
2 Bakteri yang digunakan hanya bakteri yang patogen dan non patogen pada manusia.
3 Kernel yang digunakan yaitu Gaussian Radial Basis Function (RBF), linear kernel, dan polynomial kernel.
METODE
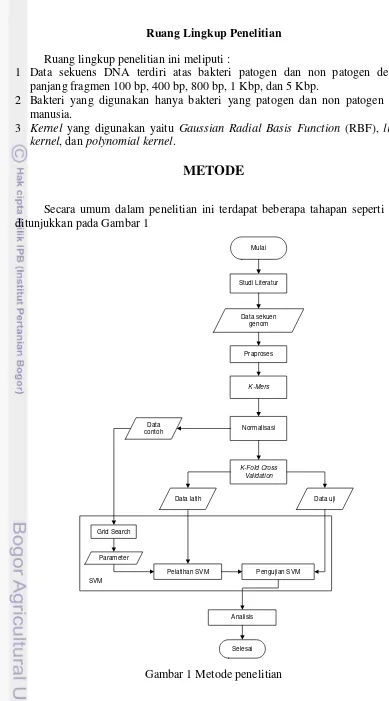
Secara umum dalam penelitian ini terdapat beberapa tahapan seperti yang ditunjukkan pada Gambar 1
Mulai
Studi Literatur
Praproses Data sekuen
genom
K-Fold Cross Validation
Data latih Data uji
K-Mers
Pelatihan SVM Pengujian SVM
Analisis
Selesai SVM
Grid Search
Parameter
Normalisasi Data
contoh
4
Studi Literatur
Pada tahap ini kegiatan yang dilakukan adalah mempelajari pustaka dan pengumpulan data yang terkait mengenai K-Mers, SVM, sekuens genom, bakteri patogen dan non patogen, identifikasi bakteri patogen secara konvensional dan molekuler. Sumber utama pada penelitian ini adalah buku, skripsi, dan jurnal.
Pengumpulan Data
Pengumpulan data diawali dengan mencari daftar spesies bakteri yang bersifat patogen dan non patogen terhadap manusia. Data yang digunakan adalah data sekuen genom pada daftar bakteri patogen dan non patogen yang dikembangkan oleh National Center for Biotechnology Information (NCBI) yang dapat diunduh pada alamat website berikut :
www.ncbi.nlm.nih.gov/genome/Bacteria/all.fna.tar,gz.
Setelah didapat data sekuen DNA dari NCBI, selanjutnya data tersebut akan diproses dengan perangkat lunak MetaSim. MetaSim merupakan perangkat lunak yang digunakan untuk mengolah data sekuen berdasarkan parameter tertentu (Richter et al. 2009). Keluaran dari MetaSim berupa fail FastA yang berisi sekuens DNA yang telah terfagmen sesuai dengan parameter yang ditelah ditentukan. Sekuen DNA tersebut terdiri atas 4 huruf yang mewakili struktur primer dari molekul DNA yaitu Adenine (A), Cytosine (C), Guanine (G), dan Timin (T).
Praproses
Praproses merupakan suatu langkah untuk menyiapkan data sesuai dengan format libSVM pada Matlab. Data pada penelitian ini merupakan data yang berbentuk serangkaian string yang sangat panjang yaitu rantai DNA. Kisaran rantai DNA mencapai 4 juta pasangan basa. Sedangkan alat sequencing saat ini masih terbatas pada panjang fragmen tertentu. Oleh karena itu, perlu dilakukan fragmentasi sekuen DNA dengan menggunakan MetaSim untuk memotong pasangan basa tersebut. Pada penelitian ini panjang fragmen yang digunakan yaitu 100 bp, 400 bp, 800 bp, 1000 bp, dan 5000 bp.
Ekstraksi Ciri K-Mers
5 Misal terdapat rantai DNA sebagai berikut : ATTAGCTACGGCATT,
ATTAGCTACGGCATT ATT
ATTAGCTACGGCATT TTA dan seterusnya sehingga K-Mers dengan K = 3 menghasilkan 64 substring dengan frekuensi sebagai berikut :
AAA: 0 AGA: 0 CAA: 0 CGA: 0 GAA: 0 GGA: 0 TAA: 0 TGA: 0
Frekuensi munculnya substring tersebut dapat digunakan sebagai penciri dari suatu kelompok string. Nilai K yang akan digunakan yaitu 3,4, dan 5.
Normalisasi
Normalisasi berfungsi untuk mengatur rentang nilai data supaya tidak terlalu jauh. Normalisasi yang digunakan menggunakan normalisasi min-max (Han J et al. 2011) dan nilai rentang yang digunakan adalah 0 sampai dengan 1 dengan rumus sebagai berikut:
vi’ =
Dimana vi’ adalah nilai yang telah dinormalisasi, vi nilai sebelum
normalisasi dilakukan, minA nilai minimum dari nilai data keseluruhan, sedangkan
maxA merupakan nilai maximum dari nilai data keseluruhan.
Pembagian Data
Untuk pembagian data uji dan data latih digunakan metode K-fold cross validation. Pada pelatihan ini data sample dibagi menjadi beberapa subsample. Saat proses pelatihan setiap 1 subsample dijadikan data uji dan subsample yang lain sebagai data latih. Penentuan subsample ini berdasarkan nilai K yaitu 5, seperti yang diilustrasikan pada Tabel 1.
Tabel 1 Ilustrasi 5-fold cross validation
Data Uji Data Latih Akurasi
Subsample_1 Subsample_2, Subsample_3, Subsample_4, Subsample_5 Akurasi_1
Subsample_2 Subsample_1, Subsample_3, Subsample_4, Subsample_5 Akurasi_2
Subsample_3 Subsample_1, Subsample_2, Subsample_4, Subsample_5 Akurasi_3
Subsample_4 Subsample_1, Subsample_2, Subsample_3, Subsample_5 Akurasi_4
Subsample_5 Subsample_1, Subsample_2, Subsample_3, Subsample_4 Akurasi_5
Dengan melalui 5-fold cross validation akan menghasilkan perbandingan data latih : data uji sebesar 80% : 20%.
6
Grid Search
Pada SVM pemilihan parameter kernel sangat berpengaruh terhadap akurasi. Pemilihan parameter kernel dapat dengan cara melakukan percobaan menggunakan parameter kernel yang berbeda-beda. Salah satu metode percobaan untuk mencari parameter kernel terbaik adalah grid search.
Metode grid search adalah proses nestedloop (proses looping di dalam looping) yang digunakan untuk mencoba berbagai nilai parameter pada pelatihan dengan sekali menjalankan program. Proses grid search pada penelitian ini menggunakan data contoh sebanyak 10% dari jumlah fragmen keselurruhan. Pengambilan data 10% ini dilakukan mengacu pada penelitian yang telah dilakukan oleh (McHardy et al. 2007). Berbagai parameter yang telah dicobakan tersebut akan dihasilkan nilai parameter terbaik yaitu parameter yang menghasilkan akurasi tertinggi pada data contoh. Dari parameter terbaik pada data contoh diharapkan menghasilkan optimum margin pada proses pelatihan SVM pada data latih.
Pada pelatihan SVM grid search dilakukan untuk menentukan parameter cost (C) , gamma ( )untuk kernel RBF serta C, gamma ( ), degree (d), dan koef 0 (r) untuk parameter pada kernelPolynomial. Namun, pada penelitian ini, nilai r menggunakan nilai default yaitu 0.
Pelatihan SVM
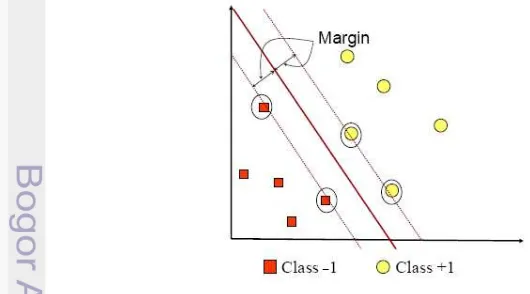
Proses pelatihan SVM dilakukan untuk data latih hasil ekstraksi ciri. Support Vector Machine (SVM) adalah metode untuk menemukan hyperplane (bidang pemisah) terbaik yang dapat memisahkan dimensi data dengan sempurna ke dalam 2 kelas (kelas +1 dan kelas -1) sehingga memperoleh margin yang maksimal antara ruang input bukan linear dengan ruang ciri menggunakan kernel. Sehingga dari label data penelitian ini yaitu kelas 1 dan kelas 2 akan berubah secara otomatis menjadi kelas +1 dan kelas -1.
Gambar 3 Support Vector Machine oleh Nugroho. et al (2003)
7 diselesaikan dengan berbagai teknik komputasi, diantaranya Lagrange Multiplier. Himpunan {x1,x2,...,xn} adalah dataset dan yi {+1, -1} adalah label kelas dari
Fungsi keputusan untuk menentukan kelas dari data uji x adalah :
dengan = koefisien Lagrange multiplier, yang bernilai nol atau positif ( .
Salah satu kendala dalam pengklasifikasian ialah ketersediaan data yang besar dan beragam yang dapat mengakibatkan data tersebut tidak dapat dipisahkan secara linear. Untuk itu, SVM menawarkan kernel yang dapat merepresentasikan atau mentransformasikan data ke dimensi lebih tinggi (lebih besar dari 2) dengan fungsi transformasi . Sehingga, data yang sudah berada di dimensi lebih tinggi tersebut dapat dengan mudah dipisahkan dengan hyperplane secara linear (Boswell 2002). Jika terdapat sebuah fungsi kernel K, maka fungsi transformasi tidak perlu diketahui secara tepat. Sehingga fungsi yang dihasilkan dari pelatihan adalah :
Beberapa fungsi kernel yang umum digunakan (Boswell 2002): 1. Linear Kernel :
K(xi,x) = . X.
2. Polynomial Kernel : K(xi,x) = ( . x+r)d
3. Radial Basic Function Kernel : K(xi,x) = exp( )
Pengujian
Dari proses K-fold cross validation menghasilkan sebuah data uji, data uji ini akan digunakan untuk menguji model yang dihasilkan oleh proses pelatihan. Karena fold yang digunakan pada penelitian ini adalah 5, makan data uji yang dihasilkan sebanyak 20% dari jumlah fragmen keseluruhan yaitu 2000 fragmen.
Proses pengujian semua organisme pada data uji menghasilkan prediksi kelas pengklasifikasian. Dari prediksi tersebut dihitung persentase keberhasilan pengklasifikasian yang menggambarkan seberapa akurat model yang didapatkan dari sebuah metode (akurasi).
8
Akurasi =
x100%
Akurasi Akhir = ; n=5
Analisis
Analisis ini dilakukan untuk mengetahui model SVM yang terbaik berdasarkan kernel, panjang fragmen dan nilai K pada proses ekstraksi ciri yang digunakan dengan membandingkan hasil akurasi yang telah didapat dari proses pengujian. Tabel 2 adalah catatan pembanding pada tiap parameter yang diteliti.
Tabel 2 Pembanding parameter Parameter Pembanding
Kernel Panjang fragmen
Panjang Fragmen Nilai K pada K-Mers
Nilai K pada K-Mers Kernel
HASIL DAN PEMBAHASAN
Pengumpulan Data
Data yang berupa sekuen DNA dalam format data FastA yang telah didapat dari NCBI dibuka dengan menggunakan MetaSim dan diambil 40 organisme bakteri yang terdiri atas 20 organisme yang bersifat patogenesis dan 20 organisme yang tidak bersifat patogenesis. Nama organisme tersebut didapat dari beberapa literatur mengenai mikrobiologi farmasi, patogenesis, dan mikrobiologi industri. Adapun daftar organisme bakteri terdapat pada Lampiran 1.
Selain data sekuen DNA pada penelitian ini diperlukan data kelas dari setiap organism, sehingga dibuat data kelas yang terdiri atas 2 kelas yaitu kelas 1 adalah kelas patogen dan kelas 2 merupakan kelas non patogen. Pembuatan data kelas ini dilakukan secara manual dengan menggunakan Ms. Excel.
Praproses
Sebelum data sekuen DNA diklasifikasikan, terlebih dahulu data sekuen diuraikan fragmennya menggunakan MetaSim. Proses penguraian fragmen ini dilakukan untuk mengambil sample dari rantai DNA suatu organisme. Panjang fragmen tiap organisme yang digunakan adalah 100 bp, 400 bp, 800 bp, 1000 bp, dan 5000 bp dengan masing-masing organisme diambil sample sebanyak 250 kali. Sehingga keluaran MetaSim dalam 1 fail mempunyai panjang fragmen yang sama menghasilkan 250 rantai DNA organisme berupa format FastA.
9 yang masing-masing fail terdiri atas 10000 organisme. Jumlah sample dapat dilihat pada Lampiran 1.
Ekstraksi Ciri
Ekstraksi ciri menggunakan K-Mers dengan nilai K = 3,4, dan 5. Proses ekstraksi ciri menghasilkan banyaknya pasangan 3-nukleotida, 4-nukleotida, dan 5-nukleotida, bergantung pada nilai K yang digunakan. Untuk K=3 menghasilkan 43 atau 64 pasangan 3-nukleotida yang terdiri atas AAA sampai GGG, sedangkan K = 4 menghasilkan 44 atau 256 pasangan 4-nukleotida yang terdiri atas AAAA sampai GGGG, dan K = 5 menghasilkan 45 atau 1024 pasangan 5-nukleotida yang terdiri atas AAAAA sampai GGGGG. Gambar 2 di bawah ini merupakan contoh hasil ekstraksi ciri K = 3 pada fragmen 100 bp :
Gambar 4 Contoh hasil ekstraksi ciri K-Mers dengan nilai K = 3 pada fragmen 100 bp
Normalisasi
Proses normalisasi menggunakan data keluaran dari ekstraksi ciri. Dan dari proses normalisasi ini menghasilkan struktur angka double dengan rentang 0 sampai dengan 1 sebanyak matriks dari hasil ekstraksi ciri, yaitu untuk 3-Mers matriks yang dihasilkan 10000 x 64, untuk 4-Mers matriks yang dihasilkan 10000 x 256, dan 10000 x 1024 untuk 5-Mers. Berikut Gambar 5 adalah hasil normalisasi dengan Persamaan 1 dan nilai 3-Mers pada fragmen 100 bp :
Gambar 5 Normalisasi dengan nilai 3-Mers pada fragmen 100 bp
Normalisasi dilakukan untuk menghindari fitur yang bernilai tinggi sehingga dapat mempermudah perhitungan selama proses pengklasifikasian.
K-Fold Cross Validation
10
Grid Search
Proses grid search menghasilkan parameter terbaik untuk kernel RBF dan Polynomial. Dari proses grid search menghasilkan salah satu parameter terbaik untuk cost = 0.5 dan gamma = 8 dengan kernel RBF dan fragmen 100 bp dan 3-Mers, sedangkan dengan kernelPolynomial dengan data yang sama menghasilkan parameter terbaik untuk cost = 0.03 gamma = 8 dan degree = 3. Adapun hasil grid search yang lain dapat dilihat pada Lampiran 2.
Pelatihan SVM
Pelatihan SVM dilakukan pada semua panjang fragmen dan K-Mers yang telah ditentukan dan dinormalisasi dengan menggunakan parameter terbaik yang dihasilkan pada proses grid search pada masing-masing data dan kernel. Data yang digunakan sebagai data latih terdiri atas 8000 data dan 2000 data uji. Pembagian data tersebut diperoleh dari proses k-fold cross validation. Dari pelatihan ini akan menghasilkan pemodelannya.
Pada setiap kernel dilakukan sebanyak 15 pelatihan dengan panjang fragmen dan nilai K-Mers yang telah ditentukan. Pelatihan ini menggunakan 3 kernel yaitu RBF, Polynomial, dan Linear, sehingga total pelatihan yang dilakukan sebanyak 45 pelatihan.
Pengujian SVM
Pengujian SVM dilakukan untuk menghitung akurasi setiap percobaan. Data yang digunakan dalam pengujian menggunakan 2000 fragmen. Dari proses pelatihan sebelumnya menghasilkan model yang dapat digunakan untuk mengklasifikasikan data uji dan mendapatkan akurasi dengan menggunakan Persamaan 2 dan Persamaan 3.
Analisis
Analisis dilakukan berdasarkan akurasi yang telah didapatkan dari pelatihan dan pengujian SVM.
Panjang Fragmen dan Kernel
Akurasi dihitung dengan percobaan pada panjang fragmen yang berbeda, yaitu: 100 bp, 400 bp, 800 bp, 1 Kbp, dan 5 Kbp. Hasil akurasi dengan 3-Mers ditunjukkan pada Tabel 3. Adapun hasil akurasi yang lain dapat dilihat pada Lampiran 3 dan Lampiran 4.
11
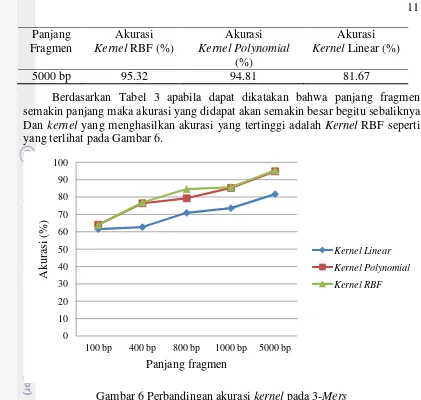
Berdasarkan Tabel 3 apabila dapat dikatakan bahwa panjang fragmen semakin panjang maka akurasi yang didapat akan semakin besar begitu sebaliknya. Dan kernel yang menghasilkan akurasi yang tertinggi adalah Kernel RBF seperti yang terlihat pada Gambar 6.
Gambar 6 Perbandingan akurasi kernel pada 3-Mers
Dari Gambar 6 terlihat bahwa selisih antara RBF dengan Polynomial hanya sedikit, tetapi selisih antara RBF dengan Linear dan Polynomial dengan Linear cukup besar berkisar antara 2.35% - 14.13% atau 47 - 283 fragmen dari 2000 fragmen. Hal ini menunjukkan bahwa data tersebut merupakan data non linear separable. Data non linear separable adalah data yang tidak dapat terpisah secara sempurna oleh hyperplane.
Nilai K pada K-Mers
Nilai K pada kernel RBF tidak berpengaruh besar. Untuk panjang fragmen 100 bp sampai dengan 1000 bp K= 3 ke K= 4 akurasi meningkat antara 0.1% hingga 2.96% dan dari K = 4 ke K = 5 akurasi menurun. Hal ini terjadi karena pada panjang fragmen tersebut dengan 5-Mers menghasilkan banyak nilai 0 untuk hasil cirinya tetapi untuk panjang fragmen 5000 bp semakin tinggi nilai K yang digunakan akurasi semakin meningkat pula seperti ditunjukkan pada Gambar 6. Pada kasus ini hasil K-Mers serupa dengan hasil penelitian oleh Mc Hardy et al. (2007), yaitu pada panjang fragmen ≤ 5000 bp akurasi yang diperoleh sangat kecil.
Dari Gambar 6 membuktikan bahwa K = 5 dengan panjang fragmen 5000 bp menghasilkan akurasi tertinggi hingga mencapai 96.61%. Untuk hasil yang lainnya dapat dilihat pada Lampiran 5 dan Lampiran 6.
12
Gambar 7 Pengaruh nilai K terhadap akurasi pada kernel RBF
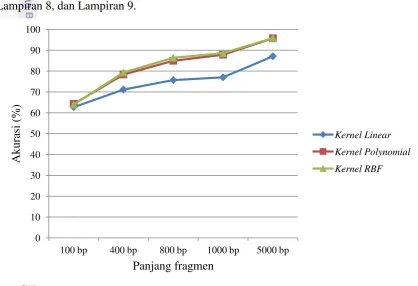
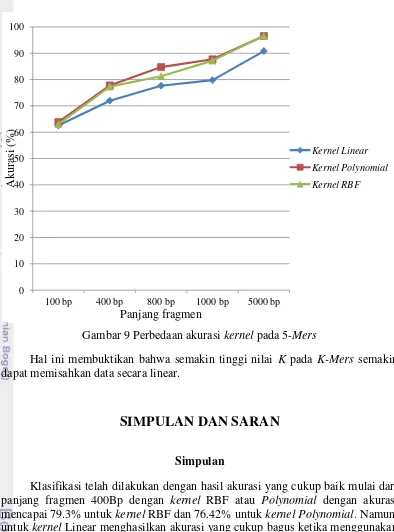
Kemudian pada 4-Mers dan 5-Mers selisih antara kernel linear dan non linear semakin berkurang yaitu berkisar antara 1.25% hingga 11.56% untuk 4-Mers dan 0.53% hingga 7.93% untuk 5-Mers, seperti yang ditunjukkan pada Gambar 8 dan Gambar 9. Adapun rincian selisih dapat dilihat pada Lampiran 7, Lampiran 8, dan Lampiran 9.
13
Gambar 9 Perbedaan akurasi kernel pada 5-Mers
Hal ini membuktikan bahwa semakin tinggi nilai K pada K-Mers semakin dapat memisahkan data secara linear.
SIMPULAN DAN SARAN
Simpulan
Klasifikasi telah dilakukan dengan hasil akurasi yang cukup baik mulai dari panjang fragmen 400Bp dengan kernel RBF atau Polynomial dengan akurasi mencapai 79.3% untuk kernel RBF dan 76.42% untuk kernelPolynomial. Namun, untuk kernel Linear menghasilkan akurasi yang cukup bagus ketika menggunakan panjang fragmen 800 bp dengan hasil akurasi mencapai 70.91%.
Penggunaan nilai K pada proses ekstraksi ciri menunjukkan bahwa nilai tertinggi pada kasus ini berada pada K = 5 dengan akurasi mencapai 96.61% pada panjang fragmen 5000 bp. Dan semakin besar nilai K maka data semakin mudah dipisahkan secara linear.
Pengaruh pemilihan kernel terhadap hasil akurasi cukup tinggi yaitu mencapai 14.13% yang menandakan bahwa data yang ada bersifat non linear separable, karena hasil dari menggunakan kernel Linear menunjukkan akurasi yang lebih rendah dari pada menggunakan kernel RBF maupun Polynomial. Dan pada kasus ini kernel terbaik adalah kernel RBF.
0 10 20 30 40 50 60 70 80 90 100
100 bp 400 bp 800 bp 1000 bp 5000 bp
Akur
asi (%
)
Panjang fragmen
Kernel Linear
Kernel Polynomial
14
Saran
Saran untuk penelitian selanjutnya adalah sebagai berikut :
1 Melakukan optimasi SVM dengan Metode Algoritma Genetika sehingga diharapkan dapat meningkatkan akurasi.
2 Melakukan ektraksi ciri dengan Space K-Mers.
DAFTAR PUSTAKA
Aris M, et al. 2013. Identifikasi molekuler bakteri patogen dan desain primer PCR. Budidaya Perairan 1: 43-50.
Boswell D. 2002. Introduction to support vector machine [Internet]. [diunduh 2014 Juni 26]. Tersedia pada: http://www.work.caltech.edu/~boswell/ IntroToSVM.pdf
Choi JH, Cho HG. 2002. Analysis of common k-mers for whole genome sequence using SSB-tree. Genome Information. 13 : 30-41
de Carvalho Junior SA. 2003. Sequence Alignment Algorithms. London. King’s College.
Han J, Kamber M, Pei J. 2011. Data Mining Concept and Techniques Third Edition. USA : Morgan Kaufmann. Hlm 113-115.
Harahap LH. 2013. Mengenal Bakteri [Internet]. [diunduh 2014 November 26]. Tersedia pada :
http://bbkpbelawan.deptan.go.id/wpcontent/uploads/2013/02/MENGENAL%2 0BAKTERI.pdf
Hidayat N, Masdiana CP, Suhartini S. 2006. Mikrobiologi Industri. Yogyakarta : Andi Offset.
Louws FJ, Cuppels DA. 2001. Appendix. Molecular techniques. Di dalam : Schaad NW. et al., editor. Laboratory Guide for Identification of PF Plant Pathogenic Bacteria. Third Edition. APS Press. St. Paul Minnesota. Hal 321-337
McHardy AC, et al. 2007. Accurate phylogonetic classification of variable-lenghth DNA fragment. Nature Methods, 4(1):63-72, doi:10.1038/nmeth976. Mims, Cedric A. 1987. The Pathogenesis of Infection Diasese. London (GB) :
Academic Press.
Nugroho AS, Wirarto AB, Handoko D. 2003. Support Vector Machine[Internet].[diunduh 2014 Juni 21]. Tersedia pada : http: //www.komputer.com
Pratiwi ST, (editor) Astikawati R, Safitri A. 2008. Mikrobiologi Farmasi. Jakarta : Erlangga.
Richter DC, et al. 2009. User manual for MetaSim V0.9.5 [Internet]. [diunduh 2014 Juni 5]. Tersedia pada:
15 Lampiran 1 Nama organisme
No Nama Bakteri Kelas No Nama Bakteri Kelas 1 Bacillus anthracis Patogen 21 Bacillus subtilis Non Patogen 2 Bartonella
bacilliformis
Patogen 22 Bifidobacterium animals
Non Patogen
3 Bordetella pertussis
Patogen 23 Bifidobacterium bifidum
Non Patogen
4 Borrelia recurrentis
Patogen 24 Bifidobacterium breve
Non Patogen
5 Brucella abortus Patogen 25 Bifidobacterium adolescentis
Non Patogen
6 Corynebacterium diphteriae
Patogen 26 Bifidobacterium longum
Non Patogen
7 Escherichia coli Patogen 27 Corynebacterium phage P1201
Non Patogen
8 Haemophilus influenzae
Patogen 28 Enterococcus faecium
Non Patogen
9 Haemophilus ducreyi
Patogen 29 Lactobacillus delbrueckii
Non Patogen
10 Legionella pneumophilla
Patogen 30 Lactobacillus acidophilus
Non Patogen
11 Listeria
monocytogenes
Patogen 31 Lactobacillus brevis Non Patogen
12 Mycobacterium tuberculosis
Patogen 32 Lactobacillus casei tp bkn yg str
Non Patogen
13 Mycobacterium leprae
Patogen 33 Lactobacillus fermentum
Non Patogen
14 Neisseria gonorrhoeae
Patogen 34 Lactobacillus reuteri
Non Patogen
15 Shigella flexneri Patogen 35 Lactobacillus plantarum
Non Patogen
16 Streptococcus salivarius
Patogen 36 Lactobacillus helveticus
Non Patogen
17 Streptococcus pyogenes
Patogen 37 Leuconostoc mesenteroides
Non Patogen
18 Streptococcus mutans
Patogen 38 Pediococcus pentosaceus
Non Patogen
19 Streptococcus agalactiae
Patogen 39 Propionibacterium freudenreichii subps shermanii
Non Patogen
20 Pseudomonas aeruginosa
Patogen 40 Streptococcus thermophillus
Non Patogen
*Setiap bakteri difragmen sepanjang 100 Bp, 400 Bp, 800 Bp, 1 Kbp, dan 5 Kbp dengan masing-masing panjang fragmen diambil sebanyak 250 kali.
16
Lampiran 2 Hasil proses grid search
Kernel RBF Kernel Polynomial
panjang
Lampiran 3 Akurasi pada tiap panjang fragmen dan kernel pada 4-Mers Panjang
17 Lampiran 5 Pengaruh nilai K terhadap akurasi pada kernelPolynomial
18
Lampiran 7 Selisih akurasi kernel pada 3-Mers Panjang
Lampiran 8 Selisih akurasi kernel pada 4-Mers Panjang
19