PEMETAAN QUANTITATIVE TRAIT LOCI (QTL) SIFAT
PERTUMBUHAN PADA POPULASI DOMBA SILANG BALIK
EKOR TIPIS DAN MERINO
ENDANG TRI MARGAWATI
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI DISERTASI DAN
SUMBER INFORMASI
Dengan ini saya menyatakan bahwa disertasi berjudul Pemetaan Quantitative Trait Loci (QTL) Sifat Produksi pada Populasi Domba Silang Balik Ekor Tipis dan Merino adalah karya saya sendiri dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir disertasi ini.
Bogor, September 2005
Endang Tri Margawati
NIM 04600013
ABSTRAK
ENDANG TRI MARGAWATI. Pemetaan Quantitative Trait Loci (QTL) Sifat Pertumbuhan pada Populasi Domba Silang Balik Ekor Tipis dan Merino. Dibimbing oleh HARIMURTI MARTOJO, MULADNO, SUBANDRIYO dan HERMAN W.H. RAADSMA
Penelitian dimaksudkan untuk mengestimasikan lokasi quantitative trait loci
(QTL) dan gen kandidat sifat pertumbuhan pada domba. Sifat pertumbuhan yang diteliti yaitu berat lahir (BL), berat umur 90 (BB90), umur 180 (BB180), umur 270
(BB270) dan umur 360 (BB360) pada populasi domba. Empat keluarga acuan half-sib
telah dirancang yang terdiri dari 381 ekor. Sebagain besar populasi didominasi oleh domba silang balik dari persilangan domba F1 jantan (persilangan domba Ekor Tipis Indonesia dan Merino) kemudian disilang balikkan dengan tetuanya (sejumlah Merino betina) dan sebagian populasi adalah F2. Penelitian ini memanfaatkan perbedaan penampilan berat badan antara domba ekor tipis dan Merino. Sebanyak 136 penciri mikrosatelit telah digunakan dalam perunutan genom yang mencakup 26 pasang kromosom autosomal. Analisis QTL dilakukan secara elektronik dengan perangkat lunak program QTL Express. Lokasi QTL, jarak QTL dan penciri apit (flanking markers) pada kromosom diperoleh dari basic analysis. Sedangkan
significance threshold dilakukan dengan uji Permutate Experiment Wide dan
Permutate Chromosome Wide pada taraf 5% (p<0.05) dan 1% (p<0.01). Identifikasi gen kandidat diperoleh dengan akses GenBank-NCBI.
Hasil penelitian mengindikasikan keberadaan 15 lokasi QTL untuk sifat pertumbuhan yang dipelajari. Tiga dari 15 lokasi QTL menunjukan keberadaan QTL pada kromosom yang kuat (p<0.01), yaitu kromosom 5 untuk BL pada 112cM (88,8-129,2cM), kromosom 7 untuk BB90 pada 8cM (0-46,9cM), untuk BB270 pada 76cM
(46,9-105,8cM) dan untuk BB360 pada 80cM (46,9-105,8cM), dan kromosom 18
untuk BB180 pada 96cM (106,4-123,9cM), untuk BB270 dan BB360 pada 104cM
(106,4-123,9cM). Lokasi QTL pada kromosom 18 tetap nyata (p<0.01) untuk BB360
dan juga untuk BB270 (p<0.05) mempertahankan pengaruhnya setelah uji experiment wide. Lokasi QTL pada kromsom 18 adalah terletak homolog dengan kromosom14 manusia pada rentangan 98,509 – 105,138 Mb. Sedangkan dua penciri pengapit (flanking markers) gen kandidat terletak pada kromosom 18 yaitu CSSM018 dan TMR1 atau AKT1. Gen kandidat sifat pertumbuhan yang teridentifikasi pada kromosom 5 yaitu gen CAST (Calpastatin), pada kromosom 7 adalah CAPN3
(Calpain3) dan SSTR1 (Somatostatin receptor1). Sementara pada kromosom 18, meskipun terdapat pengaruh kuat QTL untuk sifat pertumbuhan pada kromosom 18 namun belum ditemukan gen kandidatnya.
ABSTRACT
ENDANG TRI MARGAWATI. Quantitative Trait Loci (QTL) Mapping for Growth Traits in the Indonesian Thin Tail and Merino Backcross Sheep Populations). Under Supervision of HARIMURTI MARTOJO, MULADNO, SUBANDRIYO and HERMAN W.H. RAADSMA
This study was performed to map Quantitative Trait Loci (QTL) locations of growth traits and candidate genes. Traits considered were weights at birth, 90, 180, 270 and 360 days of age (BW, W90, W180, W270 and W360) in Indonesian Thin Tail (ITT) and
Merino sheep populations. Four half-sib reference families were designed to establish 381 heads of sheep populations. This population consisted predominantly of backcross progeny of Indonesian Thin Tail (ITT) rams crossed to Merino ewes then F1 sires were backcrossed to a number of Merino ewes and also including some F2 progeny. The study exploited differences in weight performance between Merino and ITT sheep. A total of 136 informative microsatellite markers were used in a genome-wide scan covering the 26 autosomal sheep chromosomes. QTL analysis was conducted online using the QTL Express. QTL locations, distances and flanking markers were obtained from a basic analysis. Permutate-experiment wide and permutate-chromosome wide analysis were used to analyze significance threshold differences at levels of 5% and 1%. Identification of candidate genes was obtained by accessing to GenBank-NCBI.
The study indicated the existence of 15 QTL locations for growth traits by a chromosome wide analysis while 3 out of 15 QTL locations showed a strong support (p• 0.01) of QTL locations: on chromosome 5 for BWat 112cM (88.8-129.2cM), on chromosome 7 for W90 at 8cM (0-46.9cM), for W270 at 76cM (46.9-105.8cM) and for
W360 at 80cM (46.9-105.8cM), and on chromosome 18 for W180 at 96cM
(106.4-123.9cM), for W270 and W360 at 104cM (106.4-123.9cM). Only the growth trait of
W360 retained strong support (p• 0.01) and W270 was retained less strong (p• 0.05)
QTL locations on chromosome 18 under experiment-wide significance testing. By investigating homologous human chromosomal segments, this QTL region on chromosome 18 was homologous to human chromosome 14 and resided on chromosome segment between 98.509 – 105.138 Mb and flanked by CSSM018 and TMR1/AKT1 markers. CAST (Calpastatin) and CAPN3 (Calpain3), SSTR1
(Somatostatin receptor1) genes were identified on sheep chromosome 5, and 7, respectively. While on chromosome 18, a candidate gene (s) associated with growth traits could not be shown yet. However, the effect of QTL for growth traits showed strongly on chromosome 18.
PEMETAAN QUANTITATIVE TRAIT LOCI (QTL) SIFAT
PERTUMBUHAN PADA POPULASI DOMBA SILANG BALIK
EKOR TIPIS DAN MERINO
ENDANG TRI MARGAWATI
Disertasi
Sebagai salah satu syarat untuk memperoleh gelar Doktor pada
Program Studi Ilmu Ternak
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
Judul Disertasi: Pemetaan Quantitative Trait Loci (QTL) Sifat Pertumbuhan pada Populasi Domba Silang Balik Ekor Tipis dan Merino
Nama : Endang Tri Margawati NIM : P04600013
Disetujui
Komisi Pembimbing
Prof. Dr. Harimurti Martojo, M.Sc. Dr. Ir. Muladno, MSA. Ketua Anggota
Dr. Ir. Subandriyo, M.Sc. Prof. Dr. Herman W. Raadsma Anggota Anggota
Diketahui
Ketua Program Studi Ilmu Ternak Dekan Sekolah Pascasarjana
Dr. Ir. Nahrowi, M.Sc. Prof. Dr. Ir. Syafrida Manuwoto, M.Sc.
PRAKATA
Puji dan syukur penulis panjatkan kepada Tuhan Yang Maha Esa atas segala karuniaNya sehingga karya ilmiah ini berhasil diselesaikan. Judul karya ilmiah yang dipilih dalam penelitian ini yaitu Pemetaan Quantitative Trait Loci (QTL) Sifat Produksi pada Populasi Domba Silang Balik Ekor Tipis dan Merino. Penelitian telah dimulai dari tahun 1999 sampai dengan 2004 untuk koleksi data kuantitatif pertumbuhan dan analisis molekuler.
Penulis mengucapkan terima kasih kepada Bapak Professor Dr. Harimurti Martojo, Dr. Muladno, Dr. Subandriyo dan Professor Dr. Herman W. Raadsma (Sydney University, Australia) yang telah memberi bimbingan dan pengarahan selama penyelesaian studi doktoral di Sekolah Pascasarjana, Intstitut Pertanian Bogor. Perhargaan juga disampaikan kepada Dr. Karen Fullard peneliti bidang genetika molekuler dari the Sydney University atas bimbingan dalam analisis QTL dan diskusi yang bermanfaat dalam penyelesaian karya ilmiah ini. Pekerjaan teknis di laboratorium tidak mungkin terselesaikan tanpa bantuan dari Mrs. Gina Attard dan Mrs. Marilyn Jones dari the Sydney University, Indriawati, S.Si. dari Puslit Bioteknologi – LIPI. Pekerjaan koleksi data di lapang juga tidak mungkin tertangani tanpa bantuan Sdr. Handrie, Agus Istiarto, Nugroho dan beberapa rekan di lapangan yang tidak mungkin penulis sebutkan satu persatu baik dari Puslit Bioteknologi – LIPI maupun dari Puslitbang Peternakan, Bogor yang telah membantu dalam koleksi data kuantitatif maupun koleksi darah untuk ekstraksi DNA. Ungkapan terima kasih juga disampaikan kepada para pimpinan baik di lingkungan Puslit Bioteknologi- LIPI, Cibinong, LIPI-Jakarta maupun ACIAR (the Australian Centre for International Agricultural Research) project (Dr. John Copland - Research Program Manager of the Animal Science I, Prof. Dr. Herman W. Raadsma - Australian research leader, Dr. Kusuma Diwyanto - Indonesian research leader) yang telah memberi kesempatan dan ijin melakukan penelitian bersamaan dengan ACIAR project (AS1 97/27) pada Genetic and Immunological Characterisation of High Resistance to Internal Parasites in Indonesian Thin Tail.
Semoga karya ilmiah ini memberi wawasan baru dan manfaat di bidang Ilmu Genetika Ternak modern di Indonesia.
Bogor, September 2005
RIWAYAT HIDUP
Penulis dilahirkan di Semarang, pada 30 Juni 1955 sebagai anak ke tiga dari tujuh bersaudara dari pasangan Bapak Mintardjo (Alm) dan Ibu Soentari (Almh). Pendidikan Sarjana Peternakan (Ir.) ditempuh pada Fakultas Peternakan dan Perikanan, Jurusan Peternakan, Universitas Diponegoro, Semarang, lulus tahun 1981. Tahun 1993, penulis melanjutkan studi S2 pada Department of Animal Science, the Faculty of Agriculture Science of the Massey University di Palmerston North, New Zealand dan memperoleh gelar M.Agr.Sc. pada tahun 1995. Pada 2001, penulis melanjutkan studi pascasarjana S3 pada program studi Ilmu Ternak, Institut Pertanian Bogor (IPB). Beasiswa diperoleh sebagian dari Lembaga Ilmu Pengetahuan Indonesia (LIPI) dan bantuan penelitian dari ACIAR.
Awalnya penulis bekerja sebagai peneliti di Lembaga Biologi Nasional (LBN)-LIPI di Bogor sejak tahun 1982 sampai 1986. Adanya pengembangan LBN tahun 1986, penulis berpindah dan bergabung dengan Pusat Penelitian Bioteknologi – LIPI pada bidang Reproduksi dan Genetika Ternak sampai sekarang. Tahun 1999 dan 2000, penulis mendapat pelatihan pada DNA Technology di Sydney University, Australia, sebagai bagian dari penelitian kerjasama ACIAR project: Indonesia dan Australia. Bersamaan melakukan penelitian ACIAR project, penulis melakukan riset untuk penyusunan disertasi. Sebagian besar analisis molekuler (genotyping) dilakukan di Sydney University, Australia. Penulis memperoleh pelatihan Analisis QTL pada Oktober 2004 dari Dr. Karen Fullard (Moleculer Geneticist) dari the Sydney University, Australia.
Topik disertasi yang diambil adalah Pemetaan Quantitative Trait Loci (QTL) Sifat Pertumbuhan pada Populasi Domba Silang Balik Ekor Tipis dan Merino. Selama studi pascasarjana S3, beberapa karya ilmiah telah dipresentasikan, yaitu The application of microsatellite markers for Quantitative Trait Loci (QTL) mapping of production traits in sheep, pada the 3rd Conference of Science Council of ASIA (SCA) in May 2003 di Denpasar, Bali (1); Analisis segregasi karakter berat lahir pada anak domba silang balik (Merino X Ekor Tipis X Merino) pada Simposium PERIPI (Perhimpunan Ilmu Pemuliabiakan Indonesia) di Bogor, Agustus 2004 (2); Analysis of candidate major gene for pre-weaning growth traits in sheep pada the 3rd Indonesian Biotechnology Conference (IBC) di Denpasar, Bali pada Desember 2004 (3). Satu karya ilmiah Quantitative Trait Loci (QTL) Analysis for Production Traits of Birth Weight and Weight 360 days in Backcross Sheep (Indonesian Thin Tail x Merino x Merino) telah dikirim ke Jurnal ilmiah Hayati pada Maret 2005 (4).
DAFTAR ISI
Quantitative Trait Loci (QTL)……… 5Pengertian QTL ………...…. 5
Sifat Kuantitatif……….…. 6
Gen Mayor………..….. 7
Deteksi QTL……….. 8
Prinsip Pemetaan QTL ………...… 9
Pendekatan Studi Pemetaan QTL……….………... 10
Rancangan Hewan Percobaan………... 12
Bangsa Domba……….………... 15
Analisis Keterpautan (Linkage Analysis)……….... 25
Studi Pemetaan QTL Sifat Produksi Domba………..….... 26
Aplikasi Teknologi Penciri Genetik………..…..… 27
Linked dan Direct Markers………..…..… 27
Marker-assisted selection (MAS)……….. 30
Aplikasi Studi QTL pada Kemajuan Pemuliaan……….……… 31
Bahan……….. 32
DNA Genom……….. 32
Keluarga Acuan dan Pembentukan Populasi Progeny……… 32
Penciri Mikrosatelit……… 34
Metode……… 35
Polymerase Chain Reaction (PCR)……… 35
Genotyping………. 37
Koleksi Data Fenotipe.………... 38
Analisis Genetik dan Statistik……… 38
HASIL DAN PEMBAHASAN Penapisan Penciri……… ………. 43
Genotyping Progeny ………………. 44
Identifikasi GenMayor - Analisis Segregasi ……… 46
Identifikasi QTL ………... 49
Keberadaan QTL - Significance Threshold..………. 49
Lokasi QTL dan Penciri Apit (Flanking Markers) ……….. 53
Gen Kandidat pada Kromosom 5, 7 dan 18 ………. 60
Akses GenBank .. ……… 61
Deskripsi Gen ……….. 62
Gen Kandidat pada Kromosom 5 ……… 67
Gen Kandidat pada Kromosom 7 ……… 69
Gen Kandidat pada Kromosom 18 ……….. 73
SIMPULAN ……….. 78
DAFTAR PUSTAKA ……… … 79
DAFTAR TABEL
Halaman
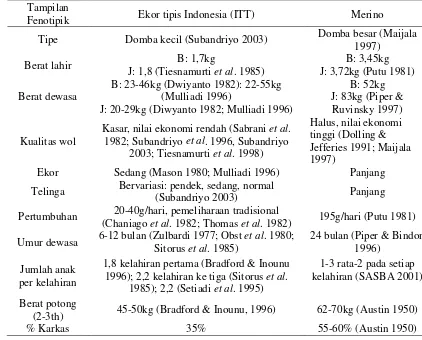
1 Tampilan fenotipik domba ekor tipis Indonesia dan domba Merino…. 17
2 Studi gen mayor dan QTL sifat produksi pada domba … …… ..……. 26
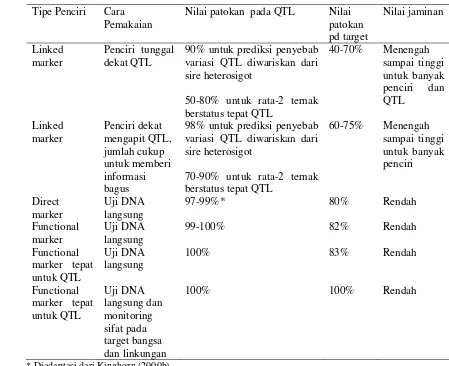
3 Daftar petunjuk penggunaan tipe penciri ……… ………… ….…….. 28
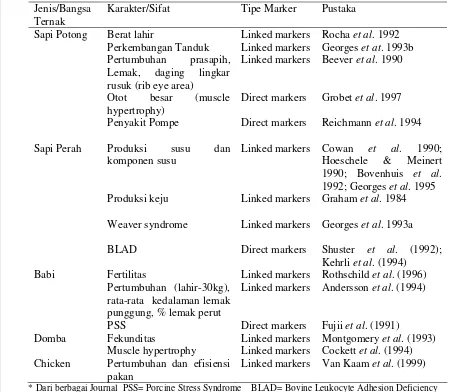
4 QTL terdeteksi pada populasi ternak …………. ……….. 29
5 Rancangan ternak percobaan ……… ………. .. 33
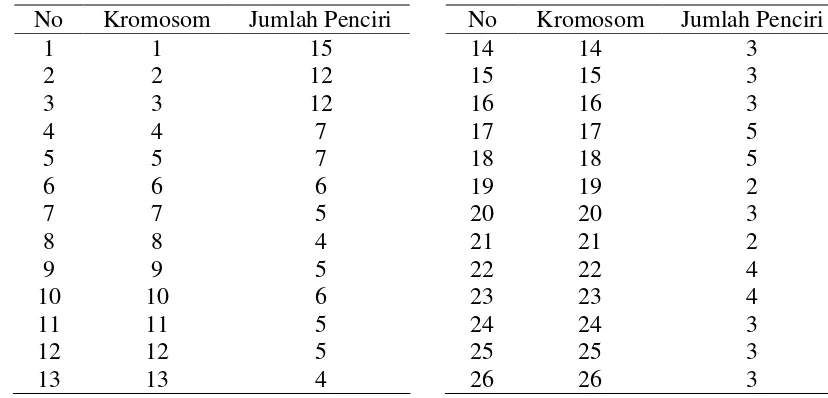
6 Penciri mikrosatelit per kromosom … ……… …. ... 34
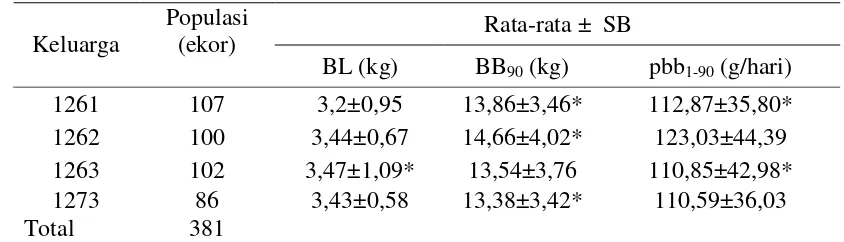
7 Rataan sifat pertumbuhan pra-sapih (BL dan BB90) pada populasi domba silang balik dari empat keluarga cuan (saudara tiri).……… … 48
8 Significance threshold berat lahir (BL)……….... .... 50
9 Significance threshold berat badan 90 (BB90)………. 51
10 Significance threshold berat badan 180 (BB180)………. 51
11 Significance threshold berat badan 180 (BB180) ………. 51
12 Significance threshold berat badan 360 (BB360)………... 51
13 Lokasi QTL dan penciri apit (Flanking Marker) sifat pertumbuhan ... 54
14 Sheep mapping website ……… ………... 62
DAFTAR GAMBAR
Halaman
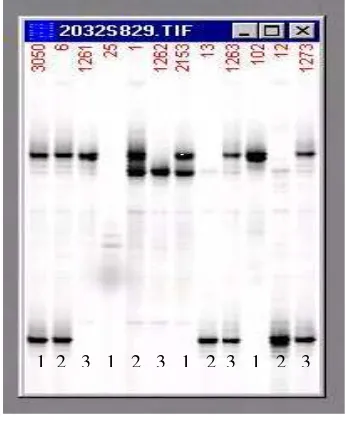
1 Hasil penapisan penciri BMS528………… ……… … 44
2 Hasil genotyping keluarga 1261………. 45
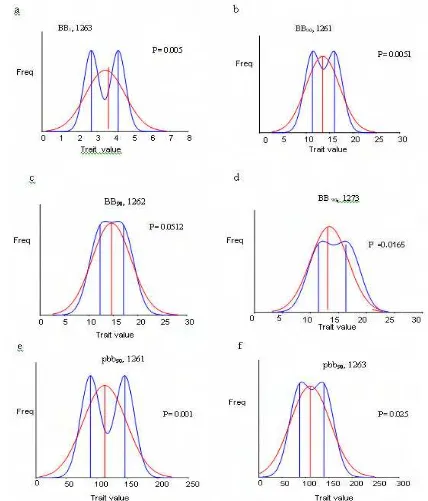
3 Kurva bi-modal ……….. 47
4 F-value kromosom 5, 7, 18 ………. 53
5 Lokasi penciri mikrosatelit pada kromosom 2………. 55
6 Lokasi penciri mikrosatelit pada kromosom 18……… 57
7 Lokasi penciri mikrosatelit pada kromosom 23……… 59
8 Lokasi gen kandidat pada kromosom 5 ……… … 63
9 Lokasi gen kandidat pada kromosom 7……… . 64
10 Lokasi gen kandidat pada kromosom 18……… .. 65
11 Ideogram lokasi gen CAST ……….. 67
12 Ideogram lokasi gen SSTR1 ……… 70
13 Ideogram lokasi gen CAPN3 ……….. 72
DAFTAR LAMPIRAN
Halaman
1 Daftar penciri per kromosom……… ………. . 94
2 Individu domba per keluarga ………. 100
3 Input data: map file …… ……… 103
4 Input data: genotype file ……….... 104
5 Input data: phenotype file ………... 105
6 Hasil uji permutasi – significance threshold... 106
7 Homologi kromosom 5 domba dengan romosom 5 Manusia ………. 107
8 Homologi kromosom 7 domba dengan kromosom 3, 14, 15 Manusia ……….. 108
9 Homologi kromosom 18 domba dengan kromosom 14 Manusia ……… ………. 116
10 Daftar gen kandidat pada segmen kromosom 5 Manusia homolog dengan kromosom 5 domba ……….. 118
11 a. Daftar gen kandidat pada segmen kromosom14 Manusia homolog dengan kromosom 7 domba……… ……… 121
b. Daftar gen kandidat pada segmen kromosom 14 dan 15 Manusia homolog dengan kromosom 7 domba………. 127
PENDAHULUAN
Latar Belakang
Populasi ternak domba di Indonesia dari tahun ke tahun mengalami
peningkatan. Data terakhir populasi domba di Indonesia pada tahun 2004, tercatat
lebih dari 8.245.000 ekor tersebar hampir di seluruh tiga puluh wilayah propinsi dan
paling banyak tersebar di Propinsi Jawa Barat (tidak termasuk Propinsi Banten), yaitu
berjumlah 3.673.812 ekor (DJBPP 2004). Hingga saat ini pengusahaan ternak domba
masih didominasi oleh peternakan rakyat dalam skala kecil dengan sistem
pemeliharaan sederhana atau tradisional. Pemeliharaan domba tersebut pada
umumnya ditujukan untuk produksi daging dan sebagian lainnya digunakan sebagai
tabungan untuk menunjang ekonomi keluarga. Dilihat dari sisi ekonomi, usaha
peternakan domba mempunyai prospek bagus karena mempunyai perputaran modal
yang lebih cepat dibandingkan dengan usaha ternak ruminansia besar seperti sapi atau
kerbau. Bahkan isu terakhir, ternak domba dari Indonesia dapat memasok sebagian
keperluan domba di Timur Tengah. Namun sampai saat ini usaha peternakan domba
belum diusahakan secara industri atau dalam skala besar. Selama ini usaha pemuliaan
telah diupayakan untuk memperbaiki sifat genetik dalam produksi (daging) tinggi,
namun hasil yang diperoleh belum maksimal. Terlebih lagi, usaha pemuliaan
produksi domba juga belum merambah untuk memikirkan produksi daging dengan
sebaran lemak yang rendah. Kualitas daging akan mempunyai nilai tambah apabila
daging yang dihasilkan tidak mengandung banyak lemak atau dihasilkan daging
kurus (leaner). Penangkar ternak juga belum belum memperhitungkan efisiensi waktu maupun jumlah ternak yang digunakan dalam program pemuliaan.
Perkembangan ilmu pengetahan dan teknologi yang semakin cepat berkembang
dewasa ini, usaha perbaikan genetik sekarang dapat lebih diarahkan untuk sifat
bernilai ekonomi tertentu. Kemajuan pengetahuan di bidang biologi molekuler dan
bidang teknologi informasi utamanya bioinformatika, maka penelitian pada bidang
Selama ini penelitian dalam perbaikan genetik ternak untuk sifat kuantitatif
bernilai ekonomi tinggi, dilakukan kurang efektif karena memerlukan populasi ternak
yang besar dan hampir semua menggunakan nilai dugaan berdasar analisis statistik.
Sekarang, penelitian seperti tersebut akan menjadi lebih cepat dan akurat karena
tersedianya peta genetik ternak dan tersedianya program komputer (software) yang
basis datanya adalah fragmen DNA (merupakan manifestasi alel) yang digunakan
sebagai parameter selain data kuantitatif. Adanya fasilitas peta genetik ternak
tersebut, kesempatan untuk mengungkap lebih jauh sifat yang sulit untuk diwariskan,
sekarang dimungkinkan untuk diupayakan perbaikan genetiknya. Penelitian pemetaan
sifat genetik akhir-akhir ini semakin penting sebagai terobosan pada pencarian sifat
kuantitatif bernilai ekonomi pada pemuliaan ternak.
Sejarah terciptanya peta genetik ternak tidak terlepas dari teori pewarisan
Mendel. Teori pewarisan sifat tersebut dilakukan oleh Bapak Ilmu Genetika Modern -
Gregor Johann Mendel pada sekitar abad 18 (Russell 1990). Kemajuan ilmu genetika
kemudian disusul dengan ditemukannya jarak antara dua gen pada kromosom oleh
Morgan. Penetapan jarak fisik antara dua gen tersebut diukur dengan satuan map units, dan akhirnya penetapan satuan jarak ini disepakati dengan nama centi-Morgan
(cM) untuk memberi kehormatan pada Morgan sebagai penemunya (Russell 1990).
Penentuan jarak antara dua gen, didasarkan pada banyaknya frekuensi terjadinya
rekombinasi. Semakin sering terjadi rekombinasi (lokasinya disebut hot spot) diantara 2 gen atau penciri genetik, ini berarti jarak atau interval antara 2 gen tersebut lebih
lebar. Sebaliknya semakin jarang kejadian rekombinasi, berarti jarak antara dua gen
tersebut adalah lebih pendek. Pada daerah hot spot tersebut dimungkinkan terjadinya segregasi, sementara jarak yang lebih pendek adalah semakin kecil terjadi segregasi.
Perkembangan genetika modern dari Mendel dan ditambah penemuan dari
Morgan tersebut kemudian mengilhami dibuatnya peta genetik pada hewan budidaya,
misal pada domba. Peta genetik tersebut awalnya dikemukakan pada tahun 1994 oleh
tahun oleh Maddox et al. (2001; 2002). Perkembangan peta genetik ternak domba tersebut sekarang menjadi acuan untuk penelitian pemetaan quantitative trait loci
(QTL) untuk sifat kuantitatif penting bernilai ekonomi. Dengan dilengkapinya peta
genetik ternak domba, hal ini menjadi kemajuan besar di bidang penelitian genetika
molekuler pada ternak domba.
Salah satu sifat kuantitatif penting pada ternak adalah produksi karkas. Sifat ini
berhubungan dengan sifat bobot badan dan pertumbuhan pada ternak. Sifat tersebut
belum diupayakan semaksimal mungkin dalam perbaikan genetik yang ekspresinya
dapat dilihat pada perbaikan tampilan fenotipenya. Perbaikan genetik melalui seleksi
ternak yang dilakukan selama ini semata-mata berdasarkan tampilan fenotipe untuk
menduga kontribusi genetik yang diwariskan atau gen yang dipindahkan dari generasi
ke generasi berikutnya. Penemuan saat ini (pada seleksi ternak) dengan melibatkan
penggunaan penciri genetik akan mempersempit estimasi karena pendugaan yang
tadinya hanya didasarkan pada parameter fenotipe sekarang dimungkinkan untuk
meduga dengan tambahan informasi sampai tingkat DNA.
Dinyatakan oleh Kinghorn et al. (1994) bahwa penggunaan teknik molekuler dapat membantu memecahkan beberapa keterbatasan dari metode yang selama ini
digunakan. Lebih lanjut diterangkan bahwa kemampuan untuk menyusun peta
genetik yang lebih lengkap untuk setiap jenis hewan memungkinkan dilakukannya
evaluasi QTL seluruh genom untuk QTL yang mempunyai efek besar terhadap
fenotipe. Informasi demikian dapat dimanfaatkan di dalam program pemuliaan.
Selain itu metode analisis segregasi telah dikembangkan untuk mendeteksi
keberadaan gen mayor (major genes) dari analisis data kuantitatif pedigree (asal usul atau silsilah) tanpa adanya informasi molekuler (Bovenhuis et al. 1997). Namun analisis segregasi ini diketahui kurang kuat dalam pembuktian keberadaan gen mayor
dibandingkan dengan studi analisis QTL yang dilengkapi dengan pemanfaatan penciri
molekuler.
Hingga saat ini di Indonesia belum dilakukan analisis QTL untuk identifikasi
gen mayor yang dikaitkan dengan sifat kuantitatif bernilai ekonomi tinggi pada
online dari internet dan tersedianya fasilitas laboratorium molekuler dapat digunakan untuk membantu analisis pemetaan QTL.
Tujuan Penelitian
Tujuan penelitian ini adalah:
1. Menerapkan metode pemetaan QTL sifat pertumbuhan dengan memanfaatkan
penciri genetik
2. Identifikasi QTL dan estimasi lokasi QTL (cM) yang mempengaruhi sifat
pertumbuhan pada berat lahir (BL), berat badan umur 90 hari (BB90), berat
umur 180 hari (BB180), berat umur 270 hari (BB270) dan berat umur 360 hari
(BB360)
3. Identifikasi penciri DNA apit (flanking markers) dalam pemetaan QTL
4. Mencari gen kandidat sifat pertumbuhan pada domba
Manfaat Penelitian
Dengan ditemukannya penciri DNA dan ketersediaan perangkat lunak dalam
komputerisasi untuk membantu analisis pemetaan QTL, maka penelitian pemetaan
QTL sifat pertumbuhan dengan beberapa karakter berat badan pada domba memberi
beberapa manfaat seperti berikut:
1. Ternak dapat diseleksi lebih akurat dengan informasi keberadaan sifat yang
dicari berlokasi relatif tepat
2. Keberadaan gen mayor yang berasosiasi dengan penciri genetik (marker)
dapat dideteksi lebih awal
3. Biaya dan waktu seleksi dapat ditekan atau program pemuliaan secara
keseluruhan dapat dilakukan secara efisien melalui Marker Assisted Selection
(MAS)
4. Metode identifikasi penciri genetik untuk sifat pertumbuhan ini dapat
TINJAUAN PUSTAKA
Quantitative Trait Loci (QTL)
Pengertian QTL
Dalam pemuliaan ternak akhir-akhir ini sering dibahas tentang istilah QTL.
Banyak pernyataan diuraikan untuk menjelaskan istilah QTL, satu dan lainnya hampir
sama artinya atau terkadang bersifat melengkapi istilah QTL lainnya. Kim & Park
(2001) menerangkan bahwa bila pada suatu lokasi dalam kromosom suatu individu
terdapat suatu gen yang bertanggung jawab terhadap variasi suatu sifat, maka tempat
tersebut disebut QTL. Studi lainnya menyebutkan bahwa QTL adalah suatu istilah
yang digunakan untuk menyebut lokusnya atau lokasinya polygene. Poligen adalah sejumlah gen yang masing-masing mempunyai efek kecil secara bersamaan
berekspresi untuk mendeterminasi fenotipe dari satu sifat kuantitatif (Zaid et al. 2001). Van der Werf (2000c) menyatakan bahwa meskipun QTL dapat saja ditempati
gen yang mempunyai pengaruh apapun namun di dalam praktek lebih ditekankan
hanya terhadap keberadaan gen mayor pada suatu QTL. Dinyatakan oleh Dominik
(2005b) bahwa QTL akan bermanfaat apabila pada loci nya ditempati oleh gen mayor. Lebih lanjut dinyatakan bahwa gen mayor sendiri sulit ditemukan namun
materi atau penciri genetik tertentu pada kromosom dapat digunakan sebagai tapak
atau landmark yang ada keterpautan dengan gen mayor tersebut. Landmark tersebut dianggap sebagai alat penciri atau tool yang dapat membantu dalam penelusuran kemungkinan apakah seekor hewan adalah pembawa suatu gen mayor.
Van den Werf (2000c) juga menerangkan bahwa QTL mencerminkan hanya
beberapa dari banyak gen yang berpengaruh pada fenotipe. Variasi pada polygene
yang terkait dengan polimorfisme QTL menentukan total variasi genetik. Walaupun
pengaruh QTL menerangkan hanya sebagian perbedaan genetik diantara hewan,
namun pengetahuan gen yang berlokasi pada QTL dapat sangat membantu dalam
estimasi suatu genotipe yang benar dari hewan. Oleh karena itu, informasi yang
2000c). Jika gen yang terdapat pada QTL berpengaruh besar, gen demikian dapat
lebih spesifik dieksploitasi pada program pemuliaan.
Sifat Kuantitatif
Kebanyakan sifat penting bernilai ekonomi pada pemuliaan ternak adalah
beragam yang berlangsung terus menerus (continuously varying), sebagai contoh adalah produksi susu, berat wol, berat badan dan produksi telur (Nicholas 1996).
Semua sifat yang menunjukkan continuous variation disebut sifat kuantitatif atau sifat yang dapat diukur (matric traits) dan variasi di dalam sifat kuantitatif disebut variasi atau keragaman kuantitatif (Zaid et al. 2001; Nicholas 1996). Dinyatakan lebih lanjut oleh Nicholas (1996), variasi kuantatif terjadi sebagai akibat adanya aksi
dari gen.
Menurut laporan Dekkers (2004) kemajuan di bidang genetika molekuler telah
memungkinkan identifikasi banyak gen (multiple genes) atau penciri genetik berhubungan dengan gen yang berpengaruh pada sifat penting ternak. Termasuk
dalam gen tersebut yaitu gen tunggal (cacat genetik, penyimpangan genetik,
penampilan) yang mempengaruhi suatu sifat dan QTL atau daerah genomik yang
mempengaruhi sifat kuantitatif. Fasilitas penciri genetik tersebut telah menyediakan
kesempatan untuk meningkatkan respons pada seleksi terutama untuk sifat yang sulit
berkembang dengan seleksi konvensional (Dekkers 2004). Termasuk dalam sifat
yang sulit berkembang tersebut yaitu sifat yang mempunyai heritabilitas rendah atau
sifat yang pengukuran fenotipenya sulit, mahal, hanya dapat dilakukan pada akhir
kehidupan atau tidak mungkin dilakukan seleksi kandidat. Beberapa karakter sifat
kuantitatif yang sulit untuk dikembangkan tersebut diistilahkan sebagai sifat
kompleks. Dinyatakan oleh Primrose (1995) bahwa sifat kompleks tersebut
diberlakukan untuk semua tampilan fenotipe yang tidak memperlihatkan adanya
pewarisan sifat hukum Mendel. Lebih lanjut diterangkan oleh Primrose (1995), sifat
kompleks yang disebabkan oleh adanya pewarisan poligenik yang memerlukan
juga dikelompokkan sebagai suatu continuous variation (Lander & Schork 1994; Zaid et al. 2001).
Gen Mayor
Telah diketahui bahwa variasi genetik pada sifat kuantitatif dikarenakan adanya
segregasi pada banyak loci. Kebanyakan sifat penting bernilai ekonomi adalah sifat kuantitatif yang kebanyakan dikontrol oleh sejumlah gen. Beberapa gen tersebut
dapat mempunyai pengaruh besar dan gen demikian disebut gen mayor atau major gene yang berlokasi pada QTL (Van der Werf 2000a). Dinyatakan oleh Montaldo et al. (1998), bahwa gen mayor menyebabkan perbedaan sifat besar diantara hewan yang menurunkan alel berbeda. Lebih lanjut dinyatakan bahwa kebalikan gen mayor
adalah polygene yang mempunyai pengaruh secara individu kecil pada fenotipe yang tidak dapat dihubungkan pada individu gen apapun.
Gen mayor dapat dideteksi dengan dua sudut pandang yang berbeda yaitu dari
sudut biologi dan teori (Lynch & Walsh 1998). Dari sudut pandang biologi,
keberadaan gen mayor menawarkan potensi untuk karakterisasi genetik dan
isolasinya. Hal tersebut merupakan suatu informasi berguna yang mendasari proses
biologi yang menurunkan variasi karakter atau sifat. Dari sudut pandang teori,
terdapat beberapa model genetika kuantitatif yang mengasumsikan sejumlah besar
loci yang mempunyai pengaruh sama secara besar (roughly equal effects). Validitas model tersebut dipercayai menunjukkan keberadaan gen mayor. Model lain dengan
pengamatan unimodal (=sebaran satu kurva) secara kontinyu dari fenotipe sering
mendukung sejumlah besar pengaruh sama secara besar. Hal tersebut dapat dianggap
sebagai asumsi jika pengaruh lingkungan cukup besar berhubungan dengan pengaruh
individu gen manapun. Apabila alel mayor pada frekuensi cukup rendah, pengaruh
segregasi gen mayor dapat benar-benar tidak jelas.
Diterangkan oleh (Lynch & Walsh 1998) bahwa gen mayor dapat pula dideteksi
dengan bentuk analisis statistik paling sederhana yaitu uji normalitas, kemudian
dengan uji yang cukup sederhana, kelompok dari keluarga yang dikenal (known
(1998), gen mayor juga dapat dideteksi dengan metode mixture-models dimana penyebaran fenotipe diasumsikan, hasil dari campuran terbobot (weighted mixture) adalah yang mendasari penyebaran (2 kurva). Hasil penyebaran gabungan dari
distribusi normal umumnya menghasilkan penyebaran tidak normal. Penyimpangan
dari distribusi normal diindikasikan terdapatnya gen mayor. Namun kekuatan dari
analisis mixture-models tersebut masih rendah.
Cara lain adalah analisis segregasi yang lebih kompleks yang melibatkan data
fenotipik dan hubungan keluarga yang kompleks (multiple generation) dengan jumlah ternak banyak (Lynch & Walsh (1998). Metode ini dimaksudkan untuk
memberi kesempatan genotipe setiap individu dalam populasi (keluarga) dengan
menggunakan semua data fenotipe. Kekuatan metode ini rendah apabila pengaruh gen
mayornya kecil. Kekuatan metode segregasi dapat lebih ditingkatkan apabila
digunakan penciri DNA, sehinga posisi gen pada kromosom dapat dideteksi (Lynch
& Walsh 1998).
Penelusuran gen mayor telah berhasil untuk beberapa sifat bernilai penting
diantaranya yaitu gen wol karpet, double muscling pada sapi, gen cekaman (stress)
pada babi, pembentukan punuk pada sapi Limousin, dan sifat resistensi terhadap
penyakit cacing pada domba (Montaldo et al. 1998). Penelusuran gen mayor dapat dilakukan melalui beberapa keluarga yang merupakan hasil persilangan balik bangsa
ternak dengan latar belakang genotipe berbeda (Raadsma et al. 2002a). Salah satu pendekatan untuk mencari keberadaan gen mayor untuk sifat tertentu yaitu melalui
analisis segregasi data fenotipe dari keluarga acuan yang tepat (LeRoy & Elsen
1992).
Deteksi QTL
Banyak sifat atau karakter biologi penting diwariskan secara kuantitatif tetapi
pengaruh dari pewarisan kuantitatif tersebut secara keseluruhan tidak dapat dideteksi
secara individu. Hal ini dikarenakan karakter kuantitatif tersebut selama ini hanya
diselesaikan dengan menggunakan prosedur biometrik (Primrose 1995). Selain itu,
diterangkan tanpa melibatkan informasi tentang gen. Identifikasi permasalahan
tersebut dapat dilakukan dengan analisis QTL. Analisis QTL dapat dilakukan dengan
fasilitas keberadaan penciri genetik yang dari waktu ke waktu semakin banyak
macamnya seperti RFLP, mikrosatelit dan lain-lain (Primrose 1995).
Dilaporkan oleh Dekkers (2004), guna tujuan aplikasi dan deteksi QTL, sifat
kuantitatif dapat dikategorikan ke dalam 3 kelompok, yaitu sifat yang perlu
pencatatan rutin, sifat yang sulit untuk dicatat (asupan pakan, kualitas produksi) dan
sifat yang tidak dapat dicatat (ketahan penyakit). Lebih lanjut dinyatakan oleh
Dekkers (2004) setiap kelompok kategori tersebut lebih lanjut dibedakan ke dalam 3
sifat, yaitu data tercatat pada kedua jenis kelamin, sifat terbatas jenis kelamin
(sex-limited) dan sifat yang dicatat pada akhir hidupnya. Kemampuan mendeteksi QTL
tergantung pada ketersediaan data fenotipik dan pengelompokkan pada ke tiga
kategori dan ke tiga sifat yang telah disebutkan. Dicontohkan oleh Dekkers (2004),
yaitu perunutan genom (genome scans) yang memerlukan lebih banyak data fenotipik dari pada analisis gen kandidat sering digunakan untuk mendeteksi QTL untuk sifat
yang dikategorikan pada pencatatan rutin sedangakn pendekatan gen kandidat lebih
sering digunakan pada identifikasi QTL untuk sifat yang tidak memerlukan pencatana
rutin (dua kategori lainnya).
Prinsip Pemetaan QTL
Seperti diketahui bahwa terdapat keterpautan antara penciri genetik dengan gen
pada locus atau QTL untuk satu sifat kuantitatif tertentu (Van der Werf 2000c; Dominik 2005a). Guna mencari hubungan tersebut, terdapat persyaratan yang perlu
dipenuhi, yaitu menyusun suatu populasi yang cukup besar dengan rancangan
tertentu, melibatkan sejumlah besar penciri genetik dengan melihat peta keterpautan
atau linkage mapping, menetapkan sifat yang akan dicari QTL nya berdasarkan data fenotipe yang diamati sebelumnya dan penggunaan analisis QTL. Saat ini analisis
QTL telah dipermudah dengan kemajuan teknologi informasi utamanya
‘bioinformatika’ yang mudah diakses secara elektronik. Disarankan oleh Bovenhuis
pemuliaan, gen yang mempengaruhi sifat penting perlu dideteksi dan efeknya perlu
diestimasikan.
Pendekatan Studi Pemetaan QTL
Pada studi Primrose (1995) dinyatakan setidaknya terdapat 2 pendekatan untuk
memetakan QTL, pertama metode Edwards et al (1987) yaitu dengan regresi linier, untuk menguji hubungan antara penampilan sifat kuantitatif dan genotipe pada
marker locus. Apabila terdapat hubungan nyata secara statistik antara penampilan sifat dan marker locus gene types, hal ini dikatakan bahwa sebuah QTL berlokasi dekat dengan lokus marker. Metode kedua yaitu dengan interval mapping. Analisis yang digunakan dalam metode kedua ini yaitu berdasarkan ukuran genom dan jumlah
marker yang dianalisis berdasarkan nilai threshold (nilai ambang) yang ditentukan. Apabila letak QTL untuk sifat tertentu yang dicari tidak diketahui, maka diperlukan
suatu rancangan dengan merunut seluruh genom atau full genome scan dengan penciri DNA polimorfik (Raadsma et al. 2002b). Selanjutnya dengan pengetahuan yang lebih baik tentang QTL, maka pengetahuan untuk mempertahankan keragaman dan
menggunakannya dengan cara yang lebih efisien, efektif dan berkelanjutan sangat
diperlukan (Nicholas 1996).
Saat ini terdapat beberapa metode (misal: metode regresi, Maximum Likelihood estimation) untuk mendeteksi QTL yang mempengaruhi sifat poligenik seperti ketahanan penyakit dan pertumbuhan telah diuraikan secara komprehensif (Lynch &
Walsh 1997). Semua metode kecuali analisis segregasi tergantung pada linkage disequilibrium antara penciri genetik dan loci yang mempengaruhi sifat tertentu (Crawford et al. 2000). Dasar teori tersebut telah diketahui beberapa tahun terakhir ini dan sekarang telah banyak penciri genetik hasil pemetaan terbaru dan yang
terakhir tahun 2004 telah dipublikasikan melalui situs
http://rubens.its.unimelb.edu.au/~jillm/jill.htm dan menghasilkan perkembangan
cepat dalam teknik analisis data QTL. Kemajuan cepat tersebut telah memungkinkan
Dasar metode untuk mendeteksi QTL tersebut dapat digunakan untuk ternak
domestik.
Dinyatakan oleh Crawford et al. (2000) metode terkini yang dipilih ditentukan oleh beberapa faktor diantaranya ketersediaan sumber dana dan populasi ternak serta
informasi QTL komparatif dari jenis ternak lainnya. Guna keperluan pemetaan QTL
sifat kuantitatif pada ternak domestik, biasanya dilakukan tahapan yang meliputi
pengukuran fenotipe, pembacaan genom atau genome scan melalui genotyping, kemudian dianalisis dengan perangkat lunak (software) yang tersedia pada internet.
Saat ini banyak perangkat lunak dibuat dan dapat diakses secara gratis melalui
internet. Salah satu diantaranya yaitu dengan menggunakan program QTL Express
melalui fasilitas website http://qtl.cap.ed.ac.uk/ yang didasarkan dengan pendekatan
regresi rentang atau jarak. Secara detail analisis tersebut dijelaskan oleh Haley &
Knott (1992) dan Seaton et al. (2002).
Dalam hubungannya dengan studi pemetaan tersebut di atas, beberapa analisis
terkait akan diperlukan untuk menentukan apakah sifat produksi dipengaruhi oleh gen
mayor. Keberadaan gen mayor dapat diketahui dengan persilangan dua populasi yang
berbeda karakter atau sifat (Lynch & Walsh 1998). Lebih lanjut diterangkan bahwa
pendekatan terkait yang digunakan untuk mendeteksi gen mayor dalam seleksi yaitu
metode seleksi dengan menggunakan populasi ternak silang balik atau disebut select-and-backcross method. Prosedur sederhananya, yaitu dua populasi berbeda karakter disilangkan untuk memperoleh F1 dengan karakter terbaik kemudian disilangkan
balik dengan individu dari populasi tetuanya yang mempunyai karakter lebih rendah.
Hal demikian dimaksudkan untuk membuang karakter yang lebih kecil untuk
kemudian memperoleh karakter yang menonjol dengan pengaruh besar atau disebut
gen mayor. Analisis untuk mendeteksi gen mayor atau analisis segregasi
dimaksudkan sebagai dasar dalam analisis berikutnya dalam pemetaan QTL.
Guna keperluan mendeteksi keberadaan QTL, maka diperlukan rancangan
penyusunan ternak penelitian atau populasi ternak yang tepat dan dalam jumlah
dapat berupa half-sib (saudara tiri) atau full-sib (saudara kandung). Populasi tersebut dipersiapkan dalam bentuk beberapa keluarga acuan atau reference family dimana satu keluarga terdiri atas kakek (Grandsire), nenek (GrandDam), bapak (Sire) dan anak atau keturunannya dalam jumlah yang cukup banyak. Dinyatakan oleh Primrose
(1995) bahwa untuk melakukan analisis QTL sebaiknya digunakan populasi
keturunan silang balik (backcross progeny) atau F2 silang dalam atau intercrossing
(F1 x F1). Sementara menurut Seaton et al. (2002) menerangkan bahwa populasi yang tepat akhir-akhir ini untuk QTL Express adalah populasi persilangan luar half-sib.
Selain populasi yang cukup, penciri genetik mikrosatelit juga diperlukan dalam
jumlah yang cukup banyak dan diharapkan dapat menunjukkan bahwa sifat
kuantitatif yang diteliti bersegregasi dan diwariskan dari tetua kepada turunannya.
Penggunaan penciri mikrosatelit akhir akhir ini semakin diminati, karena sifatnya
yang sangat polimorfik dan penyebarannya dalam genom cukup merata (Nicholas
1996). Penggunaan mikrosatelit dalam genotyping akan memberikan gambaran ada tidaknya segregasi dan menetapkan genotipe dari sifat yang dicari. Selanjutnya dapat
dibuat peta keterpautan (linkage map) dari gen dimaksud (misal untuk sifat produksi). Pemetaan ini dapat dibandingan antara jenis ternak berbeda (misal antara domba dan
sapi), hal ini dikarenakan adanya kesamaan letak peta fisik gen pada kromosom dan
linkage map (Crawford et al. 1995).
Rancangan Hewan Percobaan
Dalam studi pemetaan dan deteksi QTL diperlukan rancangan hewan percobaan
yang tepat. Hal ini sehubungan dengan penggunaan informasi penciri genetik dalam
studi tersebut. Ide dibalik penggunaan informasi penciri genetik untuk memetakan
dan mengkarakterisasi QTL adalah cukup sederhana yaitu menyilangkan dua garis
keturunan silang dalam (two inbred lines). Keterpautan disekuilibrium dibentuk
diantara loci yang berbeda diantara garis keturunan (galur). Keadaan ini membuat
Lynch & Walsh (1998) menerangkan pembentukan populasi dalam rancangan
percobaan hewan untuk studi pemetaan QTL. Lebih lanjut disarankan dua rancangan
hewan percobaan untuk studi pemetaan QTL, yaitu populasi F2 dan populasi silang
balik. Populasi progeny F2 diperoleh dengan menyilangkan dua garis tetua (parental:
P1 dan P2), sejumlah besar F1 yang dihasilkan kemudian disilangkan dengan F1
dalam satu saudara. Sementara populasi progeny silang balik (backcross) diperoleh dengan menyilangkan balik F1 dengan salah satu dari garis tetuanya. Kedua
rancangan hewan percobaan tersebut paling banyak digunakan. Rancangan
pembentukan populasi F2 mempunyai keuntungan melebihi populasi dari rancangan
silang balik, recombinant inbred lines (RILs) maupun doubled haploid lines (DHLs). Hal ini dikarenakan rancangan F2 akan menghasilkan tiga macam genotipe pada
setiap marker locus. Sementara rancangan silang balik, RILs maupun DHLs akan
menghasilkan dua genotipe pada setiap marker locus nya. Penggunaan populasi F2
ini, lebih banyak digunakan pada tanaman.
Menurut Lynch & Walsh (1998), Bovenhuis et al. (1997) dan Georges (1998) terdapat dua pendekatan dalam rancangan hewan percobaan untuk identifikasi gen.
Kedua pendekatan tersebut adalah sebagai berikut:
a. Experimental Crosses
Ini dirancang untuk identifikasi gen yang berperan pada perbedaan yang diamati
untuk satu sifat penting antara 2 galur, bangsa bahkan subspesies. Contoh dari strategi
ini yaitu pemetaan gen berdasar pada banyak perbedaan fenotipe yang diamati antara
babi jantan liar (boar) dan babi domestik sebagai hasil dari ribuan tahun domestikasi
(Anderson et al. 1994). Atau suatu usaha untuk memetakan gen yang menerangkan perbedaan fertilitas pada bangsa babi Cina dan babi Eropa.
Guna pemetaan gen, percobaan silang yang dilakukan adalah dengan
mengawinkan galur (line) dari pihak ayah (parental) yang terseleksi dan berbeda
secara genetis. Hasil individu F1 digunakan untuk menurunkan sejumlah besar F2
bersegregasi atau populasi silang balik (backcross). Banyak metode statistik untuk mendeteksi QTL pada persilangan tersebut yaitu menggunakan sejumlah penciri
fenotipe sifat penting. Cara paling umum yang digunakan yaitu melalui pendekatan
multipoint yang sering disebut sebagai interval mapping. Pendekatan ini menerangkan dimana posisi suatu hipotesis QTL digerakkan (dipindahkan) melalui
suatu peta penciri yang sudah pasti (fixed marker map). Bukti keberadaan QTL
dihitung untuk setiap posisi dengan menggunakan maximum likelihood method
(Lander & Botstein 1989), multiple regression (Haley & Knott 1992; Martinez & Curnow 1992; Haley et al. 1994) atau non-parametric rank-based tests (Kruglyak & Lander 1995).
Keuntungan menggunakan pendekatan persilangan backcross yaitu dapat memperkirakan bahwa galur berbeda nyata secara fenotipe akan mempunyai alel
QTL sangat berbeda dengan pasti atau mendekati kebenaran. Jika F1 tidak berbeda,
perlu dibuat heterosigot untuk alel QTL yang homosigot secara genetik yang
diharapkan agar menghasilkan pengaruh pengganti alel QTL relatif penting pada
generasi F2 atau backcross. Hal demikian juga berlaku bagi individu F1 yang dihasilkan dari persilangan antara garis keturunan pihak ayah yang mempunyai
kemiripan tinggi (increased likelihood) dari yang dibuat heterosigot pada penciri loci. Ini dapat meningkatkan kandungan informasi penciri. Persilangan seperti ini
umumnya dilakukan dibawah kondisi lingkungan terkontrol dengan tepat, sehingga
pengaruh non-genetik dapat dikurangi secara bersamaan .
Kelemahan menggunakan pendekatan crossing, yaitu menghasilkan persilangan jenis ternak yang sangat mahal dan memakan waktu lama. Terlebih lagi, kebanyakan
program pemuliabiakan dilakukan terus menerus pada jenis ternak dimana variasi
genetiknya terdapat pada galur (line) komersial unggul (elite). Belum diperhitungkan
bahwa loci yang menerangkan perbedaan antar galur (line) yang sangat berbeda adalah juga andil pada keberadaan variasi didalam galur suatu populasi komersial.
b. Outbred Pedigrees
Outbred pedigrees yaitu populasi keturunan yang berasal dari persilangan luar (persilangan dari 2 bangsa atau galur) yang bukan satu keluarga. Tujuan
meningkatkan variasi genetik kemudian dipetakan QTL nya. Pemetaan tersebut
berdasarkan perbedaan genetik yang diamati untuk suatu sifat penting pada populasi
komersial. Populasi ternak unggul pada percobaan ini diarahkan dengan penekanan
terhadap seleksi agar alel yang sudah pasti atau mendekati pasti dengan efek luas
dapat dipetakan dengan cepat.
Penggunaan penciri genetik yang informatif dan heterosigot akan mengurangi
jumlah atau besar populasi outbred dan dapat digunakan dalam genetic polymorphism. Perbedaan susunan loci QTL dan alel QTL akan bersegregasi pada keluarga berbeda, dengan demikian akan menambah kompleksitas genetik (perbedaan
locus dan alel) dari fenotipe yang dipelajari pada populasi outbred.
Diterangkan oleh Bovenhuis et al. (1997) bahwa terdapat beberapa perbedaan diantara rancangan percobaan penelitian untuk deteksi QTL. Perbedaan penting
tersebut yaitu analisis data antara populasi inbred lines dan populasi outbred, yaitu - Hanya kelompok grandsire akan bersifat heterosigot untuk marker dan untuk QTL - Grandsire dapat mempunyai perbedaan linkage phase dan pengaruh penciri perlu
dianalisis didalam keluarga
- QTL dapat mempunyai lebih dari 2 alel dan frekuensi alel tidak diketahui
- Linkage phase diantara marker alleles tidak diketahui
Perbedaan dalam rancangan percobaan penelitian mempunyai konsekuensi penting
untuk analisis statistik data dalam hal kekuatan dan ketepatan metodologi.
Bangsa Domba. Domba domestik yang ada saat ini di dunia maupun di
Indonesia berasal dari jenis Ovis aries, yang dipercaya sebagai hasil domestikasi sejak 9000-11.000 tahun yang lalu di Asia Barat Daya. Dinyatakan oleh Franklin
(1997), domba domestikasi (Ovis aries) dikelompokkan sebagai anggota dari suku
Bovidae dari ordo Artiodactyla. Ordo Artiodactyla adalah salah satu ordo mamalia yang paling berhasil dibandingkan dari 10 keluarga lainnya. Ovis aries atau domba domestik yang ada sekarang, dibedakan berdasarkan dari tipe liarnya dan oleh
beberapa penulis dibedakan menjadi 7 jenis (Ryder 1984). Tiga jenis diantaranya
Empat jenis lainnya yaitu Ovis amon (argali), Ovis vignei (urial), Ovis orientalis
(Asian mouflon) dan Ovis musimon. Ovis orientalis diperkirakan sebagai nenek moyang dari semua domba domestik yang ada sekarang. Ovis amon (argali) adalah domba yang penyebarannya di pegunungan Asia Tengah. Selain itu, uniknya nenek
moyang domba masih dapat ditemui dalam kehidupan liarnya dan dalam jumlah yang
banyak (Subandriyo 2003).
Domestikasi domba selama lebih dari 10.000 tahun yang lalu telah
menghasilkan peningkatan ukuran badan dan penurunan ukuran tanduk serta
perubahan dari berbulu rontok (hairy moulting fleece) sampai berbulu wool putih
(Ryder 1983). Crawford et al. (1995) menyatakan banyak bangsa domba yang
tersebar didunia ini merupakan bangsa lokal dan galur yang telah berkembang baik
pada sistem produksinya. Lebih jauh diutarakan bahwa perbaikan genetik telah terjadi
lebih dari 50 tahun dari aplikasi genetika kuantitatif dalam pemuliaan.
Sifat genetik. Sebagai salah satu syarat dalam pemetaan QTL adalah
pembentukan suatu populasi dari dua galur atau bangsa domba yang mempunyai sifat
genetik berbeda (Primrose 1995; Raadsma et al. 2002a). Persilangan dua sifat genetik berbeda dimaksudkan agar terbentuk suatu keturunan (progeny) populasi heterosigot
atau dalam genotyping akan terbentuk sebaran banyak alel dalam pedigree.
Dua galur domba yang digunakan sebagai asal-usul penurunan populasi domba
silang balik (backcross) atau keturunan F2. Dua galur tersebut yaitu domba lokal ekor
tipis Indonesia (Indonesian Thin Tail=ITT) sebagai domba tropis dan tipe domba kecil, dan domba Merino yang berasal dari daerah bersuhu dingin, dianggap sebagai
tipe domba besar. Perbedaan sifat genetik dari dua galur yang kontras ini juga
dipersyaratkan untuk analisis keberadaan gen mayor suatu sifat penting (Lynch &
Walsh 1998). Beberapa karakteristik penampilan yang berbeda dari domba ekor tipis
Indonesia dan domba Merino untuk analisis pemetaan QTL sifat pertumbuhan dalam
Tabel 1. Tampilan fenotipik domba ekor tipis Indonesia dan domba Merino
Tampilan
Fenotipik Ekor tipis Indonesia (ITT) Merino Tipe Domba kecil (Subandriyo 2003) Domba besar (Maijala
1997)
B: 23-46kg (Dwiyanto 1982): 22-55kg (Mulliadi 1996)
J: 20-29kg (Diwyanto 1982; Mulliadi 1996)
B: 52kg J: 83kg (Piper &
Ruvinsky 1997)
Kualitas wol
Kasar, nilai ekonomi rendah (Sabrani et al. 1982; Subandriyo et al. 1996, Subandriyo
2003; Tiesnamurti et al. 1998)
Halus, nilai ekonomi tinggi (Dolling & Jefferies 1991; Maijala 1997)
Ekor Sedang (Mason 1980; Mulliadi 1996) Panjang Telinga Bervariasi: pendek, sedang, normal
(Subandriyo 2003) Panjang
Pertumbuhan 20-40g/hari, pemeliharaan tradisional
(Chaniago et al. 1982; Thomas et al. 1982) 195g/hari (Putu 1981) Umur dewasa 6-12 bulan (Zulbardi 1977; Obst et al. 1980;
Sitorus et al. 1985)
24 bulan (Piper & Bindon 1996)
Jumlah anak per kelahiran
1,8 kelahiran pertama (Bradford & Inounu 1996); 2,2 kelahiran ke tiga (Sitorus et al.
1985); 2,2 (Setiadi et al. 1995)
1-3 rata-2 pada setiap kelahiran (SASBA 2001)
Berat potong
(2-3th) 45-50kg (Bradford & Inounu, 1996) 62-70kg (Austin 1950)
% Karkas 35% 55-60% (Austin 1950)
Penciri DNA
Semenjak era Mendel sampai tahun 1980 an, para ahli genetika hanya
mendapatkan penciri genetik locus tunggal berupa tampilan fenotipe (Crawford et al. 2000). Penciri tersebut diantaranya seperti warna mata pada Drosophila atau polimorfisme protein seperti dalam penggolongan darah. Lebih lanjut dijelaskan oleh
Crawford et al. (2000), penggunaan penciri tersebut pada beberapa peta keterpautan genetik secara rinci telah dikembangkan pada model jenis seperti mencit dan
Drosophila. Namun demikian terdapat beberapa keterbatasan untuk penyusunan peta keterpautan pada persilangan jenis hewan domestik. Kehadiran teknologi DNA
sekarang ini dapat dilihat banyak proyek pemetaan keterpautan untuk jenis ternak
apapun dapat direncanakan dan diimplementasikan. Selama lebih dari satu dasa warsa
terakhir ini, terdapat sejumlah penciri DNA yang secara rinci telah dideskripsikan
dalam hubungannya dengan pencarian QTL, peta keterpautan perbandingan
(comparative linkage mapping) dan pengukuran keragaman genetik. Secara garis besar penciri DNA ini dapat dibedakan menjadi dua, yaitu Multilocus marker dan
single locus marker (Crawford et al. 2000). Termasuk dalam multilocus markers
yaitu minisatelit atau variable number tandem reapeat (VNTR), randem amplified polymorphic DNA fragment (RAPD) dan amplified fragment length polymorphisms
(AFLP). Sedangkan yang termasuk single locus markers adalah restriction fragment length polymorphisms (RFLPs), Mikrosatelit dan single nucleotide polymorphisms
(SNPs). Masing-masing penciri DNA tersebut diuraikan secara jelas seperti di bawah
ini.
Minisatelit. Dikemukakan oleh Crawford et al. (2000), minisatelit merupakan penciri DNA dengan banyak alel. Ditemukan oleh Jeffreys et al. (1985), minisatelit ini merupakan penciri DNA pertama pada manusia yang cukup informatif untuk
mengemukakan genotipe unik pada setiap individu. Berdasarkan pola runutan
basanya, minisatelit ini dikelompokan sebagai molekul DNA yang bukan gen dan
menyebar disemua kromosom (Muladno 2002). Berdasarkan ukuran besar
pengulangan unit tandem atau pasangan, minisatelit mempunyai tipe pengulangan unit tandem antara 10 sampai 100 basa (Nicholas 1996). Minisatelit ini menyebar
lebih meluas pada genom dari pada satellite DNA (pengulangan unit tandem antara 5
sampai 500 pasang basa), Nicholas (1996). Penyebaran minisatelit cenderung
terkonsentrasi pada daerah tertentu seperti pada telomere (Nicholas 1996; Crawford
et al. 2000) dan pada tempat yang tidak umum yaitu daerah yang banyak terjadi frekuensi rekombinasi, daerah rekombinasi tersebut dikenal hotspots (Nicholas 1996). Lebih lanjut ditambahkan oleh Nicholas (1996), minisatelit DNA sebetulnya berperan
bermanfaat karena letaknya hanya pada daerah telomere (Crawford et al. 2000). Dilaporkan oleh Cockett et al. (1994), single locus minisatellite adalah penciri DNA pertama yang berasosiasi dengan gen Callipyge domba.
Variable Number Tandem Repeat (VNTR). VNTR adalah daerah (region)
pada genom manusia dengan tipe sekuens DNA yang sangat bervariasi dan terletak
terpisah-pisah (Van der Werf 2000b). Sebelumnya Nicholas (1996) menerangkan
pada VNTR terdapat keragaman atau variasi jumlah unit tandem pada satu tempat
dari satu kromosom yang berbeda dengan satu tempat dari kromosom homolognya
pada jenis hewan ternak yang sama. Misal, pada satu tempat dari satu kromosom
terdapat 24 kopi tandem dan pada homolog kromosomnya dari hewan yang sama
hanya terdapat 21 kopi tandem. Pengulangan tandem adalah banyak kopi dari sekuens
pasang basa yang tersusun pada tampilan kepala sampai ekor (Van der Werf 2000b).
Misal, Pengulangan tandem yang sering didapatkan adalah CA, dan satu untai terdiri
atas tipe ulangan tersebut yang dibaca CACACA.., dinotasikan sebagai (CA)n.
Sedangkan untai lain akan dibaca GTGTGT,…. Dalam contoh tersebut jumlah
pengulangan berpasangan adalah dua, namun dapat terjadi lebih dari dua. Bila jumlah
pengulangan berpasangan kurang dari empat, VNTRs disebut microsatellite dan jika pengulangan lebih panjang disebut minisatellite.
Random Amplified Polymorphic DNA Fragment (RAPD). RAPD adalah
penciri pertama yang didasarkan pada PCR untuk dapat digunakan (Williams et al. 1990). Primer berukuran kecil (8-10 basa) digunakan untuk mengamplifikasi satu
potongan acak DNA suatu genom. Ukuran primer telah disusun sedemikian sehingga
kira-kira 20 pita (bands) diamplifikasi oleh setiap reaksi PCR. Beberapa pita dapat
jadi polimorpik dan dapat digunakan sebagai penciri genetik. Penciri ini dapat
bermanfaat besar untuk menjadi sangat mudah dihasilkan dan memerlukan hanya
sedikit jumlah DNA. Oleh karena itu banyak peta keterpautan, terutama pada
tanaman menggunakan penciri RAPD. Dikarenakan individu heterosigot dan
homosigot tidak dapat dibedakan, maka penciri ini dominan. Penampakan atau tidak
PCR. Oleh karena itu penciri RAPD tidak mudah untuk diproduksi kembali (lower
reproducibility), sehingga sangat tidak menguntungkan dari penciri RAPD adalah
peta baru harus diturunkan kembali untuk setiap turunan (pedigree) baru yang akan
diuji karena tidak adanya spesifisitas locus pada primer yang digunakan. Pita yang diturunkan dari primer tertentu pada satu pedigree mungkin tidak mendukung
hubungan apapun terhadap pita yang diturunkan dari primer yang sama pada pedigree
kedua.
Amplified Fragment Length Polymorphisms (AFLP). AFLP merupakan
penciri multi locus dan telah digunakan dalam studi biodiversitas (Vos et al. 1995). Tidak seperti pada penciri RAPD, penciri AFLP diperoleh pada potongan (fragment)
yang diamplifikasi dengan menggunakan primer PCR terseleksi. Diterangkan oleh
Crawford et al. (2000), DNA genom dipotong dengan enzim restriksi endonuclease dan penghubung (linkers) diligasi (direkatkan) pada setiap ujung potongan. Primer
PCR terseleksi digunakan untuk mengamplifikasi sejumlah potongan dari campuran
potongan restriksi genom. Primer selektif pada PCR tersebut terdiri atas penghubung
yang ditambahkan pada potongan restriksi akhir dan penambahan basa pada akhir tiga
prime (3’) dari primer, dengan demikian memberikan tambahan spesifisitas. Potongan yang teramplifikasi kemudian dipisahkan menurut ukurannya. Pita-pita
yang terbentuk terdapat pada beberapa individu tetapi tidak ada pada yang lain. Pita
tersebut dapat digunakan sebagai penciri genetik. Penciri AFLP ini mempunyai
keuntungan sama seperti RAPD, yaitu dengan mudah diturunkan tetapi penciri
tersebut kurang memberi kepastian pada kondisi PCR yang sama untuk memperoleh
produk amplifikasi yang diinginkan. Lebih lanjut disarankan oleh Crawford et al. (2000), untuk memperoleh susunan baru penciri, perubahan kecil pada basa prime
tiga (3’) primer amplifikasi adalah semuanya diperlukan. Dengan demikian teknologi
ini dapat menghasilkan penciri genetik baru secara tak terbatas.
Restriction Fragment Length Polymorphisms (RFLPs). Penciri DNA ini telah berkembang lebih dulu dari perkembangan metode PCR. Penciri RFLP mendeteksi
codominant (Crawford et al. 2000). Metode RFLP menggunakan enzim pemotong atau endonuclease pada DNA genom, pemisahan berdasarkan ukurannya diekspresikan dengan gel elektroporesis, pendeteksian dan analisis sekuen DNA
dengan Southern blotting (Nicholas 1996; Crawford et al. 2000). Perbedaan pola pita pada hewan disebut sebagai RFLP. Dikatakan polymorphism, karena ada perbedaan ukuran pita lebih dari satu akibat pemotongan enzim restriksi sehingga menghasilkan
panjang fragmen DNA yang berbeda (Nicholas 1996).
Mikrosatelit. Penciri mikrosatelit, diambil dari pengertian suatu unit ulangan
terkecil 1 sampai sekitar 5 basa, misalnya basa T, AC, GGC, ATTT, ACCGG
(Nicholas 1996). Beberapa penulis memasukan Short Tandem Repeats (STRs) ke dalam kelas Mikrosatelit. Van der Werf (2000b) menyebutkan bahwa Mikrosatelit
adalah daerah DNA dengan jumlah pengulangan tandem pendek yang bervariasi
diapit oleh suatu sekuens unik. Mikrosatelit diketahui dapat membuat penciri genetik
baik karena setiap mikrosatelit mempunyai banyak alel berbeda. Alel adalah bentuk
alternatif gen (Hartl & Clark 1997, Zaid et al. 2001), didefinisikan sebagai jumlah pengulangan bentuk berbeda dari gen yang terletak pada lokasi yang sama. Dengan
banyak alel, maka kebanyakan individu adalah heterosigot. Hal ini memberikan
informasi yang kuat terhadap hubungan antara penciri alel dan penampilan (fenotipe)
pada anak keturunannya (progeny) yang mewarisia favourable linked QTL allele. Penciri mikrosatelit bersifat sangat polimorfik atau hyperpolymorphic dan sangat informatif. Oleh karenanya, penciri mikrosatelit sering digunakan dalam
pemetaan pautan gen pada organisme yang berbeda. Dengan sifat polimorfik yang
tinggi, memungkinkan individu-individu akan menjadi heterosigot dan oleh
karenanya akan lebih mudah dalam menelusuri pewarisan sifat dalam satu keluarga.
Sifat polimorfik yang tinggi ini terletak diberbagai lokasi disepanjang genom,
sehingga mikrosatelit merupakan sumber data yang ideal untuk determinasi jarak
genetik (Nicholas 1996). Seperti pada minisatelit, mikrosatelit adalah multi allelic tandem reapeats. Namun mikrosatelit dikelompokkan sebagai single locus,
(template DNA) dan relatif mudah untuk didapat dan dikarakterisasi (Crawford et al. 2000). Lebih lanjut dikatakan bahwa sebenarnya semua mikrosatelit yang didapatkan
untuk hewan ternak umumnya mempunyai sekuen AC/GT sebagai unit pengulangan.
Hal ini dikarenakan pasangan basa tersebut terdapat berlimpah pada genom ternak
dan oleh karenanya mudah mendapatkannya.
Single Nucleotide Polymorphisms (SNPs). SNPs didasarkan adanya
polimorfisme atau perubahan pada satu pasang basa tunggal (Crawford et al. 2000; Van der Werf 2000b). Lebih lanjut dijelaskan bahwa SNP adalah satu posisi yang
mana dua basa secara bergantian (alternate) berubah atau berganti pada frekuensi
cukup besar (Van der Werf 2000b). Kejadian perubahan pada satu nukleotida ini
sangat jarang, namun diperkirakan satu SNP setidaknya terjadi kira-kira sekali setiap
satu kilo basa (1.000 basa) dari runutan DNA unik pada manusia (Cooper et al. 1985) dan dapat lebih dari sekali dalam seribu pasang basa (Zaid et al. 2001). Pada hewan ternak, kejadiannya mirip, setidaknya dua sampai tiga juta SNPs masih dapat
diidentifikasi dan dikarakterisasi pada banyak jenis hewan ternak (Crawford et al. 2000).
Penciri genetik SNPs ini dihasilkan dari keragaman sekuen atau runutan basa
pada posisi tertentu didalam sekuen DNA. SNPs umumnya hasil dari perubahan
transisi (misal: basa A untuk G, T untuk C) tetapi juga transversi (G atau A untuk T
atau C) dan dilesi basa tunggal (Zaid et al. 2001). SNPs dapat dideteksi dengan banyak metode. Begitu SNPs dapat dideteksi dan dikarakterisasi, sejumlah tipe SNPs
dapat diketahui. Van der Werf (2000b) menyebutkan dengan tersedianya teknologi
baru DNA chips, SNPs dapat digunakan untuk jumlah skala skrining besar dari sejumlah besar sampel pada waktu yang sangat singkat. Perkembangan terkahir DNA chips dapat memuat sample DNA yang lebih banyak (Chee et al. 1996) dan dapat mempercepat proses analisis bahkan untuk tujuan yang lebih jauh. Sejauh ini SNPs
Linkage Mapping
Beberapa penelitian telah mengindikasikan bahwa pemanfaatan penciri genetik
yang berhubungan dengan gen-gen yang terkait dengan sifat kuantitatif ternyata dapat
meningkatkan respon seleksi dari program pemuliaan. Hal ini terutama terjadi pada
sifat kuantitatif yang sulit untuk dikembangkan apabila hanya menggunakan metode
seleksi tradisional (Smith & Simpson 1986; Stam 1986; Kashi et al. 1990; Meuwissen & van Arendonk 1992; Van der Beek & van Arendonk 1996; Meuwissen
& Goddard 1996). Dikatakan oleh Kim and Park (2001) bahwa penciri pada peta
genetik digunakan untuk identifikasi pola penurunan sifat dari linked segments genom pada populasi silsilah terstruktur. Hubungan nyata marker alleles dengan fenotipe sifat penting (interest phenotypes) menunjukan hubungan penciri pada sebuah QTL.
Disarankan oleh Lander & Botstein (1989) bahwa tahap pertama yang perlu
dilakukan untuk mengetahui sifat genetik yang komplek, yaitu perlu memanfaatkan
keragaman genetik yang luas pada domba domestik (Ovis aries) dengan melihat peta keterpautan genetik (genetic linkage map). Peta keterpautan genetik tersebut berupa
penciri genetik yang menutup sebagian besar genom domba atau pada kromosom.
Disebutkan oleh Van der Werf (2000a) dan Dominik (2005a), penyebaran penciri
genetik pada peta fisik tersebut diistilahkan sebagai landmark atau petunjuk. Hal ini karena landmark tersebut dapat digunakan sebagai petunjuk bahwa penciri genetik tersebut berdekatan atau berasosiasi dengan QTL dimana gen yang mengkode sifat
tertentu berdomisili atau terletak.
Saat ini perkembangan peta genetik domba semakin lengkap dari waktu ke
waktu yang diawali tahun 1994 dengan hanya ditemukan 17 penciri genetik (Broad et al. 1997). Kemudian disusul berturut-turut tahun 1995 dengan 246 penciri genetik (Crawford et al. 1995), ditingkatkan kelengkapannya menjadi 519 penciri genetik (de Gortari et al. 1998) selanjutnya Maddox et al. (2001) memperluas perkembangannya dan ditetapkannya 1.062 loci yang berasosiasi dengan penciri genetik. Peta genom domba dari Maddox et al. (2001) dan perkembangannya dapat diakses melalui situs: http://rubens.its.unimelb.edu.au/~jillm/jill.htm.
Analisis QTL
Menurut Lynch & Walsh (1998), estimasi dan deteksi QTL dapat dilakukan
dengan metode Maximun Likelihood (ML) dan Linear Models, namun pada kepustakaan pemetaan QTL lebih popular menggunakan metode ML. Linear models
hanya menggunakan fasilitas penciri genetik (marker means) sementara metode ML
memanfaatkan seluruh informasi dari penyebaran marker-traits, dengan demikian metode ML diakui lebih kuat (powerful). Lebih lanjut dikemukakan bahwa metode
ML menggunakan fasilitas komputer lebih intensif, memerlukan program yang lebih
khusus untuk memecahkan masalah likelihood equations. Sementara linear models
dapat dilakukan hampir dengan semua paket statistik biasa. Pada metode ML dikenal
dengan regresi Haley-Knott untuk memperkirakan ML interval mapping. Satu persoalan dengan banyak estimator ML berarti lebih tergantung dengan banyak
penghitungan. Dibandingkan dengan yang lain, metode ML membatasi daya aplikasi
atau pemanfaatan resampling methods yang memerlukan ribuan estimasi ML yang dihitung per eksperimen. Dengan adanya prosedur regresi sederhana (Haley-Knott regression), ini memberikan estimasi (approximation) yang kuat dari peta likelihood untuk ML interval mapping (Haley & Knott 1992; Martinez & Curnow 1992). Prosedur tersebut memberikan kemudahan pada persoalan besar, dengan demikian
regresi dapat dihitung dengan mudah. Pemikiran Haley & Knott (1992) adalah untuk
mengekspresikan koefisien regresi sebagai satu fungsi parameter QTL yang telah
diketahui.
Segregasi Gen. Untuk mengetahui adanya pemisahan gen dapat dilakukan
dengan cara menguji satu panel penciri genetik dengan sifat kuantitatif penting
(Primrose 1995). Analisis segregasi (tanpa penciri genetik) dimaksudkan untuk
mengetahui penyebaran fenotipe hewan, diuji untuk ketepatan terhadap konsistensi
penyebaran yang diharapkan dengan penyebaran populasi dimana gen mayor atau
major gene bersegregasi (ACIAR report 2001). Pada analisis segregasi tersebut, ukuran kuantitatif fenotipe dibuat dalam suatu kurva. Apabila ukuran kuantitatif
suatu indikasi adanya gen mayor. Selanjutnya diuji secara statistik dengan Maximum likelihood test. Apabila kurva menunjukkan tumpang tindih (overlap), maka ekspresi fenotipe tidak mampu menunjukkan keberadaan gen mayor dan berarti ukuran
fenotipe tersebut bukan pengaruh faktor genetic (ACIAR report 2001).
Segregasi gen mayor dapat dikonfirmasi melalui analisis keterpautan dengan
menggunakan polymorphic markers yang terletak mendekati gen mayor. Dalam hal lokasi gen mayor tidak diketahui, maka sejumlah besar polymorphic markers yang menutup seluruh genom perlu digunakan. Oleh karenanya perlu dilakukan genotyping
dengan suatu panel polymorphic markers. Genotyping dimaksudkan untuk mengetahui adanya alel pada progeny dari tetuanya dengan uji penciri DNA
microsatellite markers. Menurut Kinghorn (2000a), hasil uji DNA ini dengan informasi kuantitatif dapat digunakan untuk analisis segregasi dan mempunyai
kekuatan yang maximal. Analisis segregasi tanpa melibatkan sejumlah penciri
genetik dianggap kurang kuat atau less powerful.
Analisis Keterpautan (Linkage Analysis). Analisis keterpautan genetik dimaksudkan untuk memetakan lokus atau memperoleh satu lokasi kromosom dari
suatu sifat kuantitatif. Prinsip dasar dalam memetakan lokus dari sifat-sifat kuantitatif
yaitu rekombinasi kromosom (Primrose 1995). Dinyatakan lebih lanjut bahwa untuk
identifikasi sifat atau gen tertentu, para ahli genetika telah menciptakan gen penciri
(marker gene), sehingga gen dengan mudah dapat diidentifikasi. Secara genetik, gen
penciri ini diteliti keterpautannya dengan gen pembawa sifat yang dicari. Adanya
keterpautan dapat diuji dengan melakukan perkawinan silang balik (backcross) seekor hewan heterosigot ganda dengan hewan homosigot resesif untuk kedua pasang
gen (Pallawarukka 1999). Dinyatakan jika ratio rekombinasi (crossing over) kurang dari 50%, maka disimpulkan bahwa terdapat gen terpaut (Noor 1996). Crossing over
atau pindah silang terjadi antara kromatid yang bukan pasangannya dari kromosom
homolog. Kejadian ini berlangsung pada pembelahan meiosis dan tepatnya pada