ANALISIS GEROMBOL SIMULTAN DAN JEJARING
FARMAKOLOGI PADA PENENTUAN SENYAWA AKTIF
JAMU ANTI DIABETES TIPE 2
NURUL QOMARIASIH
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul “Analisis Gerombol Simultan dan Jejaring Farmakologi pada Penentuan Senyawa Aktif Jamu Anti Diabetes Tipe 2” adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apapun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir disertasi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
RINGKASAN
NURUL QOMARIASIH. Analisis Gerombol Simultan dan Jejaring Farmakologi pada Penentuan Senyawa Aktif Jamu Anti Diabetes Tipe 2. Dibimbing oleh BUDI SUSETYO dan FARIT MOCHAMAD AFENDI.
Selama ini, pembuatan obat untuk menyembuhkan suatu penyakit masih menargetkan hanya satu protein khusus yang menjadi penyebab penyakit tersebut, yang tentu hanya menggunakan satu senyawa aktif. Selain menimbulkan efek samping, obat kimia sintetis belum bisa menangani suatu penyakit dengan menyasar banyak protein sekaligus. Baru-baru ini terjadi perubahan paradigma dari “one drug, one-target” menjadi “multi-components, network target”. Paradigma baru ini telah melahirkan beberapa penelitian untuk menghasilkan formulasi jamu, dimana konsep formulasi jamu memerlukan beberapa senyawa aktif yang terlibat. Formula jamu yang diteliti sebagai upaya menyembuhkan penyakit Diabetes Melitus (DM) Tipe II terdiri dari 4 tanaman yaitu Pare (Momordica charantia), Sembung (Blumea balsamifera), Bratawali (Tinospora crispa), dan Jahe (Zingiber officinale) berdasarkan hasil penelitian Nurishmaya tahun 2014 serta berdasarkan ramuan jamu yang sedang dikembangkan di Pusat Studi Biofarmaka, Bogor. Evaluasi senyawa yang berkaitan dengan DM Tipe II dilakukan dengan terlebih dahulu menambahkan 19 obat sintetis yang ditujukan untuk DM Tipe II dari basis data Drug Bank. Sedangkan setiap tanaman juga dicari masing-masing senyawa yang terkandung di dalamnya melalui basis data Dictionary of National Products (DNP) ChemnetBase dan Take Out “Jamu” of Knapsack. Didapatkan 291 senyawa dari tanaman Jahe, 18 senyawa dari tanaman Bratawali, 41 senyawa dari tanaman Sembung, dan 245 senyawa dari tanaman Pare. Total senyawa yang didapat dari keempat tanaman ada sebanyak 595. Kesemua senyawa baik yang berasal dari tanaman maupun dari obat sintetis dicari identitasnya lebih lanjut berupa struktur kimia senyawa dan aktifitas biologisnya berupa protein yang ditargetkan. Sayangnya, tidak semua senyawa tanaman diketahui protein targetnya, sehingga data penelitian yang akan diteliti lebih lanjut berkurang menjadi sebanyak 74 senyawa aktif yang terdiri dari 55 senyawa dari keempat tanaman dan 19 senyawa dari masing-masing obat sintetis. Setiap senyawa (74 senyawa) dapat menargetkan protein yang sama di dalam tubuh manusia. Diperoleh jumlah protein sebanyak 3059, total koneksi senyawa-protein sebanyak 4082 dan total protein target unik sebanyak 478.
Didapatkan sebanyak 1250 protein unik yang semula berjumlah 7400 koneksi senyawa-protein. Skor konkordan antara 74 senyawa dengan 1250 protein tersebut kemudian digunakan dalam analisis gerombol simultan antara senyawa dan protein target. Tujuan utama melakukan analisis gerombol simultan tersebut adalah melihat penggerombolan senyawa berdasarkan protein targetnya. Senyawa tanaman yang menggerombol bersama senyawa obat sintetis dicurigai berpotensi menjadi salah satu komposisi jamu anti-diabetes. Selain itu, analisis tersebut juga digunakan untuk mendeteksi protein target mana saja yang berperan sebagai penyebab penyakit diabetes tipe 2.
Selain itu, analisis komponen utama melalui plot dua dan tiga dimensi juga dilakukan untuk memastikan bahwa hasil yang ditunjukkan melalui analisis gerombol simultan tidak berbeda jauh. Sedangkan untuk melengkapi analisis gerombol simultan, dibuat sebuah jejaring kemiripan dari senyawa tanaman yang memiliki korelasi sangat tinggi terhadap senyawa obat sintetis. Berdasarkan ketiga analisis tersebut didapat bahwa 2 dari 3 senyawa Bratawali berpotensi sebagai obat antidiabetes karena menggerombol dan berkorelasi tinggi dengan senyawa aktif obat antidiabetes, sedangkan 11 dari 44 senyawa Jahe menggerombol bersama senyawa aktif obat antidiabetes tetapi hanya 5 senyawa Jahe yang berkorelasi kuat dengan senyawa aktif obat antidiabetes. Sembung dan Pare tidak menggerombol dengan senyawa aktif obat antidiabetes namun mungkin dapat berpotensi sebagai tanaman antidiabetes bila data yang digunakan lebih banyak atau dengan analisis yang lain.
SUMMARY
NURUL QOMARIASIH. Simultaneous Clustering Analysis and Network Pharmacology in the Determination of Active Compounds of Jamu for Diabetes Type 2. Supervised by BUDI SUSETYO and FARIT MOCHAMAD AFENDI.
During this time, the manufacture of drug in order to cure a disease is still target only one specific protein that causes the disease, which of course uses only one active compound. In fact, besides causing side effects, treatment of a disease needs a lot of protein targets at once. Thus, a paradigm shifts from "one-targets, one drug" to “network target, multi-components" becomes popular. This new paradigm has inspired several studies to produce herbal formulation, while the concept of herbal formulations requires some active compounds involved. Herbal formulas studied in an effort to cure the disease of Diabetes Mellitus (DM) Type II consists of 4 plants: bitter melon (Momordica charantia), Sembung (Blumea balsamifera), Bratawali (Tinospora crispa), and ginger (Zingiber officinale) which are based on research from Nurishmaya 2014 and based on herbs that are being developed at Biopharmaca Research Centre, Bogor, Indonesia. Evaluation of the compounds associated with Type II diabetes is done by first adding 19 synthetic drugs intended to Type II DM from Drug Bank database. While compound contained in each plant also searched through the database of Dictionary of National Products (DNP) ChemnetBase and Take Out "Jamu" of Knapsack. There are 291 compounds obtained from Jahe, 18 compounds from plant Bratawali, 41 compounds from Sembung, and 245 compounds from Pare. So the total compound obtained from the four plants have as many as 595. All of the compounds whether from plants or from synthetic drugs sought further their credentials in the form of the chemical structure of the compound and its biological activities in the form of a targeted protein. Unfortunately, not all plant compounds have target protein in the body, so that the research data that will be examined further reduced to as many as 74 active compounds consisting of 55 natural compounds from four plants and 19 respectively compound of synthetic drugs. Each of the compounds (74 compounds) can target the same protein in the human body. Total unique protein obtained amounted to 3059 from a total of compound-protein connections is 4082 and a total of 478 unique protein targets.
target proteins. The main purpose of the analysis of simultaneous clustering is to see the compound cluster based on the target protein. Plant compounds which are clustered together with synthetic drug compounds suspected as the potential one to become one of the anti-diabetic Jamu composition. In addition, the analysis was also used to detect any target protein that acts as a cause of type 2 diabetes. In addition, principal component analysis through two and three-dimensional plot is also made to ensure that the results shown by the analysis of simultaneous clustering does not differ too much. As for the complete analysis of simultaneous clustering, Similarity Network was made between plant compounds which have a very high correlation to the synthetic drug compounds. Based on the three analysis, there are found 2 of the 3 Bratawali compounds as a potential antidiabetic drugs because they were gathered and correlated with active compounds of antidiabetic drugs, while 11 of the 44 Jahe compounds clustered together with the active compound of antidiabetic drugs but only 5 compounds of Jahe are correlated with the active compounds of drugs antidiabetic. Sembung and Pare did not gather with the active compound of antidiabetic drugs but may be potentially as antidiabetic plant when more data are being used or with other analyzes.
© Hak Cipta Milik IPB, Tahun 2015
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Statistika
ANALISIS GEROMBOL SIMULTAN DAN JEJARING
FARMAKOLOGI PADA PENENTUAN SENYAWA AKTIF
JAMU ANTI DIABETES TIPE 2
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2015
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih merupakan bagian dari kajian program riset pertanian pada Program Studi Statistika, yaitu pengembangan data dan analisis Jamu di Indonesia. Tesis ini diberi judul Analisis Gerombol Simultan dan Jejaring Farmakologi pada Penentuan Senyawa Aktif Jamu Anti Diabetes Tipe 2.
Terima kasih penulis ucapkan kepada Bapak Dr. Ir. Budi Susetyo, M.Sc dan Bapak Dr. Farit Mochamad Afendi, S.Si, M.Si selaku pembimbing atas kesediaannya membagi ilmu kepada penulis serta dorongan semangat yang tak terhingga dalam penyusunan tesis ini. Terimakasih kepada Direktorat Jenderal Pendidikan Tinggi (Dikti) yang telah memberikan beasiswa pascasarjana program Magister kepada penulis. Ucapan terima kasih juga penulis sampaikan sebesar-besarnya kepada seluruh Dosen Program Studi Statistika IPB yang telah mendidik penulis selama di bangku kuliah hingga berhasil menyelesaikan studi, serta seluruh staf Program Studi Statistika IPB atas bantuan, pelayanan, dan kerjasamanya selama ini.
Ucapan terima kasih yang tulus dan penghargaan setinggi-tingginya kepada Bapak Jumiran dan Mamah Juju Juarsih selaku orang tua atas kasih sayangnya membesarkan dan senantiasa mendukung di setiap langkah positif yang penulis jalani, juga kedua adik laki-laki Hadi Johar Prakoso dan Muhammad Surya yang selalu menyemangati di sela-sela pengerjaan tesis ini. Tak lupa penulis ucapkan terima kasih kepada teman seperjuangan di riset pertanian Rizal Bakri, M.Si dan Nur Hilal A. Syahrir, M.Si atas kebersamaannya menghadapi masa-masa terindah maupun tersulit dalam pencarian data penelitian ini.
Terakhir penulis sampaikan terima kasih sedalam-dalamnya kepada seluruh mahasiswa Pascasarjana Program Studi Statistika angkatan 2013 atas kebersamaannya yaitu kenangan bahagia, kenangan sedih, haru, dan pengorbanan yang kita perjuangkan bersama selama menuntut ilmu di bangku kuliah, serta semua pihak yang telah banyak membantu dan tak sempat penulis sebutkan satu per satu.
Semoga karya ilmiah ini dapat bermanfaat.
Bogor, November 2015
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
1 PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 2
2 TINJAUAN PUSTAKA 3
Diabetes Melitus (DM) Tipe 2 3
Jejaring Farmakologi 3
DrugCIPHER 3
DrugCIPHER-CS 4
DrugCIPHER-GR 6
Unweighted Breadth-First 6
Nilai konkordan 6
Analisis Komponen Utama 7
Analisis Gerombol Simultan 7
Algoritma Coupled Two-Way Clustering analysis (CTWC) 8
Metode Penggerombolan 10
Pseudo-F 11
Jejaring kemiripan (Similarity Network) 12
3 METODE PENELITIAN 13
Data 13
Metode analisis 14
4 HASIL DAN PEMBAHASAN 16
Jejaring Farmakologi 16
DrugCIPHER 16
Analisis komponen utama 19
Penggerombolan simultan 2 dimensi 20
Jejaring kemiripan 23
5 SIMPULAN DAN SARAN 24
Simpulan 24
Saran 24
DAFTAR PUSTAKA 25
LAMPIRAN 26
DAFTAR TABEL
1 Set biner antara dua senyawa 4
2 Perbandingan antara analisis gerombol klasik dan analisis gerombol
simultan (Charrad dan Ahmed 2011). 8
3 Algoritma analisis gerombol simultan 9
4 Daftar pangkalan data publikasi ilmiah 13
5 Nilai pseudo-F dari berbagai metode dan jumlah gerombol 20 6 Protein penyebab diabetes yang bersesuaian pada kotak horizontal 22
DAFTAR GAMBAR
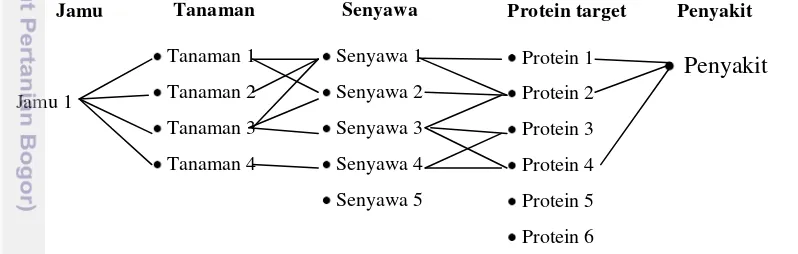
1 Jejaring farmakologi jamu 3
2 Dua struktur molekul senyawa 4
3 Prinsip drugCIPHER 5
4 Metode Breadth First Search 6
5 Reorganisasi oleh permutasi dari baris dan kolom (Govaert 1995) 8 6 Penggerombolan berhirarki 2 dimensi oleh Parsons et al. (2003) 10 7 Jejaring antara senyawa (kuning) dengan protein target (merah) 16 8 Kemiripan antar senyawa berdasarkan struktur kimia yang dihitung
melalui koefisien kemiripan Tanimoto 17
9 Jejaring interaksi antar protein target 17
10 Skor konkordan senyawa dengan protein target 18
11 Plot komponen utama 19
12 Hasil gerombol simultan Ward.D 21
13 Jejaring kemiripan senyawa sintetis dengan senyawa tanaman 23
DAFTAR LAMPIRAN
1 Senyawa sintetis antidiabetes yang telah terdaftar di Food and Drug Asociation (FDA) dengan sumber basis data Drugbank 26 2 Senyawa yang berasal dari tanaman yang diduga sebagai antidiabetes 27
3 Syntax R untuk analisis data 29
1
1
PENDAHULUAN
Latar Belakang
Seiring perkembangan jaman, kecenderungan orang memilih bahan alami untuk konsumsi sehari-hari maupun pengobatan semakin berkembang. Bidang pengobatan juga memiliki pemikiran yang mengacu pada perubahan paradigma dan menjadi topik yang mulai banyak diteliti saat ini, yakni dari "one drug, one-target" menjadi “multi-components, network target". "one drug, one-target" mengacu kepada obat kimia sintetis yang berisi satu senyawa aktif untuk mengobati satu keluhan penyakit tertentu. Namun terkadang obat seperti ini menimbulkan efek samping. Sedangkan “multi-components, network target" diterapkan salah satunya pada jamu yang memiliki konsep keterlibatan beberapa senyawa aktif untuk menargetkan beberapa protein penyebab penyakit.
Langkah pertama dalam mengidentifikasi target untuk membuat obat kimia sintetis dalam bidang farmasi yaitu penemuan obat berdasarkan target dimulai dengan mengidentifikasi fungsi dari target yang mungkin dan perananya dalam penyakit. Langkah kedua adalah validasi target, dalam langkah ini diperlukan demonstrasi bahwa molekul target secara langsung terlibat dalam proses penyakit. Langkah selanjutnya adalah identifikasi senyawa aktif yang didefinisikan sebagai molekul yang menunjukkan aktifitas biologis yang signifikan dalam uji skrining, yakni materi baru yang menghubungkan struktur kimia untuk modulasi target. Hingga langkah terakhir, yaitu langkah ke-11 yang melibatkan pengujian klinikal atas konsep obat yang telah dibuat. Berdasarkan serangkaian langkah tersebut, maka akan didapatkan molekul target (protein) yang berperan terhadap penyebab suatu penyakit tertentu dan senyawa aktif obat sintetis yang berfungsi untuk menargetkan molekul tersebut. Fungsi dari senyawa aktif adalah mengunci protein target dalam rangka penyembuhan suatu penyakit, atau keduanya diistilahkan sebagai “lock and key”.
2
untuk masing-masing senyawa. Pengelompokkan tersebut melibatkan senyawa aktif obat kimia yang telah disetujui oleh Food and Drug Administration (FDA) Amerika Serikat dan sudah diketahui fungsinya sebagai obat antidiabetes, sehingga akan didapat senyawa aktif tanaman obat yang memiliki kemiripan dengan senyawa obat kimia antidiabetes berdasarkan protein target. Metode pengelompokkan yang dipakai adalah metode complete linkage, single linkage, ward.D, ward.D2, dan average linkage. Beberapa metode tersebut akan dievaluasi menggunakan Pseudo-F untuk menentukan metode mana yang paling baik digunakan dan berapa gerombol yang sebaiknya diterapkan. Serta yang terakhir akan divisualisaskan jejaring antara senyawa aktif obat kimia sintetis dan senyawa aktif tanaman obat berdasarkan kesamaan target menggunakan Cytoscape. Hasil akhir dari analisis gerombol dua dimensi antara senyawa (senyawa aktif tanaman obat dan senyawa aktif obat sintetis) dengan protein target yaitu menghasilkan senyawa aktif tanaman yang memiliki kemiripan protein target dengan senyawa aktif obat sintetis antidiabetes.
Tujuan Penelitian
3
Penyakit
Penyakit 1
2
TINJAUAN PUSTAKA
Diabetes Melitus (DM) Tipe 2
Diabetes adalah penyakit kronis yang terjadi baik ketika pankreas tidak menghasilkan cukup insulin atau ketika tubuh tidak dapat secara efektif menggunakan insulin yang dihasilkan. Insulin merupakan hormon yang mengatur gula darah (World Health Organization 1999). Efek jangka panjang dari Diabetes adalah kerusakan serius pada sistem tubuh, terutama syaraf dan pembuluh darah. Sebagian besar DM Tipe II merupakan hasil dari kelebihan berat badan dan merupakan 90% dari penderita diabetes seluruh dunia.
Jejaring Farmakologi
Berdasarkan kemajuan dalam biologi kimia dan ilmu jaringan, jejaring farmakologi adalah pendekatan baru khusus untuk penemuan obat. Pendekatan ini melibatkan penerapan analisis jaringan untuk menentukan set protein yang paling penting dalam penyakit, dan kemudian biologi kimia untuk mengidentifikasi molekul atau senyawa yang mampu menargetkan set protein. Jejaring farmakologi berbeda dari pendekatan obat konvensional, yang umumnya didasarkan pada penargetan yang sangat spesifik dari protein tunggal (Zhang Gui-biao et al 2013)
Jejaring farmakologi sebuah jamu terdiri dari gabungan beberapa tanaman. Setiap tanaman mengandung beberapa senyawa dan masing-masing senyawa mampu menargetkan beberapa protein dalam tubuh. Sementara itu, suatu penyakit juga diakibatkan oleh aktivitas menyimpang beberapa protein dalam tubuh.
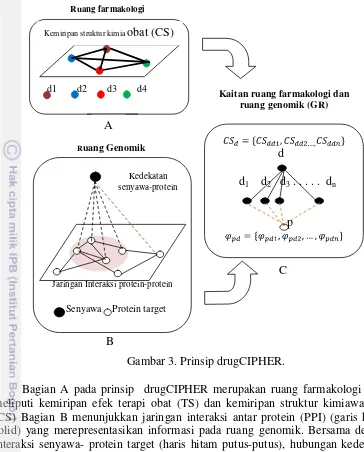
DrugCIPHER
Langkah pertama dalam menganalisis data jaringan adalah dengan metode DrugCIPHER. Metode ini merupakan ulasan tentang pengetahuan mengenai penggunaan farmasi (Wotring dan Virginia 2012). Lebih fokus Zhao dan Li (2010) mengartikan metode ini sebagai sebuah kerangka kerja komputasi berbasis
Protein target
5
Gambar 3. Prinsip drugCIPHER.
Bagian A pada prinsip drugCIPHER merupakan ruang farmakologi yang meliputi kemiripan efek terapi obat (TS) dan kemiripan struktur kimiawa obat (CS) Bagian B menunjukkan jaringan interaksi antar protein (PPI) (garis hitam solid) yang merepresentasikan informasi pada ruang genomik. Bersama dengan interaksi senyawa- protein target (haris hitam putus-putus), hubungan kedekatan (garis cokelat putus-putus) didefinisikan sebagai asosiasi senyawa dengan protein tertentu. Bagian C menunjukkan senyawa d dan protein p, dua vektor kemiripan d di dalam ruang farmakologi dan satu vektor kedekatan p adalah φp.
Pendekatan fingerprint biner merupakan salah satu metode evaluasi kemiripan. Fingerprint biner dari dua molekul (Tabel 1) dapat dibandingkan untuk dikuantifikasi jaraknya menggunakan koefisien kemiripan Tanimoto yaitu:
adalah suatu molekul tertentu, dan merupakan representasi biner
Kemiripan struktur kimia obat (CS)
6
Asosiasi ruang farmakologi dengan ruang genomik mendefinisikan kedekatan antara protein p dengan senyawa d pada basis jaringan PPI yaitu:
merupakan protein target yang dituju oleh senyawa . merupakan
jarak terpendek antara dan pada jaringan PPI pada ruang genomik
berdasarkan teori graf algoritme Dijktra. digunakan untuk mengkonversi
jarak protein-protein menjadi kedekatan protein-protein. Persamaan ini
menunjukkan bahwa kedekatan antara obat dan protein sama dengan
penjumlahan dari kedekatan antara protein dengan semua protein target dari
obat . Jika jarak dua protein sangat jauh, didefiniskan . Sedangkan
jaringan PPI merupakan data interaksi biner antar protein dalam tubuh manusia.
Unweighted Breadth-First
Breadth-first search (BFS) adalah graf dasar yang sering digunakan sebagai blok-blok pembangun pada banyak algoritma graf yang lebih komplek (Beamer et al. 2013). Metode ini akan mengunjungi terlebih dahulu semua node pada level n sebelum mengunjungi node-node pada level n+1. Pencarian dimulai dari node akar terus ke level ke-1 dari kiri ke kanan (Gambar 4).
Gambar 4. Metode Breadth First Search (Indrawati et al. 2011).
Keuntungan pertama dari metode ini yaitu tidak akan menemui jalan buntu. Keuntungan kedua adalah jika ada satu solusi maka breadth-first search akan menemukannya, namun jika ada lebih dari satu solusi, maka solusi minimum akan ditemukan. Selain keuntungan, metode ini juga memiliki kekurangan, metode ini akan menyimpan semua node dalam satu pohon, sehingga membutuhkan memori yang cukup banyak. Waktu pengerjaannya pun cukup lama, karena akan menguji n level untuk mendapatkan solusi pada level ke-(n+1).
Nilai konkordan
Nilai konkordan yang akan digunakan sebagai nilai dalam
7
Nilai konkordan ini menjelaskan derajat kontribusi dari senyawa dengan
vektor kedekatan pada protein pada persamaan sebebelumnya. Nilai ini
memiliki rentang nilai -1 hingga 1. Ketika hubungan antara dengan
positif, semakin dekat kemiripan senyawa i dengan senyawa j, maka senyawa i
akan semakin menargetkan protein target dari senyawa j.
Analisis Komponen Utama
Analisis komponen utama banyak digunakan pada berbagai bidang ilmu. Tujuan utama dari analisis komponen utama adalah mereduksi dimensi data besar dan saling berkorelasi menjadi dimensi yang lebih kecil dan saling bebas. Peubah baru ini merupakan kombinasi linier dari peubah asal dan disebut dengan komponen utama (Principal Component). Komponen utama ini dapat dibentuk berdasarkan matriks ragam peragam ataupun matriks korelasi.
Sebelum analisis penggerombolan simultan dua dimensi, data nilai konkordan dilihat penyebarannya pada plot skor komponen utama. Hal ini untuk melihat penyebaran titik-titik senyawa tanaman terhadap senyawa obat sintetis setelah jumlah peubah (protein) direduksi menjadi beberapa komponen. Penentuan jumlah komponen yang diambil dilihat melalui scree plot. Pembentukkan komponen utama berdasarkan matriks ragam peragam dapat dilakukan jika satuan pengukuran setiap peubah sama. Misalkan merupakan matriks ragam peragam , dengan pasangan akar ciri dan vektor
ciri , dengan maka
komponen utama ke-i didefinisikan sebagai berikut:
Selain itu, nilai dari proporsi kontribusi keragaman komponen utama ke-i , yaitu:
(Johnson dan Wichern 2007).
Analisis Gerombol Simultan
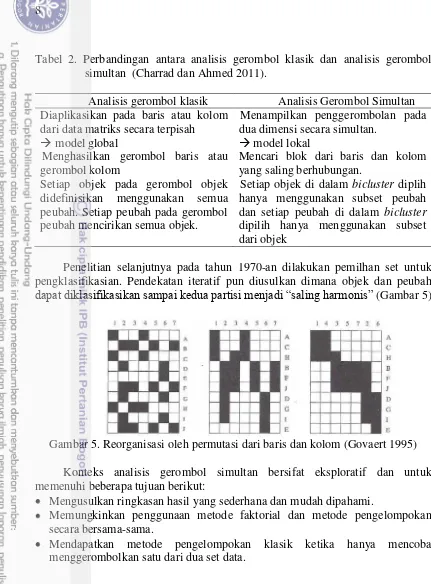
Penggerombolan simultan biasa disebut biclustering, co-clustering, 2-way clustering atau block clustering. Tujuannya adalah untuk menemukan submatrik, dimana subgrup baris dan subgrup kolom memperlihatkan korelasi tinggi. Metode ini banyak diaplikasikan pada bidang bioinformasi, web-mining, text mining dan analisis social network (Charrad dan Ahmed 2011). Perbandingan dari kedua metode penggerombolan dijelaskan pada Tabel 2.
8
Tabel 2. Perbandingan antara analisis gerombol klasik dan analisis gerombol simultan (Charrad dan Ahmed 2011).
Analisis gerombol klasik Analisis Gerombol Simultan Diaplikasikan pada baris atau kolom
dari data matriks secara terpisah model global
Menampilkan penggerombolan pada dua dimensi secara simultan.
model lokal Menghasilkan gerombol baris atau
gerombol kolom
Mencari blok dari baris dan kolom yang saling berhubungan.
Setiap objek pada gerombol objek didefinisikan menggunakan semua peubah. Setiap peubah pada gerombol peubah mencirikan semua objek.
Setiap objek di dalam bicluster diplih hanya menggunakan subset peubah dan setiap peubah di dalam bicluster dipilih hanya menggunakan subset dari objek
Penelitian selanjutnya pada tahun 1970-an dilakukan pemilhan set untuk pengklasifikasian. Pendekatan iteratif pun diusulkan dimana objek dan peubah dapat diklasifikasikan sampai kedua partisi menjadi “saling harmonis” (Gambar 5).
Gambar 5. Reorganisasi oleh permutasi dari baris dan kolom (Govaert 1995) Konteks analisis gerombol simultan bersifat eksploratif dan untuk memenuhi beberapa tujuan berikut:
Mengusulkan ringkasan hasil yang sederhana dan mudah dipahami.
Memungkinkan penggunaan metode faktorial dan metode pengelompokan secara bersama-sama.
Mendapatkan metode pengelompokan klasik ketika hanya mencoba menggerombolkan satu dari dua set data.
Algoritma Coupled Two-Way Clustering analysis (CTWC)
9
pasangan subset objek dan fitur melalui proses iteratif dengan menguji hanya pada submatriks baris (atau kolom) yang dimiliki oleh gen (atau sampel) yang telah teridentifikasi pada iterasi sebelumnya.
Proses diawali dengan matriks penuh yaitu set semua gen (g0) dan semua sampel (s0) yang digunakan sebagai fitur dan objek untuk dilakukan standard two-way clustering. Gerombol stabil dari gen dan sampel pada langkah pertama dinotasikan sebagai g1 dan s1. Setiap pasangan (gin dan sjm) akan dilakukan
two-way clustering. Hasil gerombol stabil dari gen (atau sampel) dinotasikan gk2 atau
sl2. Setiap gerombol disimpan di dalam satu dari dua “gerombol stabil register”,
gerombol gen di register G dan gerombol sampel di S. Bersama dengan gerombol baru, sebuah pointer dipasang untuk mengidentifikasi gerombol sebelumnya (gin
dan sjm) yang digunakan sebagai set objek dan fitur dalam proses penggerombolan
yang dihasilkan. Langkah tersebut terus berulang menggunakan pasangan semua gerombol yang telah ditemukan sebelumnya. Proses dihentikan ketika telah sesuai dengan posisi gerombol semula.
Tabel 3. Algoritma analisis gerombol simultan.
Algoritma Penerapan Pendekatan Tipe data
Two-way
splitting Lainnya Variance Minimization Kontinu
CROEUC Lainnya Two-way clustering Kontinu
CROKI2 Lainnya Two-way clustering Kategori
CROBIN Lainnya Two-way clustering Biner
CTWC Bioinformatika Two-way clustering Kontinu Plaid Models Bioinformatika
Probabilistic and
generative Kontinu δ-biclusters Bioinformatika Variance Minimization Kontinu δ -ks patterns Bioinformatika Variance Minimization Kontinu ITWC Bioinformatika Two-way clustering Kontinu DCC Bioinformatika Two-way clustering Kontinu OPSM Bioinformatika FLOC Bioinformatika Variance Minimization Kontinu Spectral Bioinformatika
Motif and pattern
recognition Kontinu ITWC Text Mining Bi-partite Graph Kontinu BGSP Text Mining Bi-partite Graph Kontinu cHawk Bioinformatika
Probabilistic and
generative Kategori Block-EM Lainnya Two-way clustering Kontinu
biner Block-CEM Lainnya Two-way clustering Kontinu
10
Sebagai gambaran mengenai hasil analisis gerombol simultan, Parsons et al. (2003) melakukan analisis gerombol simultan berhirarki dua-dimensi dari senyawa dan gen target yang ditampilkan seperti pada Gambar 6. Interaksi senyawa-gen target diwakili dengan warna merah. Interaksi gen diplot pada sumbu horisontal, dengan pohon gerombol gen di atas. Senyawa diplot pada sumbu vertikal, dengan pohon gerombol di sisi kiri plot. Senyawa yang mengelompok dengan gen target dan jalur hubungan keduanya ditandai oleh warna yang sama.
Gambar 6. Penggerombolan berhirarki 2 dimensi oleh Parsons et al. (2003)
Metode penggerombolan
Berbagai metode penggerombolan dapat diterapkan pada kerangka CTWC. Namun metode paling baik untuk menganalisis ekspresi gen harus memiliki beberapa sifat-sifat yaitu jumlah gerombol tidak ditentukan sebelumnya (seperti SOMS dan K-means) melainkan melalui dendogram, stabil terhadap noise, memiliki mekanisme untuk mengidentifikasi kestabilan dan kekekaran gerombol, serta dapat mengidentifikasi kepadatan dari set data (Getz et al. 2000). Beberapa metode dasar yang akan diterapkan pada penelitian ini terdiri dari metode penggerombolan agglomerative (metode penggabungan) yaitu linkage methods seperti single linkage (jarak minimum atau tetangga terdekat), complete linkage (jarak terjauh atau tetangga terjauh), dan average linkage (jarak rata-rata). Selain itu juga menggunakan metode Ward yang meminimumkan “loss of information” dari gabungan dua kelompok data, yaitu didefinisikan sebagai peningkatan kriteria jumlah kuadrat galat (ESS).
Penyelesaian metode single linkage mulanya adalah menentukan jarak terkecil lalu gabung dua objek terdekat U dan V dalam satu gerombol (UV). Jarak UV dengan gerombol lain W yaitu:
.
dan masing-masing merupakan jarak antara tetangga terdekat gerombol U-W dan V-W. Serupa pada single linkage, complete linkage berbeda pada penentuan jarak UV dengan gerombol lain W yaitu:
11
Metode average linkage, jarak UV dengan gerombol lain W ditentukan sebagai:
adalah jarak antara objek i dalam gerombol (UV) dan objek k pada gerombol W, dan masing-masing adalah jumlah objek pada gerombol UV dan gerombol W. Sedangkan akan ada dua algoritma metode Ward yang digunakan yaitu Ward.D dan Ward.D2. Keduanya memiliki konsep yang sama yaitu meminimumkan perubahan ragam atau jumlah kuadrat galat. Fion Murtagh tahun 1985 menyatakan formula untuk memperbaharui gerombol pada Ward.D yaitu:
berdasarkan jarak kuadrat Euclidean antar objek, maka
sedangkan . Pada Ward.D2, formula untuk memperbaharui gerombol yaitu:
seperti pada Ward.D, berdasarkan jarak kuadrat Euclidean antar objek, maka
sedangkan .
Semua metode di atas dicobakan untuk menggerombolkan senyawa dan protein target berdasarkan nilai konkordan antara drugCIPHER-CS dan vektor kedekatan senyawa-protein target . Lalu masing-masing metode serta jumlah
gerombol yang terbentuk dievaluasi menggunakan pseudo-F. Pseudo-F
merupakan analisis untuk mendapatkan suatu nilai perbandingan keragaman antar gerombol dengan keragaman di dalam gerombol.
Pseudo-F
Calinski dan Harabasz (1974) membuat suatu index sebagai indikasi terhadap jumlah gerombol yang benar. Indeks Calinski dan Harabasz yang disebut Variance Ratio Criterion (VRC) didefinisikan sebagai:
kriteria ini serupa dengan uji statistik F pada analisis univariat, dimana n dan k masing-masing adalah jumlah total observasi dan jumlah gerombol, WGSS merupakan jumlah kuadrat dalam gerombol yang didefinisikan sebagai:
sedangkan BGSS merupakan jumlah kuadrat antar gerombol yaitu:
12
Tujuan indeks tersebut yaitu meminimumkan WGSS dan memaksimumkan BGSS, sehingga semakin tinggi nilai suatu index mengindikasikan jumlah gerombol yang lebih baik. Hasil penggerombolan terbaik berdasarkan Pseudo-F digunakan untuk mendapatkan jejaring kemiripan antar senyawa yang merupakan langkah akhir dari analisis penentuan senyawa aktif tanaman untuk antidiabetes.
Jejaring kemiripan (Similarity Network)
Dua objek yang benar-benar mirip atau identik yaitu ketika sjj=1
sedangkan ketidakmiripan ditunjukkan sjj=0. Kemiripan merupakan kebalikan dari
13
3
METODE PENELITIAN
Data
Data yang digunakan merupakan daftar bahan aktif ramuan jamu yang sedang dikembangkan di Pusat Studi Biofarmaka untuk pengobatan diabetes mellitus tipe II yang terdiri dari Pare (Momordica charantia), Sembung (Blumea balsamifera), Bratawali (Tinospora crispa), dan Jahe (Zingiber officinale). Daftar lebih rinci mengenai bahan aktif dari tanaman-tanaman tersebut dicari lebih lanjut melalui berbagai publikasi ilmiah yang dilakukan selama bulan Januari hingga Mei 2015 seperti tertera pada Tabel 4.
Tabel 4. Daftar pangkalan data publikasi ilmiah Data Pangkalan
Data
Informasi yang diperoleh
dari pangkalan data Hasil penelusuran data Senyawa Take Out
Pubchem Daftar senyawa (tanaman & sintetis) yang memliki
14
Metode Analisis
Tahapan yang dilakukan dalam penelitian ini adalah sebagai berikut: I. Pendugaan protein target
Tahap ini menggunakan metode drugCIPHER dengan langkah-langkah sebagai berikut (Zhao dan Li 2010):
1. Mendapatkan matriks drugCIPHER-CS
a. Menggabungkan data 55 senyawa tanaman (Lampiran 2) dan 19 senyawa obat (Lampiran 1) menjadi total keseluruhan 74 senyawa dilengkapi dengan Simplified Molecular-Input Line-Entry System (Smiles).
b. Membuat set biner sepanjang 4860 bit untuk masing-masing senyawa berdasarkan fingerprint KR.
c. Menghitung nilai kemiripan struktur molekul antar senyawa menggunakan koefisien Tanimoto (Sharma dan Pranit 2011):
sehingga akan dihasilkan matriks kemiripan senyawa (0<T<1) berskala interval berukuran 74×74.
2. Menghitung drugCIPHER-GR
a. Menghitung jarak terdekat antara protein yang menjadi target (479 protein unik dari senyawa tanaman dan 43 protein dari obat) dengan seluruh protein dalam tubuh (9673 protein) yang terdokumentasi pada database HPRD menggunakan teori graf. Berdasarkan sejumlah protein tersebut diambil hanya protein uniknya saja, sehingga matriks jarak terpendek berskala rasio yang terbentuk berukuran 505×9673.
b. Menghitung drugCIPHER-GR yaitu:
Lppk merupakan jarak terpendek yang dihasilkan dari poin (a).
digunakan untuk mengkonversi jarak protein-protein menjadi
kedekatan protein-protein Dari perhitungan tersebut maka akan
dihasilkan matriks kedekatan senyawa dengan protein berskala rasio berukuran 74×9673.
3. Menghitung nilai konkordan antara drugCIPHER-CS dengan drugCIPHER-GR:
Nilai yang dihasilkan dari perhitungan di atas adalah sebuah matriks berukuran 74 senyawa × 9673 protein. Bagi masing-masing senyawa, dipilih 100 protein target dengan nilai konkordan tertinggi. Total protein yang didapat sebanyak 7400 kemudian dipilih lagi hanya yang uniknya saja, sehingga akan dihasilkan matriks berukuran 74 senyawa×1250 protein target unik.
15
II. Penggerombolan Simultan 2 Dimensi (Govaert 1995).
Sebanyak 100 protein target dari 74 senyawa yang telah didapat dari tahap sebelumnya, dipilih nilai konkordan dari protein uniknya saja, yaitu sebanyak 1.250 protein. Lalu, nilai konkordan ukuran 74×1250 dibuat penggerombolan simultan 2 dimensinya menggunakan jarak Euclid dan menggunakan metode Single Linkage, Complete Linkage, Average Linkage, serta Ward.D dan Ward.D2 (Johnson dan Wichern 2002). Sedangkan algoritma yang dipakai adalah Coupled Two-Way Clustering Analysis (Getz et al. 2000). Hasil penggerombolan dievaluasi menggunakan Pseudo-F atau Variance Ratio Criterion (VRC):
Metode dan jumlah gerombol dengan nilai pseudo-F tertinggi akan dipilih (Calinski dan Harabasz 1974). Plot skor komponen utama dan similarity network terhadap senyawa juga ditampilkan untuk mendukung hasil gerombol simultan. III. Penentuan senyawa aktif dan tanaman antidiabetes dari hasil penggerombolan
simultan 2 dimensi, plot PCA dan similarity network.
Berdasarkan hasil penggerombolan simultan 2 dimensi, dapat dilihat senyawa-senyawa tanaman yang berperan sebagai antidiabetes serta tanaman asalnya. Pada saat yang sama, dapat dilihat pula protein apa saja yang ditargetkan oleh senyawa tersebut. Sedangkan plot PCA melihat kecenderungan pengelompokkan senyawa dan similarity network memberikan gambaran kedekatan kemiripan antara senyawa sintetis dengan senyawa tanaman berdasarkan nilai similarity coefficient.
16
4
HASIL DAN PEMBAHASAN
Jejaring Farmakologi
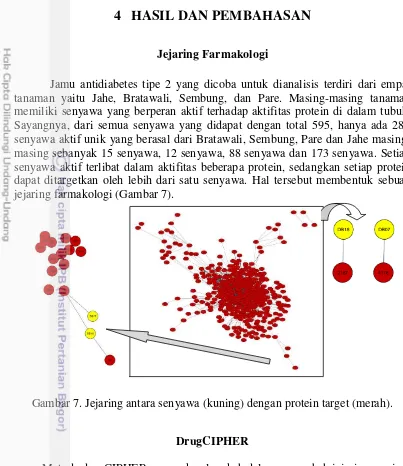
Jamu antidiabetes tipe 2 yang dicoba untuk dianalisis terdiri dari empat tanaman yaitu Jahe, Bratawali, Sembung, dan Pare. Masing-masing tanaman memiliki senyawa yang berperan aktif terhadap aktifitas protein di dalam tubuh. Sayangnya, dari semua senyawa yang didapat dengan total 595, hanya ada 288 senyawa aktif unik yang berasal dari Bratawali, Sembung, Pare dan Jahe masing-masing sebanyak 15 senyawa, 12 senyawa, 88 senyawa dan 173 senyawa. Setiap senyawa aktif terlibat dalam aktifitas beberapa protein, sedangkan setiap protein dapat ditargetkan oleh lebih dari satu senyawa. Hal tersebut membentuk sebuah jejaring farmakologi (Gambar 7).
Gambar 7. Jejaring antara senyawa (kuning) dengan protein target (merah).
DrugCIPHER
17
Pengukuran kemiripan senyawa berdasarkan struktur kimia ini penting dilakukan karena menurut Johnson dan Maggiora (1990), senyawa yang memiliki struktur kimia yang mirip akan memiliki sifat biologis yang mirip pula, sehingga senyawa yang memiliki kemiripan akan cenderung mengikat protein yang sama. Pengukuran kemiripan dilakukan dengan menghitung koefisien Tanimoto, jangkauan nilainya antara 0 hingga 1. Senyawa dengan kemiripan rendah ditunjukkan dengan warna biru pekat, sedangkan kemiripan tinggi ditunjukkan oleh warna yang lebih terang (Gambar 8).
Gambar 8. Kemiripan antar senyawa berdasarkan struktur kimia yang dihitung melalui koefisien kemiripan Tanimoto.
Garis diagonal pada Gambar 7 merupakan warna yang sangat terang, hal tersebut karena senyawa memiliki kemiripan tinggi terhadap dirinya sendiri. Selain itu, dapat dilihat bahwa senyawa ke-4 memiliki kemiripan yang cukup tinggi dengan senyawa 5 hingga senyawa 9 dan memiliki nilai kemiripan mendekati 1.
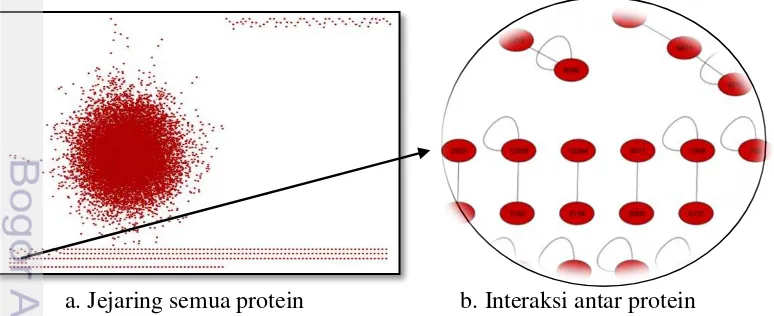
Langkah selanjutnya yaitu pada ruang genomik (drugCIPHER-GR) menghitung jarak antar protein dalam tubuh menggunakan analisis graf dengan metode Unweighted breadth-first search (Gambar 9).
18
Semakin dekat jarak antar protein, maka akan semakin cepat suatu senyawa melewati jalur tersebut untuk menargetkan protein tertentu. Terdapat sebanyak 39240 interaksi antar 9673 protein unik.
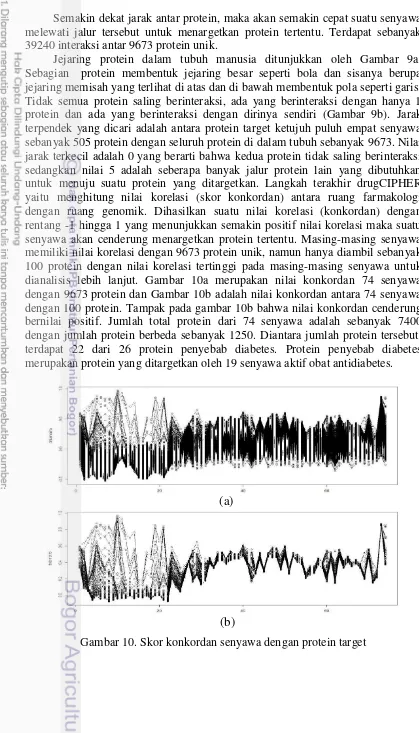
Jejaring protein dalam tubuh manusia ditunjukkan oleh Gambar 9a. Sebagian protein membentuk jejaring besar seperti bola dan sisanya berupa jejaring memisah yang terlihat di atas dan di bawah membentuk pola seperti garis. Tidak semua protein saling berinteraksi, ada yang berinteraksi dengan hanya 1 protein dan ada yang berinteraksi dengan dirinya sendiri (Gambar 9b). Jarak terpendek yang dicari adalah antara protein target ketujuh puluh empat senyawa sebanyak 505 protein dengan seluruh protein di dalam tubuh sebanyak 9673. Nilai jarak terkecil adalah 0 yang berarti bahwa kedua protein tidak saling berinteraksi sedangkan nilai 5 adalah seberapa banyak jalur protein lain yang dibutuhkan untuk menuju suatu protein yang ditargetkan. Langkah terakhir drugCIPHER yaitu menghitung nilai korelasi (skor konkordan) antara ruang farmakologi dengan ruang genomik. Dihasilkan suatu nilai korelasi (konkordan) dengan rentang -1 hingga 1 yang menunjukkan semakin positif nilai korelasi maka suatu senyawa akan cenderung menargetkan protein tertentu. Masing-masing senyawa memiliki nilai korelasi dengan 9673 protein unik, namun hanya diambil sebanyak 100 protein dengan nilai korelasi tertinggi pada masing-masing senyawa untuk dianalisis lebih lanjut. Gambar 10a merupakan nilai konkordan 74 senyawa dengan 9673 protein dan Gambar 10b adalah nilai konkordan antara 74 senyawa dengan 100 protein. Tampak pada gambar 10b bahwa nilai konkordan cenderung bernilai positif. Jumlah total protein dari 74 senyawa adalah sebanyak 7400 dengan jumlah protein berbeda sebanyak 1250. Diantara jumlah protein tersebut, terdapat 22 dari 26 protein penyebab diabetes. Protein penyebab diabetes merupakan protein yang ditargetkan oleh 19 senyawa aktif obat antidiabetes.
(a)
(b)
20
Berdasarkan grafik scree plot, komponen yang harus diambil sebanyak 5 atau 6 dimana plot terlihat mulai stabil dan persentase keragaman yang dapat dijelaskan cukup besar yaitu 91.51% hingga 93.44%, namun karena keterbatasan dimensi, plot dibuat hanya untuk dua komponen (Gambar 11b) dengan persentase kumulatif keragaman sebesar 76.79% dan tiga komponen (Gambar 11c) dengan keragaman yang mampu dijelaskan sebesar 83.97%. Baik plot 2 maupun 3 dimensi mengarah pada kesimpulan yang tidak berbeda jauh. Keduanya menunjukkan bahwa senyawa obat sintetis terlihat berkerumun; ungu pada plot 2 dimensi dan biru tua pada plot 3 dimensi. Sementara senyawa terdekat dari obat sintetik yaitu 2 senyawa dari tanaman Bratawali; merah pada plot 2 dimensi dan kuning pada plot 3 dimensi. Pada plot 2 dimensi, beberapa senyawa dari Jahe (warna hijau) terlihat berdekatan dengan senyawa obat sintetis, tetapi pada plot 3 dimensi senyawa Jahe (warna biru muda) terlihat cukup jauh. Hasil plot komponen utama ini merupakan pendeskripsian awal mengenai keadaan pengelompokkan senyawa berdasarkan komponen utama protein, lebih jauh mengenai analisis gerombol senyawa sekaligus protein yang berasosiasi dianalisis menggunakan penggerombolan simultan dua dimensi.
Penggerombolan simultan 2 dimensi
Penggerombolan simultan berhirarki dua dimensi dilakukan dengan algoritma Coupled Two-Way Clustering (CTWC) dan berbagai metode penggerombolan yang menghasilkan pengelompokkan senyawa dan protein yang berbeda (Lampiran 4-8).
Dendogram pada sisi vertikal di masing-masing hasil penggerombolan menunjukkan hasil penggerombolan protein, sedangkan dendogram pada sisi horizontal merupakan hasil penggerombolan bagi senyawa. Setiap metode menghasilkan penggerombolan yang berbeda, sehingga dilakukan evaluasi terhadap metode dan jumlah gerombol yang terbaik menggunakan nilai pseudo-F seperti ditampilkan pada Tabel 5.
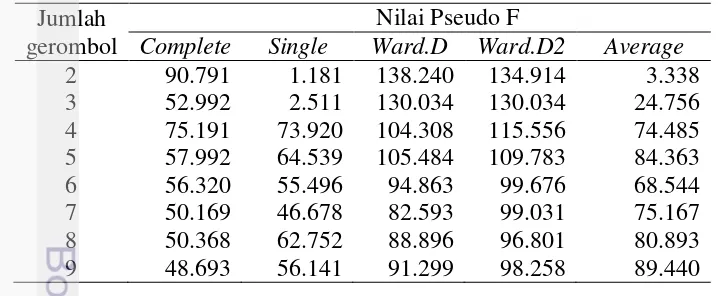
Tabel 5. Nilai pseudo-F dari berbagai metode dan jumlah gerombol Jumlah
gerombol
Nilai Pseudo F
Complete Single Ward.D Ward.D2 Average
2 90.791 1.181 138.240 134.914 3.338
21
gerombol sebanyak 2 dan 89.4403 dengan jumlah gerombol sebanyak 9. Nilai pseudo-F terbesar adalah untuk metode Ward.D sebesar 138.2402 dengan jumlah gerombol sebanyak 2.
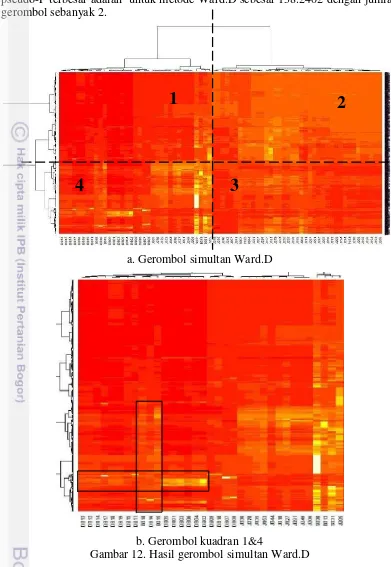
a. Gerombol simultan Ward.D
b. Gerombol kuadran 1&4
Gambar 12. Hasil gerombol simultan Ward.D
Gambar 12a menampilkan bahwa gerombol senyawa sebelah kiri cenderung bersesuaian dengan aktivitas protein di kuadran 4, sedangkan gerombol senyawa sebelah kanan lebih banyak bersesuaian dengan aktivitas protein di kuadran 2. Berdasarkan tujuan yang ingin dicapai, maka lebih ditekanka untuk mengamati penggerombolan simultan yang terdapat semua senyawa sintetis di dalamnya,
1
2
22
yaitu gerombol sebelah kiri (Gambar 12b). Terdapat total 35 senyawa yang menggerombol terdiri dari semua senyawa sintetis, 3 senyawa Bratawali, 11 senyawa Jahe dan 2 senyawa Sembung. Senyawa Bratawali B015 dan B018 menggerombol dekat dengan senyawa sintetis, sedangkan B013 menggerombol berdekatan dengan semua senyawa Jahe dan semua senyawa Sembung. Analisis gerombol simultan memberikan submatrik subset yang menyatakan aktifitas protein-protein tertentu hanya dipengaruhi oleh beberapa senyawa tertentu pula. Terdapat sebuah kotak kecil vertikal (Gambar 13b) yang menunjukkan bahwa senyawa aktif Bratawali B015 dan B018 serta senyawa aktif sintetis DB16 terlibat dalam aktifitas beberapa protein, yaitu tidak kurang dari 566 protein (dicirikan oleh warna lebih terang). Diantara protein-protein tersebut terdapat 19 dari 22 protein penyebab DM Tipe 2. Submatrik lain yang menandakan terdapat hubungan erat antara senyawa dengan aktifitas protein tertentu juga ditunjukkan oleh kotak kecil berbentuk horizontal. Kotak horizontal tersebut menarik untuk diamati karena protein-protein tersebut bersesuaian dengan gerombol senyawa sintetis dan tidak berhubungan erat dengan senyawa-senyawa pada posisi paling kanan, yaitu senyawa-senyawa yang tidak berdekatan dengan senyawa sintetis antidiabetes. Senyawa pada posisi paling kanan tersebut berhubungan erat dengan terlalu banyak protein dan cenderung kurang spesifik. Kotak kecil horizontal menjelaskan hubungan yang erat antara 19 senyawa (17 senyawa sintetis antidiabetes dan 2 senyawa tanaman Bratawali) dengan 68 protein. Protein penyebab diabetes banyak menggerombol pada lokasi tersebut yaitu sebanyak 13 dari 22 protein penyebab diabetes. Protein tersebut ditampilkan pada tabel 6.
Tabel 6. Protein penyebab diabetes yang bersesuaian pada kotak horizontal
No Nama Protein ID Protein
1 plasma membrane calcium-transporting ATPase 1
isoform 1b NP_001673.2
2 granulins precursor NP_002078.1
3
RecName: Full=Inositol 1,4,5-trisphosphate receptor type 1; AltName: Full=IP3 receptor isoform 1; Short=IP3R 1; Short=InsP3R1; AltName: Full=Type 1 inositol 1,4,5-trisphosphate receptor; Short=Type 1
InsP3 receptor
UniProtKBQ14643 .3
4 paired box protein Pax-8 isoform PAX8A NP_003457.1 5 peptidyl-glycine alpha-amidating monooxygenase
isoform a preproprotein NP_000910.2
6 ryanodine receptor 1 isoform 1 NP_000531.2
7 semenogelin-1 preproprotein NP_002998.1
8 acetyl-CoA carboxylase 1 isoform 1 NP_942131.1
9 dihydropyrimidinase NP_001376.1
10 glutamate receptor ionotropic, kainate 5 isoform 2
precursor NP_002079.3
11 RNA-binding protein Nova-1 isoform 1 NP_002506.2 12 exosome complex component RRP4 isoform 1 NP_055100.2
23
Berdasarkan hasil gerombol simultan dua dimensi, diketahui terdapat dua senyawa Bratawali menggerombol dekat dengan senyawa sintetis dan bersesuaian dengan gerombol protein yang terdapat banyak protein penyebab diabetes. Lebih lanjut dilakukan analisis jejaring kemiripan untuk menguatkan hasil penggerombolan simultan mengenai senyawa aktif antidiabetes.
Jejaring Kemiripan
Penggerombolan senyawa terhadap protein menghasilkan nilai kemiripan antara senyawa sintetis dengan senyawa tanaman. Nilai kemiripan tersebut bervariasi namun hanya nilai di atas 0.5 yang ditampilkan pada jejaring kemiripan (similarity network).
24
5
SIMPULAN DAN SARAN
Simpulan
Terdapat setidaknya 3 kesimpulan dari hasil penelitian ini, yaitu:
Analisis jejaring farmakologi dengan metode drugCIPHER mampu merubah kedekatan antar protein dan struktur kimia senyawa menjadi nilai numerik yang dapat dianalisis secara komputasi dan secara statistika.
Analisis statistika penggerombolan simultan dapat digunakan sebagai pendekatan alternatif dalam penentuan senyawa aktif tanaman obat antidiabetes tipe 2 selain pendekatan farmasi melalui laboratorium.
Jejaring kemiripan telah melengkapi hasil analisis gerombol simultan dan berhasil menunjukkan bahwa terdapat 2 dari 3 senyawa Bratawali dan 11 dari 44 senyawa Jahe yang berpotensial sebagai obat antidiabetes karena menggerombol dan berkorelasi tinggi dengan senyawa aktif antidiabetes, sebaliknya tidak demikian untuk Sembung dan Pare. Akan tetapi Sembung dan Pare mungkin berpotensi sebagai tanaman antidiabetes bila data yang digunakan lebih banyak atau dengan analisis yang lain.
Saran
Terdapat 2 hal yang dapat menjadi penelitian lanjutan dan merupakan saran dari peneliti, yaitu :
Melakukan analisis pada interaksi biner antar protein (39240 interaksi) terkait dengan interaksi protein penyebab diabetes tipe 2 dengan protein-protein lain. Hal tersebut diharapkan dapat mengefisienkan proses dan waktu untuk penentuan nilai kedekatan senyawa-protein.
25
DAFTAR PUSTAKA
Afendi F M, Darusman L K, Fukuyama M, Md. Altaf-UI-Amin, Kanaya S. 2012. A Bootstrapping approach for investigating the consistency of assignment of plants to jamu efficacy by PLS-DA model. Malaysian Journal of Mathematical Sciences. 6(2):147-164.
Afendi FM, Okada T, Yamazaki M, Hirai-Morita A, Nakamura Y , Nakamura K , Ikeda S ,Takahashi H , Altaf-Ul-Amin M, Darusman LK , Saito K, Kanaya S. 2012. KNApSAcK Family databases: Integrated metabolite–plant species databases for multifaceted plant research. Plant Cell Physiol. 53(2). 1-12. doi:10.1093/pcp/pcr165.
Beamer Scott, Buluc Aydin, Asanovic Krste, Patterson David A. 2013. Distributed Memory Breadth-First Search Revisited: Enabling Bottom-Up Search. Technical Report Electrical Engineering and Computer Sciences University of California at Berkeley, no. UCB/EECS-2013-2.
Calinski T, Harabasz J. 1974. A Dendrite Method for Cluster Analysis, Communication in Statistics. Taylor & Francis. 3, 1-27.
Definition, diagnosis and classification of diabetes mellitus and its complications. Part 1: Diagnosis and classification of diabetes mellitus. Geneva, World Health Organization, 1999 (WHO/NCD/NCS/99.2).
Getz G, Levine E, Domany E. 2000. Coupled Two-Way Clustering Analysis of Gene Microarray Data. PNAS. Vol 97, no.22, 12079-12084.
Goel R, Harsha HC, Pandey A, et al. 2012. Human Protein Reference Database and Human Proteinpedia as resources for phosphoproteome analysis. Mol Biosyst. 8(2). 453-463.
Govaert Gerard. 1995. Simultaneous clustering of rows and columns. Control and Cybernetics. vol.24 No.4.
Indrawaty Youllia, Hermana Asep Hana, Rinanto Vichy Sinar. 2011. Smulasi Pergerakan Langkah Kuda Menggunakan Metode Breadth-First Search. Jurnal Informatika. No.3, Vol. 2, September-Desember.
Johnson AM, Maggiora GM. 1990. Concepts and Applications of Molecular Similarity. New York: John Willey&Sons. ISBN 0-471-62175-7
Johnson RA, Wichern DW. 2002. Applied Multivariate Statistical Analysis, Fifth Edition. Englewood Cliffs, New Jersey: Prentice Hall.
Klekota Justin, Roth Frederick P. 2008. Chemical Substructures that Enrich for Biological Activity. Bioinformatics, 24:2518-2525.
Mudunuri U, Che A, Yi M, Stephens RM. 2009. bioDBnet: the biological database network. Bioinformatics. 25(4). 555–556. doi:10.1093/bioinformatics/btn654.
Nurishmaya MRN. 2014. Pendekatan Bioinformatika Formulasi Jamu Baru Berkhasiat Antidiabetes dengan Ikan Zebra (danio rerio) Sebagai Hewan Model [Skripsi]. Bogor(ID): Institut Pertanian Bogor.
Pairson Ainslie B, Brost Renee L et al. 2004. Integration of chemical-genetic and genetic interaction data links bioactive compounds to cellular target pathways. Nature Biotechnology. 22, 62 – 69.
26
Wang Y, Xiao J, Suzek TO, Zhang J, Wang J, Bryant SH. 2009. PubChem: a public information system for analyzing bioactivities of small molecules. Nucleic Acids Res. 2(8).1-11. doi:10.1093/nar/gkp456.
Wishart DS, Knox C, Guo AC, Cheng D, Shrivastava S, Tzur D, Gautam B, Hassanali M. 2008. DrugBank: a knowledgebase for drugs, drug actions and drug targets. Nucleic Acids Res. 2008 Jan;36(Database issue):D901-6.
Zhao S, Li S. 2010. Network-based relating pharmacological and genomic spaces for drug target identification. PLoS ONE. 5(7): e11764. doi:10.1371/journal.pone.0011764.
27
Lampiran 1 Senyawa sintetis antidiabetes yang telah terdaftar di Food and Drug Asociation (FDA) dengan sumber basis data Drugbank.
Kategori Simbol Nama senyawa Pubchem CID
Sulfonylureas drug
DB01 Tolbutamide 5505
DB02 Glyburide 3488
DB03 Glipizide 3478
DB04 Gliquidone 91610
DB05 Glimepiride 3476
DB06 Glicazide 3475
Biguanide drug
DB07 Metformin 4091
DB08 Phenformin 8249
DB09 Acarbose 441184
DB10 Voglibose 444020
DB11 Miglitol 441314
Euglycemic agent DB12 Pioglitazone 4829
DB13 Rosiglitazone 77999
Euglycemc agent (Nonsulfonylurea)
DB14 Repaglinide 65981
DB15 Starsis (TN) 443871 Aldose reductase
inhibtor
DB16 Tolrestat 53359
DB17 Alrestatin 2120
Other oral antidiabetic drugs
DB18 Sitaglipitin 4369359
28
Lampiran 2 Senyawa yang berasal dari tanaman yang diduga sebagai tanaman antidiabetes
Tanaman Simbol Nama Senyawa Pubchem
CID
BRATA-WALI
B013 (-)-Secoisolariciresinol 65373
B015 N-trans-Feruloyltyramine 5280537
B018 N-Formylanonaine 158516
JAHE
J010 Gingerdiols; [6]-Gingerdiol, 3-Epimer 5275727
J036 Gingerols; [6]-Gingerol 3473
J044 Gingerols; [6]-Gingerol, Demethoxy 9795270
J057 2-Heptanol; (ξ)-form 10976
J068 Isopropylbenzene 7406
J091 Shogaols; [8]-Shogaol 6442560
J127
1,7-Bis(3,4-dihydroxyphenyl)-3,5-heptanediol; (3ξ,5ξ)-form, 3,5-Di-Ac 11068834
J135
J193 alpha-Pinene 6654
J194 beta-Pinene 14896
J195 (+)-Camphor 159055
J196 3-Carene 26049
J198 Geraniol 637566
J199 Nerol 643820
J200 Terpinolene 11463
J205 Safrole 5144
J206 [6]-Shogaol 5281794
J207 Borneol 1201518
J208 Camphene 6616
J211 p-Cymene 7463
J212 (R)-linalool 6549
J213 alpha-Phellandrene 7460
J218 (E,E)-alpha-Farnesene 5281516
J221 (-)-beta-Sitosterol 222284
J224 (+)-S-Carvone 440917
J226 (-)-Isoborneol 6321405
29
J236 4-Terpineol 11230
J238 alpha-Terpineol 17100
J240 2-Undecanone 8163
J241 Nonanol 8914
J242 Octanal 454
J247 6-Methyl-5-hepten-2-one 9862
J248 Nonane 8141
J249 10-Shogaol 6442612
J250 1R,5R-(+)-alpha-Pinene 82227
J270 Hexahydrocurcumin 5318039
J274 Citral 638011
J275 Octane 356
J286 3-Methyl-butanal 11552
J287 n-Propanol 1031
J288 Propionaldehyde 527
J291 Methyl isobutyl ketone 7909
PARE
P044 Momordica charantia Elastase inhibitors 90677200
P183 Lycopene 446925
P195 Karaviloside I 44445582
SEM-BUNG
S002 2-Bornanol; (1S,2R)-form, Ac 6448
S030 3,4-Dihydroxybenzoic acid 72
30
Lampiran 3 Syntax R untuk analisis data
_Langkah-langkah Mencari Tanimoto Similarity_ Loading data
> amol<-load.molecules("D:/STATISTIKAS2/THESIS/Cleaning
data/datariset/smile2.sdf",aromaticity=TRUE,typing=TRUE,isotopes=TRUE, verbose=FALSE)
Mencari fingerprint (package rcdk & package fingerprint) > fps<-lapply(amol,get.fingerprint,type="pubchem") atau…
> fps<-lapply(amol,get.fingerprint,type="kr") Menampilkan koefisien tanimoto pada program R:
> fp.sim.matrix(fps, fplist2=NULL, method="tanimoto") atau…
> fp.sim.matrix(list(fps[[1]],fps[[2]]))
Eksport koefisien tanimoto Ke file xlsx: (package xlsx)
> write.xlsx(fp.sim.matrix(fps, fplist2=NULL, method="tanimoto"), "D:/tanimoto.xlsx")
Eksport koefisien tanimoto Ke file txt:
> write.table(fp.sim.matrix(fps, fplist2=NULL, method="tanimoto"), "D:/tanimoto.txt", sep="\t")
_ Langkah-langkah Mencari jarak terpendek antar protein_ Mencari Jarak terpendek antar protein (package igraph)
> g<-read.graph("D:/STATISTIKA2/THESIS_NURUL/PPI.gml",format="gml") > shortest.paths(g)
Eksport jarak terpendek Ke file xlsx: (package xlsx) > write.xlsx(shortest.paths(g),"D:/shortestpath.xlsx") _ Langkah-langkah Mencari _
> library(igraph)
> data<-as.matrix(read.table("D:/STATISTIKA S2/THESIS_NURUL/File kiriman/filenya ijal/ppi/ppi_edit.csv", sep=","))
> data1<-as.matrix(read.table("D:/STATISTIKA S2/THESIS_NURUL/File kiriman/filenya ijal/ppi/SP.csv", sep=";", header=TRUE))
> ppi<-as.vector(rbind(data[,2],data[,3])) > p<-data[1:9673,1]
> gr<-graph(ppi)
31
> write.table(hasil, file= "D:/STATISTIKA S2/THESIS_NURUL/File kiriman/filenya ijal/ppi/deket1.csv", sep=";") #file excel
_Langkah-langkah mencari nilai konkordan_
> data<-as.matrix(read.table("D:/DATA MINING/AGRICULTURE GROUP RISET/data/senyawa/regresi/cor.csv",sep=";"))
> cord<-cor(data)
> write.table(cord,file="D:/DATA MINING/AGRICULTURE GROUP RISET/data/senyawa/regresi/cordance.csv", sep=";")
_Langkah-langkah analisis komponen utama_ > data<-read.table("D:/untukPCA.csv", sep=",") > log.ir <- data[,1:1250]
> ir.species <- data[,1251]
> ir.pca <- prcomp(log.ir,center=TRUE,scale.=TRUE) > summary(ir.pca)
> g <- g + scale_color_discrete(name = '') > g <- g + theme(legend.direction = 'horizontal', legend.position = 'top')
> print(g)
> ir.pca$sdev #menampilkan eigenvalue
> head(ir.pca$rotation) #menampilkan loading factor > head(ir.pca$x) #menampilkan skor PCA 6 teratas
> scores = as.data.frame(ir.pca$x) #menampilkan semua skor PCA scores
> ggplot(data = scores, aes(x = PC1, y = PC2, label = rownames(scores))) + geom_hline(yintercept = 0, colour = "gray65") +
geom_vline(xintercept = 0, colour = "gray65") + geom_text(colour = "red", alpha = 0.8, size = 4) +
32
> ggplot(data = scores, aes(x = PC1, y = PC2, label =ir.species)) + geom_hline(yintercept = 0, colour = "gray65") +
geom_vline(xintercept = 0, colour = "gray65") +
geom_text(colour = ifelse(ir.species=="Bratawali", "red",
ifelse(ir.species=="Jahe", "dark green", ifelse(ir.species=="Pare", "Blue", ifelse(ir.species=="Sembung", "black", "purple")))), alpha = 0.8, size = 4) + ggtitle("PCA plot of Herbal Ingredients") #plot skor PCA
Menampilkan plot PCA 3 dimensi
> pca3d( ir.pca, group= ir.species, fancy= TRUE, bg= "black", axes.color= "white", new= TRUE)
_Langkah-langkah analisis gerombol simultan (package heatmap.plus)_
> data<-read.table("D:/STATISTIKA S2/THESIS_NURUL/Penggerombolan berhirarki dua dimensi bahan aktif tanaman obat dan protein target/Gambar 2-Dimensional HCA/konkordan.csv", sep=",", header=TRUE)
> datamatrix<-as.matrix(data)
> hclust.avl = function(x) hclust(x, method="ward.D") #Sesuaikan metodenya > dimensidua<-heatmap.plus(datamatrix, hclustfun=hclust.avl)
_Langkah-langkah menentukan korelasi antar senyawa untuk jejaring kemiripan_ > library(proxy)
> data<-read.table("D:/cobaeun.csv", sep=",")
33
34
35
Lampiran 6 Hasil gerombol simultan dengan metode Average Linkage
36
37
Lampiran 8 Hasil gerombol simultan dengan metode Ward.D2
38
RIWAYAT HIDUP
Penulis dilahirkan di Lampung pada tanggal 27 Februari 1991 dan merupakan anak pertama dari tiga bersaudara pasangan Jumiran dan Juju Juarsih. Penulis menyelesaikan jenjang pendidikan sekolah menengah atas jurusan Ilmu Pengetahuan Alam (IPA) pada tahun 2008 di SMA N 1 Gadingrejo, Lampung. Pada tahun yang sama penulis diterima sebagai mahasiswa di Institut Pertanian Bogor (IPB) jurusan Statistika melalui jalur undangan tanpa tes, lulus pada bulan September 2012. Penulis sempat bekerja selama 9 bulan sejak Desember 2012 hingga Agustus 2013, kemudian pada bulan September 2013 penulis memutuskan untuk melanjutkan studi pada program Magister di Jurusan Statistika IPB.
Selama perkuliahan jenjang sarjana, penulis mendapatkan beasiswa dari Yayasan Tanoto Foundation mulai dari semester 3 hingga semester 8. Sedangkan pada program Magister, penulis mendapatkan beasiswa dari Dirjen Pendidikan Tinggi (Dikti) sebagai Calon Dosen yang bernama Beasiswa Pendidikan Pascasarjana Dalam Negeri (BPP-DN). Setelah dinyatakan lulus pada program Magister, penulis akan mengabdi sebagai Dosen di Perguruan Tinggi dalam negeri. Semenjak perkuliahan jenjang sarjana hingga pascasarjana, penulis telah beberapa kali menjadi asisten Dosen untuk mengisi kelas responsi mata kuliah Metode Statistika, Perancangan Percobaan, Analisis Statistika dan Analisis Peubah Ganda. Selain itu, selama perkuliahan jenjang Magister, penulis juga aktif dalam kegiatan yang menggunakan keahlian berbahasa Inggris, seperti Summer Course Program IPB-Ibaraki University dengan tema Agriculture Sustainability pada Agustus 2015. Selain itu, penulis juga telah meraih juara Runner up pada kompetisi English Debate Competition dengan tema Climate Change, dan sekaligus menjadi salah satu dari lima peserta yang masuk nominasi sebagai Best Speaker pada ajang Pascasarjana Cup tahun 2014 yang diselenggarakan oleh Forum Wacana IPB dan Bogor Science Community (BSC).