APLIKASI MARKA MOLEKULER UNTUK SELEKSI
KETAHANAN BLAS PADA POPULASI
PADI HAPLOID GANDA
GUT WINDARSIH
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa tesis berjudul Aplikasi Marka Molekuler untuk Seleksi Ketahanan Blas pada Populasi Padi Haploid Ganda adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Tesis ini adalah bagian dari penelitian berjudul Pembentukan Padi Tahan Hama dan Penyakit Utama (Penggerek Batang, HDB, Blas, dan Tungro) yang dibiayai oleh dana DIPA APBN BB Biogen, Bogor, dengan nomor kontrak 1798.011.002.013 Tahun 2013. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor dan BB Biogen, Departemen Pertanian.
RINGKASAN
GUT WINDARSIH. Aplikasi Marka Molekuler untuk Seleksi Ketahanan Blas pada Populasi Padi Haploid Ganda. Dibimbing oleh UTUT WIDYASTUTI dan DWINITA WIKAN UTAMI.
Penyakit blas, disebabkan oleh cendawan Pyricularia grisea Sacc., merupakan salah satu penyakit yang merusak pada padi. Penggunaan varietas tahan blas adalah salah satu cara paling efisien untuk melindungi padi dari penyakit ini. Varietas tahan blas dapat diproduksi melalui program pemuliaan. Penggunaan marker-assisted selection (MAS) tersedia untuk membantu seleksi galur tahan berdasarkan gen ketahanan. Program pemuliaan varietas tahan blas dapat dikombinasikan dengan pendekatan kultur antera untuk memproduksi galur haploid ganda terfiksasi agar lebih efisien.
Ketahanan terhadap penyakit blas terjadi berdasarkan interaksi gene-to-gene antara gen ketahanan pada tanaman inang dan gen avirulen pada patogen blas. International Rice Research Institute (IRRI) telah mengembangkan set varietas padi diferensial, di mana setiap varietas memiliki gen ketahanan tunggal yang teridentifikasi. Penggunaan set varietas diferensial memungkinkan kita untuk membandingkan respons ketahanan terhadap patogen blas tertentu dan untuk mengidentifikasi gen ketahanan yang bertanggung jawab melindungi tanaman dari patogen blas berdasarkan teori gene-to-gene.
Tujuan penelitian ini yaitu: (1) membandingkan respons ketahanan galur haploid ganda dari persilangan ganda IR54/Parekaligolara//Bio110/Markuti dengan sistem diferensial standar terhadap tiga ras blas dari kawasan Indonesia terseleksi; (2) mengidentifikasi gen ketahanan blas pada populasi haploid ganda yang diuji menggunakan marka molekuler untuk gen Pi1, Pi2, Pi9, Pi33, Pib, Pir4, dan Pir7; serta (3) mengidentifikasi potensi gen-gen yang memiliki kontribusi membentuk ketahanan blas berdasarkan analisis asosiasi antara data fenotipe respons ketahanan dan data genotipe menggunakan marka molekuler.
Empat puluh sembilan galur haploid ganda dari persilangan ganda IR54/Parekaligolara//Bio110/Markuti diseleksi menggunakan marka gen Pib, Pi1, Pi2, Pi9, Pi33, Pir4, dan Pir7. Untuk membandingkan seleksi fenotipe digunakan varietas diferensial yang terdiri atas 10 galur monogenik dengan background genetik LTH. Semua tanaman diinokulasi dengan 3 isolat dari genotipe PH14.
menunjukkan gen-gen ketahanan dari varietas diferensial pada kelompok patotipe yang sama belum tentu memiliki respons patotipe yang sama terhadap ketiga isolat. Hal ini mengindikasikan galur monogenik LTH tidak dapat digunakan sebagai pembanding respons ketahanan tanaman terhadap isolat blas di Indonesia. Berdasarkan evaluasi fenotipe terhadap 49 galur haploid ganda, 2 galur tahan terhadap ras 123; 38 galur medium tahan, dan 9 galur bersifat rentan. Dua puluh satu galur tahan terhadap ras 133, 17 galur medium tahan, dan 11 galur bersifat rentan. Dua galur tahan terhadap ras 173, 21 galur medium tahan, dan 26 galur bersifat rentan.
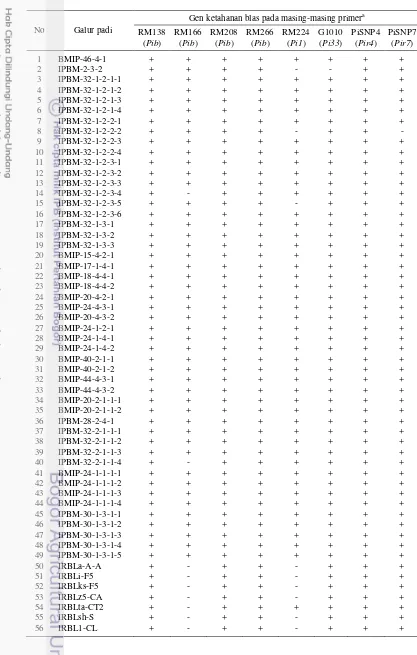
Hasil reaksi PCR menunjukkan 8 primer mampu mengamplifikasi fragmen DNA target (RM138, RM166, RM208, RM266, RM224, PiSNP4, PiSNP7, dan G1010). Namun, pita DNA target yang dihasilkan pada masing-masing primer tidak konsisten dengan respons ketahanan tanaman yang diuji terhadap ketiga isolat, kecuali PiSNP7 yang merupakan marka spesifik untuk gen Pir7. Marka PiSNP7 lebih konsisten dengan respons ketahanan terhadap ketiga ras.
Analisis asosiasi antara marka molekuler dan respons fenotipe menurut Hayashi et al. (2009) menunjukkan 3 marka berasosiasi dengan gen Pi target: RM224 berasosiasi dengan gen Pi1, RM166 berasosiasi dengan gen Pib, dan PiSNP7 berasosiasi dengan gen Pir7. Gen Pib berkontribusi membentuk ketahanan terhadap ras 123, sedangkan gen Pi1 dan Pir7 berkontribusi membentuk ketahanan terhadap ras 123 dan 133. Primer PiSNP7 paling presisi digunakan sebagai marka seleksi karena menghasilkan pita target yang berhubungan langsung dengan respons ketahanan. Sementara itu, analisis asosiasi antara marka molekuler dan respons fenotipe menurut IRRI (1996) menunjukkan gen Pib tidak berasosiasi dengan respons ketahanan terhadap ras 123, tetapi terhadap ras 133. Hasil ini menunjukkan bahwa sistem evaluasi standar dari IRRI (1996) masih diperlukan untuk menentukan tingkat ketahanan tanaman terhadap serangan blas daun. Primer RM138, RM208, RM266, PiSNP4, dan G1010 tidak berasosiasi dengan respons ketahanan terhadap ketiga ras sehingga tidak dapat digunakan sebagai marka seleksi. Tidak adanya asosiasi antara RM138, RM208, RM266, dan G1010 dengan respons ketahanan disebabkan adanya jarak genetik antara marka tersebut dengan lokus gen target sehingga memungkinkan terjadinya rekombinasi. Hal ini menunjukkan bahwa marka molekuler yang didesain untuk suatu populasi persilangan tidak selalu dapat diaplikasikan untuk populasi persilangan yang lain dengan latar belakang genetik yang berbeda. PiSNP4 merupakan primer spesifik untuk gen Pir4 yang hanya menyebabkan ketahanan terhadap isolat dari genotipe CM28. Tidak ada primer yang berasosiasi dengan respons ketahanan tanaman terhadap ras 173. Hasil ini mengindikasikan respons ketahanan terhadap ras 173 tidak dikontribusikan oleh gen Pi1, Pi2, Pi9, Pib, Pi33, Pir4, maupun Pir7.
Berdasarkan evaluasi fenotipe, 2 galur haploid ganda terseleksi paling tahan terhadap ras 123, 21 galur paling tahan terhadap ras 133, dan 2 galur paling tahan terhadap ras 173. Untuk menyeleksi galur unggul, karakteristik agronomi harus diamati seperti tinggi tanaman, umur berbunga, umur panen, dan bobot gabah. Kata kunci: marka molekuler, penyakit blas, populasi haploid ganda, Pyricularia
SUMMARY
GUT WINDARSIH. Molecular Markers Application for Blast Resistance Selection on The Double Haploid Rice Population. Supervised by UTUT WIDYASTUTI and DWINITA WIKAN UTAMI.
Blast disease, caused by fungal Pyricularia grisea Sacc., is one of the devastating diseases in rice. The using of blast-resistant varieties is one of the most efficient ways to protect rice from this disease. Blast resistant varieties can be produced through breeding program. The using of marker-assisted selection (MAS) available to support selection of resistant lines based on resistance gene. The breeding program of blast resistant varieties can be come together with anther culture approach to produce the fixed double haploid lines more efficiently.
The resistance to blast disease is governed by a gene-to-gene interaction between resistance gene in the host and avirulence gene in the blast pathogen. The International Rice Research Institute (IRRI) had developed differential rice variety set. Each variety had single resistance gene identified. The using of differential variety set allowed us to compare the resistance response to certain blast pathogen and to identify the resistance gene that responsible to protect plant from blast pathogen based on gene-to-gene theory.
The objectives of this research were: (1) to compare the resistance response of the double haploid lines from a double cross IR54/Parekaligolara//Bio110/Markuti with the standard differential system to three selected Indonesian blast races; (2) to identify blast resistance genes on the double haploid population tested using molecular markers for Pi1, Pi2, Pi9, Pi33, Pib, Pir4 and Pir7 genes; and (3) to identify the potency of gene(s) that had a contribution in form the blast resistance based on association analysis between the phenotype data of resistance response and the genotype data using molecular markers.
Forty-nine double haploid lines from a double cross between IR54/Parekaligolara//Bio110/Markuti were selected by using the markers based on Pib, Pi1, Pi2, Pi9, Pi33, Pir4 and Pir7 genes. To compare the phenotype selection, the differential varieties consisted of ten monogenic lines with LTH genetic background were used. All plants were inoculated with 3 isolates from PH14 genotype.
same pathotype response to blast isolates from Indonesia. It indicated LTH monogenic lines could not be used to compare the resistance response on plant to blast isolates in Indonesia.
Based on the phenotype evaluation on 49 double haploid lines, 2 lines were resistant to race 123; 38 lines were medium resistant and 9 lines were susceptible. Twenty-one lines was resistant to race 133, 17 lines were medium resistant and 11 lines were susceptible. Two lines were resistant to race 173, 21 lines were medium resistant and 26 lines were susceptible.
The result of PCR reaction showed 8 primers amplified the targeted DNA fragments (RM138, RM166, RM208, RM266, RM224, PiSNP4, PiSNP7 and G1010). But, the targeted DNA fragments produced on each primer were not consistent with the resistant response on plant tested to three isolates, except PiSNP7 that was a specific marker for Pir7 gene. This marker was more consistent with the resistant response to these races.
The association analysis between the molecular markers and the phenotype response by Hayashi et al. (2009) showed 3 markers were associated to Pi genes targeted: the RM224 was associated to Pi1 gene, the RM166 was associated to Pib gene and the PiSNP7 was associated to Pir7 gene. Pib gene contributed in form the resistance to race 123, while Pi1 and Pir7 genes contributed in form the resistance to races 123 and 133. Primer PiSNP7 was the most precision to be used as selection marker because produced the targeted band that directly according with the resistance response. Meanwhile, the association analysis between the molecular markers and the phenotype response by IRRI (1996) showed the Pib was not associated with the resistance response to race 123, but to race 133. This showed that the standar evaluation system from IRRI (1996) was still needed to recombination. This showed that the molecular marker designed for specific crossing population could not always be used for other crossing population from different genetic background. PiSNP4 was a specific primer for Pir4 gene that cause the resistance to isolate from CM28 genotype only. There was no primer that associated with the response of resistance on plants to race 173. This result indicated that the resistance response to race 173 was not contributed by Pi1, Pi2, Pi9, Pib, Pi33, Pir4 or Pir7 genes.
According to phenotype evaluation, 2 selected double haploid lines were the most resistant to race 123, 21 lines were the most resistant to race 133 and 2 lines were the most resistant to race 173. To select superior lines, the agronomic characteristic must be observed such as plant height, date of flowering, date to harvest and grain weight.
© Hak Cipta milik IPB dan BB Biogen, tahun 2014
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB dan BB Biogen
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Biologi Tumbuhan
APLIKASI MARKA MOLEKULER UNTUK SELEKSI
KETAHANAN BLAS PADA POPULASI
PADI HAPLOID GANDA
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2014
Judul Tesis : Aplikasi Marka Molekuler untuk Seleksi Ketahanan Blas pada Populasi Padi Haploid Ganda
Nama : Gut Windarsih
NIM : G353110191
Disetujui oleh Komisi Pembimbing
Dr.Ir. Utut Widyastuti, MSi Ketua
Dr. Dwinita Wikan Utami, MSi Anggota
Diketahui oleh
Ketua Program Studi Biologi Tumbuhan
Dr.Ir. Miftahudin, MSi
Dekan Sekolah Pascasarjana
Dr.Ir. Dahrul Syah, MScAgr
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah SWT atas segala rahmat dan karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Judul yang dipilih dalam penelitian ini adalah Aplikasi Marka Molekuler untuk Seleksi Ketahanan Blas pada Populasi Padi Haploid Ganda. Penelitian ini dilaksanakan pada bulan September 2012 sampai Oktober 2013 di Laboratorium Bioteknologi dan Biologi Molekuler, Balai Besar Bioteknologi dan Sumberdaya Genetik Pertanian (BB Biogen), Bogor. Penelitian ini merupakan bagian dari penelitian berjudul Pembentukan Padi Tahan Hama dan Penyakit Utama (Penggerek Batang, HDB, Blas, dan Tungro) yang dibiayai oleh dana DIPA APBN BB Biogen, Bogor, dengan nomor kontrak 1798.011.002.013 Tahun 2013.
Terima kasih penulis ucapkan kepada Ibu Dr.Ir. Utut Widyastuti, M.Si. dan Ibu Dr. Dwinita Wikan Utami, M.Si. selaku pembimbing serta Bapak Dr.Ir. Hajrial Aswidinnoor, MSc selaku penguji luar komisi yang telah memberikan banyak saran dan masukan. Ungkapan terima kasih juga penulis sampaikan kepada Bapak Wawan, Bapak Dadang, dan Bapak Yana Suryatna dari BB Biogen, Bogor serta Ibu Pepi Elvavina dari Laboratorium Biorin, IPB yang telah banyak membantu selama penelitian. Ungkapan terima kasih juga penulis sampaikan kepada ayah, ibu, beserta seluruh keluarga, atas doa dan semangat yang diberikan. Semoga karya ilmiah ini bermanfaat bagi dunia penelitian dan masyarakat.
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
1 PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 4
Manfaat Penelitian 5
2 BAHAN DAN METODE 5
Bahan Penelitian 5
Waktu dan Tempat Penelitian 5
Metode Penelitian 6
Analisis Data 10
3 HASIL DAN PEMBAHASAN 10
Respons Fenotipe terhadap Ras Blas 10
Evaluasi Genotipe Menggunakan Marka Molekuler 14 Keterkaitan (Asosiasi) Marka Molekuler dengan Respons Fenotipe 17
4 SIMPULAN DAN SARAN 25
DAFTAR PUSTAKA 26
DAFTAR TABEL
1 Daftar galur monogenik LTH 2
2 Sistem penentuan ras blas berdasarkan reaksi galur monogenik LTH 3 3 Nomor ras dominan berdasarkan pola reaksinya terhadap varietas
diferensial Indonesia 3
4 Deskripsi isolat blas yang digunakan 5
5 Skala skor penyakit blas daun tanaman padi (Hayashi et al. 2009) 7 6 Skala skor penyakit blas daun tanaman padi (IRRI 1996) 7 7 Primer untuk amplifikasi fragmen gen penanda ketahanan blas 9 8 Pola reaksi varietas diferensial terhadap tiga isolat blas 11
9 Pola reaksi terhadap tiga ras blas pada tetua 12
10 Pola reaksi terhadap tiga ras blas pada galur haploid ganda 13 11 Amplifikasi fragmen DNA target menggunakan marka molekuler 16 12 Analisis asosiasi marka molekuler dengan respons fenotipe menurut
Hayashi et al. (2009) 17
13 Analisis asosiasi marka molekuler dengan respons fenotipe menurut
IRRI (1996) 24
DAFTAR GAMBAR
1 Skema produksi galur haploid ganda melalui kultur antera pada F1 hasil
persilangan ganda IR54/Parekaligolara//Bio110/Markuti 4
2 Inokulasi tanaman dengan tiga ras isolat blas 6
3 Standar pemberian skor blas menurut Hayashi et al. (2009): skor 0-2 untuk sifat tahan, skor 3 untuk medium tahan, dan skor 4-5 untuk sifat
rentan 7
4 Standar pemberian sklor blas daun menurut IRRI (1996) 8 5 Amplifikasi PCR dari primer RM224 pada populasi haploid ganda
(1-49), 10 galur monogenik LTH (50-59), tetua (60=Bio110, 61=Parekaligolara, 62=Markuti, 63=IR54), dan US2 (64) 14 6 Amplifikasi PCR dari primer RM166 pada populasi haploid ganda
(1-49), 10 galur monogenik LTH (50-59), tetua (60=Bio110, 61=Parekaligolara, 62=Markuti, 63=IR54), dan US2 (64) 15 7 Amplifikasi PCR dari primer PiSNP7 pada populasi haploid ganda
(1-49), 10 galur monogenik LTH (50-59), tetua (60=Bio110, 61=Parekaligolara, 62=Markuti, 63=IR54), dan US2 (64) 15 8 Peta genetik kromosom 2 oleh Temnykh et al. (2001) (a); analisis
kandidat gen ketahanan Pir pada kromosom 2 pada posisi antara primer
RM263 dan RM250 (Utami et al. 2008) 19
9 Analisis pemetaan gen Pir4 dan Pir7 pada populasi backcross dari persilangan antara IR64 dan Oryza rufipogon; gen Pir4 terpetakan pada posisi 110.9 cM, sedangkan gen Pir7 terpetakan pada posisi 122.8 cM
10 Peta genetik kromosom 11 (a) oleh Temnykh et al. (2001); daerah antara 110.0 dan 123.2 cM (b) dilengkapi dengan informasi yang tersedia di database Gramene (www.gramene.org); analisis keterpautan marka RM123*I dan RM224 pada posisi 0.0 cM terhadap gen
ketahanan Pi1(t) (c) 21
11 Peta genetik kromosom 2 (a) oleh Temnykh et al. (2001); peta genetik marka RM166, RM208, RM266, dan RM138 pada kromosom 2 yang dekat dengan gen Pib pada populasi F12 dari persilangan Gulfmont
(tahan) dan Te-Qing (rentan) (Fjellstrom et al. 2004) 22 12 Peta genetik kromosom 8 (a) oleh Temnykh et al. (2001); pemetaan
1
PENDAHULUAN
Latar Belakang
Penyakit blas merupakan salah satu penyakit yang merusak pada padi (Kumar et al. 2010). Di Indonesia, luas serangan penyakit blas mencapai 1285 juta ha atau sekitar 12% dari total luas areal pertanaman padi (http://www.deptan.go.id, 2004). Penyakit blas disebabkan oleh cendawan Pyricularia grisea Sacc. (teleomorph: Magnaporthe grisea; sinonim: Pyricularia oryzae Cavara) (Rossman et al. 1990). Fungisida dapat digunakan untuk mengendalikan penyakit blas pada padi, namun residu kimianya dapat menyebabkan kontaminasi lingkungan. Oleh karena itu, penggunaan varietas tahan merupakan salah satu cara yang paling efisien untuk melindungi padi dari penyakit blas (Koide et al. 2009). Pemuliaan varietas padi tahan blas dapat dikombinasikan dengan pendekatan kultur antera untuk menghasilkan galur haploid ganda secara lebih efisien (Niizeki & Oono 1968).
Sistem penyerangan penyakit blas terjadi berdasarkan interaksi gene-to-gene antara gen ketahanan pada tanaman inang dan gen avirulen pada patogen blas (Silue et al. 1992). Lebih dari 70 gen ketahanan blas telah diidentifikasi melalui analisis quantitative trait locus (QTL), tetapi hanya 18 gen yang telah dikloning dan dikarakterisasi (Roychowdhury et al. 2011). Gen-gen ketahanan ini dapat dideteksi menggunakan marka molekuler, sehingga varietas tahan blas dapat diseleksi menggunakan marka molekuler yang berdekatan dengan gen ketahanan, dengan demikian proses seleksi lebih cepat (Koide et al. 2009).
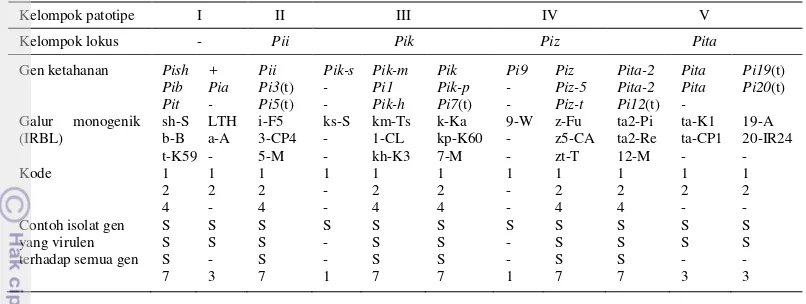
International Rice Research Institute (IRRI) telah mengembangkan varietas padi diferensial. Varietas diferensial ini terdiri atas 29 galur monogenik (monogenic lines/MLs) dengan latar belakang genetik varietas Japonica, Lijian-xin-tuan-heigu (LTH), masing-masing mengandung satu dari 24 gen ketahanan blas: Pia, Pib, Pii, Pik, Pik-h, Pik-m, Pik-p, Pik-s, Pish, Pit, Pita, Pita-2, Piz, Piz-t, Piz-5, Pi1, Pi3(t), Pi5(t), Pi7(t), Pi9, Pi11(t), Pi12(t), Pi19(t), dan Pi20(t) (Tabel 1). Penggunaan varietas diferensial berfungsi sebagai pembanding respons ketahanan terhadap suatu ras patogen tertentu dan mengidentifikasi gen ketahanan yang melindungi tanaman dari patogen blas berdasarkan teori gene-to-gene (Hayashi & Fukuta 2009).
Galur monogenik LTH dikelompokkan dalam 5 kelompok patotipe (Tabel 1). Pengelompokan patotipe tersebut dilakukan berdasarkan respons patotipe masing-masing galur terhadap isolat blas diferensial standar Jepang. Galur yang memiliki reaksi ketahanan hampir sama dikelompokkan dalam satu kelompok patotipe yang sama. Gen-gen ketahanan pada galur monogenik tersebut merupakan gen multialelik yang dikelompokkan dalam 4 lokus genetik (Tabel 2). Gen-gen ketahanan yang multialelik atau terpaut dekat dengan lokus Pii, Pik, Piz, dan Pita dikelompokkan dalam 4 lokus yang berbeda (Hayashi & Fukuta 2009).
2
satuan, sedangkan IRBLsh-B (atau IRBLsh-S), IRBLb-B, dan IRBLt-K59 di sisi puluhan. Bagian kedua memiliki satu angka dan terdiri atas IRBLi-F5, IRBL3-CP4, dan IRBL5-M. Bagian ketiga memiliki 3 angka dan terdiri atas IRBLk-Ka, IRBLkp-K60, dan IRBL7-M di sisi satuan, RBLkm-Ts, IRBL1-CL, dan IRBLkh-K3 di sisi puluhan, sedangkan IRBLks-S di sisi ratusan. Bagian keempat memiliki 2 angka dan terdiri atas IRBLz-Fu, IRBLz5-CA, dan IRBLzt-T di sisi satuan, sedangkan IRBL9-W di sisi puluhan. Bagian kelima memiliki 3 angka dan terdiri atas IRBL19-A dan IRBL20-IR24 di sisi satuan, IRBLta-K1 dan IRBLta-CP1 di sisi puluhan, sedangkan IRBLta2-Pi, IRBLta2-Re, dan IRBL12-M di sisi ratusan. Huruf i, k, z, dan ta berturut-turut disertakan di depan bagian 2 hingga 5 yang menunjukkan lokus dari multialel. Suatu nomor ras merupakan jumlah dari suatu nomor kode yang menunjukkan reaksi rentan. Huruf U sebelum nomor pada bagian pertama menunjukkan ras blas international (Hayashi & Fukuta 2009).
Tabel 1 Daftar galur monogenik LTHa
3
Sementara itu, penentuan ras blas di Indonesia secara konvensional dilakukan dengan pengujian ras pada satu set varietas diferensial Indonesia yang meliputi Asahan, Cisokan, IR64, Krueng Aceh, Cisadane, Cisanggarung, dan Kencana Bali. Ketujuh varietas diferensial tersebut memberikan reaksi yang bersifat spesifik terhadap ras-ras dominan yang ditemukan di kawasan endemik blas di Indonesia (Tabel 3). Varietas Asahan mempunyai nomor kode pola reaksi ketahanan paling tinggi. Hal ini karena pola reaksi ketahanannya bersifat tahan terhadap semua ras dominan yang diinokulasikan, kecuali terhadap ras yang paling virulen 201. Setiap isolat baru akan mempunyai nilai skor 200 apabila mampu menginfeksi varietas diferensial Asahan. Varietas diferensial berikutnya adalah Cisokan. Varietas ini menunjukkan pola reaksi rentan terhadap ras 101. Setiap isolat baru akan mempunyai nilai skor 100 apabila dapat menginfeksi varietas diferensial Cisokan. Demikian juga untuk varietas diferensial yang lain. Penentuan ras selanjutnya dilakukan berdasarkan jumlah skor rentan pada ketujuh varietas diferensial tersebut (Mogi et al. 1991).
Pyricularia grisea bersifat dinamis karena mampu beradaptasi dengan cepat terhadap kondisi tanaman inang. Patogen ini juga memiliki tingkat keanekaragaman genetik yang tinggi dan kemampuan untuk menghasilkan ras baru dengan cepat. Lebih dari 26 ras cendawan P. grisea telah diidentifikasi dari kawasan endemik penyakit blas di Indonesia, 7 ras di antaranya (ras 001, 023, 033,
Tabel 2 Sistem penentuan ras blas berdasarkan reaksi galur monogenik LTH
Kelompok patotipe I II III IV V
4
073, 101, 133, dan 173) merupakan isolat dominan yang selalu ada di setiap musim tanam (Santoso & Nasution 2009). Oleh karena itu, pengembangan varietas padi tahan blas yang mempunyai ketahanan yang tahan lama dan bersifat poligenik menjadi sangat esensial (Utami et al. 2011). Salah satu strategi untuk mengatasi patogen blas yang multi-ras adalah mengembangkan varietas tahan blas yang memiliki banyak gen ketahanan melalui pyramiding genes.
Gambar 1 Skema produksi galur haploid ganda melalui kultur antera pada F1 hasil persilangan ganda
IR54/Parekaligolara//Bio110/Markuti
Balai Besar Penelitian dan Pengembangan Bioteknologi dan Sumberdaya Genetik Pertanian (BB BIOGEN), Bogor, telah melakukan persilangan ganda dari IR54/Parekaligolara//Bio110/Markuti, kemudian pada tanaman F1 hasil
persilangan tersebut dilakukan kultur antera sehingga diperoleh 49 galur haploid ganda (Gambar 1). IR54 toleran terhadap defisiensi fosfat dan tahan penyakit blas. Galur ini dideteksi dengan marka molekuler memiliki gen ketahanan blas: Pi1, Pi2, Pi9, Pi33, dan Pib. Bio110 tahan terhadap blas dan memiliki gen ketahanan Pir4 dan Pir7. Gen-gen ini dideteksi menggunakan marka molekuler pada populasi pemetaan backcross hasil persilangan antara IR64 dan spesies padi liar Oryza rufipogon (Utami et al. 2008). Parekaligolara tahan terhadap penyakit hawar daun bakteri (bacterial leaf blight), sedangkan Markuti toleran terhadap cekaman keracunan logam Fe. Dari persilangan tersebut diharapkan akan diperoleh varietas unggul yang toleran terhadap defisiensi fosfat dan cekaman keracunan logam Fe serta tahan terhadap penyakit blas dan hawar daun bakteri.
Tujuan Penelitian
Tujuan penelitian ini adalah (1) membandingkan respons ketahanan pada populasi haploid ganda hasil persilangan IR54/Parekaligolara//Bio110/Markuti dengan varietas diferensial terhadap tiga ras blas dominan di Indonesia; (2) mengidentifikasi gen ketahanan blas pada populasi haploid ganda menggunakan marka molekuler penanda gen Pi1, Pi2, Pi9, Pi33, Pib, Pir4, dan Pir7; serta (3) mengidentifikasi potensi gen yang memiliki kontribusi membentuk ketahanan berdasarkan analisis gabungan respons ketahanan dengan data genotipe menggunakan marka molekuler tersebut.
Kultur antera
Parekaligolara x IR54 Bio110 x Markuti
F1 x F1
F1 hasil persilangan ganda
5 Manfaat Penelitian
Manfaat penelitian ini adalah memperoleh galur-galur tahan blas baik berdasarkan evaluasi fenotipe maupun gen ketahanan blas. Galur-galur terpilih tersebut merupakan kandidat galur harapan untuk selanjutnya dilakukan uji daya hasil pendahuluan (UDHP) dan uji daya hasil lanjutan (UDHL) yang pada gilirannya nanti dapat dilepas sebagai varietas unggul.
2
BAHAN DAN METODE
Bahan Penelitian
Bahan tanaman yang digunakan terdiri atas 49 galur haploid ganda hasil persilangan dari IR54/Parekaligolara//Bio110/Markuti, 10 galur monogenik dengan latar belakang genetik LTH sebagai kontrol tahan, dan varietas US2 untuk kontrol rentan. Untuk evaluasi fenotipe, setiap tanaman diuji respons ketahanannya terhadap ras 123, 133, dan 173 (Tabel 4). Ketiga isolat tersebut telah terkarakterisasi berdasarkan tingkat penyerangannya pada tanaman isogenik yang mengandung gen Pi target (ditunjukkan dalam LTH-kode patogenik), tipe perkawinan, dan gen avirulen (ACE1-avr gene). Adapun untuk evaluasi genotipe, DNA diekstrak dari daun untuk mengamplifikasi fragmen DNA penanda gen ketahanan blas target melalui proses PCR.
Waktu dan Tempat Penelitian
Penelitian ini dilakukan di Laboratorium Bioteknologi dan Biologi Molekuler, Balai Besar Penelitian dan Pengembangan Bioteknologi dan Sumberdaya Genetik Pertanian, Bogor dari September 2012 hingga Oktober 2013.
Tabel 4 Deskripsi isolat blas yang digunakan
Ras Asal
123 Lampung 2004 ID22 Cirata - - PH14
133 Lampung 2003 ID23 Cirata
6
Metode Penelitian
Penyiapan Tanaman
Analisis respons fenotipe dilakukan menurut Utami et al. (2011). Benih padi dikecambahkan pada cawan petri yang telah diberi tisu basah selama tiga hari. Selanjutnya, kecambah ditanam dalam pot plastik (ukuran: 35 cm x 25 cm x 10 cm) yang telah diisi dengan media tanah sawah. Tiga benih per galur ditanam dalam setiap baris. Varietas US2 ditanam dalam setiap pot plastik.
Penyiapan Inokulum
Penyiapan inokulum mengikuti metode Utami et al. (2007). Setiap ras P. grisea ditumbuhkan pada media potato dextrose agar (PDA) untuk memperbanyak dan memurnikan isolat. Setelah tujuh hari, isolat dipindah ke media gandum (oatmeal) untuk memproduksi konidia. Pada hari kesepuluh, koloni cendawan digosok dengan kuas dan air steril yang telah ditambah dengan streptomisin 0.01 gram/L. Isolat kemudian disimpan dalam inkubator di bawah penerangan lampu neon 20 watt selama 48 jam. Proses penggosokan dan pemberian cahaya lampu bertujuan untuk menginduksi terjadinya sporulasi pada cendawan. Isolat selanjutnya digosok kembali dengan kuas dan air steril yang mengandung 0.02% Tween 20 untuk memperoleh suspensi konidia. Kerapatan konidia yang digunakan untuk inokulasi adalah 5 x 104 konidia/ml.
Gambar 2 Inokulasi tanaman dengan tiga ras isolat blas Inokulasi dan Evaluasi Ketahanan
Setelah berumur tiga minggu, setiap galur diinokulasi dengan metode penyemprotan sebanyak 3.5 ml suspensi konidia per tanaman. Tanaman yang telah diinokulasi diletakkan di ruang lembab (suhu 25.5 oC, kelembaban udara relatif 100%) selama 48 jam untuk memberikan kondisi yang sesuai untuk spora cendawan menginfeksi tanaman, kemudian dipindah ke rumah kaca (suhu 25.5-26
o
C, kelembaban udara relatif 91-95%). Kelembaban udara dipelihara melalui pengembunan menggunakan sprinkler (Gambar 2). Reaksi ketahanan terhadap serangan blas daun pada dua minggu setelah inokulasi diskor berdasarkan sistem
Preparasi tanaman Inokulasi
7 penentuan tingkat ketahanan menurut Hayashi et al. (2009) (Gambar 3; Tabel 5) dan IRRI (1996) (Gambar 4; Tabel 6), selanjutnya hasil kedua metode tersebut dibandingkan.
0 1 2 3 4 5
Gambar 3 Standar pemberian skor blas daun menurut Hayashi et al. (2009): skor 0-2 untuk sifat tahan, skor 3 untuk medium tahan, dan skor 4-5 untuk sifat rentan
Tabel 5 Skala skor penyakit blas daun tanaman padi (Hayashi et al. 2009) Skor Gejala Sifata Keterangan
0 T Tidak ada bercak infeksi
1 T Bercak cokelat, diameter lebih kecil dari 0.5 mm, tidak terjadi sporulasi
2 T Bercak cokelat, diameter 0.5-1.00 mm, tepi bercak cokelat gelap, tidak terjadi sporulasi
3 MT Bercak berbentuk bundar hingga lonjong, diameter 1-3 mm, pusat bercak berwarna abu-abu dengan tepi bercak cokelat, mampu mengalami sporulasi
4 R Bercak berbentuk gelendong khas blas, diameter 3 mm, pusat bercak terjadi nekrotik dan berwarna abu-abu, mampu mengalami sporulasi
5 R Bercak berbentuk gelendong khas blas, pusat bercak nekrotik dan berwarna abu-abu, diameter bercak setengah dari salah satu atau dua helaian daun, mampu mengalami sporulasi
a
T = Tahan, MT = medium tahan, R = rentan
Tabel 6 Skala skor penyakit blas daun tanaman padi (IRRI 1996) Skor Gejala Sifata Keteranganb
0 T Tidak ada gejala serangan
1 T Terdapat bercak sebesar ujung jarum (LDT = 0.5%) 2 T Bercak lebih besar dari ujung jarum (LDT = 1%)
3 T Bercak keabu-abuan, berbentuk bundar dan agak lonjong, panjang 1-2 mm dengan tepi cokelat (LDT = 2%)
4 MT Bercak khas blas, panjang 1-2 mm, LDT < 5% 5 MT Bercak khas blas, LDT 5-10%
6 R Bercak khas blas, LDT 11-25% 7 R Bercak khas blas, LDT 26-50% 8 R Bercak khas blas, LDT 51-75% 9 R Bercak khas blas, LDT 76-100%
a
8
Isolasi DNA
Isolasi DNA dilakukan berdasarkan metode CTAB menurut Doyle & Doyle (1987). Daun tanaman digerus dalam nitrogen cair menggunakan mortar. Sampel ditambah dengan 750 μl larutan penyangga CTAB dan diinkubasi pada suhu 65 oC selama 30 menit. Sampel ditambah dengan 750 μl CI (kloroform:isoamil-alkohol = 24:1) dan disentrifugasi pada kecepatan 10000 rpm selama 15 menit. Supernatan 500 μl ditambah dengan 50 μl Na-asetat 2M pH 5.2 dan 1 ml etanol absolut. Setelah dipresipitasi dalam freezer semalam, sampel disentrifugasi pada kecepatan 10000 rpm selama 15 menit. Pellet ditambah dengan 500 μl alkohol 70% dan disentrifugasi pada kecepatan 10000 rpm selama 5 menit. Setelah dikeringkan, pellet ditambah dengan 50 μl TE 1x dan 10 μl RNase 10 ng/μl, selanjutnya diinkubasi pada suhu 37 oC selama satu jam dan inaktivasi RNase dilakukan dengan diinkubasi pada suhu 65 oC selama 15 menit.
0 1 2 3 4 5 6 7 8 9 Gambar 4 Standar pemberian skor blas daun menurut
9 Amplifikasi Fragmen DNA untuk Marka Gen Ketahanan Blas
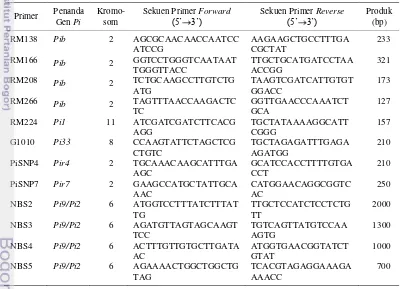
Reaksi PCR terdiri atas 5 µl KAPA Kit (larutan penyangga, dNTP, enzim Taq polymerase), 3 µl DNA 10 ng/µl, 0.5 µl primer 10 µM (forward dan reverse), dan dH2O hingga 10 µl volume total. Dua belas primer (Tabel 7) digunakan untuk
mengamplifikasi fragmen penanda gen ketahanan blas pada tanaman yang diuji. Proses PCR untuk primer RM138, RM208, RM224, dan RM266 terdiri atas tahap denaturasi awal pada suhu 94 oC selama 5 menit yang diikuti dengan 36 siklus, setiap siklus terdiri atas tahap denaturasi pada suhu 94 oC selama 1 menit, penempelan primer pada suhu 55 oC selama 1 menit, dan pemanjangan primer pada suhu 72 oC selama 2 menit. Pemanjangan akhir dilakukan pada suhu 72 oC selama 10 menit. Sementara itu, proses PCR untuk primer RM166 terdiri atas tahap denaturasi awal pada suhu 95 oC selama 3 menit yang diikuti dengan 35 siklus, setiap siklus terdiri atas tahap denaturasi pada suhu 95oC selama 30 detik, penempelan primer pada suhu 61 oC selama 30 detik, dan pemanjangan primer pada suhu 72 oC selama 1 menit. Pemanjangan akhir dilakukan pada suhu 72 oC selama 5 menit (gramene.org).
Proses PCR untuk primer PiSNP4 dan PiSNP7 terdiri atas tahap denaturasi awal pada suhu 95 oC selama 4 menit yang diikuti dengan 42 siklus. Pada 14 siklus pertama, setiap siklus terdiri atas tahap denaturasi pada suhu 95 oC selama 45 detik, penempelan primer pada suhu 61.5 oC selama 45 detik dengan penurunan suhu 0.5 oC per siklus, dan pemanjangan primer pada suhu 72 oC selama 30 detik. Pada 28 siklus berikutnya, setiap siklus terdiri atas tahap denaturasi pada suhu 95 oC selama 45 detik, pemanjangan primer pada suhu 55 oC selama 45 detik, dan pemanjangan primer pada suhu 72 oC selama 30 detik.
Tabel 7 Primer untuk amplifikasi fragmen gen penanda ketahanan blas
Primer Penanda
RM138 Pib 2 AGCGCAACAACCAATCC
ATCCG
AAGAAGCTGCCTTTGA CGCTAT
233
RM166 Pib 2 GGTCCTGGGTCAATAAT
TGGGTTACC
TTGCTGCATGATCCTAA ACCGG
321
RM208 Pib 2 TCTGCAAGCCTTGTCTG
ATG
TAAGTCGATCATTGTGT GGACC
173
RM266 Pib 2 TAGTTTAACCAAGACTC
TC
GGTTGAACCCAAATCT GCA
127
RM224 Pi1 11 ATCGATCGATCTTCACG
AGG
TGCTATAAAAGGCATT CGGG
157
G1010 Pi33 8 CCAAGTATTCTAGCTCG
CTGTC
TGCTAGAGATTTGAGA AGATGG
210
PiSNP4 Pir4 2 TGCAAACAAGCATTTGA
AGC
GCATCCACCTTTTGTGA CCT
210
PiSNP7 Pir7 2 GAAGCCATGCTATTGCA
AAC
CATGGAACAGGCGGTC AC
250
NBS2 Pi9/Pi2 6 ATGGTCCTTTATCTTTAT
TG
TTGCTCCATCTCCTCTG TT
2000
NBS3 Pi9/Pi2 6 AGATGTTAGTAGCAAGT
TCC
TGTCAGTTATGTCCAA AGTG
1300
NBS4 Pi9/Pi2 6 ACTTTGTTGTGCTTGATA
AC
ATGGTGAACGGTATCT GTAT
1000
NBS5 Pi9/Pi2 6 AGAAAACTGGCTGGCTG
TAG
TCACGTAGAGGAAAGA AAACC
10
Pemanjangan akhir dilakukan pada suhu 72 oC selama 5 menit (Utami et al. 2008). Sementara itu, proses PCR untuk primer G1010 terdiri atas tahap denaturasi awal pada suhu 95 oC selama 3 menit yang diikuti dengan 35 siklus, setiap siklus terdiri atas tahap denaturasi pada suhu 95 oC selama 30 detik, penempelan primer pada suhu 59 oC selama 30 detik, dan pemanjangan primer pada suhu 72 oC selama 1 menit. Pemanjangan akhir dilakukan pada suhu 72 oC selama 5 menit (Utami et al. 2011). Proses PCR untuk primer NBS2, NBS3, NBS4, dan NBS5 dilakukan menurut Qu et al. (2006).
Produk PCR dipisahkan pada elektroforesis gel agarosa 2% dalam TBE 0.5x pada tegangan 100 volt selama 30 menit. Pita fragmen DNA selanjutnya divisualisasi di bawah sinar UV setelah direndam dalam etidium bromida.
Analisis Data
Asosiasi antara marka molekuler dan respons fenotipe dianalisis menggunakan Program Tassel 2.0. Marka yang berasosiasi dengan respons fenotipe (p_Value kurang dari 0.05) menunjukkan bahwa marka tersebut berasosiasi dengan gen ketahanan yang menyebabkan ketahanan pada tanaman terhadap ras blas yang diinokulasikan.
3
HASIL DAN PEMBAHASAN
Respons Fenotipe terhadap Ras Blas
Hasil pengamatan respons ketahanan menurut Hayashi et al. (2009) menunjukkan varietas diferensial yang memiliki gen Pish, Pi5, Pi1, Piz, dan Pita tahan terhadap ras 123. Hal ini menunjukkan bahwa gen-gen tersebut mengenali gen Avr dari ras 123 dan melanjutkan untuk menginisiasi sistem mekanisme pertahanan dari tanaman. Sebaliknya, gen Pia, Pii, Piks, Pikm, dan Pita2 rentan terhadap ras 123. Hal ini berarti bahwa ras 123 dapat menyerang semua kelompok patotipe kecuali kelompok IV yang memiliki gen ketahanan Piz. Respons yang hampir sama juga ditunjukkan pada ras 173. Beberapa gen ketahanan: Pish, Pii, Pi5, Piks, Pikm, Pi1, dan Pita rentan terhadap ras ini. Gen-gen ini dikelompokkan dalam kelompok patotipe yang berbeda, kelompok I (Pish), II (Pii, Pi5), III (Piks, Pikm, Pi1), dan V (Pita). Hal ini mengindikasikan bahwa ras 173 tidak mampu menyerang gen ketahanan pada kelompok IV (Tabel 8). Namun demikian, ras 173 memiliki spektrum virulensi paling luas dibanding ras 123 dan 133.
Varietas diferensial yang memiliki gen Pia, Pish, Pi5, Pikm, Pi1, Pita, dan Pita2 tahan terhadap ras 133. Hal ini menunjukkan bahwa gen-gen ini berkontribusi untuk melindungi tanaman dari serangan ras 133. Dalam respons yang berbeda, beberapa gen dalam kelompok patotipe yang berbeda rentan terhadap ras 133, gen-gen ini adalah: Pii (II), Piks (III), dan Piz (IV). Hal ini mengindikasikan bahwa ras 133 memiliki spektrum virulensi paling sempit.
11 respons patotipe yang sama terhadap isolat blas diferensial standar Jepang. Namun, hasil menunjukkan gen-gen ketahanan dari kelompok patotipe yang sama belum tentu memiliki respons patotipe yang sama terhadap isolat blas di Indonesia. Gen Pia dan Pish dari kelompok patotipe I memberikan respons yang berbeda terhadap ras 123 dan 173. Begitu juga gen Pii dan Pi5 dari kelompok II, keduanya memberikan respons berbeda terhadap ras 123 dan 133. Piks, Pikm, dan Pi1 dari kelompok III memberikan respons yang berbeda terhadap ras 123 dan 133. Pita dan Pita2 dari kelompok V juga memberikan respons yang berbeda terhadap ras 123 dan 173.
Pengelompokan patotipe pada galur monogenik LTH dilakukan berdasarkan respons patotipe terhadap isolat blas diferensial standar Jepang. Perbedaan respons patotipe galur monogenik LTH terhadap isolat blas di Indonesia dengan isolat blas diferensial Jepang diduga disebabkan adanya perbedaan gen virulensi pada isolat blas yang digunakan. Hal ini mengindikasikan galur monogenik LTH tidak dapat digunakan sebagai pembanding respons ketahanan tanaman terhadap isolat blas Indonesia.
Respons ketahanan yang hampir sama juga ditunjukkan berdasarkan sistem evaluasi standar menurut IRRI (1996), kecuali terhadap ras 133. Varietas diferensial yang memiliki gen Pia rentan terhadap ras tersebut. Hal ini menunjukkan gen ini tidak berkontribusi melindungi tanaman dari serangan ras 133. Meskipun demikian, ras 133 tetap memiliki spektrum virulensi paling sempit dibanding ras 123 dan 173.
Varietas diferensial berfungsi sebagai pembanding respons ketahanan (patotipe) terhadap suatu ras patogen. Pola reaksi terhadap patogen blas pada varietas diferensial selanjutnya dibandingkan dengan respons ketahanan pada tetua persilangan dan galur haploid ganda yang diuji. Tetua persilangan menunjukkan reaksi yang beragam terhadap isolat blas yang diinokulasikan. Bio110 bersifat medium tahan terhadap ras 133, tetapi rentan terhadap ras 123 dan 173, sedangkan Markuti rentan terhadap semua ras. F1 dari persilangan kedua
tetua tersebut tahan terhadap 133. Sementara itu, IR54 medium tahan terhadap ras 133 dan 173, tetapi rentan terhadap ras 123, sedangkan Parekaligolara medium
Tabel 8 Pola reaksi varietas diferensial terhadap tiga isolat blas
No Varietas
12
tahan terhadap semua isolat. F1 dari persilangan kedua tetua tersebut tahan
terhadap semua ras. Dengan demikian, persilangan ganda tersebut dapat menghasilkan galur yang tahan terhadap ketiga ras (Tabel 9).
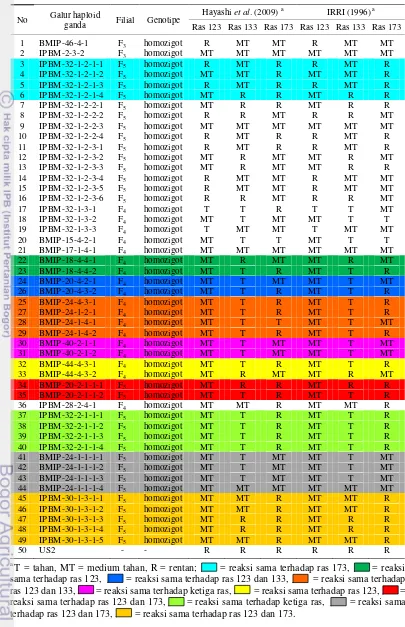
Sementara itu, hasil evaluasi fenotipe pada galur haploid ganda baik menurut Hayashi et al. (2009) maupun IRRI (1996) menunjukkan 2 galur tahan terhadap ras 123, 38 galur medium tahan, dan 9 galur bersifat rentan. Dua puluh satu galur tahan terhadap ras 133, 17 galur medium tahan, dan 11 galur bersifat rentan. Dua galur tahan terhadap ras 173, 21 galur medium tahan, dan 26 galur bersifat rentan. Namun berdasarkan kedua metode penentuan respons ketahanan tersebut, 4 galur memiliki respons yang berbeda terhadap ras 173: IPBM-32-1-2-3-3, IPBM-32-1-3-1, IPBM-32-1-3-2, dan BMIP-24-1-4-1 (Tabel 10).
Penamaan pada galur haploid ganda memiliki makna sebagai berikut. Empat huruf pada bagian pertama menunjukkan simbol varietas yang digunakan sebagai tetua betina dan tetua jantan. BMIP memiliki arti F1 dari persilangan
Bio110/Markuti merupakan tetua betina, sedangkan F1 dari persilangan
IR54/Parekaligolara merupakan tetua jantan. Sebaliknya, IPBM memiliki arti F1
dari persilangan IR54/Parekaligolara merupakan tetua betina, sedangkan F1 dari
persilangan Bio110/Markuti merupakan tetua jantan. Sementara itu, tanda hubung menunjukkan tingkat generasi F dari hasil perkawinan sendiri (selfing). Nomor kode setelah tanda hubung pertama menunjukkan nomor kalus, nomor kode setelah tanda hubung kedua menunjukkan nomor seleksi pada tanaman hijau, dan nomor kode setelah tanda hubung ketiga dan seterusnya menunjukkan tahap seleksi tanaman yang sudah dilakukan berdasarkan penampilan agronomi.
Beberapa galur haploid ganda menunjukkan reaksi yang sama terhadap ketiga isolat blas yang diinokulasikan (Tabel 10). Semakin banyak tahapan generasi, galur-galur tersebut sudah tidak mengalami segregasi karakter ketahanan blas terhadap ketiga isolat. Oleh karena itu, untuk meningkatkan efek seleksi, perlu dipertimbangkan perbanyakan tanaman pada generasi berikutnya apabila dalam seleksi tidak terlihat adanya segregasi untuk karakter ketahanan blas. Penggunaan material tanaman dapat dilakukan pada generasi terakhir yang menunjukkan respons ketahanan blas yang sama sehingga dapat meningkatkan efisiensi biaya.
13 Tabel 10 Pola reaksi terhadap tiga ras blas pada galur haploid ganda
14
Evaluasi Genotipe Menggunakan Marka Molekuler
Respons ketahanan terhadap patogen blas dikendalikan oleh gen-gen ketahanan pada tanaman inang. Keberadaan gen-gen ketahanan tersebut dapat dideteksi menggunakan marka molekuler. Pada penelitian ini, 12 primer digunakan untuk mengamplifikasi fragmen penanda gen Pi1, Pi2, Pi9, Pi33, Pib, Pir4, dan Pir7 pada populasi tanaman yang diuji. Hasil PCR menunjukkan 8 primer mampu mengamplifikasi fragmen DNA untuk marka gen ketahanan (RM138, RM166, RM208, RM266, RM224, PiSNP4, PiSNP7, dan G1010), sedangkan empat primer lainnya (NBS2, NBS3, NBS4, dan NBS5) tidak mengamplifikasi fragmen DNA untuk marka gen ketahanan target. Hasil proses PCR menggunakan primer RM166, RM224, dan PiSNP7 ditunjukkan pada Gambar 5-7, sedangkan pemberian skor produk PCR ditunjukkan pada Tabel 11.
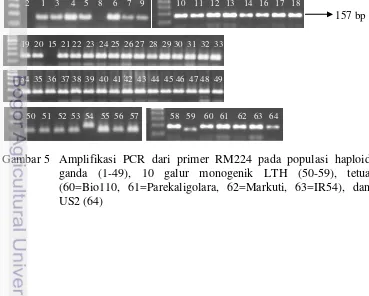
Berdasarkan hasil PCR menggunakan primer RM224 (Gambar 5) dihasilkan pita polimorfis berukuran 157 bp pada varietas diferensial IRBLks-F5 (pita nomor 54) dan IRBLta-CT2 (pita nomor 58). Namun, varietas IRBLks-F5 menunjukkan reaksi rentan terhadap semua isolat yang diinokulasikan, sedangkan IRBLta-CT2 menunjukkan reaksi medium tahan terhadap ras 123 dan 133 namun rentan terhadap ras 173. Pita polimorfis tersebut juga dimiliki oleh keempat tetua, US2, dan galur haploid ganda kecuali galur 2-3-2 (pita nomor 2), IPBM-32-1-2-2-2 (pita nomor 8), dan IPBM-32-1-2-3-5 (pita nomor 15). Galur IPBM-2-3-2 menunjukkan reaksi medium tahan terhadap ketiga ras, IPBM-32-1-2-2-2 hanya tahan terhadap ras 173, sedangkan IPBM-32-1-2-3-5 medium tahan terhadap ras 123 dan 173. Namun, varietas US2 menunjukkan respons rentan terhadap ketiga ras (Tabel 9). Hal ini dikarenakan RM224 bukan merupakan bagian dari gen, tetapi merupakan marka SSR (mikrosatelit) penanda suatu lokus. Fuentes et al. (2008) memetakan marka RM224 berjarak 0.0 cM dengan gen Pi1, namun pada penelitian ini menunjukkan RM224 tidak berhubungan langsung dengan sifat ketahanan tanaman terhadap ras 123, 133, dan 173.
Gambar 5 Amplifikasi PCR dari primer RM224 pada populasi haploid ganda (1-49), 10 galur monogenik LTH (50-59), tetua (60=Bio110, 61=Parekaligolara, 62=Markuti, 63=IR54), dan US2 (64)
157 bp
2 1 3 4 5 8 6 7 9 10 11 12 13 14 16 17 18
19 20 15 21 22 23 24 25 26 27 28 29 30 31 32 33
15
Gambar 6 Amplifikasi PCR dari primer RM166 pada populasi haploid ganda (1-49), 10 galur monogenik LTH (50-59), tetua (60=Bio110, 61=Parekaligolara, 62=Markuti, 63=IR54), dan US2 (64)
Pada proses PCR menggunakan primer RM166 (Gambar 6) dihasilkan pita polimorfis berukuran 321 bp pada tetua, US2, dan galur haploid ganda, kecuali galur IPBM-32-1-2-3-4 dan IPBM-32-2-1-1-4. Galur IPBM-32-1-2-3-4 menunjukkan reaksi medium tahan terhadap ras 133 dan 173 tetapi rentan terhadap ras 123, sedangkan IPBM-32-2-1-1-4 medium tahan terhadap ras 123, tahan terhadap ras 133, tetapi rentan terhadap ras 173. Pada semua varietas diferensial dihasilkan pita berukuran lebih besar, namun masing-masing menunjukkan reaksi ketahanan yang spesifik terhadap isolat blas yang diujikan. Sementara itu, US2 menunjukkan respons rentan terhadap ketiga ras. Hal ini dikarenakan RM166 juga bukan bagian dari gen, tetapi merupakan marka mikrosatelit penanda suatu lokus. Fjellstrom et al. (2004) memetakan RM166 terpaut dekat dengan gen Pib pada jarak 2.3 cM, namun pada penelitian ini menunjukkan primer tersebut tidak berhubungan langsung dengan sifat ketahanan pada populasi tanaman diuji terhadap ras 123, 133, dan 173.
Gambar 7 Amplifikasi PCR dari primer PiSNP7 pada populasi haploid ganda (1-49), 10 galur monogenik LTH (50-59), tetua (60=Bio110, 61=Parekaligolara, 62=Markuti, 63=IR54), dan US2 (64)
Sementara itu, hasil PCR menggunakan primer PiSNP7 menghasilkan pita spesifik berukuran 250 bp pada tetua, varietas diferensial, dan galur haploid ganda kecuali galur IPBM-32-1-2-2-2. Galur IPBM-32-1-2-2-2 menunjukkan respons rentan terhadap ras 123 dan 133, namun medium tahan terhadap ras 173. Varietas US2 tidak menghasilkan pita target saat amplifikasi PCR menggunakan marka PiSNP7 dan menunjukkan respons rentan terhadap ketiga ras. Hasil ini mengindikasikan primer PiSNP7 berhubungan langsung dengan respons ketahanan tanaman yang diuji terhadap ras 123 dan 133 karena adanya pita spesifik mengindikasikan respons tahan pada tanaman terhadap ras 123 dan 133.
1 2 3 4 5 6 7 8 9 10 14 11 12 13 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32
33 40 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 64
321 bp 50 51 52 53 54 55 56 57 58 59 60 61 62 63
1 2 3 4 5 6 7 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28
29 8 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 64
250 bp
16
Tabel 11 Amplifikasi fragmen DNA target menggunakan marka molekuler
No Galur padi
17
Keterkaitan (Asosiasi) Marka Molekuler dengan Respons Fenotipe
Marka molekuler yang berasosiasi dengan respons ketahanan terhadap patogen blas menunjukkan marka tersebut dapat digunakan sebagai marka gen ketahanan. Hal ini berarti gen-gen tersebut berkontribusi memberikan ketahanan terhadap patogen yang diujikan. Hasil asosiasi antara marka molekuler dan respons fenotipe menurut Hayashi et al. (2009) menunjukkan terdapat tiga primer yang berasosiasi dengan respons fenotipe (p_value kurang dari 0.05) (Tabel 12). Ketiga primer tersebut meliputi RM224, RM166, dan PiSNP7 yang secara berturut-turut mengamplifikasi fragmen DNA untuk marka gen Pi1, Pib, dan Pir7. Primer RM224 dan PiSNP7 berasosiasi dengan ketahanan terhadap ras 123 dan 133, sedangkan primer RM166 berasosiasi dengan ketahanan terhadap ras 123. Sementara itu, primer RM138, RM208, RM266, PiSNP4, dan G1010 tidak berasosiasi dengan respons ketahanan terhadap ras 123, 133, dan 173. Hal ini berarti kelima primer tersebut tidak dapat digunakan sebagai marka seleksi berdasarkan respons ketahanan pada populasi haploid ganda yang diuji.
Berdasarkan hasil asosiasi respons fenotipe dengan evaluasi genotipe, marka RM166 signifikan terhadap sifat ketahanan terhadap ras 123. Hal ini berarti gen Pib berkontribusi membentuk ketahanan tanaman terhadap ras 123. Gen ketahanan ini mampu mengenali protein yang dihasilkan oleh gen avirulen (Avr) dari ras 123. Adapun marka RM224 dan PiSNP7 signifikan terhadap sifat ketahanan tanaman terhadap ras 123 dan 133. Hal ini berarti gen Pi1 dan Pir7 berkontribusi membentuk ketahanan tanaman terhadap ras 123 dan 133.
No Galur padi
Gen ketahanan blas pada masing-masing primera RM138
+ = pita fragmen DNA target ada, - = pita fragmen DNA target tidak ada
Tabel 12 Analisis asosiasi marka molekuler dengan respons fenotipe menurut Hayashi et al. (2009)
Primer Penanda Pi Berasosiasi dengan ras p_value
RM224 Pi1 123 0.0203
RM224 Pi1 133 0.0252
RM166 Pib 123 0.0363
PiSNP7 Pir7 123 0.0102
18
Kemampuan penyerangan patogen dipengaruhi oleh faktor virulensi yang dimiliki oleh ras patogen blas tertentu. Mekanisme penyerangan patogen terjadi berdasarkan interaksi gene-to-gene antara gen ketahanan blas pada tanaman inang dan gen virulen (avr) pada patogen blas. Pada tanaman inang umumnya gen yang memberikan respons tahan bersifat dominan (R), sedangkan gen yang memberikan respons rentan bersifat resesif (r). Sementara itu, pada patogen, gen avirulen (tidak memiliki kemampuan untuk menginfeksi) bersifat dominan (Avr), sedangkan gen virulen (mampu menginfeksi) bersifat resesif (avr). Pada interaksi Avr-R bersifat incompatible, artinya tanaman inang memiliki gen ketahanan (R) yang mampu mengenali gen avirulen (A) dari patogen sehingga tanaman bersifat tahan. Sementara itu, pada interaksi Avr-r bersifat compatible, tanaman tidak memiliki gen ketahanan R untuk mengenali gen avirulen dari patogen sehingga patogen dapat menyerang dengan gen virulen yang lain. Interaksi avr-R menyebabkan respons rentan karena meskipun tanaman mempunyai gen ketahanan R, patogen tidak memiliki gen avirulen yang dikenali oleh gen ketahanan R sehingga mekanisme pertahanan tidak diaktifkan. Interaksi avr-r menimbulkan reaksi rentan karena tanaman tidak memiliki ketahanan dan patogen bersifat virulen sehingga patogen menyerang tanaman (Agrios 2005).
Salah satu gen avr yang telah dikarakterisasi adalah gen ACE1-avr. Gen ini mengkodekan protein poli-keto sintetase yang mengandung 4035 asam amino yang mengkatalisasi penetrasi patogen pada tanaman inang. Gen ACE1-avr memiliki dua kopi gen yang menunjukkan genotipe berbeda pada isolat blas. Ada tiga genotipe isolat blas. Pertama, genotipe Guy11 (ACE1-vir) memiliki satu kopi gen ACE1 pada kromosom 1 dari genom blas, bersifat avirulen. Kedua, genom CM28 (ACE1-vir1) memiliki satu kopi gen ACE1 pada kromosom 6, bersifat virulen. Genotipe yang lain memiliki dua kopi gen ACE1, Guy11 dan CM28, dan isolat ini merupakan genotipe PH14 (ACE1-vir2) (Couch et al. 2005).
Pir4 dan Pir7 termasuk kelompok gen QTL yang dideteksi pada populasi
19
(a) (b)
Gambar 8 Peta genetik kromosom 2 oleh Temnykh et al. (2001) (a); analisis kandidat gen ketahanan Pir pada kromosom 2 pada posisi antara primer RM263 dan RM250 (Utami et al. 2008) (b)
20
Gambar 9 Analisis pemetaan gen Pir4 dan Pir7 pada populasi backcross dari persilangan antara IR64 dan Oryza rufipogon; gen Pir4 terpetakan pada posisi 110.9 cM, sedangkan gen Pir7 terpetakan pada posisi 122.8 cM (Utami et al. 2008)
Gen Pi1 merupakan gen ketahanan tunggal yang terdapat pada kromosom 11. Dua puluh empat pasang primer untuk 19 lokus mikrosatelit (Gambar 10b) pada jarak antara 110.0-123.2 cM dari kromosom 11 diseleksi dari database Gramene (www.gramene.org) yang memiliki jarak kedekatan relatif dengan gen Pi1 berdasarkan peta genetik padi (Gambar 10c). Berdasarkan analisis keterpautan, primer RM224 terpaut di lokasi yang sama (0.0 cM) dengan gen Pi1 (Gambar 10a) yang dipetakan pada populasi F3 dari persilangan antara galur
C101LAC (tahan) dan C101A51 (rentan) (Fuentes et al. 2008).
21
(c) (b) (a)
Gambar 10 Peta genetik kromosom 11 (a) oleh Temnykh et al. (2001); daerah antara 110.0 dan 123.2 cM (b) dilengkapi dengan informasi yang tersedia di database Gramene (www.gramene.org); analisis keterpautan marka RM123*I dan RM224 pada posisi 0.0 cM terhadap gen ketahanan Pi1(t) (c)
Gen Pib terpetakan pada kromosom 2 yang terpaut dekat dengan marka RM166 dan RM138 pada posisi antara 183.0-196.8 cM (Gambar 11a). Berdasarkan analisis keterpautan pada populasi F12 dari persilangan Gulfmont
(tahan) dan Te-Qing (rentan) terhadap isolat blas ras IE-1K, marka RM208 terpaut di lokasi yang sama (0.0 cM) dengan gen Pib. Sementara itu, marka RM138, RM166, dan RM266 terpaut dekat dengan gen Pib secara berturut-turut pada posisi 3.1 cM, 2.3 cM, dan 2.2 cM (Fjellstrom et al. 2004).
Hasil menunjukkan hanya primer RM166 yang berasosiasi dengan gen Pib yang menyebabkan ketahanan pada populasi haploid ganda yang diuji. Respons tahan yang dikontribusikan oleh gen Pib pada populasi haploid ganda disebabkan oleh adanya interaksi antara gen avirulen (Avr) dari patogen dengan gen ketahanan Pib. Gen Pib mampu mengenali gen avirulen dari ras 123 dan 133 sehingga mengaktifkan sistem pertahanan pada tanaman. Tidak adanya asosiasi antara marka RM138, RM208, dan RM266 dengan respons ketahanan yang dikontribusikan oleh gen Pib menunjukkan marka molekuler yang didesain untuk suatu populasi persilangan belum tentu dapat digunakan sebagai marka seleksi untuk populasi persilangan yang lain dengan latar belakang genetik yang berbeda.
22
(a) (b)
Gambar 11 Peta genetik kromosom 2 (a) oleh Temnykh et al. (2001); peta genetik marka RM166, RM208, RM266, dan RM138 pada kromosom 2 yang dekat dengan gen Pib pada populasi F12 dari persilangan Gulfmont
(tahan) dan Te-Qing (rentan) (Fjellstrom et al. 2004)
Gen Pi33 terdapat pada lengan pendek dari kromosom 8 padi dan terpetakan dekat dengan marka SSR RM44 dan RM72 (Gambar 12a) oleh Temnykh et al. (2001). Gen yang berinteraksi dengan gen avirulen ACE1 dari P. Grisea ini kemudian dipetakan menggunakan 4 marka SSR RM44, RM72, RM404, dan RM483, 4 marka RFLP R1813, G1010, C483, dan S1633B, serta marka Y2643L sebagai probe Yeast Artificial Chromosome (YAC) pada populasi haploid ganda hasil persilangan antara IR64 (tahan) dan Azucena (rentan). Berdasarkan analisis keterpautan, primer RM72 dan RM44 berlokasi di antara lokus Pi33 pada jarak
cM
2.3
2.2 0.9
23 10.6 0.1 cM satu dengan yang lain (Gambar 12b). Sementara itu, keempat marka RFLP G1010, R1813, C483, dan S1633B berlokasi di antara RM72 dan RM44. Gen Pi33 terpetakan pada jarak 1.6 0.2 cM di antara marka RM72, G1010, dan R1813 dengan Y2643L (Berruyer et al. 2003). Namun, hasil menunjukkan bahwa primer G1010 tidak berasosiasi dengan gen Pi33 pada populasi haploid ganda. Hal ini diduga karena masih terdapat jarak genetik antara marka G1010 dengan lokus gen Pi33 yang memungkinkan terjadinya rekombinasi dengan demikian primer G1010 tidak dapat digunakan sebagai marka seleksi gen Pi33 pada populasi haploid ganda yang diuji.
(a) (b)
Gambar 12 Peta genetik kromosom 8 (a) oleh Temnykh et al. (2001); pemetaan genetik gen Pi33 pada populasi rekombinan haploid ganda hasil persilangan IR64 x Azucena menggunakan 8 marka SSR dan RFLP yang memiliki jarak genetik paling dekat dengan gen Pi33 (b)
Sementara itu, hasil asosiasi antara marka molekuler dan respons fenotipe menurut IRRI (1996) menunjukkan tiga primer: RM224, RM166, dan PiSNP7, berasosiasi dengan respons ketahanan pada galur haploid ganda yang diuji. Primer
24
RM224 dan PiSNP7 berasosiasi dengan respons ketahanan terhadap ras 123 dan 133. Sementara itu, Pib tidak berasosiasi dengan respons ketahanan terhadap ras 123, akan tetapi berasosiasi dengan ketahanan terhadap ras 133 (Tabel 13). Hasil penentuan respons ketahanan yang tidak konsisten antara Hayashi et al. (2009) dan IRRI (1996) menunjukkan bahwa sistem evaluasi standar (SES) dari IRRI (1996) masih diperlukan untuk pengujian respons ketahanan pada tanaman padi.
Asosiasi antara marka RM224 dan RM166 terhadap gen ketahanan menunjukkan tingkat signifikansi yang tinggi (p-value < 0.05). Namun, reaksi PCR menggunakan kedua marka tersebut pada varietas US2 menghasilkan pita DNA target (Tabel 11). Hasil ini menunjukkan marka RM224 dan RM166 tidak konsisten dengan respons varietas US2. Hal ini diduga karena masih adanya jarak genetik antara kedua marka tersebut dengan gen ketahanan target sehingga memungkinkan terjadinya rekombinasi genetik. RM224 dan RM166 merupakan marka SSR yang mengamplifikasi sekuen berulang (mikrosatelit) terkonservasi pada tanaman padi. Daerah mikrosatelit yang teramplifikasi tersebut diduga masih sama antartanaman yang diuji sehingga menghasilkan pita polimorfik yang tidak konsisten dengan respons ketahanan tanaman. Sementara itu, reaksi PCR menggunakan marka PiSNP7 pada varietas US2 tidak menghasilkan pita DNA target. Hasil ini menunjukkan marka PiSNP7 lebih konsisten dengan respons varietas rentan US2 terhadap isolat ras 123, 133, dan 173. PiSNP7 merupakan marka spesifik yang langsung mengamplifikasi bagian aktif dari gen Pir7. Dengan demikian, marka PiSNP7 memiliki tingkat presisi paling tinggi untuk digunakan sebagai marka seleksi pada galur haploid ganda berdasarkan gen ketahanan blas Pir7. Dengan demikian, berdasarkan hasil amplifikasi yang berhubungan dengan sifat ketahanan tersebut maka primer RM224 dan RM166 belum dapat diaplikasikan dalam proses seleksi sifat ketahanan terhadap penyakit blas, sedangkan primer PiSNP7 dapat digunakan sebagai marka seleksi sifat ketahanan pada populasi haploid ganda yang diuji.
Sementara itu, dari 8 primer yang menghasilkan pita DNA target (RM138, RM166, RM208, RM266, RM224, PiSNP4, PiSNP7, dan G1010), tidak ada primer yang berasosiasi dengan respons ketahanan tanaman terhadap ras 173. Hasil ini mengindikasikan respons ketahanan terhadap ras 173 tidak dikontribusikan oleh gen Pi1, Pi2, Pi9, Pib, Pi33, Pir4, maupun Pir7. Hal ini berarti gen-gen ketahanan tersebut tidak mengenali gen avirulen (Avr) dari ras 173 sehingga mekanisme pertahanan tanaman tidak diaktifkan.
IR54 dan Bio110 merupakan donor utama gen ketahanan blas terhadap galur haploid ganda yang diuji. Primer NBS2, NBS3, NBS4, dan NBS5 digunakan untuk mengamplifikasi fragmen DNA penanda gen Pi2/Pi9 pada IR54. Keempat
Tabel 13 Analisis asosiasi marka molekuler dengan respons fenotipe menurut IRRI (1996)
Primer Penanda Pi Berasosiasi dengan ras p_Value
RM224 Pi1 123 0.0307
RM224 Pi1 133 0.0109
RM166 Pib 133 0.0467
PiSNP7 Pir7 123 0.0040
25 primer ini merupakan primer spesifik untuk gen Pi9 yang berlokasi pada kromosom 6 pada posisi pemetaan 58.7 cM. Primer NBS2 merupakan satu-satunya primer yang mengamplifikasi fragmen fungsional yang menyebabkan ketahanan dari gen tersebut terhadap P.grisea. Gen Pi9 memiliki spektrum ketahanan yang luas dan terpaut dengan gen Pi2 (Qu et al. 2006). Namun, amplifikasi PCR menggunakan keempat primer tersebut tidak menghasilkan pita DNA target. Hal ini berarti IR54 tidak memiliki gen Pi2 maupun Pi9.
Berdasarkan evaluasi fenotipe, 2 galur menunjukkan respons paling tahan terhadap ras 123, yaitu IPBM-32-1-3-1 dan IPBM-32-1-3-3. Kedua galur tersebut terdeteksi memiliki gen Pib, Pi1, dan Pir7 menggunakan marka RM166, RM224, dan PiSNP7 (Tabel 10) yang berkontribusi membentuk ketahanan terhadap ras 123. Sementara itu, galur terpilih yang paling tahan terhadap ras 133 sebanyak 21 galur, yaitu IPBM-32-1-3-1, IPBM-32-1-3-2, BMIP-15-4-2-1, BMIP-18-4-4-2, BMIP-20-4-2-1, BMIP-24-4-3-1, BMIP-20-4-3-2, BMIP-24-1-2-1, BMIP-24-1-4-1, BMIP-24-1-4-2, BMIP-40-2-1-BMIP-24-1-4-1, BMIP-40-2-1-2, BMIP-44-4-3-BMIP-24-1-4-1, BMIP-20-2-1-1-2, IPBM-32-2-1-1-1, IPBM-32-2-BMIP-20-2-1-1-2, IPBM-32-2-1-1-3, IPBM-32-2-1-1-4, BMIP-24-1-1-1-1, BMIP-24-1-1-1-2, BMIP-24-1-1-1-3. Kedua puluh satu galur tersebut terdeteksi memiliki gen Pi1 dan Pir7 menggunakan marka RM224 dan PiSNP7 yang berkontribusi membentuk ketahanan terhadap ras 133. Sementara itu, galur terpilih yang paling tahan terhadap ras 173 sebanyak 2 galur, yaitu BMIP-15-4-2-1 dan BMIP-24-1-4-1 berdasarkan Hayashi et al. (2009), IPBM-32-1-3-2 dan BMIP-15-4-2-1 berdasarkan IRRI (1996). Gen ketahanan yang berkontribusi membentuk ketahanan pada galur-galur tersebut belum diketahui karena marka molekuler yang digunakan tidak berasosiasi dengan gen ketahanan target. Untuk pemilihan galur harapan harus memperhatikan karakter agronomi seperti tinggi tanaman, umur berbunga, umur panen, serta komponen hasil.
Penggunaan marka molekuler terbukti bermanfaat dalam membantu pemulia dalam menyeleksi galur padi tahan blas. Seleksi menggunakan marka molekuler dipandang lebih presisi dibanding seleksi berdasarkan respons fenotipe saja. Hal ini karena seleksi menggunakan marka langsung berdasarkan keberadaan gen ketahanan sehingga lebih presisi. Setiap gen ketahanan R memiliki respons yang berbeda dalam interaksi gen ketahanan dengan gen avr pada setiap ras patogen blas. Namun, marka molekuler yang didesain untuk populasi persilangan tertentu harus dievaluasi terlebih dahulu sebelum diaplikasikan sebagai marka seleksi pada populasi persilangan yang lain.
4
SIMPULAN DAN SARAN
26
ketahanan terhadap ras 133. Marka PiSNP7 memiliki tingkat presisi paling tinggi untuk digunakan sebagai marka seleksi ketahanan blas pada populasi haploid ganda hasil persilangan dari IR54/Parekaligolara//Bio110/Markuti. Tidak ada primer yang berasosiasi dengan respons ketahanan tanaman terhadap ras 173. Hal ini mengindikasikan respons ketahanan terhadap ras 173 tidak dikontribusikan oleh gen Pi1, Pi2, Pi9, Pi33, Pib, Pir4, dan Pir7. Dua galur terpilih paling tahan terhadap ras 123, 21 galur paling tahan terhadap ras 133, dan 2 galur paling tahan terhadap ras 173. Analisis sekuen basa perlu dilakukan untuk mengetahui variasi sekuen basa nukleotida gen Pi1, Pib, dan Pir7 di antara galur-galur terpilih.
DAFTAR PUSTAKA
Agrios GN. 2005. Plant Pathology. 8th ed. California (USA): Elsevier Academic Press.
Berruyer R, Adreit H, Milazzo J, Gaillard S, Berger A, Dioh W, Lebrun MH, Tharreau D. 2003. Identification and fine mapping of Pi33, the rice resistance gene corresponding to the Magnaporthe grisea avirulence gene ACE1. Theor Appl Genet. 107:1139-1147.
Couch BC, Fudal I, Lebrun MH, Tharreau D, Valent B, van Kim P, Nottéghem JL, Kohn LM. 2005. Origins of host-specific populations of the blast pathogen Magnaporthe oryzae in crop domestication with subsequent expansion of pandemic clones on rice and weeds of rice. Genetics. 170:613-630.
Doyle JJ, Doyle JL. 1987. A rapid DNA isolation from small amount of fresh leaf tissue. Phytochem Bull. 19:11-15.
Fjellstrom R, Conaway-Bormans CA, McClung AM, Marchetti MA, Shank AR, Par WD. 2004. Development of DNA markers suitable for marker assisted selection of three Pi genes conferring resistance to multiple Pyricularia grisea pathotypes. Crop Sci. 44:1790-1798.
Fuentes JL, Correa-Victoria FJ, Escobar F, Prado G, Aricapa G, Duque MC, Tohme J. 2008. Identification of microsatellite markers linked to the blast resistance gene Pi-1(t) in rice. Euphytica. 160:295-304.
Hayashi N, Fukuta Y. 2009. Development and Characterization of Blast Resistance Using Differential Varieties in Rice: Proposal for a new international system of differentiating races of blast (Pyricularia oryzae Cavara) by using LTH monogenic lines in rice (Oryza sativa L.). Tsukuba: Japan International Research Center for Agricultural Sciences.
Hayashi N, Kobayashi N, Cruz CMV, Fukuta Y. 2009. Development and Characterization of Blast Resistance Using Differential Varieties in Rice: Protocol for the sampling of diseased specimens and evaluation of blast disease in rice. Tsukuba: Japan International Research Center for Agricultural Sciences.
[IRRI] International Rice Research Institute. 1996. Standart Evaluation System for Rice. Ed ke-4. Philippines: IRRI.