VARIASI GEN
CYTOCHROME OXIDASE
I (COI) PADA LEBAH
Trigona
DI DESA BATU KUCING DAN DESA BUNGKU, JAMBI
RYZKA ANNISA BAHER
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Variasi Gen Cytochrome Oxidase I (COI) pada Lebah Trigona di Desa Batu Kucing dan Desa Bungku, Jambi adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apapun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
ABSTRAK
RYZKA ANNISA BAHER. Variasi Gen Cytochrome Oxidase I (COI) pada Lebah Trigona di Desa Batu Kucing dan Desa Bungku, Jambi. Dibimbing oleh RIKA RAFFIUDIN dan M. CHANDRA WIDJAJA.
Lebah Trigona merupakan salah satu serangga polinator yang berperan dalam ekosistem. Pendataan keanekaragaman Trigona secara morfologi telah dilakukan di Indonesia, namun pendataan secara molekular dengan variasi gen Cytochrome Oxidase I (COI) belum dilakukan. Penelitian ini bertujuan menganalisis kekerabatan antar tiga spesies Trigona dan mempelajari variasi gen Cytochrome Oxidase I (COI) pada lebah Trigona di Desa Batu Kucing dan Desa Bungku, Jambi. Jumlah sampel yang dianalisis sebanyak dua individu pada T. fuscobalteata, T. laeviceps asal Desa Batu Kucing dan Desa Bungku, Jambi. Primer gen COI di disain berdasarkan Melipona bicolor dari Genbank. Hasil sekuen gen COI Trigona secara parsial menghasilkan panjang nukleotida T. fuscobalteata dan T. laeviceps berturut-turut sebesar 724 dan 908 pb dengan komposisi AT yang tinggi. Alignment sekuen asam amino T. fuscobalteata, T. laeviceps dan T. chanchamayoensis dengan outgroup Melipona bicolor menunjukkan daerah yang conserve sebanyak 161 pb dan variasi ditemukan pada 19 pb. Analisis jarak genetik tertinggi diperoleh antara M. bicolor dengan T. laeviceps sebesar 0.023. Konstruksi filogeni dilakukan menggunakan metode Maximum Parsimony (MP) dan Neighbour Joining (NJ). Hasil kedua konstruksi filogeni menunjukkan kesamaan T. laeviceps memiliki kekerabatan yang dekat dengan T. fuscobalteata dibandingkan T. chanchamayoensis.
Kata kunci: gen COI, DNA mitokondria, Trigona
ABSTRACT
RYZKA ANNISA BAHER. Variation of Cytochrome Oxidase I (COI) Gene of Trigona Bee from Desa Batu Kucing and Desa Bungku, Jambi. Supervised by RIKA RAFFIUDIN and M. CHANDRA WIDJAJA.
Trigona stingless bees are one of many pollinator insects, which have important role in ecosystem. Morphology of the diversity of Trigona in Indonesia had been explored, however the molecular data of Trigona based on the variation of Cytochrome Oxidase I (COI) gene had not been carried out. This research was aimed to analyze the relationships between species of Trigona and to study the variation of COI gene of Trigona bees from Desa Batu Kucing and Desa Bungku, Jambi. Two samples of T. fuscobalteata, T. laeviceps from Batu Kucing Village and Bungku Village, Jambi were analysed. COI gene primer were designed based in Melipona bicolor from Genbank. The partial sequence of COI gene are 724 bp and 725 bp from T. fuscobalteata and T. laeviceps, respectively with AT rich composition. The alignment of amino acid sequen T. fuscobalteata, T. laeviceps and T. chanchamayoensi and outgroup of Melipona bicolor show that there are 161 bp of conserve and 19 bp of variation regions. Genetic distance between M. bicolor with T. fuscobalteata and M. bicolor with T. fuscobalteata.is 0.096. Phylogeny construction using Neighbour Joining (NJ) methode by using Jones-Taylor-Thornton (JTT) model showed that T. fuscobalteata had closer relationship with that of T. laeviceps compare to T. chanchamayoensis.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Sains
pada
Departemen Biologi
VARIASI GEN
CYTOCHROME OXIDASE
I (COI) PADA LEBAH
Trigona
DI DESA BATU KUCING DAN DESA BUNGKU, JAMBI
RYZKA ANNISA BAHER
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
Judul Skripsi : Variasi Gen Cytochrome Oxidase I (COI) pada Lebah Trigona di Desa Batu Kucing dan Desa Bungku, Jambi
Nama : Ryzka Annisa Baher NIM : G34090063
Disetujui oleh
Dr Ir Rika Raffiudin, MSi Pembimbing I
Drs M. Chandra Widjaja, MM Pembimbing II
Diketahui oleh
Dr Ir Iman Rusmana, MSi Ketua Departemen
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah dengan judul “Variasi Gen Cytochrome Oxidase I (COI) pada Lebah Trigona di Desa Batu Kucing dan Desa Bungku, Jambi” berhasil diselesaikan. Kegiatan penelitian ini dilaksanakan sejak bulan September 2013 sampai Februari 2014 di di laboratorium bagian Fungsi Hayati dan Perilaku Hewan Departemen Biologi, Institut Pertanian Bogor
Terima kasih penulis ucapkan kepada Ibu Dr Ir Rika Raffiudin, MSi dan Bapak Drs M. Chandra Widjaja, MM atas bimbingan dan pengarahan yang telah diberikan, serta Prof Dr Alex Hartana selaku anggota komisi pendidikan. Terima kasih kepada Bapak, Mama, serta seluruh keluarga atas setiap dukungan terbaik yang diberikan sejak lahir sampai menyelesaikan studi ini. Ungkapan terima kasih juga ditujukan kepada Andi Trisnandi, Lilia, Icha, Kak Uce, Kak Gina, Kak Eni, Bapak Adi, sahabat Wisma Balio Atas (Ulfah, Irna, Kak Ida, Lintang, Fawwarah, Dina, dan lainnya), dan teman-teman Biologi 46 untuk semangat, bantuan dan kerja samanya.
Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 2
BAHAN DAN METODE 2
Bahan dan Alat 2
Tahapan Analisis DNA 3
Ekstraksi DNA 3
Amplifikasi DNA 3
Visualisasi DNA 4
Sekuen dan Analisis Bioinformatika 5
HASIL DAN PEMBAHASAN 6
Amplifikasi DNA Gen COI Trigona 6
Sekuen DNA Gen COI Trigona dan Analisis Bioinformatika 6
Analisis Homologi Asam Amino COI Trigona 9
Analisis Jarak Genetik dan Konstruksi Filogeni 10
SIMPULAN 12
DAFTAR PUSTAKA 12
LAMPIRAN 15
DAFTAR TABEL
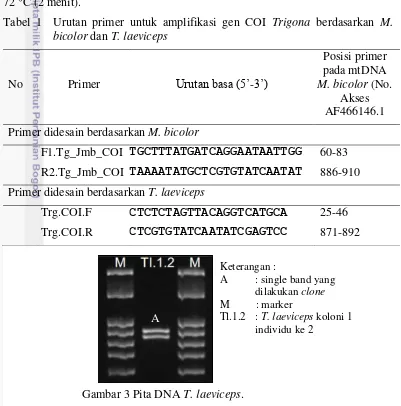
1. Urutan primer untuk amplifikasi gen COI Trigona berdasarkan M.
bicolor dan T. laeviceps 4
2. Data sampel ingroup Trigona dan outgroup Melipona dan Tetragonisca yang digunakan untuk analisis gen COI 5 3. Homologi tertinggi gen COI T. fuscobalteata dan T. laeviceps
dengan database nukleotida pada Genbank menggunakan BLAST-N
(www.ncbi. nlm.nih.gov/blastn) 9
4. Homologi tertinggi asam amino protein COI T. fuscobalteata dan T. laeviceps dengan database asam amino pada Genbank menggunakan
BLAST-P (www.ncbi. nlm.nih.gov/blastp) 9
5. Jarak genetik asam amino gen COI tiga spesies Trigona hasil
penelitian ini dengan outgroup Melipona 11
DAFTAR GAMBAR
1. Susunan sarang pada koloni T. laeviceps di ruas bambu (Wati 2013) 2 2. Morfologi Trigona: a. T. fuscobalteata, b. T. laeviceps (Soegiarso
2013) 2
3. Pita DNA T. laeviceps. 4
4. Amplikon pita DNA gen COI lebah Trigona 6
5. Sekuen gen COI T. fuscobalteata dan deduksi asam amino. 7 6. Sekuen gen COI T. laeviceps dan deduksi asam amino. 8 7. Penyejajaran asam amino putative gen COI tiga spesies Trigona dan
M. bicolor. 10
8. Konstruksi pohon filogeni berdasarkan sekuen asam amino gen COI menggunakan metode Neighbour Joining (NJ) dengan model
Jones-Taylor-Thornton (JTT). 11
9. Konstruksi pohon filogeni berdasarkan sekuen asam amino gen COI
menggunakan metode Maximum Parsimony (MP). 11
DAFTAR LAMPIRAN
1. Kromatogram sekuen gen COI DNA mitokondria lebah T. fuscobalteata menggunakan primer forward Trg.COI.F 16 2. Kromatogram sekuen gen COI DNA mitokondria lebah T.
1
PENDAHULUAN
Latar Belakang
Lebah Trigona (Hymenoptera: Apidae) merupakan spesies lebah yang tergolong dalam stingless bee atau lebah tak bersengat. Trigona tergolong dalam kelompok serangga sosial tingkat tinggi karena terdapat pembagian kasta yang jelas pada koloni (Michener 2000). Pada setiap koloni Trigona berjumlah 300 sampai 800.000 lebah. Trigona membuat sarang pada tempat berongga seperti celah-celah dinding, ruas bambu di dalam rumah atau pada rongga pada pohon yang hidup (Free 1982). Pada tanaman stroberi, penyerbukan oleh lebah Trigona laeviceps dan Apis cerana dapat meningkatkan pembentukan buah dan menurunkan persentase buah abnormal (Harahap 2010). Peran Trigona lainnya yakni pemanfaatan propolis sebagai anti mikrobial telah digunakan secara luas oleh masyarakat (Fatoni et al. 2008).
Distribusi Trigona tersebar di wilayah dataran rendah tropis dan beberapa subtropis yaitu sebagian besar Afrika Selatan, Amerika Selatan, dan Asia Tenggara (Free 1982). Distribusi Trigona yang luas mengindikasikan adanya keragaman spesies Trigona pada masing-masing wilayah. Beberapa penelitian di Indonesia menunjukkan adanya keragaman Trigona, yaitu 23 spesies Trigona ditemukan di Sumatra Barat (Sakagami et al. 1990) dan 10 spesies dari 23 spesies Trigona ditemukan di Jambi di antaranya Trigona fuscobalteata dan Trigona laeviceps (Gambar 2) (Soegiarso 2013).
Gen Cytochrome Oxydase subunit I (COI) merupakan gen mtDNA yang telah digunakan secara luas dalam teknik identifikasi molekular dalam Kingdom Animalia. Genom mtDNA memiliki pewarisan yang bersifat maternal dalam jumlah kopi salinan yang banyak dan memiliki bagian yang konservatif, sehingga banyak digunakan dalam analisis variasi genetik (Simon et al. 2006). Teknik identifikasi molekular menggunakan gen COI sebagai DNA barkode dengan standar panjang basa nukleotida sebesar 648 pb (Hebert et al. 2003a; Hebert dan Gregory 2005; Hajibabaei et al. 2006). Bagian yang konservatif pada gen COI dapat dikenali pada hampir semua filum dari divisi Animalia, sehingga dapat digunakan sebagai DNA barkode (Hebert et al. 2003a, 2003b).
Trigona merupakan salah satu serangga penting, yakni sebagai polinator dalam rantai makanan ekosistem dan sebagai bioindikator adanya transformasi ekosistem. Lebah Trigona merupakan salah satu serangga penyerbuk yang sangat potensial pada tanaman pertanian dan perkebunan. Trigona pada umumnya menjadi serangga polinator utama pada bunga yang berukuran kecil (Michener 2000). Usaha konservasi Trigona perlu dilakukan secara berdampingan baik secara fisik maupun molekuler.
2
spesimen pada fase hidup tertentu, misal telur, larva, pupa, atau imago (Herbert et al. 2003a). Pendataan terhadap keragaman gen COI Trigona baru dilakukan pada tiga spesies, diantaranya T. amalthea (No. Akses AF214669.1) (Tanaka et al. 2001), T. chancamayeonsis (No. Akses KC853315.1) dan T. spinipes (No. Akses KC853314.1) (Francoso dan Arias 2013).
Gambar 1 Susunan sarang pada koloni T. laeviceps di ruas bambu (Wati 2013)
a b
Gambar 2 Morfologi Trigona: a. T. fuscobalteata, b. T. laeviceps (Soegiarso 2013)
Tujuan Penelitian
Penelitian ini bertujuan menganalisis kekerabatan antar spesies Trigona dan mempelajari variasi gen Cytochrome Oxidase I (COI) pada lebah Trigona di Desa Batu Kucing dan Desa Bungku, Jambi .
BAHAN DAN METODE
Bahan dan Alat
3
Tahapan Analisis DNA Ekstraksi DNA
Ekstraksi DNA dilakukan menggunakan metode Cetyl Trimethyl Ammonium Brommida (CTAB) berdasarkan Sambrook et al. (1989) dengan modifikasi presipitasi etanol berdasarkan Raffiudin dan Crozier (2007). Jumlah sampel yang diekstraksi ialah 2 individu pada masing-masing spesies Trigona. Persiapan ekstraksi, sampel dimasukkan ke dalam 400 µl larutan Tris-HCL EDTA (TE) 0.5 mM (Tris HCl 2 M; EDTA 0.2 M; air destilata) sebanyak 3 kali pergantian selama selang waktu 4 jam untuk menghilangkan etanol absolut yang terdapat dalam jaringan.
Sumber DNA yang diekstraksi berasal dari bagian toraks Trigona. Sampel dipisahkan menggunakan scalpel steril dan dimasukkan ke dalam tabung 1.5 ml. Kemudian sampel direndam dalam nitrogen cair (20 menit) dan digerus sampai hancur menggunakan penggerus steril. Larutan buffer CTAB 0.2% (b/v) (7.5 ml 1M Tris-HCl pH 8; 3 ml 0.5 M NaEDTA pH 8; 6.135 g NaCl; 1.5 g CTAB; air hingga 75 ml) sebanyak 500 µl ditambahkan pada tabung untuk mendegradasi membran sel. Tabung yang berisi sampel ditambahkan 10 µl Proteinase-K (5mg/ml), kemudian diinkubasi pada suhu 55 °C (2 jam). Setelah inkubasi, tabung disentrifugasi pada 12.000 rpm (10 menit). Supernatan dipindahkan ke tabung baru, lalu ditambahkan 250 µl larutan PCI (Phenol: Chloroform: Isoamyl alcohol = 25: 24: 1). Tabung digoyang perlahan (1 menit), kemudian disentrifugasi pada 12 000 rpm (5 menit). Terbentuk dua lapisan, yaitu lapisan atas yang berisi DNA dan lapisan bawah atau lapisan PCI. Lapisan yang berisi DNA dipindahkan ke dalam tabung baru, lalu ditambahkan 400 µl larutan CIAA (Chloroform: Isoamyl alcohol = 24: 1) dan digoyang perlahan (5 menit). Tabung disentrifugasi pada 12.000 rpm (3 menit). Lapisan bagian atas yang berisi DNA dipindahkan ke tabung baru dan ditambahkan 600 µl larutan isopropanol, selanjutnya disimpan pada suhu -4 °C selama (kurang lebih) 12 jam.
Presipitasi DNA dilakukan pada suhu 4 °C dan disentrifugasi pada kecepatan 13.000 rpm (30 menit). Isopropanol yang terdapat dalam tabung dibuang, kemudian ditambahkan etanol 70%. Sentrifugasi kembali dilakukan pada kecepatan dan suhu yang sama (10 menit). Selanjutnya, etanol 70% pada tabung dibuang dan dilakukan pengeringan pelet DNA dengan vakum (30 menit). Tabung berisi DNA murni ditambahkan 20 µl larutan TE 0.5 mM dan disimpan pada suhu 4 °C.
Amplifikasi DNA
DNA hasil esktraksi diamplifikasi dengan teknik Polymerase Chain Reaction (PCR) menggunakan thermocycler (ESCO SWIFT MAXI-BLC 1). Fragmen DNA yang diamplifikasi ialah gen COI pada Trigona. Desain primer dilakukan oleh peneliti berdasarkan genom lengkap mitokondria Melipona bicolor (Silvestre et al. 2008) dengan kode akses Genbank AF466146.1. Pasangan primer hasil desain ialah primer forward F1.Tg_Jmb_COI dan primer reverse R2.Tg_Jmb_COI (Tabel 1).
4
pada pita DNA yang berukuran lebih besar (bagian atas). Desain primer dilakukan untuk kedua kalinya berdasarkan sekuen hasil klon yaitu sekuen T. laeviceps. Hasil desain primer berupa pasangan primer forward Trg.COI.F dan primer reverse Trg.COI.R (Tabel 1).
Total pereaksi yang digunakan dalam amplifikasi adalah 20 µl, terdiri atas 6.8 µl akuades steril, 0.8 µl primer forward, 0.8 µl primer reverse, 10 µl Kapa Taq Ready Mix DNA Polymerase buffer (buffer Mg2+1.5 mM, dNTP 0.4 mM, Kapa Taq DNA Polymerase 0.05 U/ µl) dengan penambahan 0.6 µl MgCl2 1.5 mM, dan 1 µl DNA hasil ekstraksi. Amplifikasi dilakukan dengan PCR dalam 5 tahapan dengan kondisi; predenaturation pada suhu 95 °C (2 menit), denaturation DNA pada suhu 95 °C (2 menit), annealing pada suhu 50 °C (30 detik), elongation pada suhu 72 °C (1 menit), dan post-elongation pada suhu
Trg.COI.F CTCTCTAGTTACAGGTCATGCA 25-46 Trg.COI.R CTCGTGTATCAATATCGAGTCC 871-892
Visualisasi DNA
5
Amonium Peroxo Sulfat (APS) 10%. Migrasi dilakukan pada tegangan 200 V selama 55 menit. DNA divisualisasi dengan metode pewarnaan perak nitrat (Byun et al. 2009).
Sekuendan Analisis Bioinformatika
Variasi genetik COI untuk T. fuscobalteata dan T. laeviceps dianalisis menggunakan metode sekuensing pada perusahaan jasa sekuen. Hasil sekuen diedit dan dianalisis menggunakan program Bioedit Sequence Alignment dan Genetyx Win Version 4.0. Sekuen DNA forward dan reverse di-alignment menggunakan program Clustal X untuk mendapatkan hasil sekuen DNA utuh. Hasil sekuen dilakukan Basic Local Alignment Search Tool–Nucleotide (BLAST-N) (http://blast.ncbi.nlm.nih.gov/) sebagai pembanding antara sekuen nukleotida hasil penelitian ini dengan nukleotida database Genbank untuk mengetahui nilai persentase homologi, e-value, dan persentase query cover. Nilai e-value merupakan parameter yang menggambarkan tingkat harapan secara eksponensial saat proses pencarian database (McEntyre & Ostell 2002). Semakin rendah nilainya, maka semakin signifikan dan dapat dipercaya. Nilai query cover merupakan persentase dari kecocokan yang meliputi sekuen input dengan sekuen yang terdapat dalam database (McEntyre & Ostell 2002)
Sekuen DNA ditranslasikan menjadi asam amino putative menggunakan program Genetyx Win Version 4.0. Hasil sekuen asam amino dilakukan atau Basic Local Alignment Search Tool – Protein, BLAST-P (http://blast.ncbi.nlm.nih.gov/)
Analisis Jarak Genetik dan Filogeni
Hasil sekuen DNA di-alignment dengan program Clustal X2.1 dan MEGA 5 untuk memperoleh variasi asam amino masing-masing spesies dan analisis pohon filogeni (Tamura et al. 2011). Penentuan jarak genetik dan analisis filogeni dilakukan berdasarkan hasil penjajaran sekuen asam amino T. fuscobalteata, T. laeviceps, T. chanchamayoensis sebagai ingroup dengan M. bicolor sebagai outgroup (Tabel 2) menggunakan model Jones-Taylor-Thornton (JTT). Penggunaan outgroup dilakukan untuk mengetahui spesies yang paling primitif. Selanjutnya, konstruksi filogeni gen COI dilakukan dengan metode Maximum Parsimony (MP) menggunakan tingkat kepercayaan bootstrap 1000x. Tabel 2 Data sampel ingroup Trigona dan outgroup Melipona dan Tetragonisca
yang digunakan untuk analisis gen COI
No Sampel Lokasi/ Asal No. Akses Genbank
Ingroup
1. T. fuscobalteata Jambi -
2. T. laeviceps Jambi -
3. T. chanchamayoensis Brazil KC853315.1 Outgroup
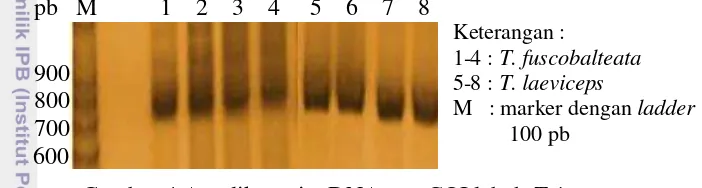
6 F1.Tg_Jmb_COI dan reverse R2.Tg_Jmb_COI berdasarkan genom mitokondria M. bicolor (AF466146.1) (Silvestre et al. 2008). Sama halnya dengan amplikon pita DNA gen COI T. fuscobalteata pada sumur 1-4 (Gambar 4) sesuai dengan prediksi gen target COI berdasarkan gen COI T. laeviceps sebesar 846 bp menggunakan primer forward Trg.COI.F dan reverse Trg.COI.R.
Gambar 4 Amplikon pita DNA gen COI lebah Trigona
Sekuen DNA Gen COI Trigona dan Analisis Bioinformatika
Hasil sekuen gen COI yang diperoleh berupa kromatogram dengan panjang basa nukleotida yang beragam yakni T. fuscobalteata sebesar 724 pb dan T. laeviceps sebesar 908 pb (Gambar 5 dan 6, Lampiran 1 dan 2). Hasil sekuen menunjukkan komposisi AT yang tinggi pada kedua spesies yakni T. fuscobalteata sebesar 66.35% dan T. laeviceps sebesar 67.58%. Hal ini menunjukkan adanya kesesuaian dengan komposisi AT yang tinggi pada DNA mitokondria lebah (Crozier 1989). Namun, kedua spesies Trigona memiliki komposisi AT yang lebih rendah dibandingkan dengan Apis, yakni Apis cerana 74.57% dan A. koschevnikovi 75.5 % (Dzulfaqor 2011 dan Nugroho 2011).
Analisis bioinformatika dengan BLAST-N dan BLAST-P dilakukan sebagai dasar dalam menentukan tingkat ketepatan suatu sekuen. Hasil BLAST-N pada T. laeviceps menunjukkan sekuen yang homolog adalah dengan gen COI M. bicolor dengan nilai kesamaan (identity) sebesar 82%, nilai e-value 0.0 dan query cover sebesar 95%, sedangkan homologi tertinggi pada T. fuscobalteata ialah Tetragonisca angustula MP100 gen COI dengan nilai kesamaan 83%, nilai e-value 2e-165 dan query cover sebesar 95% (Tabel 3).
Nilai e-value BLAST-N pada T. laeviceps dan T. fuscobalteata yang mendekati nol menandakan ketepatan yang signifikan dan bukan karena kebetulan. Nilai query cover mendekati 95% pada T. laeviceps dan T. fuscobalteata menunjukan tingginya presentase kecocokan antara sekuen tersebut dengan sekuen yang terdapat dalam database. Ketiga hal tersebut dapat digunakan sebagai dasar untuk menyatakan bahwa sekuen tersebut sebagai sekuen yang tepat berdasarkan nilai persentase homologi, nilai e-value, dan persentase query cover (McEntyre & Ostell 2002).
7
[1] : nomor nukleotida [3] : deduksi asam amino dengan singkatan [2]: sekuen nukleotida T. laeviceps satu huruf
Gambar 5 Sekuen gen COI T. fuscobalteata dan deduksi asam amino. [1]
8
[1] : nomor nukleotida [3] : deduksi asam amino dengan singkatan [2]: sekuen nukleotida T. laeviceps satu huruf
Gambar 6 Sekuen gen COI T. laeviceps dan deduksi asam amino. [1]
9
Tabel 3 Homologi tertinggi gen COI T. fuscobalteata dan T. laeviceps dengan database nukleotida pada Genbank menggunakan BLAST-N
Homologi tertinggi gen COI Trigona berdasarkan program BLAST-P menunjukkan adanya kesamaan dengan data asam amino spesies lainnya yakni sebesar 91% dengan M. melanoventer gen COI (AGO14935.1) dengan nilai query cover sebesar 100%. Perolehan nilai e-value yang berbeda pada kedua spesies yaitu T. laeviceps sebesar 2e-111 dan T. fuscobalteata sebesar 3e-111 tidak memberikan hasil yang berbeda secara signifikan (Tabel 4). Berdasarkan hasil BLAST-P menunjukkan bahwa sekuen asam amino dugaan (putative) T. laeviceps dan T. fuscobalteata adalah tepat berdasarkan nilai persentase homologi yang besar, nilai e-value yang kecil, dan persentase query cover yang mendekati 100%.
Tabel 4 Homologi tertinggi asam amino protein COI T. fuscobalteata dan T. laeviceps dengan database asam amino pada Genbank menggunakan BLAST-P (www.ncbi. nlm.nih.gov/blastp)
10
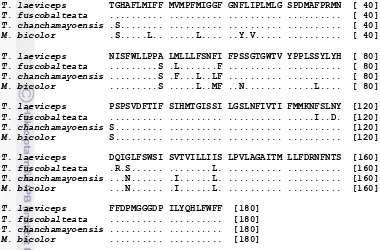
variabel sebanyak 19 pb (singelton sites 4 pb dan parsimoni informative sites 15 pb) (Gambar 7).
Gambar 7 Penyejajaran asam amino putative gen COI tiga spesies Trigona dan M. bicolor.
Analisis Jarak Genetik dan Konstruksi Filogeni
Analisis jarak genetik dilakukan sebagai input awal untuk mengetahui pengelompokan kekerabatan terdekat berdasarkan asam amino dugaan gen COI. Hasil analisis dengan metode NJ diperoleh jarak genetik tertinggi sebesar 0.023 (Tabel 5) antara M. bicolor dengan T. laeviceps dengan T. fuscobalteata, sedangkan jarak genetik terendah yaitu 0.016 antara T. laeviceps dengan T. fuscobalteata dan M. bicolor dengan T. chancamayoensis. Semakin tinggi nilai jarak genetik maka kekerabatan antar keduanya semakin jauh, begitu juga sebaliknya. Hal ini menunjukkan bahwa antara T. laeviceps dengan T. fuscobalteata memiliki kekerabatan yang paling dekat.
Konstruksi filogeni dilakukan antara T. fuscobalteata, T. laeviceps dan T. chanchamayoensis dengan M. bicolor digunakan sebagai outgroup berdasarkan metode Maximum Parsimony (MP) dan Neighbour Joining (NJ) dengan model subtitusi Jones-Taylor-Thornton (JTT). Penggunaan outgroup dilakukan untuk mengetahui spesies ingroup yang paling primitif. Panjang cabang pada pohon filogeni dengan metode NJ merefleksikan jarak genetik antar sekuen. Berbeda dengan metode MP, panjang cabang mengindikasikan posisi sekuen yang memiliki kemiripan. Hasil konstruksi filogeni dengan metode NJ menunjukkan terdapat dua kelompok utama (Gambar 8). Kelompok pertama dengan nilai ialah ingroup Trigona, yaitu T. fuscobalteata dan T. laeviceps dengan nilai bootstrap
T. laeviceps TGHAFLMIFF MVMPFMIGGF GNFLIPLMLG SPDMAFPRMN [ 40]
T. laeviceps PSPSVDFTIF SIHMTGISSI LGSLNFIVTI FMMKNFSLNY [120]
T. fuscobalteata ... ... ... ...I..D. [120]
T. chanchamayoensis S... ... ... ... [120]
M. bicolor S... ... ... ... [120]
T. laeviceps DQIGLFSWSI SVTVILLIIS LPVLAGAITM LLFDRNFNTS [160]
T. fuscobalteata .R.S... ...L. ... ... [160]
11
sebesar 98%. Kelompok berikutnya yaitu T. chanchamayoensis mengelompok dengan outgroup M. bicolor. Konstruksi filogeni dengan MP dilakukan untuk mengetahui prediksi posisi sekuen yang memiliki kemiripan. Berdasarkan metode MP, konstruksi filogeni menunjukkan adanya kelompok ingroup Trigona yang mengelompok pada satu kelompok utama (Gambar 9); T. fuscobalteata dan T. laeviceps memiliki kekerabatan yang lebih dekat dibandingkan dengan T. chanchamayoensis. Hasil dari kedua konstruksi filogeni menunjukkan adanya kesamaan, yakni bahwa T. laeviceps memiliki kekerabatan yang dekat dengan T. fuscobalteata dibandingkan dengan T. chanchamayoensis. Tabel 5 Jarak genetik asam amino gen COI tiga spesies Trigona hasil penelitian
ini dengan outgroup Melipona
Angka dalam kotak menunjukkan nilai terendah (0,000) dan nilai tertinggi (1,000)
Keterangan :
(→ ) : skala jarak genetik.
Angka pada nodus menyatakan nilai hasil bootstrap 1000x
Gambar 8 Konstruksi pohon filogeni berdasarkan sekuen asam amino gen COI menggunakan metode Neighbour Joining (NJ) dengan model Jones-Taylor-Thornton (JTT).
Keterangan :
(→ ) : skala kemiripan sekuen. Arah panah menunjukkan tingkat kemiripan semakin tinggi
Angka pada nodus menyatakan nilai hasil bootstrap 1000x
12
SIMPULAN
Hasil sekuen gen COI secara parsial menghasilkan panjang nukleotida T. fuscobalteata sebesar 724 pb dan T. laeviceps sebesar 908 pb dengan komposisi AT yang lebih tinggi dibandingkan GC. Analisis bioinformatika dengan BLAST-N dan BLAST-P pada kedua spesies menunjukkan nilai persentase homologi yang tinggi, nilai e-value yang kecil, dan persentase query cover mendekati 100%. Ketiga hal tersebut dapat digunakan sebagai dasar untuk menyatakan bahwa sekuen tersebut sebagai sekuen yang tepat.
Analisis jarak genetik tertinggi diperoleh antara M. bicolor dengan T. laeviceps, sedangkan jarak genetik terendah antara T. laeviceps dengan T. fuscobalteata. Hasil konstruksi filogeni dengan metode MP, konstruksi filogeni menunjukkan nahwa ketiga spesies Trigona membentuk satu kelompok. Konstruksi filogeni berdasarkan NJ menunjukkan terdapat dua kelompok yaitu T. fuscobalteata dan T. laeviceps dengan nilai bootstrap sebesar 98%. Kelompok berikutnya yaitu T. chanchamayoensis mengelompok dengan outgroup M. bicolor. Hasil dari kedua konstruksi filogeni menunjukkan adanya kesamaan, yakni bahwa T. laeviceps memiliki kekerabatan yang dekat dengan T. fuscobalteata dibandingkan dengan T. chanchamayoensis.
DAFTAR PUSTAKA
Byun SO, Fang Q, Zhou H, Hickford JGH. 2009. An effective method for silverstaining DNA in large numbers of polyacrilamide gels. Anal Biochem. 385:174-175.
Crozier RH, Crozier YC, Mackinlay AG. 1989. The CO-I and CO-II Region of Honeybee Mitochondrial DNA: Evidence for Variation in Insect Mitochondrial Evolutionary Rates. Mol Biol Evol 6:399-411
Dzulfaqor.2011. Cytochrome oxidase subunit I (COI) gene variations in mitochondrial DNA of Apis koschevnikovi [Skripsi]. Bogor (ID): Institut Pertanian Bogor
Fatoni A, Artika IM, Hasan AEZ, Kuswandi. 2008. Antibacterial Activity of Propolis Produced by Trigona spp. Against Campylobacter spp. HAYATI J Biosci. 15:161-164
Francoso E, Arias MC. 2013. Cytochrome c oxidase I primers for corbiculate bees: DNA barcode and mini-barcode. Mol Ecol Res. 5: 844-850
Free JB. 1982. Bees and mankind. London (UK): George Allen and Unwind. Hajibabaei et al. 2006. DNA barcode distinguish species of tropical
Lepidopteran. PNAS. 103: 968-971.
Harahap KK. 2010. Efektivitas polinasi Apis cerana Fabricus dan Trigona laeviceps Smith (Hymenoptera: Apidae) pada Fragaria x annanassa kultivar earlibrite [Tesis]. Bogor (ID): Institut Pertanian Bogor.
13
Hebert PDN, Ratnasingham S, deWaard JR. 2003b. Barcoding animal life: cythochrome c oxidase subunit 1 divergence among closely related species. Proc R Soc Lond B. 270 : S96–S99.
Hebert PDN, Gregory TR. 2005. The promise of DNA Barcoding for Taxonomy. Syst Biol. 54: 852-859.
Maddison WP, Donoghue MJ, Maddison DR. 1984. Outgroup analysis and parsimony. Syst Zool 33: 83-103.
McEntyre J, Ostell J. 2002. The NCBI Handbook. National Center for Biotechnology Information Bethesda (MD): National Center for Biotechnology Information (US)
Michener CD. 2000. The Bees of The World. Baltimore: The John Hopkins Univ Pr.
Sakagami SF, Ohgushi R, Roubik DW. 1990. Natural History of Social Wasps and Bees in Equatorial Sumatra. Sapporo (JP): Hokkaido Univ. Press.
Sambrook J, Fritsch EF, Maniatis T. 1989. Molecular Cloning: A Laboratorium Manual Second Edition. New York (US): Cold Spring Harbor Laboratory Press.
Silvestre D, Downton M, Arias MC.2008.The mitochondrial genome of the stingless bee Melipona bicolor (Hymenoptera, Apidae, Meliponini): Sequence, gene organization and a unique tRNA translocation event conserved across the tribe Meliponini. Gen Mol Biol 31: 451-460
Simon C, Buckley TR, Frati F, Stewart JB, Beckenbach AT. 2006. Incorporating molecular evolution into phylogenetic analysis, and a new compilation of conserved polymerase chain reaction primers for animal mitochondrial DNA. Annu Rev Ecol Evol Syst. 37: 545-579.
Soegiarso ATP. 2013. Keanekaragaman ordo Hymenoptera di perkebunan kelapa sawit, perkebunan karet dan hutan karet di Jambi [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Tanaka H, Roubik DW, Kato M, Liew F, Gunsalam G. 2001. Phylogenetic position of Apis nuluensis of nothern Borneo and phylogeography of Apis cerana as inferred from mitochondrial DNA sequences. Insectes Soc. 48: 44-51
15
16
Lampiran 1 Kromatogram sekuen gen COI DNA mitokondria lebah T. fuscobalteata menggunakan primer forward Trg.COI.
1
17
Lampiran 2 Kromatogram sekuen gen COI DNA mitokondria lebah T. fuscbalteata menggunakan primer reverse Trg.COI.R
18
RIWAYAT HIDUP
Penulis dilahirkan di Jakarta tanggal 21 Februari 1993 dari pasangan Bangar Sianipar dan Nanih Suherni. Penulis merupakan anak pertama dari dua bersudara. Menyelesaikan pendidikan menengah atas di SMAN 33 Jakarta pada tahun 2009, penulis melanjutkan pendidikan tinggi pada tahun yang sama di Departemen Biologi Fakultas Matematika dan IPA Institut Pertanian Bogor (IPB) melalui jalur Undangan Seleksi Masuk IPB (USMI).
Selama perkuliahan, penulis memiliki pengalaman sebagai asisten praktikum Anatomi dan Morfologi Tumbuhan (2012/2013) dan Biologi Dasar (2012/2013). Penulis juga telah melakukan penelitian mengenai Keanekaragaman Tanaman Pekarangan di Desa-desa Sekitar Gunung Walat dalam rangka program studi lapangan Departemen Biologi tahun 2011. Selain itu penulis juga telah melakukan praktik lapangan di PT. Alam Desa Tapos Salaca mengenai Pembesaran Ikan Hias Koi (Cyprinus carpio L.) di PT. Alam Desa Tapos Dalaca Kecamatan Tenjolaya Kabupaten Bogor.