PROFIL GENETIK Escherichia coli YANG DIISOLASI DARI
TEMPE BERDASARKAN ENTEROBACTERIAL REPETITIVE
INTERGENIC CONSENSUS-POLYMERASE CHAIN REACTION
(ERIC-PCR)
QURROTA A’YUN
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa tesis berjudul Profil Genetik Escherichia coli yang Diisolasi dari Tempe Berdasarkan Enterobacterial Repetitive Intergenic Consensus-Polymerase Chain Reaction (ERIC-PCR) adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, April 2015
Qurrota A’yun
RINGKASAN
QURROTA A’YUN. Profil Genetik Escherichia coli yang Diisolasi dari Tempe Berdasarkan Enterobacterial Repetitive Intergenic Consensus-Polymerase Chain Reaction (ERIC-PCR). Dibimbing oleh ANTONIUS SUWANTO dan TATI BARUS.
Tempe merupakan makanan khas Indonesia yang berasal dari fermentasi kacang kedelai yang diinokulasi menggunakan kapang Rhizopus spp. Pembuatan tempe dilakukan dengan cara yang sangat bervariasi oleh para pengrajin dan umumnya pada kondisi yang tidak terkontrol. Kondisi yang tidak higienis mengakibatkan tumbuhnya Escherichia coli yang dikenal sebagai bakteri indikator kebersihan lingkungan. Beberapa strain E. coli dapat menimbulkan diare, gastroenteritis akut atau infeksi saluran pencernaan. E. coli pada tempe kemungkinan berbeda dengan E. coli patogen pada manusia. Perbedaan genetik dapat mempengaruhi karakteristik E. coli, terutama dalam hubungannya dengan bidang medis. Analisis molekuler berdasarkan sifat genotipik penting dilakukan untuk melakukan identifikasi dan karakterisasi, mempelajari evolusi serta epidemiologi tentang patogenitas dari suatu bakteri. Salah satu metode molekuler yang digunakan untuk mengkaji keragaman mikroorganisme adalah Enterobacterial Repetitive Intergenic Consensus-Polymerase Chain Reaction (ERIC-PCR). Tujuan dari penelitian ini adalah untuk membandingkan keragaman genetik E. coli pada tempe dengan isolat medis dengan menggunakan metode ERIC-PCR.
Sampel tempe diperoleh dari pengrajin tempe EMP, DRG, WJB dan CLR di Bogor. Tiga isolat medis digunakan untuk dibandingkan dengan isolat E. coli dari tempe yaitu E. coli ATCC 25922 (koleksi Laboratorium Mikrobiologi Fakultas Kedokteran Universitas Indonesia, Jakarta), E. coli O157 (koleksi Departemen Mikrobiologi Fakultas Kedokteran Universitas Atma Jaya, Jakarta), dan EPEC K.1.1 (koleksi Laboratorium Mikrobiologi Institut Pertanian Bogor, Bogor). E. coli DH5α sebagai kontrol positif (koleksi Fakultas Teknobiologi Universitas Katolik Atma Jaya, Jakarta). Klebsiella pneumoniae yang diisolasi dari tempe digunakan sebagai outgroup. Isolasi E. coli dari tempe dilakukan dengan metode pengenceran bertingkat 10-2 sampai 10-5 dan disebar ke dalam media Eosin Methylene Blue (EMB) kemudian diuji dengan media Simmon Sitrat Agar (SSA). DNA genom E. coli diisolasi menggunakan Cetyl Trimethyl Ammonium Bromide (CTAB). Gen 16S rRNA E. coli diamplifikasi menggunakan primer 63f dan 1387r. Hasil sekuensing dianalisis menggunakan program MEGA 5.2. Sekuen ERIC E. coli diamplifikasi menggunakan primer ERIC 1R dan ERIC 2. Profil pita DNA diinterpretasikan menjadi matriks data biner untuk pembuatan pohon filogenetik dengan menggunakan program MEGA 5.2. Konstruksi pohon filogenetik menggunakan metode Unweighted Pair Groups Method Analysis (UPGMA).
berbeda dari E. coli. Dilain pihak, genotip yang sama dapat ditemukan didalam sampel tempe yang berbeda. Gen 16S rRNA setiap isolat E. coli telah berhasil diamplifikasi menggunakan primer 63f dan 1387r dengan pita DNA berukuran sekitar 1.3 kb. Hasil BLASTN sekuen gen 16S rRNA menunjukkan 33 isolat tersebut adalah E. coli dengan persentase kemiripan 96%-100% dengan E-value 0.0.
Visualisasi profil ERIC-PCR menunjukkan bahwa pola pita isolat E. coli yang berasal dari tempe berbeda dengan isolat medis. Profil pita isolat E. coli dari tempe menghasilkan pola pita DNA yang unik berukuran 0.25 kb dan 1.0 kb. Pada penelitian ini, tidak ada satupun E. coli yang diisolasi dari tempe yang menunjukkan pola pita identik dengan isolat medis.
Profil ERIC-PCR isolat E. coli dari tempe CLR menunjukkan pola yang sama sedangkan isolat E. coli dari tempe EMP memiliki pola yang beragam. Meskipun profil genetik E. coli dari tempe EMP lebih beragam daripada tempe CLR, tidak ada profil yang identik ketika isolat E. coli dari tempe dibandingkan dengan E. coli DH5α. Pohon filogenetik berdasarkan ERIC-PCR menunjukkan bahwa isolat medis dikelompokkan ke dalam kelompok terpisah dan khas sedangkan isolat E. coli dari tempe membentuk empat grup. Pada penelitian ini, menunjukkan bahwa isolat medis membentuk sebuah kelompok terpisah diluar cabang E. coli DH5α dan K. pneumoniae. Oleh sebab itu, isolat E. coli yang berasal dari tempe secara genetik berbeda dari isolat medis.
SUMMARY
QURROTA A’YUN. Genetic Profiles of Escherichia coli Isolated from Tempeh Based on Enterobacterial Repetitive Intergenic Consensus-Polymerase Chain Reaction (ERIC-PCR). Supervised by ANTONIUS SUWANTO dan TATI BARUS.
Tempeh is a famous Indonesian fermented food derived from soybeans inoculated with Rhizopus spp. Tempeh production varies depend on the producers and often conducted in an uncontrolled condition. This condition could lead to the growth of Escherichia coli which is known as bacterial indicators of environmental hygiene. Some strains of E. coli could induce diarrhea, acute gastroenteritis or gastrointestinal tract infections. E. coli in human may differ with E. coli pathogens in humans. Genetic differences may affect the characteristics of E. coli, particularly in relation to the medical field. Therefore, molecular analysis based on genotypic characteristics is important for the identification, characterization and evolutionary study and epidemiology of bacterial pathogenicity. One of molecular methods that can be used to study about microorganism diversity is Enterobacterial Repetitive Intergenic Consensus-Polymerase Chain Reaction (ERIC-PCR). The aim of this study was to compare genetic diversity isolates of E. coli derived from tempeh with medical isolates employing ERIC-PCR method.
Tempeh samples were obtained from EMP, DRG, WJB and CLR producers in Bogor. Three isolates medical were used for comparison with isolates of E. coli from tempeh i.e. E. coli ATCC 25922 (collection of Microbiology Laboratory, Faculty of Medicine, University of Indonesia, Jakarta), E. coli O157 (collection of Microbiology Laboratory, Faculty of Medicine, Atma Jaya Indonesia Catholic University, Jakarta), and EPEC K.1.1 (collection of Microbiology Laboratory, Faculty of Mathematics and Natural Science, Bogor Agricultural University, Bogor). E. coli DH5α was used as control positive (collection of Microbiology Laboratory, Faculty of Technobiology, Atma Jaya Indonesia Catholic University, Jakarta). Klebsiella pneumoniae isolated from tempeh was used for outgroup. Isolation of E. coli from tempeh was conducted by serial dilution from 10-2 until 10-5 and spreaded on Eosin Methylene Blue (EMB)
Agar then analyzed by cultivating them on Simmon’s Citrate Agar (SCA). DNA Genome of each E. coli was extracted using Cetyl Trimethyl Ammonium Bromide (CTAB) method. 16S rRNA gene was amplified using 63f and 1387r primers. Sequencing results were analyzed using MEGA 5.2 programme. ERIC sequence of E. coli was amplified using ERIC 1R and ERIC 2 primers. DNA band profiles were interpreted as binary data and analyzed using MEGA 5.2 programme. Phylogenetics tree construction was performed employing Unweighted Pair Groups Method Analysis (UPGMA).
different tempeh samples. 16S rRNA gene of each isolate of E. coli was successfully amplified using 63f and 1387r primers with DNA fragments size about 1.3 kb. Based on BLASTN, sequence of 16S rRNA gene showed that 33 isolates were E. coli with maximum identities ranging from 96% to 100%, with E-value 0.0.
Visualization of the ERIC-PCR profiles showed that DNA banding patterns isolates of E. coli derived from tempeh were different from those of medical isolates. Band profiles isolates of E. coli from tempeh generated unique DNA banding patterns of 0.25 kb and 1.0 kb in size. In this study, none of E. coli isolated from tempeh showed DNA banding patterns identical to medical isolates. ERIC-PCR profiles isolates of E. coli from CLR tempeh showed similar patterns while isolates of E. coli from EMP tempeh showed more heterogeneous patterns. Although genetic profiles of E. coli in EMP tempeh were more varied than CLR tempeh, we did not find identical profiles when isolates of E. coli from tempeh were compared to E. coli DH5α. Phylogenetic tree based on ERIC-PCR showed that medical isolates clustered into a separate and distict group while isolates of E. coli from tempeh formed four groups. This analysis showed that the medical isolates formed a separate group outside the branches which contained E. coli DH5α and K. pneumoniae. Therefore, isolates of E. coli derived from tempeh were genetically different from those of medical isolates.
©
Hak Cipta Milik IPB, Tahun 2015
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
PROFIL GENETIK Escherichia coli YANG DIISOLASI DARI
TEMPE BERDASARKAN ENTEROBACTERIAL REPETITIVE
INTERGENIC CONSENSUS-POLYMERASE CHAIN REACTION
(ERIC-PCR)
QURROTA A’YUN
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Mikrobiologi
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
Judul Tesis : Profil Genetik Escherichia coli yang Diisolasi dari Tempe Berdasarkan Enterobacterial Repetitive Intergenic Consensus-Polymerase Chain Reaction (ERIC-PCR)
Nama : Qurrota A’yun NIM : G351120371
Disetujui oleh Komisi Pembimbing
Prof Antonius Suwanto, PhD Ketua
Dr Ir Tati Barus, MSi Anggota
Diketahui oleh
Ketua Program Studi Mikrobiologi
Prof Dr Anja Meryandini, MS
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Penelitian yang dilaksanakan sejak bulan Desember 2013 sampai September 2014 ini berjudul “Profil Genetik Escherichia coli yang Diisolasi dari Tempe Berdasarkan Enterobacterial Repetitive Intergenic Consensus-Polymerase Chain Reaction (ERIC-PCR)”. Tesis ini disusun untuk memenuhi salah satu syarat untuk menyelesaikan pendidikan strata dua (S2) Program Studi Mikrobiologi, Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Terima kasih penulis ucapkan kepada Prof Antonius Suwanto, PhD dan Dr Ir Tati Barus, MSi selaku pembimbing yang telah memberikan nasehat, saran, motivasi, serta solusi dari setiap permasalahan yang penulis hadapi selama penelitian dan penyusunan karya ilmiah ini. Selain itu, penulis ucapkan terima kasih kepada penguji luar komisi Prof Dr Ir Lilis Nuraida, MSc dan Prof Dr Aris Triwahyudi, MSi selaku Perwakilan dari Ketua Program Studi Mikrobiologi IPB, yang telah memberikan saran dalam penyusunan karya ilmiah ini sehingga menjadi lebih baik. Kepada Lembaga Pengelola Dana Pendidikan (LPDP) melalui beasiswa tesis terima kasih atas kepercayaannya untuk memberikan beasiswa kepada penulis dan terima kasih atas sebagian dari dana DIPA IPB 2014 atas nama Prof Antonius Suwanto, PhD sehingga penelitian yang penulis lakukan dapat terlaksana dengan baik.
Penulis juga ingin menyampaikan terima kasih kepada Dr Susan Soka, MSc yang telah membantu mengurus segala keperluan di Laboratorium. Seluruh staf teknisi di Laboratorium Universitas Katolik Atma Jaya terutama untuk mas Bambang, mas Dahrul, kang Ridwan dan mas Tri terima kasih telah membantu penulis selama melakukan penelitian serta ucapan terima kasih kepada Lekta, Evelin dan Cidy sebagai pasangan selama penelitian. Ucapan terima kasih penulis sampaikan juga kepada Bapak Jaka (staf Laboratorium Mikrobiologi IPB), sahabat-sahabat tersayang Nezharia, Mahyarudin, Allia, Anja, Dina, Anik, Novi, Kak Heni, Hamtini, Cici Nuryanti, Ira, Mona, Wulan, Rika, Asril, dan mbak Nina terima kasih atas bantuan, saran, motivasi, serta solusi yang telah diberikan kepada penulis. Ucapan terimakasih penulis sampaikan kepada Kak Listya, Kak Ibu Tatik, Sipri, dan Kak Sari atas pengalaman dan ilmu yang telah diberikan. Kepada teman-teman Pascasarjana Mikrobiologi IPB 2012 terima kasih atas kebersamaan yang singkat dan sangat indah. Ungkapan terima kasih yang tak terhingga juga penulis ucapkan kepada orang tua tercinta Bapak Saunin, Ibu Hariroh, adik-adik tersayang, seluruh keluarga besar, serta teman-teman di bimbel BINAR atas segala doa, dukungan, motivasi dan kasih sayangnya selama ini. Semoga karya ilmiah ini dapat bermanfaat bagi kemajuan ilmu pengetahuan selanjutnya.
Bogor, April 2015
DAFTAR ISI
DAFTAR TABEL xii
DAFTAR GAMBAR xii
DAFTAR LAMPIRAN xii
PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 2
TINJAUAN PUSTAKA 3
Tempe 3
Escherichia coli 4
Pustaka Gen Penyandi 16S rRNA 4
Enterobacterial Repetitive Intergenic Consensus-Polymerase Chain Reaction
(ERIC-PCR) 5
METODE PENELITIAN 6
Kerangka Penelitian 6
Waktu dan Tempat Penelitian 6
Bahan 6
Isolasi E. coli dari Tempe 7
Isolasi DNA Genom E. coli 7
Amplifikasi Gen Penyandi 16S rRNA E. coli 8
Sekuensing Gen Penyandi 16S rRNA E. coli 8
Analisis Profil Genetik E. coli dengan Sekuen ERIC-PCR 8 Konstruksi Pohon Filogeni 9
HASIL DAN PEMBAHASAN 9
Hasil 9
Pembahasan 15
SIMPULAN DAN SARAN 18
Simpulan 18
DAFTAR PUSTAKA 18
LAMPIRAN 22
DAFTAR TABEL
1 Frekuensi keberadaan Enterobacteriaceae dan E. coli pada tempe segar 11 2 Hasil BLASTN sekuen isolat E. coli dari tempe dan isolat medis 12
DAFTAR GAMBAR
1 Tahapan kerja penelitian 6
2 Pertumbuhan bakteri pada media EMB Agar dan SSA 10 3 Perbedaan warna hijau metalik yang diduga E. coli pada tempe dalam
media EMB Agar 10
4 Hasil amplifikasi gen penyandi 16S rRNA isolat E. coli dari tempe 11 5 Profil ERIC-PCR isolat E. coli dari tempe dan isolat medis 13 6 Pohon filogenetik ERIC-PCR E. coli menggunakan metode UPGMA
melalui program MEGA 5.2 dengan 1000x bootstrap 14 7 Hasil pengulangan profil genetik ERIC-PCR E. coli 15
DAFTAR LAMPIRAN
1 Penampilan dan keadaan morfologi luar tempe segar EMP, DRG, WJB
dan CLR 22
2 Data hasil isolasi Enterobacteriaceae dalam media EMB Agar pada
tempe EMP, DRG, WJB dan CLR 23
3 Tahapan pembuatan tempe yang dilakukan oleh pengrajin tempe EMP,
PENDAHULUAN
Latar Belakang
Tempe dikenal sebagai makanan tradisional Indonesia yang terbuat dari kedelai dan difermentasi oleh Rhizopus spp. Tempe mengandung nutrisi, vitamin, fitokimia, antioksidan, sumber vitamin B12 (Keuth dan Bisping 1994; Astuti et al.
2000), asam lemak esensial (Utari 2010) serta senyawa isoflavon (Haron et al. 2009). Beberapa laporan menyatakan bahwa tempe memiliki banyak manfaat bagi kesehatan di antaranya dapat menangkal radikal bebas (Utari 2010), menurunkan resiko penyakit hati dan stroke, osteoporosis, kanker dan menopause (Babu et al. 2009).
Pembuatan tempe dilakukan dengan cara yang sangat bervariasi, dan umumnya pada kondisi yang tidak terkontrol. Proses pembuatan tempe melibatkan tiga faktor pendukung, yaitu bahan baku yang dipakai, mikroorganisme, dan keadaan lingkungan. Keberadaan Rhizopus pada tempe berperan sebagai kapang yang menyediakan substrat bagi bakteri dalam mensintesis komponen cita rasa dan komponen nutrisi lain (Seumahu 2012).
Beberapa bakteri telah diketahui berperan dalam meningkatkan kualitas tempe, seperti Citrobacter freundii dan Klebsiella pneumoniae (Keuth dan Bisping 1994), Lactobacillus reuteri (Taranto et al. 2003), Bacillus sp. (Barus et al. 2008), dan bakteri asam laktat (Efriwati et al. 2013). Selain itu, pada tempe ditemukan juga bakteri kelompok Enterobacteria yang didominasi oleh bakteri yang berwarna hijau metalik. Bakteri tersebut diduga adalah Escherichia coli (Barus et al. 2008). Kondisi yang tidak higienis mengakibatkan tumbuhnya E. coli yang dikenal sebagai bakteri indikator kebersihan lingkungan yang kemungkinan juga mengandung mikroorganisme enterik patogen lainnya (Clesceri et al. 1998).
Secara normal E. coli terdapat pada saluran pencernaan baik manusia maupun hewan. Bakteri ini juga mempunyai peran cukup penting dalam penyakit zoonosis yang disebarkan melalui makanan. E. coli patogen dapat menimbulkan sindrom klinis, yaitu gastroenteritis akut pada anak-anak dan infeksi saluran pencernaan. E. coli memiliki banyak faktor virulensi. Serotipe E. coli patogen yakni E. coli O157:H7 pernah ditemukan pada daging sapi (Suardana et al. 2007), dan VTEC (Verotoxigenic E. coli) juga pernah ditemukan pada susu dari peternakan (Suwito 2009).
2
dan diskriminatif (Olive dan Bean 1999). Metode ERIC-PCR telah berhasil menganalisis keragaman berbagai bakteri seperti Vibrio cholerae (Waturangi et al. 2012) dan Klebsiella sp. (Barus et al. 2013).
Perumusan Masalah
Teknik pembuatan tempe yang dilakukan oleh para pengrajin di Indonesia masih sangat sederhana dan dalam kondisi yang tidak terkontrol. Proses pembuatan tempe tradisional belum memiliki standar pengolahan yang higienis sehingga memungkinkan tumbuhnya E. coli. Keberadaan E. coli pada makanan memiliki peran dalam penyebaran penyakit mengingat bakteri tersebut memiliki banyak faktor virulensi. Oleh sebab itu, diperlukan adanya perhatian dalam segi keamanan pangan. Perbedaan genetik pada E. coli menentukan karakteristik E. coli terutama dalam bidang medis.
Tujuan Penelitian
Penelitian ini bertujuan membandingan profil genetik isolat E. coli yang berasal dari tempe dengan isolat medis berdasarkan sekuen ERIC-PCR.
Manfaat Penelitian
Manfaat penelitian ini merupakan informasi bahwa genetik antara isolat E. coli pada tempe berbeda dengan isolat medis serta memberikan informasi tentang keamanan pangan untuk kontrol kualitas lingkungan pada saat pengolahan tempe.
Ruang Lingkup Penelitian
3
TINJAUAN PUSTAKA
Tempe
Tempe didefinisikan sebagai hasil fermentasi Rhizopus spp. dengan bahan baku biji-bijian yang terikat bersama miselium kapang tersebut (Nout dan Kiers 2005). Tempe sudah dikenal berabad-abad lalu dan pertama kali dibuat oleh masyarakat Jawa Tengah sekitar tahun 1700-an (Astuti et al. 2000). Kata tempe sudah ditemukan sejak tahun 1660-an di dalam Serat Centhini yang berasal dari Jawa kuno dan disebutkan sebagai hidangan bernama jae santen tempe (sejenis masakan tempe dengan santan) dan kadhele tempe srundengan.
Tempe mengandung komponen antioksidan seperti isoflavon, vitamin E dan -karoten. Komponen isoflavon pada tempe juga berkontribusi pada ekspresi gen (Rimbach et al. 2008). Gamma-asam aminobutirat juga diproduksi selama proses fermentasi dan senyawa ini mempunyai efek farmakologis dalam tubuh manusia (Aoki et al. 2003). Ghozali et al. (2010) melaporkan bahwa pemberian tempe berpengaruh positif terhadap penurunan gula darah dan kecepatan kesembuhan luka pada tikus yang terkena diabetes.
Di Indonesia tempe dibuat secara tradisional pada skala industri kecil (industri rumah tangga). Pembuatan tempe dilakukan dengan cara yang sangat bervariasi oleh para pengrajin, yang berkembang secara turun temurun dan berubah karena pengalaman. Daerah yang berbeda memiliki proses pengolahan tempe yang berbeda dengan urutan proses yang berbeda juga (Barus et al. 2008). Meskipun cara pengolahan tempe setiap produsen berbeda tetapi pada dasarnya mempunyai prinsip dasar yang sama, yaitu persiapan dan fermentasi. Modifikasi yang dilakukan adalah lama perendaman, frekuensi perebusan, lama perebusan, penambahan cuka, penggilingan, pemakaian kembali kulit ari kedelai, jenis inokulum, lama fermentasi, dan tipe pengemasan (Utari 2010).
Proses pembuatan tempe kedelai terdiri atas beberapa tahap antara lain: pembersihan bahan, perendaman, pengupasan, perebusan, pencampuran laru, dan pembungkusan. Lama perendaman bervariasi, biasanya berkisar 8-12 jam, bahkan ada yang sampai 2-3 hari. Pada saat perendaman, air yang diabsorbsi oleh kedelai mendekati dua kali berat keringnya. Selama fermentasi asam oleh bakteri, pH turun hingga 5.3-4.5. Hal ini memberikan kondisi yang baik untuk pertumbuhan kapang tempe terutama Rhizopus oligosporus dan dapat mencegah perkembangan bakteri lain yang dapat membusukkan kedelai (Steinkraus 1983). Menurut Nout dan Kiers (2005) fermentasi pada tempe dapat meningkatkan kualitas nilai gizi dan karakter organoleptik.
4
Escherichia coli
E. coli merupakan bakteri fekal dari famili Enterobacteriaceae. Bakteri ini berbentuk batang dengan panjang 0.4-0.7 µm, lebar 1-3 µm, bersifat Gram negatif dan dapat berupa satu individu maupun berpasangan (Gyles et al. 2010; Songer dan Post 2005). E. coli bersifat anaerob fakultatif, memfermentasikan laktosa, sorbitol, dan menghasilkan gas. E. coli merupakan flora normal dalam saluran pencernaan hewan dan manusia. Tahun 1953 ditemukan strain E. coli yang patogen dan menyebabkan wabah diare pada bayi (Todar 2008). Galur-galur E. coli tertentu mampu menyebabkan gastroenteritis taraf sedang hingga parah pada manusia dan hewan. Kehadiran E. coli di usus manusia dan feses dapat digunakan sebagai indikasi terjadinya kontaminasi pada air, produk peternakan, dan produk pertanian (Todar 2008).
E. coli patogen memiliki virulensi dan mekanisme patogenik yang berbeda serta inang yang spesifik. Galur E. coli yang menyerang manusia diklasifikasikan ke dalam 5 grup yaitu Enteropathogenic E. coli (EPEC), Enterotoxigenic E. coli (ETEC), Enterohemorrhagic E. coli (EHEC), Enteroaggregative E. coli (EAEC) dan Enteroinvasive E. coli (EIEC) (Nataro dan Kaper 1998).
ETEC dapat menimbulkan diare seperti Vibrio cholerae yang aktif melekat pada mukosa usus kecil melalui permukaan fimbriae (pili tipe 1 dan antigen faktor kolonisasi) dan memproduksi satu atau dua enterotoksin, yaitu heat-labile toxin (LT) dan heat-stable toxin (ST). EPEC dapat menyebabkan sakit perut. EHEC mengeluarkan toksin yang disebabkan edema dan pendarahan difus di kolon dan dapat menimbulkan sindrom hemolitik oremik. Permulaan penyakit ini ditandai kejang yang akut dan diare cair yang cepat menjadi berdarah. EAEC menyebabkan diare berair pada anak-anak dan dapat berlanjut menjadi diare persisten. EIEC dapat menimbulkan demam, perut kram berlendir dan berdarah seperti disentri.
Klasifikasi serotipe E. coli terdiri atas: (1) lipopolisakarida somatik (O) bersifat tahan panas atau termostabil dan terdiri dari lipopolisakarida yang mengandung glukosamin dan terdapat pada dinding sel bakteri Gram negatif. (2) flagelar (H) merupakan alat pergerakan bakteri, bersifat tidak tahan panas atau termolabil dan akan rusak pada suhu 100 oC, dan (3) kapsula (antigen K) terdapat pada permukaan luar bakteri berfungsi untuk pertahanan diri. Komponen berupa polisakarida dan akan rusak pada suhu 100 oC selama 1 jam (Nataro dan Kaper 1998).
Pustaka Gen Penyandi 16S rRNA
5 homologus (Schluenzen et al. 2000). Gen 16S rRNA terdapat pada semua prokariot (Madigan et al. 2006) memiliki ukuran panjang antara 1.5-1.55 kb dan kaya akan basa nitrogen guanin dan sitosin dan tersusun atas daerah variabel dan konservatif (Clarridge 2004).
Dasar penggunaan gen 16S rRNA di antaranya; (1) bersifat universal (2) sekuen basa-basanya bersifat konservatif, (3) jumlahnya melimpah dalam sel, (4) memenuhi ukuran untuk perhitungan secara statistika (tidak terlalu panjang dan terlalu pendek), (5) ketersediaan informasi (data bank/data base di GeneBank) (Madigan et al. 2006). Proses amplifikasi gen 16S rRNA dapat dilakukan melalui teknik PCR. Beberapa primer universal yang umum digunakan untuk mengamplifikasi gen 16S rRNA bakteri, di antaranya 23f dan 24f serta 1392r dan 1492r (penomoran primer mengikuti konsensus sekuen 16S rRNA E. coli). Marchesi et al. (1998) mendesain dan mengevaluasi primer 63f dan 1387r untuk amplifikasi gen 16S rRNA dari domain bakteri. Pasangan primer ini mampu mengamplifikasi gen 16S rRNA dengan ukuran sekitar 1.3 kb.
Enterobacterial Repetitive Intergenic Consensus– Polymerase Chain Reaction (ERIC-PCR)
ERIC-PCR merupakan metode analisis keragaman berbasis PCR dengan cara mengamplifikasi DNA menggunakan sekuen ERIC. ERIC-PCR pertama kali ditemukan oleh Versalovic et al. (1991) pada E. coli dan Salmonella typhimurium. DNA kromosomal dari Enterobacteriaceae mengandung beberapa sekuen repetitif yaitu sekuen ERIC. Sekuen repetitif tersebut dapat ditemukan pada bakteri seperti Proteus mirabilis, K. pneumoniae, V. cholerae, Rhizobium meliloti, Bifidobacterium sp., Aeromonas sp., dan bakteri Gram positif, seperti Staphylococcus sp. dan Streptococcus sp. (Versalovic et al. 1991; de Bruijn 1992; Ventura et al. 2003; Szczuka dan Kaznowski 2004).
Primer ERIC-PCR yang digunakan bersifat spesifik, serta biasa digunakan untuk DNA fingerprinting. Primer yang digunakan dirancang untuk mengidentifikasi wilayah ERIC yang lestari dalam rangka untuk menghasilkan pola pita berdasarkan frekuensi dan orientasi urutan ERIC dalam genom bakteri (Meacham et al. 2003). Chuzcik et al. (2003) telah mengevaluasi pasangan primer ERIC 1R dan ERIC 2, dan pasangan primer tersebut mampu mengamplifikasi sekuen E. coli yang berasal dari air dengan ukuran pita 0.25 kb-4.0 kb.
6
METODE PENELITIAN
Kerangka Penelitian
Tahapan kerja yang dilakukan dalam penelitian ini, secara singkat disajikan pada Gambar 1 dibawah ini.
Gambar 1 Tahapan kerja penelitian
Waktu dan Tempat Penelitian
Penelitian dilakukan mulai bulan Desember 2013 sampai September 2014 bertempat di Laboratorium Riset Fakultas Teknobiologi Universitas Katolik Atma Jaya Jakarta.
Bahan
Bahan-bahan yang digunakan dalam penelitian ini ialah sampel tempe segar, isolat E. coli dari tempe, isolat medis yaitu E. coli ATCC 25922 (koleksi Laboratorium Mikrobiologi Fakultas Kedokteran Universitas Indonesia, Jakarta),
Isolasi E. coli pada media EMB agar
Uji konfirmasi dengan Simmon Sitrat Agar agar
Isolasi DNA genom E. coli
Pengambilan sampel tempe segar dari pengrajin
Amplifikasi gen penyandi 16S rRNA E. coli
Sekuensing gen penyandi 16S rRNA E. coli
Pensejajaran sekuen pada data base GenBank
Analisis profil genetik E. coli dengan sekuen ERIC-PCR
7 E. coli O157 (koleksi Laboratorium Mikrobiologi Fakultas Kedokteran Universitas Atma Jaya, Jakarta), dan EPEC K.1.1 (koleksi Laboratorium Mikrobiologi Institut Pertanian Bogor, Bogor) dan E. coli DH5α sebagai kontrol positif (koleksi Fakultas Teknobiologi Universitas Katolik Atma Jaya, Jakarta). K. pneumoniae yang diisolasi dari tempe digunakan sebagai outgroup.
Isolasi E. coli dari tempe
Sampel tempe diperoleh dari 4 pengrajin tempe di Bogor, yaitu tempe
EMP, DRG, WJB dan CLR (singkatan nama lokasi pengrajin tempe) (Lampiran 1). Pengambilan sampel dilakukan sebanyak 4 kali pengulangan dari
bulan Desember 2013 sampai Maret 2014.
Sebanyak 10 g tempe segar dihomogenkan ke dalam 90 mL larutan garam fisiologis 0.85% (NaCl). Pengenceran dilakukan dari 10-1 sampai 10-5 dan 100 µL dari pengenceran 10-2 sampai 10-5 disebar ke dalam media EMB (Oxoid) dan diinkubasi pada suhu 37 oC selama 24 jam. Koloni dengan ciri hijau metalik dimurnikan pada media EMB dengan metode kuadran. Koloni tersebut kemudian diuji dengan media SSA (Difco) dan diinkubasi pada suhu 37 oC selama 24 jam. Selanjutnya, isolat disimpan pada media Nutrient Agar (NA) (Oxoid) untuk dianalisis lebih lanjut.
Isolasi DNA Genom E. coli
DNA genom diisolasi dengan metode Cetyl Trimethyl Ammonium Bromide (CTAB) (Sambrook dan Russel 2001). Kultur E. coli ditumbuhkan pada media Luria Broth (LB) (Lennox) selama 16 jam pada suhu 37 oC dalam shaker dengan kecepatan 120 rpm. Sebanyak 1.5 mL kultur bakteri dipindahkan ke dalam tube dan disentrifugasi pada kecepatan 10.000 rpm selama 2 menit (dilakukan 2 kali). Supernatan dibuang dan pelet ditambahkan sebanyak 500 µL 1X Tris-EDTA (TE). Setelah proses pemanenan sel dilakukan, selanjutnya adalah proses pelisisan dinding sel bakteri yaitu sebanyak 50 µL lisozim (50 µg/mL) ditambahkan kedalam tube lalu dibolak-balik sampai keruh. Kemudian diinkubasi pada suhu 37
oC selama 60 menit. Sebanyak 50 µL SDS 10% dan 10 µL Proteinase-K (10
µg/mL) ditambahkan kedalam tube. Diinkubasi kembali pada suhu 37 oC selama 60 menit (sampai bening).
8
pada kecepatan 10.000 rpm selama 2 menit, supernatan dibuang kemudian pelet ditambahkan sebanyak 1 mL etanol 70% dan dibolak-balik perlahan untuk pencucian dan disentrifugasi cepat, supernatan dibuang dan dikeringudarakan. Pelet yang dihasilkan diresuspensi dengan 50 µL 1X TE.
DNA genom dikuantifikasi menggunakan elektroforesis pada gel agarosa 1%. Sebanyak 5µL DNA dicampur dengan 1 µL loading dye, dijalankan pada tegangan 80 volt selama 45 menit. Setelah itu, pita DNA yang terbentuk diamati dengan pewarnaan Ethidium Bromide (EtBr) dan dilihat dibawah UV transiluminator untuk didokumentasikan.
Amplifikasi Gen Penyandi 16S rRNA E. coli
Amplifikasi gen penyandi 16S rRNA menggunakan primer 6γf (5’CAG GCC TAA CAC ATG CAA GTC-γ’) dan primer 1γ87r (5’-CCC GGG AAC GTA TTC ACC GC-γ’) (Marchesi et al. 1998). Total volume PCR yang digunakan adalah 50 µL, setiap tabung terdiri atas 25 µL GoTaq Green® Master Mix (Promega), 2 µL primer forward dan reverse (25 pmol/µL-1), 19 µL nuclease free water dan 1 µL cetakan DNA. Kondisi PCR sebagai berikut: pre-denaturasi 94 oC selama 5 menit, denaturasi 94 oC selama 30 detik, annealing primer 55 oC selama 30 detik, elongasi 72 oC selama 1 menit, dan post PCR pada suhu 72 oC selama 20 menit. Penghentian reaksi dilakukan dengan penurunan suhu ke 4 oC
dan PCR berlangsung sebanyak 35 siklus.
Hasil PCR dielektroforesis dengan gel agarosa 1% (Bioline) dalam buffer 1X Tris Asetat EDTA (TAE) pada 80 volt selama 60 menit. Selanjutnya, pita-pita DNA hasil isolasi dilihat dibawah UV transluminator (UVB 36 ultralum, Carson, California).
Sekuensing Gen Penyandi 16S rRNA E. coli
Produk PCR dikirim ke perusahaan jasa sekuensing First Base Sequensing INT, Malaysia dan dianalisis menggunakan program MEGA 5.2. Hasil sekuensing disejajarkan pada data base dengan program Basic Local Alignment Search Tool Nucleotide (BLASTN) yang tersedia pada situs National Center for Biotechnology Information (NCBI) melalui website (http://www.ncbi.nlm.nih.gov/BLAST).
Analisis Profil Genetik E. coli dengan Sekuen ERIC-PCR
9 selama 1 menit, elongasi 65 oC selama 8 menit, post PCR 65 oC selama 16 menit
serta penyimpanan pada suhu 4 oC. Proses PCR berlangsung sebanyak 30 siklus. Sebanyak 3 µL hasil amplifikasi PCR diverifikasi dengan elektroforesis gel agarosa 1.5% dalam buffer 1X TAE pada 113 volt selama 120 menit.
Konstruksi Pohon Filogeni
Profil fragmen hasil amplifikasi ERIC-PCR diinterpretasikan menjadi matriks data biner dan dijadikan input untuk pembuatan pohon filogenetik dengan menggunakan program MEGA 5.2. Konstruksi pohon filogenetik menggunakan metode Unweighted Pair Groups Method Analysis (UPGMA). Topologi dari konstruksi pohon filogenetik dievaluasi menggunakan analisis bootstrap dengan 1000x.
HASIL DAN PEMBAHASAN
Hasil
Identifikasi Enterobacteriaceae dan Frekuensi Keberadaan E. coli
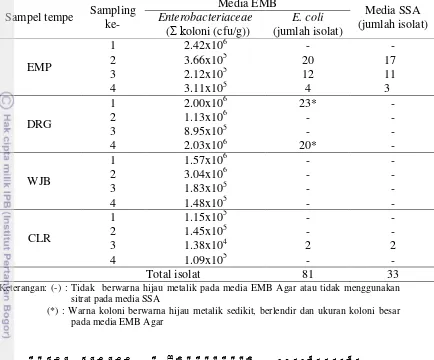
Total Enterobacteriaceae pada media EMB dari tempe EMP, DRG, WJB dan CLR beragam sekitar 105-106 cfu/g (Tabel 1). Rata-rata jumlah Enterobacteriaceae pada tempe DRG, WJB, EMP, dan CLR yaitu berturut-turut 6.2, 6.0, 5.7, 4.9 cfu/g (Lampiran 2). Koloni Enterobacteriaceae selain E. coli tidak berwarna pada media EMB Agar (Gambar 2A). Jumlah koloni yang berwarna hijau metalik banyak ditemukan pada tempe EMP dibandingkan dengan tempe dari CLR, DRG, dan WJB.
Sebanyak 81 isolat yang diduga E. coli telah berhasil diisolasi dari tempe EMP, DRG dan CLR (Tabel 1). Isolat tersebut berwarna hijau metalik pada media EMB (Gambar 2B). Namun, setelah diuji lebih lanjut dengan media SSA hanya 33 isolat yang menunjukkan positif E. coli yang ditandai dengan media berwarna hijau (Gambar 2C).
E. coli tidak selalu ditemukan pada tempe. E. coli hanya ditemukan pada tempe EMP dan CLR dengan jumlah yang bervariasi pada 4 kali pengambilan sampel dari produsen. E. coli pada tempe EMP ditemukan dari pengambilan sampel ke-2 sampai ke-4. E. coli pada tempe CLR hanya ditemukan 1 kali dari 4 kali pengambilan (Tabel 1).
10
Gambar 2 Pertumbuhan bakteri pada media EMB dan SSA. Pertumbuhan Enterobacteriaceae pada media EMB Agar (A). Isolat E. coli pada media EMB Agar (B). Isolat E. coli pada media SSA (C)
Gambar 3 Perbedaan warna hijau metalik yang diduga E. coli pada tempe dalam media EMB Agar. Terduga E. coli dari tempe DRG (A). Terduga E. coli dari tempe EMP (B). Terduga E. coli dari tempe CLR (C)
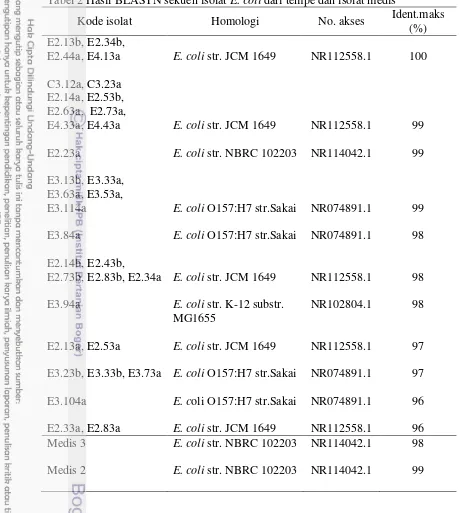
Gen 16S rRNA 33 isolat E. coli telah berhasil diamplifikasi menggunakan primer 63f dan 1387r serta menghasilkan pita DNA berukuran sekitar 1.3 kb (Gambar 4). Hasil pensejajaran sekuen pada data base di GenBank NCBI menggunakan program BLASTN menunjukkan bahwa 33 isolat tersebut merupakan E. coli dengan persentase kemiripan 96%-100% dengan E-value 0.0 (Tabel 2).
(B) (C)
(A)
(A) (B)
11 Tabel 1 Frekuensi keberadaan Enterobacteriaceae dan E. coli pada tempe segar
12
Tabel 2 Hasil BLASTN sekuen isolat E. coli dari tempe dan isolat medis
Kode isolat Homologi No. akses Ident.maks (%) E2.13b, E2.34b,
E2.44a, E4.13a E. coli str. JCM 1649 NR112558.1 100 C3.12a, C3.23a
E2.14a, E2.53b, E2.63a, E2.73a,
E4.33a, E4.43a E. coli str. JCM 1649 NR112558.1 99 E2.23a E. coli str. NBRC 102203 NR114042.1 99 E3.13b, E3.33a,
E3.63a, E3.53a,
E3.114a E. coli O157:H7 str.Sakai NR074891.1 99 E3.84a E. coli O157:H7 str.Sakai NR074891.1 98 E2.14b, E2.43b,
E2.73b, E2.83b, E2.34a E. coli str. JCM 1649 NR112558.1 98 E3.94a E. coli str. K-12 substr.
MG1655
NR102804.1 98
E2.13a, E2.53a E. coli str. JCM 1649 NR112558.1 97 E3.23b, E3.33b, E3.73a E. coli O157:H7 str.Sakai NR074891.1 97 E3.104a E. coli O157:H7 str.Sakai NR074891.1 96 E2.33a, E2.83a E. coli str. JCM 1649 NR112558.1 96 Medis 3 E. coli str. NBRC 102203 NR114042.1 98 Medis 2 E. coli str. NBRC 102203 NR114042.1 99
Analisis Profil Genetik ERIC-PCR dan Konstruksi Pohon Filogeni
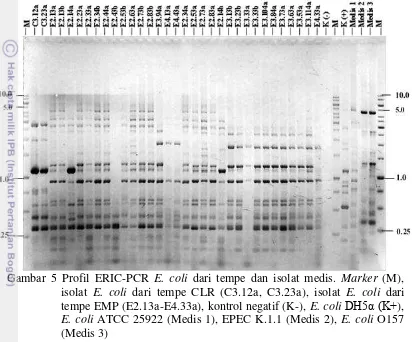
13 Profil ERIC-PCR isolat E. coli dari tempe CLR menunjukkan pola yang sama sedangkan isolat E. coli dari tempe EMP memiliki pola yang beragam (Gambar 5). Meskipun profil genetik isolat E. coli dari tempe EMP lebih beragam dari tempe CLR tidak ada profil genetik yang sama ketika isolat E. coli dari tempe tersebut dibandingkan dengan E. coli DH5α.
Gambar 5 Profil ERIC-PCR E. coli dari tempe dan isolat medis. Marker (M), isolat E. coli dari tempe CLR (C3.12a, C3.23a), isolat E. coli dari tempe EMP (E2.13a-E4.33a), kontrol negatif (K-), E. coli DH5α (K+), E. coli ATCC 25922 (Medis 1), EPEC K.1.1 (Medis 2), E. coli O157 (Medis 3)
14
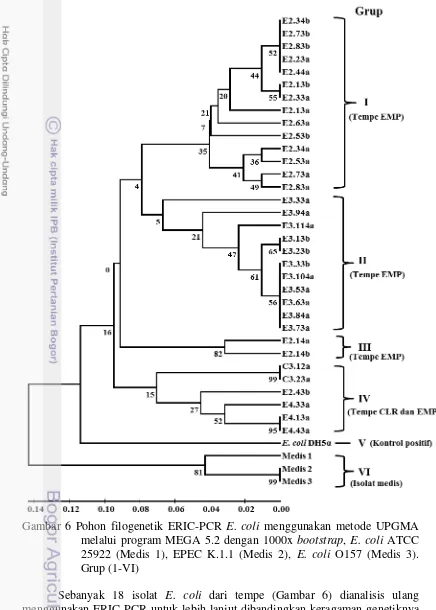
Gambar 6 Pohon filogenetik ERIC-PCR E. coli menggunakan metode UPGMA melalui program MEGA 5.2 dengan 1000x bootstrap, E. coli ATCC 25922 (Medis 1), EPEC K.1.1 (Medis 2), E. coli O157 (Medis 3). Grup (1-VI)
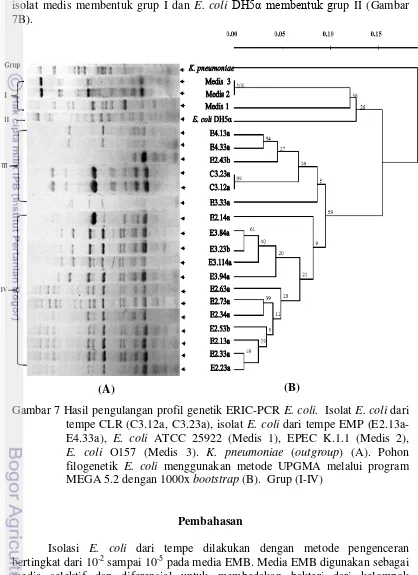
15 isolat dari tempe CLR (isolat C3.23a dan C3.12a). Grup kedua (grup IV) terdiri atas 12 isolat E. coli dari tempe EMP (isolat E2.14a, E3.84a, E3.23b, E3.114a, E3.94a, E2.63a, E2.73a, E2.34a, E2.53b, E2.13a, E2.33a, E2.33a), sedangkan isolat medis membentuk grup I dan E. coli DH5α membentuk grup II (Gambar 7B).
Gambar 7 Hasil pengulangan profil genetik ERIC-PCR E. coli. Isolat E. coli dari tempe CLR (C3.12a, C3.23a), isolat E. coli dari tempe EMP (E2.13a-E4.33a), E. coli ATCC 25922 (Medis 1), EPEC K.1.1 (Medis 2), E. coli O157 (Medis 3). K. pneumoniae (outgroup) (A). Pohon filogenetik E. coli menggunakan metode UPGMA melalui program MEGA 5.2 dengan 1000x bootstrap (B). Grup (I-IV)
Pembahasan
Isolasi E. coli dari tempe dilakukan dengan metode pengenceran bertingkat dari 10-2 sampai 10-5 pada media EMB. Media EMB digunakan sebagai media selektif dan diferensial untuk membedakan bakteri dari kelompok Enterobacteriaceae. Media ini disebut sebagai media selektif karena mampu menghambat pertumbuhan bakteri Gram positif seperti Staphylococcus spp. dan diferensial karena mengandung eosin dan methylene blue serta laktosa yang
16
berfungsi untuk membedakan bakteri yang memfermentasikan laktosa seperti Staphylococcus aureus, Pseudomonas aerugenosa, dan Salmonella. Bakteri yang memfermentasi laktosa menghasilkan koloni dengan inti berwarna gelap dengan kilap logam. Sedangkan bakteri lain yang dapat tumbuh koloninya tidak berwarna. Hal ini terjadi karena adanya penurunan pH media yang menyerap indikator sehingga warna koloni menjadi hijau metalik (Madigan dan Martinko 2006). Setelah diuji dengan media SSA hanya 33 isolat yang menunjukkan positif E. coli yang ditandai dengan media berwarna hijau (Gambar 2C). Hasil ini menunjukkan bahwa E. coli tidak menggunakan sitrat sebagai satu-satunya sumber karbon dan energi.
Hasil identifikasi koloni yang diduga E. coli dari tempe DRG setelah dilakukan sekuensing dinyatakan sebagai Enterobacter aerogenes (Data tidak ditampilkan). Isolat tersebut setelah diuji konfirmasi menggunakan media SSA menunjukkan perubahan warna media dari hijau menjadi biru. Hal tersebut mengindikasikan bahwa isolat tersebut negatif E. coli. Selain E. aerogenes ditemukan juga K. pneumoniae dari tempe EMP. Isolat K. pneumoniae tersebut kemudian dijadikan sebagai outgroup dalam analisis pohon filogenetik berdasarkan profil pita ERIC-PCR.
Keberadaan E. coli pada tempe sebelumnya telah dilaporkan oleh Barus et al. (2008). Keberadaan E. coli pada tempe bervariasi pada setiap pengambilan sampel ke-1 sampai ke-4 (Tabel 1). E. coli tidak selalu ditemukan pada keempat tempe (EMP, DRG, WJB, dan CLR) meskipun pada sampel yang sama. E. coli pada tempe kemungkinan berasal dari berbagai sumber, misalnya berasal dari bahan baku, alat yang digunakan, orang yang terlibat dalam pengolahan, dan lingkungan sekitarnya. Penyebaran E. coli dapat tejadi karena adanya kontaminasi silang secara langsung (melalui tangan) dan tidak langsung (melalui air) selama pengolahan. Keberadaan E. coli pada makanan menunjukkan bahwa makanan tersebut tercemar kotoran akibat pengolahan dan kebersihan pengolah makanan yang kurang baik. E. coli merupakan bakteri patogen yang sering dijadikan indikator sanitasi (Githiri et al. 2009).
Berdasarkan penelitian yang dilakukan Barus et al. (2008) kondisi proses pembuatan tempe WJB lebih higienis dibandingkan dengan kondisi proses pembuatan tempe EMP. Perbedaan kondisi tersebut kemungkinan yang menyebabkan tidak ditemukannya E. coli pada tempe WJB. Keberadaan E. coli pada tempe EMP dan CLR juga belum bisa dipastikan berasal dari kontaminasi silang secara langsung atau tidak langsung. Hasil pencawanan pada media EMB Agar yang diambil dari sampel air dan wadah pencampuran kedelai dengan ragi pada tempe EMP tidak ditemukan adanya pertumbuhan E. coli (Data tidak ditampilkan).
17 dilakukan 2 kali perebusan. Barus et al (2008) menyatakan bahwa perbedaan cara pengolahan berpengaruh terhadap kelimpahan bakteri pada tempe.
Pendekatan metode molekuler untuk mendeteksi keberadaan E. coli adalah dengan mengamplifikasi sekuen DNA yang mengkodekan gen 16S rRNA. E. coli diamplifikasi dengan menggunakan primer 63f dan 1387r. Selama proses amplifikasi berlangsung, primer 63f mulai mengamplifikasi cetakan DNA pada posisi basa 43-63, sedangkan primer 1387r mengamplifikasi DNA pada posisi 1387-1370 sehingga produk yang dihasilkan berukuran 1.3 kb (Marchesi et al. 1998). Sekuen gen 16S rRNA digunakan sebagai penanda molekuler karena bersifat ubikuitis dengan fungsi yang identik pada seluruh organisme dan memiliki urutan basa yang relatif konservatif dan beberapa daerah urutan basanya variatif (Pangastuti 2006).
Analisis sifat virulensi dari suatu bakteri selain secara genetik dapat diketahui dari fenotipenya (Stenutz et al. 2006). Beberapa serotipe pada E. coli diketahui memiliki faktor virulensi diantaranya: serotipe O (somatik), serotipe H (flagella), dan serotipe K (kapsular) (Nataro dan Kaper 1998). Serotipe O157: H7 adalah serotipe yang dapat menginduksi sekresi cairan tubuh secara berlebihan dan terus menerus sehingga terjadi diare dan dapat menyebabkan meningitis. E. coli O157 sebagai pembawa gen VT1 dan VT2 paling banyak ditemukan pada babi (Suardana et al. 2007).
Pada penelitian ini, fragmen multiple DNA semua strain E. coli yang di amplifikasi dengan primer ERIC berkisar antara 4-13 pita diantara ukuran 0.25 kb dan 5.0 kb (Gambar 5). Hasil ini mengindikasikan luasnya keragaman genetik pada E. coli. Menurut Meacham (2003) perbandingan jumlah dan ukuran profil pita menunjukkan keragaman genetik isolat-isolat bakteri. Teknik ERIC-PCR menggunakan primer khusus untuk mengamplifikasi sekuen DNA yang berada diantara urutan ERIC yang berulang pada bakteri enterik Gram negatif (Hulton et al. 1991).
Isolat E. coli dari tempe memiliki ciri morfologi yang sama dengan isolat medis pada media EMB, namun dapat dilihat bahwa sifat genotipik isolat ini berbeda dengan isolat medis (Gambar 5). Pada penelitian ini, isolat medis diperoleh dari rumah sakit yang berasal dari feses pasien yang mengandung E. coli patogen. Oleh sebab itu, secara genetik isolat E. coli yang diperoleh dari tempe tidak sama dengan isolat medis yang berasal dari feses manusia.
Pohon filogenetik berdasarkan profil pita ERIC-PCR yang berasal dari 2 kali pengulangan menunjukkan bahwa isolat E. coli dari tempe konsisten membentuk grup yang terpisah dengan isolat medis (Gambar 7B). Isolat E. coli dari tempe CLR tidak menunjukkan adanya keragaman genetik tetapi isolat tersebut memiliki kekerabatan dengan beberapa isolat E. coli dari tempe EMP sehingga tergabung dalam grup III (Gambar 7B). Berbeda dengan isolat E. coli dari tempe CLR, isolat E. coli dari tempe EMP menunjukkan adanya keragaman genetik. Pola hubungan kekerabatan isolat E. coli dari tempe EMP ada yang berkerabat dekat namun ada pula yang berkerabat jauh. Hal ini menunjukkan bahwa isolat E. coli dari tempe EMP memiliki keragaman yang tinggi. Sampel tempe yang sama dapat membawa genotip yang berbeda dari E. coli. Dilain pihak, genotip yang sama dapat ditemukan didalam sampel tempe yang berbeda.
18
intraspesies bakteri. Hasil penelitian ini mengindikasikan bahwa teknik ERIC-PCR telah berhasil membedakan profil genetik isolat E. coli dari tempe dengan isolat medis dan mampu bersifat diskriminatif dibandingkan dengan analisis gen 16S rRNA. Penelitian sebelumnya melaporkan bahwa ERIC-PCR telah berhasil mengamplifikasi sekuen ERIC Klebsiella spp. yang berasal dari tempe dan memperoleh hasil yang lebih diskriminatif dari sekuen 16S rRNA (Barus et al. 2013). Ayu et al. (2014) melaporkan bahwa ERIC-PCR juga mampu membedakan profil genetik K. pneumoniae dari tempe dengan K. pneumoniae medis. Selain itu, ERIC-PCR juga dapat digunakan untuk membedakan bakteri Gram positif seperti Staphylococcus sp. dan Streptococcus sp. (Versalovic et al. 1991; de Bruijn 1992; Ventura et al. 2003; Szczuka dan Kaznowski 2004). Kelemahan analisis ERIC-PCR adalah penggunaan kondisi PCR dan waktu elektroforesis yang lebih lama dibandingkan dengan PCR dan elektroforesis biasa. Namun demikian, analisis dengan teknik ERIC-PCR ini merupakan metode yang dapat digunakan untuk membandingkan intraspesies isolat E. coli dari tempe dengan isolat medis.
Hasil analisis ERIC-PCR menunjukkan bahwa secara genetik isolat E. coli dari tempe berbeda dengan isolat medis. Belum ada laporan yang menyebutkan bahwa mengkonsumsi tempe yang mengandung E. coli akan menyebabkan sakit. Secara estetika, tempe yang diproduksi dalam kondisi tidak higienis pada umumnya akan mengandung E. coli. Tetapi, itu tidak berarti bahwa tempe mengandung bakteri yang berbahaya.
SIMPULAN
Simpulan
Tidak semua tempe mengandung E. coli dan keberadaan E. coli pada tempe ditingkat produsen tidak selalu konsisten ditemukan pada setiap produksi. Identifikasi berdasarkan gen penyandi 16S rRNA menyatakan bahwa 33 isolat dari tempe EMP dan CLR merupakan E. coli dengan kemiripan sebesar 96%-100%. Hasil analisis profil DNA genetik menggunakan ERIC-PCR menunjukkan bahwa isolat E. coli dari tempe EMP dan CLR berbeda dengan E. coli patogen atau yang berasosiasi dengan isolat medis.
DAFTAR PUSTAKA
Aoki H, Uda I, Tagami K, Furuya Y, Endo Y, Fujimoto K. 2003. The production of a new tempeh-like fermented soybean containing a high level of -aminobutyric acid by anaerobic incubation with Rhizopus. Biosci Biotechnol Biochem. 67(5):1018-1023.
19 Ayu E, Suwanto A, Barus T. 2014. Klebsiella pneumoniae from Indonesian tempeh were genetically different from that of pathogenic isolates. Microbiol Indones. 8(1):9-15.doi: 10.5454/mi.8.1.2.
Babu PD, Bhakyaraj R, Vidhyalaksmi R. 2009. A low cost nutritious food “tempeh”-a review. World J Dairy food Sci. 4(1):22-27.
Barus T, Suwanto A, Wahyudi AT, Wijaya H. 2008. Role of bacteria in tempe bitter taste formation; microbiological and molecular biological analysis based on 16S rRNA gene. Microbiol Indones. 2(1):17-21.
Barus T, Hanjaya I, Sadeli J, Lay BW, Suwanto A, Yulandi A. 2013. Genetic diversity of Klebsiella spp. isolated from tempe based on enterobacterial repetitive intergenic consensus-polymerase chain reaction (ERIC-PCR). Hayati J Biosci. 20(4):171-176. doi:10.4308/hjb.20.4.171.
Case RJ, Boucher Y, Dahllof I, Holmstrom C, Doolittle WF, Kjelleberg S. 2007. Use of 16S rRNA and rpoB genes as molecular markers for microbial ecology studies. Appl Environ Microbiol. 73(1):278-88. doi:10.1128/AEM.01177-06.
Clarridge III JE. 2004. Impact of 16S rRNA gene sequence analysis for identification of bacteria on clinical microbiology and infectious diseases. J Clin Microbiol. 17(4):840-862. doi:10.1128/CMR.17.4.840-862.2004. Chudzik KB, Niedbach J, Stosik M. 2003. REP-PCR fingerprinting as a tool for
the analysis of genomic diversity in Escherichia coli strains isolated from an aqueous/freshwater environment. Cell Mol Biol Lett. 8:793-798.
Clesceri LS, Greenberg AE, Eat on AD. 1998. Standard Methods for the Examination of Water and Wastewater, 20th ed. Washington DC (US): American Public Health Associati on Washington DC.
de Bruijn FJ. 1992. Use of repetitive (repetitive extragenic poliandromic and enterobacterial repetitive intergeneric consensus) sequences and the polymerase chain reaction to fingerprint the genomes of Rhizobium meliloti isolates and other soil bacteria. Appl Environ Microbiol. 58(7):2180-2187. Efriwati, Suwanto A, Rahayu G, Nuraida L. 2013. Population dynamics of yeasts
and lactic acid bacteria (LAB) during tempeh production. Hayati J Biosci. coliforms and Staphylococcus on food at a public hospital in Kenya. J Appl Biosci. 27:1727-1731.
Gyles CL, Prescott JF, Songer G, Thoen CO. 2010. Pathogenesis of Bacterial Infections in Animals, 4th ed. Iowa (US): Wiley-Blackwell.
Haron H, Ismail A, Azlan A, Shahar S, Loh Su Peng. 2009. Daidzein and genestein contents in tempeh and selected soy products. Food Chem. 115:1350-1356.doi:10.1016/j.foodchem. 2009.01.053.
20
Keuth S, Bisping B. 1994. Vitamin B12 production by Citrobacter freundii or
Klebsiella pneumoniae during tempeh fermentation and proof of enterotoxin absence by PCR. Appl Environ Microbiol. 60(5):1495-1499.
Lupski JR, Weinstock GM. 1992. Short, interspersed repetitive DNA sequences in prokaryotic genomes. J Bacteriol. 174(14):4525-4529. genotyping large numbers of Eschericia coli isolates by enterobacterial repetitive intergenic consensus-PCR. J Clin Microbiol. 41(11):5224-5226.doi:10.1128/JCM.41.11.5224-5226.2003.
Nataro JP, Kaper JB. 1998. Diarrheagenic Escherichia coli. J Clin Microbiol Rev. 11(1):142-201.
Nout MJR, Kiers JL. 2005. Tempe fermentation, innovation and functionality: update into the third millennium [ulas balik]. Appl Environ Microbiol. 98:789-805.doi:10.1111/j.1365-2672.2004.02471.x.
Olive DM, Bean P. 1999. Principles and applications of methods for DNA-Based typing of microbial organisms [ulas balik mini]. J Clin Microbiol. 37(6):1661-1669.
Pangastuti A. 2006. Species definition of procaryotes based on 16S rRNA and protein coding genes sequences. Biodivers. 7(3):292-296.
Rademaker JLW, de Bruijn FJ. 1997. Characterization and Classification of Microbes by REP-PCR Genomic Fingerprinting and Computer Assisted Pattern Analysis. DNA Markers: Protocols, Applications and Overviews. New York (US): Wiley and Sons. hlm 151-171.
Rimbach G, Boesch-Saadatmandi C, Frank J, Fuchs D, Wenzel U, Daniel H, Hall WL, Weinberg PD. 2008. Dietary isoflavones in the prevention of cardiovascular disease- a molecular prespective. Food Chem Toxicol. 46:1308-1319.
Sahilah AM, Tuan Suraya TS. Noraida I, Ahmad Azuhairi A, Chai LC, Son R. 2010. Detection of virulence genes and enterobacterial repetitive intergenic consensus-PCR (ERIC-PCR) analysis among raw vegetables isolates of Campylobacter jejuni. Int Food Res J. 17: 681-690
Sambrook J, Russell DW. 2001. Molecular Cloning: A Laboratory Manual, 3rd ed. New York (US): Cold Spring Harbor Laboratory Pr.
Seumahu CA. 2012. Analisis metagenom untuk pencirian komunitas bakteri dan fungi pada tempe. [disertasi]. Bogor (ID): Institut Pertanian Bogor.
Schluenzen F, Tocilj A, Zarivach R, Harms J, Gluehmann M, Janell D, Bashan A, Bartels H, Agmon I, Franceschi F, et al. 2000. Structure of functionally activated small ribosomal subunit at 3.3 Å resolution. Cell. 102:615-623. Shangkuan YH, Yang JF, Lin HC, Shaio MF. 2000. Comparison of PCR-RFLP,
21 Songer JG, Post KW. 2005. Veterinary Microbiology: Bacterial and Fungal
Agents of Animal Disease. St Louis (US): Elsevier Saunders Missouri. Steinkraus KH. 1983. Handbook of Indigenous Fermented Food. New York:
Marcel Dekker, Inc. hlm 131-146.
Stenutz R, Weintraub A, Widmalm G. 2006. The structures of Escherichia coli O-polysaccharide antigens. FEMS Microbiol Rev. 30:382-403.doi:10.1111/j.1574-6976.2006.00016.x
Stephenson DP, Moore RJ, Allison GE. 2009. Comparison and utilization of repetitive-element PCR techniques for typing Lactobacillus isolates from the chicken gastrointestinal tract. Appl Environ Microbiol. 75(21):6764-6776. doi:10.1128/AEM.01150-09.
Suardana IW, Sumiarto B, Lukman DW. 2007. Isolasi dan identifikasi Escherichia coli O157:H7 pada daging sapi di Kabupaten Badung Provinsi Bali. J Vet. 8(1):16-23.
Suwito W. 2009. Escherichia coli verotoksigenik (VTEC) yang diisolasi dari susu sapi. JITV. 14(3):237-243.
Szczuka E, Kaznowski A. 2004. Typing of clinical and environmental Aeromonas sp. strain by random amplified polymorphic DNA PCR, repetitive extragenic paliandromic PCR, and enterobacterial repetitive intergenic consensus sequence PCR. J Clin Microbiol. 42(1):220-228.doi:10.1128/JCM.42.1.220-228.2004.
Taranto MP, Vera JL, Hugenholtz J, De Valdez GF, Sesma F. 2003. Lactobacillus reuteri CRL1098 produces cobalamin. J Bacteriol. 185(18):5643-5647.doi:10.1128/JB.185.185643-5647.2003.
Todar K. 2008. Pathogenic Escherichia coli. Todar’s Online Textbook of Bacteriology University of Wisconsin-Madison Department of Bacteriology.[internet].[diunduh 2013 Des 25].1-4. Tersedia pada:http//textbookofbacteriology.net/E. coli.html.
Utari DM. 2010. Kandungan asam lemak, zink, dan copper pada tempe, bagaimana potensinya untuk mencegah penyakit degeneratif?. Gizi Indon. 33(2):108-115.
Ventura M, Meylan V, Zink R. 2003. Identification and tracing of Bifidobacterium species by use of enterobacterial repetitive intergenic consensus sequences. Appl Environ Microbiol. 69(7):4296-4301.doi:10.1128/AEM.69.7.4296-4301.2003.
Versalovic J, Koeuth T, Lupski JR. 1991. Distribution of repetitive DNA sequences in eubacteria and application to fingerprinting of bacterial genomes. Nucleic Acids Res. 19(24):6823-6831.
22
LAMPIRAN
Lampiran 1 Penampilan dan keadaan morfologi luar tempe segar EMP, DRG, WJB dan CLR
Tempe segar EMP Tempe segar DRG
Tempe segar WJB Tempe segar CLR
Tempe Morfologi luar
EMP - Kedelai berwarna coklat kekuningan - Tempe segar berwarna coklat kekuningan - Air rendaman lebih kental
DRG - Tempe segar berwarna putih/ kuning bersih - Tekstur padat, lebih kompak
- Bau nya enak
WJB - Kedelai tempe berwarna kuning bersih - Tempe segar berwarna kuning bersih - Jalinan miselium kapang berwarna putih CLR - Tekstur lebih halus
23
Lampiran 2 Data hasil isolasi Enterobacteriaceae dalam media EMB Agar pada tempe EMP, DRG, WJB dan CLR
Tempe Ulangan Pengenceran Ʃ koloni
(cfu/g)
Ʃ (log
cfu/g)
Rata-rata (log cfu/g)
10-2 10-3 10-4 10-5
EMP
1 TBUD TBUD TBUD TBUD 196 188 89 60 2.42x106 6.4 5.7
2 TBUD TBUD 290 250 130 136 12 18 3.66x105 5.6
3 TBUD TBUD 175 58 133 100 0 0 2.12x105 5.3
4 TBUD TBUD 200 250 100 105 33 20 3.11x105 5.5
DRG
1 TBUD TBUD TBUD TBUD 200 150 35 56 2.00x106 6.3 6.2
2 TBUD TBUD TBUD TBUD 79 109 34 26 1.13x106 6.1
3 TBUD TBUD TBUD TBUD 90 89 20 22 8.95x105 6.0
4 TBUD TBUD TBUD TBUD 199 202 25 15 2.03x106 6.3
WJB
1 TBUD TBUD TBUD TBUD 114 199 0 0 1.57x106 6.2 6.0
2 TBUD TBUD TBUD TBUD 224 262 63 120 3.04x106 6.5
3 TBUD TBUD 146 174 35 48 4 1 1.83x105 5.3
4 TBUD TBUD TBUD TBUD 137 126 34 28 1.48x106 6.2
CLR
1 TBUD TBUD 100 83 37 33 3 14 1.15x105 5.1 4.9
2 TBUD TBUD 13 15 15 14 1 4 1.45x105 5.2
3 109 135 27 33 0 0 0 0 1.38x104 4.1
4 TBUD TBUD 99 86 30 25 0 0 1.09x105 5.0
24
Lampiran 3 Tahapan pembuatan tempe yang dilakukan oleh pengrajin tempe EMP, DRG, CLR (A) dan WJB (B)
Perebusan
Perendaman ± 18 jam
pengupasan
pencucian Penambahan inokulum
pengemasan
tempe
(A)
Perebusan pertama
pengupasan
pencucian
Perendaman ± 14 jam
Perebusan kedua
Penambahan inokulum
pengemasan
tempe
25
RIWAYAT HIDUP
Penulis dilahirkan di Jakarta pada tanggal 07 September 1986 sebagai anak pertama dari enam bersaudara, dengan ayah bernama Saunin dan ibu bernama Hariroh. Pada tahun 2004, penulis lulus dari Madrasah Aliyah Annur Bekasi dan pada tahun yang sama diterima di Jurusan Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Universitas Islam As-Syafi’iyah (UIA) Jakarta, lulus pada tahun 2008. Pada tahun 2009 penulis bekerja sebagai pengajar di Sekolah Alam Bekasi dan bekerja sebagai staff pengajar salah satu Bimbingan Belajar di Bekasi pada tahun 2010 sampai 2011. Pada tahun 2012, penulis diterima di Program Studi Mikrobiologi (MIK) Institut Pertanian Bogor, Bogor. Penulis mendapatkan dana bantuan penelitian dari Lembaga Pendidikan Pengelolaan Dana (LPDP) Departemen Keuangan RI melalui program beasiswa tesis tahun 2014.