PERBANDINGAN SKEMA PARTISI DATA ALGORITME
PAIRWISE ALIGNMENT
PARALEL PADA
SISTEM
SHARED MEMORY
AURIZA RAHMAD AKBAR
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa tesis berjudul Perbandingan Skema Partisi Data Algoritme Pairwise Alignment Paralel pada Sistem Shared Memory
adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Maret 2015
Auriza Rahmad Akbar
RINGKASAN
AURIZA RAHMAD AKBAR. Perbandingan Skema Partisi Data Algoritme
Pairwise Alignment Paralel pada Sistem Shared Memory. Dibimbing oleh HERU SUKOCO dan WISNU ANANTA KUSUMA.
Algoritme pairwise alignment (PA) dipakai dalam bioinformatika untuk menjajarkan sepasang sekuens DNA atau protein. PA merupakan penyusun dasar algoritme multiple sequence alignment (MSA) yang digunakan untuk menjajarkan lebih dari dua sekuens sekaligus untuk menemukan homologi antar-sekuens. Selain itu, PA dipakai dalam perakitan sekuens untuk menggabungkan hasil pembacaan dari sequencer yang pendek-pendek menjadi satu gen utuh. Di samping itu, PA juga dipakai untuk pencarian sekuens pada database, untuk menemukan sekuens-sekuens yang paling mirip dengan sekuens yang dicari.
Teknologi next-generation DNA sequencer terkini mampu menghasilkan data sekuens yang banyak, hingga ratusan milyar pasang basa (bp) dalam sekali jalan. Data yang besar memerlukan pemrosesan yang cepat, sehingga algoritme penjajaran perlu diparalelkan untuk mempercepat proses analisis. Beberapa penelitian sebelumnya telah memparalelkan algoritme PA dengan berbagai skema partisi data, akan tetapi belum diketahui skema yang memberikan kinerja terbaik. Skema partisi data sangat mempengaruhi kinerja PA paralel, karena algoritme ini memakai teknik pemrograman dinamis yang membutuhkan banyak sinkronisasi.
Tujuan penelitian ini adalah menerapkan algoritme PA paralel dengan berbagai skema partisi data yang berbeda pada sistem shared-memory dan menguji serta memperbaiki masing-masing skema untuk mendapatkan skema yang memberikan kinerja terbaik. Skema tersebut antara lain blocked columnwise,
rowwise, antidiagonal, dan blocked columnwise dengan penjadwalan manual dan
loop unrolling. Pengujian kinerja dilakukan pada prosesor quad-core memakai data uji sekuens DNA dengan panjang 1000 hingga 16 000 bp.
Skema antidiagonal menghasilkan kinerja terburuk dengan efisiensi 36% karena waktu sinkronisasinya dua kali lipat lebih tinggi dan pola akses memori yang non-linier. Skema rowwise menghasilkan efisiensi 80%, sedangkan skema
blocked columnwise menghasilkan efisiensi 75%. Kedua skema ini memiliki waktu sinkronisasi yang hampir sama. Seharusnya skema blocked columnwise
bisa lebih baik, namun karena keterbatasan pustaka OpenMP kinerja skema ini lebih buruk dari rowwise.
Skema blocked columnwise diperbaiki dengan menggunakan penjadwalan manual dan loop unrolling. Penjadwalan manual mengatasi batasan pustaka OpenMP, sehingga skema ini menjadi lebih efisien. Teknik loop unrolling
mengurangi waktu sinkronisasi menjadi setengahnya. Hasilnya, skema ini memberikan kinerja terbaik dengan efisiensi 89%. Hasil ini memberikan algoritme PA berkinerja tinggi dengan fine-grain parallelism yang dapat digunakan lebih lanjut untuk mengembangkan multiple sequence alignment
(MSA) paralel.
SUMMARY
AURIZA RAHMAD AKBAR. Comparison of Data Partitioning Schema of Pairwise Alignment Algorithm on Shared Memory System. Supervised by HERU SUKOCO and WISNU ANANTA KUSUMA.
The pairwise alignment (PA) algorithm is widely used in bioinformatics to analyze biological sequence. It is the foundation of the multiple sequence alignment (MSA) algorithm to align more than two sequences altogether to find homology between sequences. It is also used in sequence assembly to construct from sequencer’s short reads into the whole gene. Other than that, it is also used for database sequence searching to find the most similar sequence to the one that is given.
The next-generation DNA sequencer technology nowadays can produce a lot of sequence data, up to hundreds of billion base pair (bp) in one run. This big data needs faster processing, so the algorithm needs to be parallelized to speed up the alignment process. Many previous researches have parallelize PA algorithm using various data partitioning schema, but it is unclear which one is the best.
In this research, we parallelized PA algorithm on shared memory system using four different data partitioning schemas: blocked columnwise, rowwise, antidiagonal, and revised blocked columnwise. We tested and revised each schema to obtain the highest performance possible of parallel PA algorithm. The performance was measured on quad-core using DNA sequences with varying length of 1000–16 000 bp.
The antidiagonal schema yields the worst performance with efficiency of 36%. It is understandable because its synchronization time is doubled and its non-linear memory access pattern. The rowwise schema yields better performance than blocked columnwise schema with efficiency of 80% and 75% respectively. These two schemas have relatively same amount of synchronization time. The blocked columnwise schema could perform better, but because of OpenMP limitation this schema yields slightly worse performance than rowwise.
The blocked columnwise schema was then revised using manual loop scheduling and loop unrolling. The manual loop scheduling overcomes the OpenMP limitation and makes the synchronization more efficient. The loop unrolling technique cuts the synchronization time into half. This revised schema yields the best performance with efficiency of 89% on 4 threads. This result provided fine-grain parallelism that can be used further to develop parallel multiple sequence alignment (MSA).
© Hak Cipta Milik IPB, Tahun 2015
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Komputer
pada
Program Studi Ilmu Komputer
PERBANDINGAN SKEMA PARTISI DATA ALGORITME
PAIRWISE ALIGNMENT
PARALEL PADA
SISTEM
SHARED MEMORY
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2015
Judul Tesis : Perbandingan Skema Partisi Data Algoritme Pairwise Alignment
Paralel pada Sistem Shared Memory. Nama : Auriza Rahmad Akbar
NIM : G651114071
Disetujui oleh Komisi Pembimbing
Dr Heru Sukoco, SSi MT Ketua
Dr Wisnu Ananta Kusuma, ST MT Anggota
Diketahui oleh
Ketua Program Studi Ilmu Komputer
Dr Wisnu Ananta Kusuma, ST MT
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah ᤗ atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan, serta shalawat dan salam selalu tercurahkan kepada Nabi Muhammad ᤖ. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Oktober 2013 ini ialah pemrosesan paralel, dengan judul Perbandingan Skema Partisi Data Algoritme Pairwise Alignment Paralel pada Sistem Shared Memory.
Terima kasih penulis ucapkan kepada Bapak Dr Heru Sukoco dan Bapak Dr Wisnu Ananta Kusuma selaku pembimbing, serta Ibu Dr Sri Wahjuni yang telah banyak memberi saran. Ungkapan terima kasih juga disampaikan kepada ayah, ibu, serta seluruh keluarga, atas segala doa dan kasih sayangnya.
Penelitian ini dapat terlaksana atas bantuan dana dari program Kerjasama Kemitraan Penelitian dan Pengembangan Pertanian Nasional (KKP3N) tahun 2013 dari Kementerian Pertanian Indonesia.
Semoga karya ilmiah ini bermanfaat.
Bogor, Maret 2015
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
1 PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 1
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 2
2 TINJAUAN PUSTAKA 3
Pairwise Alignment (PA) 3
Program Paralel 3
Metriks Kinerja Program Paralel 3
3 METODE 4
Implementasi Pairwise Alignment Sekuensial 4
Implementasi Pairwise Alignment Paralel 5
Perbandingan Kinerja 7
Bahan 7
Alat 7
Prosedur Analisis Data 8
4 HASIL DAN PEMBAHASAN 9
Implementasi Pairwise Alignment Sekuensial 9
Implementasi Pairwise Alignment Paralel 11
Perbandingan Kinerja 15
5 SIMPULAN DAN SARAN 17
Simpulan 17
Saran 17
DAFTAR PUSTAKA 18
LAMPIRAN 20
DAFTAR TABEL
1 Waktu eksekusi PA sekuensial 11
2 Waktu eksekusi PA paralel skema blocked columnwise pada 4 thread 12 3 Waktu eksekusi PA paralel skema rowwise pada 4 thread 12 4 Waktu eksekusi PA paralel skema antidiagonal pada 4 thread 13 5 Waktu eksekusi PA paralel skema blocked columnwise dengan
penjadwalan manual dan loop unrolling pada 4 thread 14
DAFTAR GAMBAR
1 Metode penelitian 4
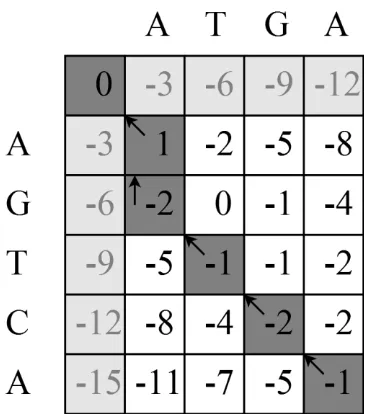
2 Matriks penjajaran sekuens AGTCA dan ATGA 5
3 Blocked columnwise 6
4 Rowwise 6
5 Antidiagonal 6
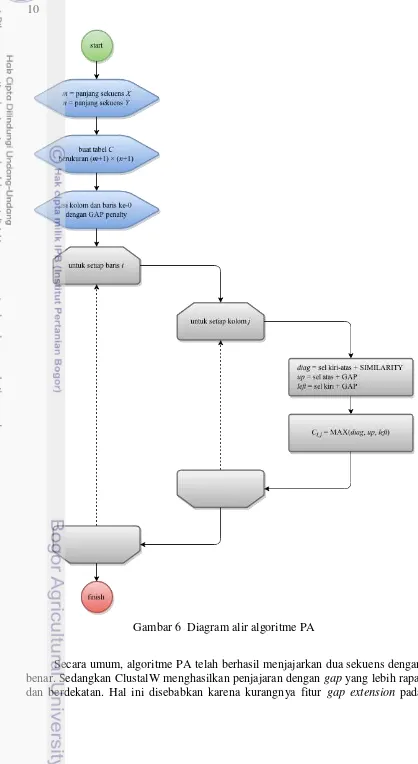
6 Diagram alir algoritme PA 10
7 Perbandingan efisiensi tiap skema pada 4 thread 16 8 Perbandingan speedup skema antidiagonal (AD), blocked columnwise (BC),
rowwise (R), dan perbaikan blocked columnwise (BC2) pada 4 thread 16
DAFTAR LAMPIRAN
1 Implementasi algoritme PA sekuensial 20
1
PENDAHULUAN
Latar Belakang
Algoritme pairwise alignment (PA) dipakai dalam bioinformatika untuk menjajarkan sepasang sekuens DNA atau protein. Hasil penjajaran digunakan untuk menentukan kemiripan antara keduanya (Cohen 2004). PA menggunakan teknik pemrograman dinamis untuk memperoleh hasil penjajaran optimal dengan kompleksitas O(n2), dengan n adalah panjang sekuens (dengan asumsi panjang kedua sekuens sama).
PA merupakan penyusun dasar algoritme multiple sequence alignment
(MSA) yang digunakan untuk menjajarkan lebih dari dua sekuens sekaligus untuk menemukan homologi antar-sekuens. Contoh aplikasi yang menggunakan algoritme ini antara lain ClustalW, T-Coffee, MAFFT, dan Muscle. Selain itu, PA dipakai dalam perakitan sekuens untuk menggabungkan hasil pembacaan dari
sequencer yang pendek-pendek menjadi satu gen utuh. Di samping itu, PA juga dipakai untuk pencarian sekuens pada database, untuk menemukan sekuens-sekuens yang paling mirip dengan sekuens-sekuens yang dicari (Rognes dan Seeberg 2000).
Teknologi next-generation DNA sequencer terkini mampu menghasilkan data sekuens yang banyak, hingga ratusan milyar pasang basa (bp) dalam sekali jalan (Liu et al. 2012). Data yang besar memerlukan pemrosesan yang cepat, sehingga algoritme penjajaran perlu diparalelkan untuk mempercepat proses analisis. Paralelisasi MSA telah banyak dilakukan, seperti pada Praline (Kleinjung
et al. 2002), ClustalW-MPI (Li 2003), MT-ClustalW (Chaichoompu et al. 2006), MAFFT (Katoh dan Toh 2010), dan pada algoritme star (Satra et al. 2014). Paralelisasi PA juga telah dilakukan meskipun tidak sebanyak MSA, seperti ParAlign (Rognes 2001) dan CudaSW (Liu et al. 2013).
Setiap penelitian di atas menggunakan suatu skema partisi data untuk memparalelkan suatu algoritme. Berbagai macam skema partisi telah digunakan, namun belum diketahui skema mana yang terbaik. Penelitian ini akan mencari tahu skema partisi data yang menghasilkan kinerja terbaik pada sistem shared memory.
Perumusan Masalah
Teknik pemrograman dinamis pada PA menyebabkan adanya dependensi data, sehingga skema partisi data sangat mempengaruhi kinerja algoritme. Skema partisi yang telah diterapkan untuk paralelisasi PA antara lain: columnwise
2
Tujuan Penelitian
Tujuan penelitian ini adalah menerapkan algoritme PA paralel dengan berbagai skema partisi data yang berbeda pada sistem shared-memory dan menguji serta memperbaiki masing-masing skema untuk mendapatkan skema yang memberikan kinerja terbaik.
Manfaat Penelitian
Penelitian ini diharapkan dapat menghasilkan algoritme PA paralel dengan
fine-grain parallelism dengan kinerja tinggi yang dapat dimanfaatkan lebih lanjut untuk mengembangkan algoritme MSA paralel pada sistem hibrida shared– distributed memory.
Ruang Lingkup Penelitian
Algoritme PA dikembangkan menggunakan pendekatan penjajaran global untuk menjajarkan sekuens DNA tanpa fitur gap extension. Paralelisasi algoritme PA dilakukan dengan memakai pustaka OpenMP dan bahasa pemrograman C pada sistem shared memory menggunakan teknik multithreading. Skema partisi yang akan diuji yaitu: blocked columnwise, rowwise, antidiagonal, dan blocked columnwise dengan penjadwalan manual dan loop unrolling. Pengujian kinerja algoritme paralel dilakukan pada sistem dengan prosesor yang memiliki 4 core
3
2
TINJAUAN PUSTAKA
Pairwise Alignment (PA)
PA adalah algoritme untuk menjajarkan sepasang sekuens DNA/protein dan menentukan tingkat kemiripan antara keduanya. PA sangat banyak dipakai pada bidang bioinformatika untuk menemukan homologi antar-sekuens, perakitan sekuens (sequence assembly), dan pencarian database sekuens.
Algoritme ini menggunakan teknik pemrograman dinamis, yaitu dengan menggunakan bantuan matriks penjajaran berukuran m × n, dengan m dan n
adalah panjang kedua sekuens. Sekuens pertama menjadi baris matriks dan sekuens kedua menjadi kolomnya. Nilai tiap sel tergantung pada nilai sel sebelah atas, kiri atas, dan kirinya. Kompleksitas algoritme ini adalah O(n2), dengan asumsi panjang kedua sekuens sama.
Program Paralel
Program paralel berjalan pada banyak prosesor secara bersamaan. Sedangkan kebalikannya, program sekuensial hanya dapat berjalan pada satu prosesor saja. Prosesor modern saat ini memiliki banyak core, sehingga dapat dimanfaatkan untuk memparalelkan suatu program agar berjalan lebih cepat.
Menurut memorinya, sistem paralel dapat dibedakan menjadi dua. Sistem paralel yang setiap proses berbagi-pakai memori yang sama disebut dengan sistem
shared memory. Sedangkan sistem paralel yang tiap proses memiliki memori masing-masing disebut sistem distributed memory.
Metriks Kinerja Program Paralel
Speedup
Speedup adalah ukuran berapa kali lipat program paralel lebih cepat dibandingkan dengan program sekuensial. Nilai speedup (S) didapatkan dari hasil bagi antara waktu eksekusi program sekuensial (Ts) dengan waktu eksekusi
Efisiensi adalah ukuran seberapa efisien penggunaan resource yang ada, dalam hal ini adalah prosesor. Efisiensi ideal ialah 100%, akan tetapi sulit untuk mencapainya. Nilai efisiensi (E) didapatkan dari hasil bagi antara speedup (S) dengan jumlah prosesor yang dipakai (p). Berikut Persamaan 2 untuk menghitung efisiensi program paralel.
4
3
METODE
Penelitian ini dibagi menjadi tiga tahapan utama yang dapat dilihat pada Gambar 1 berikut ini.
Gambar 1 Metode penelitian
Implementasi Pairwise Alignment Sekuensial
Langkah pertama adalah menerapkan algoritme PA secara sekuensial dengan menggunakan pendekatan penjajaran global. Algoritme longest common subsequence (LCS) (Cormen et al. 2009) digunakan sebagai dasar untuk pengembangan algoritme PA sekuensial. LCS merupakan algoritme penjajaran global yang sering disebut dengan algoritme Needleman–Wunsch (Needleman dan Wunsch 1970). Skor penjajaran diatur sebagai berikut: jika residu DNA pada kedua sekuens sama maka skor bertambah satu, jika berbeda maka skor dikurangi satu. Gap penalty diset dengan menginisialisasi skor pada baris dan kolom ke-0 secara menurun, yaitu nilai gap penalty dikalikan dengan jaraknya dari titik awal (pojok kiri-atas).
Contoh perhitungan tabel penjajaran dengan nilai gap penalty −3 dapat dilihat pada Gambar 2. Tabel tersebut menjajarkan sekuens AGTCA dan ATGA yang menghasilkan skor penjajaran −1 (nilai pada sel pojok kanan-bawah) dengan hasil penjajaran sebagai berikut.
AGTCA A-TGA
Kebenaran algoritme ini diverifikasi dengan menjajarkan dua sekuens DNA
SYG_YEAST dan SYG_SCHPO yang didapatkan dari benchmark suite penjajaran sekuens DNA BAliBASE (Carroll et al. 2007). Hasil penjajaran dibandingkan dengan hasil dari program ClustalW 2.1 dengan opsi default.
5
Gambar 2 Matriks penjajaran sekuens AGTCA dan ATGA
Implementasi Pairwise Alignment Paralel
Langkah kedua adalah mengembangkan versi paralel dari algoritme ini dan memverifikasi kebenarannya. Paralelisasi dilakukan menggunakan OpenMP, karena jauh lebih mudah implementasinya daripada menggunakan instruksi prosesor langsung (Rognes 2001) maupun menggunakan Pthreads (Chaichoompu
et al. 2006; Katoh dan Toh 2010). OpenMP adalah pustaka untuk memparalelkan program sekuensial menjadi multithreaded pada sistem shared memory (Dagum dan Menon 1998). Empat skema partisi yang diuji antara lain: blocked columnwise, rowwise, antidiagonal, dan blocked columnwise dengan penjadwalan manual dan loop unrolling. Kebenaran algoritme PA paralel diverifikasi dengan membandingkan keluarannya dengan keluaran algoritme PA sekuensial untuk memenuhi konsistensi sekuensial (Lamport 1979). Keluaran yang dibandingkan hanya skor penjajaran terakhirnya saja, yaitu nilai sel pada pojok kanan-bawah matriks penjajaran.
Blocked Columnwise
Skema partisi blocked columnwise membagi tabel penjajaran per blok kolom (Liu dan Schmidt 2003). Thread ke-i (ti) mendapatkan bagian bloknya dari
kolom (i.n/t)+1 hingga (i+1)n/t , dengan n adalah jumlah kolom (panjang sekuens kedua) dan t adalah jumlah thread. Misalnya jika jumlah kolom 9 dan jumlah thread 3, maka t0 mendapatkan bagian satu blok kolom 1–3, t1 mendapatkan bagian satu blok kolom 4–6, dan t2 mendapatkan bagian satu blok kolom 7–9. Ilustrasi skema partisi ini dapat dilihat pada Gambar 3.
Rowwise
Skema partisi rowwise membagi tabel penjajaran per baris (Martins et al. 2001). Thread ke-i (ti) mendapatkan bagian baris ke-(i + x.t + 1), dengan x = 0, 1,
6
maka t0 akan mendapatkan bagian baris ke-1 dan ke-4. Ilustrasi skema partisi ini dapat dilihat pada Gambar 4.
Gambar 3 Blocked columnwise
Gambar 4 Rowwise
7 Antidiagonal
Skema partisi antidiagonal membagi tabel penjajaran per antidiagonal, melintang dari kiri-bawah ke kanan-atas (Li et al. 2012). Thread ke-i (ti)
mendapatkan bagian antidiagonal ke-(i + x.t + 1), dengan x = 0, 1, 2, …, ( (m+n -1/t) -1), m adalah jumlah baris (panjang sekuens pertama), n adalah jumlah kolom (panjang sekuens kedua), dan t adalah jumlah thread. Misalnya jika jumlah baris adalah 6, jumlah kolom 9, dan jumlah thread adalah 3, maka t0 akan mendapatkan bagian antidiagonal ke-1, 4, 7, 10, dan 13. Ilustrasi skema partisi ini dapat dilihat pada Gambar 5.
Blocked Columnwise dengan Penjadwalan Manual dan Loop Unrolling
Skema blocked columnwise di atas belum optimal karena penggunaan konstruk parallelfor pada OpenMP, yang meskipun lebih simpel tetapi kurang fleksibel. Untuk mengatasi kekurangan ini, mekanisme partisi data perlu dilakukan secara manual. Perbaikan berikutnya adalah dengan mengaplikasikan teknik loop unrolling, yaitu menggabungkan beberapa iterasi perulangan menjadi satu. Teknik ini akan mengurangi instruksi prosesor dan meningkatkan lokalitas
cache (Loveman 1977; Sedgewick 1978).
Perbandingan Kinerja
Langkah terakhir adalah menguji kinerja program paralel untuk setiap skema partisi data. Beberapa data sekuens DNA uji dibangkitkan secara acak dengan panjang 1000, 2000, 4000, 8000, dan 16 000 bp sebagai masukan. Batasan panjang sekuens ini terkait dengan kapasitas memori sistem yang digunakan, yaitu hanya 4 GB. Perhitungan tabel penjajaran dicatat waktunya menggunakan fungsi
omp_get_wtime. Kinerja yang diukur adalah speedup dan efisiensi program paralel.
Bahan
Dua sekuens DNA yang digunakan untuk verifikasi kebenaran algoritme PA adalah GlyRS1 Saccharomyces cereviseae (SYG_YEAST) dan GlyRS
Schizosaccharomyces pombe (SYG_SCHPO). Selain itu, beberapa sekuens DNA dengan panjang 1000–16 000 bp dibangkitkan secara acak sebagai sekuens uji untuk menguji kinerja program paralel.
Alat
8
Prosedur Analisis Data
9
4
HASIL DAN PEMBAHASAN
Implementasi Pairwise Alignment Sekuensial
Pseudocode algoritme PA ditunjukkan sebagai sebuah prosedur bernama
PAIRWISE-ALIGN, yang mengambil dua sekuens X dan Y sebagai parameter. Skor
untuk tiap sel diperoleh dengan memilih skor tertinggi di antara tiga kemungkinan arah penjajaran: diagonal, atas, dan kiri. Skor diagonal dihitung dengan bantuan prosedur SIMILARITY: jika kedua residu DNA sama maka skor ditambahkan dengan skor MATCH, jika berbeda maka skor dikurangi dengan skor MISMATCH. Skor atas dan kiri dihitung dengan mengurangkannya dengan skor penalti GAP.
Skor penalti GAP dihitung secara linier, yaitu setiap gap memiliki skor yang
sama tanpa mempedulikan posisinya. Pemilihan nilai skor MATCH dan penalti
10
Gambar 6 Diagram alir algoritme PA
11 algoritme PA ini. Perbandingan hasil penjajaran antara ClustalW dan PA pada sekuens SYG_YEAST dan SYG_SCHPO ditampilkan sebagai berikut. Berdasarkan hasil ini, algoritme PA sederhana telah berhasil diimplementasikan dengan benar dan akan menjadi dasar untuk mengembangkan versi paralel dari algoritme PA.
> ClustalW
Hasil penghitungan waktu eksekusi dapat dilihat pada Tabel 1. Selanjutnya, algoritme ini akan menjadi dasar untuk diparalelkan.
Tabel 1 Waktu eksekusi PA sekuensial Panjang sekuens (bp) Waktu (s)
1000 0.017
2000 0.052
4000 0.203
8000 0.815
16000 3.250
Implementasi Pairwise Alignment Paralel
Blocked Columnwise
Waktu untuk sinkronisasi yang disebabkan oleh dependensi data
antar-thread untuk skema blocked columnwise adalah rendah. Hanya sel kolom pertama pada tiap baris (tanda asterik pada Gambar 3) yang harus mengecek apakah sel di sebelah kirinya telah terisi oleh thread sebelumnya ataukah belum. Jika satu
thread berjalan lebih lambat, maka thread selanjutnya harus menunggu sampai
thread itu menyelesaikan satu baris bagiannya.
Sinkronisasi seharusnya hanya dilakukan sebanyak m × t, akan tetapi karena keterbatasan OpenMP, sinkronisasi masih dilakukan sebanyak m × n. Pengunaan direktif parallel for OpenMP meskipun lebih simpel namun kurang fleksibel, sehingga pengecekan dependensi data terpaksa masih dilakukan pada tiap sel. Hal ini menyebabkan kinerja algoritme paralel kurang efisien. Berikut adalah
pseudocode untuk memparalelkan PA dengan skema ini. 1 for i = 1 to m
2 parallel for j = 1 to n // schedule(static) 3 while Ci,j-1 == null
4 wait
12
6 Ci,j = MAX├ up = Ci-1,j + GAP
7 └ left = Ci,j-1 + GAP
Skema blocked columnwise menghasilkan waktu eksekusi hingga 3.00 kali lebih cepat pada 4 thread. Hasil penghitungan waktu selengkapnya dapat dilihat pada Tabel 2. Efisiensi yang didapat untuk skema ini, yaitu speedup dibagi dengan jumlah thread, adalah 75%.
Tabel 2 Waktu eksekusi PA paralel skema blocked columnwise pada 4 thread
Panjang sekuens (bp) Waktu sekuensial (s) Waktu paralel (s) Speedup
1000 0.017 0.007 2.31
Sinkronisasi untuk skema rowwise adalah tinggi, yaitu dilakukan sebanyak
m × n. Tiap sel harus mengecek apakah sel sebelah atasnya sudah terisi oleh
thread lain atau belum. Meskipun demikian, kinerja algoritme paralel dengan skema ini tidak menjadi lebih buruk. Hal ini disebabkan karena tidak ada perpindahan data antar-thread pada sistem shared memory. Berikut adalah
pseudocode untuk memparalelkan PA dengan skema ini.
1 parallel for i = 1 to m // schedule(static,1)
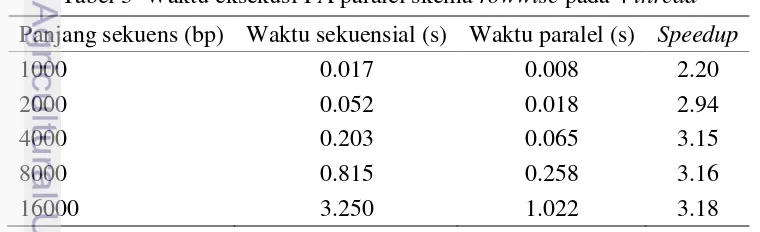
Di luar dugaan, hasil dari skema rowwise lebih baik daripada skema blocked columnwise. Skema ini menghasilkan waktu eksekusi hingga 3.18 kali lebih cepat pada 4 thread. Dengan demikian, efisiensi skema ini adalah 80%. Hasil penghitungan waktu selengkapnya dapat dilihat pada Tabel 3.
Tabel 3 Waktu eksekusi PA paralel skema rowwise pada 4 thread
Panjang sekuens (bp) Waktu sekuensial (s) Waktu paralel (s) Speedup
1000 0.017 0.008 2.20
2000 0.052 0.018 2.94
4000 0.203 0.065 3.15
8000 0.815 0.258 3.16
13
Antidiagonal
Sinkronisasi untuk skema antidiagonal sangat tinggi, dua kali lipat dari skema rowwise, yaitu sebanyak 2 × m × n. Setiap sel harus mengecek apakah sel sebelah kiri dan atasnya telah diisi oleh thread lain atau belum. Terlebih lagi, pola akses memori yang non-linier membuat kinerja algoritme paralel dengan skema ini lebih buruk. Berikut adalah pseudocode untuk memparalelkan PA dengan
Sesuai perkiraan, hasil dari skema antidiagonal adalah yang terburuk di antara skema lainnya. Skema ini menghasilkan waktu eksekusi hingga 1.44 kali lebih cepat pada 4 thread. Dengan demikian, efisiensi skema ini hanya 36%. Hasil penghitungan waktu selengkapnya dapat dilihat pada Tabel 4.
Tabel 4 Waktu eksekusi PA paralel skema antidiagonal pada 4 thread
Panjang sekuens (bp) Waktu sekuensial (s) Waktu paralel (s) Speedup
1000 0.017 0.013 1.28
2000 0.052 0.037 1.43
4000 0.203 0.145 1.40
8000 0.815 0.563 1.45
16000 3.250 2.255 1.44
Blocked Columnwise dengan Penjadwalan Manual dan Loop Unrolling
Penjadwalan manual
14
OpenMP. Dengan demikian, sinkronisasi dapat dilakukan sebanyak m × t, yaitu hanya sekali pada sel pertama tiap baris untuk setiap thread (tanda asterik pada Gambar 3). Penjadwalan manual ini membuat skema blocked columnwise lebih efisien. Berikut adalah potongan awal pseudocode untuk skema blocked columnwise yang telah diperbaiki dengan penjadwalan manual.
1 parallel
dua untuk tiap iterasinya. Sebuah mekanisme pengecekan ditambahkan di tengah perulangan untuk mengecek apakah baris terakhir sudah dicapai atau belum. Hal ini diperlukan untuk mengantisipasi jika jumlah baris (m) tidak habis dibagi dengan faktor unroll. Teknik loop unrolling faktor dua mengurangi jumlah sinkronisasi setengahnya menjadi ½ × m × t. Berikut adalah lanjutan pseudocode
untuk skema blocked columnwise yang telah diperbaiki dengan loop unrolling
faktor dua.
Tabel 5 Waktu eksekusi PA paralel skema blocked columnwise dengan penjadwalan manual dan loop unrolling pada 4 thread
Panjang sekuens (bp) Waktu sekuensial (s) Waktu paralel (s) Speedup
1000 0.017 0.006 2.56
2000 0.052 0.017 3.16
4000 0.203 0.058 3.51
8000 0.815 0.231 3.53
15 Skema blocked columnwise dengan penjadwalan manual dan loop unrolling
dengan faktor dua menghasilkan waktu eksekusi 3.54 kali lebih cepat pada 4
thread. Dengan demikian, efisiensi skema ini adalah 89%. Hasil penghitungan waktu selengkapnya dapat dilihat pada Tabel 5.
Verifikasi Kebenaran Algoritme PA Paralel
Seluruh implementasi algoritme PA paralel di atas telah diuji konsistensi sekuensialnya dengan membandingkan keluaran PA paralel dengan PA sekuensial. Keluaran yang dibandingkan adalah skor akhir penjajaran. Semua skema menghasilkan keluaran yang sama, sehingga implementasi algoritme paralel 100% sesuai dengan algoritme sekuensial. Verifikasi ini penting karena implementasi program paralel multithreaded yang kurang cermat akan menyebabkan keluaran yang tidak konsisten.
Perbandingan Kinerja
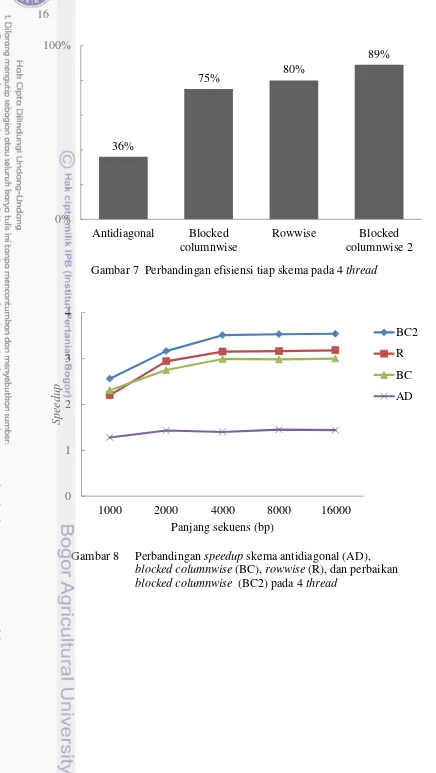
Skema antidiagonal menghasilkan kinerja terburuk dengan efisiensi hanya 36%. Hal ini dapat dimengerti karena waktu sinkronisasi dua kali lipat lebih tinggi (sebanyak 2.m.n) dan pola akses memori yang non-linier yang menyebabkan banyak terjadi cache miss.
Skema rowwise menghasilkan efisiensi 80%, sedangkan skema blocked columnwise menghasilkan efisiensi 75%. Kedua skema ini memiliki waktu sinkronisasi yang hampir sama, yaitu sebanyak m.n. Seharusnya skema blocked columnwise bisa lebih baik, namun karena keterbatasan pustaka OpenMP kinerja skema ini lebih buruk dari rowwise.
Skema blocked columnwise kedua diperbaiki dengan menggunakan penjadwalan manual dan loop unrolling. Penjadwalan manual mengatasi batasan pustaka OpenMP, sehingga skema ini menjadi lebih efisien karena sinkronisasi berkurang drastis menjadi sebanyak m.t. Teknik loop unrolling mengurangi waktu sinkronisasi menjadi setengahnya, yaitu ½.m.t. Hasilnya, skema ini memberikan kinerja terbaik dengan efisiensi 89%.
16
Gambar 7 Perbandingan efisiensi tiap skema pada 4 thread
Gambar 8 Perbandingan speedup skema antidiagonal (AD),
blocked columnwise (BC), rowwise (R), dan perbaikan
blocked columnwise (BC2) pada 4 thread
36%
75% 80%
89%
0% 100%
Antidiagonal Blocked columnwise
Rowwise Blocked
columnwise 2
0 1 2 3 4
1000 2000 4000 8000 16000
Spe
edup
Panjang sekuens (bp)
17
5
SIMPULAN DAN SARAN
Simpulan
Algoritme pairwise alignment (PA) dipakai dalam bioinformatika untuk menjajarkan sepasang sekuens DNA atau protein. Penelitian ini telah menerapkan algoritme PA paralel dengan berbagai skema partisi data yang berbeda pada sistem shared-memory. Skema tersebut antara lain blocked columnwise, rowwise, antidiagonal, dan blocked columnwise dengan penjadwalan manual dan loop unrolling.
Skema antidiagonal menghasilkan kinerja terburuk dengan efisiensi 36% karena waktu sinkronisasi tinggi dan pola akses memori non-linier. Skema
rowwise menghasilkan efisiensi 80%, sedangkan skema blocked columnwise
menghasilkan efisiensi 75%. Kedua skema ini memiliki waktu sinkronisasi yang hampir sama.
Skema blocked columnwise kedua diperbaiki dengan menggunakan penjadwalan manual dan loop unrolling. Penjadwalan manual mengatasi batasan pustaka OpenMP sehingga skema ini menjadi lebih efisien, sedangkan teknik loop unrolling memotong waktu sinkronisasi menjadi setengahnya.Hasilnya, skema ini memberikan kinerja terbaik dengan efisiensi 89%.
Dari sini dapat disimpulkan bahwa pada sistem shared memory, waktu sinkronisasi sangat menentukan baik tidaknya kinerja suatu skema partisi algoritme paralel. Secara umum, skema partisi berbasis blok kolom lebih efisien untuk paralelisasi kasus pemrograman dinamis, khususnya pada algoritme PA.
Saran
Algoritme PA ini hanya dapat menjajarkan sekuens DNA. Untuk dapat menjajarkan sekuens protein, perlu ditambahkan fitur matriks similarity khusus untuk protein, seperti BLOSUM50. Selain itu, fitur gap extension juga perlu ditambahkan pada algoritme PA ini untuk mendapatkan hasil penjajaran yang lebih optimal.
Hasil penelitian ini memberikan algoritme PA berkinerja tinggi dengan fine-grain parallelism yang dapat digunakan lebih lanjut untuk mengembangkan
18
DAFTAR PUSTAKA
Carroll H, Beckstead W, O’Connor T, Ebbert M, Clement M, Snell Q, McClellan D. 2007. DNA reference alignment benchmarks based on tertiary structure of encoded proteins. Bioinformatics. 23(19): 2648–2649.
Chaichoompu K, Kittitornkun S, Tongsima S. 2006. MT-ClustalW: multithreading multiple sequence alignment. Di dalam: Parallel and Distributed Processing Symposium, IEEE International (IPDPS). hlm 8. Cohen J. 2004. Bioinformatics—an introduction for computer scientists. ACM
Computing Surveys (CSUR). 36(2):122–158.
Cormen T, Leiserson C, Rivest R, Stein C. 2009. Introduction to Algorithms. 3rd ed. Cambridge (US): MIT Pr.
Dagum L, Menon R. 1998. OpenMP: an industry standard API for shared-memory programming. Computational Science & Engineering, IEEE. 5(1):46–55.
[DDBJ] DNA Data Bank of Japan. 2014. ClustalW help [internet]. [diperbaharui 2014 Agu 26]. Tersedia pada: http://www.ddbj.nig.ac.jp/search/help/ clustalwhelp-e.html.
Hughey R. 1996. Parallel hardware for sequence comparison and alignment.
Computer Applications in the Biosciences (CABIOS). 12(6):473–479.
Katoh K, Toh H. 2010. Parallelization of the MAFFT multiple sequence alignment program. Bioinformatics. 26(15):1899–1900.
Kleinjung J, Douglas N, Heringa J. 2002. Parallelized multiple alignment.
Bioinformatics. 18(9):1270–1271.
Lamport L. 1979. How to make a multiprocessor computer that correctly executes multiprocess programs. Computers, IEEE Transactions on. 100(9):690–691. Li KB. 2003. ClustalW-MPI: ClustalW analysis using distributed and parallel
computing. Bioinformatics. 19(12):1585–1586.
Li J, Ranka S, Sahni S. 2012. Pairwise sequence alignment for very long sequences on GPUs. Di dalam: Computational Advances in Bio and Medical Sciences, IEEE International Conference on.
Liu L, Li Y, Li S, Hu N, He Y, Pong R, Lin D, Lu L, Law M. 2012. Comparison of next-generation sequencing systems. BioMed Research International. 2012.
Liu Y, Wirawan A, Schmidt B. 2013. CUDASW++ 3.0: accelerating Smith-Waterman protein database search by coupling CPU and GPU SIMD instructions. BMC Bioinformatics. 14(1):117.
Liu W, Schmidt B. 2003. Parallel design pattern for computational biology and scientific computing applications. Di dalam: Cluster Computing, IEEE International Conference on. hlm 456–459.
Loveman DB. 1977. Program improvement by source-to-source transformation.
Journal of the ACM (JACM). 24(1):121–145.
19 Needleman SB, Wunsch CD. 1970. A general method applicable to the search for similarities in the amino acid sequence of two proteins. Journal of Molecular Biology. 48(3):443–453.
Quinn MJ. 2003. Parallel Programming in C with MPI and OpenMP. New York (US): McGraw-Hill. hlm 118–119.
Rognes T. 2001. ParAlign: a parallel sequence alignment algorithm for rapid and sensitive database searches. Nucleic Acids Research. 29(7):1647–1652. Rognes T, Seeberg E. 2000. Six-fold speed-up of Smith–Waterman sequence
database searches using parallel processing on common microprocessors.
Bioinformatics. 16(8):699–706.
Satra R, Kusuma WA, Sukoco H. 2014. Accelerating computation of DNA multiple sequence alignment in distributed environment. Telkomnika Indonesian Journal of Electrical Engineering. 12(12): 8278–8285.
Sedgewick R. 1978. Implementing quicksort programs. Communications of the ACM. 21(10):847–857.
Submitted to:
Akbar AR, Sukoco H, Kusuma WA. 2015 Jun. Comparison of data partitioning schema of parallel pairwise alignment on shared memory system.
20
Lampiran 1 Implementasi algoritme PA sekuensial
#define MATCH +1 #define MISMATCH -1 #define GAP -3
double time;
21 Lampiran 2 Implementasi algoritme PA paralel skema blocked columnwise
int** pairwise_align(const char *X, const char *Y) {
...
time = omp_get_wtime();
#pragma omp parallel private(i,diag,up,left) for (i = 1; i <= m; i++) {
#pragma omp for schedule(static) nowait for (j = 1; j <= n; j++) {
while (C[i][j-1] == NULL);
diag = C[i-1][j-1] + ((X[i-1] == Y[j-1])? MATCH : MISMATCH); up = C[i-1][j] + GAP;
left = C[i][j-1] + GAP;
if (diag >= up && diag >= left) { C[i][j] = diag;
} else if (up >= left) { C[i][j] = up;
} else {
C[i][j] = left; }
} }
time = omp_get_wtime() - time;
22
Lampiran 3 Implementasi algoritme PA paralel skema rowwise
int** pairwise_align(const char *X, const char *Y) {
...
time = omp_get_wtime();
#pragma omp parallel for schedule(static,1) private(j,diag,up,left) for (i = 1; i <= m; i++) {
for (j = 1; j <= n; j++) {
while (C[i-1][j] == NULL);
diag = C[i-1][j-1] + ((X[i-1] == Y[j-1])? MATCH : MISMATCH); up = C[i-1][j] + GAP;
left = C[i][j-1] + GAP;
if (diag >= up && diag >= left) { C[i][j] = diag;
} else if (up >= left) { C[i][j] = up;
} else {
C[i][j] = left; }
} }
time = omp_get_wtime() - time;
23 Lampiran 4 Implementasi algoritme PA paralel skema antidiagonal
int** pairwise_align(const char *X, const char *Y) {
...
time = omp_get_wtime();
24
Lampiran 5 Implementasi algoritme PA paralel perbaikan skema blocked columnwise
int** pairwise_align(const char *X, const char *Y) {
...
time = omp_get_wtime();
25 Lampiran 6 Data DNA untuk verifikasi kebenaran algoritme PA
26