RINGKASAN
MARTIANA KARTIKA DEWI. 2012. Identifikasi Keragaman Gen Calpastatin (CAST|MspI) pada Bangsa Sapi di Indonesia dengan Teknik PCR RFLP.
Skripsi. Departemen Ilmu Produksi dan Teknologi Peternakan. Fakultas Peternakan. Institut Pertanian Bogor.
Pembimbing Utama : Dr. Jakaria, S.Pt. M.Si. Pembimbing Anggota : Prof. Dr. Ir. Muladno, MSA.
Indonesia memiliki sumber daya genetik ternak yang sangat beragam, khususnya ternak sapi yang tersebar di berbagai wilayah di Indonesia. Pelestarian sumber daya genetik ternak sapi di Indonesia menjadi penting untuk dilakukan karena bangsa sapi Indonesia merupakan sumber pangan sekaligus kekayaan alam yang harus dipertahankan. Informasi genetik mengenai bangsa sapi Indonesia yang masih terbatas menjadikan identifikasi keragaman gen calpastatin (CAST|MspI) penting untuk dilakukan. Calpastatin merupakan gen yang berfungsi menghambat degradasi protein sel-sel otot. Gen calpastatin pada mamalia diduga berkaitan dengan sifat pertumbuhan otot dan keempukan daging. Penelitian ini bertujuan untuk mengetahui keragaman gen calpastatin yang terdapat di ekson 1 pada bangsa sapi Indonesia yaitu sapi bali, sapi madura, sapi pesisir, sapi aceh, dan sapi katingan.
Penelitian ini telah dilaksanakan pada September 2011 sampai dengan Februari 2012 di Laboratorium Genetika Molekuler Ternak, Fakultas Peternakan, Institut Peternakan Bogor. Ternak yang digunakan berjumlah 283 ekor yang kemudian diekstraksi hingga didapatkan sampel Deoxyribonucleic Acid (DNA). Ternak yang digunakan terdiri dari sapi bali 100 ekor, sapi madura 68 ekor, sapi pesisir 50 ekor, sapi aceh 15 ekor, dan sapi katingan 50 ekor. Amplifikasi gen calpastatin dilakukan dengan teknik Polymerase Chain Reaction (PCR), sedangkan penentuan genotipenya dilakukan menggunakan enzim restriksi MspI dengan teknik Restriction Fragment Length Polymorphism (RFLP). Analisis data yang dilakukan adalah perhitungan nilai frekuensi alel, frekuensi genotipe, serta nilai heterozigositas. Genotipe yang diperoleh pada penelitian ini adalah genotipe MM dan genotipe MN. Genotipe MM ditunjukkan dengan pita yang terpotong menjadi dua pita dan genotipe MN ditunjukkan dengan munculnya tiga pita. Frekuensi genotipe MM (1,00) diperoleh pada sapi bali, sapi madura, sapi aceh, dan sapi katingan, sedangkan sapi pesisir memiliki frekensi genotipe MM (0,92) dan genotipe MN (0,08). Frekuensi alel M tertinggi ditemukan pada sebagian besar bangsa sapi Indonesia (sapi bali, sapi madura, sapi aceh, dan sapi katingan) yaitu bernilai 1,00 kecuali pada sapi pesisir yang memiliki frekuensi alel M (0,96) dan alel N (0,04). Nilai heterozigositas baik pengamatan (Ho) maupun harapan (He) pada sebagian besar populasi bangsa sapi Indonesia bernilai 0,00 yaitu pada populasi sapi bali, sapi madura, sapi aceh, dan sapi katingan, kecuali pada sapi pesisir yaitu bernilai 0,08. Berdasarkan hasil analisis tersebut, dapat dikatakan bahwa gen calpastatin (CAST|MspI) bersifat monomorfik pada populasi bangsa sapi Indonesia yaitu pada sapi bali, sapi madura, sapi aceh, dan sapi katingan, kecuali pada sapi pesisir yang bersifat polimorfik.
ABSTRACT
Identification of Calpastatin (CAST-MspI) Gene Polymorphisms in Indonesian Cattle Breeds Using PCR-RFLP Technique
Dewi, M.K., Jakaria, and Muladno
Calpastatin (CAST) is a gene that inhibits degeredation of protein muscle. The increasing of calpastatin’s activity causes the increasing of hiperthropy, in the other hands decreasing meat tenderness. The objective of this research was to identify polymorphism of calpastatin (CAST-MspI) genes in Indonesian cattle breeds. This research used 283 Indonesian cattle breeds, they are bali cattle (100), madura cattle (68), pesisir cattle (50), aceh cattle (15), and katingan cattle (50). Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP) technique was used to identify polymorphism of calpastatin genes in Indonesian cattle breeds. The result revealed two genotypes MM and MN and two alleles M and N. Genotype frequence of bali cattle, madura cattle, aceh cattle, and katingan cattle MM (1,00) were evidenced for the CAST|MspI monomorphism, but different from pesisir cattle MM (0,92) and MN (0,08) were the evidenced of polymorphism. Allele frequence of bali cattle, madura cattle, aceh cattle, and katingan cattle is 1,00, while allele frequence M and N of pesisir cattle are 0,96 and 0,04 respectively. The highest M allele frequency was found in the bali, madura, aceh, and katingan cattle population (1,00) and the lowest one was found in pesisir cattle (0,96). The heterozygosity value of bali cattle, madura cattle, aceh cattle, and katingan cattle is 0,00, while the heterozygosity value of pesisir cattle is 0,08. Based on the analysis, Indonesian cattle breeds (bali cattle, madura cattle, aceh cattle, and katingan cattle except pesisir cattle) are monomorphic in calpastatin gene.
PENDAHULUAN
Latar Belakang
Indonesia merupakan negara yang memiliki beragam sumber daya genetik ternak, terutama ternak sapi. Sumber daya genetik ternak sapi Indonesia tersebar di berbagai wilayah di Indonesia. Populasi sapi yang semakin berkembang di Indonesia telah menghasilkan sumber daya genetik yang lebih beragam, yaitu mulai dari sapi asli seperti sapi bali, juga sapi hasil persilangan yang telah menjadi sapi lokal seperti sapi pesisir, sapi aceh, sapi madura, sapi sumba ongole (SO) dan sapi peranakan ongole (PO) (Utoyo 2002; Martojo 2003). Belum adanya perhatian dalam rangka meningkatkan produktivitas sumber daya genetik ternak sapi di Indonesia seperti sapi bali, sapi madura, sapi pesisir, sapi aceh, dan sapi katingan menyebabkan potensi sumber daya genetik tersebut belum dimanfaatkan secara optimum. Kajian mengenai populasi bangsa sapi di Indonesia belum banyak dilakukan, sehingga informasi yang dapat diperoleh mengenai bangsa sapi Indonesia tersebut masih terbatas. Informasi genetik mengenai bangsa sapi Indonesia perlu digali dalam rangka memanfaatkan dan mempertahankan sumber daya genetik ternak Indonesia sekaligus sebagai sumber pangan.
Peningkatan produktivitas ternak dapat dilakukan dengan dua pendekatan, yaitu peningkatan kualitas genetik ternak dan peningkatan manajemen lingkungan. Peningkatan kualitas genetik dapat dilakukan dengan seleksi dan persilangan. Dalam konteks seleksi, seiring dengan perkembangan ilmu dan teknologi dalam bidang bioteknologi molekuler memungkinkan seleksi dilakukan pada tingkat DNA, yaitu dengan cara mengidentifikasi keragaman pada tingkat gen yang mungkin berpengaruh terhadap sifat produksi, pertumbuhan dan reproduksi. Salah satu gen yang diduga kuat berpengaruh terhadap sifat pertumbuhan otot dan keempukan daging adalah gen calpastatin. Barendse (2002) menyatakan bahwa gen calpastatin merupakan gen kandidat kuat yang mempengaruhi keempukan daging.
2 Sumantri et al. (2008). Hal ini menjadikan identifikasi keragaman gen calpastatin penting untuk dilakukan pada bangsa sapi Indonesia.
Identifikasi keragaman gen calpastatin dapat dilakukan salah satunya melalui teknik Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP). Palmer et al. (1998) dalam penelitianya menggunakan sekuen gen calpastatin yang terletak antara ekson IC dan ekson ID untuk diamplifikasi menggunakan teknik PCR-RFLP. Goll et al. (2003) menyatakan bahwa ekspresi gen calpastatin yang terletak pada ekson 1C memiliki kemampuan yang paling efektif untuk menghambat aktivitas m-calpain. Hal tersebut mendasari penelitian ini untuk melakukan amplifikasi gen calpastatin pada ekson 1C dan 1D dengan teknik Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP).
Tujuan
TINJAUAN PUSTAKA
Sumber Daya Genetik Ternak Lokal
Secara terminologi, sumber daya genetik ternak adalah semua yang termasuk dalam spesies, bangsa, dan strain (galur) ternak yang secara ekonomi, ilmiah, dan budaya penting bagi umat manusia baik dalam bentuk makanan maupun produksi (Food Agriculture Organization, 1999). Departemen Pertanian (2006) menyatakan bahwa sumber daya genetik ternak adalah substansi yang terdapat dalam bentuk substansi individu suatu populasi rumpun ternak secara genetik unik, terbentuk dalam proses domestikasi dari masing-masing spesies yang memiliki nilai potensial serta dapat dimanfaatkan dan dikembangkan dengan baik untuk menciptakan rumpun atau galur unggul.
Sumber daya genetik ternak merupakan acuan (building block) bagi pertanian dan pengembangan varietas dan bangsa hewan ternak untuk masa yang akan datang. Keanekaragaman bangsa ternak yang mampu beradaptasi dapat menyelamatkan petani dalam menghadapi iklim yang sulit dan wilayah marjinal. Sumber daya genetik lokal dapat dimanfaatkan dengan biaya (input) minimun dan memegang peranan penting dalam budaya masyarakat pedesaan (Food Agriculture Organization, 2001).
Ternak sapi yang terdapat di Indonesia dapat dikelompokkan menjadi tiga kategori ternak, yaitu (1) ternak asli, (2) ternak yang telah beradaptasi dan (3) ternak impor (Sarbaini, 2004). MacHugh (1996) mengemukakan bahwa sapi yang terdapat di Asia khususnya di Indonesia merupakan sapi yang termasuk dalam spesies Bos bibos dan sapi persilangan (crossbreed) yang berbeda dari sapi domestikasi yang terdapat di Afrika dan Eropa, meskipun diduga bahwa pola penyebarannya berasal dari wilayah India (Bos indicus) yang merupakan tipe sapi berpunuk (Zebu). Hasil domestikasi spesies Bos (Bibos) banteng adalah sapi Bali (Bos sundaicus) atau (Bos javanicus) yang sekarang telah menjadi bangsa ternak asli Indonesia (Directorate Generale of Livestock Services, 2003; Martojo, 2003).
4 sapi pesisir, sapi aceh, sapi madura, sapi Sumba Ongole (SO) dan sapi Peranakan Ongole (PO) (Utoyo 2002; Martojo 2003).
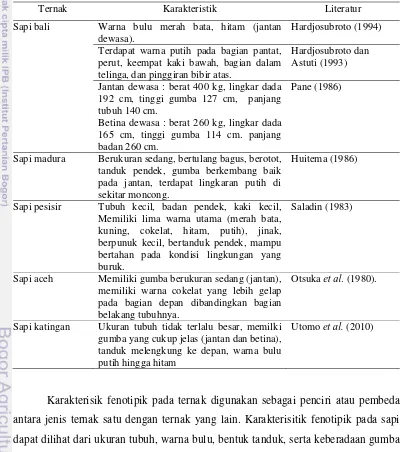
Keragaman ternak sapi di Indonesia mengakibatkan keragaman karakteristik fenotipik pada bangsa sapi Indonesia. Karakteristik fenotipik pada bangsa sapi Indonesia, yaitu sapi bali, sapi madura, sapi pesisir, sapi aceh, dan sapi katingan disajikan pada Tabel 1.
Tabel 1. Karakteristik Fenotipik Bangsa Sapi Indonesia
Ternak Karakteristik Literatur
Betina dewasa : berat 260 kg, lingkar dada 165 cm, tinggi gumba 114 cm. panjang badan 260 cm.
Pane (1986)
Sapi madura Berukuran sedang, bertulang bagus, berotot,
tanduk pendek, gumba berkembang baik pada jantan, terdapat lingkaran putih di berpunuk kecil, bertanduk pendek, mampu bertahan pada kondisi lingkungan yang buruk.
Saladin (1983)
Sapi aceh Memiliki gumba berukuran sedang (jantan),
memiliki warna cokelat yang lebih gelap pada bagian depan dibandingkan bagian belakang tubuhnya.
Otsuka et al. (1980).
Sapi katingan Ukuran tubuh tidak terlalu besar, memilki
gumba yang cukup jelas (jantan dan betina), tanduk melengkung ke depan, warna bulu putih hingga hitam
Utomo et al. (2010)
5 Gambar 1. Karakteristik Fenotipik Bangsa Sapi Indonesia. (a) sapi bali jantan; (b)
sapi bali betina; (c) sapi madura; (d) sapi aceh; (e) sapi pesisir; (f) sapi katingan (Balai Pengkajian Teknologi Pertanian Kalimantan Tengah, 2011).
(a)
(c)
(e)
(d)
6 Marker Assisted Selection (MAS)
Kemajuan teknologi bidang genetika molekuler memungkinkan seleksi dilakukan pada tingkat DNA. Identifikasi keragaman DNA yang terkait dengan sifat kuantitatif dapat dijadikan dasar untuk menerapkan Marker Assisted Selection (MAS) (Montaldo dan Herrera, 1998). Upaya seleksi pada tingkat DNA memiliki keakuratan yang lebih tinggi dibandingkan dengan seleksi secara konvensional yang melihat dari segi fenotipik dan melalui ukuran tubuh (morfometrik).
Penggunaan Marker Assisted Selection (MAS) didasarkan pada gagasan bahwa terdapat gen yang memegang peranan utama dan menjadi sasaran atau target secara spesifik dalam seleksi (Van der Werf, 2000). Gen-gen sifat kuantitatif yang memiliki pengaruh besar merupakan gen-gen yang disebut dengan gen utama (major gene) yang terletak pada lokus sifat kuantitatif (Quantitative Trait Loci).
Marker gen telah banyak digunakan untuk mengidentifikasi beberapa gen yang memiliki pengaruh utama (major gene) terhadap sifat-sifat ekonomis pada ternak, seperti kualitas (keempukan daging) (Barendse et al., 2008). Marker Assisted Selection (MAS) dapat membantu dalam meningkatkan laju peningkatan mutu genetik ternak melalui pemanfaatan informasi molekuler genetik dalam program pemuliaan (breeding programme) (Dekkers dan Hospital, 2002; Dekkers 2004).
Gen Calpastatin (CAST)
7 Gambar 2. Daerah Target Amplifikasi Gen Calpastatin (Palmer et al., 2000)
Takano et al. (1999) menyatakan bahwa struktur gen calpastatin dalam fungsinya untuk menghambat calpain ke dalam empat domain (I-IV) yang terdiri atas tiga daerah, yaitu A, B, dan C. Daerah A dan C mengikat ion kalsium (Ca2+) secara kuat, akan tetapi tidak memiliki pengaruh dalam menghambat aktivitas calpain, berbeda dengan daerah B yang memiliki pengaruh menghambat aktivitas dari calpain.
Calpain merupakan sebuah enzim proteolytic terkait dengan ion kalsium (Ca2+), yang ada dalam dua bentuk yaitu µ-calpain dan m-calpain. µ-calpain merupakan calpain yang memerlukan ion Ca2+ dalam konsentrasi rendah, sedangkan m-calpain meruapakan calpain yang memerlukan Ca2+ dalam konsentrasi tinggi. Calpain berfungsi untuk mendegradasi protein sel-sel otot (myofibril) di dalam jaringan otot (Goll et al.,1992). Koohmaraie (1995) menyatakan bahwa aktivitas calpain dalam jaringan otot postmorfem dapat menyebabkan struktur protein sel otot menjadi lemah. Hal ini berakibat pada kualitas daging menjadi lebih empuk, selain µ-calpain dan m-calpain.
Hasil pemotongan produk PCR dengan enzim MspI menghasilkan dua alel, yaitu alel M dan alel N pada domba dorset. MspI menghasilkan potongan produk 336 pb dan 286 pb (Palmer et al., 1998). Keragaman gen calpastatin pada domba lokal indonesia yang diidentifikasi menggunakan teknik PCR dan RFLP-MspI diperoleh dua genotipe, yaitu genotipe MN dan NN (Sumantri et al., 2008). Shahroudi et al. (2006) dengan menggunakan teknik yang sama pada domba Karakul ditemukan tiga genotipe, yaitu genotipe MM, MN dan NN dengan frekuensi masing-masing 0,61 dan 0,36, dan 0,03.
8 dengan menggunakan teknik PCR-RFLP dengan enzim restriksi BamHI dan EcoRI. Kubiak et al. (2004) dengan teknik PCR-SSCP menggunakan enzim restriksi AluI dan Schenkel et al. (2006) dengan teknik RFLP menggunakan enzim restriksi RsaI memperoleh tiga macam genotipe pada sapi Bos taurus, yaitu genotipe CC, GC, dan GG.
Chung et al. (1999) menemukan keragaman gen calpastatin dengan metode PCR-SSCP. Primer yang didesain dari domain 1cDNA bovine calpastatin berhasil mengamplifikasi gen calpastatin sepanjang 500 pb dan menghasilkan dua alel, yaitu alel A dan alel B. Keragaman gen calpastatin tersebut terkait erat dengan sifat pertumbuhan sapi Angus jantan. Sapi Angus dengan genotipe BB mempunyai bobot badan lebih tinggi dari pada sapi dengan genotipe AA dan AB.
Penciri Genetik Polymerase Chain Reaction-Restriction Fragment Lenght
Polymorphism (PCR-RFLP)
Restriction Fragment lenght Polymorphism (RFLP) merupakan salah satu teknik penciri genetik (genetic marker) yang dikembangkan oleh Botstein et al., (1980) yang digunakan untuk mengetahui adanya keragaman sekuens DNA. Penggunaan teknik RFLP menjadi lebih intensif setelah teknik RFLP dikombinasikan dengan teknologi Polymerase Chain Reaction (PCR) yang digunakan hingga saat ini (Mullis et al. 1986).
PCR merupakan suatu teknik untuk menggandakan jumlah molekul DNA pada ruas-ruas tertentu dan monomer-monomer nukleotida yang dilakukan secara in vitro. Proses ini berjalan dengan bantuan primer dan enzim polymerase. Primer merupakan oligonukleotida spesifik yang menempel pada bagian sampel DNA yang akan diperbanyak (Williams, 2005). Proses yang terjadi dalam mesin PCR meliputi tiga tahap utama yaitu denaturasi (pemisahan untai ganda DNA), annealing (penempelan primer), dan ekstensi (pemanjangan primer) (Muladno, 2002). Enzim polymerase merupakan enzim yang dapat mencetak urutan DNA baru. Hasil dari proses PCR dapat divisualisasikan dengan elektroforesis (Williams, 2005).
9 tidaknya situs restriksi kemudian dapat digunakan untuk mengetahui ada tidaknya mutasi. Analisis RFLP biasa digunakan untuk mendeteksi adanya keragaman pada gen yang berhubungan dengan sifat ekonomis, seperti produksi dan kualitas susu (Sumantri et al., 2007).
Keragaman genetik atau yang disebut dengan polimorfisme terjadi apabila terdapat dua alel atau lebih dalam suatu populasi (lebih dari 1%) (Nei dan Kumar, 2000). Penciri molekuler DNA restriction fragmen length polymorphism (RFLP) memiliki tingkat polimorfisme yang tinggi dan secara luas telah digunakan untuk mendapatkan gambaran populasi genetik dan juga untuk mengidentifikasi gen-gen yang mengkode sifat-sifat penting (Montaldo dan Herrera, 1998). Teknik ini semakin intensif digunakan sebagai penciri genetik karena memiliki beberapa keunggulan diantaranya yaitu perbanyakan DNA secara cepat dengan memakai Polymerase Chain Reaction (PCR) dan polimorfisme fragmennya dilakukan dengan enzim restriksi, sehingga mampu mengidentifikasi genotipe secara jelas (Jakaria et al., 2007). Teknik lain yang dapat digunakan untuk mengidentifikasi keragaman Produk PCR atau DNA amplikon antara lain SSCP, TGGE, DGGE, dan sequencing.
Single Strand Conformation Polymorphism (SSCP)
Polymerase Chain Reaction-Single Strand Conformation Polymorphism (PCR-SSCP) merupakan suatu teknik analisis keragaman DNA yang didasarkan pada asumsi bahwa perubahan yang terjadi pada fragmen DNA akan mempengaruhi bentuk fragmen DNA untai tunggalnya. Hal ini terlihat pada perubahan pola migrasi pada gel poliakrilamida non-denaturasi. Metode SSCP dapat mendeteksi adanya mutasi pada fragmen DNA, akan tetapi tidak dapat memberikan informasi mengenai posisi terjadinya fragmen DNA dan memiliki keterbatasan dalam menentukan jumlah alel (Barosso et al., 1999).
Temperature Gradient Gel Electrophoresis (TGGE) dan Denaturing Gradient Gel Electrophoresis (DGGE)
10 yang memiliki perbedaan konsentrasi bahan untuk menyamakan berat molekul (denaturing) (Liu et al., 2008).
Sequencing
MATERI DAN METODE
Lokasi dan Waktu
Penelitian ini dilaksanakan di Laboratorium Genetika Molekuler, Bagian Pemuliaan dan Genetika Ternak, Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor. Penelitian ini telah dilaksanakan pada September 2011 sampai dengan Februari 2012.
Materi
Ternak
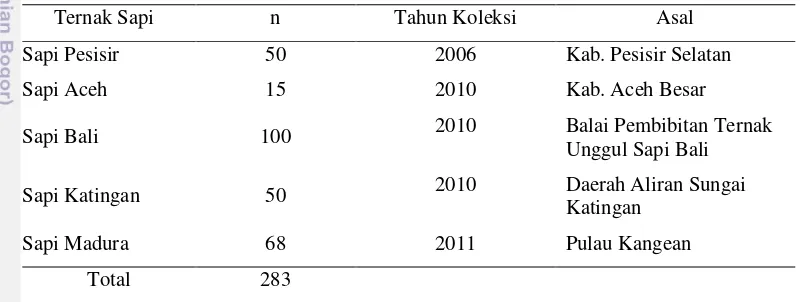
Sampel darah sapi lokal Indonesia yang digunakan sebanyak 283 sampel yang terdiri dari sampel sapi bali, sapi madura, sapi aceh, sapi pesisir, dan sapi katingan. Jumlah sampel dari masing-masing sapi lokal Indonesia yang digunakan dalam penelitian ini disajikan pada Tabel 2.
Tabel 2. Jumlah Sampel Ternak Sapi Indonesia
Ternak Sapi n Tahun Koleksi Asal
Sapi Pesisir 50 2006 Kab. Pesisir Selatan
Sapi Aceh 15 2010 Kab. Aceh Besar
Sapi Bali 100 2010 Balai Pembibitan Ternak
Unggul Sapi Bali
Sapi Katingan 50 2010 Daerah Aliran Sungai
Katingan
Sapi Madura 68 2011 Pulau Kangean
Total 283
Keterangan: n = jumlah individu
Alat dan Bahan
12 Bahan dan alat yang digunakan pada tahap elektroforesis dan genotyping antara lain produk PCR, gel agarose 1,5%, loading dye, buffer, dan enzim restriksi (MspI), elektroforesis tray vertikal, inkubator, serta UV-Transilluminator.
Prosedur
Pengambilan Sampel Darah
Sampel darah diambil dari sapi bali, sapi madura, sapi aceh, sapi katingan dan sapi pesisir melalui vena jugularis dan ditambahkan alkohol absolut selanjutnya dibawa ke Laboratorium Genetika Molekuler Ternak.
Isolasi DNA
Isolasi DNA dilakukan dari sampel darah dengan menggunakan Genomic DNA mini kit Geneaid (Lampiran 1) yang dimodifikasi untuk penggunaan sampel darah yang disimpan dalam alkohol.
Amplifikasi Gen Calpastatin
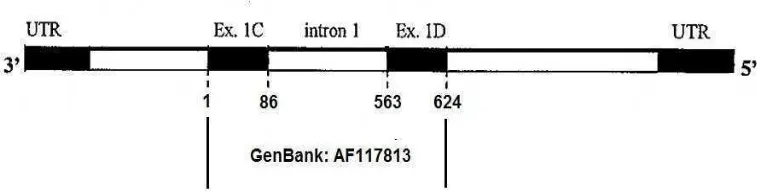
Sekuen primer yang digunakan berdasarkan Palmer et al. (1998), yaitu primer forward 5’ TGGGGCCCAATGACGCCATCGATG 3’ yang terletak di ekson 1C dan primer reverse 5’ GGTGGAGCAGCAC TTCTGATCACC 3’ yang terletak di ekson 1D (Gambar 3), dengan panjang produk PCR 624 pb.
Keterangan:
: Primer forward
: Primer reverse
Ccgghhhhhh : Situs pemotongan
Gambar 3. Tempat Penempelan Primer dan Situs Pemotongan MspI. Nomor akses GenBank: AF117813.
1 3’ tggggcccaa tgatgccatc gatgccttgt catccgactt cacctgcagt tcccctacag
61 ctgatgcaaa gaaaactgag aaagaggtat ggtttttaat gcccttaggg aagcttgtta
121 gaaactacct cccactttaa gacaacaact tttttttaaa cttcattttt cacttcactg
181 cgtcttcatt gctgtgttcg ggctttctct agttggggca agcgaggcct gttctctatt
241 tgcaattttt aggcttctgc agggggctcc tcttgttgct gggc|cggggc tctaggtgca 301 caggcttcat ttgttgtggc tcgagggctc taaaccacag gctcattggt cttggcgcac
361 gggcatggtt accccaatgc atttgggatc tcccctggcc agggagcaaa cctgtttccc
421 ctgcattgca aggcggcctc ttaaccgctg gccaccaggg aagccccaaa atgccaaggc
481 tttttacttc tggttcttac cgtttggttc atatttttcc ttcatctgcc agtcaaacct
541 tcttctgtat tttattttcc agaaatctac agaagaggct ttaaaagctc agtcagctgg
13 Amplifikasi gen calpastatin dilakukan dengan teknik Polymerase Chain Reaction (PCR) menggunakan mesin Applied Biosystem PCR thermalcycler. Amplifikasi gen calpastatin ini menggunakan campuran yang terdiri dari sampel DNA yang telah diekstraksi dari darah, primer (forward dan reverse), taq polymerase, buffer, MgCl2, dan dNTPs. Proses amplifikasi terjadi dalam empat tahap di dalam mesin Applied Biosystem PCR thermalcycler. Kondisi PCR yang digunakan berdasarkan Palmer et al., (1998) yaitu denaturasi pada suhu 95 ºC selama satu menit, penempelan pada suhu 62 ºC selama satu menit, serta pemanjangan molekul DNA yang terjadi pada suhu 72 ºC selama dua menit sebanyak 35 siklus.
Elektroforesis Produk PCR
Elektroforesis produk PCR dilakukan menggunakan 5 µl produk PCR pada gel agarose 1,5% dengan tegangan sebesar 100 volt yang dilakukan selama 60 menit. Pembuatan gel yaitu dengan mencampurkan agarose 0,45 g, 0,5 TBE 30 ml, dan 2,5 µl EtBr. Produk PCR sebanyak 5 µl dicampur dengan loading sebanyak 1 µl. Gel agarose setelah dielektroforesis dilihat panjang pita DNA dengan menggunakan UV-Transilluminator.
Genotyping
14 Gambar 4. Penentuan Genotipe Gen Calpastatin (Palmer et al., 1998). M = marker; 1, 3, 5 = Pemotongan dengan enzim MspI; 2, 4, 6 = Pemotongan dengan enzim NcoI.
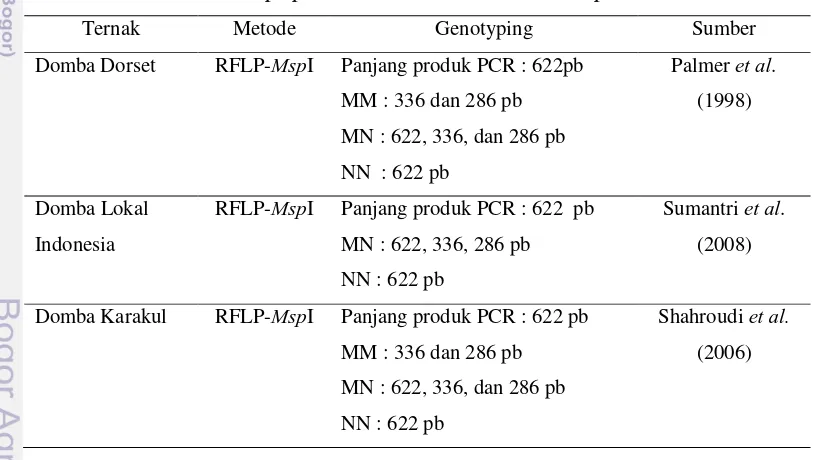
Tabel 3. Penentuan Genotipe pada Gen CAST Menurut Beberapa Penelitian
Ternak Metode Genotyping Sumber
Domba Dorset RFLP-MspI Panjang produk PCR : 622pb
MM : 336 dan 286 pb MN : 622, 336, dan 286 pb NN : 622 pb
Palmer et al.
(1998)
Domba Lokal Indonesia
RFLP-MspI Panjang produk PCR : 622 pb
MN : 622, 336, 286 pb NN : 622 pb
Sumantri et al.
(2008)
Domba Karakul RFLP-MspI Panjang produk PCR : 622 pb
MM : 336 dan 286 pb MN : 622, 336, dan 286 pb NN : 622 pb
Shahroudi et al.
15
Rancangan dan Analisis Data
Keragaman gen calpastatin setiap bangsa sapi lokal Indonesia dianalisis menggunakan pendekatan nilai frekuensi alel, frekuensi genotipe, dan nilai heterozigositas.
Frekuensi alel gen calpastatin dihitung berdasarkan rumus (Nei dan Kumar, 2000) :
Xi =
Frekuensi genotipe dihitung berdasarkan rumus (Nei dan Kumar, 2000) :
Xii =
Keterangan :
xii = frekuensi genotipe ke-ii xi = frekuensi alel ke-i
nii = jumlah individu bergenotipe ii nij = jumlah individu bergenotipe ij N = jumlah individu sampel
Nilai heterozigositas pengamatan (Ho) dihitung menggunakan rumus (Weir, 1996) :
Keterangan :
Ho = heterozigositas pengamatan (populasi) nij = jumlah individu heterozigot
N = jumlah individu yang diamati
Nilai heterozigositas harapan (He) dihitung dengan menggunakan rumus (Weir, 1996) :
Keterangan :
HASIL DAN PEMBAHASAN
Amplifikasi Gen Calpastatin (CAST|MspI)
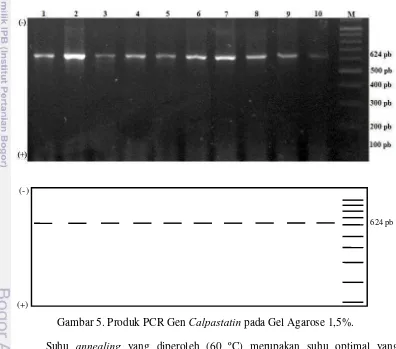
Amplifikasi fragmen gen calpastatin (CAST|MspI) pada setiap bangsa sapi dilakukan dengan menggunakan mesin thermal cycler (AB Bio System) pada suhu annealing 60 ºC selama 45 detik dengan panjang produk PCR 624 pasang basa (pb) (Gambar 5). Keberhasilan amplifikasi Gen Calpastatin (CAST|MspI) pada bangsa sapi Indonesia adalah 100%. Seluruh sampel yang digunakan yaitu sebanyak 283 sampel DNA yang berasal dari sapi bali, sapi madura, sapi pesisir, sapi aceh, dan sapi katingan berhasil diamplifikasi pada penelitian ini.
(-)
624 pb
(+)
Gambar 5. Produk PCR Gen Calpastatin pada Gel Agarose 1,5%.
17 taurus) dengan panjang produk 624 pb dengan suhu annealing 62 ºC selama 45 detik (Kubiak et al., 2004), sedangkan pada domba lokal di Indonesia gen calpastatin berhasil diamplifikasi dengan suhu annealing 48 ºC (Sumantri et al., 2008).
Keberhasilan amplifikasi sangat ditentukan oleh kondisi penempelan primer pada DNA genom (gen target), disamping faktor-faktor lainnya, seperti bahan pereaksi PCR dan kondisi mesin PCR (thermalcycler). Kisaran temperatur penempelan (annealing) adalah antara 36 ºC sampai dengan 72 ºC, namun suhu yang
biasa digunakan antara 50-60 ºC (Muladno, 2002).
Identifikasi Genotipe Gen Calpastatin
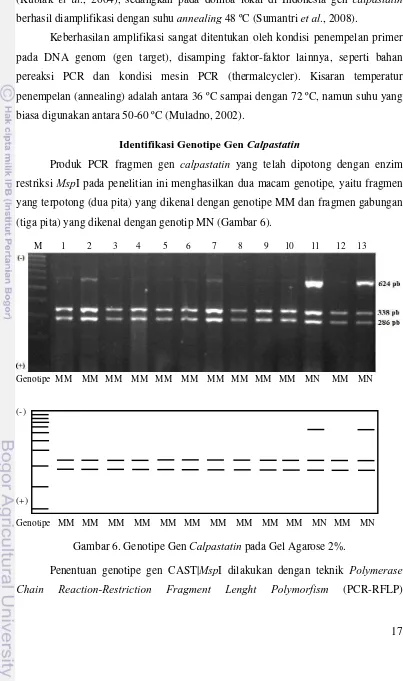
Produk PCR fragmen gen calpastatin yang telah dipotong dengan enzim restriksi MspI pada penelitian ini menghasilkan dua macam genotipe, yaitu fragmen yang terpotong (dua pita) yang dikenal dengan genotipe MM dan fragmen gabungan (tiga pita) yang dikenal dengan genotip MN (Gambar 6).
M 1 2 3 4 5 6 7 8 9 10 11 12 13
Genotipe MM MM MM MM MM MM MM MM MM MM MN MM MN
(-)
(+)
Genotipe MM MM MM MM MM MM MM MM MM MM MN MM MN
Gambar 6. Genotipe Gen Calpastatin pada Gel Agarose 2%.
18 menggunakan MspI sebagai enzim pemotong. Identifikasi keragaman gen CAST|MspI dilakukan pada produk PCR dengan panjang 624 pb. Hasil analisis keragaman gen calpastatin pada bangsa sapi Indonesia, yaitu sapi bali, sapi madura, sapi aceh, dan sapi katingan diperoleh dua genotipe, yaitu genotipe MM dan genotipe MN (Tabel 4). Genotipe MM ditunjukkan dengan panjang pita 338 dan 286 pb, genotipe MN dengan panjang pita 624, 338, dan 286 bp. Enzim MspI mengenali situs pemotongan C|CGG. Keragaman gen CAST|MspI disebabkan oleh adanya mutasi pada posisi basa ke-284 nomor akses GenBank AF117813. Subtitusi basa (transisi) basa Guanin (CCGG) – Adenin (CCGA).
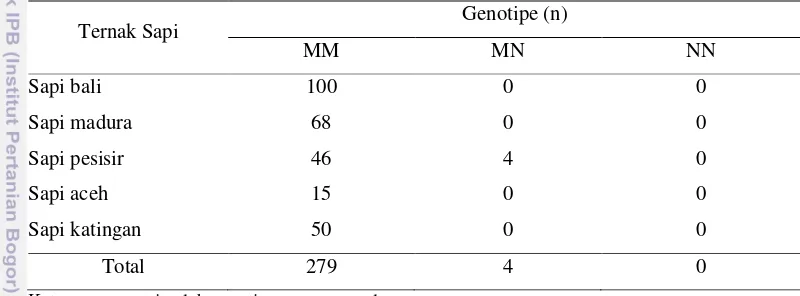
Tabel 4. Hasil Identifikasi Genotipe Gen Calpastatin pada Bangsa Sapi Indonesia
Ternak Sapi Genotipe (n)
MM MN NN
Sapi bali 100 0 0
Sapi madura 68 0 0
Sapi pesisir 46 4 0
Sapi aceh 15 0 0
Sapi katingan 50 0 0
Total 279 4 0
Keterangan: n = jumlah genotipe yang muncul
Hasil identifikasi genotipe gen calpastatin pada bangsa sapi Indonesia dengan teknik PCR-RFLP menunjukkan bahwa pada bangsa sapi Indonesia yaitu sapi bali, sapi madura, sapi pesisir, sapi aceh, dan sapi katingan tidak ditemukan genotipe NN yang ditunjukkan dengan tiga pita pada panjang 286, 338, dan 624 pb. Hasil identifikasi genotipe berbeda dengan Kubiak et al. (2004) yang melakukan amplifikasi pada sapi potong Bos taurus yang menghasilkan tiga genotipe yaitu genotipe GC, genotipe CC, dan genotipe GG. Perbedaan genotipe terjadi akibat perbedaan enzim pemotong yang digunakan, sehingga titik potong pun berbeda yang menyebabkan perbedaan dalam penentuan genotipe.
19 gen CAST|MspI sepanjang 624 pb dipotong dan menghasilkan dua alel, yaitu alel M dan N. Alel M mempunyai mempunyai nukleotida G pada posisi basa ke-284, sedangkan alel N mempunyai nukleotida A pada posisi basa yang sama. Terjadinya transisi basa G – A menyebabkan terjadinya perbedaan sekuen nukleotida dari gen CAST|MspI.
Mutasi transisi disebabkan oleh adanya subtitusi antara Adenin dengan Guanin (Purin) atau basa Sitosin dengan Timin (Pirimidin) (Paolella, 1998). Titik mutasi gen CAST|MspI diketahui berada pada daerah intron 1 yang merupakam daerah non-coding karena pada saat transkripsi bagian ini akan hilang (splicing). Proses transkripsi merupakan proses pengkodean dari kodon-kodon yang akan ditranslasikan menjadi asam-asam amino. Mutasi yang terjadi pada bagian yang tidak ditranskripsikan (intron) akan mengakibatkan silent mutation, yaitu mutasi yang tidak menyebabkan perubahan fenotipik (Paolella, 1998).
Keragaman Gen Calpastatin pada Bangsa Sapi Indonesia
Nilai frekuensi genotipe dan frekuensi alel gen CAST|MspI bangsa sapi Indonesia dapat dilihat pada Tabel 5 berikut.
Tabel 5. Nilai Frekuensi Genotipe dan Frekuensi Alel Gen CAST|MspI pada Bangsa Sapi Indonesia
Ternak Sapi n Frekuensi Genotipe Frekuensi Alel
MM MN M N
Sapi bali 100 1,00 0,00 1,00 0,00
Sapi madura 68 1,00 0,00 1,00 0,00
Sapi pesisir 50 0,92 0,08 0,96 0,04
Sapi aceh 15 1,00 0,00 1,00 0,00
Sapi katingan 50 1,00 0,00 1,00 0,00
Total 283 0,99 0,01 0,99 0,01
Keterangan: n = jumlah individu
20 pada bangsa sapi indonesia adalah alel M. Frekuensi alel pada sapi bali, sapi madura, sapi aceh, dan sapi katingan adalah 1,00, sedangkan sapi pesisir memiliki frekuensi alel M sebesar 0,96 dan alel N sebesar 0,04.
Keragaman genetik terjadi apabila terdapat dua alel atau lebih dalam suatu populasi (lebih dari 1%) (Nei dan Kumar, 2000). Hasil penelitian ini menunjukkan bahwa gen CAST|MspI bersifat monomorfik atau tidak beragam yaitu pada sapi bali, sapi madura, sapi aceh, dan sapi katingan, kecuali pada populasi sapi pesisir yang bersifat polimorfik. Populasi bangsa sapi indonesia diketahui mempunyai frekuensi alel M lebih tinggi dibandingkan frekuensi N pada semua populasi yang dianalisis. Frekuensi alel adalah frekuensi relatif dari suatu alel dalam populasi (Nei dan Kumar, 2000).
Seleksi adalah suatu tindakan untuk membiarkan ternak-ternak tertentu bereproduksi, sedangkan ternak lainnya tidak diberi kesempatan untuk bereproduksi (Noor, 2008). Seleksi dan manajemen yang dilakukan oleh peternak diduga merupakan penyebab tingginya frekuensi alel M pada populasi bangsa sapi Indonesia. Seleksi yang dilakukan adalah dengan mempertahankan individu-individu yang memiliki alel M dan tidak mempertahankan individu yang memiliki alel N. Hal tersebut berdampak pada manajemen perkawinan yang dilakukan, yaitu peternak lebih banyak melakukan perkawinan terhadap individu-individu yang memiliki alel M dibandingkan dengan individu-individu yang memikiki alel N, sehingga menghasilkan keturunan yang tentunya juga memiliki alel M yang diwariskan dari tetuanya.
Frekuensi alel dan genotipe gen calpastatin (CAST|MspI) pada bangsa sapi Indonesia memilki keragaman genetik yang cenderung rendah. Hal ini disebabkan karena ternak-ternak tersebut telah beradaptasi dengan lingkungan tropis di Indonesia sehingga pengaruh lingkungan berperan terhadap sifat produksinya. Proses adaptasi tersebut merupakan proses evolusi dan seleksi pada spesies ternak domestik. Evolusi terhadap adaptabilitas pada lingkungan tropis memberikan penyesuaian terhadap resistensi atau toleransi penyakit, stres panas, stres nutrisi dan toleransi obat-obatan (Newman dan Coffey, 1999).
21 ternak hasil seleksi alam rendah terhadap sifat produksinya dikarenakan apa yang dibutuhkan oleh hewan tersebut terbatas hanya untuk mempertahankan hidupnya (Pane, 1986). Fenomena kelenturan fenotipik sebagai hasil seleksi alam dan menyebabkan genotipe pada bangsa sapi Indonesia memiliki keragaman rendah. Noor (2002) menyatakan bahwa kemampuan suatu individu/genotipe untuk menampilkan lebih dari satu bentuk morfologi, status fisiologi dan tingkah laku sebagai respon terhadap perubahan lingkungan disebut sebagai kelenturan fenotipik.
Pewarisan sifat dari tetua yang menyumbangkan alel yang sama (homozigot) kepada keturunannya secara turun temurun mengakibatkan frekuensi gen individu-individu pada populasi bersifat monomorfik. Silang dalam merupakan suatu bentuk isolasi secara genetik. Silang dalam terjadi akibat adanya keterbatasan pilihan dalam proses perkawinan, akan tetapi silang dalam tidak akan merubah frekuensi gen awal pada proses silang dalam dimulai. Apabila terjadi perubahan frekuensi gen, maka perubahan itu disebabkan oleh adanya seleksi, mutasi, dan pengaruh sampel acak (Noor, 2008). Hal ini berkaitan dengan isolasi yang dilakukan untuk menjaga kemurnian Sapi Bali untuk kepentingan konservasi yang dilakukan oleh BPTU Sapi Bali di Pulau Bali.
Faktor lain yang diduga memiliki pengaruh terhadap rendahnya keragaman gen calpastatin (CAST|MspI) pada bangsa sapi Indonesia adalah keterbatasan sampel yang digunakan dalam penelitian ini. Pengambilan sampel dengan jumlah ternak yang terbatas mengakibatkan frekuensi gen yang diidentifikasi kurang representatif atau kurang mewakili individu-individu populasi bangsa sapi Indonesia, seperti yang terjadi padamn sampel sapi aceh yang digunakan dalam penelitian ini. Jumlah individu yang digunakan untuk sampel DNA sapi aceh hanya berjumlah 15 ekor ternak sapi.
22
Pendugaan Nilai Heterozigositas
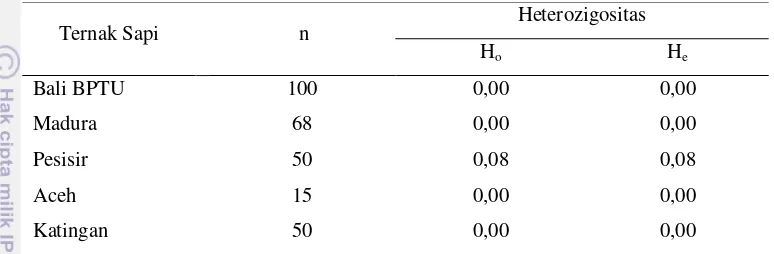
Pendugaan nilai heterozigositas pengamatan (Ho) dan nilai heterozigositas harapan (He) gen CAST-MspI pada bangsa sapi Indonesia tersaji pada Tabel 6. Tabel 6. Nilai Heterozigositas Pengamatan (Ho) dan Heterozigositas Harapan (He)
gen CAST-MspI pada Bangsa Sapi Indonesia
Ternak Sapi n Heterozigositas
Ho He
Bali BPTU 100 0,00 0,00
Madura 68 0,00 0,00
Pesisir 50 0,08 0,08
Aceh 15 0,00 0,00
Katingan 50 0,00 0,00
Keterangan: n = jumlah individu
Pendugaan nilai heterozigositas memiliki arti penting untuk diketahui, yaitu untuk mendapatkan gambaran variabilitas genetik pada suatu populasi (Marson et al., 2005), selain itu nilai heterozigositas digunakan untuk mengetahui tingkat polimorfisme suatu alel dan prospek populasi di masa yang akan datang (Falconer dan Mackay, 1996). Tabel 6 menunjukkan bahwa hasil analisis nilai heterozigositas pengamatan (Ho) dan nilai pengamatan harapan (He) tidak mengindikasikan adanya perbedaan. Hal tersebut mengindikasikan bahwa frekuensi genotipe dari populasi yang dianalisis dalam keadaan seimbang.
Tambasco et al. (2003) menyatakan bahwa jika nilai heterozigositas pengamatan (Ho) lebih rendah dari heterozigositas harapan (He) dapat mengindikasikan adanya derajat endogami (perkawinan dalam kelompok) sebagai akibat dari proses seleksi yang intensif. Moioli et al. (2004) menyatakan bahwa secara umum nilai heterozigositas, khususnya nilai heterozigositas harapan (He) merupakan indikator yang baik sebagai penciri genetik yang dapat menjelaskan keragaman genetik pada suatu populasi ternak domestik.
23 dengan kebutuhannya dalam mempertahankan hidup. Ternak memberikan respon terhadap stres dengan cara mengatur keadaan fisiologi atau morfologi sebagai upaya meredam pengaruh stres untuk mempertahankan fungsi normal tubuh agar ternak dapat bertahan dan bereproduksi dalam keadaan tersebut.
KESIMPULAN DAN SARAN
Kesimpulan
Gen calpastatin (CAST|MspI) pada bangsa sapi di Indonesia memiliki alel M dengan genotipe MM, kecuali pada sapi pesisir yang memiliki dua jenis alel yaitu M dan N dengan genotipe MM dan MN. Genotipe MM dan alel M memiliki frekuensi kemunculan paling besar pada setiap populasi. Hasil indentifikasi gen CAST|MspI pada bangsa sapi Indonesia menyatakan bahwa gen calpastatin bersifat monomorfik pada populasi sapi bali, sapi madura, sapi aceh, dan sapi katingan, kecuali pada sapi pesisir yang bersifat polimorfik.
Saran
IDENTIFIKASI KERAGAMAN GEN CALPASTATIN
(CAST|MspI) PADA BANGSA SAPI DI INDONESIA
DENGAN TEKNIK PCR-RFLP
SKRIPSI
MARTIANA KARTIKA DEWI
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
IDENTIFIKASI KERAGAMAN GEN CALPASTATIN
(CAST|MspI) PADA BANGSA SAPI DI INDONESIA
DENGAN TEKNIK PCR-RFLP
SKRIPSI
MARTIANA KARTIKA DEWI
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
RINGKASAN
MARTIANA KARTIKA DEWI. 2012. Identifikasi Keragaman Gen Calpastatin (CAST|MspI) pada Bangsa Sapi di Indonesia dengan Teknik PCR RFLP.
Skripsi. Departemen Ilmu Produksi dan Teknologi Peternakan. Fakultas Peternakan. Institut Pertanian Bogor.
Pembimbing Utama : Dr. Jakaria, S.Pt. M.Si. Pembimbing Anggota : Prof. Dr. Ir. Muladno, MSA.
Indonesia memiliki sumber daya genetik ternak yang sangat beragam, khususnya ternak sapi yang tersebar di berbagai wilayah di Indonesia. Pelestarian sumber daya genetik ternak sapi di Indonesia menjadi penting untuk dilakukan karena bangsa sapi Indonesia merupakan sumber pangan sekaligus kekayaan alam yang harus dipertahankan. Informasi genetik mengenai bangsa sapi Indonesia yang masih terbatas menjadikan identifikasi keragaman gen calpastatin (CAST|MspI) penting untuk dilakukan. Calpastatin merupakan gen yang berfungsi menghambat degradasi protein sel-sel otot. Gen calpastatin pada mamalia diduga berkaitan dengan sifat pertumbuhan otot dan keempukan daging. Penelitian ini bertujuan untuk mengetahui keragaman gen calpastatin yang terdapat di ekson 1 pada bangsa sapi Indonesia yaitu sapi bali, sapi madura, sapi pesisir, sapi aceh, dan sapi katingan.
Penelitian ini telah dilaksanakan pada September 2011 sampai dengan Februari 2012 di Laboratorium Genetika Molekuler Ternak, Fakultas Peternakan, Institut Peternakan Bogor. Ternak yang digunakan berjumlah 283 ekor yang kemudian diekstraksi hingga didapatkan sampel Deoxyribonucleic Acid (DNA). Ternak yang digunakan terdiri dari sapi bali 100 ekor, sapi madura 68 ekor, sapi pesisir 50 ekor, sapi aceh 15 ekor, dan sapi katingan 50 ekor. Amplifikasi gen calpastatin dilakukan dengan teknik Polymerase Chain Reaction (PCR), sedangkan penentuan genotipenya dilakukan menggunakan enzim restriksi MspI dengan teknik Restriction Fragment Length Polymorphism (RFLP). Analisis data yang dilakukan adalah perhitungan nilai frekuensi alel, frekuensi genotipe, serta nilai heterozigositas. Genotipe yang diperoleh pada penelitian ini adalah genotipe MM dan genotipe MN. Genotipe MM ditunjukkan dengan pita yang terpotong menjadi dua pita dan genotipe MN ditunjukkan dengan munculnya tiga pita. Frekuensi genotipe MM (1,00) diperoleh pada sapi bali, sapi madura, sapi aceh, dan sapi katingan, sedangkan sapi pesisir memiliki frekensi genotipe MM (0,92) dan genotipe MN (0,08). Frekuensi alel M tertinggi ditemukan pada sebagian besar bangsa sapi Indonesia (sapi bali, sapi madura, sapi aceh, dan sapi katingan) yaitu bernilai 1,00 kecuali pada sapi pesisir yang memiliki frekuensi alel M (0,96) dan alel N (0,04). Nilai heterozigositas baik pengamatan (Ho) maupun harapan (He) pada sebagian besar populasi bangsa sapi Indonesia bernilai 0,00 yaitu pada populasi sapi bali, sapi madura, sapi aceh, dan sapi katingan, kecuali pada sapi pesisir yaitu bernilai 0,08. Berdasarkan hasil analisis tersebut, dapat dikatakan bahwa gen calpastatin (CAST|MspI) bersifat monomorfik pada populasi bangsa sapi Indonesia yaitu pada sapi bali, sapi madura, sapi aceh, dan sapi katingan, kecuali pada sapi pesisir yang bersifat polimorfik.
ABSTRACT
Identification of Calpastatin (CAST-MspI) Gene Polymorphisms in Indonesian Cattle Breeds Using PCR-RFLP Technique
Dewi, M.K., Jakaria, and Muladno
Calpastatin (CAST) is a gene that inhibits degeredation of protein muscle. The increasing of calpastatin’s activity causes the increasing of hiperthropy, in the other hands decreasing meat tenderness. The objective of this research was to identify polymorphism of calpastatin (CAST-MspI) genes in Indonesian cattle breeds. This research used 283 Indonesian cattle breeds, they are bali cattle (100), madura cattle (68), pesisir cattle (50), aceh cattle (15), and katingan cattle (50). Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP) technique was used to identify polymorphism of calpastatin genes in Indonesian cattle breeds. The result revealed two genotypes MM and MN and two alleles M and N. Genotype frequence of bali cattle, madura cattle, aceh cattle, and katingan cattle MM (1,00) were evidenced for the CAST|MspI monomorphism, but different from pesisir cattle MM (0,92) and MN (0,08) were the evidenced of polymorphism. Allele frequence of bali cattle, madura cattle, aceh cattle, and katingan cattle is 1,00, while allele frequence M and N of pesisir cattle are 0,96 and 0,04 respectively. The highest M allele frequency was found in the bali, madura, aceh, and katingan cattle population (1,00) and the lowest one was found in pesisir cattle (0,96). The heterozygosity value of bali cattle, madura cattle, aceh cattle, and katingan cattle is 0,00, while the heterozygosity value of pesisir cattle is 0,08. Based on the analysis, Indonesian cattle breeds (bali cattle, madura cattle, aceh cattle, and katingan cattle except pesisir cattle) are monomorphic in calpastatin gene.
IDENTIFIKASI KERAGAMAN GEN CALPASTATIN
(CAST|MspI) PADA BANGSA SAPI DI INDONESIA
DENGAN TEKNIK PCR-RFLP
MARTIANA KARTIKA DEWI
D14080113
Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan pada
Fakultas Peternakan Institut Pertanian Bogor
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
Judul : Identifikasi Keragaman Gen Calpastatin (CAST|MspI) pada Bangsa Sapi di Indonesia dengan Teknik PCR RFLP
Nama : Martiana Kartika Dewi
NIM : D14080113
Menyetujui,
Mengetahui: Ketua Departemen
Ilmu Produksi dan Teknologi Peternakan
(Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc.) NIP. 19591212 198603 1 004
Tanggal Ujian: 26 Juni 2012 Tanggal Lulus: Pembimbing Utama
(Dr. Jakaria, S.Pt., M.Si.) NIP. 19660105 199303 1 001
Pembimbing Anggota
RIWAYAT HIDUP
Penulis dilahirkan di Banjarnegara pada tanggal 20 Maret 1990. Penulis adalah anak ke-dua dari tiga bersaudara pasangan Bapak Imam Gunadi dan Ibu Setiarti. Pendidikan dasar penulis diselesaikan pada tahun 2002 di SD Negeri 1 Kutabanjar, Banjarnegara. Pendidikan lanjutan menengah pertama diselesaikan pada tahun 2005 di SMP Negeri 1 Banjarnegara, dan pendidikan lanjutan tingkat atas diselesaikan pada tahun 2008 di SMA Negeri 1 Banjarnegara, Jawa Tengah. Penulis diterima sebagai mahasiswa IPB di Fakultas Peternakan IPB melalui jalur Undangan Seleksi Masuk IPB (USMI) pada tahun 2008, dan kemudian ditempatkan di Departemen Ilmu Produksi dan Teknologi Peternakan IPB pada tahun 2009.
Selama mengikuti pendidikan sebagai mahasiswa Institut Pertanian Bogor penulis aktif di berbagai organisasi, diantaranya UKM Seni Sunda Gentra Kaheman periode 2008-2010, Ikatan Mahasiswa Banyumas (IKAMAHAMAS), BEM Fakultas Peternakan IPB periode 2009-2010 dan 2010-2011, Majalah Pangan Emulsi Generasi 4 periode 2010-2011. Penulis juga aktif sebagai anggota Animal Breeding and Genetics Community (ABGSC), Himpunan Keprofesian Departemen Ilmu Produksi dan Teknologi Peternakan (HIMAPROTER), Tim Basket Fakultas Peternakan, dan Komunitas Seni Fakultas Peternakan dalam bidang seni tari dan perkusi.
KATA PENGANTAR
Puji dan syukur senantiasa dipanjatkan kepada Allah SWT, atas berkat dan rahmat-Nya skripsi ini dapat diselesaikan dengan baik. Skripsi yang berjudul
Identifikasi Keragaman Gen Calpastatin (CAST|MspI) pada Bangsa Sapi
Indonesia dengan Teknik PCR RFLP merupakan salah satu syarat untuk memperoleh gelar sarjana pada Fakultas Peternakan Insitut Pertanian Bogor.
Populasi sapi yang semakin berkembang di Indonesia telah menghasilkan sumber daya genetik yang lebih beragam. Keberagaman sumber daya genetik bangsa sapi Indonesia tersebut belum dimanfaatkan secara optimal akibat minimnya informasi mengenai sumber daya genetik bangsa sapi Indonesia. Identifikasi keragaman susunan nukleotida dalam gen yang berpengaruh terhadap sifat produksi, pertumbuhan dan reproduksi dapat digunakan sebagai dasar seleksi untuk meningkatkan produktivitas bangsa sapi Indonesia. Salah satu teknik yang dapat digunakan adalah teknik Polymerase Chain Reaction Restriction Fragment Length Polymorphism (PCR-RFLP). Gen calpastatin pada mamalia diduga berkaitan dengan sifat pertumbuhan otot dan keempukan daging. Sifat produksi dan kualitas karkas pada sapi lokal terkait dengan aktivitas calpain yang dipengaruhi oleh aktivitas gen calpastatin. Hal tersebut mendasari pentingnya dilakukannya identifikasi keragaman gen calpastatin pada bangsa sapi Indonesia yang dilakukan pada penelitian ini.
Manfaat penelitian ini adalah untuk meberikan informasi mengenai keragaman gen calpastatin pada bangsa sapi Indonesia. Informasi keragaman ini selanjutnya diharapkan menjadi dasar seleksi untuk meningkatkan sifat pertumbuhan dan kualitas karkas sapi lokal Indonesia. Karya kecil ini diharapkan bermanfaat dan memberikan kontribusi bagi kemajuan dunia peternakan khususnya di Indonesia.
DAFTAR ISI Penciri Genetik Polymerase Chain Reaction-Restriction Fragment Lenght Polymorphism (PCR-RFLP)………...
HASIL DAN PEMBAHASAN ………...… 16 Amplifikasi Gen Calpastatin………... Identifikasi Genotipe Gen Calpastatin………... Keragaman Gen Calpastatin pada Bangsa Sapi Indonesia …………. Pendugaan Nilai Heterozigositas………...
16 17 19 22
KESIMPULAN DAN SARAN ……….. 24
Kesimpulan ……….. Saran ………...
24 24
UCAPAN TERIMAKASIH ……….... 25
DAFTAR PUSTAKA ………... 26
DAFTAR TABEL
Nomor Halaman
1. Karakteristik Fenotipik Bangsa Sapi Indonesia... 4 2. Jumlah Sampel Ternak Sapi Indonesia...………….... 11 3. Penentuan Genotipe pada Gen CAST Menurut Beberapa
Penelitian………... 14 4. Hasil Identifikasi Genotipe Gen Calpastatin pada Bangsa Sapi
Indonesia……… 18
5. Nilai Frekuensi Genotipe dan Frekuensi Alel Gen CAST|MspI
pada Bangsa Sapi Indonesia... 19 6. Nilai Heterozigositas Pengamatan (Ho) dan Heterozigositas
DAFTAR GAMBAR
Nomor Halaman
1. Karakteristik Fenotipik Bangsa Sapi Indonesia. (a) sapi bali jantan; (b) sapi bali betina; (c) sapi madura; (d) sapi aceh;
DAFTAR LAMPIRAN
Nomor Halaman
1. Modifikasi Metode Isolasi DNA Menggunakan Genomic DNA
Mini Kit (Geneaid)... 31 2. Sekuen Gen Calpastatin Bos taurus dengan Nomor Akses
GenBank AF117813... 32 3. Sekuen Gen Calpastatin Bos taurus dengan Nomor Akses
GenBank AY834775... 34
PENDAHULUAN
Latar Belakang
Indonesia merupakan negara yang memiliki beragam sumber daya genetik ternak, terutama ternak sapi. Sumber daya genetik ternak sapi Indonesia tersebar di berbagai wilayah di Indonesia. Populasi sapi yang semakin berkembang di Indonesia telah menghasilkan sumber daya genetik yang lebih beragam, yaitu mulai dari sapi asli seperti sapi bali, juga sapi hasil persilangan yang telah menjadi sapi lokal seperti sapi pesisir, sapi aceh, sapi madura, sapi sumba ongole (SO) dan sapi peranakan ongole (PO) (Utoyo 2002; Martojo 2003). Belum adanya perhatian dalam rangka meningkatkan produktivitas sumber daya genetik ternak sapi di Indonesia seperti sapi bali, sapi madura, sapi pesisir, sapi aceh, dan sapi katingan menyebabkan potensi sumber daya genetik tersebut belum dimanfaatkan secara optimum. Kajian mengenai populasi bangsa sapi di Indonesia belum banyak dilakukan, sehingga informasi yang dapat diperoleh mengenai bangsa sapi Indonesia tersebut masih terbatas. Informasi genetik mengenai bangsa sapi Indonesia perlu digali dalam rangka memanfaatkan dan mempertahankan sumber daya genetik ternak Indonesia sekaligus sebagai sumber pangan.
Peningkatan produktivitas ternak dapat dilakukan dengan dua pendekatan, yaitu peningkatan kualitas genetik ternak dan peningkatan manajemen lingkungan. Peningkatan kualitas genetik dapat dilakukan dengan seleksi dan persilangan. Dalam konteks seleksi, seiring dengan perkembangan ilmu dan teknologi dalam bidang bioteknologi molekuler memungkinkan seleksi dilakukan pada tingkat DNA, yaitu dengan cara mengidentifikasi keragaman pada tingkat gen yang mungkin berpengaruh terhadap sifat produksi, pertumbuhan dan reproduksi. Salah satu gen yang diduga kuat berpengaruh terhadap sifat pertumbuhan otot dan keempukan daging adalah gen calpastatin. Barendse (2002) menyatakan bahwa gen calpastatin merupakan gen kandidat kuat yang mempengaruhi keempukan daging.
2 Sumantri et al. (2008). Hal ini menjadikan identifikasi keragaman gen calpastatin penting untuk dilakukan pada bangsa sapi Indonesia.
Identifikasi keragaman gen calpastatin dapat dilakukan salah satunya melalui teknik Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP). Palmer et al. (1998) dalam penelitianya menggunakan sekuen gen calpastatin yang terletak antara ekson IC dan ekson ID untuk diamplifikasi menggunakan teknik PCR-RFLP. Goll et al. (2003) menyatakan bahwa ekspresi gen calpastatin yang terletak pada ekson 1C memiliki kemampuan yang paling efektif untuk menghambat aktivitas m-calpain. Hal tersebut mendasari penelitian ini untuk melakukan amplifikasi gen calpastatin pada ekson 1C dan 1D dengan teknik Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP).
Tujuan
TINJAUAN PUSTAKA
Sumber Daya Genetik Ternak Lokal
Secara terminologi, sumber daya genetik ternak adalah semua yang termasuk dalam spesies, bangsa, dan strain (galur) ternak yang secara ekonomi, ilmiah, dan budaya penting bagi umat manusia baik dalam bentuk makanan maupun produksi (Food Agriculture Organization, 1999). Departemen Pertanian (2006) menyatakan bahwa sumber daya genetik ternak adalah substansi yang terdapat dalam bentuk substansi individu suatu populasi rumpun ternak secara genetik unik, terbentuk dalam proses domestikasi dari masing-masing spesies yang memiliki nilai potensial serta dapat dimanfaatkan dan dikembangkan dengan baik untuk menciptakan rumpun atau galur unggul.
Sumber daya genetik ternak merupakan acuan (building block) bagi pertanian dan pengembangan varietas dan bangsa hewan ternak untuk masa yang akan datang. Keanekaragaman bangsa ternak yang mampu beradaptasi dapat menyelamatkan petani dalam menghadapi iklim yang sulit dan wilayah marjinal. Sumber daya genetik lokal dapat dimanfaatkan dengan biaya (input) minimun dan memegang peranan penting dalam budaya masyarakat pedesaan (Food Agriculture Organization, 2001).
Ternak sapi yang terdapat di Indonesia dapat dikelompokkan menjadi tiga kategori ternak, yaitu (1) ternak asli, (2) ternak yang telah beradaptasi dan (3) ternak impor (Sarbaini, 2004). MacHugh (1996) mengemukakan bahwa sapi yang terdapat di Asia khususnya di Indonesia merupakan sapi yang termasuk dalam spesies Bos bibos dan sapi persilangan (crossbreed) yang berbeda dari sapi domestikasi yang terdapat di Afrika dan Eropa, meskipun diduga bahwa pola penyebarannya berasal dari wilayah India (Bos indicus) yang merupakan tipe sapi berpunuk (Zebu). Hasil domestikasi spesies Bos (Bibos) banteng adalah sapi Bali (Bos sundaicus) atau (Bos javanicus) yang sekarang telah menjadi bangsa ternak asli Indonesia (Directorate Generale of Livestock Services, 2003; Martojo, 2003).
4 sapi pesisir, sapi aceh, sapi madura, sapi Sumba Ongole (SO) dan sapi Peranakan Ongole (PO) (Utoyo 2002; Martojo 2003).
Keragaman ternak sapi di Indonesia mengakibatkan keragaman karakteristik fenotipik pada bangsa sapi Indonesia. Karakteristik fenotipik pada bangsa sapi Indonesia, yaitu sapi bali, sapi madura, sapi pesisir, sapi aceh, dan sapi katingan disajikan pada Tabel 1.
Tabel 1. Karakteristik Fenotipik Bangsa Sapi Indonesia
Ternak Karakteristik Literatur
Betina dewasa : berat 260 kg, lingkar dada 165 cm, tinggi gumba 114 cm. panjang badan 260 cm.
Pane (1986)
Sapi madura Berukuran sedang, bertulang bagus, berotot,
tanduk pendek, gumba berkembang baik pada jantan, terdapat lingkaran putih di berpunuk kecil, bertanduk pendek, mampu bertahan pada kondisi lingkungan yang buruk.
Saladin (1983)
Sapi aceh Memiliki gumba berukuran sedang (jantan),
memiliki warna cokelat yang lebih gelap pada bagian depan dibandingkan bagian belakang tubuhnya.
Otsuka et al. (1980).
Sapi katingan Ukuran tubuh tidak terlalu besar, memilki
gumba yang cukup jelas (jantan dan betina), tanduk melengkung ke depan, warna bulu putih hingga hitam
Utomo et al. (2010)
5 Gambar 1. Karakteristik Fenotipik Bangsa Sapi Indonesia. (a) sapi bali jantan; (b)
sapi bali betina; (c) sapi madura; (d) sapi aceh; (e) sapi pesisir; (f) sapi katingan (Balai Pengkajian Teknologi Pertanian Kalimantan Tengah, 2011).
(a)
(c)
(e)
(d)
6 Marker Assisted Selection (MAS)
Kemajuan teknologi bidang genetika molekuler memungkinkan seleksi dilakukan pada tingkat DNA. Identifikasi keragaman DNA yang terkait dengan sifat kuantitatif dapat dijadikan dasar untuk menerapkan Marker Assisted Selection (MAS) (Montaldo dan Herrera, 1998). Upaya seleksi pada tingkat DNA memiliki keakuratan yang lebih tinggi dibandingkan dengan seleksi secara konvensional yang melihat dari segi fenotipik dan melalui ukuran tubuh (morfometrik).
Penggunaan Marker Assisted Selection (MAS) didasarkan pada gagasan bahwa terdapat gen yang memegang peranan utama dan menjadi sasaran atau target secara spesifik dalam seleksi (Van der Werf, 2000). Gen-gen sifat kuantitatif yang memiliki pengaruh besar merupakan gen-gen yang disebut dengan gen utama (major gene) yang terletak pada lokus sifat kuantitatif (Quantitative Trait Loci).
Marker gen telah banyak digunakan untuk mengidentifikasi beberapa gen yang memiliki pengaruh utama (major gene) terhadap sifat-sifat ekonomis pada ternak, seperti kualitas (keempukan daging) (Barendse et al., 2008). Marker Assisted Selection (MAS) dapat membantu dalam meningkatkan laju peningkatan mutu genetik ternak melalui pemanfaatan informasi molekuler genetik dalam program pemuliaan (breeding programme) (Dekkers dan Hospital, 2002; Dekkers 2004).
Gen Calpastatin (CAST)
7 Gambar 2. Daerah Target Amplifikasi Gen Calpastatin (Palmer et al., 2000)
Takano et al. (1999) menyatakan bahwa struktur gen calpastatin dalam fungsinya untuk menghambat calpain ke dalam empat domain (I-IV) yang terdiri atas tiga daerah, yaitu A, B, dan C. Daerah A dan C mengikat ion kalsium (Ca2+) secara kuat, akan tetapi tidak memiliki pengaruh dalam menghambat aktivitas calpain, berbeda dengan daerah B yang memiliki pengaruh menghambat aktivitas dari calpain.
Calpain merupakan sebuah enzim proteolytic terkait dengan ion kalsium (Ca2+), yang ada dalam dua bentuk yaitu µ-calpain dan m-calpain. µ-calpain merupakan calpain yang memerlukan ion Ca2+ dalam konsentrasi rendah, sedangkan m-calpain meruapakan calpain yang memerlukan Ca2+ dalam konsentrasi tinggi. Calpain berfungsi untuk mendegradasi protein sel-sel otot (myofibril) di dalam jaringan otot (Goll et al.,1992). Koohmaraie (1995) menyatakan bahwa aktivitas calpain dalam jaringan otot postmorfem dapat menyebabkan struktur protein sel otot menjadi lemah. Hal ini berakibat pada kualitas daging menjadi lebih empuk, selain µ-calpain dan m-calpain.
Hasil pemotongan produk PCR dengan enzim MspI menghasilkan dua alel, yaitu alel M dan alel N pada domba dorset. MspI menghasilkan potongan produk 336 pb dan 286 pb (Palmer et al., 1998). Keragaman gen calpastatin pada domba lokal indonesia yang diidentifikasi menggunakan teknik PCR dan RFLP-MspI diperoleh dua genotipe, yaitu genotipe MN dan NN (Sumantri et al., 2008). Shahroudi et al. (2006) dengan menggunakan teknik yang sama pada domba Karakul ditemukan tiga genotipe, yaitu genotipe MM, MN dan NN dengan frekuensi masing-masing 0,61 dan 0,36, dan 0,03.
8 dengan menggunakan teknik PCR-RFLP dengan enzim restriksi BamHI dan EcoRI. Kubiak et al. (2004) dengan teknik PCR-SSCP menggunakan enzim restriksi AluI dan Schenkel et al. (2006) dengan teknik RFLP menggunakan enzim restriksi RsaI memperoleh tiga macam genotipe pada sapi Bos taurus, yaitu genotipe CC, GC, dan GG.
Chung et al. (1999) menemukan keragaman gen calpastatin dengan metode PCR-SSCP. Primer yang didesain dari domain 1cDNA bovine calpastatin berhasil mengamplifikasi gen calpastatin sepanjang 500 pb dan menghasilkan dua alel, yaitu alel A dan alel B. Keragaman gen calpastatin tersebut terkait erat dengan sifat pertumbuhan sapi Angus jantan. Sapi Angus dengan genotipe BB mempunyai bobot badan lebih tinggi dari pada sapi dengan genotipe AA dan AB.
Penciri Genetik Polymerase Chain Reaction-Restriction Fragment Lenght
Polymorphism (PCR-RFLP)
Restriction Fragment lenght Polymorphism (RFLP) merupakan salah satu teknik penciri genetik (genetic marker) yang dikembangkan oleh Botstein et al., (1980) yang digunakan untuk mengetahui adanya keragaman sekuens DNA. Penggunaan teknik RFLP menjadi lebih intensif setelah teknik RFLP dikombinasikan dengan teknologi Polymerase Chain Reaction (PCR) yang digunakan hingga saat ini (Mullis et al. 1986).
PCR merupakan suatu teknik untuk menggandakan jumlah molekul DNA pada ruas-ruas tertentu dan monomer-monomer nukleotida yang dilakukan secara in vitro. Proses ini berjalan dengan bantuan primer dan enzim polymerase. Primer merupakan oligonukleotida spesifik yang menempel pada bagian sampel DNA yang akan diperbanyak (Williams, 2005). Proses yang terjadi dalam mesin PCR meliputi tiga tahap utama yaitu denaturasi (pemisahan untai ganda DNA), annealing (penempelan primer), dan ekstensi (pemanjangan primer) (Muladno, 2002). Enzim polymerase merupakan enzim yang dapat mencetak urutan DNA baru. Hasil dari proses PCR dapat divisualisasikan dengan elektroforesis (Williams, 2005).
9 tidaknya situs restriksi kemudian dapat digunakan untuk mengetahui ada tidaknya mutasi. Analisis RFLP biasa digunakan untuk mendeteksi adanya keragaman pada gen yang berhubungan dengan sifat ekonomis, seperti produksi dan kualitas susu (Sumantri et al., 2007).
Keragaman genetik atau yang disebut dengan polimorfisme terjadi apabila terdapat dua alel atau lebih dalam suatu populasi (lebih dari 1%) (Nei dan Kumar, 2000). Penciri molekuler DNA restriction fragmen length polymorphism (RFLP) memiliki tingkat polimorfisme yang tinggi dan secara luas telah digunakan untuk mendapatkan gambaran populasi genetik dan juga untuk mengidentifikasi gen-gen yang mengkode sifat-sifat penting (Montaldo dan Herrera, 1998). Teknik ini semakin intensif digunakan sebagai penciri genetik karena memiliki beberapa keunggulan diantaranya yaitu perbanyakan DNA secara cepat dengan memakai Polymerase Chain Reaction (PCR) dan polimorfisme fragmennya dilakukan dengan enzim restriksi, sehingga mampu mengidentifikasi genotipe secara jelas (Jakaria et al., 2007). Teknik lain yang dapat digunakan untuk mengidentifikasi keragaman Produk PCR atau DNA amplikon antara lain SSCP, TGGE, DGGE, dan sequencing.
Single Strand Conformation Polymorphism (SSCP)
Polymerase Chain Reaction-Single Strand Conformation Polymorphism (PCR-SSCP) merupakan suatu teknik analisis keragaman DNA yang didasarkan pada asumsi bahwa perubahan yang terjadi pada fragmen DNA akan mempengaruhi bentuk fragmen DNA untai tunggalnya. Hal ini terlihat pada perubahan pola migrasi pada gel poliakrilamida non-denaturasi. Metode SSCP dapat mendeteksi adanya mutasi pada fragmen DNA, akan tetapi tidak dapat memberikan informasi mengenai posisi terjadinya fragmen DNA dan memiliki keterbatasan dalam menentukan jumlah alel (Barosso et al., 1999).
Temperature Gradient Gel Electrophoresis (TGGE) dan Denaturing Gradient Gel Electrophoresis (DGGE)
10 yang memiliki perbedaan konsentrasi bahan untuk menyamakan berat molekul (denaturing) (Liu et al., 2008).
Sequencing
MATERI DAN METODE
Lokasi dan Waktu
Penelitian ini dilaksanakan di Laboratorium Genetika Molekuler, Bagian Pemuliaan dan Genetika Ternak, Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor. Penelitian ini telah dilaksanakan pada September 2011 sampai dengan Februari 2012.
Materi
Ternak
Sampel darah sapi lokal Indonesia yang digunakan sebanyak 283 sampel yang terdiri dari sampel sapi bali, sapi madura, sapi aceh, sapi pesisir, dan sapi katingan. Jumlah sampel dari masing-masing sapi lokal Indonesia yang digunakan dalam penelitian ini disajikan pada Tabel 2.
Tabel 2. Jumlah Sampel Ternak Sapi Indonesia
Ternak Sapi n Tahun Koleksi Asal
Sapi Pesisir 50 2006 Kab. Pesisir Selatan
Sapi Aceh 15 2010 Kab. Aceh Besar
Sapi Bali 100 2010 Balai Pembibitan Ternak
Unggul Sapi Bali
Sapi Katingan 50 2010 Daerah Aliran Sungai
Katingan
Sapi Madura 68 2011 Pulau Kangean
Total 283
Keterangan: n = jumlah individu
Alat dan Bahan
12 Bahan dan alat yang digunakan pada tahap elektroforesis dan genotyping antara lain produk PCR, gel agarose 1,5%, loading dye, buffer, dan enzim restriksi (MspI), elektroforesis tray vertikal, inkubator, serta UV-Transilluminator.
Prosedur
Pengambilan Sampel Darah
Sampel darah diambil dari sapi bali, sapi madura, sapi aceh, sapi katingan dan sapi pesisir melalui vena jugularis dan ditambahkan alkohol absolut selanjutnya dibawa ke Laboratorium Genetika Molekuler Ternak.
Isolasi DNA
Isolasi DNA dilakukan dari sampel darah dengan menggunakan Genomic DNA mini kit Geneaid (Lampiran 1) yang dimodifikasi untuk penggunaan sampel darah yang disimpan dalam alkohol.
Amplifikasi Gen Calpastatin
Sekuen primer yang digunakan berdasarkan Palmer et al. (1998), yaitu primer forward 5’ TGGGGCCCAATGACGCCATCGATG 3’ yang terletak di ekson 1C dan primer reverse 5’ GGTGGAGCAGCAC TTCTGATCACC 3’ yang terletak di ekson 1D (Gambar 3), dengan panjang produk PCR 624 pb.
Keterangan:
: Primer forward
: Primer reverse
Ccgghhhhhh : Situs pemotongan
Gambar 3. Tempat Penempelan Primer dan Situs Pemotongan MspI. Nomor akses GenBank: AF117813.
1 3’ tggggcccaa tgatgccatc gatgccttgt catccgactt cacctgcagt tcccctacag
61 ctgatgcaaa gaaaactgag aaagaggtat ggtttttaat gcccttaggg aagcttgtta
121 gaaactacct cccactttaa gacaacaact tttttttaaa cttcattttt cacttcactg
181 cgtcttcatt gctgtgttcg ggctttctct agttggggca agcgaggcct gttctctatt
241 tgcaattttt aggcttctgc agggggctcc tcttgttgct gggc|cggggc tctaggtgca 301 caggcttcat ttgttgtggc tcgagggctc taaaccacag gctcattggt cttggcgcac
361 gggcatggtt accccaatgc atttgggatc tcccctggcc agggagcaaa cctgtttccc
421 ctgcattgca aggcggcctc ttaaccgctg gccaccaggg aagccccaaa atgccaaggc
481 tttttacttc tggttcttac cgtttggttc atatttttcc ttcatctgcc agtcaaacct
541 tcttctgtat tttattttcc agaaatctac agaagaggct ttaaaagctc agtcagctgg
13 Amplifikasi gen calpastatin dilakukan dengan teknik Polymerase Chain Reaction (PCR) menggunakan mesin Applied Biosystem PCR thermalcycler. Amplifikasi gen calpastatin ini menggunakan campuran yang terdiri dari sampel DNA yang telah diekstraksi dari darah, primer (forward dan reverse), taq polymerase, buffer, MgCl2, dan dNTPs. Proses amplifikasi terjadi dalam empat tahap di dalam mesin Applied Biosystem PCR thermalcycler. Kondisi PCR yang digunakan berdasarkan Palmer et al., (1998) yaitu denaturasi pada suhu 95 ºC selama satu menit, penempelan pada suhu 62 ºC selama satu menit, serta pemanjangan molekul DNA yang terjadi pada suhu 72 ºC selama dua menit sebanyak 35 siklus.
Elektroforesis Produk PCR
Elektroforesis produk PCR dilakukan menggunakan 5 µl produk PCR pada gel agarose 1,5% dengan tegangan sebesar 100 volt yang dilakukan selama 60 menit. Pembuatan gel yaitu dengan mencampurkan agarose 0,45 g, 0,5 TBE 30 ml, dan 2,5 µl EtBr. Produk PCR sebanyak 5 µl dicampur dengan loading sebanyak 1 µl. Gel agarose setelah dielektroforesis dilihat panjang pita DNA dengan menggunakan UV-Transilluminator.
Genotyping