VARIASI ALEL BoLA DRB 3.2 PADA SAPI MADURA
ANANDITA EKA SETIADI
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI
Dengan ini saya menyatakan bahwa tesis Variasi Alel BoLA DRB 3.2 pada Sapi Madura adalah karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada Perguruan Tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam daftar pustaka di bagian akhir tesis ini.
ABSTRACT
ANANDITA EKA SETIADI. Variation of BoLA DRB 3.2 Alleles in Madura Cattle. Supervised by ACHMAD FARAJALLAH and BAMBANG SURYOBROTO.
ANANDITA EKA SETIADI. Variasi Alel BoLA DRB 3.2 pada Sapi Madura. Dibimbing oleh ACHMAD FARAJALLAH dan BAMBANG SURYOBROTO.
Sapi madura merupakan salah satu bangsa sapi lokal Indonesia. Berdasarkan asal usulnya, sapi madura diperkirakan berasal dari persilangan sapi zebu dan banteng atau sapi bali dan sapi jawa. Sapi jawa merupakan hibrid dari sapi zebu, taurin dan banteng. Dengan demikian, komposisi dan variasi alel yang terdapat pada populasi sapi madura diperkirakan merupakan gabungan alel yang berasal dari leluhur dan alel hasil rekombinasi.
MHC yang terdapat pada sapi disebut Bovine Lymphocyte Antigen (BoLA). BoLA kelas II penyandi rantai β pada subregion DR disebut BoLA DRB. DRB 3 exon 2 merupakan lokus gen pada kompleks BoLA yang memiliki polimorfisme paling tinggi. Komposisi alel BoLA DRB 3.2 berbeda untuk masing-masing jenis sapi. Belum ada laporan mengenai variasi alel BoLA DRB 3.2 pada sapi madura.
Tujuan penelitian adalah untuk mengetahui variasi alel BoLA DRB 3.2 pada sapi madura dengan menggunakan metode PCR-RFLP serta membandingkannya dengan variasi alel BoLA DRB 3.2 pada sapi limousin dan sapi FH. Hasil penelitian ini diharapkan bisa menjadi informasi dasar untuk mengatahui komposisi alel BoLA DRB 3.2 pada populasi sapi madura, limousin dan FH di Indonesia.
Bahan yang digunakan adalah 40 sampel sapi madura (30 asal kabupaten Sampang dan 10 asal kabupaten Bangkalan). Sebagai pembanding digunakan 31 sampel sapi limousin dan 10 sampel sapi FH. Identifikasi alel BoLA DRB 3.2 didasarkan pada kombinasi pola pemotongan enzim restriksi RsaI, BstYI, dan HaeIII terhadap produk PCR ruas gen BoLA DRB3.2. Alel yang ditemukan kemudian dikonfirmasi dengan definisi alel yang dibuat oleh Van Eijk et al. (1992), Gelhaus et al. (1995), dan Maillard et al. (1999).
Hasil pemotongan amplikon dengan enzim RsaI menemukan 19 pola pemotongan dalam 35 kombinasi, dengan enzim BstYI menemukan 3 pola pemotongan dalam 3 kombinasi, dan dengan enzim HaeIII menemukan 6 pola pemotongan dalam 7 kombinasi.
Alel BoLA DRB 3.2 pada populasi sapi madura dan sapi FH memiliki keragaman yang tinggi, sedangkan pada populasi sapi limousin keragaman alelnya rendah. Analisis chi square untuk melihat kesetimbangan Hardy-Weinberg menemukan bahwa populasi sapi FH dalam keadaan setimbang, sedangkan populasi sapi madura dan limousin tidak dalam keadaan setimbang. Hal ini menunjukkan bahwa persilangan pada populasi sapi madura dan limousin tidak dilakukan secara acak.
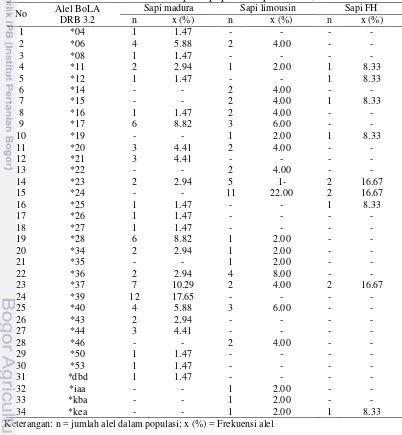
Delapan alel ditemukan pada pada populasi sapi madura dan sapi limousin, yaitu *06, *16, *17, *20, *28, *34, *36, dan *40. Dua alel ditemukan pada populasi sapi madura dan sapi FH, yaitu *11 dan *25. Sebelas alel hanya ditemukan pada populasi sapi madura, yaitu *04, *08, *21, *26, *27, *39, *43, *44, *50, *53 dan *dbd. Frekuensi alel pada populasi madura berkisar antara 1.47% - 17.65%. Alel yang paling banyak ditemukan pada populasi sapi madura adalah alel *39 dengan frekuensi 17.65% (n=68).
Variasi alel BoLA DRB 3.2 pada populasi sapi madura diperkirakan berhubungan dengan asal usulnya. Alel *06, *20, *34, dan *44 (2.94-5.88%) umum ditemukan pada populasi sapi zebu dan alel *08, *11, *12, *16, *17, *20, *21, *23, *26, *28, dan *37 (1.47-10.29%) umum ditemukan pada populasi sapi taurin.
Alel *dbd yang ditemukan pada populasi sapi madura diduga berkaitan dengan alel adaptif yang dimiliki oleh sapi madura. Alel *dbd diperkirakan berasal dari banteng sebagai leluhur sapi madura. Asumsi lain yang mungkin menjadi alasan ditemukannnya alel baru pada populasi madura berkaitan dengan spesiasi. Alel baru tersebut muncul sebagai hasil rekombinasi dari persilangan yang telah dilakukan pada populasi sapi madura.
Hak Cipta milik IPB, tahun 2010
Hak cipta dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tidak merugikan kepentingan yang wajar IPB
VARIASI ALEL BoLA DRB 3.2 PADA SAPI MADURA
ANANDITA EKA SETIADI
Tesis
Sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada
Program Studi Biosains Hewan
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
Judul Tesis : Variasi Alel BoLA DRB 3.2 pada Sapi Madura Nama : Anandita Eka Setiadi
NRP : G352080041
Disetujui Komisi Pembimbing
Dr. Ir. Achmad Farajallah, M.Si. Ketua
Dr.Bambang Suryobroto Anggota
Dr.Bambang Suryobroto Prof. Dr. Ir. Khairil A. Notodiputro, M. S. Diketahui
Ketua Program Studi Biosains Hewan
Dekan Sekolah Pascasarjana
Puji dan syukur penulis panjatkan kepada Allah SWT atas karunia-Nya sehingga karya ilmiah ini dapat diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Mei 2009 ini ialah mengenai Variasi Alel BoLA DRB 3.2 Pada Sapi Madura.
Terima kasih penulis ucapkan kepada Bapak Dr. Ir. Achmad Farajallah, M.Si. dan Bapak Dr. Bambang Suryobroto selaku pembimbing, serta Bapak Dr. Jakaria S.Pt. M.Si. selaku penguji yang telah memberikan banyak saran. Penulis juga mengucapkan terima kasih kepada Ibu Dr. Ir. Dyah Perwitasari, M.Sc. atas ijinnya untuk menggunakan sampel yang diperoleh dari proyek KKP3T Deptan tahun 2009. Terima kasih juga penulis ucapkan kepada Mbak Tini dan Mbak Ani selaku laboran bagian Fungsi Hayati dan Perilaku Hewan Biologi FMIPA IPB serta seluruh teman atas diskusi dan dukungannya. Ungkapan terima kasih juga penulis ucapkan kepada ibunda, suami dan seluruh keluarga penulis atas dukungannya. Semoga karya ilmiah ini bermanfaat.
RIWAYAT HIDUP
Penulis dilahirkan di Kalabahi pada tanggal 14 April 1980 dari ayah Paulus Detakiuk (Almarhum) dan ibu Mimin Mintarsih. Penulis menikah dengan M. Yunus Aqli S.E. dan dikaruniai seorang putri bernama Millati Nurul Hidayah.
DAFTAR ISI
Halaman
DAFTAR ISI ... xiv
DAFTAR TABEL ... xv
DAFTAR GAMBAR ... xvi
DAFTAR LAMPIRAN ... xvii
PENDAHULUAN ... Error! Bookmark not defined. Latar Belakang ... 1
Tujuan Penelitian ... 2
Manfaat Penelitian ... Error! Bookmark not defined. TINJAUAN PUSTAKA ... Error! Bookmark not defined. Tinjauan Umum Sapi Madura ... Error! Bookmark not defined. Major Histocompatibility Complex (MHC) ... Error! Bookmark not defined. Lokus Gen BoLA DRB 3.2 ... Error! Bookmark not defined. Polymerase Chain Reaction – Restriction Fragment Length Polymorphism (PCR-RFLP) ... 9
METODE PENELITIAN ... Error! Bookmark not defined. Waktu dan Tempat... Error! Bookmark not defined. Bahan ... Error! Bookmark not defined. Metode ... Error! Bookmark not defined. Ekstraksi dan Isolasi DNA ... Error! Bookmark not defined. Amplifikasi BoLA DRB 3.2 ... Error! Bookmark not defined. Pemotongan dengan Enzim Restriksi ... Error! Bookmark not defined. Visualisasi Produk PCR dan PCR-RFLP ... Error! Bookmark not defined. Konfirmasi Tipe Alel BoLA DRB 3.2 ... Error! Bookmark not defined. Analisis Data ... Error! Bookmark not defined. HASIL ... Error! Bookmark not defined. Amplifikasi BoLA DRB 3.2 dan Pola Pemotongan Enzim Restriksi ... Error! Bookmark not defined. Pola pemotongan enzim RsaI ... Error! Bookmark not defined. Pola pemotongan enzim BstYI ... Error! Bookmark not defined. Pola pemotongan enzim HaeIII ... Error! Bookmark not defined. Variasi Alel BoLA DRB 3.2 ... Error! Bookmark not defined. PEMBAHASAN ... Error! Bookmark not defined. SIMPULAN DAN SARAN... Error! Bookmark not defined. Simpulan...31
DAFTAR TABEL
Halaman 1 Sampel yang digunakan dalam penelitian ... Error! Bookmark not defined.
2 Pola pemotongan enzim RsaI dan frekuensi tipe BoLA DRB 3.2/RsaIError! Bookmark not defined.
3 Kombinasi pola pemotongan enzim RsaI ... 18
4 Pola pemotongan enzim BstYI dan frekuensi tipe BoLA DRB 3.2/BstYI ... 19
5 Kombinasi pola pemotongan enzim BstYI ... 20
6 Pola pemotongan enzim HaeIII dan frekuensi tipe BoLA DRB 3.2/HaeIII ... 20
7 Kombinasi pola pemotongan enzim HaeIII... 21
DAFTAR GAMBAR
Halaman
1 Ilustrasi kompleks BoLA (MHC mamalia) ... 7
2 Nomenklatur alel BoLA ... 8
3 Produk PCR BoLA DRB 3.2 ... 21
DAFTAR LAMPIRAN
Halaman 1 Pola pemotongan BoLA DRB 3.2 ... Error! Bookmark not defined. 2 Alel BoLA DRB 3 dengan metode SBT dan PCR-RFLP.. Error! Bookmark not defined.
3 Hasil analisis PCR-RFLP pada sampel sapi madura ... Error! Bookmark not defined.
4 Hasil analisis PCR-RFLP pada sampel sapi limousin ... Error! Bookmark not defined.
PENDAHULUAN
Latar Belakang
Perkembangan sapi di Indonesia berawal dari domestikasi banteng (Bos javanicus) yang merupakan leluhur sapi bali pada 3500 SM (Lenstra & Bradley 1999). Pada sekitar abad ke-2 M bersamaan dengan tersebarnya kebudayaan Hindu, sapi zebu (Bos indicus) dari India mulai masuk ke Indonesia (Payne & Hodges 1997). Sapi taurin (B. taurus) mulai masuk ke Indonesia pada masa penjajahan Belanda. Sapi madura diperkirakan mulai muncul setelah masuknya sapi zebu ke Indonesia. Meskipun demikian, hingga saat ini belum ada laporan yang mengungkapkan awal domestikasi sapi madura.
Sapi madura merupakan salah satu bangsa sapi lokal Indonesia yang telah terseleksi secara alamiah dan memiliki persebaran yang terbatas di pulau Madura serta pulau-pulau sekitarnya. Berdasarkan asal usulnya, sapi madura merupakan persilangan sapi zebu dan banteng atau sapi bali (Rouse 1972). Payne & Hodges (1997) menyatakan bahwa sapi madura merupakan hasil persilangan sapi bali dan sapi jawa, dimana sapi jawa sendiri merupakan hibrid dari sapi zebu, taurin dan banteng). Penelitian dengan penanda mikrosatelit menunjukkan sapi madura merupakan hasil persilangan banteng dan zebu atau taurin dan zebu (Nijman et al. 2003). Penelitian dengan penanda mtDNA menunjukkan bahwa terdapat dua tipe maternal origin sapi madura yaitu banteng dan zebu (Uggla 2008; Firdhausi 2010). Berdasarkan penanda gen SRY paternal sapi madura diperkirakan adalah banteng (Verkaar et al. 2003) atau sapi taurin (Kusdiantoro et al. 2009).
2
Persilangan pada populasi sapi madura dilakukan untuk meningkatkan mutu genetik dan produktivitas. Persilangan tersebut didasarkan pada seleksi untuk memilih sifat yang dikehendaki dan mengeliminir sifat yang tidak dikehendaki. Variasi alel yang terdapat pada populasi sapi madura saat ini, diperkirakan merupakan gabungan alel yang berasal dari leluhur dan alel hasil rekombinasi.
MHC merupakan kumpulan gen dan kendali genetik yang menentukan perbedaan reaktivitas imun pada setiap individu. Kendali genetik tersebut mempengaruhi ketahanan dan kerentanan individu terhadap suatu penyakit dalam suatu populasi (Nicholas 1987). MHC yang terdapat pada sapi disebut Bovine Lymphocyte Antigen (BoLA) (Lewin et al. 1999). BoLA terbagi menjadi tiga kelas gen. BoLA kelas II penyandi rantai β pada subregion DR disebut BoLA DRB. DRB3 exon 2 (BoLA DRB3.2) merupakan lokus gen pada kompleks BoLA yang memiliki polimorfisme paling tinggi (Gilliespie et al. 1999; Maillard et al. 1999; Takeshima et al. 2002; Behl et al. 2007; Nassiry et al. 2008).
Beberapa penelitian menunjukkan bahwa variasi alel BoLA DRB 3.2 berbeda untuk setiap jenis sapi, antara lain sapi Holstein (Dietz et al. 1997; Nassiry et al. 2008), Canadian Dairy Cattle (Sharif et al. 1998a), sapi Jersey (Gilliespie et al. 1999), Japanese Shorthorn (Takeshima et al. 2002; Takeshima et al. 2003), Brazilian Dairy Gir Cattle (da Mota 2002), Japanese Black (Takeshima et al. 2003), dan Norwegian Red Cows (Kulberg et al. 2007). Sampai saat ini, belum ada laporan mengenai variasi alel BoLA DRB 3.2 pada sapi madura. Oleh karena itu, penelitian mengenai variasi alel BoLA DRB 3.2 pada sapi madura perlu dilakukan. Sebagai pembanding untuk mengetahui hubungan antara sapi madura dengan sapi jenis taurin, dilakukan juga penelitian mengenai variasi alel BoLA DRB 3.2 pada sapi limousin dan FH.
Tujuan Penelitian
Manfaat Penelitian
TINJAUAN PUSTAKA
Tinjauan Umum Sapi Madura
Pada awal domestikasi, sapi diternakkan manusia untuk dimanfaatkan tenaganya guna membantu di bidang pertanian dan transportasi. Sapi juga digunakan masyarakat sebagai perlambang status sosial dan komoditi perdagangan. Sebagai hewan ternak, sapi juga dimanfaatkan sebagai sumber protein hewani.
Sapi domestik yang berkembang saat ini merupakan hasil domestikasi dari Bos primigenius. Leluhur sapi tersebut punah ± 2000 tahun yang lalu. Terdapat dua tipe utama sapi domestik yang berasal dari B. Primigenius, yaitu jenis kelompok sapi taurin (B. taurus) dan zebu (B. indicus). Zebu merupakan sapi berpunuk (humped) yang tersebar di Asia bagian selatan dan Afrika. Jenis sapi zebu masuk ke wilayah Asia dibawa oleh pengembara Verdic Aryan dari Irak menuju India. Berbeda dengan sapi zebu, sapi taurin merupakan sapi tanpa punuk (humpless) yang berkembang di wilayah Eropa, Asia Tengah, Afrika Barat dan Amerika (Williamson & Payne 1965; Payne & Wilson 1999).
Keragaman sapi lokal Indonesia merupakan hasil persilangan dari sapi zebu, taurin dan banteng (Rouse 1972). Domestikasi banteng yang merupakan nenek moyang sapi bali telah dimulai sejak sekitar 3500 SM (Lenstra & Bradley 1999). Menurut Uggla 2008 sebagian besar sapi lokal Indonesia berasal dari jenis sapi zebu dan sepertiganya berasal dari sapi bali.
dilakukan perbaikan mutu genetik sapi madura dengan jalan menyilangkannya dengan sapi Red Danis (Utoyo et al. 1996; Hardjosubroto 2004).
Sapi madura merupakan salah satu jenis sapi lokal Indonesia yang berkembang di pulau Madura serta pulau-pulau sekitarnya. Secara morfologi, sapi madura memiliki karakter hampir sama dengan sapi bali kecuali ukuran tubuh dan tanduknya yang lebih kecil. Warna kulit pada sapi madura jantan dan betina lebih coklat dari sapi bali, kaki bagian bawah sampai lutut dan sebagian bokongnya berwarna putih (Rouse 1972). Selain itu, sapi madura lebih tahan terhadap cuaca panas, efisien terhadap makanan, memiliki kualitas daging yang baik, dan lebih resisten terhadap parasit (Payne & Hodges 1997).
Kepastian asal domestikasi sapi madura hingga saat ini masih belum diketahui. Masih terdapat perbedaan pada beberapa hasil penelitian mengenai asal usul sapi madura. Menurut Rouse (1972) sapi madura merupakan persilangan antara jenis sapi zebu dan banteng atau sapi bali. Litelatur lain menyatakan bahwa sapi madura merupakan hasil persilangan antara sapi bali dan sapi jawa, dimana sapi jawa sendiri merupakan hibrid dari zebu, taurin, dan banteng (Payne & Hodges 1997). Penelitian Nijman et al. (2003) dengan menggunakan penanda mikrosatelit mengungkapkan bahwa sapi madura merupakan hasil persilangan antara banteng dan zebu atau taurin dan zebu. Penelitian dengan penanda mtDNA yang telah dilakukan oleh Uggla (2008) dan Firdhausi (2010) menunjukkan bahwa terdapat dua tipe maternal origin sapi madura yaitu banteng dan zebu. Sedangkan dengan penanda gen SRY pada kromosom Y paternal sapi madura diperkirakan adalah banteng (Verkaar et al. 2003) atau sapi taurin (Kusdiantoro et al. 2009).
6
mempertahankan populasi, menjaga bentuk, warna kulit, serta meningkatkan kualitas produksi sapi madura (Utoyo et al. 1996).
Major Histocompatibility Complex (MHC)
Major Histocompatibility Complex (MHC) merupakan kelompok lokus yang terdiri atas kumpulan gen penting (major) yang mengendalikan respon imun (Kuby 1997). MHC dibedakan menjadi tiga kelas gen, yaitu kelas I, II dan III. MHC kelas I berisi beberapa gen yang berperan dalam respon imun selular. MHC kelas II merupakan gabungan gen yang berperan penting dalam respon imun selular dan humoral. MHC kelas III mengandung beberapa gen yang memiliki fungsi luas, berperan dalam pembentukan komponen protein dan sistem komplemen, hanya beberapa diantaranya yang terlibat dalam respon imun (Guillemot et al. 1988).
Respon imun terutama disebabkan oleh adanya aksi limfosit yang dihasilkan dalam sel sumsum tulang. Aksi limfosit dalam sistem kekebalan dipacu oleh adanya antigen. Peranan MHC dalam sistem kekebalan seluler diawali dari masuknya antigen ke dalam tubuh melalui proses up take oleh makrofag yang kemudian memicu limfosit T untuk mematikan sel yang terinfeksi. Sementara itu, dalam sistem kekebalan humoral, MHC berperan dalam membantu pembentukan antibodi oleh limfosit B. Peranan keseluruhan MHC yang berkaitan dengan antigen adalah menentukan kemampuan individu untuk membedakan self dan non-self serta mengatur interaksi fungsi imunitas. Berkaitan dengan peranannya tersebut, MHC disebut immune response associated antigen (Nicholas 1987; Guillemot et al. 1988; Kuby 1997).
Lokus Gen BoLA DRB 3.2
MHC pada setiap spesies mempunyai sifat polimorfisme yang tinggi (Guillemot et al. 1988). Sifat polimorfisme pada BoLA menyebabkan kemampuan setiap individu untuk bereaksi terhadap antigen berbeda-beda dan sangat spesifik. Dengan kata lain, BoLA berperan dalam menentukan ketahanan dan kerentanan setiap individu dalam suatu populasi terhadap suatu penyakit secara spesifik.
Pada sapi, kompleks BoLA terdapat pada lengan pendek kromosom 23. Sebagaimana MHC pada mamalia lainnya, BoLA dibagi menjadi tiga kelas gen. BoLA kelas I memiliki tiga gen fungsional, yaitu A, B dan C. BoLA kelas II atau disebut region D dibagi menjadi tiga subregion, yaitu DP, DQ dan DR. Masing-masing subregion paling tidak memiliki dua gen fungsional, yaitu satu gen penyandi rantai α (disebut gen DPA, DQA dan DRA) dan gen lainnya penyandi rantai β (disebut gen DPB, DQB dan DRB). Sampai saat ini, telah diketahui setidaknya ada tiga lokus gen BoLA penyandi rantai β pada subregion DR, yaitu BoLA DRB1, BoLA DRB2 dan BoLA DRB3 (Gambar 1) (Nicholas 1987; Guillemot et al. 1988; Ellis & Ballingall 1999).
Gambar 1 Ilustrasi kompleks BoLA (MHC mamalia) (Guillemot et al. 1988)
8
Gambar 2 Nomenklatur alel BoLA (Buckingham & Flaws 2007)
DRB3 exon 2 (BoLA DRB3.2) merupakan lokus gen pada kompleks BoLA yang memiliki polimorfisme yang paling tinggi (Maillard et al. 1999; Gilliespie et al. 1999; Takeshima et al.2002; Behl et al. 2007; Nassiry et al. 2008). Berdasarkan metode PCR-RFLP, Van Eijk et al. (1992) telah mengidentifikasi 30 Alel BoLA DRB 3.2 berbeda pada 10 peranakan sapi Eropa. Berdasarkan metode yang sama, Gelhaus et al. (1995) menambahkan 10 alel baru yang ditemukannya pada sapi peranakan Afrika dan FH. Alel BoLA DRB3.2 yang telah diidentifikasi dengan metode PCR-RFLP hingga saat ini ada 54 tipe, dengan tambahan 14 alel baru yang ditemukan Maillard et al. (1999) pada sapi zebu Brahman. Seiring perkembangannya, metode yang digunakan untuk mengidentifikasi alel BoLA DRB 3 menjadi sangat bervariasi, salah satunya dengan metode PCR-SBT (Sequence Based Typing) yang telah mengidentifikasi 103 alel BoLA DRB 3 (Takeshima et al. 2003; Miltiadou et al. 2003).
variasi alel pada BoLA DRB3 juga berhubungan dengan karakter produksi susu (Sharif et al. 1998b).
Beberapa penelitian menunjukkan bahwa variasi dan distribusi alel BoLA DRB 3.2 berbeda untuk setiap jenis sapi, antara lain sapi Jersey (Gilliespie et al. 1999), Holstein (Dietz et al. 1997; Nassiry et al. 2008), Japanese Shorthorn (Takeshima et al.2002; Takeshima et al.2003), Japanese Black (Takeshima et al.2003), Brazilian Dairy Gir cattle ( Da Mota 2002) Canadian Dairy Cattle (Sharif et al. 1998a) dan Norwegian Red Cows (Kulberg et al. 2007). Polimorfisme pada kompleks BoLA yang dimiliki oleh populasi sapi mungkin berhubungan dengan asal usulnya (Ellis 1999). Beberapa penelitian menunjukkan bahwa distribusi alel-alel tertentu pada kompleks BoLA mewakili tipe sapi taurin dan zebu (Gilliespie et al. 1999; Maillard et al. 1999). Polimorfisme pada kompleks BoLA juga berpengaruh terhadap fitnes individu dalam populasi. Hal ini berkaitan dengan respon masing-masing gen dalam kompleks BoLA terhadap tekanan lingkungan. Polimorfisme yang tinggi dalam populasi akan memungkinkan populasi tersebut bertahan dalam berbagai kondisi lingkungan (Ellis 1999).
Polymerase Chain Reaction – Restriction Fragment Length Polymorphism (PCR-RFLP)
Polymerase Chain Reaction (PCR) merupakan metode enzimatis yang digunakan untuk melipatgandakan suatu sekuen nukleotida secara eksponensial secara in vitro (Erlich 1989). Teknik PCR dipengaruhi oleh empat komponen utama yaitu: DNA cetakan, oligonukleotida primer, deoksiribonukleotida trifosfat (dNTP) dan enzim DNA polymerase (Sambrook et al. 1989). Prinsip kerja PCR adalah denaturasi, penempelan (annaeling), dan pemanjangan (elongasi).
10
adalah pemanjangan (elongasi) pada suhu 72 °C. Proses dari denaturasi, penempelan, dan pemanjangan disebut satu siklus. Pada umumnya, proses PCR berlangsung dengan 25 – 40 siklus (Sambrook et al. 1989).
PCR-RFLP merupakan teknik PCR yang dikembangkan untuk memvisualisasikan perbedaan runutan nukleotida DNA mengunakan enzim restriksi (Park & Moran 1995). Enzim restriksi bersifat spesifik, yaitu suatu jenis enzim hanya akan memotong runutan nuleotida yang dikenalinya (situs restriksi). Profil fragmen hasil pemotongan menggambarkan variasi runutan nukleotida situs restriksi. Dengan kata lain, perbedaan runutan nukleotida pada setiap fragmen DNA akan menghasilkan pola pemotongan yang berbeda. Fragmen-fragmen DNA hasil pemotongan tersebut dapat dipisahkan dengan elektroforesis melalui matriks gel yang berbentuk pita-pita. Berdasarkan perbedaan panjang pita yang dihasilkan dapat diketahui variasi genetik antar individu dan populasi (Acharya et al. 2002).
PCR-RFLP merupakan metode yang telah terbukti cepat dan sensitif untuk mendeteksi polimorfisme pada gen BoLA DRB 3. Metode ini berguna untuk mempelajari evolusi dan polimorfisme MHC pada sapi dan Bovidae lainnya (Van Eijk et al. 1992). Metode PCR-RFLP untuk mengkarakterisasi BoLA-DRB 3 didasarkan pada banyaknya polimorfisme yang terdapat pada exon 2 gen BoLA DRB 3, polimorfisme tersebut dapat dideteksi dengan enzim restriksi. Alel BoLA DRB 3.2 yang telah diidentifikasi dengan metode PCR-RFLP hingga saat ini ada 54 alel (Van Eijk et al.1992; Gelhaus et al. 1995; Maillard et al. 1999).
Metode PCR-RFLP untuk mendeteksi variasi alel BoLA DRB 3.2 pertama kali digunakan oleh Van Eijk et al. (1992). Penelitian ini dilakukan pada 10 sapi peranakan Eropa (Angus, Ayrshire, Brown-Swiss, Gelbvieh, Guernsey, Jersey, Holstein-Friesian, Polled Hereford, Simmental dan South Devon). Enzim restriksi yang digunakan adalah RsaI, HaeIII dan BstYI. Kombinasi pola pemotongan dari ketiga enzim tersebut digunakan untuk mengidentifikasi 30 Alel BoLA DRB 3.2. Hasil konfirmasi menunjukkan bahwa alel-alel hasil identifikasi menggunakan metode PCR-RFLP cocok dengan urutan DNA yang diperkirakan.
12
METODE PENELITIAN
Waktu dan Tempat
Penelitian dilaksanakan pada bulan Mei 2009 sampai dengan April 2010, bertempat di Bagian Fungsi Hayati dan Perilaku Hewan, Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Bahan
Bahan yang digunakan dalam penelitian adalah sampel darah dan DNA sapi madura, sapi limousin dan sapi FH koleksi Laboratorium Fungsi Hayati dan Perilaku Hewan IPB. Jumlah keseluruhan sampel yang digunakan adalah 81 sampel, terdiri atas 40 sampel sapi madura, 10 sampel sapi FH, dan 31 sampel sapi limousin (Tabel 1). Sampel darah sapi madura diawetkan dalam alkohol absolut, sampel berasal dari peternakan rakyat di kabupaten Sampang dan Bangkalan, Madura. Koleksi sampel sapi limousin dan sapi FH dalam bentuk DNA. Sampel sapi FH berasal dari Koperasi Peternakan Susu (KPS) Kunak Bogor, sedangkan sampel sapi limousin berasal dari Balai Embrio Ternak (BET) Cipelang, Jawa Barat.
Tabel 1 Sampel yang digunakan dalam penelitian
Asal Sampel Jumlah Sampel Tahun Koleksi Jenis Kelamin Sapi madura
Sapi limousin Sapi FH
Sampang Bangkalan BET Cipelang KPS Kunak Bogor
30 10 31 10
2009 2009 2008 2009
♂ & ♀ ♂ & ♀
♀ ♂ & ♀
Total 81
Keterangan: ♂ = Jantan; ♀ = Betina.
Metode
Ekstraksi dan Isolasi DNA
menit. Sel-sel darah kemudian direndam dalam akuades selama 20 menit dan dilanjutkan dengan sentrifugasi 1000 g selama 10 menit. Sel-sel darah yang sudah bebas dari alkohol disuspensikan dalam bufer 1x STE (0.5 M Sodium chloride; Tris-HCl 0.2 M; EDTA 0.02 M; pH 8.0) kemudian dilisis menggunakan proteinase K 0.125 mg/ml dan sodium dodesil sulfat 1% sambil dikocok pelan dalam suhu 56 oC selama 1 jam. Tahap pemisahan dan pemurnian molekul DNA mengikuti petunjuk DNA Extraction Kit for Fresh Blood.
Amplifikasi BoLA DRB 3.2
Amplifikasi lokus BoLA DRB 3.2 dilakukan secara in vitro menggunakan metode polymerase chain reaction (PCR). Primer forward yang digunakan adalah AF135: 5’–ATCCTCTCTCTGCAGCACATTTCC–3’ dan primer reverse AF137: 5’–TCGCCGCTGCACAGTGAAACTCTC–3’. Kedua primer diatas didisain oleh Van Eijk et al. (1992) sebagai HLO30 dan HLO32 dengan target DNA hasil amplifikasi berukuran 284 bp. Pereaksi PCR terdiri atas 10-100 ng sampel DNA, primer forward dan reverse masing-masing 1 µM, dNTP mix 120 µM, MgCl2 100 µM, dan Taq polymerase RBC 1 unit beserta bufernya. Total volume pereaksi adalah 25 μl dalam tabung 0.2 ml. Reaksi PCR dilakukan menggunakan mesin thermocycler TaKaRa yang telah diprogram untuk kondisi predenaturasi pada suhu 94 oC selama 5 menit, 30 siklus denaturasi 94 oC selama 1 menit, penempelan (annealing) pada suhu 57 oC selama 1 menit, pemanjangan 64 oC selama 1 menit. Pemanjangan akhir dilakukan pada suhu 72 oC selama 2 menit.
Pemotongan dengan Enzim Restriksi
14
produk PCR oleh ketiga enzim dilakukan dalam inkubator pada suhu 37 oC selama 3 jam hingga satu malam (± 18 jam).
Visualisasi Produk PCR dan PCR-RFLP
Pemisahan produk PCR dilakukan menggunakan polyacrilamide gel electrophoresis (PAGE) 8% dan produk PCR-RFLP menggunakan PAGE 8% yang dilanjutkan dengan pewarnaan sensitif perak menurut Tegelström (1986). Elektroforesis dijalankan pada tegangan 100 V sampai pewarna bromtimol blue dari loading bufer mencapai tepi katoda gel.
Konfirmasi Tipe Alel BoLA DRB 3.2
Pola hasil pemotongan PCR-RFLP dikonfirmasikan dengan pola pemotongan yang telah diidentifikasi oleh Van Eijk et al. (1992), Gelhaus et al. (1995) dan Maillard et al. (1999) (Lampiran 1). Identifikasi alel BoLA DRB 3.2 menggunakan kombinasi pola pemotongan enzim RsaI, BstYI, dan HaeIII (Lampiran 2).
Analisis Data
Frekuensi tipe pola pemotongan dihitung dengan rumus sebagai berikut:
Keterangan:
xi = Frekuensi tipe i
ni = Jumlah tipe i dalam populasi N = Jumlah total tipe dalam populasi
Frekuensi genotipe dihitung berdasarkan rumus Nei (1987) sebagai berikut:
Keterangan:
Xii = Frekuensi genotipe ii
nii = Jumlah individu bergenotip ii
N = Jumlah total individu Xii= nii / N
Frekuensi alel dihitung berdasarkan jumlah individu bergenotipe homozigot dan heterozigot dengan menggunakan rumus Nei (1987) sebagai berikut:
Keterangan:
Xi = Frekuensi alel i (%)
nii = Jumlah individu bergenotipe ii nij = Jumlah individu bergenotipe ij N = Jumlah total individu
Analisis keragaman genetik pada populasi dilakukan dengan cara membandingkan nilai hetrozigositas hasil penelitian (Ho) dengan nilai heterozigositas yang diharapkan (He) (Nei 1973 diacu dalam Freeland 2005). Rumus yang digunakan adalah sebagai berikut:
Keterangan:
nij = jumlah individu bergenotipe ij
N = jumlah total individu Xi = frekuensi Alel i
Analisis kesetimbangan populasi menggunakan Uji Chi-squared dengan cara membandingkan frekuensi genotipe hasil pengamatan dengan frekuensi genotipe pada populasi setimbang Hardy-Weinberg. Uji Chi-squared menggunakan rumus sebagai berikut:
Keterangan:
O = jumlah individu bergenotip ii, ij,...jj hasil pengamatan E = jumlah individu bergenotip ii, ij,...jj yang diharapkan,
Kriteria Uji yang digunakan adalah: jika angka sigifikansi atau p-value lebih besar dari 0.01 atau χ2 hitung χ2tabel
,
maka populasi dalam keadaanXi= (2nii+ Σ nij)/2N x 100%
χ2= Ʃ(O –
E)2 / E
16
HASIL
Amplifikasi BoLA DRB 3.2 dan Pola Pemotongan Enzim Restriksi
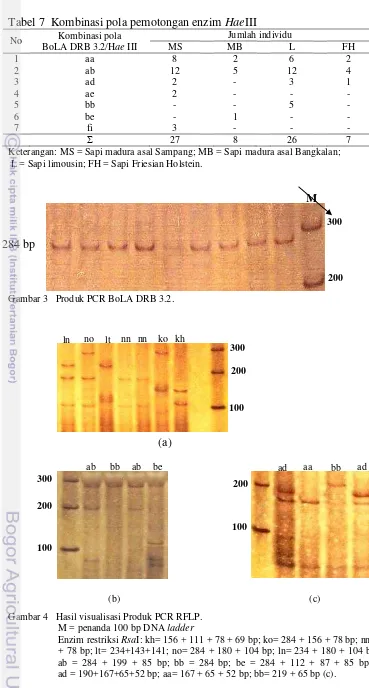
Amplifikasi lokus BoLA DRB 3.2 pada sapi madura, sapi limousin dan sapi FH menggunakan primer AF135 dan AF137. Panjang ruas DNA hasil amplifikasi adalah 284 bp.
Pola Pemotongan Enzim RsaI
Enzim restriksi RsaI memotong pada situs gt↓ac. Kesulitan yang muncul pada saat identifikasi antara lain ukuran fragmen yang terpotong sangat kecil (30, 33 dan 39 bp) dan beberapa pita memiliki perbedaan ukuran fragmen yang sangat sedikit misalnya 50, 51 dan 54 bp; 63 dan 69 bp; 90 dan 93 bp; 104 dan 111 bp.
Berdasarkan hasil pemotongan enzim RsaI, pada populasi Sampang, ditemukan 13 tipe pola pemotongan, yaitu c, d, f, h, j, k, l, n,o, t, u, x, dan y, yang terdistribusi dengan frekuensi 0.0172-0.1724 (n = 58). Tipe-tipe tersebut ditemukan dalam 14 pola kombinasi. Pada populasi Bangkalan ditemukan 8 tipe, yaitu d, g, k, l, o, p, t, dan u, yang membentuk 7 pola kombinasi. Tipe-tipe yang ditemukan pada populasi Bangkalan terdistribusi dengan frekuensi 0.0556-0.2778 (n = 18). Pada sapi limousin ditemukan 14 tipe, yaitu c,d, g, h, i, j, k, l, m, n, o, s, u, dan v, yang membentuk 20 pola kombinasi. Tipe-tipe tersebut terdistribusi dengan frekuensi 0.0167-0.2833 (n = 60). Pada sapi FH ditemukan 8 tipe, yaitu g, h, i, k, l, n, o, dan s, yang membentuk 8 pola kombinasi. Tipe-tipe tersebut terdistribusi dengan frekuensi 0.0625-0.3125 (n = 18).
Tipe k, l, dan o ditemukan pada semua populasi. Tipe d dan u ditemukan pada populasi sapi madura dan sapi limousin. Tipe t ditemukan pada populasi sapi madura. Dengan demikian, tipe d, k, l, o, t, dan u merupakan pola yang ditemukan pada kedua populasi sapi madura. Tipe pola pemotongan RsaI yang hanya ditemukan pada populasi Sampang adalah f, x, dan y, sedangkan p hanya ditemukan pada populasi Bangkalan.
18
cj, do, fy, ll, lx, dan ot hanya ditemukan pada populasi Sampang, sedangkan go, gt, lo, dan pp hanya ditemukan pada populasi Bangkalan. Selain itu, kombinasi pola cl, dl, dn, gg, hh, il,jk, jm, ln, uu, dan vv hanya ditemukan pada sapi limousin, dan ho, kn, no hanya ditemukan pada populasi sapi FH.
Tabel 2 Pola pemotongan enzim RsaI dan frekuensi tipe BoLA DRB 3.2/RsaI No Tipe BoLA
Keterangan: n= Jumlah individu; x=Frekuensi gen; MS = Sapi madura asal Sampang; MB = Sapi madura asal Bangkalan; L = Sapi limousin; FH = Sapi Friesian Holstein
Tabel 3 Kombinasi pola pemotongan enzim RsaI
No Kombinasi Pola BoLA DRB 3.2/RsaI Jumlah Individu
Lanjutan
No Kombinasi Pola BoLA DRB 3.2/RsaI Jumlah Individu
MS MB L FH limousin; FH = Sapi Friesian Holstein.
Pola Pemotongan Enzim BstYI
Enzim restriksi BstYI memotong pada situs R↓gatc. Menurut Van Eijk et al. (1992) pola pemotongan enzim BstYI teridentifikasi pada 5 tipe (a-e). Hasil visualisasi PCR-RFLP tipe a (199 dan 85 bp), c (196 dan 85 bp), dan d (197 dan 87 bp) sulit untuk dibedakan, tipe yang hampir sama tersebut diidentifikasi sebagai tipe a. Hal ini mengakibatkan, dalam penelitian ini ada over estimasi pada tipe a.
Secara keseluruhan didapatkan 3 tipe pola pemotongan dengan enzim BstYI, yaitu a, b dan e dalam 3 kombinasi (ab, bb dan be) (Tabel 4 dan Tabel 5). Tipe dan kombinasi pola terdistribusi pada semua populasi dengan frekuensi yang berbeda-beda.
Tabel 4 Pola pemotongan enzim BstYI dan frekuensi tipe BoLA DRB 3.2/BstYI No Tipe BoLA
20
Tabel 5 Kombinasi pola pemotongan enzim BstYI
No Kombinasi pola BoLA DRB 3.2/BstYI Jumlah individu
MS MB L FH limousin; FH = Sapi Friesian Holstein.
Pola Pemotongan Enzim HaeIII
Enzim restriksi HaeIII memotong pada situs gg↓cc. Ukuran fragmen hasil pemotongan enzim HaeIII adalah 219, 190, 167, 117, dan 65 bp. Besar fragmen yang terpotong tersebut teridentifikasi pada 6 tipe (a, b, d, e, f dan i) dalam 7 kombinasi (aa, ab, ad, ae, bb, be dan fi) (Tabel 6 dan Tabel 7).
Tipe a dan b ditemukan pada populasi sapi madura, sapi limousin, dan sapi FH. Tipe a merupakan tipe dengan frekuensi tertinggi di semua populasi. Tipe e ditemukan pada kedua populasi sapi madura dengan frekuensi 0.0370 pada populasi sampang (n = 54) dan 0.0625 pada populasi Bangkalan (n = 16). Tipe d, f, dan i hanya ditemukan pada populasi Sampang dengan frekuensi 0.0370, 0.0556, dan 0.0556.
Pada penelitian ini, kombinasi pola aa dan ab ditemukan pada populasi sapi madura, Limousin, dan FH. Kombinasi pola aa dan ab ditemukan juga pada populasi sapi Gir dan Kenkrej (Acharya et al. 2002) serta Iranian Holstein (Nassiry 2008). Kombinasi pola yang hanya ditemukan pada populasi Sampang adalah fi, dan be hanya ditemukan pada populasi Bangkalan. Selain itu, kombinasi pola bb hanya ditemukan pada populasi sapi limousin.
Tabel 6 Pola pemotongan enzim HaeIII dan frekuensi tipe BoLA DRB 3.2/HaeIII No Tipe BoLA
Tabel 7 Kombinasi pola pemotongan enzim HaeIII No Kombinasi pola
BoLA DRB 3.2/Hae III
Jumlah individu
Keterangan: MS = Sapi madura asal Sampang; MB = Sapi madura asal Bangkalan; L = Sapi limousin; FH = Sapi Friesian Holstein.
Gambar 3 Produk PCR BoLA DRB 3.2.
(a)
(b) (c)
22
Variasi Alel BoLA DRB 3.2
Identifikasi alel didasarkan pada pola pemotongan enzim RsaI, BstYI, dan HaeIII. Pada penelitian ini, 34 alel telah diidentifikasi, 30 diantaranya merupakan alel yang telah teridentifikasi oleh Van Eijk et al. (1992), Gelhaus et al. (1995), dan Maillard et al. (1999). Empat alel lainnya merupakan alel baru yang belum teridentifikasi sebelumnya. Alel tersebut adalah alel *dbd yang ditemukan pada populasi Sampang, alel *iaa dan *kba pada populasi sapi limousin, serta alel *kea pada populasi sapi limousin dan FH( Tabel 8).
Tabel 8 Variasi alel BoLA DRB 3.2 pada populasi sapi madura, limousin dan FH No Alel BoLA
DRB 3.2
Sapi madura Sapi limousin Sapi FH
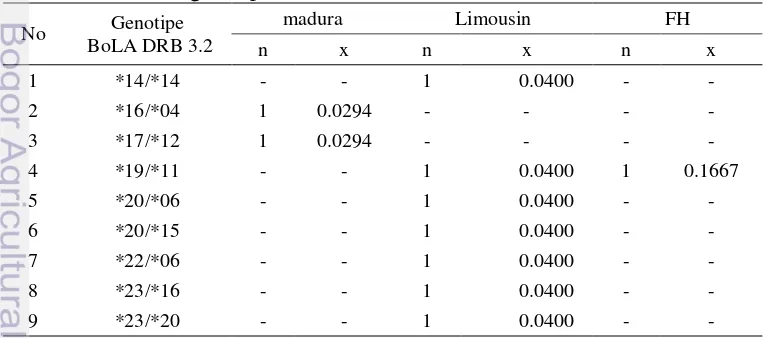
Pada penelitian ini, ditemukan 46 genotipe dari kombinasi 34 alel berbeda (Tabel 9). Dua puluh dua genotipe ditemukan pada populasi sapi madura dengan frekuensi berkisar antara 0.0294-0.1176 (n = 34). Dua puluh satu genotipe ditemukan pada populasi sapi limousin dengan frekuensi berkisar antara 0.0400-0.1600 (n = 25). Enam genotipe ditemukan pada populasi sapi FH dengan frekuensi masing-masing 0.1667 (n = 6).
Lima dari 22 genotipe pada populasi sapi madura ditemukan dengan frekuensi lebih dari 0.005, yaitu *37/*17 (0.1176), *39/*28 (0.1176), *40/*06 (0.1176), *39/*20 (0.0882), dan *44/*43 (0.0588). Tujuh belas genotipe lainnya ditemukan dengan frekuensi masing-masing 0.0294. Satu dari 22 genotipe yang ditemukan pada populasi sapi madura merupakan genotipe homozigot, yaitu *23/*23 (0.0294). Genotipe *23/*23 ditemukan juga pada populasi sapi limousin dengan frekuensi 0.0400.
Hasil analisis keragaman menunjukkan bahwa alel BoLA DRB 3.2 pada populasi sapi madura dan FH memiliki keragaman yang tinggi (Ho > He), sedangkan pada populasi sapi limousin keragaman alelnya rendah (Ho < He). Analisis Chi-squared digunakan untuk mengetahui kesetimbangan Hardy-Weinberg. Uji Chi-squared alel BoLA DRB 3.2 pada populasi sapi FH diperoleh angka signifikansi (p-value) lebih besar dari 0.01, sedangkan pada populasi sapi madura dan limousin diperoleh angka signifikansi lebih kecil dari 0.01. Hal ini berarti, populasi sapi FH dalam keadaan setimbang, sedangkan populasi sapi madura dan limousin tidak dalam keadaan setimbang.
Tabel 9 Frekuensi genotipe alel BoLA DRB 3.2 No Genotipe
BoLA DRB 3.2
madura Limousin FH
n x n x n x
1 *14/*14 - - 1 0.0400 - -
2 *16/*04 1 0.0294 - - - -
3 *17/*12 1 0.0294 - - - -
4 *19/*11 - - 1 0.0400 1 0.1667
5 *20/*06 - - 1 0.0400 - -
6 *20/*15 - - 1 0.0400 - -
7 *22/*06 - - 1 0.0400 - -
8 *23/*16 - - 1 0.0400 - -
26
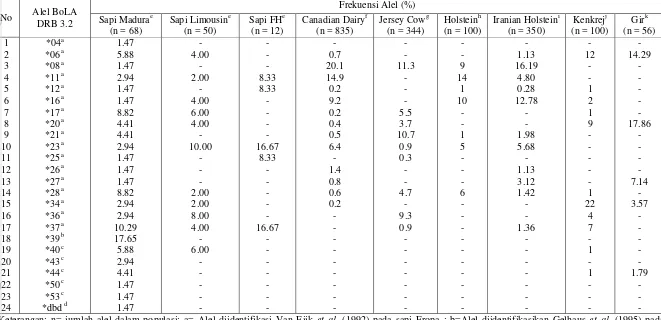
Didapatkan 11 alel yang ditemukan pada populasi sapi madura tetapi tidak ditemukan pada populasi sapi limousin dan sapi FH. Lima diantaranya ditemukan pada populasi jenis sapi lain dari penelitian lainnya (Sharif et al. 1998a; Gilliespie et al. 1999; da Mota et al. 2002; Parnian et al. 2006; Behl et al. 2007; Nassiry et al. 2008). Alel-alel tersebut adalah alel *8, *21, *26, *27, dan *44, dengan frekuensi alel berturut-turut 1.47, 4.41, 1.47, 1.47, dan 4.41%. Enam Alel lainnya hanya ditemukan pada populasi sapi madura tetapi tidak ditemukan pada populasi lainnya. Alel-alel tersebut adalah *4, *39, *43, *50, *53, dan *dbd dengan frekuensi berturut-turut 1.47, 17.65, 2.94, 1.47, dan 1.47%.
Alel *06, *12 , *16, *17, *20, *25, *27, *28, *34, *36, *37, dan *40, dengan frekuensi 55.86% dari total keseluruhan alel yang ditemukan pada populasi sapi madura, merupakan alel yang terdapat pada populasi sapi madura, sapi limousin dan sapi FH. Alel-alel tersebut ditemukan juga pada populasi sapi zebu dan taurin dari penelitian lain seperti pada populasi Canadian Dairy Cattle (Sharif et al. 1998a), Jersey cow (Gilliespie et al. 1999), Brazilian Gir (da Mota et al. 2002), Holstein bulls (Parnian et al. 2006), Kenkrej (Behl et al. 2007) dan Iranian Holstein (Nassiry et al. 2008).
Alel *08, *11, *21, *23, dan *26 terhitung 13.23% dari total alel yang ditemukan pada populasi sapi madura merupakan alel yang diidentifikasi Van Eijk et al. (1992) pada sapi Eropa dan hanya ditemukan pada populasi sapi taurin seperti Canadian Dairy Cattle (Sharif et al. 1998a), Jersey cow (Gilliespie et al. 1999), Holstein bulls (Parnian et al. 2006) dan Iranian Holstein (Nassiry et al. 2008).
PEMBAHASAN
Variasi alel BoLA DRB 3.2 pada populasi sapi madura diduga berhubungan dengan asal usulnya. Hal ini ditunjukkan dengan adanya alel sharing yang ditemukan pada populasi sapi madura, sapi limousin, dan sapi FH. Alel sharing tersebut mencapai 55.86%. Selain itu, beberapa alel yang ditemukan pada populasi sapi madura dilaporkan telah ditemukan pada populasi Canadian Dairy Cattle (Sharif et al. 1998a), Jersey cow (Gilliespie et al. 1999), Brazilian Gir (da Mota et al. 2002), Holstein bulls (Parnian et al. 2006), Kenkrej (Behl et al. 2007), dan Iranian Holstein (Nassiry et al. 2008).
Alel *06, *20, *34, dan *44 merupakan alel yang umum ditemukan pada populasi sapi zebu (Mailard et al. 1999; da Mota et al. 2002; Behl et al. 2007). Alel-alel tersebut diduga berasal dari jenis sapi zebu yang merupakan leluhur sapi madura (Rouse 1972). Ditemukannya alel sapi zebu pada populasi sapi madura mendukung hasil penelitian Firdhausi (2010) yang mengungkapkan adanya dua tipe maternal origin sapi madura yaitu banteng dan zebu. Data tersebut juga mendukung hasil penelitian Nijman et al. (2003) yang mengungkapkan bahwa sapi madura merupakan hasil persilangan antara banteng-zebu atau taurin-zebu.
Alel-alel yang ditemukan pada populasi sapi madura dan umum terdapat pada populasi sapi taurin adalah alel *08, *11, *12, *16, *17, *20, *21, *23, *26, *28, dan *37 (Sharif et al. 1998a; Gilliespie et al. 1999; Takeshima et al. 2002; Parnian et al. 2006; Nassiry et al. 2008). Alel-alel tersebut diduga berasal dari jenis sapi taurin yang merupakan salah satu leluhur sapi madura (Kusdiantoro et al. 2009).
28
Alel *39 yang diidentifikasi oleh Gelhaus et al. (1995) pada sapi peranakan Afrika merupakan alel pada populasi sapi madura dengan frekuensi paling tinggi yaitu 17.65%. Alel *04 dengan frekuensi 1.74 % merupakan alel yang diidentifikasi Van Eijk et al. (1992) pada sapi Eropa. Alel *43, *50, dan *53 dengan frekuensi berturut turut 2.94, 1.74, dan 1.74% merupakan alel yang diidentifikasi oleh Mailard et al. (1999) pada sapi Brahman. Alel-alel tersebut ditemukan pada populasi sapi madura tetapi sangat jarang ditemukan pada populasi sapi lainnya. Pada populasi sapi limousin alel *24 merupakan alel yang paling banyak ditemukan dengan frekuensi 22.00%. Alel *24 tidak ditemukan pada populasi sapi madura.
Empat alel baru yang belum diidentifikasi oleh Van Eijk et al. (1992), Gelhaus et al. (1995) dan Maillard et al. (1999) adalah *dbd, *iaa, *kba, dan *kea. Alel *dbd ditemukan pada populasi Sampang, sedangkan alel *iaa, *kba, dan *kea ditemukan pada populasi sapi limousin. Alel *iaa memiliki kesamaan dengan alel yang ditemukan pada populasi Jersey cow (Gilliespie at al. 1999) dan Holstein bulls (Parnian et al. 2006), sedangkan alel *kba ditemukan juga pada populasi Jersey cow (Gilliespie at al. 1999) dan Sarabi cow (Pashmi et al. 2006). Sampai saat ini, belum ada laporan yang menerangkan adanya kesamaan alel *dbd dan *kea dengan populasi sapi lainnya.
Alel *dbd yang ditemukan pada populasi sapi madura mungkin berkaitan dengan alel adaptif yang dimiliki oleh sapi madura. Alel baru tersebut tidak ditemukan pada jenis sapi taurin dan zebu. Diperkirakan alel *dbd merupakan alel yang berasal dari banteng sebagai leluhur sapi madura. Namun, asumsi ini belum dapat dipastikan karena tidak didukung oleh data pembanding dari variasi alel BoLA DRB 3.2 pada banteng. Asumsi lain yang mungkin menjadi alasan ditemukannnya alel baru pada populasi madura mungkin berkaitan dengan spesiasi. Alel baru tersebut muncul sebagai hasil rekombinasi dari persilangan yang telah dilakukan pada populasi sapi madura.
Eijk et al. (1992), Gelhaus et al. (1995), dan Maillard et al. (1999) (Gilliespie et al. 1999; Parnian et al. 2006; Pashmi et al. 2006; Behl et al. 2007). Lebih jauh Maillard et al. (1999) memperkirakan alel-alel baru akan banyak teridentifikasi terutama pada jenis-jenis sapi di daerah tropis.
Hasil analisis keragaman menunjukkan bahwa alel BoLA DRB 3.2 pada populasi sapi madura dan FH memiliki keragaman yang tinggi, sedangkan populasi sapi limousin keragaman alelnya rendah. Hasil analisis Chi-squared menunjukkan populasi sapi FH dalam keadaan setimbang, sedangkan populasi sapi madura dan limousin tidak dalam keadaan setimbang. Dengan kata lain alel BoLA DRB 3.2 pada populasi sapi FH terdistribusi mengikuti kesetimbangan Hardy-Weinberg, sedangkan populasi sapi madura dan limousin tidak. Hal ini mungkin dikarenakan pada populasi sapi madura dan limousin persilangan tidak dilakukan secara acak sehingga alel BoLA DRB 3.2 dipengaruhi oleh tekanan seleksi yang dikendalikan oleh manusia.
Variasi alel BoLA DRB 3.2 mengindikasikan fitness individu dalam populasi. Berdasarkan hasil penelitian, BoLA DRB 3.2 merupakan lokus gen pada kompleks BoLA yang memiliki variasi alel tinggi. Respon masing-masing alel BoLA DRB 3.2 terhadap tekanan lingkungan akan berbeda. Dengan demikian, variasi alel BoLA DRB 3.2 yang tinggi dalam populasi akan memungkinkan populasi tersebut bertahan dalam berbagai kondisi lingkungan (Ellis & Ballingall 1999).
Beberapa hasil penelitian menunjukkan alel BoLA DRB 3.2 berhubungan dengan sifat produksi dan imunitas (Dietz et al. 1997; Ballingal et al. 2004; Kulberg et al. 2007; Hameed et al. 2008; Panei et al. 2009). Namun, hubungan alel BoLA DRB 3.2 dengan sifat produksi dan imunitas tidak sama untuk semua populasi sapi. Masing-masing alel BoLA DRB 3.2 memiliki tingkat kerentanan dan ketahanan berbeda pada populasi yang berbeda (Parnian et al. 2006).
30
Coast Fever (ECF) yang disebabkan serangan kutu-parasit Theileria parva pada jenis sapi Boran.
SIMPULAN DAN SARAN
Simpulan
BoLA DRB3.2 merupakan lokus gen pada kompleks BoLA yang memiliki polimorfisme tinggi. Hasil amplifikasi dengan metode PCR menggunakan primer AF135 dan AF137 adalah sepanjang 284 bp.
Pada penelitian ini, 34 alel telah diidentifikasi, 30 diantaranya merupakan alel yang telah teridentifikasi oleh Van Eijk et al. (1992), Gelhaus et al. (1995) dan Maillard et al. (1999). Empat alel lainnya merupakan alel baru yaitu; *dbd, *iaa, *kba dan *kea. Pada populasi sapi madura didapatkan 24 alel, pada populasi sapi limousin 21 alel, dan 9 alel pada populasi sapi FH. Tiga belas alel merupakan alel bersama yang ditemukan pada populasi sapi madura, sapi limousin dan sapi FH, yaitu: alel *06, *11, *12, *16, *17, *20, *23, *25, *28, *34, *36, *37 dan *40. Sebelas alel hanya ditemukan pada populasi sapi madura, alel tersebut adalah: alel *04, *08, *21, *26, *27, *39, *43, *44, *50, *53 dan *dbd. Alel yang paling banyak ditemukan pada populasi sapi madura adalah alel *39 dengan frekuensi 17.647% (n = 68).
Saran
32
DAFTAR PUSTAKA
Acharya CP, Pipalia DL, Rank DN, Joshi CG, Solanki JV, Shah RR. 2002. Detection of BoLA DRB3 Gene Polymorphism in Gir and Kankrej Cattle using PCR-RFLP. Indian J Anim Sci 72: 680-683.
Ballingal KT, Luyai A, Rowlands GJ, Sales J, Musoke AJ, Morzaria SP, McKeever DJ. 2004. Bovine Leukocyte Antigen Major Histocompatibility Complex Class II DRB*2703 and DRB*1501 Alleles Are Associated with Variation in Levels of Protection Against Theileria parva Challenge Following Immunization with The Sporozoite p67 Antigen. Infection And Immunity 72: 2738-2741.
Behl JD, Verma NK, Behl R, Mukesh M, Ahlawat SPS. 2007. Characterization of Genetics Polymorphism of The Bovine Lymphocyte Antigen DRB3.2 Locus in Kankrej Cattle (Bos indicus). J Dairy Sci 90: 2997-3001.
Buckingham L, Flaws ML. 2007. Molecular Diagnostics: Fundamentals, Methods And Clinical Applications. Philadelphia : F. A. Davis Company.
Da Mota AM, Gabriel JE, Martinez ML, Coutinho LL. 2002. Distribution of the Bovine Lymphocyte Antigen (BoLADRB3) Alleles from Brazilian Dairy Gir cattle (Bos indicus). Eur J Immunogenet 29: 223-227.
Dietz AB, Cohen ND, Timms L, Kehrli ME. 1997. Bovine lymphocyte antigen class II alleles as risk factors for high somatic cell counts in milk of lactating dairy cow. J Dairy Sci 40: 406-412.
Diwyanto K. 2008. Pemanfaatan sumber daya lokal dan inovasi teknologi dalam mendukung pengembangan sapi potong di Indonesia. Pengembangan Informasi Pertanian 1:173-178.
Ellis SA, Ballingall KT, 1999. Cattle MHC: evolution in action?. Immunol Rev 167:159-168.
Erlich HA. 1989. PCR Technology, Principles and Aplications for DNA Amplification. New York: Stockton Press.
Firdhausi NF. 2010. Asal Usul Sapi Madura Berdasarkan Penanda DNA Mitokondria [Tesis]. Institut Pertanian Bogor.
Freeland JR. 2005. Molecular Ecology. England: J Wiley.
Gilliespie BE, Jayarao BM, Dowlen HH, Oliver SP. 1999. Analysis and Frequency of Bovine Lymphocyte Antigen DRB 3.2 Alleles in Jersey Cows. J Dairy Sci 82: 2049-2053.
Gelhaus A, Schnittger L, Mehlizt D, Horstmann RD, Meyer CG. 1995. Sequence and PCR-RFLP analysis of 14 novel BoLA DRB3 Alleles. Anim Genet 26:147-135.
Guillemot F, Auffray C, Orr HT, Strominger JL. 1988. MHC Antigen Genes. Di dalam: Hames BD, Glover DM, editor. Molecular Immunology. Oxford: IRL Press.
Hardjosubroto. 2004. Alternatif Kebijakan Pengelolaan Berkelanjutan Sumberdaya Genetik Sapi potong Lokal dalam Sistem Pembibitan Ternak Nasional. Wartazoa 14:107-115.
Kuby Janis. 1997. Immunology. Third edition. New York: W. H. Freeman and Company.
Kulberg S, Heringstad B, Guttersrud OA, Olsaker I. 2007. Study on The Association of BoLA DRB3.2 Alleles with Clinical Mastitis in Norwegian Red Cows. J Anim Breed Genet 124: 201-207.
Kusdiantoro M, Olsson M, Tol HTA, Mikko S, Vlaming BH, Andersson G, Martinez HR, Purwantara B, Paling, Colender B, Lenstra JA. 2009. The origin of Indonesian cattle. PloS ONE 4:1-5.
Lenstra JW, Bradley DG. 1999. Systematic and phylogeny of cattle. Di dalam: Fries R & Ruvinsky A, editor. The Genetics of Cattle. United Kingdom: CABI Publishing.
Lewin HA, Russell GC, Glass E. 1999. Comparative organization and function of the major histocompatibility complex of domesticated cattle. Immunol Rev 167:145-158.
Maillard JC, Berthier D, Chantal I, Thevenon S, Sidibe I, Stachurski DB, Razafindraibe H, Elsen JM. 2003. Selection Assisted by a BoLA DR/DQ Haplotype Against Susceptibility to Bovine Dermatophilosis. Genet Sel Evol 35: 193-200.
Maillard JC, Renard C, Chardon P, Chantal I, Bensaid A. 1999. Characterization of 18 New BoLA-DRB 3 Alleles. Anim Genet 30: 200-203.
34
Miltiadou D, Law AS, Russell GC. 2003. Establishment of Sequence Based Typing System for BoLA DRB3 Exon 2. Tissue Antigens 62: 55-65.
Nassiry MR, Sadeghi B, Tohidi R, Afshari JT, Khosravi M. 2008. Comparison of Bovine Lymphocyte Antigen DRB3.2 Allele Frequencies Between Two subpopulation of Iranian Holstein Cattle. Afr J Biotechnol 7: 2671-2675. Nei M. 1987. Molecular Evolutionary genetics. New York: Columbia University
Press.
Nicholas FW. 1987. Veterinary Genetics. Oxford: Clarendon Press.
Nijman IJ, Otsen M, Veekar ELC, de Ruijter C, Hanecamp E, Ochieng JW, Shamshad S, Rege JEO, Hanotte O, Barwegwn MW, Sulawati T, Lenstra JA. 2003. Hybridization of Banteng (Bos javanicus) and Zebu (Bos indicus) Revealed by Mitochondrial DNA, Satellite DNA, AFLP and Microsatellites. Heredity 90:10-16.
Panei CJ, Suzuki K, Echeverria, Serena MS, Metz GE, Gonzales ET. 2009. Association of BoLA DRB 3.2 Alleles with resistance and susceptibility to Persistent Lymphocytosis in BLV Infected Cattle in Argentina. Int J Dairy Sci 4:123-128.
Park LK, Moran P. 1995. Development in Molecular Genetics Techniques in Fisheries. Di dalam: Gary R Carvalho dan T.T Pitcher. Editor. Molecular Genetics in Fisheries. Cornwall: TJ Press Ltd.
Parnian M, Ghorashi SA, Salehi A, Pashmi M, Mollasalehi M.R. 2006. Polymorphism of Bovine Lymphocyte Antigen DRB 3.2 in Holstein Bulls of Iran using PCR-RFLP. Iran J Biotechnol 4: 197-200.
Pashmi M, Ghorashi SA, Salehi AR, Moini M, Javanmard A, Qanbari S, Yadranji-Aghdam S. 2006. Polymorphism of Bovine Lymphocyte Antigen DRB3.2 Alleles in Iranian Native Sarabi Cows. Asian-Aust J Anim Sci 6:775-778.
Payne WJA, Hodges J. 1997. Tropical Cattle: Origin, Breed, and Breeding Policies. Oxford: Blackwell Science Ltd.
Payne WJA, Wilson. 1999. An Introduction to Animal Husbandry in the Tropics. Oxford: Blackwell science Ltd.
Rouse JE. 1972. Cattle of Africa and Asia. Oklahoma: University of Oklahoma Press.
Sharif S, Mallard BA, Wilkie BN, Sargeant JM, Scott HM, Dekkers JC, Leslie KE, 1998a. Association of The Bovine Major Histocompatibility Complex DRB3 (BoLA-DRB3) alleles with Occurence of Disease and Milk Somatic Cell Score in Canadian Dairy Cattle. Anim Genet 29: 185-193.
Sharif S, Mallard BA, Wilkie BN, Sargeant JM, Scott HM, Dekkers JC, Leslie KE, 1998b. Association of The Bovine Major Histocompatibility Complex DRB3 (BoLA-DRB3) alleles with Production Trait in Canadian Dairy Cattle Anim Genet 30: 157-160.
Takeshima S, Nakai Y, Ohta M, Aida Y. 2002. Characterization of DRB3 Alleles in the MHC of Japanese Shorthorn Cattle by Polymerase Chain Reaction Sequence Based Typing. J Dairy Sci 85: 1630–1632.
Takeshima S, Saitou N, Morita M, Aida Y. 2003. The diversity of bovine MHC class II DRB3 genes in Japanese Black, Japanese Shorthorn, Jersey and Holstein cattle in Japan. Gene 316: 111–118.
Uggla CM. 2008. Investigating Genetic Variability Within Specific Indigenous Indonesian Cattle Breed [Disertasi]. Swedish University of Agricultural Science.
Utoyo DP, Djarsanto, Nasution SN. 1996. Animal Genetic Resources and Domestic. Jakarta: Ministry of Agriculture Directorate General of Livestock Services. Directorate of Livestock Breeding Development.
Verkaar ELC, Vervaecke H, Roden C, Mendoza LR, Barwegwn MW, Susilawati T, Nijman IJ, Lenstra JA. 2003. Paternally inherited markers in bovine hybrid populations. Heredity 91: 565-569.
Williamson G, Payne WJA. 1965. An Introduction To Animal Husbandry In The Tropic. London: Lougman.
Wiyono DB, Aryogi. 2007. Petunjuk Teknis Sistem Pembibitan Sapi Potong. Pasuruan: Loka Penelitian Sapi Potong Grati
Van Eijk MTJ, Stewart-Haynes JA, Lewin HA. 1992. Extensive polymorphism of the BoLA-DRB3 gene distinguished by PCR-RFLP. Anim Genet 23:483-496. http://www.projects.roslin.ac.uk/bola/drb3pcr.html. 2002. BoLA Nomenclature
36
Lampiran 1 Pola Pemotongan BoLA DRB 3.2
A. Pola Pemotongan BoLA DRB 3.2 dengan Enzim Rsa I: a.78 + 33 + 30 + 39 + 54 + 50
b.111 + 30 + 39 + 54 + 50 c.111 + 30 + 93 + 50 d.111 + 30 + 143 e.141 + 39 + 51 + 50 f.141 + 39 + 54 + 50 g.141 + 39 + 104 h.111 + 69 + 54 + 50 i.180 + 54 + 50 j.78 + 63 + 93 + 50 k.78 + 156 + 50 l.234 + 50
m. 111 + 69 + 104 n.180 + 104 o.284
p.111 + 30 + 39 + 51 + 50 q.141 + 90 + 50
r.111 + 30 + 90 +50 s.141 + 93 + 50 t.141 + 143 u.111 + 123 + 50 v.78 + 102 + 54 + 50 w. 78 + 33 + 69 + 54 + 50 x.78 + 33 + 69 + 104 y.78 + 6 + 39 + 54 + 50
B. Pola Pemotongan BoLA DRB 3.2 dengan Enzim Bst Y I:
a. 199 + 85 b. 284
c. 196 + 85 d. 87 + 197 e. 87 + 112 + 85
Pola pemotongan a - e diidentifikasi oleh van Eijk et al. (1992) (http://www.projects.roslin.ac.uk/bola/drb3pcr.html).
C. Pola Pemotongan BoLA DRB 3.2 dengan Enzim Hae III:
a. 167 + 52 + 65 b. 219 + 65 c. 167 + 49 + 65 d. 190 + 29 + 65 e. 167 + 117
f. 167 + 4 + 48 + 65 g. 164 + 55 + 65 h. 167 + 46 + 6 + 65 i. 167 + 4 + 113
Pola pemotongan a - f diidentifikasi oleh van Eijk et al. (1992); g dan h oleh Gelhaus et al. (1995); i adalah pola pemotongan untuk alel DRB3*25011 atau DRB3.2*44
38
Lampiran 2 Alel BoLA DRB 3 dengan metode SBT dan PCR-RFLP.
Alel BoLA DRB 3 Pola pemotongan
SBT PCR-RFLP RsaI BstYI HaeIII
DRB3*0501 *01 a a a
DRB3*0503 *01 a a a
DRB3*1301 *02 b b a
DRB3*1001 *03 b b b
DRB3*1002 *03 b b b
No sequence *04 c a a
DRB3*3301 *05 r c c
DRB3*2201 *06 d a a
DRB3*2202 *06 d a a
DRB3*0201 *07 e c c
DRB3*1201 *08 f a a
DRB3*0301 *09 f d a
DRB3*0302 *09 f d a
DRB3*1601 *10 f b a
DRB3*1602 *10 f b a
DRB3*0901 *11 g e a
DRB3*0902 *11 g e a
DRB3*1202 *11 g e a
DRB3*1701 *12 h a a
DRB3*1702 *12 h a a
DRB3*3201 *12 h a a
DRB3*3202 *12 h a a
DRB3*3203 *12 h a a
DRB3*0401 *13 h b a
No sequence *14 h b b
DRB3*20011 *15 i b a
DRB3*20012 *15 i b a
DRB3*2002 *15 i b a
DRB3*2003 *15 i b a
DRB3*1501 *16 j b d
DRB3*1502 *16 j b d
No sequence *17 k b b
Lanjutan
Alel BoLA DRB 3 Pola pemotongan
SBT PCR-RFLP RsaI BstYI HaeIII
DRB3*1802 *18 l b f
DRB3*2601 *19 s b b
DRB3*2301 *20 l b b
DRB3*2901 *20 l b b
DRB3*3601 *20 l b b
DRB3*0801 *21 l b e
DRB3*1101 *22 m b a
DRB3*2701 *23 n b a
DRB3*2702 *23 n b a
DRB3*2703 *23 n b a
DRB3*2705 *23 n b a
DRB3*2706 *23 n b a
DRB3*2707 *23 n b a
DRB3*0101 *24 n b b
DRB3*0102 *24 n b b
No sequence *25 o a a
DRB3*0601 *26 o a b
DRB3*14011 *27 o b f
DRB3*14012 *27 o b f
DRB3*3101 *27 o b f
DRB3*0701 *28 o b b
DRB3*4101 *29 p c c
Genotyping error *30 q c c
DRB3*2801 *31 i b f
DRB3*2401 *32 m a a
DRB3*2704 *33 n b f
DRB3*3001 *34 l a b
DRB3*3002 *34 l a b
DRB3*2101 *35 c b b
DRB05 *36 l b a
DRB07 *37 o b a
DRB18 *38 b d a
40
Lanjutan
Alel BoLA DRB 3 Pola pemotongan
SBT PCR-RFLP RsaI BstYI HaeIII
No sequence *40 u b a
DRB3*0502 *41 a b a
DRB3*1901 *41 a b a
DRB3*3801 *41 a b a
DRB3*2802 *42 h b f
DRB3*25012 *43 k b f
DRB3*25011 *44 k b i
DRB3*3401 *45 s d b
DRB3*3402 *45 s d b
DRB3*3501 *46 v b a
DRB3*1703 *47 w a a
DRB3*3901 *48 w b a
DRB3*3701 *49 w b e
DRB3*4001 *50 x b a
DRB3*4201 *51 g a a
DRB3*0303 *52 s d a
DRB3*1902 *53 y b a
DRB3*4301 *54 j d b
Alel BoLA DRB 3.2*01 – *38 diidentifikasi oleh Van Eijk et al. (1992) pada European cattle. Alel BoLA DRB 3.2*39 dan *40 diidentifikasi Gelhaus et al. (1995) pada sapi FH dan African breeds. Alel BoLA DRB 3.2*41 – *54 diidentifikasi oleh Mailard et al. (1999) pada sapi Brahman (Bos indicus) (http://www.projects.roslin.ac.uk/bola/drb3pcr.html).
Lampiran 3 Hasil Analisis PCR-RFLP pada sampel sapi madura No
Sampel
Rsa I Bst Y I Hae III
Alel
situs pemotongan pola situs pemotongan pola situs pemotongan pola
S1 78, 156, 284 ko 284 bb 48,65, 113, 167 fi 44, 27
S3 30, 143, 284 do 284 bb 52, 65, 167, 190 ad 37, *dbd
S5 104, 180 nn 284 bb 52, 65, 167 aa 23
S7 50, 78, 156 kk 284 bb 48, 65, 113, 167 fi 43, 44
S10 63, 78, 93, 111 cj 85, 199, 284 ab 52, 65, 167, 190 ad 4,16
S12 50, 234 ll 284 bb 65, 117, 167 ae 36, 21
S13 141, 143, 234 tl 284 bb 65, 167, 219 ab 20, 39
S14 69, 78, 104, 234 xl 85, 199, 284 ab 65, 167, 219 ab 50, 34
S15 50, 111, 143, 123 du 85, 199, 284 ab 52, 65, 167 aa 6, 40
S16 141, 143, 284 to 284 bb 65, 167, 219 ab 28, 39
S17 141, 143, 284 to 284 bb 65, 167, 219 ab 28, 39
S18 141, 143, 284 to 284 bb 65, 167, 219 ab 28, 39
S19 141, 143, 284 to 284 bb 52, 65, 167 aa 37, 39
S24 - 284 bb 52, 65, 167 aa -
S25 50,69, 111,156 kh 85, 199, 284 ab 65, 167, 219 ab 17, 12
S26 78, 156, 284 ko 85, 199, 284 ab 65, 167, 219 ab 17, 25
S27 141, 143, 234 tl 284 bb 52, 65, 167 aa 39, 36
S28 50, 111, 143, 123 du 85, 199, 284 ab 52, 65, 167 aa 6, 40
S28 b 50, 111, 143, 123 du 85, 199, 284 ab 52, 65, 167 aa 6, 40
S29 50, 78, 156 kk 284 bb 48,65, 113, 167 fi 43, 44
S30 78, 156, 284 ko 284 bb 65, 167, 219 ab 17, 37
42
Lanjutan No Sampel
Rsa I Bst Y I Hae III
Alel situs pemotongan pola situs pemotongan pola situs pemotongan pola
S32 50, 54, 63, 78, 141 fy 85, 199, 284 ab 52, 65, 167 aa 8, 53
S33 141, 143, 234 tl 284 bb 65, 117, 167 ae 39, 21
S34 50, 111, 123, 156 ku 85, 87, 112, 284 be - -
S36 50, 78, 156 kk 85, 87, 112, 284 be - -
S38 50, 78, 156 kk 284 bb - -
S39 141, 143, 234 tl 284 bb 65, 167, 219 ab 20, 39
S40 141, 143, 234 tl 85, 199, 284 ab 65, 167, 219 ab 39, 34
S41 141, 143, 284 to 284 bb 65, 167, 219 ab 28, 39
S43 39, 104, 141, 143 gt 85, 87, 112, 284 be 52, 65, 167 aa 39, 11
S46 78, 156, 284 ko 284 bb 65, 167, 219 ab 17, 37
S49 50, 111, 143, 123 du 85, 199, 284 ab 52, 65, 167 aa 6, 40
S50 - 85, 199, 284 ab - -
S 51 141, 143, 234 tl 284 bb 65, 167, 219 ab 20, 39
S52 50, 234, 284 lo 284 bb 65, 117, 167, 219 be 21, 28
S53 39, 104, 141, 284 go 85, 87, 112, 284 be 65, 167, 219 ab 28, 11
S54 78, 156, 284 ko 284 bb 65, 167, 219 ab 17, 37
S60 39, 50, 51, 111 pp 284 bb - -
S61 78, 156, 284 ko 284 bb 65, 167, 219 ab 17, 37
Lampiran 4 Hasil Analisis PCR-RFLP pada sampel sapi limousin No
Sampel
Rsa I Bst Y I Hae III
Alel situs pemotongan pola situs pemotongan pola situs pemotongan pola
L146 104, 180 nn 284 bb 65, 167, 219 ab 23,24
L149 50, 111, 123 uu 284 bb 52, 65, 167 aa 40
L154 50, 54, 78, 102 vv 284 bb 52, 65, 167 aa 46
L157 104, 180 nn 284 bb 52, 65, 167 aa 23
L161 - 85, 199, 284 ab - -
L168 50, 69, 111 hh 284 bb 65, 219 bb 14
L169 50, 78, 156, 180 ik 85, 199, 284 ab 65, 167, 219 ab 17 , *iaa
L171 104, 180 nn 284 bb 65, 219 bb 24
L174 104, 180 nn 284 bb 65, 219 bb 24
L119 50, 180, 234 li 284 bb 52, 65, 167 aa 36, 15
L122 50, 111, 123, 156 ku 284 bb 65, 167, 219 ab 17, 40
L128 78, 156, 284 ko 85, 87, 112, 284 be 52, 65, 167 aa 37, *kea
L135 50, 93, 104, 141 gs 85, 87, 112, 284 be 65, 167, 219 ab 11, 19
L136 284 oo 284 bb 65, 167, 219 ab 37, 28
L137 104, 180 nn 284 bb 65, 167, 219 ab 23, 24
L138 50, 111, 143, 234 dl 85, 199, 284 ab 65, 167, 219 ab 6, 20
L140 104, 111, 143, 180 nd 85, 199, 284 ab 52, 65, 167 aa 6, 22
L141 50, 180, 234 li 284 bb 65, 167, 219 ab 15, 20
L142 93, 104, 111 jm 284 bb 52, 65, 167, 190 ad 22, 16
L144 39, 104, 141 gg 85, 87, 112 ee - -
L150 50, 93, 111, 234 cl 284 bb 65, 167, 219 ab 35, 36
44
Lanjutan No Sampel
Rsa I Bst Y I Hae III
Alel situs pemotongan pola situs pemotongan pola situs pemotongan pola
L151 50, 111, 143, 123 du 85, 199, 284 ab - -
L155 104, 180, 234 ln 85, 199, 284 ab 65, 167, 219 ab 23, 34
L156 104, 180, 234 ln 284 bb 65, 167, 219 ab 20, 23
L158 - 284 bb 52, 65, 167, 190 ad -
L159 104, 180 nn 284 bb 65, 219 bb 24
L160 104, 180 nn 284 bb 65, 219 bb 24
L163 50,69, 111,156 kh 85, 199, 284 ab - -
L 148 50, 63, 78, 93 156 jk 284 bb 52, 65, 167, 190 ad 16, *kba