METODE NON-INVASIF UNTUK MENENTUKAN PEDIGRI
HARIMAU SUMATERA (
Panthera tigris sumatrae
)
BERDASARKAN PENANDA D-LOOP DAN MIKROSATELIT
DESI SETIANINGSIH
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa skripsi berjudul Metode Non-invasif untuk Menentukan Pedigri Harimau Sumatera (Panthera tigris sumatrae) berdasarkan Penanda D-loop dan Mikrosatelit adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir disertasi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Desember 2013 Desi Setianingsih
ABSTRAK
DESI SETIANINGSIH. Metode Non-invasif untuk Menentukan Pedigri Harimau Sumatera (Panthera tigris sumatrae) berdasarkan Penanda D-loop dan Mikrosatelit. Dibimbing oleh DEDY DURYADI SOLIHIN dan LIGAYA ITA TUMBELAKA.
Harimau sumatera (Panthera tigris sumatrae) merupakan subspesies harimau asal Indonesia yang masih bertahan hidup, dan termasuk hewan langka yang terancam punah. Hasil analisis karakteristik genetiknya dapat digunakan untuk merancang program guna mencegah kepunahan. Penelitian ini bertujuan untuk menganalisis pedigri pada harimau sumatera berdasarkan karakteristik genetik dengan menggunakan penanda genetik D-loop bagian HVS-I dan mikrosatelit. DNA diekstraksi dari rambut harimau sumatera (terdiri atas satu keluarga), yang diambil dari Kebun Binatang Bandung. DNA total hasil ekstraksi dilanjutkan dengan proses amplifikasi dan sekuensing. Data hasil pembacaan sekuen D-loop dianalisis menggunakan software MEGA 4.0. Hasil sekuensing disejajarkan dengan harimau sumatera asal Aceh (kode sampel A1) sebagai pembanding, dan didapat 208 nukleotida dari ketiga sampel yang diteliti (M1, W2, dan Y4) yang berbeda dari sekuen acuan harimau A1. Lokus mikrosatelit FCA 96 hasil amplifikasi digunakan sebagai penanda untuk mengetahui hubungan silsilah keturunan antar individu, sehingga dapat dianalisis karakteristik pedigrinya. Pita yang dihasilkan pada ketiga individu sampel berupa pita sejajar dengan jumlah yang sama sehingga terbukti adanya hubungan keluarga di antara ketiga individu tersebut, dengan penurunan alel yang sama dari induk jantan maupun induk betina. Kata kunci: D-loop, mikrosatelit, Panthera tigris sumatrae, pedigri
ABSTRACT
DESI SETIANINGSIH. Non-invasive Method for Determination of the Sumatran Tiger (Panthera tigris sumatrae) Pedigree based on D-loop and Microsatellite. Supervised by DEDY DURYADI SOLIHIN and LIGAYA ITA TUMBELAKA.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Sains
pada
Departemen Biologi
METODE NON-INVASIF UNTUK MENENTUKAN PEDIGRI
HARIMAU SUMATERA (
Panthera tigris sumatrae
)
BERDASARKAN PENANDA D-LOOP DAN MIKROSATELIT
DESI SETIANINGSIH
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
Judul Skripsi : Metode Non-invasif untuk Menentukan Pedigri Harimau Sumatera (Panthera tigris sumatrae) berdasarkan Penanda D-loop dan Mikrosatelit
Nama : Desi Setianingsih NIM : G34080086
Disetujui oleh
Dr Ir Dedy Duryadi Solihin, DEA Pembimbing I
Dr drh Ligaya ITA Tumbelaka SpMP MSc Pembimbing II
Diketahui oleh
Dr Ir Iman Rusmana, MSi Ketua Departemen
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga penulis dapat menyelesaikan karya ilmiah yang berjudul Metode Non-invasif untuk Menentukan Pedigri Harimau Sumatera (Panthera tigris sumatrae) berdasarkan Penanda D-loop dan Mikrosatelit.
Penulis mengucapkan rasa terima kasih kepada Dr Ir Dedy Duryadi Solihin DEA dan Dr drh Ligaya ITA Tumbelaka SpMP. M.Sc, selaku pembimbing atas bantuan, arahan dan saran yang diberikan sehingga penelitian ini dapat diselesaikan dengan baik. Penulis juga mengucapkan terima kasih kepada Dr Bambang Suryobroto selaku penguji skripsi. Terima kasih penulis sampaikan kepada Ibu Dr N.A. Butet, Bapak Heri, Bapak Jusmaldi, atas segala bantuan dan doa, terima kasih kepada seluruh staf Departemen Biologi. Di samping itu, penulis ucapkan terimakasih kepada drh Effy Sofiyanty, Bapak Dede Dani, Bapak Rukmana, Bapak Narman, Bapak Yayat, dan seluruh staf Kebun Binatang Bandung atas segala bantuan dan informasi yang diberikan. Penulis haturkan terima kasih yang sebesar-besarnya kepada kedua orang tua, Engkos Kosasih dan Nining Karningsih, Rachmat Kurnia ST, Rima Rahmawati SPd, Widiastuti Eka Wulan ST, Dudy Hamdani AMd, Alya Aprilyanti, Alifa, Azkia, Raziq, atas segala doa, dukungan dan kasih sayangnya selama ini.
Ungkapan terima kasih juga disampaikan kepada para rekan seperjuangan dan sahabat, Putri, Isna, Qila, Traya, Whendi, Mae, Ai, Wathri, Ammar, Gauz, Kang Agus, Afnan, Nia, Fatni, Indah, Rei, Windy, Ayu, Nilam, Ana, Doraemon, Kim Myungsoo, Choi Siwon, Oh Sehun, Gita, Vidar, dan seluruh keluarga besar Biologi 45 atas kebersamaan dan kenangan indah selama ini.
Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
PENDAHULUAN 1
Tujuan Penelitian 2
METODE 2
Bahan 2
Isolasi DNA 3
Amplifikasi dan Visualisasi Fragmen DNA 3
Perunutan Produk PCR (Sekuensing) 4
Pensejajaran Runutan Nukleotida (Alignment) dan Analisis Filogeni 4
HASIL DAN PEMBAHASAN 4
Hasil 4
Pembahasan 8
SIMPULAN DAN SARAN 10
Simpulan 10
Saran 10
DAFTAR PUSTAKA 11
LAMPIRAN 13
DAFTAR TABEL
1 Hasil kemurnian DNA dengan uji NanoDrop 5
2 Matrik rata-rata perbedaan nukleotida berdasarkan metode pairwise distance daerah D-loop bagian HVS-I pada harimau Sumatra M1, W2,
Y4, dan Aceh A1 7
DAFTAR GAMBAR
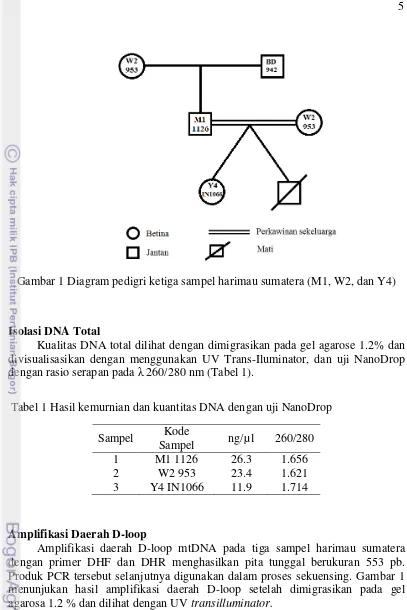
1 Diagram pedigri ketiga sampel harimau sumatera (M1, W2, dan Y4) 5 2 Hasil amplifikasi daerah D-loop pada gel agarosa 1.2% 6 3 Pohon filogeni berdasarkan runutan nukleotida daerah D-loop bagian
HVS-I dengan metode Neighbor-Joining, bootstrapped 1000x, model
p-distance 7
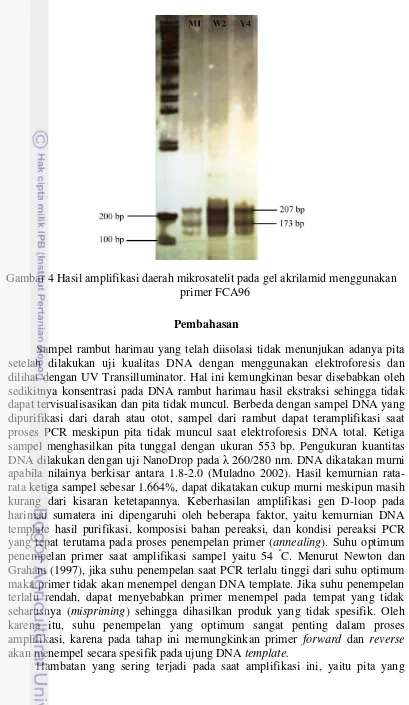
4 Hasil amplifikasi daerah mikrosatelit pada gel akrilamid menggunakan
primer FCA96 8
DAFTAR LAMPIRAN
1 Hasil BLAST-N gen sekuen D-loop 13
2 Pensejajaran berganda nukleotida (419 nt) pada gen D-loop sampel M1,
W2, Y4, dan A1 14
3 Tahapan dan komposisi pewarnaan perak (silver staining) 16
PENDAHULUAN
Harimau (Panthera tigris) adalah salah satu mamalia yang termasuk keluarga kucing (Famili Felidae). Harimau merupakan spesies kucing besar yang terbagi menjadi beberapa sub-spesies yaitu : harimau benggala (Panthera tigris tigris), harimau kaspia (Panthera tigris virgata), harimau amur (Panthera tigris altaicaa), harimau jawa (Panthera tigris sondaica), harimau china selatan (Panthera tigris amoyensis), harimau bali (Panthera tigris balica), harimau sumatera (Panthera tigris sumatrae) dan harimau indo-china (Panthera tigris corbetti) (Nowell dan Jackson 1996).
Harimau sumatera (Panthera tigris sumatrae) ialah salah satu satwa langka kebanggaan yang berasal dari Pulau Sumatera dan merupakan warisan kekayaan alam Indonesia yang saat ini masih tersisa. Penurunan populasi terjadi akibat perburuan liar, kerusakan habitat yang disengaja seperti pembukaan hutan dan peladangan berpindah, pengurangan luas habitatnya maupun bencana alam. Hal ini merupakan ancaman bagi satwa liar yang dapat menyebabkan kepunahan. Status populasi harimau sumatera berada pada tingkat yang sangat mengkhawatirkan (critically endangered) (IUCN 2008).
Survey populasi harimau sumatera dimulai pada tahun 1978 pada 26 kawasan terlindungi dengan jumlah populasi sekitar 1000 ekor. Pada tahun 1985 kembali dilakukan survey dengan jumlah populasi harimau di Sumatera sekitar 800 ekor di 26 kawasan yang sama. Menurut data terakhir diperkirakan jumlah harimau sumatera hanya tinggal 400-650 ekor di lima Taman Nasional dan dua Suaka Margasatwa yang ada di pulau Sumatera (Dirjen PHPA 1994).
Ancaman terhadap kelangsungan hidup suatu populasi harimau sumatera bukan hanya berasal dari faktor eksternal (lingkungan) tetapi juga berasal dari faktor internal (genetik). Semakin menurunnya jumlah populasi harimau sumatera juga menyebabkan menurunnya keragaman genetik populasi. Keragaman genetik rendah akan membahayakan kelestarian suatu spesies (Muazin et al. 2012). Pengkajian keragaman genetik sangat bermanfaat karena selain mencerminkan struktur genetik saat ini, juga dapat digunakan untuk menyusun langkah penyelamatan populasi dan pertimbangan untuk menetapkan strategi konservasi. Pengkajian keragaman genetik melalui penandaan molekuler menggunakan DNA, baik pada DNA inti dan DNA mitokondria (mtDNA) akan didapatkan hasil yang dapat mengungkapkan perbedaan genetik dengan lebih teliti. Perbedaan pada tingkat intra dan interspesies ini menyangkut tentang struktur, komposisi, dan organisasi genom pada tingkat DNA (Duryadi 1994).
Berdasarkan jenis gennya, genom mitokondria dibagi menjadi dua bagian, yaitu daerah penyandi (coding region) dan daerah bukan penyandi (non coding region) (Stansfield et al. 2006). Daerah penyandi terdiri atas 37 gen yaitu 13 gen penyandi protein yang berperan penting di dalam transport elektron dan fosforilasi oksidatif, dua gen penyandi ribosomal ribonucleic acid (rRNA), dan 22 gen penyandi transfer RNA (tRNA). Daerah bukan penyandi genom mitokondria hanya terdiri atas control region atau displacement loop (D-loop) (Reyes et al. 1998).
2
dari mtDNA secara umum. D-loop merupakan bagian dari mtDNA yang sangat variatif dalam substitusi nukleotida, insersi atau delesi, dan memiliki variable number tandem repeat (VNTRs) yang dinamis yang terletak pada bagian yang hipervariatif dan domain yang khusus (Fumagalli et al. 1996). Daerah D-loop dibagi menjadi tiga domain, yaitu hypervariable segment I (HVS-I), conserved sequence block (CSB), dan hypervariable segment II (HVS-II). DNA mitokondria terutama daerah D-loop, sangat baik digunakan untuk analisis hubungan kekerabatan sampai tingkatan intraspesies karena memiliki mutasi yang tinggi (Widayanti 2006).
Penanda genetik lain yang dapat digunakan untuk studi keragaman genetik selain penanda mitokondria ialah penanda mikrosatelit. Mikrosatelit merupakan rangkaian molekul DNA pendek yang susunan basanya berulang dalam bentuk salinan berdampingan (tandem), dan terdapat melimpah dalam genom eukariot. Mikrosatelit memiliki jumlah yang sangat banyak dan lokasinya tersebar di hampir semua kromosom sehingga sangat ideal untuk menganalisis fenotipe hewan. Mikrosatelit bersifat sangat polimorfisme sehingga sangat ideal untuk analisis keterpautan. Keterpautan mikrosatelit dengan daerah penyandi genom yang runutan DNA-nya cenderung conserved dapat dimanfaatkan untuk perbandingan peta genetik antar spesies (Muladno 2000). Mikrosatelit mendeteksi keragaman alel, memiliki tingkat variabilitas yang tinggi, dan mudah diaplikasikan melalui teknik PCR sehingga menjadi penanda molekul yang paling diminati oleh ahli genetika untuk mempelajari struktur genetik dan paternitas suatu populasi.
Tujuan Penelitian
Penelitian ini bertujuan menganalisis pedigri berdasarkan karakterisasi genetik menggunakan metode non-invasif pada harimau sumatera dengan marka daerah D-loop dan mikrosatelit.
METODE
Penelitian dilaksanakan di Laboratorium Biologi Molekuler PPSHB IPB dan Laboratorium Terpadu Departemen Biologi IPB, Bogor. Penelitian dimulai pada bulan Juni 2012 hingga September 2013.
Bahan
Bahan yang digunakan dalam penelitian ini antara lain rambut harimau sumatera (Panthera tigris sumatrae) sebagai sumber DNA yang diperoleh dari Kebun Binatang Bandung, diambil tiga sampel individu, yaitu induk jantan, betina dan anak (backcrossing). Bahan ekstraksi dan purifikasi DNA yang digunakan yaitu incubation buffer, DTT, proteinase K, lysis buffer (LA), etanol absolut, buffer AW1, buffer AW2, dan elution buffer. Bahan yang digunakan untuk uji kualitas DNA adalah agarosa, TBE 1x, dan etidium bromide (EtBr). Bahan untuk PCR yaitu ddH2O, buffer, enhancer, dNTP, taq polimerase, primer forward, dan
3 Isolasi DNA
Rambut harimau sumatera sebanyak 10 helai dipotong-potong ± 0.5 – 1 cm dan dimasukkan ke dalam tabung effendorf 1.5 mL. Kemudian ditambahkan incubation buffer (+ 1µL DTT) sebanyak 100µ L dan diinkubasi pada suhu 38 ºC selama 2 jam. Setelah itu, ditambahkan incubation buffer lengkap (incubation buffer, 1 µ L DTT dan proteinase K) sebanyak 100 µl dan diinkubasi semalam pada suhu 56 ºC.
Suspensi yang telah diinkubasi pada suhu 56 ºC selama semalam ditambahkan 100 µ L lysis buffer, kemudian dikocok manual pada suhu ruang selama 30 menit. Setelah itu sampel diinkubasi selama 15 menit pada suhu 70 ºC dan di swing menggunakan sentrifuse dengan kecepatan tinggi. Supernatan dipindahkan ke tube baru, ditambahkan 500 µ L etanol absolut, kemudian disimpan di freezer selama 2 jam. Sampel dipindahkan ke tube baru yang menggunakan saringan (spin column), dan disentrifugasi pada kecepatan 8000 rpm selama 1 menit kemudian cairannya dibuang. Sampel ditambahkan 500 µL buffer AW1, dan disentrifugasi pada kecepatan 8000 rpm selama 1 menit, supernatan dibuang. Sampel ditambahkan 700 µ L buffer AW2, dan disentrifugasi pada kecepatan 8000 rpm selama 1 menit, supernatan dibuang. Kemudian sampel disentrifugasi pada kecepetan 14000 rpm selama 3 menit untuk mengeringkan membran. Spin column ditempatkan pada tube baru, ditambahkan 50 µ L elution buffer, diinkubasi selama 15 menit, dan disentrifugasi pada kecepatan 14000 rpm selama 1 menit. Sampel DNA disimpan dalam freezeer sampai akan digunakan pada tahap selanjutnya.
Kualitas DNA dilihat dengan dimigrasikan pada gel agarosa 1.2% menggunakan buffer 1xTAE. Pewarnaan gel agarosa dengan EtBr (0.8% g/mL) diamati dibawah sinar ultra violet (λ= 200-400 nm). Kuantitas dan kemurnian DNA dapat diukur melalui uji spektrofotometri atau dengan NanoDrop, dengan rasio serapan pada panjang gelombang (λ) 260/280 nm.
Amplifikasi dan Visualisasi Fragmen DNA
Amplifikasi dilakukan melalui teknik PCR dengan menggunakan mesin thermocycler (ESCO). Fragmen DNA yang diamplifikasi yaitu fragmen D-loop pada harimau sumatera. Amplifikasi fragmen DNA daerah D-Loop dilakukan dengan menggunakan primer DHF dengan urutan nukleotida 5’ TAGCCCCACCATCAGCACCCAAAGC 3’ dan primer DHR dengan urutan nukleotida 5’ AATGGGCCCGGAGCGAGAAGAGGTA 3’.
Larutan pereaksi pada penelitian ini menggunakan Biolab Q5-HF. Total campuran untuk tiap reaksi PCR adalah 25 µL dengan komposisi yang terdiri atas 5 µL DNA template, masing-masing 1 µ L primer forward dan primer reverse dengan konsentrasi 10 pmol, 6.8 µL ddH2O, 5 µl 5X buffer Q5, 5 µ L 5X enhancer,
4
menggunakan buffer 1xTAE. Pewarnaan gel agarosa dengan EtBr (0.8% g/ml) divisualisasi menggunakan UV Trans-Iluminator.
Proses PCR pada mikrosatelit sama seperti pada D-loop, yang berbeda hanya pada primer yang digunakan. Amplifikasi fragmen DNA pada mikrosatelit menggunakan pasangan primer FCA 96 (Moreno et al. 2006). Urutan nukleotida primer FCA 96 (forward) 5’ CACGCCAAACTCTATGCTGA 3’ dan primer FCA 96 (reverse) 3’ CAATGTGCCGTCCAAGAAAC 5’ dengan kondisi suhu penempelan (annealing) 53 ºC. Hasil amplifikasi produk PCR mikrosatelit dilihat dengan elektroforesis gel poliakrilamid 5% dengan komposisi yang terdiri atas 19 mL aquades, 10 x TBE 2.5 mL, akrilamid 3.5 mL, APS 110 µL, dan Temed sebanyak 10 µL. Elektroforesis dilakukan dengan tegangan 85 volt selama 240 menit, kemudian dilanjutkan dengan pewarnaan perak (silver staining) untuk melihat keberadaan pita (Lampiran 3).
Perunutan Produk PCR (Sekuensing)
Prinsip analisa sekuensing DNA berbasis pada metode Sanger (1977) dan Maxam-Gilbert (Applied Biosystem 2012). DNA sekuensing menggunakan metode PCR (Polymerase Chain Reaction) sebagai pijakannya. DNA hasil PCR yang akan ditentukan urutan basanya (A,C,T,G) dijadikan sebagai cetakan (template). Produk PCR D-Loop ini berupa pita tunggal yang berukuran 553 bp. Proses amplifikasi menggunakan enzim dan bahan-bahan yang hampir sama dengan reaksi PCR, namun ada penambahan beberapa pereaksi tertentu. Proses ini dinamakan cycle sequencing.. Sekuensing dilakukan di PT Genetika Science.
Pensejajaran Runutan Nukleotida (Alignment) dan Analisis Filogeni Pensejajaran runutan nukleotida dilakukan dengan menggunakan ClustalW pada software MEGA 4.0. Hasil pensejajaran dicari homologinya dengan database Genbank menggunakan program BLAST-N (www.ncbi.nlm.niv.gov). Pohon filogeni dikonstruksi menggunakan program Neighbor-Joining Tree Method dengan nilai bootstrap 1000X (Tamura et al. 2007). Sekuen Panthera tigris sumatrae yang berasal dari Aceh (kode sampel: A1, koleksi Laboratorium Biologi Molekuler PPSHB LPPM IPB) digunakan sebagai sekuen acuan untuk membandingkan (multiple alignment) daerah D-loop parsial bagian HVS-I.
HASIL DAN PEMBAHASAN
Hasil Informasi Pedigri Sampel Harimau Sumatera
5
Isolasi DNA Total
Kualitas DNA total dilihat dengan dimigrasikan pada gel agarose 1.2% dan divisualisasikan dengan menggunakan UV Trans-Iluminator, dan uji NanoDrop dengan rasio serapan pada λ 260/280 nm (Tabel 1).
Amplifikasi Daerah D-loop
Amplifikasi daerah D-loop mtDNA pada tiga sampel harimau sumatera dengan primer DHF dan DHR menghasilkan pita tunggal berukuran 553 pb. Produk PCR tersebut selanjutnya digunakan dalam proses sekuensing. Gambar 1 menunjukan hasil amplifikasi daerah D-loop setelah dimigrasikan pada gel agarosa 1.2 % dan dilihat dengan UV transilluminator.
Tabel 1 Hasil kemurnian dan kuantitas DNA dengan uji NanoDrop Sampel Kode
Sampel ng/µl 260/280
1 M1 1126 26.3 1.656
2 W2 953 23.4 1.621
3 Y4 IN1066 11.9 1.714
6
Gambar 2 Hasil amplifikasi daerah D-loop pada gel agarosa 1.2%
Sekuens dan Alignment DNA P. t. sumatrae daerah D-Loop
Hasil pensejajaran dicari homologinya dengan menggunakan program N-BLAST. Hasil menunjukan bahwa fragmen tersebut benar merupakan control region (D-loop) dari harimau sumatera dengan persentasi kedekatan sebesar 99%. Produk PCR bagian parsial D-Loop harimau sumatera yang disekuen berada di daerah HVS-I sepanjang 419 bp dari hasil sekuen utuh sepanjang 553 bp.
Hasil pensejajaran (alignment) sekuen D-loop HVS-I, didapat 208 nukleotida dari ketiga sampel yang diteliti (M1, W2, dan Y4) yang berbeda dari sekuen acuan harimau sumatera dari Aceh (A1). Situs tersebut bersifat diagnostik sehingga dapat digunakan sebagai penciri bagi keluarga tersebut. Situs-situs tersebut di antaranya 2 delesi dan 24 insersi pada sekuens harimau sumatera. Situs nukleotida yang sama di antara individu W2 dan Y4, namun berbeda dari individu M1 maupun A1, terdapat 7 situs nukleotida. Kesamaan nukleotida antara individu M1 dan W2, tetapi bebeda dengan individu Y4 maupun A1, berjumlah 10 situs nukleotida.
Analisis Filogeni
Pohon filogeni dikonstruksi berdasarkan pairwise distance menggunakan program Neighbor-Joining Tree Method dengan nilai bootstrap 1000× (Tamura et al. 2007). Hubungan kekerabatan antara ke tiga individu sampel harimau sumatera dalam penelitian ini dapat dibandingkan berdasarkan jarak genetik (Tabel 2). Jarak genetik ketiga sampel berkisar antara 0.077-0.102. Individu M1 dengan W2, Y4, dan A1, masing-masing memiliki jarak genetik sebesar 0.077 dengan 34 nt berbeda, 0.102 dengan 44 nt berbeda, dan 0.555 dengan 244 nt berbeda. Individu W2 dengan Y4 dan A1, memiliki jarak genetik masing-masing 0.092 dengan 39 nt berbeda, dan 0.568 dengan 250 nt berbeda. Individu Y4 dengan A1 memiliki jarak genetik 0.550 dengan 242 nt berbeda.
500 bp
M1 W2 Y4
7
Rekontruksi pohon filogeni untuk mengetahui hubungan kekerabatan antara individu harimau sumatera berdasarkan jarak genetik (p-distance) dari basa-basa daerah D-loop bagian HVS-I dapat dilihat pada Gambar 3. Individu M1 berada satu kluster dengan W2. Hal ini menunjukan bahwa D-loop anaknya (M1) mirip dengan D-loop induknya (W2). Ketiga individu W2, M1, dan Y4 berada dalam satu kelompok dan berbeda dengan individu harimau Aceh A1 (berbeda > 50%).
Amplifikasi Gen Mikrosatelit
Amplifikasi daerah mikrosatelit dengan primer FCA 96 pada harimau sumatera menghasilkan pita berukuran 173 bp dan 207 bp. Kedua pita tersebut dimiliki oleh ketiga individu yang dibandingkan. Ibunya (W2) bergenotipe heterozigot 207/173 dan demikian pula dengan anaknya pada generasi pertama (F1) yaitu M1 yang bergenotipe 207/173 dan anaknya pada generasi kedua (F2) hasil backcross dengan induknya (W2) yaitu Y4, bergenotipe 207/173.
Tabel 2 Matrik rata-rata perbedaan nukleotida berdasarkan metode pairwise distance daerah D-loop bagian HVS-I pada harimau sumatra M1, W2, Y4, dan Aceh A1
8
Gambar 4 Hasil amplifikasi daerah mikrosatelit pada gel akrilamid menggunakan primer FCA96
Pembahasan
Sampel rambut harimau yang telah diisolasi tidak menunjukan adanya pita setelah dilakukan uji kualitas DNA dengan menggunakan elektroforesis dan dilihat dengan UV Transilluminator. Hal ini kemungkinan besar disebabkan oleh sedikitnya konsentrasi pada DNA rambut harimau hasil ekstraksi sehingga tidak dapat tervisualisasikan dan pita tidak muncul. Berbeda dengan sampel DNA yang dipurifikasi dari darah atau otot, sampel dari rambut dapat teramplifikasi saat proses PCR meskipun pita tidak muncul saat elektroforesis DNA total. Ketiga sampel menghasilkan pita tunggal dengan ukuran 553 bp. Pengukuran kuantitas DNA dilakukan dengan uji NanoDrop pada λ 260/280 nm. DNA dikatakan murni apabila nilainya berkisar antara 1.8-2.0 (Muladno 2002). Hasil kemurnian rata-rata ketiga sampel sebesar 1.664%, dapat dikatakan cukup murni meskipun masih kurang dari kisaran ketetapannya. Keberhasilan amplifikasi gen D-loop pada harimau sumatera ini dipengaruhi oleh beberapa faktor, yaitu kemurnian DNA template hasil purifikasi, komposisi bahan pereaksi, dan kondisi pereaksi PCR yang tepat terutama pada proses penempelan primer (annealing). Suhu optimum penempelan primer saat amplifikasi sampel yaitu 54 ºC. Menurut Newton dan Graham (1997), jika suhu penempelan saat PCR terlalu tinggi dari suhu optimum maka primer tidak akan menempel dengan DNA template. Jika suhu penempelan terlalu rendah, dapat menyebabkan primer menempel pada tempat yang tidak seharusnya (mispriming) sehingga dihasilkan produk yang tidak spesifik. Oleh karena itu, suhu penempelan yang optimum sangat penting dalam proses amplifikasi, karena pada tahap ini memungkinkan primer forward dan reverse akan menempel secara spesifik pada ujung DNA template.
9 dihasilkan multiband atau banyak pita yang bukan target. Hasil amplifikasi seringkali tidak konsisten, meskipun dengan komposisi dan kondisi yang sama. Hambatan tersebut dapat diatasi dengan melakukan modifikasi pada komposisi pereaksi dan kondisi PCR. Beberapa modifikasi di antaranya adalah menambahkan volume DNA template, mengurangi volume MgCl2, menaikkan
suhu penempelan sehingga dapat lebih spesifik.
Produk PCR D-loop harimau sumatera yang disekuen berada di daerah hypervariable segments I (HVS-I). Menurut Zhang et al (2006) diketahui bahwa daerah HVS-I pada D-loop mitokondria mempunyai variasi basa-basa nukleotida yang tinggi sehingga sangat cocok untuk membedakan antar individu baik dalam satu keluarga maupun antar keluarga. Hasil sekuensing pada produk PCR daerah D-loop bagian HVS-I pada ketiga sampel harimau sumatera dengan menggunakan primer forward DHF dan reverse DHR diperoleh hasil sekuen utuh sepanjang 553 pb. Desain primer meliputi daerah tRNA Pro, maka untuk melihat sekuen D-Loop saja daerah tRNA Pro harus dipotong sehingga panjang D-Loop utuhnya adalah 513 pb dan D-loop parsial bagian HVS-I adalah 419 bp.
Analisis keragaman basa-basa nukleotida daerah D-loop pada bagian HVS-I dilakukan dengan menambahkan data sekuen harimau yang berasal dari Aceh, yang merupakan data koleksi dari Laboratorium Biologi Molekuler PPSHB LPPM IPB. Basa nukleotida yang dibandingkan ialah 419 nt. Hasil multiple alignment tersebut didapat keragaman situs nukleotida di antara masing-masing individu dengan runutan nukleotida harimau aceh sebagai pembanding. Hal ini membuktikan bahwa D-loop bagian HVS-I merupakan daerah dengan basa nukleotida yang sangat variatif dan dapat mendeteksi variasi nukelotida yang lebih rendah pada individu dalam satu keluarga. Situs nukleotida yang bervariasi tersebut disebabkan terjadinya mutasi pada basa nukleotidanya. Mutasi yang terjadi di antaranya adalah mutasi substitusi transisi dan mutasi subtitusi transversi. Selain itu juga terjadi insersi, yaitu mutasi akibat penambahan atau adanya sisipan pasangan basa baru yang sebelumnya tidak ada, dan delesi yaitu pengurangan pasangan basa yang sebelumnya ada menjadi tidak ada.
Hubungan kekerabatan antara individu-individu sampel harimau sumatera dalam penelitian ini dapat dibandingkan berdasarkan matrik jarak genetik dari basa-basa nukleotidanya (Tabel 2). Hasil perbandingan pada Tabel 2 menunjukan individu-individu harimau sumatera yang berada dalam satu keluarga, memiliki jarak genetik yang lebih rendah dibandingkan harimau sumatera yang berasal dari Aceh (A1). Jarak genetik antara individu M1 dengan W2 ialah sebesar 0.077, M1 dengan Y4 memiliki jarak genetik 0.102, dan individu W2 dengan Y4 memiliki jarak genetik sebesar 0.092. Jarak genetik antara ketiga sampel dengan A1 ialah sebesar 0.550-0.568.
Hasil rekontruksi filogeni (Gambar 3) menunjukan ketiga sampel berada pada satu cluster yang sama (ingroup), dan terpisah cluster dengan harimau sumatera yang berasal dari Aceh (A1). Salah satu penyebab perbedaan cluster pada hasil rekonstruksi filogeni tersebut, kemungkinan karena harimau aceh dan harimau dari Kebun Binatang Bandung berasal dari daerah penyebaran geografi yang berbeda, meskipun masih termasuk ke dalam subspesies yang sama.
10
Backcross menyebabkan runutan nukleotida pada individu Y4 menjadi kembali ke awal, sebelum terjadinya mutasi pada basa-basa nukleotidanya (Frisch dan Melchinger 2005).
Amplifikasi pada mikrosatelit menggunakan pasangan primer (forward dan reverse) FCA 96, produk yang dihasilkan ialah pita berukuran 173 bp dan 207 bp. Penanda mikrosatelit digunakan secara luas sebagai penanda genetik didalam studi populasi dan verifikasi silsilah keturunan, terutama karena mikrosatelit mengandung informasi polimorfisme yang tinggi, tersebar luas di dalam genom eukariot (Tautz dan Renz 1984). Lokus-lokus pada mikrosatelit digunakan sebagai penanda untuk mengetahui hubungan evolusi antar populasi dan hubungan silsilah antar individu (Steven et al 2006).
Analisis gen mikrosatelit pada penelitian ini hanya sampai pada mengamati pita hasil elektoforesis untuk dianalisis variasi dan ukuran alelnya sehingga bisa dilihat alel yang diturunkan pada masing-masing individu. Satu pita yang muncul menunjukan alel homozigot, dan dua pita menunjukan alel yang heterozigot. Pita hasil pewarnaan (Gambar 3) menunjukan bahwa alel heterozigot yang ada pada induk jantan (M1) diturunkan pada anaknya (Y4), dan begitu juga alel heterozigot pada induk betina (W2) diturunkan kepada anaknya. Mengingat bahwa induk jantan dan betina memiliki hubungan ibu dan anak secara langsung (backcross), besar kemungkinan bahwa alel yang dimiliki induk jantan (M1) berasal juga dari induk betina (W2). Pita target yang dihasilkan pada ketiga individu memiliki ukuran yang sama, hal tersebut menunjukan bahwa individu M1, W2, Y4, dipastikan satu keluarga dan semua alel dari induk diturunkan pada anaknya.
SIMPULAN DAN SARAN
Simpulan
Karakterisasi genetik pedigri pada harimau sumatera dapat dianalisis dengan metode non-invasif menggunakan rambut dengan marka genetik daerah D-loop bagian HVS-I dan mikrosatelit. Berdasarkan analisis marka genetik daerah D-loop, terdapat 208 situs basa nukleotida yang berbeda dari harimau aceh (A1) yang bersifat diagnostik atau penciri dari keluarga harimau yang diteliti. D-loop daerah HVS-I cocok digunakan untuk membedakan antar individu dalam satu keluarga maupun antar keluarga. Lokus-lokus pada mikrosatelit dapat digunakan pula sebagai penanda hubungan silsilah antar individu, sehingga dapat dianalisis karakteristik pedigrinya dengan melihat komposisi alel-alel pada hasil amplifikasinya.
Saran
11
DAFTAR PUSTAKA
Applied Biosystems. 2012. DNA sequencing and fragment analysis by capillary electrophoresis. [internet]. [diunduh 2013 September 17]. Tersedia dari: http://www.appliedbiosystems.com.
Direktorat Jenderal Perlindungan Hutan dan pelestarian Alam. 1994. Ketentuan hukum tentang konservasi sumberdaya alam hayati. [internet]. [diunduh 2011 April 20]. Tersedia dari: http://www.dephut.go.id/files/PP13_1994.pdf.
Duryadi D. 1994. Peranan DNA mitokondria (mtDNA) dalam studi keragaman genetik dan biologi populasi pada hewan. Hayati 1(1):1-4.
Frisch M, Melchinger AE. 2005. Selection theory for marker-assisted backcrossing. Genetics 170:909-917. doi:10.1534/genetics.104.035451.
Fumagalli L, Taberlet P, Favre L, Hausser J. 1996. Origin and evolution of homologus repeated sequences in the mitochondrial DNA control region of shrews. Mol Biol Evol 13:31-46.
[IUCN] International Union for Concervation of Nature and Natural Resources. 2008. Panthera tigris ssp. sumatrae. IUCN Red List. [internet]. [diunduh 2011 Oktober 30]. Tersedia dari: http://www.redlist.org.
Moreno V, Grisolia A, Campagnari F, Milazzottp M, Adania C, Garcia J, Souza EB. 2006. Genetic variability of Herpailurus yagouaroundi, Puma concolor and Panthera onca (Mammalia, Felidae) studied using Felis catus microsatellites. Genet Mol Biol. 29(2):290-293. doi:10.1590/S1415-47572006000200017.
Muazin, Suastika P, Wandia IN. 2012. Polimorfisme lokus mikrosatelit DRB3 sapi bali di nusa dua penida. J Med Vet Indones. 1(5):621-635.
Muladno. 2000. Polimorfisme dan analisis keterpautan mikrosatelit pada genom babi. Hayati. 7(1):11-15.
_______. 2002. Seputar Teknologi Rekayasa Genetika. Bogor (ID): Pustaka Wirausaha Muda.
Newton CR, Graham A. 1997. PCR Introduction to Biotechnique. 2nd ed. Oxford (GB): ‘Bios Scientific Publisher Ltd.
Nowell K, Jackson P. 1996. Status survey and concervation action plan wild cats IUCN/SSC Cat Specialist Group. IUCN Gland. 54-65.
Reyes A, Gissi C, Pesole G, Saccone C. 1998. Asymmetrical directional mutation pressure in the mitochondrial genome of mammals. Mol Biol Evol. 15(8):957-966.
Sanger F, Nicklen S, Coulson AR. 1977. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci USA. 74:5463-5467.
Stansfield W, Cano R, Colome J.2006. Biologi Molekuler dan Sel. Amalia Safitri, editor. Terjemahan dari Molecular and Cell Biology. Jakarta (ID): Erlangga. Steven TK, Taper ML. 2006. Maximum likelihood estimation of the frequency of
null alleles at microsatellite loci. Conserv Genetics. 1-5. doi:10.1007/s10592.006.9134.9.
12
Tautz D, Renz M. 1984. Simple sequences are ubiquitous components of eukaryotics genomes. Nucleic Acids Res. 12(10):4127-4138.
Tumbelaka LITA. 2012. Studbook harimau sumatera regional Indonesia. Bogor (ID): Perhimpunan Kebun Binatang Se-Indonesia.
Widayanti R. 2006. Kajian penanda genetik gen Cytochrome B dan daerah D-loop pada Tarsius sp. [disertasi]. Bogor (ID): Institut Pertanian Bogor.
13 Lampiran 1 Hasil BLAST-N gen sekuen D-loop
Kode Aksesi Spesies
Max Score
Total Score
e-value Ident JF357970.1 Panthera tigris sumatrae
isolate SUMATRA2 mitochondrion, complete genom
994 1312 0.0 99%
DQ151550.1 Panthera tigris mitochondrion, partial genome
963 1268 0.0 98%
JQ040907.1 Panthera tigris voucher CH003 control region, partial sequence: mitochondrial
276 276 1e-70 95%
JQ040904.1 Panthera tigris voucher LA086 control region, partial sequence: mitochondrial
276 276 1e-70 95%
AY736629.1 Panthera tigris haplotype TIG6 control region, partial sequence: mitochondrial
14
Lampiran 2 Pensejajaran berganda nukleotida (419 nt) pada gen D-loop sampel M1, W2, Y4, dan A1
#MEGA
!Title Alignment M1 W2 Y4 A1; !Format
DataType=Nucleotide CodeTable=Standard NSeqs=4 NSites=419
Identical=. Missing=? Indel=-;
16
Lampiran 3 Tahapan dan komposisi pewarnaan perak (silver staining)
No Komposisi larutan Waktu Pewarnaan
1 0.2 gr CTAB 200 ml akuades
20 menit
2 200 ml akuades 15 menit
3 2.4 ml NH4OH (amoniak 25%)
200 ml akuades 20 menit
4 0.4 gr AgNO3
12 µl NaOH 200 ml akuades 20 menit
5 200 ml akuades 15 menit
6
4 gr Na2CO3
100 µl Formaldehid 200 ml akuades
sampai muncul band 7 0.2 ml as asetat
200 ml akuades
17 Lampiran 4 Gambar folikel rambut pada harimau sumatera
18
RIWAYAT HIDUP
Penulis dilahirkan di Garut pada tanggal 20 Desember 1990 dari pasangan Engkos Kosasih dan Nining Karningsih. Penulis merupakan anak ketiga dari empat bersaudara. Penulis menjalani pendidikan resmi di SDN Situ Jaya 1 Garut (1996-2002), SMPN 1 Garut (2002-2005), dan SMAN 12 Bandung (2005-2008). Tahun 2008, penulis diterima sebagai mahasiswa Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor melalui jalur Undangan Seleksi Masuk IPB (USMI).
Pada tahun 2010, penulis melakukan Studi Lapang di Taman Wisata Alam dan Cagar Alam Pangandaran Ciamis, Jawa Barat dengan judul Keberadaan Parasit pada Saluran Pencernaan Sapi Bali. Pada tahun 2011, penulis melakukan Praktik Lapang di Kebun Binatang Taman Sari Bandung dengan judul Konservasi Ex-situ Harimau Sumatera (Panthera tigris sumatrae) di Kebun Binatang Bandung.
Selama menjadi mahasiswi, penulis aktif dalam berbagai kegiatan kemahasiswaan. Penulis menjadi staff divisi Paguyuban Mahasiswa Biologi (Pamabi) Himpunan Mahasiswa Biologi (Himabio) pada tahun 2009-2010. Staff Sains dan Teknologi (Sainstek) Badan Eksekutif Mahasiswa (BEM) FMIPA pada tahun 2010-2011. Penulis juga aktif diberbagai kepanitiaan kegiatan kampus seperti Pesta Sains Nasional pada tahun 2009-2011, Olimpiade Mahasiswa IPB (OMI), IPB Art Contest (IAC), Pekan Ilmiah Mahasiswa FMIPA (PIPA), Biologi Interaktif, Bina Desa FMIPA IPB, Masa Pengenalan Departemen Biologi, dan Grand Biodiversity.