VARIASI GENETIK ROTAN BERDASARKAN PENANDA
DNA BARCODE matK, rbcL dan ITS PADA PANGKALAN
DATA GENBANK
MIRANTI ARUM PUTRI
DEPARTEMEN SILVIKULTUR
FAKULTAS KEHUTANAN
INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa skripsi berjudul Variasi Genetik Rotan
Berdasarkan Penanda
DNA Barcode
matK, rbcL
dan
ITS
Pada Pangkalan Data
GenBank adalah benar karya saya dengan arahan dari komisi pembimbing dan
belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber
informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak
diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam
Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut
Pertanian Bogor.
Bogor, Juli 2015
Miranti Arum Putri
ABSTRAK
MIRANTI ARUM PUTRI. Variasi Genetik Rotan Berdasarkan Penanda
DNA
Barcode
matK, rbcL
dan
ITS
Pada Pangkalan Data GenBank. Dibimbing oleh
ISKANDAR Z. SIREGAR.
Identifikasi jenis rotan biasa dilakukan dengan melihat karakteristik morfologi.
Seiring dengan berkembangnya ilmu pengetahuan dan teknologi, mulai diterapkan
ilmu biomolekuler sebagai alat pelengkap identifikasi jenis, salah satunya adalah
DNA barcoding
. Pada tanaman kehutanan keduanya masih jarang diterapkan
sebagai komplementer karena keterbatasan data molekuler. Penelitian ini
bertujuan untuk menganalisis struktur genetik tanaman rotan berdasarkan tiga
penanda yang ada pada pangkalan data genetik (NCBI) yaitu
matK
,
rbcL
dan
ITS
.
Pengolahan data dilakukan menggunakan perangkat lunak MEGA dan DnaSP
.
Dari hasil analisis diketahui bahwa penanda ITS memiliki sekuens yang lebih
panjang yaitu diatas 500 bp dan
GC content
(%) paling besar yaitu 63.1%.
Konstruksi filogenetik yang dilakukan belum menunjukkan penanda yang paling
akurat dalam mendiskriminasi spesies karena spesies yang berbeda masih
dikelompokkan dalam satu
clade.
Analisis variasi nukleotida dilakukan untuk
mengetahui lebih lanjut penanda yang mendekati akurat untuk membedakan
spesies. Secara keseluruhan belum dapat dipastikan penanda yang paling baik
untuk mendiskriminasi tanaman rotan.
Kata kunci: analisis molekuler,
DNA Barcode,
penanda genetik, rotan
.
ABSTRACT
MIRANTI ARUM PUTRI. Genetic Variation of Rattan Based
DNA Barcode
Marker
matK, rbcL
and
ITS
On Database GenBank. Supervised by ISKANDAR
Z. SIREGAR.
Species identification of rattan is usually done by assessing morphological
characteristics. Along with the development of science and technology,
biomolecular science began to be applied as a complementary tool identification,
one of which is DNA barcoding technique. In the forestry both are still rarely
applied as complementary to each other because of limited molecular data. This
study was aimed to analyze the genetic structure of various rattan by three DNA
Barcode markers that are available in the genetic database (NCBI), namely
matK
,
rbcL
and
ITS
. Data processing was performed using software MEGA and DnaSP.
The results showed that markers of ITS have a longer sequence that is above 500
bp and higher GC content (%) 63.1% compared to the others. Constructed
phylogeny trees do not show the most accurate marker in discriminating species
because different species are still grouped into one
clade
. Nucleotide variation
analysis was conducted to determine more accurate marker based on genetic
variation approach to differentiate Calamus and non-Calamus. However,
uncertainty in discriminating species was still observed.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar
Sarjana Kehutanan
pada
Departemen Silvikultur
VARIASI GENETIK ROTAN BERDASARKAN PENANDA
DNA BARCODE matK, rbcL dan ITS PADA PANGKALAN
DATA GENBANK
MIRANTI ARUM PUTRI
DEPARTEMEN SILVIKULTUR
FAKULTAS KEHUTANAN
INSTITUT PERTANIAN BOGOR
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah SWT atas segala
karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam
penelitian yang dilaksanakan sejak bulan Januari 2015 ini ialah
DNA
BARCODING
dengan judul Variasi Genetik Rotan Berdasarkan Penanda
DNA
Barcode
matK, rbcL
dan
ITS
Pada Pangkalan Data GenBank.
Terima kasih penulis ucapkan kepada Bapak Prof Dr Ir Iskandar Z. Siregar
MForSc selaku pembimbing. Di samping itu, penghargaan penulis sampaikan
kepada staff dan para peneliti di Laboratorium Silvikultur Bagian Genetika
Kehutanan yang telah memberi saran dan membantu dalam pelaksanaan penelitian
ini. Ungkapan terima kasih juga disampaikan kepada angkatan Departemen
Silvikultur tahun 2010, teman-teman Fakultas Kehutanan 47, senior dan junior di
Fakultas Kehutanan atas dukungannya dan kepada ayah, ibu, seluruh keluarga dan
saudara Prehadi atas segala doa dan kasih sayangnya.
Semoga karya ilmiah ini bermanfaat.
Bogor, Juli 2015
DAFTAR ISI
DAFTAR TABEL
vi
DAFTAR GAMBAR
vi
DAFTAR LAMPIRAN
vi
PENDAHULUAN
1
Latar Belakang
1
Tujuan Penelitian
2
Manfaat Penelitian
2
METODE
3
Alat
3
Bahan
3
Prosedur Analisis Data
3
HASIL DAN PEMBAHASAN
4
Struktur Genetik
4
Identifikasi efisiensi penanda menggunakan BLAST
6
Hubungan filogenetik
6
Variasi nukleotida
10
SIMPULAN DAN SARAN
11
Simpulan
11
Saran
11
DAFTAR PUSTAKA
11
LAMPIRAN
13
DAFTAR TABEL
1
Panjang sekuens basa (bp) pada ketiga penanda genetik
5
2
Persentase GC content (%) pada ketiga penanda genetik
5
3
Identifikasi efisiensi tiga penanda menggunakan BLAST
6
4
Variasi nukleotida genus Calamus dan Non Calamus pada ketiga
penanda
10
DAFTAR GAMBAR
1
Konstruksi filogenetik berdasarkan lokus matK
7
2
Konstruksi filogenetik berdasarkan lokus rbcL
8
3
Konstruksi filogenetik berdasarkan lokus ITS
9
DAFTAR LAMPIRAN
1
Hasil pengurutan basa nukleotida (
sequencing
) rotan pada penanda
matK
13
2
Hasil pengurutan basa nukleotida (
sequencing
) rotan pada penanda
rbcL
26
PENDAHULUAN
Latar Belakang
Rotan termasuk ke dalam family Palmae yang tumbuh memanjat dan banyak
tersebar di beberapa daerah beriklim tropis sampai sub tropis seperti Afrika, India,
Srilanka, kaki pegunungan Himalaya, China bagian Selatan, Malaysia, Indonesia,
Pasifik bagian Barat sampai Fiji. Rotan yang ditemukan di seluruh dunia tersebut
digolongkan berdasarkan marga, saat ini terdapat 15 marga rotan.
Keanekaragaman jenis rotan di Indonesia sendiri mencapai sekitar 306 jenis
dimana terdapat 40 jenis rotan yang bernilai ekonomi penting. Beberapa jenis
rotan banyak dijumpai tumbuh liar di hutan Indonesia ataupun dibudidayakan oleh
manusia untuk kepentingan ekonomi (Dransfield dan Manokaran 1993). Rotan
yang umum dijumpai di Indonesia yaitu berasal dari marga
Calamus, Calospatha,
Ceratolobus,
Daemonorops,
Korthalsia,
Myrialepis,
Plectocomia
dan
Plectocomiopsis.
Identifikasi jenis rotan biasanya dilakukan dengan melihat karakteristik
morfologi berupa jumlah batang per rumpun, sistem perakaran, bentuk alat
pemanjat, bentuk perkembangan dari daun, bunga dan buah (Dransfield dan
Manokaran 1993). Terdapat kelemahan tersendiri dalam mengidentifikasi jenis
rotan melalui visual karakteristik morfologi yaitu diperlukan ahli taksonomi atau
sumberdaya manusia yang berpengalaman karena terdapat beberapa jenis rotan
yang memiliki karakteristik morfologi yang sama sehingga apabila identifikasi
dilakukan oleh pemula, hasil identifikasi cenderung subjektif. Seiring dengan
berkembangnya ilmu pengetahuan dan teknologi, mulai dikenal ilmu
biomolekuler untuk makhluk hidup, salah satunya yang banyak dikembangkan
adalah
DNA
barcoding
. DNA Barcoding adalah penggunaan region DNA standar
berukuran pendek, sebagai penanda untuk identifikasi spesies yang cepat dan
akurat (Valentini 2009). Teknik
DNA barcoding
dapat mengidentifikasi dan
membedakan suatu organisme mulai tahap spesies hingga sub spesies.
Keunggulan teknik
DNA barcoding
yaitu dapat digunakan untuk identifikasi dan
karakterisasi berbagai spesies yang tidak dapat dibedakan secara morfologi
(Tudge 2000). Teknik
DNA barcoding
dapat digunakan utuk identifikasi suatu
organisme walaupun DNA dari organisme tersebut tidak dalam bentuk murni atau
utuh, bahkan DNA yang sudah mengalami degradasi dan proses pengolahan pun
dapat digunakan untuk analisis
DNA barcoding
(Hajibabaei
et al.
2006).
Pengenalan berbagai tanaman dapat dilakukan dengan hanya mengambil sedikit
bagian saja dari tubuh tanaman, tanpa harus mengamati morfologinya. Cara ini
dianggap cukup sederhana karena tidak diperlukan banyak spesimen dari lapang,
akan tetapi dibutuhkan keterampilan pada saat melakukan analisis di laboratorium.
2
ini bersifat stabil, dapat terdeteksi pada semua jaringan tanpa dipengaruhi oleh
status pertumbuhan, diferensisai, perkembangan maupun sistem pertahanan sel
(Mondini
et al
2009).
Menurut Yu
et al
(2011), kesulitan dalam memilih gen spesifik untuk
digunakan sebagai
DNA barcoding
tanaman disebabkan oleh ketidaksempurnaan
dari setiap gen pada tanaman, baik kloroplas, mitokondria atau inti genom. Gen
yang terdapat pada mitokondria tanaman perkembangannya lambat, sehingga
tidak efektif untuk membedakan antar spesies tanaman yang berbeda. CBOL
(
Consortium Barcode of Life
) dari kelompok kerja tanaman merekomendasikan
pemakaian gen pada kloroplast yaitu ribulosa-1, 5-bifosfat karboksilase
oksigenase subunit besar (
rbcL
) dan maturase K (
matK
) sebagai
barcode
standar
pada tanaman (CBOL 2009). Penanda genetik yang biasa digunakan peneliti
untuk tanaman adalah marka
rbcL
(
ribulose 1,5-biphospate carboxylase large
subunit
),
matK
(
megakaryocyte-associated tyrosine kinase
) dan
ITS
(
internal
transcribed spacer
).
Sejauh ini sudah tersedia database tanaman rotan yang terdapat di GenBank,
khususnya penanda
rbcL
,
matK
maupun
ITS
yang berfungsi sebagai
DNA
Barcode
. Walaupun demikian masih terdapat masalah dari ketiga penanda
tersebut yaitu belum diketahui jenis penanda yang paling baik dan tepat untuk
mendiskrimasi suatu spesies.
Penelitian ini mencakup kegiatan koleksi dan pengolahan seluruh data genetik
rotan yang ada pada GenBank (
http://www.ncbi.nlm.nih.gov/
). Data genetik
tersebut akan diolah untuk mendapatkan suatu pohon filogeni berdasarkan
masing-masing penanda sehingga dapat dilakukan analisis penanda yang baik
untuk mendiskriminasi spesies.
Tujuan Penelitian
Tujuan dilakukannya penelitian ini adalah untuk:
1.
Mengeksplorasi ketersediaan
DNA Barcode
(
matK, rbcL
dan
ITS
) untuk tanaman
rotan pada pangkalan data GenBank.
2.
Menganalisis variasi genetik berdasarkan ragam parameter pada masing-masing
penanda tersebut untuk diskriminasi spesies.
Manfaat Penelitian
Manfaat dilakukannya penelitian ini adalah:
1.
Memberikan informasi tentang ketersediaan data sekuens rotan yang terdapat pada
pangkalan data GenBank.
3
METODE
Waktu dan Tempat
Penelitian dilakukan pada Bulan Januari-April 2015. Analisis dan
pengolahan data dilakukan di Laboratorium Silvikultur, Bagian Genetika Hutan,
Departemen Silvikultur, Fakultas Kehutanan, Institut Pertanian Bogor.
Alat
Alat yang digunakan dalam penelitian ini berupa perangkat lunak MEGA
(
Molecular Evolutionary Genetic Analysis
) 6.0 dan DnaSP (
DNA Sequence
Polymorphism
) (Tamura
et al.
2011).
Bahan
Bahan yang digunakan dalam penelitian ini berupa koleksi data genetik
rotan (
matK, rbcL
dan
ITS
) yang berada pada pangkalan data GenBank
Internasional (
http://www.ncbi.nlm.nih.gov/
). Data yang diambil adalah data rotan
dari genus
Calamus, Ceratolobus, Daemonorops, Korthalsia, Myrialepis
dan
Plectocomia.
Data sekuens masing-masing penanda terlampir pada Lampiran 1, 2
dan 3.
Prosedur Analisis Data
Struktur Genetik
Analisis dilakukan pada data tanaman rotan yang didapatkan dari database
GenBank. Data yang digali (
data mining
) merupakan data hasil
DNA barcoding
tiga penanda genetik yang dapat digunakan pada tanaman yaitu
matK
,
rbcL
dan
ITS
. Data yang didapatkan diolah menggunakan program MEGA (
Molecular
Evolutionary Genetic Analysis
) 6.0. Program MEGA memiliki
tools
yang mampu
bekerja dalam pembacaan urutan DNA, analisis statistik DNA baik urutan basa
nukleotida atau protein dan penjajaran urutan satu sampel dengan sampel lainnya
menggunakan
ClustalW
(Kumar
et al.
2008).
4
Identifikasi efisiensi penanda menggunakan BLAST
Identifikasi efisiensi penanda dilakukan untuk mengetahui keakuratan
suatu penanda dalam membedakan spesies. Analisis ini dilakukan dengan
menggunakan
tools
yang terdapat pada pangkalan data NCBI (
National Center for
Biodtechnology Information
) yaitu BLAST (
Basic Local Alignment Search Tools
)
dengan memasukkan data sekuens setiap spesies ke dalam kolom yang terdapat
pada laman web BLAST yang kemudian akan terverifikasi persen kekerabatan
suatu spesies.
Hubungan filogenetik
Rekonstruksi filogenetik mampu menganalisa jarak gen melalui variasi
DNA dengan metode
Neighbor Joining Tree
yang mampu memperhitungkan
jarak kedekatan yang ditunjukkan dengan nilai
bootstrap
(Ward
et al.
2008).
Analisis dilakukan menggunakan program MEGA 6.0 dengan menggunakan
tools
> analysis phylogeny > construct/test Neighbor Joining Tree(s).
Variasi nukleotida genus Calamus dan Non Calamus
Analisis ini dilakukan untuk mengetahui keragaman pada masing-masing
penanda agar dapat diketahui penanda yang baik untuk diskriminasi spesies.
Program yang digunakan adalah DnaSP 5 (
DNA Sequence Polymorphism
).
HASIL DAN PEMBAHASAN
Struktur Genetik
Hasil analisis data genetik penanda molekuler
matK
,
rbcL
dan
ITS
pada
database GenBank, didapatkan sebanyak 43 spesies dari 89 individu pada lokus
matK
, 50 spesies dari 95 individu pada lokus
rbcL
dan 24 spesies dari 58 individu
pada lokus
ITS
. Sekuens basa tiap individu kemudian dikelompokkan berdasarkan
spesies terlebih dahulu, kemudian dikelompokkan lagi berdasarkan
masing-masing genus untuk memudahkan analisis pada
software.
Data tersebut diolah
menggunakan aplikasi perangkat lunak MEGA (
Molecular Evolutionary Genetic
Analysis
) 6.0, dengan terlebih dahulu dilakukan
alignment
untuk menyejajarkan
urutan basa semua individu dan untuk memudahkan dilakukan
trim
yaitu
pemotongan atau penghapusan basa.
5
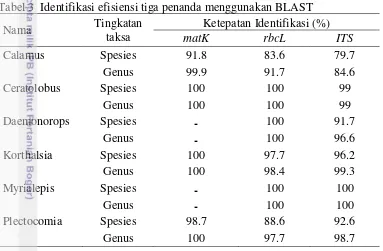
Tabel 1 Panjang sekuens basa (bp) pada ketiga penanda genetik
No.
Genus
matK
rbcL
ITS
n
bp
n
bp
n
bp
1
Calamus
82
462.1
84
507.8
20
690.6
2
Ceratolobus
1
496.0
1
514.0
2
751.0
3
Daemonorops
0
-
1
514.0
16
598.2
4
Korthalsia
1
523.0
2
514.0
11
668.3
5
Myrialepis
0
-
1
514.0
2
696.0
6
Plectocomia
4
523.0
5
514.0
6
671.2
Rata-rata
14.7
501.2
15.7
512.9
9.5
679.2
Keterangan: n = jumlah sekuensbp= base pair atau jumlah pasang basa
Tabel 1 menunjukkan pada penanda
matK
tidak terdapat data genus
Daemonorops
dan
Myrialepis.
Penanda
matK
memiliki rata-rata panjang sekuens
lebih kecil dibanding kedua penanda lainnya. Panjang basa pada
rbcL
keseluruhan
tidak berbeda secara signifikan, ini disebabkan pada masing-masing genus hanya
terdapat satu sekuens individu sehingga rata-rata sekuens genus sama dengan
panjang sekuens individu tersebut. Penanda
ITS
memiliki rata-rata panjang
sekuens basa paling besar diantara penanda lainnya yaitu di atas 500 bp. Panjang
sekuens ini berkorelasi dengan komposisi basa nukleotida pada sekuens basa.
GC content
atau komposisi basa
guanine
(G) dan
cytocyn
(C) berpengaruh
terhadap sekuens basa. Ikatan pada basa G+C lebih kuat dibanding ikatan pada
basa A+T (
adenine
+
thymyn
). Ikatan pada basa GC adalah tiga ikatan hidrogen
sedangkan pada AT adalah dua ikatan hidrogen, sehingga kemungkinan terjadinya
pelepasan basa pada GC lebih kecil dibanding pada AT karena ikatannya lebih
kuat. Semakin banyak persentase
GC content
maka sekuens basa semakin panjang
karena kemungkinan terjadinya stop kodon yang menyebabkan translasi basa
terhenti pun semakin kecil (Oliver 1996). Persentase GC content pada ketiga
penanda ditampilkan pada Tabel 2.
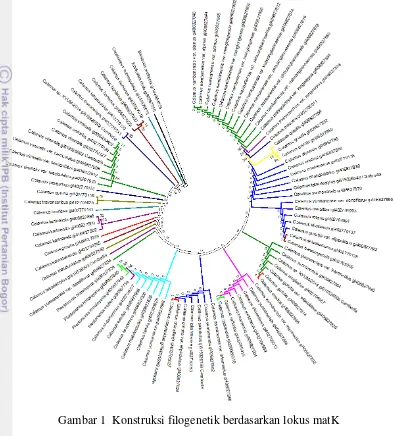
Tabel 2 Persentase GC content (%) pada ketiga penanda genetik
No.
Genus
GC Content (%)
Rata-rata
matK
rbcL
ITS
1
Calamus
35.8
43.9
42.7
40.8
2
Ceratolobus
64.3
43.7
55.8
54.6
3
Daemonorops
-
43.7
72.3
58.0
4
Korthalsia
65.0
43.9
70.5
59.8
5
Myrialepis
-
43.9
64.7
54.3
6
Plectocomia
64.6
43.9
66.1
58.2
6
Identifikasi efisiensi penanda menggunakan BLAST
Keakuratan identifikasi spesies dapat diketahui salah satunya dengan
metode BLAST (
Basic Local Alignment Search Tools
). Data sekuens basa yang
didapat kemudian diinput ke dalam
toolbox
pada laman web BLAST dan akhirnya
akan muncul spesies yang memiliki kekerabatan dengan spesies yang dimaksud.
Data kekerabatan masing-masing genus dihasilkan dalam bentuk presentase.
Semakin tinggi presentasenya atau mendekati 100% maka kekerabatannya
semakin dekat, sedangkan semakin rendah presentasenya berarti kekerabatan
semakin jauh atau berbeda sama sekali. Data hasil BLAST ketiga penanda
disajikan pada Tabel 3.
Tabel 3 Identifikasi efisiensi tiga penanda menggunakan BLAST
Nama
Tingkatan
taksa
Ketepatan Identifikasi (%)
matK
rbcL
ITS
Calamus
Spesies
91.8
83.6
79.7
Genus
99.9
91.7
84.6
Ceratolobus
Spesies
100
100
99
Genus
100
100
99
Daemonorops
Spesies
-
100
91.7
Genus
-
100
96.6
Korthalsia
Spesies
100
97.7
96.2
Genus
100
98.4
99.3
Myrialepis
Spesies
-
100
100
Genus
-
100
100
Plectocomia
Spesies
98.7
88.6
92.6
Genus
100
97.7
98.7
Tabel 3 menunjukkan tingkat keakuratan masing-masing penanda
matK
,
rbcL
dan
ITS
dalam membedakan spesies. Penanda
ITS
terlihat memiliki hasil
yang bervariasi untuk masing-masing genus sedangkan pada penanda
matK
dan
rbcL
tingkat keakuratan hampir 100%. Data yang didapat pada penanda
matK
dan
rbcL
belum dapat dikatakan akurat sepenuhnya karena ketika melakukan BLAST
banyak individu yang kekerabatannya 100% meskipun tertulis spesies yang
berbeda, ini disebabkan kemungkinan terjadinya kesalahan pada saat identifikasi
morfologi sehingga data spesies yang diinput kurang tepat. Selain itu karena
jumlah spesies pada
ITS
lebih bervariasi dibanding
matK
dan
rbcL
menyebabkan
nilai rata-rata keakuratan penanda pun bervariasi.
Hubungan Filogenetik
7
digunakan adalah
Neighbor Joining
(NJ) dengan nilai bootstrap sebesar 1000x
karena metode ini efektif untuk melakukan perhitungan tingkat kesamaan dalam
identifikasi spesies melalui kekerabatan (Ward
et al.
2008).
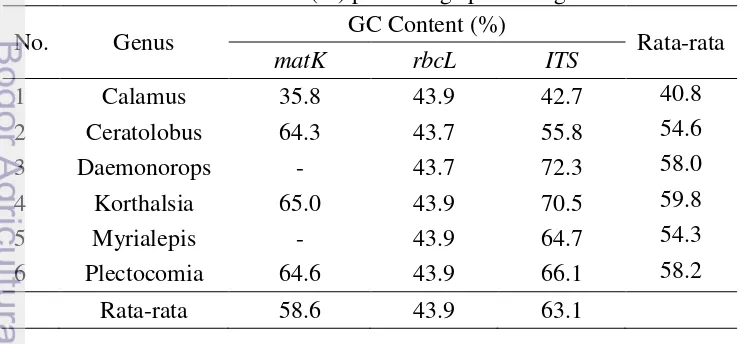
Gambar 1 menggambarkan hubungan kekerabatan spesies rotan pada lokus
matK
. Sekuens atau urutan basa dapat diketahui spesiesnya melalui percabangan
yang membentuk kelompok atau
clade
. Terdapat nilai bootstrap skala 1-100%
untuk mengetahui tingkat akurasi percabangan filogeni. Semakin tinggi nilai
bootstrap maka tingkat akurasi dan ketetapan posisi percabangan pohon filogeni
semakin tinggi.
Spesies
outgroup
diperlukan dalam konstruksi filogeni sebagai pembanding
dalam menentukan spesies. Spesies
outgroup
diperoleh dari GenBank
dengan
taksa yang tidak terlalu jauh atau terlalu dekat. Pembanding ditujukan agar
filogenetik yang terbentuk jelas dan kuat untuk mengklasifikasikan suatu
kekerabatan antar individu dan spesies.
8
Gambar 1 menggambarkan hasil terbentuknya 29 kelompok besar atau
clade
pada rotan berdasarakan lokus
matK
serta 1
clade
dari spesies
outgroup
yang
diambil dari genus
Bambusa
. Akan tetapi dari konstruksi filogeni yang terlihat,
matK
belum bisa membedakan spesies yang berbeda dengan baik, terbukti dengan
banyaknya spesies yang berbeda yang dikelompokkan menjadi satu
clade.
Contohnya pada
clade
berwarna biru di kanan atas,
Calamus aruensis, C. erectus,
C. hookerianus, C. rhabdocladus, C. tetradactylus
dan
C. yunnanensis
berada
dalam satu
clade
meskipun semuanya merupakan spesies yang berbeda.
Gambar 2 menggambarkan hubungan kekerabatan spesies rotan pada lokus
rbcL
. Konstruksi filogenetik yang terbentuk menghasilkan 12
clade
rotan serta 1
spesies
outgroup.
Seperti pada penanda
matK,
pada penanda
rbcL
pun masih
terdapat beberapa spesies yang dianggap mirip dengan spesies yang lainnya. Hal
ini ditunjukkan dari
clade
berwarna coklat di bagian kanan, dimana
Calamus
guruba, C. henryanus, C. karinensis, C. erectus, C. rhabdocladus, C. brandisii, C.
hookerianus, C. khasianus, C. moti, C. peregrinus, C. baratangensis, C.
stoloniferus, C. travancoricus, C. tenuis, C. karnatakensis, Korthalsia debilis,
9
Myrialepis paradoxa, Plectocomia himalayana
dan
Plectocomia elongate
dikelompokkan menjadi satu
clade.
Penanda
rbcL
pun belum dapat
mendiskriminasi spesies dengan baik.
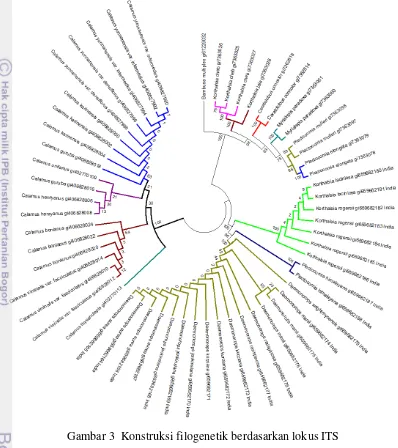
Gambar 3 menunjukkan hubungan kekerabatan spesies rotan pada lokus
ITS
.
Terbentuk 11
clade
rotan dan 1 spesies
outgroup.
Seperti kedua penanda lainnya,
pada penanda
ITS
pun masih ada spesies berbeda yang dikelompokkan ke dalam
satu
clade. Daemonorops manii, D. rarispinosa, D. kurziana, D. jenkinsiana, D.
aurea
dikelompokkan ke dalam satu
clade
berwarna coklat bagian bawah pada
Gambar 3.
Ketiga gambar tersebut menunjukkan penanda yang digunakan masih
belum cukup akurat dalam membedakan jenis karena spesies yang berbeda masih
dikelompokkan menjadi satu
clade
yang berarti tidak ada perbedaan genetik antar
spesies tersebut. Kemungkinan lainnya adalah adanya kesalahan pada saat
10
identifikasi secara morfologi masing-masing spesies sehingga kemungkinan
spesies yang sebenarnya adalah sama dianggap spesies yang berbeda.
Variasi nukleotida genus Calamus dan Non Calamus pada ketiga penanda
Variasi nukleotida dianalisis untuk mengetahui akurasi penanda dalam
membedakan spesies dengan melihat perubahan struktur genetik yang terjadi pada
sekuens. Parameter yang dapat dianalisis untuk mengetahui variasi nukleotida
diantaranya adalah jumlah site yang tersegregasi (S), jumlah insersi-delesi (indel),
jumlah
haplotype
(H),
haplotype diversity
(Hd) dan
nucleotide diversity
(π).
Analisis ini dilakukan dengan menggunakan
software
DnaSP 5 (Librado dan
Rozas 2009). Hasil analisis disajikan pada Tabel 4 untuk jenis
Calamus
dan
Non
Calamus
.
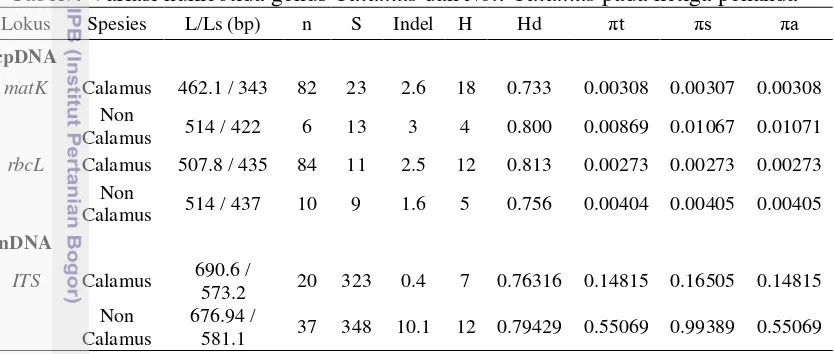
Tabel 4 Variasi nukleotida genus
Calamus
dan
Non Calamus
pada ketiga penanda
Lokus Spesies L/Ls (bp) n S Indel H Hd πt πs πa
cpDNA
matK Calamus 462.1 / 343 82 23 2.6 18 0.733 0.00308 0.00307 0.00308 Non
Calamus 514 / 422 6 13 3 4 0.800 0.00869 0.01067 0.01071
rbcL Calamus 507.8 / 435 84 11 2.5 12 0.813 0.00273 0.00273 0.00273 Non
Calamus 514 / 437 10 9 1.6 5 0.756 0.00404 0.00405 0.00405 nDNA
ITS Calamus 690.6 /
573.2 20 323 0.4 7 0.76316 0.14815 0.16505 0.14815
Non
Calamus
676.94 /
581.1 37 348 10.1 12 0.79429 0.55069 0.99389 0.55069 Keterangan: L/LS= panjang sekuens dengan gaps/jumlah silent site tanpa gaps; N= jumlah
sekuens;S= jumlah segregating site; Indel= jumlah terjadinya insersi-delesi; H= jumlah haplotype; Hd= haplotype diversity; πt= beda jumlah nukleotida per total site
dengan koreksi Jukes and Cantor (JC 1969) ; πs= beda jumlah nukleotida per silent
site dengan koreksi Jukes and Cantor (JC 1969); πa= beda jumlah nukleotida per
nonsynonymous site dengan koreksi Jukes and Cantor (JC 1969).
Tabel 4 menunjukkan bahwa pada ketiga penanda terjadi segregasi dan
ditemukan indel. Segregasi yang terbesar ditemukan pada penanda lokus ITS
sebesar 323 pada genus
Calamus
dan 348 pada
Non Calamus
. Nilai
nucleotide
diversity
atau keragaman nukleotida yang disimbolkan dengan π
tterbesar
ditemukan juga pada lokus ITS yaitu 0.14815 untuk genus
Calamus
dan 0,55069
untuk genus
Non Calamus
. Sedangkan pada penanda matK dan rbcL nilai π
t11
tersebut terdapat berbagai macam spesies yang berasal dari genus berbeda. Data
tersebut diatas menunjukkan bahwa penanda ITS memiliki potensi untuk
mendiskriminasi spesies lebih baik dibanding kedua penanda lainnya.
SIMPULAN DAN SARAN
Simpulan
Data GenBank berupa sekuens dari 6 genus tanaman rotan menunjukkan
lokus ITS memiliki sekuens yang lebih banyak dan lebih panjang dibanding lokus
matK dan rbcL. Panjang sekuens dari yang terpanjang hingga terpendek
berturut-turut adalah 679.2 bp pada ITS, 512.9 bp pada rbcL dan 501.2 bp pada matK.
Nilai persentase GC content dari mulai terbesar hingga terkecil adalah 63.1% pada
ITS, 58.6% pada matK dan 43.9% pada rbcL. Berdasarkan hasil pohon
filogenetik, ketiga penanda masih belum akurat dalam membedakan spesies
karena spesies yang berbeda masih dikelompokkan ke dalam satu
clade
. Dari hasil
analisis variasi nukleotida diketahui bahwa penanda ITS merupakan penanda yang
memperlihatkan parameter genetik yang lebih bervariasi dibanding kedua penanda
lainnya.
Saran
Dari ketiga penanda genetik tersebut masih belum dapat dipastikan yang
paling baik untuk membedakan spesies, maka dari itu perlu eksplorasi lebih lanjut
terhadap ketiga penanda tersebut. Data yang terdapat pada GenBank kurang
bervariasi, hanya terpusat pada beberapa spesies saja. Penelitian lebih lanjut
terhadap rotan yang terdapat di Indonesia dapat dilakukan agar database pada
GenBank lebih bervariasi dan bisa dijadikan referensi selanjutnya.
DAFTAR PUSTAKA
Bagali PG, Prabhu PDAH, Raghaedra K, Hittalmani S, Vadivelu JS. 2010.
Application of Molecular Markers in Plant Tissue Culture.
Asia-Pacific
Journal of Molecular Biology and Biotechnology
18 (1): 85-87.
CBOL-Plant Working Group. 2009.
A DNA barcode for land plants.
Proc Natl
Acad Sci USA. 106: 12794-12797.
Dransfield J, Manokaran N. 1993. Plant resources of South-East Asia 6: Rattans.
Wageningen, the Netherlands, Pudoc.
Hajibabaei M
et al.
2006. A minimalist barcode can identify a specimen whose
DNA is degraded.
J Compilation Blackwell Publishing.
6: 959-964.
Jukes TH, Cantor CR. 1969. Evolution of protein molecules In: Munro HN (ed)
Mammalian protein metabolism. Academic Press, New York, pp 21-132.
Kumar S, Nei M, Dudley J, Tamura K. 2008. MEGA: a biologistcentrisc software
for evolutionary analysis of DNA and protein sequences. Brief Bioinform.
9:299-306.
12
Mondini L, Noorani A, Pagnotta MA. 2009. Review: Assessing Plant Genetic
Diversity
by
Molecular
Tools.
Diversity
1:19-35.
DOI
:
10.3390/d1010019.
Nei M, Kumar S. 2000.
Molecular Evolution and Phylo
g
enetics.
New York:
Oxford University Press.
Oliver JL, Marin A. 1996. A Relationship Between GC Content and
Coding-Sequence Length. J Mol Evol 43:216-223.
Tamura K., Nei M., and Kumar S. 2004. Prospects for inferring very large
phylogenies by using the neighbor-joining method. Proceedings of the
National Academy of Sciences (USA) 101:11030-11035.
Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. 2011. Mega 5:
Molecular Evolutionary Genetics Analysis using maximum likelihood,
Evolutionary Distance and Maximum Parsimony Methods. Mol Biol
Evol.24:1596-1599.
Tamura K., Stecher G., Peterson D., Filipski A., and Kumar S. 2013. MEGA6:
Molecular Evolutionary Genetics Analysis version 6.0. Molecular Biology
and Evolution30: 2725-2729.
Tudge C. 2000.
The Variety Of Life.
New York: Oxford University Press.
Valentini A, Pompanon F, Taberlet P. 2009. DNA Barcoding for Ecologists.
Trends in Ecology and Evolution (24)2:110-117.
Ward RD, Holmes BH, White WT, Last PR. 2008. DNA barcoding Australasian
chondrichtyans: results and possible use in conservation. Mar. Freshwater
Res. 59, 57-71.
13
Lampiran 1 Hasil pengurutan basa nukleotida (
sequencing
) rotan pada penanda
matK
#Calamus yunnanensis var. intermedius gi406827892
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus yunnanensis var. intermedius gi406827890
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus yunnanensis var. intermedius gi406827888
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus yunnanensis var. densiflorus gi406827896
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus yunnanensis var. densiflorus gi406827894
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus yunnanensis gi406827870
14
#Calamus yunnanensis gi406827868
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus yunnanensis gi406827866
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus yunnanensis gi406827864
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus yunnanensis gi406827862
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus viminalis var. fasciculatus gi406827910
CACCATAATTTTTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAAATGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGC
#Calamus viminalis var. fasciculatus gi406827912
CACCATAATTTTTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAAATGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGC
#Calamus viminalis var. fasciculatus gi406827908
15
ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAAATGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGC
#Calamus viminalis gi619326562 Cambodia
CACCATAATTTTTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAAATGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGC
#Calamus viminalis gi402770127
CACCATAATTTTTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAAATGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGC
#Calamus viminalis gi386430650
CACCATAATTTTTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATTCATATAGACCAATTATCAAAATGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCAGGGCACCCTATTAGTAAGC
#Calamus vattayila gi402705015
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTTATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGC
#Calamus unifarius gi402770129
CACCATAATTTTTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAAATGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGC
#Calamus travancoricus gi402705025
16
TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus thwaitesii gi402704993
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus tetradactylus gi619328393 Cambodia
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus tetradactylus gi619326548 Cambodia
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus tetradactylus gi402770133
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGAGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus tenuis gi402705023
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus stoloniferus gi402705013
17
#Calamus sp. KYUM-2014 gi619328502 Cambodia
CACCATAATTTTTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAAATGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGC
#Calamus sp. KYUM-2014 gi619326504 Cambodia
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus salicifolius gi619326159 Cambodia
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus rotang gi402704991
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus rhabdocladus gi406827832
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus rhabdocladus gi406827830
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus rhabdocladus gi406827828
18
ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus platyacanthus var. longicarpus gi406827836
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATTCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCCATTAGTAAGCC
#Calamus platyacanthus var. longicarpus gi406827834
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATTCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCCATTAGTAAGCC
#Calamus peregrinus gi402770151
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGTATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATAATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus palustris gi402705011
CACCATAATTATTTTCAGAGGACCCTATGGGCCTTCAAGGATTCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus nambariensis var. xishuangbannaensis gi406827880
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATTCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCCATTAGTAAGCC
#Calamus nambariensis var. xishuangbannaensis gi406827878
19
#Calamus nambariensis var. xishuangbannaensis gi406827876
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATTCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCCATTAGTAAGCC
#Calamus nambariensis var. xishuangbannaensis gi406827874
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATTCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCCATTAGTAAGCC
#Calamus nambariensis var. xishuangbannaensis gi406827872
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATTCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCCATTAGTAAGCC
#Calamus nambariensis var. menglongensis gi406827886
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATTCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCCATTAGTAAGCC
#Calamus nambariensis var. menglongensis gi406827884
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATTCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCCATTAGTAAGCC
#Calamus nambariensis var. menglongensis gi406827882
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATTCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCCATTAGTAAGCC
#Calamus nambariensis var. alpinus gi406827848
20
ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCCATTAGTAAGCC
#Calamus nambariensis var. alpinus gi406827846
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATTCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCCATTAGTAAGCC
#Calamus nambariensis var. alpinus gi406827844
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATTCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCCATTAGTAAGCC
#Calamus metzianus gi402704989
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus longisetus gi402704987
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus lakshmanae gi402770153
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus khasianus gi402770137
21
#Calamus karnatakensis gi402704985
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus karinensis gi406827902
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTATGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus karinensis gi406827900
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTATGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus karinensis gi406827898
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTATGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus hookerianus gi402770135
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus henryanus gi406827906
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus henryanus gi406827904
22
ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus guruba var. elipsoideus gi406827826
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus guruba var. elipsoideus gi406827824
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus guruba var. elipsoideus gi406827822
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus guruba gi406827916
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus guruba gi406827914
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAAGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus guruba gi402770155
23
#Calamus gracilis gi406827854
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCCGTTATTCCTCTTATTGAATCA TTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCCATTAGTAAGCC
#Calamus gracilis gi406827852
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCCGTTATTCCTCTTATTGAATCA TTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCCATTAGTAAGCC
#Calamus gracilis gi406827850
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCCGTTATTCCTCTTATTGAATCA TTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCCATTAGTAAGCC
#Calamus erectus gi406827860
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus erectus gi406827858
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus erectus gi406827856
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus dransfieldii gi402705027
24
ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus delessertianus gi402770139
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus castaneus gi499069310
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus caryotoides gi384579289 Australia
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGTTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus brandisii gi402770141
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus bonianus gi406827922
CACCATAATTATTTTCAGAAGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus bonianus gi406827920
25
#Calamus bonianus gi406827918
CACCATAATTATTTTCAGAAGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus baratangensis gi402705009
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAAAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus aruensis gi90967755
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Calamus andamanicus gi402705007
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Ceratolobus subangulatus gi440579012
CACCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCATCCTATTAGTAAGCC
#Korthalsia cheb gi90967750
CGCCATAATTATTTTCAGAGGACCCTATGGTTCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTTTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAAA TGGAAATGTCACCTTGTCAATTTCTGGCAATATTATTTTCGCTTTTGGTCTCAACCGTA CAGGATCCATATAGACCAATTATATAACTGTTCTTTCTATTTTCTAGGTTATCTTTCAA GTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAAT GGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATCA TTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Plectocomia himalayana gi406827842
26
ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Plectocomia himalayana gi406827840
CGCCATAATTATTTTAAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Plectocomia himalayana gi406827838
CGCCATAATTATTTTAAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TCGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGGCACCCTATTAGTAAGCC
#Plectocomia mulleri gi90967754
CGCCATAATTATTTTCAGAGGACCCTATGGTCCTTCAAGGATCCTTTCATGCATTATGT TGGATATCAAGGAAAAGCAATTCTGGTTTCAAAGGGGGCTCATCTTCTGATGAAGAA ATGGAAATGTCACCTTGTCAATTTCTGGCAATATCATTTTCACTTTTGGTCTCAACCGT ACAGGATCCATATAGACCAATTATCAAACTGTTCTTTCTATTTTCTAGGTTATCTTTCA AGTGTATTAATAAATCTTTCGACGGTAAGGAATCAAATGCTAGAGAATTCATTTCTAA TGGATACTGTTACTAAAAAATTCGATACCAGAGTCCCAGTTATTCCTCTTATTGAATC ATTGTCTAAAGCTAAATTTTGTACCGTATCGGGACACCCTATTAGTAAGCC
#Bambusa multiplex gi144583575
TACCCAGGTTTTTTTCGGAAAACCGTATGGTTCTTTATGGATCCTCTTATGCACTATGT TCGATATCAAGGAAAGGCAATTCTTGCATCAAAAGGAACTCTTCTTTTGAAGAAGAA ATGGAAATGTTACCTTGTCAATTTCTGGCAATATTCTTTCTCTTTTTGGACTCAACCGC GAAGGATCCATCTAAACCAATTAGCAAACTCTTGCTTCGATTTTCTGGGGTACCTTTC AAGTGTACCAATAAATCCTTTGTTAGTAAGGAATCAAATGCTAGAGAATTCATTTCTA ATAGATACTCGAATGAAAAAATTCGATACCATAGTCCCCGCTACTCCCCTCATTCGAT CCTTATCAAAAGCTCAATTTTGTACTGGATCGGGGCATCCTATTAGTAAACC
Lampiran 2 Hasil pengurutan basa nukleotida (
sequencing
) rotan pada penanda
rbcL
#Calamus_thysanolepis_gi402770113
TTCCGAGTAACTCCTCAACCCGGAGTTCCGCCTGAGGAAGCAGGGGCAGCAGTAGCT GCCGAATCTTCTACTGGTACATGGACAACTGTGTGGACTGATGGACTTACCAGTCTTG ATCGTTACAAAGGACGATGCTACCACATCGAAACCGTTATCGGGGAGGAAAATCAAT ATATTGCTTATGTAGCTTATCCTTTAGACCTTTTTGAAGAAGGTTCTGTTACTAACATG TTTACTTCCATTGTGGGTAATGTATTTGGTTTCAAAGCCCTAAGAGCTCTACGTCTGGA GGATCTGCGAATTCCCACTTCTTATTCCAAAACTTTCCAAGGTCCGCCTCATGGCATCC AAGTTGAAAGAGATAAGTTGAACAAGTATGGTCGTCCTCTATTGGGATGT
#Calamus_unifarius_gi402770109
27
ATCGTTACAAAGGACGATGCTACCACATCGAAACCGTTATCGGGGAGGAAAATCAAT ATATTGCTTATGTAGCTTATCCTTTAGACCTTTTTGAAGAAGGTTCTGTTACTAACATG TTTACTTCCATTGTGGGTAATGTATTTGGTTTCAAAGCCCTAAGAGCTCTACGTCTGGA GGATCTGCGAATTCCCACTTCTTATTCCAAAACTTTCCAAGGTCCACCTCATGGCATCC AAGTTGAAAGAGATAAGTTGAACAAGTATGGTCGTCCTCTATTGGGATGT
#Calamus_bonianus_gi406828024
TTCCGAGTAACTCCTCAACCCGGAGTTCCGCCTGAGGAAGCAGGGGCAGCAGTAGCT GCCGAATCTTCTACTGGTACATGGACAACTGTGTGGACTGATGGACTTACCAGTCTTG ATCGTTACAAAGGACGATGCTACCACATCGAAACCGTTATCGGGGAGGAAAATCAAT ATATTGCTTATGTAGCTTATCCTTTAGACCTTTTTGAAGAAGGTTCTGTTACTAACATG TTTACTTCCATTGTGGGTAATGTATTTGGTTTCAAAGCCCTAAGAGCTCTACGTCTGGA GGATCTGCGAATTCCCACTTCTTATTCCAAAACTTTCCAAGGTCCGCCTCATGGCATCC AAGTTGAAAGGGATAAGTTGAACAAGTATGGTCGTCCTCTATTGGGATGT
#Calamus_bonianus_gi406828022
TTCCGAGTAACTCCTCAACCCGGAGTTCCGCCTGAGGAAGCAGGGGCAGCAGTAGCT GCCGAATCTTCTACTGGTACATGGACAACTGTGTGGACTGATGGACTTACCAGTCTTG ATCGTTACAAAGGACGATGCTACCACATCGAAACCGTTATCGGGGAGGAAAATCAAT ATATTGCTTATGTAGCTTATCCTTTAGACCTTTTTGAAGAAGGTTCTGTTACTAACATG TTTACTTCCATTGTGGGTAATGTATTTGGTTTCAAAGCCCTAAGAGCTCTACGTCTGGA GGATCTGCGAATTCCCACTTCTTATTCCAAAACTTTCCAAGGTCCGCCTCATGGCATCC AAGTTGAAAGGGATAAGTTGAACAAGTATGGTCGTCCTCTATTGGGATGT
#Calamus_bonianus_gi406828020
TTCCGAGTAACTCCTCAACCCGGAGTTCCGCCTGAGGAAGCAGGGGCAGCAGTAGCT GCCGAATCTTCTACTGGTACATGGACAACTGTGTGGACTGATGGACTTACCAGTCTTG ATCGTTACAAAGGACGATGCTACCACATCGAAACCGTTATCGGGGAGGAAAATCAAT ATATTGCTTATGTAGCTTATCCTTTAGACCTTTTTGAAGAAGGTTCTGTTACTAACATG TTTACTTCCATTGTGGGTAATGTATTTGGTTTCAAAGCCCTAAGAGCTCTACGTCTGGA GGATCTGCGAATTCCCACTTCTTATTCCAAAACTTTCCAAGGTCCGCCTCATGGCATCC AAGTTGAAAGGGATAAGTTGAACAAGTATGGTCGTCCTCTAT