KONSTRUKSI VEKTOR DAN EKSPRESI PROTEIN REKOMBINAN
LIPASE Candida antarctica (CALB) dengan Cellulose Binding
Module (CBM) pada Pichia pastoris
FEBRIANA DWI WAHYUNI
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Konstruksi Vektor dan Ekspresi Protein Rekombinan Lipase Candida antarctica (CALB) dengan
Cellulose Binding Module (CBM) pada Pichia pastoris adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir disertasi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Februari 2016
Febriana Dwi Wahyuni
RINGKASAN
FEBRIANA DWI WAHYUNI. Konstuksi Vektor dan Ekspresi Protein Rekombinan Lipase Candida antarctica (CALB) dengan Cellulose Binding Module
(CBM) pada Pichia pastoris. Dibimbing oleh SUHARSONO dan ASRUL MUHAMAD FUAD.
Gen CalBsyn telah dikonstruksi secara sintetik untuk menyandi Candida antarctica lipase B (CALB). Enzim tersebut memiliki peranan penting sebagai biokatalis yang efektif di bidang bioteknologi dan industri. Sekuens gen CalBsyn
telah dimodifikasi dengan mengintroduksi mutasi pada tiga asam amino, yaitu V210I, A281E, dan V221D untuk meningkatkan termostabilitas enzim tersebut. Fragmen gen cellulose binding module (cbm) endoglukanase I (egl1) dari
Trichoderma reesei difusikan pada ujung karboksil gen CalBsyn pada plasmid rekombinan pGAPZα-CalBsyn. CBMmerupakan suatu domain protein yang umum dijumpai pada enzim selulase dan berfungsi dalam pengikatan enzim pada matrik selulosa. Dalam penelitian ini CBM digunakan untuk memfasilitasi imobilisasi CALB pada matrik selulosa melalui fusi dari dua protein tersebut. Penelitian ini bertujuan mengkonstruksi vektor ekspresi gen CalBsyn yang difusi dengan cbm
dan mengekspresikan CALB pada Pichia pastoris SMD1168H. Baik plasmid maupun gen cbm dipotong dengan enzim ClaI. Plasmid rekombinan pGAPZα-CalBsyn-cbm dikonstruksi dengan cara meligasikan gen cbm ke dalam plasmid pGAPZα-CalBsyn. Plasmid rekombinan pGAPZα-CalBsyn-cbm diklon dan diseleksi pada Eschericia coli TOP10F’. Plasmid rekombinan pGPAZα-CalBsyn
telah berhasil diintroduksi ke dalam genom P. pastoris SMD1168H melalui metode elektroporasi. Efisiensi transformasi yang diperoleh sebesar 1,01 x 102 cfu/µg DNA. Analisis ekspresi protein rekombinan dilakukan terhadap beberapa galur rekombinan P. pastoris terpilih. Uji aktivitas lipase secara kualitatif pada media seleksi menunjukkan bahwa galur transforman P. pastoris yang diperoleh positif mensekresikan lipase rekombinan (CALB) dan memiliki aktivitas lipolitik. Uji aktivitas lipase secara kuantitatif dari salah satu galur transforman P. pastoris
menunjukkan bahwa aktivitas enzim mencapai 6,35 Units/ml pada jam ke-48. Analisis protein dengan SDS-PAGE menunjukkan bahwa CALB berhasil diekspresikan secara ekstraseluler dengan ukuran sekitar 45 kDa.
Kata kunci: Candida antarctica lipase B, cellulose binding module, recombinant
SUMMARY
CalBsyn gene was previously constructed synthetically encoding Candida antarctica lipase B (CALB). The enzyme has important role as the effective biocatalyst in biotechnology and industrial fields. Lipase from this CalBsyn gene (CALB-syn) is slightly different from wild type CALB (CALB-wt) in which it has three amino acids substitutions at different positions, including V210I, A281E, and V221D to improve thermal stability characteristics and catalytic efficiency of the enzyme. In order to immobilize the enzyme to any cellulose-based matrix, a cellulose binding module (cbm) fragment of endoglucanase-1 (egl1) from Trichoderma reesei was fused to the carboxyl end of the CALBsyn. CBM is a protein domain which is frequently found in many microbial cellulases that facilitate the binding of enzyme to cellulose matrix. This feature of cbm was used in this reseach to facilitate immobilization of CALB through fusion of both protein. This research was aimed to construct a fusion gene CalBsyn-cbm into pGPAZα expression vector and to study the expression of CALB in Pichia pastoris SMD1168H. Both plasmid and cbm gene were digested with ClaI restriction enzyme. pGAPZα-CalBsyn-cbm recombinant plasmid was constructed by ligation between cbm gene and pGAPZα -CalBsyn plasmid. pGAPZα-CalBsyn-cbm recombinant plasmid was cloned and
selected in Eschericia coli TOP10F’. pGAPZα-CalBsyn recombinant plasmid has been sccesfully introduced into P. pastoris SMD1168H using electroporation method. Transformation efficiency was 1,01 x 102 cfu/µg DNA. Recombinant protein expression was analyzed in several strains P. pastoris recombinant selected. Qualitative lipase activity assays showed that transformant P. pastoris secreted lipase recombinant (CALB) and has lipolitic activity. Quantitative lipase activity assays showed that activity of lipase was 63,5 Units/ml in 48 hours. Analysis with SDS-PAGE showed that CALB protein was expressed successfully and the recombinant protein showed a molecular size of approximately 45 kDa.
Keywords: Candida antarctica lipase B, cellulose binding module, recombinant
© Hak Cipta Milik IPB, Tahun 2016
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Bioteknologi
KONSTRUKSI VEKTOR DAN EKSPRESI PROTEIN REKOMBINAN
LIPASE Candida antarctica Lipase B (CALB) DENGAN Cellulose
Binding Module (CBM) PADA Pichia pastoris
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2016
Judul Tesis : Konstruksi Vektor dan Ekspresi Protein Rekombinan Lipase Candida antarctica (CALB) dengan Cellulose Binding Module (CBM) pada
Pichia pastoris
Nama : Febriana Dwi Wahyuni NIM : P051130241
Disetujui oleh Komisi Pembimbing
Prof. Dr. Ir. Suharsono, DEA Ketua
Dr. Asrul Muhamad Fuad Anggota
Diketahui oleh
Ketua Program Studi Bioteknologi
Prof. Dr. Ir. Suharsono, DEA
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga penulis dapat menyelesaikan penelitian yang dilaksanakan sejak November 2014 dengan judul “Konstruksi Vektor dan Ekspresi Protein Rekombinan Lipase Candida antarctica (CALB) dengan Cellulose Binding Module (CBM) pada Pichia pastoris”. Tesis ini merupakan hasil penelitian yang
dilakukan di Laboratorium Rekayasa Protein dan Sistem Penyampaian Obat, Pusat Penelitian Bioteknologi-LIPI, Cibinong. Sebagian dari hasil penelitian ini sedang dalam penelaahan untuk dipublikasikan pada Malaysian Journal of Microbiology
(MJM) dengan judul “Expression of CALB recombinant protein in Pichia pastoris”. Terima kasih penulis ucapkan kepada Bapak Prof. Dr. Ir. Suharsono, DEA dan Bapak Dr. Asrul Muhamad Fuad selaku pembimbing yang telah memberikan bimbingan dan arahan selama penelitian hingga penyusunan tesis ini. Penulis juga mengucapkan terima kasih kepada Dr. I Made Artika, M.App.Sc selaku penguji di luar komisi pembimbing yang memberikan kritik dan saran dalam penyusunan tesis ini. Di samping itu, penghargaan penulis sampaikan kepada Kepala beserta seluruh staf Laboratorium Rekayasa Protein dan Sistem Penyampaian Obat, Pusat Penelitian Bioteknologi-LIPI, Cibinong yang telah banyak membantu dalam menyelesaikan penelitian ini. Ungkapan terima kasih juga disampaikan kepada bapak, ibu, serta seluruh keluarga, dan teman-teman Pascasarjana Bioteknologi 2013 atas segala doa dan kasih sayangnya.
Penulis menyadari masih banyak kekurangan dalam penyusunan tesis ini. Penulis mengharapkan adanya saran dan kritik yang bersifat membangun. Penulis juga berharap semoga tesis ini bermanfaat.
Bogor, Februari 2016
DAFTAR ISI
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
1 PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 2
Manfaat Penelitian 2
2 TINJAUAN PUSTAKA 3
Lipase 3
Candida antarctica Lipase B (CALB) 4
Cellulose-Binding-Module (CBM) 5
Pichia pastoris 6
Vektor ekspresi pGAPZα 7
3 METODE 9
Waktu dan Tempat 9
Bahan 9
Metode Penelitian 9
4 HASIL DAN PEMBAHASAN 13
Subklon gen cbm ke dalam vektor pengklonan 13 Transformasi Pichia pastoris dengan Plasmid Rekombinan 16
Seleksi P. pastoris Transforman 17
Uji aktivitas lipase 19
Ekspresi protein rekombinan CALB 20
5 SIMPULAN DAN SARAN 22
Simpulan 22
Saran 22
DAFTAR PUSTAKA 23
LAMPIRAN 26
DAFTAR GAMBAR
1 Lipatan α/β hidrolase pada lipase 3
2 Struktur Candida antarctica lipase B (CALB) 5
3 Struktur Cellulose Binding Module 6
4 Vektor kloning pGAPZα dalam inang E. coli TOP10F’ dan Vektor
ekspresi pGAPZα dalam inang P. pastoris SMD1168H 8 5 Hasil amplifikasi gen cbm dan isolasi plasmid rekombinan
pGAPZα-CalBsyn 13
6 Hasil Site Directed Mutagenesis faktor-α 14
7 Hasil potong dengan enzim restriksi ClaI 14
8 Vektor rekombinan pGAPZα-CalBsyn-cbm yang dikonstruksi 15 9 Hasil transformasi pGAPZα-CalBsyn-cbm ke dalam E. coli TOP10F’ 15 10 Hasil PCR koloni dengan pasangan primer CBM F/R 16 11 Hasil pemotongan pGAPZα-CalBsyn dengan enzim BspHI 16 12 Hasil transformasi plasmid rekombinan pGAPZα-CalBsyn ke dalam
genom P. pastoris SMD1168H 17
13 Integrasi vektor rekombinan pada genom P. pastoris 17 14 Analisis stabilitas genetik koloni P. pastoris transforman dan non
transforman di media YPD agar dengan beberapa konsentrasi zeosin 18 15 Uji aktivitas enzim ekstrak kasar lipase secara kualitatif 19 16 Kurva pertumbuhan P. pastoris transforman 20
17 Uji aktivitas lipase secara kuantitatif 20
18 Analisis ekspresi gen CalBsyn dengan SDS-PAGE 21
DAFTAR LAMPIRAN
1 Primer yang digunakan 26
2 Komposisi larutan dan media yang digunakan beserta cara
pembuatannya 27
3 Kurva Standar ρ-Nitrophenyl Butyrate (PNPB) 30
4 Komposisi gel poliakrilamid 31
5 Analisis urutan DNA plasmid pGAPZα-CalBsyn dengan primer
GAP_F 32
6 Penentuan Efisiensi Transformasi 33
1
PENDAHULUAN
Latar Belakang
Lipase merupakan enzim yang diaplikasikan dalam proses hidrolisis triasilgliserol menjadi asam lemak, diasilgliserol, monoasilgliserol dan gliserol (Sharma et al. 2001). Pemanfaatan lipase dalam berbagai bidang sangat beragam, misalnya dalam teknologi pangan, aplikasi industri dan pengetahuan biomedik (Gupta 2004). Lipase juga memiliki sifat enzimatis dan spesifisitas substrat yang beragam sehingga banyak diaplikasikan dalam bidang industri (Blank et al. 2006). Lipase dapat diekstraksi dari berbagai mikroba, seperti bakteri, fungi, dan ragi/khamir (Gunasekaran dan Das 2005). Keuntungan lipase yang berasal dari mikroorganisme adalah memiliki aktivitas katalitik yang beragam, mudah untuk dimanipulasi secara genetik dan mudah untuk diproduksi secara massal (Hasan et al. 2006).
Candida antarctica lipase B (CALB) merupakan salah satu jenis lipase yang berasal dari mikroorganisme dan telah banyak diaplikasikan sebagai biokatalis dalam beberapa bidang industri. Pemanfaatan enzim CALB dalam bidang industri ini karena sifat enzim CALB yang dapat diaplikasikan pada suhu rendah dan dapat memiliki termostabilitas yang baik terhadap peningkatan suhu serta memiliki stabilitas yang tinggi dalam pelarut organik (Joseph et al. 2008; Pfeffer et al. 2006).
Protein rekombinan yang berasal dari eukariot telah banyak diekspresikan di dalam sistem ekspresi khamir. Khamir memiliki beberapa keunggulan sebagai sistem ekspresi untuk memproduksi protein rekombinan dibandingkan dengan bakteri. Sel khamir memungkinkan terjadinya modifikasi protein pada tahap pasca translasi. Pembentukan ikatan disulfida dan pelipatan protein yang benar meningkatkan ketahanan protein terhadap degradasi protease dan aktivitas biologis. Sel eukariot seperti khamir memiliki kemampuan untuk melakukan proses glikosilasi pada protein melalui penambahan residu gula pada asparagin (N-glikosilasi) atau serin/threonin (O-(N-glikosilasi). Struktur protein pasca translasi yang terjadi di dalam khamir dapat membentuk protein rekombinan yang aktif secara biologis (Cho et al. 1998).
Organisme yang pertama kali digunakan sebagai sistem ekspresi protein rekombinan pada khamir adalah Saccharomyces cereviciae. Namun, sistem ekspresi protein rekombinan juga dikembangkan melalui jenis khamir lainnya, misalnya Pichia pastoris (P. pastoris). P. pastoris merupakan khamir metilotropik yang mampu memanfaatkan metanol sebagai sumber karbon. Produksi protein rekombinan pada P. pastoris mempunyai beberapa keuntungan, diantaranya tingkat ekspresi protein rekombinan yang tinggi, DNA yang diintroduksi terintegrasi ke dalam genom P. pastoris sehingga stabil serta produktivitas protein yang lebih tinggi (Daly dan Hearn 2005).
2
pada khamir, seperti peptida sinyal yang berasal dari gen pho1, yeast invertase, dan sinyal sekresi alfa-mating factor (AMF). Peptida sinyal yang digunakan pada penelitian ini adalah peptida sinyal AMF karena cukup efektif dalam mensekresikan protein ke luar sel (Cereghino et al. 2013).
Cellulose-Binding Module (CBM) merupakan suatu domain protein yang umum dijumpai pada berbagai jenis enzim selulase. CBM umumnya berfungsi dalam proses pengikatan enzim selulase pada substratnya, yaitu selulosa sehingga dapat memfasilitasi kerja dari enzim selulosa tersebut (Wang et al. 2001). Fungsi CBM ini dapat dimanfaatkan untuk proses pengikatan (binding) dan imobilisasi suatu protein pada matriks selulosa. Pada penelitian ini protein khimera (protein fusi) Lipase dari Candida antarctica (CALB) difusikan dengan domain CBM dari Endoglukanase I (EGL1) dari Trichoderma reesei. Trichoderma adalah mikroorganisme penghasil sistem enzim selulase yang telah sering digunakan dalam industri terutama industri selulosa maupun dalam penelitian sebagai model dalam degradasi selulosa menggunakan fungi. Untuk menghasilkan protein rekombinan tersebut, domain CBM difusikan pada ujung C terminal dari enzim CALB.
Plasmid ekspresi yang digunakan dalam penelitian ini adalah pGAPZα. Vektor ini memiliki promoter GAP yang menyandi gliseraldehid-3-fosfat dehidrogenase (GAPDH) untuk mengekspresikan protein rekombinan pada P.pastoris dan bersifat konstitutif. Plasmid tersebut juga mengandung fragmen myc epitope untuk deteksi dan polihistidin tag untuk purifikasi yang dapat difusi pada ujung-C dari protein target. Seleksi pada vektor ini menggunakan gen penanda resistensi terhadap antibiotik zeosin (Invitrogen, 2002).
Tujuan Penelitian
Penelitian ini bertujuan mengkonstruksi vektor ekspresi gen CalB sintetik (CalBsyn) yang difusikan dengan gen penyandi cellulose binding module (cbm) pada plasmid ekspresi pGAPZα dan mengekspresikan gen CalB pada P. pastoris
SMD1168H.
Manfaat Penelitian
3
2
TINJAUAN PUSTAKA
Lipase
Lipase merupakan enzim yang diaplikasikan dalam proses hidrolisis triasilgliserol menjadi asam lemak, diasilgliserol, monoasilgliserol dan gliserol serta pada kondisi tertentu dapat mengkatalisis reaksi sebaliknya yaitu membentuk gliserida dari gliserol dan asam lemak (Sharma et al. 2001). Lipase termasuk ke dalam kelas enzim hidrolase yang anggotanya antara lain adalah protease, selulase, esterase, amilase, dan lipase. Lipase dan esterase dapat mengkatalis tipe reaksi yang sama, namun berbeda dalam spesifisitas substrat dan kinetika enzim (Fernandez 2000). Sebagian besar substrat bagi lipase adalah molekul dengan rantai yang lebih panjang dan tidak larut dalam air, sedangkan substrat bagi esterase adalah molekul yang larut dalam air seperti asam karboksilat atau ester dengan rantai yang lebih pendek (Pleiss et al. 1998). Karakteristik beberapa lipase yang tidak dimiliki esterase adalah aktivasi antarmuka, yaitu aktivitas enzimatik yang mulai teramati ketika lipase berada pada antarmuka minyak dan air (Nardini dan Dijkstra 1999; Santarossa et al. 2005). Aktivitas antarmuka terjadi karena adanya lid yang menutup sisi aktif substrat, yang akan terbuka pada keadaan aktif ketika berada pada antarmuka minyak dan air. Terbukanya lid memungkinkan masuknya substrat pada sisi aktif lipase (Kim et al. 1997).
Reaksi yang dikatalisis oleh kedua enzim ini pada lingkungan alaminya (fisiologis) adalah hidrolisis trigliserida menjadi digliserida, monogliserida, asam lemak dan gliserol. Pada lingkungan reaksi yang sesuai seperti dalam pelarut organik atau lingkungan dengan konsentrasi air yang rendah, lipase dapat mengkatalisis reaksi sebaliknya yaitu reaksi esterifikasi (Zhao et al. 2008; Iso et al2001; Ghaly et al. 2010; Mittelbach 1990). Beberapa lipase menunjukkan aktivitas yang lebih baik pada lingkungan reaksi dengan konsentrasi air yang lebih rendah. Kemampuan lipase untuk mengkatalisis reaksi esterifikasi dimanfaatkan dalam sintesis biodiesel, senyawa obat, senyawa deterjen, surfaktan, industri oleokimia, dan kosmetik (Sharma et al. 2001). Banyaknya jenis reaksi dan jenis substrat yang dapat dikatalisis oleh lipase menyebabkan enzim ini dipelajari dan digunakan secara ekstensif dalam sintesis senyawa kimia.
4
Struktur protein yang dimiliki oleh beberapa lipase termasuk dalam keluarga α/β-hydrolase fold. Struktur ini juga diadaptasi oleh hidrolase lainnya seperti protease, esterase, dehalogenase, dan epoksidase (Nardini dan Dijkstra 1999). Semua enzim yang termasuk dalam keluarga α/β-hydrolase fold mengadopsi struktur dimana terdapat delapan β-strand pada interior protein dengan diselingi adanya α-heliks diantara strand (Gambar 1). Tipe fold ini dipertahankan dengan sangat baik pada semua anggota keluarga, bahkan antara protein dengan sekuens yang berbeda jauh. Sisi aktif protein pada keluarga fold ini juga dipertahankan, yaitu katalitik triad yang bersifat nukleofil (serin, sistein, dan asam aspartat) serta residu histidin. Residu histidin dapat ditemui pada sisi aktif seluruh protein keluarga
α/β-hydrolase fold (Nardini dan Dijkstra 1999; Ollis et al. 1992).
Lipase memiliki spesifitas substrat yang beragam. Triasilgliserida bukan satu-satunya substrat yang dapat dihidrolisis oleh lipase. Beberapa lipase juga memiliki spesifitas terhadap substrat asilgliserida yang lain, seperti monoasilgliserida, diasilgliserida, dan fosfolipid. Lipase yang berasal dari Penicilium camemberti,
misalnya, memiliki aktivitas yang tinggi terhadap monoasilgliserida dan digliserida, serta tidak memiliki aktivitas terhadap trigliserida. Lipase tertentu ada yang memiliki spesifitas tinggi terhadap asam lemak, misalnya lipase yang berasal dari
Geottichum candidum. Lipase tersebut spesifik terhadap Δ9-asam lemak tidak jenuh (Pfeffer 2008).
Candida antarctica Lipase B (CALB)
Candida antarctica lipase B (CALB) merupakan salah satu enzim yang dihasilkan oleh khamir psychrophilic Candida antarctica. Mikroorganisme
psychrophilic adalah kelompok mikroorganisme yang dapat tumbuh dengan baik pada suhu 00C, memiliki temperatur pertumbuhan optimal pada suhu 150C, serta
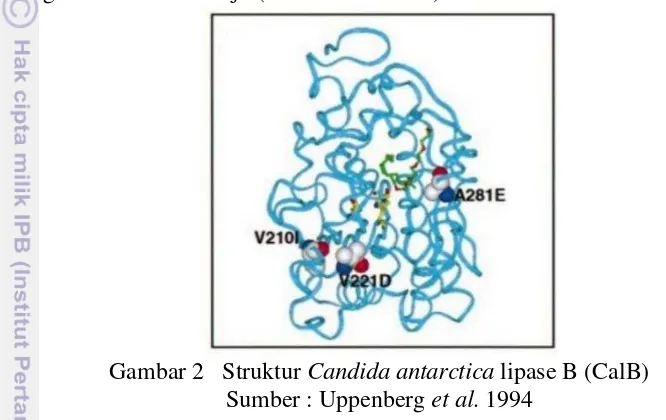
mampu hidup pada temperatur maksimum pada suhu 200C (Prescott et al. 2002). Mikroorganisme psychrophilic juga merupakan kelompok penghasil lipase yang dapat diaplikasikan pada suhu rendah, misalnya enzim CalB. Enzim CalB tersusun atas 317 asam amino dan memiliki berat molekul sebesar 33 kDa (Rotticci et al. 2000). Enzim tersebut memiliki ukuran yang lebih kecil dan bersifat lebih asam daripada enzim CalA (Joseph et al.2008). Sifat enzim CalB cenderung termostabil, khususnya di bawah kondisi tidak ada air dimana katalis tetap aktif berjam-jam dan adanya konsentrasi yang tinggi dari reaktan. Namun, enzim CalB wild type dapat terdenaturasi dengan cepat pada saat suhu meningkat sampai 400C (Zhang et al. 2003). Salah satu metode yang dapat digunakan untuk mendapatkan enzim CalB yang resisten terhadap peningkatan suhu adalah Site Directed Evolution. Melalui metode ini, enzim CalB dimodifikasi dengan melakukan mutasi pada tiga asam amino, yaitu V210I (mutasi pada asam amino ke 210 dari valin menjadi isoleusin), A281E (mutasi pada asam amino ke 281 dari alanin menjadi asam glutamat), dan V221D (mutasi pada asam amino ke 221 dari valin menjadi asam aspartat (Zhang
et al. 2003).
5 (Blank 2006). Rottici-Mulder (2001) menyatakan bahwa enzim CalB memiliki situs aktif yang dapat dijangkau pelarut. Situs aktif enzim tersebut berupa saluran yang sempit sehingga enzim CalB memiliki aktivitas yang tinggi terhadap ester asam karboksilat seperti etiloktanoat daripada terhadap trigliserida. Enzim CalB juga memiliki selektivitas hidrolisis ikatan ester pada posisi sn-3.
Enzim CalB merupakan enzim yang banyak diaplikasikan di bidang bioteknologi dan industri. Enzim CalB rekombinan telah digunakan di dalam industri detergen. Sekitar 1000 ton lipase telah digunakan dalam setahun untuk mengurangi polusi lingkungan akibat produk detergen. Enzim CalB juga digunakan dalam industri makanan, misalnya untuk menghidrolisis lemak pada susu serta meningkatkan cita rasa keju (Hasan et al. 2006).
Gambar 2 Struktur Candida antarctica lipase B (CalB) Sumber : Uppenberg et al. 1994
Cellulose Binding Module (CBM)
Polikarbohidrat tidak larut, seperti selulosa, merupakan substrat yang sulit untuk degradasi enzim. Hal ini karena sulitnya jangkauan substrat yang padat untuk situs katalitik enzim dan sebagian karena heterogenitas struktur fisik. Karakteristik substrat ini menetapkan persyaratan khusus untuk hidrolisis enzim. Salah satu solusi untuk degradasi enzim dengan substrat terlarut yaitu dihasilkan substrate-binding module yang berbeda. Contoh modul tersebut dapat ditemukan dalam
carbohydrate-active enzyme seperti selulase, hemiselulase, dan kitinase.
Cellulose-Binding Module (CBM) merupakan suatu domain protein yang umum dijumpai pada berbagai jenis enzim selulase. CBM umumnya berfungsi dalam proses pengikatan enzim selulase pada substratnya, yaitu selulosa sehingga dapat memfasilitasi kerja dari enzim selulosa tersebut (Wang 2001). Fungsi dari CBM ini dapat dimanfaatkan untuk proses pengikatan (binding) dan imobilisasi suatu protein pada matriks selulosa.
CBM awalnya diklasifikasikan sebagai Cellulose Binding Domain (CBD) berdasarkan penemuan awal dari beberapa modul yang mengikat selulosa (Gilkes
6
berada di ujung C-terminal atau N-terminal dan kadang-kadang terpusat dalam rantai polipeptida (Shoseyov et al. 2006).
CBM telah ditemukan di dalam protein hidrolitik dan non hidrolitik. Protein yang memiliki aktivitas hidrolitik (contoh: selulase dan xylanase) meliputi molekular kompleks yang berisikan modul yang mempunyai ciri-ciri tersendiri (biasanya modul katalitik), yang biasanya bergabung dengan urutan linker yang relatif tidak terstruktur (Shoseyov et al. 2006). CBM juga telah ditemukan pada beberapa polisakarida selain selulase dan xylanase. Pada Trichoderma reesei, CBM telah diidentifikasi dalam hemiselulosa, endomananase, dan acetilxylanase (Margolles et al. 1996; Stalbrand et al. 1995).
Beberapa tahun belakangan, penggunaan CBM telah dimanfaatkan dalam beberapa bidang bioteknologi. Ada beberapa sifat dasar CBM yang menjadikannya banyak diaplikasikan dalam beberapa bidang, diantaranya yaitu CBM biasanya dapat melipat sendiri dan dapat berfungsi secara otonom pada protein kimera, penempelan matrix yang berlimpah dan murah, memiliki sifat fisik dan kimia yang baik, serta dapat mengendalikan pengikatan yang spesifik (Shoseyov 2006).
Gambar 3 Struktur Cellulose Binding Module
Sumber : Lehtio 2001
Pichia pastoris
Pichia pastoris merupakan khamir metilotropik yang mampu melakukan metabolisme metanol sebagai sumber karbon. Reaksi kimia yang dilakukan P. pastoris adalah oksidasi metanol menjadi formaldehid dengan bantuan alkohol oksidase. Salah satu produk sampingan dari reaksi oksidasi metanol adalah hidrogen peroksida (H2O2) yang akan diuraikan menjadi hidrogen dan air di
peroksisom. Alkohol oksidase memiliki afinitas yang sangat rendah terhadap oksigen. P. pastoris mengatasi hal tersebut dengan menghasilkan sejumlah besar enzim (Invitrogen 2001)
7 pasca translasi (Glick dan Pasternak 2003). Keuntungan lainnya digunakannya P. pastoris sebagai sel inang untuk ekspresi protein yaitu manipulasi P. pastoris
semudah seperti manipulasi pada E. coli atau S. cerevisiae. Sistem ekspresi dengan
P. pastoris lebih cepat, mudah dan murah untuk digunakan bila dibandingkan dengan sistem ekspresi lainnya seperti Baculovirus atau sistem mamalia. P. pastoris
menggunakan beberapa manipulasi fitur molekuler dan genetik dari Saccharomyces
sehingga menambahkan keuntungan tingkat ekpresi yang lebih tinggi. Fitur dengan perawatan yang mudah, scale up dan persyaratan pertumbuhan murah membuat P. pastoris sebagai sistem ekspresi protein yang sangat berguna. Proses ini dapat ditingkatkan ke tingkat ekspresi, yaitu 10-100 kali lebih tinggi daripada E. coli
(Balamurugan et al. 2007).
Ekspresi gen asing pada P. pastoris meliputi tiga langkah dasar, yaitu penyisipan gen asing ke dalam vektor ekspresi, integrasi vektor rekombinan ke dalam genom P. pastoris dan seleksi galur P. pastoris hasil transformasi yang dapat mengekspresikan gen asing (Li et al. 2007). Studi biokimia menunjukkan bahwa pemanfaatan metanol membutuhkan jalur metabolisme yang melibatkan beberapa enzim. Enzim alkohol oksidase (AOX) mengkatalisis pada tahapan pertama di jalur metabolisme metanol. Berada di dalam peroksisom, AOX mengoksidasi metanol menjadi formaldehid dan hidrogen peroksida (Cereghino & Cregg 2000). Ada dua gen yang menyandikan alkohol oksidase dalam P. pastoris yaitu AOX1 dan AOX2. Gen AOX1 diekspresikan oleh promotor AOX1 yang sangat tergantung kepada regulasi dan induksi dari metanol yang terkandung dalam medium. Total protein yang dihasilkan dapat mencapai lebih dari 30% dari total protein terlarut ketika sel ditumbuhkan dalam medium yang mengandung metanol sebagai sumber karbon. Tingkat ekspresi dari AOX1 diatur pada tingkat transkripsi (Zhang 2009). AOX1
sebagian besar bertanggung jawab pada aktivitas alkohol oksidase dalam sel. AOX2
memiliki sekitar 97% homologi dengan AOX1 (Balamurugan et al. 2007).
Promotor alternatif yang dapat digunakan selain promotor AOX yaitu promotor GAP, FLD1, PEX8, dan YPT7. Promotor GAP merupakan promotor konstitutif yang tidak memerlukan induksi. Promotor GAP berasal dari gen yang mengkode
GAPDH (glyceraldehyde-3-phosphate dehydrogenase) dan gen tersebut merupakan salah satu housekeeping gene. GAPDH adalah enzim yang sangat diperlukan pada proses glikolisis. Enzim tersebut mengkatalis glyceraldehyde-3-phospate menjadi 1,3 biphosphoglycerate. Enzim GAPDH juga berperan pada beberapa proses non metabolisme seperti inisiasi apoptosi dan transportasi RNA
binding protein dalam aktivasi transkripsi. Enzim GAPDH terdiri dari subunit tetramer yang identik (Thanonkeo et al. 2010). Penggunaan promotor GAP dalam sistem ekspresi protein rekombinan pada P. pastoris dapat mengurangi biaya dan penggunaan metanol sebagai penginduksi promotor AOX. Sumber karbon yang dapat digunakan pada sistem ekspresi PGAP adalah glukosa, gliserol, asam oleat, dan
metanol (Zhang et al. 2009).
Vektor Ekspresi pGAPZα
8
secara konstitutif pada P. pastoris. Vektor pGAPZα dapat mengekspresikan protein
tanpa menggunakan metanol dan lebih disukai untuk ekspresi skala besar. Penggunaan promotor GAP tidak disarankan jika produksi protein toksik terhadap khamir sebagai sel inang. Vektor pGAPZα berukuran 3,1 kb, mengandung myc epitope untuk deteksi dan polyhistidine tag untuk purifikasi pada ujung C, mengandung sinyal sekresi faktor α yang berfungsi untuk sekresi protein rekombinan, memiliki multiple cloning site (MCS) dengan situs restriksi yang unik. Vektor pGAPZα juga memiliki promotor TEFI, EM7, CYCI transcription termination region, dan pUC origin. Origin pUC merupakan situs replikasi plasmid pada E. coli (Invitrogen 2008). Gambar vektor ekspresi pGAPZα dapat dilihat pada
Gambar 4.
Vektor pGAPZα mengandung gen Sh ble (Streptoalloteichus hindustanus bleomycin). Gen ini berukuran 375 bp dan memiliki resistensi terhadap antibiotik zeosin sehingga dapat digunakan untuk seleksi vektor rekombinan pada E. coli, P. pastoris dan eukariot lain. Promotor TEF1 yang berasal dari S. cerevisiae
digunakan untuk mengekspresikan gen Sh ble pada P. pastoris sebagai gen resisten zeosin. Promotor EM7 merupakan promotor konstitutif mengekspresikan gen Sh ble pada E. coli sebagai gen resisten zeosin, serta CYCI transcription termination region berasal dari S. cerevisiase yang berfungsi untuk meningkatkan stabilitas gen
Sh ble (Invitrogen 2001).
Gambar 4 Vektor kloning pGAPZα dalam inang E.coli TOP10F’ dan vektor
9
3
METODE
Waktu dan Tempat
Penelitian ini berlangsung dari November 2014 sampai Januari 2016 di Laboratorium Rekayasa Protein dan Sistem Penyampaian Obat, Pusat Penelitian Bioteknologi, Lembaga Ilmu Pengetahuan Indonesia (LIPI), Cibinong, Bogor.
Bahan
Plasmid DNA dan gen: Plasmid ekspresi yeast pGAPZα (Gambar 4) berasal dari Invitrogen, Gen CalBsyn dibuat secara sintetik dari DNA2.0 dengan optimasi kodon P. pastoris. Fragmen gen cbm dibuat secara sintetik dari IDT. Mikroorganisme Escherichia coli TOP 10F’ (Invitrogen) sebagai sel inang replikasi plasmid rekombinan, Pichia pastoris SMD 1168 H (Invitrogen) sebagai sel inang untuk ekspresi protein rekombinan. Media tumbuh dan antibiotik: LSLB (Low Salt Luria Bertani), YPD (Yeast Peptone Dextrose), Ampisilin dan Zeosin. Primer DNA berasal dari IDT. Sekuen primer DNA yang digunakan dalam penelitian ini disajikan pada Lampiran 1. Komposisi media dan larutan yang digunakan dalam penelitian ini disajikan pada Lampiran 2.
Metode Penelitian
Subklon gen cbm ke dalam Plasmid Ekspresi
Gen cbm dan plasmid pGAPZα-CalBsyn dipotong dengan enzim restriksi
ClaI dan dipurifikasi menggunakan Gel DNA Extraction Kit (GeneAid). Selanjutnya gen cbm disubklon ke dalam plasmid pGAPZα-CalBsyn pada situs ClaI menggunakan teknik umum ligasi dan diintroduksi ke dalam E.coli TOP 10F’. Transformasi pada E.coli dilakukan dengan metode kejut panas (Ausubel 2002).
E.coli transforman diseleksi dengan menggunakan medium seleksi LSLB (Low Salt Luria Bertani) agar yang mengandung antibiotik zeosin 25 µg/ml. Analisis PCR dilakukan terhadap E.coli transforman yang diperoleh menggunakan primer spesifik, CBM_F dan CBM_R dengan metode PCR koloni dan PCR plasmid. Selanjutnya plasmid rekombinan yang diperoleh dianalisis potong dengan enzim restriksi serta analisis sekuen urutan DNA.
Transformasi P. pastoris dengan Plasmid Rekombinan
Transformasi sel khamir dilakukan dengan metode elektroporasi menggunakan protokol dari invitrogen (2008). Plasmid rekombinan pGAPZα
-CalBsyn yang akan diintroduksi ke dalam sel P. pastoris SMD1168H terlebih dahulu dipotong menjadi bentuk linier dengan enzim BspHI (Invitrogen 2002). Plasmid yang telah terpotong dipurifikasi menggunakan Gel DNA Extraction Kit
10
diinokulasikan ke dalam 50 ml media YPD dan diinkubasi semalam pada suhu 30ᴼC, 250 rpm hingga OD600 mencapai 1,3-1,5. Sel dipanen dengan cara sentrifugasi (4ᴼC,
3000 g, 5 menit). Endapan sel diresuspensi dengan 50 mL akuades dingin steril dan disentrifugasi (4ᴼC, 4000 g, 7 menit). Endapan sel diresuspensi kembali dengan 25 ml akuades dingin steril dan disentrifugasi (4ᴼC, 4000 g, 7 menit). Endapan sel diresuspensi dalam 2 ml 1 M sorbitol dingin steril dan disentrifugasi (4ᴼC, 3500 g, 5 menit). Endapan sel diresuspensi kembali dalam 200 µl 1M sorbitol dingin steril. Sebanyak 70 µl sel kompeten P.pastoris dicampurkan dengan 0,5 µg plasmid linier (pGAPZα-CalB). Sel kompeten dan plasmid diinkubasi terlebih dahulu pada suhu 0ᴼC selama 10 menit. Sel kompeten dan plasmid dimasukkan ke dalam kuvet elektroporasi (2 mm gap) yang telah didinginkan. Kuvet yang mengandung sel kompeten dan plasmid diinkubasi pada suhu 0ᴼC (dalam es) selama 5 menit. Elektroporasi dilakukan dengan kondisi 2 kV, 25 µF 400Ω pada elektroporator Gene Pulser Xcell Electroporation System (Biorad). Sebanyak 150 µl 1M sorbitol dingin segera ditambahkan ke dalam kuvet tersebut. Isi kuvet ditransfer ke dalam mikrotube steril dan diinkubasi di dalam es selama 1-2 jam. Sel hasil transformasi disebar pada media agar YPDS (Yeast Potato Dextrose Sorbitol) yang mengandung zeosin dengan konsentrasi 100 µg/mL dan diinkubasi selama 3-4 hari pada suhu 30ᴼC sampai terbentuk koloni.
Seleksi Koloni P. pastoris Transforman
Koloni yang tumbuh pada media seleksi yang mengandung 100 µg/ml zeosin ditumbuhkan kembali pada media YPD agar dengan berbagai tingkat konsentrasi zeosin (100 - 1000 µg/ml) secara bertahap. Seleksi ini bertujuan untuk memperoleh koloni transforman yang memiliki stabilitas genetik yang tinggi serta koloni dengan salinan gen multikopi. Koloni transforman juga ditumbuhkan pada media YPD agar tanpa zeosin sebagai kontrol. Seleksi dilakukan dengan menggoreskan koloni transforman pada media seleksi YPD agar dengan konsentrasi zeosin 100 µg/mL dan diinkubasi selama 1-2 hari pada suhu 30ᴼC. Koloni yang tumbuh digoreskan kembali pada media YPD agar dengan zeosin 200 µg/mL. Koloni yang tumbuh pada medium tersebut digoreskan lagi pada media dengan konsentrasi zeosin yang lebih tinggi, yaitu 500 µg/mL dan 1000 µg/mL. Koloni yang hidup pada media dengan konsentrasi zeosin tertinggi digunakan untuk ekspresi protein skala kecil.
Uji Aktivitas Lipase Kualitatif dan Kuantitatif
Uji Aktivitas Lipase secara Kualitatif
Uji aktivitas lipase secara kualitatif dilakukan dengan menggunakan metode Rhodamin olive oil berdasarkan Koeker dan Jaeger (1987). Koloni P. pastoris yang tumbuh diseleksi kemampuannya dalam menghasilkan lipase. Identifikasi koloni penghasil lipase serta penentuan aktivitas lipase dilakukan menggunakan media seleksi plate Rhodamin B agar (modifikasi Kouker dan Jaeger 1987) yang mengandung PDA 39 g/L, Rhodamin B 0,01%, PVA 1,5% dan substrat olive oil
11 zona berpendar sedangkan olive oil berfungsi sebagai sumber lipida (Gupta et al. 2003).
Uji aktivitas Lipase secara Kuantitatif
Uji aktivitas lipase secara kuantitatif dilakukan dengan metode Continuous Spectrophotometric Rate Determination (Quinn et al. 1982; Sirai and Jackson 1982). Pengujian aktivitas lipase dilakukan dengan menggunakan substrat berupa ρ-Nitrophenyl Butyrate (PNPB) dan sebagai kontrol positif digunakan lipase dari
Candida rugosa (CRL). PNPB dan CRL merupakan produk dari Sigma. Reagen yang digunakan diantaranya yaitu Reagen A yang terdiri dari 100 mM Sodium Phosphate Buffer, 150 mM Sodium Chloride dan 0,5% Triton X-100. Substrat dibuat dengan cara mencampur 50 mM ρ-Nitrophenyl Butyrate (PNPB) dengan acetonitrile. Sebagai kontrol positif, lipoprotein lipase dari Candida rugosa (CRL) dilarutkan dalam reagen A. Aktivitas lipase diukur dengan melihat absorbansi pada panjang gelombang 400 nm dan penghitungan dilakukan setiap menit sampai menit ke-5. Kurva standar PNPB untuk menentukan aktivitas lipase secara kuantitatif disajikan pada Lampiran 3.
Ekspresi Protein Rekombinan CALB
Koloni P. pastoris transforman yang mengandung plasmid pGAPZα-CalBsyn
ditumbuhkan pada media agar miring YPD dengan zeosin 100 µg/mL, kemudian dilakukan uji ekspresi protein rekombinan. Uji ekspresi protein rekombinan mengikuti prosedur pada manual Pichia Expression Kit versi G (invitrogen). Koloni tunggal dari koloni transforman diinokulasikan ke dalam 10 mL media YPD. Biakan selanjutnya diinkubasi selama 24 jam pada suhu 30ᴼC dengan agitasi 250 rpm. Sebanyak 0,1 ml kultur yang tumbuh diinokulasikan ke 50 ml media YPD dan ditumbuhkan pada suhu 30ᴼC dengan agitasi 250 rpm. Pengambilan contoh untuk analisis dilakukan setiap jam ke-24, 48, dan 72. Biakan dipanen dengan cara sentrifugasi pada suhu 4ᴼC, 3000 g selama 10 menit.
Pemekatan Protein dengan metode presipitasi menggunakan larutan TCA (Trichloroacetic acid).
Presipitasi protein dilakukan untuk memekatkan protein yang terdapat pada contoh supernatan. Presipitasi protein menggunakan larutan TCA dilakukan berdasarkan prosedur dari Sanchez (2001). Sebanyak 1 mL supernatan dicampurkan dengan 250 µl larutan TCA 100%. Campuran tersebut diinkubasi selama 10 menit pada suhu 40C. Selanjutnya campuran tersebut disentrifugasi dengan kecepatan 12.000 rpm selama 5 menit pada suhu 40C. Supernatan dibuang
sedangkan protein akan terpresipitasi membentuk pelet. Pelet dicuci menggunakan 200 µl aseton dingin untuk menghilangkan larutan TCA yang tersisa dan disentrifugasi selama 5 menit dengan kecepatan 12.000 rpm pada suhu 40C. Aseton
12
Analisis protein rekombinan CALB dengan metode SDS-PAGE
13
4
HASIL DAN PEMBAHASAN
Subklon gen cbm ke dalam vektor pengklonan
Konstruksi gen cbm dirancang berdasarkan informasi sekuens gen cbm (wild type) yang terdapat pada GeneBank dengan nomor akses P07981 melalui UniProt. Sekuen gen cbm ini telah dioptimasi dengan kodon preferensi P. pastoris. Gen ini diamplifikasi dan ditambah situs restriksi ClaI dengan teknik PCR. Hasil PCR dengan primer CBM-F dan CBM-R menghasilkan produk amplifikasi berupa gen
cbm dengan ukuran 261 pb (Gambar 5). Plasmid rekombinan pGAPZα-CalBsyn
merupakan vektor ekspresi yang mengandung beberapa perangkat gen penting untuk mengekspresikan protein rekombinan, yaitu promotor GAP sebagai promotor konstitutif, fragmen MF-α(mating factor-α) sebagai gen penyandi sinyal peptida supaya protein dapat disekresikan keluar sel, gen she ble sebagai gen penyandi resisten terhadap antibiotik zeosin untuk menyeleksi transforman, pUC ORI untuk replikasi plasmid pada E. coli. Plasmid rekombinan diperbanyak di dalam sel E. coli TOP10F’ dan diperoleh dengan cara isolasi menggunakan teknik
alkali lisis (Gambar 5 ).
A B
Gambar 5 Hasil amplifikasi gen cbm dan isolasi plasmid rekombinan pGAPZα-CalBsyn. A= Hasil PCR gen cbm yang berukuran sekitar 261 pb. B= hasil isolasi plasmid rekombinan pGAPZα-CalBsyn
Sekuen sinyal peptida MFα pada plasmid rekombinan pGAPZα-CalBsyn
mengalami mutasi titik berupa delesi satu nukleotida. Site Directed Mutagenesis
(SDM) dilakukan untuk mengganti basa nitrogen pada salah satu titik yang diinginkan. SDM dilakukan untuk mengubah residu asam amino spesifik pada protein. Pada teknik mutagenesis ini, urutan DNA yang mengkode protein dan posisi asam amino yang akan diganti harus diketahui dengan akurat (Glick dan Pasternak 2003). SDM dilakukan untuk memperbaiki sekuen sinyal peptida pada
alpha-mating factor dengan melakukan satu mutasi titik. SDM dilakukan melalui amplifikasi PCR dengan menggunakan primer MFα-F dan MFα-R yang mengandung modifikasi fosforilasi pada ujung 5’. Selanjutnya dilakukan ligasi pada produk PCR dengan DNA ligase T4 yang dilanjutkan dengan transformasi ke
E. coli TOP10F’. Kemudian dilakukan verifikasi produk SDM dengan teknik
14
Gambar 6 Hasil Site Directed Mutagenesis faktor-α dengan penambahan basa nitrogen adenin
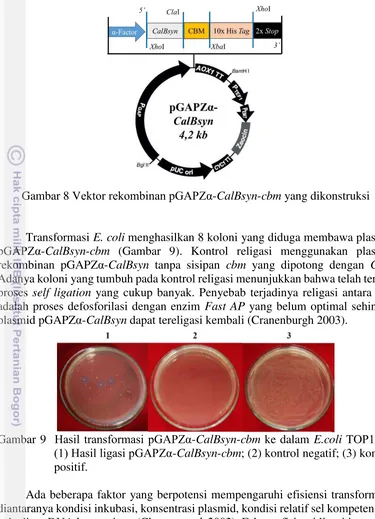
Plasmid rekombinan pGAPZα-CalBsyn-cbm dikonstruksi dengan cara menyisipkan gen cbm pada plasmid rekombinan pGAPZα-CalBsyn. Gen cbm
diligasikan pada situs restriksi ClaI. Situs restriksi ClaI diintroduksikan ke ujung 5’
dan ujung 3’ gen cbm dengan teknik PCR. Sebelum diligasi, plasmid rekombinan pGAPZα-CalBsyn dan gen cbm dipotong terlebih dahulu dengan enzim ClaI. Enzim ClaI memiliki pola pemotongan sticky end sehingga akan mempermudah proses ligasi. Vektor pGAPZα-CalBsyn yang telah dipotong, diinkubasi dengan enzim Fast AP (alkaline phosphatase) untuk mencegah terjadinya self ligation
dengan cara menghilangkan gugus fosfat pada ujung 5’ (Glick dan Pasternak 2003). Hasil potong plasmid rekombinan tersebut memperlihatkan adanya satu fragmen DNA berukuran 4236 pb sedangkan hasil potong dari gen cbm memiliki ukuran sekitar 249 pb (Gambar 7).
A B
Gambar 7 Hasil pemotongan pGAPZα-CalBsyn dan gen cbm dengan enzim ClaI. (A) M= DNA marker 1kb, 1= pGAPZα-CalBsyn dipotong dengan enzim ClaI, 2= pGAPZα-CalBsyn tidak dipotong; (B) M= DNA marker 100 pb, 1= gen cbm yang dipotong dengan enzim ClaI, 2= gen cbm
tidak dipotong.
15
Gambar 8 Vektor rekombinan pGAPZα-CalBsyn-cbm yang dikonstruksi
Transformasi E. coli menghasilkan 8 koloni yang diduga membawa plasmid pGAPZα-CalBsyn-cbm (Gambar 9). Kontrol religasi menggunakan plasmid rekombinan pGAPZα-CalBsyn tanpa sisipan cbm yang dipotong dengan ClaI. Adanya koloni yang tumbuh pada kontrol religasi menunjukkan bahwa telah terjadi proses self ligation yang cukup banyak. Penyebab terjadinya religasi antara lain adalah proses defosforilasi dengan enzim Fast AP yang belum optimal sehingga plasmid pGAPZα-CalBsyn dapat tereligasi kembali (Cranenburgh 2003).
Gambar 9 Hasil transformasi pGAPZα-CalBsyn-cbm ke dalam E.coli TOP10F’. (1) Hasil ligasi pGAPZα-CalBsyn-cbm; (2) kontrol negatif; (3) kontrol positif.
16
ukurannya pendek. Ujung kohesif kaya A,T nukleotida memliki efisiensi ligasi yang paling rendah.
Koloni E.coli kemudian diverifikasi dengan beberapa cara, diantaranya adalah melalui PCR koloni, pemotongan dengan menggunakan enzim restriksi dan analisis sekuen DNA. PCR koloni dilakukan untuk verifikasi keberadaan sisipan gen cbm di dalam plasmid rekombinan pGAPZα-CalBsyn-cbm. PCR koloni dilakukan menggunakan pasangan primer CBM_F dan CBM_R. Hasil PCR koloni menunjukkan bahwa 5 dari 8 sampel koloni yang dianalisis diduga mengandung gen cbm dengan ukuran 249 pb (Gambar 10). Sampel tersebut kemudian diverifikasi dengan penentuan arah orientasi dari struktur cbm.
Gambar 10 Hasil PCR koloni dengan pasangan primer CBM F/R. M= DNA marker
100 pb; 1-6= klon transforman pGAPZα-CalBsyn-cbm; 7= kontrol positif; 8= kontrol negatif
Transformasi Pichia pastoris dengan Plasmid Rekombinan
Plasmid rekombinan pGAPZα-CalBsyn yang akan diintroduksikan ke dalam genom P.pastoris terlebih dahulu dipotong menjadi bentuk linier dengan enzim
BspHI. Hasil potong kemudian dianalisis dengan elektroforesis gel agarosa 1% dan menunjukkan adanya pita tunggal berukuran sekitar 4236 pb (Gambar 11). Vektor rekombinan yang telah dipotong dan dipurifikasi kemudian dihitung konsentrasinya untuk selanjutnya diintroduksi ke dalam genom P.pastoris SMD1168H.
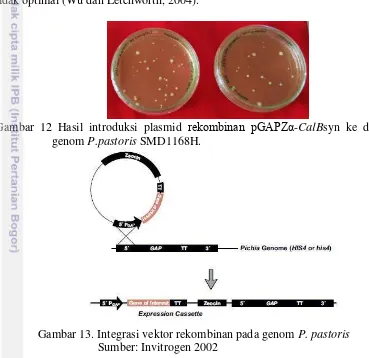
17 Plasmid rekombinan telah berhasil diintroduksikan ke dalam genom P. pastoris SMD1168H (Gambar 12). Plasmid rekombinan ini menyisip ke dalam genom P. pastoris melalui mekanisme rekombinasi homologus dengan memanfaatkan kesamaan sekuen promotor GAP yang terdapat pada genom P. pastoris dan vektor pGAPZα (Gambar 13). Vektor dirancang untuk terintegrasi ke dalam genom P. pastoris sehingga memungkinkan ekspresi protein menjadi stabil. Transformasi plasmid rekombinan menghasilkan 64 koloni tunggal transforman P. pastoris dengan efisiensi transformasi sebesar 1,01 x 102 cfu/µg DNA (lampiran 6).
Efisiensi transformasi P. pastoris tergolong rendah karena plasmid tidak hanya masuk ke dalam sel tetapi juga harus terintegrasi ke dalam genom melalui rekombinasi homolog. Efisiensi transformasi yang rendah biasanya terjadi karena situs integrasi yang tidak efisien, DNA sisipan yang sulit dan kondisi transformasi tidak optimal (Wu dan Letchworth, 2004).
Gambar 12 Hasil introduksi plasmid rekombinan pGAPZα-CalBsyn ke dalam genom P.pastoris SMD1168H.
Gambar 13. Integrasi vektor rekombinan pada genom P. pastoris
Sumber: Invitrogen 2002
Seleksi P. pastoris Transforman
Vektor telah dirancang mengandung gen penyandi resistensi zeosin, yaitu gen
18
µg/mL sekitar 90% koloni mampu tumbuh dengan baik, sedangkan pada konsentrasi zeosin 200 dan 500 µg/mL, hanya ada sekitar 40% koloni transforman yang mampu tumbuh dengan baik. Pada konsentrasi zeosin 1000 µg/mL, pertumbuhan koloni transforman mulai terhambat. Sementara P. pastoris non transforman tidak mampu tumbuh pada media yang mengandung zeosin (Gambar 14). Tingkat resistensi zeosin secara tidak langsung mencerminkan jumlah gen rekombinan yang masuk ke dalam genom P. pastoris. Menurut Norden et al. (2011), klon transforman yang resisten 100 µg/mL zeosin cenderung memiliki minimal 1 salinan plasmid, resisten 500 µg/mL zeosin minimal memiliki 4 salinan plasmid, resisten 1000 µg/mL zeosin minimal memiliki 9 salinan plasmid dan resisten zeosin lebih dari 1000 µg/mL memerlukan minimal 17 salinan plasmid yang terintegrasi pada genom. Peningkatan jumlah gen yang terintegrasi di dalam genom dapat meningkatkan ekspresi protein heterolog (Vassileva et al. 2001; Mansur et al. 2005).
P. pastoris transforman stabil secara genetik dihasilkan melalui rekombinasi homologus antara sekuen promoter GAP di dalam vektor ekspresi dengan promotor yang sama di dalam genom P. pastoris. Proses integrasi diawali dengan vektor dipotong dengan BspHI pada promotor GAP. DNA vektor yang berbentuk linier menstimulasi proses terjadinya rekombinasi homologus di lokus yang terpotong melalui peristiwa pindah silang tunggal. Fragmen DNA linier yang dihasilkan mengandung casette ekspresi dan gen penanda (zeosin) yang diapit oleh 5’ dan 3’
GAP. Kondisi ini merangsang peristiwa penggantian gen pada GAP genom sehingga menyebabkan gen GAP digantikan oleh casette ekspresi beserta gen penanda (Li et al. 2007).
19 Uji Aktivitas Lipase
Uji aktivitas lipase dilakukan untuk mendeteksi dan mengetahui kemampuan
P. pastoris rekombinan dalam menghasilkan lipase. Uji aktivitas lipase dilakukan secara kualitatif dan kuantitatif. Uji aktivitas secara kualitatif hanya mengkonfirmasi adanya aktivitas lipase yang dihasilkan tetapi tidak bisa melihat besarnya aktivitas enzim yang dihasilkannya. Pada pengujian secara kualitatif ini media yang digunakan adalah media selektif dengan penambahan Rhodamin B dan Tween 80. Pewarna Rhodamin B berfungsi sebagai pengabsorbsi zona berpendar berwarna orange ketika kapang yang menghasilkan lipase berada di bawah sinar UV. Rhodamin B akan membentuk kompleks dimer dengan monoasilgliserol, diasilgliseol dan asam lemak bebas (Kouker dan Jaeger 1987). Tween 80 berfungsi sebagai sumber lipid (Samad et al. 1989). Media pertumbuhan dalam cawan petri ini digunakan untuk mendeteksi adanya asam lemak bebas sebagai hasil dari hidrolisis lipid.
Dari hasil pengujian aktivitas enzim ekstrak kasar lipase di bawah sinar UV pada media seleksi menunjukkan bahwa P. pastoris transforman yang diujikan menghasilkan lipase. Produk asam lemak yang dibebaskan oleh enzim dari proses hidrolisis menyebabkan terbentuknya warna disekitar koloni di bawah sinar UV. Koloni P. pastoris transforman mampu membentuk zona bening disekitar media (Gambar 15). Pendaran dan zona paling tinggi dari hasil pengujian tersebut belum dapat memastikan bahwa lipase rekombinan yang dihasilkan memberikan aktivitas yang paling tinggi. Oleh karena itu untuk mengetahui potensi dan aktivitas lipase rekombinan yang dihasilkan dalam mendegradasi lemak menjadi monogliserida, digliserida, dan asam lemak dibutuhkan pengujian hidrolisis menggunakan substrat asam lemak secara kuantitatif.
Gambar 15 Uji aktivitas enzim ekstrak kasar lipase secara kualitatif di bawah sinar UV pada media modifikasi dari Koeker dan Jaeger (1987) setelah 2 hari diinkubasi pada suhu ruang
20
Gambar 16 Kurva Pertumbuhan P. pastoris transforman
Kurva pertumbuhan P. pastoris dapat digunakan untuk melihat dan menggambarkan tahap-tahap dari siklus pertumbuhannya. Pertumbuhan P. pastoris
transforman meningkat seiring dengan waktu dimana terjadi peningkatan jumlah biomassa sel hingga mencapai titik maksimumnya pada jam ke-48 (hari ke-2). Kurva pertumbuhan tersebut dapat digunakan untuk memprediksi waktu optimum tingkat produksi enzim yang dihasilkan. Analisis aktivitas enzim dari sampel yang diambil dari kultur sel pada rentang waktu berbeda diperlukan untuk mengetahui waktu produksi lipase tertinggi yang dihasilkannya. Aktivitas enzim dihitung sampai jam ke 72 (hari ke-3) Berdasarkan hasil pengamatan, aktivitas enzim mengalami penurunan setelah hari ke 2 (jam ke-48). Penghitungan aktivitas lipase disajikan pada Lampiran 7.
Gambar 17 Aktivitas Lipase
Ekspresi Protein Rekombinan CALB
Analisis ekspresi protein rekombinan CALB dilakukan dengan menggunakan metode SDS-PAGE. SDS-PAGE memberikan informasi tentang ukuran (bobot molekul) dari protein dengan cara membandingkan ukuran protein yang dihasilkan dengan ukuran protein penanda dalam satuan kilodalton (kDa). Promotor yang digunakan dalam sistem ekspresi Pichia pada penelitian ini adalah promotor GAP, yaitu promoter yang mengatur ekspresi protein gliseraldehid 3-fosfat dehidrogenase (GAP) pada sel khamir. Promotor GAP pada P. pastoris mengendalikan ekspresi
21 protein secara konstitutif yang kuat pada medium glukosa (Gaffar 2010). Penggunaan promotor ini memungkinkan sel ditumbuhkan tanpa menggunakan agen penginduksi. Salah satu keuntungan menggunakan promotor GAP dibanding promoter AOX yang juga sering digunakan pada sistem ekspresi Pichia adalah tidak dibutuhkan metanol untuk induksi dan juga tidak dibutuhkan pemindahan kultur dari satu sumber karbon ke yang lain sehingga galur tumbuh lebih cepat (Li
et al. 2007).
Berdasarkan hasil analisis SDS-PAGE, ekspresi protein CALB telah berhasil dilakukan. Sampel supernatan protein yang telah dipekatkan dengan TCA menunjukkan multiband pada visualisasi SDS-PAGE. Hal ini ditunjukkan dengan munculnya pita protein rekombinan pada P. pastoris SMD1168H rekombinan dengan ukuran sekitar 45 kDa dan 66,2 kDa (Gambar 18). Protein dengan ukuran 45 kDa diperkirakan merupakan protein CALB yang tidak mengalami glikosilasi, sedangkan protein dengan ukuran 66,2 kDa diduga merupakan protein CALB yang mengalami glikosilasi. Menurut Moore (2009), pita protein yang terbentuk dengan berat molekul lebih tinggi dari protein yang diharapkan diakibatkan adanya protein yang mengalami glikosilasi atau banyaknya residu asam amino. Hasil analisis SDS-PAGE dengan pewarnaan coomassie blue juga menunjukkan adanya pola pita protein smear. Smear yang terbentuk pada gel akrilamid akibat adanya proses hiperglikosilasi pada glikoprotein yang dihasilkan sel khamir (Conde et al. 2004).
22
5
SIMPULAN DAN SARAN
Simpulan
Vektor ekspresi pGAPZα-CalBsyn-cbm yang mengandung gen cbm telah berhasil dikonstruksi. Plasmid rekombinan pGAPZα-CalBsyn telah berhasil diintroduksikan ke dalam genom P. pastoris SMD1168H. Protein CALB telah berhasil disintesis di P. pastoris SMD1168H. Uji aktivitas lipase secara kualitatif menunjukkan bahwa P. pastoris transforman mensekresikan lipase dan dari hasil uji aktivitas lipase secara kuantitatif menunjukkan bahwa aktivitas enzim mencapai 6,35 Units/mL pada jam ke-48. Hasil analisis dengan metode SDS-PAGE menunjukkan bahwa protein rekombinan berhasil diekspresikan secara ekstraseluler dengan ukuran sekitar 45 kDa.
Saran
Analisis lebih lanjut mengenai konstruksi vektor rekombinan
23
DAFTAR PUSTAKA
Ausubel FM, Brent R, Kingston RE, Moore DD, Seidman JG, Smith JA, Sruhl K. 2002. Short Protocol in Molecular Biology. 5th Edition. New York (US): John
Wiley & Sons Inc.
Balamurugan V, Reddy GR, Suryanarayana VVS. 2007. Pichia pastoris: A notable heterologous expression system for the production of foreign proteins-vaccines.
Indian J Biotechnol. 6: 175-186.
Blank K, Morfill J, Gumpp H, Gaub HE. 2006. Functional expression of Candida antarctica lipase B in Eschericia coli. J of Biotechnol. 125:474-483.
Cereghino GPL, et al. 2013. The effect of α-Mating Factor secretion signal mutations on recombinant protein expression in Pichia pastoris. Gene. 519(2): 311-317. doi:10.1016/j.gene.2013.01.062
Cereghino JL, Cregg JM. 2000. Heterologous protein expression in the methylotrophic yeast in Pichia pastoris. FEMS Microbiol. 24(1): 45-46. Doi: 10.1111/j.1574-6976.2000.tb00532.x.
Cho BK, Kieke MC, Boder ET, Wittrup KD, Kranz DM. 1998. A yeast surface display system for the discovery of Ligands that triger cell activation. J Immunol Methods. 220:179-188.
Classen D, Lee-son NM, Lin C, Menzies JM, Yew DS. 2002. Ability of Eschericia coli to distinguish between self and foreign DNA as demonstrated by trends in transformation efficiency. JEMI. 2: 201-206.
Conde C, Cueva R, Pablo G, Polaina J, Larriba G. 2004. A search for hyperglycosylation signals in yeast glycoproteins. The Journal of Biological Chemistry. 279(42): 43789-43798.
Cranenburg. RM. 2003. An equation for calculating the volumetrix ratios required in a ligation reaction. Appl Gene and MoleculBiotechnol. 65:200-202.
Daly R, Hearn MTW. 2005. Expression of heterologous proteins in Pichia pastoris: a useful experimental tool in protein engineering and production. J Mol Recognit. 18:119–138.
Fernandez L, Beerthuyzen MM, Brown J, Siezen RJ, Coolbear T, Holland R, Kuipers OP. 2000. Cloning, characterization, Controlled overexpression and inactivation of the major tributyrin esterase gene of Lactococcus lactis. Appl Environ. Microbial 66: 1360-1368.
Gaffar S. 2010. Produksi Protein Rekombinan dalam Sistem Ekspresi Pichia pastoris. Bandung (ID): Unpad Press.
Ghaly AE, Dave D, Brooks MS, Budge S. 2010. Production of biodiesel by enzymatic transesterification: Review. Am J Biochem & Biotech. 6(2): 54-76. Gilkes NR, Warren RA, Miller RJ, Kilburn DG. 1988. Precise excision of the
cellulose binding domain from two Cellulomonas fimi celulases by a homologous protese and the effect on catalysis. J. Biol. Chem. 263: 10401-10407. Glick BR, Pasternak JJ. 2003. Molecular Biotechnology. Washington : ASM Press.
760 halaman.
Gunasekaran V, Das D. 2005. Lipase Fermentation: Progress and Prospect. Indian J of Biotechnol. 4:437-445.
24
Gupta R, Rathi P, Gupta N, Bradoo S. 2003. Review lipase assays for conventional and molecular screening. Biotechnol Appl Biochem. 37(1): 63-71.
Hasan F, Shah AA, Hameed A. 2006. Industrial Aplications of Microbial Lipase.
Enzyme and Microbial Technol. 39(2): 235-251.
Ito J, Fujita Y, Ueda M, Fukuda H, Kondo A. 2004. Improvement of cellulose-degrading ability of a yeast strain displaying Trichoderma reesei endoglucanase II by recombination of cellulose-binding domain. Biotechnol. Prog. 20: 668-691. [Invitrogen]. 2001. EasySelectTM pichia expression kit: A manual of methods for
expression of recombinant proteins using pPICZ and pPICZα in Pichia pastoris. [Invitrogen]. 2002. pGAPZα A, B, and C pGAPZα A, B, and C Pichia expression
vectors for constitutive expression and purification of recombinant protein. Iso M, Chen B, Eguchi M, Kudo T, Shrestha S. 2001. Engineering a de novo internal
disulfide bridge to improve the thermal stability of xylanase from Bacillus stearothermophilus No. 236. J of Biotechnol. 127: 300-309.
Joseph B, Ramteke PW, Thomas G. 2008. Cold Active Microbial Lipases : Some hot issues and recent development. Biotechnol Advances.26:457-470.
Kim KK, Song HK, Shin DH, Hwang KY, Suh SW. 1997. The crystal structure of a triacylglicerol lipase from Pseudomonas cepacia reveals a highly open conformation in the absence of a bound inhibitor. Structure. 5: 173-185
Koleva D, Petrova V, Hristozova T, Kujumdzeiva A. 2008. Study of catalase enzyme in methylotrophic yeasts. Biotechnology and biotechnological. 22: 762-768.
Kouker G, Jaeger KE. 1987. Specific and sensitive plate assay for bacterial lipases.
Appl Environ Microb. 3(1): 211-213.
Li P, Anumanthan A, Gao XG, Ilangovan K, Suzara, VV, Duzgunes N, Renugopalakrishnan V. 2007. Expression of recombinant proteins in Pichia pastoris. Appl Biochem Biotechnol. 142: 105-124.
Lim KH, Hwang I, Park S. 2011. Biotin-assisted folding of streptavidin on the yeast surface. Biotechnol Prog. 28:276-283.
Mansur M, Cabello C, Hernandez L, Pais J. 2005. Multiple gene copy number enhances insulin precursor secretion in the yeast Pichia pastoris. Biotechnol Lett. 27(5): 339-345. doi: 10.1007/s10529-005-1007-7
Margolles-Clark E, Tenkanen M, Soderlund H, Pentilla M. 1996. Acetyl xylan esterase from Trichoderma reesei contains an active site serine and a cellulose-binding domain. Eur J Biochem. 237: 553-560
Mittlebach M, 1990. Lipase catalyzed alcoholisis of sunflower oil. JAOCS. 67(3): 168-170.
Nardini, M., Dijkstra, B.W. 1999. α/β hydrolase fold enzymes : the family keeps growing. Current opinion in Structural Biology. 9: 732-737
Ollis DL, Cheah E, Cygler M, Dijkstra B, Frolow F, Franken SM, Harel M, Remington SJ, Silman I, Schrag J, Sussman JL, Verschueren KHG, Goldman A. 1992. The α/β hydrolase fold. Protein Engineering. 5: 197-211.
Pfeffer J, Richter S, Nieveler J, Hansen CE, Rhild RB, Schmid RD, Rusnak M. 2006. High yield expression of lipase A from Candida antarctica in the methylotrophic yeast Pichia pastoris and its purification and characterisation.
Appl Microbiol and Biotechnol. 72: 931-938
25 Prescott LM, Harley JP, Klein DA. 2002. Microbiology. 5th edition. The McGraw
Hill Companies, North America: 1026 halaman
Quinn DM, Sirai K, Jackson RL, Harmony JK. 1982. Biochemistry. 21: 6872-6879. Rotticci-Mulder JC, Gustavsson M, Holmquist M, Hult K, Martinelle M. 2001. Expression in Pichia pastoris of Candida antarctica lipase B and lipase B fused to a cellulose-binding domain. Protein Expression and Purification. 21: 386-392. Samad MYA, Razak CNA, Salleh AB, Yunus W, Ampon K, Basri M. 1989. A plate
assay for primary screening of lipase activity. J of Microbiol Methods. 9: 51-56. Santarossa G, Lafranconi PG, Alquati C, DeGioia L, Alberghina L, Fantucci P, Lotti M. 2005. Mutation in the “lid” region affect chain length specificity and thermostability of a Pseudomonas fragii lipase. FEBS Letters. 579: 2383-2386. Sharma R, Chisti Y, Banerjee UC.2001. Production, Purification, Characterization,
and Application of Lipases. Biotechnology Advances. 19: 627-662.
Shirai K dan Jackson RL. 1982. Journal of Biological Chemistry. 257: 1253-1258. Shoseyov O, Shani Z, Ley I. 2006. Carbohydrate Binding Modules: Biochemical Properties and novel applications. Microbiology and Molecular Biology. 70(2): 283-295.
Stalbrand H, Saloheimo A, Vehmaanpera J, Henrissat B, Penttila M. 1995. Cloning and expression in Saccharomyces cerevisiae of Trichoderma reesei β -mannanase gene containing a cellulose binding domain. Appl. Environ. Microbiol. 61: 1090-1097.
Tawfeek KA, Fassi FA, Ramadan EM. 1989. Selection of methylotrophic microorganism for the formation of single cell protein. Science. 1: 25-38. Thanonkeo P, Monkeang R, Saksirirat W, Thanonkeo S. 2010. Cloning and
molecular characterization of glyceraldehyde-3-phosphate dehydrogenase gene from thermotolerant mushroom Lentinus polychrous. African Journal of Biotechnology. 9(22): 3242-3251.
Vassileva A, Chugh DA, Swaminathan S, Khanna N. 2001. Expression of hepatitis B surface antigen in the methylotrophic yeast Pichia pastoris using the GAP
promoter. J Biotechnol. 88: 21-35.
Wang Y, Slade MB, Gooley AA, Atwell BJ, Williams KL. 2001. Cellulose-binding Modules from extracelluler matrix protein of Dictyostelium discoideum stalk and sheath. Eur. J. Biochem. 268: 4334-4345.
Wu S, Letchworth GJ. 2004. High efficiency transformation by electroporation of
Pichia pastoris pretreated with lithium acetat and dithiothreitol. BioTechniques. 36: 152-154.
Zhang AL, Luo JX, Zhang TY, Pan YW, Tan YH, Fu CF. 2009. Recent advances on the GAP promotor derived expression system of Pichia pastoris. Mollecular Biology Report. 36: 1611-1619.
Zhang N, Suen WC, Windsor W, Xiao L, Madison V, Zaks A. 2003. Improving tolerance of Candida antarctica Lipase B towards irreversible thermal inactivation through directed evolution. Protein Engineering. 16(8): 599-605. Zhao L, Wang J, Dou X, Cao Z. 2009. Studying the unfolding process of protein G
26
Lampiran 1 Primer yang digunakan
Primer Sekuens nukleotida (5’ → 3’)
Mfa_F 5’-/5Phos/GCA TAT GAG ATT TCC TTC AAT TTT TAC TGC TGT-3’
Mfa_R 5’-/5Phos/GTT TCG AAA TAG TTG TTC AAT TGA TTG AAA TAG GG-3’
CalB_F 5’-TTG CCT TCA GGT TCA GAC C-3’
CalB_R 5’-GTC TAG AAT CGA TAG GAG TAA CTA TAC C-3’
Cbm_F 5’-GTTACTCCTATCGATTCTAGAAGCGCTGATTACAAGGA
CGATGAT-3’
Cbm_R 5’-GATGAGTTTTTGTTCTAGAGACAAACATTGTGAGTAG
TA ATCGT TAGAGTA-3’
27 Lampiran 2 Komposisi larutan dan media yang digunakan beserta cara
pembuatannya
Media dan Larutan Komposisi dan Cara Pembuatan GTE
(Glukosa / Tris / EDTA) (Larutan 1 lisis sel E.coli)
Sebanyak 10 mL 1 M glukosa steril, 4 mL 0,5 M EDTA steril dan 5,2 mL 0,96 M Tris-Cl pH 8 steril dicampurkan dengan akuades steril hingga volume mencapai 200 mL
1 M glukosa Sebanyak 9 g glukosa dilarutkan dalam 50 mL akuades dan disterilkan dalam autoklaf pada suhu 121ᴼC, tekanan 1 atm selama 15 menit 0,96 M Tris-Cl, pH 8 Sebanyak 6,05 g Tris-basa dilarutkan dalam 30
mL akuades dan ditambahkan 3 M HCl hingga mencapai pH 8,0. Larutan ditambahkan akuades hingga mencapai volume 52 mL dan disterilkan dalam autoklaf pada suhu 121ᴼC, tekanan 1 atm selama 15 menit.
0,5 M EDTA, pH 8,0 Sebanyak 3,722 g Na2EDTA.2H2O dilarutkan
dalam 14 mL akuades dan ditambahkan 10 M NaOH hingga mencapai volume 20 mL dan disterilkan dalam autoklaf pada suhu 121ᴼC, tekanan 1 atm selama 15 menit
NaOH / SDS
(Larutan 2 lisis sel E.coli)
Sebanyak 4 mL 10 M NaOH steril dan 20 mL 10% (b/v) SDS steril dilarutkan dalam akuades steril hingga volume mencapai 200 mL. Larutan disimpan pada suhu 4ᴼC.
10 M NaOH Sebanyak 8 g NaOH dilarutkan dalam 20 mL akuades dan disterilkan dalam autoklaf pada suhu 121ᴼC, tekanan 1 atm selama 15 menit. 10 % (b/v) SDS Sebanyak 10 g SDS dilarutkan dalam 100 mL
akuades dan disterilkan dalam autoklaf pada suhu 121ᴼC, tekanan 1 atm selama 15 menit.
Buffer TAE 50x, pH 4,8 Sebanyak 242 g Tris-basa, 57,1 mL asam asetat glasial dan 37,29 g Na2EDTA dilarutkan dalam
28
Larutan disterilkan dalam autoklaf pada suhu 121ᴼC, tekanan 1 atm selama 15 menit. Untuk membuat 0,5x buffet TAE (running
elektroforesis DNA), 10 mL 50x buffer TAE diencerkan dengan 990 mL akuades steril.
Loading dye 6x Sebanyak 6 mL gliserol, 0,05 g xylene cylanol, 0,05 g bromofenol blue dilarutkan dalam 20 mL akuades steril.
dilarutkan ke dalam akuades hingga mencapai volume 100 mL. Kemudian larutan disterilkan dalam autoklaf pada suhu 121ᴼC, tekanan 1 atm selama 15 menit. Larutan disimpan pada suhu membuat media padat. Media disterilkan dalam autoklaf pada suhu 121ᴼC, tekanan 1 atm dilarutkan ke dalam 90 mL akuades. Sebanyak 2 g bacto agar ditambahkan ke dalam media jika ingin membuat media padat. Media disterilkan dalam autoklaf pada suhu 121ᴼC, tekanan 1 atm selama 15 menit. Media ditambahkan dengan 10 mL 20% D-glukosa steril (20 gram D-glukosa dilarutkan dengan 100 mL akuades dan disterilkan dengan filter) ketika media akan digunakan.
YPDS (Yeast Peptone Dextrose Sorbitol)
29 Sebanyak 1,7 g bacto agar ditambahkan ke dalam media jika ingin membuat media padat. Media disterilkan dalam autoklaf pada suhu 121ᴼC, tekanan 1 atm selama 15 menit. Media ditambahkan dengan 10 mL 20% D-glukosa steril (20 g D-glukosa dilarutkan dengan 100 mL akuades dan disterilkan dengan filter) ketika media akan digunakan.
30
Lampiran 3 Kurva Standar ρ-Nitrophenyl Butyrate (PNPB) Blanko
Kontrol positif Lipoprotein Lipase Enzyme Solution (Candida rugosa) Menit Ke Absorbansi (400nm)
Menit Ke Aktivitas enzim (U/mL)
31 Lampiran 4 Komposisi gel poliakrilamid
Bahan Volume (mL)
Gel pemisah (15%) Gel penahan (8%) Akuabides
1,5 M Tris-HCl, pH 8,8 1,5 M Tris-HCl, pH 6,8 10% SDS
40% akrilamid
10% Amonium persulfat TEMED
2375 937,5
- 50 1625
25 2,5
1380 - 250
32
Lampiran 5 Analisis urutan DNA plasmid pGAPZα-CalBsyn dengan primer GAP_F
Gen CalBsyn
Gen CalBsyn
Sinyal Sekresi faktor-α
XhoI Sinyal Sekresi faktor-α
start
Sinyal Sekresi faktor-α BglII
33 Lampiran 6 Penentuan Efisiensi Transformasi
Jumlah koloni x pengenceran x Efisiensi Transformasi =
Konsentrasi plasmid
= 64 x 1 x