IDENTIFIKASI KANDIDAT GEN
Nucleotide Binding Site
(NBS)
DARI PISANG
Musa acuminata
Colla var.
malaccensis
(Ridl.)
Nasution DAN
Musa,
AAA, sub-kelompok Cavendish
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2015
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Identifikasi Kandidat Gen
Nucleotide Binding Site (NBS) dari Pisang Musa acuminata Colla var.
malaccensis (Ridl.) Nasution dan Musa, AAA, sub-kelompok Cavendish adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada LIPI dan Institut Pertanian Bogor.
Bogor, April 2015
Diyah Martanti
RINGKASAN
DIYAH MARTANTI. Identifikasi Kandidat Gen Nucleotide Binding Site (NBS) Dari Pisang Musa acuminata Colla var. malaccensis (Ridl.) Nasution dan Musa,
AAA, sub-kelompok Cavendish. Dibimbing oleh UTUT WIDYASTUTI, YUYU SURYASARI POERBA dan RITA MEGIA.
Indonesia merupakan negara yang kaya akan keragaman pisang, baik pisang budidaya maupun pisang liar. Namun, adanya penyakit layu fusarium oleh cendawan Fusarium oxysporum f.sp.cubense menyebabkan penurunan produksi pisang Indonesia. Pengembangan varietas pisang yang tahan penyakit, secara konvensional dihambat oleh waktu generasi yang panjang, partenokarpi, poliploidi dan sterilitas dari kebanyakan kultivar.yang dapat dimakan seperti pisang Cavendish. M. acuminata var. malaccensis merupakan pisang liar yang berbiji dan diploid dan mempunyai resistensi yang tinggi terhadap penyakit layu fusarium.Sehingga, pisang ini dapat dijadikan sebagai sumber gen untuk ketahanan penyakit.
Salah satu kelas gen resistensi (R) yang paling besar yang telah dikarakterisasi adalah Nucleotide Binding Site (NBS). Motif asam amino yang
conserved pada daerah NBS mencakup motif p-loop/kinase-1, kinase-2, GLPL/kinase-3 dan RNBS A,B,C dan D. Oleh karena itu, penelitian ini bertujuan untuk mengidentifikasi kandidat gen Nucleotide Binding Site (NBS) pada aksesi pisang liar M. acuminate var. malaccensis dan Musa, AAA, subkelompok Cavendish serta uji tantang terhadap Fusarium oxysporum f.sp. cubense TR4 pada
M. acuminate var. malacensis dan Musa, AAA, sub-kelompok Cavendish
Aksesi M. acuminate var. malaccensis (II.17B#5/LIPI010) dan Musa, AAA, sub-kelompok Cavendish (II.13B#3/LIPI090) yang digunakan dalam penelitian ini berasal dari Kebun Plasma Nutfah Pisang, Cibinong Science Center, Pusat Penelitian Biologi-LIPI. Metode CTAB digunakan dalam ekstraksi DNA dari daun. Tiga pasang primer degenerate digunakan dalam amplifikasi DNA. Fragmen yang diperoleh dilanjutkan dengan kloning dan sekuensing. Analisis BLASTp digunakan untuk menentukan homologi urutan basa nukleotida dan asam amino pada Genbank dan Musa Genome. Program CHROMAS PRO digunakan untuk analisis contig. Program MEGA5 dengan metode MUSCLE digunakan dalam pensejajaran asam amino dan kontruksi pohon filogenetik. Metode double tray digunakan dalam uji tantang terhadap Fusarium oxysporum
f.sp. cubense ras 4 pada M. acuminata var. malaccensis dan Musa, AAA, subkelompok Cavendish.
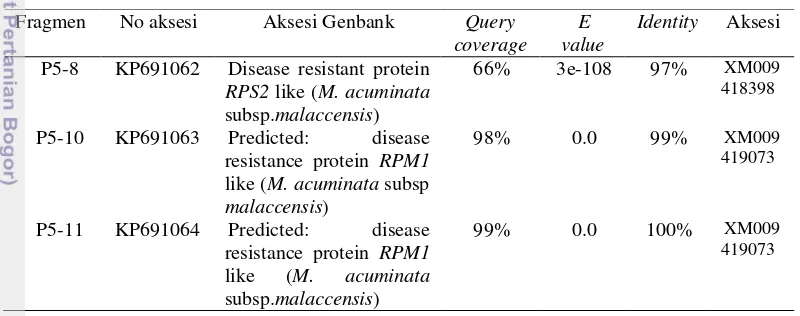
Dari tiga pasang primer degenerate, diperoleh 11 fragmen analog gen ketahanan yang berhasil diisolasi dari DNA pisang M. acuminate var. malaccensis
dan Musa, AAA, sub-kelompok Cavendish dengan produk berukuran antara 300-1000 pb. Dari 11 sampel produk PCR yang telah dikloning, semua mengandung insersi fragmen berukuran antara 323-1114 pb. Berdasarkan hasil analisis nukleotida dan deduksi asam amino dengan menggunakan analisis BLASTp yang terdapat di Genbank, ditemukan 3 sekuen yaitu P5-8, P5-10 dan P5-11 dengan ukuran berturut-turut 770, 1114 dan 791 bp.Sekuen P5-8 dan P5-10 berasal dari
disease resistance protein RPS2 pada M. acuminata var. malaccensis dengan aksesi XM009418398. Sedangkan sekuen P5-10 dan P5-11 mempunyai kesamaan identitas berturut-turut 99% dan 100% dengan disease resistance protein RPM1
pada M. acuminata var. malaccensis dengan aksesi XM009419073. BLAST pada
Musa Genome, hasilnya menunjukkan ketiga sekuen memiliki coverage > 90% dan terdapat pada kromosom nomor 9. Ketiga sekuen ini telah dideposit ke Genbank dengan nomor akses KP691062-KP691064.
Hasil pensejajaran prediksi asam amino dari ketiga fragmen dengan protein NBS-LRR tanaman pisang serta gen-R tanaman lain yang telah dideposit di Genbank menunjukkan terdapat empat struktur konservatif NBS. Keempat struktur konservatif asam amino yaitu motif P-loop/kinase-1a (GGVGKTT), kinase-2 (LVLDDIW), RNBS-B/kinase-3a (CKVLFTTRS) dan asam amino hidrofobik (hydrophobic domain) (GLPL).
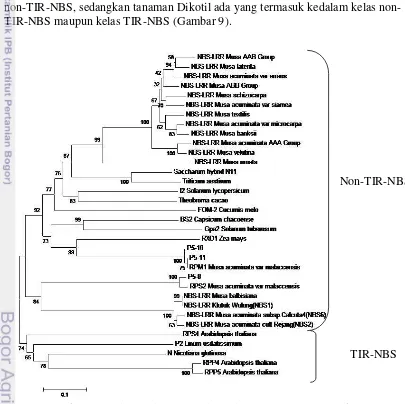
Hasil analisis filogenetik sekuen prediksi asam amino menunjukkan bahwa protein R dan protein pisang terbagi kedalam dua kelompok yaitu TIR-NBS dan non-TIR-NBS.Fragmen P5-8 tergabung satu kelompok dengan protein RPS2 dari
M. acuminate var. malaccensis dan protein NBS-LRR dari M. balbisiana. Sedangkan, fragmen P5-10 dan P5-11 tergabung satu kelompok dengan berturut-turut protein RPM1 dari M. acuminate var.malaccensis dan protein RX01 dari jagung. Fragmen yang diperoleh dari penelitian ini bersama protein pada pisang lain dan tanaman monokotil seperti tebu, gandum dan jagung termasuk kedalam kelas non-TIR-NBS.
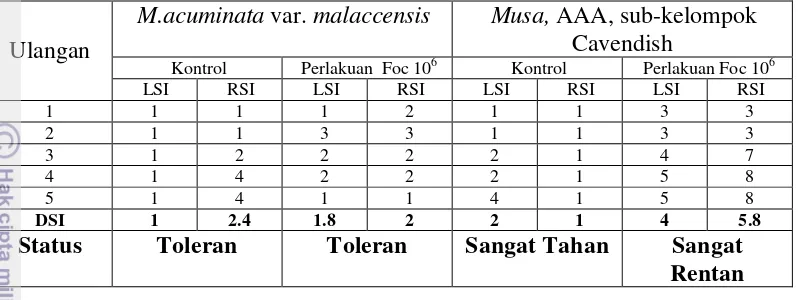
Hasil pengamatan daun dan bonggol terhadap Foc TR4menunjukkan bahwa pisang M. acuminate var. malaccensis berada pada status toleran, sedangkan
Musa, AAA, sub-kelompok Cavendish berada pada status sangat rentan. Hal ini menunjukkan bahwa M. acuminata var. malaccensis lebih tahan daripada Musa,
AAA, sub-kelompok Cavendish
Kata Kunci: Pisang, Nucleotide Binding Site, daerah konservatif, Musa acuminata
SUMMARY
DIYAH MARTANTI. Identification of Gene Candidate of Nucleotide Binding Site (NBS) from Banana Musa acuminata Colla var. malaccensis (Ridl.) Nasution and Musa, AAA, Cavendish sub-group. Supervised by UTUT WIDYASTUTI, YUYU SURYASARI POERBA and RITA MEGIA.
Indonesia is one of the centers of banana genetic diversity in the world, both cultivated and wild. However, the presence of fusarium wilt which is caused by the fungus onescan reduced banana production in Indonesia.The disease-resistant banana varieties improvement, conventionally inhibited by long time generation, parthenocarpy, polyploidy and sterility of the most cultivars like Cavendish. Musa acuminatavar.malaccensisis a wild seeded diploid banana and it has a high resistance to Fusarium oxysporum f.sp. cubense TR4. Thus, it can be used as a source of genes for disease resistance.
The largest class of resistance (R) genes is Nucleotide Binding Site (NBS). Conserved amino acid motifs in the NBS region include P-loop (1), kinase-2, GLPL (kinase-3) and RNBS A, B, C and D motifs. Therefore, the aim of this study was to identify the gene candidate of Nucleotide Binding Site (NBS) on the accession of M. acuminata var. malaccensis and Musa, AAA, Cavendish subgroup and bioassay to Fusarium oxysporum f.sp cubense in M. acuminata var.
malaccensis and Musa, AAA, Cavendish sub-group.
The banana cultivars used in this study consist of a wild banana M. acuminata var. malaccensis (II.17B#5/LIPI010) and Musa, AAA, Cavendish sub-group (II.13B#3/LIPI090) collected from Banana Germplasm Gardens, Research Center for Biology, Indonesian Institute of Sciences. CTAB method was used to extract DNA from leaves.Three pairs of degenerate primers were used to DNA amplification. The fragments were cloned and sequenced. BLASTp analysis was used to determine homology of nucleotide and amino acid in Genbank and Musa Genome. CHROMAS PRO program was used to contig analysis. MEGA5 resistance gene fragments that were isolated from M. acuminata var. malaccensis
andMusa, AAA, Cavendish sub-group. The size of the fragments between 300-1000 bp. The PCR product of the 11 fragments have been cloned. All of the fragments contained the inserted fragment between 323-1114 bp in size. Based on the analysis of nucleotide and the deduced amino acid using BLASTp analysis in Genbank, it was found that three sequences consisted of P5-8, P5-10, and P5-11 with the size of 770, 1114, and 791 bp respectively. P5-8 and P5-10 were isolated from Musa acuminata var. malaccensis and P5-11 was isolated from Musa, AAA, Cavendish sub-group.P5-8 has97% similarityto the disease resistance protein
RPS2 on M. acuminata subsp. malaccensis(XM009418398). While, P5-10 and P5-11 have 98% and 100% similarity to the disease resistance protein RPM1 on
M. acuminata subsp. malaccensis (XM009419073). BLAST in Musa genome
Contig sequences were deposited in Genbank and assigned as number KP691062 - KP691064.
Sequence allignment between amino acid of three fragments, NBS-LRR protein from bananas, and R-gene from other plants that have been deposited in Genbank showed that there were four conservative structure of NBS.The four conserved regions of amino acids wereP-loop/kinase-1a (GGVGKTT), kinase-2 (LVLDDIW), RNBS-B/kinase-3a (CKVLFTTRS) and hydrophobic amino acids (hydrophobic domain) (GLPL).
The phylogenetic analysis showed that R proteins and bananas protein are divided into two groups: TIR-NBS and non-TIR-NBS. P5-8 fragment was grouped in RPS2 protein from M. acuminata var. malaccensis. Meanwhile, P5-10 and P5-11 fragments were grouped in RPM1 protein from M. acuminata var.
malaccensis and RX01 protein from corn respectively. These fragments with another banana and protein in monocotyledonous plant such as sugarcane, wheat, and corn were included in the class of non-TIR-NBS.
The result of observation to leaves and rhizomes showed that M. acuminate
var. malaccensis was in tolerant status, meanwhile Musa, AAA,Cavendish sub-group was in highly susceptible status. That showed that M. acuminata var.
malaccensisis more resistant than Musa, AAA, Cavendish sub-group.
Key words: Banana, Nucleotide Binding Site, conserved domain, Musa acuminate
© Hak Cipta Milik IPB& LIPI, Tahun 2015
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB &LIPI
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Biologi Tumbuhan
IDENTIFIKASI KANDIDAT GEN
Nucleotide Binding Site
(NBS)
DARI PISANG
Musa acuminata
Colla var.
malaccensis
(Ridl.)
Nasution DAN
Musa,
AAA, sub-kelompok Cavendish
DIYAH MARTANTI
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
Judul Tesis : Identifikasi Kandidat Gen Nucleotide Binding Site (NBS) dari Pisang
Musa acuminata Colla var.malaccensis (Ridl.) Nasution dan Musa,
AAA, sub-kelompok Cavendish Nama : Diyah Martanti
NIM : G353110181
Disetujui oleh Komisi Pembimbing
Dr Ir Utut Widyastuti, MSi Ketua
Dr Yuyu Suryasari Poerba Anggota
Dr Rita Megia, DEA Anggota
Diketahui oleh
Ketua Program Studi Biologi Tumbuhan
Dr Ir Miftahudin, MSi
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
PRAKATA
Puji syukur penulis panjatkan kehadirat Allah SWT atas segala berkah, rahmat dan karunia-Nya, sehingga karya ilmiah ini berhasildiselesaikan dengan baik. Tema yang saya pilih dalam penelitian ini ialah genetika molekuler yang berjudul “Identifikasi Kandidat Gen Nucleotide Binding Site (NBS) dari Pisang
Musa acuminata Colla var.malaccensis (Ridl.) Nasution dan Musa acuminata
Colla cv.Cavendish (AAA). Penelitian ini telah dilaksanakan pada bulan Februari 2013 sampai dengan bulan Desember 2014, bertempat di Laboratorium Genetika Tumbuhan, Pusat Penelitian Biologi LIPI.
Penelitian ini dapat dilaksanakan atas bantuan karyasiswa LIPI 2011 dan ditunjang oleh beberapa kegiatan penelitian di Pusat Penelitian Biologi LIPI, yaitu program Kompetitif LIPI Sub-Program Domestikasi dan Pemanfaatan Terukur dengan tema „Pemuliaan pisang untuk ketahanan terhadap penyakit layu
Fusarium‟ tahun 2012 dan 2013.
Penulis menyampaikan banyak terimakasih kepada Ibu Dr Ir Utut Widyastuti, MSi., Ibu Dr Yuyu Suryasari Poerba dan Ibu Dr Rita Megia, DEA.yang telah memberikan bimbingan, saran dan masukan. Penulis juga mengucapkan banyak terimakasih kepada Bapak Dr Ir Aris Tjahjoleksono, DEA selaku dosen penguji atas saran dan masukannya. Ungkapan terimakasih juga disampaikan kepada Ibu Dr Kusumadewi Sri Yulita, Dr. Marlina Ardiyani, Achirul Nditasari, MSc., Dewi Wulansari, MSc. dan Bapak Dr Iman Hidayat yang telah memberikan saran dalam pengerjaan penelitian. Kepada seluruh staff dan teknisi Laboratorium Genetika Tumbuhan, Laboratorium Biak Sel dan Jaringan serta Laboratorium Mikrobiologi, Pusat Penelitian Biologi LIPI, penulis ucapkan terimakasih banyak atas bantuannya.
Ungkapan terimakasih dan cinta yang tak terhingga atas doa dan dukungan seluruh keluarga besar, suami Gembong Agronomianto dan putri Alya Zahra Afifatulkhonsa serta Bapak/Ibu Sugito Surodiharjo dan Bapak/Ibu Syahidin.Semoga Allah SWT senantiasa memberikan rahmat dan kemuliaan kepada semua pihak yang telah membantu dalam penelitian ini.
Semoga karya ilmiah ini memberikan manfaat bagi kemajuan ilmu pengetahuan Indonesia.
Bogor, April 2015
DAFTAR ISI
DAFTAR TABEL xiii
DAFTAR GAMBAR xiii
1 PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 3
2 TINJAUAN PUSTAKA 3
Musa acuminata Colla 3
Gen Ketahanan Nucleotide Binding Site- Leusine Rich Repeat 4
3 BAHAN DAN METODE 6
A. Identifikasi Kandidat Gen Nucleotide Binding Site (NBS) Dari Pisang
Musaacuminata Colla var.malaccensis (Ridl.) Nasution dan Musa, AAA, sub-kelompok Cavendish 6
Waktu dan Tempat Penelitian 6
Bahan Penelitian 6
Ekstraksi DNA total 6 Amplifikasi DNA 6
Analisis sekuen DNA 7
B. Uji tantang terhadap cendawan Fusarium oxysporum f.sp
cubenseTR4 7
Waktu dan Tempat Penelitian 7
Bahan tanaman dan isolat cendawan 7
Persiapan bahan uji 8
Pengujian ketahanan terhadap Fusarium oxysporum f.sp. cubenseTR4 8 Pengamatan dan analisis data 8
4 HASIL DAN PEMBAHASAN 10
Amplifikasi PCR dengan menggunakan primer degenerate 10 Analisis sekuen fragmen produk PCR dari DNA genom pisang 11 Analisis pensejajaran sekuen dan filogenetik prediksi asam amino dengan
protein RGA dan gen-R tanaman lain 13
Uji tantang terhadap Fusarium oxysporum f.sp.cubenseTR4 16
5 SIMPULAN DAN SARAN 18
UCAPAN TERIMAKASIH 18
DAFTAR PUSTAKA 19
DAFTAR TABEL
1 Primer yang digunakan untuk mengisolasi gen NBS dari genom pisang 7 2 Index kategori ketahanan 9 3 Sequence identity antara sekuen prediksi asam amino tanaman pisang
Musa acuminatavar.malaccensis dan Musa, AAA, sub-kelompok Cavendish dengan protein ketahanan penyakit Musa yang telah dideposit pada
GenBank. 11
4 BLAST di Musa genome 12
5 Status ketahanan/kerentanan Musa acuminatavar.malaccensis dan Musa, AAA, sub-kelompok Cavendish4 minggu setelah inokulasi Foc TR4
konsentrasi 106 konidia/ml 18
DAFTAR GAMBAR
1 Negara-negara penghasil pisang tertinggi tahun 2012 1 2 Total produksi buah nasional tahun 2013 2 3 Representasi dari kelas R-gen utama yang mengandung daerah
konservatif 5
4 SkorLeaf Symptom Index (LSI) 8
5 SkorRhizome Discoloration Index (RDI) 9
6 Hasil amplifikasi DNA dengan menggunakan primer degenerate 10 7 Kromosom pisang 12 8 Analisis pensejajaran sekuen prediksi asam amino dengan beberapa
protein Musa pada daerah NBS dan protein R yang terdeposit pada
genbank NCBI 14
9 Pohon filogenetik sekuen prediksi asam amino dari RGA pisang dan beberapa protein Musa dan protein tanaman lain berdasarkan
analisis pensejajaran menggunakan MUSCLE dan dibuat berdasarkan
metode NJ 15
10 Uji tantang ketahanan terhadap Fusarium oxysporum f.sp cubenseTR4
1 PENDAHULUAN
Latar Belakang
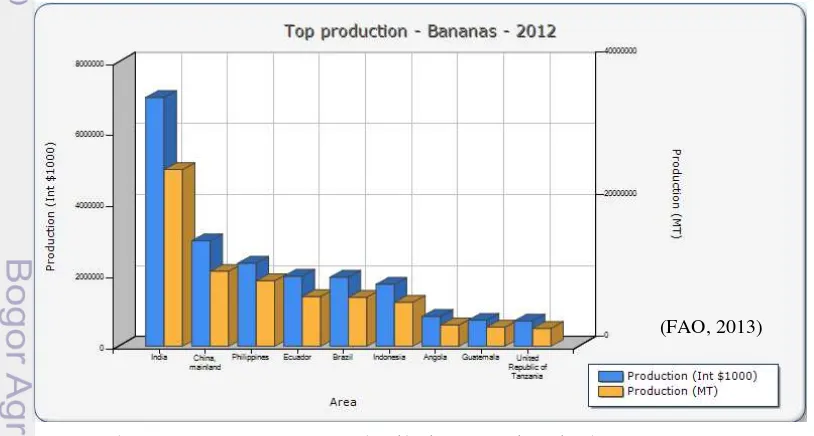
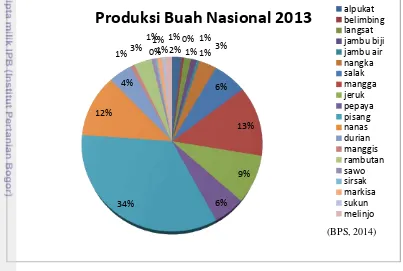
Indonesia merupakan negara yang kaya akan keragaman pisang, baik pisang budidaya maupun pisang liar. Pada tahun 2012, Indonesia menduduki peringkat ke-6 dunia dalam produksi pisang sebanyak 6.189.052 ton setelah India, China, Filipina, Ekuador dan Brazil(Gambar 1) (FAO 2013). Di Indonesia, produksi pisang mencapai 5.359.126 ton (34%) dari keseluruhan produksi buah nasional yang mencapai 15.767.472 ton pada tahun 2013 (Gambar 2) (BPS 2014). Namun, adanya penyakit layu fusarium menyebabkan 1300 hektar pisang Barangan milik petani di Sumatera Utara dan 300 hektar perkebunan pisang Cavendish untuk tujuan ekspor ke Singapura di Riau pada tahun 1995 menjadi rusak berat (Jumjunidang et al.2005). Penyakit layu fusarium disebabkan oleh cendawan
Fusarium oxysporumf.sp.cubense(Foc) yang mampu hidup di dalam tanah dalam bentuk klamidospora dan dapat bertahan di tanah dalam jangka waktu lama,akibatnya sulit untuk dikendalikan (Widono et al. 2003). Cendawan ini akan berkecambah untuk menginfeksi akar lateral ketika kontak dengan akar pisang dan akan membentuk koloni yang menyumbat sistem vaskular tanaman, sehingga menyebabkan layu dan kematian tanaman pisang. Tropical Race 4(TR4) dari cendawan ini menjadi ras yang paling virulen pada saat ini sehingga dapat menginfeksi hampir semua tanaman kultivar pisang, termasuk yang tahan terhadap ras lain dari penyakit layu fusarium (Sun et al. 2010).
Gambar 1 Negara-negara penghasil pisang tertinggi tahun 2012
Pengembangan varietas pisang yang tahan penyakit, secara konvensional dihambat oleh waktu generasi yang panjang, triploid, partenokarpi dan sterilitas dari kebanyakan kultivar. yang dapat dimakan. M. acuminata Colla liar merupakan pisangyang berbiji dan diploid. Di Indonesia, ada 15 varietas M.
2
acuminata liar tersebar dari Sumatera sampai Papua yaitu M. acuminata var.
acuminata, var. alasensis Nasution, var. bantamensis Nasution, var. breviformis
Nasution, var. cerifera Back. Nasution, var. flava (Ridl.) Nasution, var.
halabanensis (Meijer) Nasution, var. longipetiolata Nasution, var. malaccensis
(Ridl.) Nasution, var. microcarpa (Becc.) Nasution, var. nakaii Nasution, var.
rutilifes (Back.) Nasution, var. sumatrana (Becc.) Nasution, var. tomentosa
(K.Sch.) Nasution, and var. zebrina (v.Houtte) Nasution (Nasution 1991). Menurut Kayat et al. (2004) pisang liar Musa acuminata var. malaccensis
mempunyai resistensi yang tinggi terhadap penyakit layu Fusarium oxysporum
f.sp.cubense TR4. Sehingga, pisang liar ini dapat dijadikan sebagai sumber gen untuk ketahanan penyakit.
Gambar 2 Total produksi buah nasional tahun 2013
Salah satu kelas gen resistensi (R) yang paling besar yang telah dikarakterisasi adalah Nucleotide Binding Site- Leusine Rich Repeat (NBS-LRR). Daerah NBS penting untuk pengikatan ATP dan hidrolisis serta diyakini terlibat dalam sinyal transduksi yang dipicu oleh infeksi patogen. Motif asam amino yang
conserved pada daerah NBS mencakup motif p-loop/kinase-1, kinase-2, Glysine-Leusine-Proline-Leusine (GLPL)/kinase-3 dan Resistance NBS (RNBS) A,B,C dan D (Meyers et al. 1999). Daerah LRR terlibat dalam interaksi berbagai protein dan mengenali molekul elisitor dari patogen (Fluhr 2001). Dalam penelitian ini, hanya daerah NBS saja yang diidentifikasi sedangkan daerah LRR tidak. Sun et al.(2010), mendapatkan 20 fragmen gen analog ketahanan layu fusarium pada
pisang „Goldfinger‟ dengan ukuran sekitar 530 bp dan termasuk ke dalam
3 banyak dilakukan yaitu pada tomat dan Arabidopsis (Pan et al. 2000), kentang (Bendahmaneyet al. 2002) dan tebu (Glynn et al. 2008). Sedangkan penelitian pada tanaman pisang diantaranya mencakup pisang liar M. acuminata Colla (Miller et al. 2008; Peraza-Echeverria et al. 2008), pisang Cavendish (Li et al. 2012), pisang Goldfinger (Sun et al. 2010) dan Musa AAB cv. Nendran
(Augustine & Joseph 2014).
Tujuan Penelitian
Penelitian ini bertujuan untuk mengidentifikasi kandidat gen Nucleotide Binding Site (NBS) pada aksesi pisang liar M. acuminata Colla var. malaccensis
(Ridl.) Nasution dan Musa, AAA, sub-kelompok Cavendish dan uji tantang terhadap cendawan Fusarium oxysporum f.sp. cubenseTR4 pada M. acuminata
Colla var. malaccensis(Ridl.) Nasution dan Musa, AAA, sub-kelompok Cavendish
.
2 TINJAUAN PUSTAKA
Musa acuminata Colla
Genus Musa merupakan salah satu dari tiga genus yang termasuk kedalam keluarga Musaceae dari ordo Zingiberales. Berdasarkan jumlah kromosom, genus
Musa dibagi kedalam lima section. Dua section Callimusa dan Australimusa
mempunyai jumlah kromosom 2n=20, dua section, Eumusa dan Rhodochlamys
mempunyai jumlah kromosom 2n=22, dan section terakhir Ingentimusa
mempuyai jumlah kromosom 2n=14 (Horry et al. 1997).Genus Musa merupakan yang terbesar dalam section Eumusa yang termasuk kedalamnya yaitu semua pisang budidaya, M. acuminata dan M. balbisiana (Simmond 1962).
Saat ini, Hakkinen (2013) membagi genus Musa kedalam dua section, yaitu
Musa dan Callimusa berdasarkan analisis DNA. Section Musa terdiri dari subgenus Rhodochlamys dan Eumusa. Section Callimusa terdiri dari Australimusa
dan Ingentimusa.
Musa acuminata Colla berasal dari Asia Tenggara. Di Indonesia, ada 15 varietas M. acuminata liar tersebar dari Sumatera sampai Papua yaitu
M.acuminata var. acuminata, var. alasensis Nasution, var. bantamensis Nasution, var. breviformis Nasution, var. cerifera Back. Nasution, var. flava (Ridl.) Nasution, var. halabanensis (Meijer) Nasution, var. longipetiolata Nasution, var.
malaccensis (Ridl.) Nasution, var. microcarpa (Becc.) Nasution, var. nakaii
Nasution, var. rutilifes (Back.) Nasution, var. sumatrana (Becc.) Nasution, var.
4 diklasifikasikan sebagai AA, AB, AAA, AAB, ABB, AABB, AAAB, ABBB dengan sumber genom A berasal dariM. acuminata dan genom B dari M. balbisiana (Simmonds & Shepperd 1955). Pada saat ini, klasifikasi pisang budidaya menggunakan sistem genome score card untuk menentukan jenis genom pada pisangberdasarkan morfologi (Singh & Uma 1996), flowcytometer untuk menentukan tingkat ploidi dan marka molekular untuk menentukan komposisi genom.
Menurut Kayat et al. (2004) pisang liar M. acuminata var. malaccensis
mempunyai resistensi yang tinggi terhadap penyakit layu Fusarium oxysporum
f.sp. cubense TR4. Sehingga, pisang liar ini dapat dijadikan sebagai sumber gen untuk ketahanan penyakit.
Gen Ketahanan Nucleotide Binding Site-Leusine Rich Repeat
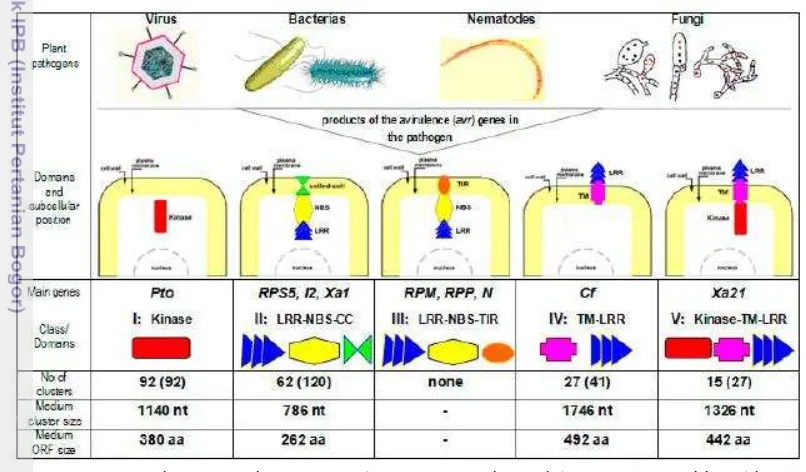
Sebagai sumber resistensi terhadap adanya patogen di dalam plasma nutfah, khususnya pada genus Musa, adanya gen ketahanan (gen-R) dalam tanaman menawarkan potensi untuk pemuliaan non-konvensional. Genotipe tanaman yang resisten dapat mencegah patogen masuk melalui gene for gene mechanism, yaitu diinisiasi melalui interaksi langsung maupun tidak langsung antara produk gen ketahanan konstitutif (gen R) dan produk gen avirulen biotropik patogen spesifik (gen Avr) atau elisitor. Interaksi ini memicu rantai transduksi sinyal yang mengaktivasi mekanisme pertahanan seperti respon hipersensitif (HR), sintesis protein dan metabolit anti mikrobia, penebalan dinding sel dan penghambatan vaskuler. Selama lebih dari 15 tahun, lebih dari 40 gen-R telah dikarakterisasi dari tanaman model dan tanaman pertanian yang penting (Martin et al. 2003).
Ada lima kelas gen ketahanan yang telah diidentifikasi berdasarkan daerah domain protein yang konservatif. Kelas yang paling banyak adalah domainprotein
Nucleotida Binding Site- Leusine Rich Repeat (NBS-LRR) (Rommens & Kishore 2000). Kelas lainnya merupakan protein dengan ekstrasitoplasmik LRRs (eLRRs) terikat pada daerah transmembran (TM), reseptor serine-threonin (Ser/thr) mirip kinase dengan ekstraseluler LRR, Ser/Thr sitoplasmik tanpa LRR) dan protein dengan membran terikat pada daerah coiled coil (CC domain) (Gambar 3).
Protein NBS-LRR merupakan protein yang paling banyak termasuk ke dalamnya lebih dari 20 gen R fungsional dari berbagai spesies tanaman. Protein ini berukuran besar, berlimpah dan terlibat dalam mendeteksi berbagai patogen termasuk bakteri, virus, cendawan, nematoda, serangga dan oomycetes (McHale
et al. 2006).
Daerah NBS penting untuk pengikatan ATP dan hidrolisis ATPserta diyakini terlibat dalam transduksi sinyal yang dipicu oleh infeksi patogen. Daerah ini mengandung beberapa motif yang khas dari transduksi sinyal ATP-ase dengan sejumlah protein Signal Ttransduction ATPases with Numerous Domains
5 Laju mutasi yang tinggi pada daerah LRR menyebabkan keragaman genetik diperlukan dalam pengenalan secara spesifik rekombinasi keanekaragaman patogen (Michelmore & Meyers 1998). Dua subfamili yang terdapat pada protein NBS-LRR berdasarkan motif N-terminal yaitu TIR dan Non-TIR. Subfamili TIR-NBS menunjukkan homologi antara motif asam amino N-terminal dan daerah
Toll Like Receptor (TLRs) pada Drosophilladan Interleukin (IL) pada mamalia sebagai faktor imunitas pada hewan. Subfamili ini terdapat terbatas pada Dikotil dan Gymnospermae (Parker et al. 1997). Subfamili Non-TIR dapat mengandung motif coiled-coil yang merupakan bagian dari Leucine Zipper Sequence (LZ), terdapat dalam Monokotil dan Dikotil. Asam amino konservatif mengandung
ikatan fosfat loop atau “P loop” (disebut Kinase 1), Kinase 2, GLPL (disebut
Kinase 3) dan motif RNBS-A,B, C dan D. Asam amino terakhir pada motif Kinase 2 dapat berbeda antara tipe TIR dan non-TIR, dengan residu asam aspartat pada TIR dan triptofan pada non-TIR (Meyers et al. 1999).
Gambar 3. Representasi dari kelas gen-R utama yang mengandung daerah konservatif.
6 pemindahan sifat tahan penyakit dari kultivar liar ke kultivar komersial yang rentan penyakit (Arvanitoyannis et al. 2008).
3
BAHAN DAN METODE
A.Identifikasi Kandidat Gen Nucleotide Binding Site (NBS) dari Pisang M. acuminata Colla var.malaccensis (Ridl.) Nasution danMusa, AAA, sub-kelompok Cavendish
Waktu dan Tempat Penelitian
Penelitian dilaksanakan mulai bulan Februari 2013-Oktober 2014 di Laboratorium Genetika Tumbuhan,Pusat Penelitian Biologi, LIPI.
Bahan Penelitian
Dua aksesi pisang yang digunakan dalam penelitian ini yaitu pisang liar M. acuminata var. malaccensis (II.17B#5/LIPI010) dan pisang budidaya Musa,
AAA, sub-kelompok Cavendish(II.13B#3/LIPI090) yang dikoleksi dari Kebun Plasma Nutfah Pisang, Pusat Penelitian Biologi LIPI. Material DNA yang digunakan berasal dari daun muda tanaman pisang yang ditanam di kebun Plasma Nutfah Pisang, Pusat Penelitian Biologi LIPI.
Ekstraksi DNA Total
DNA total diekstrak dari daun muda menggunakan metode
Cetyltrimetylammoniumbromide (Delaporta et al.1983; Syamkumar et al. 2003) yang telah dimodifikasi dengan penambahan Polyvinylpirrolidone (PVP) pada
saat penggerusan dan β-Mercaptoethanol pada buffer ekstraksi.
Amplifikasi DNA
Amplifikasi DNA menggunakan primer degenerate berdasarkan daerah konservatif gen NBS-LRR. Tiga pasang primer yang telah didesain oleh Sun et al.
(2010) digunakan untuk mengisolasi analog gen R dari DNA genom pada pisang Goldfinger (AAAB) yang tahan penyakit layu fusarium (Tabel 1).
Campuran reaksi PCR sebanyak 15 µl mengandung 7.5 µl GoTaq® Green Master Mix 2X, 0.75 µl 20 pmol primer forward dan reverse, 1 µl DNA 50 ng/µl dan ditambah air sampai 15 µl. Kondisi PCR meliputi predenaturasi pada 95oC selama 3 menit, kemudian diikuti 40 siklus dengan denaturasi pada 94oC selama 45 detik, annealing masing-masing pada 44oC (F1+F2); 54oC (F5+F6); 58oC (F9+F10) selama 30 detik dan elongasi pada 72oC selama 60 detik, dengan tahap pasca elongasi pada 72oC selama 10 menit. Proses PCR menggunakan mesin
7
μl hasil PCR (50 ng), 1 μl pGEM-T Easy vektor 25 ng, 1 μl T4 DNA ligase 5 Unit, 5 μl 2x rapid buffer ligasi dan H2O sampai volume 10 ml, kemudian
diinkubasipada 4°C semalam. Hasil ligasi ditransformasikan kedalam bakteri E. coliJM109 (Promega, USA) dengan metode heatshock dari Maniatis et al.(1982). Plasmid yang mengandung rekombinan diekstrak menggunakan High Speed Plasmid Mini Kit (GenAid) dan dikirim ke perusahaan jasa sekuensing 1st Base
untuk proses sekuensing.
Tabel 1 Primer yang digunakan untuk mengisolasi gen NBS dari genom pisang Primer
Degenerate
Peptida Sekuen Primer Referensi
F1(F) P-loop GGDGTDGGNAARACWAC Deng et al., 2000 F2(R) GLPL AANGCHAGNGGYAANCC Deng et al., 2000
F5(F) P-loop GGIGGIGTIGGIAAIACIAC Peraza-Echeverria et al.2008 F6(R) GLPL AAGIGCTAAGIGGIAAGICC Peraza-Echeverria et al.2008 F9(F) P-loop GGNGGNRTIGGIAARACIAC Sun et al., 2010
F10(R) GLPL GAGGGCNARNGGNAAICC Sun et al., 2010
catatan: kode untuk basa campuran: R=A/G, W=A/T, Y=C/T, H=A/T/C, D=A/T/G, N=A/G/T/C, I=Hypoxanthine
Analisis sekuen DNA
Analisis sekuen produk PCR yang telah teridentifikasi dengan cara membandingkan sekuen DNA dan prediksi asam amino produk PCR tersebut dengan aksesi tanaman lain yang terdapat dalam pangkalan data Genbank NCBI menggunakan algoritma Basic Local Allignment Search Tool untuk protein BLASTp (www.blast.ncbi.nlm.nih.gov/Blast.cgi) dan BLAST di Musa
Genome(http://banana-genome.cirad.fr). Analisis contig sekuen DNA
menggunakan program Chromas Pro. Analisis pensejajaran (allignment) dengan metode Multiple Sequence Allignment (MUSCLE) dan pohon filogenetik dikonstruksi dengan metode neighbor-joining dengan program Molecular Evolutionary Genetics Analysis (MEGA5) (Tamura et al. 2011). Bootstrap 1000 kali ulangan digunakan untuk mengevaluasi derajat pola pengelompokan pada pohon filogenetik.
B.Uji tantang terhadap cendawan F. oxysporum f.sp.cubenseTR4
Waktu dan Tempat Penelitian
Penelitian dilaksanakan di rumah kaca laboratorium Genetika Tumbuhan, Pusat Penelitian Biologi, LIPI pada bulan Januari-Desember 2014.
Bahan Tanaman dan Isolat Cendawan.
Tanaman yang digunakan yaitu M. acuminata var. malaccensis (II. 17B#5/ LIPI010) dan Musa, AAA, sub-kelompok Cavendish (II.13B#3/ LIPI090). Isolat
8
Persiapan Bahan Uji
Bahan tanaman yang digunakan untuk uji tantang adalah plantlet hasil perbanyakan kultur jaringan yang ditanam dalam media pertumbuhan optimum selama 2 bulan dengan tinggi mencapai 40-50 cm. Sebanyak 5 tanaman untuk setiap nomor aksesisebagai ulangan.
Isolat F. oxysporum f.sp. cubense TR4 diremajakan dalam media Potato Dextrose Broth (PDB) dan diinkubasi dalam suhu ruang selama 14 hari. Konsentrasi inokulum yang dikehendaki sebesar 106 konidia ml-1 dihitung menggunakan Haemocytometer.
Pengujian ketahanan terhadap F. oxysporum f.sp. cubenseTR4.
Pengujian berdasarkan metoda Mak et al. (2004) dengan menggunakan teknik wadah ganda (double tray). Wadah pertama dengan bagian bawah seperti saringan untuk menaruh polibag berisi tanaman. Sedangkan wadah kedua berada dibawah wadah pertama untuk menampung kelebihan air dan nutrisi.
Perlakuan meliputi kontrol dan 106 konidia ml-1. Plantlet diinokulasi dengan larutan 106 konidia ml-1 dengan cara merendam akar tanaman selama 2 jam, kemudian ditanam dalam polibag berisi pasir steril. Penyiraman dilakukan setiap hari dan pemupukan menggunakan pupuk cair Growmore dilakukan seminggu sekali.
Pengamatan dan analisis data.
Pengamatan meliputi gejala luar yang muncul pada daun (Leaf Symptom Index=LSI) dan gejala dalam pada bonggol (Rhizome Discoloration Index=RDI). Pengamatan LSI dilakukan pada minggu ke-2 dan ke-4 setelah penanaman. Pengamatan RDI dilakukan pada minggu ke-4 setelah penanaman.
Evaluasi pengamatan Leaf Symptom Index (LSI) berdasarkan Mak et al.
(2004), terdiri atas 5 skor dengan kriteria sebagai berikut (Gambar 4): 1. Tidak ada warna kuning pada daun, tanaman sehat
2. Sedikit penguningan pada daun bagian bawah
3. Penguningan pada sebagian besar daun bagian bawah 4. Penguningan meluas pada sebagian besar daun 5. tanaman mati
9 Evaluasi pengamatan Root Symptom Index (RDI) berdasarkan Mak et al.
(2004), terdiri atas 8 skor dengan kriteria sebagai berikut:
1. Tidak ada diskolorasi pada jaringan empulur bonggol atau sekitarnya
2. Tidak ada diskolorasi pada jaringan empulur bonggol; ada diskolorisasi pada daerah pertemuan akar dengan bonggol.
3. Diskolorasi sebanyak 5% pada empulur bonggol. 4. Diskolorasi sebanyak 6-20% pada empulur bonggol. 5. Diskolorasi sebanyak 21-50% pada empulur bonggol 6. Diskolorasi lebih dari 50% pada empulur bonggol 7. Diskolorasi pada seluruh empulur bonggol
8. Tanaman mati
Gambar 5SkorRhizome Discoloration Index (RDI)
Setelah dicatat LSI dan RDI, kemudian dihitung Disease Severity Index
(DSI) dengan rumus:
DSI = Σ ( skor x jumlah tanaman pada skor tersebut) jumlah tanaman yang diperlakukan
DSI terdiri atas empat kategori yaitu sangat tahan, tahan, rentan dan sangat rentan. Berdasarkan nilai DSI,tanaman tersebut dapat ditentukan masuk kedalam salah satu dari 4 kategori tersebut (Tabel 2).
Tabel 2 Indeks kategori ketahanan
Nilai DSI untuk LSI Nilai DSI untuk RDI Status ekspresi penyakit
1 1 Sangat tahan
Antara 1.1 -2 Antara 1.1 - 3 Toleran
Antara 2.1 - 3 Antara 3.1- 5 Rentan
10
4 HASIL DAN PEMBAHASAN
Amplifikasi PCR dengan menggunakan primer degenerate
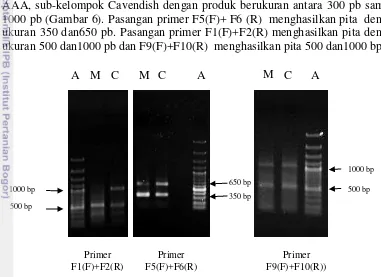
Primer degenerate telah didesain untuk mengamplifikasi daerah antara motif p-loop dan GLPL dari area Nucleotide Binding Site dari kelas gen ketahanan NBS. Dari tiga pasang primer degenerate, diperoleh 11 fragmen analog gen ketahanan yang berhasil diisolasi dari DNA pisang M. acuminata var.malaccensis dan Musa,
AAA, sub-kelompok Cavendish dengan produk berukuran antara 300 pb sampai 1000 pb (Gambar 6). Pasangan primer F5(F)+ F6 (R) menghasilkan pita dengan ukuran 350 dan650 pb. Pasangan primer F1(F)+F2(R) menghasilkan pita dengan ukuran 500 dan1000 pb dan F9(F)+F10(R) menghasilkan pita 500 dan1000 bp.
Gambar 6Hasil amplifikasi DNA dengan menggunakan primer degenerate.
Dari ketiga pasang primer, lima fragmen berasal dari M. acuminata var.
malaccensis dan enam fragmen berasal dari Musa, AAA, sub-kelompok Cavendish. Pada primer F1(F)+ F2(R), M. acuminata var. malaccensis hanya teramplifikasi satu fragmen sedangkan pada Musa, AAA, sub-kelompok Cavendish ada dua fragmen. Semua fragmen tersebut diisolasi dari gel dan dilakukan kloning ke dalam bakteri Eschericia coli.
Primer F1(F)+F2(R)
Primer F5(F)+F6(R)
Primer F9(F)+F10(R))
1000 bp
500 bp
1000 bp
500 bp 350 bp
650 bp
M C M C M C
Keterangan: A: Marker 100 bp (Fermentas), M:Musa acuminata var. malaccensis, C:Musa acuminatacv. Cavendish .
11
Analisis sekuen fragmen produk PCR dari DNA genom pisang
Dari 11 fragmen produk PCR yang telah disekuensing, semua mengandung insersi fragmen berukuran antara 323-1114 pb. Berdasarkan hasil analisis nukleotida dan deduksi asam amino dengan menggunakan BLASTp pada
Genbank, ditemukan 3 sekuen yang mengandung daerah NBS yaitu P5-8, P5-10 dan P5-11 dengan ukuran berturut-turut 770, 1114 dan 791 bp. Sekuen P5-8 dan P5-10 berasal dari M. acuminata var. malaccensis. Sedangkan, Sekuen P5-11 berasal dari Musa, AAA, sub-kelompok Cavendish. Sekuen P5-8 mempunyai kesamaan identitas 97% dengan disease resistance proteinRPS2 pada M. acuminatavar.malaccensis. Sedangkan sekuen P5-10 dan P5-11 mempunyai kesamaan identitas 99% dan 100% dengan disease resistance protein RPM1 pada
M. acuminata var. malaccensis (Tabel 3). Ketiga sekuen fragmen ini telah dideposit ke Genbank dengan nomor akses KP691062-KP691064.
Tabel 3 Sequence identity antara sekuen prediksi asam amino tanaman pisang
Musa acuminata var. malaccensis dan Musa, AAA, sub-kelompok Cavendish dengan protein ketahanan penyakit Musa yang telah dideposit pada GenBank.
Hasil penelitian Sun et al. (2010), dari 20 Resistance Gene Analog (RGA) yang berhasil diisolasi dari pisang Goldfinger (AAAB), prediksi asam aminonya berbagi homologi 28-54% dengan gen yang telah diketahui yaitu Fom2, I2C1,
I2C2 dan I2. Agustine dan Joseph (2014) mengisolasi fragmen 517 bp dari pisang AAB cv. Nendran menunjukkan homologi 97-99% dengan protein NBS-LRR varietas Musa. Sedangkan Sutanto et al. (2014), mendapatkan 17 fragmen yang berhasil diisolasi dan menunjukkan homologi 91,7-98,8% dengan protein ketahanan penyakit NBS-LRR pada M. acuminata var. malaccensis, grup AAA, AAB dan ABB.
Ketiga sekuen dilakukan BLAST pada Musa Genome ( http://banana-genome.cirad.fr), hasilnya menunjukkan ketiga sekuen memiliki coverage > 90% dan terdapat pada kromosom nomor 9 (Tabel 4 dan Gambar 7). Fragmen P5-8 memiliki kesamaan dengan disease resistance protein putatif RPS2, sedangkan P5-10 dan P5-11 memiliki kesamaan dengan disease resistance protein putatif
Fragmen No aksesi Aksesi Genbank Query coverage
E value
Identity Aksesi
P5-8 KP691062 Disease resistant protein
12
RPM1 dengan persentase coverage berturut-turut 92,76%, 96,68% dan 97,11%. Jarak antara lokus RPS1 dan RPM2 yaitu 0.156 Kbp
Pada penelitian Li et al. (2012), peningkatan ekspresi gen pada Cavendish cv. Nongke no.1 terjadi pada komplek protein RPM1/RIN4. Pada Arabidopsis thaliana, RPM1 terkait dengan ketahanan terhadap Pseudomonas syringae yang mengekspresikan avrRPM1 atau avrB (Boyes et al. 1998). AvrB dan AvrRPM1
menyebabkan hiperfosforilasi RPM1 yang saling berinteraksi dengan protein4 (RIN4) (Torres et al. 2006). Penghambatan ekspresi RIN4 oleh efektor yang belum diketahui dari F. oxysporum f.sp. cubense TR4 akan mengaktifkan jalur RPS2.
13
Analisis pensejajaran sekuen dan filogenetik prediksi asam amino dengan protein RGA dan gen-R tanaman lain.
Hasil analisis konsensus prediksi asam amino dari ketiga fragmen dengan protein NBS-LRRtanaman pisang serta gen-R tanaman lain yang telah dideposit di Genbank menunjukkan terdapat empat struktur konservatif NB-ARC yang merupakan ciri khas daerah NBS. Keempat struktur konservatif asam amino yaitu motif P-loop/kinase-1a (GGVGKTT), kinase-2 (LVLDDIW), RNBS-B/kinase-3a
NBS-LRR_Musa_acuminata_var._miGMGGVGKTTLAQQAYNHER--VKDYFHPKVWLCV.SDNFNVERLTKD--IVESITE-E---KCDLS--57
NBS-LRR_Musa_acuminata_AAA_GroGMGGVGKTTLAQQAYNHAR--VQDCFQLKVWVCV.SDNFNVERLTKE--IIESLTR-N---TCDLN--57
NBS-LRR_Musa_banksii GMGGVGKTTLAQQAYNHER--VKDYFHPKVWLCV.SDNFNVERLTKE--IIESLTR-N---KCDLN--57
NBS-LRR_Musa_acuminata_var._siGMGGVGKTTLAQQAYNHER--VKDYFHHKVWVCV.SDNFNVERLTKE--IIESLTR-N---KCDLN--57
NBS-LRR_Musa_acuminata_var._erGMGGVGKTTLAQQAYNHES--VKDYFQHEVWVCV.SYNFNVERLTKE--IIESITE-N---KCDLS--57
NBS-LRR_Musa_acuminata_var._micNLDTLQVV---VKKKLTSKRFLLVLDDVWNEDGL---KWERF--CASLRYGE---QGSKILVTTRSK 113
NBS-LRR_Musa_acuminata_AAA_Gro-NFDTLQVV---VKEKLTSKRFLPVLDDVWSEDSL---KWERF--CAPLKHGE---PGSKILVTTRSK 113
NBS-LRR_Musa_banksii - FDTLQVI---VKDMLTSKRFLLVLDDVWDEDGL---KWERF--CASLRYGE---QGSKILVTTRSK 113
NBS-LRR_Musa_acuminata_var._siaNFDTLQVV---VKEELTSKRFLLVLDDVWNEDSL---KWERF--CAPLRYGV---PGSKILVTTRSE 113
NBS-LRR_Musa_acuminata_var._errNLDTLQVV---LKKNLTSKRFLLVLDDVWNEDSL---KWERF--CAPLRYGE---PGSKILVTTRSK 113
14
Gambar 8. Analisis pensejajaran sekuen prediksi asam amino dengan beberapa protein Musa pada daerah NBS dan protein R yang terdeposit pada gen bank NCBI.
Panjang asam amino yang mencakup daerah p-loop sampai GLPL yaitu 200 asam amino. Semua RGA pisang dan protein R tanaman lain memiliki keempat daerah konservatif tersebut. Hasil ini menunjukkan konservasi evolusi gen ketahanan penyakit.
Berdasarkan penelitian Sutanto et al. (2014), perbedaan sekuen asam amino dengan fragmen P5-8, P5-10 dan P5-11 dijumpai pada daerah kinase-2 (B) terutama pada asam amino Lysine (K), Leucine (L) dan Isoleucine (I). Pada daerah Kinase 3a (C), terdapat perbedaan motif asam amino yaitu Cystein- Lysine- Leucine- Isoleucine- Leucine- Alanine- Serine- Arginine- Serine- Asparagine (CKLILASRSN) dan Serine-arginine-Valine- Valine- Threonine- Threonine- Arginine- Methionine- Gluthamine (SRVVTTRMQ). Sedangkan, pada daerah hidrofobik (D) perbedaan terletak pada asama amino Serine (S) dan Alanine (A) (Gambar 8). Perbedaan sekuen asam amino pada daerah konservatif ini memungkinkan terjadinya perbedaan respon ketahanan terhadap penyakit yang berbeda.
Fungsi dari p-loop/kinase-1a dan kinase-2 adalah untuk mengikat fosfat pada ATP, sedangkan kinase 3 berinteraksi dengan purin (Traut, 1994). Motif GLPL terlibat dalam pengikatan ADP. Motif p-loop juga diperlukan pada pengikatan nukleotida dan mutasi pada motif ini akan menyebabkan hilangnya fungsi pada beberapa protein NBS-LRR. Pada motif kinase-2, dua asam aspartat Keterangan: daerah domain terkonservasi ditandai dengan garis tebal diatas sekuen (A= P-loop atau kinase-1a, B=kinase-2, C=kinase-3a dan D= hidrophobic domain atau GLPL). Garis putus-putus menunjukkan kesamaan asam amino fragmen dengan protein RPM1 dan RPS2 serta hasil penelitian Sutanto et al (2014).
15 terakhir berinteraksi dengan fosfat ketiga dari ATP, dan berperan dalam koordinasi ion logam divalen yang diperlukan dalam reaksi transfer fosfat, contoh Mg2+ dari MgATP (McHale et al. 2006).
Hasil analisis filogenetik sekuen prediksi asam amino menunjukkan bahwa protein R dan protein NBS-LRR pisang terbagi kedalam dua kelompok yaitu TIR-NBS dan non-TIR-TIR-NBS. Diantara protein tersebut, protein RPP4, RPP5, RPS4, N
dan P2 termasuk kedalam kelompok TIR-NBS sedangkan yang lainnya masuk ke dalam kelompok non-TIR-NBS. Fragmen P5-8 tergabung satu kelompok dengan protein RPS2 dari M. acuminata var. malaccensis dan protein NBS-LRR dari M. balbisiana. Fragmen P5-10 dan P5-11 tergabung satu kelompok dengan protein
RPM1 dari M. acuminata var. malaccensis dan protein RX01 dari jagung. Fragmen yang diperoleh dari penelitian ini bersama protein pada pisang lain dan tanaman Monokotil seperti tebu, gandum dan jagung termasuk ke dalam kelas TIR-NBS, sedangkan tanaman Dikotil ada yang termasuk kedalam kelas non-TIR-NBS maupun kelas non-TIR-NBS (Gambar 9).
Gambar 9Pohon filogenetik sekuen prediksi asam amino dari RGA pisang dan beberapa protein Musa dan protein tanaman lain berdasarkan analisis pensejajaran menggunakan MUSCLE dan dibuat berdasarkan metode NJ.
Non-TIR-NBS
TIR-NBS
16 Tidak adanya daerah TIR pada protein R dapat diprediksi dengan adanya motif RNBS-A dekat dengan p-loop dan juga adanya residu tryptophan (W) di akhir motif kinase-2 (Meyers et al. 1999). Pada analisis filogenetik, secara evolusi hipotesis hilangnya daerah TIR dari NBS-LRR Monokotil kemungkinan terjadi selama penyebaran Monokotil dan Dikotil (Pan et al. 2000). Dari fungsi yang telah diketahui, pengelompokan non-TIR-NBS pada pisang kemungkinan mengkode produk gen ketahanan dengan spesifitas yang belum diketahui (Peraza-Echeverria et al. 2008). Meskipun protein pisang yang lain tidak berkorelasi dengan ketahanan penyakit pisang pada umumnya, peran potensialnya pada ketahanan penyakit dapat diuji dengan menggunakan teknologi era post-genomic
seperti RNA interference (RNAi). Teknologi ini dapat digunakan untuk men-silencing target terkait ketahanan terhadap penyakit (Waterhouse & Helliwell 2003).
Uji tantang terhadap Fusarium oxysporum f.sp. cubense TR4 pada M. acuminatavar.malaccensis dan Musa, AAA, sub-kelompok Cavendish.
Pada M. acuminata var. malaccensis, respon gejala penyakit berupa klorosis atau penguningan pada daun terjadi pada minggu ke-2 setelah inokulasi pada konsentrasi Foc TR4 106 konidia/ml. Daun yang paling tua mulai menguning dan beberapa hari kemudian seluruh daun merata mengalami klorosis. Sedangkan pada Musa, AAA, sub-kelompok Cavendishrespon gejala penyakit dimulai pada minggu pertama setelah inokulasi. Perbandingan hasil antara kontrol dan perlakuan inokulasi 106 konidia/ml pada M. acuminata var. malaccensis dan
Musa, AAA, sub-kelompok Cavendish setelah 4 minggu disajikan pada Gambar 10.
Pengamatan terhadap daun dan bonggol pisang tersebut menggunakan skoring berdasarkan Mak et al. (2004). Hasil pengamatan menunjukkan bahwa pisang M. acuminata var. malaccensis berada pada status toleran, sedangkan
Musa, AAA, sub-kelompok Cavendish berada pada status sangat rentan (Tabel 5). Hal ini menunjukkan bahwa M. acuminata var. malaccensis lebih tahan daripada
Musa, AAA, sub-kelompok Cavendish. Adapun kontrol M. acuminata var.
malaccensis yang menunjukkan status toleran dikarenakan adaptasi fisiologis dan adanya hama serangga pengebor bonggol, namun pengamatan pada daun menunjukkan status sangat tahan.
Berkaitan dengan identifikasi kandidat gen Nucleotide Binding site (NBS), uji tantang ini menunjukkan bahwa ada gen yang berperan dalam mengendalikan ketahananan terhadap penyakit layu fusarium. Tiga fragmen yang telah diidentifikasi mempunyai kesamaan dengan gen RPS2 dan RPM1, dimana M. acuminata var. malaccensis mempunyai kedua gen RPS2 dan RPM1 sedangkan
17
Gambar 10 Hasil uji tantang ketahanan terhadap F. oxysporumf.sp.cubenseTR4 pada Musa acuminata var. malaccensis dan Musa, AAA, sub-kelompok Cavendish setelah 4 minggu pengamatan.
Tanaman mempunyai sistem yang efisien untuk menahan penyebaran serangan patogen. Respon hipersensitif merupakan bentuk kematian sel yang berkaitan dengan ketahanan tanaman terhadap infeksi patogen dan terjadi pada saat interaksi dengan patogen kompatibel maupun inkompatibel. Ciri khas dari respon hipersensitif yaitu adanya kematian jaringan yang cepat dan terlokalisasi pada titik infeksi untuk membatasi penyebaran patogen.
Selama respon hipersensitif terjadi, oksigen bereaksi sangat cepat untuk memicu reaksi oksidatif dan menambah pergerakan ion K+ dan H+ melalui membran sel. Hal ini menyebabkan kerusakan membran dan hilangnya sekat selular. Tanaman memperkuat dinding selnya dengan menghasilkan senyawa fenolik, senyawa kinase, asam salisilat, sintesis senyawa toksik yang disebut fitoaleksin dan mengakumulasi protein yang mengandung senyawa antimikrobia seperti chitinase. Secara genetik, respon hipersensitif terjadi jika tanaman mengenali patogen spesifik. Reaksi ini melibatkan gen ketahanan pada tanaman (Agrios 2005; Iakimova et al. 2005).
M. acuminate var.
malaccensis
kontrol daun
Musa, AAA, sub-kelompokCavendish
kontrolbonggol perlakuan
106konidia/ml
18 Tabel 5. Status ketahanan/kerentanan Musa acuminata var. malaccensis dan
Musa, AAA, sub-kelompok Cavendish 4 minggu setelah inokulasi konsentrasi 106 konidia/ml.
Ulangan
M.acuminata var. malaccensis Musa, AAA, sub-kelompok
Cavendish
Kontrol Perlakuan Foc 106 Kontrol Perlakuan Foc 106
LSI RSI LSI RSI LSI RSI LSI RSI
1 1 1 1 2 1 1 3 3
2 1 1 3 3 1 1 3 3
3 1 2 2 2 2 1 4 7
4 1 4 2 2 2 1 5 8
5 1 4 1 1 4 1 5 8
DSI 1 2.4 1.8 2 2 1 4 5.8
Status Toleran Toleran Sangat Tahan Sangat
Rentan
Pada pisang yang terinfeksi F. oxysporum f.sp. cubense TR4, terjadi kenaikan kadar O2 dan H2O2 pada ujung akar, aktivasi Superokside Dismutase
(SOD) dan katalase (CAT), akumulasi Malonedialdehida (MDA) yang rendah dapat dikenali pada pisang yang tahan terhadap F. oxysporum f.sp. cubense TR4. Indikator ini dapat digunakan untuk seleksi plasma nutfah baru yang tahan terhadap F. oxysporum f.sp. cubense TR4 pada pemuliaan pisang (Li et al. 2011).
5 SIMPULAN
Dari pisang M. acuminata var.malaccensisdiperoleh dua fragmen DNA yaitu P5-8 dan P5-10. Sedangkan dari Musa, AAA, sub-kelompok Cavendish diperoleh satu fragmen DNA yaitu P5-11. Ketiga fragmen ini mengandung 4 motif terkonservasi dari Nucleotide Binding site yaitu p-loop, kinase-2, kinase-3a dan GLPL. Fragmen P5-8 memiliki kesamaan dengan protein RPS2 dari M. acuminata var. malaccensis, sedangkan P5-10 dan P5-11 memiliki kesamaan dengan protein RPM1 dari M. acuminata var. malaccensis. Ketiga sekuen ini telah dideposit ke dalam Genbank dengan nomor aksesi KP691062-KP691064. Secara filogenetik, semua fragmen tergabung dalam kelompok non-TIR-NBS-LRR. M. acuminata var. malaccensis lebih tahan terhadap F. oxysporum f.sp. cubense TR4 daripada Musa, AAA, sub-kelompok Cavendish.
UCAPAN TERIMAKASIH
Penelitian ini didukung oleh beasiswa Karyasiswa LIPI No. 867/H/2011 dan Program Kompetitif LIPI Sub-Program Domestikasi dan Pemanfaatan Terukur dengan tema „Pemuliaan pisang untuk ketahanan terhadap penyakit layu
19 penelitian Biologi, LIPI. Penulis mengucapkan terimakasih kepada Ibu Dr Yuyu Suryasari Poerba sebagai koordinator penelitian dan kepala Laboratorium Genetika Tumbuhan serta semua pihak yang telah membantu dalam penelitian ini.
DAFTAR PUSTAKA
[BPS] Badan Pusat Statistik. 2014. Produksi Buah-buahan di Indonesia 1995-2013. [terhubung berkala].www.bps.go.id.[20 Oktober 2014].
[FAO]. Food and Agricultural Organization. 2013. Top Production of Bananas 2012.
Agrios GN. 2005. Plant Pathology 5th edition. Elsevier Academic Press. USA. 221-223
Arvanitoyannis IS, Mavromatis AG, Avgeli GG, Sakellariou M. 2008. Banana: cultivar, biotechnological approaches and genetic transformation. Int J Food Sci Technol 43: 1871-1879
Augustine DA, Joseph AY. 2014. Isolation and Characterization of NBS_LRR Resistance gene in banana (Musa AAB cv.Nendran). Int J Adv Biotech Res. 5(3): 353-358
Bendahmaney A, Farnham G, Moffett P, Baulcombe DC. 2002. Constitutive gain of function mutants in a nucleotide binding site–leucine rich repeat protein encoded at the Rx locus of potato. The Plant J. 32:195–204.
Boyes DC, Nam J, Dangl JL. 1998. The Arabidopsis thaliana RPM1 disease resistance gene product is a pheriperal palsma membran protein that is degraded coincident with the hypersensitive response. Proc Natl Acad Sci. 95:15849-15854
Delaporta SL, Wood J, Hiks JB.1983. A plant DNA minipreparation, Version II.
Plant Mol Biol Rep. 4: 19-21
Deng Z, Huang S, Ling P, Chen C, Yu C, Weber A, Moore GA, Gmitter Jr FG. 2000. Cloning and characterization of NBS-LRR class resistance gene candidate sequence in citrus. Theor Appl Genet. 101(5-6): 814-822
Fluhr R. 2001.Sentinels of disease.plant resistance genes. Plant Physiol. 127:1367-1374.
Glynn NC, Comstock JC, Sood SG, Dang PM, Chaparro JX. 2008. Isolation of nucleotide binding site-leucine rich repeat and kinase resistance gene analogues from sugarcane (Saccharum spp.) Pest Manag Sci. 64:48–56. Hakkinen M. 2013. Reappraisal of sectional taxonomy in Musa
(Musaceae).Taxon. 62 (4):809-813
Horry JP, Ortiz R, Arnaud E, Crouch JH, Ferris RSB, Jones DR, Mateo N, Picq C, Vuylsteke D. 1997. Banana and Plantain. Di dalam Fuccillo D, Sears L, Stapleton P. In Biodiversity in Trust, Conservation and Use of Plant Genetic Resources in CGIAR Centres. Cambridge University Press. pp. 67- 81 Iakimova ET, Michalezuk L, Woltering EJ. 2005. Hypersensitive Cell Death in
20 Jumjunidang, Nasir N, Riska, Handayani H. 2005. Teknik pengujian in-vitro ketahanan pisang terhadap penyakit layu fusarium menggunakan filtrat toksin dari kultur Fusarium oxysporum f.sp.cubense. J Hort. 15:135-139. Kayat F, Javed MA, Ho YW, Othman RY. 2004. Identification of molecular
markers for disease resistance genes to Fusarium oxysporum f. sp. cubense
in Musa acuminata ssp. malaccensis for marker assisted selection (MAS).
Proceeding of The 4th Annual Seminar for National Science Fellowship (NSF). Penang 20-21 Dec 2004. Malaysia.40-44
Li CY, Deng GM, Yang J, Viljoen A, Jin Y, Kuang RB, Zuo CW, Yang QS, Sheng O, Wei YR, Hu CH, Dong T, Yi GJ. 2012. Transcriptome profilling of resistant and susceptible Cavendish banana roots following inoculation with Fusarium oxysporum f.sp. cubense tropical race 4. BMC genomic.13:374
Li WM, Qian CM, Mo YW, Hu YL, Xie JH. 2011. Tolerance of banana for fusarium wilt is associated with early H2O2 accumulation in the roots. Afr J Biotech. 10(55):11378-11387.
Mak C, Mohammed AA, Liew KW, Ho YW. 2004. Early screening technique for Fusarium wilt resistance in banana micropropagated plants. Di dalam: Mohan JS, Swennen R, editor. Banana improvement: cellular, molecular biology, and induced mutantions. Italy: FAO Science Publisher, Inc.
Maniatis T, Fritsch EF,Sambrook J. 1982. Molecular cloning. A Laboratory Manual. Cold Spring Harbor Press. Cold Spring Harbor. New York.
Martin GB, Bogdanove AJ, Sessa G. 2003. Understanding the functions of plant disease resistance proteins. Annu Rev Plant Biol 54:23-61.
McHale L, Tan X, Koehl P, Michelmore RW. 2006. Plant NBS-LRR protein: adaptable guards. Genom Biol. 7:212
Meyers B, Dickerman A, Michelmore R, Sivaramakhrisnan S, Sobral B, Young N. 1999. Plant disease resistance genes encode members of an ancient and diverse protein family within the nucleotide-binding superfamily. Plant J.
20:317-332.
Michelmore RW, Meyers BC.1998. Clusters of resistance genes in plants evolve by divergent selection and a birth-and-death process. Genome Res 8:1113-1130.
Miller RNG, Bertoli D, Baurens F, Santos C, Alves P, Martins N, Togawa R, Souza M, PappasG. 2008. Analysis of non-TIR NBS-LRR resistance gene analogs in Musa acuminata Colla: Isolation, RFLP marker development, and physical mapping. BMC Plant Biol8:15
Nasution RE. 1991. A taxonomic study of the Musa Acuminata Colla with its intraspesific taxa in Indonesia. Memoirs of the Tokyo University of Agriculture. 32: 1-22
Nogueira ACW,.Soares-Cavalcanti NM, Morais DAL, Belarmino LC, Barbosa-Silva A, Benko-Iseppon AM. 2007. Abundance and diversity of resistance genes in the sugarcane transcriptome revealed by in silico
analysis.Genet. Mol. Res. 6 (4): 866-889
21 Parker JE, Coleman MJ, Szabo V, Frost LN, Schmidt R, van der Biezen EA, Moores T, Dean C, Daniels MJ, Jones JDG. 1997. The Arabidopsis downy mildew resistance gene RPP5 shares similarity to the toll and interleukin-1 receptors with N and L6. Plant Cell 9:879-894.
Peraza-Echeverria S, Dale JL, Harding RM, Smith M, Collet C. 2008. Characterization of resistance gene candidates of the nucleotide binding site (NBS) type from banana and correlation of a transcriptional polymorphism with resistance to FOC race 4. Mol Breed. 22: 565-579. DOI 10.1007/s11032-008-9199-x
Rommens CM, Kishore GM. 2000. Exploiting the full potential of disease-resistance genes for agricultural use. Curr Opin Biotechnol11:120-125. Simmonds NW, Shepherds K. 1955. The Taxonomy and Origin of The Cultivated
Bananas. London. Linnean Soc. 55: 302-312.
Simmonds NW. 1962. The Evolution of the Banana. London. Longman.
Singh HP, Uma S. 1996. Genetic diversity of banana in India. In: Proceeding of the Conference on Challenges for Banana Production and Utilization in 21st Century, Trichy, Sept 24-25
Sun D, Hu Y, Zhang L, Mo Y, Xie J. 2010. Cloning and Analysis of Fusarium Wilt Resistance Gene Analogs in Gold Finger Banana. Mol Path. 1:1-7. Sutanto A, Sukma D, Hermanto C, Sudarsono. 2014. Isolation and
characterization of Resistance Gene Analogue (RGA) from Fusarium resistant banana cultivar. Emir J Food Agric. 26(6): 508-518
Syamkumar S, Lowarence B, Sasikumar B. 2003. Isolation and amplification of DNA from rhizomes of turmeric and ginger. Plant Mol Bio Rep. 212:171a-171e
Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. 2011. MEGA5: Molecular evolutionary genetic analysis using maximum likelihood, evolutionary distance and maximum parsimony methods. Mol Biol Evol. 28(10):2731-2739
Torres MA, Jones JDG, Dangl JL. 2006. Reactive oxygen species signaling in response to pathogens. Plant Physiol. 141: 373-378
Traut TW. 1994. The functions and consensus motifs of nine types of peptide segments that form different types of nucleotide-binding sites. Eur J Biochem.Vol.222: 9-19
Waterhouse PM, Helliwell CA. 2003. Exploring plant genomes by RNA-induced gene silencing. Nat Rev. 4: 29-38
Widono S, Sumardiyono C, Hadisuwarno B. 2003. Pengimbasan ketahanan pisang terhadap penyakit layu fusarium dengan Burkholderia cepacea.
22
RIWAYAT HIDUP
Penulis dilahirkan di Jakarta pada tanggal 26 Maret 1981 dari pasangan Syahidin dan Keminah. Penulis merupakan anak pertama dari dua bersaudara. Pada tahun 2008, penulis menikah dengan Gembong Agronomianto, S.Si dan telah dikaruniai seorang putri bernama Alya Zahra Afifatulkhonsa dan seorang putra bernama Bariq Ahmad Zein.
Pendidikan dasar sampai pendidikan menengah atas diselesaikan di kota Bekasi. Sedangkan, pendidikan S1 diselesaikan dari Fakultas Biologi, Universitas Gadjah Mada, Yogyakarta pada bulan Februari tahun 2005.