ANALISIS KERAGAMAN GENETIK TEMULAWAK (Curcuma
xanthorrhiza Roxb.) DENGAN MENGGUNAKAN PENANDA
AMPLIFIED FRAGMENT LENGTH POLYMORPHISM (AFLP)
DINI DAMAYANTI
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI
Dengan ini saya menyatakan bahwa tesis Analisis Keragaman Genetik
Temulawak (Curcuma xanthorrhiza Roxb.) Menggunakan Penanda Amplified
Fragment Length Polymorphism (AFLP) adalah karya bersama saya dengan komisi pembimbing dan belum diajukan dalam bentuk apapun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau kutipan dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan tercantum dalam Daftar Pustaka di bagian akhir tesis ini.
Bogor, November 2012
ABSTRACT
DINI DAMAYANTI. Genetic Variation Analysis of Curcuma xanthorrhiza Roxb. by Using Amplified Fragment Length Polymorphism (AFLP) Marker. Under direction of SUHARSONO and TEUKU TAJUDDIN.
Indonesia is known by its diversities, especially in herb-medicinal plants. Curcuma xanthorrhiza Roxb. is one of the potential medicinal plant belonging to Zingiberaceae family. This study was to determine genetic diversity of 32 accession Curcuma xanthorrhiza Roxb. Total DNA was extracted from leaf using Sodium Dodesyl Sulphate (SDS) modification methode. Amplified fragment length polymorphism (AFLP) was carried out according to the protocol described in AFLPTM plant mapping kit (PE Applied Biosystem) and the final polymerase chain reaction (PCR) products were separated using The Agilent 2100 Bioanalyzer. The number of fragment produced by 12 pairs primer combination of AFLP ranged from 42 to 60 with and average 0f 52. Data obtained was analyzed by the NTSys program. From the AFLP amplification on 32 DNA samples, it was proven that the accession of Curcuma xanthorrhiza Roxb. had a high degree of diversity. Based on analysis of AFLP and unweighted pair group with arithme average (UPGMA) it was shown that the accession of Curcuma xanthorrhiza Roxb. could be grouped into two cluster at relative ecludian distance of 0.10 (10%). Cluster I for accession from Palembang, Pacitan and Ciamis 2. Cluster II for accession from Makale, Pontianak, Kulonprogo, Mataram, Boyolali, Salatiga, Sumberejo, Bali, P. Seram, Sentolo, Purworejo, Samas Bantul, Ciamis1, Blora, Semarang, Poso, Kalsel, Tagari, Merapi Farm, Salakaria, NTB, Menoreh, Karang Anyar, Mangunan, Medan, Toraja, dan Solok.
RINGKASAN
DINI DAMAYANTI. Keragaman Genetik Temulawak (Curcuma xanthorrhiza Roxb.) Dengan Menggunakan Penanda Amplified Fragment Length Polymorphism (AFLP). Dibimbing oleh SUHARSONO dan TEUKU TAJUDDIN.
Temulawak (Curcuma xanthorrhiza Roxb.) merupakan tanaman obat yang dikenal sebagai Java tumeric dan secara tradisional digunakan di negara-negara Asia Tenggara untuk makanan dan obat.
Tanaman Temulawak di Indonesia tersebar di Jawa, Sumatera, Kalimantan, Bali, Sulawesi, Maluku, dan Nusa Tenggara. Temulawak yang berasal dari beberapa daerah di Indonesia tersebut, secara morfologi sulit dibedakan. Metode biologi molekuler dapat digunakan untuk analisis keragaman, karena masing-masing individu memiliki urutan DNA yang berbeda (polimorfisme). Informasi urutan DNA dapat digunakan untuk mempelajari perbedaan genetik dan hubungan kekerabatan antara individu dan jenis organisme. Informasi keragaman genetik temulawak ini diperlukan untuk mendukung kegiatan konservasi temulawak.
Ada beberapa metode penanda DNA yang dapat digunakan untuk analisis keragaman genetik, diantaranya adalah AFLP (Amplified Fragment Length Polymorphism). AFLP dapat mendeteksi variasi dan keragaman genetika pada mahluk hidup pada tingkat antar individu, spesies, dan populasi berdasarkan kesamaan atau perbedaan pola pita.
Penelitian dilaksanakan pada bulan Juni sampai Februari 2012 di Laboratorium Teknologi Gen, Balai Pengkajian Bioteknologi BPPT, Serpong. Sampel Temulawak yang digunakan dalam penelitian ini berasal dari beberapa daerah di Indonesia. Profil pita DNA diterjemahkan kedalam data biner dengan ketentuan nilai 0 untuk tidak ada pita dan 1 untuk adanya pita DNA pada satu posisi yang sama dari aksesi yang dibandingkan. Kesamaan genetik dibuat dalam bentuk matrik dengan similarity for qualitative data (SIMQUAL), kemudian klastering dilakukan dengan sub program SAHN dengan metode Unweigth Pair Group Method with Arithmatic (UPGMA) program NTSYS-pc 2.02.
Pada penelitian ini PCR dengan 12 kombinasi primer menghasilkan rata-rata 52 fragmen DNA tiap kombinasi primer yang berukuran 18bp hingga 350bp. Total jumlah fragmen yang dihasilkan dalam amplifikasi tergantung pada jumlah dan komposisi nukleotida yang digunakan. Jumlah total fragmen teramplifikasi juga tergantung pada kompleksitas genom. Jumlah fragmen yang bersifat polimorfis tergantung pada variasi genetik antar sampel yang dianalisis. Polimorfisme yang dihasilkan tidak hanya dipengaruhi oleh kombinasi pasangan basa yang digunakan dan kompleksitas genom, tetapi juga dipengaruhi oleh penggunaan enzim restriksi.
Makale, Pontianak, Kulonprogo, Mataram, Boyolali, Salatiga, Sumberejo, Bali, P. Seram, Sentolo, Purworejo, Samas Bantul, Ciamis1, Blora, Semarang, Poso, Kalsel, Tagari, Merapi Farm, Salakaria, NTB, Menoreh, Karang Anyar, Mangunan, Medan, Toraja, dan Solok. Kelompok II yang beranggotan 27 aksesi, terbagi menjadi dua sub kelompok pada koefisien 0.16.
Pengelompokkan 32 aksesi temulawak berdasarkan karakter morfologi berkisar dari 0.17-1.00 atau 17-100%. Pengelompokan pada koefisien kemiripan 0.17 (17%) membentuk dua kelompok. Kelompok I terdiri dari aksesi temulawak asal Solok, Palembang, Purworejo, Sentolo, Medan, Salakaria, Pontianak, Makale, Ciamis 1, Karang Anyar, Boyolali, P. Seram. Kelompok II terdiri dari temulawak asal aksesi Ciamis 2, Kalisanta, Semarang, Blora, Pacitan, Merapi Farm, Sumberejo, Tagari, Salatiga, Samas Bantul, Bali, Kalsel, Argomulyo, NTB, Poso, Toraja, Mataram, Mangunan, Menoreh, dan Kulonprogo.
Hasil penelitian menunjukkan bahwa klaster yang terbentuk dengan menggunakan data AFLP hasilnya berbeda dengan klaster yang terbentuk dengan menggunakan data morfologi. Penelitian menunjukkan bahwa kemiripan fenotip tidak menunjukkan kemiripan pada tingkat DNA.
© Hak cipta milik IPB, tahun 2012
Hak cipta dilindungi Undang-undang
1. Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumber
a. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik atau tinjauan suatu masalah
b. Pengutipan tersebut tidak merugikan kepentingan yang wajar dari IPB
2. Dilarang mengumumkan dan memperbanyak sebagian atau seluruh karya tulis dalam bentuk apapun tanpa izin IPB dan Balai Besar Penelitian dan Pengembangan Bioteknologi dan Sumber Daya Genetika Pertanian (BB-Biogen)
ANALISIS KERAGAMAN GENETIK TEMULAWAK (Curcuma
xanthorrhiza Roxb.) DENGAN MENGGUNAKAN PENANDA
AMPLIFIED FRAGMENT LENGTH POLYMORPHISM (AFLP)
DINI DAMAYANTI
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada
Program Studi Bioteknologi
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
Judul Tesis : Analisis Keragaman Genetik Temulawak (Curcuma xanthorrhiza Roxb.) dengan Menggunakan Penanda Amplified Fragment Lenght Polymorphism (AFLP)
Nama : Dini Damayanti
NIM : P051090111
Disetujui
Komisi Pembimbing
Prof. Dr. Ir. Suharsono, DEA. Dr. Ir. Teuku Tajuddin, M.Sc.
Ketua Anggota
Diketahui,
Ketua Program Studi/Mayor Dekan Sekolah Pasca Sarjana
Bioteknologi
Prof. Dr. Ir. Suharsono, DEA. Dr. Ir. Dahrul Syah, M.Sc, Agr.
PRAKATA
Segala puji bagi Allah yang Maha Kuasa atas rahmah dan ridho-Nya
sehingga karya ilmiah ini dapat terselesaikan. Karya ilmiah ini mengulas tentang
temulawak dengan judul Analisis keragaman genetik temulawak (Curcuma
xanthorrhiza Roxb.) dengan menggunakan penanda Amplified Fragment Length Polymorphism (AFLP). Penelitian ini bagian dari Program Insentif Kemenristek dengan judul Identifikasi temulawak (Curcuma xanthorrhiza Roxb.) unggulan lokal sebagai tanaman obat asli Indonesia dengan metode sidik jari DNA.
Terimakasih penulis ucapkan kepada Prof. Dr. Ir. Suharsono, DEA, dan Dr.
Ir. Teuku Tajuddin, M.Sc, selaku komisi pembimbing yang telah mengarahkan dan
membimbing penulis baik dalam proses penelitian maupun penulisan karya ilmiah
ini. Penulis juga mengucapkan terima kasih kepada Prof Sudarsono (Departemen
Agronomi dan Hortikultura IPB) dan Dr. Marlina Ardiyani (LIPI) atas waktu yang
telah diberikan untuk diskusi. Ucapan terimakasih juga penulis ucapkan kepada
seluruh peneliti dan staf Laboratorium Teknologi Gen Badan Pengkajian dan
Penerapan Teknologi Serpong (Pak Imam, mas Devit, mba Ana, mba Leha, Ibu
Rahma) atas bantuan dan ilmu yang telah diberikan.
Penulis juga mengucapkan terimakasih untuk keluarga tercinta, Ayah, Ibu,
Kakak dan Adikku tercinta atas kasih sayang, cinta, dukungan, dan doa yang tak
hentinya dicurahkan kepada penulis. Terimakasih kepada keponakan-keponakanku
tersayang atas keceriaannya. Terimakasih kepada teman-teman BTK 2009 atas segala
dukungan dan doanya. Terima kasih kepada sahabat-sahabat tercinta, Biologi UI
2000 yang telah memberikan semangat dan kecerian kepada penulis. Semoga karya
ilmiah ini bermanfaat.
Bogor, November 2012
RIWAYAT HIDUP
Penulis dilahirkan di Tangerang tanggal 24 Februari 1982 dari bapak Edi
Suhandi dan ibu Nani Sumarni. Penulis merupakan anak ketiga dari empat
bersaudara. Penulis menyelesaikan jenjang pendidikan menengah umum tahun 2000
di SMA Negeri 70 Jakarta dan pada tahun yang sama lulus Ujian Masuk Perguruan
Tinggi (UMPTN) di Universitas Indonesia. Penulis diterima di Jurusan Biologi,
Fakultas Matematika dan Ilmu Pengetahuan Alam dan lulus pada tahun 2006. Sejak
tahun 2006, penulis mengajar di Universitas Islam Negeri Syarif Hidayatullah.
Penulis mengampu mata kuliah Praktikum sistematika Tumbuhan. Pada tahun 2009
penulis mengikuti seleksi penerimaan mahasiswa Pascasarjana Institut Pertanian
DAFTAR ISI
Halaman
DAFTAR TABEL ... xii
DAFTAR GAMBAR ... xiii
DAFTAR LAMPIRAN ... xiv
PENDAHULUAN Latar Belakang ... 1
Tujuan Penelitian ... 3
TINJAUAN PUSTAKA Temulawak (Curcuma xanthorrhiza Roxb.) ... 4
Keragaman Genetik Temulawak ... 8
Keragaman Genetik dan Penanda Molekuler ... 9
Amplified Fragment Length Polymorphism (AFLP) ... 10
BAHAN DAN METODE Waktu dan Tempat Penelitian ... 14
Bahan ... 14
Metode ... 15
Isolasi DNA ... 15
Analisis AFLP ... 16
Pengamatan Morfologi ... 18
HASIL DAN PEMBAHASAN Karakter Morfologi ... 19
Analisis Profil Pita AFLP ... 21
Analisis Klaster Aksesi Temulawak Berdasarkan AFLP ... 26
Analisis Klaster Aksesi Temulawak Berdasarkan Karakter Morfologi ... 28
SIMPULAN ... 31
DAFTAR TABEL
Halaman
1 Komposisi rimpang temulawak ... 7
2 Kadar minyak atsiri rimpang temulawak ... 7
3 Komponen minyak temulawak ... 8
4 Aksesi temulawak yang digunakan ... 14
5 Kombinasi primer yang digunakan pada penelitian ... 15
6 Variasi warna pita ungu ibu tulang daun dan rimpang dari 32 aksesi temulawak ... 19
7 Jumlah pita hasil amplifikasi DNA dari 32 aksesi temulawak pada setiap kombinasi primer AFLP ... 23
8 Kelompok aksesi yang terbentuk berdasarkan data AFLP pada koefisien 0.10 ... 26
DAFTAR GAMBAR
Halaman
1 Tanaman dan bunga temulawak ... 5
2 Rimpang temulawak ... 6
3 Diagram alur AFLP ... 11
4 Variasi warna pita ungu tulang daun temulawak ... 20
5 Variasi warna rimpang temulawak ... 20
6 Profil DNA dari 32 aksesi temulawak hasil amplifikasi menggunakan pasangan primer EAAC-MCTC ... 24
7 Profil DNA dari 32 aksesi temulawak hasil amplifikasi menggunakan pasangan primer EAAC-MCAA ... 24
8 Kemiripan genetik antar 32 aksesi temulawak berdasarkan penanda AFLP menggunakan 12 kombinasi primer ... 26
9 Kemiripan genetik antar 32 aksesi temulawak berdasarkan karakter morfologi ... 29
DAFTAR LAMPIRAN
Halaman 1 Alur penelitian ... 152 Komposisi ligasi dan digesti ... 17
3 Komposisi PCR untuk preamplifikasi ... 17
4 Komposisi PCR untuk amplifikasi ... 17
5 Aksesi temulawak yang digunakan pada penelitian ... 28
6 Profil DNA dari 32 aksesi temulawak hasil amplifikasi menggunakan pasang primer EAAC-MCTA ……….. 24
PENDAHULUAN
Latar Belakang
Temulawak (Curcuma xanthorrhiza Roxb.) merupakan tanaman obat yang
dikenal sebagai Java tumeric dan secara tradisional digunakan di negara-negara Asia
Tenggara untuk makanan dan obat. Temulawak memiliki aroma khusus dan sedikit
rasa pahit. Rimpang temulawak ini digunakan sebagai bahan baku utama obat-obatan
karena mengandung minyak atsiri, resin, kurkumin, lemak, kamfer, serat kasar dan
kalsium klorida. Temulawak merupakan salah satu jenis tanaman obat dari famili
Zingiberaceae yang potensial untuk dikembangkan. Disamping memiliki prospek
pasar regional maupun internasional, tanaman ini juga menempati urutan pertama
sebagai tanaman yang dibutuhkan dalam jumlah besar sebagai bahan baku industri
obat tradisional, fitofarmaka, bahan makanan, minuman penyegar dan bahan
kosmetik.
Penelitian temulawak telah banyak dilakukan, baik di Indonesia maupun di
negara lain, mulai dari kandungan senyawa aktif hingga khasiatnya yang telah
terbukti secara empiris dan medis. Manfaat temulawak diantaranya adalah sebagai
sistem imunitas atau pertahanan tubuh (Hargono 1996), sebagai komponen pengatur
haid (Nurendah et al. 1996), untuk mengatasi keputihan, dan sebagai bahan kosmetika (Dzulkarnain & Wahjoedi 1996).
Tanaman Temulawak di Indonesia tersebar di Jawa, Sumatera, Kalimantan,
Bali, Sulawesi, Maluku, dan Nusa Tenggara. Tanaman dengan asal daerah yang
berbeda cenderung memiliki persentase kandungan metabolit (misal kurkumin,
demetoksikurkumin dan xanthorrhizol) yang berbeda pula (Tajuddin et al. 2008). Temulawak yang berasal dari beberapa daerah di Indonesia tersebut, secara morfologi
sulit dibedakan. Menurut Istafid (2006), temulawak sulit dibedakan secara fenotipik,
sehingga karakterisasi dilakukan secara molekuler. Metode biologi molekuler dapat
digunakan untuk analisis keragaman, karena masing-masing individu memiliki urutan
mempelajari perbedaan genetik dan hubungan kekerabatan antara individu dan jenis
organisme (Weising et al. 2005).
Informasi keragaman genetik temulawak ini juga diperlukan untuk mendukung
kegiatan konservasi temulawak Indonesia (Poerba & Yuzammi 2008), untuk
melindungi plasma nutfah Indonesia khususnya tanaman obat temulawak. Menurut
Badan Pengawasan Obat dan Makanan Republik Indonesia, temulawak telah
ditentukan sebagai salah satu dari Sembilan tanaman unggulan Indonesia (Sembiring
et al. 2006).
Ada beberapa metode penanda DNA yang dapat digunakan untuk analisis
keragaman genetik, diantaranya adalah AFLP (Amplified Fragment Length
Polymorphism). Teknik AFLP merupakan salah satu teknik untuk membuat sidik jari DNA genom. Menurut Mueller dan Wolfenbarger (1999), AFLP dapat mendeteksi
variasi dan keragaman genetika pada mahluk hidup pada tingkat antar individu,
spesies, dan populasi berdasarkan kesamaan atau perbedaan pola pita. Prinsip dasar
teknik AFLP adalah mengamplifikasi secara selektif fragmen hasil pemotongan
dengan dua enzim restriksi. Polimorfisme dapat dideteksi dari perbedaan letak situs
pemotongan dua enzim restriksi (EcoRI dan MseI) dan komposisi basa pada primer selektif (Invitrogen 2003).
Teknik AFLP sangat efisien untuk identifikasi polimorfisme DNA karena
banyak fragmen restriksi yang dapat terdeteksi (Vos et al. 1995). Hasil AFLP berupa fragmen yang terseleksi, kurang lebih 50-100 fragmen per reaksi. Fragmen tersebut
dihasilkan dari pemotongan enzim restriksi yang diikuti ligasi adaptor dan amplifikasi
dari daerah yang diapit oleh adaptor.
Keunggulan teknik AFLP adalah dapat mendeteksi polimorfisme pada tanaman
tanpa memerlukan informasi urutan basa genom. Selain itu, teknik AFLP memiliki
tingkat reproducible yang tinggi berdasarkan amplifikasi selektif fragmen hasil pemotongan genom, yaitu bila diulang cenderung menghasilkan hasil yang sama
(Mueller & Wolfenbarger 1999). Teknik AFLP mampu menganalisis genom secara
menyeluruh sehingga menghasilkan informasi yang memadai untuk menganalisis
basa-basa yang diapit oleh primer selektif yang tersebar luas pada seluruh bagian
genom. Hasil pita polimorfis yang didapatkan relatif banyak. Salah satu kelemahan
dari teknik AFLP adalah pita yang didapatkan bukan menunjukkan alel atau lokus
tertentu.
Beberapa penelitian mengenai keragaman genetik temulawak telah dilakukan.
Penelitian Santiana (2010) menunjukkan bahwa berdasarkan sekuen daerah matK dan
intergenic spacer (IGS) trnS-trnfM temulawak yang berasal dari beberapa daerah di Indonesia mempunyai keragaman genetik. Penelitian mengenai eksplorasi genetik
temulawak dengan metode AFLP juga telah dilakukan oleh Tajuddin et al. (2008). Hasil penelitian ini dapat memperkaya informasi keragaman genetik temulawak di
Indonesia.
Tujuan Penelitian
Tujuan dari penelitian ini adalah untuk menganalisis keragaman genetik 32
aksesi temulawak yang berasal dari beberapa daerah di Indonesia dengan
TINJAUAN PUSTAKA
Temulawak (Curcuma xanthorrhiza Roxb.)
Curcuma xanthorrhiza atau temulawak berasal dari bahasa Yunani, xanthos yang berarti kuning dan rhizoa yang berarti umbi akar. Temulawak memiliki sebutan
atau nama lain dari beberapa daerah, antara lain koneng gede (Sunda), temo lobak
(Madura), temulawak (Jawa), dan temulawas (Malaysia). Tanaman temulawak
tersebar di Asia Selatan dan Asia Tenggara, hingga ke Cina. Di Indonesia, tanaman
temulawak ditemukan di Jawa, Ambon dan Bali.
Klasifikasi tanaman temulawak sebagai berikut:
Kingdom : Plantae
Division : Spermatophyta
Subdivision : Angiospermae
Class : Monocotyledonae
Family : Zingiberaceae
Subfamily : Zingiberoideae
Genus : Curcuma
Species : Curcuma xanthorrhiza Roxb.
Secara alami, temulawak tumbuh dengan baik di lahan-lahan yang teduh dan
terlindung dari sinar matahari. Di habitat alaminya, rumpun tanaman ini tumbuh
subur di bawah naungan pohon bambu dan jati. Meskipun demikian, temulawak juga
dapat tumbuh di tempat yang terik, seperti di tanah tegalan. Tanaman ini memiliki
daya adaptasi yang tinggi pada berbagai cuaca di daerah beriklim tropis (Afifah &
Lentera 2003).
Temulawak merupakan herba tahunan yang tumbuh tegak dengan tinggi lebih
kurang 2 m, berwarna hijau atau coklat gelap. Tiap batang mempunyai 2 sampai
dengan 9 helai daun, berbentuk bundar memanjang sampai bangun lanset. Daun
berwarna hijau atau coklat keunguan terang sampai gelap. Daun termasuk tipe daun
sempurna, yang tersusun dari pelepah daun, tangkai daun dan helai daun. Panjang
Pada bagian midrib daun terdapat warna ungu kehitaman selebar 10 mm, tetapi sering
tidak mencapai pangkal daun.
Gambar 1 Tanaman temulawak (a) dan bunga temulawak (b)
Temulawak memiliki tipe perbungaan lateral, tangkai ramping, berbulu,
diameter tangkai berkisar 10- 37 cm. Sisik berbentuk garis, berbulu halus, panjang
sisik berkisar 4-12 cm dan lebar 2-3 cm. Bentuk bulir bulat memanjang, panjang bulir
berkisar 9-23 cm dan lebar 4-6 cm, berdaun pelindung banyak. Panjang daun
pelindung adalah melebihi atau sebanding dengan mahkota bunga, berbentuk bundar
telur sungsang sampai bangun jorong, berwarna merah, ungu atau putih dengan
sebagian dari ujungnya berwarna ungu. Bagian bawah daun pelindung berwarna hijau
muda atau keputihan, panjang 3-8 cm, dan lebar 1.5-3.5 cm. Kelopak bunga berwarna
putih berbulu dan panjang kelopak bunga 8-13 mm. Mahkota bunga berbentuk tabung
dengan panjang keseluruhan 4.5 cm. Tabung berwarna putih atau kekuningan dan
panjang 2-2.5 cm. Helaian bunga berbentuk bundar telur atau bundar memanjang,
berwarna putih dengan ujung yang berwarna merah dadu atau merah. Panjang helaian
bunga 1.25-2 cm dengan lebar 1 cm. Benang sari berwarna kuning muda, panjang
16 mm, dan lebar 10-15 mm. Panjang tangkai sari 3-4.5 mm dan lebar 2.5-4.5 mm.
Kepala sari berwarna putih, panjang 6 mm, tangkai putik panjang 3-7 mm.
Gambar 2 Rimpang temulawak
Akar rimpang temulawak terbentuk dengan sempurna dan bercabang kuat,
berwarna hijau gelap. Rimpang temulawak terdiri atas rimpang induk (empu) dan
rimpang anakan (cabang). Rimpang induknya berbentuk bulat seperti telur dan
berwarna kuning tua atau coklat kemerahan. Bagian dalamnya berwarna jingga
kecoklatan. Rimpang kedua yang lebih kecil keluar dari rimpang induk. Arah
pertumbuhannya ke samping, berwarna lebih muda dengan bentuk yang
bermacam-macam, jumlahnya sekitar 3-7 buah. Jika dibiarkan tumbuh lebih dari satu tahun,
akan tumbuh banyak rimpang lagi. Rimpang ini aromanya tajam dan rasanya pahit
agak pedas.
Umumnya tanaman temulawak tidak menghasilkan buah atau biji. Hal ini
diduga karena temulawak merupakan tanaman triploid dengan jumlah kromosom
yang besar 3n=63 sehingga menimbulkan banyak gangguan dalam proses meiosis.
Akibatnya terlihat pada ukuran dan bentuk serbuk sari yang sangat beragam.
Sebagian diantaranya adalah serbuk sari abortif yang berdinding sangat tipis dan
sangat mudah pecah dengan kesuburan sangat rendah (0-2%). Hal tersebut
menyebabkan temulawak berkembang biak secara vegetatif melalui pembentukan
tunas yang tumbuh dari mata tunas rimpang (Islam 2004).
Secara tradisional, temulawak telah banyak digunakan masyarakat antara lain
sebagai obat untuk mengatasi batu empedu, batu ginjal, demam, kolesterol tinggi,
temulawak yang dimanfatkan adalah rimpangnya. Rimpang temulawak mengandung
zat warna kurkumin, minyak atsiri, pati, protein, lemak. Suwiah (1991) menguraikan
komposisi rimpang kering temulawak dengan kadar air 10% yang ditunjukkan pada
Tabel 1. Kadar minyak atsiri rimpang temulawak dari berbagai sumber pustaka
ditunjukkan pada Tabel 2. Komponen minyak temulawak menurut Liang et al. (1985), Anang (1992), serta Dickes dan Nicholas (1976) dapat dilihat pada tabel 3.
Minyak atsiri yang terkandung dalam rimpang temulawak berpotensi sebagai
senyawa antioksidan, anti hepatotoksik, meningkatkan sekresi empedu,
antihipertensi, melarutkan kolesterol, mengeluarkan air susu (laktagoga), tonik bagi
ibu pasca-melahirkan, peluruh haid, anti bakteri, pewarna makanan dan kain, serta
bahan kosmetik. Xantorizol yang terkandung pada rimpang temulawak juga diketahui
telah digunakan dalam produk makanan dan pasta gigi untuk mencegah penyakit
pada gigi (Hwang 2008), sebagai antikanker dan antiinflamasi, memiliki aktivitas
sebagai anti fungi (Rukayadi 2006), dan sebagai anti depresi (Sidik 2008).
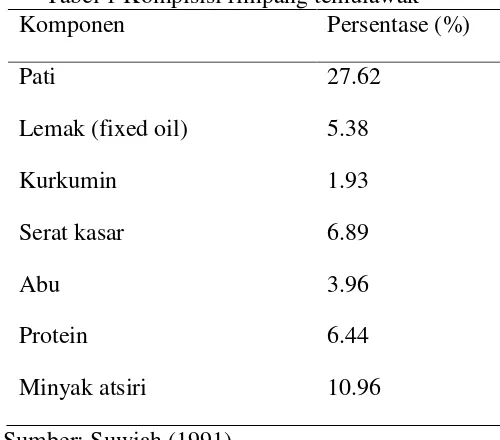
Tabel 1 Kompisisi rimpang temulawak
Komponen Persentase (%)
Pati
Lemak (fixed oil)
Kurkumin
Serat kasar
Abu
Protein
Minyak atsiri
27.62
5.38
1.93
6.89
3.96
6.44
10.96
Sumber: Suwiah (1991)
Tabel 2 Kadar minyak atsiri rimpang temulawak
Herman 1985
Nurdjanah et al. 1994
Liang et al. 1985
Sirait et al. (1985)
Rimpang temulawak berumur:
8 bulan
10 bulan
12 bulan
15 bulan
6-10
7.3-9.5
7-11
6.6
5.2
5.3
5.11
Tabel 3 Komponen minyak temulawak
- α-lumulena (25,2%)
- kamfan (21,9%)
- zerumbon (21,2%)
- α-kurkumen (0,8%)
- lumulen epolesi (4,6%)
-kamfor (4,2%) -α-pinena (3,4%) -borneol dan
α-terpineol (0,6%)
-eukaliptol (1,8%) -β-kariofilena (1,6%) -limonene (1,5%) -linaloal (0,9%) -3-karena (0,3%) -Lumulena dioksida
-β-pinena (0,6%)
1. trisiklin
2. α-pinena
3. kamfena
4. β-pinena
5. sabrinena
6. mirsena
7. felandrena
8. limonene
9. 1,8-sineol
10. δ-terpinena
11. β-simen
12. terpionlen
13. δ-elemena
14. kamfor
15. α-bergamolena
16. β-elemena
17. kariofilena
18. allo-aromadendren
19. trans-β-farnesena
20. berneol
21. gerwakrena D
22. zingibirena
23. β-bisabolen
24. β-kurkumena
*seskuiterpen
- β-kurkumena
- α-kurkumena
- 1-sikloisoprenmirsean
-zingibirena -xantorizol
-turunan bisabolen -epolisid-bisakuron -bisakuron A -bisakuron B -bisakuron C
*ketonseskuiterpena
- turmeron
- α-turmeron
- α-atlanton (0,3%)
- germakron
*monoterpena
- sineol
- d-borneol
- d-α-feladrena
25. β-kadinena
26. β-seekuifelandrena
27. ar-kurkumen
28. isofuranogermasen
29. turmeron
30. turmerol
31. ar-turmeron
32. xantorizol
Keragaman Genetik Temulawak
Tanaman temulawak di Indonesia tersebar di Jawa, Sumatera, Kalimantan,
Bali, Sulawesi, Maluku dan Nusa Tenggara. Keragaman temulawak antar daerah
terlihat dari kandungan metabolit yang dihasilkan. Tanaman dengan asal daerah yang
berbeda cenderung memiliki persentase kandungan metabolit (kurkumin,
morfologi, keragaman terlihat pada rimpang temulawak. Karakter yang dapat diamati
meliput warna daging rimpang, berat bobot kering dan bobot basah rimpang, serta
bentuk rimpang induk.
Penelitian Rahayu (2010) terhadap 20 aksesi temulawak dari beberapa daerah
di Indonesia menghasilkan jumlah fragmen DNA yang berbeda untuk setiap daerah
asal temulawak. Rata-rata polimorfisme yang dihasilkan sebesar 95.1%. Besarnya
nilai polimorfisme menunjukkan tingginya keragaman genetik pada suatu jenis.
Keragaman Genetik dan Penanda Molekuler
Menurut Soerjanegara dan Djamhuri (1979), dalam satu populasi terdapat
beberapa keragaman yaitu keragaman geografis, keragaman lokal, dan keragaman
dalam pohon serta keragaman antar pohon. Keragaman tersebut disebabkan oleh dua
faktor yaitu faktor lingkungan dan faktor genetik. Keragaman lingkungan biasanya
disebabkan oleh keadaan tempat tumbuh, sifat tanah, atau jarak tanam. Keragaman
genetik disebabkan oleh perubahan pada struktur genetik pada suatu populasi.
Keragaman genetik merupakan perbedaan sekuen nukleotida dari suatu
organism dengan organism lainnya. Keragaman genetik terjadi karena adanya mutasi,
genetic drift, dan genetic flow (Halliburton 2004). Keragaman genetik bertambah ketika turunan suatu individu menerima kombinasi gen yang unik dari induk melalui
rekombinasi gen ketika terjadi reproduksi seksual.
Keragaman genetik dapat dilihat dengan menggunakan penanda molekular.
Deteksi keragaman genetik dapat berdasarkan adanya polimorfisme protein atau
DNA. Polimorfisme adalah perbedaan sekuen DNA pada suatu kromosom atau
individu. Polimorfisme DNA memiliki sekuen unik yang dapat digunakan untuk
mempelajari keragaman genetik dan hubungan kekerabatan antar organisme (Weising
et al. 1995).
Ciri penanda molekuler yang baik adalah memiliki tingkat polimorfisme yang
tinggi, penanda alel jelas, dan marka DNA terdistribusi dalam seluruh genom, dan
populasi adalah random amplified polymorphic DNA (RAPD), restriction fragment length polymorphism (RFLP), dan amplified fragment length polymorphism (AFLP) (Edward & Mogg 2001).
AmplifiedFragmentLengthPolymorphism (AFLP)
AFLP merupakan suatu teknik untuk membuat fingerprint DNA genom. AFLP merupakan salah satu sistem DNA fingerprinting yang bersifat multilokus dan dapat menghasilkan kapasitas fingerprinting yang besar (Vos et al. 1995). AFLP merupakan penanda ideal untuk mendeteksi adanya keragaman antar individu,
populasi dan jenis (Muller & Wolfenbarger 1999).
Prinsip dasar teknik AFLP adalah mengamplifikasi secara selektif fragmen
hasil pemotongan dengan dua enzim restriksi. Polimorfisme dapat dideteksi dari
perbedaan letak situs pemotongan dengan enzim restriksi (EcoRI dan MseI) dan komposisi basa pada primer selektif (Invitrogen 2003).
Prosedur AFLP terdiri dari beberapa tahap (Gambar 3) yang dimulai dengan
pemotongan DNA genom oleh sepasang enzim restriksi. Enzim restriksi yang
digunakan terdiri dari enzim dengan situs pengenalan jarang (rare cutter) dan situs pengenalan banyak (frequent cutter). Enzim rare cutter adalah enzim EcoRI dengan 6
basa pengenalan (G↓AATTC), sedangkan enzim frequent cutter adalah enzim MseI
dengan 4 basa pengenalan (T↓TAA). Tujuan penggunaan enzim rare cutter adalah
untuk membatasi jumlah fragmen yang akan diamplifikasi karena pemotongan
dengan enzim rare cutter menghasilkan jumlah fragmen yang lebih sedikit daripada
frequent cutter dan fragmen dari pemotongan dengan rare cutter berukuran lebih besar. Kemudian, enzim frequent cutter bertujuan memotong genom dengan ukuran lebih kecil sehingga mudah diamplifikasi dan berada pada kisaran ukuran yang
optimal untuk dijalankan pada elektroforesis gel poliakrilamid. Selain itu, menurut
Vos et al. 1995, organisme eukariot memiliki komposisi basa adenin dan timin yang lebih tinggi daripada basa guanin dan sitosin, sehingga untuk menganalisis genom
basa adenin dan timin yang lebih banyak agar polimorfisme yang dihasilkan lebih
rinci.
5’ GAATTC TTAA 3’ 3’ CTTAAG AATT 5’
AATTC T G AAT
AATTCN NTAA AATTCN NTAA
AATTCA GTTA TTAAAGT CAAT
AATTCAAC TTGTTA TTAAGTTG AACAAT
PCR
+ EcoRI,
MseI
+ adaptor EcoRI Adaptor MseI
Amplifikasi preselektif dengan primer EcoRI + A primer MseI + A
Gambar 3 Diagram alur AFLP
Hasil pemotongan dengan enzim restriksi tersebut kemudian diligasi dengan
adaptor yang sesuai hasil pemotongan. Adaptor terdiri dari sekuen inti adaptor dan
sekuen spesifik enzim. Sekuen spesifik enzim akan menggabungkan adaptor dengan
fragmen restriksi sehingga dihasilkan template atau cetakan bagi primer pada proses pre-amplifikasi dengan PCR (Vuylsteke et al. 2007). Reaksi preamplifikasi
menggunakan primer selektif dengan tambahan satu basa pada ujung 3’, sehingga
fragmen yang diamplifikasi hanya fragmen yang memiliki pasangan basa tersebut.
Reaksi selanjutnya adalah amplifikasi selektif dengan prinsip PCR dan menggunakan
primer selektif dengan tambahan tiga basa pada ujung 3’ sehingga fragmen yang
diamplifikasi hanya fragmen yang memiliki pasangan basa-basa tersebut (Invitrogen
2003).
Pada dasarnya proses amplifikasi DNA menggunakan mesin PCR mengikuti
pola sintesis DNA di dalam sel. Proses sintesis DNA diawali dengan pengudaran utas
ganda menjadi utas tunggal yang disebut denaturasi dan dilanjutkan dengan sintesis
utas baru menggunakan utas tunggal sebagai cetakan. Proses sintesis mempunyai arah
5’-3’, berarti polinukleotida baru ditambahkan pada karbon ketiga (C3) yang
mengandung OH pada nukleotida sebelumnya melalui ikatan 5-3 fosfodiester.
Amplifikasi DNA secara invitro menggunakan mesin PCR juga membutuhkan enzim
polymerase DNA, primer, basa nukleotida (dGTP, dCTP, dATP, dan dTTP), MgCl2,
dan buffer sebagai kofaktor enzim serta penambahan H2O bila diperlukan untuk
memperoleh volume total yang diinginkan.
Amplifikasi selektif menggunakan teknik touchdown PCR. Prinsip dasar
teknik touchdown PCR adalah menaikkan suhu annealing ± 3oC dari suhu melting primer pada awal siklus. Suhu annealing kemudian diturunkan 1oC setiap siklus,
kemudian siklus PCR berjalan tanpa penurunan suhu annealing jika sudah mencapai
suhu normal untuk primer yang digunakan. Fungsi touchdown PCR adalah
primer, dan sekuen target yang akan diamplifikasi tidak diketahui secara pasti
(Sambrook & Russel 2001).
Menurut Muller & Wolfenbarger (1999), AFLP memiliki beberapa kelebihan
antara lain efisien dalam hal waktu, replikasi dan resolusi lebih tinggi bila
dibandingkan dengan penanda lain seperti RAPD, mikrosatelit, dan RFLP. Selain itu,
dengan AFLP jumlah karakter yang diperoleh lebih banyak karena jumlah fragmen
yang dihasilkan lebih banyak, amplifikasi DNA dapat bersifat spesifik dan stabil.
AFLP seringkali digunakan untuk berbagai analisis molekuler seperti sistematika,
genetika populasi, DNA fingerprinting, dan Quantitatif Trait Loci (QTL) (Muller &
Wolfenbarger 1999).
Keeratinijakal et al. (2010) telah menggunakan AFLP untuk mendeteksi perbedaan genetik pada 97 aksesi tanaman Curcuma comosa Roxb. Antar aksesi Curcuma comosa Roxb. tersebut sulit dibedakan secara morfologi, sehingga
digunakan AFLP dengan 9 kombinasi primer untuk membedakan beberapa Curcuma
comosa Roxb. secara genetik. Pada tanaman tomat, marka molekuler yang terpaut dengan gen Cf-9 yaitu gen yang menentukan ketahanan terhadap patogen
BAHAN DAN METODE
Waktu dan Tempat Penelitian
Penelitian dilaksanakan pada bulan Juni 2011 hingga Februari 2012 di
Laboratorium Teknologi Gen, Balai Pengkajian dan Penerapan Teknologii (BPPT),
Serpong.
Bahan
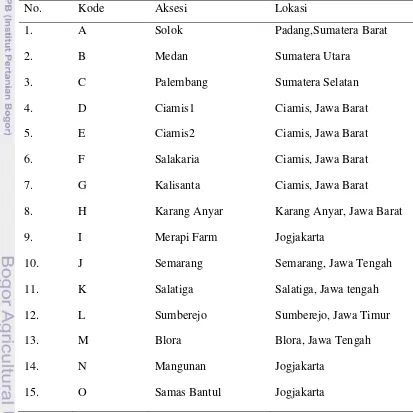
Penelitian ini menggunakan bahan tanaman temulawak yang terdiri dari 32
[image:31.612.103.516.291.704.2]aksesi (Tabel 4).
Tabel 4 Aksesi temulawak yang digunakan
No. Kode Aksesi Lokasi
1. 2. 3. 4. 5. 6. 7. 8. 9. 10. 11. 12. 13. 14. 15. A B C D E F G H I J K L M N O Solok Medan Palembang Ciamis1 Ciamis2 Salakaria Kalisanta Karang Anyar Merapi Farm Semarang Salatiga Sumberejo Blora Mangunan Samas Bantul Padang,Sumatera Barat Sumatera Utara Sumatera Selatan
Ciamis, Jawa Barat
Ciamis, Jawa Barat
Ciamis, Jawa Barat
Ciamis, Jawa Barat
Karang Anyar, Jawa Barat
Jogjakarta
Semarang, Jawa Tengah
Salatiga, Jawa tengah
Sumberejo, Jawa Timur
Blora, Jawa Tengah
Jogjakarta
16. 17. 18. 19. 20. 21. 22. 23. 24. 25. 26. 27. 28. 29. 30. 31. 32 P Q R S T U V W X Y Z AA AB AC AD AE AF Tagari Boyolali Purworejo Kulonprogo Menoreh Sentolo Pacitan
Kebun Raya Bali
Kalsel Argomulyo Pontianak NTB Poso Toraja Mataram Makale P. Seram Jawa Tengah Jawa Tengah Jawa Tengah Jawa Tengah Jawa Tengah Jogjakarta Jawa Timur Bali Kalimantan Selatan Kalimantan Timur Kalimantan Barat
Nusa Tenggara Barat
Sulawesi Tengah
Sulawesi Selatan
Nusa Tenggara Barat
Sulawesi Selatan
Maluku
Primer yang digunakan untuk AFLP disajikan pada Tabel 5.
Tabel 5 Kombinasi primer yang digunakan pada penelitian
Kombinasi primer Kode
EcoRI+AAC – MseI+CTG EAAC-MCTG
EcoRI+AAC – MseI+CAA EAAC-MCAA
EcoRI+AAC – MseI+CTA EAAC-MCTA
EcoRI+AAC – MseI+CAG EAAC-MCAG
EcoRI+AAC – MseI+CAC EAAC-MCAC
EcoRI+ACA – MseI+CTG EACA-MCTG
EcoRI+ACA – MseI+CAA EACA-MCAA
EcoRI+ACA – MseI+CTA EACA-MCTA
EcoRI+ACA – MseI+CTC EACA-MCTC
EcoRI+ACA – MseI+CAG EACA-MCAG
EcoRI+ACA – MseI+CAC EACA-MCAC
Metode
Analisis keragaman pada penelitian ini terbagi ke dalam dua bagian yaitu
analisis keragaman berdasarkan AFLP dan analisis keragaman berdasarkan karakter
morfologi. Analisis AFLP dilakukan melalui beberapa tahap yaitu isolasi DNA dan
analisis AFLP. Tahapan analisis AFLP disajikan pada Lampiran 1.
Isolasi DNA
Isolasi DNA genom temulawak dilakukan dengan metode Sodium Dodesil
Sulfat (SDS) (Angeles et al. 2005) yang dimodifikasi. Untuk itu, sampel daun temulawak dicuci dengan ddH2O. Setelah itu, 0.2 g sampel daun dipotong hingga
kecil-kecil. Potongan kecil daun tersebut kemudian ditambahi dengan 0.05 g Poly
Vinyl Poly Pirolidon (PVPP). Campuran tersebut kemudian digerus hingga halus dan
dimasukkan ke dalam tabung. Selanjutnya, 2 ml buffer ekstrak (10% CTAB, 0.5 M EDTA, 1 M Tris-HCl, 5 M NaCl, akuades steril) dan 1% merkaptoetanol
ditambahkan ke dalam suspensi jaringan daun yang sudah digerus, kemudian
campuran tersebut diinkubasi pada suhu 65 oC selama 15 menit. Campuran tersebut
ditambahkan lagi ke dalam tabung hingga volume keseluruhan menjadi 6 ml. Setelah
itu, campuran tersebut ditambahi dengan SDS 20% sebanyak 6% volume (360 µ l),
kemudian di vortex. Selanjutnya, sampel dalam tabung falcon 15 ml diinkubasi pada
suhu 65 oC selama 1 jam. Sampel kemudian disentrifugasi pada kecepatan 4000 rpm
[Beckman J2-HS, KUBOTA & Tommy] (selama 15 menit. Larutan yang jernih
(supernatan) diambil (± 2 ml), kemudian dipindahkan ke tabung falcon 15 ml yang baru. Larutan 5 M Kalium Asetat sebanyak 1/3 volume (670 µl) ditambahkan pada
supernatan. Campuran larutan tersebut dikocok dan kemudian diinkubasi selama 15
menit dalam freezer pada suhu -20 oC. Langkah berikutnya, tabung falcon 15 ml yang
mengandung sampel disentrifugasi selama 15 menit pada kecepatan 8000 rpm
(Beckman J2-HS, KUBOTA & Tommy), lalu ditambahkan isopropanol sebanyak 2/3
volume sampel. Setelah itu, tabung diinkubasi pada suhu -20oC selama 1 malam.
Kemudian, sampel dalam tabung disentrifugasi kembali pada kecepatan 8000 rpm
[Beckman J2-HS, KUBOTA & Tommy] selama 15 menit. Cairan dibuang,
endapannya ditambahi dengan 500 µl TE buffer (10 mM Tris-HCl, 1 mM EDTA).
Kontaminan RNA dalam sampel dihilangkan dengan penambahan enzim
RNAse sebanyak 1/100 volume sampel dan diinkubasi pada suhu 37 oC selama 1 jam.
Sampel tersebut kemudian ditambahi dengan larutan kloroform/isoamilalkohol (24:1)
sebanyak volume sampel. Larutan dihomogenkan dengan dibolak-balik secara
perlahan, lalu disentrifugasi pada kecepatan 11 000 rpm (Beckman J2-HS, KUBOTA
& Tommy) selama 10 menit. Cairan yang berada di lapisan atas kemudian
dipindahkan ke tabung Eppendorf 1,5 ml yang baru. Sampel kemudian ditambahi dengan isopropanol sebanyak volume sampel dan selanjutnya diinkubasi selama 1
jam pada suhu -20 oC. Setelah itu, sampel disentrifugasi kembali pada kecepatan 11
000 rpm [Beckman J2-HS, KUBOTA & Tommy] selama 10 menit. Endapan
kemudian dikeringkan, lalu ditambahi dengan 25 µl TE buffer (10 mM Tris-HCl, 1 mM EDTA). Hasil isolasi DNA dianalisis kualitasnya dengan elektroforesis di gel
agarosa 0.8% dan disimpan pada suhu -20 oC dan digunakan sebagai bahan untuk
tahap selanjutnya.
Genom DNA sebanyak 50 ng dipotong dengan sepasang enzim restriksi
(EcoR I dan Mse I). Adaptor EcoR I, Mse I dan 1 unit T4 DNA ligase kemudian
ditambahkan ke dalam reaksi pada suhu 37 oC selama 2 jam dan dilanjutkan dengan
suhu 65 oC selama 20 menit. Komposisi pemotongan dan ligasi DNA disajikan pada
Lampiran 2. Hasil ligasi diencerkan 1:10, kemudian dipakai sebagai cetakan untuk
Pre-Amplifikasi. Reaksi Pre-Amplifikasi terdiri dari 3 µl DNA hasil ligasi, 2.5 µl Taq
buffer, 1 U Taq DNA pol, 25 mM MgCl2, 25 mM dNTPs, 10 pmol primer Mse-C, 10 pmol primer Eco-A dan H2O steril. Komposisi PCR untuk pre-amplifikasi disajikan
pada Lampiran 3. Amplifikasi dilakukan menggunakan thermocycle dengan tahapan
program sebagai berikut: 72 oC selama 2 menit, 94 oC selama 30 detik, 56 oC selama
30 detik, 72 oC selama 2 menit, kembali dari tahap 94 oC selama 29 kali, dan 60 oC
selama 10 menit.
Setelah diamplifikasi, hasil PCR diencerkan 1:10, kemudian diamplikasi
selektif menggunakan 12 kombinasi primer (Tabel 5). Campuran PCR terdiri dari 5
µl hasil preamplifikasi, 2,5 µl Taq buffer (10 x), 0.04 U Ampli Taq gold, 25mM MgCl2, primer selektif masing-masing 10 pmol Mse I dan EcoR I, dan H2O steril
sampai volume 25 µl. Campuran kemudian dicampur secara perlahan dan
disentrifugasi singkat untuk menurunkan seluruh cairan di dalam tabung. Amplifikasi
dilakukan dengan menggunakan PCR. Program yang digunakan adalah sebagai
berikut: denaturasi pra-PCR 94 oC selama 2 menit, diikuti dengan denaturasi pada 94 o
C selama 30 detik, annealing 65 oC selama 30 detik dan extending 72 oC selama 2
menit. Tahap berikutnya adalah suhu annealing diturunkan 0,7 oC selama 12 siklus dan 23 siklus berikutnya PCR dilakukan dengan suhu 94 oC selama 30 detik, suhu 56
o
C selama 30 detik, dan suhu 72 oC selama 60 detik dan diakhiri dengan suhu 4 oC.
Pemisahan pita-pita hasil amplifikasi dilakukan dengan menggunakan alat Agilent
2100 Bionalyzer. Komposisi PCR untuk amplifikasi selektif disajikan pada Lampiran
4.
Profil pita DNA diterjemahkan ke dalam data biner dengan ketentuan nilai 0
untuk tidak ada pita dan 1 untuk adanya pita DNA pada satu posisi yang sama dari
similarity for qualitative data (SIMQUAL), kemudian klastering dilakukan dengan sub program SAHN dengan metode Unweigth Pair Group Method with Arithmatic (UPGMA) program NTSYS-pc 2.02 (Rohlf 1998), menggunakan koefisien
kemiripan DICE.
Pengamatan Karakter Morfologi
Karakter morfologi yang diamati pada penelitian ini adalah sebagai berikut:
1. Pita ungu ibu tulang daun
Pengamatan pita ungu pada ibu tulang daun dilakukan pada daun ke-3 dari
atas yang terdiri dari yang sangat jelas jika > 75% ibu tulang daun tertutup pita ungu,
jelas jika antara 50% hingga 75% ibu tulang daun tertutup pita ungu, kurang jelas
jika 25% hingga 50% ibu tulang daun tertutup pita ungu dan tidak jelas jika tidak
terdapat pita ungu pada ibu tulang daun.
2. Warna daging rimpang.
Pengamatan warna daging rimpang dilakukan saat panen dan dilakukan
HASIL DAN PEMBAHASAN
Karakter MorfologiHasil pengamatan karakter kualitatif berupa morfologi bentuk daun, bentuk
pangkal daun, bentuk ujung daun dan pertulangan daun pada 32 aksesi tanaman
temulawak yang ditanam pada kebun koleksi BPPT Serpong, menunjukkan tidak
terdapat perbedaan. Hasil pengamatan menunjukkan bahwa bentuk daun adalah
oblong lanceolate, bentuk pangkal daun adalah meruncing, bentuk ujung daun adalah
meruncing, dan pertulangan daun adalah menyirip. Perbedaan terletak pada warna
pita ungu tulang daun. Oleh sebab itu pengamatan karakter vegetatif terhadap 32
aksesi temulawak dilakukan terhadap warna pita ungu ibu tulang daun dan warna
rimpang. Hasil pengamatan warna pita ungu tulang daun dan warna rimpang dari 32
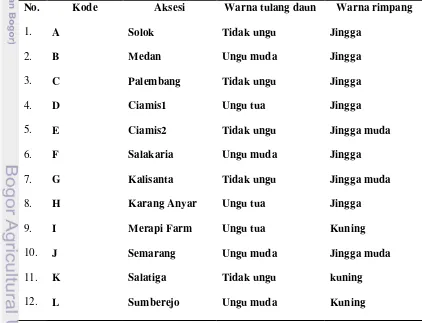
aksesi temulawak disajikan pada Tabel 6.
Tabel 6 Variasi warna pita ungu ibu tulang daun dan rimpang dari 32 aksesi temulawak
No. Kode Aksesi Warna tulang daun Warna rimpang
[image:37.612.99.521.382.705.2]13. 14. 15. 16. 17. 18. 19. 20. 21. 22. 23. 24. 25. 26. 27. 28. 29. 30. 31. 32 M N O P Q R S T U V W X Y Z AA AB AC AD AE AF Blora Mangunan Samas Bantul Tagari Boyolali Purworejo Kulonprogo Menoreh Sentolo Pacitan Bali Kalsel Argomulyo Pontianak NTB Poso Toraja Mataram Makale P. Seram Ungu muda Tidak ungu Tidak ungu Ungu muda Ungu tua Tidak ungu Ungu muda Tidak ungu Tidak ungu Ungu muda Tidak ungu Tidak ungu Tidak ungu Tidak ungu Tidak ungu Tidak ungu Tidak ungu Tidak ungu Ungu muda Ungu tua Jingga muda Kuning keputihan Kuning Kuning Jingga Jingga Kuning keputihan Kuning keputihan Jingga Jingga muda Kuning Kuning Kuning Jingga Kuning Kuning Kuning Kuning Jingga Kuning
Pengamatan terhadap karakter morfologi memperlihatkan bahwa dari 32
aksesi tanaman temulawak terdapat tiga variasi warna ungu pita ungu ibu tulang
daun, yaitu sangat jelas, jelas dan kurang jelas (Gambar 4). Pengamatan terhadap
empat variasi warna rimpang, yaitu jingga tua, jingga, kuning dan kuning keputihan
[image:39.612.241.392.141.348.2](Gambar 5).
Gambar 4 Variasi warna pita ungu ibu tulang daun temulawak, (a) sangat jelas, (b) jelas, dan (c) kurang jelas
Gambar 5 Variasi warna rimpang, (a) jingga tua (b) jingga (c) kuning (d) kuning
keputihan
Warna ungu pita ibu tulang daun dan warna rimpang merupakan variasi yang
sering ditemukan dalam temulawak. Perbedaan lingkungan tanam diduga
menimbulkan keragaman alami temulawak, walaupun tanaman ini membiak secara
vegetatif. Petunjuk adanya keragaman semacam itu pertama kali dikemukakan oleh
Prana (1985) yang menyatakan bahwa rasa dan aroma temulawak yang diambil dari
populasi “liar” tidak selalu sama dengan yang bersumber dari populasi budidaya.
d c
a b
Analisis Profil Pita AFLP
DNA genom dari 32 aksesi temulawak yang berkualitas tinggi sangat
dibutuhkan untuk analisis AFLP (Vos et al. 1995), karena DNA yang utuh dan murni
dapat memudahkan enzim endonuklease restriksi (RE) bekerja memotong DNA
sesuai dengan sisi pengenalannya (Nathans & Smith 1975). Hal ini sangat penting
karena salah satu kriteria analisis menggunakan penanda AFLP ialah persamaan dan
perbedaan situs pengenalan enzim RE yang dapat menghasilkan ukuran fragmen
DNA yang berbeda antara 50-100 pita DNA (Vos et al. 1995). Teknik AFLP dapat mendeteksi polimorfisme pada aksesi temulawak dengan menganalisis seluruh
genom. Menurut Omoto dan Lurquin (2004), polimorfisme pada AFLP dideteksi
dengan adanya perbedaan ukuran fragmen DNA. Polimorfisme yang dihasilkan
menunjukkan adanya perbedaan letak penanda AFLP (urutan basa pengenalan enzim
restriksi EcoRI dan MseI dan primer selektif) sehingga dapat diperoleh informasi perbedaan genetik pada setiap sampel. Perbedaan ukuran fragmen DNA
menghasilkan suatu pola pita DNA tertentu.
Teknik AFLP diawali dengan memotong genom temulawak dengan enzim
EcoRI dan MseI. Enzim EcoRI memiliki pengenalan sekuen 5’-G↓AATTC-3’ dan memotong antara basa Guanin (G) dan Adenin (A). Enzim MseI memiliki pengenalan
sekuen 5’-T↓TAA-3’ dan memotong antara timin (T) dan timin (T). Penggunaan
enzim EcoRI dan MseI bertujuan memperoleh polimorfisme yang lebih rinci, karena
kedua enzim tersebut lebih banyak tersusun atas basa adenin dan timin dibanding
guanin dan sitosin (Vos et al 1995).
Fragmen hasil pemotongan kemudian diligasi dengan adaptor. Adaptor
merupakan DNA untai ganda yang memiliki panjang sekitar 20 bp. Terdapat dua
jenis adaptor yaitu adaptor untuk ujung pemotongan EcoRI dengan kelebihan basa
AATT pada ujung 5’ dan ujung pemotongan MseI dengan kelebihan basa TA pada
ujung 5’. Proses ligasi dilakukan dengan bantuan enzim T4 DNA ligase yaitu membentuk ikatan fosfodiester antara ujung 5’ (ujung fosfat) dan ujung 3’ (ujung
OH) pada untai DNA (Struhl 1993). Adaptor berfungsi menyamakan dua ujung
amplifikasi selanjutnya. Hasil positif ligasi dapat dilihat pada akhir proses AFLP
karena urutan basa pada adaptor berkomplemen dengan urutan basa pada primer
sehingga bila proses ligasi gagal maka proses amplifikasi tidak akan berjalan.
Fragmen hasil ligasi kemudian diamplifikasi dengan teknik PCR. Amplifikasi
pada AFLP dibagi menjadi dua tahap yaitu preamplifikasi dan amplifikasi selektif.
Preamplifikasi dilakukan dengan primer selektif yang memiliki satu tambahan basa A
pada primer EcoRI (E-A) dan basa C pada primer MseI (M-C). Menurut Vos et al 1995, preamplifikasi bertujuan mengurangi kompleksitas fragmen hasil digesti
sehingga tidak terjadi kesalahan penempelan primer pada amplifikasi selektif.
Amplifikasi selektif dilakukan dengan primer selektif yang memiliki
tambahan 3 basa pada ujung 3’. Primer selektif adalah primer yang berfungsi
menyeleksi fragmen hasil digesti dengan adanya basa-basa selektif pada ujung 3’.
Terdapat 6 jenis primer selektif yang digunakan pada ujung potongan EcoRI dan 6 jenis primer pada ujung potongan MseI. Kombinasi 12 pasang primer selektif dapat dilihat pada Tabel 5.
Kombinasi primer digunakan untuk melihat polimorfisme (pola pita) secara
lengkap. Masing-masing primer memiliki komposisi basa-basa primer selektif.
Basa-basa selektif pada primer melekat pada fragmen hasil digesti yang memiliki Basa-basa-Basa-basa
berkomplemen. Perbedaan basa-basa selektif pada setiap primer mengakibatkan
perbedaan amplifikasi fragmen sehingga menghasilkan perbedaan pita berdasarkan
ukuran pita yang dihasilkan dari masing-masing aksesi. Amplifikasi DNA 32 aksesi
temulawak dengan 12 kombinasi primer menghasilkan jumlah pita yang terlihat pada
Tabel 7.
Tabel 7 Jumlah pita hasil amplifikasi DNA dari 32 aksesi temulawak paada setiap
kombinasi primer AFLP
Kombinasi primer Rata-rata jumlah pita hasil
amplifikasi
EAAC-MCTG 55
EAAC-MCTA 51
EAAC-MCTC 53
EAAC-MCAG 55
EAAC-MCAC 60
EACA-MCTG 51
EACA-MCAA 50
EACA-MCTA 42
EACA-MCTC 50
EACA-MCAG 47
EACA-MCAC
Jumlah
54
621
Rata-rata 52
Pada penelitian ini PCR terhadap 32 aksesi temulawak dengan 12 kombinasi
primer menghasilkan rata-rata 52 fragmen DNA tiap kombinasi primer yang
berukuran 18bp hingga 350bp (Tabel 7). Sesuai yang dikemukakan Myburg et al. 2001, bahwa total jumlah fragmen yang dihasilkan dalam amplifikasi tergantung pada
jumlah dan komposisi nukleotida yang digunakan. Jumlah total fragmen
teramplifikasi juga tergantung pada kompleksitas genom. Jumlah fragmen yang
bersifat polimorfis tergantung pada variasi genetik antar sampel yang dianalisis.
Polimorfisme yang dihasilkan tidak hanya dipengaruhi oleh kombinasi pasangan basa
yang digunakan dan kompleksitas genom, tetapi juga dipengaruhi oleh penggunaan
enzim restriksi (Bonin et al. 2005).
Banyaknya fragmen DNA yang terbentuk menunjukkan bahwa primer yang
digunakan bersifat multilokus. Kemampuan suatu primer dalam mengungkap
keragaman genetik, ditunjukkan oleh banyaknya fragmen DNA polimorfis yang
perbedaan jumlah molekul. Tingkat polimorfisme dipengaruhi oleh substitusi basa,
delesi dan insersi pada situs penempelan primer, situs enzim restriksi atau perubahan
ukuran fragmen restriksi dan hasil amplifikasi (Spooner et al. 2005).
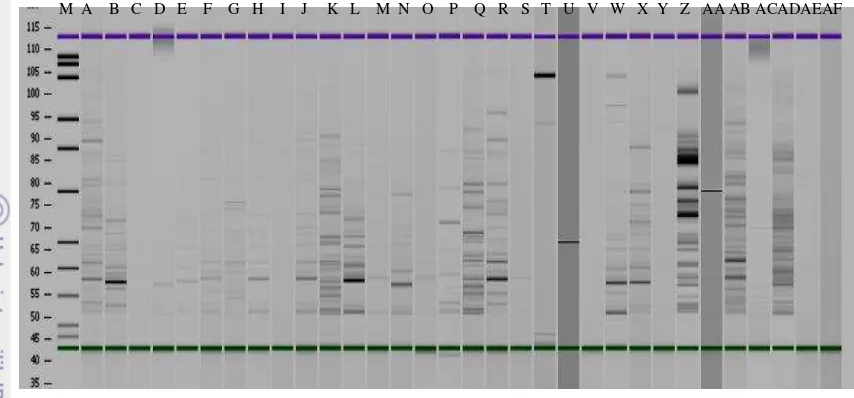
Hasil amplifikasi DNA dengan PCR menggunakan kombinasi primer
EAAC-MCTC dan EAAC-MCTG disajikan pada Gambar 6 dan 7. Pengamatan terhadap pola
pita yang dihasilkan setiap jenis kombinasi primer menghasilkan pita DNA yang
berbeda. Jumlah pita yang dihasilkan sangat bergantung pada komplementasi
terhadap DNA cetakan. Semakin banyak situs penempelan dari primer yang
digunakan, semakin banyak jumlah pita DNA yang dihasilkan. Hasil PCR pada 32
aksesi temulawak dengan primer EAAC-MCAG dan EAAC-MCTA disajikan secara
berturut-turut pada Lampiran 6 dan 7.
Gambar 6 Profil DNA dari 32 aksesi temulawak, hasil amplifikasi menggunakan pasangan primer EAAC-MCTC. M=Ladder 1kb, A-AF= aksesi temulawak (sesuai dengan kode pada tabel 4)
(bp)
1500 1000 700
500
400
300
200 150
100 50 15
(bp) 1500 1000 700
500
400
300
200
150 100
50 15
[image:43.612.95.539.33.766.2]
Gambar 7 Profil DNA dari 32 aksesi temulawak, hasil amplifikasi menggunakan pasangan primer EAAC-MCAA. M=Ladder 1kb, A-AF= aksesi temulawak (sesuai dengan kode pada tabel 4)
Perbedaan komposisi basa pada primer menghasilkan perbedaan fragmen
yang teramplifikasi. Perbedaan fragmen tersebut kemudian dibedakan berdasarkan
ukuran. Berdasarkan hasil pola pita yang diperoleh oleh setiap primer, tidak ada pola
pita yang identik antar aksesi temulawak sehinga setiap primer menghasilkan pola
pita yang unik untuk tiap aksesi. Menurut Loh et al. (2000), setiap pasangan primer
mampu menghasilkan pola pita yang spesifik untuk setiap sampel sehingga
digunakan sebagai identitas sampel. Perbedaan pola pita dapat menggambarkan
perbedaan genetik pada setiap aksesi temulawak.
Analisis Klaster Aksesi Temulawak Berdasarkan AFLP
Berdasarkan profil pita DNA setelah diinterpretasi dan diterjemahkan ke
dalam data biner, analisis klaster terhadap 32 aksesi temulawak yang didasarkan pada
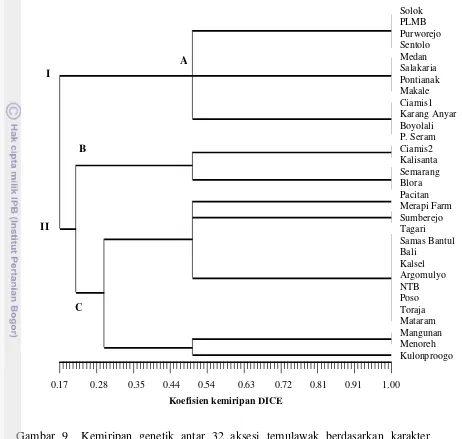
AFLP dengan menggunakan 12 kombinasi primer disajikan pada Gambar 8. Ke 32
aksesi mempunyai kemiripan yang relatif rendah yaitu antara 7-57%. Hal ini
menunjukkan bahwa ke 32 aksesi temulawak memiliki keragaman genetik yang
tinggi yaitu 43-93%. Beberapa studi menunjukkan bahwa dalam satu populasi alami
[image:44.612.113.540.95.294.2]genetik. Autosegregasi, mutasi somatik, dan instabilitas genetik diduga berperan
sebagai sumber variasi individu dalam populasi alami.
Pengelompokkan 32 aksesi temulawak pada tingkat kemiripan 0.10 (10%)
membentuk dua kelompok. Kelompok I terdiri dari 5 akesi, yaitu temulawak asal
Palembang, Pacitan, Kalisanta, Argomulyo dan Ciamis2. Kelompok I terbagi lagi
menjadi dua sub kelompok. Kelompok II terdiri dari 27 aksesi, yaitu temulawak asal
Makale, Pontianak, Kulonprogo, Mataram, Boyolali, Salatiga, Sumberejo, Bali, P.
Seram, Sentolo, Purworejo, Samas Bantul, Ciamis1, Blora, Semarang, Poso, Kalsesl,
Tagari, Merapi Farm, Salakaria, NTB, Menoreh, Karang Anyar, Mangunan, Medan,
Toraja, dan Solok. Kelompok II yang beranggotan 27 aksesi, terbagi menjadi dua sub
koefisien kemiripan DICE
0.07 0.12 0.18 0.23 0.29 0.35 0.40 0.46 0.51 0.57
[image:46.612.106.551.90.530.2]Solok Toraja Medan Mangunan KarangAnyar Menoreh NTB Salakaria MerapiFarm Tagari Kalsel Poso Semarang Blora Ciamis1 SamasBantul Purworejo Sentolo Pseram Bali Sumberejo Salatiga Boyolali Mataram Kulonprogo Pontianak Makale PLMB Pacitan Cimis2 Kalisanta Argomulyo
Gambar 8 Kemiripan genetik antar 32 aksesi temulawak berdasarkan penanda AFLP menggunakan 12 kombinasi primer.
Tabel 8 Kelompok aksesi yang terbentuk berdasarkan data AFLP pada koefisien 0.10 Kelompok Utama Sub Kelompok Aksesi
I A
B
Ciamis2, Kalisanta, Argomulyo
PLMB, Pacitan II I B A C D
0.07 0.12 0.18 0.23 0.29 0.35 0.40 0.46 0.51 0.57 Solok Toraja Medan Mangunan Karang Anyar Menoreh NTB Sakaria Merapi Farm Tagari Kalsel Poso Semarang Blora Ciamis1 Samas Bantul Purworejo Sentolo P. Seram Bali Sumberejo Salatiga Boyolali Mataram Kulonprogo Pontianak Makale PLMB Pacitan Ciamis2 Kalisanta Argomulyo
II C
D
Makale, Pontianak, Kulonprogo, Mataram, Boyolali, Salatiga, Sumberejo, Bali, P. Seram, Sentolo,
Purworejo, Samas Bantul, Blora, Ciamis1,
Semarang, Poso, Kalsel, Tagari, Merapi Farm,
Salakaria, NTB, Menoreh, Karang Anyar,
Mangunan, Medan
Solok, Toraja
Fenomena yang menarik dari hasil pengelompokkan tersebut adalah
mengelompoknya individu dari lokasi yang berlainan ke dalam satu kelompok.
Seperti halnya yang terlihat pada Kelompok I, kelompok ini memiliki anggota
temulawak yang berasal dari Palembang, Pacitan, Kalisanta, Argomulyo dan Ciamis.
Hal ini kemungkinan terjadi karena temulawak yang ada di Palembang berasal dari
Pulau Jawa. Selain itu, menurut Karuniawan et al. (2008), bahwa populasi dari habitat yang sama belum tentu memiliki hubungan kekerabatan yang dekat. Di habitat
yang sama sering terdapat juga genotip-genotip yang berbeda asalnya. Hal tersebut
dipengaruhi oleh faktor lingkungan atau adanya interaksi antara genotip dengan
lingkungan.
Rendahnya nilai kemiripan genetik berdasarkan data AFLP, menunjukkan
bahwa pada penelitian ini terdapat variasi genetik yang tinggi diantara aksesi
temulawak yang diamati. Keragaman genetik suatu populasi disebabkan karena
adanya mutasi, rekombinasi, atau migrasi gen dari satu tempat ke tempat lainnya.
Selain itu, analisis AFLP adalah sangat sensitif sehingga perbedaan kualitas DNA
sangat berpengaruh terhadap pola pita yang dihasilkan. Perbedaan kualitas DNA
dapat disebabkan oleh perbedaan metode isolasi. Untuk menghindari perbedaan pola
pita AFLP yang disebabkan oleh perbedaan kualitas DNA, maka AFLP sebaiknya
dilakukan lebih dari satu kali dengan menggunakan DNA yang diisolasi lebih dari
satu kali sampai mendapatkan pola pita yang konsisten untuk setiap aksesi.
Temulawak umumnya tidak membentuk buah atau biji, karena temulawak
(2n=3x=63). Hal ini menyebabkan temulawak berkembang biak secara vegetatif
melalui rimpang. Oleh karena persilangan seksual pada temulawak ini sangat jarang
terjadi maka keragaman yang terdapat diantara 32 aksesi kemungkinan disebabkan
oleh mutasi. Keragaman genetik yang disebabkan oleh mutasi dapat terjadi meliputi
substitusi, inversi, translokasi dan delesi. Perubahan pada tingkat DNA tersebut dapat
terdeteksi secara molekuler, sehingga menimbulkan keragaman pada tingkat DNA
(Transkley 1995). Perubahan pada tingkat gen akan menyebabkan terjadinya
perubahan pada kodon-kodon mRNA yang akhirnya menyebabkan perubahan
metabolisme serta fenotipe tanaman. Besarnya perubahan yang terjadi sangat
tergantung pada peranan asam amino yang berubah tersebut dalam menentukan
struktur akhir protein (Jusuf 2001).
Analisis Klaster Aksesi Temulawak Berdasarkan Karakter Morfologi
Berdasarkan karakter morfologi yang teramati pada penelitian ini ke 32 aksesi
temulawak mempunyai kemiripan genetik berkisar dari 0.17-1.00 atau 17-100% atau
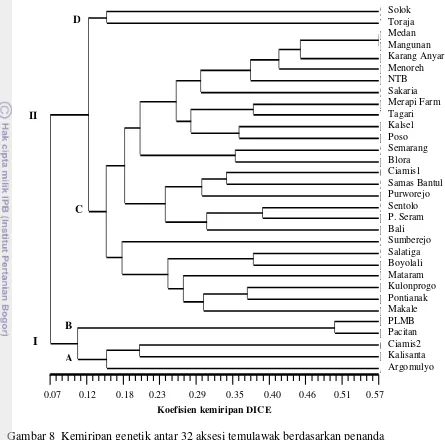
keragaman genetik yang relatif rendah yaitu 0-83%. Pengelompokan 32 aksesi
temulawak berdasarkan karakter morfologi yang teramati, pada koefisien kemiripan
0.17 (17%) membentuk dua kelompok (Gambar 9, Tabel 9). Kelompok I terdiri dari
aksesi temulawak asal Solok, Palembang, Purworejo, Sentolo, Medan, Salakaria,
Pontianak, Makale, Ciamis 1, Karang Anyar, Boyolali, P. Seram. Kelompok II terdiri
dari temulawak asal aksesi Ciamis 2, Kalisanta, Semarang, Blora, Pacitan, Merapi
Farm, Sumberejo, Tagari, Salatiga, Samas Bantul, Bali, Kalsel, Argomulyo, NTB,
Poso, Toraja, Mataram, Mangunan, Menoreh, dan Kulonprogo.
Berdasarkan kemiripan 100%, ke 32 aksesi temulawak mengelompok menjadi
10 kelompok. Dari analisis klaster (Gambar 8), terlihat ada beberapa aksesi
temulawak yang memiliki kemiripan sangat dekat (100%). Hal ini menunjukkan
secara morfologi, aksesi temulawak yang digunakan dalam penelitian ini mempunyai
kemiripan genetik yang tinggi (17-100%). Morfologi beberapa aksesi temulawak
Tabel 9 Kelompok aksesi yang terbentuk berdasarkan karakter morfologi pada koefisien 0.17
Kelompok Utama
Sub Kelompok
Aksesi
I A Solok, Palembang, Purworejo, Sentolo, Medan,
Salakaria, Pontianak, Makale, Ciamis 1, Karang Anyar, Boyolali, P. Seram
II B
C
Ciamis 2, Kalisanta, Semarang, Blora, Pacitan, Merapi Farm, Sumberejo, Tagari, Salatiga, Samas Bantul, Bali, Kalsel, Argomulyo, NTB, Poso, Toraja, Mataram
[image:49.612.92.521.73.756.2]koefisien kemiripan DICE
0.17 0.26 0.35 0.44 0.54 0.63 0.72 0.81 0.91 1.00
Solok
PLMB
Purworejo
Sentolo
Medan
Salakaria
Pontianak
Makale
Ciamis1
KarangAnyar
Boyolali
Pseram
Cimis2
Kalisanta
Semarang
Blora
Pacitan
MerapiFarm
Sumberejo
Tagari
Salatiga
SamasBantul
Bali
Kalsel
Argomulyo
NTB
Poso
Toraja
Mataram
Mangunan
Menoreh
Kulonprogo
Gambar 9 Kemiripan genetik antar 32 aksesi temulawak berdasarkan karakter morfologi.
Klaster yang terbentuk dengan menggunakan data AFLP hasilnya berbeda
dengan klaster yang terbentuk dengan menggunakan data morfologi. Analisis
kemiripan berdasarkan AFLP menunjukkan bahwa tidak ada satupun aksesi yang
mempunyai kemiripan 100% dengan aksesi lain, sedangkan berdasarkan karakter
morfologi, beberapa aksesi mempunya kemiripan 100%. Beberapa aksesi yang
mempunyai karakter morfologi warna ungu pita tulang daun dan warna rimpang yang I II A B C Solok PLMB Purworejo Sentolo Medan Salakaria Pontianak Makale Ciamis1 Karang Anyar Boyolali P. Seram Ciamis2 Kalisanta Semarang Blora Pacitan Merapi Farm Sumberejo Tagari Samas Bantul Bali Kalsel Argomulyo NTB Poso Toraja Mataram Mangunan Menoreh Kulonproogo
0.17 0.28 0.35 0.44 0.54 0.63 0.72 0.81 0.91 1.00
[image:50.612.97.559.89.528.2]sama mempunyai profil AFLP yang berbeda. Penelitian ini menunjukkan bahwa
kemiripan fenotip tidak menunjukkan kemiripan pada tingkat DNA.
Perbedaan pengelompokkan dapat disebabkan oleh perbedaan jumlah karakter
yang diamati. Semakin banyak karakter yang diamati maka hasil pengelompokkannya
menjadi semakin bagus. Pada penelitian ini pengelompokkan yang didasarkan pada
AFLP melibatkan 12 pasang primer yang menghasilkan pola pita yang sangat
beragam antar 32 aksesi. Walaupun menghasilkan keragaman yang tinggi, analisis
AFLP pada penelitian ini mempunyai kelemahan karena hanya dilakukan satu kali
sehingga pola pita yang dihasilkan belum dikonfirmasi.
Pengelompokkan yang didasarkan pada karakter morfologi pada penelitian ini
relatif kasar karena karakter yang diamati masih sangat terbatas, hanya dua karakter,
yaitu warna ungu pita tulang daun dan warna rimpang. Untuk meningkatkan
ketelitian dalam pengelompokkan, maka karakter morfologi yang diamati harus
SIMPULAN
1. Berdasarkan data AFLP, ke 32 aksesi temulawak mempunyai keragaman yang
tinggi yaitu berkisar 43-93%. Pengelompokan yang berdasarkan pada kemiripan
10% pada AFLP, ke 32 aksesi terbagi ke dalam dua kelompok. Kelompok I terdiri
dari temulawak asal Palembang, Pacitan dan Ciamis dan kelompok II terdiri dari
temulawak asal Makale, Pontianak, Kulonprogo, Mataram, Boyolali, Salatiga,
Sumberejo, Bali, P. Seram, Sentolo, Purworejo, Samas Bantul, Ciamis1, Blora,
Semarang, Poso, Kalsesl, Tagrai, Merapi Farm, Salakaria, NTB, Menoreh,
Karang Anyar, Mangunan, Medan, Toraja, dan Solok.
2. Berdasarkan karakter morfologi ke 32 aksesi temulawak mempunyai keragaman
berkisar 0-83%. Beberapa aksesi temulawak mempunyai karakter morfologi
warna pita ungu ibu tulang daun dan warna rimpang yang sama. Pada koefisien
kemiripan 0.17 (17%) ke 32 aksesi temulawak membentuk dua kelompok.
Kelompok I terdiri dari aksesi temulawak asal Solok, Palembang, Purworejo,
Sentolo, Medan, Salakaria, Pontianak, Makale, Ciamis 1, Karang Anyar,
Boyolali, P. Seram. Kelompok II terdiri dari temulawak asal aksesi Ciamis 2,
Kalisanta, Semarang, Blora, Pacitan, Merapi Farm, Sumberejo, Tagari, Salatiga,
Samas Bantul, Bali, Kalsel, Argomulyo, NTB, Poso, Toraja, Mataram,
Mangunan, Menoreh, dan Kulonprogo.
3. Klaster yang terbentuk dengan menggunakan data AFLP hasilnya berbeda dengan
klaster yang terbentuk dengan menggunakan data morfologi. Penelitian
menunjukkan bahwa kemiripan fenotip tidak menunjukkan kemiripan pada
DAFTAR PUSTAKA
Afifah, E dan Tim Lentera. 2003. Khasiat dan Manfaat Temulawak. Rimpang
Penyembuh Aneka Penyakit. Jakarta: Agromedia Pustaka.
Angeles JGC, Laurena AC, Tecson-Mendoza EM. 2005. Extraction of genomic DNA
from the lipid, polysaccharide, and polyphenol-free sugarcane DNA. Plant Mol
Biol Rep 17: 1-8.
Bonin AF, Pompanon F, Taberlet P. 2005. Use of Amplified Fragment Length
Polymorphism (AFLP) markers in surveys of vertebrata diversity. Methods Enzimol 395:145-161.
Dickes GJ, PV Nicholas. 1976. Gas chromatography in food analysis. London:
Verlay Butterworth.
Dzulkarnain B, Wahjoedi B. 1996. Informasi ilmiah kegunaan kosmetika tradisional.
Cermin Dunia Kedokteran 108: 21-