KLASIFIKASI
PROTEIN
FAMILY
MENGGUNAKAN
METODE RANTAI MARKOV
SONY MUHAMMAD
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Klasifikasi
Protein
Family
Menggunakan Metode Rantai Markov adalah benar karya saya dengan
arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada
perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya
yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam
teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut
Pertanian Bogor.
Bogor, November 2013
ABSTRAK
SONY MUHAMMAD. Klasifikasi Protein Family Menggunakan Metode Rantai Markov. Dibimbing oleh TOTO HARYANTO.
Protein adalah molekul organik yang terbentuk dari rangkaian dan kombinasi 20 asam amino. Protein memiliki banyak fungsi bagi makhluk hidup seperti melakukan katalisasi reaksi metabolisme, replikasi DNA, dan transportasi molekul. Semakin banyaknya sekuens protein baru yang berhasil ditemukan menyebabkan diperlukannya metode untuk mengklasifikasi protein ke dalam kelas yang sama. Penelitian ini bertujuan untuk menerapkan konsep rantai Markov untuk melakukan klasifikasi protein family berdasarkan sekuens asam amino. Sampel data protein diperoleh dari website Pfam protein family database. Data yang diambil sebanyak tiga kelas protein family, yaitu kelas 1-cysPrx_C, 4HBT dan ABC_Tran. Masing-masing kelas diambil sebanyak seratus data. Penelitian ini menggunakan dua cara ekstraksi protein yaitu orde satu dan orde dua. Orde satu menandakan bahwa data sekuens yang disimpan ke dalam matriks transisi adalah tiap dua sekuens yang tepat bersebelahan sedangkan orde dua menandakan bahwa data sekuens yang disimpan ke dalam matriks transisi adalah tiap dua sekuens yang saling berselang dua kali. Orde satu menghasilkan akurasi terbaik sebesar 96% dan orde dua menghasilkan akurasi terbaik sebesar 89.33%.
Kata kunci: klasifikasi, protein, rantai Markov
ABSTRACT
SONY MUHAMMAD. Protein Family Classification Based on Markov Chain Method. Supervised by TOTO HARYANTO.
Proteins are largebiological molecules that consist of one or more chains of 20 amino acids sequences. Proteins have various functions such as catalyzing metabolic reactions, replicating DNA, and transporting molecules. Many new protein sequences have been found. Therefore, a new method is required to classify proteins which have similar amino acids sequence, structure and function into the same class. The aim of this research is to apply Markov chain concept for protein family classification based on amino acid sequences. Protein data samples are obtained from Pfam protein family database website. Samples are taken from three different protein family classes, namely: 1-cysPrx_C, 4HBT and ABC_Tran, 100 data samples each. This research utilizes two different extraction methods: first order and second order Markov chain. First order indicates that the data sequence stored in the transition matrix is every two adjacent sequences, while the second order indicates that the data sequence stored in the transition matrix is every two sequences that are intermittent twice. The results show that the first order method gives 96% as its best accuracy, whereas the second order method gives 89.33% as its best accuracy.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Ilmu Komputer
pada
Departemen Ilmu Komputer
KLASIFIKASI
PROTEIN
FAMILY
MENGGUNAKAN
METODE RANTAI MARKOV
SONY MUHAMMAD
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
Penguji:
1 Dr Wisnu Ananta Kusuma, ST, MT
Judul Skripsi :Klasifikasi Protein Family Menggunakan Metode Rantai Markov Nama : Sony Muhammad
NIM : G64096058
Disetujui oleh
Toto Haryanto, SKom MSi Pembimbing
Diketahui oleh
Dr Ir Agus Buono, MSi MKom Ketua Departemen
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga penulis dapat menyelesaikan penelitian dan tulisan ini. Shalawat serta salam penulis sampaikan kepada Nabi besar Muhammad shallallahu ‘alaihi wasallam beserta keluarga, sahabat, serta umatnya hingga akhir zaman. Tulisan yang merupakan hasil penelitian yang penulis lakukan sejak November 2011 ini mengambil topic klasifikasi protein family menggunakan metode rantai Markov.
Terima kasih penulis ucapkan kepada seluruh pihak yang telah berperan membantu dalam penelitian ini, yaitu:
1 Ibunda Yeni Nuraeni, Ayahanda Herman Hamid, serta Adik Bobby Rachman atas kasih sayang, doa, dan dorongan semangat yang telah diberikan kepada penulis sehingga penelitian ini dapat terselesaikan.
2 Bapak Toto Haryanto, SKom MSi selaku pembimbing yang telah bersedia meluangkan waktunya untuk memberikan bantuan, arahan, serta kritik dan saran yang bermanfaat selama penelitian ini berlangsung.
3 Bapak Dr Wisnu Ananta Kusuma, ST, MT dan Bapak Muhammad Asyhar Agmalaro, SSi MKom selaku dosen penguji yang telah bersedia meluangkan waktunya untuk menguji serta memberikan masukan-masukan yang bermanfaat dalam penelitian ini.
4 Agni Prameswara SKom yang telah banyak membantu dalam penelitian ini. 5 Semua teman-teman Ilkom Alih Jenis.
Penulis menyadari bahwa masih terdapat banyak kekurangan dalam penulisan skripsi ini.Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL vii
DAFTAR GAMBAR vii
DAFTAR LAMPIRAN viii
PENDAHULUAN 1
Latar Belakang 1
Tujuan 2
Manfaat 2
Ruang Lingkup 2
TINJAUAN PUSTAKA 2
Protein 2
Protein Family 3
Model Rantai Markov 3
Ekstraksi Ciri Rantai Markov 4
K-Fold Cross Validation 5
METODE PENELITIAN 5
Kerangka Pemikiran 5
Pengambilan dan Praproses Data 6
Ekstraksi Ciri 6
Pembagian Data Latih dan Data Uji (K-Fold Cross Validation) 7
Data Eksternal 7
Pengujian 8
Analisa Hasil dan Model 8
Implementasi Aplikasi 8
Lingkungan Pengembangan 8
HASIL DAN PEMBAHASAN 9
Pengambilan Data 9
Praproses Data 10
Data Pelatihan dan Data Uji 10
Pengujian 10
Pengujian Orde 1 Fold 2 11
Pengujian Orde 1 Fold 3 13
Pengujian Orde 1 Fold 4 14
Pengujian Orde 2 Fold 1 16
Pengujian Orde 2 Fold 2 17
Pengujian Orde 2 Fold 3 19
Pengujian Orde 2 Fold 4 20
Pengujian Data Eksternal 22
SIMPULAN DAN SARAN 24
Simpulan 24
Saran 24
DAFTAR PUSTAKA 25
DAFTAR TABEL
1 Jenis protein yang digunakan dalam klasifikasi 9
2 Skenario k-fold cross validation 10
3 Akurasi orde 1 fold 1 11
4 Akurasi orde 1 fold 2 12
5 Akurasi orde 1 fold 3 13
6 Akurasi orde 1 fold 4 14
7 Akurasi orde 2 fold 1 16
8 Akurasi orde 2 fold 2 17
9 Akurasi orde 2 fold 3 19
10 Akurasi orde 2 fold 4 21
11 Pengujian data eksternal untuk orde satu 23
12 Pengujian data eksternal untuk orde dua 23
DAFTAR GAMBAR
1 Metode Penelitian 5
2 Contoh format FASTA 9
3 Hasil ekstraksi format FASTA 10
4 Grafik korelasi orde 1 fold 1 antara selisih error nilai matriks transisi data uji kelas 4HBT terhadap nilai matriks transisi data latih untuk
tiap-tiap kelas 11
5 Grafik korelasi orde 1 fold 2 antara selisih error nilai matriks transisi data uji kelas 4HBT terhadap nilai matriks transisi data latih untuk
tiap-tiap kelas 12
6 Grafik korelasi orde 1 fold 2 antara selisih error nilai matriks transisi data uji kelas ABC_Tran terhadap nilai matriks transisi data latih
untuk tiap-tiap kelas 12
7 Grafik korelasi orde 1 fold 3 antara selisih error nilai matriks transisi data uji kelas 1-cysPrx_C terhadap nilai matriks transisi data latih
untuk tiap-tiap kelas 13
8 Grafik korelasi orde 1 fold 3 antara selisih error nilai matriks transisi data uji kelas 4HBT terhadap nilai matriks transisi data latih untuk
tiap-tiap kelas 14
9 Grafik korelasi orde 1 fold 3 antara selisih error nilai matriks transisi data uji kelas ABC_Tran terhadap nilai matriks transisi data latih
untuk tiap-tiap kelas 14
10 Grafik korelasi orde 1 fold 4 antara selisih error nilai matriks transisi data uji kelas 1-cysPrx_C terhadap nilai matriks transisi data latih
untuk tiap-tiap kelas 15
11 Grafik korelasi orde 1 fold 4 antara selisih error nilai matriks transisi data uji kelas 4HBT terhadap nilai matriks transisi data latih untuk
tiap-tiap kelas 15
12 Grafik korelasi orde 1 fold 4 antara selisih error nilai matriks transisi data uji kelas ABC_Tran terhadap nilai matriks transisi data latih
13 Grafik korelasi orde 2 fold 1 antara selisih error nilai matriks transisi data uji kelas 4HBT terhadap nilai matriks transisi data latih untuk
tiap-tiap kelas 17
14 Grafik korelasi orde 2 fold 1 antara selisih error nilai matriks transisi data uji kelas ABC_Tran terhadap nilai matriks transisi data latih
untuk tiap-tiap kelas 17
15 Grafik korelasi orde 2 fold 2 antara selisih error nilai matriks transisi data uji kelas 4HBT terhadap nilai matriks transisi data latih untuk
tiap-tiap kelas 18
16 Grafik korelasi orde 2 fold 2 antara selisih error nilai matriks transisi data uji kelas ABC_Tran terhadap nilai matriks transisi data latih
untuk tiap-tiap kelas 18
17 Grafik korelasi orde 2 fold 3 antara selisih error nilai matriks transisi data uji kelas 1-cysPrx_C terhadap nilai matriks transisi data latih
untuk tiap-tiap kelas 19
18 Grafik korelasi orde 2 fold 3 antara selisih error nilai matriks transisi data uji kelas 4HBT terhadap nilai matriks transisi data latih untuk
tiap-tiap kelas 20
19 Grafik korelasi orde 2 fold 3 antara selisih error nilai matriks transisi data uji kelas ABC_Tran terhadap nilai matriks transisi data latih
untuk tiap-tiap kelas 20
20 Grafik korelasi orde 2 fold 4 antara selisih error nilai matriks transisi data uji kelas 1-cysPrx_C terhadap nilai matriks transisi data latih
untuk tiap-tiap kelas 21
21 Grafik korelasi orde 2 fold 4 antara selisih error nilai matriks transisi data uji kelas 4HBT terhadap nilai matriks transisi data latih untuk
tiap-tiap kelas 22
22 Grafik perbandingan nilai rata-rata antara orde satu dan orde dua
PENDAHULUAN
Latar Belakang
Protein merupakan elemen yang esensial bagi makhluk hidup. Protein secara struktur terdiri atas protein primer, protein sekunder dan protein tersier (Polanski dan Kimmel 2007). Klasifikasi protein memberikan keuntungan baik dalam informasi struktur, aktivitas dan sistem metabolisme di dalamnya (Wu et al. 2002). Klasifikasi terhadap protein family memiliki beberapa keuntungan antara lain: (1) memperbaiki identifikasi protein yang sulit dilakukan menggunakan metode pairwise alignments, (2) membantu dalam melakukan pemelihaaan pangkalan data protein family, (3) sebagai sarana yang efektif untuk dalam melakukan temu kembali terhadap informasi biologi dengan jumlah data yang besar, dan (4) merefleksikan gen dari suatu family dan melakukan analisis dalam perbandingan genom dan filogenetik.
Pangkalan data struktur primer protein yang berupa asam amino merupakan hasil translasi RNA dapat diklasifikasikan dengan beberapa kategori (Polanski dan Kimmel 2007) yaitu: (1) hierarchical family superfamilies / families pada tools PIR-PSD dan ProtoMap, (2) protein domain pada tools Pfam dan ProDom , (3) motif sekuens pada tools PROSITE dan PRINT, (4) struktur kelas protein pada tools SCOP dan CATH, dan (5) integrasi berbagai family pada tools iProClassn dan InterPro. Semua pangkalan data tersebut memiliki kegunaan yang spesifik.
2
Berdasarkan latar belakang tersebut, maka pada penelitian ini ditujukan untuk mengimplementasikan model rantai Markov untuk melakukan klasifikasi terhadap protein family.
Tujuan
Penelitian ini bertujuan untuk membuat model klasifikasi protein family menggunakan metode rantai Markov yang diharapkan dapat mengklasifikasi sekuens baru ke dalam kelas family yang memiliki kemiripan sekuens asam amino penyusunnya dengan tingkat akurasi yang cukup baik.
Manfaat
Manfaat dari penelitian ini adalah menghasilkan suatu tools untuk mengklasifikasikan suatu protein ke dalam family yang tepat sesuai dengan karakteristik dan fungsinya berdasarkan sekuens asam amino yang dimiliki.
Ruang Lingkup
Ruang lingkup penelitian ini adalah:
Struktur protein yang diklasifikasikan hanya sampai pada tingkat struktur primer. Proses klasifikasi tidak melibatkan struktur kompleks ataupun fungsi milik unidentified protein yang diujikan, tetapi hanya berdasarkan pola sekuens penyusunnya saja.
Kategori klasifikasi yang akan dilakukan adalah klasifikasi protein domain sehingga database yang digunakan adalah Pfam. Adapun data yang digunakan
dapat diunduh pada alamat sebagai berikut
ftp://ftp.sanger.ac.uk/pub/databases/pfam
Penelitian ini, melakukan klasifikasi sebanyak tiga kelas yang diambil secara acak dengan panjang sekuens yang berbeda-beda. Masing-masing kelas menggunakan seratus data.
TINJAUAN PUSTAKA
Protein
Protein (asal kata protos dari bahasa Yunani yang berarti "yang paling utama") adalah senyawa organik kompleks berbobot molekul tinggi yang merupakan polimer dari monomer-monomer asam amino yang dihubungkan satu sama lain dengan ikatan peptida. Molekul protein mengandung karbon, hidrogen, oksigen, nitrogen, dan kadang kala sulfur serta fosfor. Protein berperan penting dalam struktur dan fungsi semua sel makhluk hidup dan virus (Ussery 1998).
3 membentuk batang dan sendi sitoskeleton. Protein terlibat dalam sistem kekebalan (imun), sebagai antibodi, sistem kendali dalam bentuk hormon, sebagai komponen penyimpanan (dalam biji), dan juga dalam transportasi hara. Sebagai salah satu sumber gizi, protein berperan sebagai sumber asam amino bagi organisme yang tidak mampu membentuk asam amino tersebut (Ussery 1998).
Protein merupakan salah satu dari biomolekul raksasa selain polisakarida, lipid, dan polinukleotida yang merupakan penyusun utama makhluk hidup. Selain itu, protein merupakan salah satu molekul yang paling banyak diteliti dalam biokimia. Protein ditemukan oleh Jöns Jakob Berzelius pada tahun 1838 (Ussery 1998).
Biosintesis protein alami sama dengan ekspresi genetik. Kode genetik yang dibawa DNA ditranskripsi menjadi RNA, yang berperan sebagai cetakan bagi translasi yang dilakukan ribosom. Sampai tahap ini, protein masih "mentah", hanya tersusun dari asam amino proteinogenik. Melalui mekanisme pasca translasi, terbentuklah protein yang memiliki fungsi penuh secara biologi (Ussery 1998).
ProteinFamily
Protein-protein yang dikelompokkan ke dalam kelas yang sama, pada dasarnya memiliki keterhubungan secara evolusi. Protein-protein yang dikelompokkan ke dalam kelas yang sama, memiliki nenek moyang yang sama (ancestor), dan secara umum memiliki kemiripan, baik dalam struktur tiga dimensinya, fungsi, maupun sekuens penyusunnya. Meskipun seringkali sulit untuk mengevaluasi seberapa signifikan baik tingkat kemiripan fungsi maupun struktur antara protein yang satu dengan yang lain, pada dasarnya protein yang tidak memiliki ancestor yang sama, memiliki perbedaan yang cukup signifikan pada sekuens penyusunnya. Hal ini menyebabkan banyak dikembangkannya metode untuk mengklasifikasikan protein family berdasarkan sekuens asam amino penyusunnya. Saat ini, telah ditemukan lebih dari 60.000 kelas family (Kunin et al. 2003) meskipun masih terdapat banyaknya ambiguitas mengenai definisi yang paling tepat mengenai protein family, menyebabkan banyaknya perbedaan hasil yang ditemukan antara peneliti yang satu dengan yang lain.
Model Rantai Markov
4
sekuensial. Hal ini sesuai dengan karakterisik protein primer yang merupakan untaian sekuens dari asam amino yang membentuknya.
Ekstraksi Ciri Rantai Markov
Rantai Markov adalah suatu model stokastik yang diperkenalkan oleh matematikawan Rusia bernama A A Markov pada awal abad ke-20. Dengan menggunakan proses Markov maka dimungkinkan untuk memodelkan fenomena stokastik dalam dunia nyata yang berkembang menurut waktu. Masalah dasar dari metode stokastik dengan proses Markov adalah menentukan deskripsi state yang sesuai, sehingga proses stokastik yang berpaduan akan benar-benar memiliki apa yang akan disebut sifat Markov (Markovian property), yaitu pengetahuan terhadap state ini adalah cukup untuk memprediksi perilaku stokastik yang akan datang (Mangku 2005).
Suatu rantai Markov dikatakan diskret (discrete time Markov chain) jika ruang dari proses Markov tersebut adalah himpunan terbatas (finite) atau tercacah (countable), dengan himpunan indeks adalah T = (0, 1, 2, …) . Jika nilai suatu state pada periode tertentu hanya bergantung pada satu periode sebelumnya, maka rantai tersebut disebut rantai Markov orde satu (first order Markov chain) dan jika nilai suatu state pada periode tertentu bergantung pada periode sebelumnya, maka rantai tersebut disebut rantai Markov orde m (m order Markov chain). Rantai Markov orde satu secara matematika dirumuskan sebagai berikut.
Adapun rantai Markov orde m (m order Markov chain) secara matematika dirumuskan sebagai berikut.
Pada kasus perhitungan nilai peluang probabilitas suatu sekuens tertentu (misalnya sekuens DNA), diperlukan rumus untuk menghitung nilai peluang dari semua sekuens yang muncul pada history. Rumus yang digunakan adalah sebagai berikut.
Rumus di atas dinamakan asumsi Markov (Markov assumption). Rumus tersebut dapat digunakan untuk menghitung nilai peluang semua kombinasi sekuens yang mungkin muncul. Untuk kasus sekuens DNA, peluang semua kombinasi sekuens yang mungkin muncul adalah xi,xi-1∈ {20 monomer asam amino}. Dengan demikian, nilai probabilitas semua sekuens yang mungkin muncul dapat diketahui (Resch [tahun terbit tidak diketahui]).
P{X
n+1= j | X
n= i }
P{X
n+1= j | X
(n+1)-m= i
1, X
(n+1)-m+1= i
2,…, X
n= i
n}
n
P(x
1,…,
x
n) =
∏ P (
x
i| x
i-1)
5
K-Fold Cross Validation
Cross validation disebut juga sebagai rotation estimation. Dataset V dibagi menjadi k subset (fold) yang saling bebas secara acak, yaitu D1, D2,…Dk dengan ukuran yang sama. Pemodelan dan pengujian dilakukan sebanyak k kali, setiap kali iterasi ke-t (t=1,2,3…,k) dilatih pada D/Dt dan diuji pada Dt. Perkiraan akurasi pada cross validation dengan membagi jumlah keseluruhan klasifikasi yang benar dengan seluruh instances pada dataset (Kohavi 1995).
METODE PENELITIAN
Kerangka Pemikiran
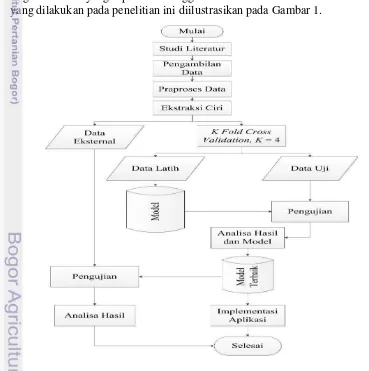
Penelitian ini dilakukan dengan beberapa tahapan proses untuk mengetahui tingkat akurasi yang diperoleh menggunakan metode rantai Markov. Tahap-tahap yang dilakukan pada penelitian ini diilustrasikan pada Gambar 1.
6
Pengambilan dan Praproses Data
Pfam merupakan pusat database komprehensif yang meliputi berbagai macam koleksi data mengenai protein family. Website ini mengoleksi hampir sekitar 12 000 data protein family yang diperoleh melalui penelitian, komputasional, eksperimen, dan penelitian biologi mengenai spesifik protein, bentuk asal serta hasil evolusinya. Pfam juga telah dipergunakan secara luas oleh berbagai macam komunitas yang bergerak di bidang structural biologi untuk mengidentifikasi jenis protein baru.
Data yang terdapat pada pfam merupakan data dengan format FASTA dan dapat diunduh di http://www.pfam.sanger.co.uk. Data yang diambil sebanyak tiga kelas protein family, yaitu kelas 1-cysPrx_C, 4HBT, dan ABC_Tran. Masing- masing kelas diambil sebanyak seratus data.
Selanjutnya, dilakukan praproses terhadap data yang telah diambil, yaitu proses memisahkan informasi identifier dengan sekuensnya atau bisa disebut dengan ekstraksi data, sehingga diperoleh sekuens data protein yang siap digunakan.
Ekstraksi Ciri
Di penelitian ini, matriks peluang transisi dibangun berdasarkan peluang-peluang munculnya asam amino tertentu berdasarkan sekuens data latih yang ditentukan. Matriks transisi yang dibangun berukuran 20 × 20 sesuai dengan banyaknya monomer atau jenis asam amino yang ada. Metode penyimpanan nilai sekuens ke dalam matriks transisi menggunakan dua cara, yaitu orde satu dan orde dua. Matriks transisi rantai Markov orde satu dibentuk dari peluang-peluang munculnya monomer asam amino tertentu setelah sebelumnya merupakan tepat satu monomer asam amino tertentu. Dengan demikian matriks transisi rantai Markov orde satu terdiri atas P(qi|qi-1), yaitu peluang munculnya asam amino qi setelah sebelumnya merupakan asam amino qi-1. Matriks transisi orde m (untuk penelitian ini digunakan m = 2) dibentuk dari peluang-peluang munculnya monomer asam amino tertentu setelah sebelumnya merupakan tepat dua monomer asam amino tertentu. Dengan demikian matriks transisi rantai Markov orde dua terdiri atas P(qi|qi-2), yaitu peluang munculnya asam amino qi setelah sebelumnya merupakan asam amino qi-2.
Berdasarkan ketentuan di atas, akan diperoleh dua metode penyimpanan
matriks transisi, yaitu matriks transisi untuk orde satu dan orde dua. Langkah selanjutnya, yaitu menghitung nilai total frekuensi kemunculan antara monomer asam amino yang satu terhadap monomer asam amino yang lain. Perhitungan ini diterapkan untuk tiap-tiap monomer asam amino yang ada pada matriks transisi. Nilai total frekuensi dihitung dengan cara berikut.
TOTAL (n) = (q1|n) + (q2 |n) + (q3|n) + … + (qn|n) dengan
n = untuk semua monomer asam amino (20 monomer)
7 Nilai total frekuensi yang telah diperoleh, selanjutnya digunakan untuk membagi nilai frekuensi kemunculan antara monomer asam amino yang satu terhadap monomer asam amino yang lain. Dengan demikian dapat diperoleh nilai probabilitas untuk tiap-tiap pasangan sekuens yang terdapat pada matriks transisi. Perhitungan untuk mendapatkan nilai probabilitas setiap elemen pada matriks transisi adalah sebagai berikut.
Guna menghindari pembagian dengan nol, ditambahkan smooth parameter dengan nilai 1×10-6 di seluruh nilai pada matriks transisi. Nilai-nilai probabilitas yang terdapat pada matriks transisi akan digunakan sebagai acuan untuk menghitung nilai akurasi, baik pada data uji maupun data latih.
Pembagian Data Latih dan Data Uji (K-Fold Cross Validation)
Data yang telah diekstrak kemudian dibagi menjadi data latih dan data uji menggunakan metode k-fold cross validation dengan k = 4. Data dibagi ke dalam 4 fold (S1, S2, S3, S4) dengan masing-masing fold memiliki jumlah anggota yang sama. Di tahapan ini proses identifikasi akan dilakukan 4 kali iterasi berdasarkan metode k-fold cross validation. Data latih dan data uji memiliki fold yang berbeda pada setiap iterasi. Di iterasi pertama, fold S2, S3 dan S4 akan digunakan sebagai data latih sedangkan fold S1 akan digunakan sebagai data uji. Akurasi dalam klasifikasi protein family dilakukan dengan menghitung jumlah kelas yang benar teridentifikasi / terklasifikasi dan membandingkan dengan banyaknya data yang diujikan pada setiap kelasnya. Persamaan untuk menghitung tingkat akurasi adalah sebagai berikut.
Data Eksternal
Data eksternal adalah data yang diambil dari website pfam selain tiga ratus data yang digunakan dalam proses k-fold cross validation. Bedanya, data eksternal langsung diujikan menggunakan model terbaik yang diperoleh dari hasil proses k-fold cross validation. Tujuan pengujian data eksternal adalah mengukur tingkat akurasi model terbaik yang diperoleh dari hasil proses k-fold cross validation terhadap data luar selain data yang digunakan dalam proses k-fold cross validation. Data yang digunakan untuk pengujian data eksternal adalah 25 data dari masing-masing kelas 1-cysPrx_C, 4HBT dan ABC_Tran, yang totalnya berjumlah 75 data.
P(q1|n) = (q1|n) / TOTAL (n) dengan
n = untuk semua monomer asam amino (20 monomer)
q = frekuensi kemunculan suatu asam amino terhadap asam amino yang lain
#Data yang teridentifikasi benar
8
Pengujian
Proses pengujian dilakukan dua kali, yaitu pengujian terhadap data yang digunakan dalam proses k-fold cross validation dan pengujian terhadap data eksternal. Pengujian data yang digunakan dalam proses k-fold cross validation dilakukan terhadap data uji sebanyak empat kali iterasi, baik untuk orde satu dan orde dua, sedangkan pengujian terhadap data eksternal dilakukan menggunakan model terbaik yang diperoleh dari hasil proses k-fold cross validation baik untuk orde satu maupun orde dua.
Analisa Hasil dan Model
Analisa hasil dan model dilakukan untuk memastikan bahwa model rantai Markov yang dihasilkan adalah model yang terbaik. Dengan model terbaik, akan menghasilkan akurasi yang paling tinggi dalam proses klasifikasi sehingga model tersebut yang nantinya akan digunakan pada saat implementasi. Model dengan tingkat akurasi terbaik juga akan digunakan untuk menghitung tingkat akurasi data eksternal.
Implementasi Aplikasi
Model terbaik yang telah dibuat kemudian diimplementasikan dalam bentuk aplikasi berbasis website. Aplikasi ini akan digunakan sebagai salah satu layanan proses klasifikasi protein family.
Lingkungan Pengembangan
Spesifikasi perangkat keras dan perangkat lunak yang digunakan untuk penelitian ini adalah sebagai berikut:
Perangkat keras:
Processor AMD Athlon x2 Dual-Core QL-65
Memory 3 GB
Hardisk 250 GB
Perangkat lunak:
Sistem operasi Microsoft Windows 7
ApacheFriends XAMPP version 1.7.4
9
HASIL DAN PEMBAHASAN
Pengambilan Data
Data protein yang digunakan pada penelitian ini merupakan kumpulan fold data yang diambil dari alamat ftp://ftp.sanger.ac.uk/pub/databases/Pfam. Data yang diambil merupakan untaian sekuens asam amino yang merupakan struktur primer protein. Adapun jenis protein yang digunakan pada penelitian ini dapat dilihat pada Tabel 1.
Tabel 1 Jenis protein yang digunakan dalam klasifikasi
No Nama Keterangan
1 1-cysPrx_C C-terminal domain dari 1-Cys peroxiredoxin
(1-cysPrx), anggota dari superfamili peroxiredoxin yang melindungi sel terhadap oksidasi membrane.
2 4HBT Berfungsi untuk melakukan katalisasi dalam
proses biosintesis.
3 ABC_Tran Atau disebut juga ABC Transporter yang
menggunakan hidrolisis ATP untuk mentranslokasi berbagai senyawa melintasi membran biologis.
Pengambilan tersebut dilakukan secara acak dengan masing-masing seratus sampel fragmen untuk setiap kelasnya. Dengan demikian terdapat tiga ratus data yang akan diambil untuk melakukan penelitian. Pembagian data latih dan data uji menggunakan metode k-fold cross validation dengan k = 4, dimana untuk tiap-tiap kelas 75 buah data digunakan sebagai data latih dan 25 buah data digunakan sebagai data uji. Data yang diperoleh memiliki format FASTA. FASTA merupakan salah satu tipe data dari data biologi yang disediakan. Adapun contoh format FASTA dapat dilihat pada Gambar 2.
>B3ARY6_ECO57/154-186 PF10417.3;1-cysPrx_C; merupakan identifier pfam dengan 1-cysPrx_C merupakan keterangan kelas protein family. Adapun deretan huruf dibawahnya merupakan sekuens asam amino atau protein.
Gambar 2 Contoh format FASTA
10
Praproses Data
Ditahap ini dilakukan ekstraksi data sehingga terjadi pemisahan antara identifier dengan sekuensnya. Lebih jelasnya dapat dilihat pada Gambar 3 berikut.
Data Pelatihan dan Data Uji
Data sebanyak tiga ratus sekuens dari tiga kelas. Setiap kelas terdiri atas 75 data pelatihan, 25 data akan dijadikan sebagai data uji. Guna menjamin bahwa setiap data yang digunakan pernah digunakan sebagai data pelatihan, digunakan k-fold cross validation. Skenario k-k-fold cross validation dapat dilihat pada Tabel 2 berikut.
Tabel 2 Skenario k-fold cross validation
Fold Data latih Data uji
1 S2 S3 S4 S1
2 S1 S3 S4 S2
3 S1 S2 S4 S3
4 S1 S2 S3 S4
Data S1 menandakan 25 data pertama dari tiap-tiap kelas. Kelas pertama atau kelas 1-cysPrx_C data S1 merupakan record data ke 1-25. Kelas kedua atau kelas 4HBT data S1 merupakan record data ke 101-125. Kelas ketiga atau kelas ABC_Tran data S1 merupakan record data ke 201-225.
Data S2 menandakan 25 data kedua dari tiap-tiap kelas. Kelas pertama atau kelas 1-cysPrx_C data S2 merupakan record data ke 26-50. Kelas kedua atau kelas 4HBT data S2 merupakan record data ke 126-150. Kelas ketiga atau kelas ABC_Tran data S2 merupakan record data ke 226-250.
Data S3 menandakan 25 data ketiga dari tiap-tiap kelas. Kelas pertama atau kelas 1-cysPrx_C data S3 merupakan record data ke 51-75. Kelas kedua atau kelas 4HBT data S3 merupakan record data ke 151-175. Kelas ketiga atau kelas ABC_Tran data S3 merupakan record data ke 251-275.
Data S4 menandakan 25 data keempat dari tiap-tiap kelas. Kelas pertama atau kelas 1-cysPrx_C data S4 merupakan record data ke 76-100. Kelas kedua atau kelas 4HBT data S4 merupakan record data ke 176-200. Kelas ketiga atau kelas ABC_Tran data S4 merupakan record data ke 276-300.
Pengujian
Pengujian dilakukan untuk mengetahui hasil akurasi dalam proses klasifikasi. Pada pengujian ini dilakukan skenario untuk membagi data menjadi
11 data latih dan data uji dengan menggunakan k-fold cross validation. Skenario penelitian ini menggunakan k = 4.
Pengujian Orde 1 Fold 1
Hasil pengujian orde1 fold 1 menunjukkan akurasi setiap kelas dengan presentase seperti dilihat pada Tabel 3.
Tabel 3 Akurasi orde1 fold 1
1-cysPrx_C 4HBT ABC_Tran Akurasi (%)
1-cysPrx_C 25 0 0 100.00
4HBT 0 12 13 48.00
ABC_Tran 0 0 25 100.00
Rata-rata 82.66
Pada Tabel 3 terlihat bahwa akurasi rata-rata yang diperoleh sebesar 82.66%. Hasil pengujian fold 1 untuk kelas 1-cysPrx_C (S1, record data ke 1-25) dan kelas ABC_Tran (S1, record data ke 201-225) dapat terklasifikasi dengan benar sesuai kelas asalnya, sedangkan hasil pengujian fold 1 untuk kelas 4HBT (S1, record data ke 101-125) menunjukkan, hanya dua belas di antaranya yang terklasifikasi dengan benar. Ketiga belas data lainnya terklasifikasi ke kelas ABC_Tran (bernilai salah).Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas 4HBT (S1, record data ke 101-125) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 4.
Pengujian Orde 1 Fold 2
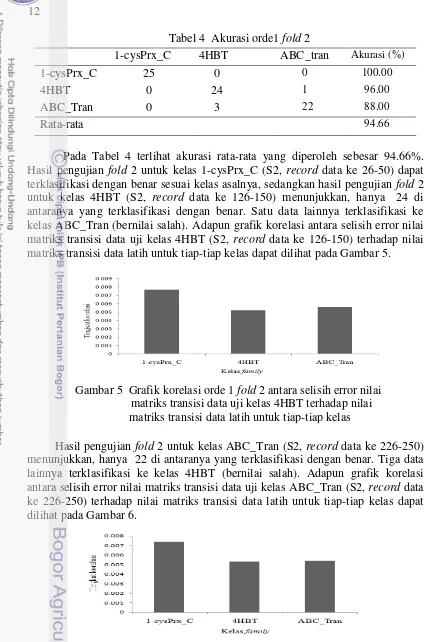
Hasil pengujian orde 1 fold 2 menunjukkan akurasi setiap kelas dengan presentase seperti dilihat pada Tabel 4.
Gambar 4 Grafik korelasi orde 1 fold 1 antara selisih error nilai matriks transisi data uji kelas 4HBT terhadap nilai matriks transisi data latih untuk tiap-tiap kelas
0 0.001 0.002 0.003 0.004 0.005 0.006 0.007 0.008
1-cysPrx_C 4HBT ABC_Tran
12
Tabel 4 Akurasi orde1 fold 2
1-cysPrx_C 4HBT ABC_tran Akurasi (%)
1-cysPrx_C 25 0 0 100.00
4HBT 0 24 1 96.00
ABC_Tran 0 3 22 88.00
Rata-rata 94.66
Pada Tabel 4 terlihat akurasi rata-rata yang diperoleh sebesar 94.66%. Hasil pengujian fold 2 untuk kelas 1-cysPrx_C (S2, record data ke 26-50) dapat terklasifikasi dengan benar sesuai kelas asalnya, sedangkan hasil pengujian fold 2 untuk kelas 4HBT (S2, record data ke 126-150) menunjukkan, hanya 24 di antaranya yang terklasifikasi dengan benar. Satu data lainnya terklasifikasi ke kelas ABC_Tran (bernilai salah). Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas 4HBT (S2, record data ke 126-150) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 5.
Hasil pengujian fold 2 untuk kelas ABC_Tran (S2, record data ke 226-250) menunjukkan, hanya 22 di antaranya yang terklasifikasi dengan benar. Tiga data lainnya terklasifikasi ke kelas 4HBT (bernilai salah). Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas ABC_Tran (S2, record data ke 226-250) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 6.
Gambar 5 Grafik korelasi orde 1 fold 2 antara selisih error nilai matriks transisi data uji kelas 4HBT terhadap nilai matriks transisi data latih untuk tiap-tiap kelas
13
Pengujian Orde 1 Fold 3
Hasil pengujian orde1 fold 3 menunjukkan akurasi setiap kelas dengan presentase seperti dilihat pada Tabel 5.
Tabel 5 Akurasi orde1 fold 3
1-cysPrx_C 4HBT ABC_tran Akurasi (%) 1-cysPrx_C 11 0 14 44.00
4HBT 0 23 2 92.00
ABC_Tran 0 2 23 92.00
Rata-rata 76.00
Pada Tabel 5 terlihat bahwa akurasi rata-rata yang diperoleh sebesar 76%. Hasil pengujian fold 3 untuk kelas 1-cysPrx_C (S3, record data ke 51-75) menunjukkan, hanya sebelas di antaranya yang terklasifikasi dengan benar. Empat belas data lainnya terklasifikasi ke kelas ABC_Tran (bernilai salah). Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas 1-cysPrx_C (S3, record data ke 51-75) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 7.
Hasil pengujian fold 3 untuk kelas 4HBT (S3, record data ke 151-175) menunjukkan, hanya 23 di antaranya yang terklasifikasi dengan benar. Dua data lainnya terklasifikasi pada ke kelas ABC_Tran (bernilai salah). Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas 4HBT (S3, record data ke 151-175) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 8.
14
Hasil pengujian fold 3 untuk kelas ABC_Tran (S3, record data ke 251-275) menunjukkan, hanya 23 di antaranya yang terklasifikasi dengan benar. Dua data lainnya terklasifikasi ke kelas 4HBT (bernilai salah). Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas ABC_Tran (S3, record data ke 251-275) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 9.
Pengujian Orde 1 Fold 4
Hasil pengujian orde1 fold 4 menunjukkan akurasi setiap kelas dengan presentase seperti dilihat pada Tabel 6.
Tabel 6 Akurasi orde1 fold 4
1-cysPrx_C 4HBT ABC_tran Akurasi (%)
1-cysPrx_C 24 0 1 96.00
4HBT 0 24 1 96.00
ABC_Tran 0 1 24 96.00
Rata-rata 96.00
Gambar 8 Grafik korelasi orde 1 fold 3 antara selisih error nilai matriks transisi data uji kelas 4HBT terhadap nilai matriks transisi data latih untuk tiap-tiap kelas
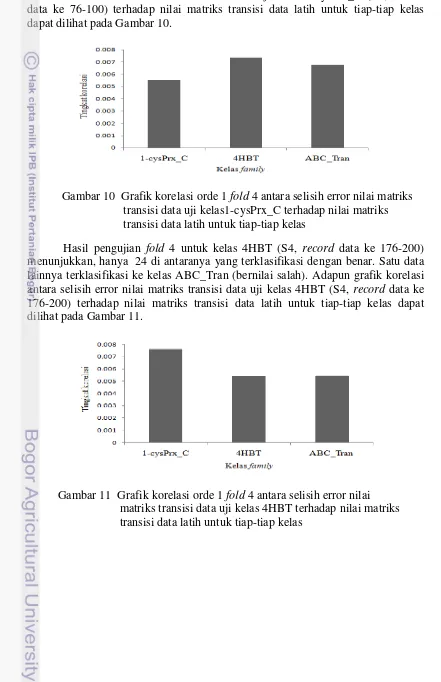
15 Pada Tabel 6 terlihat bahwa akurasi rata-rata yang diperoleh sebesar 96%. Hasil pengujian fold 4 untuk kelas 1-cysPrx_C (S4, record data ke 76-100) menunjukkan, hanya 24 di antaranya yang terklasifikasi dengan benar. Satu data lainnya terklasifikasi ke kelas ABC_Tran (bernilai salah). Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas 1-cysPrx_C (S4, record data ke 76-100) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 10.
Hasil pengujian fold 4 untuk kelas 4HBT (S4, record data ke 176-200) menunjukkan, hanya 24 di antaranya yang terklasifikasi dengan benar. Satu data lainnya terklasifikasi ke kelas ABC_Tran (bernilai salah). Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas 4HBT (S4, record data ke 176-200) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 11.
Gambar 10 Grafik korelasi orde 1 fold 4 antara selisih error nilai matriks transisi data uji kelas1-cysPrx_C terhadap nilai matriks transisi data latih untuk tiap-tiap kelas
16
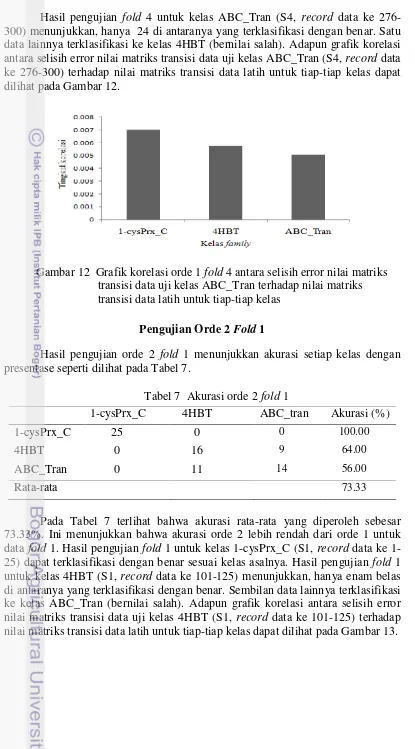
Hasil pengujian fold 4 untuk kelas ABC_Tran (S4, record data ke 276-300) menunjukkan, hanya 24 di antaranya yang terklasifikasi dengan benar. Satu data lainnya terklasifikasi ke kelas 4HBT (bernilai salah). Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas ABC_Tran (S4, record data ke 276-300) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 12.
Pengujian Orde 2 Fold 1
Hasil pengujian orde 2 fold 1 menunjukkan akurasi setiap kelas dengan presentase seperti dilihat pada Tabel 7.
Tabel 7 Akurasi orde 2 fold 1
1-cysPrx_C 4HBT ABC_tran Akurasi (%)
1-cysPrx_C 25 0 0 100.00
4HBT 0 16 9 64.00
ABC_Tran 0 11 14 56.00
Rata-rata 73.33
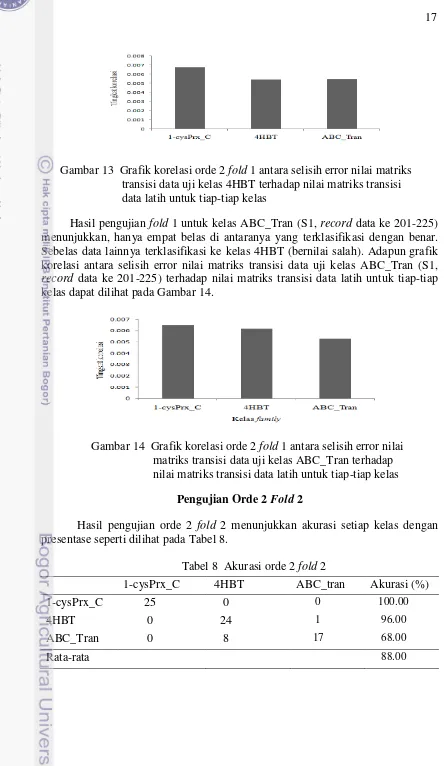
Pada Tabel 7 terlihat bahwa akurasi rata-rata yang diperoleh sebesar 73.33%. Ini menunjukkan bahwa akurasi orde 2 lebih rendah dari orde 1 untuk data fold 1. Hasil pengujian fold 1 untuk kelas cysPrx_C (S1, record data ke 1-25) dapat terklasifikasi dengan benar sesuai kelas asalnya. Hasil pengujian fold 1 untuk kelas 4HBT (S1, record data ke 101-125) menunjukkan, hanya enam belas di antaranya yang terklasifikasi dengan benar. Sembilan data lainnya terklasifikasi ke kelas ABC_Tran (bernilai salah). Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas 4HBT (S1, record data ke 101-125) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 13.
17
Hasil pengujian fold 1 untuk kelas ABC_Tran (S1, record data ke 201-225) menunjukkan, hanya empat belas di antaranya yang terklasifikasi dengan benar. Sebelas data lainnya terklasifikasi ke kelas 4HBT (bernilai salah). Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas ABC_Tran (S1, record data ke 201-225) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 14.
Pengujian Orde 2 Fold 2
Hasil pengujian orde 2 fold 2 menunjukkan akurasi setiap kelas dengan presentase seperti dilihat pada Tabel 8.
Tabel 8 Akurasi orde 2 fold 2
1-cysPrx_C 4HBT ABC_tran Akurasi (%)
1-cysPrx_C 25 0 0 100.00
4HBT 0 24 1 96.00
ABC_Tran 0 8 17 68.00
Rata-rata 88.00
Gambar 13 Grafik korelasi orde 2 fold 1 antara selisih error nilai matriks transisi data uji kelas 4HBT terhadap nilai matriks transisi data latih untuk tiap-tiap kelas
18
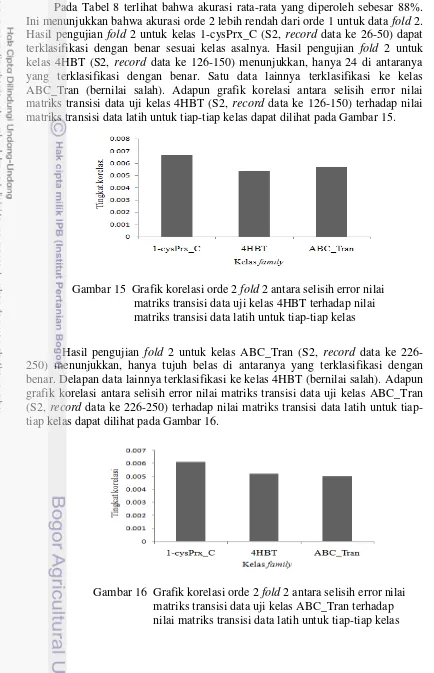
Pada Tabel 8 terlihat bahwa akurasi rata-rata yang diperoleh sebesar 88%. Ini menunjukkan bahwa akurasi orde 2 lebih rendah dari orde 1 untuk data fold 2. Hasil pengujian fold 2 untuk kelas 1-cysPrx_C (S2, record data ke 26-50) dapat terklasifikasi dengan benar sesuai kelas asalnya. Hasil pengujian fold 2 untuk kelas 4HBT (S2, record data ke 126-150) menunjukkan, hanya 24 di antaranya yang terklasifikasi dengan benar. Satu data lainnya terklasifikasi ke kelas ABC_Tran (bernilai salah). Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas 4HBT (S2, record data ke 126-150) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 15.
[image:30.595.59.493.85.758.2]Hasil pengujian fold 2 untuk kelas ABC_Tran (S2, record data ke 226-250) menunjukkan, hanya tujuh belas di antaranya yang terklasifikasi dengan benar. Delapan data lainnya terklasifikasi ke kelas 4HBT (bernilai salah). Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas ABC_Tran (S2, record data ke 226-250) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 16.
Gambar 15 Grafik korelasi orde 2 fold 2 antara selisih error nilai matriks transisi data uji kelas 4HBT terhadap nilai matriks transisi data latih untuk tiap-tiap kelas
19
Pengujian Orde 2 Fold 3
[image:31.595.89.513.47.795.2]Hasil pengujian orde 2 fold 3 menunjukkan akurasi setiap kelas dengan presentase seperti dilihat pada Tabel 9.
Tabel 9 Akurasi orde 2 fold 3
1-cysPrx_C 4HBT ABC_tran Akurasi (%)
1-cysPrx_C 11 4 10 44.00
4HBT 0 22 3 88.00
ABC_Tran 0 7 18 72.00
Rata-rata 68.00
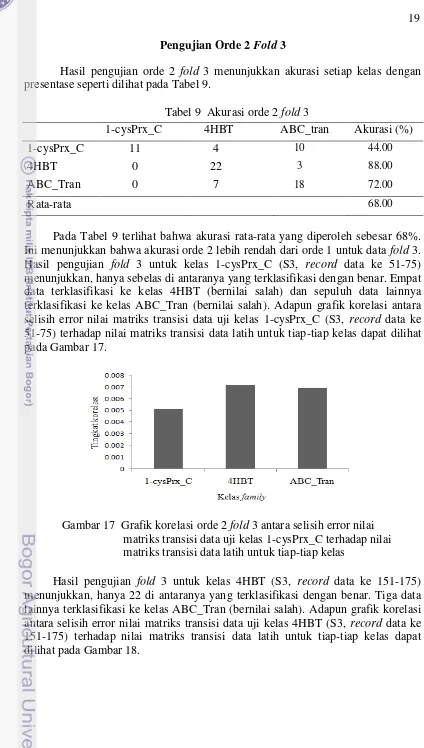
Pada Tabel 9 terlihat bahwa akurasi rata-rata yang diperoleh sebesar 68%. Ini menunjukkan bahwa akurasi orde 2 lebih rendah dari orde 1 untuk data fold 3. Hasil pengujian fold 3 untuk kelas 1-cysPrx_C (S3, record data ke 51-75) menunjukkan, hanya sebelas di antaranya yang terklasifikasi dengan benar. Empat data terklasifikasi ke kelas 4HBT (bernilai salah) dan sepuluh data lainnya terklasifikasi ke kelas ABC_Tran (bernilai salah). Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas 1-cysPrx_C (S3, record data ke 51-75) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 17.
Hasil pengujian fold 3 untuk kelas 4HBT (S3, record data ke 151-175) menunjukkan, hanya 22 di antaranya yang terklasifikasi dengan benar. Tiga data lainnya terklasifikasi ke kelas ABC_Tran (bernilai salah). Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas 4HBT (S3, record data ke 151-175) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 18.
20
Hasil pengujian fold 3 untuk kelas ABC_Tran (S3, record data ke 251-275) menunjukkan, hanya delapan belas di antaranya yang terklasifikasi dengan benar. Tujuh data lainnya terklasifikasi ke kelas 4HBT (bernilai salah). Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas ABC_Tran (S3, record data ke 251-275) dengan akurasi dari tiap-tiap kelas dapat dilihat pada Gambar 19.
Pengujian Orde 2 Fold 4
[image:32.595.50.508.71.796.2]Hasil pengujian orde 2 fold 4 menunjukkan akurasi setiap kelas dengan persentase seperti dilihat pada Tabel 10.
Gambar 18 Grafik korelasi orde 2 fold 3 antara selisih error nilai matriks transisi data uji kelas 4HBT terhadap nilai matriks transisi data latih untuk tiap-tiap kelas
21 Tabel 10 Akurasi orde 2 fold 4
1-cysPrx_C 4HBT ABC_tran Akurasi (%)
1-cysPrx_C 23 0 2 92.00
4HBT 0 19 6 76.00
ABC_Tran 0 0 25 100.00
Rata-rata 89.33
Pada Tabel 10 terlihat bahwa akurasi rata-rata yang diperoleh sebesar 89.33%. Ini menunjukkan bahwa akurasi orde 2 lebih rendah dari orde 1 untuk data fold 4. Hasil pengujian fold 4 untuk kelas 1-cysPrx_C (S4, record data ke 76-100) menunjukkan, hanya 23 di antaranya yang terklasifikasi dengan benar. Dua data lainnya terklasifikasi ke kelas ABC_Tran (bernilai salah). Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas 1-cysPrx_C (S4, record data ke 76-100) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 20.
Hasil pengujian fold 4 untuk kelas 4HBT (S4, record data ke 176-200) menunjukkan, hanya sembilan belas di antaranya yang terklasifikasi dengan benar. Enam data lainnya terklasifikasi ke kelas ABC_Tran (bernilai salah). Hasil pengujian fold 4 untuk kelas ABC_Tran (record data ke 276-300) dapat terklasifikasi dengan benar sesuai kelas asalnya. Adapun grafik korelasi antara selisih error nilai matriks transisi data uji kelas 4HBT (S4, record data ke 176-200) terhadap nilai matriks transisi data latih untuk tiap-tiap kelas dapat dilihat pada Gambar 21.
22
Adapun grafik perbandingan nilai rata-rata antara hasil orde satu dan orde dua dapat dilihat pada gambar 22 berikut.
Pengujian Data Eksternal
Pengujian data eksternal adalah pengujian untuk mengukur tingkat akurasi tool yang telah dikembangkan terhadap data eksternal atau data selain ketiga ratus data yang telah digunakan. Pengujian dilakukan menggunakan fold dengan tingkat akurasi terbaik, yaitu fold keempat baik untuk orde satu dan orde dua. Pengujian dilakukan menggunakan masing-masing 25 sampel data baik untuk kelas 1-cysPrx_C, 4HBT, dan ABC_Tran. Hasil pengujian data eksternal untuk orde satu dapat dilihat pada Tabel 11 berikut.
[image:34.595.49.500.64.716.2]Gambar 21 Grafik korelasi orde 2 fold 4 antara selisih error nilai matriks transisi data uji kelas 4HBT terhadap nilai matriks transisi data latih untuk tiap-tiap kelas
Gambar 22 Grafik perbandingan nilai rata-rata antara orde satu dan orde dua berdasarkan tiap-tiap fold
23
Tabel 11 Pengujian data eksternal untuk orde satu
1-cysPrx_C 4HBT ABC_Tran Akurasi (%)
1-cysPrx_C 24 0 1 96.00
4HBT 1 21 3 84.00
ABC_Tran 4 2 19 76.00
Rata-rata 85.33
Pada Tabel 11 terlihat bahwa akurasi rata-rata yang diperoleh sebesar 85.33%. Hasil pengujian data eksternal kelas 1-cysPrx_C, hanya 24 di antaranya yang terklasifikasi dengan benar. Satu data lainnya terklasifikasi ke kelas ABC_Tran (bernilai salah). Hasil pengujian data eksternal milik kelas 4HBT, hanya 21 di antaranya yang terklasifikasi dengan benar. Satu data lainnya terklasifikasi ke kelas 1-cysPrx_C (bernilai salah) dan tiga data lainnya terklasifikasi ke kelas ABC_Tran (bernilai salah). Hasil pengujian data eksternal milik kelas ABC_Tran, hanya 19 di antaranya yang terklasifikasi dengan benar. Empat data lainnya terklasifikasi ke kelas 1-cysPrx_C (bernilai salah) dan dua data lainnya terklasifikasi ke kelas 4HBT (bernilai salah). Hasil pengujian data eksternal untuk orde satu dapat dilihat pada Tabel 12 berikut.
Tabel 12 Pengujian data eksternal untuk orde dua
No 1-cysPrx_C 4HBT ABC_Tran Akurasi (%)
1-cysPrx_C 24 0 1 96.00
4HBT 2 18 5 72.00
ABC_Tran 5 3 17 68.00
Rata-rata 78.66
24
SIMPULAN DAN SARAN
Simpulan
Berdasarkan penelitian yang telah dilakukan, dapat ditarik kesimpulan sebagai berikut.
Model klasifikasi protein family dengan metode rantai markov telah berhasil diaplikasikan.
Berdasarkan hasil pengujian, dapat diketahui bahwa pembagian data latih dan data uji dengan tingkat akurasi paling baik, adalah fold keempat baik untuk orde satu maupun orde dua dan tingkat akurasi orde satu untuk tiap-tiap fold selalu lebih baik jika dibandingkan dengan tingkat akurasi orde dua.
Saran
Penelitian ini memungkinkan untuk dikembangkan menjadi lebih baik lagi. Beberapa saran untuk penelitian selanjutnya adalah.
Pengujian dilakukan dengan melibatkan lebih dari tiga kelas protein serta melibatkan data yang lebih besar untuk tiap-tiap kelas.
25
DAFTAR PUSTAKA
Ching Wai-Ki, Ng M.K. 2006. Markov Chain. Model, Algorithm and Applications. New York (US): Springer Science Business Media, Inc.
Eskin E, Grundy WN, Singer Y. 2000. Protein Family Classification using Sparse Markov Transducers. Columbia (US): American Association for Artificial Intelligence.
Kohavi R. 1995. A study of cross validation and bootstrap for accuracy
estimation and modern selection. Di dalam: Proceeding of the 14th International Joint Conference on Artificial Intelligence. Quebec, Kanada. hlm 1137-1193.
Kunin V, Cases I, Enright A.J, De Lorenzo V, Ouzounis C.A. 2003. Myriads of Protein Families, and Still Counting. Cambridge (GB): The European Bioinformatics Institute.
Mangku IW. 2005. Dasar-dasar Pemodelan Stokastik. Bogor (ID): Institut Pertanian Bogor.
Polanski A, Kimmel M. 2007. Bioinformatics. Berlin (DE): Springer Science. Resch B. [tahun terbit tidak diketahui]. Hidden Markov Models a Tutorial for The
Course Computational Intelligence. Styria (AT): Graz University of Technology.
Ussery D. 1998. Gene expression & regulation. [internet]. [diunduh 2012 Apr 17]. Tersedia pada: http://www.cbs.dtu.dk/staff/dave/DNA_CenDog.html Wu CH, Hongzhan H, Yeh LL, Barker WC. 2002. Protein Family Classification
and Functional Annotation. Washington DC (US): Georgetown University Medical Center and National Biomedical Research Foundation.
26