Analisis Keragaman Genetik dan Pengembangan
Profil Sidik Jari DNA 20 Varietas Cabai Lokal Indonesia
Berdasarkan Marka SSR
(Genetic Diversity Analysis and Development of DNA Fingerprints of
20 Indonesian Local Chili Pepper Varieties Based on SSR Markers)
Rerenstradika Tizar Terryana1*, Nadia Della Savitri Ayu Ningrum2, Kristianto Nugroho1, Darmawan Saptadi2, Helmi Kurniawan3, dan Puji Lestari1,4
1Balai Besar Penelitian dan Pengembangan Bioteknologi dan Sumber Daya Genetik Pertanian, Jl. Tentara Pelajar 3A, Bogor 16111 Indonesia. Telp. (0251) 8337975; Faks. (0251) 8338820; *E-mail: rerenstradika@pertanian.go.id, re2n_terryana@ymail.com
2Laboratorium Pemuliaan Tanaman, Jurusan Budi Daya Pertanian, Fakultas Pertanian, Universitas Brawijaya, Jl. Veteran, Malang 65145 Indonesia 3Balai Penelitian Tanaman Sayuran, Jl. Raya Tangkuban Perahu No. 157, Lembang, Bandung Barat 40391 Indonesia
4Balai Besar Penelitian Tanaman Padi, Jl. Raya 9, Sukamandi, Subang 41256 Indonesia
Diajukan: 15 April 2020; Direvisi: 14 Agustus 2020; Diterima: 24 Agustus 2020
ABSTRACT
Chili pepper is one of the most valuablehorticultural crops, widely cultivated in Indonesia. Analysis of its genetic diversity is needed to develop successful breeding programs of local varieties. Simple sequence repeat (SSR), a robust molecular marker used for genetic diversity analysis in plant species, offers potential, reliable DNA fingerprinting method to assess genetic variation and varietal identification of chili pepper. Fifteen SSR markers were used in this study to analyze the genetic diversity and develop profiling identification of DNA fingerprint of local chili pepper varieties. Twenty local and two improved varieties of three chili pepper species, consisting of 3, 1, and 18 varieties of Capsicum frutescens, C. chinense, and C. annuum, respectively, were assessed for their SSR polymorphism. A total of 87 alleles was obtained from the polymorphism analysis with high alleles variation (2–16 alleles) with average total allele of 5.8 and average polymorphism information content (PIC) of 0.59 (0.34–0.83). Clustering and Principle Coordinate Analyses (PCoA) classified the varieties into two groups with coefficient of similarity of 0.65 indicating their high genetic variability. Most local varieties belonged to the same cluster and separated from the two improved varieties. Based on PIC values and dendrogram with selected markers, five SSR markers, i.e. EPMS441, EPMS331, EPMS335, GPMS194, and CaSSRBio1.1, were identified as SSR marker set for DNA fingerprinting purposes. SSR marker set used in this study was successful in developing the varietal identity of local chili pepper varieties, as indicated by unique code of each variety.
Keywords: Chili pepper, SSR marker, genetic diversity, DNA fingerprinting. ABSTRAK
Cabai merupakan salah satu tanaman hortikultura penting yang banyak dibudidayakan di Indonesia. Analisis keragaman genetik cabai diperlukan untuk keberhasilan program pemuliaan varietas cabai lokal. Simple sequence repeat (SSR) merupakan marka molekuler yang telah terbukti ampuh dan handal untuk analisis keragaman genetik dan pengembangan sidik jari DNA berbagai spesies tanaman termasuk cabai. Sebanyak lima belas marka SSR digunakan pada penelitian ini untuk menganalisis keragaman genetik dan identifikasi profil sidik jari DNA varietas cabai lokal. Dua puluh varietas cabai lokal dan dua varietas unggul baru (VUB), dari tiga spesies cabai yang terdiri atas 3 varietas Capsicum frutescens, 1 varietas C. chinense, dan 18 varietas C. annuum, dianalisis dengan menggunakan marka SSR. Sebanyak 87 alel diperoleh dari analisis polimorfisme marka SSR dengan variasi alel tinggi (2–16 alel) dengan jumlah alel rata-rata 5,8 dan nilai polymorphism information content (PIC) rata-rata 0,59 (0,34–0,83). Analisis klaster dan koordinat utama (Principle Coordinate Analyses/PCoA) yang membagi varietas cabai uji menjadi dua kelompok dengan koefisien kemiripan 0,65 mengindikasikan tingkat keragaman genetik yang tinggi. Sebagian besar varietas lokal mengelompok dalam satu klaster dan terpisah dari klaster dua VUB. Berdasarkan nilai PIC dan dendrogram dengan marka SSR terseleksi, lima marka SSR, yaitu EPMS441, EPMS331, EPMS335, GPMS194, dan CaSSRBio1.1, teridentifikasi sebagai set marka SSR terpilih yang dapat digunakan untuk tujuan sidik jari DNA varietas cabai lokal. Set lima marka SSR pada penelitian ini telah berhasil digunakan untuk pengembangan identitas cabai lokal berupa kode unik untuk setiap varietas cabai lokal.
Kata kunci: Cabai, marka SSR, keragaman genetik, sidik jari DNA.
PENDAHULUAN
Cabai merupakan salah satu komoditas unggul-an hortikultura bernilai ekonomi tinggi dunggul-an sesuai dikembangkan di wilayah tropis seperti Indonesia. Tingginya pemanfaatan cabai sebagai bahan baku pangan, industri, dan obat-obatan menyebabkan tingginya permintaan cabai di Indonesia. Namun, tingginya permintaan tersebut belum diimbangi dengan ketersediaan cabai dalam jumlah cukup di pasaran karena kuantitas produksi cabai cenderung fluktuatif tiap tahunnya yang menyebabkan harganya juga berfluktuasi. Produktivitas cabai nasional dua tahun terakhir rata-rata hanya 7,49 t/ha, masih jauh dari potensi produktivitas 12 t/ha (Ditjen Hortikultura 2019). Salah satu upaya peningkatan produktivitas cabai yaitu melalui perakitan varietas unggul baru (VUB) berdaya hasil tinggi dan tahan/toleran terhadap cekaman biotik ataupun abiotik (Deviona et al. 2013).
Kegiatan perakitan VUB cabai masih terus di-lakukan dan memerlukan ketersediaan sumber daya genetik (SDG) cabai yang representatif untuk men-dukung program pemuliaan tersebut. SDG cabai dapat diperoleh dari varietas lokal, varietas unggul nasional, galur hasil pemuliaan, genotipe introduksi, dan kerabat liar (Wartono et al. 2019). Jumlah VUB cabai yang dilepas Kementerian Pertanian men-duduki peringkat tertinggi dibanding dengan varietas tanaman hortikultura lainnya, namun VUB cabai yang beredar dan dimanfaatkan masyarakat kuantitasnya masih sangat rendah. Hal ini disebabkan sebagian besar VUB tersebut merupakan hasil introduksi dari luar negeri yang daya adaptasinya relatif rendah di Indonesia (Syukur dan Yunianti 2013). Di pihak lain, varietas lokal telah dibudidayakan sejak lama dan se-cara turun-temurun sehingga cenderung mampu ber-adaptasi pada kondisi agroekosistem dan cekaman biotik dan abiotik di wilayah setempat. Oleh karena itu, penggunaan varietas lokal dalam program pemuliaan tanaman cabai sangat dianjurkan untuk memperluas latar belakang genetik VUB cabai yang dihasilkan (Sitaresmi et al. 2013).
Identifikasi genetik suatu varietas sangat penting dalam perlindungan varietas tanaman. Perlindungan atas varietas dapat diberikan jika identitas genetiknya telah terbukti berbeda dengan varietas yang telah ada sebelumnya. Adanya keterbatasan teknis menyebab-kan identifikasi varietas cabai di Indonesia selama ini lebih banyak didasarkan pada karakter morfologi yang membutuhkan observasi yang intensif dan se-ringkali sulit untuk membedakan varietas dengan tingkat kekerabatan dekat (Chaesaria et al. 2018; Saptadi et al. 2018). Seringkali keputusan yang
di-hasilkan atas identifikasi karakter suatu varietas lebih bersifat subjektif akibat kurangnya metode identifikasi yang lebih efektif dan objektif. Oleh karena itu, perlu penetapan metode identifikasi (sidik jari) varietas yang stabil, handal, objektif, dan mudah diakses untuk dapat melindungi hak kekayaan intelektual varietas tersebut secara efektif (Zhang et al. 2014).
Pemanfaatan teknologi marka molekuler meng-hasilkan sidik jari DNA yang efektif dan efisien dalam mengidentifikasi varietas pada level DNA karena lebih akurat dan tidak dipengaruhi oleh faktor lingkungan (Risliawati et al. 2015; Gao et al. 2016). Marka molekuler juga telah banyak digunakan pada analisis diversitas genetik dengan presisi tinggi dan untuk memprediksi latar belakang genetik varietas (Ribeiro et al. 2013; Lestari et al. 2016; Terryana et al. 2018). Di samping itu, marka molekuler dapat digunakan sebagai pelengkap pada uji kebaruan, keunikan, ke-seragaman, dan kestabilan (BUSS) melalui per-bandingan profil sidik jari DNA.
Salah satu teknologi marka molekuler berbasis
Polymerase Chain Reaction (PCR) yang cukup
populer pada analisis diversitas genetik dan sidik jari DNA berbagai spesies tanaman adalah marka simple
sequence repeat (SSR). SSR merupakan sekuen
pendek (2–4 nukleotida) berulang yang keberadaan-nya melimpah dalam genom organisme eukariotik (Vieira et al. 2016), bersifat kodominan, dan mudah dalam aplikasinya (Surapaneni et al. 2013). Marka SSR telah banyak digunakan pada analisis sidik jari DNA berbagai spesies tanaman, seperti kedelai (Santoso et al. 2006; Risliawati et al. 2015; Lestari et al. 2016; Tasma et al. 2018), kentang (Duan et al. 2019), kelapa sawit (Tasma dan Arumsari 2013; Tasma et al. 2013), dan ubi jalar (Meng et al. 2018). Pada cabai, marka SSR telah diaplikasikan untuk ber-bagai tujuan, di antaranya analisis keragaman genetik dan identifikasi galur cabai hasil persilangan (Dhaliwal et al. 2013), identifikasi plasma nutfah cabai koleksi French National Institute for Agricultural
Research/INRA (Nicolaï et al. 2013), karakterisasi
plasma nutfah cabai asal Meksiko (Toledo-Aguilar et al. 2016), analisis diversitas genetik cabai Indonesia (Wartono et al. 2019), dan identifikasi varietas pada uji BUSS (Kwon et al. 2005).
Namun, hingga saat ini teknologi sidik jari DNA belum banyak dimanfaatkan dalam peningkatan manfaat SDG cabai di Indonesia. Teknologi sidik jari DNA dapat digunakan untuk mengetahui informasi jarak genetik dan identifikasi varietas. Pemanfaatan teknologi sidik jari DNA diharapkan dapat memberi-kan perlindungan pada varietas cabai lokal nasional, membedakan antarvarietas dengan nama sama tapi
berasal dari daerah yang berbeda, dan mendeteksi adanya duplikasi dalam pengelolaan SDG cabai (Lestari et al. 2016). Teknologi sidik jari DNA ini akan sangat bermanfaat bagi Pusat Perlindungan Varietas Tanaman dan Perizinan Pertanian (PPVTPP) dalam penetapan keunikan varietas serta menjadi pe-lengkap informasi fenotipe varietas yang telah ter-daftar sebagai salah satu upaya perlindungan varietas tanaman.
Tujuan penelitian ini ialah menganalisis ke-ragaman genetik dan identifikasi profil sidik jari DNA varietas cabai lokal dengan menggunakan marka SSR. Mengingat marka SSR yang menghasilkan profil sidik jari DNA pada varietas cabai lokal belum ada, set markanya perlu dikembangkan untuk dapat mengidentifikasi varietas secara lebih efisien. Metode sidik jari DNA memberi informasi awal keragaman genetik untuk membantu program pemuliaan, se-dangkan informasi profil sidik jari DNA dapat diman-faatkan sebagai dasar ilmiah untuk mengidentifikasi genotipe cabai berbasis molekuler dan upaya perlin-dungan varietas cabai lokal di Indonesia.
BAHAN DAN METODE Materi Genetik dan Marka SSR
Materi genetik yang digunakan pada penelitian ini terdiri atas 20 varietas cabai lokal dan 2 VUB hasil pemuliaan sebagai pembanding. Dasar pemilihan materi genetik adalah sebaran spesies untuk men-dukung kegiatan pengelolaan SDG cabai di Balai Penelitian Tanaman Sayuran (Balitsa), Lembang,
Bandung Barat, Jawa Barat pada tahun 2019. Varietas uji terbagi ke dalam tiga kelompok spesies yang berbeda, yaitu Capsicum frutescens (1 varietas lokal dan 2 VUB), C. chinense (1 varietas lokal), dan C.
annuum (18 varietas lokal) (Tabel 1). Varietas lokal
yang digunakan merupakan koleksi Balitsa hasil eksplorasi dari beberapa provinsi di Indonesia (Jawa Barat, Jawa Tengah, dan Nusa Tenggara Barat).
Sebanyak lima belas marka SSR yang digunakan pada penelitian ini terdiri atas sembilan marka SSR hasil eksplorasi pada studi sebelumnya (Lee et al. 2004; Minamiyama et al. 2007; Nagy et al. 2007) dan enam marka SSR yang didesain berdasarkan data sekuen genom cabai yang informasinya dapat diakses pada situs PGPI (2015). Sekuen primer tiap-tiap marka SSR disajikan pada Tabel 2.
Ekstraksi, Uji Kualitatif, dan Uji Kuantitatif DNA Genomik
DNA genomik diekstraksi dari daun muda dengan prosedur ekstraksi DNA yang mengacu pada metode Doyle dan Doyle (1990) yang dimodifikasi dengan penambahan polyvinyl pyrrolidone (PVP) 2% (w/v). Sebanyak 0,5 g potongan daun muda cabai dimasukkan ke dalam tabung mikro 2 ml kemudian digerus dengan bantuan blue pestle hingga hancur, diikuti dengan penambahan 750 µl bufer ekstraksi yang mengandung Tris-HCl 100 mM (pH 8,0), NaCl 1,4 M, EDTA 20 mM (pH 8,0), cetyltrimethylammonium
bromide (CTAB) 2% (w/v), PVP 2% (w/v), dan
natrium disulfit 0,38% (w/v). Pelet DNA yang diper-oleh kemudian dicuci dengan larutan etanol 70% dan Tabel 1. Varietas cabai lokal dan varietas unggul baru (VUB) yang digunakan pada penelitian ini.
Varietas Spesies Kelompok Asal/silsilah Bentuk buah
Prima Agrihorti Capsicum frutescens VUB Seleksi dari populasi R29 Rawit Rabani Agrihorti C. frutescens VUB Seleksi dari populasi R01 Rawit
Gendot C. chinense Lokal Jawa Tengah Rawit
Saha Mpida C. frutescens Lokal Nusa Tenggara Barat Rawit
LV-8030 C. annuum Lokal Jawa Barat Besar
LV-8031 C. annuum Lokal Jawa Barat Besar
LV-8032 C. annuum Lokal Jawa Barat Keriting
LV-8038 C. annuum Lokal Jawa Tengah Besar
LV-8041 C. annuum Lokal Jawa Barat Keriting
LV-8043 C. annuum Lokal Jawa Barat Keriting
LV-8044 C. annuum Lokal Jawa Barat Keriting
LV-8045 C. annuum Lokal Jawa Barat Besar pendek
LV-1925 C. annuum Lokal Jawa Barat Besar
LV-5168 C. annuum Lokal Jawa Barat Keriting
LV-1422 C. annuum Lokal Jawa Barat Besar
LV-8051 C. annuum Lokal Jawa Barat Keriting
LV-5685 C. annuum Lokal Jawa Tengah Keriting
LV-8052 C. annuum Lokal Jawa Barat Besar
LV-5684 C. annuum Lokal Jawa Barat Keriting
LV-8060 C. annuum Lokal Jawa Barat Besar
LV-49 C. annuum Lokal Jawa Tengah Besar
dikeringkan di dalam SpeedVacTM Concentrator (Thermo Fisher Scientific, AS) pada tingkat pengeringan medium. Pelet DNA dilarutkan dalam 100 µl larutan TE (Tris 10 mM [pH 8,0] dan EDTA 1 mM) dan 2 µl RNase (10 mg/ml). Uji kuantitatif larutan stok DNA dilakukan dengan NanoDropTM
Spectrophotometer V1.0 (Thermo Fisher Scientific,
AS), sedangkan uji kualitatif dilakukan dengan teknik elektroforesis pada gel agarosa 1% di dalam tangki berisi bufer Tris-acetate-EDTA (TAE) 1× pada tegang-an 90 volt selama 30 menit. Hasil elektroforesis kemudian divisualisasi dengan UV Transiluminator (UVP, UK).
Analisis PCR dan Elektroforesis
Analisis PCR untuk DNA setiap genotipe cabai uji dilakukan dengan menggunakan lima belas marka SSR (Tabel 2). DNA setiap genotipe cabai diamplifi-kasi pada volume reaksi 10 μl yang mengandung
template DNA 10 ng sebanyak 1 μl, Kapa2G Fast
Ready Mix (KAPA Biosystem, AS) sebanyak 5 μl,
primer forward dan reverse dengan konsentrasi 10 μM masing-masing sebanyak 0,5 μl, dan 3 μl ddH2O steril. Reaksi PCR dilakukan pada mesin PCR T1
Thermocycler (Biometra, Jerman) dengan profil PCR
sebagai berikut: denaturasi awal dilakukan pada 95°C selama 5 menit, diikuti oleh sebanyak 35 siklus proses denaturasi pada 94°C selama 30 detik, tahap penempelan primer pada 55°C selama 1 menit, dan tahap perpanjangan primer pada 72°C selama 1 menit. Reaksi PCR diakhiri dengan siklus per-panjangan primer akhir pada 60°C selama 15 menit serta inkubasi pada 10°C selama 2 menit. Hasil PCR selanjutnya dielektroforesis pada gel poliakrilamida 6% untuk analisis lebih lanjut.
Analisis Data
Data dianalisis berdasarkan metode skoring pada pita DNA yang muncul dari hasil elektroforesis menggunakan gel poliakrilamida 6%. Pita-pita DNA yang tampak dengan laju migrasi yang sama di-asumsikan sebagai lokus yang homolog. Pada laju migrasi yang sama, pita DNA yang terlihat diberi skor 1, pita DNA yang tidak terlihat diberi skor 0, sedang-kan sampel yang tidak menghasilsedang-kan pita DNA diberi skor 9 dan dianggap sebagai data hilang. Jadi, hasil akhir skoring pita DNA berupa data biner. Skoring posisi pita DNA dilakukan dengan bantuan perangkat lunak GelAnalyzer. Data hasil skoring selanjutnya dianalisis dengan menggunakan Sequential Tabel 2. Marka SSR yang digunakan pada penelitian ini.
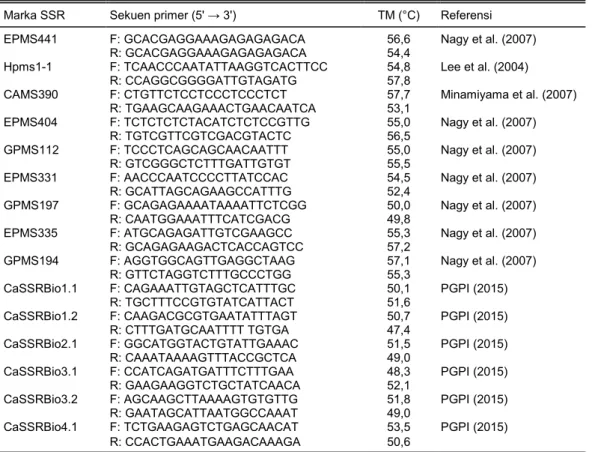
Marka SSR Sekuen primer (5' → 3') TM (°C) Referensi
EPMS441 F: GCACGAGGAAAGAGAGAGACA 56,6 Nagy et al. (2007) R: GCACGAGGAAAGAGAGAGACA 54,4
Hpms1-1 F: TCAACCCAATATTAAGGTCACTTCC 54,8 Lee et al. (2004) R: CCAGGCGGGGATTGTAGATG 57,8
CAMS390 F: CTGTTCTCCTCCCTCCCTCT 57,7 Minamiyama et al. (2007) R: TGAAGCAAGAAACTGAACAATCA 53,1
EPMS404 F: TCTCTCTCTACATCTCTCCGTTG 55,0 Nagy et al. (2007) R: TGTCGTTCGTCGACGTACTC 56,5
GPMS112 F: TCCCTCAGCAGCAACAATTT 55,0 Nagy et al. (2007) R: GTCGGGCTCTTTGATTGTGT 55,5
EPMS331 F: AACCCAATCCCCTTATCCAC 54,5 Nagy et al. (2007) R: GCATTAGCAGAAGCCATTTG 52,4
GPMS197 F: GCAGAGAAAATAAAATTCTCGG 50,0 Nagy et al. (2007) R: CAATGGAAATTTCATCGACG 49,8
EPMS335 F: ATGCAGAGATTGTCGAAGCC 55,3 Nagy et al. (2007) R: GCAGAGAAGACTCACCAGTCC 57,2
GPMS194 F: AGGTGGCAGTTGAGGCTAAG 57,1 Nagy et al. (2007) R: GTTCTAGGTCTTTGCCCTGG 55,3
CaSSRBio1.1 F: CAGAAATTGTAGCTCATTTGC 50,1 PGPI (2015) R: TGCTTTCCGTGTATCATTACT 51,6
CaSSRBio1.2 F: CAAGACGCGTGAATATTTAGT 50,7 PGPI (2015) R: CTTTGATGCAATTTT TGTGA 47,4
CaSSRBio2.1 F: GGCATGGTACTGTATTGAAAC 51,5 PGPI (2015) R: CAAATAAAAGTTTACCGCTCA 49,0
CaSSRBio3.1 F: CCATCAGATGATTTCTTTGAA 48,3 PGPI (2015) R: GAAGAAGGTCTGCTATCAACA 52,1
CaSSRBio3.2 F: AGCAAGCTTAAAAGTGTGTTG 51,8 PGPI (2015) R: GAATAGCATTAATGGCCAAAT 49,0
CaSSRBio4.1 F: TCTGAAGAGTCTGAGCAACAT 53,5 PGPI (2015) R: CCACTGAAATGAAGACAAAGA 50,6
Agglomerative Hierarchical and Nested-Unweighted
Pair Group Method with Arithmetic (SAHN-UPGMA)
pada perangkat lunak NTSYS-pc versi 2.1 (Rohlf 2000). Hasil analisis disajikan dalam bentuk dendrogram. Data hasil skoring juga dianalisis dengan mengguna-kan perangkat lunak PowerMarker V3.25 (Liu dan Muse 2005) untuk mengetahui statistik nilai frekuensi alel utama, diversitas genetik, heterozigositas, dan nilai polymorphism information content (PIC) yang dihasilkan oleh marka SSR pada penelitian ini. Data hasil skoring juga dianalisis dengan menggunakan perangkat lunak XLSTAT versi 2018 untuk analisis koordinat utama (Principal Coordinate Analysis/ PCoA). Analisis PCoA menggambarkan posisi relatif tiap-tiap individu varietas cabai. Data kodominan dihitung untuk mendapatkan nilai jarak genetik antarindividu.
Pada analisis data untuk profil sidik jari DNA terdapat beberapa tahapan dalam merancang set marka SSR untuk identifikasi varietas, meliputi seleksi kandidat marka SSR, perancangan set marka SSR, dan penyusunan identitas spesifik (ID) varietas. Pada tahap seleksi kandidat marka SSR, marka SSR diseleksi berdasarkan nilai PIC (Botstein et al. 1980). Hanya marka SSR yang masuk dalam kriteria infor-matif (nilai PIC > 0,5) berdasarkan Botstein et al. (1980) yang diseleksi. Selanjutnya, dilakukan analisis filogeni dengan menggunakan marka SSR terseleksi tersebut dengan bantuan perangkat lunak MEGA5.0. Pada analisis filogeni, diharapkan marka SSR dalam jumlah seefektif mungkin dapat membuat
keseluruh-an sampel terpisah dalam subklaster. Mengingat profil sidik jari DNA merupakan identitas yang diformulasi-kan sebagai kode spesifik dalam bentuk numerik, rancangan profil sidik jari DNA varietas cabai yang digunakan pada penelitian ini dilakukan berdasarkan hasil deteksi ukuran alel asli dengan bantuan pe-rangkat lunak GelAnalyzer. Selanjutnya, pembentuk-an kelas dibuat dengpembentuk-an kode khusus dua digit me-wakili tiap selang ukuran alel utama dari tiap-tiap lokus tertentu yang dihitung dengan formula selisih ukuran alel maksimum dengan ukuran alel minimum, dibagi dengan batas rentang sensitivitas perbedaan ukuran alel hasil deteksi dengan perangkat lunak
GelAnalyzer, sebanyak lima angka (Lazar et al. 2010).
HASIL DAN PEMBAHASAN Karakteristik Marka SSR
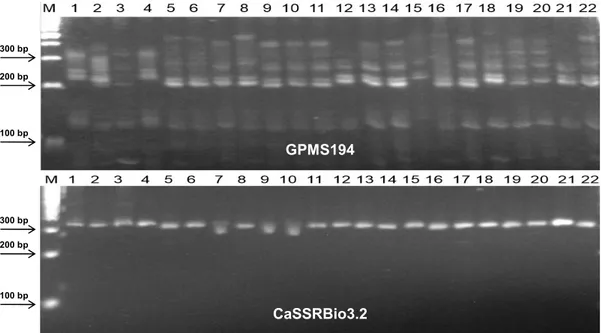
Seluruh marka SSR dapat menunjukkan polimor-fisme pada seluruh varietas cabai yang digunakan (Gambar 1). Sebanyak 87 alel terdeteksi dengan kisaran 2–16 alel per lokus berdasarkan hasil analisis menggunakan lima belas marka SSR. Ringkasan statistik analisis polimorfisme yang dihasilkan oleh marka SSR disajikan pada Tabel 3. Jumlah alel yang terdeteksi pada penelitian ini lebih tinggi dibanding dengan penelitian Carvalho et al. (2015) yang men-deteksi 55 alel berdasarkan 19 marka SSR pada 12 aksesi cabai. Namun demikian, jumlah alel yang berhasil dideteksi pada penelitian ini lebih rendah di-banding dengan hasil penelitian Yumnam et al. (2012)
Gambar 1. Contoh pola pita dua marka SSR (GPMS194 dan CaSSRBio3.2) pada produk PCR DNA genomik 20 varietas cabai lokal dan 2 VUB pembanding dengan elektroforesis pada gel poliakrilamida 6%. M = 100 bp ladder DNA, 1–22 = varietas cabai dengan urutan sesuai urutan varietas uji yang tertera pada Tabel 1.
CaSSRBio3.2 GPMS194 300 bp 200 bp 100 bp 300 bp 200 bp 100 bp CaSSRBio3.2
yang mendeteksi 134 alel berdasarkan 25 marka SSR pada 53 aksesi cabai dan Nicolaï et al. (2013) yang berhasil mendeteksi 510 alel berdasarkan 28 marka SSR pada 1.352 aksesi cabai. Perbedaan jumlah alel yang diperoleh diduga berkaitan dengan perbedaan materi genetik, termasuk jumlah dan latar belakang genetik, jumlah dan jenis marka SSR apakah marka fungsional atau marka universal (Cintamulya 2011; Wartono et al. 2019), serta resolusi separasi DNA. Dhaliwal et al. (2014) melaporkan hanya dapat men-deteksi tidak lebih dari lima alel per lokus setelah menggunakan metode separasi pada gel agarosa.
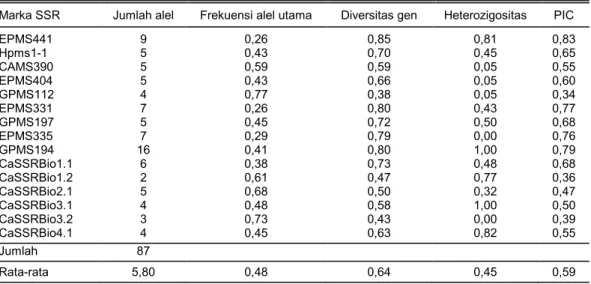
Frekuensi alel utama rata-rata yang diperoleh sebesar 48% dengan nilai tertinggi sebesar 73% (CaSSRBio3.2) dan nilai terendah 26% (EPMS441 dan EPMS331). Sementara itu, Oh et al. (2012) melapor-kan frekuensi alel utama yang lebih tinggi yaitu 69% dengan kisaran 41–96% berdasarkan hasil analisis 61 aksesi cabai menggunakan 22 marka SSR. Dari lima belas marka SSR yang diuji, marka EPMS335 dan CaSSRBio3.2 tidak dapat menghasilkan alel heterozigot karena hanya dapat mendeteksi satu alel antarindividu dalam suatu populasi. Sementara, tiga belas marka SSR lainnya mampu mendiskriminasi alel heterozigot pada seluruh varietas yang digunakan dengan nilai heterozigositas berkisar antara 0,05 (CAMS390, EPMS404, dan GPMS112) dan 1,00 (GPMS194 dan CaSSRBio3.1). Nilai heterozigositas menunjukkan peluang suatu marka yang digunakan untuk membedakan dua alel acak yang dipilih dalam suatu populasi, apabila nilai heterozigositas makin tinggi maka sebagian besar sampel yang diuji bersifat heterozigot sehingga menunjukkan keragaman dalam suatu populasi (Chaerani et al. 2011). Semakin tinggi tingkat heterozigositas, semakin tinggi kemampuan
suatu marka dalam membedakan antargenotipe secara genetik. Nilai heterozigositas yang dihasilkan dari pengujian genotipe dengan menggunakan marka SSR dapat mendeteksi tingginya perbedaan genotipe dalam suatu populasi dengan karakteristik morfologi yang hampir sama (Maranho et al. 2014).
Diversitas gen menunjukkan tingkat keragaman genetik pada varietas yang diuji. Lestari et al. (2016) mengemukakan bahwa tingkat keragaman genetik SDG yang diuji dapat diketahui dari nilai diversitas gen. Nilai diversitas gen pada penelitian ini berkisar antara 0,38 (GPMS112) dan 0,85 (EPMS441) dengan rata-rata 0,64, artinya antarvarietas terdapat keragam-an genetik ykeragam-ang tinggi. Bassil et al. (2005) melaporkkeragam-an keragaman genetik dapat diketahui dari dua indika-tor, yaitu diversitas gen yang menunjukkan peluang adanya perbedaan pada dua alel yang dipilih secara acak pada lokus yang berbeda dan nilai PIC yang menunjukkan ukuran dari peluang dua varietas yang dipilih secara acak.
Nilai PIC didefinisikan pula sebagai suatu nilai yang mencerminkan tingkat polimorfisme marka yang digunakan. Nilai PIC pada penelitian ini berkisar antara 0,34 (GPMS112) dan 0,83 (EPMS441) dengan rata-rata 0,59. Nilai PIC rata-rata pada penelitian ini lebih rendah dibanding dengan hasil penelitian Zhang et al. (2016) dengan nilai PIC rata-rata sebesar 0,60 dari 28 marka SSR yang digunakan, namun lebih tinggi daripada hasil penelitian Sharmin et al. (2018) dengan nilai PIC rata-rata hanya sebesar 0,37 dari lima marka SSR yang digunakan. Botstein et al. (1980) mengelompokkan nilai PIC menjadi tiga kategori, yaitu kategori sangat informatif (PIC > 0,5), sedang (PIC 0,25 < PIC < 0,5), dan kurang informatif (PIC < Tabel 3. Karakteristik 15 marka SSR hasil analisis 20 varietas cabai lokal dan 2 VUB pembanding.
Marka SSR Jumlah alel Frekuensi alel utama Diversitas gen Heterozigositas PIC
EPMS441 9 0,26 0,85 0,81 0,83 Hpms1-1 5 0,43 0,70 0,45 0,65 CAMS390 5 0,59 0,59 0,05 0,55 EPMS404 5 0,43 0,66 0,05 0,60 GPMS112 4 0,77 0,38 0,05 0,34 EPMS331 7 0,26 0,80 0,43 0,77 GPMS197 5 0,45 0,72 0,50 0,68 EPMS335 7 0,29 0,79 0,00 0,76 GPMS194 16 0,41 0,80 1,00 0,79 CaSSRBio1.1 6 0,38 0,73 0,48 0,68 CaSSRBio1.2 2 0,61 0,47 0,77 0,36 CaSSRBio2.1 5 0,68 0,50 0,32 0,47 CaSSRBio3.1 4 0,48 0,58 1,00 0,50 CaSSRBio3.2 3 0,73 0,43 0,00 0,39 CaSSRBio4.1 4 0,45 0,63 0,82 0,55 Jumlah 87 Rata-rata 5,80 0,48 0,64 0,45 0,59
0,25). Dari lima belas marka SSR yang digunakan pada penelitian ini, empat marka SSR di antaranya termasuk dalam kategori sedang dan sebelas marka SSR sangat informatif yang bermanfaat untuk mem-bedakan varietas cabai ke depannya.
Analisis Filogenetik
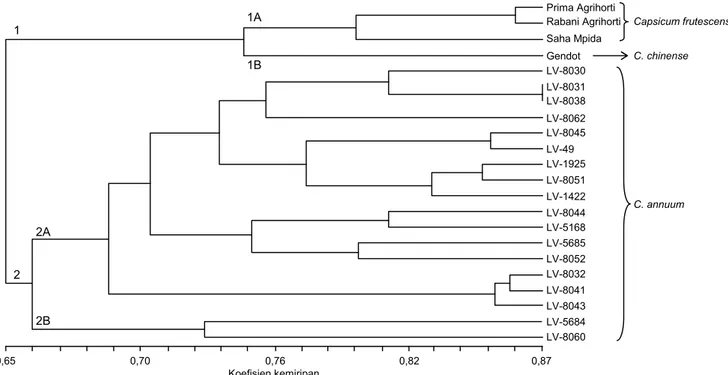
Analisis filogenetik dilakukan untuk mengidenti-fikasi jarak genetik antarvarietas (Utami et al. 2011). Analisis filogenetik 20 varietas cabai lokal Indonesia dan 2 VUB yang berbeda spesiesnya berdasarkan 15 marka SSR dilakukan berdasarkan hasil observasi terhadap keseluruhan frekuensi alel yang muncul dan hasil dendrogram pada SAHN-UPGMA. Pada tingkat kesamaan genetik 65% terbentuk dua klaster utama (Gambar 2). Klaster pertama terbagi lagi menjadi dua subklaster dengan subklaster pertama terdiri atas dua VUB (Prima Agrihorti dan Rabani Agrihorti) dan satu varietas lokal (Saha Mpida) yang seluruhnya merupa-kan spesies C. frutescens, sedangkan subklaster kedua hanya terdiri atas satu varietas lokal (Gendot) yang merupakan spesies C. chinense. Klaster kedua terdiri atas 18 varietas lokal (8030, 8031, LV-8032, LV-8038, LV-8041, LV-8043, LV-8044, LV-8045, LV-1925, LV-5168, LV-1422, LV-8051, LV-5685, LV-8052, LV-5684, LV-8060, LV-49, dan LV-8062) yang seluruh-nya merupakan spesies C. annuum.
Hasil analisis filogenetik ini menunjukkan bahwa lima belas marka SSR yang digunakan dapat
menge-lompokkan varietas cabai berdasarkan status varie-tas, yaitu VUB dan varietas lokal dalam spesies yang sama, serta berdasarkan spesiesnya, yaitu C.
chinense, C. frutescens,dan C. annuum. Penelitian ini
memberikan hasil yang serupa dengan penelitian Cheng et al. (2016) yang dapat mengelompokkan 21 aksesi cabai dengan 65 marka SSR polimorfis ber-dasarkan spesiesnya, yaitu C. baccatum, C. chinense,
C. frutescens, dan C. annuum. Secara umum, marka
SSR yang digunakan pada penelitian ini cukup aplikatif dan efektif dalam membedakan varietas cabai berdasarkan spesiesnya.
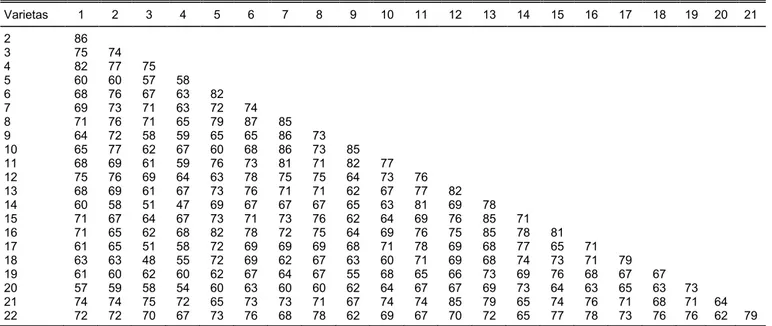
Berdasarkan hasil analisis keragaman genetik (Tabel 4), terlihat bahwa varietas lokal LV-8031 asal Jawa Barat dan varietas lokal LV-8038 asal Jawa Tengah memiliki tingkat kesamaan genetik sebesar 87%. Tingginya nilai kesamaan genetik tersebut mengindikasikan adanya kemiripan antarvarietas sehingga terdapat hubungan kekerabatan yang dekat (Hadiati 2003; Mustofa et al. 2013). Sementara, varietas lokal Saha Mpida (C. frutescens) asal NTB dan varietas lokal LV-5168 (C. annuum) asal Jawa Barat diduga memiliki tingkat kekerabatan terjauh dengan nilai kesamaan genetik sebesar 47% sehingga kedua varietas lokal tersebut sangat potensial untuk dijadikan calon tetua persilangan. Persilangan genotipe cabai dengan jarak genetik jauh, bahkan antarspesies, berpotensi untuk mendapatkan progeni dengan keragaan fenotipe superior. Sebagai contoh,
Gambar 2. Dendrogram 20 varietas cabai lokal dan 2 VUB berdasarkan hasil analisis menggunakan 15 marka SSR yang dikonstruksi menggunakan program SAHN-UPGMA dengan perangkat lunak NTSYS-pc.
0,65 1A 1B 1 2A 2 2B 0,70 0,76 0,82 0,87 Koefisien kemiripan Prima Agrihorti Rabani Agrihorti LV-8041 Saha Mpida Gendot LV-8030 LV-8031 LV-8038 LV-8062 LV-8045 LV-49 LV-1925 LV-8051 LV-1422 LV-8044 LV-5168 LV-5685 LV-8052 LV-8032 C. annuum LV-8043 LV-5684 LV-8060 Capsicum frutescens C. chinense
persilangan interspesies cabai (C. frutescens dengan
C. annuum) telah dilakukan untuk menghasilkan
cabai hias dengan karakter unik (Monteiro et al. 2011; Kurniawan et al. 2015; Wei et al. 2019). Persilangan antarcalon tetua dengan jarak genetik jauh akan memaksimalkan kesempatan dalam memperoleh segregan transgresif di antara progeni persilangan (Rubiyo 2013; Sutoro et al. 2017).
Analisis PCoA sangat berguna untuk mengana-lisis keragaman genetik antarvarietas tanaman (Shankar et al. 2009) dan penting untuk mengetahui informasi kedekatan antarindividu berdasarkan ke-miripan karakter melalui penyederhanaan dimensi (Shankar et al. 2009; Kristamtini et al. 2014). Analisis PCoA telah digunakan sebelumnya oleh Oh et al.
(2012), Rai et al. (2013), dan Terryana et al. (2018) untuk mengonfirmasi hasil analisis filogenetik dalam studi keragaman genetik cabai berdasarkan marka SSR. Diagram dua dimensi menunjukkan bahwa koordinat utama pertama dan kedua menjelaskan ke-ragaman genetik total berdasarkan marka SSR ber-turut-turut sebesar 21,15% dan 14,10%. Dengan demi-kian, kedua komponen tersebut menjelaskan kera-gaman genetik seluruh varietas cabai yang dianalisis pada penelitian ini sebesar 35,25% (Gambar 3).
Hasil analisis PCoA menunjukkan kesesuaian dengan hasil analisis filogenetik, varietas cabai yang digunakan mengelompok berdasarkan spesiesnya. Posisi koordinat 20 varietas cabai lokal dan 2 VUB pembanding yang dianalisis pada ruang dua dimensi Tabel 4. Matriks persentase kesamaan genetik 20 varietas cabai lokal dan 2 VUB hasil analisis menggunakan 15 marka SSR.
Varietas 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 2 86 3 75 74 4 82 77 75 5 60 60 57 58 6 68 76 67 63 82 7 69 73 71 63 72 74 8 71 76 71 65 79 87 85 9 64 72 58 59 65 65 86 73 10 65 77 62 67 60 68 86 73 85 11 68 69 61 59 76 73 81 71 82 77 12 75 76 69 64 63 78 75 75 64 73 76 13 68 69 61 67 73 76 71 71 62 67 77 82 14 60 58 51 47 69 67 67 67 65 63 81 69 78 15 71 67 64 67 73 71 73 76 62 64 69 76 85 71 16 71 65 62 68 82 78 72 75 64 69 76 75 85 78 81 17 61 65 51 58 72 69 69 69 68 71 78 69 68 77 65 71 18 63 63 48 55 72 69 62 67 63 60 71 69 68 74 73 71 79 19 61 60 62 60 62 67 64 67 55 68 65 66 73 69 76 68 67 67 20 57 59 58 54 60 63 60 60 62 64 67 67 69 73 64 63 65 63 73 21 74 74 75 72 65 73 73 71 67 74 74 85 79 65 74 76 71 68 71 64 22 72 72 70 67 73 76 68 78 62 69 67 70 72 65 77 78 73 76 76 62 79 1 = Prima Agrihorti, 2 = Rabani Agrihorti, 3 = Gendot, 4 = Saha Mpida, 5 = LV-8030, 6 = LV-8031, 7 = LV-8032, 8 = LV-8038, 9 = LV-8041, 10 = 8043, 11 = 8044, 12 = 8045, 13 = 1925, 14 = 5168, 15 = 1422, 16 = 8051, 17 = 5685, 18 = 8052, 19 = LV-5684, 20 = LV-8060, 21 = LV-49, 22 = LV-8062.
Gambar 3. Analisis koordinat utama (Principle Coordinate Analyses/PCoA) 20 varietas cabai lokal dan 2 VUB pembanding berdasarkan pola pita DNA dengan 15 marka SSR.
8 6 4 2 0 -2 -4 -6 -4 -2 0 0 2 4 6 Koordinat utama 1 (21,15%) K o o rd in a t u ta m a 2 ( 1 4 ,1 0 % ) C. chinense C. annuum Capsicum frutescens
hasil analisis PCoA tersebut telah memberikan peluang untuk memperketat tahap seleksi dalam program pemuliaan. Varietas dengan posisi koordinat yang berdekatan hendaknya dihindari untuk dijadikan tetua persilangan dan sebaiknya pemulia mencari genotipe cabai dengan koordinat yang berjauhan agar diperoleh segregasi progeni yang prospektif pada lingkungan tertentu (Salimi et al. 2013; Kumar et al. 2015).
Profil Sidik Jari DNA Varietas Cabai Lokal Uji Analisis sidik jari DNA pada penelitian ini selain untuk estimasi keragaman genetik juga bertujuan membedakan seluruh varietas uji, baik menurut status varietas, spesies, maupun karakter penciri spesifik. Adanya perbedaan pada tiap-tiap varietas dapat menjadi suatu penciri atau sidik jari yang membuktikan bahwa tidak ada satupun varietas yang memiliki kemiripan secara genetik. Hasil analisis sidik jari DNA nantinya akan dapat dimanfaatkan pada ke-giatan identifikasi varietas khususnya pada penyusun-an identitas, pelengkap dokumen pendaftarpenyusun-an varie-tas, serta perlindungan varietas sebagai salah satu bentuk kekayaan intelektual (Risliawati et al. 2015).
Pembuatan profil sidik jari DNA terdiri atas beberapa tahapan, yaitu seleksi kandidat marka, perancangan set marka, dan penyusunan ID varietas
dalam bentuk kode numerik. Berdasarkan nilai PIC, indeks keragaman genetik, serta estimasi jarak genetik, pada awalnya dipilih sebelas marka SSR dengan nilai PIC yang termasuk dalam kategori sangat informatif berdasarkan Botstein et al. (1980) dari lima belas marka SSR sebagai set marka dasar. Kesebelas marka SSR tersebut adalah CAMS390, CaSSRBio1.1, CaSSRBio3.1, CaSSRBio4.1, EPMS331, EPMS335, EPMS404, EPMS441, GPMS194, GPMS197, dan Hpms1-1. Risliawati et al. (2015), Lestari et al. (2016), dan Reflinur dan Lestari (2016) mengemuka-kan bahwa marka SSR dengan nilai PIC tinggi dan masuk dalam kategori informatif dapat dijadikan set marka dalam identifikasi varietas.
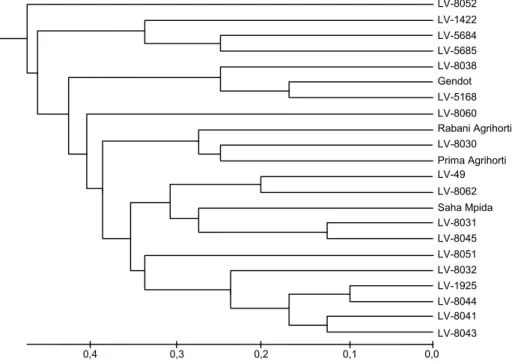
Set marka SSR bersifat efektif dalam identifikasi varietas jika mampu menghasilkan profil sidik jari DNA yang spesifik pada suatu varietas dan dapat menjadi pembeda antarvarietas (Lestari et al. 2016). Adapun jumlah marka SSR dalam formulasi identitas dan proteksi varietas seminimal mungkin namun efisien (Ritschel et al. 2004). Pada penelitian ini, set marka dirancang berdasarkan ukuran alel asli yang secara kuantitatif diobservasi dengan bantuan pe-rangkat lunak GelAnalyzer. Berdasarkan analisis filogenetik dengan lima marka terpilih dengan nilai PIC tertinggi, yaitu EPMS441, EPMS331, GPMS335, GPMS194, dan CaSSRBio1.1, seluruh varietas cabai dapat terpisah dalam subklaster (Gambar 4). Dengan
Gambar 4. Dendrogram 20 varietas cabai lokal dan 2 VUB pembanding hasil analisis berdasarkan set lima marka SSR. Data dianalisis dengan menggunakan program SAHN-UPGMA dan perangkat lunak
MEGA5.0. Varietas lokal pada dendrogram ditandai dengan dua huruf LV diikuti dengan nomor setiap varietas cabai lokal. Dua VUB pembanding yang digunakan, yaitu Rabani Agrihorti dan Prima Agrihorti. 0,0 0,1 0,2 0,3 0,4 Prima Agrihorti Rabani Agrihorti LV-8041 Saha Mpida Gendot LV-8030 LV-8031 LV-8038 LV-8062 LV-8045 LV-49 LV-1925 LV-8051 LV-1422 LV-8044 LV-5168 LV-5685 LV-8052 LV-8032 LV-8043 LV-5684 LV-8060
demikian, dapat diasumsikan bahwa kelima marka SSR tersebut dapat mendiskriminasikan antarindividu varietas uji, baik varietas lokal maupun VUB, tanpa ada kesamaan secara genetik berdasarkan marka molekuler.
Mengingat profil sidik jari DNA diformulasikan dalam bentuk kode numerik, pembentukan kelas dilakukan dengan kode khusus dua digit mewakili tiap selang ukuran alel asli dari setiap lokus tertentu (Tabel 5). Berdasarkan jumlah total motif yang deteksi, diperoleh kode 00–10 dari ukuran alel ter-kecil hingga alel terbesar untuk lima lokus marka SSR sehingga setiap marka SSR memiliki jumlah kelas kode yang berbeda. Contohnya, EPMS441 menghasil-kan empat kelas kode, EPMS331 menghasilmenghasil-kan enam kelas kode, EPMS 335 dan GPMS194 masing-masing menghasilkan sebelas kelas kode, serta CaSSRBio1.1
menghasilkan tujuh kelas kode.
Pemberian kode numerik pada setiap varietas dengan set marka merupakan upaya pemberian identitas bagi varietas agar dapat dibedakan secara genetik dengan sistem digitalisasi. Sebagai contoh, marka EPMS441, yang mendeteksi empat alel dengan kisaran ukuran alel 101–140, akan menghasilkan empat kode numerik yang merupakan representasi ukuran alel homozigot hasil amplifikasi DNA oleh lokus marka SSR tersebut. Teknik pemberian kode numerik dilakukan pada kelima lokus marka SSR dalam set marka untuk identitas.
Berdasarkan ukuran alel menggunakan kelima marka SSR tersebut, diperoleh ID dalam bentuk kode numerik pada setiap varietas cabai (Tabel 6). Setiap varietas memiliki sepuluh digit kode numerik ber-dasarkan transformasi ukuran alel pada lima lokus Tabel 5. Kode yang diberikan untuk setiap selang ukuran alel yang diformulasikan untuk lima
marka SSR sebagai kandidat set marka dalam identifikasi varietas cabai lokal.
Kode
Kisaran alel
EPMS441 EPMS331 EPMS335 GPMS194 CaSSRBio1.1
00 101–105 166–170 125–130 201–205 201–205 01 116–120 171–175 131–135 211–215 211–215 02 131–135 176–180 141–145 216–220 216–220 03 136–140 181–185 161–165 221–225 221–225 04 186–190 166–170 226–230 226–230 05 191–195 176–180 236–240 236–240 06 181–185 241–245 241–245 07 191–195 246–250 08 196–200 256–260 09 216–220 261–265 10 236–240 266–270
Tabel 6. Identitas spesifik (ID) 20 varietas cabai lokal dan 2 VUB pembanding berdasarkan profil sidik jari dari set lima marka SSR hasil penelitian ini.
Varietas Spesies Asal Kelompok cabai Kode ID
Prima Agrihorti Capsicum frutescens Koleksi Balitsa Rawit 00000007** Rabani Agrihorti C. frutescens Koleksi Balitsa Rawit 0000010802 Gendot C. chinense Jawa Tengah Rawit **02000900
Saha Mpida C. frutescens NTB Rawit 0001010603
LV-8030 C. annuum Jawa Barat Besar 0000030200
LV-8031 C. annuum Jawa Barat Besar 0001020001
LV-8032 C. annuum Jawa Barat Keriting 0002040101 LV-8038 C. annuum Jawa Tengah Besar 0002020300 LV-8041 C. annuum Jawa Barat Keriting 0001040106 LV-8043 C. annuum Jawa Barat Keriting **01040104 LV-8044 C. annuum Jawa Barat Keriting 0001040100 LV-8045 C. annuum Jawa Barat Besar pendek 0001**0301
LV-1925 C. annuum Jawa Barat Besar 0001080100
LV-5168 C. annuum Jawa Barat Keriting 010207**00
LV-1422 C. annuum Jawa Barat Besar 0203060501
LV-8051 C. annuum Jawa Barat Keriting 0005040002 LV-5685 C. annuum Jawa Tengah Keriting 020503**03
LV-8052 C. annuum Jawa Barat Besar 0303090402
LV-5684 C. annuum Jawa Barat Keriting 0202060203 LV-8060 C. annuum Jawa Barat Besar 0004051005
LV-49 C. annuum Jawa Tengah Besar 0003100303
marka SSR tersebut. Sebagai contoh, VUB Rabani Agrihorti mempunyai ukuran alel 101, 169, 131, 256, 301 bp secara berurutan untuk lokus EPMS441, EPMS331, EPMS335, GPMS194, dan CaSSRBio1.1. Ber-dasarkan transformasi ke kode numerik (Tabel 5), keempat lokus marka SSR dari set marka tersebut diberi kode secara berurutan 00, 00, 01, 08, 02. Dengan demikian, ID VUB Rabani Agrihorti adalah 0000010802. Sementara, varietas lokal LV-8030 yang memiliki ukuran alel 101, 170, 164, 217, 292 bp secara berurutan dikodekan 00, 00, 03, 02, 00 untuk keempat lokus marka SSR dari set marka. Dengan demikian, ID varietas lokal LV-8030 adalah 0000030200. Prosedur yang sama juga diterapkan untuk pembuatan ID varietas cabai lainnya, namun jika terdapat alel yang tidak teramplifikasi maka diberikan kode “**”. Kelima marka SSR dalam set marka tersebut telah dapat membedakan seluruh varietas cabai yang digunakan, baik varietas lokal maupun VUB. Hal ini ditunjukkan oleh tidak adanya varietas dengan ID yang sama. Penetapan ID tersebut akan sangat bermanfaat dalam penelusuran latar belakang genetik dan kemurnian genetik varietas tersebut.
Penetapan identitas genetik khususnya pada varietas lokal akan sangat bermanfaat dalam mem-bantu perlindungan varietas lokal di Indonesia dari eksploitasi komersial dan klaim negara lain (Reflinur et al. 2016), menghindari kemungkinan duplikasi varietas (Chaerani et al. 2011; Lestari et al. 2012), mendiskriminasikan varietas dengan nama dan ka-rakter fenotipe yang identik, dan membantu penge-lolaan SDG khususnya varietas lokal di Indonesia (Santoso et al. 2006). Identitas genetik tersebut dapat pula digunakan sebagai alat penelusuran jika ditemu-kan varietas lokal yang memiliki nama yang sama namun berasal dari daerah yang berbeda (Lestari et al. 2016). Tersedianya identitas genetik untuk suatu varietas lokal akan sangat membantu jika varietas lokal tersebut memiliki karakter fenotipe yang superior sehingga akan dilepas menjadi varietas ung-gul. Informasi identitas genetik varietas cabai lokal Indonesia pada penelitian ini akan sangat berguna sebagai referensi kode identitas genetik dalam basis data pengelolaan SDG cabai.
Dengan demikian, set marka SSR yang diperoleh pada penelitian ini sangat bermanfaat untuk mendapatkan profil sidik jari DNA khususnya untuk varietas lokal yang dapat diidentifikasi secara spesifik dan terpisah dari kelompok varietas lokal lain dan VUB. Metode pemberian kode numerik sebagai profil sidik jari varietas cabai lokal Indonesia berdasarkan set marka SSR pada studi ini serupa dengan peneliti-an ypeneliti-ang telah dilakukpeneliti-an sebelumnya. Kode numerik
tersebut diberikan dalam pengembangan set marka SSR untuk identitas spesifik varietas kedelai lokal (Lestari et al. 2016), varietas kedelai unggul Indonesia (Risliawati et al. 2015), dan varietas kacang hijau unggul Indonesia (Reflinur et al. 2016). Meskipun susunan set marka SSR tersebut kemungkinan dapat berubah sejalan dengan penambahan jumlah varie-tas, metode pengembangan set marka SSR dan pem-berian identitas spesifik tersebut dapat diaplikasikan secara konsisten. Set marka SSR serta metode analisis sidik jari DNA yang dikembangkan pada pe-nelitian ini sangat bermanfaat untuk mengidentifikasi varietas cabai lokal secara molekuler guna meleng-kapi informasi fenotipe sebagai salah satu bentuk upaya perlindungan varietas cabai lokal di Indonesia, perlindungan konsumen, pendaftaran VUB, serta penyelesaian perselisihan terkait peredaran benih.
KESIMPULAN
Tingkat keragaman genetik 20 varietas cabai lokal dan 2 VUB pembanding pada penelitian ini cukup tinggi berdasarkan hasil analisis klaster dan koordinat utama menggunakan 15 marka SSR yang mampu membedakan varietas berdasarkan spesies-nya. Informasi keragaman genetik berdasarkan marka SSR dapat menjadi informasi awal dalam pe-milihan tetua persilangan dalam program pemuliaan cabai. Satu set marka SSR untuk identifikasi sidik jari DNA varietas cabai telah berhasil dikembangkan pada penelitian ini. Set marka SSR tersebut terdiri atas lima marka SSR (EPMS441, EPMS331, EPMS335, GPMS194, dan CaSSRBio1.1) yang mampu membeda-kan setiap individu varietas cabai lokal. Seluruh varietas cabai lokal pada penelitian ini telah memiliki identitas genetik dalam sepuluh digit kode unik yang spesifik untuk setiap varietas. Set marka SSR ini dapat digunakan sebagai pelengkap pada uji BUSS varietas cabai.
UCAPAN TERIMA KASIH
Penulis mengucapkan terima kasih kepada Meddy Saputra atas bantuan teknis laboratorium. Penelitian ini didanai melalui DIPA-APBN Balai Besar Penelitian dan Pengembangan Bioteknologi dan Sumber Daya Genetik Pertanian, Badan Penelitian dan Pengembangan Pertanian, Kementerian Pertani-an TA 2018. Penulis juga mengucapkPertani-an terima kasih kepada Dr. I Made Tasma atas masukan dan saran beliau dalam proses penulisan naskah ini.
KONTRIBUTOR PENULISAN
RTT: kontributor utama, mengumpulkan data, menganalisis data, dan menulis manuskrip. NDSAN: kontributor utama, analisis genotyping, dan mem-bantu menulis manuskrip. KN: kontributor anggota, membantu menulis manuskrip, dan analisis
genotyping. DS: kontributor anggota, review
ma-nuskrip. HK: kontributor anggota, penyedia materi genetik, dan informasi fenotipe. PL: kontributor anggota, menyusun desain dan ide penelitian, dan memfinalkan manuskrip.
DAFTARPUSTAKA
Bassil, N.V., Botta, R. & Mehlenbacher, S.A. (2005) Microsatellite markers in hazelnut: Isolation, characterization, and cross-species amplification.
Journal of the American Society for Horticultural Science, 130 (4), 543–549.
Botstein, D., White, R.L. & Davis, R.W. (1980) Construction of a genetic linkage map in man using restriction fragment length polymorfism. The American Journal of Human Genetics, 32, 314–331.
Carvalho, S.I.C., Ragassi, C.F., Oliveira, I.B., Amaral, Z.P.S., Reifschneider, F.J.B., Faleiro, F.G. & Buso, G.S.C. (2015) Transferability of microsatellite markers of Capsicum annuum L. to C. frutescens L. and C. chinense Jacq. Genetics and Molecular Research, 14 (3), 7937–7946.
Chaerani, C., Hidayatun, N. & Utami, D.W. (2011) Keragaman genetik 50 aksesi plasma nutfah kedelai berdasarkan sepuluh penanda mikrosatelit. Jurnal AgroBiogen, 7 (2), 96–105.
Chaesaria, N., Sobir & Syukur, M. (2018) Analisis keragaan cabai rawit merah (Capsicum frutescens) lokal asal Kediri dan Jember. Buletin Agrohorti, 6 (3), 388–396. Cheng, J., Zhao, Z., Li, B., Qin, C., Wu, Z., Trejo-Saavedra,
D.L., Luo, X., Cui, J., Rivera-Bustamante, R.F., Li, S. & Hu, K. (2016) A comprehensive characterization of simple sequence repeats in pepper genomes provides valuable resources for marker development in
Capsicum. Scientific Reports, 6, 1–12.
Cintamulya, I. (2011) Aplikasi penanda molekuler mikrosatelit/SSRs (simple sequence repeats) untuk menunjang program pemuliaan tanaman. Berkala Penelitian Hayati Edisi Khusus, 7A, 161–165.
Deviona, Syukur, M., Nurbaiti, Zuhry, E. & Cahya, E.B.N. (2013) Pendugaan keragaman genetik 20 genotipe cabai (Capsicum annuum) di lahan gambut. Dalam:
Seminar Nasional dan Rapat Tahunan Dekan Bidang Ilmu Pertanian BKS-PTN Wilayah Barat Tahun 2013. Pontianak, Universitas Tanjungpura, hlm. 371–377. Dhaliwal, M.S., Jindal, S.K., Gaikwad, A.K. & Singh, K.
(2013) Genetic diversity analysis and DNA fingerprinting of elite chilli pepper lines using SSR markers. International Journal of Vegetable Science, 19 (July), 207–216.
Dhaliwal, M.S., Yadav, A. & Jindal, S.K. (2014) Molecular characterization and diversity analysis in chilli pepper using simple sequence repeats (SSR) markers. African Journal of Biotechnology, 13 (31), 3137–3143. Ditjen Hortikultura (2019) Produksi cabai besar dan cabai
rawit tahun 2018. [Online] Direktorat Jenderal Hortikultura, Kementerian Pertanian. Tersedia pada: https://www.pertanian.go.id/home/?show=page&act=vi ew&id=61 [Diakses 8 Oktober 2019].
Doyle, J.J. & Doyle, J.L. (1990) Isolation of plant DNA from fresh tissue. Focus, 12, 13–15.
Duan, Y., Liu, J., Xu, J., Bian, C., Duan, S., Pang, W., Hu, J., Li, G. & Jin, L. (2019) DNA fingerprinting and genetic diversity analysis with simple sequence repeat markers of 217 potato cultivars (Solanum tuberosum
L.) in China. American Journal of Potato Research, 96 (1), 21–32.
Gao, L., Jia, J. & Kong, X. (2016) A SNP-based molecular barcode for characterization of common wheat. PLOS Genetics, 1–12.
Hadiati, S. (2003) Pendugaan jarak genetik dan hubungan kekerabatan nanas berdasarkan analisis isozim. Jurnal Hortikultura, 13 (2), 87–94.
Kristamtini, Taryono, Basunanda, P. & Murti, R.H. (2014) Keragaman genetik kultivar padi beras hitam lokal berdasarkan penanda mikrosatelit. Jurnal AgroBiogen, 10 (2), 69–76.
Kumar, A., Pandey, A., Aochen, C. & Pattanayak, A. (2015) Evaluation of genetic diversity and interrelationships of agro-morphological characters in soybean (Glycine max) genotypes. Proceedings of the National Academy of Sciences, India Section B: Biological Sciences, 85 (2), 397–405.
Kurniawan, A.C., Purwantoro, A. & Basunanda, P. (2015) Evaluasi karakter kualitatif cabai hias generasi F1 hasil
persilangan Capsicum annuum × Capsicum frutescens. Vegetalika, 4 (1), 10–14.
Kwon, Y., Lee, J., Yi, G., Yi, S., Kim, K., Soh, E., Bae, K., Park, E., Song, I. & Kim, B. (2005) Use of SSR markers to complement tests of distinctiveness, uniformity, and stability (DUS) of pepper (Capsicum annuum L.) varieties. Molecules and Cells, 19 (3), 428–435.
Lazar, I. (2010) GelAnalyzer 2010 user’s manual. [Online] Tersedia pada: http://www.gelanalyzer.com/downloads/
users_manual_2010.pdf [Diakses 20 Oktober 2017]. Lee, J., Nahm, S., Kim, Y. & Kim, B. (2004)
Characterization and molecular genetic mapping of microsatellite loci in pepper. Theoretical and Applied Genetics, 108 (4), 619–627.
Lestari, P., Risliawati, A. & Koh, H. (2012) Identifikasi dan aplikasi marka berbasis PCR untuk identifikasi varietas padi dengan palatabilitas tinggi. Jurnal AgroBiogen, 8 (2), 69–77.
Lestari, P., Risliawati, A., Utami, D.W., Hidayatun, N., Santoso, T.J. & Chaerani (2016) Pengembangan
identitas spesifik berbasis marka SSR pada 29 varietas kedelai lokal Indonesia. Jurnal Biologi Indonesia, 12 (2), 219–230.
Liu, K. & Muse, S.V. (2005) PowerMarker: An integrated analysis environment for genetic marker analysis.
Bioinformatics, 21 (9), 2128–2129.
Maranho, R.C., Augusto, R., Mangolin, C.A. & Machado, M.F.P.S. (2014) Use of differential levels of mean observed heterozygosity in microsatellite loci of commercial varieties of sugarcane (Saccharum spp.).
Genetics and Molecular Research, 13 (4), 10130– 10141.
Meng, Y., Zhao, N., Li, H., Zhai, H., He, S. & Liu, Q. (2018) SSR fingerprinting of 203 sweetpotato (Ipomoea batatas [L.] Lam.) varieties. Journal of Integrative Agriculture, 17 (1), 86–93.
Minamiyama, Y., Tsuro, M., Kubo, T. & Hirai, M. (2007) QTL analysis for resistance to Phytophthora capsici in pepper using a high density SSR-based map. Breeding Science, 57 (2), 129–134.
Monteiro, C.E. da S., Pereira, T.N.S. & de Campos, K.P. (2011) Reproductive characterization of interspecific hybrids among Capsicum species. Crop Breeding and Applied Biotechnology, 11 (3), 241–249.
Mustofa, Z., Budiarsa, I.M. & Samdas, G. (2013) Variasi genetik jagung (Zea mays L.) berdasarkan karakter fenotipik tongkol jagung yang dibudidaya di desa Jono Oge. e-Jipbiol, 1, 33–41.
Nagy, I., Stágel, A., Sasvári, Z., Röder, M. & Ganal, M. (2007) Development, characterization, and transferability to other Solanaceae of microsatellite markers in pepper (Capsicum annuum L.). Genome, 50 (7), 668–688.
Nicolaï, M., Cantet, M., Lefebvre, V., Sage-Palloix, A. & Palloix, A. (2013) Genotyping a large collection of pepper (Capsicum spp.) with SSR loci brings new evidence for the wild origin of cultivated C. annuum
and the structuring of genetic diversity by human selection of cultivar types. Genetic Resources and Crop Evolution, 60 (8), 2375–2390.
Oh, S., Song, J., Lee, J., Lee, G., Ko, H., Stoilova, T., Krasteva, L., Kim, Y., Rhee, J., Gwag, J., Ro, N., Hur, O. & Lee, M. (2012) Evaluation of genetic diversity of red pepper landraces (Capsicum annuum L.) from Bulgaria using SSR markers. Journal of the Korean Society of International Agriculture, 24 (5), 547–556. PGPI (2015) Pusat Genom Pertanian Indonesia. [Online]
Tersedia pada: http://genom.litbang.pertanian.go.id [Diakses 12 Desember 2018].
Rai, V.P., Kumar, R., Kumar, S., Rai, A., Kumar, S., Singh, M., Singh, S.P., Rai, A.B. & Paliwal, R. (2013) Genetic diversity in Capsicum germplasm based on microsatellite and random amplified microsatellite polymorphism markers. Physiology and Molecular Biology of Plants, 19 (4), 575–586.
Reflinur, Lestari, P. & Lee, S. (2016) The potential use of SSR markers to support the morphological identification of Indonesian mungbean varieties.
Indonesian Journal of Agricultural Science, 17 (2), 65– 74.
Ribeiro, C.A.G., Tanure, J.P.M., Maciel, T.E.F.M. & de Barros, E.G. (2013) Molecular characterization of soybean cultivars by microsatellite markers with universal tail sequence. Pesquisa Agropecuaria Brasileira, 48 (3), 270–279.
Risliawati, A., Riyanti, E.I., Lestari, P., Utami, D.W. & Silitonga, T.S. (2015) Development of SSR marker set to identify fourty two Indonesian soybean varieties.
Jurnal AgroBiogen, 11 (2), 49–58.
Ritschel, P.S., Lins, T.C.de L., Tristan, R.L., Buso, G.S.C., Buso, J.A. & Ferreira, M.E. (2004) Development of microsatellite markers from an enriched genomic library for genetic analysis of melon (Cucumis melo L.).
BMC Plant Biology, 14, 1–14.
Rohlf, F.J. (2000) NTSYS-pc: Numerical taxonomy and multivariate analysis system, version 2.1. Exeter Software Setauket, New York, USA.
Rubiyo (2013) Teknologi perbaikan bahan tanam kakao di Indonesia. Buletin RISTRI, 4 (3), 199–214.
Salimi, S., Lahiji, H.S., Abadi, G.M., Salimi, S. & Salaheddin, M. (2013) Genetic diversity in soybean genotypes under water stress and normal condition using factor analysis and cluster analysis. World Applied Sciences Journal, 8 (49), 6529–6536.
Santoso, T.J., Utami, D.W. & Septiningsih, E.M. (2006) Analisis sidik jari DNA plasma nutfah kedelai menggunakan markah SSR. Jurnal AgroBiogen, 2 (1), 1–7.
Saptadi, D., Liana, N., Waluyo, B., Purnamaningsih, S.L., Ardiarini, N.R. & Agustina, N.I. (2018) Evaluasi potensi keragaman genetik cabai rawit lokal untuk mendukung kegiatan seleksi. Dalam: Waluyo, B. & Saptadi, D. (ed.) Seminar Nasional Peripi Komda Jatim 2017: Sumbangan Ilmu Pemuliaan dalam Optimalisasi Pemanfaatan Sumberdaya Genetik Lokal Menjadi Varietas Unggul. Malang, Fakultas Pertanian Universitas Brawijaya dan Perhimpunan Ilmu Pemuliaan Indonesia, hlm. 91–104.
Shankar, R., Bagle, B.G. & More, T.A. (2009) Diversity analysis of bitter gourd (Momordica charantia L.) germplasm from tribal belts of India. The Asian and Australasian Journal of Plant Science and Biotechnology, 3 (1), 21–25.
Sharmin, A., Hoque, Md.E., Haque, Md.M. & Khatun, F. (2018) Molecular diversity analysis of some chilli (Capsicum spp.) genotypes using SSR markers.
American Journal of Plant Sciences, 9 (3), 368–379. Sitaresmi, T., Wening, R.H., Rakhmi, A.T., Yunani, N. &
Susanto, U. (2013) Pemanfaatan plasma putfah padi varietas lokal dalam perakitan varietas unggul. Iptek Tanaman Pangan, 8 (1), 22–30.
Surapaneni, M., Vemireddy, L.R., Begum, H., Reddy, B.P., Neetasri, C., Nagaraju, J., Anwar, S.Y. & Siddiq, E.A. (2013) Population structure and genetic analysis of different utility types of mango (Mangifera indica L.) germplasm of Andhra Pradesh state of India using
microsatellite markers. Plant Systematics and Evolution, 299 (7), 1215–1229.
Sutoro, Lestari, P. Risliawati, A, Nugroho, K. & Iriany, R.N. (2017) Evaluasi keragaman genetik jagung inbrida berdasarkan sepuluh marka simple sequence repeat.
Jurnal AgroBiogen, 13 (2), 89–90.
Syukur, M. & Yunianti, R. (2013) Pemanfaatan sumber daya genetik lokal dalam perakitan varietas unggul cabai (Capsicum annuum) tahan terhadap penyakit antraknosa yang disebabkan oleh Colletotrichum sp.
Jurnal Ilmu Pertanian, 18 (2), 67–72.
Tasma, I.M. & Arumsari, S. (2013) Analisis diversitas genetik aksesi kelapa sawit Kamerun berdasarkan marka SSR. Jurnal Littri, 19 (4), 194–202.
Tasma, I.M., Warsun, A., Satyawan, D., Syafaruddin & Martono, B. (2013) Analisis kekerabatan 50 aksesi kelapa sawit (Elaeis guineensis Jacq.) asal Kamerun berdasarkan marka mikrosatelit. Jurnal AgroBiogen, 9 (1), 19–27.
Tasma, I.M., Yani, N.P.M.G., Purwaningdyah, R., Satyawan, D., Nugroho, K., Lestari, P., Trijatmiko, K.R. & Mastur (2018) Genetic diversity analysis and F2 population
development for breeding of long juvenile trait in soybean. Jurnal AgroBiogen, 14 (1), 11–22.
Terryana, R.T., Nugroho, K., Rijzaani, H. & Lestari, P. (2018) Karakterisasi keragaman genetik 27 genotipe cabai berdasarkan marka SSR (simple sequence repeat). Berita Biologi, 17 (2), 183–194.
Toledo-Aguilar, R., López-Sánchez, H., Santacruz-Varela, A., Valadez-Moctezuma, E., López, P.A., Aguilar-Rincón, V.H., González-Hernández, V.A. & Vaquera-Huerta, H. (2016) Characterization of genetic diversity of native ‘Ancho’ chili populations of Mexico using microsatellite markers. Chilean Journal of Agricultural Research, 76 (1), 18–26.
Utami, D.W., Sutoro, Hidayatun, N., Risliawati, A. & Hanarida, I. (2011) Keragaman genetik 96 aksesi plasma nutfah padi berdasarkan 30 marka SSR terpaut gen pengatur waktu pembungaan (HD genes).
AgroBiogen, 7 (2), 76–84.
Vieira, M.L.C., Santini, L., Diniz, A.L. & Munhoz, A.de F. (2016) Microsatellite markers: What they mean and why they are so useful. Genetics and Molecular Biology, 39 (3), 312–328.
Wartono, Wiyono, S., Syukur, M., Giyanto, Nugroho, K. & Lestari, P. (2019) Genetic diversity analysis of 41 chili pepper genotypes (Capsicum annuum L.) based on SSR markers. Jurnal AgroBiogen, 15 (2), 65–74. Wei, J., Zheng, J., Yu, J., Zhao, D., Cheng, Y., Ruan, M.,
Ye, Q., Yao, Z., Wang, R., Zhou, G., Yang, Y., Li, Z. & Wan, H. (2019) Production and identification of interspecific hybrids between pepper (Capsicum annuum L.) and the wild relative (Capsicum frutescens
L.). Journal of Agricultural Science and Technology, 21 (3), 761–769.
Yumnam, J.S., Tyagi, W., Pandey, A., Meetei, N.T. & Rai, M. (2012) Evaluation of genetic diversity of chilli landraces from North Eastern India based on morphology, SSR markers and the Pun1 locus. Plant Molecular Biology Reporter, 30 (6), 1470–1479. Zhang, C.C., Wang, L.Y., Wei, K. & Cheng, H. (2014)
Development and characterization of single nucleotide polymorphism markers in Camellia sinensis
(Theaceae). Genetics and Molecular Research, 13 (3), 5822–5831.
Zhang, X.M., Zhang, Z.H., Gu, X.Z., Mao, S.L., Li, X.X., Chadœuf, J., Palloix, A., Wang, L.H. & Zhang, B.X. (2016) Genetic diversity of pepper (Capsicum spp.) germplasm resources in China reflects selection for cultivar types and spatial distribution. Journal of Integrative Agriculture, 15 (9), 1991–2001.