IDENTIFIKASI SPESIES RUMINANSIA DENGAN METODE
PCR-RFLP PADA GEN SITOKROM b MITOKONDRIA
JUITA SIREGAR
FAKULTAS KEDOKTERAN HEWAN INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi dengan judul Identifikasi Spesies Ruminansia dengan Metode PCR-RFLP pada Gen Sitokrom b Mitokondria adalah karya saya sendiri dengan arahan pembimbing dan belum diajukan dalam bentuk apapun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
ABSTRAK
JUITA SIREGAR. Identifikasi Spesies Ruminansia dengan Metode PCR-RFLP pada Gen Sitokrom b Mitokondria. Dibimbing oleh WAHONO ESTHI PRASETYANINGTYAS dan KUSDIANTORO MOHAMAD.
Tujuan dari penelitian ini adalah untuk melakukan identifikasi spesies ruminansia dengan metode PCR-RFLP pada gen sitokrom b dari DNA mitokondria. Sampel jaringan sapi, domba, kambing, dan kerbau diperoleh dari pasar di sekitar Bogor; sampel rusa, anoa, dan banteng dari koleksi Laboratorium Embriologi; serta sampel kijang dan kancil dari koleksi Laboratorium Anatomi. DNA diisolasi dengan menggunakan metode presipitasi amonium asetat. PCR-RFLP menggunakan primer universal sitokrom b dengan panjang fragmen 359 bp dan amplikon dipotong dengan enzim restriksi Hinf I dan Alu I. Hasil dari penelitian ini menunjukkan bahwa DNA dari sampel sapi, domba, kambing, kerbau, dan anoa dapat diamplifikasi. DNA dari sampel rusa, kijang, kancil, dan banteng tidak dapat diamplifikasi. Enzim Hinf I dapat memotong amplikon sapi, domba, kambing, dan anoa tetapi tidak untuk kerbau. Enzim Alu I dapat memotong amplikon sapi, kerbau, dan anoa tetapi tidak untuk kambing dan domba. Penelitian ini dapat membedakan antara sampel anoa dengan sampel ruminansia domestik lainnya. Hasil dari penelitian ini diharapkan dapat digunakan sebagai informasi dasar untuk identifikasi spesies ruminansia, terutama untuk anoa.
ABSTRACT
JUITA SIREGAR. Species Identification of Ruminants Using PCR-RFLP Analysis of Mitochondrial Cytochrome b Gene. Supervised by WAHONO ESTHI PRASETYANINGTYAS and KUSDIANTORO MOHAMAD.
The aim of this research is to identify the species of ruminants using PCR-RFLP analysis of mitochondrial cytochrome b gene. Cattle, sheep, goat, and buffalo tissues collected from Bogor market; bull, deer, and anoa tissues from collection of Embryology Laboratorium; kancil and muntjak tissues from collection of Anatomy Laboratorium. DNA were extracted using ammonium acetate precipitation. PCR-RFLP were performed using universal primers of cytochrome b gene with a 359 bp of fragment and then the amplicon were digested using restriction enzymes of Hinf I and Alu I. The result showed that DNA from cattle, sheep, goat, buffalo, and anoa could be amplified, and DNA from deer, kancil, muntjak, and bull couldn’t be amplified. Hinf I were capable to digest cattle, sheep, goat, and anoa amplicons but not buffalo amplicon. Alu I were capable to digest cattle, buffalo, and anoa amplicon but not sheep and goat amplicons. The assay was succesfully discriminated between anoa and the other domestic ruminants. The results of this study is expected to be used as basic information for tissue ruminant species identification, especially for anoa.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Kedokteran Hewan
pada
Fakultas Kedokteran Hewan
IDENTIFIKASI SPESIES RUMINANSIA DENGAN METODE
PCR-RFLP PADA GEN SITOKROM b MITOKONDRIA
FAKULTAS KEDOKTERAN HEWAN INSTITUT PERTANIAN BOGOR
PRAKATA
Segala puji dan syukur penulis ucapkan kepada Tuhan Yesus atas segala kasih karunia-Nya sehingga dapat menyelesaikan skripsi hasil penelitian yang berjudul Identifikasi Spesies Ruminansia dengan Metode PCR-RFLP pada Gen Sitokrom b Mitokondria.
Penulis mengucapkan terima kasih kepada ibu Drh Wahono Esthi Prasetyaningtyas, MSi PAVet selaku pembimbing skripsi pertama, bapak Drh Kusdiantoro Mohamad, MSi PAVet selaku pembimbing skripsi kedua, ibu Dr Drh Ita Djuwita, Mphil PAVet (K) (alm) untuk teladan dan ilmunya, ibu Dr Drh Ni Luh Putu Ika Mayasari yang telah memberikan bantuan bahan penelitian, Bapak Wahyudin, Amd selaku staf Laboratorium Embriologi FKH IPB, staf Laboratorium Pendidikan dan Layanan Terpadu Fakultas Kedokteran Hewan IPB, bapak penulis Djaparo Siregar (alm), ibu penulis Romina Simbolon untuk doanya yang luar biasa, semua saudara-saudara penulis untuk dukungan dan semangatnya, Dwi Budiono, Putri Ekandini, Fitri Susana, Siti Khadijah, Deny Putra Romadhon, dan Fatimah sebagai teman sepenelitian, Komisi Kesenian PMK IPB sebagai wadah untuk bertumbuh, serta semua pihak yang telah membantu yang tidak bisa disebutkan satu persatu.
Penulis menyadari bahwa karya tulis ini masih banyak terdapat kekurangan. Oleh karena itu penulis mengharapkan saran dan kritik dari semua pihak. Semoga karya tulis ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL xii
DAFTAR GAMBAR xii
DAFTAR LAMPIRAN xii
PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 2
Manfaat Penelitian 2
TINJAUAN PUSTAKA 2
Ruminansia 2
PCR-RFLP 2
DNA mitokondria 3
METODE PENELITIAN 4
Tempat dan Waktu Penelitian 4
Alat dan Bahan Penelitian 4
Prosedur Penelitian 4
Pengumpulan Sampel 4
Ekstraksi DNA Genom dari Jaringan 5
Amplifikasi DNA Mitokondria dengan PCR 5
Pemotongan Amplikon dengan Enzim Restriksi Hinf I dan Alu I 6 Pembacaan Hasil Menggunakan Elektroforesis 6
Penghitungan Ukuran Fragmen Restriksi 6
HASIL DAN PEMBAHASAN 7
Isolasi DNA 7
Amplifikasi DNA 7
Restriksi Amplikon 8
SIMPULAN 11
DAFTAR PUSTAKA 11
LAMPIRAN 14
DAFTAR TABEL
1 Taksonomi sembilan jenis ruminansia 2
2 Ukuran fragmen hasil pemotongan dengan menggunakan enzim
pemotong 9
DAFTAR GAMBAR
1 Hasil elekroforesis ekstraksi DNA pada gel agarosa 1% 7 2 Amplikon hasil amplifikasi DNA dengan menggunakan primer
universal sitokrom b pada gel agarosa 1,5% 8
3 Visualisasi fragmen pemotongan amplikon sitokrom b menggunakan
enzim Hinf I pada gel agarosa 2% 9
4 Visualisasi fragmen pemotongan amplikon sitokrom b menggunakan
enzim Alu I pada gel agarosa 2% 10
DAFTAR LAMPIRAN
PENDAHULUAN
Latar Belakang
Indonesia merupakan negara yang memiliki jenis ruminansia beragam. Berdasarkan data Badan Pusat Statistik tahun 2013, jumlah ruminansia domestik yang diternakkan meningkat dalam sepuluh tahun terakhir. Ruminansia yang telah didomestikasi biasanya digunakan sebagai sumber protein hewani bagi manusia. Beberapa jenis ruminansia yang telah didomestikasi di antaranya adalah sapi (Bos taurus), kerbau (Bubalus bubalis), domba (Ovis aries), dan kambing (Capra hircus). Selain itu juga terdapat beberapa jenis ruminansia liar di Indonesia yang sifatnya dilindungi di antaranya kancil (Tragalus javanicus), kijang (Muntiacus muntjak), rusa (Cervus timorensis), banteng (Bos javanicus), dan anoa (Bubalus quarlesi).
Beberapa tahun terakhir populasi ruminansia liar di Indonesia semakin menurun karena adanya penyakit, perburuan, dan perdagangan ilegal. Ancaman terhadap perdagangan ilegal dari hewan ruminansia liar dari Indonesia harus ditekan serendah mungkin agar keanekaragaman hayati Indonesia tetap terjaga. Salah satu cara untuk mempertahankan keanekaragaman hayati adalah dengan mengetahui data molekuler dari spesies tersebut. Data molekuler hewan berguna untuk mempelajari ekologi dan manajemen satwa liar (DeYoung dan Honeycutt 2005). Selain itu, data molekuler juga dapat digunakan untuk identifikasi spesies pada daging atau bagian tubuh hewan yang diperdagangkan secara ilegal.
Salah satu cara untuk mengetahui data molekuler dari spesies hewan adalah dengan menggunakan metode Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP) menggunakan gen sitokrom b dari DNA mitokondria. Analisis PCR-RFLP adalah salah satu teknik pertama yang digunakan untuk mendeteksi variasi pada tingkat sekuens DNA (Fatchiyah dan Arumingtyas 2011). Metode PCR-RFLP berdasarkan gen sitokrom b dari DNA mitokondria sudah digunakan secara luas untuk mengetahui data molekuler hewan, misalnya untuk identifikasi spesies burung (Wibowo 2009), ikan (Saputra 2009), dan sapi-sapi asli Indonesia(Mohamad et al. 2009).
2
Tujuan
Tujuan dari penelitian ini adalah untuk melakukan identifikasi spesies ruminansia berdasarkan perbedaan fragmen restriksi pada gen sitokrom b dari DNA mitokondria melalui metode PCR-RFLP.
Manfaat
Penelitian ini bermanfaat untuk memberikan informasi dasar untuk identifikasi spesies ruminansia dengan memotong amplikon menggunakan enzim restriksi Hinf I dan Alu I, terutama untuk anoa.
TINJAUAN PUSTAKA
Ruminansia
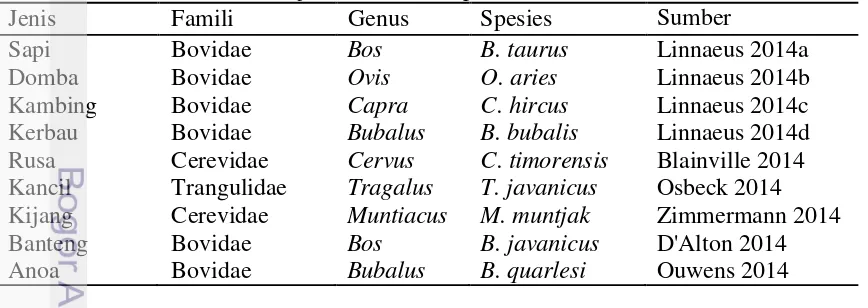
Ruminansia merupakan hewan mamalia berkuku genap (ungulata). Kata ruminan berasal dari bahasa latin yaitu “ruminare” yang artinya mengunyah kembali atau memamah biak. Hewan ruminansia umumnya herbivora sehingga sebagian besar makanannya adalah selulose, hemiselulose, dan bahkan lignin yang semuanya dikategorikan sebagai serat kasar. Ruminansia juga sering disebut polygastric animal karena lambungnya terdiri dari rumen, retikulum, omasum, dan abomasum. Sapi, domba, kambing, kerbau, rusa, kancil, kijang, banteng, dan anoa termasuk ke dalam filum chordata, kelas mamalia, ordo artiodactyla dengan famili, genus dan spesies dapat dilihat pada Tabel 1.
Tabel 1 Taksonomi sembilan jenis ruminansia penelitian
Jenis Famili Genus Spesies Sumber
Sapi Bovidae Bos B. taurus Linnaeus 2014a
Domba Bovidae Ovis O. aries Linnaeus 2014b
Kambing Bovidae Capra C. hircus Linnaeus 2014c
Kerbau Bovidae Bubalus B. bubalis Linnaeus 2014d Rusa Cerevidae Cervus C. timorensis Blainville 2014 Kancil Trangulidae Tragalus T. javanicus Osbeck 2014 Kijang Cerevidae Muntiacus M. muntjak Zimmermann 2014
Banteng Bovidae Bos B. javanicus D'Alton 2014
Anoa Bovidae Bubalus B. quarlesi Ouwens 2014
PCR-RFLP
3 DNA secara in vitro dengan bantuan enzim polymerase dan oligonukleotida sebagai primer untuk menggandakan jumlah molekul DNA secara spesifik melalui perubahan suhu. Proses PCR terdiri dari tiga tahapan: 1) Denaturasi, yaitu perubahan struktur DNA utas ganda menjadi utas tunggal, 2) Annealing, yaitu penempelan primer pada sekuens DNA komplementer yang akan diperbanyak, 3) Ekstensi yaitu pemanjangan primer oleh DNA polymerase, yang dilakukan dalam satu siklus amplifikasi (Sambrook et al. 1989).
Deteksi restriction fragment length polymorphism setelah dilakukan PCR (PCR-RFLP) dilakukan berdasarkan adanya kemungkinan untuk membandingkan profil pita-pita yang dihasilkan setelah dilakukan pemotongan oleh enzim restriksi terhadap DNA target atau individu yang berbeda. Perbedaan panjang fragmen dapat dilihat setelah dilakukan elektroforesis pada gel, hibridisasi, dan visualisasi. Aplikasi teknik PCR-RFLP biasa digunakan untuk mendeteksi diversitas genetik, hubungan kekerabatan, sejarah domestikasi, asal dan evolusi suatu spesies, aliran genetik (genetic drift) dan seleksi, pemetaan keseluruhan genom, pengamanan gen-gen target yang akan diekspresikan (tagging gene), mengisolasi gen-gen yang berguna dari spesies liar serta mengonstruksi pustaka DNA (Fatchiyah et al. 2011). PCR-RFLP merupakan metode yang mempunyai akurasi tinggi dan mudah ditransfer antar laboratorium.
Menurut Yuwono (2006), terdapat empat komponen utama pada proses PCR yaitu: 1) DNA cetakan, yaitu fragmen DNA yang akan dilipatgandakan, 2) oligonukleotida primer, yaitu sekuen oligonukleotida pendek (15-20 basa nukleotida) yang digunakan untuk mengawali sintesa rantai DNA, 3) deoksiribonukleotida trifosfat (dNTP), terdiri atas dATP, dCTP, dGTP, dTTP, serta 4) enzim DNA polymerase, yaitu enzim yang melakukan katalis reaksi sintesis terhadap rantai DNA. Komponen lain yang juga penting adalah senyawa buffer. Teknologi PCR mensyaratkan bagian tertentu dari sekuen DNA yang akan dilipatgandakan harus diketahui terlebih dahulu sebelum proses pelipatgandaan tersebut dilakukan. Metode PCR-RFLP memanfaatkan urutan nukleotida yang dikenali oleh enzim restriksi dan disebut dengan situs restriksi. Jika situs restriksi mengalami mutasi maka enzim restriksi tidak mampu mengenalinya. Analisis PCR-RFLP biasa digunakan untuk mendeteksi ada atau tidaknya keragaman pada gen yang berhubungan dengan sifat ekonomis (Sumantri et al. 2007).
DNA Mitokondria
4
lebih beragam dan dapat mengidentifikasi spesies yang memiliki kekerabatan yang dekat (Brown et al. 1996).
METODE PENELITIAN
Tempat dan Waktu Penelitian
Penelitian ini dilaksanakan di Laboratorium Pendidikan dan Layanan, Fakultas Kedokteran Hewan, Institut Pertanian Bogor dalam kurun waktu Januari–Juni 2014.
Alat dan Bahan Penelitian
Alat yang digunakan dalam penelitian ini antara lain alat diseksi, timbangan analitik, mikropipet 0.5–10 μl, mikropipet 10–100 μl, mikropipet 100–1000 μl, mikrotip, tabung eppendorf 1.5 ml, tabung PCR, tabung erlenmeyer, gelas piala, vortex mixer, sentrifus, microwave, waterbath, electrophoresis, ultraviolet transluminator, automatic thermal cycler GeneAMP® PCR System 9700 (Applied Biosystem), dan lemari pendingin 4 ºC dan -20 ºC.
Bahan-bahan yang digunakan dalam penelitian ini adalah daging sapi, kerbau, domba, kambing, kancil, kijang, rusa, anoa dan jaringan kulit banteng. Bahan lain yang digunakan adalah pure water (milli-Q), larutan lysis buffer (Tris-HCl 10 mM, EDTA 25 mM, NaCl 100 mM, SDS 0.5% pada pH 8), Proteinase K, larutan RNAse, larutan ammonium asetat 5 M, isopropanol, etanol 70%, larutan TAE (Tris Asetat EDTA) 1 X, loading dye 6 X (Bromophenol blue 0.25%, Xylene cyanol RF 0.25% sucruze dalam H2O 40%), ethidium bromida, Invitrogen® PCR
5 Ekstrasi DNA Genom dari Jaringan
Sampel daging dan kulit diekstraksi dengan menggunakan metode presipitasi ammonium asetat (Sambrook et al. 1982). Untuk jaringan yang dipreservasi dalam formalin, jaringan terlebih dahulu dimasukkan ke dalam larutan alkohol 70% selama 48 jam dengan penggantian alkohol dua kali, selanjutnya jaringan dimasukkan ke dalam larutan lisis buffer selama 48 jam dengan penggantian lisis buffer dua kali. Sementara untuk jaringan kulit yang dipreservasi dalam alkohol 70% dimasukkan ke dalam larutan lisis buffer selama 24 jam tanpa penggantian larutan lisis buffer (Shiozawa et al. 1992).
Sebanyak 100 mg jaringan diambil dan dihancurkan dengan menggunakan penumbuk hingga halus kemudian dimasukkan ke dalam tabung eppendorf 1.5 ml. Jaringan tersebut kemudian dilarutkan dalam larutan lisis buffer sebanyak 500 l dan diinkubasi dengan menggunakan waterbath selama 1 jam pada suhu 55 ºC. Kemudian ke dalam larutan tersebut ditambahkan 6 µl proteinase K (10 mg/ml) dan diinkubasi kembali selama 3 jam pada suhu 55 ºC atau satu malam.
Sebanyak 3 µl RNAse solution (20 mg/ml) ditambahkan dan dicampur merata dengan cara membolak-balikkan larutan sebanyak 25 kali, setelah itu diinkubasi pada suhu 37 ºC selama 15-30 menit. Larutan yang ada dalam tabung disimpan dan didiamkan di dalam pecahan es selama 30 menit, kemudian disentrifugasi dengan kecepatan 9 000 g selama 30 menit pada suhu 4 ºC.
Setelah dilakukan sentrifugasi, larutan tersebut akan terpisah menjadi endapan di bagian bawah dan supernatan di bagian atas tabung. Supernatan tersebut diambil dan dimasukkan ke dalam tabung eppendorf yang baru. Larutan ammonium acetate 5 M sebanyak 500 µl dimasukkan ke dalam tabung eppendorf yang berisi supernatan, kemudian dihomogenkan dengan menggunakan vortex pada kecepatan maksimum selama 20 detik dan didiamkan selama 10 menit. Setelah itu, larutan tersebut disentrifugasi pada kecepatan 3000 rpm, pada suhu 4 ºC selama 15 menit. Supernatan hasil sentrifugasi dipindahkan ke dalam tabung eppendorf baru yang sebelumnya telah diisi 600 µl isopropanol absolut pada suhu ruang. Larutan dihomogenkan dengan cara membolak-balik tabung kemudian didiamkan selama satu malam pada suhu ruangan hingga terbentuk endapan DNA.
Setelah satu malam, larutan disentrifugasi pada kecepatan 3 000 rpm pada suhu 4 ºC selama 5 menit sehingga DNA akan terlihat sebagai endapan putih kecil. Supernatan dibuang dan apabila diperlukan endapan dapat dilakukan sentrifugasi ulang untuk membuang sisa isopropanol. Endapan yang tersisa kemudian ditambahkan 500 µl alkohol 70% dingin, kemudian dihomogenkan dengan cara membolak-balikkan tabung beberapa kali untuk membantu pencucian DNA. Setelah itu larutan tersebut disentrifugasi pada kecepatan 16 000 g selama 1 menit. Supernatan (sisa alkohol) dibuang secara hati-hati supaya endapan tidak terikut. Endapan yang tersisa kemudian dikeringkan pada suhu kamar atau pada suhu 75 ºC selama 5 menit. Setelah endapan tersebut kering, ke dalam tabung ditambahkan 3050 µl ddH2O (MQ) dan didiamkan selama semalam atau diinkubasi pada suhu 60 ºC selama 3 menit atau 55 ºC selama 2030 menit untuk melarutkan DNA.
Amplifikasi Fragmen DNA Mitokondria dengan PCR
6
menggunakan primer universal sitokrom b dengan urutan basa cyt b-1 -5’CCA TCC AAC ATC TCA GCA TGA TGA AA-3’ dan cyt b-2 -5’CCC CTC AGA ATG ATA TTT GTC CTC TCA-3’ (359 bp). Larutan reaksi terdiri dari 3 μl templat DNA, primer sitokrom b1 (10 μM) dan primer sitokrom b2 (10 μM) masing-masing 1 μl, danInvitrogen® PCR Reagen Supermix45 μl, sehingga total volume dalam 1× reaksi PCR adalah 50 μl.
Tabung PCR yang sudah berisi larutan tersebut dimasukan ke dalam mesin PCR GeneAMP® PCR System 9700 yang sudah diprogram. Siklus yang digunakan dimulai dengan 1 siklus denaturasi awal pada suhu 95 °C selama 5 menit, dilanjutkan dengan 35 siklus yang terdiri dari (i) denaturasi pada suhu 95 °C selama 45 detik, (ii) annealing primer pada suhu 55 °C selama 45 detik, (iii) extention pada suhu 72 °C selama 1 menit, kemudian diakhiri dengan 1 siklus extention akhir pada suhu 72 °C selama 3 menit.
Pemotongan Amplikon dengan Enzim Restriksi Hinf I dan Alu I
Proses pemotongan DNA hasil amplifikasi (amplikon) dilakukan dengan menggunakan enzim Hinf I dan Alu I. Reagen yang digunakan dalam pemotongan DNA hasil PCR dengan Hinf I dan Alu I secara berurutan adalah ddH2O sebanyak 12 µl, kemudian larutan buffer sebanyak 2.5 µl, setelah itu dicampurkan enzim Hinf I atau Alu I (10 U/µl) sebanyak 0.5 µl (5 unit), serta DNA hasil PCR sebanyak 10 µl sehingga diperoleh volume total 25 µl. DNA hasil PCR yang dipotong selanjutnya diinkubasi pada suhu 37 ºC selama 2 jam untuk pemotongan dengan Hinf I dan selama 1 jam pada suhu 37 ºC untuk pemotongan dengan Alu I. Pembacaan Hasil Menggunakan Elektroforesis
Hasil ekstraksi dibaca secara kualitatif dengan menggunakan elektroforesis gel agarosa 1% dalam 1× TAE buffer. Hasil PCR dibaca dengan menggunakan elektroforesis gel agarosa 1.5% dan hasil pemotongan amplikon dengan enzim Hinf I dan Alu I menggunakan elektroforesis gel agarosa 2%. Ultraviolet transluminator digunakan untuk melihat pendar pita setelah elektroforesis.
Penghitungan Ukuran Fragmen Restriksi
7 HASIL DAN PEMBAHASAN
Isolasi DNA
Hasil elektroforesis DNA pada agarosa 1% menunjukkan DNA dari sapi, domba, kambing, kerbau, dan banteng terlihat jelas, sedangkan DNA dari rusa dan anoa terlihat sangat tipis. Hasil ini menunjukkan bahwa konsentrasi DNA sampel mencukupi untuk dilihat dengan elektroforesis. DNA dari sampel kancil dan kijang tidak terlihat pada hasil elektroforesis. Hal ini menunjukkan bahwa konsentrasi DNA tidak ada atau belum mencukupi untuk dilihat dengan elektroforesis.
Secara umum masalah yang sering dialami ketika melakukan ekstraksi DNA adalah ukuran DNA pendek, DNA patah-patah selama proses isolasi, dan adanya metabolit sekunder yang ikut terisolasi (Fatchiyah et al. 2011). Kondisi penyimpanan sampel dan metode ektraksi DNA dapat mempengaruhi kualitas dan jumlah DNA sampel yang diperoleh. Hasil ekstraksi DNA menggunakan kit lebih baik dibandingkan dengan ekstraksi secara konvensional (Shrivastava et al. 2012). Selain itu, jenis preservasi sampel juga berpengaruh terhadap hasil ekstraksi DNA. Sampel yang dipreservasi dalam formalin lebih sulit diekstraksi daripada sampel yang dipreservasi pada DMSO 25% atau alkohol 70%. Kandungan formalin yang terdapat pada sampel dapat mengganggu proses presipitasi DNA sehingga dibutuhkan perlakuan khusus untuk mengurangi kandungan formalin dalam sampel (Shiozawa et al. 1992).
Amplifikasi DNA
Amplifikasi DNA dengan menggunakan primer sitokrom b menghasilkan amplikon dengan panjang fragmen 359 bp. Ukuran panjang fragmen yang dihasilkan sesuai dengan primer yang digunakan. Hasil elektroforesis menunjukkan bahwa DNA yang diekstrak dari sampel sapi, domba, kambing,
8
kerbau, dan anoa memiliki DNA yang cukup dan berhasil diamplifikasi dengan PCR. Sedangkan DNA dari sampel rusa dan banteng tidak ada atau belum mencukupi sehingga tidak berhasil diamplifikasi dengan PCR.
Kegagalan dalam amplifikasi pada sampel rusa dan banteng dapat disebabkan beberapa faktor seperti konsentrasi ekstraksi DNA yang sedikit dan kontaminan di dalam DNA sehingga mengganggu proses PCR. Kontaminan yang umum ditemukan di antaranya polisakarida yang dapat menghambat aktivitas Taq polimerase atau polifenol teroksidasi yang akan mengikat DNA secara kovalen (Fatchiyah et al. 2011). Selain itu, lama preservasi dan jenis preservasi sampel juga mempengaruhi kualitas dan jumlah DNA yang diperoleh (Shrivastava et al. 2012).
Amplifikasi DNA dengan metode PCR pada penelitian ini menggunakan gen sitokrom b (cyt b). Sitokrom b adalah bagian dari sitokrom pada transpor elektron yang terletak di rantai respirasi mitokondria dan dikodekan oleh DNA mitokondria. DNA mitokondria lebih banyak digunakan karena lebih cepat dalam mengidentifikasi spesies daripada DNA nuklear, selain itu juga mengandung sekuen yang lebih beragam jika dibandingkan dengan DNA nuklear dan juga dapat mengidentifikasi spesies yang memiliki kekerabatan yang dekat (Brown et al. 1996).
Restriksi Amplikon
Amplikon hasil PCR yang dipotong dengan menggunakan enzim Hinf I dan Alu I menunjukkan fragmen pemotongan yang sesuai menurut Meyer et al. (1995) untuk sampel sapi, domba, kambing, dan kerbau (Tabel 2). Sedangkan untuk anoa belum ada yang melaporkan dan dilakukan penghitungan secara manual (Lampiran 1 dan 2).
Hasil pemotongan dengan enzim Hinf I menunjukkan amplikon sapi dapat dipotong menjadi tiga fragmen yaitu 198 bp, 117 bp, dan 44 bp, amplikon domba dan kambing menjadi dua fragmen yaitu 198 bp dan 161 bp, amplikon anoa Gambar 2 Amplikon hasil amplifikasi DNA dengan menggunakan primer
9 menjadi dua fragmen yaitu 200 bp dan 159 bp, dan amplikon kerbau tidak terpotong (Tabel 2, Gambar 3). Fragmen amplikon anoa yang terpotong berbeda dengan fragmen ruminansia domestik lainnya.
Tabel 2 Ukuran fragmen hasil pemotongan dengan menggunakan enzim restriksi
Spesies
Fragmen DNA (bp) setelah pemotongan dengan enzim
restriksi Sumber
Hinf I Alu I Sapi (Bos taurus) 198, 117, 44 190, 169
Meyer et al. 1995
Domba (Ovis aries) 198, 161 -
Kambing (Capra hircus) 198, 161 - Kerbau (Bubalus bubalis) - 190, 169
Anoa (Bubalus quarlesi) 200, 159 180, 179 Penelitian* *Penghitungan manual
Hasil pemotongan dengan enzim Alu I menunjukkan amplikon sapi dan kerbau dapat dipotong menjadi dua fragmen yaitu 190 bp dan 169 bp, amplikon anoa menjadi dua fragmen yaitu 180 bp dan 179 bp. Sedangkan amplikon domba dan kambing tidak dapat dipotong karena tidak memiliki titik restriksi (Tabel 2, Gambar 4).
10
Pemotongan amplikon dengan menggunakan enzim Hinf I menunjukkan bahwa enzim tersebut dapat memotong amplikon sapi, domba, kambing, dan anoa. Domba dan kambing memiliki ukuran fragmen amplikon yang sama jika dipotong dengan enzim Hinf I sehingga enzim Hinf I tidak bisa digunakan untuk membedakan sampel kambing dan domba. Enzim Hinf I juga tidak dapat digunakan untuk mendeteksi sampel kerbau karena tidak dapat memotong amplikon. Pemotongan amplikon dengan menggunakan enzim Hinf I dapat digunakan untuk membedakan sampel sapi, kerbau, dan anoa dengan sampel domba atau kambing. Sampel anoa dapat dibedakan dari semua jenis sampel ruminansia domestik dengan enzim Hinf I.
Enzim Alu I tidak dapat digunakan untuk membedakan sampel kerbau dan sapi karena amplikon yang terpotong memiliki ukuran fragmen yang sama. Enzim Alu I juga tidak dapat digunakan untuk mendeteksi sampel kambing dan domba karena tidak dapat memotong amplikon. Tetapi enzim ini sangat efektif untuk membedakan sampel anoa dengan sampel yang lain.
Identifikasi spesies dengan metode PCR-RFLP pada sitokrom b DNA mitokondria telah banyak dilakukan untuk keperluan forensik (Bravi et al. 2004). Teknik ini dapat digunakan untuk mengetahui campuran dari daging yang diperjualbelikan di pasar. Daging kerbau dan anoa sangat mungkin dipalsukan karena masih dalam satu genus. Sebelumnya telah terdapat penelitian mengenai analisis diversitas kerbau domestik dengan anoa berdasarkan sekuensing sitokrom b DNA mitokondria (Kikkawa et al. 1999). Identifikasi sampel anoa dan kerbau dapat dilakukan dengan metode PCR-RFLP karena lebih mudah dan membutuhkan waktu yang singkat dibandingkan dengan sekuensing. Sejauh ini, PCR-RFLP dengan enzim Hinf I dan Alu I menjadi pertama kali dilaporkan dapat secara efektif membedakan sampel anoa dengan kerbau dan juga dengan ruminansia domestik lainnya seperti sapi, domba, dan kambing.
11
SIMPULAN
Enzim Hinf I dapat digunakan untuk mengidentifikasi DNA sapi, domba, kambing, dan anoa karena dapat memotong amplikon dengan ukuran fragmen yang berbeda tetapi tidak dapat digunakan untuk mengidentifikasi kerbau. Enzim Alu I dapat digunakan untuk mengidentifikasi sapi, kerbau, dan anoa karena dapat memotong amplikon. Enzim Alu I tidak dapat digunakan untuk mengidentifikasi kambing dan domba. Enzim Hinf I dan Alu I dapat digunakan untuk membedakan anoa dengan ruminansia lainnya seperti sapi, domba, kambing, dan kerbau.
DAFTAR PUSTAKA
[BPS] Badan Pusat Statistik. 2013. Populasi ternak di Indonesia. [Internet]. [diunduh 17 April 2014]. Tersedia pada: http://www.bps.go.id/tab_ sub/view.php.
Blainville. 2014. Cervus timorensis. [Internet]. [diunduh 9 September 2014]. Tersedia pada: http://www.iucnredlist.org/details/41789/0.
Bravi CM, Liron JP, Mirol PM, Ripoli MV, Garcia PP, Giovambattista G. 2004. A simple method for domestic animal identification in Argentina using PCR-RFLP analysis of cytochrome b gene. Leg Med. 6:246251.
Brown JR, Beckenbach K, Beckenbach AT, Smith MJ. 1996. Length variation, heteroplasmy and sequence divergence in the mitochondrial DNA of four species of sturgeon (Acipenser). Genetics. 142:525-535.
D'Alton. 2014. Bos javanicus. [Internet]. [diunduh 9 September 2014]. Tersedia pada: http://www.iucnredlist.org/details/2888/0.
DeYoung RW, Honeycutt RL. 2005. The molecular toolbox: genetic techniques in wildlife ecology and management. J Wildlife Manage. 69(4):1362-1384. Fatchiyah, Arumingtyas EL. 2011. Restriction Fragment Length Polymorphism
(RFLP). Di dalam: Fatchiyah, Arumingtyas EL, Widyarti S, Rahayu S. Biologi Molekular-Prinsip Dasar Analisis. Jakarta (ID): Erlangga. hlm 42– 46.
Fatchiyah, Rahayu S, Arumingtyas EL. 2011. Isolasi DNA. Di dalam : Fatchiyah, Arumingtyas EL, Widyarti S, Rahayu S. Biologi Molekular-Prinsip Dasar Analisis. Jakarta (ID): Erlangga. hlm 21–32.
Hsieh HM, Chiang HL, Tsai LC, Lai SY, Huang NE, Linnere A, Lee LCI. 2001. Cytochrome b gene for species identification of the conservation animals. Foren Sci Int. 122:7-18.
Kikkawa Y, Yonekawa H, Suzuki H, Amano T. 1999. Analysis of genetic diversity of domestic water buffaloes and anoas based on variations in the mitochondrial gene for cytochrome b. Anim Genet. 28:195201.
Linnaeus. 2014a. Bos taurus. [Internet]. [diunduh 9 September 2014]. Tersedia pada: http://arctos.database.museum/name/Bos%20taurus#NCBI.
12
Linnaeus. 2014c. Capra hircus. [Internet]. [diunduh 9 September 2014]. Tersedia pada: http://arctos.database.museum/name/Capra%20hircus#NCBI.
Linnaeus. 2014d. Bubalus bubalis. [Internet]. [diunduh 9 September 2014]. Tersedia pada: http://arctos.database.museum/name/Bubalus%20bubalis-#NCBI.
Meyer R, Hofelein C, Luthy J, Candrian U. 1995. Polymerase chain reaction-restriction fragment length polymorphism analysis: a simple method for species identification in food. J AOAC Int. 78(6):1542–1551.
Mohamad K, Ollson M, van Tol HTA, Mikko S, Vlamings BH, Andersson A, Martinez HR, Purwantaro B, Paling RW, Colenbrander B, et al. 2009. On the origin of Indonesia cattle. Plos One. 4(5):16.
Muladno. 2010. Teknologi Rekayasa Genetika. Edisi Kedua. Bogor (ID): IPB Pr. Osbeck. 2014. Tragalus javanicus. [Internet]. [diunduh 9 september 2014].
Tersedia pada: http://www.iucnredlist.org/details/41780/0
Ouwens. 2014. Bubalus quarlesi. [Internet]. [diunduh 9 september 2014]. Tersedia pada: http://www.iucnredlist.org/details/3128/0
Pfeiffer I, Burger J, Brenig B. 2004. Diagnostic polymorphism in the mitochondrial cytochrome b gene allow discrimination between cattle, sheep, goat, roe buck, and deer by PCR-RFLP. BMC Genetics. 5:30-34. Prusak B, Grzybowski T, Bednarek J. 2005. Cytochrome b gene (cytb) in analysis
of anonymous biological traces and its application in veterinary diagnostics and animal conservation. Anim Sci Pap Rep. 23(4):229–236.
Sambrook J, Maniatis T, Fritsch FF. 1982. Molecular Clonning: A Laboratory Manual. New York (US): Cold Spring Harbour Lab Pr.
Sambrook J, Fritsch EF, Manuatis T. 1989. Molecular Cloning, A Laboratory Manual. 3rd Ed. New York (US): Cold Spring Harbour Lab Pr.
Saputra D. 2009. Gambaran restriction fragment length polymorphism (RFLP) gen sitokrom b DNA mitokondria dari sembilan spesies ikan air tawar konsumsi [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Sartika T, Duryadi D, Mansjoer SS, Gunawan B. 2000. Genetic diversity of native chicken based on analysis of D-Loop mtDNA marker. JITV. 5(2):100-106.
Shiozawa DK, Kudo J, Evans RP, Woodward SR, Williams RN. 1992. DNA extraction from preserved trout tissues. Great Basin Nat. 52(1):29-34. Shrivastava K, Thakur MS, Tomar MPS, Shrivastav AB, Parmar SNS. 2012.
Extraction of genomic DNA from formaline fixed tissues of different wild avian species. Ann Biol Res. 3(7):3174-3177.
Sumantri C, Anggraeni A, Farajallah A, Perwitasari D. 2007. Keragaman mikrosatelit DNA sapi perah FH di balai pembibitan ternak unggul Baturaden. JITV. 12: 124-133.
Wibowo DA. 2009. Restriction fragment length polymorphism (rflp) gen sitokrom b DNA mitokondria dari delapan spesies burung [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Williams JL. 2005. The use of marker-assisted selection in animal breeding and biotechnology. Rev Sci Tech of Int Epiz. 24:379-391.
13 Zimmermann. 2014. Muntiacus muntjak. [Internet]. [diunduh 9 September 2014].
14
Lampiran 1 Penghitungan manual fragmen restriksi Hinf I pada sampel anoa
Keterangan :
DNA ladder 100 bp : 2 cm 200 bp : 1.5 cm 300 bp : 1.3 cm
Fragmen restriksi anoa 1 : 1.5 cm (200 bp) Fragmen restriksi anoa 2 : 1.7 cm (159 bp)
B
er
at m
olekul (
bp)
15 Lampiran 2 Penghitungan manual fragmen restriksi Alu I pada anoa
Keterangan :
DNA ladder 100 bp : 2 cm 200 bp : 1.5 cm 300 bp : 1.3 cm
Fragmen restriksi anoa 1 : 1.6 cm (180 bp) Fragmen restriksi anoa 2 : 1.62 cm (179 bp)
B
er
at m
olekul (
bp)
16
RIWAYAT HIDUP
Penulis dilahirkan di Sumatera Utara pada 13 Desember 1992 dari ayah Djaparo Siregar (alm) dan ibu Romina Simbolon. Penulis adalah anak kesepuluh dari sepuluh bersaudara. Penulis menghabiskan masa sekolah SD sampai SMA di Muara. Lulus dari SD 175795 Lobutangga tahun 2004 kemudian melanjutkan pendidikan di SMP Negeri 1 Muara lulus pada tahun 2007. Tahun 2010 lulus dari SMA Negeri 1 Muara dan pada tahun yang sama penulis diterima di Institut Pertanian Bogor (IPB) melalui jalur Undangan Seleksi Masuk IPB dan diterima di Fakultas Kedokteran Hewan IPB.