CLUSTERING FRAGMEN METAGENOME MENGGUNAKAN
SOM DENGAN EKSTRAKSI FITUR GRAY LEVEL
CO-OCCURRENCE MATRIX (GLCM) PADA
VARIASI PANJANG FRAGMEN
DANIALDI WAHYU PRATAMA
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Clustering Fragmen Metagenome Menggunakan SOM dengan Ekstraksi Fitur Gray Level Co-occurrence Matrix (GLCM) Pada Variasi Panjang Fragmen adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
ABSTRAK
DANIALDI WAHYU PRATAMA. Clustering Fragmen Metagenome Menggunakan SOM dengan Ekstraksi Fitur Gray Level Co-occurrence Matrix (GLCM) Pada Variasi Panjang Fragmen. Dibimbing oleh AZIZ KUSTIYO dan TOTO HARYANTO.
Penelitian di bidang bioinformatika berkembang semakin pesat, terutama penelitian mengenai metagenome. Namun, pada penelitian metagenome masih terdapat kelemahan, yaitu proses sequencing secara langsung akan memungkinkan bercampurnya fragmen sehingga dapat mempengaruhi perakitan fragmen. Oleh sebab itu, penelitian ini menggunakan teknik clustering untuk mengantisipasi kesalahan perakitan tersebut. Data yang digunakan dalam penelitian ini adalah data metagenome yang diunduh dari situs National Center for Biotechnology Information (NCBI) sebanyak 100 organisme dari 10 genus. Terdapat 4 variasi panjang fragmen yang digunakan, yaitu 200 bp, 1 Kbp, 3 Kbp, dan 10 Kbp. Clustering pada fragmen dilakukan dengan menggunakan metode Self Organizing Maps (SOM) dan pada tahap ekstraksi fitur menggunakan metode Gray Level Co-occurrence Matrix (GLCM). Proses clustering yang telah dilakukan mencapai akurasi 92% - 93.6%. Nilai spesifisitas berada pada rentang 85.9% - 87.4%, sedangkan nilai sensitivitas berada pada rentang 61% - 65%. Dari penelitian yang telah dilakukan, tidak terlihat adanya pengaruh panjang fragmen terhadap hasil akurasi clustering.
Kata kunci: clustering, fragmen, GLCM, metagenome, SOM
ABSTRACT
DANIALDI WAHYU PRATAMA. Metagenome Fragment Clustering Using SOM With Gray Level Co-occurrence Matrix (GLCM) Feature Extraction on the Fragment Length Variation. Supervised by AZIZ KUSTIYO and TOTO HARYANTO.
Research in bioinformatics has developed rapidly, especially research about metagenome. However, metagenome studies still have weaknesses, for example direct sequencing process could mix fragments that will affect the assembly of the fragments. Therefore, in this research we use clustering method to anticipate that problem. This research uses data downloaded from National Center for Biotechnology Information (NCBI) website, consisting of 100 organisms from 10 genuses. There are 4 variations of fragment length that we use: 200 bp, 1 Kbp, 3 Kbp and 10 Kbp. The clustering method used in this is the Self Organizing Maps (SOM). For the feature extraction stage, we used the Gray Level Co-occurrence Matrix (GLCM) method. Based on clustering process, the accuracy of this research was 92% - 93.6%. The specificity value was 85.9% - 87.4%, and sensitivity value was 61% - 65%. Based on the result, fragment length did not affect clustering accuracy.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Komputer
pada
Departemen Ilmu Komputer
CLUSTERING FRAGMEN METAGENOME MENGGUNAKAN
SOM DENGAN EKSTRAKSI FITUR GRAY LEVEL
CO-OCCURRENCE MATRIX (GLCM) PADA
VARIASI PANJANG FRAGMEN
DANIALDI WAHYU PRATAMA
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Desember 2014 ini ialah clustering metagenome, dengan judul Clustering Fragmen Metagenome Menggunakan SOM dengan Ekstraksi Fitur Gray Level Co-occurrence Matrix (GLCM) pada Variasi Panjang Fragmen. Penulis juga menyampaikan terima kasih kepada seluruh pihak yang telah berperan dalam penelitian ini, yaitu:
1 Kedua orang tua, adik, dan keluarga atas doa, motivasi, dan kasih sayangnya untuk menyelesaikan penelitian ini.
2 Bapak Aziz Kustiyo, SSi, MKom dan Bapak Toto Haryanto, SKom, MSi selaku dosen pembimbing yang telah memberi ide, saran, dan bantuan hingga penelitian ini selesai.
3 Bapak Dr Eng Wisnu Ananta Kusuma, ST, MT selaku dosen penguji yang telah memberi saran dalam penelitian ini.
4 Rekan satu topik penelitian, yaitu Hilda Sudawani atas kerjasama dan saran selama ini.
5 Rekan satu bimbingan, Selma Siti Lutfiah, Fitri, dan Ulfa Nikmatiya atas bantuannya selama ini.
6 Rahmanda Wibowo, Randolph Wibowo, Dede Nur Fitriansyah, Firdaus Saptahadi Pratama, dan rekan-rekan Ilmu Komputer 48 atas segala kebersamaan, bantuan, dan dukungan selama menjalani studi.
Semoga karya ilmiah ini bermanfaat.
DAFTAR TABEL
1 Confusion matrix 7
2 Rincian panjang data genus yang digunakan 8
3 Perbandingan panjang fragmen dan jumlah fragmen 9
4 Hasil clustering pada output 9 cluster 12
5 Hasil clustering pada output 10 cluster 12
6 Hasil clustering pada output 11 cluster 12
7 Perbandingan akurasi clustering dan Indeks Davies Bouldin 14 8 Confusion matrix panjang fragmen 200 bp dengan output 9 cluster 14
DAFTAR GAMBAR
1 Diagram alir penelitian 3
2 Ilustrasi pemetaan fragmen ke dalam matrix co-occurrence 4
3 Arsitektur SOM 6
4 Potongan fragmen hasil praproses data 9
5 Proses pemetaan sequence DNA menjadi matrix co-occurrence 10 6 Ilustrasi matrix co-occurrence yang telah dinormalisasi 10
7 Hasil ekstraksi fitur genus Clostridium 11
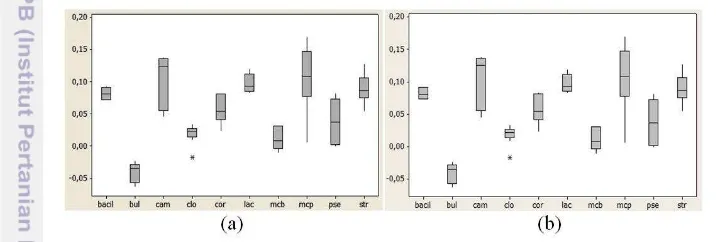
8 Boxplot fitur correlation pada panjang fragmen (a) 200 bp dan (b) 1
Kbp 13
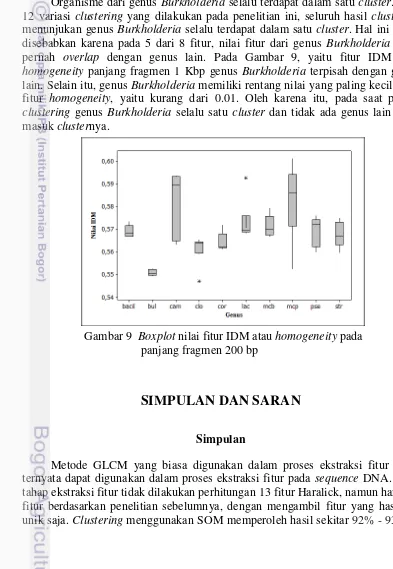
9 Boxplot nilai fitur IDM atau homogeneity pada panjang fragmen 200 bp 15
DAFTAR LAMPIRAN
1 Potongan hasil ekstraksi fitur panjang fragmen 200 bp pada 30
organisme 17
2 Confusion matrix hasil clustering 18
PENDAHULUAN
Latar Belakang
Ilmu pengetahuan di bidang bioinformatika berkembang semakin pesat. Bidang bioinformatika yang saat ini banyak dianalisis adalah genome dan metagenome. Metagenome adalah kumpulan data genome dari suatu komunitas mikrob yang terdapat di alam. Studi yang mempelajari metagenome disebut metagenomics. Studi metagenomics tidak memerlukan pure clonal culture atau dengan kata lain suatu sequence DNA tidak perlu dibiakkan pada preparat melalui proses laboratorium.
Fragmen-fragmen yang diperoleh dari pendekatan metagenomics masih memiliki kelemahan, yaitu memungkinkan bercampurnya suatu fragmen dengan fragmen lain. Hal ini dapat disebabkan oleh keanekaragaman mikroba dalam suatu ekosistem mikro yang sangat tinggi. Fragmen yang bercampur akan mempengaruhi proses perakitan fragmen. Untuk mengantisipasi kesalahan tersebut, proses binning dilakukan dengan metode supervised learning atau unsupervised learning. Fragmen-fragmen dari proses sequencing akan dikelompokkan berdasarkan level taksonomi tertentu, contohnya pada level genus (Meyerdierks dan Glockner 2010).
Salah satu penelitian terkait fragmen metagenome dilakukan McHardy et al. (2007). Penelitian tersebut menggunakan K-mers dan support vector machine (SVM) untuk ekstraksi fitur dan tahap klasifikasi. Penelitian McHardy et al. (2007) mencapai hasil yang cukup tinggi, yaitu antara 60%-90% untuk panjang ≥ 5 Kbp. Selanjutnya akurasi terus menurun jika menggunakan panjang fragmen yang pendek. Pada panjang < 3 Kbp, akurasi yang diperoleh sebesar 40% dan panjang 1 Kbp sebesar < 10%. Namun, karena menggunakan K-mers dengan k=5, waktu yang dibutuhkan dalam perhitungan kernel cukup lama. Selain itu, pemodelan menggunakan SVM cukup kompleks karena pada umumnya SVM digunakan untuk menyelesaikan masalah klasifikasi dengan dua kelas. Untuk menyelesaikan masalah dengan dua kelas atau lebih perlu digunakan pendekatan lain, salah satunya adalah pendekatan one versus rest. Oleh karena itu, pada penelitian ini menggunakan alternatif lain dalam ekstraksi fitur, yakni menggunakan metode Gray Level Co-occurrence Matrix (GLCM) dan metode Self Organizing Maps (SOM) untuk pengelompokkan.
2
Penelitian ini menggunakan data yang lebih banyak dari penelitian Aliefiya (2014) dan Dhira (2014), yaitu 100 organisme dari 10 genus yang berbeda. Penambahan data ini dilakukan untuk menguji kedua penelitian sebelumnya yang memiliki hasil sempurna, dengan jumlah data yang lebih banyak akan menghasilkan akurasi yang sama atau berbeda, terlepas dari perbedaan metode yang digunakan.
Pada tahap clustering, penelitian ini menggunakan metode SOM, salah satu metode clustering yang dikembangkan oleh Teuvo Kohonen dari Finlandia. SOM adalah metode yang efektif untuk memvisualisasikan data dengan dimensi tinggi (Kohonen 2001). Proses ekstraksi fitur menggunakan metode GLCM. GLCM adalah metode untuk menganalisis tekstur atau menemukan fitur pada citra (Haralick et al. 1973). Dengan menggunakan GLCM, data genome sebesar apapun akan diekstrak menjadi hanya beberapa fitur. Penelitian ini juga membahas pengaruh panjang fragmen terhadap hasil akurasi clustering.
Perumusan Masalah
Perumusan masalah yang menjadi bahan analisis penelitian ini adalah: 1 Bagaimana menerapkan metode GLCM untuk ekstraksi fitur pada penelitian
ini?
2 Bagaimana mengimplementasikan SOM sebagai teknik clustering pada penelitian ini?
3 Apakah panjang fragmen akan mempengaruhi hasil clustering? 4 Bagaimana karakteristik fragmen metagenome dari hasil clustering?
Tujuan Penelitian
Tujuan dari penelitian ini, yaitu:
1 Menerapkan metode GLCM pada proses ekstraksi fitur fragmen.
2 Melakukan clustering dengan metode SOM terhadap fragmen metagenome 3 Mengetahui pengaruh panjang fragmen terhadap hasil clustering.
4 Memperoleh karakteristik fragmen metagenome dari hasil clustering.
Manfaat Penelitian
3 Ruang Lingkup Penelitian
Ruang lingkup penelitian ini meliputi:
1 Data metagenome terdiri atas 100 organisme. Data tersebut dipilih dari genus Bacillus, Burkholderia, Campylobacter, Clostridium, Corynebacterium, Lactobacillus, Mycobacterium, Mycoplasma, Pseudomonas, dan Streptococcus.
2 Variasi panjang fragmen yang digunakan, yaitu 200 bp, 1 Kbp, 3 Kbp, dan 10 Kbp.
3 Orientasi sudut pada proses GLCM terbatas pada sudut 0° dengan jarak adalah 1.
4 Fragmen yang digunakan merupakan hasil simulasi Ilumina sequencer dari perangkat lunak MetaSim.
5 Sequence DNA direpresentasikan sebagai 4 karakter A, T, G, dan C yang mewakili basa nitrogen adenin, timin, guanin, dan sitosin.
METODE
Penelitian ini dimulai dengan tahap pengumpulan data, praproses data, ekstraksi fitur, proses clustering dan evaluasi. Ilustrasi metode penelitian dapat dilihat pada Gambar 1.
Pengambilan Data
Data yang digunakan dalam penelitian ini adalah data metagenome yang diunduh langsung dari situs National Center for Biotechnology Information (NCBI) sebanyak 100 organisme dari 10 genus. Data metagenome ini merupakan sequence DNA organisme dengan format fastA. Data ini selanjutnya diproses menggunakan perangkat lunak MetaSim. Alamat untuk mengunduh data ini yaitu ftp://ftp.ncbi.nih.gov/genomes/Bacteria/.
4
Praproses Data
Pada tahap praproses data, sequence DNA metagenome yang telah diunduh dari situs NCBI diuraikan menggunakan perangkat lunak MetaSim. Hasil dari penguraian sequence DNA menggunakan MetaSim adalah fragmen. Pada penelitian ini dipilih 10 organisme dari 10 genus. Panjang fragmen yang ditetapkan untuk setiap kali pengolahan yaitu 200 bp, 1 Kbp, 3 Kbp, dan 10 Kbp. Setelah dilakukan pemilihan data, diperoleh 100 fail fastA dari 10 genus yang berbeda. Namun terdapat dua organisme yang mengandung error. Organisme yang error tersebut mengandung nilai N. Pada data yang error, nilai N tidak dihitung pada tahap pembuatan matrix co-occurrence.
Ekstraksi Fitur
Pada tahap ini dilakukan ekstraksi fitur fragmen yang telah diuraikan menggunakan perangkat lunak MetaSim. Teknik ekstraksi fitur yang digunakan pada penelitian ini adalah Gray Level Co-occurrence Matrix (GLCM). Sequence DNA setiap fragmen akan dipetakan ke dalam sebuah matriks sesuai banyaknya fragmen dengan jarak 1 dan sudut orientasi 0°. Matriks inilah yang disebut dengan matrix co-occurrence. Ukuran matrix co-occurrence pada penelitian ini adalah 4x4 yang merupakan representasi dari matriks ACGT x ACGT. Ilustrasi membuat matrix co-occurrence dapat dilihat pada Gambar 2.
Setelah diperoleh matriks total dari tiap fragmen, perlu dilakukan normalisasi pada matriks tersebut. Normalisasi dilakukan dengan cara membagi nilai setiap elemen matriks dengan total nilai matriks, normalisasi ini bertujuan agar nilai elemen matriks jika dijumlahkan hasilnya 1. Selain itu, ekstraksi fitur sequences DNA hanya dapat dilakukan jika matrix co-occurrence telah dinormalisasi.
Menurut Haralick et al. (1973) terdapat berbagai fitur ciri tekstural yang dapat diekstraksi dari matrix co-occurrence. Beberapa dari ekstraksi ciri tersebut merupakan perhitungan untuk pengenalan karakteristik citra meliputi homogenitas, kontras dan keberadaan tekstur pada suatu citra. Dalam penelitian ini akan
5 dihitung 8 fitur dari 13 fitur yang diusulkan oleh Haralick. Beberapa perhitungan tersebut antara lain:
1 Angular Second Moment (ASM) atau disebut juga energy. Menunjukkan ukuran sifat homogenitas.
ASM = ∑ ∑ { , } (1) 2 Contrast. Menunjukkan ukuran penyebaran (Moment Inertia) elemen
matriks citra.
Con = ∑ | − | ,, (2) 3 Correlation. Menunjukkan ukuran ketergantungan linear derajat keabuan
citra.
Cor = ∑ ∑ −�� �−� � , (3)
4 Inverse Different Moment atau disebut juga homogeneity. Menunjukkan kehomogenan citra yang berderajat keabuan sejenis.
IDM = ∑ , � ,+| − | (4)
5 Entropy. Menunjukkan ukuran ketidakteraturan bentuk.
ENT = − ∑, , log , (5)
6 Sum Entropy
SENT = ∑=��� + � {�+ } (6) 7 Information Measures of Correlation 1
IMC = max − , (7)
8 Information Measures of Correlation 2
IMC = − exp [ . � − � ] / (8)
6
kompetisi, setiap input node dihitung jaraknya dengan bobot penelitian dengan menggunakan jarak Euclidean seperti formula di bawah ini:
= √∑ ( , − � ) =
Output yang memiliki jarak terkecil akan menjadi node pemenang. Output node pemenang pada setiap iterasi akan diperbarui bobotnya dengan menggunakan formula berikut:
� − = � + η � − �
Proses clustering dilakukan hingga mencapai kriteria pemberhentian. Kriteria pemberhentian dapat berupa pembatasan jumlah iterasi atau ketika jumlah
η = . Penelitian ini menggunakan 3 variasi kelas output, yaitu output 9 kelas, 10 kelas dan 11 kelas. Ilustrasi arsitektur SOM dapat dilihat pada Gambar 3. Hasil perhitungan Haralick akan menjadi input, sehingga terdapat 8 ciri untuk setiap organisme. Selanjutnya keseluruhan input akan dibandingkan kemiripannya dengan n output.
Analisis dan Evaluasi
Tahap evaluasi menggunakan 3 pendekatan. Pendekatan pertama menggunakan Indeks Davies Bouldin. Pendekatan pengukuran ini untuk memaksimalkan jarak inter-cluster di antara cluster Ci dan Cj dan pada waktu yang sama mencoba untuk meminimalkan jarak antar titik dalam sebuah cluster (Edward 2006). Formula Indeks Davies Bouldin adalah:
= ∑ � ≠ { �� + ��
|| − || }
=
7 Pada formula tersebut var(Ci) dan var(Cj) adalah jarak intra-cluster dan ||ci – cj|| adalah jarak inter-cluster. Hasil clustering yang optimal menurut Indeks Davies Bouldin adalah yang memiliki nilai Indeks Davies Bouldin paling kecil.
Evaluasi kedua menggunakan confusion matrix. Confusion matrix digunakan untuk melihat sebaran data hasil clustering. Confusion matrix adalah sebuah matriks yang menyimpan nilai aktual dan nilai prediksi dari klasifikasi atau clustering yang dilakukan (Kohavi dan Provost 1998). Data hasil clustering menggunakan SOM akan dibandingkan dengan genus metagenome pada data awal. Keluaran dari evaluasi menggunakan confusion matrix ini adalah berapa banyak data hasil cluster yang sesuai dengan data awal. Ilustrasi confusion matrix dapat dilihat pada Tabel 1.
Selain menggunakan confusion matrix, pada tahap evaluasi juga akan dilakukan perhitungan akurasi, spesifisitas dan sensitivitas untuk memperkuat hasil dari penelitian. Sensitivitas menyatakan hasil prediksi positif jika diberikan data aktual positif, sedangkan spesifisitas menyatakan hasil prediksi negatif jika diberikan data aktual negatif. Nilai akurasi didapatkan dengan menggunakan persamaan 9. Setiap genus dicari nilai akurasinya dengan menjumlahkan nilai true positive (TP) dan true negative (TN) lalu dibagi dengan nilai total dari true positive (TP), true negative (TN), false positive (FP) dan false negative (FN). Nilai akurasi untuk setiap panjang fragmen didapat dari total nilai akurasi setiap genus yang dibagi dengan n, dengan n adalah jumlah genus yang digunakan. Nilai spesifisitas dan sensitivitas didapatkan dengan menggunakan persamaan 10 dan persamaan 11. Nilai spesifisitas dan sensitivitas untuk setiap panjang fragmen didapatkan dari jumlah nilai spesifisitas dan sensitivitas 10 genus yang dirata-ratakan.
Spesifikasi perangkat keras yang digunakan dalam penelitian ini adalah prosesor Intel Core i3-380M 2.53 GHz, memori 4 GB, dan harddisk 320 GB. Adapun untuk perangkat lunak yang digunakan yaitu MetaSim 0.9.1 sebagai
8
perangkat lunak untuk membangkitkan sequence, Notepad++, Matlab R2008a, Microsoft Excel 2010, dan Weka 3.7.12 sebagai software alternatif untuk melakukan clustering.
HASIL DAN PEMBAHASAN
Pengambilan Data
Data yang digunakan pada penelitian ini adalah data metagenome yang diunduh dari situs NCBI dengan alamat ftp://.ncbi.nih.gov/genomes/Bacteria. Data tersebut terdiri atas 100 organisme dari 10 genus yang berbeda. Data ini selanjutnya akan diproses menggunakan perangkat lunak MetaSim pada tahap praproses data. Rincian data genus yang digunakan pada penelitian ini dapat dilihat pada Tabel 2.
Praproses Data
Data DNA yang telah diunduh dari situs NCBI dibangkitkan menggunakan perangkat lunak MetaSim. Panjang fragmen yang digunakanpada penelitian ini adalah 200 bp, 1 Kbp, 3 Kbp, dan 10 Kbp. Untuk menentukan banyak fragmen yang dibutuhkan pada setiap panjang fragmennya, perlu digunakan formula berikut:
Coverage= �.
n : banyak fragmen yang dibutuhkan. l : panjang fragmen yang dibutuhkan.
L : total rata-rata dari seluruh panjang mikroorganisme.
Tabel 2 Rincian panjang data genus yang digunakan Nomor Nama genus Total panjang
mikroorganisme
Jumlah organisme
1 Bacillus 47477478 10
2 Burkholderia 31648210 10 3 Campylobacterium 17612241 10
4 Clostridium 37650188 10
5 Corynebacterium 26786828 10 6 Lactobacillus 23652389 10 7 Mycobacterium 51574566 10
8 Mycoplasma 10392924 10
9 Pseudomonas 59316339 10
9 Coverage adalah rata-rata sekuens DNA yang merepresentasikan sebuah nukleotida dalam perakitan DNA tersebut. Coverage yang digunakan pada penelitian ini adalah 5. Nilai coverage tersebut digunakan karena terbilang cukup untuk merepresentasikan suatu mikroorganisme. Total rata-rata dari seluruh panjang mikroorganisme didapat dari seluruh panjang tiap mikroorganisme yang digunakan adalah 3273146. Dengan demikian untuk perbandingan panjang fragmen dan banyak fragmen yang dibutuhkan dapat dilihat pada Tabel 3.
Setelah didapatkan banyak fragmen, nilai dari panjang fragmen dan banyak fragmen digunakan untuk membangkitkan sequence melalui MetaSim. Contoh output dari MetaSim dapat dilihat pada Gambar 4. Gambar 4 adalah potongan fragmen dari organisme Lactobacillus fermentum CECT 5716 dengan panjang fragmen 200 bp yang telah dibangkitkan menggunakan perangkat lunak MetaSim. Namun, terdapat dua organisme yang mengandung error, yaitu pada genus Bacillus dan Campylobacter. Organisme yang error tersebut mengandung nilai N. Pada data yang error, nilai N tidak dihitung pada tahap pembuatan matrix co-occurrence.
Gambar 4 Potongan fragmen hasil praproses data Tabel 3 Perbandingan panjang fragmen dan
10
Ekstraksi Fitur
Output MetaSim berupa sequence DNA yang telah dipotong ke dalam beberapa variasi panjang fragmen, diproses untuk mendapatkan matrix co-occurrence. Matrix co-occurrence dihitung berdasarkan jarak dan sudut yang telah ditentukan. Sequence DNA tersebut dihitung pasangan antar elemennya dengan jarak 1 dan dengan sudut orientasi 0°. Penggunaan sudut 0° pada penelitian ini dikarenakan sequence DNA merupakan data 1 dimensi dengan ukuran 1xN dengan N adalah panjang sequence DNA yang digunakan. Jumlah dari banyaknya pasangan tersebut dimasukkan ke dalam matriks yang berukuran 4x4.
Gambar 5 adalah proses pemetaan sequence DNA menjadi matrix co-occurrence pada potongan sequence organisme Lactobacillus fermentum CECT 5716 dengan panjang fragmen 200 bp. Setiap karakter dipasangkan dengan karakter tetangganya kemudian dihitung jumlah kemunculan setiap pasangan karakter tersebut. Matriks yang berisi total kemunculan pasangan karakter disebut dengan matrix co-occurrence. Matrix co-occurrence yang telah didapat selanjutnya dilakukan proses normalisasi. Matrix co-occurrence yang telah dinormalisasi dari setiap organisme akan digunakan dalam perhitungan fitur Haralick. Ilustrasi matrix co-occurrence yang telah dinormalisasi dapat dilihat pada Gambar 6 yang merupakan matrix co-occurrence yang telah dinormalisasi dari organisme Lactobacillus fermentum CECT 5716 dengan panjang fragmen 200 bp.
Proses ekstraksi fitur menggunakan GLCM melibatkan 13 fitur Haralick. Namun, berdasarkan penelitian yang telah dilakukan Dhira (2014) dan Aliefiya
Gambar 5 Proses pemetaan sequence DNA menjadi matrix co-occurrence
11 (2014) terdapat beberapa fitur yang hasilnya serupa, terlihat dari boxplot 13 fitur Haralick. Fitur yang hasilnya sama direduksi sehingga hanya satu fitur saja yang digunakan. Hingga pada akhirnya hanya 8 fitur yang digunakan pada penelitian ini untuk mewakili 13 fitur Haralick. Fitur yang digunakan pada penelitian ini adalah Angular Second Moment, Contrast, Correlation, Entropy, Sum Entropy, Invers Different Momment, Information Measure of Correlation 1, dan Information Measure of Correlation 2. Potongan hasil ekstraksi fitur genus Clostridium pada panjang fragmen 1 Kbp dapat dilihat pada Gambar 7. Hasil ekstraksi fitur pada beberapa genus dengan panjang fragmen 200 bp dapat dilihat pada Lampiran 1.
Clustering
Fitur Haralick dari setiap organisme akan menjadi pencirinya masing-masing dalam proses clustering menggunakan SOM. Proses clustering dilakukan dengan bantuan perangkat lunak Weka. Terdapat 100 input yang berupa organisme. Setiap input memiliki 8 penciri. Pada tahap ini digunakan 3 variasi kelas output, yaitu output 9 cluster, 10 cluster, dan 11 cluster. Variasi banyaknya output digunakan untuk membandingkan output yang memiliki akurasi paling baik. Pemilihan tiga variasi tersebut berdasarkan trial and error yang telah dilakukan.
Analisis dan Evaluasi
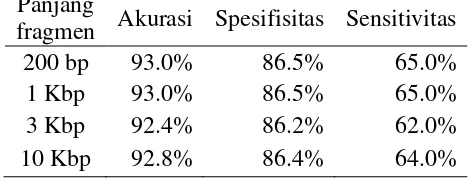
Dari proses clustering yang telah dilakukan, pada output 9 cluster nilai akurasi paling baik sebesar 93% yaitu pada panjang fragmen 200 bp dan 1 Kbp. Begitu pula pada nilai spesifisitas dan sensitivitas. Nilai tertinggi terdapat pada panjang fragmen 200 bp dan 1 Kbp. Pada Tabel 4 terlihat bahwa terdapat hubungan antara nilai akurasi, spesifisitas, dan sensitivitas. Ketika nilai akurasi tinggi, nilai spesifisitas dan sensitivitas juga tinggi. Bahkan saat terjadi penurunan nilai akurasi pada panjang fragmen 3 Kbp, nilai spesifisitas dan sensitivitas ikut turun.
12
Seperti yang terlihat pada Tabel 5, nilai spesifitas tertinggi terdapat pada panjang fragmen 10 Kbp, sama dengan nilai akurasi tertinggi. Saat nilai akurasi mengalami penurunan pada panjang fragmen 3 Kbp, nilai spesifitas juga ikut turun. Nilai sensitivitas cenderung tetap, hanya saja mengalami kenaikan pada panjang fragmen 10 Kbp.
Tabel 6 menyajikan secara keseluruhan nilai akurasi pada hasil clustering 11 cluster memiliki nilai yang sama, yaitu 93.6%. Namun, seperti yang terjadi pada hasil clustering output 9 dan 10 cluster, terjadi penurunan nilai akurasi pada panjang fragmen 3 Kbp. Pada panjang fragmen 10 Kbp nilai akurasi kembali naik menjadi 93.6% , sama dengan panjang fragmen 200 bp dan 1 Kbp. Begitu juga dengan nilai spesifisitas dan sensitivitas yang mengalami penurunan pada panjang fragmen 3 Kbp. Nilai spesifititas tertinggi terdapat pada panjang fragmen 200 bp dan 1 Kbp, sedangkan nilai sensitivitas tertinggi terdapat pada panjang fragmen 10 Kbp.
Jika dilihat secara keseluruhan, nilai akurasi memiliki rentang antara 92% -93.6%, nilai spesifitas berada pada rentang 85.9% - 87.4%, dan nilai sensitivitas berada pada rentang 61% - 65%. Terdapat penurunan nilai akurasi terutama pada panjang 3 Kbp dengan selisih kurang dari 1%. Seharusnya secara eksplisit,
Tabel 4 Hasil clustering pada output 9 cluster Panjang
fragmen Akurasi Spesifisitas Sensitivitas
200 bp 93.0% 86.5% 65.0%
1 Kbp 93.0% 86.5% 65.0%
3 Kbp 92.4% 86.2% 62.0%
10 Kbp 92.8% 86.4% 64.0%
Tabel 5 Hasil clustering pada output 10 cluster Panjang
fragmen Akurasi Spesifisitas Sensitivitas
200 bp 92.0% 85.9% 61.0%
1 Kbp 92.8% 86.7% 61.0%
3 Kbp 92.0% 85.9% 61.0%
10 Kbp 93.6% 87.4% 62.0%
Tabel 6 Hasil clustering pada output 11 cluster Panjang
fragmen Akurasi Spesifisitas Sensitivitas
200 bp 93.6% 87.3% 63.0%
1 Kbp 93.6% 87.3% 63.0%
3 Kbp 93.1% 86.9% 62.0%
13 semakin panjang fragmen yang digunakan maka nilai akurasi yang didapatkan lebih baik.
Salah satu faktor yang menyebabkan hal ini terjadi adalah proses agregasi pada saat ekstraksi fitur. Pada tahap ekstraksi fitur penelitian ini, nilai dari setiap fragmen diagregasi sehingga satu matrix co-occurrence didapatkan untuk satu organisme. Setelah matrix co-occurrence didapatkan, matriks tersebut dinormalisasi dan dilakukan proses perhitungan Haralick, sehingga setiap organisme hanya diwakili 8 fitur Haralick. Akan lebih baik jika setiap fragmen dihitung menggunakan fitur Haralick, karena nilai setiap fragmen memiliki informasi yang berbeda. Jika nilai fragmen tersebut diagregasi atau dijumlahkan menjadi satu matriks untuk setiap organisme, mengakibatkan informasi tersebut hilang sehingga mempengaruhi tahap clustering.
Dari Tabel 4, Tabel 5 dan Tabel 6 dapat dilihat bahwa terdapat beberapa panjang fragmen memiliki nilai akurasi, spesifitas dan sensitivitas yang sama. Jika mengacu pada Gambar 8 yang memuat boxplot nilai fitur correlation pada panjang fragmen 200 bp dan 1 Kbp, dapat disimpulkan bahwa kedua panjang fragmen memiliki pola yang sama dan nilai yang hampir sama. Hal ini dapat menyebabkan beberapa panjang fragmen memiliki hasil akurasi yang sama, karena pola tersebut terjadi pada beberapa fitur lain.
Setelah didapatkan hasil clustering, dilanjutkan dengan evaluasi hasil clustering menggunakan Indeks Davies Bouldin. Indeks Davies Bouldin paling baik terdapat pada panjang fragmen 200 bp dengan output 10 cluster, sedangkan yang paling buruk terdapat pada panjang fragmen 10 Kbp dengan output 10 cluster. Terdapat pola yang unik pada hasil Indeks Davies Bouldin, dimana nilai yang paling buruk selalu terdapat pada panjang fragmen 10 Kbp. Nilai akurasi yang sama bahkan memiliki nilai Indeks Davies Bouldin yang berbeda. Selain itu, nilai akurasi yang paling rendah memiliki nilai Indeks Davies Bouldin paling baik. Nilai Indeks Davies Bouldin pada panjang fragmen 10 Kbp selalu paling tinggi atau kurang optimal diantara panjang fragmen lain. Hal ini dapat disebabkan karena keragaman antar cluster semakin kecil. Nilai Indeks Davies Bouldin yang besar dapat disebabkan oleh nilai intra-cluster yang semakin besar atau nilai inter-cluster yang semakin kecil. Jika melihat boxplot fitur Haralick, terlihat bahwa jarak intra-cluster setiap genus tetap, sehingga dapat disumpulkan nilai Indeks Davies Bouldin pada panjang fragmen 10 Kbp lebih tinggi dari panjang fragmen lain disebabkan oleh nilai inter-cluster yang semakin kecil. Jika melihat hasil
14
Indeks Davies Bouldin pada Tabel 7, dapat disimpulkan bahwa nilai Indeks Davies Bouldin tidak berbanding lurus dengan akurasi clustering.
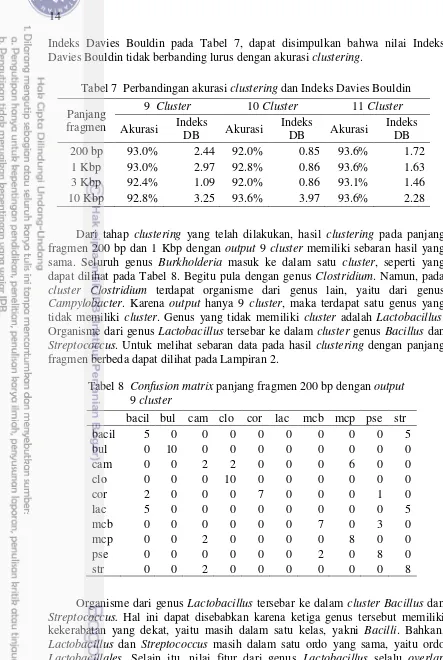
Dari tahap clustering yang telah dilakukan, hasil clustering pada panjang fragmen 200 bp dan 1 Kbp dengan output 9 cluster memiliki sebaran hasil yang sama. Seluruh genus Burkholderia masuk ke dalam satu cluster, seperti yang dapat dilihat pada Tabel 8. Begitu pula dengan genus Clostridium. Namun, pada cluster Clostridium terdapat organisme dari genus lain, yaitu dari genus Campylobacter. Karena output hanya 9 cluster, maka terdapat satu genus yang tidak memiliki cluster. Genus yang tidak memiliki cluster adalah Lactobacillus. Organisme dari genus Lactobacillus tersebar ke dalam cluster genus Bacillus dan Streptococcus. Untuk melihat sebaran data pada hasil clustering dengan panjang fragmen berbeda dapat dilihat pada Lampiran 2.
Organisme dari genus Lactobacillus tersebar ke dalam cluster Bacillus dan Streptococcus. Hal ini dapat disebabkan karena ketiga genus tersebut memiliki kekerabatan yang dekat, yaitu masih dalam satu kelas, yakni Bacilli. Bahkan, Lactobacillus dan Streptococcus masih dalam satu ordo yang sama, yaitu ordo Lactobacillales. Selain itu, nilai fitur dari genus Lactobacillus selalu overlap dengan genus Bacillus dan Streptococcus. Pada Lampiran 3 terlihat dari semua fitur yang digunakan pada penelitian ini, seluruh fitur menunjukkan ketiga genus tersebut memiliki rentang nilai fitur yang sama atau saling beririsan.
Tabel 7 Perbandingan akurasi clustering dan Indeks Davies Bouldin Panjang
15 Genus Lactobacillus tidak memiliki cluster pada hasil clustering output 9 cluster. Pada hasil clustering output 10 cluster genus Lactobacillus hanya memiliki cluster pada hasil clustering dengan panjang fragmen 200 bp dan 3 Kbp. Sisanya organisme dari genus Lactobacillus menyebar pada cluster Bacillus, Streptococcus dan Corynobacter. Pada hasil clustering output 11 cluster nilai akurasi pada genus Lactobacillus dapat dikatakan lebih baik. Genus Lactobacillus memiliki cluster pada setiap panjang fragmen. Saat panjang fragmen 200 bp, 1 Kbp dan 3 Kbp terdapat 5 organisme yang tercluster dengan benar. Hanya saja pada panjang fragmen 10 Kbp turun menjadi 4 organisme yang tercluster dengan benar.
Organisme dari genus Burkholderia selalu terdapat dalam satu cluster. Dari 12 variasi clustering yang dilakukan pada penelitian ini, seluruh hasil clustering menunjukan genus Burkholderia selalu terdapat dalam satu cluster. Hal ini dapat disebabkan karena pada 5 dari 8 fitur, nilai fitur dari genus Burkholderia tidak pernah overlap dengan genus lain. Pada Gambar 9, yaitu fitur IDM atau homogeneity panjang fragmen 1 Kbp genus Burkholderia terpisah dengan genus lain. Selain itu, genus Burkholderia memiliki rentang nilai yang paling kecil pada fitur homogeneity, yaitu kurang dari 0.01. Oleh karena itu, pada saat proses ternyata dapat digunakan dalam proses ekstraksi fitur pada sequence DNA. Pada tahap ekstraksi fitur tidak dilakukan perhitungan 13 fitur Haralick, namun hanya 8 fitur berdasarkan penelitian sebelumnya, dengan mengambil fitur yang hasilnya unik saja. Clustering menggunakan SOM memperoleh hasil sekitar 92% - 93.6%,
16
nilai spesifitas berada pada rentang 85.9% - 87.4%, dan nilai sensitivitas berada pada rentang 61% - 65%. Akurasi hasil clustering yang paling baik pada panjang fragmen 200 bp, 1 Kbp dan 3 Kbp dengan output 11 cluster. Dari hasil clustering tidak terlihat pengaruh panjang fragmen. Karena hasil clustering tidak menunjukan adanya pola yang tetap saat menggunakan panjang fragmen besar maupun kecil.
Genus Burkholderia menjadi genus yang paling konsisten karena dari hasil clustering empat variasi panjang fragmen yang berbeda, semua organisme dari genus Burkholderia selalu masuk dalam satu cluster. Genus Lactobacillus memiliki akurasi yang paling buruk diantara genus lain. Hal ini disebabkan nilai fitur dari Lactobacillus selalu overlap dengan dengan nilai fitur dari genus lain, terutama dengan genus Bacillus dan Streptococcus. Selain itu, genus Lactobacillus memiliki kekerabatan yang sama pada ordo dengan Bacillus dan Streptococcus.
Saran
Beberapa saran untuk penelitian berikutnya:
1 Menggunakan jarak GLCM yang beragam (jarak 1, 2, 3, dan 4). 2 Menghitung fitur GLCM pada setiap fragmen.
DAFTAR PUSTAKA
Aliefiya M. 2014. Klasifikasi fragmen metagenome menggunakan KNN dan PNN dengan ekstraksi fitur gray level co-occurrence matrix (GLCM) pada variasi jumlah fragmen [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Dhira M. 2014. Klasifikasi fragmen metagenome menggunakan KNN dan PNN dengan ekstraksi fitur gray level co-occurrence matrix (GLCM) pada variasi panjang fragmen [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Edward. 2006. Clustering menggunakan self organizing maps studi kasus:data PPMB IPB. [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Haralick MR, Shanmugan K, Dinstein I. 1973. Textural feature for image classification. IEEE Transactions on Systems, Man, and Cybernetics. 3(6):610-621. doi: 10.1109/tmsc.1973.4309314.
Kohavi R, Provost F. 1998. Machine Learning. Boston (US): Springer . Kohonen T. 2001. Self Organizing Maps: 3rd Edition. Berlin (DE): Springer. McHardy AC, Martin HG, Tsirigos A, Hugenholtz P, Rigoutsos I. 2007. Accurate
phylogonetic classification of variable-length DNA fragments. Nature Methods. 4(1):63-72. doi: 10.1038/nmeth976.
17 Lampiran 1 Potongan hasil ekstraksi fitur panjang fragmen 200 bp pada 30
18
Lampiran 2 Confusion matrix hasil clustering
Confusion matrix hasil clustering output 9 cluster panjang fragmen 200bp bacil bul cam clo cor lac mcb Mcp pse str
Confusion matrix hasil clustering output 9 cluster panjang fragmen 1Kbp bacil bul cam clo cor lac mcb Mcp pse str
19 Lanjutan
Confusion matrix hasil clustering output 9 cluster panjang fragmen 10Kbp bacil bul cam clo cor lac mcb mcp pse str
Confusion matrix hasil clustering output 10 cluster panjang fragmen 200bp bacil bul cam clo cor lac mcb mcp pse str
Confusion matrix hasil clustering output 10 cluster panjang fragmen 1Kbp
20
Lanjutan
Confusion matrix hasil clustering output 10 cluster panjang fragmen 3Kbp bacil bul cam clo cor lac mcb mcp pse str
Confusion matrix hasil clustering output 10 cluster panjang fragmen 10Kbp
bacil bul cam clo cor lac mcb mcp pse str
Confusion matrix hasil clustering output 11 cluster panjang fragmen 200bp
21 Lanjutan
Confusion matrix hasil clustering output 11 cluster panjang fragmen 1Kbp
bacil bul cam clo cor lac mcb mcp pse str
Confusion matrix hasil clustering output 11 cluster panjang fragmen 3Kbp
bacil bul cam clo cor lac mcb mcp pse str
Confusion matrix hasil clustering output 11 cluster panjang fragmen 10Kbp
22
Lampiran 3 Boxplot seluruh fitur untuk kesepuluh genus
Boxplot fitur angular second moment atau energy panjang fragmen 200bp
Boxplot fitur contrast panjang fragmen 200bp
23 Lanjutan
Boxplot fitur invers difference moment atau homogeneity panjang fragmen 200bp
Boxplot fitur entropy panjang fragmen 200bp
24
Lanjutan
Boxplot fitur information measures of correlation 1 panjang fragmen 200bp
Boxplot fitur information measures of correlation 2 panjang fragmen 200bp
25 Lanjutan
Boxplot fitur contrast panjang fragmen 1Kbp
Boxplot fitur correlation panjang fragmen 1Kbp
26
Lanjutan
Boxplot fitur entropy panjang fragmen 1Kbp
Boxplot fitur sum entropy panjang fragmen 1Kbp
27 Lanjutan
Boxplot fitur information measures of correlation 2 panjang fragmen 1Kbp
Boxplot fitur angular second moment atau energy pada panjang fragmen 3Kbp
28
Lanjutan
Boxplot fitur correlation pada panjang fragmen 3Kbp
Boxplot fitur invers difference moment atau homogeneity pada panjang fragmen 3Kbp
29 Lanjutan
Boxplot fitur sum entropy pada panjang fragmen 3Kbp
Boxplot fitur information measures of correlation 1 pada panjang fragmen 3Kbp
30
Lanjutan
Boxplot fitur angular second moment atau energy pada panjang fragmen 10Kbp
Boxplot fitur contrast pada panjang fragmen 10Kbp
31 Lanjutan
Boxplot fitur invers difference moment atau homogeneity pada panjang fragmen 10Kbp
Boxplot fitur entropy pada panjang fragmen 10Kbp
32
Lanjutan
Boxplot fitur information measures of correlation 1 pada panjang fragmen 10Kbp
33
RIWAYAT HIDUP
Penulis dilahirkan di Bogor pada tanggal 4 Januari 1993. Penulis merupakan anak pertama dari tiga bersaudara pasangan Untung Wahyudi dan Dhiny Kartika. Penulis mengenyam pendidikan dasar di SD Negeri Polisi 1 Kota Bogor (1999-2005). Penulis melanjutkan pendidikan menengah pertama di SMP Negeri 1 Kota Bogor (2005-2008). Kemudian, penulis melanjutkan pendidikan menengah atas di SMA Negeri 1 Bogor (2008-2011). Penulis berkesempatan melanjutkan studi di Institut Pertanian Bogor melalui jalur Seleksi Nasional Masuk Perguruan Tinggi Negeri (SNMPTN) Undangan di Departemen Ilmu Komputer, Fakultas Matematika dan Ilmu Pengetahuan Alam.