418 ISSN: 2302-8467 Jurnal Riset Sains dan Kimia Terapan PRIMER SPESIFIK GEN HEAT SHOCK PROTEIN 70 SEBAGAI ALAT DETEKSI BAKTERI S. TYPHI
DENGAN POLYMERASE CHAIN REACTION Muktiningsih Nurjayadi, Irma Ratna Kartika dan Nilam Pratiwi
Department of Chemistry, Faculty of Mathematis and Science,Universitas Negeri Jakarta. Jalan Pemuda No. 10, Rawamangun 13220, Jakarta, Indonesia
Corresponding author: muktiningsih@unj.ac.id ABSTRAK
Salmonella typhi merupakan bakteri patogen penyebab demam tifoid pada manusia. Mekanisme patogenesitas Salmonella typhi merupakan sistem yang kompleks dan diatur oleh sejumlah gen dan faktor-faktor virulen. Salah satu gen-nya adalah dnaK yang mengatur sintesis Heat Shock Protein 70 (HSP70). Penelitian ini bertujuan untuk merancang dan menguji primer HSP70 Salmonella typhi dalam mendeteksi bakteri Salmonella typhi dari biakan murni. Metode yang digunakan adalah metode eksperimen dengan teknik Polymerase Chain Reaction (PCR). Penelitian ini telah berhasil merancang dan mensintesis tiga pasang primer yaitu primer HSP70-1, primer HSP70-2, dan primer HSP70-3, hal ini ditunjukan dengan hasil elektroforesis, bahwa ketiga primer tersebut dapat mengamplifikasi daerah gen HSP70 Salmonella typhi berukuran 302 bp, 273 bp, dan 213 bp. Hasil penelitian ini dapat digunakan untuk studi pengembangan metode deteksi bakteri S. typhi berbasis HSP sebagai bakteri penyebab demam tifoid dan metode deteksi bakteri Salmonella typhi sebagai bakteri penyebab keracunan makan yang spesifik dengan metode PCR. ABSTRACT
Salmonella typhi is a pathogen causing typhoid fever in humans. Pathogenicity mechanism of Salmonella typhi is complex and regulated by a number of genes and virulence factors. DnaK is the one that regulated the synthesis of Heat Shock Protein 70 (HSP 70). This research purpose to designing and testing the primer pairs in the detection of Salmonella typhi bacteria from pure cultures. The method used was experimental by Polymerase Chain Reaction (PCR). This research has successfully designed three primaries namely HSP70-1, HSP70-2, and HSP70-3 were in electrophoresis results showed that the three primary can amplify HS 70 of Salmonella typhi with sized 302 base pairs, 273 base pairs and 213 base pairs. The results could be used to study the development of Salmonella typhi bacteria detection method based HSP as the bacteria which cause typhoid fever and Salmonella bacteria detection method as the bacteria that cause food poisoning by specific PCR method.
Keywords: Salmonella typhi, HSP70, detection methods, PCR.
1.
Introduction/PendahuluanDemam tifoid merupakan penyakit sistemik yang disebabkan oleh infeksi bakteri Salmonella typhi. Terdapat sekitar 17 juta kasus demam tifoid di seluruh dunia. Kasus ini terjadi di seluruh provinsi Indonesia dengan insidensi
kematian mencapai 180/100.000
penduduk/tahun [13]. Diagnosis klinis demam tifoid menjadi tantangan medis karena kemiripannya dengan penyakit demam lainnya [4]. Oleh sebab itu, metode laboratorium yang cepat dan sensitif untuk diagnosis demam tifoid sangat penting. Meskipun telah diterapkan beberapa tes serologi, namun tes yang terbukti sangat sensitif dan spesifik untuk mendeteksi demam tifoid masih terus dikembangkan. Salah satu metode alternatif untuk mendeteksi
demam tifoid adalah Polymerase Chain Reaction (PCR). Metode genetik yang didasarkan pada PCR cukup sensitif untuk mendeteksi 100 CFU/ml bakteri, bahkan pada saat yang sama dapat mendeteksi sel-sel mati setelah pengolahan karena sensitivitasnya yang tinggi [5]. Penelitian sebelumnya juga telah membuktikan bahwa dengan metode PCR menggunakan pasangan primer Fim-C-S. typhi mampu mendeteksi 1.295 x 10-39 μg/mL DNA kromosom dan memiliki spesifisitas yang tinggi [8, 10].
Penelitian sebelumnya telah berhasil mengamplifikasi daerah variabel gen HSP 70 S. typhi berukuran 1,9 kb [11]. Namun sebagai alat deteksi molekuler daerah variabel gen HSP70 S. typhi tersebut masih memiliki tingkat
Jurnal Riset Sains dan Kimia Terapan ISSN: 2302-8467 419 homologi yang tinggi bila dibandingkan dengan
bakteri lain. Oleh sebab itu, dilakukan pencarian daerah variabel gen HSP70 S. typhi yang memiliki tingkat homologi rendah terhadap bakteri lain serta dilakukan perancangan dan pengujian primer spesifiknya dengan metode PCR sehingga metode deteksi yang dikembangkan ini diharapkan dapat mendeteksi keberadaan bakteri S. typhi baik dari biakan murni maupun dari sampel pasien atau sampel pangan. Hasil penelitian ini diharapkan dapat berkontribusi dalam pengembangan ilmu pengertahuan dan pengembangan metode deteksi penyebab penyakit typhus yang pada akhirnya dapat meningkatkan kualitas hidup masyarakat Indonesia.
2. Experimental/Metodologi Penelitian
2.1. Isolasi DNA dan KarakterisasinyaBakteri S. typhi, S. typhimurium, V. cholerae, E. coli, dan S. dysentriae dalam medium padat miring diperoleh dari Laboratorium Mikrobiologi FK-UI di kultur dalam media LB Broth suhu 37°C [1]. Media selektif SSA digunakan untuk memeriksa hasil kultur S. typhi dan S. typhimurium [2]. DNA bakteri diisolasi menggunakan kit wizard purifikasi DNA (Promega). Konsentrasi DNA hasil isolasi diukur pada nilai rasio A260/A280 dengan nanodrop (Maestro Gen) [9].
Gambar 1. Hasil Elektroforesis DNA (1) Marker 1 kb (2) DNA Genom S. typhi, (3) S. typhimurim, (4) E. coli, (5) Vibrio cholerae, dan (6) Shigella dysentriae.
Elektroforesis dilakukan pada tegangan 100 volt, selama 30 menit, dan pewarnaan dengan ethidium
bromide
2.2. Perancangan dan Sintesis Primer Gen HSP 70 S. typhi
Sekuen data gen HSP 70 S. typhi diperoleh dari GenBank NCBI [10]. Daerah yang menunjukkan persentase “Query Cover 0%” bila dibandingkan dengan bakteri S. typhimurium, V. cholerae, E. coli, dan S. dysentriae adalah parameter yang dipilih untuk menjadi DNA template yang kemudian akan dirancang primernya. Parameter perancangan primer dipilih karena daerah tersebut tidak memiliki kesamaan dengan gen HSP 70 S. typhi sehingga pada saat hasil akhir proses PCR hanya gen HSP 70 S. typhi 70 yang dapat teramplifikasi dan akan menghasilkan pita saat pengecekan dengan menggunakan elektroforesis gel agarosa [3]. Primer hasil rancangan selanjutnya di sintesis di Laboratorium komersial Macrogen, Inc – Korea [8]. Selanjutnya primer hasil sintesis akan diuji spesifisitasnya.
Gambar 2. Optimasi Suhu Annealing Primer HSP70-1 pada Template DNA S. typhi. Optimasi pada
suhu annealing (1) Marker DNA (2) 48°C, (3) 50°C, (4) 52°C, (5) 54°C, (6) 56°C, (7) 58°C, (8) 60°C, (9) 62°C (10) 64°C, (11) 66°C, (12) 68°C, (13) 70°C, (14) 72°C, (15) Kontrol (-). Elektroforesis dilakukan pada tegangan 100 volt, selama 30 menit, dan pewarnaan
dengan ethidium bromide. Primer Hasil Rancangan
Primer hasil rancangan yaitu primer HSP70-1 dengan urutan (5’-GGA TCC GAC GCA TGG CTT GAT GTG AA-3’) (forward) dan 5’–AAG CTT AAA GTA CCA CCA CC GAG GTC-3’ (reverse) dengan lokasi penempelan lokus STY0012 11891-12192, dan menghasilkan amplikon berukuran 302 pasang basa (pb); Primer
420 ISSN: 2302-8467 Jurnal Riset Sains dan Kimia Terapan HSP70-2 dengan urutan 5’-GGA TCC AAG ACT
TCG ATA CCC GCC TG -3’ (forward) dan 5’–AAG CTT TCA GCG GCT CGA TAG AAC G-3’(reverse) dengan lokasi penempelan lokus STY0012 12282-12536, dengan menghasilkan amplikon berukuran 273 pasang basa (pb); Primer HSP70-3 dengan urutan 5’-GGA TCC TAC TGC TGC TGG ACG TTA CC-3’ (forward) dan 5’–AAG CTT CCA GGT TGA ACT GAC CCA GA -3’(reverse) dengan lokasi penempelan lokus STY0012 12759-12852, dengan menghasilkan amplikon berukuran 213 pasang basa (pb).
2.3. Optimasi Kondisi Suhu Annealing PCR dengan Template S. typhi dan Karakterisasinya
Diuji beberapa kondisi suhu annealing dengan rentang suhu 46 °C - 72 °C dengan metode. Proses PCR dilakukan selama30 siklus, dengan kondisi: denaturasi pada suhu 95oC selama 1 menit, anneling pada rentang suhu 46 °C - 72 °C selama 1 menit, pemanjangan rantai pada suhu 72°C selama 1 menit, serta pemantapan hasil reaksi pada akhir siklus pada suhu 72°C selama 4 menit. Proses pelaksaaan PCR menggunakan mesin PCR gradient (Biometra-T Advanced) [5]. Hasil PCR dikarakterisasi dengan elektroforesis gel agarosa 1.5 % menggunakan pewarna ethidium bromide [11].
2.4. Uji Spesifikasi Primer dengan Metode PCR dan Karakterisasinya
Ketiga primer hasil rancangan diuji spesifikasinya dengan menggunakan PCR dengan sampel hasil isolasi DNA. Hasil PCR dikarakterisasi dengan elektroforesis agarosa 1.5 % dan dibandingkan. Primer dikatakan spesifik jika hanya dapat mengamplifikasi DNA genom S. typhi, tetapi tidak dapat mengamplifikasi bakteri uji lainnya [1].
3. Results
and discussion/Hasil Dan Pembahasan3.1. Isolasi DNA Genom dan Karakterisasinya
DNA genom bakteri berasal dari pembiakan bakteri dalam medium padat miring diperoleh dari laboratorium Mikrobiologi UI yaitu bakteri S. typhi, S. typhimurium, V. cholerae, E. coli, dan S. dysentriae. Selanjutnya bakteri-bakteri tersebut dibiakkan di Laboratorium Biokimia-Bioteknologi FMIPA UNJ menggunakan media LB Broth. Pellet bakteri yang telah dipisahkan ditambah dengan Nuclei Lysis Solution untuk memecahkan inti sel, lalu ditambahkan dengan RNase Solution untuk menghancurkan RNA. Protein Precipitation Solution ditambahkan setelahnya untuk mengendapkan protein [8].
DNA yang telah terpisah dengan protein lalu diendapkan dan dipekatkan dengan penambahan isopropanol. Pellet DNA yang telah didapat kemudian ditambahkan dengan etanol 70% untuk mencuci benang DNA yang telah didapat. Setelah etanol dibuang, pellet DNA yang didapat dikering anginkan agar benar-benar bebas dari etanol karena dapat mengganggu kerja enzim pada proses amplifikasi. Setelah itu pellet DNA ditambahkan dengan DNA Rehydration Solution sehingga didapat larutan DNA genom. Hasil isolasi kemudian disimpan pada suhu sekitar -4˚C yang selanjutnya akan digunakan pada amplifikasi genom bakteri [8].
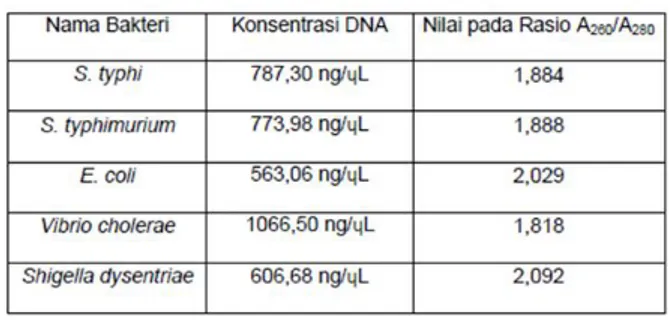
Kemurnian DNA genom hasil isolasi selanjutnya diukur dengan spektrofotometer Nanodrop. Hasil pengukuran disajikan pada tabel 1.
Tabel 1. Hasil Pengukuran Konsentrasi DNA dengan Nanodrop
Pita DNA menyerap spektrum UV pada ʎ= 260nm, sedangkan kontaminan protein dan fenol menyerap UV pada ʎ= 270nm. Oleh
Jurnal Riset Sains dan Kimia Terapan ISSN: 2302-8467 421 karena itu kemurnian sampel DNA dapat diukur
dengan menghitung nilai absorbansi pada ʎ 260nm dan ʎ 280nm. Nilai kemurnian DNA yang baik adalah yang berada pada rentang nilai 1,80-2,00. Jadi sampel DNA hasil isolasi memiliki kemurnian yang baik [9].
Gambar 3. Optimasi Suhu Annealing Primer HSP70-2 pada template DNA S. typhi. Optimasi pada suhu annealing (1) Marker DNA (2) 48°C, (3) 50°C, (4) 52°C, (5) 54°C, (6) 56°C, (7) 58°C, (8) 60°C, (9) 62°C (10) 64°C, (11) 66°C, (12) 68°C, (13) 70°C, (14) 72°C,
(15) Kontrol (-). Elektroforesis dilakukan pada tegangan 100 volt, selama 30 menit, dan pewarnaan
dengan ethidium bromide.
Hasil isolasi DNA genom bakteri-bakteri selanjutnya dikarakterisasi untuk memastikan keberhasilan isolasi dengan elektroforesis gel agarosa 1,5% pada arus 100 Volt selama 30 menit dengan menggunakan pewarna etidium bromida[11].. Karakterisasi menggunakan DNA Ladder 1 kb sebagai marker DNA, dan DNA genom bakteri S. typhi, S. typhimurim, E. coli, Vibrio cholerae dan Shigella dysentriae hasil isolasi sebagai sampel. Dokumentasi hasil karakterisasi dengan elektroforesis agarosa disajikan pada gambar 1.
Berdasarkan data hasil elektroforesis pada gambar 1 diketahui bahwa DNA genom bakteri-bakteri telah berhasil diisolasi. Hal ini ditandai dengan munculnya pita-pita DNA pada gel. Pita DNA semua bakteri terletak pada posisi yang lebih tinggi dari garis pertama marker DNA atau lebih dari 1,5 kb. Munculnya pita DNA pada ukuran >1,5kb menunjukkan bahwa hasil ini sesuai dengan hasil pencarian informasi tentang ukuran DNA genom bakteri dari beberapa genom database. Hasil analisis data
dari beberapa literatur menunjukkan bahwa ukuran DNA genom bakteri S. typhi sebesar 4,81 mega base pairs (mb), S. typhimurium sebesar 4,86 mb, E. coli sebesar 4,8 mb, Vibrio cholerae sebesar 4,03 mb, dan Shigella dysentriae sebesar 4,34 mb [12].
3.2. Optimasi Kondisi Suhu Annealing PCR Primer Hasil Rancangan
Optimasi suhu annealing menunjukkan bahwa semua suhu annealing 46°C-72°C menghasilkan pita spesifik secara jelas pada template DNA genom bakteri S. typhi dari hasil amplifikasi dengan ketiga primer. Foto hasil optimasi suhu anneling menggunakan pasangan primer HSP70-1, HSP70-2, dan HSP70-3 dengan masing-masing amplikon berukuran 302 pasang basa (pb), 273 pb dan 213pb disajikan pada gambar 2, gambar 3 dan gambar 4.
Gambar 4. Optimasi Suhu Annealing Primer HSP70-3 pada template DNA S. typhi. Optimasi pada
suhu annealing (1) Marker DNA (2) 48°C, (3) 50°C, (4) 52°C, (5) 54°C, (6) 56°C, (7) 58°C, (8) 60°C, (9) 62°C (10) 64°C, (11) 66°C, (12) 68°C, (13) 70°C, (14) 72°C, (15) Kontrol (-). Elektroforesis dilakukan pada tegangan 100 volt, selama 30 menit, dan pewarnaan
dengan ethidium bromide.
Hasil optimasi suhu annealing menggunakan pasangan primer HSP70-1 menunjukkan adanya pita DNA yang tebal pada rentang suhu 46-72oC, berdasarkan hal tersebut maka optimasi pasangan primer lainnya dilakukan dengan rentang suhu annealing 46°C-72°C. Seperti diketahui bahwa suhu annealing yang ideal berkaitan dengan Tm primer yang
422 ISSN: 2302-8467 Jurnal Riset Sains dan Kimia Terapan Gambar 5. Hasil Amplifikasi dengan Pasangan
Primer Gen HSP70-1. (1) Marker DNA, (2) Hasil PCR DNA Genom S. typhi, (3) Hasil PCR DNA genom S. typhimurium, (4) Hasil PCR DNA genom
E. Coli, (5) Hasil PCR DNA genom Shigella dysentriae, dan (6) Hasil PCR DNA genom Vibrio cholera, (7) Kontrol (-). Elektroforesis dilakukan digunakan untuk proses PCR. Suhu annealing yang digunakan dapat dihitung berdasarkan (Tm –5) °C sampai
dengan (Tm + 5) °C. Secara
teoritis Tm primer dapat dihitung dengan menggunakan rumus [2(A+T)+4(C+G)]. Sehingga rentang suhu 46°C - 72°C merupakan suhu yang ideal untuk proses annealing primer [3], sehingga dari hasil yang diperoleh dapat disimpulkan bahwa proses amplifikasi genom S. typhi dari biakan murni telah berhasil dilakukan pada rentang suhu tersebut. Selanjutnya akan dipilih satu suhu yang paling optimum untuk tahap uji selanjutnya sesuai standar pengembangan alat deteksi molekular.
3.3. Uji Spesifikasi Primer HSP70-1, HSP70-2 dan HSP70-3 dengan Metode PCR dan Karakterisasinya
Uji spesifisitas bertujuan untuk mengecek pasangan primer berfungsi secara selektif dan dapat membedakan template bakteri S. typhi dengan template bakteri lainnya. Tahap ini merupakan salah satu syarat standar uji untuk alat deteksi molekular yang harus dilakukan. Primer dapat dikatakan spesifik apabila hanya dapat mengamplifikasi DNA genom biakan
murni S. Typhi. Uji spesifisitas pada penelitian ini dilakukan pada suhu annealing 72°C. Hal ini didasarkan pada amplikon yang dihasilkan pada suhu tersebut memiliki ketebaln pita yang optimum.
Gambar 6.Hasil Amplifikasi dengan Pasangan Primer Gen HSP70-2. (1) Marker DNA, (2) Hasil PCR DNA Genom S. typhi, (3) Hasil PCR DNA Genom S. typhimurium, (4) Hasil PCR DNA Genom E. Coli, (5) Hasil PCR DNA Genom Shigella dysentriae, dan (6) Hasil PCR DNA Genom Vibrio cholera, (7) Kontrol (-). Elektroforesis dilakukan pada tegangan 100 volt, selama 30 menit, dan pewarnaan dengan ethidium bromide.
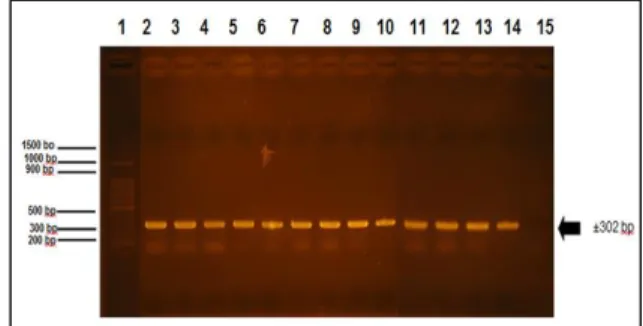
Amplikon berukuran ± 302 bp diperoleh dari DNA genom bakteri S typhi dan S typhimurium pada pasangan primer HSP70-1. Hal ini ditunjukkan dengan munculnya pita DNA pada hasil karakterisasi dengan elektroforesis gel agarosa 2%. Pita DNA tunggal (tanpa smear) untuk hasil PCR dengan template DNA genom S. typhi dan S typhimurium hasil isolasi muncul pada daerah ± 0,3 kb atau 302 pb sesuai dengan ukuran pita DNA S. Typhi yang dihitung secara in silico. Untuk template DNA bakteri uji lainnya yaitu E. Coli, Shigella dysentriae dan Vibrio cholera tidak dihasilkan pita pada ukuran tertentu.
Hasil uji spesfisitas dengan pasangan primer HSP70-1 menunjukkan bahwa telah berhasil di rancang pasangan primer HSP-70 yang dapat membedakan template bakteri S. Typhi dengan bakteri E. Coli, Shigella dysentriae dan Vibrio cholera sebagi template,
Jurnal Riset Sains dan Kimia Terapan ISSN: 2302-8467 423 namun belum dapat membedakan bakteri S.
Typhi dengan S. Typhimurium. Hasil analisis literature menunjukkan bahwa urutan genome bakteri S. Typhi dan S. Typhimurium memiliki kesamaan 98% [6,7,8, 10], sehingga pada fragmen gen HSP-70 diperkirakan juga memiliki tingkat homologi atau kesamaan urutan sekuens nukletioda yang tinggi. Foto hasil elektroforesis uji spesifisitas pasangan primer HSP70-1 disajikan pada gambar 5.
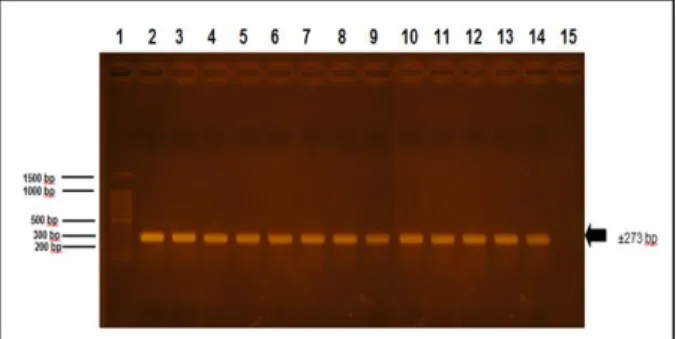
Amplikon berukuran ± 273 bp diperoleh pada DNA genom bakteri S typhi dan S typhimurium pada pasangan primer HSP70-2. Hal ini ditunjukkan dengan munculnya pita DNA pada hasil karakterisasi dengan elektroforesis gel agarosa 2%. Pita DNA tunggal (tanpa smear) untuk hasil PCR dengan template DNA genom S. typhi dan S typhimurium hasil isolasi muncul pada daerah ± 0,3 kb atau 273 pb. Sama dengan pasangan primer HSP70-1, maka hasil uji spesfisitas dengan pasangan primer HSP70-2 menunjukkan bahwa telah berhasil di rancang pasangan primer HSP-70 yang dapat membedakan template bakteri S. Typhi dengan bakteri E. Coli, Shigella dysentriae dan Vibrio cholera sebagi template, namun belum dapat membedakan bakteri S. Typhi dengan S. Typhimurium. Hasil analisis literature menunjukkan bahwa urutan Genome bakteri S. Typhi dan S. Typhimurium memiliki kesamaan 98% [8, 10], sehingga pada fragmen gen HSP-70 diperkirakan juga memiliki tingkat homologi atau kesamaan urutan sekuens nuknletida yang tinggi. Foto hasil elektroforesis uji spesifisitas pasangan primer HSP70-2 disajikan pada gambar 6.
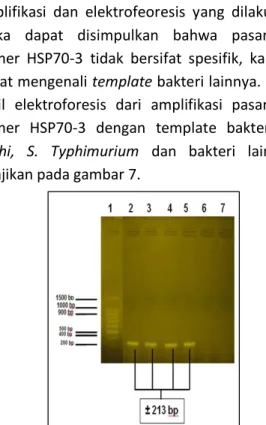
Amplikon berukuran ± 213 bp diperoleh pada DNA genom bakteri S typhi, S typhimurium, E. Coli dan Shigella dysentriae pada hasil apmlifikasi dengan pasangan primer HSP70-3. Hal ini ditunjukkan dengan munculnya pita DNA pada hasil karakterisasi dengan elektroforesis gel agarosa 2%. Pita DNA tunggal (tanpa smear) untuk hasil PCR dengan template DNA genom S typhi, S typhimurium, E. Coli dan
Shigella dysentriae hasil isolasi muncul pada daerah ± 0,2 kb atau 213 pb. Berdasarkan hasil amplifikasi dan elektrofeoresis yang dilakukan maka dapat disimpulkan bahwa pasangan primer HSP70-3 tidak bersifat spesifik, karena dapat mengenali template bakteri lainnya. Foto hasil elektroforesis dari amplifikasi pasangan primer HSP70-3 dengan template bakteri S. Typhi, S. Typhimurium dan bakteri lainnya disajikan pada gambar 7.
Gambar 7. Hasil Amplifikasi dengan Pasangan Primer Gen HSP70-3. (1) Marker DNA, (2) Hasil PCR DNA Genom S. typhi, (3) Hasil PCR DNA Genom S. typhimurium, (4) Hasil PCR DNA Genom E. Coli, (5) Hasil PCR DNA Genom Shigella dysentriae, dan (6) Hasil PCR DNA Genom Vibrio cholera, (7) Kontrol (-). Elektroforesis dilakukan pada tegangan 100 volt, selama 30 menit, dan pewarnaan dengan ethidium bromide.
4.
KesimpulanPenelitian ini telah berhasil menemukan dua pasangan primer spesifik yang dapat mengamplifikasi gen HSP 70 S typhi dan S. typhimurium berukuran 302 bp dan 273 bp dengan sampel DNA genom dari biakan murni, menemukan satu pasangan primer yang dapat mengamplifikasi gen HSP 70 S. typhi, S. typhimurium, E coli, dan Shigella dysentriae berukuran 213 bp dengan sampel DNA genom dari biakan murni.Hasil penelitian ini dapat digunakan untuk studi pengembangan metode deteksi bakteri S. typhi berbasis Heat Shock Protein sebagai bakteri penyebab demam tifoid dan metode deteksi bakteri S. typhimurium dan bakteri lainnya sebagai bakteri penyebab keracunan makan (food poisoning) yang spesifik
424 ISSN: 2302-8467 Jurnal Riset Sains dan Kimia Terapan dengan metode Polymerase Chain Reaction
(PCR). Uji tahap selanjutnya yang berkaitan dengan sensitivitas dan uji terhadap sampel pasien sedang dalam proses pengembangan.
5.
Ucapan Terima KasihUcapan terimakasih kami haturkan kepada DP2M Kemenristek Dikti yang telah mendanai penelitian ini dengan skim Hibah Penelitian
Unggulan Perguruan Tinggi (PUPT 2016), Rektor UNJ, Ketua lembaga Penelitian UNJ, Prodi Kimia, Fakultas Matematika dan Ilmu Pengetahuan Alam, Universitas Negeri Jakarta, dan PT Sentra Biodinamika Sains atas segala saran dan bantuannya, serta Tim salmonella 2010, 2011, dan 2012 yang dengan semangat tinggi, dan kolaborasinya sehingga penelitian dapatberjalanlancar.
Daftar Pustaka
[1]
Hatta, M., and Smits, L. H. Journal of Tropical Medicine Hygine, Vol. 76, No. 1, (2007) 139-143.[2]
Hardy Diagnostics. 2015. Instruction for Use SS Agar. https://catalog. hardydiagnostics.com/cp_prod/Content/hugo/SSAgar.html, Accessed 12 Juli, at 23.28 GMT+7 (2015).[3]
Innis, M. A. and Gelfand, H. 1990. Optimization of PCR: PCR Protocols. A Guide to Methods and Applications, 30. Academic Press Inc, California.[4]
Khan, S., Harish, N. B G., Menezes, A. G., Acharya, S. N., and Parija, C. S. Indian Journal of Medicine Res, Vol. 136, (2012) 850-854.[5]
McPherson, M., and Moller, S. PCR Second Edition. Vol. 2 (2006) 1-20.[6]
Muktiningsih, Dewi, F. K., Sukmawati, D. S., Sandra, R. N., dan Wulansari, F., (2009). Laporan Penelitian. Lembaga Penelitian UNJ, Jakarta.[7]
Muktiningsih Nurjayadi1, Fera Kurniadewi, Irma Ratna Kartika, Suhartono, Restu Nidia Sandra, Fitri Wulandari, Taufan Ardianto, Dalia Sukmawati, and Wibowo Mangunwardoyo, AIP Conf. Proc. 1729, (2015) 020060-1–020060-4; doi: 10.1063/1.4946963.[8]
Muktiningsih, Fera Kurniadewi, Imanuelle Orchidea R.P., Jurnal Kimia Dan Pendidikan Kimia (JKPK), Vol.1, No.1(2016) 32-40.[9]
NanoDrop Technologies Inc. 2007. 260/280 and 260/230 Ratios. Wilmington-Delaware, USA.[10]
NCBI. Basic Local Alignment Search Tools, http://blast.ncbi.nlm.nih.gov/Blast.cgi, Accessed 24 December, at 22.20 GMT+7 (2011).[11]
Pal, G. 2013. Principles of Electrophoresis. http://elte.prompt.hu/sites/de fault/files/tananyagok/practical_biochemistry/ch07.html, Accessed 2 November at 15.03 GMT+7 (2014).[12]
Parkhil,l. J., Dougan, G., James, K. D., Thomson, N. R., Pickard, D., Wain, J., Churcher, C., Mungall, K. L., Bentley, S. D., Holden, M.T., Sebaihia, M., Baker, S., Basham, D., Brooks, K., Chillingworth, T., Connerton, P., Cronin, A., Davis, P., Davies, R. M., Dowd, L., White, N., Farrar, J., Feltwell, T., Hamlin , N., Haque, A., Hien, T. T., Holroyd, S., Jagels, K., Krogh, A., Larsen, T. S., Leather, S., Moule, S., O'Gaora, P., Parry, C., Quail, M., Rutherford, K., Simmonds, M., Skelton, J., Stevens, K., Whitehead, S., and Barrell, B. G. 2001. Complete genome sequence of a multiple drug resistant Salmonella enterica serovar Typhi CT18. Journal of Nature, Vol. 413, 848-852.[13]
WHO. 2014. Focus on Typhoid Feverhttp://www.wpro.who.int/philippines/typhoon_haiyan/media/Typhoid_fever.pdf?ua=1, Accessed 11 November, at 15.03 GMT+7 (2014).