(Ananas comosus (L.) Merr.) HASIL PERSILANGAN
BERDASARKAN PENANDA MORFOLOGI DAN RAPD
Abstrak
Tiga puluh genotipe nenas hibrida dan tetuanya ‘Queen’ dan ‘Smooth Cayenne’ dari Pusat Kajian Buah-buahan Tropika (PKBT) IPB telah dianalisis keragamannya berdasarkan penanda morfologi dan RAPD. Analisis ini dilakukan
untuk mengevaluasi jarak genetik dan pola hubungan antar hibrida tersebut. Di dalam studi ini dilakukan pengamatan terhadap 20 karakter vegetatif-generatif
dan digunakan 12 primer (RAPD dan E-RAPD). Hasil analisis similaritas menunjukkan rentang koefisien kemiripan hibrida berbeda berdasarkan penanda morfologi (0.06-0.86) dan RAPD (0.38- 0.81). Hasil analisis gerombol terhadap 30 hibrida dapat dibentuk dua kelompok hibrida, baik berdasarkan penanda morfologi dan RAPD yang dipisahkan masing-masing pada koefisien kemiripan 0.30 dan 0.61. Meskipun sama-sama dapat dibentuk 2 kelompok, kedua penanda menunjukkan pola pengelompokan yang berbeda. Masing-masing penanda menunjukkan pengelompokan genotipe yang tidak sesuai dengan komposisi karakternya. Hasil analisis gerombol berdasarkan data gabungan menghasilkan koefisien kemiripan berkisar 0.47-0.76 dan dendrogramnya yang dipisahkan pada koefisien kemiripan 0.47 dapat dibentuk 2 kelompok utama. Pengelompokan berdasarkan data gabungan juga menunjukkan pengelompokan hibrida yang tidak sesuai dengan komposisi karakternya. Berdasarkan hasil analisis komparasi antar matrik koefisien kemiripan dari kedua penanda menunjukkan nilai korelasi yang lemah (r = 0.7769). Hasil analisis korelasi parsial antara karakter kualitatif dan profil RAPD (primer) pada taraf kepercayaan 95-99%, menunjukkan bahwa fragmen DNA OPE7 baris 5 yang cenderung berasosiasi dengan karakter kelopak warna putih perak dan buah kuning emas. Sedangkan fragmen SBH8 pita 3 yang cenderung berasosiasi dengan karakter duri tidak merata. Penanda-penanda yang digunakan ini, belum cukup optimum sebagai penanda untuk mengelompokkan berdasarkan genetiknya, dan belum dapat merefleksikan keanekaragaman genetik yang sesungguhnya. Pemilihan karakter morfologi dan primer polimorfik yang tepat mungkin dapat mengelompokkan hibrida ke dalam kelompok genotipik yang sesungguhnya.
Kata kunci : morfologi, RAPD, E-RAPD, genotipe, similaritas, gerombol Abstract
Genetic diversity analysis were conducted on 30 hybrids Ananas comosus (L).Merr and two parentals, which are collected by The Centre for Tropical and Fruit Studies based on phenotypic performance and RAPD markers to evaluation
the genetic distance and relationship pattern among these accessions. Twenty phenotypic traits and 12 (RAPD and E-RAPD) primers had been used in this study. Similarity analysis showed different similarity coefficient of phenotypic
(0.06-0.86) and RAPD (0.38- 0.81) markers. Two primary groups could be distingiused at similarity coeficient of 0.30 dan 0.61, respectively, for phenotypic
coefficient ranged from 0.47-0.76, which fall into Two primary groups at similarity of coefficient 0.52, however, dendrogram, as shown in phenotypic and
RAPD, showed any accession excluded from their genomic groups. In general, clustering analysis result based on RAPD seem to be closer to the clustering pattern of its genomic composition than the other clusters. Base on comparison
analysis between phenotypic and RAPD similarity matrix indicated a poor fit correlation (r=0.7769). Partial correlation analysis between qualitative traits and
DNA profile, at 95-99% confidence revealed OPE7 line 5 tends to associated with silvery-white of sepal colour and golden yellow of fruit color when ripe. DNA SBH8 line 3, significally, associated with spines occur irregularly along both margin of distribution of spines. These markers were not optimum to classify
30 hybrids and its parental, and neither reflects their real genetic diversity yet. Choosing more appropriate traits and polymorphic primers may be able to cluster
the hybrids and its parental into the genomic composition groups precisely. Key words : phenotypic, RAPD, E-RAPD,, genotipe, similarity, clustering
Pendahuluan
Studi keragaman genetik tanaman memegang peranan penting dalam upaya meningkatkan efektivitas dalam program pemuliaan tanaman. Informasi mengenai
keragaman genetik tanaman nenas hasil persilangan sangat diperlukan dalam kegiatan seleksi.
Keberhasilan dalam program pemuliaan tanaman nenas sangat ditentukan dari keberhasilan dalam memilih invidu-individu tanaman plus yang digunakan sebagai sumber material genetik yang memiliki keragaman genetik total tinggi. Beberapa metode sering digunakan pada studi keragaman genetik tanaman nenas
seperti analisis karakter morfologi, fisiologi dan biokimia serta beberapa marker atau penanda molekuler. Dari sejumlah metode tersebut, jenis penanda yang didasarkan pada tingkat molekuler DNA dianggap cukup handal dan lebih dapat
dipercaya karena tidak dipengaruhi oleh faktor lingkungan.
Analisis keragaman pada tanaman nenas oleh beberapa peneliti didasarkan pada penanda morfologi, penanda isozim dan penanda molekuler. Hadiati (2002) telah melakukan analisis kekerabatan pada 24 nomor aksesi nenas dengan mempergunakan penanda morfologi. Hasil yang diperoleh menunjukkan bahwa dengan derajat kemiripan 0.84 persen dari 24 nomor aksesi yang dipergunakan dapat dikelompokkan menjadi sembilan kelompok. Hadiati dan Sukmajaya (2002) memperoleh empat kelompok kekerabatan dari 30 aksesi nenas yang dianalisis berdasarkan penanda isozim pada derajat kemiripan 0.63. Duval et al., (2001)
dengan menggunakan RFLP berhasil membuktikan bahwa Ananas comosus dengan spesies lainnya, seperti Pseudananas sagenarius mempunyai polimorfisme yang tinggi, yaitu 58.7 persen, sedangkan Ananas lucidus, Ananas ananassoides dan Ananas parguazensis relatif homogen. Ruas et al., (2001) melalui penanda RAPD telah memperoleh koefisien kemiripan rata-rata aksesi Ananas comosus sebesar 0.85, sementara Ananas lucidus sebesar 0.75. Popluechai et al., (2007) berhasil mengelompokkan tiga kelompok kultivar nenas di Thailand melalui penanda RAPD dengan kemiripan 0.64 hingga 0.96. Cecilia et al., ( 2005) dengan penanda AFLP memperoleh koefisien kemiripan genetik sebesar 0.55 hingga 0.97 dari 100 aksesi, 48 aksesi dari Ananas comosus dan 14 aksesi dari spesies yang ada.
Metode RAPD (random amplified polymorphic DNA) merupakan salah satu metode yang akhir-akhir ini banyak digunakan dalam analisis keragaman genetik tanaman, karena relatif lebih cepat dan lebih mudah. Selain itu, untuk keperluan analisis keragaman, teknik RAPD cukup potensial karena mampu menghasilkan karakter yang tidak terbatas jumlahnya. Liu dan Furnier (1993) melaporkan bahwa penggunaan analisis RAPD selalu memperlihatkan keragaman
yang tinggi dibanding isozim dan RFLP.
Beberapa alasan penting lainnya sehingga memilih teknik RAPD ini, yaitu (1) tidak diperlukan pengetahuan latar belakang genom yang dipelajari, (2) secara cepat hasil RAPD dapat diperoleh terutama jika dibandingkan dengan analisis RFLP yang memerlukan banyak tahapan, dan (3) beberapa jenis atau set universal primer acak yang umum secara komersial telah tersedia dan dapat digunakan untuk analisis genomik pada hampir semua jenis organisme (Wels dan McCleland, 1990; William et al., 1990). Teknik ini telah banyak membantu pemulia tanaman seperti seleksi dan evaluasi keragaman genetik nenas.
Penelitian ini bertujuan untuk mengetahui keragaman fenotipik dan genotipik nenas hibrida berdasarkan penanda morfologi dan penanda RAPD melalui analisis similaritas, analisis gerombol, dan analisis komponen utama.
Bahan dan Metode Waktu dan Tempat
Penelitian dimulai Januari 2005 sampai Desember 2007 yang terdiri dari dua bagian yaitu observasi fenotipik dan observasi genotipik. Observasi fenotipik dilaksanakan di Kebun Percobaan Pasir Kuda PKBT IPB. Observasi genotipik yang meliputi isolasi, pemurniaan, penetapan kuantitas DNA, seleksi primer dan reaksi amplifikasi dilaksanakan di Laboratorium Molekuler PKBT Kampus IPB Baranangsiang Bogor dan visualisasi hasil elektroforesis dilaksanakan di Laboratorium Biologi Molekuler Pusat Penelitian Bioteknologi PAU IPB, Kampus Darmaga, Bogor.
Material Tanaman
Material tanaman yang digunakan adalah 30 tanaman nenas hibrida dan sepasang tetuanya JBBMQH6 (Queen) x JBSMSC3 (Smooth Cayenne) yang
berasal dari Kebun Percobaan Pasir Kuda PKBT IPB. Metode Penelitian
Penelitian terdiri atas dua bagian, yaitu observasi fenotipik di lapang dan observasi genotipik di laboratorium. Observasi fenotipik dilakukan dengan mengamati penampilan morfologi tanaman dan observasi genotipik melalui analisis pola pita DNA dengan menggunakan teknik RAPD.
Pelaksanaan Penelitian dan Pengamatan Observasi Fenotipik
Observasi fenotipik nenas dimulai dengan penanaman hibrida di lapang, kemudian pemeliharaan dan pengamatan, serta analisis data.
Teknik pengamatan pada 21 karakter morfologi dilakukan dengan menggunakan panduan deskriptor nenas (IBPGR, 1991). Pengamatan dilakukan terhadap kedudukan daun, warna daun, kehadiran duri pada daun, distribusi duri,
kekakuan duri, warna duri, warna sepal, dan warna petal. Pada saat panen dilakukan pengamatan terhadap bentuk permukaan buah, warna buah sebelum matang, warna buah setelah matang, bentuk permukaan mahkota, orientasi daun
mahkota, warna daun mahkota, kehadiran duri pada daun mahkota, orientasi spiral, aroma luar buah, warna daging buah, tekstur daging buah, dan profil mata
Observasi Genotipik
Observasi genotipik dilakukan dengan menggunakan analisis pola pita DNA berdasarkan teknik RAPD. Tahapan pelaksanaan terdiri dari isolasi, pemurniaan, penetapan kuantitas DNA, reaksi amplifikasi dan seleksi primer.
Isolasi DNA
Isolasi DNA dilakukan mengikuti metode CTAB Doyle dan Doyle (1987). Bahan yang dianalisis adalah daun muda (bagian pangkal) dari hasil persilangan sebanyak 0.5 g dan dipotong kecil-kecil. Potongan daun dimasukkan ke dalam mortal, lalu ditambah PVPP dan nitrogen cair, kemudian digerus sampai halus. Hasil gerusan dimasukkan ke dalam tabung eppendorf yang telah berisi 600 µl larutan buffer ekstrak CTAB (100 mM Tris-HCl pH 8.0, 1.4 M NaCl, 20 mM
ethylene diamine extraacetic acid (EDTA) pH 8.0, 2% (m/v) cetyltrimethylamonium Bromide (CTAB), dan 0,2% β-mercapto-ethanol), campuran dikocok dan dipanaskan dalam waterbath selama 30 menit pada suhu
65oC (setiap 10 menit campuran dibolak-balik). Campuran dibiarkan hingga mencapai suhu kamar lalu ditambahkan 600 µl larutan khloroform-isoamil alkohol
(24:1) dan dikocok. Sentrifuse campuran pada suhu ruang dengan kecepatan 12000 rpm selama 15 menit sehingga terbentuk dua fase cair. Fase cair bagian
atas (supernatan) dituang ke dalam tabung baru, kemudian ditambahkan isopropanol dingin dengan volume yang sama dan disimpan dalam frezer selama
semalam. Campuran dicairkan pada suhu kamar dan disentrifuse dengan kecepatan 12.000 rpm selama 15 menit. Cairan dibuang dengan hati-hati dan pellet ditambah etanol 70% dingin sebanyak 100 µl, kemudian disentrifuse dengan
kecepatan 12.000 rpm selama 5 menit. Cairan dibuang dan pellet dikeringkan dengan cara membalikkan tabung. Pellet yang telah kering ditambahkan 100 µl air
bebas ion dan dikocok sampai larut. Pemurniaan DNA
Pemurniaan DNA dengan menggunakan metode Sambrook et al. (1989). Larutan DNA ditambah 1 µl RNAase dan dibiarkan pada suhu kamar selama 2 jam. Selanjutnya tambahkan fenol kloroform isoamilalkohol dingin sebanyak 100
µL dan disentrifuse dengan kecepatan 12.000 rpm selama 15 menit. Supernatan dipipet ke dalam tabung baru dan ditambahkan kloroform isoamilalkohol dengan
volume sama, kemudian disentrifuse dengan kecepatan 12.000 rpm selama 15 menit. Supernatan dipipet ke dalam tabung baru dan ditambahkan natrium asetat 3
M pH 5.2 sebanyak 1/10 volume dan isopropanol dingin sebanyak 2.5 volume. Larutan dikocok hingga homogen dan disimpan dalam freezer semalam. Larutan disentrifuse dengan kecepatan 12.000 rpm selama 15 menit, pellet yang diperoleh
ditambah etanol 70% sebanyak 100 µl dan disentrifuse dengan kecepatan 12.000 rpm selama 5 menit. Cairan dibuang dan pellet dikeringkan dengan cara membalikkan tabung. Pellet yang telah kering ditambahkan 100 µl air bebas ion.
Penetapan kuantitas DNA
Penetapan kualitas DNA diestimasi melalui elektroforesis dan dibandingkan dengan standar DNA lamda. Sebanyak 5 µl masing-masing larutan
DNA yang diperoleh dicampur dengan 1 µl loading dye (10:2) dimasukkan ke dalam sumur gel agarose 1% dan di running pada bak elektroforesis selama 30 menit dengan tegangan 100 volt. Gel yang telah dielektroforesis direndam dalam
larutan ethium bromida 1% selama 15-20 menit, dibilas dengan aquades, dan selanjutnya pita DNA hasil isolasi divisualisasi pada UV transiluminator, dan
dipotret dengan kamera digital. Konsentrasi DNA ditentukan dengan membandingkan ketebalan DNA sampel dengan DNA lamda. DNA cetakan kemudian diencerkan sampai konsentrasi 25 ng dan siap digunakan untuk reaksi
amplifikasi.
Reaksi amplifikasi dan elektroforesis
Amplifikasi DNA nenas dilakukan menurut metode Williams et al. (1990). Reaksi amplifikasi dilakukan menggunakan microtube volume 0.5 nl yang berisi 25 µl campuran larutan yang terdiri atas: 12.5 µl Go tag mix, 10.5 µl air bebas ion
(ion free), 1 µl primer acak, dan 1 µl DNA. Volume akhir campuran reaksi amplifikasi adalah 25 µl.
Selanjutnya tabung-tabung tersebut dimasukkan ke dalam blok mesin PCR (Applied Biosystem Thermal Cycler version 2.00), yang diprogramkan dengan
tahapan sebagai berikut :
Tahap I. Pre-PCR : 94oC selama 4 menit sebanyak satu siklus;
Tahap II. PCR : Denaturasi 94oC selama 30 detik, annealing (penempelan primer) pada suhu 36oC selama 1 menit; dan extention
No. Primer 5’ to 3’ No. Primer 5’ to 3’ 1 2 3 4 5 6 OPE-07 OPE-11 SBR-04 SBR-08 SBN-05 SBN-13 AGATGCAGCC GAGTCTCAGG AATCGGGCTG GTGACGTAGG ACTGAACGCC AGCGTCACTC 7 8 9 10 11 12 SBH-02 SBH-07 SBH-08 SOB-01 SOB-02 SOB-03 TCGGACGTGA GGAAGTCGCC ACCTCAGCTC AGATGCAGCCG AGATGCAGCCA AGATGCAGCCT (perpanjangan) pada suhu 72oC selama 1 menit sebanyak
40 siklus;
Tahap III Post PCR : Perpanjangan akhir pada suhu 72oC selama 5 menit sebanyak satu siklus.
Setelah reaksi amplifikasi berakhir produk amplifikasi diberi loading dye, dan selanjutnya dielektroforesis pada 1.2% gel agarose dalam larutan TBE 1x.
Elektroforesis dilakukan selama 120 menit pada tegangan 60 volt pada suhu ruang. Pengamatan pita hasil amplifikasi dilakukan menggunakan alat
dokumentasi gel (gel dok) dan direkam ke dalam disket. Seleksi primer
Seleksi primer bertujuan untuk menyeleksi primer yang dapat menghasilkan produk amplifikasi, yang dilakukan terhadap 40 jenis primer RAPD dan 4 primer
e-RAPD dengan menggunakan DNA cetakan dari satu sampel tanaman hibrida yang memiliki stok DNA yang cukup. Seleksi primer dilakukan melalui reaksi amplifikasi dan elektroforesis. Primer yang memberikan hasil amplifikasi yang polimorfik berupa pita DNA yang jelas dan tajam, akan dipilih untuk digunakan
dalam analisis selanjutnya.
Sebanyak sembilan primer RAPD dan tiga primer e-RAPD hasil seleksi terhadap 44 primer telah diketahui dapat memberikan amplifikasi yang polimorfis
pada tanaman nenas. Hasil disajikan pada Tabel 8.
Tabel 8. Primer-primer RAPD dan E-RAPD hasil seleksi yang digunakan dalam penelitian
Analisis Data Data Fenotipik
Data fenotipik yang diperoleh dianalisis menggunakan program NTSYS-pc (Numerical Taxonomy and Multivariate Analysis) versi 2.02 (Rohlf, 1993).
(Similarity for Qualitative Data) pada program NTSYS-pc versi 2.02 dan dihitung berdasarkan rumus Nei dan Li (1979) atau koefisien Dice (S) yaitu :
S= 2nab/(na+ nb);
Keterangan: S = Koefisien kemiripan a dan b = dua individu yang dibandingkan
nab = jumlah subkarakter yang sama posisinya baik pada individu a maupun b
na = jumlah subkarakter pada individu a nb = jumlah subkarakter pada individu b
Hasil skoring data biner morfologi disajikan pada Lampiran 3. Untuk analisis gerombol (Clustering analysis) dipilih metode Sequential, Agglomerative,
Hierarchical, and Nested (SAHN)-UPGMA (Unweighted pair-group method, arithmetic average) pada program NTSYS-pc versi 2.02. Untuk analisis komponen utama (Principal Componen Analysis) digunakan prosedure analisis
Ordination dalam program NTSYS-pc versi 2.02.
Untuk kebutuhan analisis, data fenotipik terlebih dahulu diubah ke dalam bentuk data biner. Setiap karakter dibagi ke dalam sub karakter yang memungkinkan. Sub karakter yang terdapat pada genotipe tersebut diberi nilai 1 dan yang tidak tampak diberi nilai 0. Penetapan kriteria sub karakter berdasarkan
pedoman “Descriptors for pineapple” diterbitkan oleh International Board for Plant Genetic Resources (IBPGR, 1991). Pembagian karakter menjadi sub
karakter dan data binernya disajikan pada Lampiran 3.
Selanjutnya untuk mengetahui korelasi antar karakter fenotipik dilakukan analisis korelasi melalui program NTSYS-pc versi 2.02 dengan menggunakan
analisis perbandingan (Comparison) fungsi MXCOMP. Data Genotipik
Data genotipik yang diperoleh dari visualiasi hasil RAPD adalah pola pita DNA dengan ukuran tertentu. Ukuran potongan DNA genom dilakukan dengan membandingkannya dengan berat molekul standar 1 kb DNA Ladder. Perbedaan
antar tanaman ditunjukkan oleh jumlah pita dan jarak migrasinya. Apabila tidak terdapat perbedaan antara pola pita DNA tanaman berarti tidak terdapat keragaman genetik dan apabila sebaliknya berarti terdapat keragaman genetik. Untuk kebutuhan analisis, penilaian (skoring) dilakukan terhadap pita-pita tegas
dan tipis secara konsisten. Pita-pita yang dimiliki bersama diberi nilai skor 1 (ada), dan jika tidak diberi skor 0. Hasil skoring dalam bentuk data biner disajikan
pada Lampiran 4. Analisis similaritas, analisis gerombol dan analisis komponen utama pada data RAPD digunakan prosedur yang sama dengan analisis data
fenotipik.
Matriks kemiripan genotipik dihitung berdasarkan koefisien Dice dengan rumus :
S= 2nab/(na+nb)
Keterangan: S = Koefisien kemiripan a dan b = dua individu yang dibandingkan
nab = jumlah pita DNA yang sama posisinya baik pada individu a maupun b
na = jumlah pita DNA pada individu a
nb = jumlah pita DNA pada individu b
Selanjutnya korelasi antar primer dihitung melalui analisis perbandingan fungsi MXCOMP.
Data Gabungan
Analisis similaritas, analisis gerombol dan analisis komponen utama pada data RAPD digunakan prosedur yang sama pada analisis data fenotipik dan data
genotipik.
Untuk mengetahui tingkat keselarasan antara penampilan fenotipik dan pola pita RAPD, matriks kemiripan fenotipik dan matriks kemiripan genetik dibandingkan melalui uji korelasi fungsi MXCOMP pada program NTSYS-pc
versi 2.1 (Rohlf, 1993). Nilai korelasi Spearman dihitung berdasarkan rumus berikut (Gasperz, 1995): ) 1 ( 2 2
1
− Σ−
=
=
n n d jk jk iy
x
ρ
Keterangan : di : selisih setiap pasangan rank yang berkaitan dengan pasangan data (Xi, Yi)
n : banyaknya pasangan rank
Korelasi antara pasangan dua matriks yang diuji dengan statistik Z Mantel (Mantel, 1967 dalam Beer et al. 1993) sebagai berikut :
jk jk
j k x y
Keterangan :
x
jk:
elemen baris matrik ke j dan kolom ke k dari X:
jk
y
elemen baris matrik ke j dan kolom ke k dari YNilai Z kemudian ditransformasi melalui Z mantel dan diperoleh nilai korelasi sebagai berikut :
jk jk
j k x y
Z =
∑ ∑
Keterangan : xjk : hasil transformasi
x
jkyjk : hasil transformasi
y
jkHipotesis : H0 : nilai xjk tidak berkorelasi dengan yjk H1 : nilai xjk berkorelasi dengan yjk Dasar pengambilan keputusan :
1. Jika probabilitas > 0.05, maka H0 diterima 2. Jika probabilitas < 0.05, maka H0 ditolak 3. Jika t hitung < t(n-2): α/2, maka H0 diterima 4. Jika t hitung > t(n-2): α/2, maka H0 ditolak
Keselarasan pengelompokan ditentukan dari kriteria goodness of fit, berdasarkan nilai korelasi menurut Rohlf (1993) yaitu : sangat sesuai (r > 0.9 ),
sesuai (0.8 < r ≤ 0.9), tidak sesuai (0.7 ≤ r ≤ 0.8), sangat tidak sesuai (r < 0.7). Analisis Korelasi antara Karakter Kualitatif dan Primer
Analisis korelasi untuk melihat tingkat kecenderungan terpaut antara karakter kualitatif dan DNA hasil amplifikasi masing-masing primer, dilakukan
dengan menggabungkan data kualitatif morfologi dengan data DNA. Analisis dilakukan berdasarkan prosedur analisis similaritas dalam NTSYS-pc versi 2.02, kemudian dilanjutkan dengan analisis korelasi Pearson dalam MINITAB release 14. Hasil analisis korelasi diuji tingkat signifikasinya menggunakan uji t-student
pada taraf kepercayaan 95-99%. H a s i l
Penanda Fenotipik Analisis Kemiripan
Matriks koefisien kemiripan morfologi antara 30 hibrida dan kedua tetuanya diturunkan dari matriks simqual menunjukkan rentang nilai kemiripan berkisar antara 0.06-0.86 (Lampiran 5). Nilai koefisien kemiripan fenotipik (KF) tertinggi yaitu 0.86 diperoleh pada hibrida H05 dengan H06, yang memiliki perbedaan
pada duduk daun. Nilai KF terendah diperoleh hibrida H03 dengan H17 yaitu 0.06, yang memiliki perbedaan pada karakter duduk daun, warna daun, keberadaan duri pada daun, warna duri daun, warna kelopak, bentuk buah, warna buah sebelum matang, warna buah setelah matang, bentuk mahkota, duduk daun mahkota, warna daun mahkota, duri pada daun mahkota dan warna daging buah.
Hibrida H21 memiliki koefisien kemiripan fenotipik tertinggi sebesar 0.55 dengan tetua JBBMQH6 (T1) yang memiliki kesamaan pada karakter duduk daun, warna daun, kekakuan duri, warna kelopak, warna petal, bentuk buah, duduk daun mahkota, warna daun mahkota, karakter mahkota dan aroma buah. Hibrida H16 memiliki koefisien kemiripan terendah sebesar 0.15 dengan tetua JBBMQH6 (T1) yang memiliki kesamaan pada karakter duduk daun, warna petal dan karakter mahkota.
Hibrida H14 dan H19 memiliki koefisien tertinggi sebesar 0.70 dengan tetua JBSMSC3 (T2) yang dimiliki kesamaan pada karakter warna daun, keberadaan duri pada daun, warna duri, kekakuan duri, warna kelopak, warna petal, warna buah setelah matang, duduk daun mahkota, karakter mahkota, warna daging buah dan permukaan kulit buah. Hibrida H07 memiliki koefisien kemiripan terendah sebesar 0.20 dengan tetua JBSMSC3 (T2) yang memiliki kesamaan pada karakter duduk daun, warna daun, warna petal dan warna buah sesudah matang.
Nilai koefisien kemiripan yang tingginya antara hibrida nomor H14 dan H19 dengan H17 dengan JBSMSC3 (T2) menunjukkan terjadi segregasi beberapa karakter dari tetua jantan (T2) ke dalam dua hibrida tersebut. Adanya segregasi pada turunan hasil persilangan berarti terjadi keragaman genetik yang selanjutnya perlu diseleksi dan dievaluasi sesuai dengan kebutuhannya (Poespodarsono, 1988).
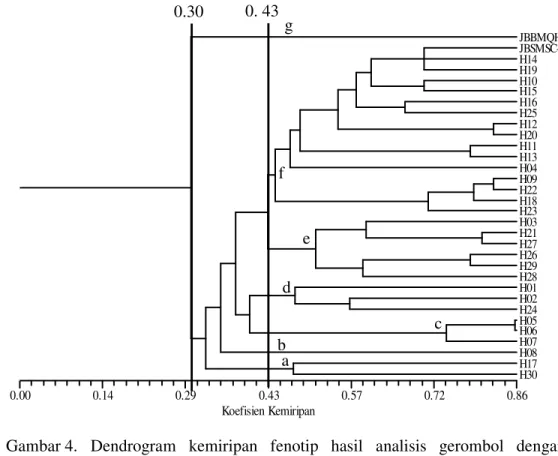
Analisis Gerombol
Analisis gerombol terhadap 87 subkarakter morfologi menghasilkan dendrogram dengan koefisien kemiripan berkisar antara 30–86% atau terdapat keragaman morfologi sebesar 14-70% (Gambar 4). Pada koefisien kemiripan 30% dapat dibentuk dua kelompok besar dimana tetua JBBMQH6 memisah dari turunannya (semua hibrida).
Gambar 4. Dendrogram kemiripan fenotip hasil analisis gerombol dengan metode pengelompokan UPGMA berdasarkan 87 subkarakter morfologi.
Pada koefisien kemiripan 43% terbentuk 7 subkelompok yaitu, kelompok pertama (a) hibrida H17 dan H30 yang memiliki kesamaan pada karakter warna daun, warna duri, warna kelopak, warna buah sebelum dan sesudah matang, warna daun mahkota, aroma buah, warna daging buah dan tekstur daging buah. Kelompok kedua (b) hibrida H08, kelompok ketiga (c) hibrida H05, H06 dan H07 yang memiliki kesamaan pada karakter warna daun, keberadaan duri pada daun, warna petal, bentuk buah, warna buah sebelum dan sesudah matang, kedudukan daun mahkota, duri pada mahkota, karakter mahkota, orientasi spiral, warna daging buah, tekstur dan mata buah, kelompok keempat (d) hibrida H01, H04 dan H24 yang memiliki kesamaan pada karakter warna petal, orienatasi spiral, aroma buah dan warna daging buah. Kelompok kelima (e) hibrida H03, H21, H26, H27, H28, dan H29 yang memiliki kesamaan pada karakter warna kelopak, warna petal dan tekstur daging buah.
Kelompok hibrida keenam (f) hibrida H04, H09, H10, H11, H12, H13, H14, H15, H16, H18, H19, H20, H22, H23 dan H25 yang memiliki kesamaan pada
1 2 h b c d e f g a Koefisien Kemiripan 0.00 0.14 0.29 0.43 0.57 0.72 0.86 JBBMQH6 JBSMSC-3 H14 H19 H10 H15 H16 H25 H12 H20 H11 H13 H04 H09 H22 H18 H23 H03 H21 H27 H26 H29 H28 H01 H02 H24 H05 H06 H07 H08 H17 H30 0.30 0. 43 a b c d e f g
karakter kedudukan daun, warna kelopak, bentuk buah, warna buah sebelum matang, kedudukan daun mahkota, warna mahkota, duri pada daun mahkota, orientasi spiral, aroma buah, warna daging buah dan tekstur dagung buah, warna petal dan warna buah sebelum matang dan kelompok ketujuh (g) adalah tetua JBBMQH6.
Nilai korelasi matriks kesamaan MxComp r = 0.825. Artinya dendrogram yang dihasilkan goodness of fit sesuai menggambarkan pengelompokan 30 hibrida tersebut (Rohlf, 1993).
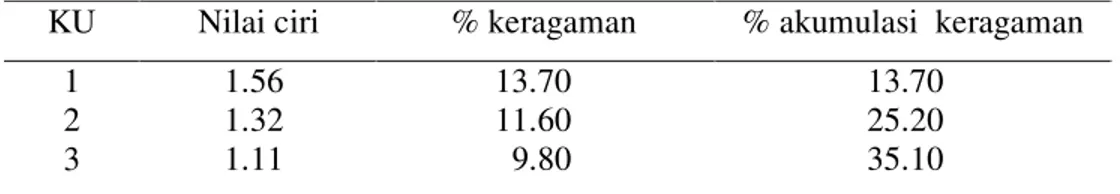
Analisis Komponen Utama
Analisis komponen utama merupakan teknik eksplorasi data yang digunakan sangat luas ketika menghadapi data peubah ganda. Analisis ini memberikan gambaran berupa besarnya pengaruh persentase nilai keragaman dari beberapa komponen utama (biasanya 3 komponen utama) yang dapat dibentuk dari minimal 70% keragaman yang dimiliki oleh karakter-karakter pada populasi yang dikarakterisasi. Menurut Diyarti, 2003, konsep analisis komponen utama adalah pereduksian dimensi sekumpulan peubah asal menjadi peubah baru yang berdimensi lebih kecil dan saling bebas.
Hasil analisis komponen utama menunjukkan bahwa hanya 25.20% dari total 100% keragaman data dapat dijelaskan menggunakan dua komponen utama
dan 35.1% dari total 100% keragaman data dapat dijelaskan menggunakan tiga komponen utama (Tabel 9). Hal ini menunjukkan bahwa nilai akumulasi keragaman yang diperoleh tidak memenuhi batas minimum 70% untuk tiga komponen pertama utama. Dengan demikian 87 subkarakter yang diamati tidak diperoleh karakter yang dapat dijadikan komponen utama untuk mengelompokkan
30 tanaman nenas hibrida dan kedua tetuanya. Rendahnya akumulasi keragaman pada populasi hibrida ini disebabkan karakter-karakter yang diamati memberikan
keragaman yang relatif sama.
Tabel 9. Nilai akar ciri tiga komponen utama (KU) 87 subkarakter morfologi
KU Nilai ciri % keragaman % akumulasi keragaman
1 2 3 1.56 1.32 1.11 13.70 11.60 9.80 13.70 25.20 35.10
Penanda Genotipik Analisis DNA
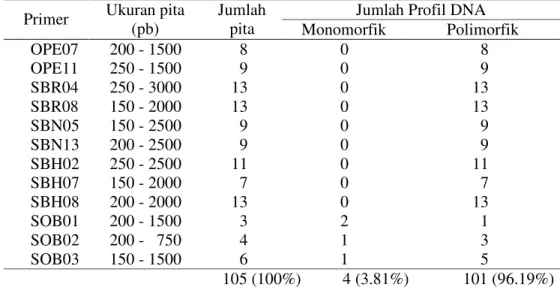
Pada Tabel 10 menunjukkan hasil amplifikasi DNA yang dilakukan terhadap 30 nomor nenas hibrida dan tetuanya dengan menggunakan 12 primer yang telah diseleksi untuk melihat polimorfisme DNA pada A. comosus (L.) Merr. Hasil seleksi diperoleh primer OPE7, OPE11, SBR04, SBR08, SBN05, SBN13, SBH02, SBH07, SBH08, SOB01, SOB02, dan SOB03 yang menghasilkan 105 pola pita dengan ukuran fragmen DNA yang teramplifikasi berkisar 150 bp – 3000 bp. Ukuran fragmen DNA yang teramplifikasi tergantung daerah yang diapit oleh dua primer dalam arah bolak-balik (McPherson et al. 1992).
Berdasarkan hasil yang diperoleh pita DNA terbagi dalam dua kelompok, yakni pita yang menunjukkan polimorfik dan pita monomorfik. Secara umum, hasil amplifikasi dengan 12 primer ini sudah memperlihatkan polimorfisme DNA. Primer yang menghasilkan jumlah pita terkecil adalah primer SOB01, sedangkan yang terbanyak adalah primer SBR04, SBR08, dan SBH08. Jumlah pita yang dihasilkan tergantung pada berapa banyak potongan DNA yang dihasilkan dari PCR. Pola pita yang paling bervariasi ditunjukkan oleh amplifikasi dengan primer SBR04, SBR08, dan SBH08, yakni masing-masing 13 pola pita. Sedangkan pola pita yang keragamannya paling rendah diperoleh dari hasil amplifikasi dengan primer SOB01, yakni 3 pola pita. Keragaman pola pita ini menunjukkan keragaman individu nenas hibrida dan tetuanya pada tingkat DNA.
Pada Tabel 10 terlihat bahwa dari 12 primer yang digunakan diperoleh 105 pita DNA. Tingkat polimorfisme primer yang digunakan tinggi, terdapat sembilan primer yang memiliki tingkat polimorfisme 100%. Dari 105 pola pita yang dihasilkan oleh ke 12 primer diperoleh 101 (96.19%) pola pita polimorfik dan 4 (3.81%) pola pita monomorfik. Tapia et al. (2005) dengan menggunakan penanda AFLP memperoleh 95% pita polimorfik dari 100 pita yang dihasilkan dari sampel 40 aksesi nenas koleksi plasma nutfah Campo Experimental del Papaloapan, Veracruz. Menurut McGregor et al., (2000), polimorfisme merupakan gambaran amplifikasi yang diperoleh dari perbedaan fragmen DNA yang terobservasi.
Jumlah Profil DNA Primer Ukuran pita
(pb)
Jumlah
pita Monomorfik Polimorfik
OPE07 OPE11 SBR04 SBR08 SBN05 SBN13 SBH02 SBH07 SBH08 SOB01 SOB02 SOB03 200 - 1500 250 - 1500 250 - 3000 150 - 2000 150 - 2500 200 - 2500 250 - 2500 150 - 2000 200 - 2000 200 - 1500 200 - 750 150 - 1500 8 9 13 13 9 9 11 7 13 3 4 6 0 0 0 0 0 0 0 0 0 2 1 1 8 9 13 13 9 9 11 7 13 1 3 5 105 (100%) 4 (3.81%) 101 (96.19%) Tabel 10. Data primer dan jumlah profil DNA hasil analisis RAPD dan E-RAPD
Contoh pola pita RAPD hasil amplifikasi primer SBN5 menggunakan DNA tetua JBBMQH6 dan JBSMSC3 serta 30 hibrida dan tetuanya dapat dilihat pada Gambar 5.
Gambar 5. Pola pita penanda RAPD yang dibangkitkan menggunakan primer SBN5 pada genotipe tetua (T1=JBBMQH6; T2= JBSMSC3) dan 30 F1 hasil persilangan tetua T1 x T2.
Perbedaan ada tidaknya pita dapat digunakan untuk menduga jumlah pasang kopi basa pada setiap pita RAPD dan e-RAPD. Ada tidaknya pita DNA hasil amplifikasi pada setiap primer sangat dipengaruhi:
a. Kemurnian dan konsentrasi DNA cetakan (template). DNA cetakan mengandung senyawa-senyawa seperti polisakarida dan senyawa fenolik serta konsentrasi DNA cetakan yang terlalu kecil sering menghasilkan pita DNA amplifikasi yang samar-smar atau tidak jelas.
b. Sebaran situs penempelan primer pada DNA cetakan.
M T1 T2 1 2 3 4 5 6 7 8 9 10 11 12 13 14 M 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 250 500 1000 1500 10000 3000
c. Adanya kompetisi tempat penempelan primer pada DNA cetakan yang menyebabkan satu fragment diamplifikasi dalam jumlah banyak dan fragment lainnya sedikit
Primer yang tidak menghasilkan pita DNA mengindikasikan bahwa primer-primer tersebut tidak mempunyai homologi dengan DNA cetakan, karena terbentuknya fragmen pita DNA bergantung pada sekuen primer dan genotipe dari DNA cetakan. Perbedaan jumlah dan polimorfisme pita DNA yang dihasilkan dari setiap primer menggambarkan kekompleksan genom tanaman yang diamati. Pita RAPD merupakan hasil berpasangannya nukleotida primer dengan nukleotida genom tanaman, maka semakin banyak primer yang digunakan akan semakin terwakili bagian-bagian genom, sehingga semakin tergambar keadaan genom tanaman yang sesungguhnya.
Polimorfisme yang dideteksi oleh RAPD pada prinsipnya merupakan hasil dari beberapa tipe peristiwa, yaitu: 1). Insersi DNA di antara dua situs penempelan primer, 2) Delesi pada bagian genom yang mengandung situs penempelan dan 3). Substitusi nukleotida pada situs penempelan primer (Weising et al., 1995). Insersi DNA yang berukuran besar diantara dua situs penempelan primer menyebabkan ketidakmampuan DNA polimerase untuk mensintesis DNA sehingga daerah tersebut tidak dapat diamplifikasi. Delesi pada genom yang mengandung situs penempelan menyebabkan primer tidak dapat menempel pada daerah tersebut, sehingga daerah tersebut tidak dapat diamplifikasi. Delesi di antara dua situs penempelan menyebabkan perubahan panjang dan ukuran daerah yang diamplifikasi.
Analisis Kemiripan
Matriks koefisien kemiripan genetik antara 30 hibrida dan kedua tetuanya diturunkan dari matriks simqual menunjukkan rentang nilai kemiripan berkisar antara 0.38-0.81 (Lampiran 6). Nilai koefisien kemiripan genotipik (KG) tertinggi yaitu 0.81, diperoleh pada hibrida H25 dengan H27 dan hibrida nomor H27 dengan H28, kemudian disusul hibrida H23 dengan H26 dan H23 dengan H27 dengan nilai 0.78. Nilai KG terendah diperoleh H01 dengan H30 yaitu 0.38.
Matriks kemiripan genetik dihitung berdasarkan jarak genetik antara tanaman yang satu dengan tanaman yang lainnya. Nilai kemiripan genetik 0.38
bearti jarak genetik antara hibrida nomor H01 dengan H30 paling jauh, hal ini berarti kedua individu sangat berbeda. Sedangkan nilai 0.81 menunjukkan bahwa antara pasangan hibrida nomor H25 dengan H27 dan pasangan hibrida nomor H27 dengan H28 memiliki jarak genetik yang sama dan rendah, yaitu 0.19.
Hibrida H13 memiliki koefisien kemiripan genetik tertinggi dengan tetua JBBMQH6 (T1) sebesar 0.68 dan hibrida H01 memiliki koefisien kemiripan genetik terendah dengan tetua JBBMQH6 (T1) sebesar 0.43. Hibrida H22 memiliki koefisien kemiripan genetik tertinggi dengan tetua JBSMSC3 (T2) sebesar 0.73 dan hibrida H30 memiliki koefisien kemiripan genetik terendah dengan tetua JBSMSC3 (T2) sebesar 0.45. Ini berarti hibrida H13 memiliki karakter yang sama lebih banyak dengan tetua JBBMQH6 (T1) dan hibrida H01 memiliki paling sedikit karakter yang sama dengan tetua JBBMQH6 (T1). Demikian pula hibrida H22 memiliki karakter sama yang lebih banyak dengan tetua JBSMSC3 (T2) dan H30 memiliki karakter yang sama lebih sedikit dengan tetua JBSMSC3 (T2).
Analisis Gerombol
Analisis gerombol terhadap 105 pola pita DNA menghasilkan dendrogram dengan koefisien kemiripan berkisar antara 61–88% atau terdapat keragaman genetik sebesar 12-39% (Gambar 6).
Pada koefisien kemiripan genetik (KKG) 61% terbentuk dua kelompok besar. Kelompok pertama terdapat empat hibrida, sedangkan kelompok kedua terdapat 26 hibrida dan kedua tetuanya.
Pada KKG 72% terbentuk tujuh subkelompok. Subkelompok pertama (a) memiliki KKG sekitar 83% atau memiliki kisaran jarak genetik 17% terdiri dari nomor hibrida H05 dan H02, subkelompok kedua (b) memiliki KKG 73% atau meiliki jarak genetik 27% terdiri dari nomor hibrida H01 dan H20. Subkelompok tiga (c) dan keempat (d) masing-masing terdiri dari hibrida 30 dan 24. Sub kelompok lima (e) memiliki KKG 74% memiliki anggota hibrida nomor H15, H16, H17, H18, H19, H22, H23, H25, H26, H27, H28, dan H29. Subkelompok keenam (f) dengan KKG 72% memiliki anggota hibrida nomor H08, H09, H10, H11, H12, H13, H14, H21, dan tetua JBSMSC3. Sedang subkelompok ketujuh (g) dengan KKG 73% memiliki anggota hibrida nomor H03, H04, H06 dan H07 serta
tetua JBBMQH6. Berdasarkan pola pengelompokan ini terlihat bahwa belum terungkap penciri karakter morfologi tertentu, baik tingkat kelompok maupun pada tingkat subkelompok.
Gambar 6. Dendrogram kemiripan genotipik hasil analisis gerombol dengan metode pengelompokan UPGMA berdasarkan 105 pola pita DNA. Nilai korelasi matriks kesamaan MxComp r = 0.767. Artinya dendrogram yang dihasilkan goodness of fit kurang sesuai menggambarkan pengelompokan
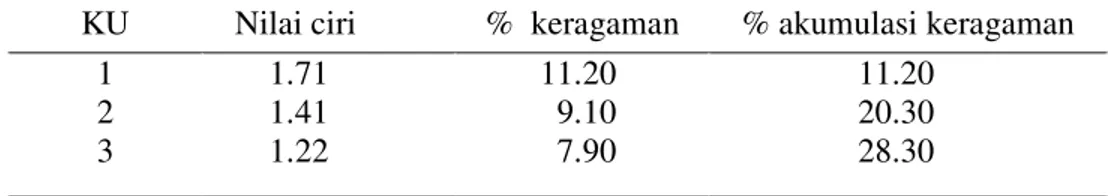
tersebut di atas (Rohlf, 1993). Analisis Komponen Utama
Hasil analisis komponen utama menunjukkan bahwa hanya 20.30% dari total 100% keragaman data dapat dijelaskan menggunakan dua komponen utama
dan 28.30% dari total 100% keragaman data dapat dijelaskan menggunakan tiga komponen utama (Tabel 11). Hal ini menunjukkan bahwa nilai akumulasi keragaman yang diperoleh tidak memenuhi batas minimum 70% untuk tiga komponen utama pertama. Dengan demikian 105 pola pita DNA yang diperoleh
tidak ada yang dapat dijadikan komponen utama untuk mengelompokkan 30 tanaman nenas hibrida dan tetuanya.
1 2 a b c e f g Koefisien Kemiripan 0.50 0.54 0.58 0.63 0.67 0.71 0.75 0.80 0.84 0.88 JBBMQH6 H03 H04 H06 H07 JBSMSC-3 H09 H10 H13 H14 H11 H12 H08 H21 H15 H17 H16 H19 H22 H23 H26 H29 H27 H28 H18 H25 H24 H30 H01 H20 H02 H05 0.61 0. 72 e a b d c f g
Tabel 11. Nilai akar ciri tiga komponen utama (KU) 105 pola pita DNA
Penanda Data Gabungan Analisis Kemiripan
Analisis kemiripan data gabungan penanda morfologi dan RAPD menghasilkan nilai koefisien kemiripan (KK) dengan kisaran antara 0.29 sampai 0.76 (Lampiran 8).
Nilai KK paling rendah terdapat pada pasangan nomor hibrida H01 dengan H30 yakni 0.29. Selain itu terdapat nilai KK 0.38 dari pasangan nomor hibrida antara H20 dengan H30. Nilai KK data gabungan paling tinggi terdapat pada pasangan H18 dengan H22 dan H26 dengan H29 sebesar 0.76, selain itu terdapat nilai KK 0.75 antara pasangan nomor hibrida H28 dengan H29.
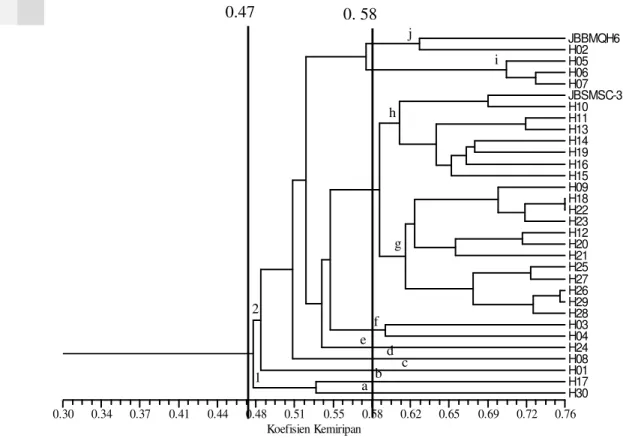
Analisis Gerombol
Hasil analisis gerombol data gabungan didapatkan rentang koefisien kemiripan 47-76% (Gambar 7) atau terdapat tingkat keragaman 0.24-0.53. Berdasarkan dendrogram ini pada koefisien kemiripan 47% dapat dibentuk 2 kelompok hibrida. Pada koefisien kemiripan 58% dapat dibentuk 10 subkelompok. Subkelompok pertama (a), kedua (b), ketiga (c), keempat (d) dan kelima (e) masing-masing terdapat hibrida nomor H30, H17, H01, H08 dan H24. Subkelompok keenam (f) memiliki kemiripan 59%, terdapat nomor hibrida H03 dan H04. Subkelompok ketujuh (g) memiliki kemiripan 61% terdapat nomor hibrida H09, H12, H18, H19, H20, H21, H22, H23, H25, H26, H27, H28, dan H29. Subkelompok delapan (h) dengan koefisien kemiripan 60% memiliki anggota hibrida nomor H10, H11, H13, H14, H15, H16, dan H19 dan tetua JBSMSC3. Subkelompok sembilan (i) dengan kemiripan genetik 58% terdapat anggota hibrida nomor, H05, H06 dan H07 serta tetua JBSMSC3. Subkelompok kesepeluh (j) terdapat hibrida nomor H02, H05 dan JBBMQH6.
KU Nilai ciri % keragaman % akumulasi keragaman
1 2 3 1.71 1.41 1.22 11.20 9.10 7.90 11.20 20.30 28.30
Berdasarkan data gabungan, pola pengelompokan ini terlihat bahwa belum terungkap penciri karakter morfologi tertentu, baik tingkat kelompok maupun pada tingkat sub kelompok.
Gambar 7. Dendrogram kemiripan data gabungan hasil analisis gerombol dengan metode pengelompokan UPGMA berdasarkan 87 karakter morfologi dan 105 pola pita RAPD.
Analisis Komponen Utama
Hasil analisis komponen utama menunjukkan bahwa hanya 18.00% dari total 100% keragaman data dapat dijelaskan menggunakan dua komponen utama
dan 25.40% dari total 100% keragaman data dapat dijelaskan menggunakan tiga komponen utama (Tabel 12). Hal ini menunjukkan bahwa nilai akumulasi keragaman yang diperoleh tidak memenuhi batas minimum 70% untuk tiga komponen utama pertama. Dengan demikian data gabungan yang diperoleh tidak
ada yang dapat dijadikan komponen utama untuk mengelompokkan 30 tanaman nenas hibrida dan tetuanya.
b c Koefisien Kemiripan 0.30 0.34 0.37 0.41 0.44 0.48 0.51 0.55 0.58 0.62 0.65 0.69 0.72 0.76 JBBMQH6 H02 H05 H06 H07 JBSMSC-3 H10 H11 H13 H14 H19 H16 H15 H09 H18 H22 H23 H12 H20 H21 H25 H27 H26 H29 H28 H03 H04 H24 H08 H01 H17 H30 1 2 a h 0.47 0. 58 f g d e i j b c
Tabel 12. Nilai akar ciri 3 komponen utama (KU) data gabungan
KU Nilai ciri % keragaman % Akumulasi keragaman
1 2 3 2.82 1.99 1.98 10.50 7.50 7.40 10.50 18.00 25.40 Analisis Perbandingan antara Penanda Fenotipik dan RAPD
Berdasarkan analisis perbandingan antara matriks kemiripan dua penanda (morfologi dan RAPD) menunjukkan nilai korelasi r = 0.7769. Nilai korelasi ini berdasarkan kriteria goodness of fit, yakni tingkat keselarasan nilai matriks pada 2 data, diinterpretasikan (poor fit, 0.7< r <0.8). Berdasarkan uji statistic Z mantel α 0.05 didapatkan korelasi yang sangat tidak nyata, karena pada tingkat korelasi tersebut didapatkan ρ = 1, dimana nilai ρ > 0.05 menunjukkan nilai korelasi yang diperoleh tidak nyata (Rohfl. 1993). Hal ini berarti pola antara keragaman fenotipik tidak selaras dengan pola keragaman profil DNA RAPD.
Faktor utama yang menyebabkan ketidakselarasan memungkinkan adalah penanda morfologi yang diamati dan profil DNA RAPD yang teramplifikasi bukan merupakan satu bagian yang saling berhubungan, atau hanya berhubungan sebagian, artinya penanda RAPD yang diperoleh belum tentu merupakan DNA yang menjadi penyandi karakter morfologi yang diamati. Hasil analisis yang tidak selaras yang dihasilkan dalam penelitian ini, terdapat pula pada hasil penelitian Apriani (2005), dengan menggunakan penanda morfologi (berdasarkan data vegetatif dan generatif) plasma nutfah nenas bersama empat primer RAPD, dimana diperoleh tingkat keselarasan yang tidak sesuai antara kedua penanda. Analisis Korelasi antar Karakter Kualitatif
Hasil analisis korelasi parsial antar 87 varian subkarakter didapatkan 22 varian nyata saling berkaitan pada taraf nyata 95-99%. Secara khusus didapatkan korelasi yang sangat nyata (99-100% kepercayaan) antar 14 karakter kualitatif (Tabel 13), dimana diperoleh korelasi 100% antara warna duri orange dengan duduk daun mahkota horisontal, antara warna kelopak putih perak dengan warna daging buah emas, dan antara warna buah sebelum matang (kuning pudar) dengan warna buah sesudah matang (kuning dalam sampai jingga tua).
Kode Karakter 14 18 23 32 38 43 49 19 - 0.84 - - - - - 46 - - - - 1.00 - - 53 1.00 - - - - 64 - - - 0.85 65 - - - 0.80 - - - 77 - - - 0.80 - 79 - - 1.00 - - -
Tabel 13. Nilai koefisien korelasi antar karakter kualitatif yang signifikan
Kode karakter : 14. warna duri orange; 18. duri daun kaku; 19. warna kelopak hijau dengan kuning bercorak merah; 23.kelopak putih perak; 32. Bentuk buah silinder tajam memanjang; 38. warna buah sebelum matang (kuning pudar); 43. Warna buah sesudah matang (hijau pudar); 46. Warna buah sesudah matang (kuning dalam sampai jingga tua); 49. bentuk mahkota kubus membujur; 53. duduk daun mahkota horisontal; 64. mahkota ganda; 65. mahkota tunggal kecil; 77. warna daging buah kuning dalam.. 79. warna daging buah emas tajam.
Analisis Korelasi Primer dengan Karakter
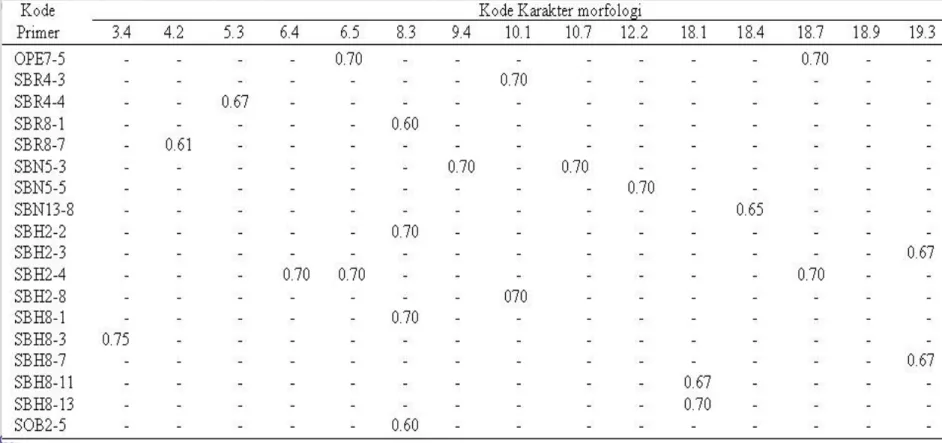
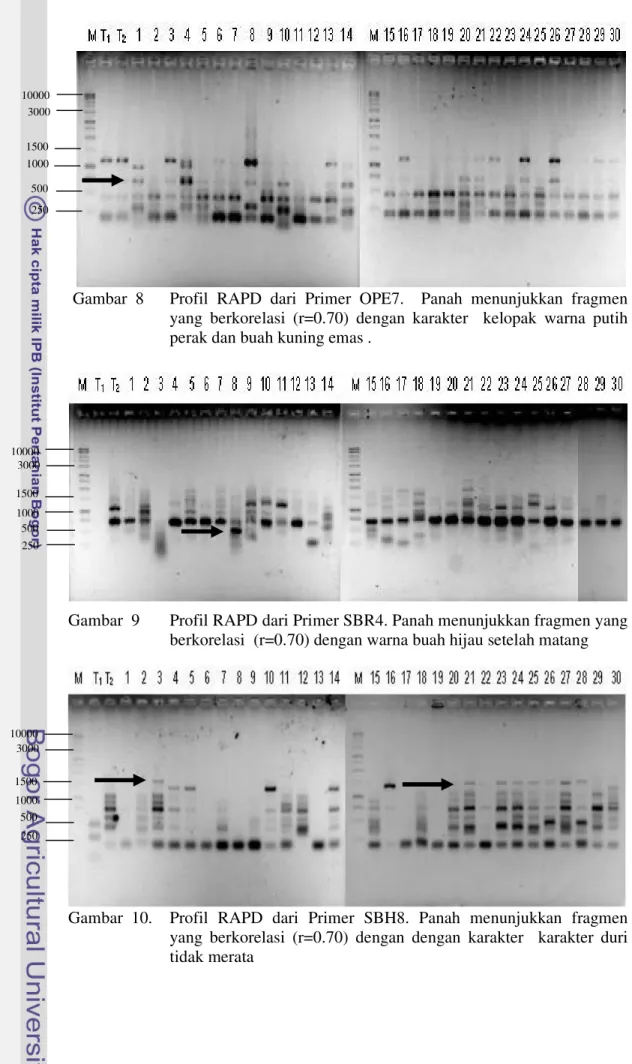
Hasil analisis korelasi menunjukkan bahwa terdapat 15 varian karakter kualitatif yang nyata berkaitan dengan 8 primer dari 12 primer RAPD dan e_RAPD yang digunakan. Delapan primer tersebut diwakili oleh 18 profil pita DNA. Berdasarkan analisis korelasi ini diperoleh 15 varian karakter semuanya sangat nyata (100% kepercayaan) berkorelasi anrtara 60-75% dengan 18 profil RAPD dari 12 primer yang berbeda (Tabel 14). Primer OPE7 pita 5 ukuran 750 pb berkorelasi dengan karakter kelopak warna putih perak (r=0.70), juga berkorelasi dengan warna daging buah kuning emas (r=0.70) yang terdapat pada hibrida H01, primer SBR4 pita 3 ukuran 500 pb berkorelasi dengan warna buah hijau setelah matang (r=0.70) yang terdapat hanya pada hibrida H08, primer SBN5 pita 3 ukuran 500 pb berkorelasi dengan warna buah kuning pudar sebelum matang (r=0.70) dan juga berkorelasi dengan warna buah kuning sampai jingga tua (r=0.70) yang hanya terdapat pada hibrida H21, primer SBH2 pita 8 berkorelasi dengan warna buah hijau setelah matang (r=0.70) yang terdapat pada hibrida H29, primer SBH8 pita 3 ukuran 1500 pb berkorelasi dengan karakter duri tidak merata (r=0.75) yang terdapat pada hibrida H03, H021, H023, H024, H025, H026, H027, dan H028. Profil DNA dari primer OPE7, SBR4, dan SBH8 masing-masing disajikan pada Gambar 8, 9 dan 10.
Tabel 14. Karakter morfologi berkorelasi nyata dengan profil RAPD
Kode Karakter : 3.4-Letak duri tidak merata; 4.2-Warna duri orange; 5.3-Duri kaku; 6.4-Kelopak warna merah keunguan; 6.5-Kelopak warna putih perak; 8.3-
Bentuk buah kerucut; 9.4--Warna buah kuning pudar sebelum matang; 10.1-Warna buah hijau setelah matang; 10.7-Warna buah kuning sampai jingga tua setelah matang; 12.2-Daun mahkota semi tegak; 18.1-Warna daging buah putih; 18.4- Warna daging kuning pucat; 18.7- Warna daging buah kuning emas; 18.9- Warna
daging buah orange ; 19.3-Tekstur buah kasar. 7
Gambar 8 Profil RAPD dari Primer OPE7. Panah menunjukkan fragmen yang berkorelasi (r=0.70) dengan karakter kelopak warna putih perak dan buah kuning emas .
Gambar 9 Profil RAPD dari Primer SBR4. Panah menunjukkan fragmen yang berkorelasi (r=0.70) dengan warna buah hijau setelah matang
.
Gambar 10. Profil RAPD dari Primer SBH8. Panah menunjukkan fragmen yang berkorelasi (r=0.70) dengan dengan karakter karakter duri tidak merata 10000 3000 1500 500 250 1000 1000 10000 3000 1500 500 250 1000 10000 3000 1500 500 250
Pembahasan Pembadingan Hasil Analisis Kemiripan
Hasil analisis penanda morfologi dan RAPD menunjukkan terdapat perbedaan rentang nilai koefisien kemiripan, dimana koefisien penanda morfologi menunjukkan rentang yang lebih besar dibandingkan nilai koefisien kemiripan RAPD (Tabel 15).
Tabel 15. Nilai koefisien kemiripan tertinggi dan terendah pada penanda morfologi, RAPD, dan data gabungan.
Koefisien
Kemiripan Data morfologi Data RAPD Data gabungan
Nilai tertinggi (kode nenas hibrida) 0.86 H05- H06. 0.81 H25-H27 dan H27- H28, 0.76 H28-H29. Nilai terendah (Kode nenas hibrida) 0.06 H03- H17 0.38 H01-H30 0.29 H01-H30
Analisis terhadap data gabungan menunjukkan adanya perbedaan hasil analisis antara data gabungan dengan masing-masing penanda (Tabel 15). Nilai koefisien tertinggi pada data gabungan mendekati nilai koefisien tertinggi pada data RAPD, demikian pula nilai koefisien terendah pada data gabungan cenderung mendekati nilai koefisien terendah data RAPD.
Pembadingan Hasil Analisis Kelompok
Hasil analisis gerombol menunjukkan pola pengelompokan yang berbeda antara morfologi, RAPD, maupun data gabungan. Pembandingan hasil pengelompokan masing-masing gerombol menunjukkan pola yang berbeda (Tabel 16). Namun demikian, data gabungan cenderung mengikuti pola pengelompokan penanda RAPD. Hal ini dimungkinkan karena RAPD lebih dominan dalam pembentukan kelompok dibandingkan penanda morfologi dan ada keterkaitan komponen karakter yang dilibatkan dalam pembentukan kelompok pada RAPD dan data gabungan.
Tabel 16. Pengelompokan hibrida dan tetuanya berdasarkan penanda morfologi, RAPD, dan data gabungan dengan analisis gerombol.
Morfologi RAPD Data gabungan
1. a.. H17 dan H30 b. H08 2. c. H05, H06, dan H07 d. H01, H04 dan H24 e. H03, H21, H26, H27, H28 dan H29 f. H09, H18, H22 dan H23 g. H04, H10, H11, H12, H13, H14, H15, H16, H19, H20, H25 dan JBSMSC3 h. JBBMQH6 1. a. H05 dan H02 b. H01 dan H20. 2. c. H24 dan H30 d. H15, H16, H17, H18, H19, H22, H23, H25, H26, H27, H28, dan H29 e. H08, H09, H10, H11, H12, H13, H14, H21, dan tetua JBSMSC3 . f. H03, H04, H06 dan H07 serta JBBMQH6 1 a. H17 dan H30 2 b. H03 dan H04 c. H09, H12, H18, H19, H20, H22, H23, H25, H26, H27, H28, dan H29 d. H10, H11, H13, H14, H15, H16, dan H19 dan tetua JBSMSC3 e. H02, H05, H06 dan H07 serta JBBMQH6 Perbandingan hasil analisis komponen utama.
Berdasarkan hasil analisis komponen utama terlihat bahwa baik data morfologi maupun data RAPD menunjukkan bahwa ketiganya tidak mencapai nilai minimum akumulasi 70% keragaman. Dengan demikian analisis lebih lanjut dengan menggunakan analisis komponen utama tidak diperlukan.
Perbandingan antara matriks kemiripan penanda morfologi dan RAPD Hasil analisis gerombol telah menunjukkan bahwa terdapat perbedaan pola pengelompokan nenas hibrida dan tetuanya antara penanda morfologi dan RAPD. Setelah dilakukan analisis komparasi antara matriks koefisien kemiripan kedua penanda didapatkan nilai korelasi keduanya r = 0.786 yang dikategorikan sebagai korelasi yang lemah. Korelasi yang rendah ini menunjukkan bahwa pola kemiripan di antara dua penanda sangat sedikit yang selaras.
Faktor utama yang menyebabkan ketidak selarasan kemungkinan adalah antara penanda morfologi yang diamati dan profil DNA RAPD yang teramplifikasi bukan merupakan satu bagian yang saling berhubungan, atau hanya berhubungan sebagian, artinya penanda RAPD yang diperoleh belum tentu merupakan DNA yang menyandi karakter morfologi yang diamati. Hal ini terlihat jelas pada hasil amplifikasi DNA kedua tetua, dimana sebagian besar primer yang digunakan tidak bisa membedakan antara genotipe tetua berduri dan tidak
berduri. Hasil analisis yang tidak selaras antara morfologi dan DNA yang menyandi karakter morfologi yang dihasilkan pada penelitian ini, terdapat pula pada hasil penelitian Crouch et al. (2002) dan Robi’ah (2004).
Korelasi Parsial antara Primer dengan Karakter
Analisis perbandingan antara matrik kemiripan penanda morfologi dan RAPD menunjukkan tingkat korelasi yang rendah antara keduanya. Hasil ini diperkuat oleh hasil analisis korelasi parsial antara karakter kualitatif dan profil DNA, dimana dari analisis ini menunjukkan bahwa hanya 15 varian karakter kualitatif (dari 87 varian yang dianalisis) yang berkorelasi nyata dengan 7 primer yang digunakan dalam RAPD. Dari hasil analisis ini secara khusus didapatkan satu varians karakter yang sangat berkorelasi 75% dengan 1 profil RAPD, yakni primer SBH8 pita 3 yang berkorelasi dengan letak duri tidak merata pada tepian daun.
Kesimpulan Bertdasarkan hasil penelitian dapat disimpulkan :
1. Pengelompokan 30 hibrida nenas dan tetuanya berdasarkan penanda morfologi dan RAPD dan gabungan (morfologi dan RAPD) menggunakan analisis kemiripan dan analisis gerombol diperoleh hasil pengelompokan yang berbeda.
2. Penanda morfologi melalui analisis kemiripan diperoleh kisaran koefisien kemiripan antara 0.06 sampai 0.86, penanda RAPD memiliki rentang lebih kecil dibandingkan morfologi, yakni berkisar 0.38 sampai 0.81, sedang data gabungan menghasilkan koefisien kemiripan antara 0.29-0.76.
3. Hasil analisis gerombol dengan menggunakan penanda morfologi terbentuk dua kelompok dan 8 subkelompok dengan kisaran koefisien kemiripan 0.30 sampai 0.86, penanda RAPD terbentuk dua kelompok dan enam subkelompok dengan kisaran koefisien kemiripan antara 0.61 hingga 0.88, dan penanda data gabungan terbetuk dua kelompok dan lima subkelompok dengan kisaran koefisien kemiripan antara 0.47 hingga 0.76.
4. Hasil analisis keselarasan diperoleh bahwa penanda morfologi memiliki nilai r yang sesuai, RAPD memiliki nilai r yang lemah (kurang sesuai), sedangkan data gabungan memiliki nilai r kurang sesuai (lemah).
5. Belum terungkap karakter yang dapat dijadikan sebagai komponen utama untuk mempelari keragaman genetik dari 30 populasi nenas hibrida dan tetuanya.
Saran
Disarankan dalam karakterisasi selain melibatkan karakter morfologi, juga melibatkan karakter yang tepat, terutama karakter-karakter yang berasosiasi dengan karakter unggul nenas. Sedangkan karakterisasi yang menggunakan penanda RAPD sebaiknya digunakan primer yang secara spesifik dapat menghasilkan profil DNA yang polimorfik yang juga berasosiasi dengan karakter penting nenas. Dengan melakukan pemilihan penanda yang tepat kemungkinan akan memberikan hasil karakterisasi yang lebih akurat dan dapat mengelompokkan nenas ke dalam kelompok-kelompok karakter utama.