Sensitivitas metode PCR (Polymerase chain reaction)
dalam mendekteksi isolat klinis Mycobacterium tuberculosis
Maria Lina Rosilawati*, Pratiwi Sudarmono**, Fera Ibrahim**
* Pusat Penelitian dan Pengembangan Teknologi Isotop dan Radiasi, BATAN, Jakarta ** Bagian Mikrobiologi Fakultas Kedokteran Universitas Indonesia
ABSTRACT
The PCR method was carried out to detect Mycobacterium tuberculosis H37Rv, clinical isolates of M.
tuberculosis from sputum of tuberculosis patients, and atypical mycobacteria strains. The bacteria were cultured
on Lowenstein Jensen medium. DNA extraction was done by using phenol-chloroform method after lysing the bacterial cells with lyzozyme, proteinase-K, and SDS. Serial dilution of extracted DNA was done to determine the sensitivity of the PCR assay. Primers used for DNA amplification were Pt8 and Pt9. The primers were designed from insertion sequence (IS)6110 of M. tuberculosis. Amplification product was analysed by gel agarose electrophoresis. Gel was stained with ethidium bromide solution and visualized under ultraviolet transilluminator. Results showed that the detection limits of amplified DNA of M. tuberculosis isolates were 100 fg - 500 pg, equivalent to 20 - 100.000 bacteria cells. PCR assay on isolate IMt3 and isolate IMt4, resulted the highest and the lowest sensitivities, respectively. The ampllified fragment DNA of 541 bp was resulted by using the primers mentioned above. Amplification of DNA did not occur on atypical mycobacteria strains.The result shows that the PCR using Pt8 and Pt9 primers is a specific assay.
Key words : PCR method, M. tuberculosis H37Rv, clinical isolates of M. tuberculosis, atypical Mycobacteria, sensitivity
ABSTRAK
Metode PCR digunakan untuk mendeteksi M. tuberculosis H37Rv, isolat M. tuberculosis dari sputum penderita tuberkulosis, dan strain mikobakteria atipik. Pembiakan bakteri dilakukan dalam medium Lowensen Jensen. Metode fenol-kloroform digunakan untuk mengekstraksi DNA bakteri setelah sel dilisiskan dengan lisosim, proteinase-K, dan SDS. Untuk mengetahui sensitivitas uji PCR, DNA hasil ekstraksi diencerkan dalam beberapa pengenceran. Amplifikasi DNA dilakukan dengan menggunakan primer oligonukleotida Pt8 and Pt9 yang disintesis dari sekuens sisipan 6110 M. tuberculosis. Teknik elektroforesis gel agarosa digunakan untuk mendeteksi hasil amplifikasi. Gel kemudian diwarnai dengan larutan etidium bromida dan divisualisasi dengan ultraviolet transilluminator. Hasil penelitian menunjukkan batas deteksi DNA isolat klinis M. tuberculosis hasil amplifikasi berkisar 100 fg - 500 pg yang setara dengan 20-100.000 sel bakteri. Uji sensitivitas PCR tertinggi adalah pada isolat klinis IMt3 dan terendah pada isolat IMt4. Besarnya fragmen DNA hasil amplifikasi adalah 541 bp. Amplfikasi DNA beberapa strain mikobakteria atipik menggunakan primer Pt8 and Pt9 cukup spesifik, karena tidak ada amplifikasi DNA strain-strain bakteri tersebut.
Kata kunci : Metode PCR, M. tuberculosis H37Rv, isolat M. tuberculosis, mikobakteria atipik, sensitivitas PENDAHULUAN
Perkembangan biologi molekuler di bidang kedokteran sangat cepat khususnya pada
pengendalian penyakit infeksi yang meliputi diagnosis, pengobatan, epidemiologi, dan
pencegahan. Diagnosis penyakit infeksi dengan biologi molekuler adalah mendeteksi asam nukleat mikroorganisme penyebab dengan menggunakan
pelacak DNA,(1) PCR,(2,3) dan LCR (ligase chain
reaction).(4) Sekuens DNA target spesifik yang
berbeda pada tiap organisme merupakan dasar penggunaan pelacak DNA. Sementara itu metode PCR dan LCR dapat memperbanyak jumlah salinan DNA target sehingga dapat mendeteksi mikroorganisme meskipun dalam jumlah sedikit.
Tuberkulosis adalah suatu penyakit infeksi yang disebabkan M. tuberculosis dan ditemukan lebih dari 100 tahun yang lalu. Namun, sampai saat ini penyakit tersebut masih merupakan masalah kesehatan yang sangat penting terutama di negara berkembang. WHO menyatakan sekitar 1,9 milyar manusia yaitu kurang lebih sepertiga penduduk dunia telah terinfeksi kuman tuberkulosis (TB) dan setiap tahunnya terdapat sekitar 8 juta penderita dengan 3 juta orang yang meninggal. Pada tahun 2000 diperkirakan di seluruh dunia penderita baru lebih dari 10,2 juta dengan 3,5 juta kematian akibat
penyakit tersebut.(5,6) Berdasarkan Survei Kesehatan
Rumah Tangga (SKRT) tahun 1992, tuberkulosis paru di Indonesia menduduki peringkat kedua
setelah penyakit kardiovaskular.(7) Data terakhir
WHO memperkirakan di Indonesia setiap tahun terdapat 550.000 kasus TB dan 175.000 kematian
akibat penyakit tersebut.(5)
Salah satu alasan gagalnya program pengendalian tuberkulosis di negara berkembang karena kelemahan diagnostik untuk mendeteksi
kasus infeksi pada saat dini.(8) Pada umumnya
metode diagnostik penyakit tuberkulosis dilakukan secara konvensional seperti pemeriksaan mikroskopik, kultur, dan serologi. Namun, metode tersebut mempunyai banyak kelemahan. Pemeriksaan mikroskopik di samping memerlukan kuman/bakteri minimal 10.000 sel/ml, juga tidak
dapat mendeteksi spesies mikobakteria.(9,10)
Pemeriksaan kultur kuman mempunyai sensitivitas dan spesifisitas cukup tinggi, akan tetapi memerlukan waktu yang cukup lama yaitu berkisar
3-8 minggu.(10,11) Sensitivitas metode tersebut
mendekati 100% dan dapat dilakukan pada sampel klinis yang mempunyai kandungan bakteri 10-100
sel.(12,13) Spesifisitas dan sensitivitas uji serologi
juga masih kurang memuaskan.(14) Penggunaan
pelacak DNA untuk deteksi M. tuberculosis juga telah banyak diteliti, spesifitas metode tersebut tinggi tetapi sensitivitas tidak berbeda jauh dengan pemeriksaan mikroskopik yang memerlukan sel
kuman dalam jumlah banyak.(15)
Untuk mengatasi keterbatasan cara diagnostik tersebut, sejumlah penelitian mengembangkan metode berdasarkan pada amplifikasi DNA
menggunakan metode PCR(2,3,16) ataupun metode
PCR yang dilanjutkan dengan teknik hibridisasi menggunakan sekuens DNA spesifik yang dilabel
dengan radioaktif/non radioaktif.(17,3) Metode
tersebut adalah metode yang cepat, sensitif dan spesifik jika dibandingkan dengan pembiakan yang merupakan metode baku emas (gold standard).
Penelitian pada tahap pertama bertujuan untuk mengetahui batas deteksi uji PCR pada beberapa isolat klinis M. tuberculosis menggunakan primer oligonukleotida Pt8 dan Pt9 yang spesifik dapat mendeteksi kuman tersebut.
BAHAN DAN CARA Pembiakan bakteri
Strain bakteri yang digunakan adalah M.
tuberculosis H37Rv sebagai kuman standar, M.
tuberculosis hasil isolasi sputum penderita
tuberkulosis sebanyak 6 isolat (IMt1, IMt2, IMt3,
IMt4, IMt5, IMt6) yang diperoleh dari salah satu
rumah sakit di Jakarta. Sebagai pembanding digunakan strain mikobakteria atipik yaitu M.
smegmatis, M. phlei, M. chelonae, M. terrae, M. scrofulaceum, dan M. fortuitum. Isolat M. tuberkulosis, kuman standar, M. scrofulaceum, dan M. terrae ditumbuhkan dalam medium miring
Lowestein-Jensen, diinkubasi pada suhu 37oC
selama 3-4 minggu, sedangkan untuk mikobakteria atipik yang lain diinkubasi selama ± 3 hari.
Ekstraksi DNA bakteri
Bakteri yang sudah tumbuh, dibuat suspensi dengan menambahkan larutan NaCl 0,9% (b/v) ke dalam kultur tersebut. Suspensi kemudian dipanaskan, disentrifugasi, dan dicuci sebanyak 2
kali. Pelet bakteri kemudian dilarutkan dengan larutan penyangga TE/Tris-EDTA pH 8,0. Ekstraksi DNA dilakukan sesuai dengan prosedur
peneliti terdahulu(18) yang secara singkat dapat
dijelaskan sebagai berikut. Sel bakteri dilisis dengan lisosim, proteinase-K, dan SDS (sodium dodecyl
sulfate). Suspensi tersebut ditambah dengan larutan
fenol-kloroform-isoamilalkohol dengan volume sama, digoyang supaya menjadi homogen (tanpa divorteks), kemudian disentrifugasi. Fase DNA yang didapat dipisahkan dan diendapkan dengan larutan NaCL 5M, etanol absolut dan etanol 70%
dingin, dan disentrifugasi pada suhu 10oC dengan
kecepatan tinggi (13.000 rpm). Pelet DNA yang diperoleh setelah dikeringkan, dilarutkan dengan larutan 1 x TE. Konsentrasi dan kemurnian DNA hasil ekstraksi ditentukan dengan spektrofotometer pada l 260 nm dan 280 nm. Untuk mengetahui batas deteksi DNA yang diamplifikasi dengan primer oligonukleotida yang digunakan, dilakukan pengenceran DNA hasil ekstraks dengan beberapa konsentrasi. Pengenceran DNA isolat M.
tuberculosis yang berasal dari sputum penderita TB
adalah pada konsentrasi 1ng/ml, 500 pg/ml, 100pg/ ml, 50pg/ml, 10pg/ml, 1pg/ml, 500fg/ml, 100fg/ml, 50fg/ml, 10fg/ml, dan 1fg/ml. Penentuan konsentrasi tersebut adalah berdasarkan penelitian
terdahulu.(19) Sebagai kontrol positif dan negatif
digunakan DNA M. tuberculosis H37Rv dan M.
smegmatis 1008.
Proses amplifikasi DNA
Amplifikasi DNA dilakukan dengan metode PCR menggunakan primer oligonukleotida Pt8 GTGCGGATGGTCGCGAGAT-3') dan Pt9 (5'-CTGGATGCC CTC ACG GTTCA-3'). Sekuens ini masing-masing terletak pada pasangan basa 105 sampai 124 dan 626 sampai 645 dari sekuens sisipan (IS: insertion sequence) 6110 M.
tuberculosis.(18) Komponen yang dipakai untuk
proses PCR adalah larutan penyangga (10mM
Tris-HCl pH 8,3 + 50 mM KCl), 2,0 mM MgCl2, 0,01%
(b/v) gelatin, dNTP (dATP, dCTP, dGTP, dTTP) masing 0,2 mM, primer Pt8 dan Pt9 masing-masing 0,2 mM, dan 1 unit Taq DNA polimerase. Campuran tersebut sebanyak 40 ml ditambah dengan 10 ml DNA sampel dan 50 ml minyak
mineral. Proses PCR dilakukan dalam DNA
thermocycler (Perkin-Elmer) dengan tahap
denaturasi 1,5 menit pada suhu 94oC, annealing
pada suhu 65oC, 2 menit, tahap extension 3 menit
pada suhu 72oC, dan extended extension pada suhu
72oC selama 7 menit, untuk tiap siklus. Jumlah
siklus yang diperlukan 40 siklus.
Analisis DNA hasil amplifikasi
Teknik elektroforesis gel agarosa digunakan untuk menganalisis DNA hasil amplifikasi dengan konsentrasi agarosa 1,5% (b/v). Proses ini dilaksanakan dalam larutan penyangga TBE (Tris-Borat-EDTA). Pewarnaan DNA hasil elektroforesis dilakukan dengan menggunakan larutan etidium bromida dengan konsentrasi 0,5 mg/ml. Setelah diwarnai, gel kemudian divisualisasi melalui
ultraviolet transilluminator. Untuk menentukan
ukuran fragmen DNA, digunakan penanda berat molekul HaeIII∅X174 yang dinyatakan dengan pasangan basa (basepair = bp). Hasil positif ditunjukkan dengan adanya pita fragmen DNA pada gel agarosa setelah diwarnai dan divisualisasi.
HASIL
Konsentrasi dan tingkat kemurnian DNA hasil ekstraksi yang dinyatakan dengan nilai rasio absorbansi (l 260 nm/280 nm) dari M. tuberculosis
H37Rv sebagai strain standar, isolat M. tuberculosis,
dan mikobakteria atipik, dapat dilihat pada Tabel 1. Nilai rasio absorbansi DNA strain standar dan isolat M. tuberculosis berkisar 1,500 - 2,000, sedangkan untuk DNA mikobakteria atipik adalah 1,413 - 1,844. Sampel DNA dinyatakan murni apabila nilai rasio absorbansi (l 260 nm / 280 nm)
= 1,8 - 1,9.(18,20)
Batas deteksi uji PCR, yaitu jumlah DNA minimum yang memberikan hasil positif dengan metode PCR dan elektroforesis gel agarosa, ditunjukkan dengan adanya pita DNA pada gel dari 6 isolat M. tuberculosis diperlihatkan pada Tabel 2.
Sensitivitas uji PCR yang ditunjukkan dengan batas deteksi tersebut untuk 6 isolat yang digunakan dalam penelitian ini bervariasi. Pada umumnya makin tinggi tingkat kemurnian DNA, sensitivitas
uji PCR makin meningkat. Namun, apabila
dibandingkan hasil amplifikasi DNA isolat IMt3
yang mempunyai nilai rasio absorbansi 2,000
dengan isolat IMt1 dan IMt2 dengan nilai rasio
masing-masing 1,829 dan 1,827, terlihat senstivitas
uji PCR untuk isolat IMt3 lebih tinggi. Batas deteksi
DNA isolat IMT3, isolat IMt1, dan isolat IMt2
masing-masing adalah 100 fg, 1 pg, dan 5 pg setara dengan 20, 200, dan 1000 sel bakteri. Menurut Kolk
et al,(3) DNA M. tuberculosis 5 fg setara dengan 1
sel bakteri. Demikian juga sensitivitas uji PCR pada
DNA isolat IMt6 dengan tingkat kemurnian paling
rendah, sensitivitasnya lebih tinggi dari isolat IMt4
dan IMt5 yang mempunyai tingkat kemurnian lebih
tinggi. Berdasarkan batas deteksi DNA, sensitivitas tertinggi dan terendah hasil uji PCR dalam penelitian
ini masing-masing adalah dari isolat IMt3 dan IMt4
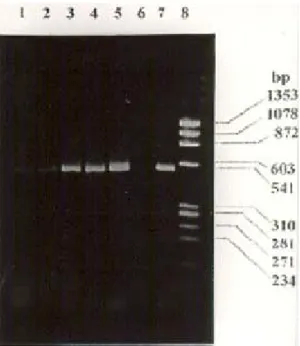
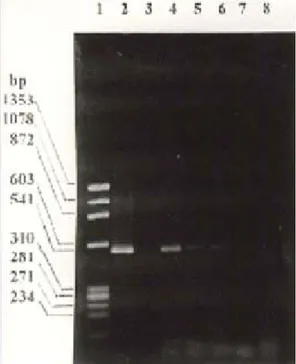
dengan batas deteksi 100 fg (Tabel 2, Gambar 1) dan 500 pg (Tabel 2, Gambar 2). Jumlah tersebut setara dengan 20 dan 100.000 sel bakteri. Besarnya fragmen DNA hasil amplifikasi menggunakan primer Pt8 dan Pt9 yang digunakan dalam penelitian adalah 541 bp.Gambar 3 menunjukkan hasil amplifikasi dan analisis dengan teknik elektroforesis dari strain mikobakteria atipik dengan jumlah DNA yang diamplifikasi cukup besar yaitu 100 ng. Amplifikasi DNA strain tersebut juga dilakukan untuk jumlah DNA yang bervariasi. Dari gambar tersebut dengan jumlah DNA yang cukup besar
Tabel 1. Konsentrasi dan tingkat kemurnian DNA hasil ekstrasi M. tuberculosis strain H37Rv,
isolat klinis M. tuberculosis, dan mikobakteria atipik
Strain bakteri Konsentrasi DNA Rasio absorbansi (ng/µ1) (λλλλλ 260 nm/280 nm)
M. tuberculosis H37Rv 1160 1,901
M. tuberculosis isolat IMt1 310 1,829
M. tuberculosis isolat IMt2 278 1,827
M. tuberculosis isolat IMt3 78 2,000
M. tuberculosis isolat IMt4 138 1,761
M. tuberculosis isolat IMt5 100 1,682
M. tuberculosis isolat IMt6 58 1,500
M. smegmatis 1068 1,940 M. phlei 765 1,842 M. chelonae 634 1,627 M. terrae 118 1,511 M. scrofulaceum 88 1,500 M. fortuitum 168 1,413
maupun dengan jumlah DNA yang bervariasi menggunakan primer yang sama (Pt8 dan Pt9) tetap menunjukkan tidak adanya amplifikasi DNA strain mikobakteria tersebut (tidak ada pita DNA pada gel agarosa).
PEMBAHASAN
Konsentrasi dan tingkat kemurnian hasil ekstraksi DNA dinyatakan dengan nilai rasio absorbansi (l 260 nm/280 nm) dalam penelitian ini,
bervariasi. Menurut Hill et al,(22) DNA M.
tuberculosis hasil ekstraksi yang diendapkan dengan
CTAB (cetyltrimethyl-amonium bromide) mempunyai rasio absorbansi 1,88 dibanding dengan nilai sebelum penambahan yaitu 1,75. Ekstraksi Tabel 2. Jumlah DNA minimun isolat klinis M.
tuberculosis yang memberikanhasil positif dengan
metode PCR dan elektroforesis gel agarosa Sampel DNA Jumlah DNA
M. tuberculosis isolat IMt1 1 pg
M. tuberculosis isolat IMt2 5 fg
M. tuberculosis isolat IMt3 100 fg
M. tuberculosis isolat IMt4 500 pg
M. tuberculosis isolat IMt5 100 pg
DNA dalam peneltian ini adalah menggunakan metode yang tidak memakai CTAB. Hal lain yang mungkin menyebabkan berkurangnya tingkat kemurnian DNA hasil ekstraksi dengan nilai rasio di bawah 1,8-1,9, adalah adanya kontaminasi bahan yang terdapat pada medium pertumbuhan pada saat penyediaan suspensi sel bakteri di samping bahan lain pada waktu proses ekstraksi. Nilai rasio absorbansi di bawah 1,8 - 1,9 menunjukkan adanya kontaminasi protein, fenol, SDS dan
polisakarida.(20,21)
Sensitivitas uji PCR pada 6 isolat M.
tuberculosis yang dipakai dalam penelitian ini, juga
bervariasi. Seperti dijelaskan sebelumnya, sensitivitas tersebut sangat dipengaruhi tingkat kemurnian DNA. Berdasarkan penelitian Sjobring
et al,(12) sensitivitas PCR sangat dipengaruhi metode
ekstraksi DNA . Dari hasil penelitian yang telah dikemukakan terlihat sensitivitas PCR pada isolat
IMt3 lebih tinggi dari pada isolat IMt1 dan IMt2,
sedangkan tingkat kemurnian DNA IMt3 yang
diamplifikasi lebih rendah dari ke dua isolat
tersebut. Demikian juga isolat IMt6 dengan tingkat
kemurnian DNA paling rendah (nilai rasio absorbansi = 2,000), mempunyai batas deteksi uji PCR lebih tinggi yaitu 5 pg setara 1000 sel bakteri
dibandingkan dengan isolat IMt4 dan IMt5 dengan
batas deteksi 100 pg dan 500 pg yang masing-masing setara dengan 20.000 dan 100.000 sel bakteri. Perbedaan sensitivitas tersebut kemungkinan besar disebabkan banyaknya salinan/ fragmen DNA target yang terdapat berulang (IS6110) dalam genom antara strain M.
tuberculosis, berbeda secara alamiah. Salah satu
faktor yang menentukan sensitivitas PCR adalah jumlah salinan sekuens DNA sasaran yang terdapat
dalam genom suatu mikroorganisme.(22) Beberapa
strain bakteri tersebut di Asia hanya mempunyai 1
salinan IS6110 dalam genomnya.(23) Dari hasil
penelitian terdahulu(19) didapatkan batas deteksi
DNA M. tuberculosis H37Rv yang diamplifikasi
dengan primer yang sama (Pt8 & Pt9) adalah 10 fg setara dengan 2 sel bakteri. Jumlah IS6110 M.
tuberculosis H37Rv yang merupakan sekuens DNA sasaran primer Pt8 & Pt9 berjumlah 16 salinan
dalam genomnya.(24) Berdasarkan penelitian Kox
et al,(2) diperoleh jumlah DNA yang dapat
diamplifikasi/batas deteksi uji PCR pada strain-strain M. tuberculosis menggunakan primer yang sama juga bervariasi, yaitu 10 fg pada 6 uji dari 8 uji sedangkan 2 uji lainnya menunjukkan hasil lebih sensitif yaitu 1 fg.
Gambar 1. Hasil amplifikasi DNA M. tuberculosis isolat IMt3 dengan metode PCR
dan elektroforesis gel agarosa
Lajur 1 : DNA isolat IMt3 100 fg
Lajur 2 : DNA isolat IMt3 1 pg
Lajur 3 : DNA isolat IMt3 5 pg
Lajur 4 : DNA M. tuberculosis H37Rv 10 ng
(Kontrol+)
Lajur 5 : DNA isolat IMt3 10 pg
Lajur 6 : Kontrol – (tanpa DNA)
Lajur 7 : DNA isolat IMt3 100 pg
Lajur 8 : Marker (penanda berat molekul) HaeIII∅X174
Gambar 3. Hasil amplifikasi DNA mikobakteria atipik dengan metode PCR dan elektroforesis gel agarosa.
Lajur 1 : Marker (penanda berat molekul) HaeIII∅X174
Lajur 2 : DNA M.chelonae 100 ng Lajur 3 : DNA M. fortuitum 100 ng Lajur 4 : DNA M. phlei 100 ng
Lajur 5 : DNA M. scrofulaceum 100 ng Lajur 6 : DNA M. smegmatis 100 ng Lajur 7 : DNA M. terrae 100 ng
Lajur 8 : DNA M. tuberculosis H37Rv 10 ng
(Kontrol +)
Gambar 2. Hasil Aplifikasi DNA M. tuberculosis isolat IMt4 dengan metode PCR dan elektroforesis gel agarosa
Lajur 1 : Marker (penanda berat molekul)
HaeIII∅X174 DNA
Lajur 2 : DNA M. tuberculosis H37Rv 10 ng
(Kontrol+)
Lajur 3 : Kontrol – (tanpa DNA)
Lajur 4 : DNA isolat IMt3 5 ng
Lajur 5 : DNA isolat IMt3 1 pg
Lajur 6 : DNA isolat IMt3 500 pg
Lajur 7 : DNA isolat IMt3 100 pg
Uji PCR dengan primer Pt8 & Pt9 dalam penelitian ini cukup spesifik untuk mendeteksi kuman M. tuberculosis, karena tidak ada amplifikasi DNA mikobakteria atipik (Gambar 3). Hasil ini
sesuai dengan hasil peneliti lain(2) yang memperoleh
hasil PCR negatif menggunakan primer yang sama pada 12 sampel klinis yang mengandung
mikobakteria bukan M. tuberculosis. Kent et al(25)
menyatakan 24 strain dari 35 strain mikobakteria nontuberkulosis yang diuji dengan teknik PCR menggunakan primer yang dirancang dari IS6110 menunjukkan hasil positif. Hal tersebut disebabkan karena beberapa strain mikobakteria nontuberkulosis mempunyai suatu sekuens DNA yang homolog dengan IS6110. Sebaliknya, hasil
penelitian Hellyer et al,(26) menunjukkan tidak ada
sekuens DNA dalam mikobakteria nontuberkulosis yang homolog dengan IS6110. Kemungkinan yang menyebabkan hasil berbeda dari ke dua penelitian tersebut adalah daerah elemen IS6110 yang menjadi DNA sasaran, berbeda. Kemungkinan lain jumlah DNA sasaran yang digunakan dalam penelitian Kent
et al terlalu banyak, sehingga besar kemungkinan
terjadi hasil positif semu yang dihasilkan DNA M.
tuberculosis sebagai kontaminan dan atau amplikon
yang terdapat dalam jumlah kecil. Oleh karenanya, untuk menghindari reaksi silang dengan spesies bukan M. tuberculosis kompleks, diperlukan ketelitian mendisain primer berdasarkan sekuens dalam IS6110, di samping persyaratan yang memenuhi untuk menghindari kontaminasi.
KESIMPULAN
Metode PCR menggunakan primer oligonukleotida Pt8 & Pt9 pada isolat klinis M.
tuberculosis mempunyai kemampuan mendeteksi
jumlah DNA sebesar 100 fg yang setara dengan 20 sel bakteri.
Primer oligonukleotida Pt8 & Pt9 untuk uji PCR cukup spesifik untuk mendeteksi bakteri M.
tuberculosis. Mikobakteria atipik yang digunakan
dalam penelitian ini tidak terdeteksi dengan teknik PCR menggunakan primer tersebut.
Daftar Pustaka
1. Lebrun L, Espinase F, Poveda JD, Frebault VVL.
Evaluation of non radioactive DNA probe for identification of mycobacteria. J Clin Microbiol 1992; 30:2476-78.
2. Kox, LFF, Rienthong, D, Miranda AM,
Udomsantisuk N, Ellis K, van Leeuwen J, et al A more reliable PCR for detection of Mycobacterium tuberculosis in clinical samples J Clin Microbiol 1994; 32:672-78.
3. Kolk AHJ, Schuitema ARJ, Kuijper S, van
Leeuwen J, Hermans PWM, van Embden JDA, et al. Detection of Mycobacterium tuberculosis in clinical samples by using polymerase chain reaction and a non radioactive detection system. J Clin Microbiol 1992; 30:2567-75.
4. Lindbrathen A, Gaustad P, Hovig B, TÆnjun T.
Direct detection of Mycobacterium tuberculosis complex in clinical samples from patients in Norway by ligase chain reaction. J Clin Microbiol 1997; 35:3248-53.
5. Tjandra JA, Priyanti ZS. Tuberkulosis. Diagnosis, Terapi, dan Masalahnya. Edisi III Jakarta: Lab. Mikobakteriologi RSUP Persahabatan/WHO Collaborating Center for Tuberculosis; 2000.
6. Dollin PJ, Raviglione MC, Kochi A. Global
tuberculosis incidence and mortality during 1990-2000. Bull WHO 1994; 72:213-20.
7. Departemen Kesehatan R.I. Survei Kesehatan
Rumah Tangga 1992. Jakarta: Badan Penelitian dan Pengembangan Kesehatan RI; 1992. 8. Styblo K. Overview and epidemiologic assesment
of the current global tuberculosis situation with an emphasis on control in developing countries. Rev Infect Dis 1989; 11:339-46.
9. Koneman EW, Allen SD, Janda WM,
Schreckenberger PC, Winn WC, (ed). Color atlas and textbook of diagnostic microbiology. 4th ed.
Philadelphia: JB Lippincott Company; 1990. p.703-14
10. Plorde JJ. Mycobacteria. In: Sherris JC, Ryan KJ, editor. Medical microbiology. An introduction to infectious disease. London: Prentice-Hall International Inc. (UK) Limited; 1994.p.401-15. 11. Brooks GF, Butel JS, Ornston LN, Jawetz E, Melnick JL, Adelberg EA. Medical microbiology 20th ed. Norwalk, Connecticut: Appleton & Lange;
1995. p.263-71.
12. Sjobring JH, Mecklenburg M, Andersen AB, Miorner H. Polymerase chain reaction for detection of Mycobacterium tuberculosis. J Clin Microbiol 1990; 28:2200-04.
13. Hobby GT, Holman AP, Iseman MD, Jones JM. Enumeration of tubercle bacilli in sputum of patients with pulmonary tuberculosis. Antimicrob Agents Chemother 1973; 4:97-104.
14. Daniel TM, Debanne SM. The serodiagnosis of tuberculosis and other mycobacterial disease by enzyme-linked immunosorbent assay. Am Rev Respir Dis 1987; 135:1137-51.
15. Oka GMS, Okuzumi K, Kimura S, Shimada K. Evaluation of acridinium-esterlabeled DNA probe for identification of Mycobacterium tuberculosis and Mycobacterium avium-Myco-bacterium intracellular complex in culture. J Clin Microbiol 1991; 29:2473-76.
16. Beavis KG, Lichty MB, Jungkind DL, Giger O. Evaluation amplicor PCR for direct detection of Mycobacterium tuberculosis from sputum specimens. J Clin Microbiol 1995; 33:2582-86. 17. Altamirano M, Kelly MT, Wong A, Bessuille ET,
Black WA, Smith JA. Characterization of DNA probe for detection of Mycobacterium tuberculosis complex in clinical samples by polymerase chain reaction. J Clin Microbiol 1992; 30:2173-76. 18. Kolk AHJ, Kox LFF, van Leeuwen J, Kuijper S.
Polymerase chain reaction for the M. tuberculosis complex. Laboratory of Tropical Hygiene, Department of Biomedical Research Royal Tropical Institute, Amsterdam, The Netherland; 1995.
19. Lina MR, Sudarmono P, Ibrahim F, Soebandrio
A. Deteksi Mycobacterium tuberculosis H37Rv
dengan reaksi berantai polimerase (PCR). Maj Kedokt Indon 1999; 49:250-255.
20. Brown TA. Gene cloning: an introduction. Workingham, Berkshire, England: Van Nostrand Reinhold (UK) Co Ltd Molly Millars Lane; 1986.p. 26.
21. Hill EB, Wayne LG, Gross WM. Curent practices in mycobacteriology: result of a survey of public health laboratories. J Bacteriol 1972; 112:1033-39.
22. Thierry D, Brisson-Noel A, Frebault VVL, Nguyen S, Jeanuc G, Gicquel B. Characterization of a Mycobacterium tuberculosis insertion sequence, IS6110, and its application in diagnosis. J Clin Microbiol 1990; 28:2668-74.
23. Yuen LK, Ross BC, Jackson KM, Dwyer B. Characterization of Mycobacterium tuberculosis strains from Vietnamese patients by southern blot hybridization. J Clin Microbiol 1993; 131:1615-18.
24. Philipp WJ, Poulet S, Eiglmeier K, Pascopella L, Balasu BV, Heym B et al. An integrated map of the genom of the tubercle bacillus, Mycobacterium
tuberculosis H37Rv, and comparison with
Mycobacteriunm leprae.
25. Kent L, McHugh Td, Billington O, Dale JW, Gillespie SH. Demonstration of homolog between IS6110 of Mycobacterium tuberculosis and DNAs of other Mycobacterium spp. J Clin Microbiol 1995; 33:2290-93.
26. Hellyer TJ, DesJardin LE, Assaf MK, Bates JH, Cave MD, Eisenach KD. Specificity of IS6110 based amplification assay for Mycobacterium tuberculosis complex. J Clin Microbiol 1996; 34:2843-46.